Микробиология, 2020, T. 89, № 3, стр. 308-314
Филогенетика и биогеография важных для биотехнологии метилотрофных дрожжей Komagataella
Е. С. Наумова a, *, К. Л. Боунди-Миллс b, Г. И. Наумов a
a Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов” Национального исследовательского центра “Курчатовский Институт” (НИЦ “Курчатовский Институт” – ГосНИИгенетика)
117545 Москва, Россия
b Phaff Yeast Culture Collection, Department of Food Science and Technology, University of California
CA, Davis 95616, USA
* E-mail: lena_naumova@yahoo.com
Поступила в редакцию 30.09.2019
После доработки 30.10.2019
Принята к публикации 29.11.2019
Аннотация
Проведено молекулярно-генетическое изучение 22 штаммов метанол-ассимилирующих дрожжей Komagataella из коллекции UCDFST (США). Мультигенный филогенетический анализ показал, что штаммы, ранее идентифицированные на основании стандартных таксономических тестов как “Pichia pastoris”, в действительности относятся к четырем разным видам: K. pastoris, K. phaffii, K. mondaviorum и K. ulmi. Семь видов рода Komagataella практически неразличимы на основании стандартных морфологических и физиологических тестов, а также имеют сходные D1/D2-последовательности. Установлено, что для достоверной дифференциации всех видов Komagataella необходимо использовать мультигенный филогенетический анализ. Обращает на себя внимание биогеография дрожжей Komagataella. Виды K. mondaviorum, K. kurtzmanii, K. phaffii, K. populi и K. ulmi обнаружены только в Северной Америке, тогда как K. pastoris и K. pseudopastoris характерны для Европы.
Метилотрофные дрожжи Komagataella, относимые ранее к полифилетическому роду Pichia, характеризуются способностью расти на метаноле как на единственном источнике углерода. Обладая сильными индуцируемыми метанолом промоторами, эти дрожжи широко используются в биотехнологии для получения высококачественных рекомбинантных белков медицинского и микробиологического назначения (Cregg et al., 1988, 1993; Chen et al., 2012; Love et al., 2016; Zahrl et al., 2017). Экспрессионная система “Pichia pastoris” была коммерциализирована компанией “Invitrogen” и широко используется для научных и промышленных целей (Cregg et al., 1993).
Большое прикладное значение этих дрожжей способствовало их тщательной классификации и идентификации. Впервые эти дрожжи были описаны как Zygosaccharomyces pastori на единичном штамме, выделенном из каштана во Франции (Guilliermond, 1919). В 1950-х гг. Герман Фафф изолировал несколько родственных штаммов из черных дубов в Калифорнии, и переименовал вид в Pichia pastoris (Phaff, 1956). Очередная ревизия гетерогенного рода Pichia привела к описанию нового монотипного рода Komagataella, в который был перенесен вид Pichia pastoris (Yamada et al., 1995).
После открытия европейского вида Komagataella pseudopastoris, изначально описанного как Pich-ia pseudopastoris (Dlauchy et al., 2003), род Komagataella стал общепризнанным (Kurtzman, 2005, 2011). На основе сравнительного анализа нуклеотидных последовательностей домена D1/D2 гена 26S рРНК был описан третий вид K. phaffii, к которому были отнесены американские изоляты (Kurtzman, 2005). Было установлено, что важные с биотехнологической точки зрения дрожжи “Pichia pastoris” в действительности представлены двумя видами: K. pastoris и K. phaffi, а используемый в коммерчески доступном наборе для экспрессии “Invitrogen” штамм “Pichia pastoris” NRRL Y-48124 относится к K. phaffi (Kurtzman, 2009). Большинство промышленных штаммов относятся к K. phaffii (Gasser, Mattanovich, 2018). На материале отдельных штаммов были описаны еще три новых вида Komagataella: K. populi, K. ulmi и K. kurtzmanii (Kurtzman, 2012; Naumov et al., 2013). Недавно нами был описан седьмой вид рода Komagataella: K. mondaviorum (Naumov et al., 2018). Важными с биотехнологической точки зрения являются также дрожжи K. kurtzmanii, которые по ряду характеристик превосходят коммерческие штаммы K. phaffii и K. pastoris (Tyurin et al., 2013). Недавно проведено секвенирование полноразмерных геномов типовых культур K. pastoris, K. phaffii, K. populi и K. pseudopastoris (Love et al., 2016; Valli et al., 2016).
В настоящей работе проведено молекулярно-генетическое изучение и реидентификация штаммов Komagataella, хранящихся в коллекции UCDFST (Phaff Yeast Culture Collection, Department of Food Science and Technology, University of California, Дэвис, США), с целью расширения научного и прикладного использования естественного генофонда этих дрожжей.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Штаммы и среды. Использованные в работе штаммы и их происхождение представлены в табл. 1. Большинство штаммов получены из дрожжевой коллекции UCDFST (http://phaffcollection.ucdavis.edu/) и выделены в разные годы из различных природных источников. Дрожжи выращивали при 28°С на полной среде YPD (г/л): бакто-агар фирмы “Difco” (США) – 20; глюкоза фирмы “Merck” (Германия) – 20; дрожжевой экстракт “Difco” – 10; бакто-пептон “Difco” – 20.
Таблица 1.
Происхождение изученных штаммов Komagataella
| Номер штамма | Источник, место и год выделения | Оригинальное видовое название |
|---|---|---|
| K. pastoris | ||
| NRRL Y-1603T | Сокотечение каштана, Франция, 1919 | K. pastoris |
| UCDFST 60-58 | Сокотечение каштана, Нидерланды, 1960 | K. pastoris |
| UCDFST 68-773.2 | Сокотечение березы Betula sp., Британская Колумбия, Канада, 1968 | K. pastoris |
| LU2T88 | Почва, Тайвань, 2011 | K. pastoris |
| LE2B88 | Насекомое, Московская обл., 2011 | K. pastoris |
| K. phafii | ||
| NRRL Y-7556T | Сокотечение дуба Quercuskelloggii, Калифорния, США, 1956 | K. phafii |
| UCDFST 54-11.214 | Сокотечение дуба Quercuskelloggii, Калифорния, США, 1954 | K. pastoris |
| UCDFST 54-11.229 | Сокотечение дуба Quercuskelloggii, Калифорния, США, 1954 | K. pastoris |
| UCDFST 56-57 | Сокотечение дуба Quercuskelloggii, Калифорния, США, 1956 | K. pastoris |
| UCDFST 68-1033.1 | Сокотечение дуба Quercuskelloggii, Калифорния, США, 1968 | K. pastoris |
| UCDFST 52-155 | Drosophila pseudoobscura, Калифорния, США, 1952 | K. pastoris |
| K. kurtzmanii | ||
| ВКПМ Y-727T | Сокотечение пихты Abies sp., Южная Аризона, США, 1976 | K. kurtzmanii |
| K. mondaviorum | ||
| UCDFST 71-1024T | Сокотечение тополя, Калифорния, США, 1971 | K. mondaviorum |
| UCDFST 54-11.16 | Сокотечение дуба Quercus sp., Калифорния, США, 1954 | K. mondaviorum |
| UCDFST 54-11.141 | Сокотечение дуба Q. kelloggii, Калифорния, США, 1954 | K. mondaviorum |
| UCDFST 68-967.1 | Буровая мука ивы Salex sp., штат Вашингтон, США, 1968 | K. mondaviorum |
| UCDFST 68-692.1 | Сокотечение тополя P. tremuloides, Аляска, США, 1968 | K. pastoris |
| UCDFST 68-974.1 | Сокотечение клена Acer sp., штат Вашингтон, США, 1968 | K. pastoris |
| UCDFST 68-891.2 | Сокотечение P. trichocarpa, Британская Колумбия, Канада, 1968 | K. pastoris |
| UCDFST 72-1033 | Сокотечение тополя, Калифорния, США, 1972 | K. pastoris |
| UCDFST 74-1030 | Сокотечение каркаса Celtis sp., Калифорния, США, 1974 | K. mondaviorum |
| UCDFST 77-1019 | Источник неизвестен, Калифорния, США, 1977 | K. pastoris |
| K. ulmi | ||
| NRRL YB-407T | Сокотечение вяза Ulmus americana, Иллинойс, США, 1945 | K. ulmi |
| UCDFST 49-54 | Сокотечение вяза, Миссури, США, 1949 | K. pastoris |
| UCDFST 67-1008 | Сокотечение дуба Quercus sp., Калифорния, США, 1967 | K. pastoris |
| UCDFST 71-1016 | Сокотечение вяза, Калифорния, США, 1971 | K. pastoris |
| UCDFST 71-1036 | Сокотечение дуба Q. suber, Калифорния, США, 1971 | K. pastoris |
| UCDFST 72-1017 | Сокотечение дуба Q. lobata, Калифорния, США, 1972 | K. pastoris |
| UCDFST 72-256 | Источник неизвестен, Калифорния, США, 1972 | K. pastoris |
| UCDFST 96-151.1 | Сокотечение клена Acer saccharum, Калифорния, США, 1996 | K. pastoris |
| UCDFST 96-151.2 | Сокотечение клена Acer saccharum, Калифорния, США, 1996 | K. pastoris |
| K. populi | ||
| NRRL YB-455T | Сокотечение тополя Populus deltoides, Иллинойс, США, 1945 | K. populi |
| K. pseudopastoris | ||
| NRRL Y-27603T | Гниющая древесина ивы Salix alba, Венгрия, 2001 | K. pseudopastoris |
| NRRL Y-27602 | Гниющая древесина ивы Salix alba, Венгрия, 2001 | K. pseudopastoris |
| NRRL Y-27604 | Гниющая древесина ивы Salix alba, Венгрия, 2001 | K. pseudopastoris |
| NRRL Y-27605 | Гниющая древесина ивы Salix alba, Венгрия, 2001 | K. pseudopastoris |
Примечание. Сокращенные названия коллекций: ВКПМ – Всероссийская коллекция промышленных микроорганизмов, Москва; NRRL – USDA-ARS Culture Collection, National Center for Agricultural Utilization Research, Пеория, США; UCDFST – Phaff Yeast Collection, University of California, Дэвис, США. T – типовая культура ВКПМ Y-727= UCDFST 76-20.
Полимеразную цепную реакцию осуществляли на ДНК-амплификаторе ”Bio-Rad” (США). Дрожжевую ДНК выделяли с помощью набора Genomic DNA Purification Kit (“Fermentas”, Литва). Для амплификации домена D1/D2 26S рДНК, 5.8S-ITS-фрагмента (ген 5.8S РНК и внутренние транскрибируемые спейсеры ITS1/ITS2), фактора элонгации трансляции EF-1α и гена субъединицы RPB1 РНК полимеразы II использовали стандартные праймеры (Kurtzman, Robnett, 1998, 2003; Kurtzman, 2009). ПЦР проводили в 30 мкл буфера, содержащего 2.5 мМ MgCl2, 0.1 мМ каждого дНТФ, 50 пмоль каждого праймера, 2.5 единицы Taq-полимеразы (“Синтол”, Россия), 20–200 нг ДНК. Начальную денатурацию осуществляли при 94°С в течение 3 мин, затем 30 циклов в следующем режиме: денатурация при 94°С – 45 с, отжиг праймеров при 52°С – 30 с, синтез ДНК при 72°С – 120 с; конечная достройка при 72°С – 10 мин. Продукты амплификации подвергали электрофорезу в 1%-ном агарозном геле при 60–65 В в 0.5× TBE буфере (45 мМ трис, 10 мМ ЭДТА, 45 мМ борная кислота, рН 8.0) в течение 2–3 ч. Гель окрашивали бромистым этидием, промывали в дистиллированной воде и фотографировали в ультрафиолетовом свете на транс-иллюминаторе Vilber Lourmat (Франция). В качестве маркера молекулярных весов использовали 1kb DNA Ladder (“Fermentas”, Литва).
Секвенирование. Амплифицованныe фрагменты элюировали из геля с помощью набора DNA Extraction Kit (“Fermentas”, Литва) согласно протоколу фирмы изготовителя. Нуклеотидные последовательности домена D1/D2, 5.8S-ITS-участка, EF-1α и RPB1 определяли по двум цепям с помощью прямого секвенирования по методу Сенгера на автоматическом секвенаторе “Applied Biosystems 3730” (США).
Филогенетический анализ. Полученные нуклеотидные последовательности анализировали при помощи программы SeqMan package (“DNA Star Inc.”, США). Поиск гомологии с известными нуклеотидными последовательностями проводили в базе данных GenBank (http://www.ncbi.nlm.nih.gov/ genbank/) с помощью программы BLAST. Множественное выравнивание изученных нуклеотидных последовательностей осуществляли с использованием программы BioEdit (http://www.mbio.ncsu.edu/ BioEdit/bioedit.html). Филогенетические деревья строили методом ближайших соседей (Neighbor-Joining) в программе MEGA 6 (Tamura et al., 2013). В качестве внешней группы использовали типовые культуры Ogataea glucozyma NRRL YB-2185 и Pichia membranifaciens NRRL Y-2026. Индексы бутстрепа, определяющие статистическую достоверность выделения групп, определяли для 1000 псевдореплик.
РЕЗУЛЬТАТЫ
Современная классификация аскомицетовых дрожжей основана на филогенетическом анализе ряда молекулярных маркеров, прежде всего домена D1/D2 гена 26S рРНК (Kurtzman, Robnett, 1998; Kurtzman, 2011). Мы провели секвенирование домена D1/D2 у 22 коллекционных штаммов Komagataella, которые на основании стандартных таксономических тестов были ранее отнесены к Komagataella (Pichia) pastoris (табл. 1). Полученные нуклеотидные последовательности сравнили с последовательностями D1/D2-участка типовых культур семи известных видов Komagataella: K. pastoris (NRRL Y-1603), K. phafii (NRRL Y-7556), K. kuztzmanii (ВКПМ Y-727), K. ulmi (NRRL Y-407), K. populi (NRRL YB-455), K. pseudopastoris (NRRL Y-27603) и K. mondaviorum (UCDFST 71-1024). В анализ были включены еще четыре штамма K. mondaviorum: UCDFST 54-11.16, UCDFST 54‑11.141, UCDFST 68-967.1 и UCDFST 74-1030 (табл. 1).
Семь изолированных в Калифорнии штаммов (UCDFST: 67-1008, 71-1016, 71-1036, 72-1017, 72-256, 96-151.1 и 96-151.2) и UCDFST 49-54, выделенный из сокотечения вяза в штате Миссури, имели идентичные D1/D2-последовательности с типовой культурой K. ulmi NRRL Y-407. Согласно проведенному анализу, пять штаммов (UCDFST: 54-11.214, 54-11.229, 56-57, 52-155 и 68-1033.1) относятся к K. phaffii (табл. 1). Подтверждена принадлежность четырех штаммов (UCDFST 60-58, UCDFST 68-773.2, LE2B88 и LU2T88) к виду K. pastoris. Штаммы UCDFST 54-11.16, UCDFST 54-11.141 и UCDFST 68-967.1 имели идентичные D1/D2-последовательности, которые отличались от соответствующих последовательностей дрожжей UCDFST 71-1024 (типовая культура K. mondaviorum), UCDFST 74-1030, UCDFST 72-1033 и UCDFST 77-1019 одной нуклеотидной заменой. Последовательности домена D1/D2 штаммов UCDFST 68-692.1, UCDFST 68-974.1 и UCDFST 68-891.2 отличались от соответствующих последовательностей указанных семи штаммов 1‒2 нуклеотидными заменами. Ранее нами было показано, что виды K. populi, K. pseudopastoris и K. mondaviorum не могут быть дифференцированы только на основании D1/D2-последовательностей (Naumov et al., 2018).
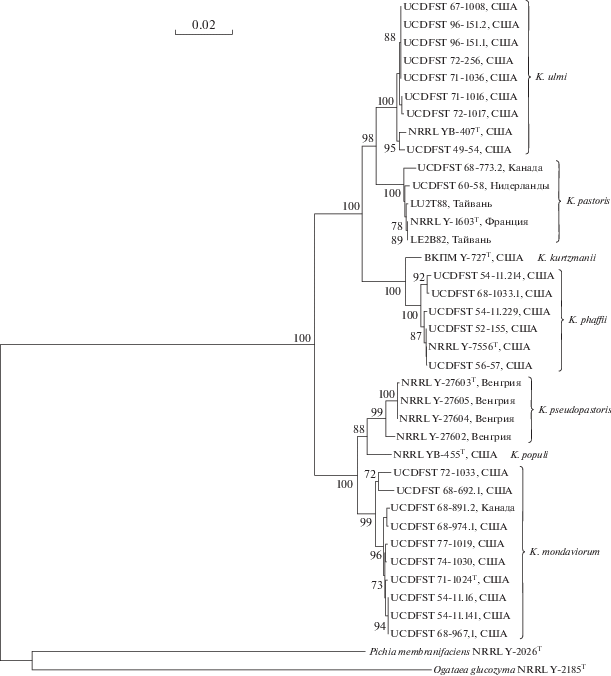
Для установления филогенетического родства 22 изученных штаммов Komagataella и выяснения таксономического статуса штаммов UCDFST 68-692.1, UCDFST 68-974.1, UCDFST 68-891.2, UCDFST 72-1033, UCDFST 77-1019 мы провели сравнительный анализ нуклеотидных последовательностей домена D1/D2, ITS-участка, генов EF-1α и RPB1. На основании полученных нуклеотидных последовательностей было построено филогенетическое древо (рис. 1).
Изученные штаммы Komagataella разделились на два основных кластера. В свою очередь, первый кластер включает три субкластера. Типовая культура K. ulmi NRRL Y-407 и еще 8 штаммов, имеющие идентичные последовательности домена D1/D2 и очень сходные последовательности ядерных генов EF-1α и RPB1, сформировали первый субкластер. Основные различия между штаммами касались ITS-последовательностей. Во втором субкластере со 100%-ной статистической поддержкой объединились штаммы K. pastoris различного происхождения (табл. 1). Штаммы LU2T88, LE2B82 и UCDFST 60-58 не отличались по последовательностям домена D1/D2, ITS-участка и гена RPB1 от типовой культуры NRRL Y-1603, выделенной из сокотечения каштана во Франции. Третий субкластер включает типовую культуру K. phaffii NRRL Y-7556 и еще пять штаммов, изолированных в Калифорнии. Шесть штаммов имеют идентичные D1/D2 и EF-1α последовательности. Единичные нуклеотидные замены были обнаружены только по ITS-последовательностям и гену RPB1. Пять штаммов этого субкластера изолированы из сокотечений черного дуба Quercus kellogii, а UCDFST 52-155 из Drosophila pseudoobscura. К этому субкластеру примыкает типовая культура K. kurtzmanii ВКПМ Y-727, которая существенно отличается от штаммов K. phaffii по последовательностям всех четырех анализируемых молекулярных маркеров.
Второй кластер разделен на два субкластера. Первый включает четыре штамма K. pseudopastoris, выделенные из гниющей древесины ивы Salix alba в Венгрии. К этому субкластеру примыкает типовая культура K. populi NRRL YB-455, изолированная из сокотечения тополя в штате Иллинойс.
Во втором субкластере (99% статистической поддержки) объединились 10 штаммов K. mondaviorum (рис. 1). Большинство штаммов этого субкластера выделены в Калифорнии из сокотечений тополя и черного дуба. Штаммы UCDFST 68-967.1 и UCDFST 68-974.1 изолированы в штате Вашингтон, UCDFST 68-692.1 на Аляске и UCDFST 68-891.2 в Канаде (табл. 1). Это наиболее гетерогенный субкластер. Штаммы UCDFST 54-11.16, UCDFST 54-11.141 и UCDFST 68-967.1 имеют идентичные D1/D2, ITS, EF-1α и RPB1 нуклеотидные последовательности. Практически идентичные последовательности всех четырех молекулярных маркеров имеют типовая культура UCDFST 71-1024 и штаммы UCDFST 74-1030, UCDFST 77-1019. Остальные четыре штамма UCD72-1033, UCD68-692.1, UCDFST 68-974.2 и UCDFST 68-891.2 отличались друг от друга и от остальных штаммов K. mondaviorum по последовательностям ITS-участка, EF-1α и RPB1. Различия между 10 штаммами K. mondaviorum по последовательностям генов EF-1α и RPB1 составили, соответственно, 0–4 и 0–9 нуклеотидных замен. В то же время, EF-1α и RPB1 последовательности штаммов K. mondaviourum отличаются от соответствующих последовательностей видов K. populi и K. pseudopastoris более чем 10 и 28 нуклеотидными заменами.
ОБСУЖДЕНИЕ
С помощью мультигенного филогенетического анализа мы провели кардинальную реидентификацию штаммов Komagataella, которые были выделены в разные годы (с 1919 по 1996) из различных природных источников (табл. 1). Из 22 штаммов, полученных как K. pastoris, указанная видовая принадлежность была подтверждена только у UCDFST 60-58 и UCDFST 68-773.2, LU2T88 и LE2B88 (табл. 1, рис. 1). Восемь штаммов были реидентифицированы как K. ulmi, по пять как K. phaffii и K. mondaviorum (табл. 1). Следует отметить, что среди изученных дрожжей нам не удалось обнаружить новых штаммов K. kurtzmanii, K. populi и K. pseudopastoris.
Рис. 1.
Филогенетический анализ нуклеотидных последовательностей домена D1/D2, 5.8S-ITS-участка, фактора элонгации EF-1α и гена субъединицы RPB1 РНК полимеразы II дрожжей рода Komagataella. В качестве внешней группы использовали типовые культуры Ogataea glucozyma NRRL YB-2185 и Pichia membranifaciens NRRL Y-2026. Шкала соответствует 20 нуклеотидным заменам на 1000 нуклеотидных позиций. Приводятся значения бутстрепа >70%. T – типовая культура.
Проведенное исследование выявило большое филогенетическое родство дрожжей Komagataella различного происхождения, имеющих сходные D1/D2-последовательности. В то же время, различие по домену D1/D2 с филогенетически ближайшим родом Phaffomyces составляет более 80 нуклеотидных замен (Наумов, 2015). Значительные различия между штаммами семи видов Komagataella обнаружены по последовательностям ITS-участка, EF-1α и RPB1. Более вариабельными являются ITS1-последовательности изученных штаммов, тогда как ITS2-последовательности характеризуются внутривидовой консервативностью. Так, все штаммы, отнесенные к K. pastoris, K. phaffii, K. ulmi и K. mondaviorum, имели идентичные ITS2-последовательности с соответствующими типовыми культурами. Молекулярные маркеры EF-1α и RPB1 также характеризовались низким внутривидовым полиморфизмом.
Следует отметить, что семь видов Komagataella практически неразличимы на основании стандартных морфологических и физиологических тестов. Исключением является K. kurtzmanii, который в отличие от остальных шести видов не способен ассимилировать трегалозу (Naumov et al., 2013, 2018). Учитывая изменчивость физиологических признаков и то, что K. kurtzmanii и K. populi представлены единичными штаммами, невозможно дифференцировать все семь видов с помощью стандартных таксономических тестов. Следовательно, для достоверной видовой идентификации штаммов Komagataella необходимо использовать мультигенный филогенетический анализ.
Установленный филогенетическим анализом род Komagataella полностью соответствует концепции генетического рода у аскомицетовых грибов: составляющие его виды имеют общую систему типов спаривания, позволяющую им скрещиваться (Наумов, 1978, 2015). Виды рода Komagataella обладают пост-зиготической изоляцией и формируют стерильные гибриды, имеющие нежизнеспособные аскоспоры (Наумов и соавт., 2016). Обращает на себя внимание биогеография дрожжей Komagataella. Штаммы пяти видов (K. mondaviorum, K. kurtzmanii, K. phaffii, K. populi и K. ulmi) встречаются в Северной Америке, тогда как K. pastoris и K. pseudopastoris характерны для Европы (Dlauchy et al., 2003; Kurtzman, 2011; настоящее исследование). Идентифицированные нами как K. pastoris штаммы LU2T88 (Тайвань) и UCDFST 68-773.2 (Канада), по-видимому, является инвазивными из Европы. Это хорошо согласуется с тем, что, несмотря на многолетние исследования дрожжей, только единичные изоляты Komagataella были обнаружены в Японии (Phaff et al., 1972; Kodama, 1974; Banno, Mikata, 1981).
Следует также отметить экологические особенности видов Komagataella – ассоциацию с широколиственными деревьями (табл. 1). Исключением является типовая культура K. kurtzmanii, выделенная из течи хвойного дерева – пихты в Южной Аризоне (США). Проведенное нами исследование не подтвердило уникальность видового эпитета дрожжей K. ulmi, типовая культура которого NRRL Y-407 выделена из вяза. Среди штаммов, отнесенных нами к этому виду, есть также изоляты, выделенные из сокотечений различных видов дубов и клена (табл. 1). Все известные четыре штамма K. pseudopastoris были изолированы в Венгрии из гниющей древесины ивы Salix alba (Dlauchy et al., 2003). Интересно отметить, что дрожжи K. pseudopastoris не были обнаружены ни в одной из 50 взятых с дуба проб, тогда как штаммы K. pastoris выделялись с высокой частотой. По-видимому, это связано с повышенной чувствительностью K. pseudopastoris, по сравнению с K. pastoris, к дубильной кислоте, содержащейся в дубах Quercus spp. (Dlauchy et al., 2003; Peter et al., 2019).
Принимая во внимание большое биотехнологическое и научное значение дрожжей Komagataella целесообразно продолжить изучение экологической генетики и биогеографии дрожжей рода Komagataella. Молекулярно-генетическое изучение штаммов Komagataella (Pichia) pastoris из различных дрожжевых коллекций и выделение новых штаммов из природы позволит обнаружить новые виды и дополнительные штаммы уже известных видов этого рода.
Список литературы
Наумов Г.И. Генетическая концепция рода у грибов // ДАН СССР. 1978. Т. 241. С. 952‒954.
Naumov G.I. Genetic concept of genus in fungi // Dokl. Biol. Sciences. 1979. V. 241. P. 345‒347.
Наумов Г.И. Дрожжи Komagataella – генетический род согласно межвидовой гибридизации // Микробиология. 2015. Т. 84. С. 449–455.
Naumov G.I. The yeast Komagataella: a genetic genus in accordance with interspecies hybridization // Microbiology (Moscow). 2015. V. 84. P. 538–543.
Наумов Г.И., Кондратьева В.И., Мещеряковова Е.В., Наумова Е.С. Генетическое изучение метилотрофных дрожжей рода Komagataella: новый биологический вид K. kurtzmanii // Генетика. 2016. Т. 52. С. 431–435.
Naumov G.I., Kondratieva V.I., Meshcheryakova E.V., Naumova E.S. Taxonomic genetics of methylotrophic yeast genus Komagataella: new biological species K. kurtzmanii // Russ. J. Genet. 2016. V. 52. P. 378–382.
Banno I., Mikata K. Ascomycetous yeasts isolated from forest materials in Japan // IFO Res. Comm. 1981. V. 10. P. 10‒19.
Chen M.T., Lin S., Shandil I., Andrews D., Stadheim T.A., Choi B.K. Generation of diploid Pichia pastoris strains by mating and their application for recombinant protein production // Microb. Cell Fact. 2012. V. 11. P. 91.
Cregg J.M., Madden K.R. Development of the methylotrophic yeast, Pichia pastoris, as a host system for the production of foreign proteins // Dev. Ind. Microbiol. 1988. V. 29. P. 33–41.
Cregg J.M., Vedvick T.S., Raschke W.C. Recent advances in the expression of foreign genes in Pichia pastoris // Biotechnol. 1993. V. 11. P. 905–910.
Dlauchy D., Tornai-Lehoczki J., Fülöp L., Péter G. Pichia (Komagataella) pseudopastoris sp. nov., a new yeast species from Hungary // Antonie van Leeuwenhoek. 2003. V. 83. P. 327–332.
Gasser B., Mattanovich D. A yeast for all seasons ‒ is Pichia pastoris a suitable chassis organism for future bioproduction? // FEMS Microbiol. Lett. 2018. V. 365. P. 1–4.
Guilliermond A. Sur une nouvelle levure à copulation hétérogamique // C.R. Soc. Biol. 1919. № 82. P. 466–470.
Kodama K. Ascosporogenous yeasts isolated from tree exudates in Japan // Ann. Microbiol. Enzimol. 1974. № 24. P. 215–231.
Kurtzman C.P. Description of Komagataella phaffii sp. nov. and the transfer of Pichia pseudopastoris to the methylotrophic yeast genus Komagataella // Int. J. Sys. Evol. Microbiol. 2005. V. 55. P. 973–976.
Kurtzman C.P. Biotechnological strains of Komagataella (Pichia) pastoris are Komagataella phaffii as determined from multigene sequence analysis // J. Ind. Microbiol. Biotechnol. 2009. V. 36. P. 1435–1438.
Kurtzman C.P. Komagataella populi sp. nov. and Komagataella ulmi sp. nov., two new methanol assimilating yeasts from exudates of deciduous trees // Antonie van Leeuwenhoek. 2012. V. 101. P. 859–868.
Kurtzman C.P. Komagataella Y. Yamada, Matsuda, Maeda & Mikata 1995 // The Yeast. A Taxonomic Study / Eds. Kurtzman C.P., Fell J., Boekhout T. 5th ed. Amsterdam: Elsevier, 2011. V. 2. P. 491–495.
Kurtzman C.P., Robnett C.J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences // Antonie Van Leeuwenhoek. 1998. V. 73. P. 331–371.
Kurtzman C.P., Robnett C.J. Phylogenetic relationships among yeasts of the “Saccharomyces complex” determined from multigene sequence analyses // FEMS Yeast Res. 2003. V. 3. P. 417–432.
Love K.R., Shah K.A., Whittaker C.A., Wu J., Bartlett M.C., Ma D., Leeson R.L., Priest M., Borowsky J., Young S.K., Love J.C. Comparative genomics and transcriptomics of Pichia pastoris // BMC Genomics. 2016. V. 17. P. 1–17.
Naumov G.I., Naumova E.S., Boundy-Mills K.L. Description of Komagataella mondaviorum sp. nov., a new sibling species of Komagataella (Pichia) pastoris // Antonie van Leeuwenhoek. 2018. V. 111. P. 1197–1207.
Naumov G.I., Naumova E.S., Tyurin O.V., Kozlov D.G. Komagataella kurtzmanii sp. nov., a new sibling species of Komagataella (Pichia) pastoris in accordance with multigene sequence analysis // Antonie van Leeuwenhoek. 2013. V. 104. P. 339‒347.
Péter G., Nagy E.S., Dlauchy D. Systematics, diversity and ecology of the genus Yarrowia and the methanol-assimilating yeasts // Non-Conventional Yeasts: from Basic Research to Application / Ed. Sibirny A. Switzerland AG.: Springer Nature, 2019. P. 297–339.
Phaff H.Y. A proposal for amendment of the diagnosis of the genus Pichia Hansen // Antonie van Leeuwenhoek. 1956. V. 22. P. 113–116.
Phaff H.J., Miller M.W., Yoneyama M., Soneda M. A comparative study of the yeast florae associated with trees from the Japanese islands and on the west coast of North America // Fermentation Technology Today: Proceedings IV IFS / Ed. Terui G. Osaka. Society of Fermentation Technology, 1972. P. 759–774.
Tamura K., Stecher G., Peterson D., Filipski A., Kumar S. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0 // Mol. Biol. Evol. 2013. V. 30. P. 2725–2729.
Tyurin O., Gubaidullin I., Cheperegin S., Efremov B., Naumova E., Naumov G., Kozlov D. The new expression system based on a novel yeast species of the genus Komagataella // FEBS J. 2013. V. 280. Suppl. 1. P. 602.
Valli M., Tatto N.E., Peymann A., Gruber C., Landes N., Ekker H., Thallinger G.G., Mattanovich D., Gasser B., Graf A.B. Curation of the genome annotation of Pichia pastoris (Komagataella phaffii) CBS 7435 from gene level to protein function // FEMS Yeast Res. 2016. V. 16. pii: fow051.
Yamada Y., Matsuda M., Maeda K., Mikata K. The phylogenetic relationships of methanol-assimilating yeasts based on the partial sequences of 18S and 26S ribosomal RNAs: the proposal of Komagataella gen. nov. (Saccharomycetaceae) // Biosci. Biotechnol. Biochem. 1995. V. 59. P. 439–444.
Zahrl R.J, Peña D.A, Mattanovich D., Gasser B. Systems biotechnology for protein production in Pichia pastoris // FEMS Yeast Res. 2017. V. 17. P. 1–15. https://doi.org/10.1093/femsyr/fox068
Дополнительные материалы отсутствуют.


