Микробиология, 2020, T. 89, № 6, стр. 646-657
Молекулярно-генетический и функциональный анализ плазмиды PBS72 природных бактерий Bacillus subtilis
А. С. Гуринович a, М. А. Титок a, *
a Белорусский государственный университет, биологический факультет, кафедра микробиологии
220030 Минск, Республика Беларусь
* E-mail: ma_titok@bsu.by
Поступила в редакцию 01.04.2020
После доработки 07.07.2020
Принята к публикации 13.07.2020
Аннотация
В результате анализа полной нуклеотидной последовательности плазмиды pBS72 природных бактерий Bacillus subtilis 72 размером 102 254 п.н. охарактеризованы генетические локусы, ответственные за ее вертикальный и горизонтальный перенос. Установлено, что репликоны, подобные плазмиде pBS72, содержатся в клетках бактерий B. subtilis, выделенных из различных природных источников на территории Пакистана и Нидерландов (сходство Rep-белков составляло 99%). Показано, что релаксаза плазмиды pBS72, обеспечивающая ее конъюгационный перенос, относится к семейству MobL. Всего в составе генома плазмиды pBS72 определено 142 открытые рамки считывания, большинство из которых (93) кодировали синтез гипотетических полипептидов. Среди известных белков бактерий Bacillus, детерминируемых генами плазмиды pBS72, выявлены полипептиды, определяющие взаимодействие бактериальной клетки с внешней средой, защиту от чужеродной ДНК и повреждений, участвующие в клеточном метаболизме и выполняющие регуляторные функции. Создана система, позволяющая установить роль плазмиды в жизнедеятельности природных и коллекционных бактерий B. subtilis. Показано, что в среде с добавлением 13% NaCl, при значениях рН 4.5 и рН 11 присутствие плазмиды увеличивало выживаемость природных бактерий B. subtilis 72 и коллекционных бактерий B. subtilis 168 в 4.33 и 13 раз, в 1.75 и 1.95 раза, в 1.75 и 9.12 раза соответственно. В результате ультрафиолетового облучения выживаемость плазмидсодержащих бактерий B. subtilis 72 возрастала в 2.25 раза, а бактерий B. subtilis 168 – в 4.5 раза. При этом природные бактерии B. subtilis 72 характеризовались более выраженными адаптивными свойствами, что может свидетельствовать о наличии в их хромосоме дополнительных генетических детерминант, продукты которых при взаимодействии с белками плазмидного происхождения обеспечивали устойчивость клеток к повышенной осмолярности, высокой и низкой концентрации ионов водорода, а также УФ-облучению.
Повсеместно распространенные в природной среде обитания бактерии Bacillus subtilis, способные утилизировать широкий спектр органических и неорганических субстратов и продуцировать во внешнюю среду биологически активные соединения, широко применяются в биотехнологии в качестве продуцентов ферментов, антибиотиков, стимуляторов роста растений и животных (Harwood, 2018). Генетический аппарат микроорганизмов данной таксономической группы может быть представлен генами хромосомного и внехромосомного происхождения. При этом присутствующие в клетках этих микроорганизмов внехромосомные генетические элементы могут копироваться в соответствии с механизмом “разматывающегося рулона” (RCR-типа) либо иметь тета-тип репликации. Следует отметить, что все описанные плазмиды RCR-типа бактерий B. subtilis относятся к семейству рС194, представители которого широко распространены среди грамположительных бактерий разных систематических групп (в частности, Staphylococcus aureus, Streptococcus faecalis, Shigella sonnei, Lactobacillus plantarum, Bacillus coagulans, B. cereus, B. pumilus, Lactococcus lactis, Clostridium butyricum и многих других). Отличительной особенностью данных плазмид бактерий B. subtilis является наличие в их составе mob/pre локусов, детерминирующих их конъюгативный перенос путем мобилизации, а также генов клеточного метаболизма, в частности, определяющих процесс секреции белков (sip), регуляции процесса споруляции (rap), а также обеспечивающих синтез белков теплового шока (hsp) (Meijer, 1998). В настоящее время для бактерий B. subtilis охарактеризовано несколько плазмид тета-типа (pLS20, pLS32 и pBS72). Плазмиды pLS32 размером 85 603 п.н. и pLS20 размером 65 774 п.н. имеют системы инициации репликации, характерные для внехромосомных генетических элементов бактерий Enterococcus faecalis и Bacillus thuringiensis соответственно (Meijer, 1995; Tanaka, 1998). Присутствие плазмиды pLS32 влияло на компетентность, споруляцию и образование биопленок у бактерий B. subtilis (Tanaka, 1998). Конъюгативная плазмида pLS20 обеспечивала содержащим ее бактериям устойчивость к стрессовым факторам среды и содержала уникальную систему конъюгационного переноса, определяющую горизонтальный перенос генов (Rösch, 2014; Ramachandran, 2017). Плазмида pBS72, широко распространенная среди природных бактерий B. subtilis, выделенных из почвы на территории Беларуси, обладала уникальной системой инициации репликации и с высокой частотой обеспечивала конъюгационный перенос плазмид семейства рС194 (Titok, 2003; Poluektova, 2004). Наличие полной нуклеотидной последовательности позволяет изучить особенности генетической организации данной плазмиды, а также в полной мере манипулировать данным репликоном для установления его роли в жизнедеятельности бактериальной клетки.
Целью настоящего исследования являлось изучение особенностей генетической организации плазмиды pBS72 и ее влияния на жизнеспособность бактерий B. subtilis в стрессовых условиях среды.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
В работе использовали природные бактерии B. subtilis, содержащие плазмиду pBS72 (Titok, 2003), штаммы B. subtilis 168 (Anagnostopoulos, 1961) и E. coli XL1-Blue (Bullock, 1987), а также векторы pMTL21E (Chambers, 1988) и pAL1 (Лагодич, 2005). Бактерии выращивали в полноценной среде LB при 37°С (Harwood, 1990). Агаризованные среды содержали 1.5% агара, источником углерода служила глюкоза в концентрации 0.2%. В работе использовали коммерческие препараты ампициллина, хлорамфеникола и эритромицина в концентрациях 100, 5 и 10 мкг/мл соответственно. Изопропилтио-β-D-галактозид (IPTG) и 5-бромо-4-хлоро-3-индоил-β-D-галактопиранозид (X-Gal) производства “Thermo Scientific” (ЕС) готовили в соответствии с рекомендациями изготовителя и использовали в концентрации 0.5 мM и 50 мкг/мл соответственно.
Тотальную ДНК выделяли саркозиловым методом (te Riele, 1986). Плазмидную ДНК выделяли с использованием набора реактивов Fast-n-Easy Plasmid Mini-Prep Kit производства “Jena Bioscience” (Германия).
Трансформацию бактерий E. coli и B. subtilis осуществляли согласно методам, приведенным в работах (Sambrook, 1989; Harwood, 1990).
Скрещивание бактерий B. subtilis осуществляли согласно методике, приведенной в работе (Poluektova, 2004).
Для амплификации использовали HF ДНК-полимеразу производства “Thermo Scientific” (ЕС) и праймеры производства ОДО “Праймтех” (Республика Беларусь). Реакционная смесь для ПЦР (50 мкл) содержала около 100 нг ДНК-матрицы, 0.2 ммоль/л каждого дНТФ, 0.5 мкмоль/л каждого праймера, 2.5 ед. ДНК-полимеразы и соответствующий буфер.
Для амплификации фрагмента плазмиды pBS72 размером 1063 п.н. (orf 52) использовали праймеры F1 (5'-TTA GCA ACA TTC TCA GCT TGC-3') и R1 (5'-GTT CAT TAG TCG CAG ATC TG-3') при режиме амплификации: 94°С – 5 мин (1 цикл); 94°С – 30 с, 50°С – 45 c, 72°С – 1 мин 10 с (5 циклов); 94°С – 30 с, 55°С – 30 с, 72°С – 1 мин 10 с (20 циклов); 72°С – 5 мин (1 цикл). Полученные продукты ПЦР очищали с помощью набора Agarose Gel Extraction Kit производства “Jena Bioscience” (Германия). Очищенные продукты лигировали с суицидальным вектором pMTL21E, предварительно обработанным рестриктазой SmaI.
Рестрикцию плазмиды pMTL21E, содержащей фрагмент плазмиды pBS72 размером 1063 п.н., проводили ферментами XbaI и KpnI в условиях, рекомендуемых фирмой-изготовителем (“Thermo Scientific”, ЕС).
Электрофоретический анализ ДНК осуществляли общепринятыми методами, приведенными в руководстве (Sambrook, 1989). Размер фрагментов ДНК устанавливали на основании их электрофоретической подвижности в агарозном геле, в качестве реперной ДНК использовали GeneRuler™ DNA Ladder Mix производства “Thermo Scientific” (ЕС).
Жизнеспособность бактерий определяли на основании значений ОП600 и КОЕ/мл после воздействия на бактериальную культуру в логарифмической фазе роста стрессовых факторов (рН 11, рН 4.5, 13% NaCl) в течение 1, 2, 3 и 4 ч, а также после воздействия ультрафиолетом в течение 5, 10, 15, 20, 25 и 30 с. В зависимости от времени доза облучения составляла от 75 до 450 Дж. Статистическую обработку данных (3 повторности каждого эксперимента) проводили с использованием программы Microsoft Excel 2010.
Для анализа генов и детерминируемых ими белков, входящих в состав плазмиды pBS72 (регистрационный номер в ГенБанке NCBI – KX711616), использовали программу BLASTN2.10.0 (сайт: https://blast.ncbi.nlm.nih.gov).
Для поиска релаксаз семейств MobL, MobP, MobQ и MobV использовали типовые аминокислотные последовательности релаксаз плазмид pLS20 (MobL), RP4 (MobP), RSF1010 (MobQ) и pMV158 (MobV). С помощью программы USEARCH (версия 11.0.667_i86linux32) удаляли избыточные последовательности (Edgar, 2010).
Для поиска функциональных доменов в составе релаксазы плазмиды pBS72 использовали программу MEME (сайт http://meme-suite.org/index.html, версия 5.1.1.) с пороговым значением E ниже 1e-27. Для поиска использовали до десяти консервативных последовательностей, характерных для каждого типа релаксаз.
Для построения филогенетического дерева релаксаз семейства MobL размером около 300 аминокислотных остатков применяли метод ближайших соседей (1000 бутстрепов) в программе MEGA7 (Kumar, 2016). С помощью программы USEARCH для построения использовались аминокислотные последовательности, отличающиеся между собой более чем на 50%.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Наличие полной нуклеотидной последовательности плазмиды pBS72 позволило установить некоторые особенности ее генетической организации и сравнить с известными входящие в ее состав гены и детерминируемые ими белки. В результате сравнительного анализа генома данной плазмиды было установлено, что она представляет собой самый крупный внехромосомный генетический элемент бактерий B. subtilis. Ее размер составил 102 254 п.н. Более крупные плазмиды выявлялись в клетках бактерий рода Bacillus других таксономических групп (в частности, B. cereus, B. anthracis, B. thuringiensis, B. weihenstephanensis) (Shintani, 2015). Геном плазмиды pBS72 содержал 35% Г + Ц, что на 10% меньше ГЦ-состава хромосомы бактерий хозяев. Следует отметить, что низкое содержание пар Г + Ц, снижающее энергетические затраты клетки на их стабильное поддержание, характерно для внехромосомных генетических элементов грамположительных бактерий (Shintani, 2015). В составе плазмиды выявлено 142 открытые рамки считывания, большинство из которых (93) определяли синтез гипотетических полипептидов с неизвестными функциями. Определенный интерес представляли детерминанты, обеспечивающие инициацию репликации и конъюгационный перенос данного внехромосомного генетического элемента.
Анализ нуклеотидной последовательности rep-гена позволил выявить гомологию между С-концевой последовательностью кодируемого им белка и N-концевой последовательностью DnaA-белка грамположительных и грамотрицательных бактерий, обеспечивающего инициацию репликации хромосомы. Известно, что данный функциональный домен DnaA-белка (локализовался между 275 и 330 аминокислотными остатками) определяет его олигомеризацию в области oriC и последующее взаимодействие с репликативной геликазой, что ведет к образованию затравочного комплекса при репликации хромосомы грамположительных и грамотрицательных бактерий (Weigel, 1999; Messer, 2002). Кроме того, белковый продукт, детерминируемый rep-геном, содержал НТН-последовательность (локализовался между 88 и 141 аминокислотными остатками), определяющую взаимодействие с молекулой ДНК. Следует отметить, что Rep-белок плазмиды pBS72, имея сходство с белками инициации репликации бактериальных хромосом, не обнаруживал сходство с гомологичными полипептидами других внехромосомных генетических элементов, на основании чего был отнесен к новому типу репликонов (Titok, 2003). Поскольку информация о полных нуклеотидных последовательностях бактериальных геномов обновляется достаточно быстро, был проведен сравнительный анализ Rep-белка плазмиды pBS72 с известными, депонированными в ГенБанк NCBI. Как видно из данных, приведенных в табл. 1 и на рис. 1, идентичные на 99% белки выявлены в геномах пяти штаммов бактерий B. subtilis, выделенных на территории Пакистана и Нидерландов (B4071, MB378, MB415, EH11, DH12). Анализируя эти близкородственные полипептиды, можно выявить некоторые закономерности. Во-первых, для генов, детерминирующих синтез этих белков, не установлена плазмидная локализация (они аннотированы в составе отдельных фрагментов геномов), и данные белки отнесены к разряду гипотетических. Во-вторых, штаммы, содержащие данные детерминанты, не привязаны к специфическим природным источникам. Идентифицированные как B. subtilis, они выделены из разных мест (кари суп, лесная и луговая почва, загрязненная нефтью почва) (табл. 1). Скорее всего, присутствие в изолированных бактериях плазмид не обеспечивало содержащим их микроорганизмам определенных фенотипических признаков (например, деградативных свойств либо устойчивости к определенным соединениям), что создавало сложности в обнаружении внехромосомных генетических элементов такого большого размера. В-третьих, подобно Rep-белку плазмиды pBS72, гомологичные полипептиды содержали два функциональных домена с одинаковой локализацией. Это НТН-домен (расположен между аминокислотными остатками 88 и 141) и последовательность между аминокислотными остатками 275 и 345, имеющая сходство с N-концевой последовательностью белка DnaA. При этом данные белки отличались от Rep-белка плазмиды pBS72 четырьмя несинонимическими заменами, три из которых в позиции 255 (T → P), 310 (S → N) и 318 (K → Q) должны сказываться на функциональной активности данных полипептидов (рис. 1).
Таблица 1.
Сходство белков плазмиды pBS72, определяющих процесс инициации репликации и конъюгационного переноса, с известными белками
| Штамм | Источник выделения | Страна выделения | Идентичность белков плазмиды pBS72 с известными (в %)/номер нуклеотидной последовательности в ГенБанке NCBI | |
|---|---|---|---|---|
| Rep (orf1) | релаксаза (orf37) | |||
| B. subtilis 19 | Смешанный лес | Беларусь | 100 EF506610 |
100 FJ434456 |
| B. subtilis MB378 | Загрязненная почва | Пакистан | 99 MBPE01000006 (NODE_15) |
100 MBPE01000006 (NODE_15) |
| B. subtilis MB415 | Загрязненная нефтью почва | Пакистан | 99 MQSR01000011 (NODE_14) |
100 MQSR01000011 (NODE_14) |
| B. subtilis B4071 | Кари суп | Нидерланды | 99 JXHN01000026 (NODE_32) |
99 JXHN01000031 (NODE_37) |
| B. subtilis EH11 | Почва луговая | Нидерланды | 99 RPHE01000007 (NODE_7) |
99 NZ_RPHE01000007 (Node_7) |
| B. subtilis DH12 | Почва луговая | Нидерланды | 99 RQPH01000007 (NODE_7) |
99 NZ_RQPH01000007 (Node_7) |
| B. subtilis SX01705 плазмида pSX01705-1 |
Субстрат для грибов | Китай | 62 CP022288 |
61 CP022288 |
| B. licheniformis YNP5-TSUWRT508 |
Трава в парке | США | 62 MEDD01000008 |
60 MEDD01000008 |
| B. licheniformis YNP1-TSU | Трава в парке | США | 62 MIGE01000004 contig04 |
60 MIGE01000004 contig04 |
| B. licheniformis SRCM103529 плазмида 1 |
Пищевой продукт | Южная Корея |
62 CP035229 |
60 CP035229 |
Рис. 1.
Аминокислотная последовательность Rep-белка плазмиды pBS72 в сравнении с гомологичными белками из ГенБанка NCBI. Верхняя последовательность соответствует Rep-белку плазмиды pBS72, нижняя – гомологичным белкам бактерий B. subtilis (штаммы B4071, MB378, MB415, EH11, DH12). Жирным шрифтом выделены аминокислотные замены.

Кроме того, белки, отличающиеся от Rep-белка плазмиды pBS72 в большей степени (идентичность составила 62%), выявлены в составе плазмиды pSX01705-1 размером 79 987 п.н. бактерий B. subtilis SX01705 (CP022288), плазмиды 1 размером 96 005 п.н. бактерий B. licheniformis SRCM103529 (CP035229) и в составе геномов B. licheniformis (штаммы YNP1-TSU и YNP1-TSU). Данные белки, как и в предыдущем случае, отнесены к разряду гипотетических (табл. 1).
Таким образом, в результате проведенного анализа можно заключить, что репликоны, подобные плазмиде pBS72, широко распространены среди бактерий B. subtilis и B. licheniformis.
Помимо систем репликации, обеспечивающих вертикальный перенос генов, не менее важными являются системы конъюгационного переноса. Данным системам в настоящее время уделяется особое внимание, поскольку именно они определяют горизонтальный перенос генетического материала, играющий большую роль в распространении генов антибиотикорезистентности среди бактерий природных популяций. В отличие от систем репликации, характеризующихся большим разнообразием (в системе грамположительных бактерий на основании организации rep-областей выделяют 18 групп несовместимости) (Shintani, 2015), системы конъюгационного переноса характеризуются более выраженным консерватизмом. Наиболее изученные системы конъюгации содержат определенный набор ключевых генетических детерминант, определяющих перенос внехромосомных генетических элементов между бактериями природных популяций. Во-первых, это гены, детерминирующие синтез белков, образующих канал, по которому передается плазмидная ДНК (VirB-подобные белки системы T4SS). Во-вторых, релаксазы, обеспечивающие разрез плазмиды в области oriТ c образованием свободных концов, один из которых передается в реципиентную клетку (5'-конец), а второй служит затравкой для репликации плазмиды в клетке донора (3'-конец). И, наконец, белок, обеспечивающий взаимодействие между релаксированной плазмидной ДНК и белками канала, пронизывающих поверхностные структуры клетки (белок T4CP). На основании сходства белков T4SS, Т4СР и релаксаз строится система классификации конъюгативных и мобилизуемых плазмид грамположительных и грамотрицательных бактерий. В частности, на основании гомологии релаксаз все плазмиды делят на 6 основных семейств (MobР, MobH, MobQ, MobF, MobV, MobC) (Smillie, 2010). Детальный анализ релаксазы плазмиды pLS20 бактерий B. subtilis позволил отнести ее к новому семейству, обозначенному как MobL (Ramachandran, 2017).
Анализ белка, кодируемого открытой рамкой считывания (orf37) плазмиды pBS72, позволил выявить функционально значимые домены, характерные для релаксаз, и сравнить их с гомологичными последовательностями классификационных групп MobL, MobР, MobQ и MobV. Подобно релаксазам семейства MobL, в исследуемом белке выявлено 8 консервативных последовательностей (Ramachandran, 2017). При этом в N-концевой части молекулы выявлено три характерных функциональных домена (His-домен, Tyr-домен и Ser-домен), которые необходимы для активации релаксазы (His-домен), разрезания плазмиды в сайте oriТ (Tyr-домен) и связывания с молекулой плазмидной ДНК в области свободного 3'-конца, образовавшегося после разрезания oriТ-сайта (Ser-домен) и выполняющего функцию затравки для репликации по типу “катящегося кольца”. В отношении анализируемых релаксаз (MobL, MobР, MobQ и MobV) наиболее консервативным являлся His-домен, содержащий функционально значимые аминокислотные остатки гистидина, активирующие каталитический центр белка после взаимодействия с ионами металлов (Garcillán-Barcia, 2004) (рис. 2а). В то же время Ser-домен анализируемого белка плазмиды pBS72, включающий реакционно способную аминокислоту серин, был сходен с релаксазами семейства MobL и MobР (в семействе MobQ активность домена обеспечивается аминокислотным остатком глутамата, а для семейства MobV данный домен не выявлен) (Garcillán-Barcia, 2009) (рис. 2б). И, наконец, Tyr-домен, обладающий эндонуклеазной активностью за счет аминокислоты тирозин, обнаруживал сходство с релаксазами семейства MobL, MobР и MobQ (в релаксазах семейства MobV функциональный центр представлен аминокислотой гистидин) (Pluta, 2017) (рис. 2в). Исходя из приведенного анализа, можно заключить, что релаксаза плазмиды pBS72 имеет наибольшее сходство с гомологичными белками семейства MobL. Подтверждением этому могут служить результаты филогенетического анализа релаксаз семейства MobL. Следует отметить, что релаксазы этого семейства весьма гетерогенны. Каждая из четырех филогенетических групп включает разнообразные по аминокислотному составу белки, образующие многочисленные подгруппы (рис. 3). При этом релаксаза плазмиды pLS20 и плазмиды pBS72 входят в разные филогенетические ветви, что свидетельствует о значительном отличии их аминокислотных последовательностей. Действительно, сравнительный анализ функциональных доменов релаксаз плазмиды pBS72 и pLS20 позволил установить, что среди 26 аминокислот, входящих в Tyr-домен, они отличаются между собой 15 аминокислотами, 12 из которых представляют собой функционально значимые несинонимические замены. Такая же картина наблюдается для Ser- и His-доменов (из 36 аминокислот выявляется по 15 несинонимических замен, соответственно, 11 и 9 из которых могут влиять на функциональную активность белка) (рис. 4). Полученный результат объясняет отсутствие сходства между этими белками при поиске гомологичных полипептидов с использованием программы Blast. Гомология выявлялась с ограниченным количеством белков, причем гены, кодирующие данные полипептиды, обнаружены в геномах, способных кодировать гомологичные плазмиде pBS72 Rep-белки (табл. 1). Тем не менее, на основе проведенного анализа можно заключить, что релаксаза плазмиды pBS72, содержащая характерные аминокислотные остатки в пределах функциональных доменов (тирозин, серин и гистидин), относится к семейству MobL.
Рис. 2.
Аминокислотные последовательности доменов релаксаз семейства MobL, MobР, MobQ и MobV: а – His-доменов; б – Ser-доменов; в – Tyr-доменов.
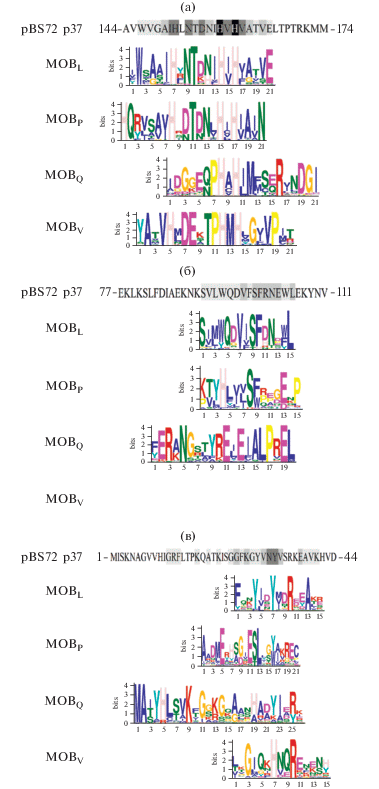
Рис. 3.
Филогенетическое дерево релаксаз семейства MobL. Релаксаза плазмиды pBS72 выделена подчеркиванием.
Рис. 4.
Аминокислотные последовательности Tyr-, Ser- и His-доменов плазмиды pBS72 и pLS20 с указанием функционально значимых (*) и незначимых несинонимических замен (**).

Следует отметить, что гены, детерминирующие синтез наименее сходных между собой релаксаз семейства MobL и MobV, широко распространены среди бактерий группы Firmicutes (обнаружены в геномах 800 и 5765 штаммов соответственно) (Ramachandran, 2017; Pluta, 2017). При этом наиболее часто детерминанты, кодирующие релаксазы семейства MobV, выявлялись в геномах золотистого стафилококка (72% изолятов Staphylococcus aureus содержат MobV релаксазы) и других представителей животной микрофлоры, способных вызывать различные заболевания (Enterococcus faecium – 68%, Enterococcus faecalis – 45%, Staphylococcus agalactiae – 56%). Следует отметить, что гены, кодирующие данные белки, обнаруживались не только в составе мобилизуемых плазмид RCR-типа, но и входили в состав транспозонов размером до 30 т.п.н., половина из которых содержала гены, кодирующие устойчивость к антибиотикам или другие признаки вирулентности (Pluta, 2017). Учитывая способность конъюгативных плазмид, содержащих MobL-релаксазы, передаваться из клетки в клетку, а также обеспечивать с высокой частотой перенос мобилизируемых плазмид, в состав которых входят детерминанты, определяющие синтез MobV-релаксаз (Meijer, 1995; Poluektova, 2004), можно представить масштабы горизонтального переноса генетического материала среди бактерий группы Firmicutes. В подтверждение этому можно привести данные о высокой эффективности переноса генов в почве, воде и в организме человека между бактериями рода Bacillus (Popa, 2011). При этом геномы представителей этой таксономической группы достаточно сильно отличаются по размеру (до 1 млн п.н.) и их становление происходило путем объединения чужеродных фрагментов ДНК (до 90% генов в геномах получены путем горизонтального переноса) (Dagan, 2008).
Анализ других белков, кодируемых генами плазмиды pBS72, позволил выявить ряд полипептидов, способных играть важную роль в жизнедеятельности бактериальной клетки. В частности, Rap-белок (кодируется orf 130), играющий ключевую роль в процессах спорообразования, формирования биопленок и компетентности, а также участвующий в синтезе вторичных метаболитов (Yang, 2015); метилтрансферазы (кодируются orf 58, orf 72, orf 84); белок теплового шока Hsp40 (orf 59); белки UmuC и UmuD (orf 132 и orf 133), способные обеспечивать защиту клетки от чужой ДНК, противостоять стрессу и участвовать в репарации. Особое внимание заслуживает orf 94, кодирующая синтез тРНК цистеина. Следует отметить, что в составе плазмидных репликонов достаточно редко встречаются гены, кодирующие синтез тРНК (Shintani, 2015). Присутствие данной детерминанты может свидетельствовать об участии плазмиды в метаболизме бактериальной клетки, а также играть определенную роль при восстановлении дисульфидных связей в нарушенных под действием стрессовых факторов белках (Guillen, 2017).
Распространение систем репликации и конъюгационного переноса сходных с таковыми плазмиды pBS72, а также присутствие в ее составе генов, детерминирующих функционально значимые белки, дает основание предполагать, что ее присутствие может играть определенную роль в бактериальной клетке.
Для изучения влияния плазмиды на жизнеспособность бактерий в стрессовых условиях среды использовали природный штамм B. subtilis 72, содержащий плазмиду pBS72, и коллекционный штамм B. subtilis 168, а также их производные, сконструированные с использованием молекулярно-генетических методов. Это штамм B. subtilis 72, из клеток которого плазмида pBS72 была элиминирована путем введения ее минимизированного варианта pAL1 методом трансформации с последующим отбором клонов, спонтанно утративших мини-репликон в результате культивирования в неселективных условиях. Кроме того, был получен штамм B. subtilis 168, содержащий плазмиду pBS72 путем конъюгационного переноса данного внехромосомного генетического элемента, в межгенную область которого был введен ген устойчивости к эритромицину. Маркирование плазмиды геном устойчивости к эритромицину осуществляли путем гомологичной рекомбинации. Для этого с использованием полимеразной цепной реакции с праймерами F1-R1 амплифицировали фрагмент плазмидного репликона размером 1063 п.н., который очищали из геля и клонировали в состав плазмиды pMTL21E. Полученную гибридную плазмиду вводили трансформацией в плазмидсодержащие бактерии B. subtilis 72. Поскольку использованная гибридная конструкция не способна копировать в клетках B. subtilis (содержит ColE1-репликон), появление трансформантов на среде с эритромицином могло происходить за счет гомологичной рекомбинации между идентичными последовательностями, присутствующими в составе вектора pMTL21E и плазмиды pBS72. Доказательством встраивания гибридной конструкции в состав плазмиды pBS72 служили эксперименты по ее конъюгационному переносу в клетки бактерий B. subtilis 168, устойчивых к рифампицину (в качестве селектирующего маркера использовали эритромицин, а в качестве контрселектирующего – рифампицин). Присутствие и отсутствие плазмиды в клетках бактерий B. subtilis доказывали методом ПЦР с использованием праймеров M13 Forward и F1, обеспечивающих амплификацию фрагмента вектора pMTL21E и orf 52 плазмиды pBS72.
Изучение динамики роста плазмидного и бесплазмидного вариантов при температурах 37, 45 и 54°С не выявило различий. Вне зависимости от температуры культивирования фиксировали лаг-фазу, длившуюся приблизительно 1 ч, фазу логарифмического роста в интервале от 1 до 5 ч, затем происходило замедление роста и наступала стационарная фаза.
Для изучения влияния плазмиды на выживаемость бактерий под действием стрессовых факторов плазмидные и бесплазмидные варианты бактерий B. subtilis 72 и B. subtilis 168, находящиеся в логарифмической фазе роста, культивировали в присутствии 13% NaCl, в среде с низким и высоким значением рН (4.5 и 11), а также облучали ультрафиолетом. Следует отметить, что использование в данных экспериментах бактериальной культуры в активной фазе роста исключало ее устойчивость к стрессовым факторам за счет присутствия спор, обеспечивающих выживание в экстремальных условиях среды. В результате было установлено, что в среде с повышенной осмолярностью (13% NaCl) преимущество имели бактерии, содержащие плазмиду pBS72 (рис. 5а). Через 4 ч культивирования жизнеспособность плазмидсодержащих вариантов бактерий B. subtilis 168 и B. subtilis 72 снижалась до 0.16 и 1.08% соответственно, тогда как присутствие плазмиды pBS72 обеспечивало увеличение выживаемости содержащим ее бактериям (2.08 и 4.68% жизнеспособных бактерий B. subtilis 72 и B. subtilis 168 соответственно).
Рис. 5.
Выживаемость бактерий B. subtilis 72 (А) и бактерий B. subtilis 168 (Б) в среде, содержащей 13% NaCl (а); в среде c рН 4.5 (б); в среде c рН 11 (в); после облучения УФ (г). 1 – Кривые выживаемости бактерий, содержащих плазмиду pBS72; 2 – не содержащих плазмиду pBS72. Доверительный интервал средней рассчитывали с использованием t-теста для двустороннего распределения Стьюдента с вероятностью Р < 0.05 (*) и Р < 0.01 (**).
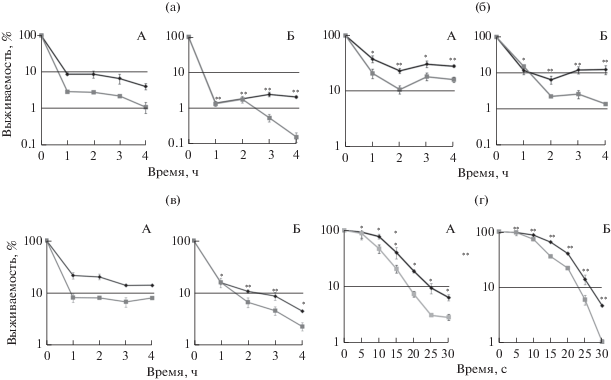
Присутствие плазмиды достоверно увеличивало выживаемость бактерий в среде со значением рН 4.5. После 4 ч культивирования выживаемость плазмидного варианта бактерий B. subtilis 72 составила 30.57%, а бесплазмидного – 18.16%. В таких же условиях фиксировали 12.32% жизнеспособных плазмидсодержащих бактерий B. subtilis 168 и 2.63% бесплазмидных вариантов (рис. 5б).
В щелочной среде (рН 11) после культивирования в течение 4 ч выживаемость плазмидсодержащего варианта бактерий B. subtilis 72 составила 14.06%, а бесплазмидного варианта – 8.04%. В присутствии плазмиды pBS72 в клетках B. subtilis 168 фиксировали 4.34% жизнеспособных клеток, тогда как в ее отсутствии только 2.24% бактерий формировали колонии на поверхности агаризованной среды (рис. 5в).
В результате ультрафиолетового облучения выживаемость плазмидсодержащего варианта бактерий B. subtilis 72 составила 6.33%, а бесплазмидного – 2.81%. При таких же дозах облучения выживаемость бактерий B. subtilis 168 с плазмидой составила 4.63%, а бесплазмидного – 1.03% (рис. 5г).
Таким образом, присутствие плазмиды pBS72 увеличивает жизнеспособность бактерий B. subtilis под действием всех исследованных факторов. При этом наименьший стресс клетки B. subtilis испытывали в среде с высокой концентрацией ионов водорода (рН 4.5). Полученные результаты позволяют предположить, что повсеместно распространенные бактерии B. subtilis в активном состоянии (в отсутствии спор) могут сохранять жизнеспособность в далеких от физиологической нормы условиях, а при наличии в их клетках плазмид, подобных плазмиде pBS72, – обеспечивать активный горизонтальный перенос генов, в том числе, антибиотикорезистентности между микроорганизмами природных популяций. Протекторный эффект плазмиды pBS72 наиболее выражен в клетках исходного штамма B. subtilis 72 по сравнению с коллекционным штаммом B. subtilis 168, что может свидетельствовать о наличии в хромосоме природных бактерий дополнительных детерминант, определяющих устойчивость к стрессу.
Список литературы
Лагодич А.В., Черва Е.А., Штанюк Я.В., Прокулевич В.А., Фомичёв Ю.К., Прозоров А.А., Титок М.А. Создание векторной системы для молекулярного клонирования в клетках Bacillus subtilis и Escherichia coli // Мол. биология. 2005. Т. 39. С. 345–348.
Lagodich A.V., Cherva E.A., Shtaniuk Y.V., Prokulevich V.A., Fomichev Y.K., Prozorov A.A., Titok M.A. Construction of a vector system for molecular cloning in Bacillus subtilis and Escherichia coli // Mol. Biol. 2005. V. 39. P. 306–309.
Anagnostopoulos C., Spizizen J. Requirements for transformation in Bacillus subtilis // J. Bacteriol. 1961. V. 81. P. 741–746.
Bullock W.O., Fernandez J.M., Short J.M. XL1-Blue: a high efficiency plasmid transforming recA Escherichia coli strain with beta-galactosidase selection // BioTechniques. 1987. V. 5. P. 376–378.
Chambers S.P., Prior S.E., Barstow D.A., Minton N.P. The pMTL nic-cloning vectors. I. Improved pUC polylinker regions to facilitate the use of sonicated DNA for nucleotide sequencing // Gene. 1988. V. 68. P. 139–149.
Dagan T., Artzy-Randrup Y., Martin W. Modular networks and cumulative impact of lateral transfer in prokaryote genome evolution // PNAS. 2008. V. 105. P. 10039–10044.
Edgar R.C. Search and clustering orders of magnitude faster than BLAST // Bioinformatics. 2010. V. 26. P. 2460–2461.
Garcillán-Barcia M.P., Francia M.V., de la Cruz F. The diversity of conjugative relaxases and its application in plasmid classification // FEMS Microbiol. Rev. 2009. V. 33. P. 657–687.
Guillen C.J, Jones G.H., Saldaña Gutiérrez C., Hernández-Flores J.L., Cruz Medina J.A., Valenzuela Soto J.H., Pacheco Hernández S., Romero Gómez S., Morales Tlalpan V. Critical minireview: The fate of tRNA Cys during oxidative stress in Bacillus subtilis // Biomolecules. 2017. V. 7. Art. 6. https://doi.org/10.3390/biom7010006
Harwood C.R., Cutting S.M. Molecular Biological Methods for Bacillus. N.Y.: Chichester, 1990. P. 75–174.
Harwood C.R., Mouillon J.M., Pohl S., Arnau J. Secondary metabolite production and the safety of industrially important members of the Bacillus subtilis group // FEMS Microbiol. Rev. 2018. V. 42. P. 721–738.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874.
Meijer W.J., de Boer A.J., van Tongeren S., Venema G., Bron S. Characterization of the replication region of the Bacillus subtilis plasmid pLS20: a novel type of replicon // Nucl. Acids Res. 1995. V. 23. P. 3214–3223.
Meijer W.J., Wisman G.B., Terpstra P., Thorsted P.B., Thomas C.M., Holsappel S., Venema G., Bron S. Rolling-circle plasmids from Bacillus subtilis: complete nucleotide sequences and analyses of genes of pTA1015, pTA1040, pTA1050 and pTA1060, and comparisons with related plasmids from gram-positive bacteria // FEMS Microbiol. Rev. 1998. V. 21. P. 337–368.
Messer W. The bacterial replication initiator DnaA. DnaA and oriC, the bacterial mode to initiate DNA replication // FEMS Microbiol. Rev. 2002. V. 26. P. 355–374.
Popa O. Dagan T. Trends and barriers to lateral gene transfer in prokaryotes // Curr. Opin. Microbiol. 2011. V. 14. P. 615–623.
Pluta R., Boer D.R., Lorenzo-Díaz F., Russi S., Gómez H., Fernández-López C., Pérez-Luque R., Orozco M., Espinosa M., Coll M. Structural basis of a histidine-DNA nicking/joining mechanism for gene transfer and promiscuous spread of antibiotic resistance // Proc. Natl. Acad. Sci. USA. 2017. V. 114. e: 6526–6535.
Poluektova E.U., Fedorina E.A., Lotareva O.V., Prozorov A.A. Plasmid transfer in bacilli by a self-transmissible plasmid p19 from a Bacillus subtilis soil strain // Plasmid. 2004. V. 52. P. 212–217.
Ramachandran G., Miguel-Arribas A., Abia D., Singh P.K., Crespo I., Gago-Córdoba C., Hao J.A., Luque-Ortega J.R., Alfonso C., Wu L.J., Boer D.R., Meijer W.J. Discovery of a new family of relaxases in Firmicutes bacteria // PLoS Genet. 2017. V. 13. e: 1006586.
Rösch T.C., Golman W., Hucklesby L., Gonzalez-Pastor J.E., Graumann P.L. The presence of conjugative plasmid pLS20 affects global transcription of its Bacillus subtilis host and confers beneficial stress resistance to cells // Appl. Environ. Microbiol. 2014. V. 80. P. 1349–1358.
Sambrook J., Fritsch E., Maniatis T. Molecular Cloning: A Laboratory Manual, 2nd ed. N.Y.: Cold Spring Harbor Laboratory, Cold Spring Harbor Publications, 1989. 468 p.
Shintani M., Sanchez Z.K., Kimbara K. Genomics of microbial plasmids: classification and identification based on replication and transfer systems and host taxonomy // Front. Microbiol. 2015. V. 6. Art. 242.
Smillie C., Garcillán-Barcia M.P., Francia M.V., Rocha E.P., de la Cruz F. Mobility of plasmids // Microbiol. Mol. Biol. Rev. 2010. V. 74. P. 434–452.
Tanaka T., Ogura M. A novel Bacillus natto plasmid pLS32 capable of replication in Bacillus subtilis // FEBS Lett. 1998. V. 422. P. 243–246.
te Riele H., Michel B., Ehrlich S.D. Single-stranded plasmid DNA in Bacillus subtilis and Staphylococcus aureus // Proc. Natl. Acad. Sci. USA. 1986. V. 83. P. 2541–2545.
Titok M.A., Chapuis J., Selezneva Y.V., Lagodich A.V., Prokulevich V.A., Ehrlich S.D., Jannière L. Bacillus subtilis soil isolates: plasmid replicon analysis and construction of a new theta-replicating vector // Plasmid. 2003. V. 49. P. 53–62.
Weigel C., Schmidt A., Seitz H., Tüngler D., Welzeck M., Messer W. The N-terminus promotes oligomerization of the Escherichia coli initiator protein DnaA // Mol. Microbiol. 1999. V. 34. P. 53–66.
Yang Y., Wu H.J., Lin L., Zhu Q.Q., Borriss R., Gao X.W. A plasmid-born Rap-Phr system regulates surfactin production, sporulation and genetic competence in the heterologous host, Bacillus subtilis OKB105 // Appl. Microbiol. Biotechnol. 2015. V. 99. P. 7241–7252.
Дополнительные материалы отсутствуют.


