Микробиология, 2021, T. 90, № 4, стр. 413-420
Азотный метаболизм аноксигенной нитчатой фототрофной бактерии Oscillocholris trichoides DG-6
Р. Н. Ивановский a, *, Н. В. Лебедева a, О. И. Кеппен a, Т. П. Турова b
a Московский государственный университет им. М.В. Ломоносова,
биологический факультет
119191 Москва, Россия
b Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
* E-mail: mguru@mail.ru
Поступила в редакцию 21.02.2021
После доработки 23.03.2021
Принята к публикации 24.03.2021
Аннотация
Источниками азота для Oscillochloris trichoides DG-6 – типового штамма семейства Oscillochloridaceae, могут служить аммоний, N2, глутамат, а также аминокислоты аспарагин, глицин, и глутамин. Ассимиляция молекулярного азота происходит при участии нитрогеназы, структурный ген которой, nifH, находится в составе генного кластера, включающего также гены субъединиц нитрогеназы nifD и nifK и вспомогательный ген nifB. Учитывая, что nifHBDK кластеры аннотированы также в геномах других представителей семейства Oscillochloridaceae, в том числе некультивируемых и таксонов-кандидатов, можно предположить, что способность к азотфиксации является свойством, имманентным для всего этого семейства. Пути ассимиляции аммония у клеток, выросших с использованием различных источников азота, могут различаться. Osc. trichoides DG-6 при росте на среде с аммонием ассимилирует его при участии глутаматдегидрогеназы, которая детерминируется единственным геном. Продукт экспрессии этого гена имеет двойную специфичность и может использовать для реализации реакции как НАД, так и НАДФ. При росте Osc. trichoides DG-6 на среде с глутаматом в качестве единственного источника азота в клетках обнаруживаются все ферменты, необходимые для реализации ГС‑ГОГАТ пути. Однако для осуществления глутаминсинтетазной реакции, участвующей в этом пути, необходим отсутствующий в среде аммоний, источником которого может служить глутаматдегидрогеназная реакция.
Все известные в настоящее времяаноксигенные нитчатые фототрофные бактерии (АНФБ) входят в филум Chloroflexi. Представители порядка Chloroflexales образуют монофилетическую линию, включающую семейства Chloroflexaceae, Roseiflex-a-ceae и Oscillochloridaceae. АНФБ, принадлежащие этим семействам, различаются по строению фотохимического аппарата, способности к автотрофии, а также по температурным режимам роста. Кроме того, между представителями этих семейств имеются различия и в осуществлении метаболизма азота (Hanada, 2014).
У представителей семейства термофильных фототрофов Roseiflexaceae способность использовать молекулярный азот в качестве источника азота не показана (Thiel et al., 2017). Однако в геномах Roseiflexus sp. RS1 иRoseiflexus castenholzii был обнаружен кластер из четырех совместно локализованных nifHBDK генов, которые, как предполагается, кодируют структурные гены Mo-нитрогеназы. При этом было высказано предположение, что неспособность к росту этих бактерий в диазотрофных условиях связана с отсутствием дополнительных генов, обычно участвующих в биосинтезе и созревании функционального апопротеина нитрогеназы (van der Meer et al., 2010; Klatt et al., 2013; Thiel et al., 2017; Nishihara et al., 2018).
Представители семейства Chloroflexaceae в качестве единственного источника азота могут использовать аммоний и некоторые аминокислоты – аланин, глутамат, глутамин, аспартат, глицин и серин, но не нитраты (Heda, Madigan, 1986; Hanada, 2014). Способность к азотфиксации и активность нитрогеназы не были обнаружены у четырех штаммов Chloroflexus aurantiacus, что коррелирует с их термофилией (Heda, Madigan, 1986; Hanada, 2014). Впоследствии эти данные были подтверждены при анализе генома Cfl. aurantiacus, в котором не были выявлены структурные гены нитрогеназы и нитратредуктазы (Tang et al., 2011).
Фотоавтотрофные бактерии семейства Oscillochloridaceae, являющиеся основным объектом данного исследования, являются мезотрофными микроорганизмами с оптимумом роста при 28–30°C (Кеппен и соавт., 1993). Основным источником азота для роста Oscillochloris trichoides DG-6 – типового штамма семейства Oscillochloridaceae, является аммоний, а также аминокислоты аспарагин, глицин, глутамат, глутамин (Кеппен и соавт., 1989). Отличительной особенностью Osc. trichoides от бактерий, принадлежащих к родам Roseiflexus и Chloroflexus, является его способность использовать для роста N2 в качестве единственного источника азота (Кеппен и соавт., 1989). Впоследствии были проведены амлификация и секвенирование гена nifH, детерминирующего редуктазу нитрогеназы, ключевой фермент нитрогеназного комплекса на матрице ДНК трех штаммов Oscillochloris, включая Osc. trichoides DG-6 (Tourova et al., 2006).
Целью данной работы являлся сравнительный анализ геномных данных, касающихся предположительных реакций азотного метаболизма у культивируемых АФНБ порядка Chloroflexales, а также экспериментальное подтверждение осуществления этих реакций бактериями штамма Osc. trichoides DG-6.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Бактерии и условия их культивирования. Объектом исследования была аноксигенная нитчатая фототрофная бактерия (АНФБ) Osc. trichoides DG-6. Культуры выращивали на модифицированной среде DGN с комплексом витаминов, 0.1% ацетата, 0.1% бикарбоната и 0.1% сульфида (Keppen et al., 1994). Культивирование бактерий проводили в анаэробных условиях на свету на магнитной мешалке (300 об./мин) при температуре 28–30°С и освещенности 2000 лк во флаконах на 500 мл с завинчивающимися пробками. Если в качестве источника азота использовали N2, флаконы содержали 300 мл среды и молекулярный азот в качестве газовой фазы. Когда в качестве источника азота использовали аммоний или глутамат, флаконы заполняли средой полностью.
Получение экстрактов клеток. Для получения бесклеточных экстрактов культуры, находящиеся в конце экспоненциальной фазы роста, центрифугировали (9000 об./мин при 6–8°С), отмывали 0.05 М трис-HCl буфером (рН 7.5) и разрушали клетки на Х-прессе (“LKB”, Швеция) при избыточном давлении 10 т/см2. Неразрушенные клетки и крупные фрагменты отделяли центрифугированием при 35 000 g в течение 1 ч. Получившейся супернатант использовали для определения ферментативной активности.
Определение активности ферментов. Реакции и катализирующие их ферменты путей N-метаболизма Osc. trichoides DG-6, активность которых измерялась в данной работе, представлена в табл. 1. Активность ферментов определяли на спектрофотометре Hitachi 200 (“Hitachi”, Япония) по окислению НАД(Ф)Н и восстановлению НАД(Ф) при 340 нм.
Таблица 1.
Реакции и катализирующие их ферменты путей N-метаболизма Osc. trichoides DG-6, активность которых измерялась в данной работе
| Фермент | Метка локуса гена в геноме (locus_tag) | Реакция |
|---|---|---|
| Нитрогеназа EC 1.18.6.1 | OSCT_0062 OSCT_0063 OSCT_0064 OSCT_0065 | N2 + 8H+ + 8e−16АТФ → 2NH3 + H2 + 16АДФ + 16Фн |
| Глутаматдегидрогеназа (ГДГ) ЕС 1.4.1.2; ЕС 1.4.1.4 |
OSCT_0145 | Глутамат + НАД(Ф)+ + Н2О → α‑кетоглутарат + НАД(Ф)Н + NH3 |
| Глютамин синтаза (ГС) ЕС 6.3.1.2 |
OSCT_1286 | Глутамат + NH4 + АТФ → глутамин + АДФ + Фн |
| Глютаматсинтаза (ГОГАТ) ЕС 1.4.1.13 |
OSCT_0551 | 2Глутамат + НАДФ+ → глутамин + α-кетоглутарат + НАДФН |
| Глютамат-оксалоацетат трансаминаза (ГОТ) ЕС 2.6.1.1 |
OSCT_0733 OSCT_1058 OSCT_1297 OSCT_3177 |
Аспартат + α-кетоглутарат → оксалоацетат + глутамат |
| Глутамат-пируват
трансаминаза (ГПТ) EC 2.6.1.2 |
OSCT_0566 | α-Кетоглутарат + аланин → глутамат + пируват |
Реакционные смеси для определения активностей ферментов содержали соответственно:
(1) глутаматдегидрогеназы (ГДГ): кетоглутарат – 5 × 10–3 М, NH4Cl – 4 × 10–2 М, НАД(Ф)Н – 2.5 × × 10–4 М; трис-НCl (рН 7.5) – 5 × 10–2 М;
(2) глутаматсинтазы (ГОГАТ): кетоглутарат – 5 × 10–3 М, глутамин – 5 × 10–3 М, НАД(Ф)Н – 2.5 × 10–4 М; трис-НCl (рН 7.5) – 5 × 10–2 М;
(3) глутамат-оксалоацетат трансаминазы (ГОТ): НАД – 2.5 × 10–4 М, кетоглутарат – 4 × 10–3 М, аспартат – 4 × 10–3 М, фосфатный буфер (рН 7.4) – 0.1 М, малатдегидрогеназу – 1.25 мг/мл;
(4) глутамат-пируват трансаминазы (ГПТ): НАД – 2 × 10–4 М, кетоглутарат – 4 × 10–3 М, фосфатный буфер (рН 7.5) – 0.1, аланин – 8 × 10–3 М, лактатдегидрогеназа – 1.25 × 10–3 мг/мл;
(5) глутаминазы (ГА): глутамин 4 × 10–3 М, трис-НCl (рН 7.5) – 5 × 10–2 М, глутаматдегидрогеназа – 0.15 ед./мл.
Активность глутаминсинтетазы (ГС) измеряли, как описано ранее (Ivanovsky, Khatipov, 1994).
Во все реакционные смеси вносили экстракт клеток, содержащий 1.0–2.0 мг/мг белка.
Биоинформатический анализ. Реконструкцию возможных путей метаболизма азота на основании сравнения геномов культивируемых штаммов АФНБ порядка Chlorofexales проводили с использованием онлайн-сервисов NCBI (https:// www.ncbi.nlm.nih.gov/genome/) PATRIC (https:// www.patricbrc.org) и KEGG (https://www.genome.jp/ kegg/pathway.html). Сравнение кластеров nif-генов проводили с использованием онлайн-сервиса Gene Graphics (https://katlabs.cc/genegraphics/app).
Белок измеряли по методу Лоури.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Потенциальные функциональные характеристики азотного метаболизма культивируемых штаммов АФНБ порядка Cloroflexales на основе сравнения их геномов. Было проведено сравнение представленности генов, детерминирующих ферменты путей метаболизма азота (“Nitrogen methabolism”) и аланина, аспартата и глютамата (“Alanine, aspartate and glutamate methabolism”) на основании базы данных KEGG в геномах культивируемых штаммов родов Oscillochloris, Chloroflexus и Roseiflexus (рис. S1a, S1b ).
На тепловой карте предполагаемых ферментов метаболизма азота, согласно геномным данным, у всех сравниваемых штаммов представлены ферменты преобразования глутамата: глутаматсинтаза (ГОГАТ) (NAD(Ф)H) (EC 1.4.1.13); глутамин синтетатаза (ГС) (EC 6.3.1.2) и глутаматдегидрогеназа (ГДГ) в двух функциональных вариантах – NADH (EC 1.4.1.2) и NAD(P)H (EC 1.4.1.4). Эти же ферменты представлены у всех сравниваемых штаммов так же и на тепловой карте ферментов метаболизма аланина, аспартата и глутамата.
Представленность остальных предполагаемых ферментов метаболизма азота заметно различается в зависимости от таксономической принадлежности сравниваемых штаммов. Нитрогеназа (EC 1.18.6.1), обеспечивающая фиксацию молекулярного азота, аннотирована только в геномах штаммов Oscillochloris и Roseiflexus, так же, как и карбоангидраза (ЕС 4.2.1.1). Гидроксиламинредуктаза (ЕС 1.7.99.1) аннотирована только для штаммов Oscillochloris, а нитратредуктаза (EC 1.7.99.4) – только для штаммов Roseiflexus. Большинство ферментов метаболизма аланина, аспартата и глюконата аннотированы в геномах всех сравниваемых штаммов, однако аланиндегидрогеназа (ЕС 1.4.1.1) аннотирована только у штаммов Roseiflexus, а аланинтрансаминаза (ЕС 2.6.1.2) – только у штаммов Oscillochloris.
Отсутствие генов, детерминирующих ферменты ассимиляционной и диссимиляционной нитратредукции в геномах культивируемых штаммов родов Chloroflexales и Oscillochloris, объясняет ранее полученные экспериментальные данные о неспособности АФНБ этих родов расти на средах, содержащих окисленные соединения азота. В то же время, подтверждаются сведения об использовании восстановленных соединений азота (аммония, мочевины, глутамата и ряда аминокислот) для роста штаммов родов Chloroflexus и Oscillochloris (Heda, Madigan, 1986; Кеппен и соавт., 1993; Keppen et al., 2000). Результатам сравнительного геномного анализа соответствуют также ранее полученные данные о неспособности АФНБ рода Chlorofelexus к фиксации молекулярного азота (Heda, Madigan, 1986) и, наоборот, способности к этому пути метаболизма азота АФНБ рода Oscillochloris (Кеппен и соавт., 1989; Ivanovsky, Khatipov, 1994; Keppen et al., 2000).
Источники азота, используемые Osc. trichoides DG-6. Источниками азота для Osc. trichoides DG-6 могут служить аммоний, N2, а также аминокислоты: аспарагин, глицин, глутамат, глутамин (табл. 2).
Ассимиляция молекулярного азота у Osc. tri-choides DG-6. Присутствие гена nifH, детерминирующего ключевой фермент фиксации молекулярного азота, было выявлено ранее в экспериментах по амплификации этих генов не только для типового штамма DG-6, но и для нескольких близких к нему штаммов, также отнесенных к виду Osc. trichoides (Tourova et al., 2006). Функциональная активность нитрогеназы была ранее подтверждена ростом культуры в присутствии N2, как единственного источника азота (Кеппен et al., 1989). В данной работе, на основании способности клеточной суспензии к восстановлению ацетилена, показана активность нитрогеназы при росте штамма DG-6 на N2, которая репрессируется при добавлении восстановленных соединений азота (табл. 3). Фермент нитрогеназа представлен на карте метаболизма азота в базе данных KEGG на основании данных анализа генома Osc. trichoides DG-6 (рис. S2a ). Согласно этим данным, ранее обнаруженный ген редуктазы нитрогеназы nifH находится в составе генного кластера (OSCT_0062, OSCT_0063, OSCT_0064, OSCT_0065), включающего также гены субъединиц нитрогеназы (nifD и nifK) и вспомогательный гена nifB (рис. 1). Таким образом, в настоящее время Osc. trichoides DG-6 является единственным представителем порядка Chloroflexales, способность к азотфиксации которого доказана как геномными данными, так и экспериментально. Однако геномные данные свидетельствуют о том, что вновь выделенные виды АНФБ “Candidatus Oscillochloris kuznetsovii” и “Candidatus Oscillochloris fontis”, “Candidatus Chloroploca asiatica” и “Candidatus Viridilinea mediisalina”, возможно, способны к диазотрофии, благодаря наличию у них кластера генов нитрогеназы (Grouzdev et al., 2018, 2019; Gaisin et al., 2019a, 2019b, 2020), гомологичного кластеру nifHBDK штамма Osc. trichoides GD-6. Учитывая, что nifHBDK кластеры аннотированы также в геномах других представителей семейства Oscillochloridaceae, в том числе, некультивируемых и таксонов-кандидатов, можно предположить, что способность к азотфиксации является свойством, имманентным для всего этого семейства.
Таблица 3.
Активность нитрогеназы у Osc. trichoides DG-6 (нмоль/мин мг белка)
| Фермент/субстрат в ростовой среде выращивания | Глутамат, 0.1% | ${\text{NH}}_{4}^{ + }$, 0.1%+ | N2 |
|---|---|---|---|
| Нитрогеназа | 0.9 | 0.0 | 3.2 |
Рис. 1.
Структура геномных кластеров, содержащих аннотированные nif-гены у представителей семейств Oscillochloridaceae и Roseiflexaceae.
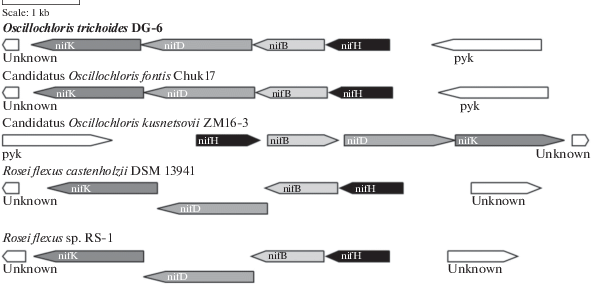
В то же время, в геномах культивируемых штаммов АФНБ рода Roseiflexus также анннотирован кластер нитрогеназных генов, сходный по структуре с nifHBDK-кластером представителей рода Oscillochloris, за исключением обнаруженного в нем перекрывания генов (рис. 1). При этом сведений о функциональной активности нитрогеназы у культивируемых штаммов Roseiflexus spp. не имеется, а типовой штамм Rof. castenholzii не способен расти на N2 в качестве единственного источника азота (Thiel et al., 2017). Однако высказанное ранее предположение о том, что это связано с отсутствием других генов, nifE и nifN, детерминирующих минимальный набор вспомогательных белков для созревания нитрогеназы (Dos Santos et al., 2012), становится сомнительным, принимая во внимание функциональность аналогичного nifHBDK-кластера у штаммов Oscillochloris. Можно предположить, что перекрывание генов в кластере свидетельствует о мутационном процессе, связанном с потерей функциональности генов нитрогеназы у штаммов рода Roseiflexus в связи приспособлением их к термофильным условиям обитания.
Можно отметить, что экспериментальные свидетельства существования экстремально упрощенного, но функционального кластера генов нитрогеназы может быть существенным для потенциальных биотехнологических исследований по имплантации этих генов в геном растений.
Ассимиляция восстановленных соединений азота у Osc. trichoides DG-6. Как и у большинства фототрофных бактерий, лучший рост Osc. trichoides DG-6 наблюдается на средах с восстановленными соединениями азота (табл. 2). Карты путей метаболизма азота и аланина, аспартата и глутамата, основанные на анализе генома Osc. trichoides DG-6 в базе данных KEGG, показаны на рис. S2a, S2b . В данной работе исследовалась активность этих ферментов в клетках Osc. trichoides DG-6, выросших на средах с разными источниками азота (табл. 2). Полученные результаты показывают, что, несмотря на присутствие в анализируемом геноме всех генов, детерминирующих ферменты путей ассимиляции аммония, реализация этих путей у клеток, выросших с использованием различных источников азота, может различаться (рис. 2). Так же, как и у других АФНБ, аммоний может включаться в метаболизм Osc. trichoides DG-6, используя ГС‑ГОГАТ путь. Согласно геномным данным, ферменты этого пути глютаминсинтетаза (ГС – ЕС 6.3.1.2) и глутаматсинтаза (ГОГАТ – ЕС 1.4.1.13), детерминированные каждая одним геном (OSCT_1286 и OSCT_0551 соответственно), присутствуют на рассматриваемых картах метаболизма (рис. S2 а, S2b). Однако активность ГС обнаруживалась только при росте на среде с глутаматом (табл. 4), но не на средах с аммонием, в отличие от Cfl. aurantiacus, у которого измеренная активность этого фермента была даже более высокой на средах с аммонием, чем на средах с глутаматом (Kaulen, Klemme, 1983).
Таблица 4.
Активность ферментов, участвующих в ассимиляции аммония у Osc. trichoides DG-6 (нмоль/мин мг белка)
| Фермент/ источник азота | Глутамат, 0.1% | ${\text{NH}}_{4}^{ + }$, 0.1%+ |
|---|---|---|
| ГДГ НАДН | 3.2 | 6.5 |
| ГДГ НАД(Ф)Н | 3.2 | 2.0 |
| ГОГАТ НАДН | 1.9 | 2.0 |
| ГОГАТ НАД(Ф)Н | 0.9 | 2.0 |
| ГС | 20.2 | 0.0 |
| ГА | 4.0 | 4.9 |
| ГОТ | 42.1 | 70.5 |
| ГПТ | 47.3 | 7.8 |
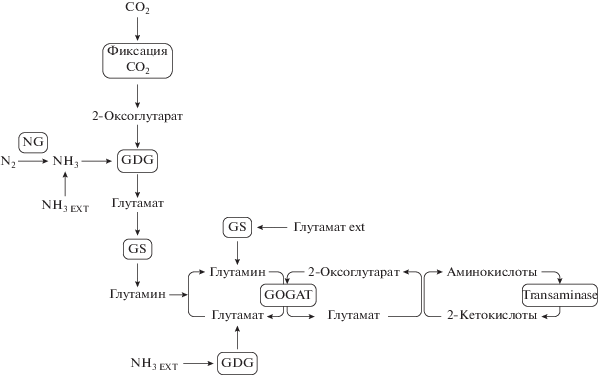
Согласно полученным в данной работе данным, штамм Osc. trichoides DG-6 при росте на среде с аммонием использует вариант ассимиляции аммония при участии глутаматдегидрогеназы (рис. 2). Глутаматдегидрогеназы (ГДГ – ЕС 1.4.1.2 и 1.4.1.4), детерминируются единственным геном (OSCT_0145), аннотированном в геноме Osc. trichoides DG-6 (рис. S2 а, S2b). Продукт экспрессии этого гена имеет двойную субстратную специфичность и может использовать для реализации реакции как НАД, так и НАДФ. Двукратное увеличение активности НАД-зависимой изоформы фермента в экстрактах клеток, выросших на среде с аммонием, может указывать на ее преимущественное участие в ассимиляции данного субстрата (табл. 4).
При росте Osc. trichoides DG-6 на среде с глутаматом в качестве единственного источника азота, в клетках обнаруживаются все ферменты, необходимые для реализации ГС‑ГОГАТ пути (табл. 4). Однако для осуществления глутаминсинтетазной реакции необходим отсутствующий в среде аммоний, источником которого может служить глутаматдегидрогеназная реакция (рис. 2), поскольку активность обоих изоферментов ГДГ обнаружена и в клетках, выросших на среде с глутаматом (табл. 4).
По-видимому, ГС‑ГОГАТ путь у Osc. trichoides реализуется также в процессе азотфиксации. Однако для доказательства этого утверждения требуются дополнительные исследования.
Ген аланиндегидрогеназы (АДГ – ЕС 1.4.1.1) у Osc. trichoides не аннотирован, что исключает участие этого фермента в процессе ассимиляции аммония у этой АФНБ, так же как и у Cfl. aurantiacus, в геноме которого этот ген также не аннотирован, а активность фермента не обнаружена (Kaulen, Klemme, 1983). Однако у Osc. trichoides аннотирован ген аланинтрансаминазы (ГПТ – ЕС 2.6.1.2) что отличает Osc. trichoides от других культивируемых штаммов представителей порядка Cloroflexales. Активность этого фермента была более высокой на среде с глутаматом, чем на средах с аммонием, аналогично другому ферменту – аспартаттрансаминазе (ГОТ – ЕС 2.6.1.1) (табл. 4). Высокие активности ферментов трансаминирования согласуются со способностью Osc. trichoides использовать для роста аминокислоты в качестве источников азота.
Полученные в данной работе данные об особенностях азотного метаболизма Osc. trichoides DG-6 при использовании различных источников азота суммированы в рис. 2.
Можно заключить, что комплексные исследования генетических и ферментативных аспектов органического N-метаболизма позволяют прояснить экологические и эволюционные особенности АФНБ семейства Oscillochloridaceae и возможность использования их метаболический потенциал в биотехнологии.
Список литературы
Кеппен О.И., Лебедева Н.В., Трошина О.Ю., Родионов Ю.В. Нитрогеназная активность нитчатой фототрофной зеленой бактерии // Микробиология. 1989. Т. 58. С. 520–521.
Keppen O.I., Lebedeva N.V., Troshina O.Y., Rodionov Y.V. The nitrogenase activity of filamentous phototrophic green bacterium // Microbiology (Moscow). 1989. V. 58. P. 520–521.
Кеппен О.И., Баулина О.И., Лысенко А.М., Кондратьева Е.Н. Новая зеленая бактерия, относящаяся к семейству Chloroflexaceae // Микробиология. 1993. V. 62. P. 267–275.
Keppen O.I., Baulina O.I., Lysenko A.M., Kondratieva E.N. A new green bacterium belonging to the Chloroflexaceae family // Microbiology (Moscow). 1993. V. 62. P. 179–185.
Dos Santos P.C., Fang Z., Mason S.W., Setubal J.C., Dixon R. Distribution of nitrogen fixation and nitrogenase-like sequences amongst microbial genomes // BMC Genomics. 2012. V. 13. Art. 162. https://doi.org/10.1186/1471-2164-13-162
Gaisin V.A., Burganskaya E.I., Grouzdev D.S., Ashikhmin A.A., Kostrikina N.A., Bryantseva I.A., Koziaeva V.V., Gorlenko V.M. “Candidatus Viridilinea mediisalina”, a novel phototrophic Chloroflexi bacterium from a Siberian soda lake // FEMS Microbiol. Lett. 2019a. V. 366. Art. fnz043. https://doi.org/10.1093/femsle/fnz043
Gaisin V.A., Burganskaya E.I., Grouzdev D.S., Osipova N.S., Ashikhmin A.A., Sinetova M.A., Krutkina M.S., Bryantseva I.A., Sukhacheva M.V., Kochetkova T.V., Koziaeva V.V., Kalashnikov A.M., Gorlenko V.M. “Candidatus Oscillochloris fontis”: a novel mesophilic phototrophic Chloroflexota bacterium belonging to the ubiquitous Oscillochloris genus // FEMS Microbiol. Lett. 2019b. V. 366. Art. fnz097. https://doi.org/10.1093/femsle/fnz097
Gaisin V.A., Grouzdev D.S., Krutkina M.S., Ashikhmin A.A., Sinetova M.A., Osipova N.S., Koziaeva V.V., Gorlenko V.M. “Candidatus Oscillochloris kuznetsovii” a novel mesophilic filamentous anoxygenic phototrophic Chloroflexales bacterium from Arctic coastal environments // FEMS Microbiol. Lett. 2020. V. 367. Art. fnaa158. https://doi.org/10.1093/femsle/fnaa158
Grouzdev D.S., Burganskaya E.I., Krutkina M.S., Sukhacheva M.V., Gorlenko V.M. Genome sequence of “Candidatus Viridilinea halotolerans” Chok-6, isolated from a saline sulfide-rich spring // Microbiol. Resour. Announc. 2019. V. 8. Art. e01614-18. https://doi.org/10.1128/mra.01614-18
Grouzdev D.S., Rysina M.S., Bryantseva I.A., Gorlenko V.M., Gaisin V.A. Draft genome sequences of “Candidatus Chloroploca asiatica” and “Candidatus Viridilinea mediisalina”, candidate representatives of the Chloroflexales order: phylogenetic and taxonomic implications // Stand. Genom. Sci. 2018. V. 13. https://doi.org/10.1186/s40793-018-0329-8
Hanada S. The phylum Chloroflexi, the family Chloroflexaceae, and the related phototrophic families Oscillochloridaceae and Roseiflexaceae // The Prokaryotes: Other Major Lineages of Bacteria and the Archaea / Eds. Rosenberg E., DeLong E.F., Lory S., Stackebrandt E., Thompson F. Berlin, Heidelberg: Springer Berlin Heidelberg, 2014. P. 515–532.
Heda S.D., Madigan M.T. Utilization of amino acids and lack of diazotrophy in the thermophilic anoxygenic phototroph Chlorolexus aurantiacus // J. Gen. Microbiol. 1986. V. 132. P. 2469–2473. https://doi.org/10.1093/femsle/fnaa158
Ivanovsky R.N., Khatipov E.-A.A. Evidence of covalent modification of glutamine synthetase in the purple sulfur bacterium Thiocapsa roseopersicina // FEMS Microbiol. Lett. 1994. V. 122. P. 115–119. https://doi.org/10.1111/j.1574-6968.1994.tb07153.x
Kaulen H., Klemme J.-H. No evidence of covalent modification of glutamine synthetase in the thermophilic phototropic bacterium Chloroflexus aurantiacus // FEMS Microbiol. Lett. 1983. V. 201. P. 75–79. https://doi.org/10.1111/j.1574-6968.1983.tb00092.x
Keppen O.I., Baulina O.I., Kondratieva E.N. Oscillochloris trichoides neotype strain DG-6 // Photosyn. Res. 1994. V. 41. P. 29–33. https://doi.org/10.1007/BF02184143
Keppen O.I., Tourova T.P., Kuznetsov B.B., Ivanovsky R.N., Gorlenko V.M. Proposal of Oscillochloridaceae fam. nov. on the basis of a phylogenetic analysis of the filamentous anoxygenic phototrophic bacteria, and emended description of Oscillochloris and Oscillochloris trichoides in comparison with further new isolates // Int. J. Syst. Evol. Microbiol. 2000. V. 50. P. 1529–1537. https://doi.org/10.1099/00207713-50-4-1529
Klatt C.G., Liu Z., Ludwig M., Kühl M., Jensen S.I., Bryant D.A., Ward D.M. Temporal metatranscriptomic patterning in phototrophic Chloroflexi inhabiting a microbial mat in a geothermal spring // ISME J. 2013. V. 7. P. 1775–1789. https://doi.org/10.1038/ismej.2013.52
Nishihara A., Thiel V., Matsuura K., McGlynn S.E., Haruta S. Phylogenetic diversity of nitrogenase reductase genes and possible nitrogen-fixing bacteria in thermophilic chemosynthetic microbial communities in Nakabusa hot springs // Microb. Environ. 2018. V. 33. P. 357–365. https://doi.org/10.1264/jsme2.ME18030
Tang K.-H., Barry K., Chertkov O., Dalin E., Han C.S., Hauser L.J., Honchak B.M., Karbach L.E., Land M.L., Lapidus A., Larimer F.W., Mikhailova N., Pitluck S., Pierson B.K., Blankenship R.E. Complete genome sequence of the filamentous anoxygenic phototrophic bacterium Chloroflexus aurantiacus // BMC Genomics. 2011. V. 12. P. 334. https://doi.org/10.1186/1471-2164-12-334
Thiel V., Hügler M., Ward D.M., Bryant D.A. The dark side of the Mushroom Spring microbial mat: Life in the shadow of chlorophototrophs. II. Metabolic functions of abundant community members predicted from metagenomic analyses // Front. Microbiol. 2017. V. 8. Art. 943. https://doi.org/10.3389/fmicb.2017.00943
Tourova T.P., Spiridonova E.M., Slobodova N.V., Boulygina E.S., Keppen O.I., Kuznetsov B.B., Ivanovskii R.N. Phylogeny of anoxygenic filamentous phototrophic bacteria of the family Oscillochloridaceae as inferred from comparative analyses of the rrs, cbbL, and nifH genes // Microbiology (Moscow). 2006. V. 752. P. 192–200. https://doi.org/10.1134/S0026261706020135
van der Meer M.T.J., Klatt C.G., Wood J., Bryant D.A., Bateson M.M., Lammerts L., Schouten S., Sinninghe Damsté J.S., Madigan M.T., Ward D.M. Cultivation and genomic, nutritional, and lipid biomarker characterization of Roseiflexus strains closely related to predominant in situ populations inhabiting Yellowstone hot spring microbial mats // J. Bacteriol. 2010. V. 192. P. 3033–3042. https://doi.org/10.1128/JB.01610-09
Дополнительные материалы
- скачать ESM_Page_2.jpg
- Рисунок S1(А, Б). Сравнение представленности ферментов на основании анализа геномов культивируемых штаммов родов Oscillochloris, Chloroflexus и Roseiflexus: А – ферменты пути «Метаболизм азота» (“Nitrogen metabolism”);
- скачать ESM_Page_3.jpg
- Рисунок S1(А, Б). Сравнение представленности ферментов на основании анализа геномов культивируемых штаммов родов Oscillochloris, Chloroflexus и Roseiflexus: В - ферменты пути «Метаболизм аланина, аспартата и глютомата» (“Alanine, aspartate and glutamate metabolism”).;
- скачать ESM_Page_4.jpg
- Рисунок S2. Ферменты исследуемых путей метаболизма, представленные в геноме Oscillochloris trichoides DG-6 (выделены зеленым цветом). А – карта пути «Метаболизм азота» (“Nitrogen metabolism”);
- скачать ESM_Page_5.jpg
- Рисунок S2. Ферменты исследуемых путей метаболизма, представленные в геноме Oscillochloris trichoides DG-6 (выделены зеленым цветом). В - карта пути «Метаболизм аланина, аспартата и глютомата» (“Alanine, aspartate and glutamate metabolism”).