Микробиология, 2021, T. 90, № 4, стр. 400-412
Сравнение таксономического состава бактериальных обрастаний, развивающихся на поверхности различных материалов, экспонированных в водных средах
Т. П. Турова a, *, Д. Ш. Соколова a, Т. Н. Назина a, А. Б. Лаптев b
a Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b Федеральное государственное унитарное предприятие “Всероссийский научно-исследовательский институт авиационных материалов”
105005 Москва, Россия
* E-mail: tptour@rambler.ru
Поступила в редакцию 29.12.2020
После доработки 12.02.2021
Принята к публикации 05.03.2021
Аннотация
Целью работы являлось выявление потенциальных деструкторов полиэтилентерефталата (ПЭТ), полистирола (ПС) и стали 20 на основании сравнения такcономического состава сообществ бактериальных обрастаний на образцах этих материалов, помещенных в водные среды различного происхождения (морские, природные пресные и промышленные) в разных климатических регионах. Полученные методом высокопроизводительного секвенирования V3–V4 региона гена 16S рРНК библиотеки группировались, преимущественно, согласно типам водных сред. При этом состав и количественное соотношение таксонов, как на уровне филумов/классов, так и на родовом уровне заметно различались для каждой из групп полученных библиотек, а внутри групп менялись в основном в зависимости от времени экспозиции, в промышленной воде – от проведения антимикробной обработки и, в меньшей степени, от типа материала образцов. Полученные результаты позволили предположить, что промышленная оборотная вода является более агрессивной средой по отношению к полимерным материалам, а потенциальные штаммы-деструкторы полимеров (ПЭТ и ПС) могут быть обнаружены среди представителей родов Pseudomonas, Acidovorax и Arenimonas. В морской воде в биокоррозии стали наиболее вероятно участвуют представители серо- и железоокисляющих бактерий родов Sulfurimonas и Mariprofundus.
В настоящее время созданы полимерные материалы, максимально стойкие в различных агрессивных средах, что обусловливает необходимость разработки методов их утилизации с наименьшим ущербом для окружающей среды. Полиэтилентерефталат (ПЭТ) и полистирол (ПС) наиболее широко производятся и используются в Европе и мире и составляют значительную часть твердых бытовых отходов на суше и основную массу загрязнителей в морских экосистемах. Отмечена тенденция перехода большинства предприятий на “экологически чистую” биологическую утилизацию отходов. Основой таких очистных сооружений являются бактериальные сообщества, которые с течением времени эволюционируют и приспосабливаются к использованию различных химических соединенийиз отходов предприятий, в том числе, и синтетических полимеров. При этом микроорганизмы-биодеструкторы в сточных водах не учитываются и сливаются в реки в неограниченном количестве. В частности, в результате исследования состава микробных сообществ в 250 образцах природных и антропогенных сред обитания, включая почву, осадки водоемов и промышленные стоки, первая и пока единственная бактерия-деструктор ПЭТ Ideonella sakaiensis была выделена японскими учеными именно из активного ила предприятия по переработке пластиковых бутылок (Yoshida et al., 2016). С экологической точки зрения, такие штаммы микроорганизмов-биодеструкторов можно эффективно использовать для утилизации бытовых отходов и промышленных изделий из пластических материалов, загрязняющих окружающую среду. Но с технологической точки зрения, для обеспечения длительных сроков эксплуатации конструкций и изделий из полимерных материалов необходимо предусмотреть меры по контролю за их использованием в потенциально агрессивных средах.
Устойчивость полимеров к биодеградации зависит от различных факторов: типа, химической структуры и физического состояния полимеров, а также физико-химических условий среды, в которой они эксплуатируются. Полимеры с гидролизуемыми химическими связями в основной цепи, такие как ПЭТ, более подвержены биодеградации (Webb et al., 2013), чем такие как ПС, в структуре которого отсутствуют гидролизуемые функциональные группы (Krueger et al., 2017). В частности, не было обнаружено никаких признаков разрушения ПС-листа, закопанного в почве в течение 32 лет (Otake et al., 2020). Однако вспененный ПС может подвергаться интенсивной биодеградации с участием симбиоза личинок насекомого Tenebrio molitor Linnaeus и выделенного из их желудка штамма-деструктора ПС Exiguobacterium sp. YT2 (Yang et al., 2015).
В водных системах на поверхности погруженных материалов и конструкций неизбежно образуются микробные биопленки. Биопленки усиливают микробную активность, защищают микроорганизмы от ультрафиолета, радиации, высокой солености, тяжелых металлов и антибиотиков (de Carvalho et al., 2018). Однако пока отсутствует понимание, чем в большей степени определяется состав формирующихся биопленок – физико-химическими факторами и микрофлорой окружающей водной среды или свойствами экспонируемых материалов. Имеющиеся в научной литературе данные, полученные в результате изучения био-пленок, развивающихся на различных материалах в водных средах, пока не позволяют однозначно ответить на этот вопрос. В частности, было установлено, что микробные скопления на пластике и стекле при экспозиции в Северном море в течение 6 недель формировались в основном под влиянием сезонных и географических факторов, а не в зависимости от поверхностных свойств материала (Oberbeckmann et al., 2018). Обнаружено, что основным фактором, влияющим на микробные сообщества, развивающиеся на различных природных и искусственных материалах в пресных водах, загрязненных антропогенным мусором, является место отбора пробы; при этом, однако, существуют некоторые различия между составом биопленок в зависимости от типа субстрата (Hoellein et al., 2014). Недавно было показано, что состав бактериальных сообществ на поверхности двух типов микропластика и древесных частиц больше зависит от места экспозиции в Северном море, чем от типа материала, но в то же время значительно отличается от состава сообществ сестона (естественной взвеси микрочастиц) и свободноживущих сообществ (Kesy et al., 2019). Аналогично обнаружены различия в таксономическом составе бактериальных сообществ с образцов взвеси микропластика и сообществ сестона, а также свободноживущих сообществ в западной части Средиземноморского бассейна, при этом были выявлены специфичные для микропластика филотипы бактерий (Dussud et al., 2018).
В наших предыдущих работах было показано заметное влияние типа водной среды обитания на состав сообществ микробных обрастаний, развивающихся на образцах ПЭТ (Турова и соавт., 2020) и ПС (Tourova et al., 2020), помещенных в водные среды различного происхождения (морские, природные пресные и промышленные) в разных климатических регионах.
Для обнаружения возможных микроорганизмов-биодеструкторов необходимы сравнительные комплексные исследования распространенности тех или иных микроорганизмов в разных средах, их способности к деструкции различных материалов, включая полимерные, а также путей распространения в окружающей среде микроорганизмов, наиболее агрессивных к материалам технологических конструкций.
Целью работы являлось выявление потенциальных деструкторов полиэтилентерефталата (ПЭТ), полистирола (ПС) и стали 20 (CT) на основании сравнения такcономического состава сообществ микробных обрастаний на образцах этих материалов, помещенных в водные среды различного происхождения (морские, природные пресные и промышленные) в разных климатических регионах.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Подготовка образцов. Для сравнительного анализа использовали следующие материалы: полиэтилентерефталат (ПЭТ) (ГОСТ Р 51695-2000) – распространенный полимерный материал, как широко используемый для изготовления некоторых промышленных конструкций, так и наиболее интенсивно загрязняющий окружающую среду; полистирол (ПС) (ГОСТ 20282-86) – стойкий к химическим воздействиям и биоповреждениям полимер, широко применяемый как для изготовления бытовых изделий, так и в промышленных конструкциях; сталь 20 (СТ) (ГОСТ 1050-2013) – наиболее распространенный материал для изготовления несущих конструкций, трубопроводов и метизов.
Специально подготовленные образцы материалов размером 100 × 10 × 2 ммпомещали в водные среды на глубину 1 м в разных климатических регионах России. В г. Геленджик образцы экспонировали в соленой морской воде в прибрежной зоне Черного моря в естественных природных условиях (группа “Marine”); в Подмосковье (г. Можайск) образцы помещали в пресноводное Можайское водохранилище в районе д. Горетово (группа “Fresh”). Все образцы группы “Fresh” экспонировали 10 сут, а образцы группы “Marine” экспонировали 10, 20, 40 и 60 сут (ПЭТ и СТ) и 10, 40, 60 сут (ПС).
В г. Уфа образцы помещали в водоблоки с пресной оборотной охлаждающей водой промышленного назначения завода “Уфаоргсинтез” (группа “Industrial”). Образцы этой группы экспонировали 10, 20, 40 и 60 сут. Особенностями охлаждающей воды являются незначительно изменяющаяся температура, недостаток освещенности и периодическое насыщение солями (в основном, хлоридами и сульфатами Na+, Ca2+ и Mg2+), продуктами нефтепереработки, а также взвешенными неорганическими и органическими частицами, которые являются благоприятной средой для развития микроорганизмов. Кроме того, на заводском водоблоке для корректировки роста микроорганизмов используется обработка химическими антимикробными реагентами. При превышении численности микроорганизмов в воде свыше 105 кл./мл подается 10%-ный щелочной раствор гипохлорита натрия, который обладает антисептическим и дезинфицирующим действием и широко используется в качестве бытового и промышленного дезинфектанта, средства очистки и обеззараживания воды. Такая обработка производилась примерно на 17–19 сут экспозиции образцов.
Химический состав водных сред определяли с помощью Biotronic ion хроматографа. Соленость, общую жесткость и общее содержание Fe2+/Fe3+ определяли с использованием, соответственно, Aquamerk alkalinity analytic kit, Aquamerck Total Hardness kit и Iron Test kit (“Merck”, Darmstadt, Германия). Значение pH определяли с использованием Аnion ionometer (Новосибирск, Россия) с соответствующими электродами. Определение индекса химического потребления кислорода (ХПК) производили согласно ГОСТ 31859-2012 (Российский Госстандарт) с использованием стандартных процедур. Измерения проводились в 3-х повторностях.
Выделение ДНК, высокопроизводительное секвенирование фрагментов гена 16S рРНК и подготовка их к анализу были проведены, как описано ранее (Турова и соавт., 2020).
Анализ библиотек фрагментов генa 16S рРНК. Библиотеки фрагментов генa 16S рРНК бактериальных сообществ анализировали с использованием онлайн-ресурсов SILVA (https://www.arb-silva.de/ngs/) и RDP (http://pyro.cme.msu.edu). Для анализа библиотек последовательности предварительно объединяли в операционные таксономические единицы (OTE) c уровнем сходства 97%. Неидентифицированные последовательности (риды) и единичные риды (синглтоны), представленные только в одной из анализируемых библиотек, были исключены из дальнейшего анализа. Индексы разнообразия были рассчитаны с использованием программы EstimateS (http://purl.oclc.org/ estimates). Для создания диаграмм Венна использовали онлайн-ресурс InteractiVenn (http://www.interactivenn.net). Онлайн-ресурс ClustVis (https:// biit.cs.ut.ee/clustvis/) был использован для создания графика Principal Component Analysis (PCA) и тепловых карт (Heatmaps) членов сообщества на родовом уровне. Для определения таксонов-маркеров, различающих библиотеки согласно применяемым параметрам сравнения (тип материала образцов и характер среды экспозиции), был использован метод эффекта размера в линейном дискриминантном анализе (Linear discriminant analysis effect size method, LEfSe) (Segata et al., 2011) на основании таблицы относительной представленности ОТЕ в сравниваемых библиотеках. Использовали настройки параметров по умолчанию с минимальным значением размера 4.0 и применением стратегии сравнения “всех-против-всех” (all-against-all).
Микроскопические методы. При исследовании бактериальных обрастаний и поверхности образцов ПЭТ применяли сканирующий микроскоп TESCAN VEGA 3 XMU (Чехия) и систему энергодисперсионного микроанализа (ADVANCED AZTEC Energy, включая Inca Energy 350) на базе безазотного энергодисперсионного детектора X-MAX 50 STANDARD (“Oxford Instruments NanoAnalysis”, Великобритания).
Библиотеки фрагментов генa 16S рРНК бактерий из микробных обрастаний образцов, полученные в результате секвенирования, депонированы в NCBI SRA под номерами PRJNA545821 (ПЭТ), PRJNA613231 (ПС) и PRJNA660935 (СТ).
РЕЗУЛЬТАТЫ
Физико-химические условия водных сред. Физико-химические условия в местах экспозиции образцов материалов приведены в табл. 1. Общая соленость и жесткость оборотной воды нефтехимического предприятия превышали аналогичные показатели пресной воды Можайского водохранилища, но были значительно ниже соответствующих значений морской воды. В местах экспозиции образцов в море и в пресноводном водоеме изменения температуры определялись суточными колебаниями и были в интервале значений температуры промышленной оборотной воды. Величины солености, жесткости и химического потребления кислорода (ХПК) промышленной воды в ходе эксперимента снижались, вероятно, в результате циклической очистки оборотных вод от вредных примесей в этот период, связанной с разбавлением их чистой водой согласно технологическому протоколу. Величина рН пресной воды составляла 7.4, а пробы промышленной и морской воды были более щелочными (рН 7.8–8.5).
Таблица 1.
Химический состав промышленной оборотной, морской и природной пресной воды, в которой экспонировали образцы
| Местообитание/время экспозиции | Общая соленость, мг/л | pH | Температура воды, °С | Жесткость общая, мг-экв/л | Железо общее, мг/л | Щелочность общая, мг-экв/кг | ХПК*, мг О2/дм3 |
|---|---|---|---|---|---|---|---|
| Промышленная вода | |||||||
| 10 сут | 1098 | 8.5 ± 0.1 | 24.0–27.0 | 13.8 | 0.45 | 6.4 | 101 |
| 20 сут | 1100 | 8.5 ± 0.1 | 21.5–25.5 | 12.6 | 0.37 | 6.5 | 103 |
| 40 сут | 918 | 8.4 ± 0.1 | 19.0–23.0 | 9.8 | 0.21 | 4.3 | 72 |
| 60 сут | 676 | 8.1 ± 0.1 | 14.0–18.0 | 6.7 | 0.61 | 2.4 | 90 |
| Чeрное море**, образцы 10–60 сут | 18 600 | 7.8 ± 0.1 | 26.8–24.1 | 66.9 | Нд*** | Нд | Нд |
| Пресная вода, 10 сут | 90–500 | 7.4 ± 0.1 | 17–23 | 4.3 | Нд | Нд | Нд |
Статистический анализ. В результате применения высокопроизводительного секвенирования V3–V4 региона гена 16S рРНК были получены библиотеки фрагментов гена 16S рРНК для каждой из 26 проб бактериальных обрастаний на подложке исследуемых материалов. Общее количество последовательностей (ридов) для 26 библиотек после исключения неидентифицированных ридов и синглтонов составило 828 925, из которых было сформировано 10 214 ОТЕ (филотипов) с уровнем сходства последовательностей ≥97%.
Уровень покрытия для всех библиотек составлял >97%, что подтверждало их репрезентативность. Об этом же свидетельствовали низкие значения индексов Chao1 (табл. 2). Индексы разнообразия различались для библиотек из разных мест экспонирования образцов. Для библиотек, полученных из образцов, экспонировавшихся в воде Черного моря и в природной пресной воде Можайского водохранилища, индексы разнообразия Шеннона–Уивера и Симпсона оказались высокими, а индексы доминирования Бергера–Паркера, наоборот, низкими. При этом в морской воде наибольшее разнообразие на образцах пластика наблюдалось на начальной стадии экспозиции (10 сут), а на образцах стали на следующей стадии (20 сут) с постепенным снижением к окончанию экспозиции. Для образцов, экспонировавшихся в пресной промышленной оборотной воде, варьирование индексов разнообразия и доминирования было выражено значительно сильнее, причем в некоторых случаях разнонаправленно для различных материалов. Так, на стадии 20 сут, т.е. после проведения антимикробной обработки, для образцов ПЭТ индексы разнообразия существенно падали, а индекс доминирования, соответственно, возрастал. И наоборот, для образцов ПС на этом же этапе индексы разнообразия резко вырастали, а индекс доминирования падал. Для образцов СТ прослеживалась аналогичная тенденция, но менее резко выраженная. При этом в группах “Industrial” и “Marine” к окончанию экспозиции индексы разнообразия становились ниже, а индекс доминирования выше их максимального и минимального значений соответственно, что свидетельствовало о постепенном действии отбора специфической микрофлоры в обрастаниях.
Таблица 2.
Индексы разнообразия* библиотек фрагментов гена 16S рРНК из обрастаний на поверхности подложек из различных материалов, экспонированных в водных средах
| Среда | Образец | Библиотека | Риды | ОТЕ | Chao1 | H | 1/S | D |
|---|---|---|---|---|---|---|---|---|
| Группа “Industrial” (промышленная оборотная вода) |
PET | PET10I | 12 421 | 365 | 377 ± 6 | 4.7 | 53.4 | 0.08 |
| PET20I | 16 647 | 256 | 292 ± 13 | 1.8 | 2.0 | 0.70 | ||
| PET40I | 13 690 | 226 | 249 ± 10 | 3.3 | 8.6 | 0.30 | ||
| PET60I | 10 001 | 281 | 299 ± 8 | 4.1 | 20.9 | 0.15 | ||
| PS | PS10I | 24 282 | 575 | 618 ± 12 | 3.4 | 6.1 | 0.39 | |
| PS20I | 59 621 | 1446 | 1457 ± 5 | 5.8 | 131.4 | 0.04 | ||
| PS40I | 37 938 | 779 | 803 ± 8 | 4.4 | 26.4 | 0.11 | ||
| PS60I | 24 789 | 596 | 633 ± 12 | 4.0 | 11.9 | 0.26 | ||
| ST | ST10I | 35 475 | 1219 | 1278 ± 13 | 5.4 | 85.4 | 0.05 | |
| ST20I | 62 539 | 1571 | 1601 ± 8 | 5.7 | 105.5 | 0.04 | ||
| ST40I | 31 814 | 554 | 569 ± 7 | 4.7 | 45.6 | 0.08 | ||
| ST60I | 41 464 | 699 | 716 ± 15 | 4.5 | 17.8 | 0.20 | ||
| Группа “Marine” (морская вода) |
PET | PET10M | 5688 | 266 | 281 ± 8 | 4.8 | 72.9 | 0.04 |
| PET20M | 4338 | 284 | 296 ± 7 | 4.8 | 59.6 | 0.06 | ||
| PET40M | 5351 | 192 | 191 ± 1 | 4.4 | 45.2 | 0.07 | ||
| PET60M | 10 881 | 256 | 271 ± 9 | 4.3 | 30.3 | 0.13 | ||
| PS | PS10M | 20 996 | 930 | 950 ± 7 | 5.8 | 162.7 | 0.03 | |
| PS40M | 15 852 | 574 | 581 ± 4 | 5.4 | 99.6 | 0.05 | ||
| PS60M | 46 101 | 1061 | 1069 ± 4 | 5.2 | 51.0 | 0.10 | ||
| ST | ST10M | 18 501 | 192 | 197 ± 4 | 2.7 | 8.7 | 0.20 | |
| ST20M | 23 973 | 980 | 1007 ± 8 | 5.2 | 43.6 | 0.10 | ||
| ST40M | 61 537 | 1186 | 1208 ± 8 | 5.5 | 88.7 | 0.05 | ||
| ST60M | 61 246 | 1066 | 1082 ± 7 | 4.8 | 32.7 | 0.13 | ||
| Группа “Fresh” (природная пресная вода) | PET | PET10F | 9568 | 469 | 481 ± 5 | 5.0 | 57.3 | 0.03 |
| PS | PS10F | 27 423 | 1721 | 1724 ± 4 | 6.2 | 85.3 | 0.09 | |
| ST | ST10F | 44 734 | 2000 | 2021 ± 6 | 6.1 | 124.9 | 0.05 |
Таксономический состав сообществ бактерий на исследуемых образцах на уровне филумов/классов. Во всех анализируемых библиотеках были выявлены только бактерии, а архейный компонент не был обнаружен. Таксономическое распределение филотипов бактерий на уровне филумов, а для Proteobacteria – на уровне классов, показано на рис. S1 .
Большинство последовательностей в библиотеках генов 16S рРНК, представленных для всех исследуемых типов образцов материалов (ПЭТ, ПС и СТ), относилось к филуму Proteobacteria, однако состав классов в этом филуме различался как между библиотеками с разных типов образцов, так и из разных сред. Значительную часть разнообразия бактерий во всех библиотеках составляли Gammaproteobacteria, при этом их количественное соотношение и изменения в процессе экспонирования были специфичными не для сравниваемых материалов, а для водных сред, в которых эти материалы экспонировались. Для всех сравниваемых образцов материалов в промышленной оборотной воде наблюдалось значительное увеличение доли гаммапротеобактерий на последних стадиях экспозиции (c 13–19% для 1–2 этапов до 37–53% на 3–4 этапах), в то время как в морской воде их доля на различных стадиях либо оставалась примерно одинаковой (23–30% для СТ), либо немного уменьшалась на последних стадиях (20–10% для ПЭТ и 24–13% для ПС). Представители Betaproteobacteria в заметных количествах были преимущественно выявлены только в пресных водах, т.е. в промышленной оборотной (5–27%) и в природной пресной воде Можайского водохранилища (l6–28%). Присутствие представителей филума Cyanobacteria было выявлено только в морской и природной пресной воде, а в промышленной оборотной воде цианобактерии отсутствовали, по-видимому, из-за недостаточной освещенности. Наиболее значительным различием между образцами из пластика и стали являлось присутствие в бактериальных сообществах последних, при этом исключительно в морской воде, значительного количества представителей филумов Epsilon- (16–18%) и Zetaproteobacteria (6–8%).
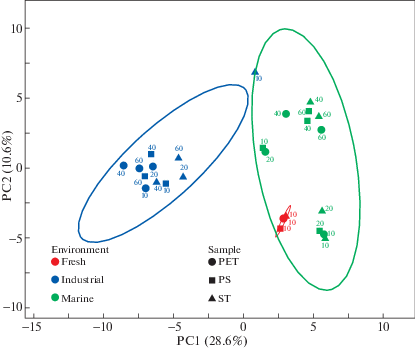
Сравнение состава сообществ обрастаний на уровне родов. В результате классификации 10 214 ОТЕ в общем составе 26 библиотек фрагментов генов 16S рРНК были выявлены представители 778 родов бактерий. Группирование библиотек на родовом уровне приведено на графике PCA, построенном с помощью программы ClustVis (рис. 1). Результаты этого анализа свидетельствуют о разделении библиотек на 2 области, соответствующие экспозиции образцов в морской воде и в обоих типах пресных вод. При этом наблюдалось внутреннее группирование библиотек из пресных вод, отделяющее библиотеки группы “Fresh”, а также отдельные библиотеки группы “Industriаl” друг от друга. Группирование последних коррелировало со временем экспозиции, т.е. библиотеки, полученные на поздних стадиях экспозиции (40–60 сут) (после антимикробной обработки), преимущественно группировались отдельно от библиотек, полученных на ранних стадиях экспозиции (10–20 сут).
Рис. 1.
График, показывающий группирование образцов ПЭТ, ПС и СТ в различных средах в соответствии с составом бактериальных сообществ их обрастаний, полученный методом главных компонент (Principal Components Analysis, PCA) с использованием параметра SVD в программе ClustVis. Эллипсы обозначают области, внутрь которых с вероятностью 0.95 попадет новый объект из той же группы.
Тепловая карта распределения всех 778 родов бактерий, созданная с использованием программы ClustVis, позволяет проследить распределение библиотек по двум параметрам, а именно, типу материала образца и характеру среды экспозиции. На тепловой карте (рис. S2 ) видны 3 четко различающиеся области, коррелирующие с типами сред обитания. Такое распределение библиотек свидетельствует о кардинальных различиях в родовом составе бактериальных сообществ при экспонировании образцов в соответствующих водных средах, хотя пресноводные сообщества были более близки по составу между собой, чем с морскими сообществами. При этом внутри областей в группе “Marine” наблюдалась тенденция к группированию библиотек по типу материала образцов, а для группы “Industrial” такой тенденции не прослеживалось.
На тепловой карте 25 родов, преобладающих в общем составе библиотек, можно отметить формирование только двух областей, коррелирующих с морской и пресноводными типами сред, что подтверждает относительное сходство библиотек групп “Industrial” и “Fresh” (рис. 2). В результате этого анализа были выявлены различия в родовой структуре преобладающих компонентов сообществ бактерий в морской и пресноводных средах.
Рис. 2.
Тепловая карта (Heatmap) распределения 25 доминантных родов бактерий по общей сумме ридов во всех 26 библиотеках, представляющих бактериальные сообщества с образцов ПЭТ, ПС и СТ. Представленность родов рассчитана как отношение количества ридов, принадлежащих представителям данного рода, к общему количеству ридов в каждой библиотеке. Как строки, так и столбцы группировались с использованием программы ClustVis. Сравнительное количество фрагментов последовательностей гена 16S рРНК бактерий определенных родов в библиотеках показано цветом.
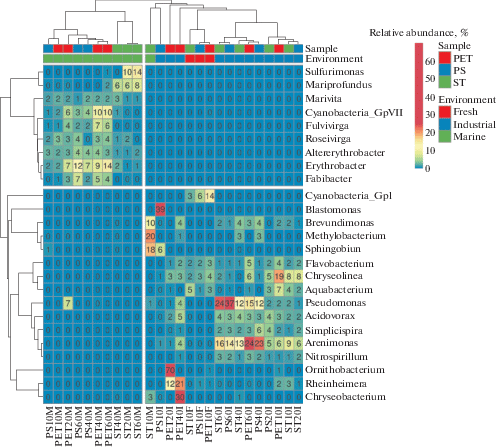
В промышленной оборотной воде на образцах всех типов материалов наиболее представленными были бактерии родов Arenimonas и Pseudomonas (Gammaproteobacteria). Там же присутствовали бактерии родов Acidovorax и Simplispira (Betaproteobacteria); на некоторых стадиях экспозиции выявлялись также Brevundimonas и Nitrospirillum (Alp-haproteobacteria). Специфичными для библиотек ПЭТ на средних стадиях экспонирования (20–40 сут) в промышленной воде были бактерии рода Reinheimera, а для библиотеки с образца ПС на начальной стадии экспонирования (10 сут) – бактерий родов Blastomonas и Sphingobium (Alphaproteobacteria).
В морской воде универсально представленными на образцах всех типов материалов были бактерии родов Мarivita, Erythrobacter и Altererythrobacter (Alphaproteobacteria) и Roseivirga (Bacteroidetes). Представители доминантных родов Fulvivirga и Fabibacter филума Bacteroidetes были выявлены только в библиотеках с образцов пластика. В то же время специфичными для библиотек с образцов стали были бактерии рода Sphingobium (Alphaproteobacteria) на начальной стадии экспонирования (10 сут), а родов Sulfurimonas (Epsilonproteobacteria) и Mariprofundus (Zetaproteobacteria) – на последующих стадиях (20–60 сут).
Поскольку было выявлено заметное изменение состава исследуемых библиотек на родовом уровне в соответствии с типом среды экспонирования, было дополнительно проведено 2 варианта анализа, в которых сравнивали состав доминирующих родов (топ-25) индивидуально по каждой среде экспозиции для каждого из типов материалов (табл. S1 ).
В первом варианте этого анализа выявленные доминирующие роды сравнивались по группированию согласно типу материала образцов (рис. 3_Sample). При сравнении диаграмм Венна, построенных для каждого материала, прослеживались тенденции, одинаковые для всех трех материалов. Так, ни на одной диаграмме не было обнаружено общих родов для каждой из трех групп сред, в которой экспонировались образцы данного типа материалов. Кроме того, при попарном сравнении между пресноводными средами и морской средой для каждого типа материалов количество общих родов было минимальным (0–3). И только при сравнении обеих пресноводных сред для каждого типа материалов было обнаружено заметное количество общих родов (5–7). Соответственно, количество родов, индивидуальных для каждого типа материалов, было значительным (16–24).
Рис. 3.
Диаграммы Венна, показывающие количество общих и уникальных родов из доминантных (топ-25) родов по количеству ридов представителей каждого рода в сумме ридов по каждому типу материала (ПЭТ, ПС и СТ) в каждой среде экспозиции (группы “Industrial”, “Marine” и “Fresh”). Sample – соотношения родов при сравнении по типу материала образцов; Environment – соотношение родов при сравнении по типу среды экспозиции.
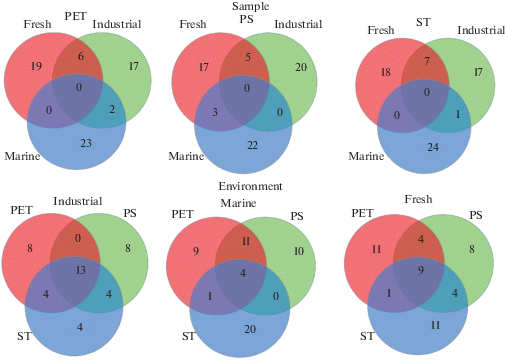
Во втором варианте анализа наборы топ-25 родов сравнивались по группированию согласно среде экспозиции (рис. 3_Environment). При сравнении диаграмм Венна, построенных для каждой среды, количество общих родов для каждого типа материала при группировании по типам сред было значительно выше, чем в первом варианте анализа, но при этом варьировало для каждой среды. Оно было наименьшим для морской воды (4), средним для природной пресной воды (9) и наибольшим для промышленной оборотной воды (13). Соответственно варьировало и количество индивидуальных для каждой среды родов, которое было минимальным для группы “Industrial” (4–8), средним для группы “Fresh” (8–11) и максимальным для группы “Marine” (10–20). Попарное сравнение образцов материалов также варьировало для каждой из сред. Для обоих типов пресной воды количество общих родов было незначительно как для обоих образцов пластика (0–4), так и образцов пластика со сталью (1–4). В то же время в случае инкубации в морской воде было выявлено значительное количество общих родов для образцов пластика (10) и низкое при сравнении образцов пластика и стали (0–1).
Выявление таксонов-маркеров в бактериальных сообществах. Чтобы выявить таксоны (маркеры), которые достоверно различают сравниваемые сообщества по сравнительной обильности, был применен метод LefSe. Из общего количества для всех библиотек были отобраны 2777 ОТЕ (каждая из ОТЕ объединяла в своем составе >0.1% от общего состава 26 библиотек), классифицированные от филума до рода.
При сравнении библиотек согласно различиям по средам экспозиции (рис. S3 _Environment) было выявлено всего 40 таксонов уровня от филума до рода, представленность которых была специфична для каждой из сред экспозиции, в том числе, 23 таксона – для морской воды, 13 – для промышленной оборотной воды и 14 – для природной пресной воды. При этом в группe “Marine” были наиболее представлены бактерии филума Planсtomycetes и классов Alphaproteobacteria, Epsilonproteobacteria и Zetaproteobacteria, а также порядка Alteromonadales; для группы “Industrial” – бактерии порядков Pseudomonadales и Xanthomonadales. Бактерии филума Bacteroidetes и класса Betaproteobacteria были относительно обильны в группах “Industrial” и “Fresh”, а филума Cyanobacteria/Chloroplast – в группах “Marine” и “Fresh”.
Выявленные с помощью программы LefSe роды-маркеры, для группы “Industrial” – Pseudomonas, Arenimonas и Acidovorax, а для группы “Marine” – Erythrobacter, Altererythrobacter и Mariprofundus, входили в топ-25 родов по сумме представленности во всех библиотеках (рис. 2), что свидетельствовало о специфичности этих доминирующих в сообществах родов бактерий для сравниваемых сред экспозиции. Распределение родов-маркеров по средам экспозиции и по типу материала образцов представлены на рис. S4 .
При сравнении библиотек согласно типам материала образцов (рис. S3 _Sample) результаты LefSe анализа существенно отличались от рассмотренных выше. Было выявлено всего 8 таксонов уровня от филума до рода, представленность которых была специфична для каждого из типов материала: 4 таксона для образцов ПЭТ, 3 – для образцов ПС и всего 1 – для образцов СТ. Дифференцирующая относительная обильность таксонов-маркеров для образцов ПЭТ была выявлена на уровне семейства Flavobacteraceae, для образцов ПC – на уровне рода Blastomonas и филума Actinobacteria, а для образцов СТ – только на уровне филума Proteobacteria.
Микроскопия микробных обрастаний на образцах пластика. Методом сканирующей электронной микроскопии были исследованы микробные обрастания на поверхности полимерных образцов (ПЭТ и ПС), экспонировавшихся в водных средах, а также поверхность образцов, отмытых от обрастаний. Обрастания на образцах полимеров в промышленной оборотной воде были в основном представлены рыхлыми конгломератами бактериальных колоний, а структурированные микроэукариоты в них были единичны (рис. 4а, 4д). При этом на микрофотографиях отмытых от обрастаний образцов обоих типов полимеров в этой среде видны хотя и локальные, но заметные разрушения подложки (рис. 4б, 4е).
Рис. 4.
Микрофотографии обрастаний на образцах полимеров после 60 сут экспозиции: ПЭТ (а–г) и ПС (д–з), экспонированных в промышленной (а, д) и в морской воде (в, ж); а также отмытых от обрастаний образцов ПЭТ и ПС, экспонированных в промышленной (б, е) и в морской воде (г, з). Сканирующий электронный микроскоп TESCAN VEGA 3 XMU.

На образцах обоих типов полимеров в морской воде были обнаружены более разнообразные обрастания, значительную часть которых составляли различные структурированные микроэукариоты (рис. 4в, 4ж). Возможные конгломераты бактериальных колоний были представлены либо в виде нижнего слоя на поверхности подложек, либо на элементах микроэукариотных обрастаний. Однако микрофотографии отмытых от обрастаний образцов ПЭТ и ПС продемонстрировали различное влияние обрастаний на эти типы материалов. Образец ПЭТ после экспонирования в морской воде остался интактным, в то время как на поверхности образца ПС были обнаружены незначительные точечные повреждения (рис. 4г, 4з).
ОБСУЖДЕНИЕ
Различные способы анализа таксономического состава обрастаний на поверхности двух полимерных материалов (ПЭТ и ПС), а также стали показали, что влияние среды экспонирования на состав микробных сообществ с поверхности подложек из различных материалов было значительнее, чем влияние природы материала. Наиболее заметное влияние на состав сообществ обнаружено для промышленной оборотной воды. Среди общих для этой среды таксонов особое место занимали бактерии рода Pseudomonas, представленность которых заметно возрастала к концу экспозиции всех образцов материалов в данной среде. Бактерии этого рода являются модельным объектом для изучения биодеградации ряда ароматических и полициклических соединений, в том числе, бензола, катехола и других (Nogales et al., 2017). Многие представители этого рода являются деструкторами синтетических пластических материалов, включая ПЭТ и ПС (Wilkes, Aristilde, 2017), а также участвуют в биокоррозии стали (Abdolahi et al., 2014). Известно, что представители вида Pseudomonas putida могут разлагать как обработанный ультрафиолетом ПЭТ (Vague et al., 2019), так и его мономеры – этиленгликоль (Franden et al., 2018) и терефталевую кислоту (Kenny et al., 2008). Были выделены штаммы P. putida, способные деградировать мономер ПС – стирол (Gassner, 2019). Штаммы P. putida входили также в состав микроконсорциума, осуществляющего коррозию стали (Aruliah, Ting, 2014). Проведенные нами микроскопические исследования подтвердили протекание процессов биодеструкции полимеров преимущественно в промышленной оборотной воде, поскольку после экспонирования в этой среде были наиболее заметны повреждения подложек из этих материалов. Таким образом, эта среда может быть наиболее агрессивной в отношении промышленных изделий и конструкций, изготовленных из пластика. По-видимому, это связано с тем, что поступление в оборотную воду различных химических примесей, образующихся в производственных процессах нефтесинтеза, способствует отбору штаммов-деструкторов полимеров и/или их мономерных составляющих. Кроме того, антимикробная обработка промышленной воды, проводимая согласно программе корректировки, приводит к смене состава микробного сообщества и появлению наиболее устойчивой к обработке микрофлоры. Такие циклические изменения также влияют и на структуру подложки, возможно, делая ее более доступной для биодеструкции (Лаптев и соавт., 2019).
Наиболее заметные изменения таксономического состава бактериальных сообществ в результате антимикробной обработки промышленной оборотной воды прослеживались на образцах ПС. На начальной стадии (10 сут) преобладающими в библиотеке с образца ПС являлись бактерии рода Blastomonas (Alphaproteobacteria), которые отсутствовали во всех остальных сравниваемых библиотеках. Возможно, это объясняется способностью бактерий рода Blastomonas продуцировать в пресной водной среде адгезины для формирования биопленок с другими бактериями (Rickard et al., 2004), что особенно актуально для образцов ПС с наиболее твердой и гладкой поверхностью. Еще одним специфическим доминантным родом для этой стадии экспозиции ПС в промышленной воде оказался Sphingobium, многие штаммы которого способны к деградации широкого круга ароматических соединений, включая стирол (Verma et al., 2014). Однако, по-видимому, представители этих родов оказались наиболее чувствительными к антимикробной обработке, поэтому в дальнейшем таксономический состав этих библиотек изменялся с накоплением к концу экспозиции бактерий родов Arenimonas и Pseudomonas. Накопление бактерий рода Pseudomonas на всех образцах в промышленной воде может быть результатом их участия в процессах как биодеградации полимеров и/или продуктов их деполимеризации, так и биокоррозии стали в этой среде. Для бактерий рода Arenimonas показано активное участие в деградации нефтепродуктов (Gao et al., 2019), что может способствовать накоплению их в загрязненной продуктами нефтехимического синтеза промышленной оборотной воде. Не исключена также возможность использования этими бактериями мономерных продуктов деградации пластических материалов.
Несмотря на то, что по уровню солености промышленная оборотная вода значительно ближе к пресной, чем к морской воде, общими компонентами библиотек для обоих типов пресноводных сред являются только бактерии доминантных родов Flavobacterium и Chryseolinea (Bacteroidetes), а также бактерии рода Aquabacterium (Alphaproteobacteria). Таким образом, особенности таксономического состава бактериальных сообществ обрастаний на поверхности полимерных материалов и стали в промышленной оборотной воде в основном связаны со специфическими физико-химическими условиями этой среды: повышенной соленостью, жесткостью и, особенно, значительным количеством продуктов нефтесинтеза.
Таксономический состав доминантных родов (топ-25) библиотек группы “Marine” был кардинально отличным от такового в группе “Industrial”. Общими для всех библиотек доминантными бактериями в группе “Marine” были морские альфапротеобактерии родов Marivita, Erythrobacter и Altererythrobacter. Доля бактерий родов Erythrobacter и Altererythrobacter была более высокой на образцах пластика (до 14 и 4% на последних этапах экспонирования соответственно). Это совпадало с результатами, полученными ранее при экспонировании образцов ПЭТ и ПС в Балтийском море, на основании чего предполагалось, что обильно представленные на них бактерии рода Erythrobacter катаболизируют не сам пластик, а ассоциированные с ним полициклические ароматические углеводороды, сорбированные из окружающей водной среды (Oberbeckmann et al., 2018). В то же время в геноме одного из штаммов Alterythrobacter (АС AP018498) (Maeda et al., 2018) представлены гены деградации стирола, что не исключает возможности участия представителей этого рода в деградации ПС. Кроме того, возможно участие в деградации стирола минорных компонентов сообщества, относящихся к родам Tropicibacter и Marinobacter (Tourova et al., 2020). Это не противоречит нашим результатам микроскопии, согласно которым на подложках ПС, экспонированных в морской воде, наблюдались точечные повреждения, в то время как на подложках ПЭТ в этой среде никаких повреждений не обнаружено (рис. 4).
Выявлены значительные различия таксономического состава микроорганизмов на образцах стали на первоначальном (10 сут) и последующих этапах (20–40 сут) экспонирования в морской воде. На первом этапе типичная морская микробиота отсутствовала, но при этом преобладали бактерии родов Methylobacterium и Sphingobium, для которых показано участие в формировании биопленок на металлических поверхностях (Ogawa et al., 2020). Возможно, это связано с избирательностью первичной адгезии микроорганизмов на металлической поверхности в соленой морской воде (обусловленной электростатическим взаимодействием с поверхностью) по сравнению с адгезией на поверхности пластика. Особый состав этой библиотеки обусловил ее группирование на графике PCA и на тепловых картах с библиотеками бактерий из пресноводных местообитаний, а не с остальными морскими (рис. 1, S2 и 2 ). На последующих этапах экспонирования (20–60 сут) таксономический состав библиотек с образцов стали кардинально изменялся с появлением в нем родов морских альфапротеобактерий, общих для всех остальных библиотек группы “Marine”. Главным отличием этих сообществ от сообществ, развивающихся на образцах пластика, стало присутствие бактерий доминантных родов Sulfurimonas (Epsilonproteobacteria) и Mariprofundus (Zetaproteobacteria). Представители этих таксонов были выявлены ранее при анализе бактериальных сообществ морских местообитаний (Dang et al., 2011; McBeth, Emerson, 2016). Предполагается, что бактерии рода Sulfurimonas участвуют в окислении восстановленных соединений серы и, возможно, водорода, образующихся в процессах биокоррозии (Little, Lee, 2007). В состав филума Zetaproteobacteria входят литотрофные железоокисляющие бактерии, которые присутствуют в различных морских экосистемах и быстро появляются в биопленках на поверхности стальных материалов, в результате чего предлагается рассматривать их в качестве маркеров протекающих в этой среде процессов Fe-окисления (Emerson, 2018). Обогащение обрастаний на поверхности стали в морской воде серо- и железоокисляющими бактериями, в отличие от одновременно экспонируемых образцов пластика, свидетельствует о влиянии этих бактерий на процессы биокоррозии и, возможно, о большей агрессивности этой среды в отношении металлоконструкций по сравнению с промышленной водой.
Полученные результаты могут найти применение в последующих исследованиях по выделению эффективных бактерий-деструкторов полимерных материалов для использования их в процессах утилизации бытовых и промышленных отходов.
Список литературы
Лаптев А.Б., Николаев Е.В., Куршев Е.В., Горяшник Ю.С. Особенности биодеструкции термопластов на основе полиэфиров в различных климатических зонах // Труды ВИАМ. 2019. № 7. С. 84–91. https://doi.org/10.18577/2307-6046-2019-0-7-84-91
Турова Т.П., Соколова Д.Ш., Назина Т.Н., Груздев Д.С., Лаптев А.Б. Филогенетическое разнообразие микробных сообществ с поверхности полиэтилентерефталатных материалов при экспозиции в водных средах // Микробиология. 2020. Т. 89. С. 99–110. https://doi.org/10.31857/S0026365620010152
Tourova T.P., Sokolova D.S., Nazina T.N., Grouzdev D.S., Laptev A.B. Phylogenetic diversity of microbial communities from the surface of polyethylene terephthalate materials exposed to different water environments // Microbiology (Moscow). 2020. V. 89. P. 96–106. https://doi.org/10.1134/S0026261720010154
Abdolahi A., Hamzah E., Ibrahim Z., Hashim S. Microbially influenced corrosion of steels by Pseudomonas aeruginosa // Corros. Rev. 2014. V. 32. P. 129–141.https://doi.org/10.1515/corrrev-2013-0047
Aruliah R., Ting Y.-P. Characterization of corrosive bacterial consortia isolated from water in a cooling tower // ISRN Corros. 2014. V. 2014. Art. 803219. https://doi.org/10.1155/2014/803219
Caporaso J.G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F.D., Costello E.K., Fierer N., Pena A.G., Goodrich J.K., Gordon J.I., Huttley G.A., Kelley S.T., Knights D., Koenig J.E., Ley R.E., Lozupone C.A., McDonald D., Muegge B.D., Pirrung M., Reeder J., Sevinsky J.R., Turnbaugh P.J., Walters W.A., Widmann J., Yatsunenko T., Zaneveld J., Knight R. QIIME allows analysis of high-throughput community sequencing data // Nat. Methods. 2010. V. 7. P. 335–336. https://doi.org/10.1038/nmeth.f.303
de Carvalho C.C.C.R. Marine biofilms: A successful microbial strategy with economic implications // Front. Mar. Sci. 2018. V. 5. Art. 126. https://doi.org/10.3389/fmars.2018.00126
Dang H., Chen R., Wang L., Shao S., Dai L., Ye Y., Guo L., Huang G., Klotz M.G. Molecular characterization of putative biocorroding microbiota with a novel niche detection of Epsilon- and Zetaproteobacteria in Pacific Ocean coastal seawaters // Environ. Microbiol. 2011. V. 13. P. 3059–3074. https://doi.org/10.1111/j.1462-2920.2011.02583.x
Dussud C., Meistertzheim A.L., Conan P., Pujo-Pay M., George M., Fabre P., Coudane J., Higgs P., Elineau A., Pedrotti M.L., Gorsky G., Ghiglione J.F. Evidence of niche partitioning among bacteria living on plastics, organic particles and surrounding seawaters // Environ. Pollut. 2018. V. 236. P. 807–816. https://doi.org/10.1016/j.envpol.2017.12.027
Emerson D. The role of iron-oxidizing bacteria in biocorrosion: a review // Biofouling. 2018. V. 34. P. 989–1000. https://doi.org/10.1080/08927014.2018.1526281
Franden M.A., Jayakody L.N., Li W.-J., Wagner N.J., Cleveland N.S., Michener W.E., Hauer B, Blank L.M., Wierckx N., Klebensberger J., Beckham G.T. Engineering Pseudomonas putida KT2440 for efficient ethylene glycol utilization // Metab. Eng. 2018. V. 48. P. 197–207. https://doi.org/10.1016/j.ymben.2018.06.003
Gao S., Liang J., Teng T., Zhang M. Petroleum contamination evaluation and bacterial community distribution in a historic oilfeld located in loess plateau in china // Appl. Soil Ecol. 2018. V. 136. P. 30–42. https://doi.org/10.1016/j.apsoil.2018.12.012
Gassner G.T. The styrene monooxygenase system // Methods Enzymol. 2019. V. 620. P. 423–453. https://doi.org/10.1016/bs.mie.2019.03.019
Hoellein T., Rojas M., Pink A., Gasior J., Kelly J. Anthropogenic litter in urban “Fresh” water ecosystems: Distribution and microbial interactions // PLoS One. 2014. V. 9. e98485. https://doi.org/10.1371/journal.pone.0098485
Kenny S.T., Nikodinovic-Runic J., Kaminsky W., Woods T., Babu R.P., Keely C.M., Blau W., O’Connor K.E. Up-Cycling of PET (polyethylene terephthalate) to the biodegradable plastic PHA (polyhydroxyalkanoate) // Environ. Sci. Technol. 2008. V. 42. P. 7696–7701. https://doi.org/10.1021/es801010e
Kesy K., Oberbeckmann S., Kreikemeyer B., Labrenz M. Spatial environmental heterogeneity determines young biofilm assemblages on microplastics in Baltic Sea mesocosms // Front. Microbiol. 2019. V. 10. Art. 1665. https://doi.org/10.3389/fmicb.2019.01665
Krueger M.C., Seiwert B., Prager A., Zhang S., Abel B., Harms H., Schlosse D. Degradation of polystyrene and selected analogues by biological Fenton chemistry approaches: Opportunities and limitations // Chemosphere. 2017. V. 173. P. 520–528. https://doi.org/10.1016/j.chemosphere.2017.01.089
Little B.J., Lee J.S. Microbiologically Influenced Corrosion. Hoboken, NJ: John Wiley & Sons Inc., 2007. Ch. 1e4.
Maeda A.H., Nishi S., Ishii S., Shimane Y., Kobayashi H., Ichikawa J., Kurosawa K., Arai W., Takami H., Ohta Y. Complete genome sequence of Altererythrobacter sp. strain B11, an aromatic monomer-degrading bacterium, isolated from deep-sea sediment under the seabed off Kashima, Japan // Genome Announc. 2018. V. 22. e00200-18. https://doi.org/10.1128/genomeA.00200-18
McBeth J.M., Emerson D. In situ microbial community succession on mild steel in estuarine and “Marine” environments: exploring the role of iron-oxidizing bacteria // Front. Microbiol. 2016. V. 7. Art. 767. https://doi.org/10.3389/fmicb.2016.00767
Nogales J., García J.L., Díaz E. Degradation of aromatic compounds in Pseudomonas: A systems biology view // Aerobic Utilization of Hydrocarbons, Oils and Lipids / Ed. Ro-jo F. Handbook of Hydrocarbon and Lipid Microbiology. Springer, Cham. 2017. P. 1–49. https://doi.org/10.1007/978-3-319-39782-5_32-1
Oberbeckmann S., Kreikemeyer B., Labrenz M. Environmental factors support the formation of specific bacterial assemblages on microplastics // Front. Microbiol. 2018. V. 8. Art. 2709. https://doi.org/10.3389/fmicb.2017.02709
Ogawa A., Takakura K., Hirai N., Kanematsu H., Kuroda D., Kougo T., Sano K., Terada S. Biofilm formation plays a crucial rule in the initial step of carbon steel corrosion in air and water environments // Materials. 2020. V. 13. Art. 923. https://doi.org/10.3390/ma13040923
Otake Y., Kobayashi T., Asabe H., Murakami K., Ono K. Biodegradation of low-density polyethylene, polystyrene, polyvinyl chloride, and urea formaldehyde resin buried under soil for over 32 years // J. Appl. Polym. Sci. 1995. V. 56. P. 1789–1796. https://doi.org/10.1002/app.1995.070561309
Rickard A.H., Gilbert P., Handley P.S. Influence of growth environment on coaggregation between freshwater biofilm bacteria // J. Appl. Microbiol. 2004. V. 96. P. 1367–1373. https://doi.org/10.1111/j.1365-2672.2004.02297.x
Segata N., Izard J., Waldron L., Gevers D., Gevers D., Miropolsky L., Garrett W.S., Huttenhower C. Metagenomic biomarker discovery and explanation // Genome Biol. 2011. V. 24. Art. R60. https://doi.org/10.1186/gb-2011-12-6-r60
Tourova T., Sokolova D., Nazina T., Grouzdev D., Kurshev E., Laptev A. Biodiversity of microorganisms colonizing the surface of polystyrene samples exposed to different aqueous environments // Sustainability. 2020. V. 12. Art. 3624. https://doi.org/10.3390/su12093624
Vague M., Chan G., Roberts C., Swartz N.A., Jay L., Mellies J.L. Pseudomonas isolates degrade and form biofilms on polyethylene terephthalate (PET) plastic // bioRxiv. 2019. 647321. https://doi.org/10.1101/647321
Verma H., Kumar R., Oldach P., Sangwan N., Khurana J.P., Gilbert J.A., Rup L. Comparative genomic analysis of nine Sphingobium strains: insights into their evolution and hexachlorocyclohexane (HCH) degradation pathways // BMC Genomics. 2014. V. 15. Art. 1014. https://doi.org/10.1186/1471-2164-15-1014
Webb H., Arnott J., Crawford R., Ivanova E. Plastic degradation and its environmental implications with special reference to poly(ethylene terephthalate) // Polymers. 2013. V. 5. P. 1–18. https://doi.org/10.3390/polym5010001
Wilkes R., Aristilde L. Degradation and metabolism of synthetic plastics and associated products by Pseudomonas spp.: capabilities and challenges // J. Appl. Microbiol. 2017. V. 123. P. 582–593. https://doi.org/10.1111/jam.13472
Yang Y., Yang J., Wu W.-M., Zhao J., Song Y., Gao L., Yang R., Jiang L. Biodegradation and mineralization of polystyrene by plastic-eating mealworms: part 2. Role of gut microorganisms // Environm. Sci. Technol. 2015. V. 49. P. 12087–12093.https://doi.org/10.1021/acs.est.5b02663
Yoshida S., Hiraga K., Takehana T., Taniguchi I., Yamaji H., Maeda Y., Toyohara K., Miyamoto K., Kimura Y., Oda K. A bacterium that degrades and assimilates poly(ethylene terephthalate) // Science. 2016. V. 353. P. 759–759. https://doi.org/10.1126/science.aad6359
Дополнительные материалы
- скачать ESM.docx
- Таблица S1. Список доминантных (топ-25) родов по относительному количеству ридов представителей каждого рода в сумме ридов по каждому типу материала (ПЭТ, ПС и СТ) в каждой среде экспозиции (группы “Industrial”, “Marine” и “Fresh”).
- скачать ESM_Page_4.jpg
- Рисунок S1. Относительная доля последовательностей гена 16S рРНК (ридов) бактерий на уровне филумов/классов в библиотеках обрастаний на образцах полиэтилентерефталата (PET), полистирола (PS) и стали 20 (ST), экспонированных в промышленной оборотной (группа “Industrial”), морской (группа “Marine”) и природной пресной (группа “Fresh”) водных средах.
- скачать ESM_Page_5.jpg
- Рисунок S2. Тепловая карта (heatmap) распределения 778 родов бактерий по 26 библиотекам обрастаний с образцов ПЭТ (PET), ПС (PS) и СТ (ST).
- скачать ESM_Page_6.jpg
- Рисунок S3. Результаты анализа LefSe сравнения бактериальных сообществ из обрастаний с поверхности исследуемых материалов (ПЭТ (PET), ПС (PS) и СТ (ST)) в различных водных средах (группы “Industrial”, “Marine” и “Fresh”).
- скачать ESM_Page_7.jpg
- Рисунок S4. Распределение родов-маркеров, специфичных для групп “Industrial” и “Marine”, по типам материала образцов и сред экспозиции.


