Микробиология, 2021, T. 90, № 4, стр. 492-499
Молекулярно-генетический полиморфизм дрожжей Kluyveromyces dobzhanskii
Е. С. Наумова a, *, Ч.-Ф. Ли b, Г. И. Наумов a
a Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”
117519 Москва, Россия
b Department of Applied Science, National Tsing Hua University of Education
30014 Hsinchu, Taiwan
* E-mail: lena_naumova@yahoo.com
Поступила в редакцию 03.03.2021
После доработки 10.03.2021
Принята к публикации 11.03.2021
Аннотация
На материале штаммов различного экологического и географического происхождения изучен внутривидовой полиморфизм диких дрожжей Kluyveromyces dobzhanskii – ближайших родственников культурных молочных дрожжей K. lactis и K. marxianus. С помощью микросателлитного типирования, филогенетического анализа нуклеотидных последовательностей внутренних транскрибируемых спейсеров ITS1/ITS2 и митохондриального гена COX2 установлено, что вид K. dobzhanskii имеет сложный состав и представлен, по крайней мере, тремя географическими популяциями: североамериканской (включая типовую культуру CBS 2104), европейской и дальневосточной. Штаммы различного географического происхождения характеризуются уникальными нуклеотидными заменами в ITS1/ITS2-участке и митохондриальном гене COX2. У европейских штаммов обнаружена корреляция (GTG)5-профилей с источником выделения. Штаммы, изолированные в Испании из насекомых, имеют уникальные паттерны.
Дрожжи Kluyveromyces – второй по значимости после Saccharomyces объект фундаментальных и прикладных исследований. Они имеют большое биотехнологическое значение и используются для производства различных гетерологичных белков медицинского и пищевого значения, а также в качестве биологических агентов, подавляющих развитие вредных микроорганизмов, и продуцентов биоэтанола из лигноцеллюлозных отходов сельского хозяйства и деревообрабатывающей промышленности (Fonseca et al., 2008; Suzuki et al., 2014). В настоящее время род Kluyveromyces включает семь видов: наземные K. lactis, K. marxianus, K. dobzhanskii и K. wickerhamii, а также морские K. aestuarii, K. nonfermentas и K. siamensis (Kurtzman, 2003, Lachance, 2011). Дрожжи K. lactis и K. marxianus – постоянные компоненты различных молочнокислых продуктов и могут использоваться в качестве пробиотических микроорганизмов (Maccaferri et al., 2012; Romanin et al., 2016; Наумова и соавт., 2017). Их ближайшим филогенетическим родственником являются дикие дрожжи K. dobzhanskii, которые распространены во многих частях света и выделяются исключительно из природных источников: растений, насекомых, почвы, шляпочных грибов и др. Эти дрожжи представляют собой удобную модель для популяционно-генетических исследований. В отличие от молочных штаммов Kluyveromyces, дрожжи K. dobzhanskii практически не изучены. Имеющиеся в литературе сведения по электрофорезу изоферментов и ПДРФ-анализу митохондриальной ДНК отдельных штаммов указывают на возможную генетическую гетерогенность вида K. dobzhanskii (Sidenberg, Lachance, 1986; Belloch et al., 1997).
Современная классификация аскомицетовых дрожжей основана на филогенетическом анализе нуклеотидных последовательностей ряда молекулярных маркеров (баркодов), прежде всего домена D1/D2 гена 26S рРНК и 5.8S–ITS-фрагмента, включающего ген 5.8S РНК и внутренние транскрибируемые спейсеры ITS1/ITS2 (Kurtzman, Robnett, 1998; Kurtzman, 2003). ITS-район характеризуется значительной межвидовой дивергенцией, его длина постоянна у штаммов одного и того же вида, а последовательность может варьировать (James et al., 1998). Поэтому этот участок рДНК используется не только для разделения близкородственных видов, но и для изучения генетической изменчивости дрожжей на популяционном уровне. Используемый в современной таксономии дрожжей митохондриальный ген COX2 обладает значительной вариабельностью и больше подходит для дифференциации отдельных штаммов, чем для разграничения видов. На ряде грибных таксонов отмечено, что видовая филогения, построенная на основании анализа ядерных и митохондриальных генов, может не совпадать (Schoch et al., 2012; Vu et al., 2016).
Микросателлитные маркеры имеют множественную локализацию в геноме дрожжей, что позволяет сравнивать большое количество полиморфных локусов. На дрожжах Saccharomyces было показано, что с помощью микросателлитного маркера (GTG)5 можно дифференцировать отдельные виды этого рода, а также идентифицировать отдельные штаммы (Baleiras Couto et al., 1996; Naumova et al., 2000, 2011). Ранее мы использовали этот маркер для изучения внутривидового полиморфизма дрожжей K. lactis (Наумова и соавт., 2005).
Целью настоящей работы было изучение молекулярного полиморфизма дрожжей K. dobzhanskii на материале штаммов различного экологического и географического происхождения.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Штаммы и среды. Использованные в работе штаммы и их происхождение представлены в табл. 1. Дрожжи культивировали при 28°С на полной среде YPD (г/л): бакто-агар (“Difco”, США) – 20; глюкоза (“Merck”, Германия) – 20; дрожжевой экстракт (“Difco”) – 10; бакто-пептон (“Difco”) – 20.
Полимеразную цепную реакцию осуществляли на ДНК-амплификаторе ”Bio-Rad” (США). Дрожжевую ДНК выделяли с помощью набора Genomic DNA Purification Kit (“Fermentas”, Литва). Для амплификации домена D1/D2 гена 26S рРНК и 5.8S-ITS-фрагмента (ген 5.8S РНК и внутренние транскрибируемые спейсеры ITS1/ITS2) использовали стандартные праймеры (Kurtzman, Robnett, 2003), а для амплификации митохондриального гена СОХ2 использовали праймеры СОХ23 (5'-GGTATTTTAGAATTCATGA-3') и СОХ25 (5'-ATTTATTGTTCGTTTAATCA-3') (Belloch et al., 2000). ПЦР проводили в 30 мкл буфера, содержащего 2.5 мМ MgCl2, 0.1 мМ каждого дНТФ, 50 пмоль каждого праймера, 2.5 единицы Taq-полимеразы (“Синтол”, Россия), 20–200 нг ДНК. Начальную денатурацию осуществляли при 94°С в течение 3 мин, затем 30 циклов в следующем режиме: денатурация при 94°С, 45 с; отжиг праймеров при 52°С, 30 с; синтез ДНК при 72°С, 120 с; конечная достройка при 72°С, 10 мин. Продукты амплификации подвергали электрофорезу в 1%-ном агарозном геле при 60–65 В в 0.5× TBE буфере (45 мМ трис, 10 мМ ЭДТА, 45 мМ борная кислота; рН 8.0) в течение 2 ч.
Амплификацию ДНК с микросателлитным праймером (GTG)5 проводили в 30 мкл реакционной смеси, содержащей 3 мМ MgCl2, 0.3 мМ дНТФ, 1.25 ед. акт. Taq-полимеразы (“Синтол”, Россия), 30 пмоль праймера, 20–200 нг анализируемой геномной ДНК. ПЦР (30 циклов) осуществляли в режиме: денатурация ДНК при 94°С, 30 с; отжиг праймера при 52°С, 30 с; синтез ДНК при 72°C, 1 мин. Разделение амплифицированной ДНК проводили в 1.6%-ном агарозном геле при 55–60 В в 0.5× ТВЕ в течение 3.5–4 ч.
Гели окрашивали бромистым этидием, промывали в дистиллированной воде и фотографировали в ультрафиолетовом свете на трансиллюминаторе Vilber Lourmat (Франция). В качестве маркера молекулярных весов использовали препарат 1 kb DNA Ladder (“Thermo Fisher Scientific”, США).
Секвенирование. Амплифицованныe фрагменты домена D1/D2, 5.8S-ITS-фрагмента и гена COX2 элюировали из геля с помощью набора Silica Beads DNA Gel Extraction Kit (“Thermo Fisher Scientific”, США), согласно протоколу фирмы-изготовителя. Нуклеотидные последовательности домена D1/D2, 5.8S-ITS-участка и гена COX2 определяли по двум цепям с помощью прямого секвенирования по методу Сенгера на автоматическом секвенаторе “Applied Biosystems 3730” (США).
Филогенетический анализ. Полученные нуклеотидные последовательности анализировали при помощи программы SeqMan package (“DNA Star Inc.”, США). Поиск гомологии с известными нуклеотидными последовательностями проводили в базе данных GenBank (http://www.ncbi.nlm.nih.gov/ genbank/) с помощью программы BLAST. Множественное выравнивание изученных нуклеотидных последовательностей осуществляли с использованием программы BioEdit (http://www.mbio.ncsu.edu/BioEdit/bioedit.html). Филогенетические деревья строили методом объединения соседей (Neighbor-Joining) в программе MEGA 7 (Kumar et al., 2013). В качестве внешней группы использовали типовую культуру дрожжей Kluyveromyces marxianus CBS 712. Индексы бутстрепа, определяющие статистическую достоверность выделения групп, определяли для 1000 псевдореплик.
Филогенетические связи между штаммами K. dobzhhanskii устанавливали также путем сравнения профилей ПЦР-продуктов, амплифицированных с микросателлитным праймером (GTG)5. Дендрограмму, основанную на матрице различий по (GTG)5-профилям, строили с помощью программы Neighbor-Joining из компьютерного пакета TREECON (van der Peer, de Wachter, 1994). Индексы бутстрепа, определяющие статистическую достоверность выделения групп, определяли для 100 псевдореплик. В качестве внешней группы использовали типовую культуру дрожжей K. marxianus CBS 712.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Объектом исследования служили 29 штаммов Kluyveromyces, выделенные из различных природных источников в Европе, Северной Америке и на Дальнем Востоке России (табл. 1). Штаммы получены из отечественных и международных коллекций. Согласно нуклеотидным последовательностям домена D1/D2 26S рДНК, все изученные штаммы относятся к виду K. dobzhanskii. В качестве контроля была использована типовая культура CBS 2104, выделенная из Drosophila pseudoobscura в США. Генетическое родства штаммов K. dobzhanskii различного происхождения изучали с помощью микросателлитного типирования и филогенетического анализа нуклеотидных последовательностей 5.8S-ITS участка и митохондриального гена COX2.
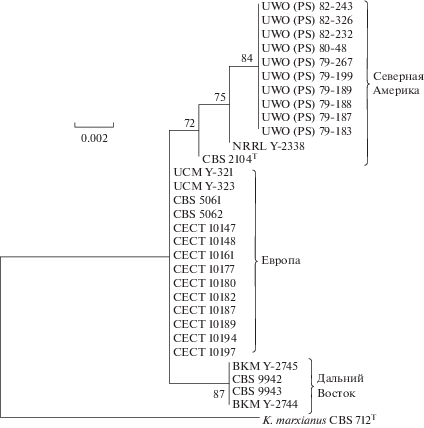
ПЦР с микросателлитным праймером (GTG)5. ПЦР-профили изученных штаммов K. dobzhanskii значительно отличались от паттерна тест-штамма K. marxianus CBS 712 (рисунок не приводится). На основании полученных (GTG)5-профилей была построена дендрограмма (рис. 1). Штаммы K. dobzhanskii сформировали отдельный кластер относительно тест-штамма K. marxianus CBS 712. Внутри этого кластера выделяются три четкие группы, объединяющие штаммы с наиболее похожими паттернами. Внутри каждой группы обнаружена вариация по ПЦР-профилям, проявляющаяся в наличии или отсутствии отдельных фрагментов.
Рис. 1.
Дендрограмма родства штаммов Kluyvromyces dobzhanskii, основанная на матрице различий по ПЦР-профилям с микросателлитным праймером (GTG)5. В качестве внешней группы использовали типовую культуру K. marxianus CBS 712. Приводятся значения бутстрепа >50%. T – типовая культура.

Первую группу сформировали североамериканские штаммы. Внутри этой группы выделяются три подгруппы. Первая включает типовую культуру CBS 2104 и штамм NRRL Y-2338 (США), а остальные две подгруппы представлены канадскими изолятами. Следует отметить, что штаммы, выделенные из галлов Prunus virginiana в 1979 г., отличаются по (GTG)5-паттернам от остальных канадских штаммов. Европейские штаммы объединились во вторую группу, при этом изолированные из различных насекомых в Испании штаммы отличались по ПЦР-профилям от штаммов, выделенных с поверхности грибов во Франции (CBS 5061, CBS 5062) и листьев дуба на Украине (UCM Y-321, UCM Y-323). Дальневосточные изоляты CBS 9942, CBS 9943, ВКМ Y-2744 и ВКМ Y-2745 сформировали третью группу (рис. 1).
Таким образом, обнаружена корреляция (GTG)5-паттернов с географическим происхождением штаммов, а для европейских штаммов и с источником выделения.
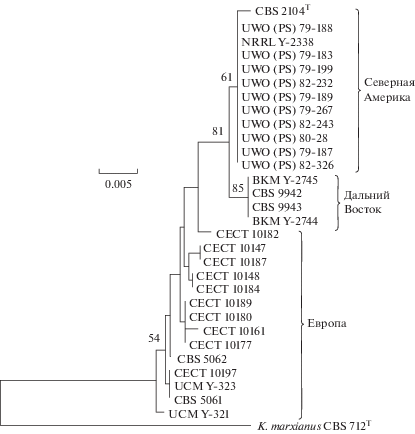
Филогенетический анализ нуклеотидных последовательностей 5.8S-ITS-фрагмента рДНК. Мы провели секвенирование 5.8S-ITS-района у 26 изученных штаммов. У трех европейских штаммов (СЕСТ 10177, СЕСТ 10180, СЕСТ 10187) нуклеотидные последовательности этого участка рДНК были определены ранее (Belloch et al., 2002). Полученные нуклеотидные последовательности сравнили между собой и с последовательностями 5.8S-ITS-участка типовой культуры K. dobzhanskii CBS 2104, а также тест-штамма K. marxianus CBS 712.
На основании полученных нуклеотидных последовательностей было построено филогенетическое древо, на котором выделяются три группы штаммов (рис. 2). Первая группа образована североамериканскими штаммами. Десять канадских штаммов имеют идентичные нуклеотидные последовательности 5.8S-ITS-участка, которые отличаются одной заменой от штамма NRRL Y-2338 и двумя заменами от CBS 2104, соответственно, в 69 и 199 позициях (нумерация приводится по последовательности типовой культуры CBS 2104). Вторую группу сформировали 14 европейских штаммов, имеющих идентичные 5.8S-ITS-последовательности. Дальневосточные штаммы CBS 9942, CBS 9943, BKM Y-2745 и BKM Y-2744, также имеющие идентичные последовательности, вошли в третью группу. Различия по 5.8S-ITS-последовательностям между штаммами трех групп составили от трех до пяти нуклеотидных замен. В тоже время, 5.8S-ITS-последовательности штаммов K. dobzhanskii и типовой культуры K. marxianus CBS 712 различаются более чем 20 нуклеотидными заменами.
Рис. 2.
Филогенетическое родство штаммов Kluyveromyces dobzhanskii различного географического происхождения, основанное на анализе нуклеотидных последовательностей 5.8S-ITS-фрагмента рДНК. Приводятся значения бутстрепа >50%. Шкала соответствует 2 нуклеотидным заменам на 1000 нуклеотидных позиций. В качестве внешней группы использовали типовую культуру K. marxianus CBS 712. T – типовая культура.
Штаммы различного географического происхождения характеризуются уникальными нуклеотидными заменами. В отличие от североамериканских штаммов, у всех европейских и дальневосточных штаммов имеется замена нуклеотида G на A в 41 позиции. Дальневосточные штаммы характеризуются двумя дополнительными транзициями G–A в 38 и 564 позициях.
Филогенетический анализ нуклеотидных последовательностей митохондриального гена COX2. У 29 изученных штаммов была определена нуклеотидная последовательность фрагмента гена COX2 размером 585 п.н. Полученные последовательности сравнили с последовательностями гена COX2 типовых культур K. dobzhanskii CBS 2104 и K. marxianus CBS 712, имеющимися в GenBank. На основании сравнительного анализа нуклеотидных последовательностей было построено филогенетическое древо (рис. 3).
Рис. 3.
Филогенетическое родство штаммов Kluyveromyces dobzhanskii различного географического происхождения, основанное на анализе нуклеотидных последовательностей митохондриального гена COX2. Приводятся значения бутстрепа >50%. Шкала соответствует 5 нуклеотидным заменам на 1000 нуклеотидных позиций. В качестве внешней группы использовали типовую культуру K. marxianus CBS 712. T – типовая культура.
Изученные штаммы K. dobzhanskii разделились на три кластера. В первый кластер вошли североамериканские изоляты. Одиннадцать штаммов имели идентичные последовательности гена COX2 и отличались от типовой культуры CBS 2104 одной нуклеотидной заменой. Второй кластер образовали четыре дальневосточных штамма, имеющие идентичные последовательности. Европейские штаммы сформировали третий кластер. Этот кластер является наиболее гетерогенным, входящие в него 14 европейских штаммов различаются 2–7 нуклеотидными заменами. Несмотря на указанные различия, все 14 европейских штаммов характеризуются двумя уникальными заменами: трансверсией T–A и транзицией C–T соответственно, в 177 и 182 позициях (нумерация приводится по последовательности типовой культуры CBS 2104). Дальневосточные штаммы имеют уникальную замену C–T в пятой позиции. Различия в последовательностях гена COX2 европейских и остальных штаммов K. dobzhanskii составили от 8 до 11 замен, а различия с последовательностью этого гена типовой культуры K. marxianus CBS 712 превышали 28 нуклеотидов.
Таблица 1.
Изученные штаммы дрожжей Kluyveromyces dobzhanskii и их происхождение
| Штамм | Источник выделения | Место выделения |
|---|---|---|
| CBS 2104 (T) | Drosophila pseudoobscura | США |
| CECT 10147 | Euoxa segetum | Испания |
| CECT 10148 | Noctua pronuba | Испания |
| CECT 10161 | Trigonophora meticulosa | Испания |
| CECT 10177 | Noctua pronuba | Испания |
| CECT 10180 | Taenicompa gotica | Испания |
| CECT 10182 | Noctua pronuba | Испания |
| CECT 10187 | Synvaleria jaspidea | Испания |
| CECT 10189 | Synvaleria jaspidea | Испания |
| CECT 10194 | Antitype flavicincta | Испания |
| CECT 10197 | Rhizotype venusta | Испания |
| CBS 5061 | Hymenochaete rubiginosa | Франция |
| CBS 5062 | Fistulina hepatica | Франция |
| NRRL Y-2338 | Папоротник | США |
| UCM Y-321 | Листья, Quercus sp. | Украина |
| UCM Y-323 | Листья, Quercus sp. | Украина |
| UWO (PS) 79-183 | Галл, Prunus virginiana | Мельбурн, Канада |
| UWO (PS) 79-187 | Галл, Prunus virginiana | Мельбурн, Канада |
| UWO (PS) 79-188 | Галл, Prunus virginiana | Мельбурн, Канада |
| UWO (PS) 79-189 | Галл, Prunus virginiana | Мельбурн, Канада |
| UWO (PS) 79-199 | Галл, Prunus virginiana | Пинери, Канада |
| UWO (PS) 79-267 | Галл, Prunus virginiana | Пинери, Канада |
| UWO (PS) 80-28 | Галл, Prunus virginiana | Пинери, Канада |
| UWO (PS) 82-232 | Drosophila sp. | Пинери, Канада |
| UWO (PS) 82-243 | Drosophila sp. | Пинери, Канада |
| UWO (PS) 82-326 | Drosophila sp. | Пинери, Канада |
| CBS 9942 | Chloranthus japonicus | Владивосток, Россия |
| CBS 9943 | Actinida arguta | Владивосток, Россия |
| ВКМ Y-2744 | Kalopanoax septemlobus | Владивосток, Россия |
| ВКМ Y-2745 | Schisandra chinensis | Владивосток, Россия |
Примечание. Сокращенные названия коллекций: ВКМ – Всероссийская коллекция микроорганизмов (Пущино, Россия); CBS – The Westerdijk Fungal Biodiversity Institute (Утрехт, Нидерланды); CECT – Spanish Type Culture Collection, University of Valencia (Валенсия, Испания); NRRL – USDA-ARS Culture Collection, National Center for Agricultural Utilization Research, Пеория, США; UCM – Украинская коллекция микроорганизмов, Институт микробиологии и вирусологии НАН, Киев, Украина; UWO (PS) – Culture collection of the Departament of Biology, University of Western Ontario (Онтарио, Канада).
Полученные данные свидетельствуют о значительном полиморфизме дрожжей K. dobzhanskii, связанным с географическим происхождением штаммов. Результаты, полученные с помощью микросателлитного типирования, филогенетического анализа нуклеотидных последовательностей внутренних транскрибируемых спейсеров ITS1/ITS2 и митохондриального гена COX2, хорошо согласуются. По всем трем молекулярным маркерам изученные штаммы K. dobzhanskii разделились на четкие группы, соответствующие трем географическим популяциям: североамериканской, европейской и дальневосточной. Обнаружена корреляция (GTG)5-профилей европейских штаммов с источником их выделения: изоляты из насекомых имеют уникальные паттерны. Европейская популяция также гетерогенна по нуклеотидным последовательностям митохондриального гена COX2. В пределах каждой из трех географических популяций штаммы имели практически идентичные 5.8S-ITS-последовательности, тогда как штаммы из разных популяций отличались по последовательностям этого участка рДНК. У штаммов, выделенных в Северной Америке и Европе, замены обнаружены в ITS1, тогда как ITS2-последовательности у них были идентичными. Напротив, у дальневосточных штаммов замены выявлены также в ITS2-участке. Все обнаруженные нуклеотидные замены являются уникальными и коррелируют с географическим происхождением штаммов.
Следует отметить, что в отечественных и международных коллекциях имеется ограниченное количество дрожжей K. dobzhanskii, поэтому выделение новых штаммов этого вида из природы в Дальневосточной Азии и других регионах мира позволит более полно изучить внутривидовую структуру этих дрожжей и обнаружить новые географические популяции.
Список литературы
Наумова Е.С., Сухотина Н.Н., Наумов Г.И. Молекулярные маркеры, дифференцирующие молочные дрожжи Kluyveromyces lactis var. lactis от их ближайших диких родственников – европейской популяции “krassilnikovii” // Микробиология. 2005. Т. 74. С. 387–393.
Naumova E.S., Sukhotina N.N., Naumov G.I. Molecular markers for differentiation between the closely related dairy yeast Kluyveromyces lactis var. lactis and wild Kluyveromyces lactis strains from the European “krassilnikovii” population // Microbiology (Moscow). 2005. V. 74. P. 329–335.
Наумова Е.С., Садыкова А.Ж., Михайлова Ю.В., Наумов Г.И. Полиморфизм лактозных генов молочных дрожжей Kluyveromyces marxianus, потенциальных проиботических микроогранизмов // Микробиология. 2017. Т. 86. С. 335–343.
Naumova E.S., Sadykova A.Zh., Michailova Yu.V., Naumov G.I. Polymorphism of lactose genes in the dairy yeasts Kluyveromyces marxianus, potential probiotic microorganisms // Microbiology (Moscow). 2017. V. 86. P. 363–369.
Baleiras Couto M.M., Eijsma B., Hofstra H., Huis in’t Veld J.H., van der Vossen J.M. Evaluation of molecular typing techniques to assign genetic diversity among Saccharomyces cerevisiae strains // Appl. Environ. Microbiol. 1996. V. 62. P. 41–46.
Belloch C., Barrio E., Uruburu F., Garcia M.D., Querol A. Characterization of four species of the genus Kluyveromyces by mitochondrial DNA restriction analysis // Syst. Appl. Microbiol. 1997. V. 20. P. 397–408.
Belloch C., Querol A., Garcia M.D., Barrio E. Phylogeny of the genus Kluyveromyces inferred from the mitochondrial cytochrome-c oxidase II gene // Int. J. System. Evol. Microbiol. 2000. V. 50. P. 405–416.
Belloch C., Fernandes-Espinar T., Querol A., Garcia M.D., Barrio E. An analysis of inter- and intraspecific genetic variabilities in the Kluyveromyces marxianus group of yeast species for the reconcideration of the K. lactis taxon // Yeast. 2002. V. 19. P. 257–268.
Fonceca G.G., Heizle E., Wittmann C., Gormbert A.K. The yeast Kluyveromyces marxianus and its biotechnological potential // Appl. Microbiol. Biotechnol. 2008. V. 79. P. 339–354.
James S.A., Collins M.D., Roberts I.N. Use of an rRNA internal transcribed spacer region to distinguish phylogenetically closely related species of the genera Zygosaccharomyces and Torulaspora // Int. J. Syst. Bacteriol. 1996. V. 46. P. 189–194.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874.
Kurtzman C.P. Phylogenetic circumscription of Saccharomyces, Kluyveromyces and other members of the Saccharomycetaceae, and the proposal of the new genera Lachancea, Nakaseomyces, Naumovia, Vanderwaltozyma and Zygotorulospora // FEMS Yeast Res. 2003. V. 4. P. 233–245.
Kurtzman C.P., Robnett C.J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences // Antonie van Leeuwenhoek. 1998. V. 73. P. 331–371.
Lachance M.-A. Kluyveromyces van der Walt (1971) // The Yeasts. A Taxonomic Study / Eds. Kurtzman C.P., Fell J.W., Boekhout T. Amsterdam: Elsevier, 2011. P. 471–482.
Maccaferri S., Klinder A., Brigidi P., Cavina P., Costabileb A. Potential probiotic Kluyveromyces marxianus B0399 modulates the immune response in CACO-2 cells and peripheral blood mononuclear cells and impacts the human gut microbiota in an in vitro colonic model system // Appl. Environ. Microbiol. 2012. V. 78. P. 956–964.
Naumova E.S., Naumov G.I., Molina F.I. Genetic variation among European strains of Saccharomyces paradoxus: results from DNA fingerprinting // Syst. Appl. Microbiol. 2000. V. 23. P. 86–92.
Naumova E.S., Naumov G.I., Michailova Yu.V., Martynenko N.N., Masneuf-Pomarède I. Genetic diversity study of the yeast Saccharomyces bayanus var. uvarum reveals introgressed subtelomeric Saccharomyces cerevisiae genes // Res. Microbiol. 2011. V. 162. P. 204–213.
Romanin D.E., Llopis S., Genovés S., Martorell P., Ramón V.D., Garrote G.L., Rumbo M.1 Probiotic yeast Kluyveromyces marxianus CIDCA 8154 shows anti-inflammatory and anti-oxidative stress properties in in vivo models // Beneficial Microbes. 2016. V. 7. № 4. P. 83–93
Schoch C.L., Seifert K.A., Huhndorf S., Robert V., Spouge J.L., Levesque C.A., Chen W. Fungal Barcoding Consortium. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi // Proc. Natl. Acad. Sci. USA. 2012. V. 109. P. 6241–6246.
Sidenberg D.G., Lachance M.-A. Electophoretic isoenzyme variation in Kluyveromyces populations and revision of Kluyveromyces marxianus (Hansen) van der Walt. // Int. J. Syst. Bacteriol. 1986. V. 36. P. 94–102.
Suzuki T., Hoshino T., Matsushika A. Draft genome sequence of Kluyveromyces marxianus strain DMB1, isolated from sugarcane bagasse hydrolysate // Genome Announc. 2014. V. 2. e00733–14.
Van de Peer Y., De Wachter R. TREECON for Windows: a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment // Comp. Appl. Biosci. 1994. V. 10. P. 569–570.
Vu D., Groenewald M., Szo’ke S., Cardinali G., Eberhardt U., Stielow B., de Vries M., Verkleij G.J., Crous P.W., Boekhout T., Robert V. DNA barcoding analysis of more than 9000 yeast isolates contributes to quantitative thresholds for yeast species and genera delimitation // Stud. Mycol. 2016. V. 85. P. 91–105.
Дополнительные материалы отсутствуют.


