Микробиология, 2021, T. 90, № 5, стр. 564-573
Gemmata palustris sp. Nov., новый планктомицет, выделенный из низинного болота севера России
А. А. Иванова a, *, И. С. Куличевская a
a Институт микробиологии им. С.Н. Виноградского, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” РАН
119071 Москва, Россия
* E-mail: ivanovastasja@gmail.com
Поступила в редакцию 22.04.2021
После доработки 13.05.2021
Принята к публикации 13.05.2021
Аннотация
Планктомицеты являются типичными обитателями северных болотных экосистем. В настоящей работе сообщается о выделении нового планктомицета рода Gemmata, штамм G18T из низинного болота северо-запада России. Этот изолят был представлен одиночными или собранными в небольшие группы сферическими клетками, обладающими подвижностью в экспоненциальной фазе роста. Штамм G18T являлся хемоорганотрофным, психротолерантным мезофилом с оптимумом роста при pH 7.0 и температуре 15–20°C. В качестве источников углерода и энергии новый планктомицет предпочитал полисахариды пектин, ксилан, лихенан, ксантановую камедь и фитагель. Нуклеотидная последовательность гена 16S рРНК штамма G18T обнаруживала 95.7–98.9% сходства по сравнению с отмеченной у пресноводных планктомицетов Gemmata obscuriglobus DSM 5831Т и “Gemmata massiliana” IIL30. Геном штамма G18T, размером 9.23 × 106 п.о., включал 7631 белок-кодирующих генов и 3 копии оперона рРНК (генов 5S, 16S и 23S рРНК). Содержание G + C в ДНК составляло 65 мол. %. Согласно калькулятору ANI, сходство генома G18Т с ранее описанными представителями рода Gemmata составляло 86.0% с “G. massiliana” IIL30 и 79.7% с G. obscuriglobus DSM 5831Т. Ряд различий в фенотипических и генотипических свойствах позволил классифицировать штамм G18T в качестве нового вида рода Gemmata – Gemmata palustris sp. nov. с типовым штаммом G18T (=KCTC 82682 = VKM B-3541T).
Семейство Gemmataceae объединяет почкующиеся аэробные хемоорганотрофные планктомицеты с клетками сферической или овальной формы, которые встречаются одиночно или собраны в розетки и дендриформные структуры (Kulichevskaya et al., 2017). На текущий момент семейство насчитывает 7 родов: Gemmata (Franzmann, Skerman, 1984), Zavarzinella (Kulichevskaya et al., 2009), Tuwongella (Seeger et al., 2017), Telmatocola (Kulichevskaya et al., 2012), Limnoglobus (Kulichevskaya et al., 2020b), Fimbriiglobus (Kulichevskaya et al., 2017), Frigoriglobus (Kulichevskaya et al., 2020a) и Thermogemmata (Elcheninov et al., 2021). Одной из отличительных черт представителей данного семейства являются большие размеры геномов, в среднем 9 × 106 п.о., а у болотного планктомицета Fimbriiglobus ruber SP5T – 12.3 × 106 п.о. (Ravin et al., 2018). Анализ геномных данных выявил высокое содержание в геномах планктомицетов гликозилгидролаз, что может свидетельствовать о наличии способности данных микроорганизмов к деструкции различных полисахаридов (Kulichev-sk-aya et al., 2020b). Согласно экспериментальным данным слабая целлюлозолитическая активность была показана для планктомицета Telmatocola sphagniphila SP2T (Kulichevskaya et al., 2012) и Fri-goriglobus tundricola PL17T (Kulichevskaya et al., 2020a). Хитинолитические свойства были выявлены у болотного планктомицета Fimbriiglobus ruber SP5T. Интересно, что рост микроорганизма был возможен, когда хитин использовался в качестве единственного источника азота, а не углерода (Ravin et al., 2018). Дополнительным свидетельством способности Gemmata-подобных планктомицетов к гидролизу хитина является увеличение доли этой группы бактерий в образцах торфа верхового болота, обогащенных хитином, выявленного методом метатранскриптомного анализа (Ivanova et al., 2018).
Планктомицеты семейства Gemmataceae встречаются повсеместно. Несколько представителей новых родов данного семейства были выделены из пресноводных водоемов (Franzmann, Skerman, 1984; Seeger et al., 2017; Kulichevskaya et al., 2020b). В недавней работе был охарактеризован первый термофильный планктомицет рода Thermogemmata, изолированный из горячего источника Камчатки (Elcheninov et al., 2021). С введением в практику методов молекулярного анализа Gemmata-подобные планктомицеты были обнаружены в верховых сфагновых болотах (Куличевская и соавт., 2006; Ivanova, Dedysh, 2012; Dedysh, Ivanova, 2019). Попытки выделения новых микроорганизмов из данных экосистем привели к описанию четырeх новых родов семейства Gemmataceae (Kulichevskaya et al., 2009, 2012, 2017, 2020a). Согласно исследованию, направленному на сравнение микробного разнообразия верховых и низинных болот, в последних Gemmata-подобные планктомицеты также составляли существенную долю бактериального сообщества (Ivanova et al., 2020).
Настоящая работа посвящена описанию первого планктомицета, выделенного из низинного болота северо-западной части России. Согласно сравнительному анализу генов 16S рРНК, ближайшими родственниками нового изолята были пресноводная G. obscuriglobus DSM 5831Т и “G. massiliana” IIL30, выделенная из воды больничной сети водоснабжения с оптимумом роста 30°С.
Таким образом, целью настоящего исследования явилось изучение совокупности экофизиологических и геномных характеристик нового штамма G18T и определение его таксономического статуса.
ОБЪЕКТЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Процедура выделения и условия культивирования. Штамм G18T был выделен из образца торфа (рН 7.6), отобранного из низинного болота Радионское (Вологодская обл., 59°47′08′′ N, 37°52′08′′ E) на глубине 5–10 см. Изолят был получен путем высева пробы водной суспензии торфа на среду DSM 629 следующего состава (г/л дистиллированной воды): пептон – 5, дрожжевой экстракт – 0.5, циклогексимид – 0.05, раствор солей Хатнера – 20 мл; раствор витаминов (Staley et al., 1992) – 1 мл; рН 7.0. В качестве желирующего агента использовали 1% раствор полисахарида микробного происхождения фитагеля (PhytaGel, “Fluka”). Бактериальный газон, образовавшийся на твердой среде через 2 нед., микроскопировали и регулярно рассевали методом истончающего штриха до получения изолированных колоний.
Идентификация микроорганизма. Выделение тотальной ДНК из клеток планктомицета производили с использованием набора FastDNA SPIN kit for soil (“Biol 101”, США) в соответствии с рекомендацией производителя. Полученную ДНК использовали в качестве матрицы в ПЦР-амплификации генов 16S рРНК со стандартными бактериальными праймерами 9F/1492R (Lane, 1991). Очистку ПЦР-продуктов осуществляли с использованием набора Wizard® SV Gel and PCR Clean-Up System (“Promega”, США). Секвенирование проводили на базе ЦКП “Биоинженерия” ФИЦ Биотехнологии РАН. Редактирование полученных нуклеотидных последовательностей проводили с помощью программы BioEdit. Построение филогенетических дендрограмм производили с использованием программного пакета MEGAX (Kumar et al., 2018) методом максимального правдоподобия. Статистическую достоверность дендрограмм рассчитывали с помощью “bootstrap”-анализа путем построения 100 альтернативных деревьев. Определенная в работе последовательность гена 16S рРНК планктомицета депонирована в GenBank под номером MW542577.
Геномное секвенирование и аннотирование. Выделение ДНК для геномного секвенирования осуществлялось по стандартному протоколу CTAB – фенол/хлороформ. Часть геномной ДНК секвенировали на ячейке R9.4 прибора MinION (“Oxford Nanopore”, Великобритания), используя Ligation Sequencing kit 1D, согласно рекомендациям производителя. Другая часть геномной ДНК была отсеквенирована на платформе Illumina MiSeq. Подготовка библиотеки и процедура секвенирования осуществлялась на базе фирмы “ReaGen” (Москва, Россия). Гибридная сборка прочтений Illumina и Nanopore была выполнена с использованием программы Unicycler (Wick et al., 2017) и BWA-MEM (Li, Durbin, 2009) с последующим сравнением качества в программах Quast (Gurevich et al., 2013) и Busco. Последовательность генома штамма G18T депонирована в GenBank под номером JAGKQQ000000000.
Аннотацию генома проводили с помощью программного пакета PROKKA (Seemann, 2014) и BLASTKoala (Kanehisa et al., 2016). Поиск вторичных метаболитов осуществляли в программе AntiSmash (Medema et al., 2011). Построение геномного дерева производили в программе GTDB-Tk, путем множественного выравнивания 120 маркерных генов. В анализ так же были взяты геномы других охарактеризованных представителей семейства Gemmataceae.
Оценка ростовых характеристик культуры планктомицета. Определение ростовых характеристик изолята проводили при культивировании в жидкой среде, указанной выше, во флаконах на качалке (120 об./мин) в диапазоне температур от 4 до 35°С, значений рН от 4.0 до 8.0 и концентрации NaCl в среде 0–3.0% . Окислительное и ферментативное использование углеводов определяли с помощью набора API 20NE kit (“bioMérieux”). Энзиматическую активность проверяли с использованем набора API ZYM (“bioMérieux”). В качестве ростовых субстратов тестировали сахара (глюкоза, сахароза, ксилоза, лактоза, манноза, раффиноза) и полимерные соединения (фитагель, крахмал, пектин, ксилан, ксантановая камедь, лихенан, хитин и микрокристаллическая целлюлоза (МКЦ)). Тестируемые субстраты вносили в концентрации 0.5 г/л. При этом в исходную среду № 629 не вносили пептон, а в качестве источника азота добавляли NH4NO3 в концентрации 0.1 г/л. Рост оценивали путем регулярных измерений оптической плотности культуры на спектрофотометре Eppendorf Biophotometer AG 22331 (Германия) при длине волны 600 нм. Перед измерением культуру активно встряхивали с целью ее гомогенизирования. Чувствительность штамма G18T к антибиотикам проверяли на твердой среде путем наложения на газоны культуры тест-дисков с различными антибиотиками и последующим измерением зон подавления роста.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Выделение, морфология и идентификация нового микроорганизма. Исследуемый штамм G18T был выделен из пробы водной суспензии торфа низинного болота на твердой среде № 629 с фитагелем. При культивировании на твердой среде изолят формировал небольшие отдельные колонии розового цвета. Микроскопический анализ показал, что колонии образованы шаровидными клетками размером 1.2–2.8 мкм (рис. 1а). В жидкой среде № 629 планктомицет рос в виде бледно-розового слизевого агрегата (рис. 1б). Микроорганизм размножался почкованием. Клетки обладали подвижностью только в экспоненциальной фазе роста. В культуре встречались как одиночные клетки, так и их скопления в виде бесформенных агрегатов.
Рис. 1.
а – Морфология клеток штамма G18T; фазовый контраст, маркер – 10 мкм; б – рост культуры в жидкой среде с формированием слизевого агрегата.

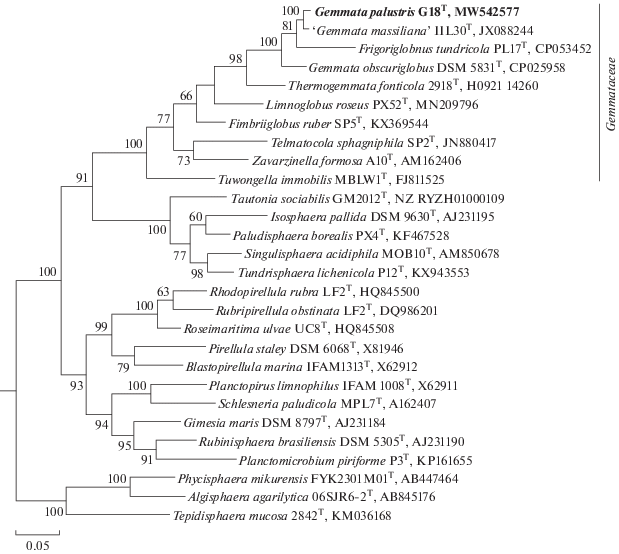
Идентификация и сравнительный анализ последовательности гена 16S рРНК штамма G18T выявил 98.9% сходства с геном невалидированного планктомицета рода Gemmata – “Gemmata massiliana” IIL30, выделенного из воды сети водоснабжения в госпитале во Франции (Aghnatios et al., 2015) и 95.7% сходства с пресноводным планктомицетом G. obscuriglobus DSM 5831T (Franzmann, Skerman, 1984) (рис. 2). Сходство 97.1% по генам 16S рРНК также было выявлено между штаммом G18T и представителем другого рода геммат – Frigoriglobus tundricola PL17T (Kulichevskaya et al., 2020a). Однако последующий филогеномный анализ позволил отнести новый изолят G18T к планктомицетам рода Gemmata.
Рис. 2.
Филогенетическая дендрограмма, построенная методом максимального подобия на основе сравнительного анализа нуклеотидных последовательностей гена 16S рРНК штамма G18T и других представителей филума Planctomycetes. В качестве внешней группы использованы нуклеотидные последовательности генов 16S рРНК пяти anammox-планктомицетов (AF375994, AF375995, AY254883, AY254882, AY257181). Показаны значения бутстрэп-анализа >60.
Физиологические характеристики. Штамм G18T рос в интервале pH среды от 6.0 до 8.0 (оптимум рН 7.0) и солености среды от 0 до 0.8% NaCl. Температурный диапазон роста планктомицета составил 4–28°С, с оптимумом при 15–20°С. Штамм не рос анаэробно в глубине столбика среды с фитагелем, что показывает, что он является строгим аэробом. Штамм обладал оксидазной активностью, каталазная и уреазная активности не были обнаружены.
Анализ ферментативных свойств штамма G18T, проведенный с использованием стандартного набора API ZYM (“bioMérieux”), показал наличие активности ряда ферментов, включая щелочную фосфатазу, эстеразу (С4), липазу (С8), лейцин-, валин-цистин-ариламидазу, трипсин, α-химотрипсин, кислую фосфатазу и нафтол-AS-BI-фосфогидролазу. Отрицательный результат был показан для липазы (C14), α- и β-галактозидазы, β-глюкуронидазы, α- и β-глюкозидазы, N-ацетил-β-глюкозаминидазы, α-маннозидазы и α-фукозидазы. По результатам тестов системы API 20NE новый планктомицет не способен к брожению, а также к восстановлению нитратов до нитритов и молекулярного азота.
Проверка спектра используемых субстратов показала, что предпочтительными источниками углерода и энергии являются полисахариды пектин, ксилан, фитагель, ксантантановая камедь, лихенан, а также пептон и дрожжевой экстракт. На сахарах лактозе, маннозе, сахарозе, ксилозе и раффинозе рост планктомицета был значительно слабее. Крахмал, хитин и МКЦ ингибировали рост изолята (табл. 1).
Таблица 1.
Отличительные признаки штамма G18Т и других видов рода Gemmata
| Признак | Штамм G18Т | G. obscuriglobus DSM 5831 Т | ‘G. massiliana’ IIL30 |
|---|---|---|---|
| Размер клеток, мкм | 1.2–2.8 | 1.4–3.0 | 1.1–2.1 |
| Гомогенный рост | – | + | ND |
| pH, диапазон (оптимум) | 6.0–8.0 (7.0) | 7.8–8.8 (ND) | 6.0–8.0 (8.0) |
| T, °C, диапазон (оптимум) | 4–28 (15–20) | 16–35 (ND) | 25–37 (30) |
| Солeность, NaCl% | 0–0.8 | 0–0.6 | 0–1.25 |
| Оксидаза | + | – | – |
| Каталаза | – | + | – |
| Устойчивость к антибиотикам: | |||
| линкомицин | S | R | ND |
| неомицин | S | R | ND |
| стрептомицин | S | R | ND |
| ампициллин | S | R | ND |
| рифампицин | S | R | S |
| Источники углерода: | |||
| сахароза | +/– | + | ND |
| ксилоза | +/– | + | ND |
| лактоза | +/– | + | ND |
| манноза | +/– | + | ND |
| раффиноза | +/– | – | ND |
| пектин | ++ | + | ND |
| крахмал | – | + | ND |
| Размер генома, ×106 п.о. | 9.23 | 9.06 | 9.25 |
| Г + Ц, мол. % | 65 | 67.4 | 64.1 |
| Количество CDS | 7631 | 7266 | 7985 |
| Кластеры вторичных метаболитов, шт. | 11 | 9 | 12 |
| Источник выделения | Торф низинного болота, Россия | Пресный водоем, Австралия | Вода из больничной сети водоснабжения Франция |
Проверка устойчивости штамма G18T к спектру антибиотиков выявила резистентные свойства к хлорамфениколу, олеандомицину, имипенему, цефотаксиму, амоксиклаву, фосфомицину и ванкомицину. Разница с другими представителями рода Gemmata была обнаружена по отношению к линкомицину, неомицину, стрептомицину, ампициллину и рифампицину. Штамм G18Т оказался чувствительным к вышеперечисленным антибиотикам, в то время как G. obscuriglobus DSM 5831Т проявляла резистентные свойства к данным веществам (табл. 1) (Ivanova et al., 2021).
Геномные характеристики. По итогам секвенирования на платформе Nanopore было получено 145 171 прочтений с общей длиной 1.4 × 109 п.о. Дополнительный раунд секвенирования на платформе Illumina MiSeq сгенерировал в общей сложности 3 061 576 парных прочтений со средней длиной считывания 150 п.о. Программой Unicycler удалось собрать последовательности с обеих платформ в 3 контига длинами 8 313 494, 905 837 и 8905 п.о. Другой подход, осуществленный с помощью ассемблера BWA-MEM, привел к сборке единственного кольцевого контига длиной 9 268 081 п.о. Однако сборка генома первым методом согласно программам Quast и Busco оказалась более качественной и была взята для дальнейшего анализа. Содержание G + C в геноме штамма G18T составило 65 мол. % (табл. 1). Геномным аннотатором Prokka было предсказано 7631 потенциальных белок-кодирующих последовательностей, 3 копии оперона рРНК и 97 генов тРНК.
ДНК–ДНК гибридизация выявила следующие сходства штамма G18Т с двумя представителями рода Gemmata: 31.2 ± 2.5% c ближайшим гомологом “G. massiliana” IIL30 и с 21.8 ± 2.2% G. obscuriglobus DSM 5831Т. Согласно калькулятору ANI (average nucleotide identity) сходство генома G18T с представителями рода Gemmata составляло: 86.0% (“G. massiliana” IIL30) и 79.7% (G. obscuriglobus DSM 5831Т). Согласно таксономическим стандартам такие показатели геномного анализа достаточны для отнесения изолята к новому виду (Chun et al., 2018). Проведенный филогеномный анализ показал, что штамм G18Т кластеризуется с другими представителями рода Gemmata в семействе Gemmataceae (рис. 3).
Рис. 3.
Филогеномная дендрограмма, основанная на анализе 120 конкатенированных последовательностей консервативных маркерных генов штамма G18T и других представителей филума Planctomycetes. В качестве внешней группы использованы геномы anammox-планктомицетов, доступные в базе данных GTDB. Показаны значения бутстрэп-анализа >60.

Гены, кодирующие основные метаболические пути хемоорганотрофных бактерий, такие как гликолиз, ЦТК, пентозо-фосфатный путь и окислительное фосфорилирование, присутствуют у штамма G18Т. Также у планктомицета имеется геномный потенциал для синтеза всех аминокислот. Большинство генов, ответственных за хемотаксис, включая cheB, cheR и cheW обнаружены в геноме G18Т. У других представителей рода Gemmata данные гены также присутствуют. Как и у всех планктомицетов, у новой гемматы G18Т не обнаружен ген FtsZ, ответственный за бинарное деление бактериальных клеток (Wiegand et al., 2018). Согласно недавнему исследованию для представителей семейства Gemmataceae характерны следующие гены, отвечающие за синтез пептидогликана: mraW, lysM и murB (Mahajan et al., 2020). У штамма G18Т они также были обнаружены.
Анализ вторичных метаболитов in situ. Планктомицеты обладают высоким потенциалом к синтезу вторичных метаболитов (Jeske et al., 2013). Недавно группой немецких ученых впервые было идентифицировано вещество стирелиацин, синтезируемое планктомицетом семейства Pirellulaceae, ответственное за формирование микроорганизмом биопленок (Kallscheuer et al., 2020).
В геноме штамма G18Т также был произведен поиск генных кластеров, необходимых для синтеза вторичных метаболитов. Вместе с исследуемым планктомицетом были проанализированы геномы ближайших родственных организмов – “G. massiliana” IIL30 и G. obscuriglobus DSM 5831Т. По итогам анализа в геноме изолята G18Т было выявлено 11 кластеров, потенциально кодирующих синтез вторичных метаболитов (табл. 1, рис. 4). Из них два кластера отвечают за синтез нерибосомных пептид-синтетаз (NRPS), которые, наравне с поликетидсинтазами (PKS), являются ключевыми ферментами синтеза биологически активных веществ (Donadio et al., 2007). Ближайшие гомологи двух NRPS, кодируемые в геноме штамма G18Т, были обнаружены у планктомицета “G. massiliana” IIL30 (21 и 57% сходства). Один генный кластер отвечал за синтез резорцинол-подобного вещества. Классический резорцинол используют в производстве красителей, обеззараживающих средств и противоопухолевой терапии. Производные данного вещества в основном синтезируется растениями (Miyanaga, Horinouchi, 2009). Ближайший родственник к данному генному кластеру (52% сходства) был найден у “G. massiliana” IIL30. Также обнаружен единичный кластер генов, кодирующих синтез поликетидсинтаз (PKS) третьего типа. Ближайший гомологичный белок был у планктомицета семейства Gemmataceae – Frigoriglobus tundricola PL17T (27% сходства) (Kulichevskaya et al., 2020a). Четыре кластера отвечали за производство терпенов – широкой группы органических веществ с антибиотическими, противоопухолевыми и гормональными свойствами (Yamada et al., 2012; Jeske et al., 2013). Гомологами в случае терпеновых кластеров являлись белки разных представителей семейства Gemmataceae co сходством от 64 до 89% (рис. 4). Три последних кластера (hgIE-KS и RiPP-like), также присутствующих в геноме G18T, относились к разным типам поликетидсинтаз, согласно классификации программы AntiSmash. Ближайшие родственники к ним были обнаружены у “G. massiliana” IIL30 и Gemmata sp. SH-PL17 со сходством от 71 до 81% (рис. 4).
Рис. 4.
Состав генных кластеров, отвечающих за синтез вторичных метаболитов, обнаруженных в геноме штамма G18T. Для каждого кластера приведены соответствующие ближайшие гены-гомологи других микроорганизмов.
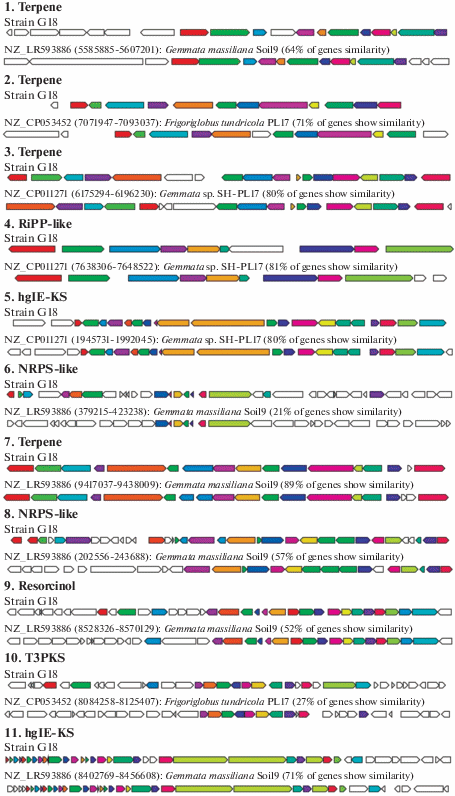
В геноме G. obscuriglobus DSM 5831Т было найдено 9 метаболических кластеров, в то время как у “G. massiliana” IIL30 обнаружили 12 кластеров (табл. 1). Ближайшие гомологи шести из 12-и кластеров, обнаруженных у “G. massiliana” IIL30, были выявлены в геноме штамма G18Т.
Проведенный сравнительный анализ морфологических, физиолого-биохимических и генетических характеристик нового изолята G18T с известными представителями рода Gemmata позволил выявить ряд существенных различий, приведенных в табл. 1. Филогенетически новый штамм наиболее близок к выделенному из активированного ила невалидированному планктомицету “Gemmata massiliana” IIL30, но, в отличие от последнего, имеет оптимум роста при 15–20°С и предпочитает более низкие значения pH. C G. obscuriglobus DSM 5831Т различия были обнаружены в спектре устойчивости к ряду антибиотиков, ростовых субстратов, оксидазной и каталазной активностям. Значения ДНК–ДНК гибридизации и ANI соответствуют стандартам таксономической классификации (Chun et al., 2018) для отнесения штамма G18T к новому виду рода Gemmata.
Диагноз нового вида – Gemmata palustris sp. nov.
Gemmata palustris sp. nov. (pa. lus´ tris. L. n. palus болото; M.L. adj. palustris обитающий в болотах). Шаровидные клетки диаметром 1.2–2.8 мкм, одиночные или объединенные в бесформенные скопления. Колонии розово-пигментированные. Дочерние клетки подвижные, материнские нет. В жидкой культуре рост наблюдался в виде слизевого агрегата. Облигатные аэробы. Психротолерантные мезофилы и нейтрофилы с оптимумом роста при 15–20°С и рН 7.0. Предпочтительные ростовые субстраты – полисахариды, в том числе пектин, ксилан, фитагель и ксантантановая камедь. Слабый рост на сахарозе, глюкозе, ксилозе, лактозе, раффинозе и маннозе. Оксидазо-положительный, каталаза- и уреаза-отрицательный. Обнаружены активности ряда ферментов: щелочной фосфатазы, эстеразы (С4), липазы (С8), лейцин-, валин-, цистин-ариламидазы, трипсина, α-химотрипсина, кислой фосфатазы и нафтол-AS-BI-фосфогидролазы. Отрицательный результат был показан для липазы (C14), α- и β-галактозидазы, β-глюкуронидазы, α- и β-глюкозидазы, N-ацетил-β-глюкозаминидазы, α-маннозидазы и α-фукозидазы. Размер генома штамма G18T составляет 9.23 × 106 п.о., содержание G + C в ДНК составляло 65 мол. %. Типовой штамм G18T нового таксона депонирован в двух международных коллекциях микроорганизмов – КСTC и ВКМ: под номерами КСTC 82682 и VKM B-3541T. Местообитания – низинные болота.
Список литературы
Куличевская И.С., Панкратов Т.А., Дедыш С.Н. Анализ бактериального сообщества, развивающегося при разложении сфагнума // Микробиология. 2007. Т. 76. С. 702–710.
Kulichevskaya I.S., Belova S.E., Kevbrin V.V., Dedysh S.N., Zavarzin G.A. Analysis of the bacterial community developing in the course of sphagnum moss decomposition // Microbiology (Moscow). 2007. V. 76. P. 621–629.
Aghnatios R., Cayrou C., Garibal M., Robert C., Azza S., Raoult D., Drancourt M. Draft genome of Gemmata massiliana sp. nov., a water-borne Planctomycetes species exhibiting two variants // Stand Genomic Sci. 2015. V. 10. Art. 120.
Chun J., Oren A., Ventosa A., Christensen H., Arahal D.R., da Costa M.S., Rooney A.P., Yi H., Xu X.-W., De Meyer S., Trujillo M.E. Proposed minimal standards for the use of genome data for the taxonomy of prokaryotes // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 461–466.
Dedysh S.N., Ivanova A.A. Planctomycetes in boreal and subarctic wetlands: diversity patterns and potential ecological functions // FEMS Microbiol. Ecol. 2019. V. 95. fiy227.
Donadio S., Monciardini P., Sosio M. Polyketide synthases and nonribosomal peptide synthetases: the emerging view from bacterial genomics // Nat. Prod. Rep. 2007. V. 24. P. 1073–1109.
Elcheninov A.G., Podosokorskaya O.A., Kovaleva O.L., Novikov A.A., Toshchakov S.V., Bonch-Osmolovskaya E.A., Kublanov I.V. Thermogemmata fonticola gen. nov., sp. nov., the first thermophilic planctomycete of the order Gemmatales from a Kamchatka hot spring // Syst. Appl. Microbiol. 2021. V. 44. Art. 126157.
Franzmann P.D., Skerman V.B. Gemmata obscuriglobus, a new genus and species of the budding bacteria // Antonie Van Leeuwenhoek. 1984. V. 50. P. 261–268.
Fuerst J.A., Gwilliam H.G., Lindsay M., Lichanska A., Belcher C., Vickers J.E., Hugenholtz P. Isolation and molecular identification of planctomycete bacteria from postlarvae of the giant tiger prawn, Penaeus monodon // Appl. Environ Microbiol. 1997. V. 63. P. 254–262.
Gurevich A., Saveliev V., Vyahhi N., Tesler G. QUAST: quality assessment tool for genome assemblies // Bioinformatics. 2013. V. 29. P. 1072–1075.
Ivanova A.A., Beletsky A.V., Rakitin A.L., Kadnikov V.V., Philippov D.A., Mardanov A.V., Ravin N.V., Dedysh S.N. Closely located but totally distinct. highly contrasting prokaryotic diversity patterns in raised bogs and eutrophic fens // Microorganisms. 2020. V. 8. Art. 484.
Ivanova A.A., Wegner C.-E., Kim Y., Liesack W., Dedysh S.N. Metatranscriptomics reveals the hydrolytic potential of peat-inhabiting Planctomycetes // Antonie Van Leeuwenhoek. 2018. V. 111. P. 801–809.
Ivanova A.A., Miroshnikov K.K., Oshkin I.Y. Exploring antibiotic susceptibility, resistome and mobilome structure of planctomycetes from Gemmataceae family // Sustainability. 2021. V. 13. Art. 5031.
Ivanova A.O., Dedysh S.N. Abundance, diversity, and depth distribution of Planctomycetes in acidic northern wetlands // Front. Microbiol. 2012. V. 3. Art. 5.
Jeske O., Jogler M., Petersen J., Sikorski J., Jogler C. From genome mining to phenotypic microarrays. Planctomycetes as source for novel bioactive molecules // Antonie Van Leeuwenhoek. 2013. V. 104. P. 551–567.
Kallscheuer N., Jeske O., Sandargo B., Boedeker C., Wiegand S., Bartling P., Jogler M., Rohde M., Petersen J., Medema M.H., Surup F., Jogler C. The planctomycete Stieleria maiorica Mal15T employs stieleriacines to alter the species composition in marine biofilms // Commun. Biol. 2020. V. 3. Art. 303.
Kanehisa M., Sato Y., Morishima K. BlastKOALA and GhostKOALA. KEGG tools for functional characterization of genome and metagenome sequences // J. Mol. Biol. 2016. V. 428. P. 726–731.
Kulichevskaya I.S., Baulina O.I., Bodelier P.L.E., Rijpstra W.I.C., Sinninghe Damsté J.S., Dedysh S.N. Zavarzinella formosa gen. nov., sp. nov., a novel stalked, Gemmata-like planctomycete from a Siberian peat bog // Int. J. Syst. Evol. Microbiol. 2009. V. 59. P. 357–364.
Kulichevskaya I.S., Ivanova A.A., Baulina O.I., Rijpstra W.I.C., Sinninghe Damsté J.S., Dedysh S.N. Fimbriiglobus ruber gen. nov., sp. nov., a Gemmata-like planctomycete from Sphagnum peat bog and the proposal of Gemmataceae fam. nov. // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 218–224.
Kulichevskaya I.S., Ivanova A.A., Naumoff D.G., Beletsky A.V., Rijpstra W.I.C., Damste J.S.S., Mardanov A.V., Ravin N.V., Dedysh S.N. Frigoriglobus tundricola gen. nov., sp. nov., a psychrotolerant cellulolytic planctomycete of the family Gemmataceae from a littoraltundra wetland // Syst. Appl. Microbiol. 2020. V. 43. Art. 126129.
Kulichevskaya I.S., Naumoff D.G., Miroshnikov K.K., Ivanova A.A., Philippov D.A., Hakobyan A., Rijpstra W.I.C., Sinninghe Damsté J.S., Liesack W., Dedysh S.N. Limnoglobus roseus gen. nov., sp. nov., a novel freshwater planctomycete with a giant genome from the family Gemmataceae // Int. J. Syst. Evol. Microbiol. 2020. V. 70. P. 1240–1249.
Kulichevskaya I.S., Serkebaeva Y.M., Kim Y., Rijpstra W.I.C., Damsté J.S.S., Liesack W., Dedysh S.N. Telmatocola sphagniphila gen. nov., sp. nov., a novel dendriform planctomycete from northern wetlands // Front. Microbiol. 2012. V. 3. Art. 146.
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X. Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evol. 2018. V. 35. P. 1547–1549.
Lane D.J. 16S/23S rRNA sequencing // Nucleic Acid Techniques in Bacterial Systematics / Ed. Stackebrandt E., Goodfellow M. N.Y.: John Wiley & Sons, 1991. P. 115–175.
Li H., Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform // Bioinformatics. 2009. V. 25. P. 1754–1760.
Mahajan M., Seeger C., Yee B., Andersson S.G.E. Evolutionary remodeling of the cell envelope in Bacteria of the Planctomycetes phylum // Genome Biol. Evol. V. 12. P. 1528–1548.
Medema M.H., Blin K., Cimermancic P., De Jager V., Zakrzewski P., Fischbach M.A., Weber T., Takano E., Breitling R. AntiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences // Nucleic Acids Res. 2011. V. 39. P. W339–W346.
Miyanaga A., Horinouchi S. Enzymatic synthesis of bis-5-alkylresorcinols by resorcinol-producing type III polyketide synthases // J. Antibiot. (Tokyo). 2009. V. 62. P. 371–376.
Ravin N.V., Rakitin A.L., Ivanova A.A., Beletsky A.V., Kulichevskaya I.S., Mardanov A.V., Dedysh S.N. Genome analysis of Fimbriiglobus ruber SP5T, a planctomycete with confirmed chitinolytic capability // Appl. Environ. Microbiol. 2018. V. 84. https://doi.org/10.1128/AEM.02645-17
Seeger C., Butler M.K., Yee B., Mahajan M., Fuerst J.A., Andersson S.G.E. Tuwongella immobilis gen. nov., sp. nov., a novel non-motile bacterium within the phylum Planctomycetes // Int. J. Syst. Evol. Microbiol. 2017. V. 67. P. 4923–4929.
Simão F.A., Waterhouse R.M., Ioannidis P., Kriventseva E.V., Zdobnov E.M. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs // Bioinformatics. 2015. V. 31. P. 3210–3212.
Staley J.T., Fuerst J.A., Giovannoni S., Schlesner H. The order Planctomycetales and the genera Planctomyces, Pirellula, Gemmata, and Isosphaera // The Prokaryotes: A Handbook on the Biology of Bacteria: Ecophysiology, Isolation, Identification, Applications / Ed. Balows A., Trüper H.G., Dworkin M., Harder W., Schleifer K.-H. N.Y.: Springer, 1992. P. 3710–3731.
Wick R.R., Judd L.M., Gorrie C.L., Holt K.E. Unicycler: resolving bacterial genome assemblies from short and long sequencing reads // PLoS Comput. Biol. 2017. V. 13. e1005595.
Wiegand S., Jogler M., Jogler C. On the maverick Planctomycetes // FEMS Microbiol. Rev. 2018. V. 42. P. 739–760.
Yamada Y., Cane D.E., Ikeda H. Diversity and analysis of bacterial terpene synthases // Methods in Enzymology, 1st ed. / Ed. Hopwood D.A. Academic Press, 2012. P. 123–162.
Дополнительные материалы отсутствуют.


