Микробиология, 2022, T. 91, № 2, стр. 191-203
Анализ геномов двух бактериобионтов лишайников, Lichenibacterium ramalinae и Lichenibacterium minor: токсин‒антитоксин системы и белки секреции
Т. А. Панкратов a, *, П. А. Никитин b, Е. О. Патутина c
a Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b Факультет биоинженерии и биоинформатики, МГУ им. М.В. Ломоносова
119192 Москва, Россия
c Институт биоинженерии им. К.Г. Скрябина, ФИЦ Биотехнологии РАН
119071 Москва, Россия
* E-mail: tpankratov@gmail.com
Поступила в редакцию 31.10.2021
После доработки 15.11.2021
Принята к публикации 15.11.2021
- EDN: NBEXBM
- DOI: 10.31857/S0026365622020112
Аннотация
Проведен анализ геномов двух бактериобионтов кустистого эпифитного лишайника Ramalina pollinaria – Lichenibacterium ramalinae и L. minor. Выявлены генетические детерминанты, потенциально определяющие интеграцию и адаптацию этих бактерий в талломах лишайников. Впервые проведена оценка генных детерминант факторов реакции на стресс и систем секреции лишайниковых бактериобионтов. Гены, кодирующие белки токсин‒антитоксин (ТА) систем VapCB, имели уровень гомологии >60% с генами известных симбионтов растений – Bradyrhizobium, Sinorhizobium, Agrobacterium, Mesorhizobium и Ralstonia, а также Bartonella – патогеном человека. Гены, кодирующие белки системы секреции II типа, найдены в геномах обоих видов. Гены, кодирующие белки секреции VI типа, обнаружены только в геноме L. ramalinae, при этом они гомологичны таковым у эпифитного Methylobacterium, патогена растений Agrobacterium и корневых симбионтов растений Rhizobium и Neorhizobium. Степень гомологии генов, кодирующих белки ТА и белков систем секреции, с генами бактерий, ассоциированных с растениями, составляет более 60%. Это позволяет предположить, что основной мишенью инвазии могут быть зеленые водоросли. Обнаружение в геномах лишайниковых бактериобионтов генов синтеза уреаз позволило сформулировать гипотезу, согласно которой разложение мочевины приводит к дополнительному снабжению компонентов симбиоза аммонием и гидрокарбонатом. Последний может быть потенциально использован фототрофными эукариотами и прокариотами как источник дополнительного углерода. Анализ геномов бактериобионтов лишайников L. ramalinae и L. minor показал, что их стратегии выживания могут различаться: L. ramalinae теснее интегрирован в симбиоз, в то время как L. minor характеризуется большим количеством черт автономности.
Микробные сообщества лишайников активно исследуются на протяжении последних 20 лет. Произошел существенный сдвиг в восприятии лишайников как комплексных систем. Доказано присутствие в них бактерий (Панкратов и соавт., 2017), формирующих микроколонии среди гиф микобионта и клеток фотобионта (водорослей), то есть во внутренней среде таллома. Эти бактерии не относятся к филуму Cyanobacteria и найдены в водорослевых лишайниках. Методами флуоресцентной микроскопии с мечеными зондами, денатурирующего градиентного гель-электрофореза, а также методом молекулярного клонирования было показано доминирование в лишайниковых талломах представителей класса Alphaproteobacteria (Cardinale et al., 2008). Позднее (Erlacher et al., 2015) показано доминирование представителей порядков Hyphomicrobiales (бывший Rhizobiales) и Rhodospirillales. Была обнаружена специфическая для лишайников группа клонов LAR1 (Lichen Associated Rhizobiales) (Hodkinson, Lutzoni, 2009), которые формировали изолированный филогенетический кластер в порядке Hyphomicrobiales. Недавно из антарктического лишайника Psoroma sp. был выделен и охарактеризован представитель нового семейства Lichenihabitantaceae, Lichenihabitans psoromatis, филогенетически близкий клонам группы LAR1 (Noh et al., 2019). Одновременно из лишайника Ramalina pollinaria был описан и второй представитель кластера LAR1 – Lichenibacterium, с двумя видами L. ramalinae и L. minor, внутри нового семейства Lichenibacteriaceae (Pankratov et al., 2020). Представители этого кластера найдены также и в ряде образцов, не связанных с лишайниковыми симбиозами (Li et al., 2012). Стабильное обнаружение бактерий рода Lichenibacterium в лишайниках различных видов, например, Cladonia (Панкратов, неопубл. данные), позволило предположить, что бактерии этого рода специфически приурочены к лишайниковым симбиозам и представляют интерес как группа автохтонных для лишайников бактерий.
Роль бактерий в лишайниковых симбиозах постепенно становится более понятной благодаря выделению чистых культур и анализу их физиологических характеристик (Grube et al., 2009), в результате анализа метагеномных данных (Wicaksono et al., 2020) и данных транскрипционного анализа (Nurhani et al., 2013). Наиболее часто обсуждаемыми и частично доказанными являются данные о фиксации молекулярного азота, антибиотической и солюбилизирующей активности бактериобионтов лишайников (Gonzáles et al., 2005; Grube, Berg, 2009), а также о синтезе гормонов и факторов защиты от токсических соединений (Erlacher et al., 2015).
Другой важный аспект ‒ интеграция и селекция бактерий в лишайниковый симбиоз. В этой связи возникают вопросы: 1) как бактерии избегают отторжения со стороны микобионта и фотобионта; почему только некоторые виды способны к интеграции в лишайниковый таллом; 2) как протекает процесс селекции. Механизмы и факторы, обеспечивающие селекцию бактериальных эндобионтов лишайников, а также их адаптацию в симбиозе, пока совсем не исследованы (неизвестны). В этой связи, поиск генетических детерминант адаптации и инвазии в бактериях, которые существуют в глубоких слоях таллома и специфически приурочены именно к лишайникам, выглядит логичным и обоснованным.
В число факторов, которые могут оказаться значимыми для формирования и поддержания симбиотических отношений бактерий и микобионта (фотобионта), следует включить: белки систем нодуляции (играющие ключевую роль во взаимодействии ризобиальных бактерий с корнями растений); белки систем мобильности (флагеллины и пилины); белки адгезии (адгезины и пилины); системы транспорта (белки секреции и киназы); токсин‒антитоксин системы; сигнальные белки или их продукты; белки секреции сидерофоров; разнообразные факторы вирулентности. В данной статье обсуждается поиск и филогеномный анализ генов, кодирующих токсин−антитоксин системы, белки секреции и уреазу.
Белки секреции присутствуют у большинства бактерий, взаимодействующих с хозяйскими организмами, и некоторые из них, наряду с пилинами, являются частью систем, отвечающих за вирулентность и паразитизм (Depluverez et al., 2016; Green, Mecsas, 2021). Если белки секреции I, II и III типов широко распространены среди прокариот и характеризуются большим функциональным спектром, то типы IV, V и VI имеют ограниченное распространение среди бактерий. В частности, V тип ‒ это, в основном, системы секреции патогенных и паразитических бактерий, а IV и VI могут иметь как бактерии-патогены человека и животных (например, Pseudomonas aeruginosa, Vibrio cholerae и Serratia marcescens), так и патогены растений (Agrobacterium tumefaciens) (Green, Mecsas, 2021).
Токсин–антитоксин системы присутствуют практически во всех микроорганизмах и локализованы на плазмидах или в хромосоме. Их основная функция – регуляция процессов деления, программируемая гибель клетки и другие процессы, связанные с контролем популяционной плотности (Hayes, 2003). Однако в последнее время появляются данные о том, что ТА системы могут участвовать в контроле вирулентности при патогенезе растений (Shavit et al., 2016).
Впервые роль уреазы, как фактора вирулентности, была установлена в работе Сох и соавт. при изучении экспериментального криптококкоза (Cox et al., 2000). При этом уровень активности этого фермента мог быть видовым и/или специфичным для конкретного вида и/или места инфекции. Известно также, что механизм транспорта и гидролиза мочевины лежит в основе вирулентности у Helicobacter pylori. Процесс осуществляется благодаря механизму снижения рН периплазмы H. pylori, в результате которого активируется канал мочевины с протонным захватом (UreI), импортирующим мочевину в бактериальный цитозоль, где она гидролизуется уреазой. Далее аммиак и бикарбонат используются для буферизации протонов внутри периплазмы до значений рН, приемлемых для выживания бактерии (Ansari, Yamaoka, 2016).
Целью данной работы было выявление и сравнительный анализ в геномах L. ramalinae и L. minor генетических детерминант, влияющих на межбактериальную коммуникацию и их взаимодействие с эукариотическими компонентами лишайникового симбиоза: уреаза, токсин–антитоксин системы и комплекс белков секреции.
ОБЪЕКТЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Объекты исследования. Штаммы RmlP001T и RmlP026T рода Lichenibacterium выделены ранее (Pankratov et al., 2020) из отмытых от эпифитной микрофлоры гомогенатов лишайника Ramalina pollinaria и охарактеризованы как два новых вида – L. ramalinae и L. minor.
Анализ геномов. Аннотированные в GenBank геномные сборки (L. ramalinae, GCA_004137085; L. minor, GCA_004137685) анализировали с использованием программ Unipro UGENE 38.1 (Okonechnikov et al., 2012) и BIOEDIT v. 7.0.5.3 (Hall, 1999). Выравнивание амнокислотных и нуклеотидных последовательностей проводили с использованием алгоритма MUSCLE в программе MEGA X (Kumar et al., 2018). Филогененетический анализ и построение деревьев осуществляли с использованием программ MEGA X, Unipro UGENE 38.1, сервиса IQ Tree (http://iqtree.cibiv.univie.ac.at/) (Trifinopoulos et al., 2016) и FigTree 1.4.4 с использованием конкатенированных аминокислотных последовательностей целевых белков. Для визуализации геномных кластеров и оперонов пользовались пакетами DNA Features Viewer (Zulkower, Rosser, 2020) и matplotlib (Hunter, 2007) языка программирования Python3 (Van Rossum, Drake, 2009). Для поиска ортологичных генов использовали базу данных COG (https://www.ncbi.nlm.nih.gov/research/ cog) (Tatusov et al., 1997).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Общая характеристика геномов. Оба генома имели средние значения величины – около 6 Мб (табл. 1). Плазмидные последовательности не обнаружены. У типового вида L. ramalinae размер генома оказался меньше, чем у L. minor.
Таблица 1.
Характеристики геномов L. ramalinae и L. minor
| Показатель | L. ramalinae | L. minor |
|---|---|---|
| Размер (Mб) | 5.8 | 5.92 |
| Г + Ц, % | 70.8 | 70.5 |
| Белки | 5299 | 5493 |
| рРНК | 3 | 3 |
| тРНК | 50 | 55 |
| нкРНК | 4 | 4 |
| Геномное покрытие | 32× | 78× |
| Гены (общее кол-во) | 5442 | 5674 |
| Кодирующие регионы | 5385 | 5612 |
| Кодирующие гены | 5299 | 5493 |
| Гены РНК | 57 | 62 |
| Псевдогены (без белков) | 86 | 119 |
Размер геномов не предполагает их облигатно паразитический стиль жизни, так как у большинства истинных паразитов, например, микоплазмы, бореллии или хламидии, он не превышает 2 Мб. Факультативные симбионты Hamiltonella defensa и Regiella insecticola имеют размеры геномов немногим более 2 Мб, а факультативный симбионт мухи цеце Sodalis glossinidius – 4.2 Мб (Clark et al., 2010). Вместе с тем, геномы обоих штаммов Lichenibacterium меньше, чем геномы некоторых ризобиальных симбионтов и свободноживущих ризобий. Так, например, у различных видов Rhizobium размер генома варьирует в пределах от 4.98 Мб у Rhizobium rhizoryzae DSM 29514 до 7.5 Мб у Rhizobium sophorae BCCM/LMG 27901. У Rhizobium leguminosarum размер генома может достигать, в зависимости от штамма, 8 Мб.
Количество кодирующих генов у L. minor на 194 больше, чем у L. ramalinae. 976 белков L. ramalinae и 1164 белков L. minor не были аннотированы и имеют статус гипотетического белка. Виды различаются также количеством транспортных РНК – у L. ramalinae их выявлено 50, а у L. minor – 55, что пропорционально разнице в размерах геномов.
Токсин–антитоксин системы (ТА). Среди генов, которые участвуют в формировании токсинов и факторов их инактивации, выделяются белки VapCB, относящиеся к классу тРНК-специфичных эндонуклеаз (Fraikin et al., 2020). В геноме L. ramalinae найдено семь паралогичных последовательностей гена, кодирующего белок VapC, т.е. собственно токсин. У L. minor их четыре. Один из паралогов этого гена у L. minor оказался в одном кластере с генами симбиотических бактерий родов Bradyrhizobium и Rhizobium, ассоциированных с корнями растений, а также оппортунистического патогена человека Brucella anthropi (рис. 1). Большинство паралогов этого гена у L. ramalinae кластеризуются в группах генов свободноживущих бактерий. И только один из них оказались в кластерах, сформированных генами ризобиальных бактерий (рис. 2). Филогеномный анализ выявил родство генов, кодирующих VapC белки, как генам бактерий – симбионтов растений, так и свободноживущих бактерий, выделенных из почв и иных субстратов.
Рис. 1.
Эволюционный анализ филогении гена vарС Lichenibacterium minor методом максимального правдоподобия. Эволюционная история была определена с помощью метода максимального правдоподобия и модели Jones et al. w/freq. Исходные деревья для эвристического поиска были получены автоматически путем применения алгоритмов Neighbor-Join и BioNJ к матрице парных расстояний, оцененных с помощью модели JTT, и последующего выбора топологии с лучшим значением log-вероятности.
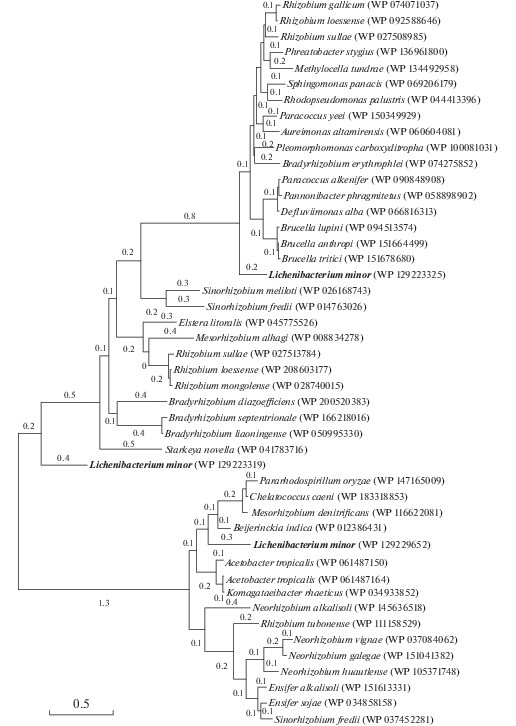
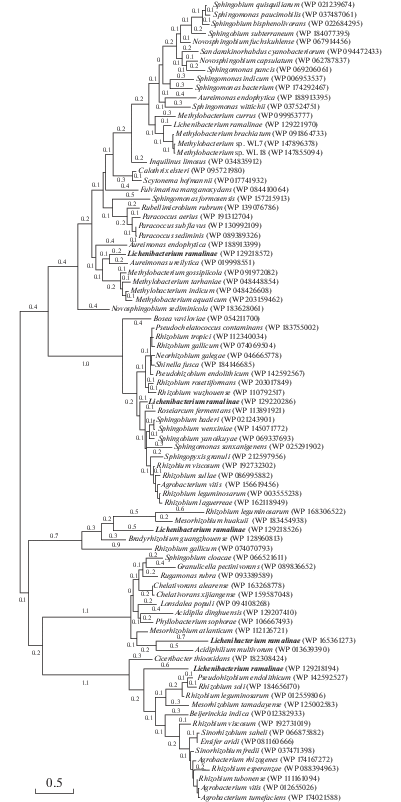
Рис. 2.
Эволюционный анализ филогении гена vарС Lichenibacterium ramalinae методом максимального правдоподобия. Показано дерево с наибольшим логарифмическим правдоподобием (‒12422.15). Исходные деревья для эвристического поиска были получены автоматически путем применения алгоритмов Neighbor-Join и BioNJ к матрице парных расстояний, оцененных с помощью модели JTT, и последующего выбора топологии с наибольшим значением логарифмического правдоподобия. Дерево нарисовано в масштабе, длина ветвей измеряется в количестве замен на сайт. В данном анализе участвовали 87 аминокислотных последовательностей. Всего в окончательном наборе данных было 172 позиции.
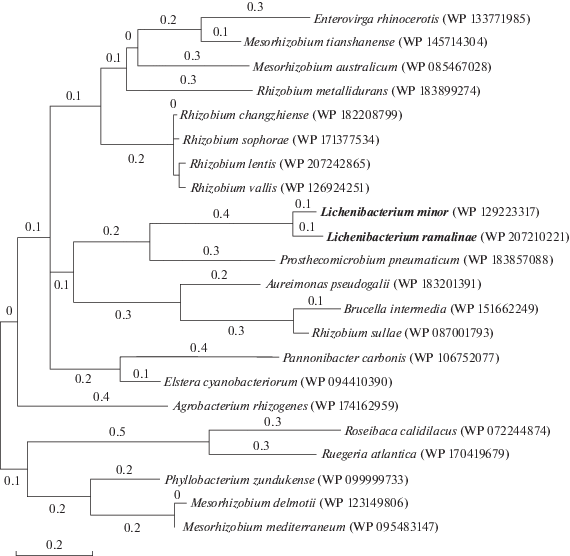
Ген, кодирующий белок VapB (антитоксин) в геноме L. ramalinae представлен двумя паралогами, а у L. minor – только одним геном. Эволюционный анализ этого гена в обеих культурах показал его родство как со свободноживущим Prosthecomicrobium pneumaticum, так и с паразитической Brucella intermedia и симбионтом Rhizobium sullae (рис. 3).
Рис. 3.
Эволюционный анализ филогении гена vapB у L. ramalinae и L. minor методом максимального правдоподобия. Эволюционная история была определена с помощью метода максимального правдоподобия и модели Jones et al. w/freq. Дерево нарисовано в масштабе, длина ветвей измеряется в количестве замен на сайт. В данном анализе участвовали 22 аминокислотные последовательности. Всего в окончательном наборе данных было 66 позиций.
Ранее у патогена растений Acidovorax citrulli показана активация оперона VapCB системы во время инфекционного процесса (Shavit et al., 2016). Было установлено, что экспрессия vapB отстает от экспрессии токсина на несколько суток. В отличие от патогенов животных, большинство патогенных бактерий растений, включая A. citrulli, колонизируют внеклеточные пространства внутри растительной ткани (Alfano, Collmer, 1996). Инвазия таких бактерий соответствует взаимодействию с растительной тканью, а не внутриклеточному поведению. Аналогичная ситуация возможна и для Lichenibacterium, когда факторы VapCB участвуют в активной колонизации лишайника. Тем более что данные, полученные Shavit et al. (2016) подтвердили, что в процессе колонизации проростков после заражения культурой A. citrulli численность клеток вырастала на три порядка, что указывало на отсутствие остановки деления клеток во время экспрессии vapC. Таким образом, белки VapBC не останавливали рост бактериальной популяции, а наоборот, способствовали более активной колонизации.
В геномах обоих штаммов найден антитоксин Phd. Этот белок имеет различный уровень гомологии у двух видов Lichenibacterium. Если ген антитоксинового белка L. minor кластеризуется в группу, представленную Azospirillum, Segnochrobacter, Methylorubrum и наиболее близок к Nitrobacter, то в случае L. ramalinae он имеет высокий уровень гомологии с геном паразитического Ensifer adhaerens, а также симбионта Aureimonas endophytica (рис. 4). В этом же кластере находится Sphingomonas, который, как было показано ранее, является одним из наиболее распространенных в лишайниках представителей альфапротеобактерий (Jiang et al., 2017). Ген токсина doc найден только у L. ramalinae. Система ТА Phd/Doc ранее была обнаружена в геноме патогенной для растений бактерии Pseudomonas syringae (Guérout et al., 2013).
Рис. 4.
Эволюционный анализ филогении гена phd Lichenibacterium ramalinae и L. minor методом максимального правдоподобия. Эволюционная история была определена с помощью метода максимального правдоподобия и модели Jones et al. w/freq. Исходные деревья для эвристического поиска были получены автоматически путем применения алгоритмов Neighbor-Join и BioNJ к матрице парных расстояний, оцененных с помощью модели JTT, и последующего выбора топологии с лучшим значением log-вероятности.
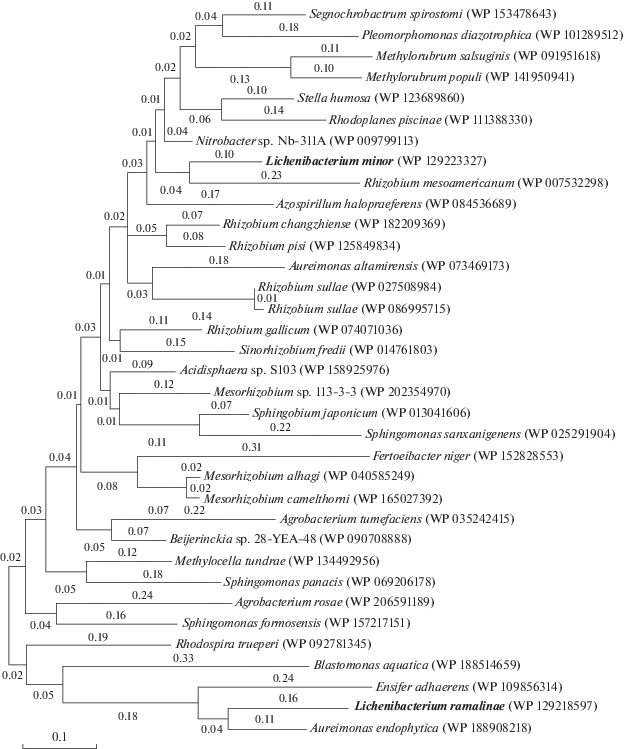
Интересно, что токсин HicA, ген которого также обнаружен в геномах обеих бактерий, у L. ramalinae не имеет антитоксина HicB. Можно предположить, что продукт трансляции гена hicA с помощью систем секреции выбрасывается за пределы клетки, где его мишенями могут являться РНК других микроорганизмов, в отношении которых может проявляться его специфичность. В то же время, hicAB присутствуют в хромосоме L. minor, у которого нет системы секреции VI типа.
Ранее для Burkholderia pseudomallei было показано влияние сверхэкспрессии токсина HicA на остановку роста и увеличение количества персистирующих клеток, устойчивых к ципрофлоксацину или цефтазидиму (Butt et al., 2014). Со стимулированием персистенции и формированием биопленочного фенотипа связывают и активность других ТА систем, например, RelBE и ParDE, которые также обнаружены в геномах обеих культур. Кроме указанных, в геномах обоих штаммов присутствуют ТА гены pemK/mazF, brnT/brnA, регулирующие ответ на стресс у ряда патогенных бактерий, например, Brucella abortus (Heaton et al., 2012). Таким образом, наличие токсин‒антитоксин систем разного типа может свидетельствовать как о наличии устойчивости к стрессовым факторам хозяйского организма (антибиотики, лишайниковые кислоты, пептиды и т.д.), так и возможном участии исследуемых бактерий в формировании бактериобиома лишайника посредством контроля численности и активности собственных и чужеродных бактериальных популяций. Разнообразие ТА систем у Lichenibacterium может свидетельствовать о физиологической и экологической пластичности этого рода бактериобионтов лишайников.
Белки секреции I, II, IV, VI типов. Геномы L. ramalinae и L. minor включают в себя гены, кодирующие белки секреции I, II, IV и VI типов. Причем гены белков секреции первого типа представлены только ортологами hlyD. Они, по всей вероятности, являются уникальными, так как для большинства генов в нуклеотидных базах данных не удалось найти близкие гены, удовлетворяющие условиям гомологии. Исключение составляют лишь некоторые белки. Например, белок HlyD (NCBI no WP_165361460) имеет сходство с ортологами из бактерий рода Methylobacterium (выделены с листьев пшеницы), на уровне 74% (при 100% покрытии) и на уровне 61–63% ‒ с ортологами из симбиотических Mesorhizobium и Bradyrhizobium (также при 100% покрытии).
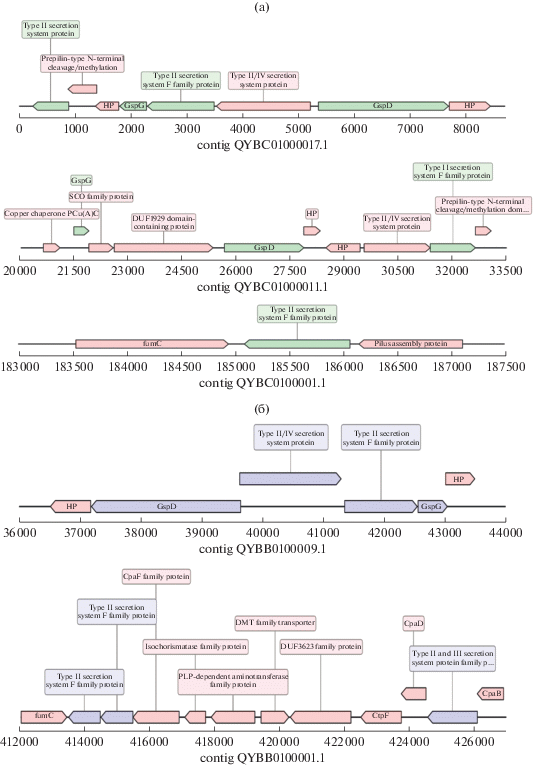
Гены белков секреции второго типа представлены в геноме L. ramalinae 11 генами, из которых 5 объединены в одну кассету (рис. 5а), а остальные располагаются в различных участках генома. Часть из них представлена паралогичными генами псевдопилинов/секретинов gspDG. У L. minor гены, кодирующие белки секреции II типа, расположены в двух кластерах (рис. 5б), в которых также присутствуют гены сборки пилинов и псевдопилинов. Системы секреции II типа являются наиболее распространенными, и обеспечивают транспорт разнообразных классов веществ через наружную мембрану грамотрицательных бактерий (Korotkov et al., 2012). Известно (Ursula et al., 2017), что система секреции II типа (T2SS) вносит свой вклад в жизнеспособность патогенной бактерии Acinetobacter baumannii in vivo. В частности, была показана роль T2SS в защите A. baumannii от человеческого комплемента. Мутанты с делецией гена T2SS ∆gspD были в 100 раз менее жизнеспособны при инкубации с человеческой сывороткой. Возможно, что у Lichenibacterium может функционировать сходный механизм защиты от хозяйского организма (микобионта).
Рис. 5.
Организация генов белков секреции II типа в геноме L. ramalinae (a) и L. minor (б). HP – гипотетический белок.
Среди белков, секретируемых растительными патогенами и патогенами человека в процессе заражения, присутствуют в основном ферменты протеазы, липазы, хитиназы, а также эндотоксины (Sandkvist, 2001). Ранее при изучении метаболизма обоих штаммов Lichenibacterium мы обнаружили у L. ramalinae способность использовать пектин и ламинарин в качестве источников углерода (Pankratov et al., 2020). В то же время хитинолитическая и целлюлозолитическая активности не были обнаружены. Эти данные могут также указывать на то, что Lichenibacterium в лишайниковом симбиозе ассоциирован не с микобионтом, а с фотобионтом (ламинарин и пектины – полисахариды растительного происхождения).
Cреди генов гликозилгидролаз, присутствующих в геноме L. ramalinae, найдены представители пяти семейств (1, 15, 16, 37 и 127), а в геноме L. minor – четырех семейств (1, 15, 16, 127). Семейство 16 – это гликозидгидролазы, субстратами которых являются полимеры, содержащие β‑1,4- или β-1,3-гликозидные связи, 1,3-β-галактаны, 1,3-β-глюканы, 1,3(4)-β-глюканы, лихенаны, β-агарозы, β-порфираны and κ-каррагинаны. Некоторые из этих полисахаридов являются компонентами клеточных стенок грибов и водорослей. Ранее (Mathews et al., 2019) было показано, что гены гликозидгидролаз необходимы для факторов вирулентности Agrobacterium tumefaciens на Bryophyllum daigremontiana и Solanum lycopersicum. В работе d’Enfert и соавт. (1987) была впервые показана роль второй системы секреции во внеклеточном транспорте пуллуланазы. Вероятно, что системы секреции II типа у Lichenibacterium вносят свой вклад в транспорт гликозилгидролаз, участвующих в ассоциации с микобионтом или фотобионтом.
Если гены синтеза белков секреции II типа характерны для обоих видов Lichenibacterium, то гены синтеза белков VI типа (T6SS) найдены только у L. ramalinae, что указывает на функциональные различия двух видов. Пластичность кластера T6SS предполагает, что эволюционно эта система могла быть приобретена L. ramalinae путем горизонтального переноса (Navarro-Garcia et al., 2019). Известно, что T6SS грамотрицательных бактерий представляют собой устройства эффекторной транслокации, напоминающие перевернутое устройство для прокалывания у бактериофагов, состоящее, по крайней мере, из 13 белков, называемых ядерными компонентами. Бактерии используют быстрое сокращение длинной оболочки секреторной системы типа VI (T6SS) для доставки эффекторов в клетку-мишень. Они осуществляют широкий набор функций, включая вирулентность и антибактериальную активность (Pukatzki et al., 2006, 2009; Sana et al., 2012), а также являются компонентом систем межбактериальной коммуникации (Gallique et al., 2017). В геноме L. ramalinae гены этой системы организованы в два оперона, первый из которых включает гены белков сборки канала секреции ‒ гены, кодирующие белки базальной пластинки tssEFGK, наконечник Hcp и VgrG – гемолизин-корегулируемый белок (белок прокалывания клетки) (рис. 6a). Второй содержит гены, кодирующие белки мембранного комплекса (рис. 6б).
Рис. 6.
Положение генов, кодирующие белки секреции VI типа в геноме L. ramalinae: a – оперон белков сборки канала секреции; б – гены, кодирующие мембранные белки секреции VI типа. HP – гипотетический белок.

Ранее было показано, что система секреции T6SS используется бактериями для атаки на другие бактерии (Basler et al., 2013). Нельзя исключить, что бактерии L. ramalinae могут участвовать в атаках на грамотрицательные бактерии, поддерживая таким способом стабильность лишайникового бактериобиома. Ближайшими гомологами белков Hcp и VgrG являются белки эпифитных Methylobacterium и корневых симбионтов растений Rhizobium, Agrobacterium, Neorhizobium (>72% идентичности). Уровень сходства гомологичных генов, кодирующих белки базальной пластинки, оказался меньше, и составил около 60–62% с ортологами из эпифитных Methylobacterium. Можно предположить, что разные гены, кодирующие белки комплекса T6SS у L. ramalinae были приобретены независимо, путем горизонтального переноса.
Уреаза. Обе культуры Lichenibacterium имеют все гены для экспрессии белков, участвующих в гидролизе мочевины, включая системы транспорта мочевины в клетку. В геномах также найдены системы транспорта никеля – кофактора уреаз (HoxN/HupN/NixA, белок бисинтеза кофактора никель-пинчера LarB и белок вставки никеля LarC).
Вероятно, что присутствие уреазы у этих бактерий обусловлено необходимостью адаптации к кислой среде, характерной для всех кладониевых лишайников и рамалины (рН водных эктрактов талломов от 3.5 до 4.5 в апикальной части таллома (Pankratov, 2012). Следствием активности уреазы может быть понижение рН микроокружения, увеличение пула доступного иона аммония, а также формирование дополнительного бикарбоната, который может быть использован как самими бактериями, так и водорослями в цикле Кальвина. На фоне данных, указывающих на ассоциацию Lichenibacterium с водорослями, предположение об участии уреаз в стимулировании фототрофии у водорослей может быть достойным последующей экспериментальной проверки.
Таким образом, анализ геномов двух бактериобионтов лишайников Lichenibacterium ramalinae и L. minor показал наличие генетических детерминант, экспрессия которых может обусловить направленную инвазию этих бактерий в лишайниковый симбиоз. Наличие ТА систем двух типов предполагает возможность вовлечения этих бактерий в процессы регуляции состава и численности микроокружения во внутренней среде таллома лишайника, а также регулировки численности собственной популяции в ответ на стресс. Четыре системы секреции потенциально способны обеспечивать многоуровневую коммуникацию в микробных ценозах лишайников с микроорганизмами-соседями и способствовать формированию биопленочных фенотипов Lichenibacterium. Отсутствие системы секреции III типа предполагает, что эти бактерии не имеют механизмов, обеспечивающих инвазию в клетки эукариот, следовательно, паразитизм на эукариотах можно исключить. Подтверждением этому является спектр потребляемых субстратов, в который, прежде всего, входят полиолы и сахара (Pankratov et al., 2020), что характерно для свободноживущих бактерий. Вероятнее всего, мутуализм является основной стратегией Lichenibacterium в симбиозе с лишайником. Позитивная роль Lichenibacterium может заключаться в контроле численности нежелательных бактерий и создании микросреды, в которой происходит обогащение продуктами распада мочевины – гидрокарбоната и аммония. Наличие разнообразных систем секреции, ТА систем и уреазы позволяет этим бактериям проявлять экологическую и физиологическую пластичность в разных микроусловиях, возникающих при формировании и дальнейшем росте таллома лишайника.
Два вида этого рода отличаются по набору генов, обеспечивающих обсуждаемые функции. Для L. minor характерно отсутствие системы секреции VI типа, но при этом более активно выражены процессы образования каротиноидов – β-каротина и ликопина (Pankratov et al., 2020). Кроме того, в геноме L. minor обнаружены гены, отвечающие за синтез бактериохлорофилла (2-винил бактериохлорофиллид гидратаза, ферредоксин:протохлорофиллид редуктаза, хлорофилл синтаза ChlG и другие) и белков фотосистемы II, что предполагает, при наличии возможной фотозависимой фиксации СО2, автономное существование этого вида в лишайниковом симбиозе. У L. ramalinae есть все системы для проявления антибактериальных свойств, что подразумевает более активное участие этих бактерий в регуляции состава бактериобиома лишайников.
Итак, анализ геномов бактериобионтов лишайников L. ramalinae и L. minor показал, что стратегии выживания двух видов могут отличаться. L. ramalinae приобретает черты более глубоко интегрированного в лишайниковый симбиоз организма, а L. minor, приобретя, по-видимому, путем горизонтального переноса фотосистему II и гены синтеза бактериохлорофилла, стремится к автономии, но сохраняет способность противостоять атакам со стороны бактериального микроокружения и эукариот.
Номера доступа DDBJ/ENA/GenBank для последовательностей, созданных в этом исследовании, следующие: QYBC000000 (геном штамма RmlP001T) и QYBB000000 (геном штамма R-mlP026T).
Пользовательские скрипты, использованные в данном исследовании для визуализации кластеров генов, были размещены на Github (https:// github.com/nikitin-p/Lichenibacterium_analysis).
Список литературы
Alfano J.R., Collmer C. Bacterial pathogens in plants: life up against the wall // Plant Cell. 1996. V. 8. P. 1683–1698. https://doi.org/10.1105/tpc.8.10.1683
Ansari S., Yamaoka Y. Survival of Helicobacter pylori in gastric acidic territory // Helicobacter. 2017. V. 22. Art. 10.1111/ hel.12386. https://doi.org/10.1111/hel.12386
Basler M., Ho B.T., Mekalanos J.J. Tit-for-tat: type VI secretion system counterattack during bacterial cell‒cell interactions // Cell. 2013. V. 152. P. 884‒894. https://doi.org/10.1016/j.cell.2013.01.042
Butt A., Higman V.A., Williams C., Crump M.P., Hemsley C.M., Harmer N., Titball R.W. The HicA toxin from Burkholderia pseudomalleihas a role in persister cell formation // Biochem. J. 2014. V. 459. P. 333–344. https://doi.org/10.1042/BJ20140073333
Cardinale M., de Castro J.V., Mueller H., Berg G., Grube M. In situ analysis of the bacterial community associated with the reindeer lichen Cladonia arbuscula reveals predominance of Alphaproteobacteria // FEMS Microbiol. Ecol. 2008. V. 66. P. 63–71. https://doi.org/10.1111/j.1574-6941.2008.00546.x
Clark E.L., Karley A.J., Hubbard S.F. Insect endosymbionts: manipulators of insect herbivore trophic interactions? // Protoplasma. 2010. V. 244. P. 25‒51. https://doi.org/10.1007/s00709-010-0156-2
Cox G.M., Mukherjee J., Cole G.T., Casadevall A., Perfect J.R. Urease as a virulence factor in experimental cryptococco-sis // Infect Immun. 2000. V. 68. P. 443‒448. https://doi.org/10.1128/IAI.68.2.443-448.2000
Depluverez S., Devos S., Devreese B. The role of bacterial secretion systems in the virulence of gram-negative airway pathogens associated with cystic fibrosis // Front. Microbiol. 2016. V. 7. Art. 1336. https://doi.org/10.3389/fmicb.2016.01336
d’Enfert C., Ryter A., Pugsley A.P. Cloning and expression in Escherichia coli of the Klebsiella pneumoniae genes for production, surface localization and secretion of the lipoprotein pullulanase // EMBO J. 1987. V. 6. P. 3531–3538.
Erlacher A., Cernava T., Cardinale M., Soh J., Sensen C.W., Grube M., Berg G. Rhizobiales as functional and endosymbiotic members in the lichen symbiosis of Lobaria pulmonaria L. // Front. Microbiol. 2015. V. 6. Art. 53. https://doi.org/10.3389/fmicb.2015.00053
Fraikin N., Goormaghtigh F., Van Melderen L. Type II toxin-antitoxin systems: evolution and revolutions // J. Bacteriol. 2020. V. 202. e00763-19. https://doi.org/10.1128/JB.00763-19
Gallique M., Bouteiller M., Merieau A. The type VI secretion system: a dynamic system for bacterial communication? // Front. Microbiol. 2017. V. 28. Art. 1454. https://doi.org/10.3389/fmicb.2017.01454
Gonzáles I., Ayuso-Sacido A., Anderson A., Genilloud O. Actinomycetes isolated from lichens: evaluation of their diversity and detection of biosynthetic gene sequences // FEMS Microbiol. Ecol. 2005. V. 54. P. 401–415. https://doi.org/10.1016/j.femsec.2005.05.004
Green E.R., Mecsas J. Bacterial secretion systems: an overview // Microbiol. Spectr. 2021. V. 4. https://doi.org/10.1128/microbiolspec.VMBF-0012-2015
Grube M., Berg G. Microbial consortia of bacteria and fungi with focus on the lichen symbiosis // Fungal Biol. Rev. 2009. V. 23. P. 72–85. https://doi.org/10.1016/j.fbr.2009.10.001
Grube M., Cardinale M., de Castro J., Müller H., Berg G. Species-specific structural and functional diversity of bacterial communities in lichen symbioses // ISME J. 2009. V. 3. P. 1105–1115. https://doi.org/10.1038/ismej.2009.63
Guérout A.-M., Iqbal N., Mine N., Ducos-Galand M., Van Melderen L., Mazel D. Characterization of the phd-doc and ccd toxin‒antitoxin cassettes from Vibrio superintegrons // J. Bacteriol. 2013. V. 195. P. 2270‒2283.
Hall T.A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Acids Symp. 1999. V. 41. P. 95‒98.
Hayes F. Toxins‒antitoxins: plasmid maintenance, programmed cell death, and cell cycle arrest // Science. 2003. V. 301. P. 1496–1499. https://doi.org/10.1126/science.1088157
Heaton B.E., Herrou J., Blackwell A.E., Wysocki V.H., Crosson S. Molecular structure and function of the novel B-rnT/BrnA toxin‒antitoxin system of Brucella abortus // J. Biol. Chem. 2012. V. 287. P. 12098‒12110. https://doi.org/10.1074/jbc.M111.332163
Hodkinson B.P., Lutzoni F. A microbiotic survey of lichen-associated bacteria reveals a new lineage from the Rhizobiales // Symbiosis. 2009. V. 49. P. 163–180. https://doi.org/10.1007/s13199-009-0049-3
Hunter J.D. Matplotlib: A 2D graphics environment // Comput. Sci. Eng. 2007. V. 9. P. 90–95. https://doi.org/10.1109/mcse.2007.55
Jiang D.-F., Wang H.-Y., Si H.-L., Zhao L., Liu C.-P., Zhang H. Isolation and culture of lichen bacteriobionts // The Lichenologist. 2017. V. 49. P. 175–181. https://doi.org/10.1017/S0024282917000044
Korotkov K., Sandkvist M., Hol W. The type II secretion system: biogenesis, molecular architecture and mechanism // Nat. Rev. Microbiol. 2012. V. 10. P. 336–351. https://doi.org/10.1038/nrmicro2762
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evolut. 2018. V. 35. P. 1547‒1549. https://doi.org/10.1093/molbev/msy096
Li E., Hamm C.M., Gulati A.S., Sartor R.B., Chen H., Wu X., Zhang T., Rohlf F.J., Zhu W., Gu C., Robertson C.E., Pace N.R., Boedeker E.C., Harpaz N., Yuan J., Weinstock G.M., Sodergren E., Frank D.N. Inflammatory Bowel diseases phenotype, C. difficile and NOD2 genotype are associated with shifts in human ileum associated microbial composition // PLoS One. 2012. V. 7. e26284. https://doi.org/10.1371/journal.pone.0026284
Mathews S.L., Hannah H., Samagaio H., Martin C., Rodriguez-Rassi E., Matthysse A.G. Glycoside hydrolase genes are required for virulence of Agrobacterium tumefaciens on Bryophyllum daigremontiana and tomato // Appl. Environ. Microbiol. 2019. V. 85. e00603-19. https://doi.org/10.1128/AEM.00603-19
Navarro-Garcia F., Ruiz-Perez F., Cataldi Á., Larzábal M. Type VI secretion system in pathogenic Escherichia coli: structure, role in virulence, and acquisition // Front. Microbiol. 2019. V. 10. Art. 01965. https://doi.org/10.3389/fmicb.2019.01965
Noh H.J., Baek K., Hwang C.Y., Shin S.C., Hong S.G., Lee Y.M. Lichenihabitans psoromatis gen. nov., sp. nov., a member of a novel lineage (Lichenihabitantaceae fam. nov.) within the order of Rhizobiales isolated from Antarctic lichen // Int. J. Syst. Evol. Microbiol. 2019. V. 69. P. 3837‒3842. https://doi.org/10.1099/ijsem.0.003695
Nurhani A.R.S., Abdul Munir A.M., Mohd Wahid S., Farah Diba A.B. A preliminary transcriptomic analysis of lichen Dirinaria sp. // AIP Conference Proceedings. 2013. V. 1571. P. 258. https://doi.org/10.1063/1.4858665
Okonechnikov K., Golosova O., Fursov M., the UGENE team. Unipro UGENE: a unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. P. 1166–1167. https://doi.org/10.1093/bioinformatics/bts091
Pankratov T.A., Grouzdev D.S., Patutina E.O., Kolganova T.V., Suzina N.E., Berestovskaya J.J. Lichenibacterium ramalinae gen. nov, sp. nov., Lichenibacterium minor sp. nov., the first endophytic, beta-carotene producing bacterial representatives from lichen thalli and the proposal of the new family Lichenibacteriaceae within the order Rhizobiales // Antonie van Leeuwenhoek. 2020. V. 113. P. 477‒489. https://doi.org/10.1007/s10482-019-01357-6
Pankratov T.A., Kachalkin A.V., Korchikov E.S., Dobrovol’skaya T.G. Microbial communities of lichens // Microbiology (Moscow). 2017. V. 86. P. 293–309. https://doi.org/10.1134/S0026261717030134
Pukatzki S., Ma A.T., Sturtevant D., Krastins B., Sarracino D., Nelson W.C., Heidelberg J.F., Mekalanos J.J. Identification of a conserved bacterial protein secretion system in Vibrio cholerae using the Dictyostelium host model system // Proc. Natl. Acad. Sci. USA. 2006. V. 103. P. 1528–1533. https://doi.org/10.1073/pnas.0510322103
Pukatzki S., McAuley S.B., Miyata S.T. The type VI secretion system: translocation of effectors and effector-domains // Curr. Opin. Microbiol. 2009. V. 12. P. 11–17. https://doi.org/10.1016/j.mib.2008.11.010
Sana T.G., Hachani A., Bucior I., Soscia C., Garvis S., Termine E., Engel J., Filloux A., Bleves S. The second type VI secretion system of Pseudomonas aeruginosa strain PAO1 is regulated by quorum sensing and fur and modulates internalization in epithelial cells // J. Biol. Chem. 2012. V. 287. P. 27095–27105. https://doi.org/10.1074/jbc.M112.376368
Sandkvist M. Type II secretion and pathogenesis // Infect. Immun. 2001. V. 69. P. 3523‒3535. https://doi.org/10.1128/IAI.69.6.3523-3535.2001
Shavit R., Lebendiker M., Pasternak Z., Burdman S., Helman Y. The vapB–vapC operon of Acidovorax citrulli functions as a Bona-fide toxin–antitoxin module // Front. Microbiol. 2016. V. 6. Art. 1499. https://doi.org/10.3389/fmicb.2015.01499
Tatusov R.L., Koonin E.V., Lipman D.J. A genomic perspective on protein families // Science. 1997. V. 278. P. 631‒637. https://doi.org/10.1126/science.278.5338.631
Trifinopoulos J., Nguyen L.T., von Haeseler A., Minh B.Q. W-IQ-TREE: a fast online phylogenetic tool for maximum likelihood analysis // Nucl. Acids Res. 2016. V. 44. P. 232‒235. https://doi.org/10.1093/nar/gkw256
Ursula W., Johnson T.L., Chedid Kh., Xi Ch., Simmons L.A., Mobley H.L.T., Sandkvist M. Targeting the type II secretion system: development, optimization, and validation of a high-throughput screen for the identification of small molecule inhibitors // Front. Cell. Infect. Microbiol. 2017. V. 7. Art. 380. https://doi.org/10.3389/fcimb.2017.00380
Van Rossum G., Drake F.L. Python 3 Reference Manual. . Scotts Valley, CA: CreateSpace. 2009.
Wicaksono W.A., Cernava T., Grube M., Berg G. Assembly of bacterial genomes from the metagenomes of three lichen species // Microbiol. Resour. Announc. 2020. V. 9. e00622-20. https://doi.org/10.1128/MRA.00622-20
Zulkower V., Rosser S. DNA Features Viewer, a sequence annotations formatting and plotting library for Python // Bioinformatics. 2020. V. 36. P. 4350–4352. https://doi.org/10.1093/bioinformatics/btaa213
Дополнительные материалы отсутствуют.


