Микробиология, 2022, T. 91, № 3, стр. 285-299
Геномные и физиологические характеристики галофильных бактерий родов Halomonas и Marinobacter из нефтяных пластов
Т. П. Турова a, Д. Ш. Соколова a, Е. М. Семенова a, А. П. Ершов a, Д. С. Груздев b, Т. Н. Назина a, *
a Институт микробиологии им. С.Н. Виноградского, ФИЦ Биотехнологии РАН
119071 Москва, Россия
b SciBear OU
10115 Tallinn, Estonia
* E-mail: nazina@inmi.ru
Поступила в редакцию 25.01.2022
После доработки 27.01.2022
Принята к публикации 27.01.2022
- EDN: CQWXRI
- DOI: 10.31857/S0026365622300036
Аннотация
При эксплуатации нефтяных пластов с высокоминерализованными водами, отделенная от нефти вода закачивается обратно в пласты, что в ряде случаев приводит к увеличению солености среды и ограничивает возможность применения биотехнологий повышения нефтеизвлечения. Микробное сообщество таких пластов сравнительно мало изучено. Два штамма галофильных углеводородокисляющих бактерий Halomonas titanicae TAT1 и Marinobacter lutaoensis KAZ22 выделены из нефтяных пластов с минерализованной пластовой водой, определены их физиологические и геномные характеристики, детерминирующие их присутствие в этих местообитаниях, и возможность биотехнологического применения. Штаммы H. titanicae TAT1 и M. lutaoensis KAZ22 растут аэробно на сырой нефти в интервале температуры от 4 до 42 и от 22 до 55°С соответственно и имеют оптимумы роста при солености 2–12 и 5–10% NaCl (вес/об.). Феноменологические наблюдения деградации н-алканов нефти подтверждены обнаружением трех генов (аlkB1, аlkB2 и almA), кодирующих алкан-1-монооксигеназы, в геноме штамма KAZ22, и одного гена alkB в геноме штамма TAT1. В геноме штамма H. titanicae TAT1 аннотированы все гены образования осмопротекторов бетаина, эктоина и гидроксиэктоина, а у штамма M. lutaoensis KAZ22 – гены образования эктоина и гидроксиэктоина. Рост штамма KAZ22 на нефти сопровождается снижением поверхностного натяжения среды более чем на 20 мН/м. Вследствие отсутствия гена нитритредуктазы nirK анаэробный рост штамма H. titanicae ТАТ1 в среде с ацетатом и нитратом приводит к накоплению около 100 мг нитрита/л, который может подавлять рост сульфидогенов. Биологические особенности штаммов и анализ геномов позволяют рекомендовать нагнетание нитрата и штамма H. titanicae ТАТ1 для биоаугментации нефтяных резервуаров с минерализованной пластовой водой для подавления коррозии и снижения содержания сульфида, а M. lutaoensis KAZ22 – для очистки от нефтяных загрязнений и увеличения нефтеотдачи пластов.
Галофильные органотрофные гаммапротеобактерии родов Halomonas и Marinobacter часто встречаются в составе микробных сообществ местообитаний с высокой соленостью, включая морские водоемы, глубинные подземные горизонты, гидротермальные венты, нефтяные пласты, сточные воды нефтедобывающих предприятий, загрязненные нефтью почвы и поверхностные соленые озера (Huu et al., 1999; Mnif et al., 2009; Kaye et al., 2010; Al-Mailem et al., 2010; Fathepure, 2014; Luo et al., 2015). Широкоераспространение этих бактерий свидетельствует о потенциальном вкладе в биогеохимические циклы элементов в высокосоленых местообитаниях. Особый интерес представляют галофильные углеводородокисляющие бактерии, обитающие в нефтяных пластах. Пластовая вода многих нефтяных месторождений имеет высокую соленость, что осложняет нефтедобычу, очистку и утилизацию нефтезагрязненных вод. Применение микробных технологий на таких месторождениях ограничено вследствие негативного воздействия соли на микроорганизмы, включая разрушение клеточной стенки, денатурацию ферментов, а также низкой растворимости кислорода в этих условиях (Fathepure, 2014). Положительное воздействие микроорганизмов на вытеснение нефти обусловлено жизнедеятельностью углеводород-окисляющих бактерий, рост которых на нефти всегда сопровождается образованием поверхностно-активных веществ (Беляев и соавт., 2004). Некоторые углеводородокисляющие бактерии образуют не только био-ПАВ, но и экзополисахариды, что может быть использовано также в биотехнологиях повышения нефтеотдачи.
Из нефтяных пластов с высокоминерализованными водами были выделены галофильные и галотолерантные бактерии родов Halomonas, M-arinobacter, Bacillus (Huu et al., 1999; Ben Ali Gam et al., 2007; Mnif et al., 2009; Fathepure, 2014; An et al., 2017) и археи Haloferax, Halorubrum (Halobacterium), Halococcus (Звягинцева и соавт., 1995), использующие углеводороды нефти. Бактерии росли в интервале температуры, рН и солености, характерных для нефтяного пласта; использовали широкий спектр углеводородов, жирных кислот, спиртов и сырую нефть. Сообщалось о выделении галотолерантных углеводородокисляющих бактерий родов Rhodococcus, Gordonia, Dietzia и Pseudomonas из нефти и пластовых вод месторождений Татарстана, Западной Сибири и Вьетнама (Борзенков и соавт., 2006).
Хотя бактерии родов Halomonas и Marinobacter имеют похожие физиологические характеристики и используют широкий круг субстратов и сырую нефть, в нефтяных пластах обычно преобладают бактерии одного из родов. Например, на высокотемпературном нефтяном месторождении Узень, заводняемом морской водой, бактерии рода Ma-rinobacter были минорными компонентами микробных сообществ пластовой воды и становились доминирующей группой в посевах проб воды на среды с нитратом и органическими субстратами (Sokolova et al., 2021). В обработанном нитратом нефтяном пласте Северного моря наряду с доминирующими нитратредуцирующими бактериями рода Terasakiella присутствовали бактерии рода Marinobacter, способные расти на спиртах и жирных кислотах, восстанавливая нитраты (Bødtker et al., 2009). Галофильные/галотолерантные бактерии родов Halomonas, Marinobacter, Halanaerobium, Modicisalibacter и Chromohalobacter входили в состав культур денитрифицирующих бактерий, растущих при высокой солености среды (2.5 M NaCl), полученных из пластовой воды месторождения в Канаде (Bakken field, An et al., 2017). Внесение нитрата в хемостат с культурой сульфатредуцирующих бактерий из пластовой воды этого месторождения сопровождалось восстановлением нитрата до нитрита бактериями рода Halomonas, что приводило к подавлению роста сульфидогенов.
Из нефтяных месторождений в Тунисе выделен штамм Halomonas sp. C2SS100 и галофильная бактерия Modicisalibacter tunisiensis LIT2T семейства Halomonadaceae, которые росли в среде с нефтью в присутствии 0–14 и 1–25% NaCl (вес/об.) соответственно и имели оптимумы температуры при 37°С (Ben Ali Gam et al., 2007; Mnif et al., 2009). Из пластовой воды нефтяного месторождения во Вьетнаме выделена галотолерантная углеводородокисляющая бактерия Marinobacter aquaeolei (позднее отнесенная к виду Marinobacter hydrocarbonoclasticus и затем к Marinobacter nauticus), которая росла с среде, содержащей от 0 до 20% NaCl (оптимум 5% NaCl) при температуре от 13 до 50°С (оптимум 30°С) (Huu et al., 1999).
Таким образом, бактерии родов Halomonas и Marinobacter в аэробных условиях могут расти на сырой нефти с образованием биоПАВ, а в анаэробных условиях расти гетеротрофно на органических субстратах, восстанавливая нитрат до нитрита или молекулярного азота. Какие биологические особенности позволяют им населять нефтяные пласты с минерализованными водами и разной температурой, и как эти бактерии могут быть использованы в биотехнологиях очистки от нефтяных загрязнений, увеличения нефтеотдачи или подавления коррозии, представляет научный и практический интерес.
В настоящее время известны четыре системы монооксигеназ, позволяющие микроорганизмам использовать н-алканы нефти: трансмембранные алкан-гидроксилазы (монооксигеназы), AlkB, флавин-зависимые монооксигеназы, окисляющие длинноцепочечные алканы, AlmA и LadA, а также цитоплазматические цитохром Р450-монооксигеназы. Некоторые бактерии, выделенные из нефтезагрязненных вод, в частности, Amycolicicoccus subflavus DQS3-9A1T, содержат в геноме все известные системы алкан-гидроксилаз (Nie et al., 2013).
Из пластовой воды Ромашкинского нефтяного месторождения (Россия) и из нефтяного месторождения Узень (Республика Казахстан) нами были выделены два штамма ‒ Halomonas titanicae TAT1 и Marinobacter lutaoensis KAZ22 соответственно.
Целью настоящего исследования было изучение физиологии и геномных детерминант деградации н-алканов и устойчивости к повышенной солености у галофильных бактерий родов Halomonas и Marinobacter, выделенных из нефтяных пластов.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Бактериальные штаммы. В работе использовали бактериальные штаммы Halomonas titanicae TAT1 (VKM B-3500D) и Marinobacter lutaoensis KAZ22 (VKM B-3501D), выделенные ранее из проб пластовой воды залежи 302 Ромашкинского нефтяного месторождения (Республика Татарстан, Россия) и нефтяного месторождения Узень (Республика Казахстан) соответственно (Grouzdev et al., 2020). Характеристика проб пластовой воды, используемой для выделения штаммов, приведена в табл. S1 (дополнительные материалы). Нефтяные пласты залежи 302 располагаются на глубине 493–515 м ниже уровня моря, имеют температуру 17–23°С. Пластовая вода имеет общую соленость от 17.0 до 23.4 г/л. Вода содержит сульфаты и сульфид (от 150 до 290 мг/л); рН варьирует от 6.8 до 7.9 (Nazina et al., 2013). Для поддержания пластового давления в залежь нагнетают пресную речную воду, смешанную с пластовой водой. Нефтяные пласты месторождения Узень залегают на глубине 1080–1370 м, имеют температуру около 57–68°С, заводняются сточной нефтепромысловой водой, смешанной с водой Каспийского моря. Штамм KAZ22 выделен из пробы воды, изливающейся из призабойной зоны нагнетательной скважины 2755, где температура пласта снижалась до 40–45°С; пластовая вода относилась к хлоркальциевому типу, ее минерализация составляла 51.4 г/л, pH 6.6 (Sokolova et al., 2021).
Состав сред и условия культивирования бактерий. Бактерии культивировали на минеральной среде, содержащей (г/л): K2HPO4 – 1.5; KH2PO4 – 0.75; NH4Cl – 1.0; MgSO4 ∙ 7H2O – 0.1; CaCl2 ∙ 2H2O – 0.02; KCl – 0.1; дрожжевой экстракт – 0.5; NaCl – 100.0; раствор микроэлементов; pH 6.8–7.2. В качестве источника углерода и энергии в среду вносили сырую тяжелую нефть (1%, об./об.) плотностью 0.932 г/cм3, содержащую 23.5% насыщенных углеводородов, 42.3% ароматических углеводородов, 30.1% смол и 4.1% асфальтенов. Бактерии культивировали на качалке (110 об./мин) при температуре 28°C, если не указано иное. Спектр используемых субстратов определяли в минеральной среде того же состава; сахара, пептон, триптон и дрожжевой экстракт добавляли в концентрации 0.5% (вес/об.), спирты, соли органических кислот – 0.2% (вес/об.), аминокислоты – 0.1–0.2% (вес/об.). Контролем служила инокулированная среда без субстрата. Анаэробный рост исследовали, используя среду с ацетатом в качестве источника углерода и энергии, и нитратом натрия (0.85 г/л) в качестве акцептора электронов. Оптимум солености и диапазоны роста определяли на указанной выше среде, содержащей от 0 до 20% NaCl (вес/об.). Интервал температуры для роста определяли в среде, оптимальной по солености, после 7 сут инкубации. Рост оценивали по изменению величины оптической плотности среды (OD660) и микроскопированием посевов. С использованием наборов API 50CH и API ZYM (“Biomerieux”, Франция) анализировали спектр используемых субстратов и ферментативную активность штаммов.
Определение использования н-алканов нефти. Биодеградацию нефти оценивали после инкубации штаммов в указанной выше минеральной среде, содержащей 0.5 г/л нефти и 0.05 г/л дрожжевого экстракта, после 7 сут инкубации при 37°С. Изменение содержания н-алканов в деградированной нефти определяли газохроматографическим методом по сравнению с контролем (в %), как описано ранее (Борзенков и соавт., 2006).
Методы молекулярной идентификации штаммов и детекции генов алкан-монооксигеназ. ДНК выделяли из биомассы штаммов с использованием набора реактивов “DIAtomTMDNA Prep100” (“Биоком”, Россия) согласно рекомендациям фирмы изготовителя. Очищенный препарат ДНК использовали в качестве матрицы для проведения ПЦР. Чистые культуры идентифицировали методом анализа гена 16S рРНК с использованием универсальных праймеров (Lane, 1991). Гены алкан-монооксигеназ (alkB, ladAα, ladAβ, ladB, cyp153A) амплифицировали с использованием праймеров, приведенных в табл. S2 (дополнительные материалы). Секвенирование ДНК проводили по методу Сэнгера на автоматическом секвенаторе ABI 3730 DNA Analyzer, с использованием набора реактивов ABI PRISM BigDyeTM Terminator v 3.1 (“Applied Biosystems”, США). При детекции генов алкангидроксилаз использовали штаммы из коллекции лаборатории нефтяной микробиологии ФИЦ Биотехнологии РАН, а также штамм Geobacillus icigianus G1w1T (=VKM B-2853T = DSM 28325T), предоставленный сотрудниками Института цитологии и генетики СО РАН (Bryanskaya et al., 2015).
Секвенирование и анализ геномов. Штамм KAZ22 выращивали аэробно в среде LB с 5% (вес/об.) NaCl при температуре 40°С, клетки собирали центрифугированием через 7 сут культивирования. ДНК очищали от клеточной биомассы с помощью цетилтриметиламмоний бромида (Wilson, 2001). Библиотеки ДНК были подготовлены с помощью набора реагентов (NEBNext DNA library prep для Illumina, “New England Biolabs”). Секвенирование геномной ДНК проводили на HiSeq 2500 (“Illumina Inc.”, США) в формате парно-концевых прочтений 2 × × 150 п.н. Качество последовательностей проверяли с помощью FastQC v. 11.7 (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/). Последовательности были обрезаны с помощью Trimmomatic v. 0.36 (Bolger et al., 2014). Отфильтрованные по качеству риды были de novo собраны с помощью SPAdes v. 3.13.0 с использованием настроек по умолчанию (Bankevich et al., 2012). Полноту и контаминацию геномов оценивали с использованием программы CheckM v1.0.18 (Parks et al., 2015). Собранные геномы анализировали с помощью GTDB-Tk 1.0.2 для определения филогенетически близких таксонов (Chaumeil et al., 2019). Среднюю нуклеотидную идентичность (ANI) оценивали с помощью FastANI v. 1.3 (Jain et al., 2018). Цифровую ДНК‒ДНК-гибридизацию (dDDH) геномов проводили с использованием Genome-to-Genome Distance Calculator (GGDC) v. 2.1 (Meier-Kolthoff et al., 2013). Идентификацию белок-кодирующих последовательностей и первичную аннотацию геномов проводили с помощью NCBI Prokaryotic Genome Automatic Annotation Pipeline (PGAP) (Tatusova et al., 2013).
Геном штамма TAT1 был секвенирован и расшифрован ранее, как описано в статье (Grouzdev et al., 2020).
Биоинформатический анализ. Реконструкцию возможных путей метаболизма алканов и осмопротекторов проводили на основании сравнения геномов исследуемых штаммов с использованием онлайн-сервисов NCBI (https://www.ncbi.nlm.nih.gov/ genome/), PATRIC (https://www.patricbrc.org) и KEGG (https://www.genome.jp/kegg/pathway.html). Сравнение кластеров генов деградации алканов и синтеза осмопротекторов проводили с использованием онлайн-сервиса Gene Graphics (https:// katlabs.cc/genegraphics/app).
Hуклеотидные последовательности гена 16S рРНК штаммов H. titanicae TAT1 и M. lutaoensis KAZ22 были помещены в GenBank под номерами MK478935 и MT277027, геномы депонированы в DDBJ/EMBL/GenBank под номерами JABWTB010000000 и JABWTC000000000 соответственно.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Физиологические характеристики выделенных галотолерантных бактерий и деградация нефти. Выделенные штаммы TAT1 и KAZ22 были идентифицированы на основании BLAST-анализа генов 16S рРНК с использованием базы данных GenBank. В результате анализа был обнаружен 100% уровень идентичности гена 16S рРНК штамма TAT1 с соответствующим геном типового штамма Halomonas titanicae ВН1T (АС NR_117300), а штамма KAZ22 – с типовым штаммом Marinobacter lutaoensis T5054T (АС NR_025116), на основании чего новые штаммы были отнесены к соответствующим видам. Штаммы Halomonas titanicae TAT1 и Marinobacter lutaoensis KAZ22 были представлены подвижными палочками, окрашивающимися по Граму отрицательно (табл. 1, рис. S1 , дополнительные материалы). Фенотипически штаммы проявляли похожие свойства. В аэробных условиях использовали сахара, органические кислоты, аминокислоты, н-алканы нефти. Штамм H. titanicae ТАТ1 был способен также расти в анаэробных условиях в среде с ацетатом, восстанавливая нитрат с образованием около 100 мг нитрита/л. Штаммы H. titanicae TAT1 и M. lutaoensis KAZ22 имеют оптимумы роста при солености 2.0–12 и 5–10% NaCl (вес/об.) соответственно (табл. 1). Эти значения совпадают с оптимумами солености (0.5–2.5 M) группы умеренных галофилов, согласно классификации (Edbeib et al., 2016). Способность штамма TAT1 расти в отсутствие соли в среде позволяет отнести его к группе галотолерантных бактерий, тогда как штамм KAZ22 является галофилом. Штамм H. titanicae TAT1 проявлял психротолерантные свойства и рос при температуре от 4 до 42°С, а штамм M. lutaoensis KAZ22 был термотолерантным и рос при более высокой температуре (22–55°С). Интервалы температуры и солености для роста штаммов TAT1 и KAZ22 соответствовали условиям их местообитания в нефтяных пластах. Штамм H. titanicae TAT1 проявлял слабый рост на нефти, используя длинноцепочечные C20–C23 и C26–C32н-алканы (рис. S2 а). Штамм M. lutaoensis KAZ22 использовал C14–C19н-алканы нефти (рис. S2 б). Штамм ТАТ1 рос на тяжелой нефти, снижая поверхностное натяжение и межфазное натяжение среды (против гексадекана) более чем на 10 мН/м по сравнению со стерильной средой. Штамм KAZ22 проявлял реологические свойства как при росте на нефти, так и на растворимых субстратах; он снижал проверхностное натяжение более чем на 20 мН/м и межфазное (против гексадекана) – на 10 мН/м, вызывая эмульгирование среды (индекс эмульгирования Е24 составлял 20%). Время удвоения µ штамма KAZ22 составляло 0.58 сут–1 в среде с бакто-триптоном (5.0 г/л), дрожжевым экстрактом (2.5 г/л), глюкозой (1.0 г/л) и 60 г NaCl/л при 37°С. Способность расти в широком интервале температуры, вероятно, обусловливает присутствие штамма KAZ22 в призабойной зоне нагнетательной скважины высокотемпературного нефтяного месторождения Узень. Нагнетание сточной нефтепромысловой воды, смешанной с водой Каспийского моря, приводит к локальному снижению температуры пласта с 57–68°С до 40–45°С (Sokolova et al., 2021). Таким образом, температурный режим и общая минерализация пластовой воды (51.4 г/л) в зоне нагнетательной скважины 2755 были благоприятны для роста штамма KAZ22. Поступление кислорода, растворенного в закачиваемой воде, поддерживает рост популяции аэробных нефтеокисляющих бактерий в зоне нагнетания.
Таблица 1.
Фенотипические признаки галофильных/галотолерантных бактерий M. lutaoensis KAZ22 и H. titanicae TAT1, выделенных из нефтяных пластов
| Признак | TAT1 | KAZ22 |
|---|---|---|
| Морфология клеток | Палочки | Палочки |
| Размер клеток (мкм) | 0.6 × 2.0 | 2.0–3.5 × 0.6–1.0 |
| Подвижность | + | + |
| Интервал (оптимум) NaCl для роста, % (вес/об.) | 0–20 (5–10) | 0.5–14 (2.0–12) |
| Интервал (оптимум) температуры для роста, °С | 4–42 (37) | 22–55 (28–37) |
| Образуют кислоту из: | ||
| мальтоза | + | + |
| манноза | – | + |
| маннит, сахароза | + | – |
| Используют: | ||
| н-алканы нефти | + | + |
| ацетат | +/– | + |
| формиат, бутират, бензоат | – | – |
| валерат | – | +/– |
| цитрат | + | – |
| пируват, сукцинат, фумарат, глутамат | + | + |
| DL-фенилаланин | + | – |
| нафталин | + | |
| ${\text{NO}}_{3}^{ - }$ → ${\text{NO}}_{2}^{ - }$ | + | – |
| ${\text{NO}}_{3}^{ - }$ → N2 | – | – |
| Образование H2S | – | – |
| Уреаза | + | – |
| Активность ферементов (API Zym): | ||
| щелочная фосфатаза, эстераза (С4), лейцин ариламидаза, валин ариламидаза, цистин ариламидаза, нафтол-AS-BI-фосфогидролаза β-глюкозидаза | + | + |
| α-химитрипсин, β-галактозидаза, β-глюкуронидаза, α-фукозидаза | – | – |
| эстераза липаза (С8), липаза (С14), N-ацетил-β-глюкозаминидаза, α-маннозидаза | – | + |
| трипсин, кислая фосфатаза, α-глюкозидаза | + | – |
| Г + Ц генома, % | 54.6 | 63.4 |
| Филогенетически близкий вид | Halomonas titanicae BHT | Marinobacter lutaoensis T5054T |
| Сходство генов 16S рРНК, % | 99.73 | 99.9% |
Детекция генов окисления н-алканов. С использованием тотальной ДНК и вырожденных праймеров, приведенных в табл. S1 , был выполнен поиск генов деградации алканов (alkB, ladAα, ladAβ, ladB, cyp153A и almA) у галофильных бактерий H. titanicae TAT1 и M. lutaoensis KAZ22. Ожидаемая длина ПЦР-продуктов генов alkB – 500 п.н., ladAα и ladAβ – около 1500 п.н., ladB – около 1000 п.н., cyp153 – 800 п.н. и almA – около 1100 п.н. В качестве положительного контроля использовали штамм Geobacillus icigianus G1w1T, у которого ранее были обнаружены гены alkB, ladAα, ladAβ и ladB (Tourova et al., 2018).
В результате ПЦР с использованием праймеров, специфичных гену alkB, ПЦР-продукт ожидаемой длины (около 500 п.н.) был получен для M. lutaoensis KAZ22, H. titanicae TAT1 и G. icigianus G1w1T (рис. S3 а). С использованием праймеров к гену ladA, кодирующему алкан-монооксигеназу, участвующую в деструкции длинноцепочечных алканов (гены ladAα, ladAβ, ladB), специфичный ПЦР-продукт был получен только для контрольного штамма G. icigianus G1w1T (рис. S3 б‒S3г). У других исследуемых штаммов бактерий продукт не был детектирован. С праймерами к гену цитохром-подобной монооксигеназы (cyp153) специфичная амплификация наблюдалось только с ДНК R. erythropolis HO-KS22 (рис. S3 д). Специфичный ПЦР-продукт гена almA (около 1100 п.н.) получен для всех исследованных штаммов маринобактеров (рис. S3 е).
Характеристика геномов Halomonas titanicae TAT1 и Marinobacter lutaoensis KAZ22. Геном H. titanicae TAT1 состоял из 51 скаффолда общим размером 5 303 463 п.н., имел среднее покрытие 285× и G + C содержание 54.6% (табл. 2). Значения ANI и dDDH с близкородственным геномом H. titanicae BH1T составили 99.8 и 99.2% соответственно, что подтверждает идентификацию штамма как представителя этого вида (Grouzdev et al., 2020). Геном содержал 4910 генов, из которых 4757 были белок-кодирующими генами и 67 кодирующими РНК. Функциональная аннотация генома, выполненная с использованием RASTtk, выявила 409 генов связанных с метаболизмом аминокислот и их производных, 273 гена связанных с углеводным обменом, 212 генов – с метаболизмом кофакторов, витаминов, пигментов и 169 генов ассоциированных с белковым обменом. Анализ генома, основанный на результатах функционального предсказания белков с помощью BlastKOALA, позволяет предположить, что геном штамма TAT1 содержит все ключевые гены, отвечающие за полные пути углеводного обмена: гликолиз (путь Эмбдена‒Мейергофа), окисление пирувата, цитратный цикл (ЦТК), пентозофосфатный путь, путь Энтнера‒Дудорова, деградация D‑глюкуроната и D-галактоната, глиоксилатный цикл. Кроме того, геном содержит гены деградации бензоата (benABCDE) и катехола (catABC), организованные в единый генный кластер. Геном TAT1 несет гены cysND, cysC, cysH, cysI, ответственные за ассимиляционную сульфатредукцию.
Таблица 2.
Характеристика геномов бактерий H. titanicae TAT1 и M. lutaoensis KAZ22
| Характеристика | H. titanicae TAT1 | M. lutaoensis KAZ22 |
|---|---|---|
| Биопроект | PRJNA637646 | PRJNA638879 |
| Номер в Генбанке | JABWTB010000000 | JABWTC000000000 |
| Размер генома (п.н.) | 5 303 463 | 3 460 169 |
| Г + Ц генома, % | 54.6 | 63.4 |
| Полнота сборки/контаминация, % | 100.0/0.9 | 99.56/0.29 |
| Количество парноконцевых ридов | 8 878 727 | 5 406 706 |
| N50 (п.о.) | 381 158 | 111 938 |
| Покрытие (×) | 285 | 460 |
| Общее количество генов | 4908 | 3231 |
| Количество белок-кодирующих генов | 4743 | 3066 |
| Количество генов (РНК) | 67 | 60 |
| Ближайший родственный вид (номер геномной сборки) | H. titanicae BH1T (GCF_000336575) | M. lutaoensis T5054T (GCF_001981305) |
| ANI/dDDH | 99.8%/99.2% | 99.3%/94.7% |
В результате секвенирования и сборки генома штамма KAZ22 было получено 70 скаффолдов общим размером 3 460 169 п.н. и содержанием G + C 63.4%. Значения ANI и dDDH для KAZ22 и ближайшего штамма M. lutaoensis T5054T составили 99.3 и 94.7% соответственно, что указывает на принадлежность изолированного штамма к этому виду. Геном штамма M. lutaoensis KAZ22 содержал 3236 генов, из них 3059 белок-кодирующих последовательностей, 117 псевдогенов и 60 генов РНК. Анализ с помощью RASTtk показал, что 183 гена связаны с углеводным обменом, 308 генов – с метаболизмом аминокислот и их производных, 189 генов – с метаболизмом белков, 127 генов – с метаболизмом кофакторов, витаминов и пигментов. Геном штамма KAZ22 несет полные наборы генов для гликолиза, окисления пирувата, цикла трикарбоновых кислот, пентозофосфатного пути, пути Энтнера‒Дудорова, биосинтеза гликогена. Геном также имеет гены, необходимые для ассимиляционной сульфатредукции, а также сульфид:хиноноксидоредуктазу.
Филогенетический анализ и структура генов окисления алканов. Экспериментальные данные по ПЦР-детекции генов алкан-моноокигеназ, которые продемонстрировали амплификацю генов alkB и almA на матрице ДНК штамма M. lutaoensis KAZ22 и гена alkB на матрице ДНК штамма H. titanicae TAT1, были подтверждены на основании анализа их геномов.
В геноме штамма M. lutaoensis KAZ22 было аннотировано 2 гена алкан-1-монооксигеназ (аlkB1 и аlkB2) длиной 1284 и 1140 п.н. соответственно. В базе данных GenBank транслированные аминокислотные последовательности этих генов (427 и 379 а.о.) были близки к аналогичным последовательностям разных видов и неидентифицированных штаммов рода Marinobacter. На филогенетическом дереве (рис. 1) эти последовательности составляли отдельные близкородственные кластеры в составе гаммапротеобактерий. Кластер видов, содержащих alkB1 ген, включал также штаммы Marinobacter sp. UBA856 и Marinobacter sp. UBA7807 (100% идентичности генов alkB1), типовой штамм M. lutaoensis T5054T (97% идентичности с единственным геном alkB), а также M. salsuginis NBRC 109114 (87% идентичности генов alkB1). В кластер, к которому относился alkB2 ген штамма KAZ22, также входили alkB гены штаммов Marinobacter sp. UBA856 и Marinobacter sp. UBA7807 (100% идентичности) и M. salsuginis NBRC 109114 (83% идентичности) с высоким уровнем бутстреп-поддержки (100%). Однако в геноме типового штамма M. lutaoensis T5054T ген alkB2 отсутствовал.
Рис. 1.
Филогенетическое дерево транслированных аминокислотных последовательностей генов alkB различных филумов бактерий, включая бактерии родов Marinobacter и Halomonas. Дерево построено с использованием алгоритма maximum likelihood. Жирным шрифтом выделены последовательности, исследованные в данной работе, рамкой – штамм M. salsuginis NBRC 109114, в геноме которого представлено 3 alkB-гена. Точками обозначены ветви дерева с бутстреп-поддержкой больше 80%. Масштаб показывает эволюционное расстояние, соответствующее 10 заменам на каждые 100 аминокислотных остатков.
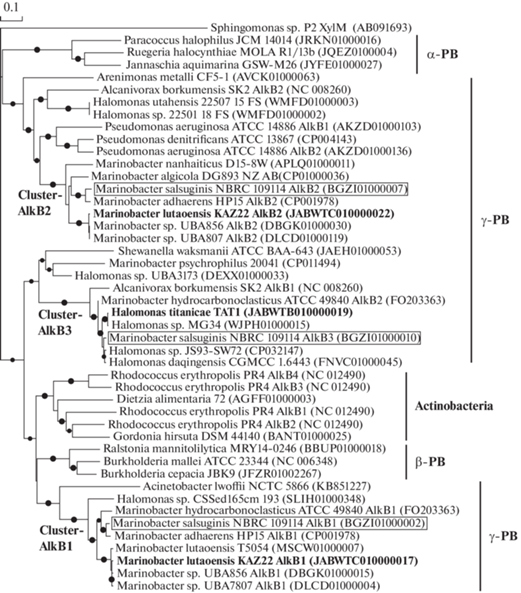
В результате аннотирования генома штамма H. titanicae TAT1 был выявлен единственный ген алкан-1-моноокисигеназы (alkB) длиной 1224 п.н., хотя в геноме типового штамма H. titanicae BH1T этот ген отсутствует. В базе данных GenBank транслированная аминокислотная последовательность alkB гена H. titanicae TAT1 (407 а.о.) была наиболее близка к аналогичным последовательностям из геномов гамма-протеобактерий родов Halomonas и Marinobacter. На филогенетическом дереве транслированных аминокислотных последовательностей генов alkB (рис. 1) последовательность штамма TAT1 относилась к близкородственному кластеру, включающему гомологичные последовательности Halomonas sp. MG34, Halomonas sp. JS92-SW72 и H. daqingensis CGMCC 1.6443 (96–100% идентичности), а также M. hydrocarbonoclasticus ATCC 49840 (alkB2) (96% идентичности) и M. salsuginis NBRC 109114 (alkB3) (83% идентичности), с бутстреп-поддержкой 99%, что свидетельствует о достоверности ветвления.
Таким образом, alkB-гены штаммов H. titanicae TAT1 и M. lutaoensis KAZ22 распредилились на филогенетическом дереве по трем кластерам, которые были обозначены как “Marinobacter-AlkB1, AlkB2 и AlkB3” в соответствии с тройным набором alkB-генов в геноме штамма M. salsuginis NBRC 109114.
Кроме генов алкан-1-монооксигеназ, в геноме штамма M. lutaoensis KAZ22 был также аннотирован ген almA длиной 1527 п.н., кодирующий флавин-зависимую монооксигеназу (AlmA). В базе данных GenBank транслированная аминокислотная последовательность almA гена (508 а.о.) была наиболее близка к аналогичным последовательностям из геномов гамма-протеобактерий рода Marinobacter. На построенном филогенетическом дереве (рис. 2) almA ген штамма KAZ22 входил в близкородственный кластер генов разных видов рода Marinobacter с высоким уровнем бутстреп-поддержки (100%). При этом наиболее близкими к нему также оказались гены штаммов Marinobacter sp. UBA856, Marinobacter sp. UBA7807 и типового штамма M. lutaoensis T5054T (100% идентичности).
Рис. 2.
Филогенетическое дерево транслированных аминокислотных последовательностей генов almA различных филумов бактерий, включая бактерии рода Marinobacter. Дерево построено с использованием алгоритма maximum likelihood. Жирным шрифтом выделена последовательность, исследованная в данной работе. Точками обозначены ветви дерева с бутстреп-поддержкой больше 80%. Масштаб показывает эволюционное расстояние, соответствующее 10 заменам на каждые 100 аминокислотных остатков.

Гены алкан-монооксигеназ присутствуют в большинстве геномов бактерий рода Marinobacter (alkB гены в 90% и almA гены в 86% из 362 геномов), при этом многие из них содержат больше одного гена на геном. Это свидетельствует о том, что способность к использованию н-алканов является важной частью метаболизма представителей этого рода. При этом некоторые штаммы сохранили полный набор генов алкан-монооксигеназ (alkB, alkB2, alkB3 и almA), аналогично штамму M. salsuginis NBRC 109114, а другие утрачивали гены из полного набора, что, возможно, было связано с особенностями их сред обитания. Штамм M. lutaoensis KAZ22, как и многие бактерии рода Marinobacter, входящие в “Marinobacter-AlkB1 и ‑AlkB2” филогенетические кластеры, характеризуется присутствием нескольких alkB-генов, расположенных в разных областях генома и не связанных с другими генами деградации н-алканов. Генная организация AlkB1-области была идентична для штаммов KAZ22, Marinobacter sp. UBA856, Marinobacter sp. UBA7807 и M. lutaoensis T5054T. Для этих штаммов alkB1-ген фланкирован противоположно направлеными генами, детерминирующими неизвестный белок и 3-метилмеркаптопропионил-КоА дегидрогеназу (рис. 3а). Похожая организация AlkB1-области выявлена для M. salsuginis NBRC 109114 и других штаммов Marinobacter-AlkB1 кластера.
Рис. 3.
Сравнение структуры геномных кластеров, содержащих аннотированные гены алкан-моноксигеназ у штаммов галофильных бактерий. Обозначения: (a) – 1 – 3-метилмеркаптопропионил-КоА дегидрогеназа (DmdC), 2, 3 – неизвестный белок, 4 – предполагаемая фосфатаза; (б) – 1 – предполагаемая L-лактатдегидрогеназа, 2 – субъединица Yk-gG неизвестного белка, 3 – пермеаза, 4 – лактоилглютатион лиаза (GloA), 5 – транскрипционный регулятор семейства GntR; (в) – 1 – FAD/FMN-зависимая оксидоредуктаза, 2 – регулятор транскрипции, 3 – белок внешней мембраны, 4 – тиоредоксин, 5 – неизвестный белок, 6 – предположительно трансглютаминаза; (г) – 1 – 3-метилмеркаптопропионил-КоА лигаза, 2 – алкогольдегидрогеназа (AlkJ), 3 – альдегиддегидрогеназа (AlkH), 4 – рубредоксин (Rub), 5 – активатор транскрипции alkB-кластера, 6 – ферредоксин-редуктаза.

Генная организация AlkB2-области также идентична для штамма KAZ22 и штаммов Marinobacter sp. UBA856, Marinobacter sp. UBA7807 и M. salsuginis NBRC 109114. В этой области alkB2-ген фланкирован геном лактоилглутатионлиазы (gloA) и противоположно направленным геном транскрипционного регулятора семейства GntR (рис. 3б). Такая структура генной области AlkB2 характерна для многих представителей рода Ma-rinobacter, обладающих гомологичным alkB-геном. Можно отметить, что в геноме типового штамма M. lutaoensis T5054T имеются аналогично направленные гены лактоилглутатионлиазы и транскрипционного регулятора семейства GntR, хотя ген alkB2 между ними отсутствет, что, видимо, связано с его потерей в процессе эволюции.
Генная организация AlmA-области штамма KAZ22 была похожа на таковую штамма M. lutaoensis T5054T, а также неидентифицированного штамма Oceanospirillales bacterium SP333. Ген almA был расположен одиночно и фланкирован геном неизвестного белка и противоположно направленным геном, предположительно сходным с трансглютаминазой (рис. 3в). Остальные бактерии рода Marinobacter, включая M. salsuginis NBRC 109114, входящие в родственный кластер на филогенетическом дереве, имели другую структуру AlmA-области генома.
Исходя из сходства последовательностей генов деградации н-алканов штамма M. lutaoensis KAZ22, а также сходства структуры областей расположения этих генов в геномах, можно предположить эволюционную взаимосвязь его способности к деградации н-алканов с таковой других бактерий рода Marinobacter, из которых самым близким к нему является типовой штамм вида M. lutaoensis T5054T.
Из 464 проанализированных геномов бактерий рода Halomonas всего в 5% геномов были аннотированы гены алкан-1-моноксигеназ (alkB), причем в каждом из этих геномов, как и в геноме штамма H. titanicae TAT1, был представлен единственный alkB ген. При этом структура AlkB-областей в геномах отдельных бактерий рода Halomonas была различной (рис. 3г). В области расположения гена alkB в геноме штамма H. titanicae TAT1 присутствовали и другие гены, участвующие в деградации н-алканов: ген рубредоксина, ген альдегидгидрогеназы alkH, ген алкогольдегидрогеназы alkJ, а также противоположно направленный ген активатора транскрипции alkB генного кластера. Аналогичной генной структурой в этой области обладают геномы бактерий рода Halomonas (Halomonas sp. MG34 и H. daqingensis CGMCC 1.6443), нескольких штаммов рода Marinobacter, относящихся к филогенетическому кластеру “Marinobacter-AlkB3”, включая M. salsuginis NBRC 109114 (alkB3) и M. hydrocarbonoclasticus ATCC 49840, а также представителей других родов Gamma- и Alphaproteobacteria (Alcanivorax, Sulfitobacter, Hyphomonas, Bradyrhizobium).
Сходство структуры достаточно протяженного участка генома позволяет предположить возможность появления детерминант деградации н-алканов и приобретения соответствующих процессов метаболизма у штамма H. titanicae TAT1 и близких к нему штаммов за счет межвидового горизонтального переноса генов, вероятно, из геномов бактерий рода Marinobacter, для которых эта способность является характерной. Вероятно, такой перенос обусловлен приспособлением штамма H. titanicae TAT1 к существованию в загрязненных нефтью водных средах, что существенно отличает его от типового штамма H. titanicae BH1T (Sanchez-Porro et al., 2010) и других штаммов, идентифицированных как H. titanicae, которые выделены из морских сред и не имеют в геномах alkB генов.
Гены метаболизма н-алканов. Согласно базе данных KEGG, процесс деградации алканов включен в путь метаболизма “Деградация жирных кислот” (рис. S4 а, S4б). Фермент алкан-моноксигеназа (ЕС 1.14.15.3) участвует в этом процессе как на начальной стадии окисления алканов, так и на терминальной стадии окисления ω-гидроксижирных кислот. Исходя из аннотации геномов M. lutaoensis KAZ22 и H. titanicae TAT1 потенциальное присутствие этого фермента предполагается для обоих штаммов, при этом для M. lutaoensis KAZ22 показано присутствие двух форм фермента, как негемовой железо-содержащей формы (AlkB), так и флавин-зависимой формы (AlmA), а для H. titanicae TAT1 только формы AlkB (рис. S4 а, S4б). Вместе с алкан-монооксигеназой в состав гидролазной системы окисления алканов входят белок рубредоксин и фермент рубредоксинредуктаза (ЕС 1.18.1.1), гены которых также представлены в геномах обоих штаммов. Однако локализация этих генов в геномах исследуемых штаммов различается. У H. titanicae TAT1 ген рубредоксина (rub) входит в один кластер с единственным геном alkB, а ген рубредоксинредуктазы (rubB) расположен на другом участке генома (рис. 4а, 4б). У M. lutaoensis KAZ22 оба этих гена составляют кластер, расположенный отдельно от каждого из генов алкан-моноокисгеназы (alkB1, alkB2 и almA). Другие гены метаболизма алканов, алкогольдегидрогеназа (ЕС 1.1.99.20 и ЕС 1.1.1.1) и альдегиддегидрогеназа (ЕС 1.2.1.3), расположены в одном кластере с геном alkB у H. titanicae TAT1 и на отдельных участках генома у M. lutaoensis KAZ22. Дополнительные сведения о генах деградации н-алканов представлены в табл. S3 .
Рис. 4.
Комплексы генов деградации н-алканов (а, б) и генов синтеза осмопротекторов (в, г) в геномах бактерий H. titanicae TAT1 (а, в) и M. lutaoensis KAZ22 (б, г). Обозначения генов: (а) – HT661-23455 – алкогольдегидрогеназа; НТ661_23480 – альдегиддегидрогеназа; rub – рубредоксин; alkB – алкан-1-моноокcигеназа; НТ661_23475 – активатор транскрипции alkB-кластера; rubB – рубредоксинредуктаза; (б) – alkB1, alkB2, almA – алкан-1-моноокcигеназа; rub – рубредоксин; rubB – рубредоксинредуктаза; HUS22_00130, HUS22_11055 – альдегиддегидрогеназа; HUS22_11065 – алкогольдегидрогеназа; (в) – betA – холиндегидрогеназа; betB – бетаин-альдегиддегидрогеназа; ectA – диаминобутиратацетилтрансферазa; ectB – диаминобутираттрансаминазa; ectC – эктоинсинтаза; ectD1, ectD2 – эктоингидролаза; (г) – ectA – диаминобутиратацетилтрансферазa; ectB – диаминобутираттрансаминазa; ectC – эктоинсинтаза; ask – аспартаткиназа.

Гены метаболизма осмопротекторов. В геномах штаммов H. titanicae TAT1 и M. lutaoensis KAZ22 выполнен поиск генов образования основных осмопротекторов – бетаина и эктоина, которые в базе данных KEGG включены в путь “Метаболизм глицина, серина и треонина”. В геноме H. titanicae TAT1 обнаружен кластер генов betIBA (табл. S4 , рис. S5 а), детерминирующих ферменты окисления холина до бетаинового альдегида (холиндегидрогеназа, ЕС 1.1.99.1) и далее до бетаина (бетаин-альдегиддегидрогеназа, (ЕС 1.2.1.8), что свидетельствует о потенциальной возможности синтеза бетаина. Кроме того, в геноме H. titanicae TAT1 присутствуют все гены образования осмопротекторов эктоина и гидроксиэктоина из аспартата и треонина через промежуточный метаболит аспартат-полуальдегид. Гены синтеза эктоина (ectA, ectB и ectC), как и в геномах других бактерий рода Halomonas, объединены в кластер, кодирующий диаминобутиратацетилтрансферазу (ЕС 2.3.1.178) (EctA), диаминобутираттрансаминазу (ЕС 2.6.1.76) (EctB) и эктоинсинтазу (EC 4.2.1.108) (EctC) (рис. 4в). Кроме того, в геноме штамма TAT1 аннотированы 2 гена (ectD1 и ectD1), предположительно детерминирующие эктоиндегидрогеназу (ЕС 1.14.11.55), которые расположены каждый в различных участках генома (рис. 4в). Аналогичные системы образования осмопротекторов были подробно рассмотрены у Chromohalobacter salexigens (ранее Halomonas elongata DSM 3043) (Vargas et al., 2008).
В геноме штамма M. lutaoensis KAZ22 отсутствовали гены образования бетаина. В то же время в геноме этого штамма также аннотированы все гены образования осмопротекторов эктоина и гидроксиэктоина (рис. S5 б). Локализация этих генов в геноме похожа на локализацию аналогичных генов в геноме M. hydrocarbonoclasticus (ранее M. aquaeoli) (Vargas et al., 2008). Гены ectA и ectB сгруппированы в единый кластер вместе с геном ask аспартаткиназы (EC 2.7.2.4), а единственные гены ectС и ectD локализованы отдельно на различных участках генома (рис. 4г). Дополнительные сведения о генах синтеза осмопротекторов представлены в табл. S4 .
Гены метаболизма бензоата у исследованных галофильных бактерий. В геноме штамма M. lutaoensis KAZ22 наряду с несколькими генами деградации н-алканов, присутствовало большое количество генов разложения ароматических соединений, таких как бензол, катехол, бензоат и фенилацетат. Бензоат является центраболитом деградации многих ароматических соединений. В геноме H. titanicae TAT1 представлены гены, кодирующие ферменты аэробной деградации бензоата – бензоат-1,2-диоксигеназу (EC 1.14.12.10) и дигидроксициклогексадиен-дегидрогеназу (EC 1.3.1.25), которые катаболизируют превращения бензоата до катехола. Кроме того, в его геноме аннотированы гены всех ферментов орто-расщепления катехола, начиная с катехол 1,2-диоксигеназы (ЕС 1.13.11.1), как до сукцинил-КоА, так и до пирувата/ацетил-КоА, а также гены фермента деградации фенола и толуола – толуол/фенол-2-монооксигеназы (ЕС 1.14.13.244) (рис. S6 ). Отметим, что у галофильной бактерии Amycolicicoccus subflavus DQS3-9A1T при мульткопийности генов деградации н-алканов, отсутствовали гены деградации ароматических соединений (Nie et al., 2013). В отличие от этой бактерии, в геноме Polymorphum gilvum SL003B-26A1T были обнаружены гены ферментов полных путей катаболизма ароматических соединений, но только единственный ген ladA кодировал деградацию н-алканов (Nie et al., 2012), хотя оба микроорганизма были выделены из загрязненных нефтью сред обитания.
Гены метаболизм азота у штамма H. titanicae TAT1. В анаэробных условиях штамм H. titanicae TAT1 рос гетеротрофно на органических субстратах, восстанавливая нитрат до нитрита. Феноменологические наблюдения восстановления нитрата до нитрита штаммом ТАТ1 подтвердились при анализе его генома, в котором были аннотированы гены ферментов, участвующих в диссимиляционном восстановлении нитрата до аммония: нитратредуктаза (гены narG, narZ, nxrA) (EC 1.7.5.1; 1.7.99.4), которая катализирует восстановление нитрата до нитрита, и нитритредуктаза (ген nirВ) (EC 1.7.1.15), завершающая этот процесс восстановлением нитрита до аммония. Кроме того, в геноме H. titanicae TAT1, так же как в геноме типового штамма H. titanicae BH1T, были аннотированы гены неполного пути денитрификации, в котором отсутствовал ключевой ген нитритредуктазы (nirK) (EC 1.7.2.1), кодирующий восстановление нитрита до оксида азота (рис. S7 а). Это свойство ранее отмечалось для ряда штаммов группы H. titanicae (Wang, Shao, 2021).
Штамм M. lutaoensis KAZ22, как и типовой штамм M. lutaoensis T5054T, был неспособен расти анаэробно и восстанавливать нитрат, что подтверждалось отсутствием в его геноме генов нитратредуктаз. Однако в нем был аннотирован ген нитритредуктазы (EC 1.7.1.4), восстанавливающей нитрит до аммония в процессе ассимиляционной нитратредукции (рис. S7 б).
Приведенные результаты изучения физиологии и анализ геномов галофильных/галотолерантных бактерий Marinobacter lutaoensis KAZ22 и Halomonas titanicae ТАТ1, выделенных из нефтяных пластов, свидетельствует о приспособленности штаммов к высокой солености среды местообитания. Штаммы способны расти при температуре нефтяного пласта, используя сырую нефть в аэробных условиях. Наличие трех генов alkB1, alkB2 и almA алкан-монооксигеназ у штамма M. lutaoensis KAZ22 позволяет ему эффективно деградировать н-алканы нефти, тогда как использование н-алканов нефти штаммом H. titanicae ТАТ1, детерминированное геном алкан-1-монооксигеназы alkB, было слабым. Рост при высокой солености штамма ТАТ1 был обусловлен наличием генов синтеза осмопротекторов бетаина, эктоина и гидроксиэктоина, а штамма KAZ22 – наличием генов синтеза эктоина и гидроксиэктоина.
Способность штамма ТАТ1 накапливать нитрит в среде, обусловленная отсутствием гена нитритредуктазы nir, может быть использована в биотехнологии подавления образования сероводорода в нефтяном пласте. Сероводород вызывает коррозию стального оборудования, порчу нефти и осаждение сульфидов, что, в свою очередь, приводит к снижению проницаемости пласта и приемистости нагнетательных скважин (An et al., 2017). Известно, что нитрит подавляет рост бактерий-продуцентов сероводорода посредством ингибирования активности α-субъединицы сульфитредуктазы (dsrA), а также химически окисляет сульфид, снижая его концентрацию в среде. Кроме того, денитрифицирующие бактерии имеют конкурентное преимущество над сульфидогенами в использовании доступных субстратов, поскольку восстановление нитрата дает больший выход энергии, чем восстановление сульфата до сульфида. Таким образом, штамм H. titanicae ТАТ1 может быть использован совместно с нитратами для биоаугментации нефтяных пластов с высокой минерализацией пластовой воды с целью подавления коррозии и снижения содержания сульфида, а штамм M. lutaoensis KAZ22 – для очистки от нефтяных загрязнений и увеличения нефтеотдачи пластов.
Список литературы
Беляев С.С., Борзенков И.А., Назина Т.Н., Розанова Е.П., Глумов И.Ф., Ибатуллин Р.Р., Иванов М.В. Использование микроорганизмов в биотехнологии повышения нефтеизвлечения // Микробиология. 2004. Т. 73. С. 687–697.
Belyaev S.S., Borzenkov I.A., Nazina T.N., Rozanova E.P., Glumov I.F., Ibatullin R.R., Ivanov M.V. Use of microorganisms in the biotechnology for the enhancement of oil recovery // Microbiology (Moscow). 2004. V. 73. P. 590–598.
Борзенков И.А., Милехина Е.И., Готоева М.Т., Розанова Е.П., Беляев С.С. Свойства углеводородокисляющих бактерий, изолированных из нефтяных месторождений Татарстана, Западной Сибири и Вьетнама // Микробиология. 2006. Т. 75. С. 82–89.
Borzenkov I.A., Milekhina E.I., Gotoeva M.T., Rozanova E.P., Belyaev S.S. The properties of hydrocarbon-oxidizing bacteria isolated from the oilfields of Tatarstan, Western Siberia, and Vietnam // Microbiology (Moscow). 2006. V. 75. P. 66–72.https://doi.org/10.1134/S0026261706010127
Звягинцева И.С., Беляев С.С., Борзенков H.A., Кострикина H.A., Милехина H.H., Иванов М.В. Галофильные архебактерии из нефтяного месторождения Каламкасс // Микробиология. 1995. Т. 64. С. 83–87.
Zvyagintseva I.S., Belyaev S.S., Borzenkov I.A., Kostrikina N.A., Milekhina E.I., Ivanov M.V. Halophilic archaebacteria from the Kalamkass oil field // Microbiology (Moscow). 1995. V. 64. P. 67–71.
Турова Т.П., Назина Т.Н., Михайлова Е.М., Родионова Т.А., Екимов А.Н., Машукова А.В., Полтараус А.Б. Гомологи alkB гена термофильных бактерий рода Geobacillus // Мол. биология. 2008. Т. 42. С. 247–257.
Tourova T.P., Nazina T.N., Mikhailova E.M., Rodionova T.A., Ekimov A.N., Mashukova A.V., Poltaraus A.B. AlkB homologs in thermophilic bacteria of the genus Geobacillus // Mol. Biol. 2008. V. 42. P. 217–226.
Турова Т.П., Соколова Д.Ш., Семенова Е.М., Полтараус А.Б., Назина Т.Н. Разнообразие генов биодеградации н-алканов alkB у термофильных углеводородокисляющих бактерий родов Geobacillus, Parageobacillus и Aeribacillus // Микробиология. 2018. Т. 87. С. 225–232.https://doi.org/10.7868/S0026365618030011
Tourova T.P., Sokolova D.Sh., Semenova E.M., Poltaraus A.B., Nazina T.N. Diversity of the alkB genes of n-alkane biodegradation in thermophilic hydrocarbon-oxidizing bacteria of the genera Geobacillus, Parageobacillus, and Aeribacillus // Microbiology (Moscow). 2018. V. 87. P. 301–307.https://doi.org/10.1134/S002626171803013X
Al-Mailem D.M., Sorkhoh N.A., Al-Awadhi H., Eliyas M., Radwan S.S. Biodegradation of crude oil and pure hydrocarbons by extreme halophilic archaea from hypersaline coasts of the Arabian Gulf // Extremophiles. 2010. V. 14. P. 321–328. https://doi.org/10.1007/s00792-010-0312-9
An B.A., Shen Y., Voordouw G. Control of sulfide production in high salinity Bakken shale oil reservoirs by halophilic bacteria reducing nitrate to nitrite // Front. Microbiol. 2017. V. 8. Art. 1164.https://doi.org/10.3389/fmicb.2017.01164
Bankevich A., Nurk S., Antipov D., Gurevich A.A., Dvorkin M., Kulikov A.S., Lesin V.M., Nikolenko S.I., Pham S., Prjibelski A.D., Pyshkin A.V., Sirotkin A.V., Vyahhi N., Tesler G., Alekseyev M.A., Pevzner P.A. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing // J. Comput. Biol. 2012. V. 19. P. 455–477.https://doi.org/10.1089/cmb.2012.0021
Ben Ali Gam Z., Abdelkafi S., Casalot L., Tholozan J.L., Oueslati R., Labat M. Modicisalibacter tunisiensis gen. nov., sp. nov., an aerobic, moderately halophilic bacterium isolated from an oilfield-water injection sample, and emended description of the family Halomonadaceae Franzmann et al. 1989 emend Dobson and Franzmann 1996 emend. Ntougias et al. 2007 // Int. J. Syst. Evol. Microbiol. 2007. V. 57. P. 2307–2313.https://doi.org/10.1099/ijs.0.65088-0
Bødtker G., Lysnes K., Torsvik T., Bjørnestad E.Ø., Sunde E. Microbial analysis of backflowed injection water from a nitrate-treated North Sea oil reservoir // J. Ind. Microbiol. Biotechnol. 2009. V. 36. P. 439–450.
Bolger A.M., Lohse M., Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30. P. 2114–2120.
Boonmak C., Takahashi Y., Morikawa M. Cloning and expression of three ladA-type alkane monooxygenase genes from an extremely thermophilic alkane-degrading bacterium Geobacillus thermoleovorans B23 // Extremophiles. 2014. V. 18. P. 515–523.
Bryanskaya A.V., Rozanov A.S., Slynko N.M., Shekhovtsov S.V., Peltek S.E. Geobacillus icigianus sp. nov., a thermophilic bacterium isolated from a hot spring // Int. J. Syst. Evol. Microbiol. 2015. V. 65. P. 864–869.
Chaumeil P.-A., Mussig A.J., Hugenholtz P., Parks D.H. GTDB-Tk: a toolkit to classify genomes with the Genome Taxonomy Database // Bioinformatics. 2019. V. 36. P. 1925–1927.https://doi.org/10.1093/bioinformatics/btz848
Edbeib M.F., Wahab R.A., Huyop F. Halophiles: biology, adaptation, and their role in decontamination of hypersaline environments // World J. Microbiol. Biotechnol. 2016. V. 32. P. 135.https://doi.org/10.1007/s11274-016-2081-9
Fathepure B.Z. Recent studies in microbial degradation of petroleum hydrocarbons in hypersaline environments // Front. Microbiol. 2014. V. 5. Art. 173. P. 1–16.https://doi.org/10.3389/fmicb.2014.00173
Grouzdev D.S., Sokolova D.S., Poltaraus A.B., Nazina T.N. Draft genome sequence of Halomonas titanicae strain TAT1, a hydrocarbon-oxidizing halophilic bacterium isolated from a petroleum reservoir in Russia // Microbiol. Resour. Announc. 2020. V. 9. e01255-20.https://doi.org/10.1128/MRA.01255-20
Huu N.B., Denner E.B., Ha D.T., Wanner G., Stan-Lotter H. Marinobacter aquaeolei sp. nov., a halophilic bacterium isolated from a Vietnamese oil-producing well // Int. J. Syst. Bacteriol. 1999. V. 49. P. 367–375.https://doi.org/10.1099/00207713-49-2-367
Jain C., Rodriguez R. L.M., Phillippy A.M., Konstantinidis K.T., Aluru S. High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries // Nat. Commun. 2018. V. 9. P. 5114.https://doi.org/10.1038/s41467-018-07641-9
Kaye J.Z., Sylvan J.B., Edwards K.J., Baross J.A. Halomonas and Marinobacter ecotypes from hydrothermal vent, subseafloor and deep-sea environments // FEMS Microbiol. Ecol. 2010. V. 75. Art 1. P. 123–133.https://doi.org/10.1111/j.1574-6941.2010.00984.x
Lane D.J. 16S/23S rRNA sequencing // Nucleic Acid Techniques in Bacterial Systematics / Eds. Stackebrandt E., Goodfellow M. N.Y.: John Wiley& Sons, 1991. P. 115–175.
Luo Y.J., Xie B.S., Lv X.L., Cai M., Wang Y.N., Cui H.L., Cai H., Wu X.L. Marinobacter shengliensis sp. nov., a moderately halophilic bacterium isolated from oil-contaminated saline soil // Antonie Van Leeuwenhoek. 2015. V. 107. P. 1085–1094.https://doi.org/10.1007/s10482-015-0401-y
Meier-Kolthoff J.P., Auch A.F., Klenk H.P., Göker M. Genome sequence-based species delimitation with confidence intervals and improved distance functions // BMC Bioinform. 2013. V. 14. Art. 60.https://doi.org/10.1186/1471-2105-14-60
Mnif S., Chamkha M., Sayadi S. Isolation and characterization of Halomonas sp. strain C2SS100, a hydrocarbon-degrading bacterium under hypersaline conditions // J. Appl. Microbiol. 2009. V. 107. P. 785–794.https://doi.org/10.1111/j.1365-2672.2009.04251.x
Nazina T.N., Shestakova N.M., Pavlova N.K., Tatarkin Y.V., Ivoilov V.S., Khisametdinov M.R., Sokolova D.Sh., Babich T.L., Tourova T.P., Poltaraus A.B., Belyaev S.S., Ivanov M.V. Functional and phylogenetic microbial diversity in formation waters of a low-temperature carbonate petroleum reservoir // Int. Biodeter. Biodegr. 2013. V. 81. P. 71–81.https://doi.org/10.1016/j.ibiod.2012.07.008
Nie Y., Tang Y.-Q., Li Y., Chi C.-Q., Cai M., Wu X.-L. The genome sequence of Polymorphum gilvum SL003B-26A1T reveals its genetic basis for crude oil degradation and adaptation to the saline soil // PLoS One. 2012. V. 7. Art. e31261.https://doi.org/10.1371/journal.pone.0031261
Nie Y., Fang H., Li Y., Chi C.-Q., Tang Y.-Q., Wu X.-L. The genome of the moderate halophile Amycolicicoccus subflavus DQS3-9A1T reveals four alkane hydroxylation systems and provides some clues on the genetic basis for its adaptation to a petroleum environment // PLoS One. 2013. V. 8. Art. e70986.https://doi.org/10.1371/journal.pone.0070986
Parks D.H., Imelfort M., Skennerton C.T., Hugenholtz P., Tyson G.W. CheckM: assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes // Genome Res. 2015. V. 25. P. 1043–1055.https://doi.org/10.1101/gr.186072.114
Sanchez-Porro C., Kaur B., Mann H., Ventosa A. Halomonas titanicae sp. nov., a halophilic bacterium isolated from the RMS Titanic // Int. J. Syst. Evol. Microbiol. 2010. V. 60. P. 2768‒2774.https://doi.org/10.1099/ijs.0.020628-0
Sokolova D.S., Semenova E.M., Grouzdev D.S., Bidzhieva S.K., Babich T.L., Loiko N.G., Ershov A.P., Kadnikov V.V., Beletsky A.V., Mardanov A.V., Zhaparov N.S., Nazina T.N. Sulfidogenic microbial communities of the Uzen high-temperature oil field in Kazakhstan // Microorganisms. 2021. V. 9. P. 1818. https://doi.org/10.3390/microorganisms9091818
Tatusova T., Di Cuccio M., Badretdin A., Chetvernin V., Ciufo S., Li W. Prokaryotic Genome Annotation Pipeline // The NCBI Handbook, 2nd ed. National Center for Biotechnology Information, Bethesda, MD. 2013. http://www.ncbi.nlm.nih.gov/books/NBK174280
Vargas C., Argandoña M., Reina-Bueno M., Rodríguez-Moya J., Fernández-Aunión C., Nieto J.J. Unravelling the adaptation responses to osmotic and temperature stress in Chromohalobacter salexigens, a bacterium with broad salinity tolerance // Saline Syst. 2008 V. 4. Art. 14.https://doi.org/10.1186/1746-1448-4-14
Wang W., Shao Z. Diversity of flavin-binding monooxygenase genes (almA) in marine bacteria capable of degradation long-chain alkanes // FEMS Microbiol. Ecol. 2011. V. 80. P. 523–533.
Wang L., Shao Z. Aerobic denitrification and heterotrophic sulfur oxidation in the genus Halomonas revealed by six novel species characterizations and genome-based analysis // Front. Microbiol. 2021. V. 12. Art. 652766.https://doi.org/10.3389/fmicb.2021.652766
Wang L., Wang W., Lai Q., Shao Z. Gene diversity of CYP153A and AlkB alkane hydroxylases in oil-degrading bacteria isolated from the Atlantic Ocean // Environ. Microbiol. 2010. V. 12. P. 1230–1242.
Wilson K. Preparation of genomic DNA from bacteria // Curr. Protoc. Mol. Biol. 2001. Ch. 2. Unit 2.4. https://doi.org/10.1002/0471142727.mb0204s56
Дополнительные материалы
- скачать ESM.docx
- Таблица S1 - Таблица S4.
Рис. S1. - Рис. S7.


