Микробиология, 2022, T. 91, № 4, стр. 451-465
Молекулярно-генетический и функциональный анализ системы конъюгации плазмиды pBS72 природных бактерий Bacillus subtilis
А. С. Гуринович a, М. А. Титок a, *
a Кафедра микробиологии, биологический факультет, Белорусский государственный университет
220030 Минск, Республика Беларусь
* E-mail: m_titok@bsu.by
Поступила в редакцию 14.02.2022
После доработки 22.03.2022
Принята к публикации 27.03.2022
- EDN: JRTMLY
- DOI: 10.31857/S0026365622300188
Аннотация
Конъюгационная система плазмиды pBS72, широко распространенная среди внехромосомных генетических элементов природных бактерий рода Bacillus, является уникальной и может быть отнесена к новому семейству. Новая систематическая группа, обозначенная как MpfS, имея сходство с системой конъюгации хорошо изученной плазмиды pLS20 бактерий B. subtillis nato, характеризовалась особенностями молекулярно-генетической организации и функциональной активности. В частности, ключевые белки конъюгации VirB4, VirB6, VirB11, VirD4 и Adg плазмид pLS20 и pBS72, сходные в области функциональных доменов, достоверно отличались между собой, образуя отдельные филогенетические группы. Гены (rco, rap, phr), определяющие белки регуляции конъюгации плазмиды pLS20, в плазмиде pBS72 имели иную локализацию, а сходство между аминокислотными последовательностями не превышало 40.21% для белка Rco, 24.41% для белка Rap и отсутствовало для белка Phr. В отличие от плазмиды pLS20, частота конъюгационного переноса плазмиды pBS72 и мобилизуемой плазмиды pCB16 не зависела от стадии роста и концентрации донорных клеток и определялась составом среды и генетическими особенностями бактерий реципиентов. Полученные результаты являются основой для изучения механизма распространения pBS72-подобных плазмид в природной среде обитания и создания на их основе системы генетического анализа биотехнологически значимых бактерий рода Bacillus.
Распространение генов антибиотикорезистентности среди природных микроорганизмов представляет серьезную проблему. В медицинской практике данный феномен рассматривается как новый вид инфекции, осложняющий лечение бактериальных заболеваний дорогостоящими антибактериальными препаратами. Усилия многих научных лабораторий направлены на изучение путей ее распространения и механизмов возникновения. В этом плане ключевой интерес представляют гены антибиотикорезистентности, локализованные в составе плазмид, которые могут попадать в клетки природных микроорганизмов путем конъюгации, трансформации или трансдукции (Davies, 2010). Следует отметить, что только бактерии способны использовать системы генетического обмена для горизонтального переноса генов между неродственными видами, что обеспечивает их быструю адаптацию в изменяющихся условиях внешней среды.
Среди систем горизонтального переноса генов ключевой интерес представляет процесс конъюгации, за счет которого осуществляется эффективная передача не только конъюгативных плазмид, но и любых внехромосомных генетических элементов (передаются за счет конъюгативных плазмид или путем временного объединения двух плазмидных репликонов в клетке донора, с последующим разъединением в клетке реципиента) (Xiaoming, 1997). В связи с этим, конъюгативные системы являются объектом пристального внимания. В частности, для них активно разрабатывается классификация, основанная на особенностях организации ключевых белков, определяющих конъюгационный перенос. Накопленные данные позволяют систематизировать знания о функциональной организации этих систем и служат основой для изучения механизмов распространения плазмид в природной среде обитания.
Структурная и функциональная организация белков, определяющих конъюгационный перенос, наиболее полно изучена для Ti-плазмиды бактерий Agrobacterium tumefaciens. Для остальных плазмид проводят анализ отдельных белков в сравнении с таковыми плазмиды Ti, позволяющий судить об их функциональной активности. В результате такого подхода были выявлены наиболее консервативные белки, которые используются в качестве маркерных для классификации внехромосомных генетических элементов. В первую очередь, это релаксаза (Mob-белок), входящая в систему всех без исключения плазмид, передающихся путем конъюгации (конъюгативные и мобилизуемые плазмиды). Она является ключевым белком конъюгации, распознающим oriT-сайт, локализованным в цис-положении на передающейся плазмиде, и катализирует начальное расщепление плазмидной ДНК в oriT-сайте в клетке донора и восстановление передаваемой плазмиды в клетке реципиентa. На основании сходства релаксаз, конъюгативные и мобилизуемые плазмиды отнесены к семи классификационным группам или семействам (MOBF, MOBH, MOBQ, MOBC, MOBP, MOBV и MOBL) (Ramachandran, 2017). Следует отметить, что релаксазы семейства MOBL характерны только для конъюгативных плазмид грамположительных бактерий группы Firmicutes.
Поскольку перенос ДНК из клетки донора в клетку реципиента требует энергетических затрат, все конъюгативные плазмиды кодируют белки с АТФ-азной активностью. Ключевыми АТФ-азами являются белок VirB4, входящий в Mpf-систему секреции IV типа и определяющий перенос ДНК через цитоплазматическую мембрану (для плазмид грамотрицательных бактерий дополнительно определяет синтез половых пилей), и белок VirD4 (coupling protein – связывающий белок T4CP), играющий роль связывающего белка между Mpf-системой и релаксасомой (Тra-система). На основании сходства белков VirB4 выделено восемь классификационных групп (MPFT, MPFF, MPFG, MPFC, MPFB, MPFFA, MPFFATA, MPFI) (Guglielmini et al., 2014).
Следует отметить, что ограниченный круг внехромосомных генетических элементов (плазмиды семейств MPFT и MPFI) кодируют синтез еще одной АТФ-азы VirB11, взаимодействующей с белком VirB4 и выполняющей сходные с ним функции. Во время переноса данных плазмид одна цепь ДНК последовательно контактирует со связывающим белком VirD4, а затем с АТФ-азой VirB11 и белками внутренней мембраны VirB6 и VirB8 и, наконец, с белковым комплексом, образующим переплазматический канал для переноса (VirB7, VirB9 и VirB10) (Cascales, 2003). Системы конъюгации грамположительных бактерий не содержат генов, кодирующих белки, обеспечивающие образование пилей (VirB5 и VirB2) и перенос ДНК через периплазму (VirB7, VirB9 и VirB10) (Alvarez-Rodriguez, 2020). Контакт между конъюгирующими клетками обеспечивается адгезинами (Grohmann, 2017).
Конъюгативные и мобилизуемые плазмиды грамположительных бактерий группы Firmicutes представляют большой научный и практический интерес, поскольку эти повсеместно распространенные микроорганизмы являются патогенами для человека и животных (например, Staphylococcus, Streptococcus и др.), входят в состав нормальной микробиоты человека (например, Lactobacillus) и широко используются в промышленном производстве в качестве продуцентов биологически активных соединений, ферментов, антибиотиков, стимуляторов роста растений и животных (Harwood, 2018). Ряд представителей этой обширной группы (например, B. subtilis) отнесены к абсолютно безопасным для человека микроорганизмам (имеют статус GRAS). Отсутствие принципиальных генетических барьеров между представителями Firmic-utes предполагает возможность горизонтального переноса генов с помощью конъюгативных плазмид. Известно, что многие неконъюгативные плазмиды, в том числе и антибиотикорезистентные патогенных бактерий (например, Staphylococcus и Streptococcus), имеющие разные системы репликации, способны стабильно поддерживаться в клетках B. subtilis (Grohmann, 2003). Следовательно, данные повсеместно распространенные и безопасные микроорганизмы при наличии в их клетках конъюгативных плазмид могут играть ключевую роль в распространении генов, в том числе антибиотикорезистентности, среди бактерий разных систематических групп. В настоящее время для бактерий B. subtilis описано лишь две конъюгативные плазмиды (pLS20 и pBS72) (Meijer, 1995; Titok, 2003). Установлено, что плазмиды, содержащие идентичные плазмиде pBS72 системы репликации, содержатся в клетках бактерий B. subtilis, циркулирующих на территории Беларуси, Пакистана и Нидерландов (Гуринович, 2020). Присутствие плазмиды pBS72 повышает устойчивость содержащих ее природных и коллекционных бактерий B. subtilis к стрессовым факторам среды (высокая осмолярность, низкие и высокие значения рН среды, УФ-облучение) (Гуринович, 2020). Для одной из плазмид подобных pBS72 (p19) показана способность эффективно передаваться в клетки близкородственных бактерий и мобилизовать перенос плазмид, содержащих релаксазу семейства MOBV (Poluektova, 2004). Вышеперечисленные свойства плазмиды pBS72 позволяют рассматривать ее в качестве модельного объекта для изучения особенностей организации системы конъюгации.
Целью настоящей работы являлся сравнительный молекулярно-генетический и функциональный анализ системы конъюгации плазмиды pBS72 природных бактерий B. subtilis.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
В работе использовали природные бактерии B. subtilis 16, B. licheniformis FD9, B. vezelensis 7IA3 и B. subtilis 72 из коллекции кафедры микробиологии БГУ, коллекционный штамм B. subtilis 168 (Anagnostopoulos, 1961) и его рифампицинрезистентный вариант (Гуринович, 2020), а также плазмида pBS72, маркированная геном эритромицинрезистентности (Гуринович, 2020), и плазмида pCB16, определяющая устойчивость к тетрациклину (Bernhard, 1978). Бактерии выращивали в полноценной среде LB (Миллер, 1976) или минимальной среде Spizizen (Anagnostopoulos, 1961) при 37°С с аэрацией (240 об./мин). Для роста бактерий B. subtilis 168 на среде Spizizen добавляли триптофан в концентрации 50 мкг/мл. Агаризованные среды содержали 1.5% агара, источником углерода служила глюкоза в концентрации 0.5%. В работе использовали коммерческие препараты тетрациклина, эритромицина, стрептомицина и рифампицина в концентрациях 10, 20, 25 и 100 мкг/мл соответственно.
Тотальную ДНК выделяли саркозиловым методом (te Riele, 1986).
Скрещивание бактерий. В качестве донорных использовали бактерии B. subtilis 72, содержащие маркированную геном эритромицинрезистентности плазмиду pBS72, и B. subtilis 168, содержащие маркированную геном эритромицинрезистентности плазмиду pBS72 и вектор pBC16, детерминирующий устойчивость к тетрациклину. В качестве реципиентов использовали штамм B. subtilis 168 устойчивый к рифампицину (спонтанный мутант) и природные штаммы B. subtilis 16, B. licheniformis FD9 и B. vezelensis 7IA3, устойчивые стрептомицину (устойчивость природного происхождения). Для скрещивания в жидкой среде ночную культуру бактерий, выращенную в среде LB или Spizizen, разводили в 10 раз, соответственно, в свежей среде LB или Spizizen и выращивали с аэрацией в течение 3 или 24 ч. Для скрещивания на плотной среде ночные культуры донора и реципиента в соотношении 1 : 1 наносили на мембранные фильтры, помещенные на поверхность агаризованной среды LB или Spizizen, и культивировали в течении 3 или 24 ч. По истечении 3 или 24 ч скрещивания клетки высевали на селективные среды определенного состава. При переносе плазмиды pBS72 в клетки бактерии B. subtilis 168 трансконъюганты отбирали на полноценной агаризованной среде LB, содержащей в качестве селективного и контрселективного маркеров, соответственно, эритромицин и рифампицин в концентрации 10 и 100 мкг/мл. При переносе плазмиды pBC16 в природные бактерии B. subtilis 16, B. licheniformis FD9 и B. vezelensis 7IA3 трансконъюганты отбирали на минимальной среде Spizizen, не содержащей триптофан, с добавлением стрептомицина в концентрации 25 мкг/мл (контрселективные маркеры) и тетрациклина в концентрации 20 мкг/мл (селективный маркер) с последующим анализом отобранных клонов на наличие маркера эритромицинрезистентности, кодируемого плазмидой pBS72. Частоту переноса определяли отношением числа трансконъюгантов к количеству клеток реципиента. Присутствие в клетках трансконъюгантов плазмид дополнительно устанавливали с использованием ПЦР-анализа.
Для амплификации использовали HF ДНК-полимеразу производства “Thermo Scientific” (ЕС) и праймеры производства ОДО “Праймтех” (Беларусь). Реакционная смесь для ПЦР (50 мкл) содержала около 100 нг ДНК-матрицы, 0.2 ммоль/л каждого дНТФ, 0.3 мкмоль/л каждого праймера, 2.5 ед. ДНК-полимеразы и соответствующий буфер.
Для рестрикционного анализа использовали ферменты рестрикции при режимах, рекомендуемых фирмой изготовителем (“Thermo Scienti-fic”, ЕС).
Для идентификации плазмиды pBS72 амплифицировали фрагмент размером 685 п.н. с использованием праймеров F2 (5'-TAA ATC GCT TCC CTC CTT C-3') и R2 (5'-GTT TCC ACT GTC CAG GAG-3') при режиме амплификации: 94°С – 5 мин (1 цикл); 94°С – 30 с, 50°С – 30 с, 72°С – 42 с (25 циклов); 72°С – 5 мин (1 цикл).
Для идентификации плазмиды pBC16 амплифицировали фрагмент размером 1861 п.н. с праймерами Fmob (5'-GCG CGA TTG CTG AAT AAA AGA TAC-3') и Rmob (5'-CAC TTC AAC GCA CCT TTC AG-3') при режиме амплификации: 94°С – 5 мин (1 цикл); 94°С – 30 с, 50°С – 30 с, 72°С – 1 мин 52 с (25 циклов); 72°С – 5 мин (1 цикл).
Для локализации регуляторных генов rap и phr амплифицировали фрагмент плазмиды pBS72 размером 3269 п.н. с праймерами Frap (5'-AGA GAA ATG GTT CAA TGC CAG-3') и Rrap (5'-ACG GTT AGG AGA ACT CGA C-3') при режиме амплификации: 94°С – 5 мин (1 цикл); 94°С – 30 с, 52°С – 30 с, 72°С – 3 мин 20 с (25 циклов); 72°С – 5 мин (1 цикл) с последующим рестрикционным анализом ферментом рестрикции SacI, Eco53k1, AflIII.
Для локализации гена rco амплифицировали фрагмент плазмиды pBS72 размером 2428 п.н. с праймерами Frco (5'-TTC CCT TTC CTC GTC ATC G-3') и Rrco (5'-TCG CTC ACT ACG ACT TTT AG-3') при режиме амплификации: 94°С – 5 мин (1 цикл); 94°С – 30 с, 52°С – 30 с, 72°С – 2 мин 25 с (25 циклов); 72°С – 5 мин (1 цикл) с последующим рестрикционным анализом ферментом рестрикции EcoRV, XhoII, NdeI.
Для анализа генов и детерминируемых ими белков, входящих в состав плазмиды pBS72 (регистрационный номер в ГенБанке NCBI – KX711616) и плазмиды pLS20 (регистрационный номер в ГенБанке NCBI – AB615352), использовали программу BLASTN2.10.0 (сайт: https://blast.ncbi.nlm.nih.gov).
Построение филогенетических деревьев полноразмерных аминокислотных последовательностей осуществляли методом ближайших соседей (1000 бутстрепов) в программе MEGA7 (Kumar, 2016).
Поиск гомологичных плазмиде pBS72 белков конъюгации осуществляли в базе данных NCBI с использованием программы psi-blast. Для сравнения использовали типовые аминокислотные последовательности белков плазмид pTiBo542 (MPFT), R64 (MPFI), R27 (MPFF), ICEhin1056 (MPFG), pCW3 (MPFFA), pGO1 (MPFFATA), pCC7120alpha (MPFC) и транспозона CtnDOT (MPFB). С помощью программы USEARCH (версия 11.0.667_i86linux32) для построения филогенетических деревьев использовали аминокислотные последовательности, отличающиеся между собой более чем на 50% (Edgar, 2010).
Аминокислотные последовательности гомологичных белков выравнивали с помощью программы Clustal Omega (сайт https://www.ebi.ac.uk/ Tools/common/tools/help/index.html?tool=clustalo), для графического изображения которых использовали программу JalView.
Поиск функциональных доменов проводили с помощью программ TMHMM v2.0, MOTIF Search и Phyre2 сервера Hhpred и баз данных NCBI и Uni-prot.
Для сравнительного анализа структурных и регуляторных генов, определяющих белки конъюгации, использовали депонированные в ГенБанк NCBI нуклеотидные последовательности бактерий B. subtilis MB378 (MBPE01000006), B. subtilis MB415 (MQSR01000011), B. subtilis DH12 (NZ_RQPH01000007), B. subtilis EH11 (NZ_RPHE01000007), B. subtilis 18-3 (NZ_N-JRE01000013), B. subtilis B4071 (NZ_-JXHN01000026, NZ_JXHN01000027, NZ_-JXHN01000031), B. rugosus SPB7 (NZ_-JABUXO010000030), B. licheniformis YNP5-SUWRT508 (MEDD01000008), B. licheniformis YNP1-TSU (MIGE01000004), B. paralicheniformis G-1 (NZ_AZSK01000003, NZ_AZSK01000013), Bacillus sp. CX-120 (NZ_NCTZ01000020) и полные нуклеотидные последовательности плазмиды SX01705-1 бактерий B. subtilis (CP022288) и плазмиды 1 бактерий B. licheniformis (CP035229).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Наличие полной нуклеотидной последовательности плазмиды pBS72 позволило провести сравнительный анализ генов и детерминируемых ими белков конъюгации с известными. Плазмида pBS72, размером 102 254 п.н., является самым крупным внехромосомным генетическим элементом, выявленным в клетках бактерий B. subtilis. В состав ее генома входит 142 открытые рамки считывания, из которых 49 кодируют белки с определенными функциональными свойствами, остальные (93) определяют синтез гипотетических полипептидов. Около 30% плазмидного репликона представлено генами, предположительно определяющими белки конъюгации (orf14–оrf 51), среди которых выявленные белки VirB4 (orf 31), VirB11 (orf 26), Mob (orf 37) и VirD4 (or f24) являются ключевыми и используются для характеристики и классификации систем конъюгации внехромосомных генетических элементов грамположительных и грамотрицательных бактерий (Guglielmini et al., 2014; Ramachandran, 2017).
На первом этапе осуществляли поиск pBS72-подобных плазмид на основании сходства белков VirB4, VirB11 и VirD4 с известными. В результате данные белки, идентичные на 98–100% таковым плазмиде pBS72, были выявлены у природных бактерий B. subtilis, выделенных из загрязненной нефтью почвы на территории Пакистана (штаммы MB378 и MB415), из луговой почвы в Нидерландах (штаммы EH11 и DH12), из кари супа в Нидерландах (штамм B4071), коммерческого штамма B. subtilis, использующегося в качестве пробиотического препарата для животных в Китае (штамм 18-3), природных бактерий B. rugosus, изолированных из морской воды на территории Индии (штамм SPB7). Гомологи VirB4, VirB11 и VirD4, идентичные на 60–80% плазмиде pBS72, обнаружены у бактерий Bacillus sp., выделенных из компоста в Китае (штамм CX-120), B. licheniformis, изолированных из травы в парках США (штаммы YNP5-SUWRT508 и YNP1-TSU), B. paralicheniformis, выделенных из сухого молока в Австралии (штамм G-1), а также плазмиды SX01705-1 бактерий B. subtilis, изолированных из субстрата для выращивания грибов в Китае и плазмиды 1 бактерий B. licheniformis, выделенных из пищевого продукта в Южной Корее. Ранее для ряда вышеперечисленных геномов выявлены rep- и mob-гены, кодирующие, соответственно, белки инициации репликации и релаксазы, идентичные плазмиде pBS72 на 99–100 или 62% (Гуринович, 2020). Полученные данные однозначно свидетельствовали о присутствии в клетках внехромосомных генетических элементов. Следует отметить, что геномы всех вышеперечисленных бактерий депонированы в ГенБанк NCBI в виде отдельных контигов, анализ которых позволил установить, что некоторые из них можно представить в виде кольцевых молекул ДНК (фрагменты одной и той же рамки считывания присутствовали на 5'- и 3'-концах) и, следовательно, определить размер плазмидных репликонов. В частности, в клетках бактерий B. subtilis MB378 и B. subtilis MB415, выделенных на территории Пакистана, присутствовали плазмиды размером 88 605 и 94 937 п.н. соответственно, а в клетках бактерий B. subtilis EH11 и B. subtilis DH12, выделенных на территории Нидерландов – плазмиды более крупного размера –122 937 и 117 972 п.н. соответственно. Для остальных бактерий размер внехромосомных генетических элементов установить не представлялось возможным, поскольку их контиги не перекрывались. Среди обнаруженных плазмидсодержащих штаммов определенный интерес представляли бактерии B. rugosus, изолированные из морской воды на территории Индии, поскольку все детерминанты, кодирующие белки инициации репликации и конъюгации, были идентичны таковым плазмиде pBS72 на 100%.
Таким образом, на основании полученных данных можно утверждать, что бактерии рода Bacillus, содержащие pBS72-подобные плазмиды, широко распространены по всему миру (Беларусь, Нидерланды, Китай, Индия, Пакистан, Южная Корея, США, Австралия). При этом, наиболее сходные плазмиде pBS72 репликоны выявлены в клетках B. subtilis (исключение составил штамм, идентифицированный как B. rugosus). Данный факт является весьма примечательным и обосновывает детальный анализ систем конъюгации данных внехромосомных генетических элементов.
Как указывалось выше, консервативные белки VirB4, MOB и VirD4 являются маркерными и используются для классификации конъюгативных плазмид. Ранее был проведен анализ функциональных доменов релаксазы плазмиды pBS72 и установлено, что они имеют наибольшее сходство с таковыми плазмиды pLS20 группы MOBL, но полная аминокислотная последовательность белка достоверно отличалась от релаксазы pLS20 (сходство составляло 34.93%) и представлена отдельной филогенетической группой (Гуринович, 2020).
В ходе настоящей работы был проведен сравнительный молекулярно-генетический и филогенетический анализ белков VirB4, VirB11, VirD4, VirB6 плазмиды pBS72. Предполагали, что детальный анализ достаточно большой выборки белковых молекул позволит более точно охарактеризовать систему конъюгации плазмид, подобных pBS72, и определить их место в существующей системе классификации.
Анализ белка VirB4 (orf31) плазмиды pBS72 позволил выявить в N- и С-концевой части молекулы консервативные аминокислотные последовательности, определяющие характерную для данных белков АТФ-азную активность (Walker A и Walker B, Мотив D и E). В то же время отсутствовали характерные для данного типа белков трансмембранные домены, что могло свидетельствовать о неспособности белка VirB4 плазмиды pBS72 непосредственно взаимодействовать с белками, входящими в состав секреторного канала, через который передается однонитевая молекула ДНК (Sgro, 2019). Возможно, подобно некоторым другим системам конъюгации данный белок обеспечивал связывание с внутренней мембраной посредством взаимодействий с другими компонентами системы секреции IV-типа (Green, 2016). Сравнительный анализ функциональных доменов белков VirB4 с АТФ-азной активностью, кодируемых конъюгативными плазмидами разных систематических групп, позволил установить, что они характеризуются полиморфностью и содержат ряд несинонимических замен (рис. 1).
Рис. 1.
Сравнительный анализ функциональных доменов белков VirB4 плазмиды pBS72 и pLS20 с известными представителями классификационных групп MPFT (плазмида pTiBo542), MPFI (плазмида R64), MPFF (плазмида R27), MPFG (плазмида ICEhin1056 MPFFATA), MPFFA (плазмида pCW3), MPFFATA (плазмида pGO1), MPFC (плазмида pCC7120alpha), MPFB (транспозон CtnDOT). Стрелками на рисунке обозначены отсутствующие локусы с множественными делециями. Цветом выделены идентичные и консервативные аминокислоты.

Филогенетический анализ белков VirB4 позволил установить, что наиболее сходные полипептиды кодируются плазмидой pLS20. Однако, несмотря на 95% покрытие, данные белки достоверно отличались друг от друга (сходство составляло 35.32%) и входили в разные филогенетические группы. Филогенетически близкими данным полипептидам являлись белки VirB4 конъюгативных плазмид семейства MPFFATA, присутствующих в клетках, удаленных в таксономическом отношении микроорганизмов (Firmicutes, Actinobacteria, Tenericutes и Archaea) (рис. 2).
Особый интерес представлял белок VirB11 плазмиды pBS72, кодируемый orf 26, являющийся достаточно уникальным и характерным для 20% известных Mpf-систем. Только внехромосомные генетические элементы семейств MPFT (Ti-плазмида) и MPFI (плазмида R64) детерминировали синтез данных полипептидов. Он не обнаружен в системах секреции IV-типа, кодируемых F-плазмидой E. coli и плазмидами грамположительных бактерий и архей (Christie, 2017). Белки VirB11 являются членами AAA-суперсемейства АТФ-аз. Локализуясь в цитоплазме клеток, они способны временно взаимодействовать с белками секреции в ответ на связывание субстрата или другой сигнал (Sexton, 2004). Анализ аминокислотной последовательности белка VirB11 плазмиды pBS72 выявил наличие консервативных доменов с АТФ-азной активностью (Walker A, Walker B), а также Asp- и His-доменов, определяющих связывание молекул ДНК (Sagulenko, 2001). Кроме плазмиды Ti и RP4, гомологичный белок VirB11 с неполным покрытием (74%) и сходством, не превышающим 36%, характерен для плазмиды pLS20 (код доступа BAJ76915.1 в ГенБанке NCBI). Сравнительный анализ функциональных доменов белков VirB11 плазмиды pBS72 и pLS20 выявил наличие несинонимических замен во всех функционально значимых участках. При этом наиболее консервативным являлся His-домен, в котором выявлялись только функционально незначимые аминокислотные замены. Все остальные домены содержали значимые для функциональной активности изменения. Наиболее выраженным полиморфизмом характеризовались междоменные локусы (рис. 3). Филогенетический анализ позволил установить, что белок VirB11, кодируемый плазмидой pBS72, представлен отдельной филогенетической группой, достоверно отличающейся от гомологичных белков, кодируемых плазмидой pLS20 и внехромосомными генетическими элементами семейств MPFT и MPFI (рис. 4).
Рис. 3.
Сравнительный анализ функциональных доменов белков VirB11 плазмид pBS72, pLS20 и представителей семейств MPFT и MPFI, MPFI. Цветом выделены идентичные и консервативные аминокислоты.

Анализ белка VirD4 (orf24) обнаружил типичное строение, характерное для белков T4CP. В N‑концевой последовательности данного белка локализовалось два трансмембранных домена (NTD) из 34 аминокислот, способных формировать периплазматическую петлю, обеспечивающую взаимодействие с секреторными белками. Центральная часть белка включала нуклеотид-связывающий домен (NBD) и мотивы Walker A и Walker B, обладающие АТФ-азной активностью. C-концевая последовательность белка содержала нуклеотид-связывающий домен (NBD) и вариабельный альфа-домен (AAD), обеспечивающий связывание субстратов (Li, 2018) (рис. 5). Анализ белка VirD4 плазмиды pBS72 позволил установить, что наибольшее сходство в области функционально значимых участков данный белок проявлял с гомологичным полипептидом плазмиды pLS20. Однако их полные аминокислотные последовательности достоверно отличались друг от друга (покрытие составило 79%, сходство не превышало 33.89%) и входили в разные филогенетические группы. Следует отметить, что белки VirD4 плазмид pBS72 и pLS20 имели наибольшее филогенетическое сходство с гомологичными белками плазмид семейств MPFТ и MPFFATA (рис. 6).
Рис. 5.
Сравнительный анализ функциональных доменов белков VirD4 системы секреции IV типа плазмид разных систематических групп. Стрелками на рисунке обозначены отсутствующие локусы с множественными делециями. Цветом выделены идентичные и консервативные аминокислоты.

Рис. 6.
Филогенетическое дерево белков VirD4 (белок VirD4 плазмиды pBS72 и плазмиды pLS20 выделены подчеркиванием).

Таким образом, сравнительный анализ консервативных белков конъюгации VirB4 и VirD4, кодируемых плазмидой pBS72, позволил установить, что они наиболее сходны с плазмидой pLS20 и внехромосомными генетическими элементами классификационной группы MPFFATA или MPFFATA и MPFТ соответственно. В то же время гомологичный белок VirB11 имел наибольшее сходство только с плазмидами, подобными pLS20 (рис. 3). В связи с этим, анализ других белков проводили в сравнении с таковыми плазмиды pLS20. Для этого анализировали белки, связанные с поверхностными структурами, которые, как известно, являются крайне специфическими, и наличие сходства между ними может однозначно свидетельствовать в пользу филогенетического родства.
Поиск белков, образующих секреторный канал, позволил обнаружить полипептид, кодируемый открытой рамкой считывания orf28 плазмиды pBS72, способный выполнять данные функции. В пределах N-концевой последовательности этого достаточно крупного белка было выявлено 7 трансмембранных доменов, характерных для белка VirB6 системы секреции IV типа. Гомологичный белок, имеющий сходные функционально значимые аминокислотные последовательности (шесть трансмембранных доменов), выявлен для плазмиды pLS20 (orf28). При этом данные белки в значительной степени отличались друг от друга (покрытие составило 43%, сходство не превышало 30.28%), и сходство обнаруживалось только в области трансмембранных доменов.
Для плазмиды pLS20 был показано, что ген orf11 детерминирует белок адгезии (Adg), обеспечивающий контакт между донорскими и реципиентными клетками (Gago-Córdoba, 2021). Поиск гомологичного белка для плазмиды pBS72 позволил установить, что полипептид, обладающий сходными свойствами, кодируется открытой рамкой считывания orf14. В N-концевой части данного белка был выявлен трансмембранный домен (между 12 и 29 аминокислотой) и сигнальная последовательность, содержащая между 37 и 38 аминокислотными остатками сайт VEA-AS, способный расщепляться пептидазой 1. Наличие данных последовательностей свидетельствовало о выведении данного белка за пределы клетки (Sec-зависимый путь секреции). Однако функционально значимые аминокислотные последовательности, характерные для поверхностных белков адгезии, имели крайне низкое сходство с таковыми плазмиды pLS20. Сходство MucBP-, Cna- и LPXTG-доменов составило 33, 20 и 28% соответственно.
Таким образом, на основании филогенетического и сравнительного анализа функциональных доменов белков VirB4, VirB11, VirB6, VirD4 и Adg с белками внехромосомных генетических элементов известных классификационных групп можно утверждать, что система конъюгации плазмид, подобных pBS72, наиболее близка по организации плазмиде pLS20. Однако присутствие несинонимических замен в функционально значимых последовательностях и практически отсутствие сходства между полными аминокислотными последовательностями исследуемых белков, приводящих к достоверным филогенетическим отличиям, позволяют считать, что конъюгационная система плазмиды pBS72 относится к отдельной систематической группе (обозначена как MpfS). Отличительной особенностью конъюгационной системы плазмид, подобных pBS72, является ее сходство с таковыми плазмид бактерий группы Firmicutes (подобных pLS20 и MPFFATA), что представляется весьма логичным и может свидетельствовать об их едином происхождении. Наличие значимых изменений в белках конъюгации, свидетельствует о процессах дивергенции, которые могли быть обусловлены, в том числе, особенностями их копирования в бактериальных клетках. Следует отметить, что плазмиды pLS20 и pBS72 имеют разные системы инициации репликации (Titok et al., 2003).
Ранее было установлено, что плазмида р19, содержащая абсолютно идентичную плазмиде pBS72 систему конъюгации (регистрационный номер в ГенБанке NCBI FJ434456), передавалась в жидкой среде LB с частотой 10–3 в бактерии B. subtilis 168 и с такой же частотой обеспечивала мобилизационный перенос плазмиды pUB110, содержащей релаксазу mobV-типа. При этом частота переноса плазмиды р19 увеличивалась на два порядка при отсутствии в клетках реципиента системы рестрикции‒модификации. В то же время частота мобилизации плазмиды pUB110 сохранялась на прежнем уровне (10–3). Однако в изогенной системе скрещивания (между природным плазмидсодержащим штаммом и его бесплазмидным вариантом) частота переноса плазмиды р19 возрастала до 10–1, а частота мобилизации плазмиды pUB110 по-прежнему сохранялась на уровне 10–3. Во всех скрещиваниях максимальную частоту фиксировали через 3 ч, когда скрещиваемые бактерии находились в логарифмической фазе роста (Poluektova, 2004). Несколько иная картина наблюдалась при изучении кинетики переноса плазмиды pLS20. В изогенной системе скрещивания (между B. subtilis nato) она передавалась с максимальной частотой 10–3‒10–4 в узком временном диапазоне. После 3 ч скрещивания в жидкой среде LB частота переноса плазмиды pLS20 снижалась до значений 10–7–10–8 и возрастала до 10−2 в отсутствии белков репрессоров Rco и Phr, а также при увеличенной экспрессии белка Rap (Singh, 2013). На основании функционального анализа генов rap, prh и rco была предложена модель регуляции конъюгации плазмиды pLS20 (Meijer, 2021). Согласно данной модели, конъюгационный перенос плазмиды pLS20 негативно регулировался продуктом гена rco. Репрессия снималась белком Rap путем его взаимодействия с белком Rco. Однако через 3 ч скрещивания в результате увеличения концентрации белка Phr происходила инактивация позитивного регулятора Rap и, как следствие, частота конъюгационного переноса снижалась до предельно низких значений 10–7–10–8. Приведенные данные могут свидетельствовать в пользу разной функциональной активности систем конъюгации плазмид pLS20 и pBS72. Для проверки этого предположения был проведен сравнительный анализ регуляторных белков, кодируемых генами rco, rap и phr, а также определены частоты переноса плазмиды pBS72 и ее способность мобилизовать перенос плазмиды pBC16 в средах разного состава в зависимости от времени.
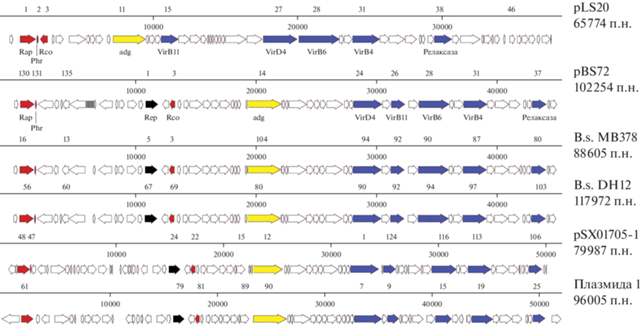
В результате было установлено, что в геноме плазмид pLS20 и pBS72 регуляторные гены rco, rap и phr имеют разную локализацию. Все эти регуляторные детерминанты в плазмиде pLS20 локализованы в непосредственной близости от tra-оперона. В частности, промотор негативного регулятора rco перекрывался с промотором первого гена системы tra, а противоположно ему транскрибируемые с одного промотора гены rap и phr локализовались непосредственно выше по течению. В плазмиде pBS72 данные детерминанты присутствовали в разных частях генома. При этом гомология между регуляторными белками плазмид pLS20 и pBS72 составила 40.21% для белка Rco (покрытие 86%) и 24.41% для белка Rap (покрытие 98%), а сходство между белками Phr отсутствовало. Для подтверждения локализации регуляторных генов и исключения ошибки при сборке полной нуклеотидной последовательности плазмиды pBS72 с использованием полимеразной цепной реакции были амплифицированы фрагменты плазмиды pBS72, в пределах которых находились гены rap (orf130), phr (orf132) и rco (orf3). В результате были получены ампликоны искомого размера, рестрикционный анализ которых позволил подтвердить локализацию исследуемых локусов, соответствующую аннотированной нуклеотидной последовательности, депонированной в ГенБанк NCBI. Дополнительным доказательством выявленных отличий являлась одинаковая локализация регуляторных генов rap, phr и rco в составе pBS72-подобных плазмид бактерий B. subtilis MB378, MB415, EH11, DH12 и B. rugosus SPB7, а также плазмиды SX01705-1 бактерий B. subtilis и плазмиды 1 бактерий B. licheniformis (рис. 7). Следует отметить, что в составе плазмиды pBS72 между регуляторными генами rap, phr и генами tra, помимо дополнительных открытых рамок считывания, выявлено 14 повторов СTAC (размером 46 п.н.), расположенных в межгенной области между генами orf136 и orf137. Такие же повторы присутствовали в составе pBS72-подобных плазмид. Наличие повторяющихся последовательностей является примечательным фактом, который требует дальнейшего изучения.
Рис. 7.
Схема генетической организации локусов, содержащих гены, определяющие белки регуляции конъюгации Rco, Rap, Phr (выделены красным цветом), конъюгационного переноса VirB4, VirB11, VirB6, VirD4, релаксазы (выделены синим цветом), адгезии (выделены желтым цветом) и инициации репликации Rep (выделены черным цветом). Номерами обозначены открытые рамки считывания.
Поскольку разное расположение регуляторных генов в составе плазмид pLS20 и pBS72 не может являться прямым доказательством разной функциональной активности их систем конъюгации, была изучена кинетика конъюгационного и мобилизационного переноса, детерминированного плазмидой pBS72. При этом ставилась задача определить способность pBS72 передаваться в бактерии B. subtilis 168 и обеспечивать перенос плазмиды pBC16 в бактерии рода Bacillus при разных условиях (плотная полноценная и минимальная среды или жидкая полноценная и минимальная среды) в зависимости от продолжительности скрещивания (3 и 24 ч соответственно). Предполагали, что в случае функционального сходства систем конъюгации плазмид pBS72 и pLS20, частота конъюгационного переноса должна снижаться при увеличении времени скрещивания вне зависимости от состава среды. При этом наибольшее снижение частоты переноса ожидали при достижении донорными и реципиентными бактериями стационарной фазы роста (через 24 ч скрещивания в жидкой среде или через 3‒24 ч скрещивания на плотной среде). Предполагали, что при таких условиях в клетках донора должны накапливаться регуляторные белки, ключевым из которых, согласно предложенной модели регуляции конъюгации плазмиды pLS20, являлся белок Phr, увеличение концентрации которого приводило к ингибированию конъюгационного переноса (Singh, 2013; Meijer, 2021).
В результате проведенных скрещиваний было установлено, что частота переноса исходной плазмиды pBS72 в клетки бактерий B. subtilis 168 в жидкой и на плотной среде LB через 24 ч снижалась в 100 раз (через 3 ч, соответственно, составляла 2.6 × 10–3 и 3.5 × 10–4, а через 24 ч – 2.6 × 10–5 и 6.3 × 10–6). Однако при скрещивании в жидкой и на плотной минимальной среде Spizizen частота переноса pBS72 через 24 ч увеличивалась в 5–10 раз (через 3 ч, соответственно, составляла 2.0 × 10–6 и 6.88 × 10–5, а через 24 ч – 2.1 × 10–5 и 1.7 × 10–4) (табл. 1). Полученные данные свидетельствовали в пользу того, что частота переноса в большей степени зависела не от времени скрещивания, а от состава среды.
Таблица 1.
Частота переноса плазмиды pBS72 в клетки бактерий B. subtilis 168 при разных условиях скрещивания
| Донор | Условия скрещивания | Частота переноса плазмиды pBS72 | ||
|---|---|---|---|---|
| среда | время скрещивания, ч | в жидкой среде | на плотной среде | |
| B. subtilis 72 | Spizizen | 3 | 1.8 × 10–5 | 9.8 × 10–5 |
| 24 | 2.1 × 10–5 | 1.7 × 10–4 | ||
| LB | 3 | 2.6 × 10–3 | 3.5 × 10–4 | |
| 24 | 2.6 × 10–5 | 6.3 × 10–6 | ||
Для анализа частоты мобилизационного переноса в качестве донора использовали штамм B. subtilis 168, содержащий, соответственно, конъюгативную и мобилизуемую плазмиды pBS72 и pBC16. В качестве реципиентов использовали природные бактерии рода Bacillus, активно синтезирующие антимикробные и поверхностно-активные соединения (B. subtilis 16, B. licheniformis FD9, B. am-yloliquefaciens 7IA3). Частоту переноса плазмиды pBC16 определяли при разных условиях и продолжительности скрещивания. В результате было установлено, что при увеличении времени скрещивания в жидкой среде LB и Spizizen частота переноса плазмиды pBC16 через 24 ч скрещивания либо не менялась, либо возрастала в 10–100 раз. При скрещивании на плотной среде, вне зависимости от ее состава, перенос плазмиды pBC16 через 3 ч либо не регистрировался, либо происходил с гораздо более низкой частотой (частота переноса через 24 ч увеличивалась в 10–100 раз) (табл. 2). На основании полученных данных можно заключить, что частота мобилизационного переноса плазмиды pBC16 зависела от условий скрещивания (плотность и состав среды) и видовой принадлежности бактерий реципиентов.
Таблица 2.
Частота мобилизационного переноса плазмиды pBC16 в клетки бактерий B. subtilis d16, B. velezensis 7I3A и B. licheniformes FD9 при разных условиях скрещивания
| Штамм реципиент | Условия скрещивания | Частота переноса плазмиды pBC16 | ||
|---|---|---|---|---|
| среда | время скрещивания. ч | в жидкой среде | на плотной среде | |
| B. subtilis d16 | Spizizen | 3 | 5.0 × 10–7 | 0 |
| 24 | 9.9 × 10–7 | 7.5 × 10–6 | ||
| LB | 3 | 6.3 × 10–6 | 1.8 × 10–7 | |
| 24 | 6.1 × 10–6 | 4.0 × 10–6 | ||
| B. licheniformes FD9 | Spizizen | 3 | 9.6 × 10–6 | 1.3 × 10–6 |
| 24 | 2.4 × 10–6 | 1.2 × 10–4 | ||
| LB | 3 | 1.1 × 10–4 | 6.8 × 10–5 | |
| 24 | 2.5 × 10–4 | 2.6 × 10–3 | ||
| B. velezensis 7I3A | Spizizen | 3 | 5.5 × 10–7 | 0 |
| 24 | 2.5 × 10–6 | 2.9 × 10–5 | ||
| LB | 3 | 4.2 × 10–6 | 0 | |
| 24 | 4.7 × 10–4 | 1.8 × 10–4 | ||
Примечание. В конъюгационных скрещиваниях в качестве донора использовали типовой штамм B. subtilis 168, содержащий конъюгативную плазмиду pBS72 и мобилизуемую плазмиду pBC16; частота переноса определялась относительно количества клеток реципиента. Трансконъюганты, отобранные на среде, содержащей тетрациклин, дополнительно проверялись на присутствие мобилизующей плазмиды pBS72 и мобилизуемой плазмиды pBC16. В результате установлено, что все они содержали плазмиду pBC16 (в реакции ПЦР с использованием специфических праймеров, обеспечивающих амплификацию гена mob, регистрировали образование ампликона искомого размера 1861 п.н.) и не содержали плазмиду pBS72 (чувствительны к эритромицину, в реакции ПЦР не регистрировали образование специфических продуктов амплификации размером 685 п.н. при использовании праймеров, комплементарно связывающихся в области плазмиды pBS72 между генами orf138 и orf139).
На основании приведенных данных можно утверждать, что генетическая организация и функциональная активность системы конъюгации плазмиды pBS72 является уникальной для грамположительных бактерий. Подтверждением этому являются результаты, полученные для pBS72-подобной плазмиды р19, которая в изогенной системе скрещивания передавалась с частотой 10–1 (Poluektova, 2004). Только для плазмид с дерепрессированной системой конъюгации (например, F-плазмида) характерна такая высокая частота переноса.
Таким образом, в результате проведенного анализа было установлено, что генетическая организация и функциональная активность системы конъюгационного переноса плазмиды pBS72 является уникальной. Несмотря на сходство с ключевыми структурными и регуляторными белками конъюгации плазмиды pLS20 в области функционально значимых доменов, она характеризовалась рядом отличий. Во-первых, сходство ключевых белков конъюгации ограничивалось областью функциональных доменов, в остальном белки имели значительные отличия (гомология не превышала 40%). Во-вторых, гены, детерминирующие предположительно регуляторные белки (Rco, Rap, Phr), имели разную локализацию в плазмиде pLS20 и pBS72 (соответственно, находились в непосредственной близости от tra-оперона, либо в разных локусах генома). В-третьих, у плазмид pBS72 и pLS20 отсутствовало сходство между регуляторными белками Phr. И, наконец, в отличие от плазмиды pLS20 функциональная активность конъюгационной системы плазмиды pBS72 не зависела напрямую от времени скрещивания, а определялась составом среды и генетическими особенностями бактерий реципиентов. Проведенный анализ позволяет отнести систему конъюгации плазмиды pBS72 к новому семейству (обозначено как MpfS) и является основой для изучения механизма распространения pBS72-подобных плазмид в природной среде обитания и создания на их основе системы генетического анализа биотехнологически значимых бактерий рода Bacillus.
Список литературы
Гуринович А.С., Титок М.А. Молекулярно-генетический и функциональный анализ плазмиды pBS72 природных бактерий Bacillus subtilis // Микробиология. 2020. Т. 89. С. 646–657.
Gurinovich A.S., Titok M.A. Molecular genetic and functional analysis of the pBS72 plasmid from Bacillus subtilis environmental isolates // Microbiology (Moscow). 2020. V. 89. P. 660‒669.
Миллер Дж. Эксперименты в молекулярной генетике / Пер. с англ.: Зографа Ю.Н. и др.. Под ред. и с предисл. Алиханяна С.И. М.: Мир, 1976. 436 с.
Alvarez-Rodriguez I., Arana L., Ugarte-Uribe B., Gomez-Rubio E., Martin-Santamaria S., Garbisu C., Alkorta I. Type IV coupling proteins as potential targets to control the dissemination of antibiotic resistance // Front. Mol. Biosci. 2020. V. 7. P. 201. https://doi.org/10.3389/fmolb.2020.00201
Anagnostopoulos C., Spizizen J. Requirements for transformation in Bacillus subtilis // J. Bacteriol. 1961. V. 81. P. 741–746.
Bernhard K., Schrempf H., Goebel W. Bacteriocin and antibiotic resistance plasmids in Bacillus cereus and Bacillus subtilis // J. Bacteriol. 1978. V. 133. P. 897–903.
Cascales E., Christie P.J. The versatile bacterial type IV secretion systems // Nat. Rev. Microbiol. 2003. V. 1. P. 137–149.
Christie P.J., Gomez V.L., Buchrieser C. Biological diversity and evolution of type IV secretion systems // Curr. Top. Microbiol. Immunol. 2017. V. 413. P. 1–30.
Davies J., Davies D. Origins and evolution of antibiotic resistance // Microbiol. Mol. Biol. Rev. 2010. V. 74. P. 417–433.
Edgar R.C. Search and clustering orders of magnitude faster than BLAST // Bioinform. 2010. V. 26. P. 2460–2461.
Gago-Cordoba C., Val-Calvo J., Abia D., Diaz-Talavera A., Miguel-Arribas A., Aguilar Suarez R., van Dijl J.M., Wu L.J., Meijer W.J.J. A conserved class II type thioester domain-containing adhesin is required for efficient conjugation in Bacillus subtilis // mBio. 2021. V. 12. e00104-21. https://doi.org/10.1128/mBio.00104-21
Green E.R., Mecsas J. Bacterial secretion systems: an overview // Microbiol. Spectr. 2016. V. 4. https://doi.org/10.1128/microbiolspec.VMBF-0012-2015
Grohmann E., Keller W., Muth G. Mechanisms of conjugative transfer and Type IV secretion-mediated effector transport in gram-positive bacteria // Curr. Top. Microbiol. Immunol. 2017. V. 413. P. 115–141.
Grohmann E., Muth G., Espinosa M. Conjugative plasmid transfer in gram-positive bacteria // Microbiol. Mol. Biol. Rev. 2003. V. 67. P. 277–301.
Guglielmini J., Neron B., Abby S.S., Garcillan-Barcia M.P., de la Cruz F., Rocha E.P. Key components of the eight classes of type IV secretion systems involved in bacterial conjugation or protein secretion // Nucleic Acids Res. 2014. V. 42. P. 5715–5727.
Harwood C.R., Mouillon J.M., Pohl S., Arnau J. Secondary metabolite production and the safety of industrially important members of the Bacillus subtilis group // FEMS Microbiol. Rev. 2018. V. 42. P. 721–738.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. EV. 2016. V. 33. P. 1870–1874.
Li Y.G., Christie P.J. The Agrobacterium VirB/VirD4 T4SS: mechanism and architecture defined through in vivo mutagenesis and chimeric systems // Curr. Top. Microbiol. Immunol. 2018. V. 418. P. 233–260.
Meijer W.J., de Boer A.J., van Tongeren S., Venema G., Bron S. Characterization of the replication region of the Bacillus subtilis plasmid pLS20: a novel type of replicon // Nucleic Acids Res. 1995. V. 23. P. 3214–3223.
Meijer W.J.J., Boer D.R., Ares S., Alfonso C., Rojo F., Luque-Ortega J.R., Wu L.J. Multiple layered control of the conjugation process of the Bacillus subtilis plasmid pLS20 // Front. Mol. Biosci. 2021. V. 8. P. 648468. https://doi.org/10.3389/fmolb.2021.648468
Poluektova E.U., Fedorina E.A., Lotareva O.V., Prozorov A.A. Plasmid transfer in bacilli by a self-transmissible plasmid p19 from a Bacillus subtilis soil strain // Plasmid. 2004. V. 52. P. 212–217.
Ramachandran G., Miguel-Arribas A., Abia D., Singh P.K., Crespo I., Gago-Córdoba C., Hao J.A., Luque-Ortega J.R., Alfonso C., Wu L.J., Boer D.R., Meijer W.J. Discovery of a new family of relaxases in Firmicutes bacteria // PLoS Ge-net. 2017. V. 13. e10065.
Sagulenko E., Sagulenko V., Chen J., Christie P.J. Role of Agrobacterium VirB11 ATPase in T-pilus assembly and substrate selection // J. Bacteriol. 2001. V. 183. P. 5813‒5825.
Sexton J.A., Pinkner J.S., Roth R., Heuser J.E., Hultgren S.J., Vogel J.P. The Legionella pneumophila PilT homologue DotB exhibits ATPase activity that is critical for intracellular growth // J. Bacteriol. 2004. V. 186. P. 1658–1666.
Sgro G.G., Oka G.U., Souza D.P., Cenens W., Bayer-Santos E., Matsuyama B.Y., Bueno N.F., Dos Santos T.R., Alvarez-Martinez C.E., Salinas R.K., Farah C.S. Bacteria-killing type IV secretion systems // Front. Microbiol. 2019. V. 10. P. 1078. https://doi.org/10.3389/fmicb.2019.01078
Singh P.K., Ramachandran G., Ramos-Ruiz R., Peiro-Pastor R., Abia D., Wu L.J., Meijer W.J. Mobility of the native Bacillus subtilis conjugative plasmid pLS20 is regulated by intercellular signaling // PLoS Genet. 2013. V. 9. e1003892. https://doi.org/10.1371/journal.pgen.1003892
te Riele H., Michel B., Ehrlich S.D. Single-stranded plasmid DNA in Bacillus subtilis and Staphylococcus aureus // Proc. Natl. Acad. Sci. USA. 1986. V. 83. P. 2541–2545.
Titok M.A., Chapuis J., Selezneva Y.V., Lagodich A.V., Prokulevich V.A., Ehrlich S.D., Jannière L. Bacillus subtilis soil isolates: plasmid replicon analysis and construction of a new theta-replicating vector // Plasmid. 2003. V. 49. P. 53–62.
Yin X., Stotzky G. Gene transfer among bacteria in natural environments // Adv. Appl. Microbiol. 1997. V. 45. P. 153–212.
Дополнительные материалы отсутствуют.




