Микробиология, 2022, T. 91, № 5, стр. 537-546
Филогения и свойства новых штаммов Pseudomonas sp. из ризосферы бобовых растений Южного Урала
О. В. Чубукова a, *, Л. Р. Хакимова a, Е. С. Акимова a, З. Р. Вершинина a
a Институт биохимии и генетики – обособленное структурное подразделение
Федерального государственного бюджетного научного учреждения Уфимского федерального исследовательского центра Российской академии наук
450054 Уфа, Россия
* E-mail: chubukova@bk.ru
Поступила в редакцию 19.03.2022
После доработки 27.04.2022
Принята к публикации 28.04.2022
- EDN: RPUMJS
- DOI: 10.31857/S0026365622100196
Аннотация
Многие представители рода Pseudomonas обладают ростостимулирующими свойствами и способностью к биологическому контролю, что позволяет использовать их для создания биопрепаратов и экологически безопасных удобрений. Целью данной работы было выделение новых штаммов псевдомонад из ризосферы бобовых растений Южного Урала и исследование их свойств. Проведен молекулярно-генетический анализ данных бактериальных штаммов. По результатам исследования нуклеотидной последовательности генов 16S pРНК и rpoD было выявлено 4 штамма, относящихся к роду Pseudomonas. Один из них, Pseudomonas sp. OBA 2.4.1, по уровню гомологии исследованных генов наиболее близок к типовому штамму Pseudomonas fluorescens, а штаммы Pseudomonas sp. OBA 2.9, GOR 4.17, STA 3 – к типовому штамму Pseudomonas koreensis. Все изучаемые бактерии способны формировать биопленки на инертных поверхностях и на корнях растений, а также обладают антагонистической активностью в отношении фитопатогенных грибов. Кроме того, у трех штаммов показана способность к растворению неорганического фосфата. Обработка семян клевера лугового штаммом Pseudomonas sp. OBA 2.4.1 приводила к повышению всхожести семян на 18% и увеличению сухой биомассы растений на 21% относительно контроля. Полученные данные позволяют рассматривать исследованные микроорганизмы как бактерии, которые могут оказывать положительное влияние на рост и развитие растений.
Род Pseudomonas на сегодняшний день включает наибольшее число описанных видов грамотрицательных бактерий (Gomila et al., 2015). Вследствие широкого спектра метаболической активности представители этого рода повсеместно распространены в природе, что позволяет им заселять различные экологические ниши и успешно выдерживать природную конкуренцию (Sitaraman, 2015). Микроорганизмы Pseudomonas sp. являются типичными представителями почвенного биоценоза, среди которых есть как патогены животных и растений, так и бактерии, которые вступают в различные симбиотические взаимодействия с растениями (Espinosa-Urgel, 2004). В почве псевдомонады концентрируются в богатой питательными веществами прикорневой зоне растения (ризосфере), а также на поверхности корней (ризоплане), образуя с ними прочные ассоциации (Vejan et al., 2016). По данным литературы значительная часть микроорганизмов в ризосфере и ризоплане представлена флуоресцентными псевдомонадами, такими как Pseudomonas fluorescens, P. putida, P. chlororaphis (Qessaoui et al., 2019; Bertani et al., 2021). Также псевдомонады найдены среди эндофитов – бактерий, которые колонизируют внутренние ткани растений, не оказывая негативного воздействия на растение-хозяина (Dudeja, Giri, 2014; Mengistu et al., 2020). Известно, что многие бактерии, ассоциированные с растениями, могут оказывать положительное влияние на его рост и продуктивность за счет целого комплекса механизмов, основными из которых являются синтез гормонов, сидерофоров, веществ с антимикробными и антифунгальными свойствами, фосфатмобилизующей активности, развития системной устойчивости. Такие микроорганизмы, способные улучшать рост растения и повышать его урожайность, объединяют в группу с общим названием plant growth-promoting rhizobacteria (PGPR) (Compant et al., 2010; Vejan et al., 2016). Повсеместная распространенность псевдомонад, а также их способность утилизировать и синтезировать разнообразные соединения вызывают значительный интерес исследователей к бактериям данного рода как к потенциальным объектам агробиотехнологии. В настоящее время PGPR-штаммы почвенных псевдомонад, относящихся к видам P. fluorescens, P. protegens, P. kilonensis, P. chlororaphis, P. simiae, используют в сельском хозяйстве в качестве биопрепаратов, которые позволяют повысить урожайность сельскохозяйственных растений и снизить долю вносимых химических удобрений (Almario et al., 2017; Gu et al., 2020). Также найдены штаммы псевдомонад, служащие продуцентами биологически активных веществ (антибиотиков, гормонов, витаминов), которые находят применение, как в сельском хозяйстве, так и в медицине (Rosas et al., 2012; Nikel et al., 2018).
Таким образом, весьма актуальным является поиск и изучение новых штаммов псевдомонад, обладающих свойствами PGPR, которые потенциально могут использоваться в качестве экологически безопасных удобрений и средств защиты растений.
Целью данной работы был поиск новых ризосферных штаммов бактерий рода Pseudomonas, определение их таксономической принадлежности и изучение свойств, которые могут быть полезны для растений.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Выделение бактериальных изолятов. Объектом исследования являлись микроорганизмы, выделенные из ризосферы дикорастущих бобовых растений Южного Урала. Для получения бактериальных изолятов образцы почв, собранные с корней растений, гомогенизировали в стерильной среде LB (мас. % в водном растворе: бактотриптон – 1, дрожжевой экстракт – 0.5, NaCl – 0.5). Через 30 мин отбирали 200 мкл жидкости и рассеивали на чашки с агаризованной средой LB (Desoky et al., 2020). В дальнейшем отобранные штаммы Pseudomonas sp. выращивали на жидкой и агаризованной среде LB при температуре 28°C (Desoky et al., 2020).
ДНК из бактерий выделяли лизированием клеток в 1% Triton X100 и 1%-ной суспензии Chelex100. Надосадочную жидкость брали в качестве матрицы для ПЦР (Чубукова и соавт., 2021).
Молекулярно-генетическая идентификация бактериальных штаммов. Генетическое разнообразие собранных изолятов исследовали с помощью RAPD анализа (Random Amplified Polymorphic DNA) с использованием следующих “случайных” праймеров: 1) LMBD 5'-GGGCGCTG-3'; 2) AFK 5'-ACGGTGGACG-3' (Баймиев и соавт., 2010).
Для амплификации гена 16S рРНК были использованы универсальные праймеры fD1 5'-cccgggatccaagcttaaggaggtgatccagcc-3' и rD1 5'-ccgaattcgtcgacaacagagtttgatcctggctcag-3', фланкирующие фрагмент гена размером около 1500 п.н. (Баймиев и соавт., 2019). Для амплификации фрагмента гена rpoD были использованы праймеры PsEG30F5'-ATYGAAATCGCCAARCG-3' и PsEG790R5'-CGGTTGATKTCCTTGA-3' с размером продукта 736 п.н. (Mulet et al., 2009).
Определение нуклеотидных последовательностей проводили на автоматическом секвенаторе Applied Biosystems 3500 (“Applied Biosystems, Inc.”, США) с использованием наборов “Big Dye Terminator v. 3.1”.
Анализ нуклеотидных последовательностей проводили с помощью пакета компьютерных программ Lasergene (“DNASTAR, Inc.”, США). Нуклеотидные последовательности для сравнительного анализа были взяты из базы данных GenBank (www.ncbi.nlm.nih.gov).
Компьютерный анализ нуклеотидных последовательностей проводили с использованием метода множественного выравнивания Clustal W в программе Megalign Lasergene (“DNASTAR”, США).
Статистическую достоверность ветвления (bootstrap-анализ) оценивали с использованием соответствующей функции программы Megalign на основе 1000 альтернативных деревьев.
Анализ ризобиальных биопленок, формируемых на инертных поверхностях. Для получения био-пленок использовали среду LB, YM (мас. % в водном растворе: маннитол – 1, дрожжевой экстракт – 0.04, NaCl – 0.01, MgSO4 – 0.01, K2HPO4 ∙ 3H2O – 0.05, агар – 1) и MH (мас. % в водном растворе: кислый казеиновый пептон – 1.75, крахмал – 0.15, вытяжка из говядины – 0.2) с применением 24-луночных пластиковых планшетов (полистирол) (“Corning Inc.”, США). Для этого штаммы Pseudomonas sp. выращивали 24 ч в жидкой среде на качалке при 28°С и 140 об./мин до концентрации 108–109 КОЕ/мл. Затем культуру разводили свежей средой до 106 КОЕ/мл и переносили по 1 мл в лунки планшета. Планшеты герметизировали Parafilm (“Amcor”, США) и инкубировали при температуре 28°С и 50 об./мин в течение 7 сут. Для определения относительных показателей плотности биопленки использовали метод окрашивания генцианом фиолетовым (“Агат-Мед”, Россия) (Вершинина и соавт., 2021). Оптическую плотность образцов измеряли с помощью прибора Enspire Model 2300 Multilabel Microplate Reader (“Perkin Elmer”, США).
Исследование влияния бактерий на прорастание семян и биомассу растений. Семена растений клевера лугового в течение 1 мин стерилизовали в 70% спирте, а после 20 мин в 5% растворе гипохлорита натрия. Далее семена были обработаны суточными культурами псевдомонад, которые предварительно выращивали на качалках в жидкой среде LB (перед инокуляцией плотность суспензии ризобий была измерена на спектрофотометре и доведена до 105 КОЕ/мл). Затем инокулированные семена раскладывали на влажную фильтровальную бумагу в чашки Петри. Часть семян оставили неинокулированными в качестве контроля. Инкубировали в темноте при комнатной температуре в течение 3 сут и проводили подсчет проросших и не проросших семян. Всхожесть определяли по формуле: (число проросших семян/число посеянных семян) × 100%. Далее проросшие семена выращивали в течение 1 нед. в предварительно прокаленном вермикулите в условиях светоплощадки (освещение 5000 лк, световой день 16 ч), после чего измеряли биомассу контрольных и опытных растений. Для этого растения целиком высушивали в сушильном шкафу до постоянного веса, а затем взвешивали.
Микроскопирование корней растений. Для визуализации взаимодействия бактерий с корнями растений штаммы Pseudomonas sp. были трансформированы ранее сконструированной генетической конструкцией на основе вектора pJN105, в которую был добавлен ген флуоресцентного белка TurboGFР (Баймиев и соавт., 2011). В дальнейшем полученными флуоресцентными штаммами были обработаны корни проростков томата.
Подготовка электрокомпетентных клеток и их трансформация, а также электрофорез фрагментов ДНК проводились согласно (Sambrook et al., 1989).
Стерильные семена томата сорта Грунтовый Грибовский 1180 (“СибНИИРС”, Россия) проращивали в течение 1 нед. на влажной фильтровальной бумаге в чашках Петри. Далее проростки выдерживали при покачивании (50 об./мин) в суспензии ризобий (107 КОЕ/мл) в течение 1 сут. Для приготовления суспензий использовали суточные культуры бактерий, выращенные в жидкой среде LB.
Визуальное наблюдение меченых микроорганизмов на корнях растений проводили на флуоресцентном микроскопе AxioImagerM1 (“CarlZeiss”, Германия).
Выявление фосфатмобилизующей активности при росте бактерий на агаризованной питательной среде. Определение способности штаммов Pseudomonas sp. к мобилизации неорганического фосфора проводили по образованию зоны просветления (гало) вокруг колоний на чашках со средой Муромцева (мас. % в водном растворе: глюкоза – 1, аспарагин – 1, K2SO4 – 0.02, MgSO4 – 0.02, кукурузный экстракт – 0.002, агар – 2; рН 6.8), содержащей нерастворимый фосфат. В качестве источника фосфора в среду добавляли Ca3(PO4)2 в концентрации 5 г/л (Егоршина и соавт., 2011). Суточную культуру бактерий наносили в виде капли на поверхность агаризованной среды. Чашки инкубировали при температуре 28°С. Фосфатмобилизующую активность бактерий оценивали количественно как площадь зон гало через 1–4 сут.
Оценка антагонистической активности бактерий. Антагонистическую активность изучаемых микроорганизмов по отношению к фитопатогенным грибам оценивали, используя метод двойной культуры (Whipps, 1987). Cтепень подавления роста грибов определяли через три дня по формуле (R2 – R1)/R2 × 100%, где R1 ‒ радиус колоний грибов в направлениях к бактериям, а R2 – к краю чашки.
В качестве тест-организмов для определения антифунгальной активности использовали Fusarium culmorum ВКМ 844, F. graminearum ВКМ 1668, F. nivale ВКМ 3106, F. solani ВКМ 142.
Статистическая обработка результатов. Результаты обрабатывали с использованием пакета Microsoft Office Excel 2010, доверительные интервалы определяли для 95% уровня значимости.
Депонирование штаммов. Последовательности исследованных штаммов были зарегистрированы в базе данных GenBank под номерами OK039351, OK040062, OM835809, OM846603 для гена 16S рРНК и OM641958, OM641959, OM678573, OM678574 для гена rpoD соответственно.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Нами были исследовано 80 изолятов бактерий, полученных из ризосферы бобовых растений остролодочника башкирского (Oxytropis baschkiriensis Knjas.), козлятника восточного (Galéga orientális Lam.), стальника колючего (Ononis spinosa L.). Отобранные чистые культуры изучаемых микроорганизмов предварительно проверяли на гетерогенность с помощью RAPD-анализа с применением “произвольных” олигонуклеотидных праймеров AFK и LMBD. В дальнейшем образцы, которые отличались по RAPD-профилю, были взяты для определения их филогении.
Для установления филогенетической принадлежности исследуемых штаммов были секвенированы нуклеотидные последовательности фрагментов консервативного гена 16S рРНК, который традиционно используется для идентификации и классификации бактерий (Баймиев и соавт., 2010, 2019). По результатам сравнительного анализа полученных нуклеотидных последовательностей данного гена с известными структурами из GenBank (http://www.ncbi.nlm.nih.gov/genbank) среди изучаемых микроорганизмов были отобраны 4 штамма, относящиеся к Pseudomonas sp.: OBA 2.4.1, OBA 2.9, GOR 4.17, STA 3. Однако в настоящее время признано, что анализа только одного консервативного гена недостаточно для дифференциации родов бактерий, которые включают генетически близкие виды, как, например, род Pseudomonas (Janda, Abbott, 2007). Поэтому в качестве альтернативного гена для филогенетических исследований был использован ген rpoD, кодирующий бактериальный фактор инициации транскрипции. В ряде исследований было показано, что секвенирование последовательности данного гена является более эффективным для точной идентификации штаммов Pseudomonas sp. по сравнению с геном 16S рРНК (Girard et al., 2020; Lauritsen et al., 2021). На основе сравнительного анализа полученных нуклеотидных последовательностей фрагментов гена rpoD с уже известными аналогичными структурами из базы данных GenBank было также установлено, что штаммы OBA 2.4.1, OBA 2.9, GOR 4.17, STA 3. относятся к бактериям рода Pseudomonas. На филогенетических деревьях сходства, построенных на основе данных анализа генов 16S рРНК и rpoD типовых представителей видов Pseudomonas sp. и анализируемых штаммов, последние разделились на две группы (рис. 1 и 2). На обоих рисунках штамм Pseudomonas sp. OBA 2.4.1 входит в отдельный кластер с типовым штаммом P. fluorescens IAM 12022T, сходство с которым по гену 16S рРНК составило 99.7%, а по гену rpoD 91.1%. Остальные анализируемые штаммы вошли в общий кластер, в котором штамм Pseudomonas sp. GOR 4.17 показал на 16S рРНК-дендрограмме наибольший уровень сходства с P. koreensis LMG 21318T (99.2%), а штаммы Pseudomonas sp. OBA 2.9 и STA 3 образовали отдельную ветвь с близкородственным к виду P. koreensis типовым штаммом P. jessenii CIP 105274Т (97.9 и 98.9% соответственно).
Рис. 1.
Филогенетическое древо бактерий, построенное на основании сравнительного анализа последовательностей гена 16S рРНК. Цифрами показана статистическая достоверность порядка ветвления, определенная с помощью “bootstrap”-анализа (показаны величины показателя “bootstrap”-анализа от 80%). На горизонтальной оси приведен вес данного выравнивания, выраженный в количестве замен нуклеотидов (×100). В качестве внешней группы использована нуклеотидная последовательность гена 16S рРНК E. coli ATCC 11775T.

Рис. 2.
Филогенетическое древо бактерий, построенное на основании сравнительного анализа последовательностей гена rpoD. Цифрами показана статистическая достоверность порядка ветвления, определенная с помощью “bootstrap”-анализа (показаны величины показателя “bootstrap”-анализа от 80%). На горизонтальной оси приведен вес данного выравнивания, выраженный в количестве замен нуклеотидов (×100). В качестве внешней группы использована нуклеотидная последовательность гена rpoD E. coli ATCC 11775T.

Причем на rpoD-дендрограмме штаммы Pseudomonas sp. OBA 2.9, GOR 4.17, STA 3 образуют отдельную ветвь с типовым штаммом P. koreensis LMG 21318T c гомологией 95.6, 94 и 91.9% соответственно, тогда как с типовым штаммом P. jessenii CIP 105274T гомология ниже (90.9, 90.2 и 89.5% соответственно). Поскольку дискриминационная способность гена rpoD считается более высокой для дифференциации близкородственных видов, можно предположить, что три вышеуказанных штамма наиболее близки к штамму P. koreensis LMG 21318T (Lauritsen et al., 2021).
Анализ биопленок, формируемых штаммами псевдомонад на инертных поверхностях. Для большинства бактерий формирование биопленок является основной стратегией выживания в неблагоприятных условиях среды (Вершинина и соавт., 2021). Для почвенных микроорганизмов способность к биопленкообразованию является залогом конкурентоспособности и эффективной колонизации ризосферы. Также использование в агротехнологиях предпосевной обработки семян PG-PR-бактериями в целях улучшения всхожести и роста растений будет результативным при успешном образовании ими биопленок.
Нами было изучено биопленкообразование штаммами Pseudomonas sp. OBA 2.4.1, OBA 2.9, GOR 4.17, STA 3 на поверхности полистироловых планшетов в различных жидких питательных средах: белковые среды MH, LB и углеводная, безбелковая среда YM. Исследования показали, что все 4 штамма псевдомонад были способны к образованию биопленок на инертных поверхностях во всех средах. При этом наиболее эффективно они формировались в среде MH, содержащей большое количество питательных веществ. Относительная биомасса биопленок при культивировании на среде MH у всех штаммов превышала соответствующие показатели на среде LB и YM в 1.5 раза (рис. 3). Однако все штаммы показали хорошую способность к биопленкообразованию и на бедной питательными веществами среде YM.
Рис. 3.
Относительная биомасса биопленок штаммов Pseudomonas sp. после 7 сут культивирования при 28°C в зависимости от среды. По оси ординат – оптическая плотность кристаллического фиолетового, десорбированного после окрашивания биопленок, образованных на пластиковых планшетах. Обозначения штаммов Pseudomonas sp. (1–4): 1 – OBA 2.4.1; 2 ‒ OBA 2.9; 3 – GOR 4.17; 4 – STA 3.

Полученные результаты свидетельствуют о способности исследованных штаммов псевдомонад образовывать зрелые биопленки в условиях как богатых, так и бедных питательных сред. При этом лучшую способность к биопленкообразованию во всех средах показал штамм Pseudomonas sp. OBA 2.4.1, относительная биомасса биопленок которого превышала соответствующие показатели других штаммов более чем в 1.2 раза.
По данным флуоресцентной микроскопии, после 1 сут совместного культивирования штаммы Pseudomonas sp. ОВА 2.4.1, OBA 2.9, GOR 4.17, STA 3 формировали микроколонии и биопленки на поверхности корней томатов. Причем характерной особенностью штаммов Pseudomonas sp. ОВА 2.4.1 и STA 3 оказалось образование биопленок как на корневых волосках, так и на корневой поверхности, тогда как у штаммов Pseudomonas sp. ОВА 2.9 и GOR 4.17 бактерии преимущественно локализовались на поверхности ризодермы, а на корневых волосках наблюдались единичные колонии (рис. 4).
Рис. 4.
Образование биопленок на поверхности корней проростков томата через 24 ч совместного культивирования: 1 – Pseudomonas sp. OBA 2.4.1; 2 – Pseudomonas sp. STA 3; 3 – Pseudomonas sp. OBA 2.9; 4 – Pseudomonas sp. GOR 4.17.

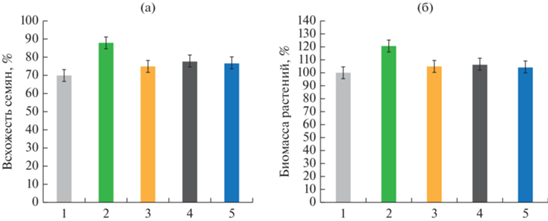
Исследование влияния бактерий на прорастание семян и биомассу растений. По данным литературы положительное влияние PGPR-бактерий на растения проявляется, в том числе, в улучшении всхожести семян (Bharathi, 2004; Qessaoui et al., 2019). В нашей работе было показано, что предварительная инокуляция семян растений клевера лугового штаммом Pseudomonas sp. ОВА 2.4.1 приводила к повышению прорастания семян на 18% больше относительно необработанного контроля (рис. 5а). Сухая биомасса растений, инокулированных штаммом Pseudomonas sp. ОВА 2.4.1, была на 21% больше по сравнению с необработанными контрольными растениями. Со штаммами псевдомонад Pseudomonas sp. OBA 2.9, GOR 4.17, STA 3 достоверно значимых различий во всхожести семян и в биомассе относительно контроля не было выявлено (рис. 5б).
Рис. 5.
Влияние штаммов Pseudomonas sp. на всхожесть семян растений клевера лугового (по оси ординат всхожесть в %) (а) и сухую биомассу растений клевера лугового (по оси ординат изменение сухой биомассы растений в %) (б). Обозначения штаммов Pseudomonas sp. (1–5): 1 – контроль; 2 – OBA 2.4.1; 3 — OBA 2.9; 4 –GOR 4.17; 5 – STA 3.
Выявление способности к растворению фосфатов. Фосфор является чрезвычайно значимым элементом для нормального роста и развития растения, однако большая часть фосфора в почве находится в недоступной для растений форме (Lambers et al., 2006). Одной из важнейших характеристик PGPR-штаммов бактерий считается способность к растворению труднодоступных фосфатов и, как следствие, улучшение фосфорного питания растения (Vejan et al., 2016). Поэтому нами были исследованы фосфатмобилизующие свойства новых штаммов псевдомонад.
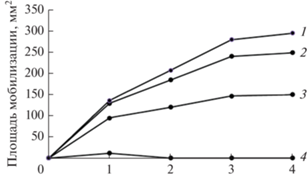
Оценка фосфатмобилизующей активности исследуемых нами штаммов псевдомонад по наличию или отсутствию прозрачных зон “гало” на агаризованной среде Муромцева показала, что все они, кроме штамма Pseudomonas sp. ОВА 2.4.1, способны к растворению неорганического фосфата в виде Ca3(PO4)2 (рис. 6). Наиболее высокий показатель фосфатмобилизующей активности был получен у штамма Pseudomonas sp. STA 3.
Рис. 6.
Динамика мобилизации неорганического фосфата штаммами Pseudomonas sp.: 1 – STA 3; 2 – ОВА 2.9; 3 – GOR 4.1; 4 – OBA 2.4.1. По оси абсцисс ‒ сутки, на которые измеряли площадь мобилизации фосфата; по оси ординат ‒ значения площади зон мобилизации фосфата в мм2.
Оценка антагонистической активности бактерий. По данным литературы многие штаммы Pseudomonas sp. с помощью различных механизмов, таких как, например, синтез сидерофоров и антибиотиков, индукция системной устойчивости, способны подавлять или замедлять рост фитопатогенных грибов (Bharathiс et al., 2004; Dutta, Podile, 2010). Данные свойства позволяют использовать подобные штаммы-антагонисты в качестве биологического контроля для защиты растений от заболеваний.
Изучение влияния исследуемых микроорганизмов на различные виды фитопатогенных грибов Fusarium sp. показало наличие антагонистической активности у всех штаммов бактерий (табл. 1). Ингибирование роста F. nivale в двойной культуре варьировало в диапазоне 20.7–30.2%, F. culmorum ‒ 15.1–30.7%, F. graminearum ‒ 33.9–34.1%, F. solani ‒ 20.6–37.5%. Наибольший антагонизм проявил штамм Pseudomonas sp. 2.4.1.
Таблица 1.
Ингибирование роста грибов различными штаммами псевдомонад
| Штаммы бактерий | Грибы | |||
|---|---|---|---|---|
| Fusarium nivale | Fusarium culmorum | Fusarium graminearum | Fusarium solani | |
| Pseudomonas sp. ОВА 2.4.1 | 22.9 ± 2.35 | 30.7 ± 2.63 | 33.9 ± 2.58 | 37.5 ± 2.01 |
| Pseudomonas sp. ОВА 2.9 | 30.2 ± 2.83 | 12.3 ± 1.34 | – | 31.3 ± 2.32 |
| Pseudomonas sp. GOR 4.17 | 25.4 ± 2.52 | 15.7 ± 1.75 | – | 22.7 ± 1.87 |
| Pseudomonas sp. STA 3 | 20.7 ± 1.88 | 15.1 ± 1.58 | 34.1 ± 2.89 | 20.6 ± 2.12 |
В настоящее время в мире остро стоит проблема повышения урожайности сельскохозяйственных культур. Однако традиционные агротехнологии часто приводят к значительному загрязнению почв и, впоследствии, сельскохозяйственной продукции минеральными удобрениями. Используемые для борьбы с вредителями культурных растений антифунгальные препараты, в свою очередь, могут накапливаться в продуктах, а также провоцировать возникновение новых устойчивых штаммов фитопатогенов. Применение PGPR-микроорганизмов может служить альтернативой препаратам химического происхождения.
Нами было идентифицировано четыре новых штамма, относящихся к роду Pseudomonas, среди бактериальных изолятов, полученных при выделении из ризосферы бобовых растений. Исследования показали у всех штаммов наличие таких хозяйственно-полезных признаков, как антагонистическая активность в отношении фитопатогенных грибов рода Fusarium, у 3 штаммов бактерий выявлена способность к мобилизации неорганического фосфата. Также было показано, что обработка семян клевера лугового штаммом Pseudomonas OBA 2.4.1 улучшала всхожесть семян и увеличивала накопление сухой биомассы проростков. Ранее нами было продемонстрировано, что инокуляция корней проростков томата сорта Грунтовый Грибовский 1180 штаммом Pseudomonas OBA 2.4.1 через 30 дней приводила к значительному увеличению сухой биомассы растений и содержанию пигментов в листьях томата в сравнении с контрольными неинокулированными растениями (Масленникова и соавт., 2021). В дальнейшем планируется исследование биохимических свойств новых штаммов Pseudomonas sp. для уточнения видовой принадлежности и изучения влияния данных бактерий на рост и развитие различных видов растений.
Список литературы
Баймиев Ан.Х., Акимова Е.С., Гуменко Р.С., Владимирова А.А., Чемерис А.В., Баймиев Ал.Х. Генетическое разнообразие и филогения клубеньковых бактерий, выделенных из клубеньков растений рода Lupinaster, произрастающих на Южном Урале // Генетика. 2019. Т. 55. С. 55–59.
Baymiev An.K., Akimova E.S., Gumenko R.S., Vladimirova A.A., Muldashev A.A., Chemeris A.V., Baymiev Al.K. Genetic diversity and phylogeny of root nodule bacteria isolated from nodules of plants of the Lupinaster genus inhabiting the southern Urals // Russ. J. Genet. 2019. V. 55. P. 45–51.
Баймиев Ан.Х., Ямиданов Р.С., Матниязов Р.Т., Благова Д.К., Баймиев Ал.Х., Чемерис А.В. Получение флуоресцентно меченных штаммов клубеньковых бактерий дикорастущих бобовых для их детекции in vivo и in vitro // Мол. биология. 2011. Т. 45. С. 984–991.
Baymiev An.K., Yamidanov R.S., Matniyazov R.T., Blagova D.K., Chemeris A.V. Preparation of fluorescent labeled nodule bacteria strains of wild lefumes for their detection in vivo and in vitro // Mol. Biol. (Moscow). 2011. V. 45. P. 904–910.
Баймиев Ан.Х., Птицын К.Г., Баймиев Ал.Х. Влияние интродукции караганы древовидной на состав ее клубеньковых бактерий // Микробиология. 2010. Т. 79. С. 123–128.
Baymiev An.K., Ptitsyn K.G., Baimiev Al.K. Influence of the introduction of Caragana arborescens on the composition of its root-nodule bacteria // Microbiology (Moscow). 2010. V. 79. P. 115–112.
Вершинина З.Р., Чубукова О.В., Никоноров Ю.М., Хакимов Л.Р., Лавина А.М., Каримова Л.Р., Баймиев Ан.Х., Баймиев Ал.Х. Влияние сверхэкспрессии гена rosR на образование биопленок бактериями Rhizobium leguminosarum // Микробиология. 2021. Т. 90. С. 191–203.
Vershinina Z.R., Chubukova O.V., Nikonorov Y.M., Khakimova L.R., Lavina A.M., Karimova L.R., Baymiev An.K., Baymiev Al.K. Effect of rosR gene overexpression on biofilm formation by Rhizobium leguminosarum // Microbiology (Moscow). 2021. V. 90. P. 198–209.
Егоршина А.А., Хайруллин Р.М., Лукьянцев М.А., Курамшина З.М., Смирнова Ю.В. Фосфат-мобилизующая активность эндофитных штаммов Bacillus subtilis и их влияние на степень микоризации корней пшеницы // Журн. Сибирского фед. ун-та. Биология. 2011. Т. 4. № 2. С. 172–182.
Масленникова Д.Р., Чубукова О.В., Вершинина З.Р., Емелина А.А., Насырова К.Р., Хакимова Л.Р. Михайлова Е.В. Влияние ростостимулирующих бактерий на рост и содержание фотосинтетических пигментов в листьях растений томата // Biomics. 2021. Т. 13. С. 274‒279. https://doi.org/10.31301/2221-6197.bmcs.2021-18
Чубукова О.В., Вершинина З.Р., Матниязов Р.Т., Баймиев Ан.Х., Баймиев Ал.Х. Создание индуцируемой векторной системы на основе промотора гена nodA ризобий // Экологическая генетика. 2021. Т. 19. С. 13–21.
Almario J., Bruto M., Vacheron J., Prigent-Combaret C., Moenne-Loccoz Y., Muller D. Distribution of 2,4-diacetylphloroglucinol biosynthetic genes among the Pseudomonas spp. reveals unexpected polyphyletism // Front. Microbiol. 2017. V. 8. Art. 1218. https://doi.org/10.3389/fmicb.2017.01218
Bertani I., Zampieri E., Bez C., Volante A., Venturi V., Monaco S. Isolation and characterization of Pseudomonas chlororaphis strain ST9; rhizomicrobiota and in planta studies // Plants. 2021. V. 10. P. 1466. https://doi.org/10.3390/plants10071466
Bharathi R., Vivekananthan R., Harish S., Ramanathan A., Samiyappan R. Rhizobacteria-based bio-formulations for the management of fruit rot infection in chillies // Crop Protection. 2004. V. 23. P. 835–843.
Compant S., Clément C., Sessitsch A. Plant growth-promoting bacteria in the rhizo- and endosphere of plants: their role, colonization, mechanisms involved and prospects for utilization // Soil Biol. Biochem. 2010. V. 42. P. 669–678.
Desoky E.S.M., Merwad A.R.M., Semida W.M., Ibrahim S.A., El-Saadony M.T., Rady M.M. Heavy metals-resistant bacteria (HM-RB): Potential bioremediators of heavy metals-stressed Spinacia oleracea plant // Ecotoxicol. Environ. Saf. 2020. V. 198. P. 110685. https://doi.org/10.1016/j.ecoenv.2020.110685
Dudeja S.S., Giri R. Beneficial properties, colonization, establishment and molecular diversity of endophytic bacteria in legumes and non legumes // Afr. J. Microbiol. Res. 2014. V. 8. P. 1562–1572.
Dutta S., Podile A.R. Plant growth promoting rhizobacteria (PGPR): the bugs to debug the root zone // Crit. Rev. Microbiol. 2010. V. 36. P. 232–244. https://doi.org/10.3109/10408411003766806
Espinosa-Urgel M. Plant-associated Pseudomonas populations: molecular biology, DNA dynamics, and gene transfer // Plasmid. 2004. V. 52. P. 139–150.
Girard L., Lood C., Rokni-Zadeh H., van Noort V., Lavigne R., De Mot R. Reliable identification of environmental Pseudomonas isolates using the rpoD gene // Microorganisms. 2020. V. 8. Art. 1166. https://doi.org/10.3390/microorganisms8081166
Gomila M., Peña A., Mulet M. M., Lalucat J., García-Valdés E. Phylogenomics and systematics in Pseudomonas // Front. Microbiol. 2015. V. 6. Art. 214. https://doi.org/10.3389/fmicb.2015.00214
Gu Y., Ma Y.N., Wang J., Xia Z., Wei H.L. Genomic insights into a plant growth-promoting Pseudomonas koreensis strain with cyclic lipopeptide-mediated antifungal activity // MicrobiologyOpen. 2020. V. 9. Art. e1092. https://doi.org/10.1002/mbo3.1092
Janda J.M., Abbott S.L. 16S rRNA gene sequencing for bacterial identification in the diagnostic laboratory: pluses, perils, and pitfalls // J. Clin. Microbiol. 2007. V. 45. P. 2761–2764.
Lambers H., Shane M.W., Cramer M.D., Pearse S.J., Veneklaas E.J. Root structure and functioning for efficient acquisition of phosphorus: matching morphological and physiological traits // Ann. Bot. 2006. V. 98. P. 693–713.
Lauritsen J.G., Hansen M.L., Bech P.K., Jelsbak L., Gram L., Strube M.L. Identification and differentiation of Pseudomonas species in field samples using an rpoD amplicon sequencing methodology // mSystems. 2021. V. 6. e0070421. https://doi.org/10.1128/mSystems.00704-21
Mengistu A.A. Endophytes: colonization, behaviour, and their role in defense mechanism // Int. J. Microbiol. 2020. V. 2020. Art. 6927219. https://doi.org/10.1155/2020/6927219
Mulet M., Bennasar A., Lalucat J., García-Valdés E. An rpoD-based PCR procedure for the identification of Pseudomonas species and for their detection in environmental samples // Mol. Cell Probes. 2009. V. 23. P. 140–147. https://doi.org/10.1016/j.mcp.2009.02.001
Nikel P.I., de Lorenzo V. Pseudomonas putida as a functional chassis for industrial biocatalysis: from native biochemistry to trans-metabolism // Metab. Eng. 2018. V. 50. P. 142–155. https://doi.org/10.1016/j.ymben.2018.05.005
Qessaoui R., Bouharroud R., Furze J.N., El Aalaoui M., Akroud H., Amarraque A., Vaerenbergh J.V., Tahzima R., Mayad E.H., Chebli B. Applications of new rhizobacteria Pseudomonas isolates in agroecology via fundamental processes complementing plant growth // Sci. Rep. 2019. V. 9. Art. 12832. https://doi.org/10.1038/s41598-019-49216-8
Rosas S.B., Pastor N.A., Guiñazú L.B., Andrés J.A., Carlier E., Vogt V., Bergesse J., Rovera M. Efficacy of Pseudomonas chlororaphis subsp. aurantiaca SR1 for improving productivity of several crops // Crop Production Technologies / Ed. P. Sharma. InTech, 2012. P. 199–210.
Sambrook J., Fritsch E., Maniatis T. Molecular Cloning: a Laboratory Manual. N.Y.: Cold Spring Harbor Lab. Press, 1989. 1626 p.
Sitaraman R. Pseudomonas spp. as models for plant-microbe interactions // Front. Plant Sci. 2015. V. 6. Art. 787. https://doi.org/10.3389/fpls.2015.00787
Vejan P., Abdullah R., Khadiran T., Ismail S., Nasrulhaq Boyce A. Role of plant growth promoting rhizobacteria in agricultural sustainability ‒ a review // Molecules. 2016. V. 21. Art. 573. https://doi.org/10.3390/molecules21050573
Whipps J.M. Effect of media on growth and interactions between a range of soil-borne glasshouse pathogens and antagonistic fungi // New Phytologyst. 1987. V. 107. P. 127–142.
Дополнительные материалы отсутствуют.


