Микробиология, 2023, T. 92, № 1, стр. 14-23
Sulfurospirillum Tamanensis sp. nov. – факультативно анаэробная алкалифильная бактерия из наземного грязевого вулкана
А. А. Фролова a, *, А. Ю. Меркель a, В. В. Кевбрин a, Д. С. Копицын b, А. И. Слободкин a
a ФИЦ Биотехнологии РАН, Институт микробиологии им. С.Н. Виноградского
119071 Москва, Россия
b РГУ нефти и газа им. И.М. Губкина
119991 Москва, Россия
* E-mail: romana2804@gmail.com
Поступила в редакцию 15.07.2022
После доработки 27.07.2022
Принята к публикации 29.07.2022
- EDN: NLZORV
- DOI: 10.31857/S0026365622600560
Аннотация
Из наземного грязевого вулкана Таманского полуострова выделена алкалифильная факультативно анаэробная бактерия (штамм T05bT). Клетки изолята представляют собой подвижные спириллы толщиной 0.5 мкм и длиной 1.5–2.5 мкм. Штамм T05bT растет при температуре 6–42°C (оптимум ‒ 30°C), pH 8.0–11.0 (оптимум ‒ 9.0), концентрации NaCl 0–14% (вес/об.); использует лактат, формиат, малат, пируват, молекулярный водород, элементную серу, сульфит, тиосульфат и сульфид в качестве донора электронов и нитрат, фумарат, элементную серу, сульфит, тиосульфат, ДМСО, арсенат и кислород в качестве акцептора электронов. Продуктом восстановления нитрата является аммоний. Сбраживает малат, пируват, фумарат. Способен к микроаэробному (до 3% О2, об./об.) росту. Штамм T05bT не использует сульфат и Fe(III) в качестве акцептора электронов, не диспропорционирует элементную серу, тиосульфат и сульфит и не сбраживает глюкозу, фруктозу, сахарозу, лактат, формиат, малеинат, дрожжевой экстракт и пептон. В жирнокислотном составе клеток преобладают C16:1 ω7 (45.9%), C16:0 (25.8%) и C18:1 ω7 (20.9%). Геном штамма T05bT имеет размер 2.46 Мп и содержание Г + Ц 45.5%. В геноме содержатся гены, кодирующие ферменты энергетического метаболизма: периплазматический комплекс нитратредуктазы Nap, тиосульфат/полисульфид редуктазу Phs/Psr, сульфид:хинон оксидоредуктазу Sqr и дыхательную арсенатредуктазу Arr. Анализ нуклеотидной последовательности гена 16S рРНК штамма T05bT показал 98.61% сходства с типовым штаммом Sulfurospirillum alkalitolerans (филум “Campylobacterota”). На основании фенотипических характеристик и данных филогенетического анализа предлагается отнести данный изолят к новому виду рода Sulfurospirillum, как Sulfurospirillum tamanensis sp. nov. с типовым штаммом T05bT (=DSM 112596T = VKM B-3538T).
Грязевые вулканы ‒ это геологические структуры, образованные выбросами частиц глины, брекчий, жидкостей и газов из глубоких осадочных слоев. Грязевой вулканизм играет существенную роль в балансе метана в атмосфере (Mazzini, Etiope, 2017). Наземные грязевые вулканы геологически связаны с подземными залежами нефти и природного газа и содержат различные неорганические и органические соединения, которые могут использоваться в качестве доноров и акцепторов электронов в микробном метаболизме. Вероятно, этим объясняется значительное филогенетическое разнообразие микроорганизмов, как детектированных молекулярными методами (Cheng et al., 2012; Yang et al., 2012; Merkel et al., 2021), так и выделенных в чистые культуры из наземных грязевых вулканов (Alain et al., 2006; Kokoschka et al., 2015; Khomyakova et al., 2020; Ratnikova et al., 2020; Slobodkina et al., 2020; Frolova et al., 2021a, 2021b).
Таманский полуостров является одним из регионов с наиболее интенсивным грязевым вулканизмом. Керченско-Таманская грязевулканическая провинция насчитывает свыше 100 действующих наземных грязевых вулканов (Шнюков и соавт., 1986; Холодов и соавт., 2012). Флюиды многих из этих вулканов имеют pH > 8.5, что способствует развитию алкалифильных микроорганизмов. Алкалифильные анаэробные бактерии, принадлежащие к разным филогенетическим группам, обладают значительным биотехнологическим потенциалом и населяют различные природные и антропогенные среды (Sorokin et al., 2014; Preiss et al., 2015), но разнообразие алкалифильных анаэробов в наземных грязевых вулканах изучено слабо.
В настоящей работе приводится описание штамма факультативно анаэробного алкалифильного микроорганизма, выделенного из наземного грязевого вулкана полуострова Тамань и отнесенного нами к новому виду рода Sulfurospirillum.
МАТЕРИАЛЫ И МЕТОДЫ ИССЛЕДОВАНИЯ
Источник выделения. Образец грязевулканического флюида, содержащего твердую и жидкую фракции, был отобран в мае 2017 г. из активного грифона наземного грязевого вулкана Гнилая Гора (Таманский полуостров, Краснодарский край, РФ). GPS координаты ‒ 45.251° N, 37.436° E. Температура в месте отбора проб составляла 21°C, pH 8.5, концентрация Cl– ‒ 15.7 мМ, концентрация ${\text{SO}}_{4}^{{2 - }}$ ‒ 5.3 мМ. Образец был отобран анаэробно в пластиковую пробирку с плотно закручивающийся крышкой и в таком виде перевезен в лабораторию для дальнейших экспериментов.
Среды и культивирование. Для выделения и рутинного культивирования штамма T05bT использовали анаэробную восстановленную солоноватую среду следующего состава (на 1 л дистиллированной воды): 0.33 г KH2PO4, 0.33 г NH4Cl, 0.33 г KCl, 0.33 г CaCl2 · 6H2O, 2.00 г NaHCO3, 0.33 г MgCl2 · · 6H2O, 10.00 г NaCl, 0.66 г Na2S · 9H2O, 0.001 г резазурин, 1 мл витаминов (Wolin et al., 1963) и 1 мл микроэлементов (Slobodkin et al., 2012). Среду готовили кипячением и охлаждением под непрерывным током N2, после чего добавляли восстанавливающий агент (Na2S · 9H2O). Приготовленную среду разливали по 10 мл в пробирки Хангейта объемом 17 мл и автоклавировали при 121°C в течение 60 мин. После стерилизации pH среды был 9.0. Нитрат калия (10 мМ) и лактат натрия (10 мМ) добавляли из стерильных концентрированных растворов перед инокуляцией образца.
Фенотипические характеристики. Изучение морфологии и подвижности клеток проводили в жидкой среде после 48 ч инкубации, используя микроскоп Zeiss Primo Star с фазово-контрастным устройством. Ростовые эксперименты проводились в трехкратной повторности. Для морфологической, физиологической и метаболической характеристики штамм T05bT культивировали на среде, применявшейся для выделения, пока не указано иное. Определение диапазонов температуры, pH и солености проводились на восстановленной среде с лактатом и нитратом. Диапазоны солености измеряли при концентрации NaCl 0–15% (вес/об.). Различные значения pH создавали путем внесения в отдельные пробирки стерильных растворов 3 М HCl или 10% NaOH. Эксперименты с серосодержащими соединениями и кислородом проводили на невосстановленной среде.
Состав клеточных жирных кислот. Состав жирных кислот определяли, как указано ранее (Slobodkina et al., 2020), используя прямое метилирование лиофильно высушенной биомассы бактерий и хромато-масс-спектрометрический анализ; содержание определяли по методу внутренней нормализации по площади пиков полного ионного тока метиловых эфиров жирных кислот.
Выделение ДНК, секвенирование и анализ полного генома. Выделение ДНК для определения нуклеотидной последовательности гена 16S рРНК и полногеномного секвенирования проводили с помощью FastDNA Spin Kit (“MP Bio”), следуя протоколу производителя. Ген 16S рРНК амплифицировали, используя универсальные бактериальные праймеры 27F, 357F, 530F, 1114F, 342R, 519R и 1492R (Weisburg et al., 1991). Секвенирование продуктов ПЦР проводили методом Сэнгера. Предварительный филогенетический скрининг сходства последовательностей генов 16S рРНК проводили по базе данных GenBank (Benson et al., 1999) с помощью программы BLAST (Altschul et al., 1990). Для более точного определения филогенетического положения изолята нуклеотидную последовательность гена 16S рРНК выравнивали с последовательностями референтных штаммов ближайших родственных микроорганизмов с помощью программы Clustal W (Thompson, 1997). Филогенетический анализ выполняли при помощи программы MEGA 7.0 (Kumar et al., 2016). Статистическую достоверность ветвления оценивали с помощью “bootstrap-анализа” 1000 альтернативных филограмм (Felsenstein, 1985), построенных методами, входящими в пакет программ для филогенетического анализа MEGA 7.
Геном штамма T05bT секвенировали, используя систему MiSeq (“Illumina”, San Diego, California, США). Поиск генов и аннотацию проводили с использованием сервера RAST (Brettin et al., 2015). Средство просмотра SEED использовалось для распределения предсказанных генов по категориям подсистем (Overbeek et al., 2014).
Нуклеотидная последовательность гена 16S рРНК штамма T05bT депонирована в GenBank/EMBL под номером MW872671.
Полногеномная последовательность штамма T05bT депонирована в GenBank/EMBL под номером JAFHKK010000000.
РЕЗУЛЬТАТЫ
Получение чистой культуры. Накопительные культуры были получены путем внесения природного образца (около 10%, об.) в стерильную анаэробную среду, содержащую лактат и нитрат. После инкубации накопительных культур при 30°C в течение 2 сут, наблюдался значительный микробный рост. После трех последовательных пересевов (5%, об.) культуру рассевали методом 10-кратных серийных разведений на жидкой среде того же состава. В последнем разведении, показавшем рост (10–9), наблюдался только один морфологический тип клеток. Десятикратные разведения были повторены еще дважды, и культура, выросшая в последнем разведении, получила обозначение штамм T05bT. Результаты секвенирования гена 16S рРНК подтвердили чистоту культуры. Попытки получить рост колоний в анаэробных условиях на твердой среде методом roll-tube или в толще 1% Gelrite gellan gum или 1% агара, не привели к положительным результатам.
Клетки штамма T05bT представляют собой спириллы толщиной 0.5 мкм и длиной 1.5–2.5 мкм, растущие одиночно или парами; подвижны за счет одиночного полярного жгутика. Образования эндоспор не наблюдалось на протяжении 30 сут культивирования.
Физиология роста. Штамм T05bT способен расти при температурах от 10 до 42°C, с оптимумом при 30°C. Рост не наблюдался при 50°C и выше и при 6°C и ниже при инкубации в течение 20 сут. Диапазон pH для роста составляет 8.0–11.0 (оптимум 9.0), роста не отмечалось при pH 7.5 и ниже или 11.5 и выше. Штамм T05bT способен расти при концентрации NaCl в среде от 0 до 14.0% (вес/об.); при 15% NaCl и выше роста не происходило.
Доноры и акцепторы электронов. Штамм T05bT способен использовать широкий спектр органических и неорганических соединений в качестве доноров и акцепторов электронов для роста (табл. 1). Добавление дрожжевого экстракта (0.2 г/л) не стимулировало рост. Максимальная концентрация клеток (около 7 × 107 кл./мл) наблюдалась при росте с лактатом. Водород использовался только с фумаратом, в этих условиях штамм T05bT не нуждался в дополнительном источнике углерода для роста. Элементная сера, тиосульфат и сульфит могут быть использованы в качестве доноров электронов с нитратом или фумаратом в качестве акцептора. Восстановление нитрата с соединениями серы требует присутствия малата или сукцината как источника углерода, причем рост с сульфитом был значительно лучше, чем с элементной серой или тиосульфатом. Сульфид (в концентрациях до 5 мМ) использовался в качестве донора электронов для восстановления малата, фумарата (10 мМ каждый) и нитрата (1 мМ) без добавления дополнительных источников углерода с образованием элементной серы, сукцината и ацетата (в соотношении 5 : 1 при росте с малатом и 6.7 : 1 при росте с фумаратом). Продуктом восстановления нитрата со всеми протестированными донорами электронов является аммоний. Штамм T05bT способен к дисмутации малата и фумарата. Основными продуктами дисмутации малата и фумарата являются сукцинат и ацетат. Соотношение сукцинат/ацетат при сбраживании малата составило 2.2, а при сбраживании фумарата 4.6. Штамм T05bT способен к росту в микроаэробных (до 3% кислорода) условиях; роста при концентрации кислорода выше 3% или в аэробных условиях не наблюдалось при культивировании в течение трех недель.
Таблица 1.
Использование доноров и акцепторов электронов штаммом T05bT
| Акцептор/донор | Лактат | Формиат | Малат | Пируват | Водород | Элементная сера | Сульфит | Тиосульфат | Сульфид |
|---|---|---|---|---|---|---|---|---|---|
| Нитрат | + | ‒ | + | ‒ | ‒ | +* | +* | +* | + |
| Фумарат | + | + | + | + | + | + | + | + | + |
| Элементная сера | + | ‒ | + | + | ‒ | ‒ | ‒ | ‒ | ‒ |
| Сульфит | + | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ | ‒ |
| Тиосульфат | ‒ | ‒ | ‒ | + | ‒ | ‒ | ‒ | ‒ | ‒ |
| ДМСО | + | ‒ | ‒ | + | ‒ | ‒ | ‒ | ‒ | ‒ |
| Арсенат | + | + | + | + | ‒ | ‒ | ‒ | ‒ | ‒ |
| Кислород | + | ‒ | + | + | ‒ | ‒ | ‒ | ‒ | ‒ |
Примечание. (+) – рост >2 × 106 кл./мл. (‒) – отсутствие роста. Концентрация всех растворимых доноров и акцепторов – 10 мМ. Элементная сера – 5 г/л, ДМСО ‒ 10%, кислород ‒ 3% (об./об.) в газовой фазе, водород ‒ 80% (об./об.) в газовой фазе. * В присутствии малата или сукцината как источника углерода.
Штамм T05bT не использует сульфат (10 мМ) и ферригидрит (слабокристаллический оксид Fe(III), 90 мМ) в качестве акцепторов электронов с протестированными донорами электронов и не способен к диспропорционированию элементной серы, тиосульфата и сульфита. Штамм T05bT не способен к сбраживанию глюкозы, фруктозы, сахарозы, лактата, формиата, малеината (каждый субстрат 10 мМ), дрожжевого экстракта (1 г/л) и пептона (2.5 г/л).
Состав клеточных жирных кислот. Клеточные жирные кислоты штамма T05bT были представлены смесью неразветвленных насыщенных и ненасыщенных кислот: C16:1ω7 (45.9%), C16:0 (25.8%) и C18:1 ω7 (20.9%). Также были обнаружены C14:1 ω7 (3.1%), C14:0 (1.9%), C15:0 (0.3%), C16:1 ω5 (0.7%) и C18:0 (1.4%).
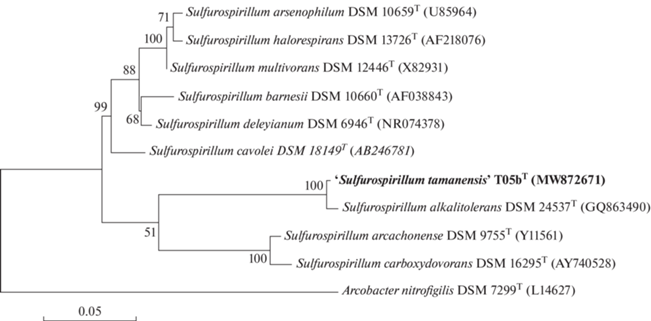
Филогения. Нуклеотидные последовательности гена 16S рРНК штамма T05bT, полученные путем амплификации с универсальными бактериальными праймерами и в ходе полногеномного секвенирования, были идентичны. Сравнение 1473 нуклеотидов последовательности гена 16S рРНК штамма T05bT с нуклеотидными последовательностями, размещенными в базе данных GenBank (Benson et al., 1999) показали, что изолят принадлежит к роду Sulfurospirillum класса Epsilonproteobacteria с 98.61% сходства с геном 16S рРНК с S. alkalitolerans (Sorokin et al., 2013). Реконструкция филогенетического дерева гена 16S рРНК показала, что штамм T05bT представляет собой монофилетическую ветвь, четко отделенную от наиболее близких видов (рис.1).
Рис. 1.
Филогенетическое дерево, основанное на последовательностях гена 16S рРНК, показывающее положение штамма T05bT и родственных ему микроорганизмов. Дерево было реконструировано методом maximum-likelihood. Деревья, построенные с помощью алгоритмов neighbor-joining и minimum-evolution, отображали одинаковую топологию. Каждое число указывает значение начальной загрузки из 1000 повторностей. Масштабная метка ‒ 0.020 замен на нуклеотидное положение. Идентификационные номера GenBank указаны в скобках.
Общие характеристики генома. Геном штамма T05bT, собранный из 63 контигов, имеет общую длину 2 456 696 нт и значение N50 73 734 нт. Содержание Г + Ц геномной ДНК составляет 45.5%. Геном содержит 2568 нуклеотидных последовательностей кодирующих белки и 39 генов РНК. Большинство аннотированных генов отвечает за синтез аминокислот и производных (182), метаболизм белков (104), дыхание (93), кофакторы, витамины, простетические группы и образование пигментов (86).
Углеродный метаболизм. В отличие от других представителей рода Sulfurospirillum (Ross et al., 2016), геном штамма T05bT не содержит полного набора генов цикла трикарбоновых кислот. Однако функцию отсутствующей малатдегидрогеназы может выполнять мембранно-связанная малат:хинон оксидоредуктаза (WP_205459850), которая содержит ФАД в качестве кофактора и отдает электроны хинонам, которые впоследствии окисляются компонентами электрон-транспортной цепи (Kather et al., 2000). Геном штамма T05bT содержит гены фумаратгидратазы I типа (WP_205460080), что является характерным для рода Sulfurospirillum (Ross et al., 2016). Гены пируваткарбоксилазы, присутствующие у всех представителей рода Sulfurospirillum, в геноме штамма T05bT отсутствуют. Превращение пирувата в оксалоацетат происходит, по-видимому, с помощью оксалоацетатдекарбоксилазы (WP_205458054‒WP_205458055). Наличие всех генов фосфатацетилтрансферазного/ацетаткиназного пути, вероятно, позволяет штамму T05bT использовать лактат в качестве донора электронов, превращая ацетат в ацетил-КоА.
Геном штамма T05bT содержит все гены неокислительной части пентозофосфатного пути, что является характерным для всех представителей рода Sulfurospirillum (Ross et al., 2016), а также все гены нефосфорилирующего пути Энтнера‒Дудорова.
Сопряжение окисления органических субстратов с цепью переноса электронов происходит через респираторный комплекс I. Геном штамма T05bT кодирует два типа НАДН:хиноноксидоредуктазоподобных комплексов: комплекс ε-протеобактериального типа (WP_205458496‒WP_205458507), который может связывать окисление пирувата с цепью переноса электронов, используя ферредоксин/флаводоксин в качестве переносчика электронов, и комплекс с высокой степенью схожести с nuoEF E. coli (WP_205457951‒WP_205457964), который может связывать множественные дегидрогеназы с цепью переноса электронов (Goris et al., 2014). Наличие комплекса ε-протеобактериального типа (ε-NADH I) характерно для всех представителей рода Sulfurospirillum (Ross et al., 2016).
Азотный метаболизм. Геном штамма T05bT содержит генный кластер, кодирующий дыхательный периплазматический комплекс нитратредуктазы Nap типа – napAGHBFLD (WP_205459257‒WP_205459263), характерный для Epsilonproteobacteria (Simon et al., 2003), и расположенный рядом с ним генный кластер нитритредуктазы nrfHAIJ (WP_205459266‒ WP_205459269). Гены редуктаз, восстанавливающих оксид азота и закись азота (nor и nos), в геноме штамма T05bT отсутствуют. Наиболее вероятно, что штамм T05bT осуществляет диссимиляционное восстановление нитрата с образованием аммония за счет канонических энзиматических систем Nap и Nrf типа.
В геноме штамма T05bT закодированы все компоненты нитрогеназного комплекса для фиксации молекулярного азота nifHDKE (WP_205459985‒ WP_205459999).
Серный метаболизм. Геном штамма T05bT не содержит генов диссимиляционной сульфитредуктазы Dsr типа и окисляющих серные соединения белков Sox. Возможно, окисление элементной серы и тиосульфата происходит с участием серии ферментов, включающих роданазу и Hdr-подобный комплекс (Zhang et al., 2021); биохимия и генетические детерминанты этих метаболических путей в настоящее время изучены недостаточно. В щелочных условиях вероятно также окисление S0, связанное с образованием растворимых полисульфидов (Ghosh, Dam, 2009). Способность изолята восстанавливать тиосульфат и элементную серу, вероятно, вызвана активностью тиосульфат/полисульфид редуктазы Phs/Psr, три субъединицы которой (PhsABC) закодированы в генном кластере WP_205459460‒ WP_205459462. Окисление сульфида, возможно, осуществляется сульфид:хинон оксидоредуктазой Sqr (WP_205458630).
Метаболизм мышьяка. Геном штамма T05bT кодирует две энзиматические системы, восстанавливающие арсенат: Arr ‒ для анаэробного дыхания с As(V) в качестве конечного акцептора электронов и Ars, обуславливающую устойчивость к ионам мышьяка. Субъединицы арсенатредуктазы ArrA (WP_205459496) и ArrB (WP_205459497) имеют 50 и 52% идентичности аминокислотных последовательностей с субъединицами хорошо изученной дыхательной арсенатредуктазы из Shewanella ANA-3 (Malasarn et al., 2008). Система детоксикации мышьяка представлена опероном, состоящим из двух копий тиоредоксин-связанной арсенатредуктазы ArsC, белками устойчивости к мышьяку и арсенит/антимонит H+ антипортером ArsB (WP_205460022‒WP_205460027).
Гидрогеназы. Геном штамма T05bT содержит один генный кластер, кодирующий две разные NiFe гидрогеназы (WP_205458701‒WP_205458715). Согласно классификатору гидрогеназ HydDB (Søndergaard et al., 2016), каталитическая единица одной из них (WP_205458711) принадлежит к [NiFe] Group 1b, которая включает дыхательные водород-поглощающие гидрогеназы, участвующие в процессах анаэробного дыхания с фумаратом, нитратом и сульфатом, и широко представленную среди Epsilonproteobacteria. Малая (WP_205458712) и цитохром b-содержащие (WP_205458710) субъединицы этой гидрогеназы обеспечивают перенос электронов к пулу хинонов. Большая субъединица второй гидрогеназы HyaB (WP_205458714) относится к [NiFe] Group 2d, которая включает дыхательные водород-поглощающие гидрогеназы, участвующие в аэробном и фумаратном дыхании. Этот тип гидрогеназ встречается у бактерий различных филогенетических групп, однако у представителей Epsilonproteobacteria ее функции не охарактеризованы. Малая субъединица этой гидрогеназы HyaA также закодирована в геноме (WP_205458715). Кроме этого, кластер содержит гены белков участвующих в сборке гидрогеназ hy-pAEDCB.
ОБСУЖДЕНИЕ
Штамм T05bT выделен из наземного грязевого вулкана Таманского полуострова. Он представляет собой факультативно анаэробную алкалофильную мезофильную бактерию.
Филогенетический анализ на основе гена 16S рРНК показал, что штамм T05bT образует отдельную линию в пределах рода Sulfurospirillum (Schumacher et al., 1993), относящегося к семейству Campylobacteraceae филума “Campylobacterota”. На момент публикации род Sulfurospirillum представлен восемью видами с валидно опубликованными именами: S. alkalitolerans (Sorokin et al., 2013), S. arcachonense (Finster et al., 1997), S. arsenophilum, S. barnesii (Stolz et al., 1999), S. cavolei (Kodama et al., 2007), S. deleyianum (Schumacher et al., 1993), S. halorespirans и S. multivorans (Luijten et al., 2003). Согласно современной практике в систематике прокариот, для установления видовой принадлежности микроорганизма рекомендуется сравнение идентичности нуклеотидных последовательностей (ANI) и уровня ДНК‒ДНК гибридизации in silico полных геномов. Однако сравнить геномы штамма T05bT и его ближайшего родственника S. alkalitolerans (DSM 24537T) в настоящее время не представляется возможным ввиду отсутствия данных секвенирования полного генома S. alkalitolerans в общедоступных базах данных. Вместе с этим, полноценным основанием для отнесения изолята к новому виду считается уровень сходства по генам 16S рРНК ниже 98.7% (Chun et al., 2018). Идентичность нуклеотидных последовательностей гена 16S рРНК между штаммом T05bT и S. alkalitolerans (DSM 24537T) составляет 98.61%, таким образом, штамм T05bT может быть отнесен к новому виду рода Sulfurospirillum.
Представители рода Sulfurospirillum широко географически распространены и были обнаружены в разнообразных экосистемах, включая нефтяные месторождения, чистые и загрязненные грунтовые воды и морские метановые сипы (GBIF Secretariat (2021). GBIF Backbone Taxonomy. Checklist dataset https://doi.org/10.15468/39omei; Jensen, Finster, 2005; Hubert et al., 2007; Cornish Shartau et al., 2009; Rowland et al., 2009; Sercu et al., 2013). Штамм T05bT является первым представителем рода, выделенным из наземного грязевого вулкана. Новый изолят имеет диапазоны pH, температуры и солености для роста, близкие к параметрам в его среде обитания.
Метаболический потенциал, кодируемый в геноме штамма T05bT, соответствует фенотипическим данным. Разнообразие используемых доноров и акцепторов электронов характерно для всех видов рода Sulfurospirillum и для представителей “Campylobacterota” в целом (van der Stel et al., 2019). Дифференцирующие характеристики штамма T05bT и членов рода Sulfurospirillum представлены в табл. 2. Наиболее заметными отличиями являются более высокий pH оптимум и более высокий диапазон солености для роста. Таким образом, на основании проведенных исследований мы предлагаем отнести штамм T05bT к новому виду рода Sulfurospirillum ‒ Sulfurospirillum tamanensis sp. nov.
Таблица 2.
Дифференцирующие характеристики штамма T05bT и представителей рода Sulfurospirillum
| Характеристика | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 |
|---|---|---|---|---|---|---|---|---|---|
| Т опт., °C | 30 | Н.д. ‒ 41 (н.д.) | Н.д. – 30 (20) | 8–30 (26) | Н.д. – 30 (н.д.) | 20–40 (30) | 20–36 (н.д.) | Н.д. (25–30) | Н.д. (30) |
| pH опт. | 9.0 | 8.5 | 7.0–7.4 | 7.5 | 7.5 | 7.0 | 7.1 | Н.д. | 7–7.5 |
| NaCl, % (вес/объем) | 0‒14 | 3.5–10.2 | 0.1 | 0.6‒4 | 0–0.8 | <1.0 | <0.2 | Н.д. | Н.д. |
| Доноры электронов | |||||||||
| Сульфид | + | + | Н.д. | Н.д. | Н.д. | Н.д. | + | Н.д. | + |
| Лактат | + | + | + | + | + | + | ‒ | + | + |
| Малат | + | ‒ | Н.д. | + | Н.д. | + | + | Н.д. | Н.д. |
| Акцепторы электронов | |||||||||
| Тиосульфат | + | + | + | ‒ | + | + | + | ‒ | + |
| Сульфит | + | ‒ | Н.д. | ‒ | ‒ | + | + | ‒ | Н.д. |
| ДМСО | + | ‒ | Н.д. | ‒ | ‒ | + | + | Н.д. | + |
| Нитрат | + | + | + | ‒ | + | + | + | + | + |
| Арсенат | + | + | ‒ | + | + | + | + | + | + |
| Кислород 3% (вес/объем) | + | ‒ | ‒ | + | + | + | + | + | Н.д. |
| Брожение | |||||||||
| Лактат | ‒ | ‒ | ‒ | Н.д. | Н.д. | ‒ | Н.д. | Н.д. | Н.д. |
| Малат | + | ‒ | Н.д. | ‒ | Н.д. | + | + | Н.д. | Н.д. |
| Пируват | + | + | ‒ | Н.д. | Н.д. | + | Н.д. | + | + |
| Фумарат | + | + | + | + | + | + | + | + | Н.д. |
| Содержание Г + Ц (мол. %) | 45.5 | 47.6 | 40.9 | 32 | 40.8 | 42.7 | 38.4 | 41.8 | 41.5 |
| Источник выделения | Грязевой вулкан | Биореактор | Пресноводныe отложения, загрязненныe мышьяком | Морской осадок | Пресноводное болото, загрязненное селенатом | Подземные воды, загрязненные нефтью | Осадок лесного пруда | Анаэробная почва, загрязненная хлорированными алифатическими соединениями | Активный ил |
Примечание. 1 ‒ T05bT (настоящее исследование); 2 ‒ S. alkalitolerans (Sorokin et al., 2013); 3 ‒ S. arsenophilum (Stolz et al., 1999); 4 ‒ S. arcachonense (Finster et al., 1997); 5 ‒ S. barnesii (Stolz et al., 1999); 6 ‒ S. cavolei (Kodama et al., 2007); 7 ‒ S. deleyianum (Schumacher et al., 1992); 8 ‒ S. halorespirans (Luijten et al., 2003); 9 ‒ S. multivorans (Luijten et al., 2003). Все штаммы используют водород, пируват, формиат, фумарат в качестве донора электронов и элементную серу, фумарат в качестве акцептора электронов. Ни один из штаммов не использует сульфат как акцептор электронов. Н.д. – данные в литературных источниках отсутствуют.
Sulfurospirillum tamanensis (ta.ma.nén.sis – лат. прил., обитающий на Тамани)
Клетки представляют собой подвижные спириллы, 0.5 мкм толщиной и 1.5–2.5 мкм длиной. Рост при температуре 6–42°C (оптимум ‒ 30°C), pH 8.0–11.0 (оптимум ‒ 9.0), концентрации NaCl 0–14% (вес/об.); использует лактат, формиат, малат, пируват, молекулярный водород, элементную серу, сульфит, тиосульфат и сульфид в качестве донора электронов и нитрат, фумарат, элементную серу, сульфит, тиосульфат, ДМСО, арсенат и кислород в качестве акцептора электронов. Продуктом восстановления нитрата является аммоний. Сбраживает малат, пируват, фумарат. Способен к микроаэробному (до 3% О2; об./об.) росту. Не использует сульфат и Fe(III) в качестве акцептора электронов. Не сбраживает глюкозу, фруктозу, сахарозу, лактат, формиат, малеинат, дрожжевой экстракт и пептон. Не диспропорционирует элементную серу, тиосульфат и сульфит.
Типовой штамм T05bT (=DSM 112596T = VKM B-3538T) выделен из наземного грязевого вулкана полуострова Тамань. Размер генома – 2.46 Мп. Содержание Г + Ц в геномной ДНК 45.5% (полногеномое секвенирование).
Список литературы
Гнатенко Г.И., Кутний В.А., Науменко П.И., Соболевский Ю.В., Шнюков Е.Ф. Грязевые вулканы Керченско-Таманской области (атлас) // Киев: Наукова Думка, 1986. С. 149.
Холодов В.Н. Грязевые вулканы: распространение и генезис // Геология и полезные ископаемые Мирового океана. 2012. Т. 4. С. 5–27.
Alain K., Holler T., Musat F., Elvert M., Treude T., Krüger M. Microbiological investigation of methane- and hydrocarbon-discharging mud volcanoes in the Carpathian Mountains, Romania // Environ. Microbiol. 2006. V. 8. P. 574–590.
Altschul S.F., Gish W., Miller W., Myers E.W., Lipman D.J. Basic local alignment search tool // J. Mol. Biol. 1990. V. 215. P. 403‒410.
Benson D.A., Boguski M.S., Lipman D.J., Ostell J., Ouellette B.F.F., Rapp B.A., Wheeler D.L. GenBank // Nucleic Acids Res. 1999. V. 27. P. 12–17.
Bonch-Osmolovskaya E.A., Sokolova T.G., Kostrikina N.A., Zavarzin G.A. Desulfurella acetivorans gen. nov. and sp. nov. ‒ a new thermophilic sulfur-reducing eubacterium // Arch. Microbiol. 1990. V. 153. P. 151–155.
Brettin T., Davis J.J., Disz T., Edwards R.A., Gerdes S., Olsen G.J., Olson R., Overbeek R., Parrello B., Pusch G.D., Shukla M., Thomason J.A., Stevens R., Vonstein V., Wattam A.R., Xia F. RASTtk: A modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes // Sci. Rep. 2015. V. 5. P. 8365.
Cheng T.W., Chang Y.H., Tang S.L., Tseng C.H., Chiang P.W., Chang K.T., Sun C.H., Chen Y.G., Kuo H.C., Wang C.H., Chu P.H., Song S.R., Wang P.L., Lin L.H. Metabolic stratification driven by surface and subsurface interactions in a terrestrial mud volcano // ISME J. 2012. V. 6. P. 2280–2290.
Chun J., Oren A., Ventosa A., Christensen H., Arahal D.R., da Costa M.S., Rooney A.P., Yi H., Xu X.-W., De Meyer S., Trujillo M.E. Proposed minimal standards for the use of genome data for the taxonomy of prokaryotes // Int. J. Syst. Evol. Microbiol. 2018. V. 68. P. 461–466.
Cornish Shartau S.L., Yurkiw M., Lin S., Grigoryan A.A., Lambo A., Park H.S., Lomans B.P., van der Biezen E., Jetten M.S.M., Voordouw G. Ammonium concentrations in produced waters from a mesothermic oil field subjected to nitrate injection decrease through formation of denitrifying biomass and anammox activity // Appl. Environ. Microbiol. 2010. V. 76. P. 4977–4987.
Dimitrov L.I. Mud volcanoes – a significant source of atmospheric methane // Geo-Mar. Lett. 2003. V. 23. P. 155‒161.
Felsenstein J. Confidence limits on phylogenies: an approach using the bootstrap // Evolution. 1985. V. 39. P. 783‒791.
Finster K., Liesack W., Tindall B.J. Sulfurospirillum arcachonense sp. nov., a new microaerophilic sulfur-reducing bacterium // Int. J. Syst. Evol. Microbiol. 1997. V. 47. P. 1212‒1217.
GBIF Secretariat (2021). GBIF Backbone Taxonomy. Checklist dataset.https://doi.org/10.15468/39omei
Ghosh W., Dam B. Biochemistry and molecular biology of lithotrophic sulfur oxidation by taxonomically and ecologically diverse bacteria and archaea // FEMS Microbiol. Rev. 2009. V. 33. P. 999–1043.
Goris T., Schubert T., Gadkari J., Wubet T., Tarkka M., Buscot F., Adrian L., Diekert G. Insights into organohalide respiration and the versatile catabolism of Sulfurospirillum multivorans gained from comparative genomics and physiological studies // Env. Microb. Special Issue: Sulfur Cycle Ecology. 2014. V. 16. I. 11. P. 3562‒3580.
Grissa I., Vergnaud G., Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats // BMC Bioinf. 2007. V. 8. P. 172.
Gross R., Pisa R., Sänger M., Lancaster C.R.D., Simon J. Characterization of the menaquinone reduction site in the diheme cytochrome b membrane anchor of Wolinella succinogenes NiFe-hydrogenase // J. Biol. Chem. 2004. V. 279. P. 274‒281.
Hazkani-Covo E., Graur D. A comparative analysis of numt evolution in human and chimpanzee // Mol. Biol. Evol. 2007. V. 24. P. 13–18.
Hubert C., Voordouw G. Oil field souring control by nitrate-reducing Sulfurospirillum spp. that outcompete sulfate-reducing bacteria for organic electron donors // Appl. Environ. Microbiol. 2007. V. 73. P. 2644–2652.
Jensen A., Finster K. Isolation and characterization of Sulfurospirillum carboxydovorans sp. nov., a new microaerophilic carbon monoxide oxidizing epsilon Proteobacterium // Ant. van Leeuwenhoek. 2005. V. 87. P. 339‒353.
Kather B., Stingl K., van der Rest M.E., Altendorf K., Molenaar D. Another unusual type of citric acid cycle enzyme in Helicobacter pylori: the malate:quinone oxidoreductase // J. Bacteriol. 2000. V. 182. P. 3204‒3209.
Khomyakova M.A., Merkel A.Y., Petrova D.A., Bonch-Osmolovskaya E.A., Slobodkin A.I. Alkalibaculum sporogenes sp. nov., isolated from a terrestrial mud volcano and emended description of the genus Alkalibaculum // Int. J. Syst. Evol. Microbiol. 2020. V. 70. P. 4914–4919.
Kodama Y., Ha L.T., Watanabe K. Sulfurospirillum cavolei sp. nov., a facultatively anaerobic sulfur-reducing bacterium isolated from an underground crude oil storage cavity // Int. J. Syst. Evol. Microbiol. 2007. V. 57. P. 827‒831.
Kokoschka S., Dreier A., Romoth K., Taviani M., Schäfer N., Reitner J., Hoppert M. Isolation of anaerobic bacteria from terrestrial mud volcanoes (Salse di Nirano, Northern Apennines, Italy) // Geomicrobiol. 2015. V. 32. P. 355–364.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874.
Luijten M.L., de Weert J., Smidt H., Boschker H.T., de Vos W.M., Schraa G., Stams A.J. Description of Sulfurospirillum halorespirans sp. nov., an anaerobic, tetrachloroethene-respiring bacterium, and transfer of Dehalospirillum multivorans to the genus Sulfurospirillum as Sulfurospirillum multivorans comb. nov. // Int. J. Syst. Evol. Microbiol. 2003. V. 53. P. 787‒793.
Malasarn D., Keeffe J.R., Newman D.K. Characterization of the arsenate respiratory reductase from Shewanella sp. strain ANA-3 // J. Bacteriol. 2008. V. 190. P. 135–142. https://doi.org/10.1128/JB.01110-07
Merkel A.Y., Chernyh N.A., Pimenov N.V., Bonch-Osmolovskaya E.A., Slobodkin A.I. Metabolic potential of the terrestrial mud volcano microbial community with a high abundance of archaea mediating the anaerobic oxidation of methane // Life. 2021. V. 11. P. 953.
Mazzini A., Etiope G. Mud volcanism: An updated review // Earth-Science Rev. 2017. V. 168. P. 81–112.
Overbeek R., Olson R., Push G.D., Olsen G.J., Davis J.J., Disz T., Edwards R.A., Gerdes S., Parrello B., Shukla M., Vonstein V., Wattam A.R., Xia F., Stevens R. The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST) // Nuclear Acids Res. 2014. V. 42. P. D206–D214.
Preiss L., Hicks D.B., Suzuki S., Meier T., Krulwich T.A. Alkaliphilic bacteria with impact on industrial applications, concepts of early life forms, and bioenergetics of ATP synthesis // Front. Bioeng. Biotechnol. 2015. V. 3. P. 75.
Ratnikova N.M., Slobodkin A.I., Merkel A.Y., Kopitsyn D.S., Kevbrin V.V., Bonch-Osmolovskaya E.A., Slobodkina G.B. Sulfurimonas crateris sp. nov., a facultative anaerobic sulfur-oxidizing chemolithoautotrophic bacterium isolated from a terrestrial mud volcano // Int. J. Syst. Evol. Microbiol. 2020. V. 70. P. 487–492.
Ross D.E., Marshall C.W., May H.D., Norman R.S. Comparative genomic analysis of Sulfurospirillum cavolei MES reconstructed from the metagenome of an electrosynthetic microbiome // PLoS One. 2016. V. 11. P. e0151214.
Rousseau C., Gonnet M., Le Romancer M., Nicolas J. CRI-SPI: a CRISPR interactive database // Bioinf. 2009. V. 25. P. 3317–3318.
Rowland H.A.L., Boothman C., Pancost R., Gault A.G., Polya D.A., Lloyd J.R. The role of indigenous microorganisms in the biodegradation of naturally occurring petroleum, the reduction of iron, and the mobilization of arsenite from West Bengal Aquifer Sediments // J. Env. Qual. 2009. V. 38. P. 1598‒1607.
Rzhetsky A., Nei M. A simple method for estimating and testing minimum-evolution trees // Mol. Biol. Evol. 1992. V. 9. P. 945–967.
Sercu B., Jones A.D.G., Wu C.H., Escobar M.H., Serlin C.L., Knapp T.A., Andersen G.L., Holden P.A. The influence of in situ chemical oxidation on microbial community composition in groundwater contaminated with chlorinated solvents // Microb. Ecol. 2013. V. 65. P. 39–49.
Schumacher W., Kroneck P.M.H., Pfennig N. Comparative systematic study on “Spirillum” 5175, Campylobacter and Wolinella species. Description of “Spirillum” 5175 as Sulfurospirillum deleyianum gen. nov., spec. nov. // Arch. Microbiol. 1992. V. 158. P. 287‒293.
Slobodkin A.I., Reysenbach A.-L., Slobodkina G.B., Baslerov R.V., Kostrikina N.A., Wagner I.D., Bonch-Osmolovskaya E.A. Thermosulfurimonas dismutans gen. nov., sp. nov. a novel extremely thermophilic sulfur-disproportionating bacterium from a deep-sea hydrothermal vent // Int. J. Syst. Evol. Microbiol. 2012. V. 62. P. 2565‒2571.
Slobodkina G.B., Merkel A.Y., Novikov A.A., Bonch-Osmolovskaya E.A., Slobodkin A.I. Pelomicrobium methylotrophicum gen. nov., sp. nov. a moderately thermophilic, facultatively anaerobic, lithoautotrophic and methylotrophic bacterium isolated from a terrestrial mud volcano // Extremophiles. 2020. V. 24. P. 177–185.
Søndergaard D., Pedersen C.N.S., Greening C. HydDB: A web tool for hydrogenase classification and analysis // Sci. Rep. 2016. V. 6. P. 34212. https://doi.org/10.1038/srep34212
Sorokin D.Y., Berben T., Melton E.D., Overmars L., Vavorakis C.D., Muyzer G. Microbial diversity and biogeochemical cycling in soda lakes // Extremophiles. 2014. V. 18. P. 791‒809.
Sorokin D.Y., Tourova T.P., Muyzer G. Isolation and characterization of two novel alkalitolerant sulfidogens from a Thiopaq bioreactor, Desulfonatronum alkalitolerans sp. nov., and Sulfurospirillum alkalitolerans sp. nov. // Extremophiles. 2013. V. 17. P. 535–543.
Stolz J.F., Ellis D.J., Blum J.S., Ahmann D., Lovley D.R., Oremland R.S. Sulfurospirillum barnesii sp. nov. and Sulfurospirillum arsenophilum sp. nov., new members of the Sulfurospirillum clade of the ε-Proteobacteria // Int. J. Syst. Bacteriol. 1999. V. 49. P. 1177‒1180.
Thompson J.D., Gibson T.J., Plewniak F., Jeanmougin F., Higgins D.G. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools // Nucleic Acids Res. 1997. V. 25. P. 4876‒4882.
van der Stel A.-X., Wösten M.M.S.M. Regulation of respiratory pathways in Campylobacterota: a review // Front. Microbiol. 2009. V. 10. P. 1719. https://doi.org/10.3389/fmicb.2019.01719
Weisburg W.G., Barns S.M., Pelletier D.A., Lane D.J. 16S ribosomal DNA amplification for phylogenetic study // J. Bacteriol. 1991. V. 173. P. 697–703.
Wolin E.A., Wolin M.J., Wolfe R.S. Formation of methane // J. Franklin Inst. 1963. V. 176. P. 737.
Yang H.M., Lou K., Sun J., Zhang T., Ma X.L. Prokaryotic diversity of an active mud volcano in the Usu City of Xinjiang, China // J. Basic Microbiol. 2012. V. 52. P. 79–85.
Yun J.H., Sung H., Kim H.S., Tak E.J., Kang W., Lee J.-Y., Hyun D.-W., Kim P.S., Bae J.-W. Complete genome sequence of the halophile bacterium Kushneria konosiri X49T, isolated from salt-fermented Konosirus punctatus // Stand. Genomic Sci. 2018. 13:19. https://doi.org/10.1186/s40793-018-0324-0
Zhang L., Qiu Y.Y., Zhou Y., Chen G.H., van Loosdrecht M.C.M., Jiang F. Elemental sulfur as electron donor and/or acceptor: Mechanisms, applications and perspectives for biological water and wastewater treatment // Water Res. 2021. V. 202. P. 117373.
Дополнительные материалы отсутствуют.


