Молекулярная биология, 2019, T. 53, № 1, стр. 64-73
Усиление трансляции РНК с помощью длинной некодирующей РНК путем привлечения ILF3 и eIF4A1 на мРНК-мишени млекопитающих
S.-H. Jin a, b, c, *, Y. Yao a, b, c, **, Q. Wu a, b, c, ***
a MOE Key Lab. Bioinformatics, Tsinghua University
100084 Beijing, China
b Center for Synthetic and Systems Biology, Tsinghua University
100084 Beijing, China
c School of Life Sciences, Tsinghua University
100084 Beijing, China
* E-mail: jinshouhong@phalab.org
** E-mail: yaoyi@phalab.org
*** E-mail: wuqiong@tsinghua.edu.cn
Поступила в редакцию 10.03.2018
После доработки 13.06.2018
Принята к публикации 15.06.2018
Аннотация
Разработанная ранее технология повышения уровня РНК (RNA enhancement, RNAe) обеспечивает усиление посттранскрипционной экспрессии специфических генов, действуя через длинные некодирующие РНК (lncRNA). Однако механизм, посредством которого ее функциональный элемент SINEB2 усиливает трансляцию, понятен не до конца. В настоящей работе RNAe-lncRNA получена методом медленного охлаждения в дважды деионизированной воде. Более того, с использованием этой структурированной lncRNA и шести потенциальных RNAe lncRNA-связывающих белков проведен РНК pull-down-анализ белков. Методом определения аффинности РНК-связывающих белков и анализа сдвига электрофоретической подвижности (EMSA) подтверждено прямое взаимодействие RNAe-lncRNA с фактором 3, связывающим энхансер гена интерлейкина (ILF3), и с эукариотическим фактором инициации трансляции 4A-I (eIF4A1) in vivo и in vitro соответственно. Мы полагаем, что RNAe-lncRNA может усиливать трансляцию путем таргетинга белка ILF3, который, в свою очередь, рекрутирует полисомы или факторы трансляции, и привлечения дополнительных белков eIF4A1, способных ускорять расплетание вторичной структуры на 5'-конце мРНК в ходе инициации трансляции. Это позволяет понять механизм, посредством которого lncRNA могут регулировать трансляцию специфических мРНК, рекрутируя белки, ассоциированные с трансляцией.
ВВЕДЕНИЕ
Длинные некодирующие РНК (lncRNA) – группа некодирующих РНК, состоящих обычно более чем из 200 н. и транскрибируемых РНК-полимеразой II. lncRNA играют важную роль в таких процессах, как эмбриогенез, метастазирование и апоптоз [1]. Однако механизмы регуляторного действия lncRNA, число которых постоянно возрастает, а функции активно изучаются, установлены только в незначительном числе случаев.
В 2012 г. был открыт новый функциональный класс lncRNA, антисмысловых по отношению к мРНК С-концевой убиквитин-гидролазы L1 (AS Uchl1), которые могут способствовать посттранскрипционной регуляции трансляции смысловой мРНК путем рекрутирования полисом на мРНК [2]. В нашей лаборатории [3], а также в других работах [4] на основе AS Uchl1 разработан универсальный инструмент для усиления трансляции специфических мРНК млекопитающих, названный “RNA enhancement” (RNAe) и SINEUP соответственно. Кроме того, с использованием медаки в качестве модели болезней человека показано, что SINEUP-cox7B позволяет избежать проявлений гаплонедостаточности [5], а у трансгенных мышей RNAe-mGH (гормон роста мыши) повышен размер и вес [6]. Результаты этих исследований показывают, что RNAe (SINEUP) можно использовать in vivo, раскрывая потенциал их применения в генотерапии для усиления экспрессии генов-мишеней при так называемых болезнях гаплонедостаточности. Кроме того, создан набор методов скрининга оптимизированных доменов RNAe-lncRNA, которые расширяют возможности применения этого подхода к любому представляющему интерес гену-мишени [7].
RNAe-lncRNA содержит два важных элемента – сегмент длиной 72 н., узнающий мРНК-мишень, и функциональный элемент 167 н. SINEB2. SINEB2 относится к подсемейству SINE, представленному ретротранспозонами, охарактеризованными у различных многоклеточных организмов [8]. Насколько нам известно, нет данных о том, что SINEB2 регулируют трансляцию. Исключением является AS Uchl1. Выяснение механизма этого особого типа регуляции важно для получения дополнительных знаний о семействе SINE.
Важную роль в функционировании RNAe-lncRNA играет вторичная структура lncRNA. Чтобы получить RNAe-lncRNA в функциональном состоянии, мы подобрали условия фолдинга RNAe-lncRNA в опытах по рефолдингу. Механизм действия RNAe-lncRNA изучали, анализируя вторичную структуру SINEB2 [9]. В представленной работе биохимические основы процесса RNAe изучены путем поиска lncRNA-связывающих белков, сопровождающих процесс трансляции.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Культуры клеток. Клетки линии HEK 293T культивировали в среде Игла, модифицированной Дульбекко (DMEM) (“Gibco”, C11885500BT, США), содержащей 10% фетальной сыворотки крупного рогатого скота (FBS) (“Gibco”, 16000-044) и 1× раствор пенициллина-стрептомицина (“Corning”, 30-002-CI) при 37°C в атмосфере с 5% CO2.
Клетки Escherichia coli. Клетки E. coli Trans1-T1 (“Transgene Biotech.”, Индия, CD501-03) и BL21 (DE3) (“Transgene Biotech.”, CD601-02) культивировали в среде Luria-Bertani (LB), содержащей соответствующие антибиотики, при постоянном перемешивании при 200 об./мин при 37°C.
Конструирование плазмид. Детали конструирования плазмид приведены в дополнительной табл. 1S (см. Приложение на сайте http://www.molecbio.ru/ downloads/2019/1/supp_Jin_rus.pdf). Ферменты для манипуляций с ДНК получены в “Fermentas” (США).
Таблица 1.
Идентифицированные методом масс-спектрометрии (MS) белки, потенциально связывающие RNAe-lncRNA
| MS-балл | Acc. No. | Ген | Белок | ||||
|---|---|---|---|---|---|---|---|
| NC | FL-RNAe | minRNAe | SINEB2 | minRNAe + + GFP-PS | |||
| 7.6 | 69.1 | 10.1 | ND | 11.6 | P68104 | eEF1A1 | Фактор элонгации 1альфа-1, EF-1альфа-1, EF-Tu, eEF1A1. |
| ND | 28.2 | 44.8 | 42.3 | 21.0 | Q12905 | ILF2 | Фактор 2, связывающий энхансер гена интерлейкина; ядерный фактор активированных T-клеток 45 кДа |
| ND | 61.7 | 112.1 | 32.4 | 62.7 | Q12906-5 | ILF3 | Фактор 3, связывающий энхансер гена интерлейкина; DRBP76, MPP4, NFAR, NF-AT-90, TCP80 |
| ND | 53.6 | 71.5 | 12.6 | 13.7 | P19338 | NCL | Нуклеолин, белок C23 |
| ND | 11.0 | 23.6 | ND | ND | Q9NZI8 | IGF2BP1 | Белок 1, связывающий мРНК инсулиноподобного фактора роста 2, IMP-1, CRD-BP, белок 1, связывающий мРНК IGF-II, член 1 семейства VICKZ, ZBP-1 |
| ND | 47.6 | ND | ND | ND | P60842 | eIF4A1 | eIF-4AI, ATP-зависимая РНК-хеликаза |
Трансфекция клеток. Клетки HEK 293T пассировали до достижения 70–90% монослоя. Проточную цитометрию (FCM) проводили, используя клетки, трансфицированные 0.2 мкг плазмиды pEGFP-C1 и 1.2 мкг плазмидной ДНК для RNAe на лунку 12-луночного планшета. Аффинность РНК-связывающих белков определяли с использованием 9.0 мкг плазмиды для определения белка и 6.0 мкг плазмиды pminRNAe или p8×MS2bs на чашку диаметром 100 мм. EMSA проводили, используя клетки, трансфицированные 15 мкг плазмидной ДНК, экспрессирующей белок, на чашку диаметром 100 мм. Трансфекцию проводили с использованием линейного полиэтиленимина (PEI) (мол. масса 25 000, “Polysciences” ФРГ, 23966-2) при соотношении ДНК : PEI = 1 : 2). Клетки инкубировали в течение 48 ч, а затем собирали.
Проточная цитометрия (FCM). Через 48 ч после трансфекции клетки обрабатывали трипсином и ресуспендировали в фосфатно-солевом буфере (PBS). Флуоресценцию зеленого флуоресцентного белка (GFP) измеряли на цитометре Flow Cytometry instrument (“BD Biosciences”, США, BD LSRFortessa) при длине волны возбуждения и эмиссии 488 и 501 нм соответственно.
Транскрипция in vitro. В качестве матриц для транскрипции in vitro использовали расщепленные HindIII линеаризованные плазмиды (pFL-RNAe, pminRNAe-HindIII, pSINEB2, pGFP-PS). Транскрипцию проводили, используя набор HiScribe™ T7 High Yield RNA Synthesis Kit (“New England Biolabs”, США, E2050S). Транскрибированные РНК экстрагировали, используя TRIzol® LS Reagent (“Ambion”, США 10296010), в соответствии с протоколом производителя. Затем РНК денатурировали и подтверждали ее целостность с помощью электрофореза в 1%-ном агарозном геле (РНК > 1 т.н.) или в 10%-ном денатурирующем полиакриламидном геле (PAGE) (РНК < 1 т.н.).
Опыты по рефолдингу РНК. В ходе первых опытов оценивали пригодность протоколов рефолдинга РНК. С этой целью определяли состояние фолдинга в 6%-ном PAGE с использованием следующих буферов и протоколов.
Буферы для рефолдинга: дважды деионизированная вода (DDW) и Трис-буфер получали в системе обработки воды Milli-Q (“Millipore”, США). Tрис-буфер: 50 мM Tрис-HCl (pH 8.0), 150 мM NaCl, 3 мM MgCl2.
Процедуры рефолдинга: быстрое охлаждение, резкое охлаждение, медленное охлаждение.
Быстрое охлаждение: раствор РНК подвергали денатурации при 80°C в нагревательном блоке в течение 5 мин, а затем немедленно охлаждали во льду.
Резкое охлаждение: РНК после денатурации при 80°C в течение 5 мин в нагревательном блоке сразу же охлаждали во льду в течение 5 мин, а затем инкубировали при комнатной температуре в течение 20 мин.
Медленное охлаждение: после денатурации при 80°C в течение 5 мин в нагревательном блоке РНК медленно охлаждали до комнатной температуры, инкубируя в выключенном нагревательном блоке, после чего выдерживали при комнатной температуре в течение 20 мин.
РНК pull-down анализ белков. РНК транскрибировали in vitro и тагировали по 3'-концу, используя набор Pierce™ RNA 3' End Desthiobiotinylation Kit (“Thermo Scientific”, 20163, CША). Полученную биотинилированную РНК подвергали рефолдингу в DDW, используя протокол медленного охлаждения, и проводили “pull down” цитоплазматических белков с помощью Pierce™ Magnetic RNA-Protein Pull-Down Kit (“Thermo Scientific”, 20164). Цитоплазматические белки клеток HEK 293T экстрагировали, используя набор PARIS™ Kit (“Ambion”, AM1921), в соответствии с инструкциями производителя.
Минорные преципитированные белки детектировали, используя окрашивание серебром (“Beyotime”, P0017s), после разделения в 10%-ном SDS-PAGE. Кроме того, после SDS-PAGE и окрашивания Сoomassie brilliant blue (“Huaxingbio”, 04808274, Китай) основные белки отправляли в Центр биомедицинского анализа Университета Цингуа (Center of Biomedical Analysis, Tsingua University) для идентификации методом LC-MS/MS-масс-спектрометрии (“Thermo Scientific”, Q-Exactive). Критерием отсечения для идентификации потенциальных RNAe-lncRNA-связывающих белков считали 10 [10].
Вестерн-блот-анализ. Белки, полученные методом “pull-down”, анализировали с помощью Вестерн-блотинга. Белки разделяли в 10%-ном SDS-PAGE. Анти-eEF1A1, -ILF3, -NCL и -eIF4A1-антитела мыши получали от фирмы “Santa Cruz Biotechnology” (sc-377315, sc-377406, sc-8031, sc-377315). Моноклональные антитела мыши с His-тагом и вторые антимышиные антитела козы, конъюгированные с пероксидазой хрена (HRP), получены от “Huaxingbio” (HX1804, HX2032). Блоты проявляли, используя реагенты для хемилюминесценции (ECL) (“Huaxiobio”, HX1868) и имиджер (GE, ImageQuant LAS 4000) для визуализации.
Аффинность РНК-связывающих белков. Через 48 ч после трансфекции плазмидами, обеспечивающими экспрессию РНК и белков, клетки HEK 293T подвергали УФ-облучению (254 нм, 2000 Вт, 400 мДж/cм2), дважды промывали холодным PBS (4 мл на чашку диаметром 100 мм), после чего добавляли 1 мл лизирующего буфера 1 (50 мM фосфат натрия pH 7.4, 300 мM NaCl, 2% Tритона X-100, 10% глицерина, 1 мM фенилметилсульфонилфторила (PMSF) (“Beyotime”, Китай, ST506-10 ml) и 400 ед./мл ингибитора РНКаз (“TaКaRa Bio”, КНР, 2313A), лизаты инкубировали с HisPur™ Cobalt Resin (“Thermo Scientific”, 89964) в течение 30 мин при 4°C. После промывки 20 мM раствором имидазола, оставшиеся белки элюировали 150 мM имидазолом. Затем РНК экстрагировали из элюата, используя TRIzol® LS Reagent (“Ambion”, 10296010). кДНК для анализа РНК синтезировали с помощью Fastquant RT Kit (“Tiangen”, Китай). Полуколичественную полимеразную цепную реакцию в реальном времени (RT-PCR) проводили с использованием SuperReal PreMix Plus (“SYBR Green”) master mix (“Tiangen”, FP205-02) на 7500 Fast Real-Time PCR System (“Applied Biosystems”, США, 7500 fast). В качестве внутреннего контроля при детекции РНК использовали 18S рРНК. RT-PCR проводили со следующими праймерами:
18S рРНК: F – CGAACGTCTGCCCTATCAACTT и R – ACCCGTGGTCACCATGGTA; minRNAe: F – TTGCTCACCATGGTGGCG и R – CAGTGTTTGGAACTCAATATCCATC; MS2bs: F – ACACGACGCTCTTCCGATCT и R – CACCATGGAAACAGACT.
Очистка белков. Через 48 ч после трансфекции плазмидами, обеспечивающими экспрессию белков с His-таг (p6×His-eEF1A1/ILF3/NCL/eIF4A1 соответственно), клетки HEK 293T снимали с подложки, добавляли 1 мл лизирующего буфера 2 (50 мM фосфата натрия, pH 7.4, 300 мM NaCl и 2% Tритона X-100, 10% глицерина, 1 мM PMSF). Лизат инкубировали с HisPur™ Cobalt Resin (“Thermo Scientific”, 89964) в течение 30 мин при 4°C. Отмывали 20 мM имидазолом, а затем оставшиеся белки элюировали 150 мM имидазолом и концентрировали ультрафильтрацией через Amicon® Ultra 3K Centrifugal Filter Device (“Millipore”, UFC501096) в соответствии с протоколом производителя, и определяли концентрацию белка, используя BCA Protein Assay Kit (“Tiangen”, 03522) и бычий сывороточный альбумин (ВSА) в качестве стандарта.
Очистка белков из клеток E. coli BL21 (DE3). После трансформации плазмидами, обеспечивающими экспрессию тагированных белков (p6×His-eEF1A1/ILF3/NCL/eIF4A1-BL21 соответственно), мы выбрали единичные колонии и перенесли их в 5 мл среды LB, а затем в 100 мл среды. После культивирования в течение 12 ч и индукции в течение 6 ч IPTG (“Sigma”, США), клетки собирали центрифугированием при 4000 g и обрабатывали ультразвуком в лизирующем буфере 3 (50 мM фосфат натрия pH 7.4, 300 мM NaCl, 0.1% Tритона X-100, 5% глицерина и 1 мM PMSF). Супернатант после центрифугирования лизата наносили на HisPur™ Cobalt Resin (“Thermo Scientific”, 89964). Промывали 20 мM имидазолом, а затем элюировали связавшиеся белки 150 мM имидазолом, концентрировали ультрафильтрацией через Amicon® Ultra 3K Centrifugal Filter Device (“Millipore”, UFC501096) и определяли концентрацию белков, используя BCA Protein Assay Kit (“Tiangen”, 03522) c BSA в качестве стандарта.
Анализ сдвига электрофоретической подвижности (EMSA). Транскрибированную in vitro РНК (0.5 мкг) подвергали рефолдингу и инкубировали с белками (6.0, 3.6, 1.8, 0.6, 0.0 мкг), очищенными из клеток HEK 293T или E. coli BL21 (DE3), в связывающем буфере (50 мM фосфат натрия pH 7.4, 100 мM NaCl, 5 мM MgCl2) при 4°C в течение 30 мин. Затем комплексы разделяли в 6%-ном PAGE на льду при 200 мВ в течение 1–2 ч. Нуклеиновые кислоты и белки окрашивали по отдельности SYBR® Green и SYPRO® Ruby, соответственно, используя Electrophoretic Mobility Shift Assay (EMSA) Kit (“Molecular Probes”, E33075, США), и визуализировали с помощью системы имиджинга (Ultra-Violet Products, GelDoc-It Ts) с УФ-эпииллюминатором при 254 нм.
Статистический анализ. Статистический анализ проводили, используя парный t-тест Стъюдента. Результаты представлены в виде средних значений (n ≥ 3) ± стандартная ошибка (SD). Значимыми считали величины при P < 0.05, P < 0.01 или P < 0.001 (отмечено одной, двумя или тремя звездочками соответственно).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Медленное охлаждение в DDW – подходящий протокол фолдинга RNAe-lncRNA
SINEB2 – функциональный элемент RNAe-lncRNA, с помощью которого в комбинации со спаривающимся сегментом minRNAe-lncRNA может выполнять свои функции (рис. 1a, б) [3, 4]. Мы транскрибировали полноразмерную RNAe (FL-RNAe) lncRNA, minRNAe-lncRNA и SINEB2 in vitro (рис. 1a) и после подтверждения чистоты полученных РНК (рис. 1в) проверяли возможность использования для их рефолдинга DDW и Трис-буфера (pH 7.4) (рис. 1г). Мониторинг состояния РНК осуществляли с помощью PAGE, в котором из-за формирования компактной 3D структуры “свернутые” РНК должны мигрировать быстрее, чем “развернутые”. Полученные результаты свидетельствуют о том, что при использовании DDW или Трис-буфера отсутствуют различия в фолдинге FL-RNAe-lncRNA и SINEB2. Однако minRNAe-lncRNA не может полностью “сворачиваться” в Трис-буфере, что подтверждается появлением четырех полос “несвернутых” РНК в дорожке геля (рис. 1г). DDW можно считать наиболее подходящей для системой для РНК, имеющих отношение к RNAe.
Рис. 1.
Определение условий фолдинга RNAe-lncRNA. а – Схема FL-RNAe-lncRNA, minRNAe-lncRNA их мишени – мРНК egfp. Показан паттерн спаривания. б – 0.2 мкг pEGFP-C1 и 1.2 мкг ДНК pRNAe-mock, pFL-RNAe или pminRNAe на лунку 12-луночного планшета использовали для котрансфекции клеток HEK 293T. После инкубации в течение 48 ч клетки собирали и детектировали флуоресценцию GFP с помощью FCM для оценки влияния FL-RNAe-egfpc1 и minRNAe-egfpc1 на мРНК egfpc1. в – Проверка чистоты РНК, транскрибированных in vitro, с помощью электрофореза в 1%-ном агарозном геле (FL-RNAe-lncRNA) или в 10%-ном денатурирующем PAGE (minRNAe-lncRNA и SINEB2). г – Анализ фолдинга РНК в DDW и Трис-буфере (50 мM Трис-HCl pH 8.0, 150 мM NaCl, 3 мM MgCl2) с помощью PAGE в 6%-ном геле. U – несвернутая РНК (отсутствие рефолдинга), F – свернутая РНК. д – Анализ рефолдинга РНК с использованием различных процедур (F1, F2 и F3) с помощью PAGE в 6%-ном нативном геле. U – несвернутая РНК (отсутствие рефолдинга); F1 – быстрое охлаждение (после денатурации при 80°C в течение 5 мин в нагревательном блоке раствор РНК немедленно охлаждали во льду); F2 – резкое охлаждение (после денатурации при 80°C в течение 5 мин в тепловом блоке РНК немедленно охлаждали во льду в течение 5 мин с последующей инкубацией при комнатной температуре в течение 20 мин; F3 – медленное охлаждение (после денатурации при 80°C в течение 5 мин в нагревательном боксе РНК медленно охлаждали до комнатной температуры, инкубируя в выключенном нагревательном боксе, после чего выдерживали в течение 20 мин при комнатной температуре. е – Проверка влияния DMSO и магния на рефолдинг RNAe-lncRNA. U – несвернутая РНК, инкубированная в DDW, 0/3/10; в растворе, содержащем 0/3/10 мM магния; D – в растворе, содержащем 25% DMSO.
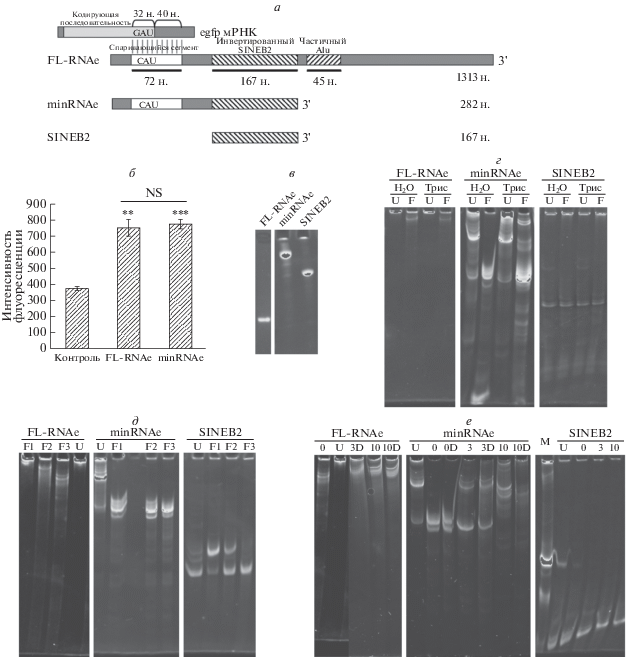
Мы сравнили также три разных метода отжига при рефолдинге РНК в DDW: быстрое, резкое и медленное охлаждение (F1, F2, F3 на рис. 1д). Обнаружено, что полнота рефолдинга изменяется в следующем порядке при разных режимах охлаждения: медленное > резкое > быстрое. РНК, имеющие отношение к RNAe, особенно SINEB2, могут сворачиваться в режиме медленного охлаждения с образованием единственной формы (рис. 1д). Напротив, быстрое охлаждение дает худший результат, поскольку при низкой (комнатной) температуре РНК остается в денатурированном состоянии (расплавленном при нагревании до 80°C). Обнаружено, что большая часть нативного SINEB2 (несвернутая группа) уже находится в свернутом состоянии (рис. 1д). Кроме того, выше свернутой РНК находится небольшое количество отстающих полос, содержащих несвернутые РНК.
С целью улучшения фолдинга разных видов РНК в буфер для фолдинга (DDW) добавляли 25% диметилсульфоксида (DMSO) или магний в различной концентрации, чтобы проверить могут ли эти агенты способствовать фолдингу RNAe-lncRNA (рис. 1е). Однако DMSO и магний не влияли на фолдинг РНК, имеющих отношение к RNAe. Таким образом, медленное охлаждение в DDW – наиболее подходит для фолдинга РНК, связанных с RNAe.
Обнаружение шести белков, потенциально связывающих RNAe-lncRNA
Известно, что RNAe действует на посттранскрипционном уровне, но детали этого процесса не установлены. Однако RNAe-lncRNA не может активировать инициацию трансляции, так как не действует на некепированную РНК (данные не приведены). Поэтому мы предположили, что RNAe-lncRNA может ускорять трансляцию мРНК-мишени, действуя преимущественно как гидовая РНК, привлекающая и связывающая белки, имеющие отношение к трансляции, к 5'-концу мРНК-мишени. Чтобы понять детали этого механизма, мы начали изучение белков, связывающих RNAe-lncRNA. С помощью in vitro транскрипции были получены FL-RNAe-lncRNA, minRNAe-lncRNA, SINEB2 и GFP-PS (сегмент спаривания: сегмент мРНК egfpc1 длиной 72 н., спаривающийся с RNAe-egfpc1) (GFP-PS и minRNAe-lncRNA образуют другую группу) (рис. 2a). Чтобы предотвратить интерференцию ядерных белков, которые могут не вовлекаться в трансляцию, из клеток НЕК 293Т экстрагировали цитоплазматические белки. Затем 3'-концы этих РНК биотинилировали с помощью набора для биотинилирования. Методом медленного охлаждения провели рефолдинг полученных тагированных РНК в DDW и pull-down-анализ РНК с помощью набора RNA-protein pull-down kit. Полученные результаты проанализировали также с помощью SDS-PAGE с окрашиванием серебром для подтверждения отмывки неспецифически связавшихся белков (Suppl. Fig. 1 , см. Приложение на сайте http:// www.molecbio.ru/downloads/2019/1/supp_Jin_rus.pdf). Из гелей вырезали полосы, содержащие белки-мишени (рис. 2б), и идентифицировали их с помощью масс-спектрометрии (MS).
Рис. 2.
Поиск и идентификация RNAe-lncRNA-связывающих белков. а – Схема РНК, использованных в опытах по “RNA pull-down protein”. б – SDS-PAGE и окрашивание серебром элюатов каждой группы. в – Клетки HEK 293T котрансфицировали, используя 9.0 мкг pEGFP-C1, p6×His-eEF1A1, p6×His-ILF3, p6×His-NCL или p6×His-eIF4A1 и 6.0 мкг pminRNAe на чашку диаметром 100 мм. После инкубации в течение 48 ч клетки подвергали УФ-облучению и собирали для анализа аффинности РНК-связывающих белков. Результаты RT-PCR показывают повышение относительного содержания minRNAe-lncRNA с помощью белков, предположительно связывающих RNAe-lncRNA. В качестве внутреннего контроля использовали 18S рРНК, и каждую группу нормировали по индивидуальной РНК. г – Вестерн-блот-анализ фракций в ходе очистки белков-мишеней, представленных на рис. 2в.
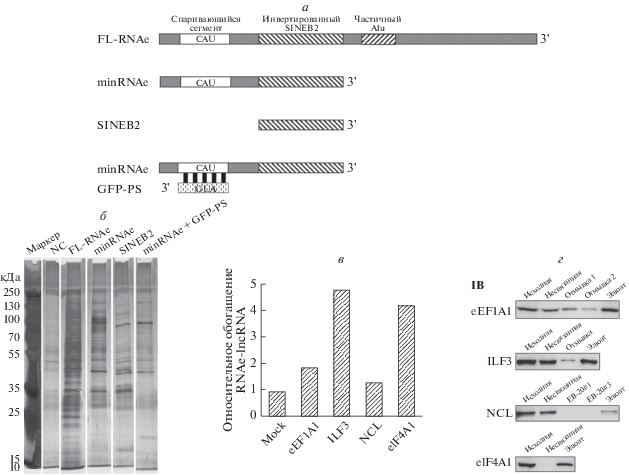
Белки, способные связывать РНК на посттранскрипционной стадии, выбирали на основе их аннотаций в базе данных UniProt [11] и подразделили на пять групп (табл. 1). В группе негативного контроля (NC) обнаружено несколько РНК-связывающих белков, но только один белок – фактор элонгации 1альфа1 (eEF1A1) – найден во всех пяти группах. Три других белка – фактор 3, связывающий энхансер гена интерлейкина (ILF3), фактор 2, связывающий энхансер гена интерлейкина (ILF2) и нуклеолин (NCL) – обнаружены в четырех экспериментальных группах и характеризуются высокими значениями MS, что указывает на высокую вероятность связывания с RNAe-lncRNA. В группах minRNAe и FL-RNAe обнаружен белок 1, связывающий мРНК инсулиноподобного фактора роста 2 (IGF2BP1). Напротив, несмотря на то, что эукариотический фактор инициации 4A-I (eIF4A1) появляется только в группе FL-RNAe, его роль в процессинге мРНК хорошо соответствует характеру процесса RNAe, поэтому мы решили, что этот фактор может быть кандидатом на роль RNAe-lncRNA-связывающего белка.
ILF2, один из шести потенциальных RNAe-lncRNA-связывающих белков всегда образует комплекс с ILF3, необходимый для выполнения его функций [12], поэтому ILF2 мог быть непрямо выявлен с помощью pull-down-анализа через ILF3. IGF2BP1 рекрутирует транскрипты-мишени на цитоплазматические мРНП, обеспечивая их транспорт и временное хранение [13]. После связывания с ними мРНК секвестрируются из пулов деградации и трансляции, что не согласуется с RNAe. Поэтому для дальнейшего изучения мы выбрали eEF1A1, ILF3, NCL и eIF4A1.
ILF3 и eIF4A1 могут взаимодействовать с RNAe-lncRNA in vivo и in vitro
Проверять взаимодействие белков и РНК in vivo можно методом анализа аффинности РНК-связывающих белков. Используя MS2 и MS2-связывающие сайты (MS2bs) в качестве положительного контроля (Suppl. Fig. 2 ), мы определили аффинность связывания RNAe-lncRNA с eEF1A1, ILF3, NCL и eIF4A1 соответственно. Обнаружено, что ILF3 и eIF4A1 существенно усиливают связывание, что свидетельствует о том, что RNAe-lncRNA действительно взаимодействует с этими белками. Напротив, eEF1A1 и NCL характеризовались слабым связыванием (рис. 2в, г).
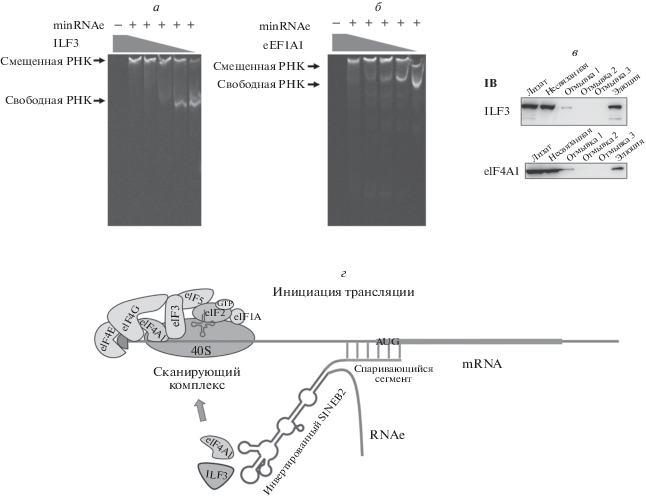
После этого мы провели EMSA с целью проверки возможности прямого взаимодействия ILF3/eIF4A1 с RNAe-lncRNA. Белки сверхэкспрессировали и выделяли из клеток HEK 293T (рис. 3в). Затем белки инкубировали со свернутой minRNAe-lncRNA в течение 30 мин при 4°C и разделяли комплексы в 6%-ном нативном PAGE. Из рис. 3a,б видно, что по мере снижения количества ILF3/eIF4A1 количество свободной minRNAe-lncRNA возрастает, а количество “смещенной” minRNAe-lncRNA снижается. К таким же результатам привел анализ белков, продуцируемых в E. coli (Suppl. Fig. 3a ,б). Наконец, тот факт, что миграция minRNAe-lncRNA не изменяется в присутствии разных количеств BSA, подтверждает, что minRNAe-lncRNA не взаимодействует неспецифически со случайными белками (Suppl. Fig. 4a ). Кроме того, ILF3 (Suppl. Fig. 4b ) и eIF4A1 (Suppl. Fig. 4c ) не влияют на миграцию РНК MS2bs, не связанной с RNAe-lncRNA, т.е. они не взаимодействуют со случайными РНК. Таким образом, результаты EMSA показали, что minRNAe-lncRNA может взаимодействовать с ILF3 и eIF4A1 in vitro, и это взаимодействие является прямым и специфическим.
Рис. 3.
Идентификация прямого взаимодействия minRNAe-lncRNA с ILF3 и eIF4A1. а, б – Клетки НЕК 293Т котрансфицировали плазмидной ДНК – 15 мкг p6×His-ILF3 или p6×His-eIF4A1 на чашку диаметром 100 мм. После инкубации в течение 48 ч из клеток выделяли белки ILF3 или eIF4A1, очищали их и использовали для EMSA с транскрибированной in vitro minRNAe. minRNAe окрашивали SYBR Green после инкубации с ILF3 (a) или eIF4A1 (б) и разделения в 6%-ном PAGE. В дорожках “–” отсутствуют minRNAe-lncRNA, в дорожки “+” добавляли 0.5 мкг minRNAe-lncRNA. Количество экспрессированных в клетках HEK 293T белков ILF3 и eIF4A1, нанесенных на дорожки, составляло 6.0, 6.0, 3.6, 1.8, 0.6 и 0.0 мкг соответственно. в – Вестерн-блотинг фракций в ходе очистки белков-мишеней из а и б. г – Гипотетический механизм RNAe: спаривающийся сегмент узнает мРНК-мишень, SINEB2 усиливает трансляцию, рекрутируя ILF3 и eIF4A1 для ускорения сборки сканирующего комплекса и процесса инициации трансляции.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Наши попытки применить RNAe в клетках дрожжей (данные не приведены) были безуспешными, что может указывать на отсутствие в клетках дрожжей некоторых факторов, необходимых для RNAe, таких как ILF3. Белок ILF3, названный также NF90, DRBP76, MPP4 и NFAR, экспрессируется в клетках животных, но не дрожжей [14]. Это белок, связывающий дцРНК (DRBP), который содержит два мотива для взаимодействия с дцРНК (RBM). ILF3 связан с функциями РНК, включая регуляцию транскрипции и трансляции [15], репликацию и трансляцию вирусного генома [16], а также с функциями некодирующих РНК [17]. ILF3 способен связываться с 3'-нетранслируемыми участками (3'-UTR) определенных мРНК, способствуя загрузке полисом на мРНК и облегчая рекрутирование факторов (40S и eIF2), необходимых для инициации трансляции в определенных условиях [18]. Поэтому мы предположили, что ILF3 может связываться с SINEB2 для рекрутирования полисом или факторов инициации трансляции, и усиливать таким образом трансляцию мРНК.
EIF4A1 – компонент комплекса eIF4F, играющий важную роль в инициации трансляции. Обладая активностью ATPaзы с DEAD-боксом и ATP-зависимой РНК-хеликазы, этот фактор может расплетать вторичную структуру вблизи 5'-конца мРНК с помощью eIF4G, eIF4B или eIF4H, что способствует связыванию преинициаторного комплекса 43S и скринингу стартового кодона [19]. Нами показано, что SINEB2 может прямо связывать eIF4A1 и in vitro, и in vivo, что указывает на возможную способность RNAe-lncRNA рекрутировать дополнительные белки eIF4A1 на мРНК-мишени для ускорения расплетания 5'-структуры и формирования комплексов 43S.
Ранее предполагали, что такие регуляторные белки, как ILF3 и eIF4A1 могут только связываться с 5'- или 3'-UTR мРНК, а петля кеп-поли(А) способствует тому, что белок, связывающий 3'‑UTR, может регулировать трансляцию. Другими словами, полагали, что только сама мРНК является единственной РНК, участвующей в регуляции собственной трансляции. Однако полученные нами результаты позволяют думать, что другая lncRNA также может усиливать трансляцию мРНК, действуя как гидовая РНК, которая направляет регуляторные белки на мРНК-мишени или даже частично замещает функции 5'/3'-UTR (рис. 3г). Предложенный механизм дает новое представление о регуляции трансляции. Более того, поскольку lncRNA этого типа, регулирующие инициацию трансляции, уникальны среди lncRNA, регулирующих экспрессию генов, выяснение механизма особого типа регуляции позволит понять, каким образом в регуляцию трансляции вовлечены какие-то еще неизвестные lncRNA.
Авторы признательны Hongwei Wang (Университет Цингуа, Китай) и Xiaohua Shen (Университет Цингуа, Китай) за обсуждение и советы, Lele Wang (Университет Цингуа, Китай) и Chuya Niu (Университет Цингуа, Китай) за помощь в проведении опытов по рефолдингу, Lichao Liu (Университет Цингуа, Китай) за помощь в определении аффинности РНК-связывающих белков.
Работа поддержана Natural Sciences Foundation of China (гранты № 31470933 31670991) и National High Technology Research and Development (863 Program) (грант № 2013AA020301).
Список литературы
Marchese F.P., Raimondi I., Huarte M. (2017) The multidimensional mechanisms of long noncoding RNA function. Genome Biol. 18, 206.
Carrieri C., Cimatti L., Biagioli M., Beugnet A., Zucchelli S., Fedele S., Pesce E., Ferrer I., Collavin L., Santoro C., Forrest A.R., Carninci P., Biffo S., Stupka E., Gustincich S. (2012) Long non-coding antisense RNA controls Uchl1 translation through an embedded SINEB2 repeat. Nature. 491, 454–457.
Yao Y., Jin S., Long H., Yu Y., Zhang Z., Cheng G., Xu C., Ding Y., Guan Q., Li N., Fu S., Chen X.J., Yan Y.B., Zhang H., Tong P., Tan Y., Yu Y., Fu S., Li J., He G.J., Wu Q. (2015) RNAe: an effective method for targeted protein translation enhancement by artificial non-coding RNA with SINEB2 repeat. Nucl. Acids Res. 43, e58.
Zucchelli S., Fasolo F., Russo R., Cimatti L., Patru-cco L., Takahashi H., Jones M.H., Santoro C., Sblattero D., Cotella D., Persichetti F., Carninci P., Gustincich S. (2015) SINEUPs are modular antisense long non-coding RNAs that increase synthesis of target proteins in cells. Front. Cell Neurosci. 9, 174.
Indrieri A., Grimaldi C., Zucchelli S., Tammaro R., Gustincich S., Franco B. (2016) Synthetic long non-coding RNAs [SINEUPs] rescue defective gene expression in vivo. SCI REP-UK. 6, 27315.
Long H.Z., Yao Y., Jin S.H., Yu Y.T., Hu X.B., Zhuang F.F., Zhang,H.S., Wu Q. (2017) RNAe in a transgenic growth hormone mouse model shows potential for use in gene therapy. Biotechnol. Lett. 39, 179–188.
Takahashi H., Kozhuharova A., Sharma H., Hirose M., Ohyama T., Fasolo F., Yamazaki T., Cotella D., Santo-ro C., Zucchelli S., Gustincich S., Carninci P. (2018) Identification of functional features of synthetic sineups, antisense lncrnas that specifically enhance protein translation. Plos One. 13, e0183229.
Kramerov D.A., Vassetzky N.S. (2005) Short retroposons in eukaryotic genomes. Int. Rev. Cytol. 247, 165–221.
Podbevšek P., Fasolo F., Bon C., Cimatti L., Reißer S., Carninci P., Bussi G., Zucchelli S., Plavec J., Gustincich S. (2018) Structural determinants of the sine b2 element embedded in the long non-coding rna activator of translation as uchl1. SCI REP-UK. 8, 3189.
Guo Q., Goto S., Chen Y., Feng B., Xu Y., Muto A., Himeno H., Deng H., Lei J., Gao N. (2013) Dissecting the in vivo assembly of the 30S ribosomal subunit reveals the role of RimM and general features of the assembly process. Nucl. Acids Res. 41, 2609–2620.
Apweiler R., Bairoch A., Wu C.H., Barker W.C., Boeckmann B., Ferro S., Gasteiger E., Huang H., Lopez R., Magrane M., Martin M.J., Natale D.A., O’Donovan C., Redaschi N., Yeh L.S. (2004) UniProt: the Universal Protein knowledgebase. Nucl. Acids Res. 32, D115–D119.
Guan D.Y., Altan-Bonnet N., Parrott A.M., Arrigo C.J., Li Q., Khaleduzzaman M., Li H., Lee C.G., Péery T., Mathews M.B. (2008) Nuclear factor 45 (NF45) is a regulatory subunit of complexes with NF90/110 involved in mitotic control. Mol. Cell Biol. 28, 4629–4641.
Stohr N., Lederer M., Reinke C., Meyer S., Hatzfeld M., Singer R.H., Huttelmaier S. (2006) ZBP1 regulates mRNA stability during cellular stress. J. Cell Biol. 175, 527–534.
Castella S., Bernard R., Corno M., Fradin A., Larc-her J.-C. (2015) Ilf3 and NF90 functions in RNA biology. Wires RNA. 6, 243–256.
Vumbaca F., Phoenix K.N., Rodriguez-Pinto D., Han D.K., Claffey K.P. (2008) Double-stranded RNA-binding protein regulates vascular endothelial growth factor mRNA stability, translation, and breast cancer angiogenesis. Mol. Cell Biol. 28, 772–783.
Li Y., Masaki T., Shimakami T., Lemon S.M. (2014) hnRNP L and NF90 Interact with hepatitis C Virus 5'‑terminal untranslated RNA and promote efficient replication. J. Virol. 88, 7199–7209.
Sakamoto S., Aoki K., Higuchi T., Todaka H., Morisawa K., Tamaki N., Hatano E., Fukushima A., Taniguchi T., Agata Y. (2009) The NF90-NF45 complex functions as a negative regulator in the microRNA processing pathway. Mol. Cell Biol. 29, 3754–3769.
Hoque M., Shamanna R.A., Guan D., Péery T., Mathews M.B. (2011) HIV-1 replication and latency are regulated by translational control of cyclin T1. J. Mol. Biol. 410, 917–932.
Svitkin Y.V., Pause A., Haghighat A., Pyronnet S., Witherell G., Belsham G.J., Sonenberg N. (2001) The requirement for eukaryotic initiation factor 4A (eIF4A) in translation is in direct proportion to the degree of mRNA 5' secondary structure. RNA. 7, 382–394.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


