Молекулярная биология, 2019, T. 53, № 2, стр. 311-323
Низкомолекулярные ингибиторы путей репарации двухцепочечных разрывов ДНК усиливают противовирусное действие системы CRISPR/Cas9 на моделях вируса гепатита B
А. П. Костюшева a, Д. С. Костюшев a, *, С. А. Брезгин a, b, Д. Н. Зарифьян a, Е. В. Волчкова c, В. П. Чуланов a, c
a Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора
111123 Москва, Россия
b Государственный научный центр “Институт иммунологии”
Федерального медико-биологического агентства России
115478 Москва, Россия
c Первый Московский государственный медицинский университет
им. И.М. Сеченова Минздрава России
119048 Москва, Россия
* E-mail: dk@rcvh.ru
Поступила в редакцию 05.09.2018
Принята к публикации 20.09.2018
Аннотация
Системы нуклеаз CRISPR/Cas9 могут эффективно подавлять репликацию вируса гепатита В, хотя кольцевая ковалентно замкнутая вирусная ДНК (ккзДНК) продолжает персистировать в ядре инфицированных клеток. Двухцепочечные разрывы в (double-strand breaks, DSBs) ккзДНК, образовавшиеся под действием CRISPR/Cas9, как правило, подвергаются репарации по пути соединения негомологичных концов (NHEJ), в результате чего в последовательности ккзДНК возникают мутации по типу вставок/делеций. Модуляция путей репарации DSBs с помощью малых молекул усиливает действие систем CRISPR/Cas9 и может способствовать элиминации ккзДНК вируса гепатита В. Проанализирована противовирусная активность системы CRISPR/Cas9 при действии ингибиторов процессов гомологичного соединения концов ДНК (homologous recombination, HR), RI-1, и NHEJ, NU7026, а также их комбинации на двух моделях инфекции вируса гепатита В in vitro: в клетках HepG2-1.1merHBV с геномом вируса гепатита В под tet-on-регулируемым цитомегаловирусным промотором и HepG2-1.5merHBV с конститутивной продукцией прегеномной РНК вируса гепатита В под промотором дикого типа. Обработка растворами низкомолекулярных соединений RI-1 или NU7026 приводила к значительному снижению уровней ккзДНК в сравнении с контрольной группой (обработка зараженных клеток DMSO) после лентивирусной трансдукции системы CRISPR/Cas9. В зависимости от модели клеток, под действием RI-1 и NU7026 уровни ккзДНК снижались в 5.0–6.5 раз по сравнению с контрольными образцами. Использование как ингибиторов HR, так и ингибиторов NHEJ способствует элиминации ккзДНК вируса гепатита В системами CRISPR/Cas9 in vitro, что в дальнейшем может быть использовано в исследованиях in vivo и рассматриваться как потенциальный инструмент для создания таргетных препаратов для лечения хронического гепатита В.
ВВЕДЕНИЕ
Хронический гепатит В (ХГВ) – тяжелое заболевание печени, связанное с хронической персистенцией вируса гепатита В (ВГВ) в гепатоцитах. Это одно из самых распространенных вирусных заболеваний человека – более 250 млн по всему миру; из которых более 1 млн ежегодно умирает от ассоциированных с ХГВ заболеваний печени: гепатоцеллюлярной карциномы и цирроза. По обновленной статистике, опубликованной в журнале Lancet в 2017 году [1], в смертности от инфекционных заболеваний лидируют вирусные гепатиты.
Основная причина персистенции ВГВ – наличие суперспирализованной кольцевой ковалентно замкнутой ДНК (ккзДНК) в ядре. Эта ДНК служит матрицей для транскрипции всех мРНК ВГВ, включая основную форму, прегеномную РНК (пгРНК) ВГВ (3.5 тыс. нуклеотидов), с которой происходит синтез минус-цепи ДНК ВГВ.
До последнего времени изучение цикла ВГВ и разработка противовирусных препаратов была во многом ограничена отсутствием пригодных моделей in vitro [2]. Одними из первых были суррогатные модели гепатита В (гепатит В уток и сурка) [3, 4] и модели первичных гепатоцитов человека [5]. На суррогатных моделях изучены фундаментальные аспекты цикла ВГВ, но, так как это все-таки модели гепатита В животных, их использование для разработки препаратов прямого противовирусного действия нерационально. Первичные гепатоциты человека труднодоступны и быстро теряют собственный фенотип в культуре клеток (в частности, экспрессию NTCP-рецептора) [6], поэтому их использование в исследованиях также затруднено, хотя последние разработки в области трехмерных культур клеток [7], совместного культивирования гепатоцитов человека в микропаттернах (организованных двухмерных островках) с фибробластами животных [8] и получение гепатоцитов из стволовых клеток человека [9] отчасти решают эти проблемы. Клетки человека HepaRG исторически считаются первой перевиваемой линией клеток, чувствительной к заражению ВГВ и наиболее близкой по своим свойствам и транскриптому к гепатоцитам. Тем не менее, использование HepaRG связано с длительной дифференцировкой клеток и плохой воспроизводимостью результатов экспериментов [10]. Главное достоинство HepaRG состоит в возможности изучения ранних этапов инфекции, то есть на стадии проникновения вируса в клетку. Изначально нечувствительные к инфекции линии клеток HepG2 [11] со вставками генома ВГВ, HepAD38 и HepG2.2.15, и модель трансфекции HepG2 плазмидой, кодирующей геном ВГВ, линейной или кольцевой ДНК ВГВ – наиболее простые и надежные модели ВГВ-инфекции. Их использование позволяет получать воспроизводимые данные по репликации вируса, при этом уровни ккзДНК ВГВ остаются довольно низкими (менее 1 копии на клетку) [12]. В проведенной нами работе использованы как классические модели ВГВ (трансфекция клеток HepG2 плазмидой, кодирующей ВГВ), так и новые модели со стабильной репликацией ВГВ (вставка генома ВГВ 1.5-mer под промотором дикого типа, то есть с низкой репликативной активностью) и с индуцируемой тетрациклином транскрипцией ВГВ (вставка генома ВГВ 1.1-mer под цитомегаловирусным промотором).
Ни одно из современных лекарственных средств не действует напрямую на ккзДНК ВГВ. Однако известно, что напрямую деградировать эту ДНК могут APOBEC-дезаминазы [13] и сайтспецифичные нуклеазы систем CRISPR/Cas9, ZFNs (нуклеазы с цинковыми пальцами), TALENs (эффекторные нуклеазы, подобные активаторам) [14–16].
Работа системы CRISPR/Cas9 основана на привлечении нуклеазы Cas9 к целевому сайту ДНК, определяемому специальным РНК-проводником [17]. Специфичность и эффективность системы CRISPR/Cas9 сравнимы с TALENs и ZFNs, однако простота работы этой системы и возможность “усовершенствования” белков Cas9 – это те факторы, которые обусловливают ее широкое использование при разработке новых лекарственных препаратов [18]. Недавно подтверждена [19] концепция, согласно которой CRISPR/Cas9 с РНК-проводниками к геному ВГВ может оказывать мощное противовирусное действие, снижая все показатели ВГВ-инфекции на различных моделях клеток. Однако, несмотря на практически полную остановку цикла ВГВ вскоре после введения CRISPR/Cas9, число копий ккзДНК значительно снижалось только к 4 неделе после трансдукции лентивирусов [14]. Считается, что следствием нуклеолитического расщепления ккзДНК системами CRISPR/Cas9 могут быть различные мутации, в том числе со сдвигом рамок считывания [20]. Хотя значительное снижение репликации ВГВ – важное достижение, о полном излечении ХГВ можно будет говорить только при полной элиминации ккзДНК. В частности, персистентность ккзДНК, даже в условиях постоянного ингибирования вирусного цикла аналогами нуклеот(з)идов, сопровождается продукцией белка НВх – основного мутагенного фактора ВГВ [21]. Таким образом, встает задача изменения судьбы двухцепочечных разрывов (double-strand breaks, DSBs) в ккзДНК с репарации на путь деградации. Известно, что репарация DSBs после нуклеолитического расщепления системами CRISPR/Cas9 реализуется по механизму соединения негомологичных концов ДНК (NHEJ) и, в редких случаях, гомологичным соединением концов ДНК (HR) [19]. Модуляция путей репарации с помощью низкомолекулярных соединений, антител и отдельных белков, многие из которых уже используются в клинической практике [22] или находятся на стадиях клинических испытаний [23], усиливает эффекты, связанные с действием CRISPR/Cas9. В частности, подавление NHEJ усиливает эффективность редактирования генома с помощью CRISPR/Cas9 [24].
Считается, что NHEJ и HR связаны реципрокным взаимодействием. В упрощенном варианте можно сказать, что подавление активности NHEJ усиливает HR и наоборот. Однако механизмы репарации DSBs гораздо более сложны, и возможность модуляции активности путей репарации DSBs с целью усиления анти-ВГВ-активности CRISPR/Cas9 требует отдельного изучения.
Нами разработана система CRISPR/Cas9, эффективно блокирующая транскрипцию и репликацию ВГВ. Показано, что низкомолекулярное соединение RI-1, которое препятствует сборке филаментной структуры RAD51 и подавляет путь HR [24], и низкомолекулярное соединение NU7026, необратимо связывающееся с каталитической субъединицей ДНК-протеинкиназы (DNA-PKcs) [23], а также их смесь не токсичны для клеток. Обработка этими соединениями клеток, зараженных ВГВ, приводит к усилению противовирусного действия систем CRISPR/Cas9.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Линии клеток. Клетки гепатомы человека HepG2, а также линии HepG2-1.1merHBV и HepG2-1.5merHBV (предоставлены проф. Dieter Glebe, Университет Гиссена, Германия) с геномом ВГВ 1.1-mer и 1.5-mer генотипа D, под tet-on-регулируемым промотором цитомегаловируса (CMV) и промотором дикого типа соответственно, культивировали в среде DMEM с высоким содержанием глюкозы (4.5 г/л) (“Thermo Fisher Scientific”, США), 1% эмбриональной сыворотки крупного рогатого скота (FBS; “Gibco”), 100 ед./мл пенициллина, 100 мкг/мл стрептомицина и 2 мМ L-глутамина при 37°С в среде с 5% СО2. Клетки рассевали в 6-луночные планшеты с 50%-ной плотностью, на следующие сутки цикл ВГВ на модели HepG2-1.1merHBV активировали доксициклином (100 нг/мл) в течение 24 ч, после чего клетки дважды промывали фосфатным буфером и трансдуцировали лентивирусами (MOI = 3.5) с Cas9-P2A-EGFP (плазмида Addgene #63592 предоставлена PhD Phil Sharp и PhD Feng Zhang) и РНК-проводником под U6-промотором (плазмида Addgene #50662 предоставлена PhD Eric Lander и PhD David Sabatini) (MOI = 3.0) либо нецелевым РНК-проводником (контроль) в смеси с полибреном (8 мкг/мкл). Через 48 ч после трансдукции клетки обрабатывали растворами ингибиторов репарации в DMSO, в качестве контроля использовали обработку DMSO в полной среде DMEM. Инкубацию с низкомолекулярными соединениями вели в течение 72 ч, меняя среду с ингибиторами каждые 24 ч.
Получение ПЦР-продукта, кодирующего РНК-проводник. Мишень в геноме ВГВ подбирали с использованием сервиса Broad Institute Genetic Perturbation Platform (portals.broadinstitute.org) и программы Geneious 7.1.9. ПЦР-продукт с U6-промотором, кодирующий РНК-проводник, получали в двухэтапной ПЦР с высокоточной полимеразой Q5 (“New England Biolabs”, США) с проведением амплификации с плазмиды pLX-sgRNA (плазмида Addgene #50662). РНК-проводник нацелен на мишень в геноме ВГВ и содержит шпильку, необходимую для распознавания белком Cas9 Streptococcus pyogenes.
Низкомолекулярные соединения. В работе использованы: специфический ингибитор каталитической субъединицы ДНК-протеинкиназы, принимающей участие в NHEJ-пути репарции ДНК, NU7026 (“Abcam Biochemicals”, Великобритания) в концентрации 20 мкМ; специфический ингибитор белка RAD51 HR-пути репарации ДНК RI-1 (“Abcam Biochemicals”) в концентрации 10 мкМ и комбинация RI-1+Nu7026 (10 мкМ и 20 мкМ соответственно). Концентрированные растворы низкомолекулярных соединений хранили в DMSO, растворяя в полной среде DMEM непосредственно перед использованием.
Анализ пролиферации и цитотоксичности методом колориметрии. Измерение пролиферации клеток HepG2-1.1merHBV под действием растворов низкомолекулярных соединений проводили с помощью набора BrdU Cell Proliferation kit (“Abcam Biochemicals”) по инкорпорации BrdU в делящиеся клетки, используя колориметрическую детекцию при 450 нм. Исследование динамики пролиферации клеток HepG2-1.1merHBV и их жизнеспособности проводили с помощью набора Cell Cytotoxicity Assay kit (“Abcam Biochemicals”), который предназначен для оценки пролиферации и жизнеспособности клеток по запатентованной технологии восстановления флуоресцентного субстрата делящимися клетками, по протоколу производителя. Эксперименты проводили в 96-луночных планшетах, рассевая клетки с 30%-ной плотностью в 8 технических повторах; для детекции флуоресцентного сигнала использовали планшетный спектрофотометр iMark (“Bio-Rad Laboratories, Inc.”, США).
Клонирование, накопление и очистка лентивирусных векторов. Клетки HEK-293T выращивали в среде DMEM, содержащей 4.5 г/л глюкозы, 1% FBS, 100 ед./мл пенициллина, 100 мкг/мл стрептомицина и 2 мМ L-глутамина. За сутки до трансфекции клетки с 70%-ной плотностью рассаживали в Т175-флаконы. Трансфекцию плазмиды dHelp (“Clontech”, США) для упаковки лентивирусов, VSV-G (“Clontech”) и плазмид, кодирующих Cas9-P2A-EGFP, РНК-проводник или нецелевой РНК-проводник, проводили с помощью полиэтиленимина (7.5 мМ) с NaCl (150 мМ). Через 24 ч среду меняли на OptiMem (“Thermo Fisher Scientific”), еще через 48 ч среду OptiMem отбирали из флаконов, пропускали через 0.45-мкм фильтр (“Corning”, США), наслаивали на стерильный 2%-ный раствор сахарозы (“Sigma”, США) в пробирки для ультрацентрифугирования и центрифугировали в роторе SW28 в течение 2 ч при 100 000 × g (ультрацентрифуга LE-80K; “Beckman Coulter”, США). По окончании центрифугирования надосадочную жидкость сливали, осадок подсушивали при комнатной температуре, добавляли 300 мкл полной среды DMEM и инкубировали при 4°С в течение 2 ч, после чего осадок лентивирусных частиц тщательно ресуспендировали и, отобрав 20-мкл аликвоты, замораживали при –70°С.
Определение титра лентивирусов. Клетки суспензионной культуры SupT1 инкубировали в среде RPMI-1640 (“Thermo Fisher Scientific”) c 2% FBS и 2 мМ L-глутамина, без антибиотиков. В день трансдукции клетки высеивали на 48-луночный планшет по 1 × 105 клеток на лунку и добавляли лентивирус в различных разведениях (в трех повторах каждое). Через 24 ч клетки промывали фосфатным буфером и культивировали в полной среде RPMI-1640 в течение 64 ч, после чего клетки осаждали в течение 5 мин при 500 × g и ресуспендировали в 300 мкл фосфатного буфера. Процентное cодержание GFP-позитивных клеток определяли на флуоресцентном проточном цитометре NovoCyte (“ACEA Biosciences”, США). Число трансдуцирующих единиц в 1 мл определяли, как описано ранее (Lentiviral Transduction Protocol, “Sigma Aldrich”).
Микроскопия. Клетки в 6-луночных планшетах анализировали на 3 сутки после трансдукции, используя технологии флуоресцентной микроскопии (при длине волны 488 нм) и микроскопии в светлом поле (Olympus IX71; “Olympus”, Япония),
Выделение нуклеиновых кислот. Выделение нуклеиновых кислот проводили с помощью набора РИБО-преп (“ИнтерЛабСервис”, Россия) по протоколу производителя, добавляя лизирующий буфер непосредственно к клеткам после удаления среды и двукратного промывания фосфатным буфером. Для выделения пгРНК и мРНК S-белка (S-РНК) ВГВ нуклеиновые кислоты обрабатывали ДНКазой I без РНКазной активности (“Thermo Fisher Scientific”) в течение 30 мин при 37°С, после чего повторяли выделение РНК с помощью набора РИБО-преп и проводили реакцию обратной транскрипции, используя набор РЕВЕРТА-L (“ИнтерЛабСервис”). Для выделения ккзДНК ВГВ полученную ДНК обрабатывали ферментом Plasmid-safe ATP-dependent DNase (“Epicentre”, США) в течение 12 ч при 37°С (для разрушения всех форм ДНК, кроме ккзДНК) и инактивировали фермент в течение 30 мин при 70°С.
Полуколичественный ПЦР-анализ. Полуколичественный ПЦР-анализ проводили со специфическими праймерами и зондами TaqMan для пгРНК ВГВ, S-РНК ВГВ, GAPDH, ДНК ВГВ, ккзДНК ВГВ (таблица) на приборе Rotor Gene 6000. Праймеры на ген β-глобина использовали из коммерчески доступного набора АмплиСенс® HBV-FL (“ИнтерЛабСервис”).
Таблица 1.
Праймеры и зонды, использованные в работе
| Мишень | Типa | Последовательностьb | Тотжига, °C |
|---|---|---|---|
| ккзДНК | f | 5'-CCGTGTGCACTTCGCTTCA-3' | 60 |
| r | 5'-GCACAGCTTGGAGGCTTGA-3' | ||
| p | (FAM) 5'-CATGGAGACCACCGTGAACGCCC-3' (BHQ1) | ||
| пгРНК | f | 5'-GGTCCCCTAGAAGAAGAACTCCCT-3' | 62 |
| r | 5'-CATTGAGATTCCCGAGATTGAGAT-3' | ||
| p | (FAM) 5'-TCTCAATCGCCGCGTCGCAGA-3' (BHQ1) | ||
| S-РНК | f | 5'-TCCTCCAAСTTGTCCTGGTTATC-3' | 60 |
| r | 5'-AGATGAGGCATAGCAGCAGGAT-3' | ||
| p | (FAM) 5'-ATGATAAAACGCCGCAGACACATCCAGC-3' (BHQ1) | ||
| мРНК Cas9 S. pyogenes | f | 5'-TCCTGCTGAGCGACATCCTG-3' | 56 |
| r | 5'-GCGTAGCCGTTCTTGCTCTG-3' | ||
| p | (FAM) 5'-TCAGGCAGCTGCTGCCGCACGA-3' (BHQ1) | ||
| GAPDH | f | 5'-CAACGGATTTGGTCGTATTGG-3' | 51 |
| r | 5'-GCAACAATATCCACTTTACCAGAGTTAA-3' | ||
| p | (FAM) 5'-CGCCTGGTCACCAGGGCTGC-3' (BHQ1) | ||
| РНК-проводник | f | 5'-TAAAGAATTTGGAGCTACTGGTTTTAGAGCTAGAAATAG-3' | 55 |
| r | 5'-CAGTAGCTCCAAATTCTTTACGGTGTTTCGTCCTTTC-3' | ||
| Незначащий РНК-проводник | f | 5'-GGGGCCACTAGGGACAGGATGTTTTAGAGCTAGAAATAG-3' | 55 |
| r | 5'-TATATAGCTAGCAAAAAAAGCACCGACTCGG-3' |
Определение уровней HBsAg. Определение секретируемого антигена ВГВ, HBsAg, в культуральной среде проводили с помощью иммуноферментного анализа, используя набор Architect HBsAg (“Abbott Laboratories”) на приборе Abbott Architect HBsAg (“Abbott Laboratories”) после пропускания среды через 0.2-мкм фильтры (“Corning”) для удаления погибших клеток. Данные по количествам HBsAg представлены в международных единицах/мл (МЕ/мл) с учетом количества клеток в образце на момент забора культуральной среды.
Статистика. Статистическую обработку проводили с помощью t-критерия Стьюдента или однофакторного дисперсионного анализа (где применимо) с использованием программного обеспечения IBM SPSS Statistics 19.0.0.0; попарные апостериорные сравнения производили с помощью критерия Тьюки. Различия считали статистически достоверными при p < 0.05.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Система CRISPR/Cas9 значительно подавляет транскрипцию и репликацию ВГВ на модели котрансфекции
С целью определения эффективности противовирусного действия системы CRISPR/Cas9 с одиночным РНК-проводником клетки HepG2 котрансфицировали с помощью реагента Lipofec-tamine 2000 (“Thermo Fisher Scientific”) плазмидой 1.1merHBV (предоставлено Dieter Glebe, Гиссен, Германия), плазмидой Cas9-P2A-EGFP (Addgene #63592) и ПЦР-продуктом, кодирующим РНК-проводник к геному ВГВ или незначащий РНК-проводник (рис. 1а). Через 24 ч клетки отмывали дважды фосфатным буфером, добавляли полную среду и снимали через 48 ч. Противовирусный эффект оценивали по экспрессии пгРНК, основного транскрипта ккзДНК ВГВ и S-РНК ВГВ на 3, 4 и 7 сутки после трансфекции, а также по количеству секретируемых в культуральную среду HBsAg и ДНК ВГВ в сравнении с контролем на 4 сутки после трансфекции. Кодируемый РНК-проводник ложится на область core/pre-core генома ВГВ – ключевой регион для транскрипции пгРНК (рис. 1б).
Рис. 1.
Влияние системы CRISPR/Cas9 на транскрипцию и репликацию ВГВ в клетках HepG2. а – Схема эксперимента (см. пояснения в тексте). б – Карта генома ВГВ и целевой регион РНК-проводника. Pol – ген полимеразы, preC/C – гены кор- и прекор-регионов, EnhI/EnhII – регионы энхансеров I и II, штрихом отмечен регион Х-гена. в – Уровни пгРНК и S-РНК ВГВ на 3, 4 и 7 сутки после трансфекции системы CRISPR/Cas9 с целевым РНК-проводником (РНКп) или с незначащим (К). Данные воспроизведены в 5 независимых экспериментах. Уровни пгРНК и S-РНК нормализованы относительно экспрессии мРНК Cas9 в трансфицированных клетках. Уровни экспрессии секретируемой ДНК ВГВ (n = 3) (г) и белка HBsAg (n = 3) (д) при действии системы CRISPR/Cas9 относительно контрольного образца через 4 суток после трансфекции. Относительные уровни рассчитывали с помощью метода ΔΔCt. Планки погрешностей соответствуют стандартным отклонениям. **** p < 0.0001.
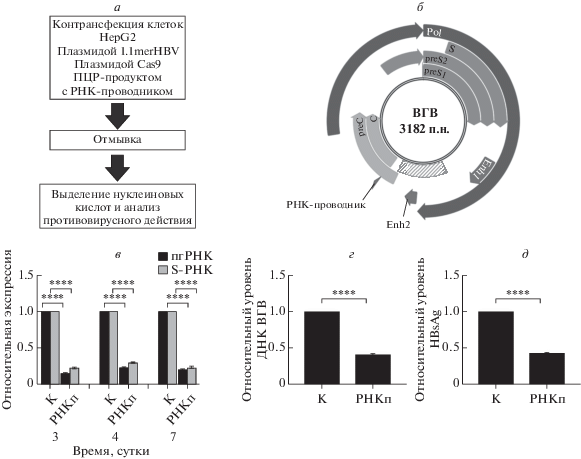
Таким образом, трансфекция системы CRISPR/ Cas9 в клетки HepG2 вызывала снижение транскрипции ВГВ более чем на 90% (рис. 1в). Важно заметить, что транскрипция ВГВ фактически прерывалась за счет CRISPR/Cas9 и не восстанавливалась к 7 суткам после трансфекции. На 4 сутки после трансфекции измеряли уровни секретируемой ДНК ВГВ и HBsAg. Оба параметра были более чем в 2 раза ниже в клетках с CRISPR/ Cas9, чем в контрольном образце (рис. 1г, д). Ввиду особенностей выделения ккзДНК ВГВ (см. раздел “Экспериментальная часть”) оценить ее уровни на модели котрансфекции невозможно, поэтому влияние CRISPR/Cas9 системы и растворов низкомолекулярных соединений на экспрессию ккзДНК ВГВ исследовали на моделях стабильных клеточных культур.
Сравнительный анализ транскрипции и репликации ВГВ на моделях клеток гепатомы
Клетки HepG2-1.1merHBV и HepG2-1.5merHBV, содержащие интегрированные трансгены генома ВГВ 1.1mer и 1.5mer под цитомегаловирусным промотором и промотором дикого типа соответственно, воспроизводят полноценный инфекционный цикл ВГВ, включая образование ккзДНК, экспрессию РНК ВГВ и секрецию частиц ВГВ (рис. 2а). Tet-on-регулируемая экспрессия пгРНК в клетках HepG2-1.1merHBV позволяет запускать цикл вируса при добавлении доксициклина и прекращать экспрессию пгРНК с геномной вставки при изъятии доксициклина из культуральной среды. Таким образом, после удаления доксициклина ВГВ транскрибируется только с образованных de novo матриц ккзДНК. В клетках HepG2-1.5merHBV происходит конститутивная экспрессия пгРНК со вставки генома ВГВ и с эписомальной ккзДНК. S-РНК и X-РНК (мРНК X‑белка) транскрибируются с внутренних промоторов и не регулируются механизмами tet-on.
Рис. 2.
Сравнительный анализ транскрипции и репликации ВГВ в линиях клеток гепатомы. а – Цикл ВГВ (HBcAg – кор-антиген ВГВ, ЭР – эндоплазматический ретикулум, kb – тыс. нуклеодидов). б – Динамика транскрипции ВГВ (пгРНК и S-РНК, нормализация на мРНК GAPDH) в клетках HepG2-1.1merHBV (1.1) и HepG2-1.5merHBV (1.5) на 3, 4 и 7 сутки. в – Динамика изменения уровней ДНК ВГВ и ккзДНК (нормализация на ген β-глобина) в клетках HepG2-1.1merHBV (1.1) и HepG2-1.5merHBV (1.5). г – Уровни экспрессии HBsAg в линиях клеток гепатомы. Измерения проведены в трех биологических повторах. Планки погрешностей соответствуют стандартным отклонениям. **p < 0.01, ***p < 0.001, ****p < 0.0001.
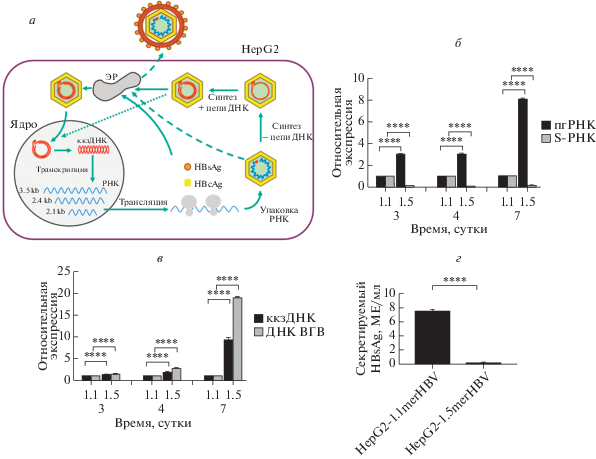
В результате анализа транскрипции и репликации ВГВ в динамике выявлено, что уровни пгРНК (рис. 2б), а также ккзДНК и ДНК ВГВ (рис. 2в) стабильно и статистически значимо выше в клетках HepG2-1.5merHBV по сравнению с HepG2-1.1merHBV. На 3 сутки экспрессия пгРНК между клетками мало отличалась, так как к этому моменту в клетках HepG2-1.1merHBV с мощного CMV-промотора успевает наработаться сравнимое с HepG2-1.5merHBV количество транскрипта. Позже уровни транскрипции пгРНК и репликации вируса в клетках 1.5HBV становятся существенно выше, так как транскрипция пгРНК в этих клетках продолжается и с генома, и с ккзДНК, в то время как в клетках 1.1merHBV пгРНК может нарабатываться только с ккзДНК, образованной в первые сутки после активации трансгена ВГВ. В клетках HepG2-1.1merHBV наблюдается более активная транскрипция S-РНК (приблизительно в 10 раз; см. рис. 2б) и секреция HBsAg (приблизительно в 50 раз; см. рис. 2г), что связано с масштабной продукцией S-РНК со вставки генома.
Лентивирусная трансдукция CRISPR/Cas9 снижает репликацию ВГВ на моделях хронической инфекции
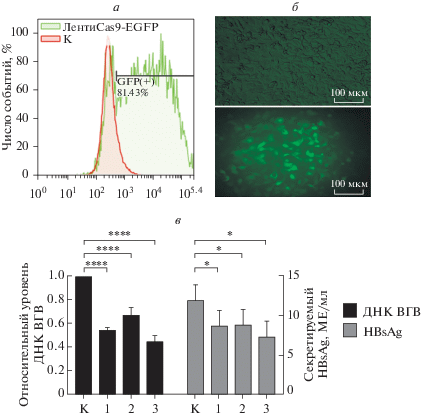
Клетки HepG2-1.1merHBV обрабатывали доксициклином (100 нг/мл) для активации tet-on-промотора, через 24 ч отмывали дважды фосфатным буфером и трансдуцировали лентивирусными векторами с помощью полибрена. На следующие сутки после трансдукции среду с лентивирусами удаляли и добавляли полную среду. По результатам проточной цитофлуориметрии (рис. 3а) и флуоресцентной микроскопии (рис. 3б), эффективность лентивирусной трансдукции при MOI 3.5 составила 83.8 ± 3.11%. Наименьшие количества лентивирусов с РНК-проводником и нецелевым РНК-проводником, способные подавлять вирусный цикл, выбирали, основываясь на изменении в секреции HBsAg и ДНК ВГВ на 3 сутки после трансдукции (рис. 3в). В связи с тем, что существенных отличий в этих показателях не обнаружили для лентивирусных частиц с РНК-проводником при значениях MOI, равных 3 (1), 6 (2) и 9 (3), мы использовали наименьшую дозу.
Рис. 3.
Выбор условий трансдукции клеток гепатомы лентивирусными векторами с CRISPR/Cas9. а –Трансдуцированные лентивирусом (ЛентиCas9-EGFP) клетки, продуцирующие EGFP, детектировали на канале 495/519 нм как зеленые флуоресцентные клетки. Контроль (нетрансдуцированные клетки) отмечен красным, трансдуцированные клетки отмечены зеленым. Зеленые флуоресцентные клетки выделены ограничительной линией. б – Микрофотографии трансдуцированных клеток в светлом поле (сверху) и на зеленом канале (снизу); увеличение 10×. в – Подбор оптимального количества лентивируса с РНК-проводником на основании данных по влиянию на уровни секретируемой ДНК ВГВ (черные столбцы), нормированные на экспрессию гена β-глобина, и секретируемого HBsAg (серые столбцы) относительно показателей в контрольных клетках. Контрольная группа клеток (К) трансдуцирована лентивирусом с Cas9-P2A-EGFP (MOI = 3.5); экспериментальные группы клеток трансдуцированы лентивирусом с Cas9-P2A-EGFP (MOI = 3.5) и лентивирусом с РНК-проводником при MOI 3 (1); 6 (2) или 9 (3).
Ингибиторы NHEJ/HR не оказывают цитотоксического действия и вызывают незначительные изменения в транскрипции ВГВ
Низкомолекулярное соединение NU7026 необратимо связывается с фактором DNA-PKcs, тем самым подавляя активность пути NHEJ. Соединение RI-1 ингибирует путь HR, препятствуя сборке филаментной структуры RAD51. Выбранные концентрации RI-1 и NU7026, а также их комбинация не оказывали цитотоксического действия на клетки HepG2-1.1merHBV. Это продемонстрировано по результатам трех различных тестов, включая анализ пролиферации клеток (рис. 4а,б,д) и анализ жизнеспособности клеток (рис. 4в,г) при инкубации с этими соединениями в течение 24 и 72 ч. Во всех временных точках изученные параметры не отличались значимо от контрольных величин.
Рис. 4.
Анализ цитотоксичности RI-1 и NU7026 и их влияния на репликацию ВГВ. Анализ пролиферации клеток HepG2-1.1merHBV при обработке RI-1 (10 мкМ), NU7026 (20 мкМ) или их комбинацией (RI-1/NU7026 в концентрациях 10 мкМ/20 мкМ) в течение 48 ч (а) и 72 ч (б) с использованием набора Cytotoxicity kit. Число клеток прямо пропорционально оптическому поглощению при 490 нм (А490). Контрольные клетки обрабатывали DMSO (0.115% в культуральной среде, что соответствует максимальному содержанию DMSO в группе RI-NU). Анализ жизнеспособности клеток HepG2-1.1merHBV относительно контрольного образца при обработке препаратами RI-1, NU7026 или RI-1/NU7026 в течение 24 (в) и 72 ч (г) с использованием набора Cytotoxicity kit. д – Анализ пролиферация клеток HepG2-1.1merHBV при обработке RI-1, NU7026 илиRI-1/NU7026 в течение 24 и 72 ч. Скорость пролиферации клеток оценивали по включению в них бромуридина (BrdU) в конечной временной точке, измеряя оптическое поглощение при 450 нм (A450). Контрольные клетки обрабатывали DMSO. Планки погрешностей соответствуют стандартным отклонениям. Отличия считались значимыми при p < 0.05. е – Клетки HepG2-1.1merHBV и HepG2-1.5merHBV обрабатывали RI-1, NU7026 или RI-1/NU7026 в течение 24 и 72 ч и оценивали репликацию ВГВ по экспрессии пгРНК и уровню ккзДНК. Измерения проведены в трех биологических повторах. Планки погрешностей соответствуют стандартным отклонениям. *p < 0.05, ****p < 0.0001.
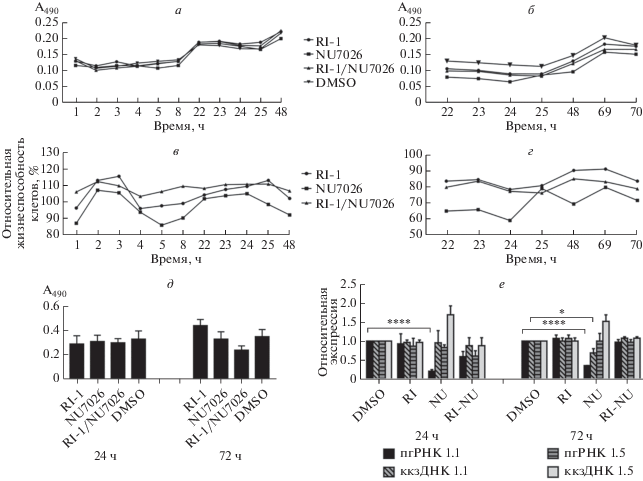
Ранее показано, что ВГВ использует сигнальные пути, активируемые DSBs и одноцепочечными разрывами, для собственной репликации [25]. Нами исследовано влияние ингибиторов DNA-PKcs (NU7026) и RAD51 (RI-1) на уровни ккзДНК и экспрессию пгРНК в обеих клеточных линиях. В выбранных условиях RI-1 практически не оказывал влияния на инфекционный цикл ВГВ, в то время как NU7026 снижал экспрессию пгРНК в клетках HepG2-1.1merHBV на 80% (p < < 0.001) через 24 ч и на 65% (p < 0.001) через 72 ч. Обработка клеток NU7026 приводила к незначительному снижению уровня ккзДНК в клетках HepG2-1.1merHBV через 72 ч (рис. 4е).
Ингибиторы репарации усиливают противовирусное действие CRISPR/Cas9 на ВГВ
С целью проанализировать действие исследуемых ингибиторов на противовирусную активность CRISPR/Cas9, клетки HepG2-1.1merHBV и HepG2-1.5merHBV через 48 ч после трансдукции обрабатывали RI-1 и NU7026 в выбранных концентрациях и каждые 24 ч заменяли культуральную среду на свежую с теми же концентрациями ингибиторов. Инкубацию проводили в течение 3 суток, после чего культуральную среду использовали для анализа HBsAg. Клетки промывали дважды фосфатным буфером и лизировали с последующим анализом компонентов жизненного цикла ВГВ (рис. 5а).
Рис. 5.
Влияние RI-1 и NU7026 на противовирусную активность CRISPR/Cas9. а – Схема эксперимента (см. пояснения в тексте). б – Схематическое изображение действия RI-1 и NU7026 как ингибиторов HR (I) и NHEJ (II). При репарации двухцепочечных разрывов ДНК RI-1 ингибирует фактор RAD51 и блокирует путь HR; NU7026 необратимо связывается с DNA-PKcs и блокирует путь NHEJ. Ферменты системы CRISPR/Cas9 могут вносить двухцепочечные разрывы в ккзДНК ВГВ (III) по целевым мишеням. MRN, CtIP, RPA, RAD51, BRCA/BRCA2 – белки и белковые комплексы пути HR. Ku70/80, DNA-PKcs, Artemis, DNA pol – белки и белковые комплексы пути NHEJ. в – Экспрессия пгРНК ВГВ относительно мРНК GAPDH после трансдукции клеток HepG2-1.1merHBV (1.1) и HepG2-1.5merHBV (1.5) лентивирусами с Cas9 с нецелевым РНК-проводником (контроль) и Cas9 c целевым РНК-проводником (РНКп) и обработки NU7026, RI-1 или NU7026/RI-1. г – Уровни ккзДНК относительно гена β-глобина. д – Уровни секретируемого HBsAg относительно контрольного образца с нецелевым проводником. е – Уровни секретируемой ДНК ВГВ относительно контрольного образца. Эксперименты проведены в трех биологических повторах. Планки погрешностей соответствуют стандартным отклонениям.*p < 0.05.
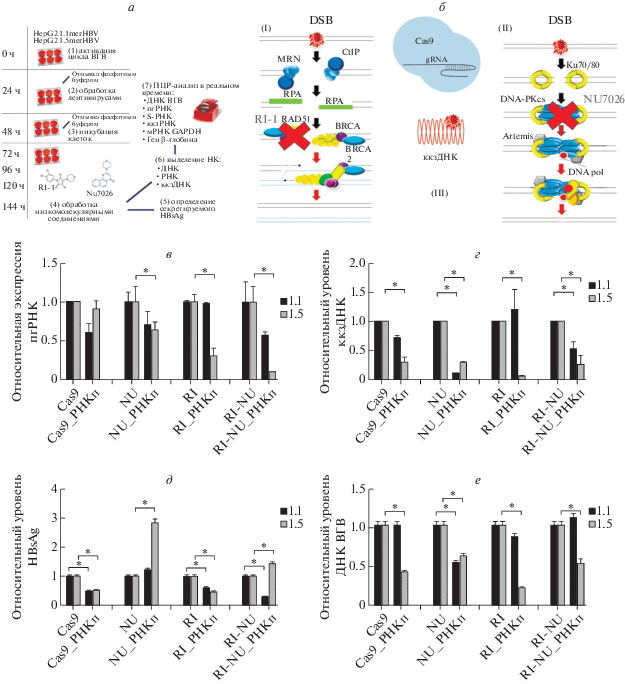
Нуклеазы системы CRISPR/Cas9 могут вносить DSBs в две основные формы ДНК ВГВ: в линейную двухцепочечную ДНК и ккзДНК, – которые локализуются в ядре инфицированных клеток (рис. 5б). При репарации DSBs в геноме ВГВ преимущественно используется NHEJ-путь (рис. 5б (II)) [19].
В условиях эксперимента лентивирусная трансдукция модельных клеток снижает уровни всех исследованных маркеров ВГВ в клетках 1.1 и 1.5: пгРНК на 39% (p < 0.05) и 10%; ккзДНК на 28% (p < 0.05) и 70% (p < 0.05); HBsAg на 51% (p < 0.05) и 48% (p < 0.05) соответственно (рис. 5в–д). Обработка клеток 1.1 и 1.5 соединением NU7026 давала выраженный эффект по ккзДНК: снижение на 89% (p < 0.05) и 70% (p < 0.05) соответственно (рис. 5г). Кроме того, использование NU7026 на клетках 1.5 приводило к статистически значимому снижению пгРНК (рис. 5в). Уровни HBsAg не снижались, а, наоборот, несколько возрастали при совместном действии системы CRISPR/Cas9 и NU7026 на клетки 1.1 и 1.5: в 1.22 (p < 0.05) и 2.83 раза (p < 0.05) соответственно (рис. 5д). При обработке клеток 1.5 RI-1 действие CRISPR/Cas9 усиливалось по отношению к ккзДНК (снижение уровня на 94%, p < 0.01; рис. 5г) и пгРНК (на 69%, p < 0.05; рис. 5в). Следует заметить, что в клетках 1.1 уровни пгРНК и HBsAg практически не отличались от контроля с РНК-проводником в DMSO. Обработка клеток комбинацией ингибиторов, RI-1/NU7026, давала неоднозначный эффект. Так, экспрессии пгРНК снижалась на 42% и 89% (p < 0.01) в клетках 1.1 и 1.5 соответственно, уровни ккзДНК снижались соответственно на 47 и 74% (p < 0.05), а содержание HBsAg снижалось на 70% (p < 0.05) в клетках 1.1 и увеличивалось в 1.43 раза в клетках 1.5 (p < 0.05) в сравнении с контрольными клетками (рис. 5в–д). Изменения в уровнях секретируемой ДНК ВГВ в целом отображали влияние CRISPR/Cas9 на уровни ккзДНК в тех же клетках (рис. 5е). В группе контроля уровни секретируемой ДНК ВГВ снижаются на клетках 1.5 более чем в 2 раза (p < 0.05). В целом, уровни ДНК ВГВ практически не отличались от контроля во всех экспериментальных группах на клетках 1.1 за исключением клеток, обработанных NU7026. При обработке клеток 1.1 и 1.5 NU7026 уровни ДНК ВГВ были на уровне 50% от контрольных значений (p < 0.05).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Ранее показано, что системы CRISPR/Cas9 могут вносить двухцепочечные разрывы в геном ВГВ и прерывать цикл вируса; хотя при этом уровни ккзДНК ВГВ, высокоперсистентной формы генома и ключевого компонента цикла вируса, мало изменяются [14, 19]. В подобранных нами условиях при первичной оценке противовирусного действия системы CRISPR/Cas9 транскрипция и репликация ВГВ в клетках HepG2 значительно снижались (от 50 до 90% для разных параметров). Согласно опубликованным данным, CRISPR/Cas9-система с отдельными РНК-проводниками эффективно блокировала транскрипцию и репликацию ВГВ [14, 26, 27]. Мишенью выбранного нами РНК-проводника системы CRISPR/Cas9 служит pre-core/core регион генома ВГВ, необходимый для считывания пгРНК – основного транскрипта ВГВ, который служит матрицей для синтеза кольцевой частично двухцепочечной ДНК (кчдДНК), секретирумой в составе вирионов формой генома. Внесение разрывов в ккзДНК ВГВ с помощью системы CRISPR/Cas9 может приводить к частичной деградации ккзДНК, а также внесению мутаций со сдвигами рамок считывания и блокированию образования транскриптов. Важно отметить, что помимо снижения уровней пгРНК, использование CRISPR/Cas9 снижало уровень S-РНК, считываемой с отдельной рамки. Кроме того, выявлено снижение продукции HBsAg и секретируемой ДНК ВГВ. Из этого следует, что действие сайтспецифичных нуклеаз в регионе pre-core/core фактически нарушает образование РНК ВГВ с нескольких кодирующих регионов ккзДНК, существенно подавляет репликацию ВГВ и продукцию вирусных частиц.
Уровни ккзДНК после лентивирусной трансдукции CRISPR/Cas9 снижались на 30%–70% в зависимости от использованной модели. Мы наблюдали значительно более сильное снижение уровней ккзДНК ВГВ, чем описано ранее – Ramanan с соавт. [14] регистрировали снижение пула ккзДНК на 70 ± 7% через 21 сутки после трансдукции.
Известно, что, наряду с деградацией ккзДНК, нуклеазы системы CRISPR/Cas9 могут вносить разрывы, которые репарируются по пути HR и NHEJ с формированием мутаций, сдвигающих или не сдвигающих рамки считывания [28]. Считается, что ингибирование пути NHEJ приводит к активации системы HR и наоборот. Таким образом, теоретически, действие NU7026 будет способствовать репарации ккзДНК по бесшовному механизму HR либо приводить к деградации ккзДНК, в то время как RI-1 будет усиливать частоту мутаций по типу вставок/делеций. Известны случаи использования низкомолекулярных соединений для повышения эффективности генетического редактирования с помощью нуклеаз CRISPR/Cas9 [23, 29]. Но никто не исследовал, как изменяются уровни ккзДНК ВГВ при одновременном действии CRISPR/Cas9 и ингибиторов NHEJ/HR. По результатам нашего исследования подавление активности путей NHEJ/HR репарации DSBs способствует снижению уровней ккзДНК на 70–89% при обработке NU7026 и на 58–94% при обработке RI-1. Обработка клеток смесью этих соединений не давала синергетического эффекта (снижение на 47–74%).
Из работы Seeger с соавт. [28] известно, что значительная доля мутаций, индуцируемых CRISPR/ Cas9 в ккзДНК ВГВ, не влияет на транскрипцию и репликацию вируса. Наиболее вероятно, что при подавлении активности NHEJ/HR CRISPR/Cas9 более эффективно блокирует транскрипцию вирусного генома за счет образования сложных мутаций, вносимых в ккзДНК. Снижение уровней ккзДНК при использовании NU7026 и RI-1 может происходить за счет подавления образования ккзДНК de novo (снижения репликации вируса) или за счет усиленной деградации ккзДНК под действием CRISPR/Cas9.
Несмотря на многообещающие данные по влиянию двух исследованных нами соединений, NU7026 и RI-1, на уровни ккзДНК, при обработке NU7026 происходило устойчивое увеличение секреции HBsAg ВГВ; при этом уровни ДНК ВГВ в культуральной среде при использовании CRISPR/ Cas9 снижались. Вирионы ВГВ заключены в оболочку, на поверхности которой экспонирован белок S, а внутренний капсид содержит кчдДНК. Таким образом, увеличение продукции HBsAg при действии CRISPR/Cas9 совместно с NU7026 не связано с усилением продукции вирионов (содержащих ДНК ВГВ). Увеличение продукции HBsAg может быть обусловлено особенностями моделей трансгенных клеток, а именно с усилением транскрипции мРНК белка S, экспрессия которой происходит с отдельного промотора, или с усилением экспорта вирусных частиц из клетки. В целом, это можно рассматривать как нежелательный эффект молекулы NU7026. Связано ли усиление продукции HBsAg с особенностями именно NU7026 или это эффект, общий для всех ингибиторов пути NHEJ, при действии CRISPR/ Cas9, еще предстоит выяснить.
Работа выполнена при поддержке гранта РНФ № 16-15-10426.
Список литературы
GBD 2016 Causes of Death Collaborators. (2017) Global, regional, and national age-sex specific mortality for 264 causes of death, 1980–2016: a systematic analysis for the Global Burden of Disease Study 2016. Lancet. 390 (10100), 1151–1210.
Dahari H., Shudo E., Ribeiro R.M., Perelson A.S. (2009) Modeling complex decay profiles of hepatitis B virus during antiviral therapy. Hepatology. 49(1), 32–38.
Chen Z.-Y., Cheng A.-C., Wang M.-S., Xu D.-W., Zeng W., Li Z. (2007) Antiviral effects of PNA in duck hepatitis B virus infection model. Acta Pharmacol. Sin. 28(10), 1652–1658.
Tennant B. C., Gerin J. L. (2001) The woodchuck model of hepatitis B virus infection. ILAR J. 42(2), 89–102.
Schulze-Bergkamen H., Untergasser A., Dax A., Vogel H., Buchler P., Klar E., Lehnert T., Friess H., Buchler M.W., Kirschfink M., Stremmel W., Kram-mer P.H., Muller M., Protzer U. (2003) Primary human hepatocytes – a valuable tool for investigation of apoptosis and hepatitis B virus infection. J. Hepatol. 38(6), 736–744.
Konig A., Doring B., Mohr C., Geipel A., Geyer J., Glebe D. (2014) Kinetics of the bile acid transporter and hepatitis B virus receptor Na+/taurocholate cotransporting polypeptide (NTCP) in hepatocytes. J. Hepatol. 61(4), 867–875.
Ortega-Prieto A.M., Skelton J.K., Wai S.N., Large E., Lussignol M., Vizcay-Barrena G., Hughes D., Fleck R.A., Thursz M., Catanese M.T., Dorner M. (2018) 3D microfluidic liver cultures as a physiological preclinical tool for hepatitis B virus infection. Nat. Commun. 9(1), 682.
March S., Ramanan V., Trehan K., Ng S., Galstian A., Gural N., Scull M.A., Schlomai A., Mota M.M., Fleming H.E., Khetani S.R., Rice C.M., Bhatia S.N. (2015) Micropatterned coculture of primary human hepatocytes and supportive cells for the study of hepatotropic pathogens. Nat. Protoc. 10(12), 2027–2053.
Xia Y., Carpentier A., Cheng X., Block P.D., Zhao Y., Zhang Z., Protzer U., Liang T.J. (2017) Human stem cell-derived hepatocytes as a model for hepatitis B virus infection, spreading and virus-host interactions. J. Hepatol. 66(3), 494–503.
Gripon P., Rumin S., Urban S., Le Seyec J., Glaise D., Cannie I., Guyomard C., Lucas J., Trepo C., Guguen-Guillouzo C. (2002) Infection of a human hepatoma cell line by hepatitis B virus. Proc. Natl. Acad. Sci. USA. 99, 15655–15660.
Wang L. Y., Li Y.G., Chen K., Li L., Qu J.L., Qin D.D., Tang H. (2012) Stable expression and integrated hepatitis B virus genome in a human hepatoma cell line. Genet. Mol. Res. 11(2), 1442–1448.
Verrier E. R., Colpitts C. C., Schuster C., Zeisel M. B., Baumert T.F. (2016) Cell culture models for the investigation of hepatitis B and D virus infection. Viruses. 8(9), 261.
Lucifora J., Xia Y., Reisinger F., Zhang K., Stadler D., Cheng X., Sprinzl M.F., Koppensteiner H., Makowska Z., Volz T., Remouchamps C., Chou W.M., Thasler W.E., Huser N., Durantel D., Liang T.J., Munk C., Heim M.H., Browning J.L., Dejardin E., Dandri M., Schindler M., Heikenwalder M., Protzer U. (2014) Specific and nonhepatotoxic degradation of nuclear hepatitis B virus cccDNA. Science. 343(6176), 1221–1228.
Ramanan V., Shlomai A., Cox D., Schwartz R.E., Michailidis E., Bhatta A., Scott D.A., Zhang F., Rice C.M., Bhatia S.N. (2015) CRISPR/Cas9 cleavage of viral DNA efficiently suppresses hepatitis B virus. Sci. Rep. 5, 10833.
Bloom K., Ely A., Mussolino C., Cathomen T., Arbuthnot P. (2013) Inactivation of hepatitis B virus replication in cultured cells and in vivo with engineered transcription activator-like effector nucleases. Mol. Ther. 21(10), 1889–1897.
Weber N.D., Stone D., Sedlak R.H., De Silva Fee-lixge H.S., Roychoudhury P., Schiffer J.T., Aubert M., Jerome K.R. (2014) AAV-mediated delivery of zinc finger nucleases targeting hepatitis B virus inhibits active replication. PLoS One. 9(5), e97579.
Банников А.В., Лавров А.В. (2017) CRISPR/Cas9 – король геномного редактирования. Молекуляр. биология. 51(4), 582–594.
He Z., Proudfoot C., Whitelaw C.B., Lillico S.G. (2016) Comparison of CRISPR/Cas9 and TALENs on editing an integrated EGFP gene in the genome of HEK293FT cells. Springerplus. 5(1), 814.
Li H., Sheng C., Liu H., Liu G., Du X., Du J., Zhang L., Li P., Yang C., Qi L., Wang J., Yang X., Jia L., Xie J., Wang L., Hao R., Xu D., Tong Y., Zhou Y., Zhou J., Sun Y., Li Q., Qiu S., Sogn H. (2016) An effective molecular target site in hepatitis B virus S gene for Cas9 cleavage and mutational inactivation. Int. J. Biol. Sci. 12(9), 1104–1113.
Allweiss L., Dandri M. (2017) The role of cccDNA in HBV maintenance. Viruses. 9(6), pii: E156. doi 10.3390/v9060156
Jekimovs C., Bolderson E., Suraweera A., Adams M., O’Byrne K.J., Richard D.J. (2014) Chemotherapeutic compounds targeting the DNA double-strand break repair pathways: the good, the bad, and the promising. Front. Oncol. 4, 86.
Davidson D., Amrein L., Panasci L., Aloyz R. (2013) Small molecules, inhibitors of DNA-PK, targeting DNA repair, and beyond. Front. Pharmacol. 4, 5.
Robert F., Barbeau M., Éthier S., Dostie J., Pelletier J. (2015) Pharmacological inhibition of DNA-PK stimulates Cas9-mediated genome editing. Genome Med. 7(1), 93.
Budke B., Logan H.L., Kalin J.H., Zelivianskaia A.S., Cameron M.W., Miller L.L., Stark J.M., Kozikowski A.P., Bishop D.K., Connell P.P. (2012) RI-1: a chemical inhibitor of RAD51 that disrupts homologous recombination in human cells. Nucleic Acids Res. 40(15), 7347–7357.
Zhao F., Hou N.B., He X., Zheng Z.R., Ma Q.J., Li L., Zhang Y.H., Zhong H. (2008) Cellular DNA repair cofactors affecting hepatitis B virus infection and replication. World J. Gastroenterol. 14(32), 5059–5065.
Zhu W., Zie K., Xu Y., Wang L., Chen K., Zhang L., Fang J. (2016) CRISPR/Cas9 produces anti-hepatitis B virus effect in hepatoma cells and transgenic mouse. Virus Res. 217, 125–132.
Liu Y., Zhao M., Gong M., Xu Y., Xie C., Deng H., Li X., Wu H., Wang Z. (2018) Inhibition of hepatitis B virus replication via HBV DNA cleavage by Cas9 from Staphylococcus aureus. Antiviral Res.152, 58–67.
Seeger C., Sohn J.A. (2016) Complete spectrum of CRISPR/Cas9-induced mutations on HBV cccDNA. Mol. Ther. 24(7), 1258–1266.
Yu C., Liu Y., Ma T., Liu K., Xu S., Zhang Y., Liu H., La Russa M., Xie M., Ding S., Qi L.S. (2015) Small molecules enhance CRISPR genome editing in pluripotent stem cells. Cell Stem Cell. 16(2), 142–147.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


