Молекулярная биология, 2019, T. 53, № 3, стр. 421-429
Группа гиперметилированных генов микроРНК при раке молочной железы: диагностический потенциал
Е. А. Филиппова a, В. И. Логинов a, b, И. В. Пронина a, Д. С. Ходырев c, А. М. Бурдённый a, Т. П. Казубская d, Э. А. Брага a, b, *
a Научно-исследовательский институт общей патологии и патофизиологии
125315 Москва, Россия
b Медико-генетический научный центр
115478 Москва, Россия
c Федеральный научно-клинический центр специализированных видов медицинской помощи и медицинских технологий Федерального медико-биологического агентства России
115682 Москва, Россия
d Национальный медицинский исследовательский центр онкологии им. Н.Н. Блохина
Министерства здравоохранения Российской Федерации
115478 Москва, Россия
* E-mail: eleonora10_45@mail.ru
Поступила в редакцию 29.11.2018
После доработки 12.12.2018
Принята к публикации 12.12.2018
Аннотация
Ранее мы обнаружили аберрантное метилирование группы генов микроРНК в опухолях яичников и молочной железы, а также потенциальную возможность использования гиперметилированных генов микроРНК для диагностики рака яичников. В данной работе предпринята попытка оценить диагностический потенциал группы гиперметилируемых генов микроРНК (MIR-124-1, MIR-124-3, MIR-125B-1, MIR-127, MIR-132, MIR-193a и MIR-34b/c) на представительной выборке из 70 образцов рака молочной железы (РМЖ) и 17 образцов тканей молочной железы “доноров” (умерших от неонкологических заболеваний). Методом метилспецифичной ПЦР определены частоты метилирования семи указанных генов, составившие 26‒76% в образцах опухолей, 1‒27% ‒ в прилежащих к ним условно нормальных тканях, 0‒18% в тканях молочной железы “доноров”. На выборке из 38 образцов РМЖ методом количественной ОТ-ПЦР показано снижение уровней экспрессии этих микроРНК, выявлена корреляция снижения с гиперметилированием генов этих микроРНК. С помощью ROC-анализа определены 3- и 4-маркерные потенциальные системы для диагностики РМЖ с высокой чувствительностью (76‒93%) и специфичностью (88‒100%), основанные на статусе метилирования генов MIR-124-1, MIR-125B-1, MIR-127 и MIR-34b/c. Обнаружено, что ген MIR-127 гиперметилирован только в образцах опухолей от пациенток с метастазами, т.е. он представляет собой потенциальный высокоспецифичный маркер метастазирования РМЖ. Выявленные особенности метилирования генов микроРНК могут найти применение в разработке подходов к диагностике, прогнозированию и выбору тактики лечения РМЖ.
Метилирование регуляторных последовательностей ДНК и взаимодействие между микроРНК и мРНК генов-мишеней играют важную роль в динамической регуляции активности генов. Аберрации в этих механизмах могут привести к нарушению функционирования клеточных сигнальных путей, что и наблюдается в злокачественных опухолях [1‒3]. В последнее десятилетие продолжает возрастать интерес к поиску новых мишеней метилирования, новых регуляторных микроРНК и их генов-мишеней, вовлеченных в онкогенез. Благодаря таким исследованиям достигнут прогресс в персонализированной терапии многих злокачественных опухолей [4, 5].
Рак молочной железы (РМЖ) – самый распространенный вид рака у женщин, он составляет 1/10 от всех злокачественных опухолей и считается наиболее частой причиной смерти женщин от онкологических заболеваний [6]. Ежегодно в мире регистрируют 1.6 млн. случаев РМЖ, а его распространенность постоянно растет [6]. В России в 2017 г. выявлено более 70500 новых случаев РМЖ [7]. Кроме того, в последние годы наблюдается тенденция к омоложению РМЖ. Отсутствие эффективной диагностики на ранних стадиях заболевания и высокая частота летальных исходов указывают на необходимость поиска новых маркеров. В настоящее время широко обсуждаются перспективы применения микроРНК как маркеров, позволяющих диагностировать РМЖ на ранней стадии, прогнозировать его течение и оценивать индивидуальный ответ организма на терапию [8‒10].
Системную роль в регуляции работы генов-мишеней микроРНК играют факторы, изменяющие уровень экспрессии самих микроРНК, в частности, аберрантное метилирование регуляторных CpG-островков [11]. За последнее десятилетие установлена роль гиперметилирования широкого круга генов микроРНК в развитии опухолей разных локализаций [12]. Эпигеномные исследования показали, что доля подверженных метилированию генов микроРНК в несколько раз выше, чем белоккодирующих генов. Это указывает на важную роль метилирования в дерегуляции генов микроРНК в опухолях и делает гиперметилированные микроРНК перспективными диагностическими маркерами [13, 14].
Ранее мы обнаружили аберрантное метилирование обширной группы генов микроРНК, вовлеченное в патогенез рака яичников и РМЖ [15‒18], и показали потенциальную применимость гиперметилированных генов микроРНК для диагностики рака яичников [19]. В данной работе на представительной выборке из 70 образцов РМЖ и 17 образцов тканей молочной железы “доноров” (умерших от неонкологических заболеваний) изучена роль гиперметилирования группы генов микроРНК (MIR-124-1, MIR-124-3, MIR-125B-1, MIR-127, MIR-132, MIR-193a и MIR-34b/c) и оценен диагностический потенциал этих генов.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Образцы РМЖ собраны и клинически охарактеризованы в НИИ клинической онкологии НМИЦ онкологии им. Н.Н. Блохина. Использовали парные образцы опухолей и гистологически неизмененных (условно нормальных) тканей молочной железы, полученные от 70 больных РМЖ; клинико-гистологические данные пациенток приведены в табл. 1.
Таблица 1.
Клинические и гистологические характеристики 70 образцов рака молочной железы
| Характеристика опухоли | Гистологический тип рака | Число образцов |
|---|---|---|
| ИП-РМЖ | 49 | |
| ИД-РМЖ | 21 | |
| Клиническая стадия | I | 9 |
| II | 37 | |
| III | 23 | |
| IV | 1 | |
| Степень дифференцировки | Высокодифференцированный (hd) | 6 |
| Умереннодифференцированный (md) | 51 | |
| Низкодифференцированный (ld) | 13 | |
| Метастазирование | N0/M0 | 26 |
| N1–2 | 44 | |
| M1 | 1 | |
| Размер и степень инвазии опухоли | T1 | 13 |
| T2 | 43 | |
| T3 | 5 | |
| T4 | 9 |
Работа проведена с соблюдением принципов добровольности и конфиденциальности в соответствии с “Основами законодательства РФ об охране здоровья граждан”, получено разрешение этического комитета НМИЦ онкологии им. Н.Н. Блохина, а также информированное согласие больных. Исследование проведено в соответствии с принципами Хельсинкской декларации Всемирной медицинской ассоциации [20]. Проанализированы образцы злокачественных опухолей больных, которые до операции не получали специфического лечения (лучевую или химиотерапию). Все опухоли молочной железы были классифицированы в соответствии с TNM-классификацией Международного противоракового союза [21] и гистологически охарактеризованы на основании критериев классификации Всемирной Организации Здравоохранения [22]. Для отбора образцов с высоким содержанием опухолевых клеток (не менее 70‒80%) проведен дополнительный гистологический анализ микросрезов (3‒5 мкм), окрашенных эозином и гематоксилином.
Кроме того, проведен анализ 17 образцов тканей молочной железы женщин, умерших от неонкологических заболеваний и не имевших онкологических заболеваний в анамнезе (так называемые доноры).
Образцы тканей хранили при –70°С. Замороженную в жидком азоте ткань измельчали с помощью гомогенизатора-диспергатора SilentCrusher S (“Heidolph”, Германия).
Выделение ДНК и РНК, обратная транскрипция. Высокомолекулярную ДНК из тканей выделяли с использованием экстракции фенолом в соответствии со стандартными протоколами. Суммарную РНК выделяли по протоколу экстракции с использованием гуанидинтиоцианата, фенола и хлороформа, как описано ранее [23].
Концентрацию суммарной РНК определяли спектрофотометрически при 260 нм. Качество РНК также оценивали спектрофотометрически с использованием коэффициентов поглощения при 260 против 230 нм и 260 против 280 нм. Сохранность РНК определяли по соотношению интенсивностей полос 28S рРНК и 18S рРНК при электрофорезе в 1%-ном денатурирующем агарозном геле. Перед использованием все образцы РНК обрабатывали ДНКазой, свободной от РНКазы. Препараты кДНК синтезировали из 1 мкг суммарной РНК с использованием обратной транскриптазы M-MuLV и случайных (random) наномеров в соответствии с протоколом производителя (“Thermo Fisher Scientific”, США).
Количественную ПЦР (для оценки изменения содержания семи микроРНК в образцах РМЖ) проводили с использованием кДНК, полученной, как описано ранее [16]. Использовали наборы TaqMan MicroRNA Assays (“Applied Biosystems”, США): miR-124-3p (Assay ID: 001182), miR-125b-5p (Assay ID: 000449), miR-127-5p (Assay ID: 002229), miR-132-3p (Assay ID: 000457), miR-193a-5p (Assay ID: 002281), miR-34b-3p (Assay ID: 002102) и miR-34c-3p (Assay ID: 241009). Для нормализации применяли RNU48 (Assay ID: 001006) и RNU6 (Assay ID: 001093). Все реакции повторяли трижды. В качестве отрицательного контроля использовали пробы без кДНК. Данные анализировали с использованием относительной количественной оценки по ΔΔCt-методу. Изменения уровня микроРНК менее чем в 2 раза (|ΔΔCt| ≤ 2) рассматривали как отсутствие изменений [16].
Бисульфитную конверсию ДНК и метилспецифичную ПЦР (МС-ПЦР) проводили, как описано ранее [15, 16]. ПЦР проводили на амплификаторe DNA Engine Dyad Cycler Т-100 (“Bio-Rad”, США) с использованием олигонуклеотидов и описанных условий амплификации [15‒18]. Анализировали от трех до шести CpG-динуклеотидов в каждом гене. Ложноположительные результаты из-за неполной бисульфитной конверсии ДНК исключали на стадии подбора праймеров по отсутствию продукта МС-ПЦР на необработанной бисульфитом ДНК. Препарат метилированной ДНК человека (#SD1131, “Thermo Scientific”) использовали как контроль для метилированного аллеля, а препарат ДНК человека (#G1471, “Promega”, США) – как контроль для неметилированного аллеля. Продукты ПЦР разных генов разделяли одновременно с использованием 2%-ного агарозного геля.
Статистический анализ проводили с применением точного критерия Фишера в программе BioStat 6.1. Изменения считали значимыми при p ≤ 0.05. Изменения уровня экспрессии и метилирования генов с определением коэффициента корреляции Спирмена (rs) сопоставляли с помощью корреляционного анализа. Оптимальные системы маркеров подбирали по результатам ROC (Receiver Operator Characteristic)-анализа [24]. Учет множественного сравнения проводили с помощью поправки Бенджамини‒Хохберга; результат считали значимым при FDR (false discovery rate) ≤ 0.05.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Гиперметилированные микроРНК как потенциальные специфичные маркеры рака маркерной железы
На представительной выборке из 70 парных образцов (опухоль/условная норма) РМЖ методом МС-ПЦР проведен анализ метилирования семи генов микроРНК: MIR-124-1, MIR-124-3, MIR-125B-1, MIR-127, MIR-132, MIR-193a и MIR-34b/c. В табл. 2 суммированы результаты определения частоты метилирования этих генов в образцах тканей молочной железы, полученных от 70 больных РМЖ и 17 “доноров” (умерших от неонкологических заболеваний).
Таблица 2.
Частота метилирования семи генов микроРНК при раке молочной железы
| Ген микроРНК | Опухоль | Норма | p | Доноры |
|---|---|---|---|---|
| MIR-124-1 | 76% (53/70) | 27% (19/70) | 1 × 10–8 | 12%, 2/17 |
| MIR-124-3 | 39% (27/70) | 3% (2/70) | 1 × 10–7 | 12%, 2/17 |
| MIR-125B-1 | 49% (34/70) | 6% (4/70) | 1 × 10–8 | 0%, 0/17 |
| MIR-127 | 30% (21/70) | 1% (1/70) | 2 × 10–6 | 0%, 0/17 |
| MIR-132 | 26% (18/70) | 9% (6/70) | 0.01 | 0%, 0/17 |
| MIR-193a | 59% (41/70) | 13% (9/70) | 2 × 10–8 | 18%, 3/17 |
| MIR-34b/c | 39% (27/70) | 9% (6/70) | 4 × 10–5 | 0%, 0/17 |
Из табл. 2 видно, что в опухолях наиболее часто метилированы два гена (MIR-124-1 и MIR-193 ‒ 76 и 59% соответственно), но их метилирование часто выявляют в условно нормальной ткани (27 и 13%) и в тканях молочной железы “доноров” (12 и 18%). Отмечено, что четыре гена (MIR-125B-1, MIR-127, MIR-132, MIR-34b/c) метилированы в опухолях с частотой 26‒49%, 1‒9% в условной норме, тогда как в тканях молочной железы 17 “доноров” метилирование этих генов не обнаружено. Эти четыре гена можно предложить в качестве достаточно специфичных диагностических маркеров РМЖ.
Гиперметилирование вносит вклад в подавление экспрессии группы генов микроРНК при раке молочной железы
Чтобы проверить функциональную роль метилирования, определили изменение уровня экспрессии генов микроРНК, подверженных гиперметилированию при РМЖ. В табл. 3 приведены частоты снижения и повышения содержания зрелых микроРНК (продуктов данных генов) на подвыборке из 38 образцов РМЖ, входящих в общую выборку из 70 образцов. Из табл. 3 видно, что в образцах РМЖ преобладает снижение содержания всех зрелых микроРНК, транскрибированных с этих гиперметилированных генов. Так, miR-124-3p, -125b-5p, -127-5p, -132-3p, -193a-5p, -34b-3p и -34с-3p преимущественно ингибированы, что свойственно опухолевым супрессорам, и это ингибирование ассоциировано с гиперметилированием кодирующих их генов, как следует из результатов корреляционного анализа между изменениями статуса метилирования и экспрессии, выполненного на общей подвыборке из 38 образцов (табл. 4).
Таблица 3.
Частота изменений уровня микроРНК при раке молочной железы
| микроРНК | Снижение | Повышение | Нет изменений |
|---|---|---|---|
| miR-124-3p | 68% (26/38) | 13% (5/38) | 18% (7/38) |
| miR-125b-5p | 76% (29/38) | 8% (3/38) | 16% (6/38) |
| miR-127-5p | 39% (15/38) | 8% (3/38) | 53% (20/38) |
| miR-132-3p | 50% (19/38) | 5% (2/38) | 45% (17/38)) |
| miR-193a-5p | 79% (30/38) | 3% (1/38) | 18% (7/38) |
| miR-34b-3p | 47% (18/38) | 11% (4/38) | 42% (16/38) |
| miR-34с-3p | 37% (14/38) | 16% (6/38) | 47% (18/38) |
Таблица 4.
Корреляция между изменением статуса метилирования гена микроРНК и содержанием микроРНК при анализе 38 парных образцов рака молочной железы
| Ген микроРНК | микроРНК | rs | p |
|---|---|---|---|
| MIR-124-1 | miR-124-3p | 0.61 | 4 ×10–5 |
| MIR-124-3 | miR-124-3p | 0.24 | 0.15 |
| MIR-125B-1 | miR-125b-5p | 0.77 | 2 ×10–8 |
| MIR-127 | miR-127-5p | 0.50 | 2 ×10–3 |
| MIR-132 | miR-132-3p | 0.60 | 7 ×10–5 |
| MIR-193a | miR-193a-5p | 0.64 | 2 ×10–5 |
| MIR-34b/c | miR-34b-3p | 0.51 | 10–5 |
| MIR-34b/c | miR-34c-3p | 0.38 | 0.02 |
Выявлена высокая корреляция между изменениями метилирования и экспрессии шести из семи генов; коэффициент корреляции Спирмена (rs) находился в интервале между 0.38 и 0.61, p ≤ 0.02. Единственный ген, MIR-124-3, показал слабую корреляцию между изменением статуса метилирования и экспрессией miR-124-3p. Можно предположить, что синтез miR-124-3p обеспечивается преимущественно геном MIR-124-1, но не MIR-124-3. Таким образом, можно заключить, что гиперметилирование регуляторных областей генов MIR-124-1, -125b-1, -127, -132, -193a и -34b/c вносит значимый вклад в подавление экспрессии этих генов и синтез соответствующих зрелых микроРНК при РМЖ. Эти данные подтверждают участие аберрантного метилирования в патогенезе РМЖ.
Диагностический потенциал группы генов микроРНК при раке молочной железы
На основании данных о статусе метилирования семи генов микроРНК в образцах РМЖ (70) и ткани молочной железы “доноров” (17) методом ROC-анализа определены две оптимальные потенциальные диагностические системы маркеров для выявления РМЖ (табл. 5).
Таблица 5.
Потенциальные диагностические системы генов-маркеров микроРНК
| Набор генов микроРНК |
AUC (95% CI) |
Criterion | Sn, % (95% CI) |
Sp, % (95% CI) |
p |
|---|---|---|---|---|---|
| MIR-125B-1 MIR-127 MIR-34b/c |
0.88 (0.79‒0.94) |
>0 | 76 (64‒85) |
100 (80‒100) |
<10–4 |
| MIR-124-1 MIR-125B-1 MIR-127 MIR-34b/c |
0.94 (0.87‒0.98) |
>0 | 93 (84‒98) |
88 (64‒98) |
<10–4 |
Первая система составлена из трех генов, метилирование которых не выявлено ни в одном образце ткани молочной железы “доноров” (табл. 2). Cпецифичность этой системы равна 100% при чувствительности только 76% и величине AUC (Area Under ROC-Curve), интегрально характеризующей надежность системы, 0.88. Добавление в систему маркера MIR-124-1 с высокой частотой метилированного в образцах опухолей (76%, табл. 2) и низкой частотой метилирования в тканях “доноров” (12%, 2/17) позволило нам составить наиболее оптимальную систему. Эта система из четырех генов характеризуется высокой величиной AUC (0.94), чувствительностью 93% при специфичности 88%.
Таким образом, систему из четырех маркеров можно предложить как потенциальную диагностическую систему, причем обнаружения метилирования хотя бы одного из генов этой системы достаточно для отнесения образца к РМЖ.
Ген MIR-127 гиперметилирован только в образцах опухолей от пациенток с метастазами и представляет собой высокоспецифичный маркер метастазирования рака молочной железы
Изучена связь между статусом метилирования каждого из семи генов (MIR-124-1, MIR-124-3, MIR-125B-1, MIR-127, MIR-132, MIR-193a, MIR-34b/c) и клинико-гистологическими характеристиками 70 образцов РМЖ. При этом установлена значимая ассоциация метилирования гена MIR-127 с более поздними клиническими стадиями (p < 10–4), метастазированием (главным образом в регионарные лимфатические узлы) (p < 10–4) и размером опухоли (p < 10–4, рис. 1). Между метилированием этого гена и потерей дифференцировки опухоли наблюдалась менее значимая связь (p ≤ 0.05).
Рис. 1.
Зависимость между частотой метилирования гена MIR-127 и прогрессией рака молочной железы. Клиническая стадия: I/II против III/IV. Метастазирование: N0/M0 – группа без метастазов, N1–2/M1 – группа с метастазами. Размер и степень инвазии опухоли: T1, T2, T3, T4. Дифференцировка: hd – высокодифференцированная, md – умереннодифференцированная; ld – низкодифференцированная; С учетом поправки Бенджамини‒Хохберга на множественное сравнение значения p ≤ 10–4 статистически значимы при FDR = 0.01.
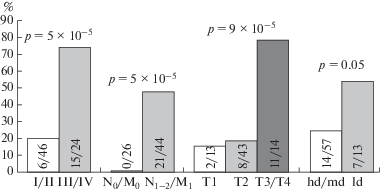
Обнаружено, что ген MIR-127 метилирован исключительно в группе образцов опухолей от пациенток, у которых выявлены метастазы (в половине случаев, 21/44, рис. 1). При этом метилирование MIR-127 обнаружено в единственном условно нормальном образце (1/70, табл. 2). Причем, как выяснилось, этот образец получен от пациентки с клинической стадией III заболевания, низкой степенью дифференцировки опухоли и метастазами, обнаруженными в двух лимфатических узлах (T2N2M0). Опухоль с такой степенью прогрессии может проявлять значительную инвазию в близлежащие ткани помимо регионарных лимфатических узлов, и парная прилежащая ткань может включать опухолевые клетки, которые и показали метилирование этого гена при анализе условно нормальной ткани.
Таким образом, из семи генов микроРНК, метилирование которых изучено нами на выборке из 70 парных образцов, с прогрессией РМЖ связан только один ‒ ген MIR-127. Причем этот ген гиперметилирован только в образцах опухолей от пациенток с метастазами. Таким образом, ген MIR-127 представляет собой потенциальный высокоспецифичный маркер, который может использоваться для выявления или прогнозирования метастазирования опухоли у пациентки, у которой обнаружено его метилирование.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В нашей работе показана значимая роль гиперметилирования в подавлении экспрессии семи генов микроРНК (MIR-124-1, MIR-124-3, MIR-125B-1, MIR-127, MIR-132, MIR-193a, MIR-34b/c) и снижении содержания зрелых микроРНК при РМЖ. Установлена корреляция между метилированием шести генов и синтезом соответствующих зрелых микроРНК. Эти данные подтверждают функциональную роль аберрантного метилирования в патогенезе РМЖ.
Гиперметилирование и снижение экспрессии указанных генов согласуется с супрессорной ролью кодируемых ими микроРНК (miR-124, -125b, -127, -132, -193a-5p, -34b/c) при РМЖ, что установлено, главным образом, с применением клеточных культур [25‒30]. Сниженный уровень экспрессии этих микроРНК рассматривают как маркер РМЖ. Кроме того, выявлена связь miR-124, -125b, -127, -132, и -193a-5p с прогрессией РМЖ, инвазией и метастазированием [26, 28, 30‒32].
В качестве маркеров РМЖ нами рассмотрено гиперметилирование шести генов микроРНК, функциональная роль которого подтверждена корреляцией между изменениями их метилирования и экспрессии.
Составлена потенциальная система маркеров для диагностики РМЖ, основанная на анализе метилирования генов микроРНК, а именно MIR-124-1, MIR-125B-1, MIR-127 и MIR-34b/c. Система характеризуется высокой чувствительностью, 93%, и специфичностью, 88%, (AUC = = 0.94). Эти параметры рассчитаны на основе данных для представительной выборки из 70 образцов РМЖ относительно абсолютной нормы (17 “доноров” без онкологических заболеваний в анамнезе).
В качестве диагностических систем исследуют профили экспрессии микроРНК, например, при раке предстательной железы, мочевого пузыря, легкого, колоректального рака и рака гортани [33‒38]. Предлагаемые панели маркеров характеризуются высокими показателями чувствительности, специфичности и величины AUC (>0.9). Не менее активно разрабатываются маркеры на основе статуса метилирования генов микроРНК. Такие панели предложены, например, для колоректального рака, рака мочевого пузыря и предстательной железы [39‒41]. В результате системного анализа метилирования генов супрессорных микроРНК нашей группой разработаны системы маркеров для диагностики рака легкого, почки и яичников [19, 42, 43], а в данной работе – для диагностики РМЖ. Это первая известная нам работа, в которой предложен набор диагностических маркеров РМЖ, отобранных на основании анализа гиперметилированных генов микроРНК.
Интересно, что нами обнаружен ген MIR-127, часто гиперметилированный в образцах опухолей, полученных от пациенток, у которых выявлены метастазы (21 из 44), и неметилированный ни в одном образце опухолей от пациенток, у которых метастазы не выявлены (0 из 26). Таким образом, ген MIR-127 представляет достаточно уникальный потенциальный высокоспецифичный прогностический маркер метастазирования РМЖ.
Связь сниженной экспрессии miR-127 с метастазированием РМЖ отмечали ранее [32, 44]. Сведения о мишенях miR-127 немногочисленны: при РМЖ к мишеням miR-127 отнесен протоонкоген BCL6 [32, 44]. Ядерный фактор-κB (NF-κB) рассматривается как предполагаемая мишень miR-127 в гепатоклеточной карциноме [45], что подтверждает супрессорные свойства этой микроРНК, но никак не объясняет ее антиметастатическую активность. Удивительно, что при раке легкого miR-127 не подавляет, а напротив, индуцирует эпителиально-мезенхимальный переход [46]. Таким образом, молекулярный механизм антиметастатической активности miR-127 и активирующий эффект метилирования гена MIR-127 на диссеминацию РМЖ требуют дальнейшего изучения. В то же время, содержание miR-127 в крови коррелирует с присутствием циркулирующих в крови опухолевых клеток, что делает miR-127 потенциальным неинвазивным клинически значимым маркером [47].
Таким образом, выявленные особенности метилирования семи генов микроРНК, уникальный прогностический потенциал гиперметилирования гена MIR-127 и предложенная система диагностических маркеров РМЖ, основанная на статусе метилирования генов микроРНК, могут найти клиническое применение при разработке современных подходов к диагностике, прогнозированию и выбору тактики лечения РМЖ.
Работа выполнена за счет средств Российского научного фонда (№ 14-15-00654).
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Llinàs-Arias P., Esteller M. (2017) Epigenetic inactivation of tumour suppressor coding and non-coding genes in human cancer: an update. Open Biol. 7(9). pii: 170152. doi 170152https://doi.org/10.1098/rsob
Musavi Shenas M.H., Eghbal-Fard S., Mehrisofiani V., Abd Yazdani N., Rahbar Farzam O., Marofi F., Yousefi M. (2018) MicroRNAs and signaling networks involved in epithelial-mesenchymal transition. J. Cell. Physiol. Nov. 11. doi https://doi.org/10.1002/jcp.27489
Moutinho C., Esteller M. (2017) MicroRNAs and epigenetics. Adv. Cancer Res. 135, 189‒220. doi https://doi.org/10.1016/ bs.acr.2017.06.003
Stefansson O.A., Esteller M. (2013) Epigenetic modifications in breast cancer and their role in personalized medicine. Am. J. Pathol. 183, 1052‒1063. doi https://doi.org/10.1016/ j.ajpath.2013.04.033
Bertoli G., Cava C., Castiglioni I. (2015) MicroRNA: new biomarkers for diagnosis, prognosis, therapy prediction and therapeutic tools for breast cancer. Theranostics. 5, 1122‒1143. doi https://doi.org/10.7150/thno.11543
Dey S. (2014) Preventing breast cancer in LMICs via screening and/or early detection: the real and the surreal. World J. Clin. Oncol. 5, 509–519. doi https://doi.org/10.5306/ wjco.v5.i3.509
Злокачественные новообразования в России в 2017 году (заболеваемость и смертность). (2018) Ред. Каприн А.Д., Старинский В.В., Петрова Г.В. М.: “МНИОИ им. П.А. Герцена” Минздрав. России, 250 с.
Takahashi R., Miyazaki H., Ochiya T. (2015) The roles of microRNAs in breast cancer. Cancers (Basel). 7, 598‒616. doi https://doi.org/10.3390/cancers7020598
Khordadmehr M., Shahbazi R., Ezzati H., Jigari-Asl F., Sadreddini S., Baradaran B. (2018) Key microRNAs in the biology of breast cancer; emerging evidence in the last decade. J. Cell. Physiol. doi https://doi.org/10.1002/jcp.27716
Campos-Parra A.D., Mitznahuatl G.C., Pedroza-Torres A., Romo R.V., Reyes F.IP., López-Urrutia E., Pérez-Plasencia C. (2017) Micro-RNAs as potential predictors of response to breast cancer systemic therapy: future clinical implications. Int. J. Mol. Sci. 18(6). pii: E1182. doi https://doi.org/10.3390/ijms18061182
Vrba L., Muñoz-Rodríguez J.L., Stampfer M.R., Futscher B.W. (2013) miRNA gene promoters are frequent targets of aberrant DNA methylation in human breast cancer. PLoS One. 8(1), e54398. doi https://doi.org/10.1371/ journal.pone.0054398
Логинов В.И., Рыков С.В., Фридман М.В., Брага Э.А. (2015) Метилирование генов микроРНК и онкогенез. Биохимия. 80, 184‒203.
Kunej T., Godnic I., Ferdin J., Horvat S., Dovc P., Calin G.A. (2011) Epigenetic regulation of microRNAs in cancer: an integrated review of literature. Mutat. Res. 717, 77‒84. doi https://doi.org/10.1016/j.mrfmmm.2011.03.008
Piletič K., Kunej T. (2016) MicroRNA epigenetic signatures in human disease. Arch. Toxicol. 90, 2405‒2419. doi https://doi.org/10.1007/s00204-016-1815-7
Логинов В.И., Бурденный А.М., Пронина И.В., Хоконова В.В., Куревлёв С.В., Казубская Т.П., Кушлинский Н.Е., Брага Э.А. (2016) Идентификация новых генов микроРНК, гиперметилированных при раке молочной железы. Молекуляр. биология. 50, 797‒802.
Pronina I.V., Loginov V.I., Burdennyy A.M., Fridman M.V., Senchenko V.N., Kazubskaya T.P., Kushlinskii N.E., Dmitriev A.A., Braga E.A. (2017) DNA methylation contributes to deregulation of 12 cancer-associated microRNAs and breast cancer progression. Gene. 604, 1‒8. doi https://doi.org/10.1016/j.gene.2016.12.018
Логинов В.И., Бурдённый А.М., Филиппова Е.А., Пронина И.В., Казубская Т.П., Кушлинский Д.Н., Ермилова В.Д., Рыков С.В., Ходырев Д.С., Брага Э.А. (2018) Гиперметилирование генов микроРНК miR-107, miR-130b, miR-203, miR-1258 ассоциировано с развитием и метастазированием рака яичников. Молекуляр. биология. 52, 801–809.
Loginov V.I., Pronina I.V., Burdennyy A.M., Filippova E.A., Kazubskaya T.P., Kushlinsky D.N., Utkin D.O., Khodyrev D.S., Kushlinskii N.E., Dmitriev A.A., Braga E.A. (2018) Novel miRNA genes deregulated by aberrant methylation in ovarian carcinoma are involved in metastasis. Gene. 662, 28‒36. doi https://doi.org/10.1016/ j.gene.2018.04.005
Брага Э.А., Логинов В.И., Филиппова Е.А., Бурдённый А.М., Пронина И.В., Казубская Т.П., Уткин Д.О., Ходырев Д.С., Кушлинский Д.Н., Адамян Л.В., Кушлинский Н.Е. (2018) Диагностическое значение группы генов микроРНК, гипер-метилированных в карциноме яичников. Бюлл. эксп. биологии и медицины. 166, 213‒217.
World Medical Association. (2013) Declaration of Helsinki: Ethical Principles for Medical Research Involving Human Subjects. J. Am. Med. Association. 310, 2191–2194.
Sobin L.H., Gospodarowicz M.K., Wittekind Ch. (2010) International Union against Cancer. TNM Classification of Malignant Tumours. 7th ed. 2009. Chichester, West Sussex, UK; Hoboken, NJ: Wiley-Blackwell, 2010. pp. 332. https://www.ncbi.nlm.nih.gov/nlmcatalog/101511218.
WHO Classification of Tumours of Female Reproductive Organs. 4th edn. (2014) Eds Kurman R.J., Carcangiu M.L., Herrington C.S., Young R.H. Lyon: IARC Press.
Пронина И.В., Логинов В.И., Ходырев Д.С., Казубская Т.П., Брага Э.А. (2012) Уровень экспрессии гена RASSF1А в первичных эпителиальных опухолях разной локализации. Молекуляр. биология. 46(2), 260‒268. doi https://doi.org/10.1134/S0026893312010189
Göksülük D., Korkmaz S., Zararsiz G., Karaagaoglu A.E. (2016) EasyROC: An interactive Web-tool for ROC curve analysis using R language environment. The R. J. 8, 213‒230.
Wang Y., Chen L., Wu Z., Wang M., Jin F., Wang N., Hu X., Liu Z., Zhang C.Y., Zen K., Chen J., Liang H., Zhang Y., Chen X. (2016) miR-124-3p functions as a tumor suppressor in breast cancer by targeting CBL. BMC Cancer. 16(1), 826. PMID: https://doi.org/27842510
Li Y., Wang Y., Fan H., Zhang Z., Li N. (2018) miR-125b-5p inhibits breast cancer cell proliferation, migration and invasion by targeting KIAA1522. Biochem. Biophys. Res. Commun. 504, 277‒282. doi: https://doi.org/10.1016/ j.bbrc.2018.08.172
Chen J., Wang M., Guo M., Xie Y., Cong Y.S. (2013) miR-127 regulates cell proliferation and senescence by targeting BCL6. PLoS One. 8(11), e80266. doi https://doi.org/10.1371/ journal.pone.0080266
Zhang Z.G., Chen W.X., Wu Y.H., Liang H.F., Zhang B.X. (2014) MiR-132 prohibits proliferation, invasion, migration, and metastasis in breast cancer by targeting HN1. Biochem. Biophys. Res. Commun. 454, 109‒114. doi https://doi.org/10.1016/j.bbrc.2014.10.049
Liu X., Feng J., Tang L., Liao L., Xu Q., Zhu S. (2015) The regulation and function of miR-21-FOXO3a-miR-34b/c signaling in breast cancer. Int. J. Mol. Sci. 16, 3148‒3162. doi https://doi.org/10.3390/ijms16023148
Xie F., Hosany S., Zhong S., Jiang Y., Zhang F., Lin L., Wang X., Gao S., Hu X. (2017) MicroRNA-193a inhibits breast cancer proliferation and metastasis by downregulating WT1. PLoS One. 12(10), e0185565. doi https://doi.org/10.1371/journal.pone.0185565
Zhang L., Chen X., Liu B., Han J. (2018) MicroRNA-124-3p directly targets PDCD6 to inhibit metastasis in breast cancer. Oncol. Lett. 15, 984‒990. doi https://doi.org/10.3892/ ol.2017.7358
Wang S., Li H., Wang J., Wang D., Yao A., Li Q. (2014) Prognostic and biological significance of microRNA-127 expression in human breast cancer. Dis. Markers. 2014, 401986. doi https://doi.org/10.1155/2014/401986
Huang Z., Zhu D., Wu L., He M., Zhou X., Zhang L., Zhang H., Wang W., Zhu J., Cheng W., Chen Y., Fan Y., Qi L., Yin Y., Zhu W., Shu Y., Liu P. (2017) Six serum-based miRNAs as potential diagnostic biomarkers for gastric cancer. Cancer Epidemiol. Biomarkers Prev. 26, 188‒196. doi https://doi.org/10.1158/1055-9965.EPI-16-0607
Urquidi V., Netherton M., Gomes-Giacoia E., Serie D.J., Eckel-Passow J., Rosser C.J., Goodison S. (2016) A microRNA biomarker panel for the non-invasive detection of bladder cancer. Oncotarget. 7, 86290‒86299. doi https://doi.org/10.18632/oncotarget.13382
Daniel R., Wu Q., Williams V., Clark G., Guruli G., Zehner Z. (2017) A panel of microRNAs as diagnostic biomarkers for the identification of prostate cancer. Int. J. Mol. Sci. 18(6). pii: E1281. doi https://doi.org/10.3390/ijms18061281
Zhu M., Huang Z., Zhu D., Zhou X., Shan X., Qi L.W., Wu L., Cheng W., Zhu J., Zhang L., Zhang H., Chen Y., Zhu W., Wang T., Liu P. (2017) A panel of microRNA signature in serum for colorectal cancer diagnosis. Oncotarget. 8, 17081‒17091. doi https://doi.org/10.18632/oncotarget.15059
Zhang H., Zhu M., Shan X., Zhou X., Wang T., Zhang J., Tao J., Cheng W., Chen G., Li J., Liu P., Wang Q., Zhu W. (2018) A panel of seven-miRNA signature in plasma as potential biomarker for colorectal cancer diagnosis. Gene. Nov 17. pii S0378-1119(18)31197-1. doi https://doi.org/10.1016/j.gene.2018.11.055
Chang Y.A., Weng S.L., Yang S.F., Chou C.H., Huang W.C., Tu S.J., Chang T.H., Huang C.N., Jong Y.J., Huang H.D. (2018) a three-microrna signature as a potential biomarker for the early detection of oral cancer. Int. J. Mol. Sci. 19(3). pii E758. doi https://doi.org/10.3390/ ijms19030758
Shimizu T., Suzuki H., Nojima M., Kitamura H., Yamamoto E., Maruyama R., Ashida M., Hatahira T., Kai M., Masumori N., Tokino T., Imai K., Tsukamoto T., Toyota M. (2013) Methylation of a panel of microRNA genes is a novel biomarker for detection of bladder cancer. Eur. Urol. 63, 1091‒100. doi https://doi.org/10.1016/ j.eururo.2012.11.030
Toiyama Y., Okugawa Y., Tanaka K., Araki T., Uchida K., Hishida A., Uchino M., Ikeuchi H., Hirota S., Kusunoki M., Boland C.R., Goel A. (2017) A panel of methylated microRNA biomarkers for identifying high-risk patients with ulcerative colitis-associated colorectal cancer. Gastroenterology. 153(6), 1634‒1646. e8. doi https://doi.org/10.1053/j.gastro.2017.08.037
Torres-Ferreira J., Ramalho-Carvalho J., Gomez A., Menezes F.D., Freitas R., Oliveira J., Antunes L., Bento M.J., Esteller M., Henrique R., Jerónimo C. (2017) MiR-193b promoter methylation accurately detects prostate cancer in urine sediments and miR-34b/c or miR-129-2 promoter methylation define subsets of clinically aggressive tumors. Mol. Cancer. 16, 26.
Рыков С.В., Ходырев Д.С., Пронина И.В., Казубская Т.П., Логинов В.И., Брага Э.А. (2013) Новые гены микроРНК, подверженные метилированию в опухолях легкого. Генетика. 49(7), 896‒901. doi https://doi.org/10.1134/S1022795413070119
Loginov V.I., Beresneva E.V., Kazubskaya T.P., Braga E.A., Karpukhin A.V. (2017). Methylation of 10 miRNA genes in clear cell renal cell carcinoma and their diagnostic value. Cancer Urology. 13, 27‒33. doi https://doi.org/10.17650/1726-9776-2017-13-3-27-33
Zhao X., Duan Z., Liu X., Wang B., Wang X., He J., Yao Z., Yang J. (2013). MicroRNA-127 is downregulated by Tudor-SN protein and contributes to metastasis and proliferation in breast cancer cell line MDA-MB-231. Anat. Rec. (Hoboken). 296, 1842‒1849. doi https://doi.org/10.1002/ ar.22823
Huan L., Bao C., Chen D., Li Y., Lian J., Ding J., Huang S., Liang L., He X. (2016) MicroRNA-127-5p targets the biliverdin reductase B/nuclear factor-κB pathway to suppress cell growth in hepatocellular carcinoma cells. Cancer Sci. 107, 258‒266. doi https://doi.org/10.1111/ cas.12869
Shi L., Wang Y., Lu Z., Zhang H., Zhuang N., Wang B., Song Z., Chen G., Huang C., Xu D., Zhang Y., Zhang W., Gao Y. (2017) miR-127 promotes EMT and stem-like traits in lung cancer through a feed-forward regulatory loop. Oncogene. 36, 1631‒1643. doi https://doi.org/10.1038/ onc.2016.332
Alunni-Fabbroni M., Majunke L., Trapp E.K., Tzschaschel M., Mahner S., Fasching P.A., Fehm T., Schneeweiss A., Beck T., Lorenz R., Friedl T.W.P., Janni W., Rack B., SUCCESS Study Group. (2018) Whole blood microRNAs as potential biomarkers in post-operative early breast cancer patients. BMC Cancer. 18(1), 141. doi https://doi.org/10.1186/s12885-018-4020-7
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


