Молекулярная биология, 2019, T. 53, № 3, стр. 513-523
Ферментативное получение модифицированных ДНК: изучение кинетики ПЦР в режиме реального времени
С. А. Лапа a, *, А. С. Павлов a, b, В. Е. Кузнецова a, В. Е. Шершов a, М. А. Спицын a, b, Т. О. Гусейнов a, b, С. П. Радько b, c, А. С. Заседателев a, А. В. Лисица c, А. В. Чудинов a, b
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b ООО “ИБМХ-ЭкоБиоФарм”
119121 Москва, Россия
c Институт биомедицинской химии им. В.Н. Ореховича, Российской академии наук
119121 Москва, Россия
* E-mail: lapa@biochip.ru
Поступила в редакцию 03.12.2018
После доработки 24.12.2018
Принята к публикации 24.12.2018
Аннотация
Изучено влияние модифицированных дезоксиуридин-5'-трифосфатов (mod-dUTPs) с различными заместителями, связанными по С5-положению пиримидинового цикла транс-алкеновой связью, на кинетику ПЦР с ДНК-полимеразами Taq и Vent (exo-). Заместители в mod-dUTPs включают карбоксамидную группу и группы, входящие в состав боковых цепей аланина, валина, лейцина, фенилаланина, триптофана и тирозина. Измерен выход целевого продукта для каждого из исследованных модифицированных производных dUTP при полном замещении природного dTTP. В качестве матриц для амплификации использовали фрагмент бактериальной ДНК с фиксированной нуклеотидной последовательностью и синтетическую комбинаторную ДНК-библиотеку с вырожденной последовательностью нуклеотидов. Исследована корреляция между эффективностью амплификации и выходом целевого продукта для каждой комбинации mod-dUTP–матрица–полимераза. Показана зависимость кинетики накопления продукта ПЦР как от использованной матрицы, так и от структуры модифицированного субстрата. Каталитическая активность Taq-полимеразы выше для mod-dUTPs с короткими алифатическими заместителями и ниже для производных с длинными алифатическими, фенильными и индольными заместителями. Vent (exo-)-полимераза менее чувствительна к химическому строению mod-dUTPs. Найденные динамические характеристики накопления модифицированных ДНК могут быть использованы для оптимизации температурно-временных профилей ПЦР индивидуально для каждого производного dUTP. Все исследованные производные могут с разной степенью эффективности использоваться в сочетании с Vent (exo-)-полимеразой для получения модифицированных ДНК в методе селекции модифицированных аптамеров (mod-SELEX).
ВВЕДЕНИЕ
Модифицированные ДНК играют важную роль при конструировании биосенсоров, ДНК-зондов и аптамеров для диагностики и терапии [1]. Ферментативный способ введения в ДНК модифицированных нуклеотидов относится к наиболее универсальным, но его использование ограничено совместимостью субстратной специфичности применяемых полимераз со структурой модифицированных dNTPs (mod-dNTPs) [2].
Общепринятым и наиболее распространенным методом ферментативного получения модифицированных аптамеров на сегодняшний день остается реакция удлинения праймера (primer extension, PEX) [3, 4]. Однако амплификация целевого продукта в случае PEX происходит в арифметической прогрессии, значительно уступая ПЦР, где продукт накапливается в геометрической прогрессии. Именно поэтому при получении модифицированных аптамеров приходится использовать составные циклы их отбора: ПЦР с немодифицированными dNTPs, за которым следует PEX с одним из модифицированных dNTP, и лишь затем инкубация ДНК-библиотеки с молекулярной мишенью – и так на протяжении всех раундов SELEX.
Амплификация модифицированных ДНК-библиотек посредством ПЦР без применения стадии PEX остается весьма привлекательной альтернативой. Первые успешные попытки получения модифицированных аптамеров с применением ПЦР были осуществлены еще в 90-х годах прошлого века. Описан аптамер к человеческому тромбину, полученный методом ПЦР с использованием 5-(1-пентинил)-2'-дезоксиуридин-5'-трифосфата и Vent-ДНК-полимеразы, но со сравнительно высокой константой диссоциации (400 нМ) [5]. Модифицирующий заместитель содержал алкинильный фрагмент, что обусловливало преимущественно гидрофобные взаимодействия этого аптамера с белковой мишенью. Позже для другой белковой мишени (фактора роста PDGF-BB) показали, что включение в модифицирующий заместитель карбоксамидной группы дополняет характер взаимодействия с белковой мишенью как гидрофобными взаимодействиями, так и водородными связями, что позволило добиться снижения константы диссоциации (около 1 нМ) [6].
Структурный мотив заместителя, связанного с пиримидиновым основанием, влияет не только на природу межмолекулярных взаимодействий аптамера с мишенью, но и на его достаточно сложную уникальную трехмерную структуру [7]. Важно заметить, что mod-dNTP с модифицирующими группами, способными к различным межмолекулярным взаимодействиям, проявляют ингибирующий эффект в ПЦР [8–11]. Для получения аптамеров с низкой константой диссоциации приходится осуществлять поиск структурного мотива заместителя нуклеотида и совместимой с ним ДНК-полимеразы, а также использовать реакцию PEX вместо ПЦР [6].
В случае PEX проблема ингибирования решается увеличением времени элонгации праймера. Тот же прием применительно к ПЦР возможен с существенными ограничениями, так как необходимое число циклов амплификации должно укладываться в приемлемые временные рамки. В случае применения ПЦР для амплификации модифицированных комбинаторных ДНК-библиотек важно учитывать кинетику встраивания модифицированных нуклеотидов. Кроме того, важно контролировать наличие полноразмерного продукта. Tolle и соавт. [12] не удавалось обогащать библиотеки из-за интенсивной конкурентной амплификации нецелевых продуктов. В таком и подобных случаях хорошая кинетика амплификации в ПЦР с детекцией в режиме реального времени относится не только к целевым, но и к побочным продуктам. Следовательно, исследования кинетики амплификации необходимо совмещать с электрофоретическим анализом размера получаемых продуктов. В результате Tolle и соавт. [12] пришли к выводу о том, что в подобных случаях лучше использовать эмульсионную ПЦР. Именно этот подход использован нами ранее [10], но и в этом случае остаются актуальными вопросы как исследования кинетики амплификации, так и контроля размера получаемых продуктов.
В представленной работе изучена динамическая характеристика накопления продукта – эффективность амплификации – при проведении ПЦР с модифицированным субстратом (mod-dUTP), измерены выходы целевого продукта для каждого варианта модифицированного dUTP и проведено сравнение эффективности двух полимераз, принадлежащих семействам А и B, с отсутствующей корректирующей 3'-5'-экзонуклеазной активностью. В качестве матрицы для амплификации использованы как бактериальная ДНК с фиксированной нуклеотидной последовательностью, так и комбинаторная ДНК-библиотека с вырожденной последовательностью нуклеотидов, применяемая как исходная при отборе аптамеров.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материалы и оборудование. В работе использовали немодифицированные dNTPs (“Thermo Scientific”, США), полимеразы Taq (“Thermo Scientific”) и Vent (exo-) (“New England Biolabs”, США). ПЦР в режиме реального времени проводили в амплификаторе iCycler iQ5 (“Bio-Rad Laboratories”, США). В качестве флуоресцентного красителя для ПЦР в режиме реального времени использовали EvaGreen (“Biotium”, США). Для разделения фрагментов ДНК использовали электрофорез в агарозном геле. Для окрашивания гелей использовали бромид этидия (ООО “Хеликон”, Россия). В качестве маркера длин ДНК использовали 50 bp DNA Ladder (“Thermo Scientific”, США).
В работе использованы амплификатор PTC-220 (“Bio-Rad Laboratories”), амплификатор для ПЦР в режиме реального времени IQ5 (“Bio-Rad Laboratories”), ПЦР-анализатор Light Cycler 96 (“Roche”, Швейцария), центрифуга-вортекс “Микроспин” FV-2400 (“Biosan”, Латвия), высокоскоростная центрифуга для микропробирок Frontier 5515 (“Ohaus”, США), система для горизонтального электрофореза (ООО “Хеликон”, Россия), транс-иллюминатор TCP-20.МС 254/312 нм (“Vilber Lourmat”, Франция), цифровой фотоаппарат EOS 500D (“Canon”, Япония) с оптическим фильтром SYBR photographic filter S7569 (“Molecular Probes”, США). Олигонуклеотиды синтезировали с помощью автоматического синтезатора ABI 394 DNA/RNA (“Applied Biosystems”, США). Измерение концентрации олигонуклеотидов и ДНК проводили с помощью спектрофотометра для микрообъемов NanoDrop 1 000 (“Thermo Scientific”).
Модифицированные дезоксинуклеозидтрифосфаты. Использованные в работе модифицированные dUTPs синтезированы по методу, описанному ранее [13].
Бактериальная матрица для ПЦР и праймеры для ее амплификации. Бактериальная матрица представляла собой ПЦР-продукт фрагмента гена rpoB из деконтаминированной геномной ДНК Mycobacterium tuberculosis, штамм H37rv, полученной с использованием следующих праймеров:
105f (прямой) 5'-CGTGGAGGCGATCACACCGCAGACGTTG-3' и
293r (обратный) 5'-AGTGAGACGGGTGCACGTCGCGGACCT-3', впервые описанных ранее [14]. Полученный ПЦР-продукт очищали на спин-колонках (“Bio-Rad Laboratories”), растворяли в деионизованной воде качества milliQ и доводили концентрацию до 104 копий/мкл.
Для ПЦР на бактериальной матрице применяли “вложенные” праймеры, использование которых позволяет получать более короткий ПЦР-продукт [14, 15]:
1272f (прямой) 5'-CGCCGCGATCAAGGAG-TTCT-3',
1398r (обратный) 5'-TCACGTGACAGACCGCCGGG-3'.
Синтетическая вырожденная матрица и праймеры для ее амплификации. В качестве вырожденной матрицы использовали “условно двухцепочечную” ДНК, составленную из двух синтетических олигонуклеотидов с вырожденной центральной частью и взаимно комплементарными фланкирующими частями.
Это Matrix_1f-06:
5'-CTGTCAGCTCCATACTGGTAGCC(N)40GC-GTTCGAATCTAGACGGTACGA-3'
и Matrix_1r-06:
5'-TCGTACCGTCTAGATTCGAACGC(N)40GG-CTACCAGTATGGAGCTGACAG-3'.
Для амплификации матричных вырожденных олигонуклеотидов использовали праймеры:
Forw_1-06 5'-CTGTCAGCTCCATACTGGTA-GCC-3'
и Rev_1-06 5'-TCGTACCGTCTAGATTCGAA-CGC-3'
Использованная в работе составная синтетическая матрица и особенности ее конструирования и синтеза описаны ранее [10].
Для проверки обнаруженного эффекта волнообразной амплификации вырожденных матриц (см. раздел “Обсуждение результатов”) использовали несоставные матрицы, то есть индивидуальные синтетические олигонуклеотиды Matrix_1f-06 и Matrix_1r-06 раздельно в независимых реакциях. В реакционной смеси присутствовало 106 копий матричного олигонуклеотида с вырожденной центральной частью. Расчет концентрации проводили по среднему арифметическому молярного поглощения нуклеотидов (A, T, G и C) по каждой позиции вырожденного олигонуклеотида, исключая фиксированные фланкирующие последовательности, для которых расчет производили с учетом их последовательности.
ПЦР в режиме реального времени. Кинетику амплификации изучали методом ПЦР в режиме реального времени по накоплению флуоресцентного сигнала от интеркалирующего красителя EvaGreen для каждого флуоресцентно меченного dUTP на приборе BioRad iQ5. ДНК-полимеразы Taq и Vent (exo-) использовали в концентрациях, рекомендованных производителем. Реакционная смесь содержала 200 мкM каждого из немодифицированных dNTP (dATP, dCTP, dGTP, dTTP) или один из mod-dUTPs при полной или частичной замене dTTP. Ингибирующий эффект mod-dUTPs оценивали в диапазоне концентраций от 0 (только dTTP в смеси) до 200 мкМ (полная замена dTTP на модифицированный аналог).
Температурно-временной профиль ПЦР состоял из предварительной денатурации при 95°С в течение 3 мин, за которой следовали 40 циклов: 95°С (денатурация ДНК) в течение 30 с, 64°С (отжиг праймеров) в течение 30 с, 72°С (достройка праймеров) в течение 40 с – и завершающая инкубация при 72°С в течение 5 мин.
Эффективность реакции определяли по углу наклона прямого участка S-образной кривой накопления ДНК, представленной в логарифмическом масштабе. Использовали следующую формулу для расчета эффективности: Е = 10tgα, где E – эффективность амплификации (динамический показатель накопления флуоресцентного сигнала), tgα – тангенс угла наклона линейного участка кривой накопления флуоресцентного сигнала.
Расчет выхода продуктов амплификации. Для определения выхода продуктов амплификации проводили ПЦР без добавления интеркалирующего красителя в реакционную смесь. Температурно-временной режим был аналогичен примененному для ПЦР в реальном времени, за исключением числа циклов амплификации – 32 цикла.
Полученные ПЦР-продукты разделяли в 4%-ном агарозном геле, выход реакции оценивали по оптической плотности полос, соответствующих полноразмерным продуктам, с использованием программы ImageJ (https://imagej.nih.gov/ij/).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
В работе исследована субстратная эффективность 13 mod-dUTPs, содержащих заместители различной химической природы.
Для анализа кинетики накопления сигнала в процессе ПЦР (расчетная эффективность амплификации “Е”) проводили ПЦР при полном замещении природного dTTP на каждый из mod-dUTPs с ДНК-полимеразами Taq (семейство A) и Vent (exo-) (семейство B). Кинетику реакции исследовали с помощью ПЦР в режиме реального времени. В качестве матриц для амплификации использовали фрагмент бактериальной ДНК (M. tuberculosis) [14] и вырожденную комбинаторную ДНК-библиотеку [10].
Бактериальная ДНК-матрица представляла собой ПЦР-продукт фрагмента гена rpoB длиной 215 п.н. Для амплификации более короткого внутреннего фрагмента длиной 126 п.н. использовали “вложенные” праймеры.
В качестве вырожденной ДНК использовали комбинаторную ДНК-библиотеку, представляющую собой эквимолярную смесь из двух 86-членных олигонуклеотидов с вырожденной центральной частью и взаимокомплементарными фланкирующими участками: Matrix_1f-06 и Matrix_1r-06. Особенности конструирования и синтеза такой комплексной (составной) библиотеки описаны ранее [10].
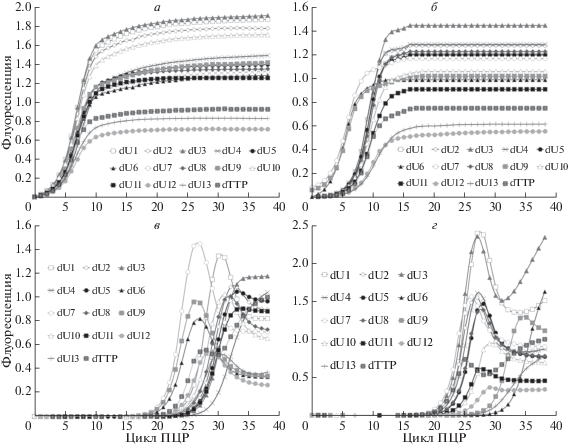
Для каждого из mod-dUTPs проводили амплификацию в 3–5 повторах. Результирующие усредненные кривые накопления флуоресцентного сигнала, полученные с применением интеркалирующего флуоресцентного красителя EvaGreen, приведены на рис. 1. Полученные кривые использовали для расчета эффективности амплификации по наклону прямого участка S-образной кривой накопления сигнала, представленной в логарифмическо масштабе [16, 17]. В случае вырожденной ДНК-библиотеки, при амплификации которой характер накопления сигнала носил волнообразный характер, вычисления осуществляли по первичному увеличению интенсивности сигнала (см. рис. 1в и г).
Рис. 1.
Кинетика накопления продуктов амплификации с включением mod-dUTPs. ПЦР в режиме реального времени проводили на бактериальной ДНК с фиксированной последовательностью с использованием Taq- (а) или Vent (exo-)-полимеразы (б) или на вырожденной синтетической ДНК с теми же полимеразами (в и г соответственно). Визуализация с помощью красителя EvaGreen. Расчетные значения эффективности амплификации “Е” приведены в таблице.
Для определения выхода целевого продукта проводили ПЦР в 3 повторах для каждого mod-dUTP без применения интеркалирующего красителя в реакционной смеси. Визуализацию продуктов реакции осуществляли путем окрашивания бромистым этидием агарозного геля с электроферетически разделенными продуктами ПРЦ. Выход целевого продукта реакции рассчитывали по оптической плотности соответствующих участков электрофореграммы Примеры электрофореграмм для каждой комбинации матрица–полимераза–mod-dUTP приведены на рис. 2.
Рис. 2.
Электрофореграммы продуктов ПЦР, проходившей при полном замещении dTTP на mod-dUTP. 4%-ный агарозный гель окрашен бромистым этидием. Ladder – маркер длин ДНК “GeneRuler 50bp”, NC – отрицательный контроль. Расчетные выходы полноразмерных продуктов приведены в таблице.

Результаты расчета эффективности амплификации и выхода реакции для каждого mod-dUTP приведены в сводной таблице.
Таблица.
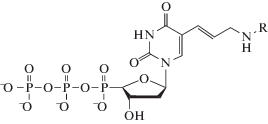
Эффективность амплификации и выход целевого продукта при использовании в качестве субстрата модифицированных производных dUTP вместо dTTP
 |
|||||
| dN | Ra | Полимераза | |||
| Taq | Vent (exo-) | ||||
| Е (Mt/RL)b | η (Mt/RL)c | Е (Mt/RL)b | η (Mt/RL)c | ||
| dTd | – | 1.76/1.76 | 1.00/1.00 | 1.77/1.85 | 1.00/1.00 |
| dU1 |  |
1.74/177 | 1.05/0.79 | 1.75/1.93 | 1.09/1.11 |
| dU2 |  |
1.68/1.76 | 0.74/0.79 | 1.77/1.94 | 0.95/1.03 |
| dU3 | 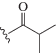 |
1.68/1.74 | 1.05/0.85 | 1.75/1.94 | 0.98/1.01 |
| dU4 | 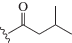 |
1.68/1.65 | 0.85/0.71 | 1.78/1.89 | 0.97/0.95 |
| dU5 | 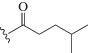 |
1.61/1.65 | 0.87/0.69 | 1.77/1.92 | 0.98/0.64 |
| dU6 |  |
1.57/1.60 | 0.63/0.27 | 1.70/1.74 | 0.85/0.73 |
| dU7 | 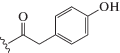 |
1.62/1.78 | 0.54/0.52 | 1.70/1.85 | 0.92/0.75 |
| dU8 | 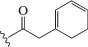 |
1.68/1.75 | 0.42/0.68 | 1.75/1.91 | 0.83/0.77 |
| dU9 | 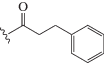 |
1.59/1.62 | 0.32/0.19 | 1.71/1.79 | 0.46/0.68 |
| dU10 |  |
1.60/1.61 | 0.65/0.54 | 1.71/1.82 | 0.74/0.68 |
| dU11 | 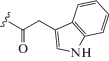 |
1.69/1.73 | 0.35/0.58 | 1.67/1.79 | 0.75/0.57 |
| dU12 | 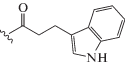 |
1.59/1.65 | 0.39/0.28 | 1.62/1.64 | 0.44/0.41 |
| dU13 | 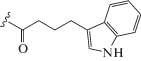 |
1.58/1.67 | 0.36/0.32 | 1.55/1.62 | 0.42/0.55 |
a Структура заместителя R в общей формуле mod-dUTP, приведенной в верхней части таблицы. b Е – эффективность амплификации (Е = 10tgα, где tgα – тангенс угла наклона прямого участка S-образной кривой накопления флуоресцентного сигнала, представленной в логарифмическом масштабе [16]), Mt – фиксированная последовательность бактериальной ДНК (M. tuberculosis, фрагмент гена rpoB, длина 126 п.н.), RL – вырожденная составная ДНК-библиотека (длина 86 н.). c η – выход продукта, нормированный на выход немодифицированной ДНК (ПЦР с использованием dTTP в качестве субстрата). d Природный тимидин, т.е. CH3-группа при С5 азотистого основания.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Для расчета эффективности амплификации (E) общеприняты два метода: по последовательным разведениям образца с определением значения порогового цикла и по углу наклона прямого участка кривой накопления, представленной в логарифмическом масштабе [16], а также их производные, теоретически позволяющие повысить точность вычислений [17–19]. У каждого метода есть как достоинства, так и недостатки [16, 18]. Ramakers и соавт. [16] провели сравнение двух основных методов для определения эффективности ПЦР в режиме реального времени. Отмечено, что метод последовательных разведений требователен к подготовке образцов. Кроме того, для его осуществления необходимо проведение отдельных расчетов для каждого используемого разбавления образца. В методе расчета по углу наклона прямого участка S-образной кривой используют только один образец для вычислений, что может негативно отражаться на точности получаемых данных. Однако преимущества этого метода очевидны при анализе большого числа исследуемых образцов, когда приготовление последовательных разведений затруднительно, – поэтому его часто выбирают в качестве единственного инструмента для оценки эффективности амплификации. Еще одно преимущество метода расчета по углу наклона – непосредственное вычисление эффективности амплификации из отрезка прямой линии регрессии.
Ранее нами проведено сравнение методов последовательных разведений и расчета тангенса угла наклона для определения эффективности амплификации с использованием в качестве субстрата в ПЦР флуоресцентно меченных производных dUTP [20]. При масштабном сравнении этих двух методов выявлен высокий уровень корреляции между ними. В представленной здесь работе для определения эффективности амплификации мы выбрали метод расчета тангенса угла наклона прямого участка S-образной кривой накопления сигнала для всех исследуемых mod-dUTPs.
В случае амплификации бактериальной ДНК (фиксированная нуклеотидная последовательность) получены графики накопления флуоресцентного сигнала, соответствующие канонической S-образной кривой с выходом на плато после определенного числа циклов амплификации (рис. 1а, б).
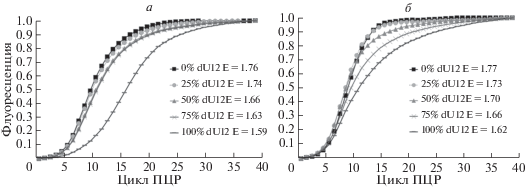
Использование нормировки сигналов (к выбранной константе при выходе кривых на плато) позволяет визуально оценить степень пологости кривых накопления сигнала, которая и служит показателем эффективности амплификации. На рис. 3 приведены выборочные кривые накопления сигнала для различных mod-dUTPs. Видно, что характер наклона кривых (а значит, и эффективность амплификации) варьирует в зависимости от химической природы заместителя в молекуле mod-dUTP.
Рис. 3.
Нормированные кривые накопления ПЦР-продуктов. Выборочные примеры для нескольких mod-dUTPs. В качестве матрицы использован фрагмент бактериальной ДНК с Taq- (а) или полимераза Vent (exo-) (б).
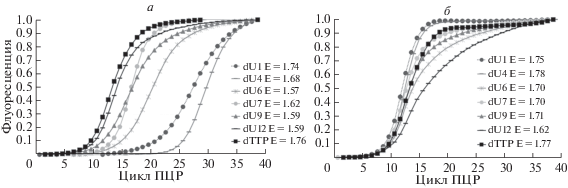
На примере “сложного” субстрата dU12 показано влияние концентрации mod-dUTP на ингибирование ДНК-полимеразы (рис. 4). Тенденция отчетливо прослеживается в случае обеих исследованных полимераз: Taq и Vent (exo-). Наиболее выраженное ингибирующее действие dU12 на амплификацию наблюдали при полном замещении им природного dTTP.
Рис. 4.
Влияние “сложных субстратов” на кинетику амплификации на примере dU12 (структура соединения приведена в таблице). В качестве матрицы использован фрагмент бактериальной ДНК и Taq- (а) или Vent (exo-)-полимераза (б).
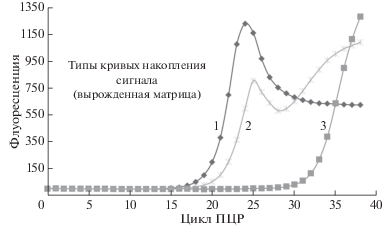
Характер кривых накопления сигнала при амплификации на вырожденных библиотеках воспроизводимо отличался от канонического вида кривых амплификации на бактериальной матрице. Эти кривые можно условно классифицировать на 3 типа (см. рис. 5). Тип 1 характеризуется наличием максимума, за которым следует спад флуоресцентного сигнала с его стабилизацией на плато; тип 2 – появлением второго максимума; тип 3 – поздним появлением порогового цикла, что не дает возможности проследить дальнейшее развитие кривой. Такой вид кривых воспроизводимо наблюдали лишь с применением вырожденной матрицы. Наблюдаемый эффект не был связан с использованными полимеразами и модификацией dUTP (для dTTP характер накопления сигнала был аналогичным). На матрицах с фиксированными последовательностями для всех исследованных mod-dUTPs и полимераз форма кривых амплификации имела канонический тип.
Рис. 5.
Характерные кривые накопления сигнала при амплификации составной (комплексной) вырожденной матрицы. 1, 2 и 3 – различные типы кривых накопления сигнала.
Для того, чтобы исключить влияние приборной части на характер получаемых кривых накопления сигнала, эксперимент повторяли на другом амплификаторе для проведения ПЦР в режиме реального времени (Light Cycler 96). Характер кривых накопления сигнала от вырожденных матриц оставался прежним.
Для проверки влияния составного (комплексного) типа вырожденной матрицы на характер накопления сигнала мы провели ряд экспериментов с индивидуальными синтетическими олигонуклеотидами, входящими в состав матрицы. Понятие “индивидуальный олигонуклеотид” в данном случае применено с определенными оговорками – оно используется для того, чтобы подчеркнуть, что синтез был проведен с применением одной и той же твердофазной колонки и каждый, в прямом смысле, индивидуальный олигонуклеотид из вырожденной библиотеки имел одни и те же фланкирующие последовательности для праймеров. При ПЦР с применением отдельных синтетических библиотек выявлены те же типы кривых накопления сигнала, что и для составной (комплексной) вырожденной библиотеки.
Уменьшение интенсивности флуоресценции после прохождения первого максимума на кривых накопления может быть связано с изменением состава вырожденной библиотеки в процессе амплификации, то есть происходит перераспределение интеркалирующего красителя с образованием структур, препятствующих его дальнейшему включению (это могут быть G-квадруплексы, сложные трехмерные структуры и т.п.). Это и приводит к снижению интенсивности флуоресцентного сигнала, а в ряде случаев ко вторичному росту флуоресценции при дальнейшей амплификации ПЦР-продуктов.
При амплификации вырожденных матриц в классическом варианте ПЦР в одном общем объеме (без использования изолированных реакционных объемов, как в эмульсионной ПЦР [10]) практически неизбежно происходит перераспределение процентного соотношения различных последовательностей из-за конкурентной амплификации; при этом определенную роль в изменении интенсивности флуоресцентного сигнала может играть избирательность интеркалирующего красителя к GC-составу образующихся ПЦР-продуктов. Известно, что взаимодействие интеркалирующего красителя SYBR Green I с ДНК зависит от ряда факторов [21]. Для использованного нами красителя EvaGreen фирмой, его производящей, “Biotium” (США), проведено масштабное исследование, в котором показана низкая зависимость этого красителя от GC-состава ДНК – вплоть до практически полного отсутствия [22], в то время как для SYBR Green I выявлена слабая избирательность в отношении AT-богатых последовательностей. В независимых исследованиях показано, что корреляция между концентрацией введенной в реакцию матрицы и значением порогового цикла носит более линейный характер в случае применения EvaGreen, чем в случае применения SYBR Green I [23]. Определенное влияние на распределение интеркалирующего красителя между продуктами ПЦР могут оказывать образующиеся в процессе амплификации побочные продукты, что характерно при проведении селекции аптамеров и, как следствие, затрудняет получение целевых последовательностей [12]. Детальное исследование факторов, влияющих на формирование того или иного типа кривой амплификации на вырожденных ДНК-библиотеках, не входило в задачи представленного исследования.
При изучении mod-dUTPs как субстратов ДНК-полимераз кинетика реакции не может считаться единственным показателем эффективности работы фермента и его субстратной специфичности и должна быть дополнена другими методами контроля образующихся ПЦР-продуктов. Для анализа образовавшихся продуктов амплификации использовали электрофорез в агарозном геле (см. рис. 2). Контроль целевых продуктов методом электрофореза необходим, кроме прочего, из-за проблемы образования побочных продуктов, что особенно характерно для амплификации вырожденных ДНК (рис. 2). На основании полученных электрофореграмм оценивали выход целевого продукта. Объединенные данные по эффективности амплификации и по выходу целевого продукта для 13 исследованных mod-dUTPs приведены в таблице.
Из табл. 1 видно, что при использовании в качестве субстратов полимераз Taq и Vent (exo-) mod-dUTPs эффективность амплификации снижается по мере увеличения длины (и гидрофобности) цепи насыщенного алифатического углеводорода в позиции С5 уридина. В ряду ароматических и гетероциклических углеводородов эффективность амплификации уменьшается еще более, причем зависимость от размера гидрофобного линкера сохраняется.
Эта тенденция еще более четко выражена при анализе выхода целевых продуктов. Как по эффективности амплификации, так и по выходу целевого продукта полимераза Vent (exo-) более эффективна по сравнению с Taq-полимеразой, для которой относительно хорошими субстратами были лишь производные с короткими фрагментами насыщенных алифатических углеводородов и которая практически непригодна для амплификации ДНК, модифицированных гетероциклическими производными, из-за очень низкого выхода целевых продуктов. В отличие от Taq-полимеразы, Vent (exo-) работала практически со всеми изученными mod-dUTPs, хотя и с разной эффективностью.
На вырожденной матрице средняя эффективность ПЦР выше для обеих исследованных полимераз. Возможно, это связано с тем, что происходит избирательная амплификация последовательностей, содержащих либо меньше A (пар A-T), либо повторов (A)n (пар (A-T)n).
Таким образом, изучение кинетики амплификации позволит индивидуально оптимизировать температурно-временные профили ПЦР для различных модифицированных нуклеозидтрифосфатов при получении модифицированных ДНК в препаративных масштабах для осуществления методологии SELEX.
ВЫВОДЫ
При получении модифицированных ДНК методом ПЦР с матрицей из природных нуклеотидов при полной замене природного dTTP на аналоги dUTP с различными заместителями по С5-положению азотистого основания выявлена корреляция между эффективностью амплификации и выходом полноразмерного целевого продукта.
Показана зависимость эффективности работы полимераз от гидрофобности (длины) углеводородного фрагмента, ковалентно связанного с С5-позицией уридина в молекуле mod-dUTP, а также от химической природы заместителя. Выявлено влияние химической природы заместителя на степень ингибирования ПЦР, показано возрастание ингибирующего эффекта при увеличении концентрации mod-dUTP на примере производных, относящихся к “плохими” субстратам для ДНК-полимераз.
Исследованные соединения пригодны для ферментативного получения модифицированных ДНК, в том числе для отбора аптамеров методом SELEX, с помощью ДНК-полимеразы Vent (exo-).
Работа выполнена при финансовой поддержке Министерства образования и науки Российской Федерации (соглашение № 14.576.21.0096, уникальный идентификатор проекта RFMEFI57617X0096).
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Zhuo Z., Yu Y., Wang M., Li J., Zhang Z., Liu J., Wu X., Lu A., Zhang G., Zhang B. (2017) Recent advances in SELEX technology and aptamer applications in biomedicine. Int. J. Mol. Sci. 18, e2142.
Lapa S.A., Chudinov A.V., Timofeev E.N. (2016) The toolbox for modified aptamers. Mol. Biotechnol. 58, 79–92.
Gold L., Ayers D., Bertino J., Bock C., Bock A., Brody E.N., Carter J., Dalby A.B., Eaton B.E., Fitzwater T., Flather D., Forbes A., Foreman T., Fowler C., Gawande B., Goss M., Gunn M., Gupta S., Halladay D., Heil J., Heilig J., Hicke B., Husar G., Janjic N., Jarvis T., Jennings S., Katilius E., Keeney T.R., Kim N., Koch T.H., Kraemer S., Kroiss L., Le N., Levine D., Lindsey W., Lollo B., Mayfield W., Mehan M., Mehler R., Nelson S.K., Nelson M., Nieuwlandt D., Nikrad M., Ochsner U., Ostroff R.M., Otis M., Parker T., Pietrasiewicz S., Resnicow D.I., Rohloff J., Sanders G., Sattin S., Schneider D., Singer B., Stanton M., Sterkel A., Stewart A., Stratford S., Vaught J.D., Vrkljan M., Walker J.J., Watrobka M., Waugh S., Weiss A., Wilcox S.K., Wolfson A., Wolk S.K., Zhang C., Zichi D. (2010) Aptamer-based multiplexed proteomic technology for biomarker discovery. PLoS One. 5, e15004.
Gold L. (2015) SELEX: how it happened and where it will go. J. Mol. Evol. 81, 140–143.
Latham J.A., Johnson R., Toole J.J. (1994) The application of a modified nucleotide in aptamer selection: novel thrombin aptamers containing 5-(1-pentynyl)-2'-deoxyuridine. Nucleic Acids Res. 22, 2817–2822.
Davies D.R., Gelinas A.D., Zhang C., Rohloff J.C., Carter J.D., O’Connell D., Waugh S.M., Wolk S.K., Mayfield W.S., Burgin A.B., Edwards T.E., Stewart L.J., Gold L., Janjic N., Jarvis T.C. (2012) Unique motifs and hydrophobic interactions shape the binding of modified DNA ligands to protein targets. Proc. Natl. Acad. Sci. USA. 109, 19971–19976.
Копылов А.М., Спиридонова В.А. (2000) Комбинаторная химия нуклеиновых кислот: SELEX. Молекуляр. биология. 34, 1097–1113.
Kuwahara M., Hanawa K,Ohsawa K, Kitagata R, Ozaki H., Sawai H. (2006) Direct PCR amplification of various modified DNAs having amino acids: convenient preparation of DNA libraries with high-potential activities for in vitro selection. Bioorg. Med. Chem. 14, 2518–2526.
Masud M.M., Kuwahara M., Ozaki H., Sawai H. (2004) Sialyllactose-binding modified DNA aptamer bearing additional functionality by SELEX. Bioorg. Med. Chem. 12, 1111–1120.
Лапа С.А., Ромашова К.С., Спицын М.А., Шершов В.Е., Кузнецова В.Е., Гусейнов Т.О., Заседателева О.А., Радько С.П., Тимофеев Э.Н., Лисица А.В., Чудинов А.В. (2018) Получение модифицированных комбинаторных ДНК-библиотек методом ПЦР в обратной эмульсии с последующим разделением цепей. Молекуляр. биология. 52, 984–996.
Vaught J.D., Bock C., Carter J., Fitzwater T., Otis M., Schneider D., Rolando J., Vaugh S., Wilcox S.K., Eaton B.E. (2010) Expanding the chemistry of DNA for in vitro selection. J. Am. Chem. Soc. 132, 4141–4151.
Tolle F., Wilke J., Wengel J., Mayer G. (2014) By-product formation in repetitive PCR amplification of DNA libraries during SELEX. PLoS One. 9, e114693.
Чудинов А.В., Киселева Я.Ю., Кузнецова В.Е., Шершов В.Е., Спицын М.А., Гусейнов Т.О., Лапа С.А., Тимофеев Э.Н., Арчаков А.И., Лисица А.В., Радько С.П., Заседателев А.С. (2017) Ферментативный синтез ДНК с высокой степенью модификации. Молекуляр. биология. 51, 534–544.
Mikhailovich V., Lapa S., Gryadunov D., Sobolev A., Strizhkov B., Chernyh N., Skotnikova O., Irtuganova O., Moroz A., Litvinov V., Vladimirskii M., Perelman M., Chernousova L., Erokhin V., Zasedatelev A., Mirzabekov A. (2001) Identification of rifampin-resistant Mycobacterium tuberculosis strains by hybridization, PCR, and ligase detection reaction on oligonucleotide microchips. J. Clin. Microbiol. 39, 2531–2540.
Gryadunov D., Mikhailovich V., Lapa S., Roudinskii N., Donnikov M., Pan’kov S., Markova O., Zasedatelev A., Mirzabekov A., Kuz’min A., Chernousova L., Skotnikova O., Moroz A. (2005) Evaluation of hybridisation on oligonucleotide microarrays for analysis of drug-resistant Mycobacterium tuberculosis. Clin. Microbiol. Infect. 11, 531–539.
Ramakers C., Ruijter J.M., Deprez R.H., Moorman A.F. (2003) Assumption-free analysis of quantitative real-time polymerase chain reaction (PCR) data. Neurosci. Lett. 339, 62–66.
Peirson S.N., Butler J.N., Foster R.G. (2003) Experimental validation of novel and conventional approaches to quantitative real-time PCR data analysis. Nucleic Acids Res. 31, e73.
Liu W., Saint D.A. (2002) A new quantitative method of real time reverse transcription polymerase chain reaction assay based on simulation of polymerase chain reaction kinetics. Anal. Biochem. 302, 52–59.
Ahmed M., Kim D.R. (2018) PCR: an R package for quality assessment, analysis and testing of qPCR data. Peer J. 6, e4473.
Лапа С.А., Волкова О.С., Спицын М.А., Шершов В.Е., Кузнецова В.Е., Гусейнов Т.О., Заседателев А.С., Чудинов А.В. (2019) Эффективность амплификации и субстратные свойства флуоресцентно меченных трифосфатов дезоксиуридина в ПЦР с ДНК-полимеразами, не обладающими 3′-5′ экзонуклеазной активностью. Биоорг. химия. (в печати).
Ioannou A.K., Alexiadou D.K., Kouidou S.A., Voul-garopoulos A.N., Girousi S.T. (2010) Electroanalytical study of SYBR Green I and ethidium bromide intercalation in methylated and unmethylated amplicons. Anal. Chim. Acta. 657, 163–168.
Mao F., Leung W.Y., Xin X. (2007) Characterization of EvaGreen and the implication of its physicochemical properties for qPCR applications. BMC Biotechnol. 7, 76.
Khan S.A., Sung K., Nawaz M.S.(2011) Detection of aacA-aphD, qacEδ1, marA, floR, and tetA genes from multidrug-resistant bacteria: comparative analysis of real-time multiplex PCR assays using EvaGreen® and SYBR® Green I dyes. Mol. Cell. Probes. 25, 78–86.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


