Молекулярная биология, 2019, T. 53, № 4, стр. 561-573
Элонгационный фактор P: новые механизмы работы и эволюционное разнообразие регуляции трансляции
А. А. Голубев a, Ш. З. Валидов a, К. С. Усачев a, М. М. Юсупов a, b, *
a Институт фундаментальной медицины и биологии, Казанского (Приволжского) федерального университета
420008 Казань, Россия
b Institut de Génétique et de Biologie Moléculaire et Cellulaire
67404 Strasbourg, France
* E-mail: marat@igbmc.fr
Поступила в редакцию 04.03.2019
После доработки 15.03.2019
Принята к публикации 15.03.2019
Аннотация
Синтез белков (трансляция) в клетке происходит в рибосомах с участием дополнительных белковых факторов. Один из таких факторов – фактор элонгации P (EF-P) – трехдоменный белок, который связывается с рибосомой между P- и Е-сайтами вблизи Р-тРНК пептидилтрансферазного центра и Е-сайт-кодоном мРНК. EF-P помогает рибосоме процессировать специализированные трудносинтезируемые аминокислотные последовательности, например полипролиновые мотивы. В первой части обзора рассмотрено эволюционное разнообразие EF-P, возможность регуляции, осуществляемой EF-P, и роль EF-P в поддержании белкового баланса клетки в разных физиологических состояниях. Несмотря на то что функции EF-P хорошо изучены, некоторые важные аспекты его функционирования еще требуют разрешения. Так, получены данные, которые противоречат существующим моделям работы EF-P. Во второй части рассмотрены результаты, согласно которым EF-P участвует в каждой транслокации, а не только в связанных с синтезом полипролина. Это предположение противоречит некоторым положениям известной модели элиминации Е-тРНК в процессе транслокации. В третьей части описана еще мало изученная функция домена III и расширена идея об участии EF-P в предотвращении сдвига рамки считывания, обсуждается также происхождение EF-P и e/aIF5А.
ВВЕДЕНИЕ
Процесс трансляции происходит на рибосомах – сложных молекулярных машинах, обеспечивающих синтез полипептидных цепей в соответствии с последовательностью нуклеотидов в мРНК. Механизмы трансляции у эукариот и прокариот имеют существенные различия, однако структурные элементы, отвечающие за процесс декодирования и синтез полипептидной цепи, высококонсервативны. Для обеспечения точности и эффективности синтеза требуются не только рибосомы, но и специальные белковые факторы, необходимые на разных этапах трансляции. У прокариот одним из таких факторов является EF-P [1], у эукариот имеется аналог этого фактора – eIF5А [2]. Эти факторы часто рассматривают совместно, перенося данные, полученные для одного фактора, на другой, что в большинстве случаев верно и полезно. Однако необходимо принимать во внимание некоторые структурные особенности, которые непременно приводят и к функциональным различиям. В данном обзоре мы сконцентрировались на прокариотическом факторе элонгации EF-P, при этом данные для эукариотического аналога использовали только в качестве дополнения и получения более полной картины.
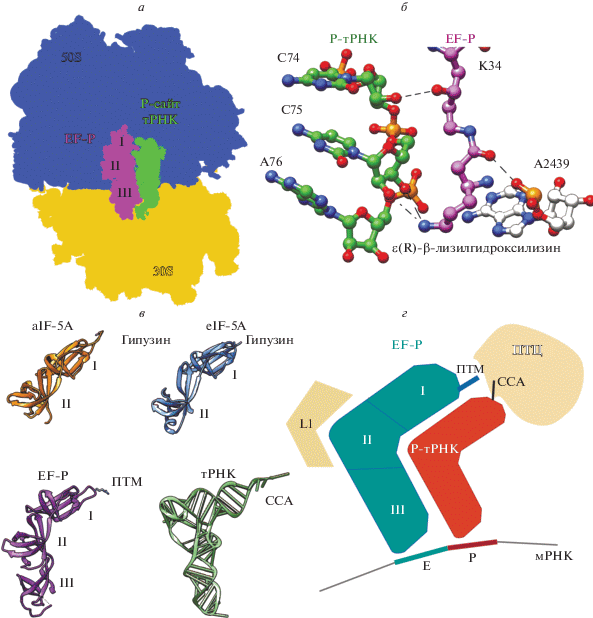
EF-P был открыт в 1975 году при проведении метионин-пуромицинового теста как стимулирующий фактор – обнаружено его положительное влияние на синтез первой пептидной связи [3]. Методом кристаллографии решена структура комплекса EF-P с рибосомой Thermus thermophilus. Оказалось, что EF-P связывается с рибосомой между P- и Е-сайтами и вплотную примыкает к тРНК в Р-сайте (P-тРНК) [4]. EF-P – трехдоменный белок, по форме напоминающий тРНК [5, 6] (рис. 1). Домен I располагается вблизи акцепторного конца P-тРНК (CCA) и пептидилтрансферазного центра (ПТЦ). Домен II контактирует с белком L1, необходимым, по всей видимости, для удаления фактора из рибосомы. Домен III находится вблизи антикодоновой петли P-тРНК и кодона мРНК в Е-сайте.
Рис. 1.
а – Место связывания EF-P с рибосомой. б – Взаимодействие EF-P (обозначен розовым) с ССА-концом P-тРНК (выделено зеленым цветом). Пунктирной линией показаны возможные водородные связи между ε(R)-β-лизилгидроксильной модификацией К34 EF-P с ССA-концом P-тРНК и А2439. в – Структура EF-P в сравнении с его аналогами у архей и эукариот (aIF5A и eIF5A соответственно). Домены I и II сравниваемых структур обладают высоким сходством. В верхнем правом углу домена I располагается уникальная посттранстрансляционная модификация. Структурное сходство EF-P c тРНК – пример молекулярной мимикрии. г – Схематичное изображение расположения EF-P в соответствии с контактирующими структурами внутри рибосомы.
Считается, что EF-P ускоряет трансляцию специализированных аминокислотных последовательностей, в состав которых входят полипролиновые участки, такие как, например PPP и АPP [7, 8] (рис. 2). Получены данные, согласно которым для этого необходимы контакты EF-P с D-петлей P-тРНКfMET и P-тРНКPRO, однако подобная функция не подтверждена в случае тРНК с другими аминоацильными остатками [9, 10].
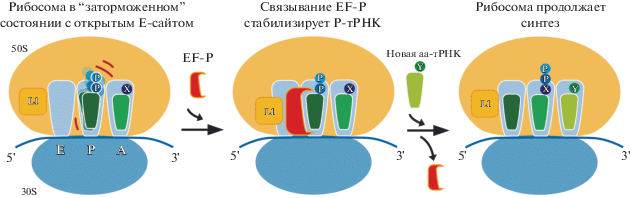
Рис. 2.
Механизм работы EF-P по устранению остановки или замедления трансляции в случае синтеза мотивов полипролинового типа. Во время замедления трансляции EF-P связывается со свободным Е-сайтом рибосомы, создавая условия для возобновления (либо ускорения) синтеза, предположительно путем стабилизации Р-тРНК.
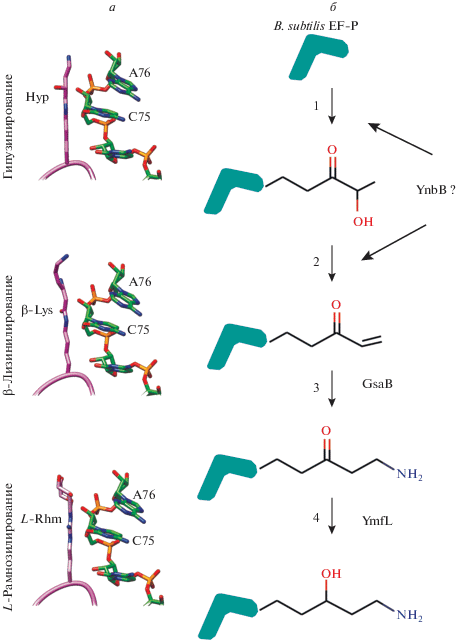
Для максимально эффективного осуществления функции ускорения синтеза мотивов полипролинового типа необходима специальная посттрансляционная модификация в консервативном регионе петли домена I EF-P (Lys32 у Escherichia coli и Staphylococcus aureus; Arg34 у Pseudomonas aeruginosa), наиболее сближенной с ПТЦ. На сегодняшний день известно четыре различных типа уникальных модификаций: B-лизинилирование [11], 5-аминопентанолирование [12], рамнозилирование [13] у EF-P, гипузинирование – у его эукариотического аналога – eIF5A [14]. Данные модификации позволяют удлинить EF-P в сторону ССА-конца Р-тРНК и ПТЦ. Известны полные системы ферментов, осуществляющих три типа модификаций EF-P. Недавно на примере Bacillus subtilis была предложена система белков для модификации по типу 5-аминопентанола [15] (рис. 3), которая, по всей видимости, присутствует у большой части грамположительных бактерий (Firmicutes), в том числе у патогенного S. aureus. Интересно, что гомологи ферментов-модификаторов всех систем найдены лишь у трети всех известных бактерий. Сведения о наличии и типах модификации EF-P у других прокариот в настоящее время отсутствуют.
Рис. 3.
Известные типы модификаций EF-P/IF5A относительно ССА-конца Р-тРНК: гипузин, β-лизинилирование, L-рамнозилирование (a), и предполагаемая схема посттрансляционной модификации EF-P – 5-аминопентанилирования по данным [15] (б). YnbB предположительно переносит субстрат с ацил-переносящего белка с образованием первого интермедиата в процессе формирования 5-аминопентанола (шаг 1 или 2). GsaB (паралог глутамат-1-полуальдегид-аминотрансферазы HemL) предположительно добавляет аминогруппу к молекуле пентенона (шаг 3). Ymfl участвует в восстановлении 5-аминопентанона до 5-аминопентанола на последнем этапе (шаг 4) [16].
В представленном обзоре рассмотрены некоторые стороны эволюционного разнообразия EF-P и его возможная функциональная и регуляторная роль в изменении баланса белков в клетке при различных функциональных состояниях. Подробно описаны результаты последних исследований, их вклад в понимание функций EF-P и представления об общих механизмах синтеза белков у прокариот. В следующей части обзора рассмотрена функция EF-P, связанная с ролью третьего домена в стабилизации рамки считывания.
УЧАСТИЕ EF-P В ИЗМЕНЕНИИ СОСТОЯНИЙ КЛЕТКИ
EF-P найден в клетках всех известных бактерий, включая самую маленькую – свободно живущую Mycoplasma genitalium, а также эндосимбиотических бактерий, за исключением трех видов: Carsonella ruddii, Hodgkinia cicadicola и Nasuia deltocephalinicola [17]. При этом делеция гена efp не летальна для большинства исследованных видов: P. aeruginosa и Shewanella oneidensis [18], E. coli [19], Salmonella enterica [20–22], B. subtilis [23, 24], Brucella abortus [25], Agrobacterium tumefaciens [26]. Однако для M. tuberculosis этот ген оказался жизненно важным [27]. Эукариотический аналог EF-P – eIF5A – абсолютно необходим для жизнеспособности организма [28–30]. При этом в клетках эукариот общее количество белков с полипролиновыми (PPP, PPG) мотивами превышает 10% и растет при увеличении сложности организма [31]. Количество кодирующих последовательностей с тремя последовательно расположенными остатками пролина у M. tuberculosis равно 420, что составляет более 10% от общего количества кодирующих последовательностей (4034), тогда как у E. coli K12 только около 2% белков содержат РРР [28]. Предполагается, что жизненная необходимость в гене efp определяется количеством РРР-белков. Следует сказать, что мы приводим расчеты, сделанные до того момента, когда были найдены новые типы трудносинтезируемых аминокислотных последовательностей, поэтому проведение повторного анализа может расширить наше понимание этой взаимосвязи.
Можно предположить, что по мере усложнения организации организмов полипролиновые мотивы в белках используются все чаще, а наличие специализированных факторов (EF-P/eIF5А) и систем их модификации становится критически необходимым. Организмы с малым количеством аминокислотных последовательностей полипролинового типа в кодирующих областях имеют возможность координировать скорости синтеза некоторых белков с помощью EF-P/eIF5А. По всей видимости, подобная координация может происходить не только за счет изменения интенсивности синтеза EF-P/eIF5А, но и в результате использования изоформ и посттрансляционных модификаций фактора. Интересно, что у большинства эукариот, в том числе у дрожжей и человека, известны две изоформы eIF5А, тогда как у некоторых растений, например у Arabidopsis thaliana, таких изоформ четыре [32]. Можно предположить, что изоформы eIF5А в данном случае могут заменять действие специализированной модификации – два белка могут различаться сродством к рибосоме, или же выполнять две разные функции. В настоящее время из прокариот только у Lactobacillus jenseni выявлены две формы EF-P, гомологичные на 47% [33]. Если облегчение синтеза полипролиновых мотивов это единственная функция фактора элонгации P, то непонятно, зачем в клетках L. jenseni синтезируются две изоформы EF-P, поскольку геном этой лактобациллы кодирует всего один белок с полипролиновым мотивом [33].
Предполагается, что регуляция скорости синтеза белков с помощью EF-P могла бы достигаться путем включения системы специфической модификации в условиях, когда необходимы белки с трудносинтезируемыми аминокислотными последовательностями, и выключения в других обстоятельствах. Немодифицированное состояние EF-P может быть доминирующим, при этом немодифицированный EF-P может облегчать трансляцию полипролиновых мотивов и выполнять при этом другие функции. Мы предполагаем, что при необходимости возможно увеличение синтеза ферментов-модификаторов, что позволяет ускорить получение набора белков с трудносинтезируемыми последовательностями. Таким образом, скорость синтеза белков могла бы зависеть от уровня синтеза самого EF-P и регуляции уровня синтеза его модификаторов.
Некоторые микроорганизмы, например Pseudohongiella spirulinae, Thalassolutuus oleivorans MIL-1 и Nitrincola nitratireducens, имеют одновременно две системы модификации EF-P: системы рамнозилирования и β-лизинилирования [34–36]. Зачем нужны этим бактериям две системы модификации пока остается загадкой, однако можно предположить, что различные модификации по-разному влияют на проявление функций EF-P. Возможно, изучение функциональной активности EF-P одного организма с различными типами модификаций, и структурные исследования комплексов таких химерных белков с рибосомой позволят узнать, почему модификации столь различны, каким образом они влияют на баланс белков с трудносинтезируемыми аминокислотными последовательностями, насколько количественно отличается активность EF-P c разными модификациями, и какими структурными особенностями это можно объяснить.
Интересной представляется также роль трудносинтезируемых аминокислотных последовательностей – являются ли они структурно значимыми, либо выполняют лишь регуляторную роль при синтезе. Полипролиновые мотивы часто находятся в междоменных пространствах [37]. Этот факт можно интерпретировать двояко – мы не можем с уверенностью определить одну исключительную роль таких мотивов. Находясь в междоменном пространстве, трудносинтезируемые последовательности полипролинового типа могут обладать как структурной функцией – создавать изгиб в аминокислотной цепи, разрушая монотонные структуры, так и регуляторной – останавливая трансляцию после синтеза крупного домена, что помогает правильно свернуться уже синтезированной части белка.
Роль пауз в трансляции на специфических участках мРНК может заключаться в необходимости регуляции синтеза во время элонгации. Синтез белка может регулироваться с помощью удлинения или укорочения паузы, а EF-P/eIF5А могут, вероятно, служить регуляторами такого рода [38]. Способность к регуляции скорости синтеза определенного набора белков (особенно, если этот набор содержит регуляторы транскрипции) дает возможность изменять общий белковый баланс клетки. Потенциально EF-P может служить переключателем состояний клетки, например, вводить клетку в состояние ответа на стрессовые условия или переход к патогенному состоянию [39]. При этом удаление efp нелетально в благоприятных условиях, но делает невозможным переключение между разными состояниями [21, 39–42]. Возможно, что эукариотический фактор eIF5А отвечает за процессирование большого количества регулирующих мотивов и специализируется именно на регуляции [43]. Учитывая большое количество зависимых от eIF5А белков с трудносинтезируемыми последовательностями, не удивительно, что удаление этого фактора приводит к летальному эффекту. На сегодняшний день мы мало знаем о том, как выглядит сеть влияния EF-P/eIF5А на синтез других белков в разных условиях, поэтому исследования в этой области позволят уточнить роль данных факторов в регуляции баланса белков в клетке.
НОВЫЕ ДАННЫЕ О РАБОТЕ EF-P
Наряду с расширением числа исследований в рамках модели, в которой функция EF-P заключается преимущественно в помощи трансляции мотивов полипролинового типа, в последние годы стали появляться работы, предлагающие пересмотреть некоторые положения этой модели. Прежде всего, при исследовании eIF5А был значительно расширен список трудносинтезируемых аминокислотных последовательностей [43]. Из 29 самых эффективных трудносинтезируемых трипептидов 18 не содержали последовательно расположенных остатков пролина, а четыре мотива не содержали пролин. При этом прокариотический фактор EF-P не ускоряет трансляцию большинства таких мотивов, что, по всей видимости, оставляет за ним функцию преимущественно полипролинового акселератора. В случае EF-P некоторые неполипролиновые мотивы – TyrIleArgTyrIleArg, GlySerCysGlyProGly – также рассматриваются, как трудносинтезируемые [44], но тесты in vivo и in vitro не показывают значительного влияния данных мотивов на замедление синтеза белка [8]. По всей видимости, роль eIF5А в облегчении синтеза мотивов такого рода гораздо шире, что согласуется с его абсолютной необходимостью для жизни эукариотической клетки, в отличие от EF-P. Некоторые предположения о структурной природе сложности синтеза полипролинововых пептидов уже обсуждались ранее [45], однако на данный момент не известно, каким образом другие аминокислотные последовательности вызывают остановку рибосомы в процессе трансляции.
В 2017 году появились данные другого исследования [46], которые подрывают теорию ускорения синтеза полипролиновых мотивов. С помощью технологии флуоресцентной микроскопии удалось рассчитать частоту ассоциации EF-P с рибосомой. Показано, что EF-P связывается с рибосомой гораздо чаще, чем необходимо для устранения возникающих полипролиновых пауз [46].
Исходя из сложившейся теории, согласно которой EF-P ускоряет образование пептидной связи в остановившейся рибосоме при синтезе последовательностей полипролинового типа, пустой Е-сайт можно считать обязательным условием для работы EF-P [17]. При этом ранее показали, что при нормальной скорости трансляции свободная диффузия тРНК из Е-сайта при транслокации явление редкое, поэтому вероятность того, что Е-сайт будет свободен, мала [47]. Таким образом, рибосома может иметь свободное место для EF-P в двух случаях: при остановке синтеза, когда из-за возникшей паузы тРНК уходит из Е-сайта, и при синтезе первой пептидной связи, когда E-сайт еще не занят. Согласно [46], в нормальных условиях EF-P должен взаимодействовать с 25–100% транслирующих рибосом. Это число варьирует в зависимости от того, какие данные использовали для расчета количества копий EF-P в клетке: 25% в случае данных 1980 года [48] и 100% – в случае 2016 года [49]. Результаты дальнейших работ по уточнению количества копий EF-P в клетках могут помочь в разрешении вопроса о точном числе транслокаций с участием EF-P.
Новая количественная оценка взаимодействий EF-P с рибосомой противоречит данным о количестве рибосом, транслирующих мотивы XProProX в единицу времени – 0.05% (подробные расчеты приведены в [46]). По-видимому, EF-P cканирует рибосомы гораздо чаще, чем если бы задержанные полипролиновым мотивом-рибосомы были его единственной мишенью. Здесь существует несколько вариантов: 1) либо количество пауз трансляции намного превышает расчетные значения; 2) либо EF-P взаимодействует с рибосомой независимо от пауз. В первом случае, по-видимому, удастся уложиться в нижнюю планку данных (25% транслокаций) при добавлении в расчеты найденных новых трудносинтезируемых аминокислотных последовательностей, а также при учете пауз, возникающих по причинам, не связанным напрямую с синтезом аминокислотных мотивов, но при которых E-сайт непременно будет открыт для посадки EF-P. Во втором случае EF-P, по всей видимости, может не обладать специфичностью к пролиновым остановкам и взаимодействовать в любых случаях с открытым Е-сайтом, что вступает в противоречие с некоторыми аспектами существующих положений о процессе транслокации [47]. Интересно, что по данным флуоресцентной микроскопии Е-сайт становится свободным гораздо чаще, чем считалось ранее. Видимо, при транслокации деацилированная тРНК может свободно выходить из Е-сайта без необходимости ее высвобождения в акте транслокации, что противоречит данным о частоте событий свободной диффузии тРНК, упоминаемым ранее [47]. Причем процесс выхода должен происходить достаточно быстро, чтобы EF-P успел связаться в Е-сайте и выйти из него, возможно, за счет транслокационного состояния рибосомы (рис. 4). Таким образом, исходя из новых данных, часто открытый Е-сайт предполагает, что транслокация не нужна для диффузии E-тРНК, а EF-P связывается с рибосомой при каждой транслокации.
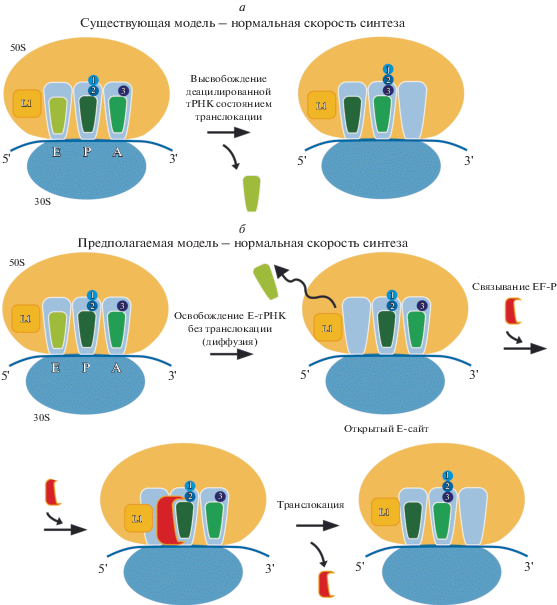
Рис. 4.
Новая предполагаемая схема транслокации с участием EF-P по данным [50]. a – По существующей модели диффузия Е-тРНК при нормальной скорости синтеза ограничена, и удаление Е-тРНК происходит во время транслокации [47]. б – По новой модели при нормальной скорости синтеза E-тРНК свободно освобождается из рибосомы, и образуется пустой Е-сайт – необходимое условие для связывания EF-P. После связывания EF-P осуществляет свои функции и высвобождается из рибосомы либо при помощи последующей транслокации, либо свободно с помощью диффузии до транслокационных событий. В данной модели EF-P участвует при каждой транслокации.
По всей видимости, большинство событий связывания EF-P не относятся к функциональным, но при этом они безвредны для синтеза белков. Возможно, что частое связывание EF-P позволяет фактору постоянно находиться на месте функционирования и экстренно устранять внезапно возникающие паузы. С другой стороны, участие EF-P в столь большом количестве транслокаций может скрывать другие функции. Имеющиеся данные не дают никаких оснований для исключения возможности взаимодействия EF-P с рибосомой в других случаях пауз, не связанных с трансляцией трудносинтезируемых аминокислотных последовательностей. Таким образом, дальнейшие исследования EF-P помогут лучше понять не только специализированную роль этого фактора, но и общие механизмы элонгационного цикла у бактерий.
ОДИН БЕЛОК – ДВЕ ФУНКЦИИ
Подавляющее большинство работ по исследованию EF-P направлено на выяснение функций первого домена: поиск новых типов модификаций, уточнение и поиск новых трудносинтезируемых аминокислотных последовательностей, выяснение механизма возникновения и снятия паузы. Мы бы хотели показать другую функциональную сторону EF-P, которую может нести на себе домен III.
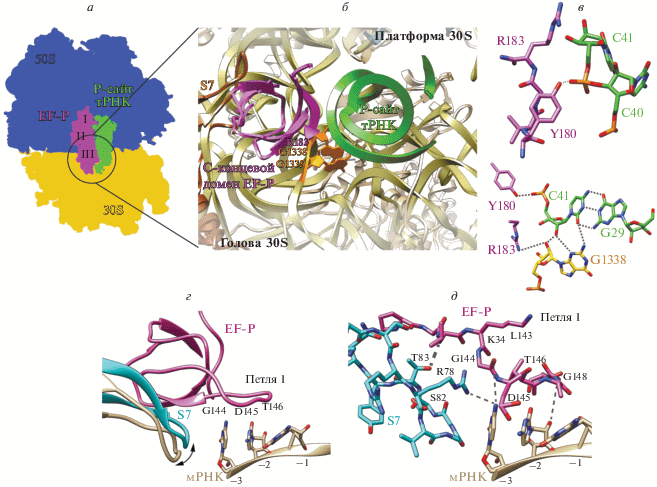
В большей части структурных исследований комплексов EF-P с рибосомой мы наблюдаем низкое разрешение в районе домена III EF-P, возможно, ввиду его большой подвижности. Однако имеющиеся структурные данные [9] позволяют предположить возможную функцию домена III. Домен III частично занимает Е-сайт около антикодоновой петли P-тРНК, а петля № 1 домена III тянется в сторону мРНК. Предполагается, что взаимодействие третьего домена в этой области может ограничивать преждевременное движение тРНК в Е-сайт. Остатки Y180 и R183 домена III стабилизируют A-малые взаимодействия между основаниями антикодоновой петли и основаниями A1339 и G1338 16S рРНК. Эти контакты должны разорваться при транслокации, поэтому предполагают, что нуклеотиды A1339 и G1338 действуют наподобие “ворот” между Е- и P-сайтами [51] (рис. 5). Получены данные с хорошим разрешением в области дезорганизованной петли № 1 домена III [45]. Обнаружено, что домен III контактирует с P-тРНК, рибосомным белком S7, а также непосредственно с кодоном мРНК в Е-сайте (рис. 5). Предполагается, что EF-P может в какой-то мере стабилизировать и позиционировать мРНК в Е-сайте. Более того, имеется вероятность, что EF-P может обладать кодон-специфичностью наподобие факторов терминации RF1 и RF2 [52].
Рис. 5.
Стабилизация рамки считывания с помощью EF-P. а‒в – Расположение С-домена EF-P позволяет стабилизировать антикодоновую петлю Р-тРНК и нуклеотиды A1339 и G1338 рРНК, которые служат своеобразными “воротами” между А- и Е-сайтами (по данным [9]). г, д – Петля I предположительно имеет контакты с мРНК в Е-сайте и белком S7. Показано расположение белка S7 в присутствии и в отсутствие EF-P. Таким образом, EF-P, по-видимому, стабилизирует рамку считывания, предотвращая преждевременное продвижение P-тРНК в Е-сайт.
Известно, что замедление скорости трансляции и возникновение пауз приводит к различным негативным для правильного синтеза белков последствиям (подробный обзор в [53]). При остановке трансляции часто возникает сдвиг рамки считывания. По всей видимости, сдвиг рамки вызван скольжением на какой-то промежуточной стадии транслокации, когда из А- и P-сайтов уже частично вышли тРНК, но они все еще остаются связанными с мРНК. И, очевидно, что в таком положении взаимодействие тРНК с мРНК ослаблено, разрешая тем самым сдвиг рамки считывания ([38], стр. 261). Остановки могут возникать в целях регуляции синтеза белка, сворачивания уже синтезированной части полипептида [54, 55]. Чтобы паузы смогли помочь белку свернуться, не вызывая при этом побочных эффектов, должны существовать механизмы, которые помогают избегать сдвига рамки считывания или проскока. С другой стороны, сдвиг рамки считывания может происходить как трансляционная ошибка. Кроме того, многие ретровирусы и РНК-содержащие вирусы используют программируемый сдвиг рамки считывания для синтеза большего набора белков с одного участка РНК/ДНК [4]. В работах на E. coli показано, что при нехватке аа-тРНК для обслуживания некоторых кодонов, например, в условиях стресса или голодания, происходят частые сдвиги рамки считывания, обусловленные появлением большого количества пауз – это приводит к образованию нонсенс-продуктов [56, 57]. У E. coli паузы также могут индуцировать трансляционный проскок – рибосома пропускает часть участка мРНК и продолжает трансляцию, когда удается восстановить взаимодействие с кодоном ниже по цепи мРНК [58]. Суммируя, можно сказать, что существует обширный ряд событий, при которых возникают паузы, которые, в свою очередь, приводят к сдвигу рамки считывания. При этом при скорости синтеза 10–20 аминокислот в секунду рибосома E. coli делает менее одной ошибки сдвига рамки считывания на 30 000 аминокислот, что 10 раз меньше относительно других трансляционных ошибок. На данный момент не до конца понятно, каким образом рамка считывания остается настолько стабильной.
EF-P потенциально может принимать участие в устойчивости к ошибкам трансляции: показано участие EF-P в предотвращении сдвига рамки считывания в некоторых условиях [59]. Установлено, что EF-P может помогать избегать дефектов транслокации при ошибках декодирования в A-сайте [60]. По данным этих исследований выяснилось, что наличие специфической модификации EF-P положительно влияет на предотвращение негативных эффектов. Интересно, что при анализе мутантов S. enterica с нокаутом EF-P [61] выяснилось, что штаммы без EF-P отличаются повышенной чувствительностью к гентамицину, который присоединяется к 30S субъединице в области декодирующего центра и приводит к неправильному кодированию.
Предполагается, что EF-P может служить стабилизирующим фактором при возникновении разного рода пауз, потому что он, по нашим представлениям, взаимодействует с рибосомой, когда Е-сайт пуст, что случается при синтезе первой пептидной связи, а также при паузах или снижении скорости трансляции, когда деацилированная тРНК выходит из Е-сайта, а новая там еще не появилась. Если предположить, что EF-P связывается с рибосомой независимо от пауз при каждой транслокации (согласно модели, описанной выше), то нет никаких противоречий с идеей, согласно которой EF-P может помогать стабилизировать рамку именно при паузах. Более того, при таком подходе EF-P теоретически должен контролировать рамку считывания при каждой транслокации, что, несомненно, важно для правильного синтеза белка и жизнедеятельности клетки. Данные об участии EF-P в 25–100% событий транслокаций предоставляют свободную нишу для другой функции EF-P в дополнение к функции улучшения синтеза аминокислотных последовательностей полипролинового типа. Предполагается, что EF-P может выполнять роль “стража рамки считывания”. Удаление EF-P в таком случае может и не приводить к немедленной гибели бактерии из-за недостатка белков с трудносинтезируемыми аминокислотными последовательностями, но должно увеличивать число ошибок трансляции (как уже показано в [59]. Проведение большего числа исследований по влиянию EF-P на количество ошибок рамки считывания могло бы расширить наше понимание работы EF-P. Рассматривая функции EF-P, мы можем предположить возможность появления специфичных модификаций фактора. Гипотетическая функция “стража рамки считывания” могла появиться первой для обеспечения правильного кодирования, далее эффективность связывания фактора повышалась с помощью модификации. Затем в силу того, что предковый EF-P уже функционировал в рибосоме, в том числе и во время пауз, стали использоваться новые возможности модификации, направленные на улучшение синтеза мотивов полипролинового типа. Видимо, модификации появлялись относительно независимо у разных организмов, что привело к их многообразию. Интересно, что у eIF5А нет третьего домена и, предположительно, стабильность рамки считывания должна обеспечиваться за счет других механизмов: особенностей строения рибосом либо механизмов, опосредованных сложностью контроля и регуляции синтеза белка у эукариот. Все cказанное позволяет решить проблему, связанную с эволюцией предкового белка EF-P/eIF5А (рис. 6). Здесь имеются две возможности – либо трехдоменный предковый белок EF-P потерял второй домен в результате делеции и появился eIF5А, либо путем дупликации домена II двухдоменный предковый белок eIF5А получил новой домен и появился EF-P.
Рис. 6.
Предполагаемая схема эволюции факторов EF-P и eIF5A. Два варианта событий: а – появление современного eIF5A путем делеции одного из доменов B у древнего EF-P; б – появление современного EF-P путем дупликации домена B у древнего eIF5A. в – Особенности гомологии EF-P и eIF5A. Домены II и III у EF-P сходны и могли произойти друг от друга путем дупликации, однако известно, что второй домен современного eIF5A (обозначен розовым и римской цифрой III) гомологичен третьему домену современного EF-P (также показан розовым и римской цифрой III), а не второму домену (бирюзовый цвет, римская цифра II).
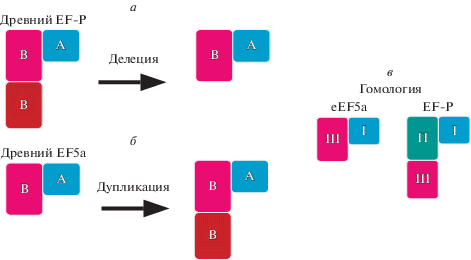
Принимая во внимание эволюционную первичность прокариот, а также тот факт, что часть С-домена eF5А крайне сходна с III доменом EF-P [62], более предпочтительным считается образование eEF5А из трехдоменного предкового EF-P путем делеции региона b10–b14 (домен II) EF-P. Кроме того, стабилизация рамки считывания, видимо, появилась раньше функции обеспечения синтеза сложных мотивов которую у современных архей и эукариот выполняют aIF5A и eIF5А соответственно.
ЗАКЛЮЧЕНИЕ
Как мы видим, изучение механизмов работы EF-P еще далеко от завершения – остается много неразрешенных вопросов, касающихся не только механизмов действия непосредственно EF-P, но и деталей элонгационного цикла у прокариот в целом, а также механизмов возникновения ошибок рамки считывания. Для разрешения всех противоречий необходимы новые данные. В первую очередь, требуется точно установить процент событий транслокаций с участием EF-P. Эти расчеты опираются на данные о количестве копий EF-P в клетках, поэтому нужно оценить число молекул-EF-P у различных видов бактерий. Критически важно также получение новых данных о влиянии EF-P на снижение числа ошибок сдвига рамки считывания. Другая широкая область исследований – поиск новых типов модификаций EF-P и систем белков-модификаторов. На данный момент мы не знаем, как 2/3 всех прокариот модифицируют свой EF-P, и модифицируют ли они его вообще. Интересные подробности могут открыть исследования регуляторных сетей EF-P: определение наборов белков с трудносинтезируемыми аминокислотными последовательностями и их функций, скорость синтеза которых предположительно должна регулироваться EF-P, а также выявление изменений баланса белков при различных условиях.
Не остается в стороне и прикладной аспект. Рибосома и факторы трансляции – хорошая мишень для антибиотиков. Анализ вовлеченности EF-P в общие процессы патогенности и процессы ответа на стрессовые условия, а также выявление видовых особенностей структуры EF-P позволят разработать новые способы терапии, что особенно актуально, учитывая все возрастающую антибиотикорезистентность патогенных микроорганизмов.
Работа выполнена в рамках гранта РНФ17-74-20009.
Настоящая статья не содержит каких-либо исследований с участием людей в качестве объектов исследований.
Настоящая статья не содержит каких-либо исследований с использованием животных в качестве объектов.
Конфликт интересов: Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Rajkovic A., Ibba M. (2017) Elongation factor P and the control of translation elongation. Annu. Rev. Microbiol. 8(71), 117–131.
Rossi D., Kuroshu R., Zanelli C.F., Valentini S.R. (2014) eIF5A and EF-P: two unique translation factors are now traveling the same road. Wiley Interdiscip. Rev. RNA. 5(2), 209–222.
Glick B.R., Ganoza M.C. (1975) Identification of a soluble protein that stimulates peptide bond synthesis. Proc. Natl. Acad. Sci. USA. 72, 4257–4260.
Jacks T., Madhani H.D., Masiarz F.R., Varmus H.E. (1988) Signals for ribosomal frameshifting in the Rous sarcoma virus gag-pol region. Cell. 55(3), 447–458.
Hanawa-Suetsugu K., Sekine S., Sakai H., Hori-Takemoto C., Terada T., Unzai S., Tame J.R., Kuramitsu S., Shirouzu M., Yokoyama S. (2004) Crystal structure of elongation factor P from Thermus thermophilus HB8. Proc. Natl. Acad. Sci. USA. 101, 9595–9600.
Choi S., Choe J. (2011) Crystal structure of elongation factor P from Pseudomonas aeruginosa at 1.75 Å resolution. Proteins. 79, 1688–1693.
Doerfel L.K., Wohlgemuth I., Kothe C., Peske F., Urlaub H., Rodnina M.V. (2012) EF-P is essential for rapid synthesis of proteins containing consecutive proline residues. Science. 339, 85–88.
Peil L., Starosta A.L., Lassak J., Atkinson G.C., Virumae K., Spitzer M., Tenson T., Jung K., Remme J., Wilson D.N. (2013) Distinct XPPX sequence motifs induce ribosome stalling, which is rescued by the translation elongation factor EF-P. Proc. Natl. Acad. Sci. USA. 110, 15265–15270.
Blaha G., Stanley R.E., Steitz T.A. (2009) Formation of the first peptide bond: The structure of EF-P bound to the 70S ribosome. Science. 325(5943), 966–970.
Katoh T., Wohlgemuth I., Nagano M., Rodnina M.V., Suga H. (2016) Essential structural elements in tRNA(Pro) for EF-P-mediated alleviation of translation stalling. Nat. Commun. 7, 11657.
Bullwinkle T.J., Zou S.B., Rajkovic A., Hersch S.J., Elgamal S., Robinson N., Smil D., Bolshan Y., Navar-re W.W., Ibba M. (2013) (R)-β-lysine-modified elongation factor P functions in translation elongation. J. Biol. Chem. 288, 4416–4423.
Rajkovic A., Hummels K.R., Witzky A., Erickson S., Gafken P.R., Whitelegge J.P., Faull K.F., Kearns D.B., Ibba M. (2016) Translation control of swarming proficiency in Bacillus subtilis by 5-amino-pentanolylated elongation factor P. J. Biol. Chem. 291(21), 10976–10985.
Lassak J., Keilhauer E.C., Furst M., Wuichet K., Godeke J., Starosta A.L., Chen J.M., Sogaard-Andersen L., Rohr J., Wilson D.N., Haussler S., Mann M., Jung K. (2015) Arginine-rhamnosylation as new strategy to activate translation elongation factor P. Nat. Chem. Biol. 11, 266–270.
Dever T.E., Gutierrez E., Shin B.S. (2014) The hypusine-containing translation factor eIF5A. Crit. Rev. Biochem. Mol. Biol. 49(5), 413–425.
Witzky A., Hummels K.R., Tollerson R. 2nd, Rajkovic A.1, Jones L.A., Kearns D.B., Ibba M. (2018) EF-P Posttranslational modification has variable impact on polyproline translation in Bacillus subtilis. MBio. 9(2), e00306–18.
Hummels K.R., Witzky A., Rajkovic A., Tollerson R., Jones L.A., Ibba M., Kearns D.B. (2017) Carbonyl reduction by YmfI in Bacillus subtilis prevents accumulation of an inhibitory EF-P modification state. Mol. Microbiol. 106(2), 236–251.
Lassak J., Wilson D.N., Jung K. (2016) Stall no more at polyproline stretches with the translation elongation factors EF-P and IF-5A. Mol. Microbiol. 99(2), 219–235.
Lassak J., Keilhauer E.C., Furst M., Wuichet K., Godeke J., Starosta A.L., Chen J.M., Sogaard-Andersen L., Rohr J., Wilson D.N., Haussler S., Mann M., Jung K. (2015) Arginine-rhamnosylation as new strategy to activate translation elongation factor P. Nat. Chem. Biol. 11(4), 266–270.
Baba T., Ara T., Hasegawa M., Takai Y., Okumura Y., Baba M., Datsenko K.A., Tomita M., Wanner B.L., Mori H. (2006) Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006.0008.
Bearson S.M., Bearson B.L., Brunelle B.W., Sharma V.K., Lee I.S. (2011) A mutation in the poxA gene of Salmonella enterica serovar Typhimurium alters protein production, elevates susceptibility to environmental challenges, and decreases swine colonization. Foodborne Pathog. Dis. 8(6), 725–732.
Navarre W.W., Zou S.B., Roy H., Xie J.L., Savchenko A., Singer A., Edvokimova E., Prost L.R., Kumar R., Ibba M., Fang F.C. (2010) PoxA, yjeK, and elongation factor P coordinately modulate virulence and drug resistance in Salmonella enterica. Mol. Cell. 39(2), 209–221.
Zou S.B., Hersch S.J., Roy H., Wiggers J.B., Leung A.S., Buranyi S., Xie J.L., Dare K., Ibba M., Navarre W.W. (2012) Loss of elongation factor P disrupts bacterial outer membrane integrity. J. Bacteriol. 194(2), 413–425.
Ohashi Y., Inaoka T., Kasai K., Ito Y., Okamoto S., Satsu H., Tozawa Y., Kawamura F., Ochi K. (2003) Expression profiling of translation-associated genes in sporulating Bacillus subtilis and consequence of sporulation by gene inactivation. Biosci. Biotechnol. Biochem. 67(10), 2245–2253.
Kearns D.B., Chu F., Rudner R., Losick R. (2004). Genes governing swarming in Bacillus subtilis and evidence for a phase variation mechanism controlling surface motility. Mo. lMicrobiol. 52(2), 357–369.
Iannino F., Ugalde J.E., Inon de Iannino N. (2012) Brucella abortus efp gene is required for an efficient internalization in HeLa cells. Microb. Pathog. 52(1), 31–40.
Peng W.T., Banta L.M., Charles T.C., Nester E.W. (2001) The chvH locus of Agrobacterium encodes a homologue of an elongation factor involved in protein synthesis. J. Bacteriol. 183(1) 36–45.
Sassetti C.M., Boyd D.H., Rubin E.J. (2003) Genes required for mycobacterial growth defined by high density mutagenesis. Mol. Microbiol. 48(1), 77–84.
Starosta A.L., Lassak J., Peil L., Atkinson G.C., Woolstenhulme C.J., Virumae K., Buskirk A., Tenson T., Remme J., Jung K., Wilson D.N. (2014) A conserved proline triplet in Val-tRNAsynthetase and the origin of elongation factor P. Cell. Rep. 9(2), 476–483.
Schnier J., Schnier J., Schwelberger H.G., Smit-McBride Z., Kang H.A., Hershey J.W. (1991) Translation initiation factor 5A and its hypusine modification are essential for cell viability in the yeast Saccharomyces cerevisiae. Mol. Cell. Biol. 11(6), 3105–3114.
Wohl T., Klier H., Ammer H., Lottspeich F., Magdolen V. (1993) The HYP2 gene of Saccharomyces cerevisiae is essential for aerobic growth: characterization of different isoforms of the hypusine-containing protein Hyp2p and analysis of gene disruption mutants. Mol. Gen. Genet. 241(3-4), 305–311.
Mandal A., Mandal S., Park M.H. (2014) Genome-wide analyses and functional classification of proline repeat-rich proteins: potential role of eIF5A in eukaryotic evolution. PLoS One. 9(11), e111800.
Jenkins Z.A., Haag P.G., Johansson H.E. (2001) Human eIF5A2 on chromosome 3q25-q27 is a phylogenetically conserved vertebrate variant of eukaryotic translation initiation factor 5A with tissue-specific expression. Genomics. 71(1), 101–119.
Karlyshev A.V., Nadarajah S., Abramov V.M. (2013) Draft genome sequence of Lactobacillus jensenii strain MD IIE-70(2). Genome Announc. 1(6), e01005–13.
Rahim R., Ochsner U.A., Olvera C., Graninger M., Messner P., Lam J.S., Soberon-Chavez G. (2001) Cloning and functional characterization of the Pseudomonas aeruginosa rhlC gene that encodes rhamnosyltransferase 2, an enzyme responsible for di-rhamnolipid biosynthesis. Mol. Microbiol. 40(3), 708–718.
Singh A., Vaidya B., Tanuku N.R., Pinnaka A.K. (2015) Nitrincola nitratireducens sp. nov. isolated from a haloalkaline crater lake. Syst. Appl. Microbiol. 38(8), 555–562.
Park S., Jung Y.T., Park J.M., Yoon J.H. (2014) Pseudohongiella acticola sp. nov., a novel gammaproteobacterium isolated from seawater, and emended description of the genus Pseudohongiella. Antonie van Leeuwenhoek. 106(4), 809–815.
Qi F., Motz M., Jung K., Lassak J., Frishman D. (2018) Evolutionary analysis of polyproline motifs in Escherichia coli reveals their regulatory role in translation. PLoS Comput. Biol. 14(2), e1005987.
Spirin A.S. (1999) Ribosomes. Springer
Tollerson II R., Witzky A., Ibba M. (2018) Elongation factor P is required to maintain proteome homeostasis at high growth rate. Proc. Natl. Acad. Sci. USA. 115(43), 11072–11077.
Roy H., Zou S.B., Bullwinkle T.J., Wolfe B.S., Gilreath M.S., Forsyth C.J., Navarre W.W., Ibba M. (2011) The tRNA synthetase paralog PoxA modifies elongation factor-P with (R)-beta-lysine. Nat. Chem. Biol. 7(10), 667–669.
Kaniga K., Compton M.S., Curtiss R. 3rd, Sundaram P. (1998) Molecular and functional characterization of Salmonella enterica serovar typhimurium poxA gene: effect on attenuation of virulence and protection. Infect. Immun. 66(12), 5599–5606.
Choi E., Nam D., Choi J., Park S., Lee J.S., Lee E.J. (2018) Elongation factor P controls translation of the mgtA gene encoding a Mg2+ transporter during Salmonella infection. Microbiologyopen. e680, 1–9.
Schuller A.P., Wu C.C., Dever T.E., Buskirk A.R., Green R. (2017) eIF5A functions globally in translation elongation and termination. Mol. Cell. 66(2), 194–205.e5.
Hersch S.J., Wang M., Zou S.B., Moon K.M., Foster L.J., Ibba M., Navarre W.W. (2013) Divergent protein motifs direct elongation factor P-mediated translational regulation in Salmonella enterica and Escherichia coli. MBio. 4(2), e00180–13.
Huter P., Arenz S., Bock L.V., Graf M., Frister. J.O., Heuer A., Peil L., Starosta A.L., Wohlgemuth I., Peske F., Novacek J., Berninghausen O., Grubmuller H., Tenson T., Beckmann R., Rodnina M.V., Vaiana A.C., Wilson D.N. (2017) Structural basis for polyproline-mediated ribosome stalling and rescue by the translation elongation factor EF-P. Mol. Cell. 68(3), 515–527.e6.
Mohapatra S., Choi H., Ge X., Sanyal S., Weisshaar J.C. (2017) Spatial distribution and ribosome-binding dynamics of EF-P in live Escherichia coli. MBio. 8(3), e00300–17.
Chen C., Stevens B., Kaur J., Smilansky Z., Cooperman B.S., Goldman Y.E. (2011) Allosteric vs. spontaneous exit-site (E-site) tRNA dissociation early in protein synthesis. Proc. Natl. Acad. Sci. USA. 108(41), 16980–16985.
An G., Glick B.R., Friesen J.D., Ganoza M.C. (1980) Identification and quantitation of elongation factor EF-P in Escherichia coli cell-free extracts. Can. J. Biochem. 58(11), 1312–1314.
Schmidt A., Kochanowski K., Vedelaar S., Ahrne E., Volkmer B., Callipo L., Knoops K., Bauer M., Aebersold R., Heinemann M. (2016) The quantitative and condition-dependent Escherichia coli proteome. Nat. Biotechnol. 34(1), 104–110.
Tollerson R., Witzky A., Ibba M. (2017) Elongation Factor P interactions with the ribosome are independent of pausing. MBio. 8(4), e01056–17.
Selmer M., Dunham C.M., Murphy F.V. 4th, Weixlbaumer A., Petry S., Kelley A.C., Weir J.R., Ramakrishnan V. (2006) Structure of the 70S ribosome complexed with mRNA and tRNA. Science. 313(5795), 1935–1942.
Zhou J., Korostelev A., Lancaster L., Noller H.F. (2012) Crystal structures of 70S ribosomes bound to release factors RF1, RF2 and RF3. Curr. Opin. Struct. Biol. 22(6), 733–742.
Buchan J.R., Stansfield I. (2007) Halting a cellular production line: responses to ribosomal pausing during translation. Biol. Cell. 99(9), 475–487.
Adamski F.M., Donly B.C., Tate W.P. (1993) Competition between frameshifting, termination and suppression at the frameshift site in the Escherichia coli release factor-2 mRNA. Nucl. Acids Res. 21(22), 5074–5078.
McNulty D.E., Claffee B.A., Huddleston M.J., Porter M.L., Cavnar K.M., Kane J.F. (2003) Mistranslational errors associated with the rare arginine codon CGG in Escherichia coli. Protein Expr. Purif. 27(2), 365–374.
Fu C., Parker J. (1994) A ribosomal frameshifting error during translation of the argI mRNA of Escherichia coli. Mol. Gen. Genet. 243(4), 434–441.
Gurvich O.L., Baranov P.V., Gesteland R.F., Atkins J.F. (2005) Expression levels influence ribosomal frameshifting at the tandem rare arginine codons AGG_AGG and AGA_AGA in Escherichia coli. J. Bacteriol. 187(12), 4023–4032.
Gallant J.A., Lindsley D. (1998) Ribosomes can slide over and beyond “hungry” codons, resuming protein chain elongation many nucleotides downstream. Proc. Natl. Acad. Sci. USA. 95(23), 13771–13776.
Gamper H.B., Masuda I., Frenkel-Morgenstern M., Hou Y.M. (2015) Maintenance of protein synthesis reading frame by EF-P and m(1)G37-tRNA. Nat. Commun. 6, 7226.
Alejo J.L., Blanchard S.C. (2017) Miscoding-induced stalling of substrate translocation on the bacterial ribosome. Proc. Natl. Acad. Sci. USA. 114(41), E8603–E8610.
Zou S.B., Hersch S.J., Roy H., Wiggers J.B., Leung A.S., Buranyi S., Xie J.L., Dare K., Ibba M., Navarre W.W. (2012) Loss of elongation factor P disrupts bacterial outer membrane integrity. J. Bacteriol. 194(2), 413–425.
Hanawa-Suetsugu K., Sekine S., Sakai H., Hori-Takemoto C., Terada T., Unzai S., Tame J.R., Kuramitsu S., Shirouzu M., Yokoyama S. (2004) Crystal structure of elongation factor P from Thermus thermophilus HB8. Proc. Natl. Acad. Sci. USA. 101(26), 9595–9600.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


