Молекулярная биология, 2019, T. 53, № 6, стр. 933-953
Сродство подобий: механизмы самоассоциации и компартментализации эукариотического хроматина
С. А. Григорьев a, *, Е. Ю. Попова b
a Department of Biochemistry and Molecular Biology, Penn State University College of Medicine
PA 17033 Hershey, USA
b Department of Neural and Behavioral Sciences, Penn State University College of Medicine
PA 17033 Hershey, USA
* E-mail: sag17@psu.edu
Поступила в редакцию 16.05.2019
После доработки 10.06.2019
Принята к публикации 12.06.2019
Аннотация
Упаковка хроматина в хромосомах эукариотической клетки традиционно рассматривалась как иерархический процесс, в котором цепи нуклеосом складываются в спиральные фибриллы, образующие, в свою очередь, последовательно усложняющиеся регулярные структуры. Однако результаты микроскопического анализа хроматина и изучения контактов ДНК хромосом в трехмерном пространстве клеточного ядра заставили кардинально пересмотреть иерархическую модель укладки хроматина. Согласно новым данным цепь нуклеосом обладает свободной пространственной конфигурацией при отсутствии в большинстве типов клеток регулярных спиральных фибрилл. Общая пространственная организация ДНК в ядре включает петли хроматина и контактные домены размером до нескольких миллионов пар нуклеотидов. Внутри клеточного ядра отдельные структурно-функциональные участки хроматина, обладающие сходными типами модификации гистонов и функциональными состояниями, могут сливаться вместе и образовывать хромосомные субкомпартменты, приспособленные к локальной активации или репрессии генов. Это “сродство подобий” может происходить за счет непосредственной самоассоциации цепей нуклеосом, а также при посредстве архитектурных белков, образующих олигомерные белковые “мостики” между участками хроматина и способствующих разделению жидких фаз, включающих определенные структурно-функциональные домены хроматина. Дальнейшее изучение механизмов самоассоциации и компартментализации структурно-функциональных доменов хроматина потребует объединения молекулярных, микроскопических и компьютерных подходов, способных описать трехмерную организацию генома эукариотической клетки с нуклеосомным разрешением.
ВВЕДЕНИЕ
Молекулы ДНК в эукариотических хромосомах связаны с белками, образуя цепи из повторяющихся ДНК-белковых частиц – нуклеосом. Внутри каждой нуклеосомы участок ДНК размером 145‒147 п.н. делает около 1.7 левого суперспирального оборота вокруг октамера из четырех пар гистонов: H2A, H2B, H3 и H4 [1]. Эти структуры, также называемые нуклеосомными кор-частицами, соединяются линейными участками ДНК переменного размера (от 10 до 100 п.н.), образуя цепи нуклеосом с характерной структурой типа “бусы-на-нитке”. Структура открытой нуклеосомной цепи, изученная с помощью электронной микроскопии [2, 3], хорошо известна как зигзагообразная цепь кор-частиц, соединенных относительно прямой линкерной ДНК. В метафазных хромосомах делящихся клеток нуклеосомные цепи уложены в структуры высшего порядка, обеспечивающие 10 000-кратную упаковку ДНК [4]. В хроматине интерфазных ядер упаковка также достигает весьма значительного уровня, в 400‒1000 раз большей, чем в ее полностью вытянутой конфигурации [5].
Вплоть до последнего времени упаковку хроматина рассматривали как иерархический процесс, в котором нуклеосомы (структуры первичного уровня), взаимодействуя со своими ближайшими соседями в цепи (от одной до шести соседних нуклеосом), компактизуются продольно в регулярные спиральные фибриллы диаметром 30 нм (вторичные структуры), и эти фибриллы, взаимодействуя между собой латерально, образуют структуры третичного и более высоких уровней, обеспечивая в конечном итоге примерно 10000-кратное сокращение длины ДНК в хромосомах. Латеральные взаимодействия не зависят от расстояния между нуклеосомами вдоль одной цепи. Молекулярный процесс, лежащий в основе латеральных взаимодействий между цепями нуклеосом, мы будем называть самоассоциацией хроматина.
In vitro рентгеноструктурные исследования и крио-ЭМ выявили укладку зигзагообразных нуклеосомных цепей в спиральные 30 нм фибриллы, образуемые двумя стопками нуклеосом [6‒9]. Однако существование каких-либо регулярных структур высоких порядков (больших, чем первичный) в клеточном хроматине in vivo вызывает большие сомнения. С одной стороны, ни 30 нм фибриллы, ни вообще какие-либо регулярные структуры не обнаружены в ядрах большинства эукариотических клеток при исследовании с помощью электронной микроскопии [10‒13], а также флуоресцентной микроскопии сверхвысокого разрешения [14, 15]. Эти данные согласуются с представлениями о полностью неорганизованной структуре хроматина, находящегося в состоянии так называемого жидкого полимера [16, 17]. С другой стороны, изучение контактов между соседними нуклеосомами в клетках методами Micro-C [18, 19], EMANIC [20] и RICC [21] показало наличие зигзагообразной укладки нуклеосом. Небольшие кластеры, образованные контактами нескольких соседних нуклеосом, наблюдали с помощью флуоресцентной микроскопии сверхвысокого разрешения [14, 22, 23]. Складки или петли, свидетельствующие о латеральной самоассоциации цепей нуклеосом, обнаружены в интерфазном хроматине с помощью электронно-микроскопической томографии [12], в метафазных хромосомах методом EMANIC [20] и крио-ЭМ [24]. Эти данные показывают, что отдельные участки хроматина, несмотря на отсутствие регулярной организации, могут включать в себя элементы первичной, вторичной и третичной структуры.
Существенные отличия в механизмах укладки нуклеосомной цепи in vitro и in vivo обусловлены в том числе тем, что в ядрах различных интерфазных клеток цепи нуклеосом связаны с различными внутриядерными структурами, участвующими в укладке и компартментализации хромосом. На макроуровне трехмерная организация интерфазного хроматина определяется ДНК-связывающим белком CTCF и белком когезином, образующим кольцевые структуры, соединяющие основания петель ДНК [25]. Контакты и петли хроматина совместно приводят к образованию топологически-ассоциированных доменов (ТАДов) размером до нескольких миллионов пар нуклеотидов. Другие ядерные структуры, обеспечивающие компактность хроматина в клеточном ядре, ‒ полимерные рибонуклеопротеиновые фибриллы [26] и ядерная ламина [27]. Наконец, существенными факторами считаются архитектурные белки хроматина, которые за счет способности к самоассоциации при связывании с определенными доменами хроматина вызывают динамическую конденсацию и способствуют фазовому разделению хромосомных доменов внутри клеточного ядра [28, 29].
В этом обзоре мы рассмотрим механизмы самоассоциации хроматина, определяемые как непосредственным взаимодействием между цепями нуклеосом, так и архитектурными белками. Наша исследовательская группа, созданная И.А. Крашенинниковым на кафедре молекулярной биологии Биологического факультета МГУ, была одним из пионеров исследований в этой области [30‒33]. За последнее время развитие новых методов определения конформации хромосом, микроскопии, и биохимического анализа белковых комплексов привело к значительному прогрессу в изучении компартментализации и фазового состояния хроматина. Понимание механизмов самоассоциации хроматина, определяющего ключевые биологические процессы, необходимо для обнаружения новых классов генетических регуляторов и синтеза фармакологически активных соединений, способных изменять или исправлять генетические или эпигенетические дефекты развития и функционирования клеток и тканей эукариотического организма.
САМОАССОЦИАЦИЯ МОНОНУКЛЕОСОМ В РАСТВОРЕ
Нуклеосомные кор-частицы, содержащие около 147 п.н. ДНК, могут быть выделены из природного хроматина или реконструированы из ДНК и октамеров гистонов. В отличие от свободной ДНК, кор-частицы легко ассоциируют в растворах с физиологической ионной силой, образуя кристаллические или псевдокристаллические комплексы различного типа. Впервые такие комплексы детально описали J. Dubochet и M. Noll [34], которым с помощью ЭМ удалось показать укладку нуклеосомных дисков в стопки, образующие арки и спирали, с преобладанием взаимодействий между плоскими поверхностями нуклеосомных дисков. Впоследствии изучение самоассоциированных структур, образуемых стопками кор-нуклеосом, с помощью крио-ЭМ и дифракции рентгеновских лучей выявило многообразие типов их укладки [35, 36], свидетельствующее о множественности механизмов электростатического взаимодействия между нуклеосомами в растворе. В последнее время развитие методов крио-ЭМ позволило изучать взаимодействие индивидуальных пар кор-нуклеосом в растворе. Обнаружено, что пары нуклеосом образуют множественные структуры, связанные как с разными типами относительно устойчивых взаимодействий, так и с динамическими конформационными переходами отдельных нуклеосом [37].
Важная информация о разнообразии возможных контактов между нуклеосомами получена также в ходе рентгеноструктурных исследований кристаллов кор-нуклеосом. Кристаллография, в отличие от ЭМ, требует минимальной гетерогенности при укладке молекул в кристалле. Однако сравнение разных кристаллов может выявить существенные различия между положением молекул в кристаллической решетке. Так, в первой рентгеновской структуре нуклеосом высокого разрешения [38] была обнаружена кристаллическая упаковка кор-нуклеосом с параллельной ориентацией диадных осей, которая соответствовала вероятной упаковке нуклеосом в фибриллах хроматина. Напротив, в более поздней структуре такого же разрешения (но отличающейся использованием природных, а не рекомбинантных гистонов) выявлена антипараллельная упаковка нуклеосом, которая не вписывается ни в одну из возможных моделей фибрилл хроматина [39]. Последующие определение рентгеновских структур нуклеосом, содержащих различные гистоны, а также негистоновые белки, подтвердило множественные формы упаковки нуклеосом при сохранении консервативной структуры ДНК и гистонов внутри каждой нуклеосомы [40‒42]. Хотя отдельные мононуклеосомы не являются полностью адекватной моделью хроматина, например в силу отсутствия линкерной ДНК, их способность к образованию ряда различных форм упаковки может отражать структурное многообразие природного хроматина, важное для понимания межнуклеосомных взаимодействий в более сложных, в том числе живых, системах.
САМОАССОЦИАЦИЯ ПРИРОДНОГО ХРОМАТИНА
Более адекватными моделями для изучения строения хроматина являются природные цепи нуклеосом, сохраняющие природные гистоны и их модификации, а также цепи нуклеосом, реконструированные из ДНК и гистонов. Как природные, так и реконструированные цепи нуклеосом способны к кооперативной самоассоциации при повышении концентрации одно- и двухвалентных катионов в растворе до значений, близких к физиологическим. В ходе изучения конденсации хроматина с помощью ЭМ J. Widom показал, что при увеличении концентрации катионов цепи нуклеосом сначала складываются в фибриллы диаметром 30 нм, затем ассоциируют в структуры большего диаметра (45‒50 нм) и в итоге образуют агрегаты, выпадая из раствора [43]. Впоследствии D. Сlark и Т. Kimura теоретически проанализировали механизм компактизации и самоассоциации цепей хроматина в растворе, представив фазовую диаграмму структурных переходов хроматина в зависимости от присутствия одно- и двухвалентных катионов и гистона Н1 [44]. Результаты этого анализа указывали на преимущественно электростатический механизм конденсации и самоассоциации хроматина, согласующийся с теорией конденсации полиэлектролитов [45]. Проведенное в нашей лаборатории сравнение хроматина, выделенного из активно пролиферирующих и зрелых дифференцированных клеток, выявило существенные отличия в степени его самоассоциации. При одинаковой концентрации соли хроматин из делящихся клеток представлял собой открытые зигзагообразные цепи нуклеосом, в то время как хроматин из зрелых дифференцированных клеток обладал сильно конденсированной структурой [46]. Анализ такого хроматина методом крио-ЭМ позволил впервые увидеть конденсированные структуры, в которых отсутствовали регулярные фибриллы диаметром 30 нм, но были заметны открытые поверхности нуклеосом на периферии конденсированного хроматина (рис. 1). На основании этих данных впоследствии мы предложили механизм конденсации хроматина за счет латеральной самоассоциации и интердигитации (сцепления пальцев) между дисками нуклеосом [47, 48], принципиально отличный от иерархической структуры, в которой 30 нм фибриллы укладываются в третичные структуры, которые, в свою очередь, дают более высокие уровни организации.
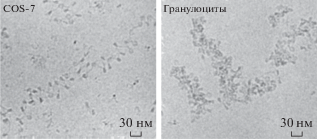
Рис. 1.
Степень конденсации нуклеосомных цепей различается в хроматине из делящихся и дифференцированных клеток. Крио-ЭМ хроматина из пролиферирующих клеток COS-7 (слева) выявляет нуклеосомы типа “бусы на нитке” с открытой линкерной ДНК. Хроматин, выделенный из терминально дифференцированных гранулоцитов, содержит плотно упакованные нуклеосомы в центре фибриллы и отдельные диски нуклеосом на поверхности фибриллы (справа). Рисунок воспроизведен и модифицирован из статьи С.А. Григорьева и соавт. [46], опубликованной в The Journal of Biological Chemistry © American Society for Biochemistry and Molecular Biology.
Дальнейшие ЭМ исследования нуклеосомных контактов, образуемых в метафазных хромосомах при частичной фиксации формальдегидом (EMANIC) [20], выявили структуры, образованные ассоциированными зигзагообразными цепями нуклеосом и принципиально отличающиеся от компактных 30 нм фибрилл в растворе, а также в некоторых типах дифференцированных клеток. На основании этих экспериментов и компьютерного моделирования нами была предложена модель трех основных типов укладки нуклеосомной цепи. В этой модели цепи нуклеосом имеют зигзагообразную конформацию во всех состояниях хроматина, но подвергаются продольной укладке в 30 нм фибриллы только в некоторых типах дифференцированных клеток. В конденсированных метафазных хромосомах преобладает латеральная самоассоциация относительно открытых нуклеосомных цепей (рис. 2). Проведенное совсем недавно наблюдение внутренней структуры хромосом с помощью крио-ЭМ подтвердило возможность латеральной интердигитации нуклеосомных цепей в конденсированных метафазных хромосомах [24].
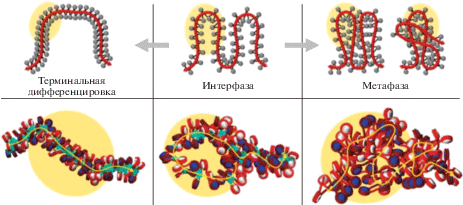
Рис. 2.
Три основных структурных состояния хроматина. Схематическое изображение (вверху) и компьютерное моделирование (внизу), иллюстрирующие три основных типа укладки нуклеосомной цепи: 30 нм фибрилла, наблюдаемая в определенных дифференцированных клетках, открытые нуклеосомные цепи и петли в интерфазных ядрах пролиферирующих клеток, конденсированные нуклеосомные петли, образующиеся в метафазной хромосоме. Рисунок воспроизведен из статьи С.А. Григорьева и соавт. [20], опубликованной в Proc. Natl. Acad. Sci. USA © United States National Academy of Sciences.
Таким образом, анализ как нативного хроматина, так и метафазных хромосом позволяет предположить, что латеральная самоассоциация нуклеосомных цепей, а не продольное и иерархическое укладывание спиральных фибрилл, является наиболее общим механизмом конденсации эукариотического хроматина. В дальнейшем нам предстоит установить, присутствуют ли аналогичные третичные структуры в ядрах интерфазных клеток и каков молекулярный механизм взаимодействия нуклеосом в этих структурах.
МЕХАНИЗМЫ САМОАССОЦИАЦИИ РЕКОНСТРУИРОВАННОГО ХРОМАТИНА
Поскольку значительный вклад в укладку цепей природных нуклеосом вносят разнообразные одно-, двух- и поливалентные катионы, а также архитектурные белки хроматина, существенно варьирующие в клетках различных тканей и организмов, основной моделью хроматина для изучения общего механизма упаковки и самоассоциации стали реконструированные системы, в которых нуклеосомы собираются из природных или рекомбинантных гистонов и ДНК, обладающей повышенным сродством к октамерам гистонов, например ДНК гена 5S рРНК морского ежа [49] или искусственно синтезированной ДНК клона 601 [50]. Реконструированные цепи нуклеосом сделали возможным изучение хроматина в биохимически определенной системе с охарактеризованными размерами ДНК, источником гистонов (нативных или рекомбинантных), установленным числом нуклеосом и точным расстоянием между соседними нуклеосомами.
Используя ДНК гена 5S рРНК морского ежа, J.C. Hansen и его группа разработали простую и надежную систему анализа самоассоциации в растворе [51]. В этой системе цепи нуклеосом (наиболее часто из 12 тандемно расположенных нуклеосом) при добавлении катионов Mg2+ претерпевают как линейную компактизацию, так и обратимую самоассоциацию. В самоассоциированном состоянии нуклеосомы осаждают с помощью низкоскоростного центрифугирования (например, в течение 10 мин при 14 000 g), а количественный анализ ДНК в осадке и в растворе позволяет определить точку полуосаждения, т.е. такую концентрацию Mg2+ (~3 мM), при которой 50% ДНК переходит в осадок. Следует отметить, что величина полуосаждения Mg2+ является устойчивой характеристикой базового хроматина, состоящего из ДНК и гистонов. Используя ДНК клона 601, которая задает межнуклеосомные расстояния с точностью до одного нуклеотида, наша группа выявила существенную зависимость вторичной, но не третичной структуры хроматина от расстояния между нуклеосомами [52]. Величина полуосаждения цепей из линейных и кольцевых нуклеосом также оказалась практически одинаковой, показывая, что свободные концевые нуклеосомы, отсутствующие в хромосомах живой клетки, не определяют самоассоциацию нуклеосомных цепей [53].
Использование реконструированных нуклеосомных цепей с различными делециями или модификациями аминокислот в гистонах сделало возможным изучение специфических факторов, изменяющих конформационную динамику хроматина (см. обзоры [54, 55]). Так установлено, что положительно заряженные N-концевые домены всех четырех коровых гистонов, а также С-концевые домены гистонов Н2А и Н2В, которые не участвуют в структуре нуклеосомного кора [1] и являются высокомобильными и деструктурированными в сильно концентрированном хроматине [56], вносят существенный вклад как во вторичную укладку хроматина [54, 57], так и в самоассоциацию нуклеосомных цепей [58, 59], образуя внутри- и межнуклеосомные контакты [55] (см. рис. 3). При этом удаление N-концевых доменов двух из четырех кор-гистонов влияет на компактизацию нуклеосомных цепей (вторичный уровень упаковки) сильнее, чем на самоассоциацию (третичный уровень), свидетельствуя о том, что самоассоциация хроматина не зависит от его компактизации [60]. Этот вывод подтвержден в более поздних исследованиях с использованием ДНК клона 601 [61].
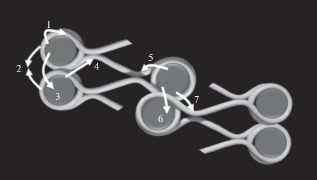
Рис. 3.
Множественные взаимодействия N-концевых доменов гистонов с нуклеосомами могут участвовать в конденсации и самоассоциации нуклеосомных цепей. Схематическое изображение, составленное по результатам компьютерного моделирования [20, 67, 68], показывает возможные взаимодействия между N-доменом и ДНК внутри той же нуклеосомы (1), между двумя N-доменами из двух разных нуклеосом в той же цепи (2), между N-доменом и поверхностью другой нуклеосомы в той же цепи (3), между N-доменом и линкерной ДНК другой нуклеосомы в той же цепи (4), между N-доменом и линкерной ДНК в той же нуклеосоме (5), между N-доменом и поверхностью нуклеосомы в другой цепи (6), между N-доменом и линкерной ДНК нуклеосомы в другой цепи (7).
Первый молекулярный механизм межнуклео-сомных контактов с участием N-концевого домена гистона Н4 и отрицательно заряженной поверхности нуклеосомного диска, образованной так называемыми “кислотными” участками гистонов Н2А и Н2В, обнаружили в ходе рентгеноструктурных исследований [38] и экспериментально подтвердили с помощью химической сшивки и ЭМ, показавших роль этих взаимодействий в поддержании структуры 30 нм фибриллы (вторичный уровень компактизации) [6]. Однако последующие эксперименты с химическими сшивками показали, что N-концевые домены гистонов Н3 и Н4 способны взаимодействовать как внутри одной цепи нуклеосом, так и между цепями, участвуя и во вторичной укладке, и в третичной самоассоциации [62‒64]. В то же время, эксперименты с изменением молекулярной поверхности нуклеосомного диска с помощью замены природных вариантов гистона Н2А в нуклеосоме [65] или мутациями в кислотном участке гистона Н2А [66] показали, что поверхность нуклеосомного диска также важна для межнуклеосомных взаимодействий, в частности как акцептора N‑концевых доменов, и ее изменение может усиливать транс-взаимодействия, приводящие к самоассоциации цепей нуклеосом. Множественный характер взаимодействия N-концевых доменов гистонов внутри цепей нуклеосом подтверждается также данными компьютерного моделирования [67‒69]. На схеме, составленной на основе компьютерных моделей (рис. 3), типы взаимодействия 2, 3 и 4 вызывают продольную конденсацию внутри данной цепи нуклеосом, а взаимодействия 6 и 7 способствуют латеральной самоассоциации между различными нуклеосомными цепями.
Аминокислоты N-концевых доменов гистонов подвергаются множественным посттрансляционным модификациям. Эти модификации влияют на структуру хроматина как непосредственно, изменяя заряд гистонов, так и путем рекрутирования негистоновых архитектурных белков, участвующих в конденсации хроматина [70, 71]. В реконструированной системе наиболее изученным оказалось влияние ацетилирования гистонов. Так J.C. Hansen и его группа показали, что ацетилирования 12 аминокислотных остатков в гистоновом октамере достаточно для полного предотвращения самоассоциации в присутствии Mg2+ [72]. Ацетилирование даже одного, но ключевого, лизина-16 в N-концевом домене гистона Н4 способно ослаблять как линейную компактизацию, так и самоассоциацию цепей нуклеосом [73]. Ацетилирование гистона Н4 также негативно влияло на самоассоциацию, индуцированную линкерным гистоном Н1 [63]. Путем систематического изменения заряда аминокислот в N-концевом домене гистона Н4 и в отрицательно заряженном участке гистона Н2А показано, что механизм самоассоциации нуклеосомных цепей в основном согласуется с неспецифическим электростатическим механизмом, который, в отличие от вторичной компактизации, не требует непосредственных контактов между N-концевым доменом гистона Н4 и кислотным участком гистонов Н2А/Н2В [74].
Изучение нуклеосомных цепей, реконструированных с ДНК клона 601, показало, что самоассоциация нуклеосомных комплексов в присутствии ионов Mg2+ приводит к образованию полимерных глобул диаметром 50–1000 нм, внутри которых в качестве структурных интермедиатов не выявляются элементы вторичной структуры, в частности 30 нм фибриллы. При этом связывание линкерного гистона Н1 помогает образованию самоассоциированных глобул [61]. Предполагается, что внутри самоассоциированных частиц нуклеосомы имеют полностью дезорганизованную упаковку, как и цепи нуклеосом в метафазных хромосомах. Сравнительно недавно показано, что С-концевой домен гистона Н1 в комплексе с ДНК способен образовывать жидкие коацерватные капельки, весьма похожие на глобулы самоассоциированного хроматина [75]. Аналогичные Mg2+-зависимые глобулы изучали в одной из самых последних работ, где динамику хроматина внутри глобул наблюдали с помощью флуоресцентно меченных гистонов и подтвердили частичное ингибирование самоассоциации хроматина при ацетилировании гистонов [76]. Сделан вывод о сходстве самоассоциированных глобул и так называемых “жидких капелек”, формирующихся в процессе разделения жидких фаз хроматина внутри клеточного ядра [28, 29], которое мы обсудим ниже. Следует отметить, что электростатический механизм образования глобул самоассоциированного хроматина в растворе скорее напоминает процесс коацервации заряженных биологических полимеров, в свое время интенсивно изучавшийся в лаборатории А.И. Опарина и Т.Н. Евреиновой на кафедре биохимии растений МГУ [77], чем гидрофобные взаимодействия, образующие жидкие фазы в клетке. В настоящее время открытым остается вопрос о соответствии механизма самоассоциации хроматина в растворе фазовому разделению и образованию “жидких капелек” в клеточном ядре.
САМОАССОЦИАЦИЯ ТРАНСКРИПЦИОННО-РЕПРЕССИРОВАННОГО ХРОМАТИНА
Отдельные участки эукариотического генома могут резко отличаться как по уровню транскрипционной активности, так и по наличию определенных маркеров, в первую очередь, посттрансляционных модификаций гистонов, определяющих структурно-функциональные состояния доменов хроматина [78]. Впервые существенные структурные отличия между двумя классами хроматина, которые различаются уровнем транскрипции генов, обнаружили H. Weintraub и M. Groudine в ходе изучения доступности ДНК в ядрах для ДНКазы I. Оказалось, что протяженные домены транскрибируемого хроматина отличаются большей скоростью деградации под действием ДНКазы I [79]. Участки хроматина, чувствительного к ДНКазе I, могут иметь протяженность более 100000 п.н. и включать как активно транскрибируемые гены, так и нетранскрибируемые участки генома. Эти эксперименты позволили предположить, что структура активного хроматина имеет более открытую конформацию, и поставили вопрос о свойствах нуклеосом и/или архитектурных белков хроматина, которые отвечают за поддержание этих структурных различий. В последующих биохимических исследованиях H. Weintraub показал, что транскрипционно-активный и репрессированный хроматин сохраняют структурные различия в растворе; при этом репрессированный хроматин образует так называемые А‑частицы (рис. 4), обладающие повышенной способностью к самоассоциации, наблюдаемой при электрофорезе [80]. На основании этих данных выдвинута гипотеза о том, что молекулярные мостики между цепями нуклеосом, вызывающие самоассоциацию, могут приводить к конденсации хроматина и ингибировать его доступность для факторов транскрипции [81].
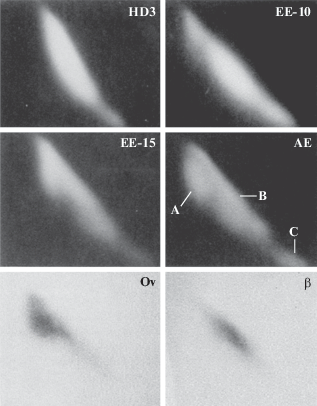
Рис. 4.
Разделение активного и репрессированного хроматина методом двумерного электрофореза. Вверху: четыре агарозных геля, окрашенных бромистым этидием, показывают отсутствие А-частиц в хроматине эритробластов (HD3) и эритроцитов 10-дневных эмбрионов курицы (ЕЕ-10), но образование А-частиц в хроматине эритроцитов 15-дневных эмбрионов (ЕЕ-15) и взрослой курицы (АЕ). Две нижние панели ‒ радиоавтографы геля АЕ после блотинга по Саузерну, гибридизации с репрессированным геном овальбумина (Ov) и транскрипционно активным геном β-глобина (β). Рисунок воспроизведен из статьи С.А. Григорьева и соавт. [33], опубликованной в Experimental Cell Research © Elsevier.
С помощью гибридизационного картирования J.O. Thomas и ее коллеги изучили соответствие между чувствительными к ДНКазе I доменами и способностью к самоассоциации. Показано, что как активно транскрибируемые “взрослые” бета-глобиновые гены, так и молчащие, но чувствительные к ДНКазе I “эмбриональные” гены устойчивы к самоассоциации. Участки генома с повышенной способностью к самоассоциации на краях бета-глобинового кластера обладают повышенной устойчивостью к ДНКазе I [82, 83]. Хроматин этих двух различных типов отделен друг от друга пограничными элементами ДНК, или инсуляторами, способными как препятствовать распространению активного хроматина, так и ингибировать взаимодействие между энхансерами и промоторами транскрибируемых генов [84].
Сохранение структурных отличий разных фракций нативного хроматина в растворе позволило впервые изучить специфические модификации гистонов, негистоновые белки и другие компоненты хроматина, связанные с активным и репрессированным состоянием. Так, активный хроматин, устойчивый к самоассоциации и осаждению [85], обогащен гиперацетилированными гистонами H4, H2A.Z и H2B, а также убиквитинированными гистонами H2A и H2B. В нашей группе на кафедре молекулярной биологии МГУ показано, что репрессированный хроматин А-частиц отличается повышенной устойчивостью линкерной ДНК к нуклеазам и может сохранять линкерные гистоны, обуславливающие избирательную самоассоциацию [30, 32]. Впоследствии из А-частиц нами был выделен негистоновый белок MENT ‒ первый тканеспецифический негистоновый белок, способный индуцировать конденсацию хроматина [31, 33], механизм работы которого мы рассмотрим ниже.
САМОАССОЦИАЦИЯ И СЕГРЕГАЦИЯ ЭУХРОМАТИНА И ГЕТЕРОХРОМАТИНА В ХОДЕ КЛЕТОЧНОЙ ДИФФЕРЕНЦИРОВКИ
На клеточном уровне основные работы по взаимосвязи между конденсацией и компартментализацией хроматина проведены в рамках изучения двух морфологически и функционально различных типов хроматина: эухроматина и гетерохроматина. E. Heitz, первым описавший морфологию относительно развернутого эухроматина (настоящий хроматин) и конденсированного гетерохроматина (другой хроматин) в 1928 году [86], предполагал, что гетерохроматин соответствует участкам хромосом, которые не полностью разворачиваются при переходе от метафазы к интерфазе. Участки хромосом, располагающиеся вблизи центромер и теломер, находятся в состоянии гетерохроматина независимо от типов тканей и степени клеточной дифференцировки и называются конститутивным гетерохроматином [87, 88]. Для микроскопического изучения пространственного расположения конститутивного гетерохроматина удобными ориентирами служат так называемые хромоцентры ‒ крупные кластеры хроматина, содержащие слившиеся вместе перицентромерные участки нескольких хромосом [89]. Хромоцентры выявляются в большинстве клеток высших эукариот с помощью иммунофлуоресценции с антителами против гистона Н3, триметилированного по лизину-9 (Н3К9ме3) [90]. Кроме того, в клетках мыши конститутивный гетерохроматин содержит АТ-богатые сателлитные ДНК и легко выявляется с помощью АТ-специфичных флуоресцентных ДНК-интеркаляторов, таких как Hoechst 33258 (см. рис. 5д, е).
Другой вид гетерохроматина, факультативный, образуется только в определенных типах клеток [88]. Примером факультативного гетерохроматина служит хроматин неактивной Х-хромосомы в соматических клетках млекопитающих женского пола [91]. Факультативный гетерохроматин наиболее представлен в ядрах терминально дифференцированных клеток, таких как клетки крови, печени, тимуса, сетчатки глаза (рис. 5а‒в) и в других тканях, где он занимает большую часть пространства клеточного ядра [48, 92]. Распространение факультативного гетерохроматина сопровождается (в разной степени в разных клетках) глобальным изменением уровня и расположения посттрансляционных модификаций гистонов [48, 93‒95], а также структуры хроматина – увеличением длины межнуклеосомных линкеров и уровня линкерного гистона [94, 96, 97], а также появлением 30 нм фибрилл хроматина, наблюдаемых in situ [98, 99].
Рис. 5.
Компартментализация гетерохроматина в пролиферирующих и терминально дифференцированных клетках мыши. а–в ‒ Электронная микроскопия срезов палочек сетчатки, выделенных на первом (а), 15-м (б) и 56-м (в) днях постэмбрионального развития мыши. Е – эухроматин; Н – гетерохроматин. г ‒ Флуоресцентная микроскопия срезов палочек сетчатки взрослой мыши с тройным нокаутом генов гистона Н1с, Н1е и Н1о, окрашенных антителами, специфическими к факультативному гетерохроматину (Н3К9mе2, красный), активному хроматину (H3K4me2, зеленый), и интеркалятором Hoechst 33258, связывающимся с ДНК перицентромерного гетерохроматина (синий). Панели а‒г воспроизведены и модифицированы из статьи Е.Ю. Поповой, С.А. Григорьева и соавт. [94], опубликованной в The Journal of Biological Chemistry © American Society for Biochemistry and Molecular Biology. д‒з ‒ Флуоресцентная микроскопия клеток культуры NIH/3T3, окрашенных антителами к гистону Н3К9mе3, специфичными к конститутивному гетерохроматину (д ‒ красный), интеркалятором Hoechst 33258 (е, ж, з ‒ синий), а также антителами к гистону H4K12ас, специфичными к активному хроматину (ж ‒ зеленый), и антителами к гистону Н3К9mе2, специфичными к факультативному гетерохроматину (з ‒ красный). и, к ‒ Флуоресцентная микроскопия лимфоцитов селезенки мыши, окрашенных антителами к гистону H4K12ас, специфичными к активному хроматину (и ‒ зеленый), антителами к гистону Н3К9mе2, специфичными к факультативному гетерохроматину (к ‒ красный), и интеркалятором Hoechst 33258 (и, к ‒ синий). Панели д ‒ к воспроизведены и модифицированы из статьи С.А. Григорьева и соавт. [100], опубликованной в Journal of Cell Science © The Company of Biologists.
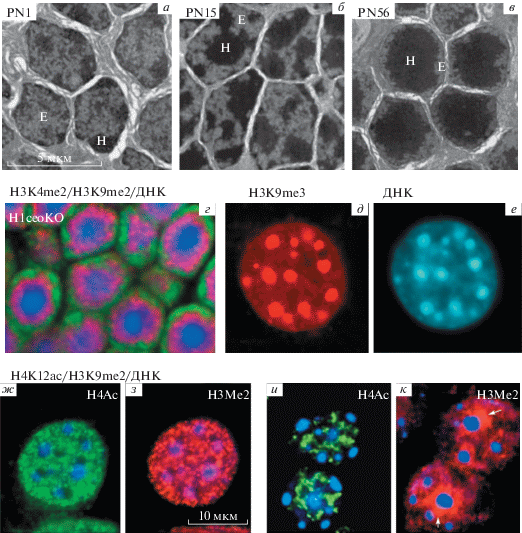
Многие гены, инактивированные в процессе терминальной дифференцировки, перемещаются внутри ядра к перицентромерному гетерохроматину или ядерной периферии [101‒103]. Ассоциация репрессированного гена с гетерохроматином сопровождается уменьшением общего уровня ацетилирования гистонов и глобальной конденсации хроматина [102]. Анализ хромосомного локуса, содержащего ген Dntt, репрессируемый в ходе терминальной дифференцировки Т-лимфоцитов мыши, выявил связь между общим падением уровня ацетилирования на протяжении всего домена, увеличением уровня диметилирования гистона Н3 по лизину-9 (Н3К9mе2) и перицентромерной транслокации всего протяженного хроматинового домена [104]. A.S. Belmont и его группа наглядно показали самоассоциацию и разделение функционально различных участков хроматина на генном уровне [105]. Они сконструировали искусственные хромосомные локусы, содержащие повторяющиеся сегменты генов человека или мыши, разделенные участками ДНК бактериального вектора, и поместили их в эмбриональные стволовые клетки мыши. В этой системе эукариотические сегменты приобрели маркеры эухроматина и сформировали отдельные территории на поверхности компактного локуса. Напротив, бактериальные сегменты приобрели маркеры гетерохроматина и сформировали компактные зоны внутри общего локуса.
При изучении конденсации хроматина в зрелых клетках крови мы показали, что эта конденсация связана с массовым изменением компартментализации и сегрегацией основных функциональных состояний хроматина [48]. Например, в ядре дифференцированных лимфоцитов хроматин, связанный с модификацией Н3К9mе2 гистона Н3, характерной для факультативного гетерохроматина, образует так называемые апоцентромерные зоны (рис. 5к, стрелка) на поверхности перицентромерного гетерохроматина, в то время как хроматин, связанный с активаторной модификацией (ацетилированием гистона Н4), образует отдельные кластеры, находящиеся на удалении от хромоцентров (рис. 5и, к). Такая внутриядерная локализация различных структурно-функциональных состояний хроматина резко контрастирует с равномерным распределением этих же модификаций в ядре активно пролиферирующих клеток NIH/3T3 (рис. 5ж, з) [100]. В дальнейшем мы выявили сходные глобальные переходы структуры хроматина, маркированного Н3К9mе2, при терминальной дифференцировке других типов клеток крови – эритроидных [93] и миелоидных [106, 107].
Наиболее выраженное образование сегрегированных зон хроматина проявляется в ядрах клеток палочек сетчатки взрослой мыши, в которых конденсированный гетерохроматин сливается в один общий кластер в середине клеточного ядра (рис. 5г), что, согласно гипотезе И. Соловей и ее сотрудников, может быть связано с адаптацией к ночному видению [108]. В этих клетках зоны факультативного гетерохроматина (Н3К9mе2) и репрессированного хроматина (Н3К27mе3) располагаются на поверхности перицентромерного кластера, а на самой периферии клеточного ядра находится транскрипционно-активный хроматин, маркированный гистоном Н3 метилированным по лизину-4 (рис. 5г) При этом линкерный гистон Н1 увеличивает степень конденсации хроматина, а частичный нокаут генов Н1 позволяет выявить концентрическое расположение зон гетерохроматина [94]. Таким образом, в ядрах дифференцированных клеток наблюдается ярко выраженная компартментализация хроматина по принципу сродства подобий, где одинаково модифицированные участки хроматина соединяются, образуя функционально и пространственно разделeнные компартменты.
БЕЛКОВЫЕ “МОСТИКИ” И ДРУГИЕ ФАКТОРЫ, ИНДУЦИРУЮЩИЕ САМОАССОЦИАЦИЮ И КОМПАРТМЕНТАЛИЗАЦИЮ ХРОМАТИНА
Какие же архитектурные факторы вызывают слияние доменов репрессированного гетерохроматина и их компартментализацию в ядре эукариотической клетки? В составе гетерохроматина наиболее изучены белки группы НР1 (heterochromatin protein 1) [89], в структуре которых присутствует хромодомен, способный связываться с N‑концевым доменом гистона Н3, содержащим ди- и триметилированный лизин-9. НР1 также обладает ДНК-связывающей активностью, независимой от метилирования гистонов. Вариант НР1 из клеток позвоночных, HP1α, обладает наибольшим сродством к ДНК и хроматину [109], конкурируя при этом с гистоном Н1 за линкерную ДНК [110]. В дополнение к связыванию с нуклеосомами белки НР1 способны олигомеризоваться, что опосредовано их так называемым хромотеневым доменом [111, 112], и образовывать белковые мостики, усиливающие ассоциацию нуклеосомных цепей in vitro [113, 114]. Недавно с помощью крио-ЭМ показано, что димеры НР1 способны образовывать белковые мостики между открытыми поверхностями нуклеосомных дисков [115]. In vivo роль НР1 как фактора, вызывающего образование гетерохроматина, показана в экспериментах, в которых искусственная ассоциация белка НР1 с хромосомным локусом индуцировала конденсацию хроматина и появление маркеров гетерохроматина [116], а также приводила к установлению контактов между удаленными участками хромосом [113].
Вызывает удивление, что резкое увеличение степени конденсации хроматина в ходе терминальной дифференцировки может сопровождается столь же резким падением уровня НР1 [93, 94, 117, 118]. При этом оставшийся НР1 взаимодействует с транскрипционно-активными генами, а не с гетерохроматином [119]. Таким образом, снижение экспрессии и уровня белка НР1 противоречит его возможной роли в структурной организации хроматина в дифференцированных клетках, указывая на участие каких-то других факторов в процессе конденсации факультативного гетерохроматина.
Один из важных факторов, вызывающих конденсацию хроматина, ‒ линкерный гистон Н1. Несколько вариантов этого гистона, включая Н5, характерный для ядерных эритроцитов птиц и других позвоночных (за исключением млекопитающих, эритроциты которых теряют ядро при созревании), связаны с терминальной дифференцировкой. Линкерные гистоны могут связываться с линкерной ДНК и при связывании (одна молекула Н1 на нуклеосому) стабилизировать 30 нм фибриллу хроматина [120, 121]. Уровень гистона Н1 сильно варьирует в клетках разного типа [122], при этом гистон Н1 в соотношении менее одной молекулы на нуклеосому дестабилизирует 30 нм фибриллу, но усиливает образование нерегулярных петлевых структур [20]. В ядрах некоторых дифференцированных клеток, таких как эритроциты птиц [97] и палочки сетчатки мыши [94], уровень гистона Н1 больше, чем одна молекула на нуклеосому; именно для этих двух типов клеток характерна глобальная конденсация хроматина и образование 30 нм фибрилл, наблюдаемых in situ [98, 99]. Интересно, что при делеции двух из шести вариантов гистона Н1 происходит частичная деконденсация хроматина и увеличение связывания специфичных для факультативного хроматина антител к гистону Н3К9mе2 [94]. Однако в ряде других дифференцированных клеток с высоким уровнем конденсации хроматина не обнаружено увеличения уровня линкерного гистона [93, 100, 106, 107], что свидетельствует о том, что такое увеличение не является универсальным фактором, вызывающим конденсацию факультативного гетерохроматина.
При анализе белков А-частиц, соответствующих репрессированному хроматину из эритроцитов курицы (рис. 4), нашей группой обнаружен негистоновый белок MENT (Myeloid and Erythroid Nuclear Termination stage-specific protein), экспрессия которого совпадает с терминальной дифференцировкой и образованием конденсированного хроматина [31, 33]. Впоследствии этот белок нашли также в хроматине других клеток крови курицы – лимфоцитах и гранулоцитах [123]. Клонирование и анализ последовательности гена, кодирующего MENT, выявили его принадлежность к семейству серпинов (SERPIN, serine protease inhibitor). MENT, в отличие от других серпинов, обладает уникальным ДНК-связывающим доменом [46]. Биохимическое и рентгеноструктурное изучение MENT позволило в дальнейшем открыть новый механизм конденсации хроматина, при котором связывание белка с нуклеосомами индуцировало переход так называемого RСL-домена из α-спиральной конформации в β-структуру, что приводило к олигомеризации белка MENT и образованию белковых мостиков, вызывающих самоассоциацию нуклеосомных цепей [124, 125] (рис. 6). Параллельные исследования показали, что MENT предпочтительно связан с гетерохроматином, маркированным модификацией гистона Н3К9mе2. Транскрипционно-активные гены β-глобина курицы защищены при этом от распространения белка MENT инсуляторными элементами на границе β‑глобинового кластера [117]. Интересно, что геном курицы содержит наиболее сильные инсуляторы, широко используемые в векторах, предназначенных для генной терапии человека [126]. Возможно, что высокая эффективность этих инсуляторов возникла в ходе эволюции генома птиц как адаптация к высокому уровню конденсации хроматина, вызванному белком MENT. В геноме млекопитающих серпин, полностью гомологичный белку MENT и обладающий подобным сродством к ДНК, не обнаружен, несмотря на то что в составе конденсированного хроматина млекопитающих присутствуют другие близкородственные серпины [106, 107, 127]. Известен также ряд других, более широко распространенных архитектурных белков, таких как PRC1, MeCP2 и L3MBTL1, способных образовывать белковые мостики и стабилизировать хроматин в конденсированном состоянии (см. обзор [71]). В частности, показано накопление белка MeCP2 и его непосредственное участие в процессе конденсации гетерохроматина при дифференцировке мышечных клеток [128, 129].
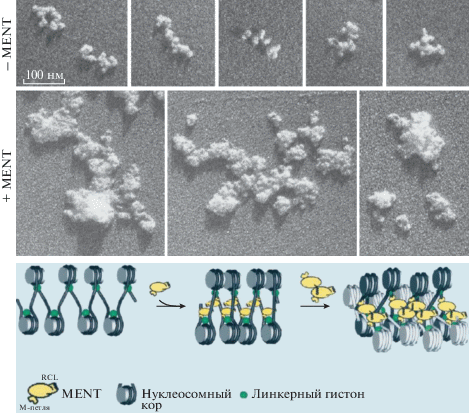
Рис. 6.
Негистоновый белок MENT вызывает самоассоциацию нуклеосомных цепей. Вверху: электронная микроскопия реконструированных цепей нуклеосом до (– MENT) и после (+ MENT) восстановления с белком MENT. Внизу ‒ модель механизма конденсации хроматина, опосредованной белком: MENT кооперативно связывается с нуклеосомами за счет М-петли, что влечет за собой конформационный переход его RCL-домена из α-спирали в β-структуру, вызывая олигомеризацию MENT и способствуя самоассоциации цепей нуклеосом. Рисунок воспроизведен из статьи Springhetti E.M. и соавт. [124], опубликованной в The Journal of Biological Chemistry © American Society for Biochemistry and Molecular Biology.
Белок конденсин, играющий основную роль в образовании и поддержании структуры конденсированных метафазных хромосом, также участвует в образовании факультативного хроматина. Так, в процессе конденсации хроматина в ядрах Т-лимфоцитов ДНК становится недоступной для активаторов транскрипции. Однако мутация в конденсине II ослабляет конденсацию хроматина и делает ДНК более доступной для транскрипции [130], указывая на возможную роль конденсина как фактора, промотирующего конденсацию хроматина в интерфазной клетке, что подтверждает раннюю гипотезу E. Heitz о сходном механизме конденсации метафазных хромосом и интерфазного гетерохроматина [86].
Зрелые лимфоциты, обладающие высокой степенью конденсации хроматина, могут проводить длительное время в неделящемся состоянии (фаза G0 клеточного цикла), после чего реактивироваться в процессе иммунного ответа и претерпевать несколько клеточных делений. В ходе реактивации происходит резкое увеличение клеточного ядра и деконденсация хроматина [130]. Недавно с применением геномного анализа и микроскопии сверхвысокого разрешения показано, что в ходе реактивации В-лимфоцитов происходит как общая деконденсация хроматина на уровне ядерных компартментов, так и разворачивание отдельных кластеров (нанодоменов) из нескольких нуклеосом. При этом общая деконденсация хроматина зависела от уровня ацетилирования гистонов, в то время как факторы разворачивания нуклеосомных нанодоменов включали в себя АТР (источник энергии) и белок Myc, известный регулятор клеточной пролиферации, который ранее не числился среди архитектурных белков хроматина [23].
Таким образом, в клетках разного типа конденсированный хроматин содержит различные архитектурные белки, что свидетельствует о независимой и поздней эволюции этого механизма в клетках позвоночных. При этом наиболее общее свойство ядер терминально дифференцированных клеток ‒ контраст между очень низким уровнем ацетилирования гистонов в конденсированном хроматине и повышенным уровнем метилирования гистона Н3К9 [23, 93, 106], а также Н4К20 [95]. Это указывает на то, что потеря ацетилирования гистонов может приводить к конденсации и самоассоциации факультативного хроматина за счет повышения электростатических взаимодействий, в соответствии со способностью ацетилирования гистонов ингибировать самоассоциацию хроматина in vitro [72, 73]. При этом конденсация хроматина в ходе терминальной дифференцировки может быть обусловлена как активностью гистоновых деацетилаз, показанной, например, при мышечной [131] и эритроидной дифференцировке [93], так и активностью ферментов, вызывающих метилирование гистонов Н3 [132] и Н4 [95], которое может блокировать ацетилирование по тому же или соседнему аминокислотному остатку.
РАЗДЕЛЕНИЕ ЖИДКИХ ФАЗ КАК МЕХАНИЗМ КОМПАРТМЕНТАЛИЗАЦИИ ХРОМАТИНА
Ядро клетки не содержит мембранных компартментов, но имеет характерные внутриядерные тельца, такие как ядрышко, спеклы, параспеклы и др., которые образуют отдельные структуры с определенными биохимическими и биофизическими свойствами. Такие структуры формируются при локальном концентрировании белков и нуклеиновых кислот, что приводит к разделению жидких фаз (англ. liquid-liquid phase separation, LLPS) и образованию так называемых “жидких капелек”, избирательно включающих в себя определенные типы белков и нуклеиновых кислот [133, 134]. Основным механизмом, приводящим к образованию жидких капелек, является самоассоциация и олигомеризация белков, содержащих неструктурированные полипептидные домены [135]. К общим свойствам разделенных жидких фаз относятся подвижное (“жидкое”) состояние белковых молекул, повышенная чувствительность к температуре, а также к определенным реагентам, разрушающим слабые гидрофобные взаимодействия, таким как 1,6-гександиол [134, 136]. Большинство ядерных структур, опосредованных LLPS, включает белки и РНК и образуется во внутриядерном пространстве с низкой концентрацией ДНК и хроматина [137]. Однако изучение механизмов образования крупных кластеров гетерохроматина и суперэнхансеров показало, что процесс разделения жидких фаз может быть движущей силой динамической конденсации и компартментализации хроматина в клеточном ядре [28, 29, 138].
В ядрах делящихся клеток кластеры конститутивного гетерохроматина (рис. 5д, е), наряду с гистоном Н3, триметилированным по лизину-9 (Н3К9mе3), обогащены белком НР1 [87]. Как уже отмечено, белки НР1 способны олигомеризоваться в растворе и образовывать межнуклеосомные белковые мостики [111, 112]. Недавно была выявлена способность белка НР1 образовывать при определенной концентрации гидрогели, или жидкие капельки. Сливаясь вместе, эти капельки не только способствуют объединению и компартментализации гетерохроматина, но также могут вызывать фазовый переход, который делает этот компартмент доступным для одних белков и недоступным для других [143]. Исследования in vitro показали, что фосфорилирование неструктурированного белкового домена НР1, которое приводит к олигомеризации НР1, а также добавление насыщающей концентрации ДНК достаточны для разделения жидких фаз [143]. Совсем недавно установили, что разделение жидких фаз сопровождает образование динамических конденсатов другого фактора репрессии хроматина ‒ белка PRC1 [139]. Весьма вероятно, что будет показано непосредственное участие в процессе LLPS и других архитектурных белков хроматина, образующих межнуклеосомные мостики и упомянутых в предыдущем разделе.
Компартментализация и самоассоциация хроматина в клеточном ядре не является прерогативой репрессированных генов. Транскрипционные факторы и коактиваторы транскрипции также способны собираться в жидкие капельки при помощи неструктурированных белковых доменов [29]. Эти ядерные субкомпартменты могут включать в себя РНК-полимеразу и активировать транскрипцию. Так, транскрипционные факторы FUS, EWS, TAF15 и SP1 самоассоциируют и образуют жидкие капельки, видимые в живой клетке [140]. Белок BRD4, содержащий бромодомен, связывающийся с ацетилированными гистонами, вносит существенный вклад в поддержание структуры активного хроматина [141]. Связывание BRD4, а также белка медиаторного комплекса MED1 с ДНК транскрипционных энхансеров вызывает их самоассоциацию и образование суперэнхансерных кластеров в составе динамических конденсатов или гидрогелей [29, 142], подобных гидрогелям, образуемым белком НР1 [28], и другим структурам, образующимся за счет раздела жидких фаз.
В настоящее время не установлено окончательно, необходимо ли разделение жидких фаз для нормального функционирования хроматина, или же образование динамических конденсатов обусловлено избыточным накоплением неструктурированных белков и нуклеиновых кислот при определенных условиях. Белки гетерохроматина [116] и активного хроматина [140] могут выполнять свои регуляторные функции в концентрациях, существенно меньших, чем необходимо для разделения жидких фаз. Предстоит выяснить, какие пороговые концентрации белков требуются как для фазового перехода, так и для соответствующих функций, а также есть ли общий механизм для процесса LLPS в клеточном ядре и самоассоциации хроматина в растворе, индуцированной добавлением катионов [61, 76] или олигомерными белковыми мостиками (рис. 6). В этой связи удачной концепцией представляется выделение в механизме фазового разделения трех взаимосвязанных, но отдельных процессов: а) кооперативное связывание архитектурных белков; б) образование белковых мостиков и конденсированных полимерных фаз (PPPS); в) образование настоящих изолированных жидких фаз (LLPS) [138].
ХРОМОСОМНЫЕ КОНТАКТНЫЕ ДОМЕНЫ РЕГУЛИРУЮТ САМОАССОЦИАЦИЮ ХРОМАТИНА В ИНТЕРФАЗНОМ ЯДРЕ
Важно и то, какие механизмы или ядерные структуры ограничивают и локализуют самоассоциацию хроматина и распространение жидких фаз на все участки генома [143]. Ведущую роль в пространственной организации хроматина в метафазных хромосомах и интерфазных клеточных ядрах играют белки группы SMC (structural maintenance of chromosomes), в первую очередь конденсин и когезин [144]. Основные свойства этих белков ‒ образование колец, замыкающихся вокруг молекул ДНК [145, 146], а также способность к направленному перемещению вдоль ДНК за счет энергии АТР [147], что приводит к выпетливанию молекул ДНК [148] и, вероятно, к образованию протяженных и устойчивых петлевых доменов хроматина в результате так называемой экструзии петель [149, 150].
В последнее десятилетие технологический прорыв в области высокоэффективного секвенирования геномной ДНК привел к революционному скачку в понимании пространственно-временной организации хроматина в ядре интерфазной клетки. Геномное картирование модификаций гистонов, а также транскрипционно-регуляторных белков методом иммунопреципитации хроматина и секвенирования (ChIP-seq) в рамках проекта Энциклопедии элементов ДНК (ENCODE) привело к созданию детальных карт различных эпигенетических состояний хроматина в геномах человека и мыши. Эти карты широко используются как стандартные базы данных для сравнения транскрипционной активности и эпигенетической маркировки индивидуальных генов и хромосомных доменов [151]. Другая революционная технология ‒ метод пан-геномного анализа хромосомных контактов (Hi-C), выявила геномные домены с резко обозначенными границами, внутри которых участки хроматина обладают повышенной частотой контактов между собой. При этом границы контактных доменов совпадают с границами эпигенетических состояний хроматина, что позволило условно разделить контактные домены на типы А (активный хроматин) и В (репрессированный хроматин) [152]. Наиболее крупные контактные домены, так называемые ТАДы, размером до нескольких миллионов пар нуклеотидов, образуют микроскопически различимые хромосомные субкомпартменты в трехмерном пространстве клеточного ядра [153, 154]. Анализ генома Drosophila melanogaster методом Hi-C, проведенный группой С. Разина, также показал, что границы контактных доменов соответствуют границам между активным и репрессированным хроматином, причем ТАДы соответствуют хорошо известным конденсированным дискам политенных хромосом, указывая на ведущую роль структурно-функциональной сегрегации активного и репрессированного хроматина в пространственной организации генома Drosophila [155, 156].
Путем увеличения числа секвенированных контактов ДНК до нескольких миллиардов и развития методов компьютерного анализа данных Hi-C Е. Liеberman Aiden и его группа выявили множественные контактные домены хроматина существенно меньшего размера, чем ТАДы, в основании которых находятся ДНК-связывающий белок CTCF и SMC белок когезин, формирующий кольцевые структуры, соединяющие основания петель хроматина. При сопоставлении с ранее картированными эпигенетическими состояниями хроматина были обнаружены эпигенетические домены шести типов: два типа активного хроматина (А1 и А2) и четыре ‒ репрессированного (В1‒В4), границы которых в целом, совпадают с границами петлевых и контактных доменов, что указывает на ведущую роль петель в структурно-функциональной организации интерфазного хроматина [25]. Впоследствии, однако, было показано, что при инактивации как CTCF [157], так и когезина [158, 159] ТАДы и петлевые домены исчезают, в то время как эпигенетические состояния хроматина сохраняются. При некоторых из этих состояний хроматина после потери когезина наблюдалось слияние отдельных доменов в более крупные комплексы. Интересно, что с помощью компьютерного моделирования удалось наблюдать сегрегацию хромосомных доменов, опосредованную N-концевыми доменами гистонов и регулируемую их посттрансляционными модификациями [159]. Таким образом, становится ясно, что петлевые домены могут препятствовать спонтанной ассоциации и пространственной сегрегации структурно-функциональных доменов хроматина в клеточном ядре. Этот вывод согласуется с результатами анализа структурных изменений хроматина в ходе активации лимфоцитов, когда число CTCF-зависимых петель резко увеличивается, а конденсированный хроматин разворачивается в открытые цепи нуклеосом [23]. Напротив, способность к самоассоциации и сегрегации отдельных хромосомных доменов, по-видимому, не требует наличия ТАДов и петель и определяется сродством сходных типов хроматина, таких как гетерохроматин [156] или кластеры суперэнхансеров [159].
Наряду с ТАДами и петлями, локализация конденсированного гетерохроматина на периферии клеточного ядра обеспечивается взаимодействием между белками ядерной ламины и так называемыми ламина-ассоциированными доменами хроматина [27]. Потеря ассоциации хроматина с ядерной ламиной приводит к “инверсии” хроматина [160], когда зоны гетерохроматина перемещаются от периферии к центру клеточного ядра (рис. 5a–в). Самое последнее исследование пространственной организации генома в ходе инверсии хроматина в клетках сетчатки глаза, выполненное с помощью метода Hi-C, показало, что и в этом случае способность гетерохроматина к самоассоциации является движущей силой пространственной организации и компартментализации хроматина в клеточном ядре [161].
ЗАКЛЮЧЕНИЕ
Несмотря на существенные успехи в изучении трехмерной упаковки интерфазного хроматина, мы еще далеки от понимания всего комплекса физиологических условий, влияющих на структуру и укладку хроматина на нуклеосомном уровне. В этом обзоре мы рассмотрели два разных процесса, приводящих к конденсации хроматина: самоассоциацию цепей нуклеосом in vitro и компартментализацию и сегрегацию хромосомных доменов in vivo. Есть ли связь между этими двумя процессами? С одной стороны, хроматин in vitro претерпевает кооперативный фазовый переход в узких пределах концентрации двухвалентных катионов, и мы относительно хорошо представляем физический (электростатический) механизм этого процесса, опосредованного транс-нуклеосомными взаимодействиями N-концевых доменов гистонов. С другой стороны, хромосомные домены in vivo подвергаются структурным переходам в существенно разной степени в зависимости от модификаций гистонов, физиологического состояния и типа клетки. Поскольку недавние эксперименты показали, что внутреннее сродство или самоассоциация подобных состояний хроматина усиливается при ослаблении внутриядерных структур, таких как петли, ТАДы, и ядерная ламина, мы можем предположить, что структура и компартментализация хроматина определяется балансом между способностью нуклеосомных цепей к самоассоциации и их взаимодействиями с внутриядерными структурами, поддерживающими нуклеосомные цепи в развернутом состоянии.
Чтобы лучше понять эти механизмы и выявить роль свойств самой нуклеосомной цепи в процессе внутриядерной компартментализации, нам необходимо более точно знать ионные условия и степень нейтрализации ДНК внутри ядра живой клетки, в частности, изменение концентрации двух- и многовалентных катионов в ходе клеточной дифференцировки. Нам необходимо также выяснить топологию и состав нуклеосомных цепей, соответствующих отдельным эпигенетическим состояниям хроматина, используя методы биохимического фракционирования хроматина в сочетании с ЭМ [20] или масс-спектрометрией [127].
Поскольку структура хроматина многообразна и не может быть описана единой регулярной моделью, такой как спиральная 30 нм фибрилла, необходимо дальнейшее развитие методов компьютерного моделирования, включающих конформационное многообразие и способность к самоассоциации нуклеосомной цепи. Так, например, недавнее моделирование структуры хроматина группой Т. Schlick, основанное на электростатических взаимодействиях N-концевых доменов гистонов, регулируемых ацетилированием, и гибкости хроматиновых фибрилл, регулируемой линкерным гистоном, смогло воспроизвести трехмерную сегрегацию и компартментализацию активных и неактивных доменов хроматина [159, 162]. В обозримом будущем комбинирование методов молекулярного и структурного анализа нуклеосомных контактов, ЭМ и микроскопии сверхвысокого разрешения, а также компьютерного моделирования должно привести к полномасштабному картированию трехмерной организации интерфазного генома с нуклеосомным уровнем разрешения и созданию ресурсов для успешного молекулярного дизайна новых соединений, способных регулировать эпигенетические состояния хроматина в живой клетке.
Список литературы
Richmond T.J., Davey C.A. (2003) The structure of DNA in the nucleosome core. Nature. 423, 145‒150.
Thoma F., Koller T., Klug A. (1979) Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin. J. Cell Biol. 83, 403‒427.
Woodcock C.L., Grigoryev S.A., Horowitz R.A., Whitaker N. (1993) A chromatin folding model that incorporates linker variability generates fibers resembling the native structures. Proc. Natl. Acad. Sci. USA. 90, 9021‒9025.
Alberts B., Johnson A., Lewis J., Morgan D., Raff M., Roberts K., Walter P. (2015) Molecular Biology of the Cell. 6th ed. New York: Garland Science.
Hu Y., Kireev I., Plutz M., Ashourian N., Belmont A.S. (2009) Large-scale chromatin structure of inducible genes: transcription on a condensed, linear template. J. Cell Biol. 185, 87‒100.
Dorigo B., Schalch T., Kulangara A., Duda S., Schroeder R.R., Richmond T.J. (2004) Nucleosome arrays reveal the two-start organization of the chromatin fiber. Science. 306, 1571‒1573.
Schalch T., Duda S., Sargent D.F., Richmond T.J. (2005) X-ray structure of a tetranucleosome and its implications for the chromatin fibre. Nature. 436, 138‒141.
Song F., Chen P., Sun D., Wang M., Dong L., Liang D., Xu R.M., Zhu P., Li G. (2014) Cryo-EM study of the chromatin fiber reveals a double helix twisted by tetranucleosomal units. Science. 344, 376‒380.
Garcia-Saez I., Menoni H., Boopathi R., Shukla M.S., Soueidan L., Noirclerc-Savoye M., Le Roy A., Skoufias D.A., Bednar J., Hamiche A., Angelov D., Petosa C., Dimitrov S. (2018) Structure of an H1-bound 6-nucleosome array reveals an untwisted two-start chromatin fiber conformation. Mol. Cell. 72, 902‒915.
Fussner E., Ching R.W., Bazett-Jones D.P. (2011) Living without 30 nm chromatin fibers. Trends Biochem. Sci. 36, 1‒6.
Eltsov M., Maclellan K.M., Maeshima K., Frangakis A.S., Dubochet J. (2008) Analysis of cryo-electron microscopy images does not support the existence of 30-nm chromatin fibers in mitotic chromosomes in situ. Proc. Natl. Acad. Sci. USA. 105, 19732‒19737.
Ou H.D., Phan S., Deerinck T.J., Thor A., Ellisman M.H., O’Shea C.C. (2017) ChromEMT: visualizing 3D chromatin structure and compaction in interphase and mitotic cells. Science. 357, 370.
Cai S., Chen C., Tan Z.Y., Huang Y., Shi J., Gan L. (2018) Cryo-ET reveals the macromolecular reorganization of S. pombe mitotic chromosomes in vivo. Proc. Natl. Acad. Sci. USA. 115, 10977‒10982.
Ricci M.A., Manzo C., Garcia-Parajo M.F., Lakadamyali M., Cosma M.P. (2015) Chromatin fibers are formed by heterogeneous groups of nucleosomes in vivo. Cell. 160, 1145‒1158.
Nozaki T., Imai R., Tanbo M., Nagashima R., Tamura S., Tani T., Joti Y., Tomita M., Hibino K., Kanemaki M.T., Wendt K.S., Okada Y., Nagai T., Maeshima K. (2017) Dynamic organization of chromatin domains revealed by super-resolution live-cell imaging. Mol. Cell. 67, 282‒293. e7.
McDowall A.W., Smith J.M., Dubochet J. (1986) Cryo-electron microscopy of vitrified chromosomes in situ. EMBO J. 5, 1395‒1402.
Joti Y., Hikima T., Nishino Y., Kamda F., Hihara S., Takata H., Ishikawa T., Maeshima K. (2012) Chromosomes without a 30-nm chromatin fiber. Nucleus. 3, 404‒410.
Hsieh T.H., Weiner A., Lajoie B., Dekker J., Friedman N., Rando O.J. (2015) Mapping nucleosome resolution chromosome folding in yeast by micro-C. Cell. 162, 108‒119.
Hsieh T.-H.S., Slobodyanyuk E., Hansen A.S., Cattoglio C., Rando O.J., Tjian R., Darzacq X. (2019) Resolving the 3D landscape of transcription-linked mammalian chromatin folding. bioRxiv. https://doi.org/10.1101/638775
Grigoryev S.A., Bascom G., Buckwalter J.M., Schubert M.B., Woodcock C.L., Schlick T. (2016) Hierarchical looping of zigzag nucleosome chains in metaphase chromosomes. Proc. Natl. Acad. Sci. USA. 113, 1238‒1243.
Risca V.I., Denny S.K., Straight A.F., Greenleaf W.J. (2017) Variable chromatin structure revealed by in situ spatially correlated DNA cleavage mapping. Nature. 541, 237‒241.
Fang K., Chen X., Li X., Shen Y., Sun J., Czajkowsky D.M., Shao Z. (2018) Super-resolution imaging of individual human subchromosomal regions in situ reveals nanoscopic building blocks of higher-order structure. ACS Nano. 12, 4909‒4918.
Kieffer-Kwon K.R., Nimura K., Rao S.S.P., Xu J., Jung S., Pekowska A., Dose M., Stevens E., Mathe E., Dong P., Huang S.C., Ricci M.A., Baranello L., Zheng Y., Tomassoni Ardori F., Resch W., Stavreva D., Nelson S., McAndrew M., Casellas A., Finn E., Gregory C., St Hilaire B.G., Johnson S.M., Dubois W., Cosma M.P., Batchelor E., Levens D., Phair R.D., Misteli T., Tessarollo L., Hager G., Lakadamyali M., Liu Z., Floer M., Shroff H., Aiden E.L., Casellas R. (2017) Myc regulates chromatin decompaction and nuclear architecture during B cell activation. Mol. Cell. 67, 566‒578. e10.
Chicano A., Crosas E., Oton J., Melero R., Engel B.D., Daban J.R. (2019) Frozen-hydrated chromatin from metaphase chromosomes has an interdigitated multilayer structure. EMBO J. 38, e99769.
Rao S.S., Huntley M.H., Durand N.C., Stamenova E.K., Bochkov I.D., Robinson J.T., Sanborn A.L., Machol I., Omer A.D., Lander E.S., Aiden E.L. (2014) A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159, 1665‒1680.
Nozawa R.S., Boteva L., Soares D.C., Naughton C., Dun A.R., Buckle A., Ramsahoye B., Bruton P.C., Saleeb R.S., Arnedo M., Hill B., Duncan R.R., Maciver S.K., Gilbert N. (2017) SAF-A regulates interphase chromosome structure through oligomerization with chromatin-associated RNAs. Cell. 169, 1214‒1227. e18.
van Steensel B., Belmont A.S. (2017) Lamina-associated domains: Links with chromosome architecture, heterochromatin, and gene repression. Cell. 169, 780‒791.
Klosin A., Hyman A.A. (2017) Molecular biology: A liquid reservoir for silent chromatin. Nature. 547, 168‒170.
Plys A.J., Kingston R.E. (2018) Dynamic condensates activate transcription. Science. 361, 329‒330.
Спирин К.С., Григорьев С.А., Крашенинников И.А. (1988) Механизм образования ассоциатов олигонуклеосом при электрофорезе. Молекуляр. биология. 22, 530‒538.
Григорьев С.А., Спирин К.С., Соловьева В.О., Крашенинников И.А. (1990) Новый положительно заряженный негистоновый белок участвует в конденсации неактивного хроматина на терминальной стадии дифференцировки эритроцитов цыплeнка. Докл. АН СССР. 312, 1266‒1271.
Grigoryev S.A., Spirin K.S., Krasheninnikov I.A. (1990) Loosened nucleosome linker folding in transcriptionally active chromatin of chicken embryo erythrocyte nuclei. Nucl. Acids Res. 18, 7397‒7406.
Grigoryev S.A., Solovieva V.O., Spirin K.S., Krasheninnikov I.A. (1992) A novel nonhistone protein (MENT) promotes nuclear collapse at the terminal stage of avian erythropoiesis. Exp. Cell. Res. 198, 268‒275.
Dubochet J., Noll M. (1978) Nucleosome arcs and helices. Science. 202, 280‒286.
Leforestier A., Dubochet J., Livolant F. (2001) Bilayers of nucleosome core particles. Biophys. J. 81, 2414‒2421.
Mangenot S., Leforestier A., Durand D., Livolant F. (2003) X-ray diffraction characterization of the dense phases formed by nucleosome core particles. Biophys. J. 84, 2570‒2584.
Bilokapic S., Strauss M., Halic M. (2018) Cryo-EM of nucleosome core particle interactions in trans. Sci. Rep. 8, 7046.
Luger K., Mader A.W., Richmond R.K., Sargent D.F., Richmond T.J. (1997) Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature. 389, 251‒260.
Harp J.M., Hanson B.L., Timm D.E., Bunick G.J. (2000) Asymmetries in the nucleosome core particle at 2.5 A resolution. Acta Crystallogr. D Biol. Crystallogr. 56, 1513‒1534.
White C.L., Suto R.K., Luger K. (2001) Structure of the yeast nucleosome core particle reveals fundamental changes in internucleosome interactions. EMBO J. 20, 5207‒5218.
Andrews A.J., Luger K. (2011) Nucleosome structure(s) and stability: variations on a theme. Annu. Rev. Biophys. 40, 99‒117.
McGinty R.K., Tan S. (2016) Recognition of the nucleosome by chromatin factors and enzymes. Curr. Opin. Struct. Biol. 37, 54‒61.
Widom J. (1986) Physicochemical studies of the folding of the 100 A nucleosome filament into the 300 Å filament. Cation dependence. J. Mol. Biol. 190, 411‒424.
Clark D.J., Kimura T. (1990) Electrostatic mechanism of chromatin folding. J. Mol. Biol. 211, 883‒896.
Manning G.S. (1978) The molecular theory of polyelectrolyte solutions with applications to the electrostatic properties of polynucleotides. Q. Rev. Biophys. 11, 179‒246.
Grigoryev S.A., Bednar J., Woodcock C.L. (1999) MENT, a heterochromatin protein that mediates higher order chromatin folding, is a new serpin family member. J. Biol. Chem. 274, 5626‒5636.
Grigoryev S.A. (2004) Keeping fingers crossed: heterochromatin spreading through interdigitation of nucleosome arrays. FEBS Lett. 564, 4‒8.
Grigoryev S.A., Bulynko Y.A., Popova E.Y. (2006) The end adjusts the means: heterochromatin remodelling during terminal cell differentiation. Chromosome Res. 14, 53‒69.
Simpson R.T., Thoma F., Brubaker J.M. (1985) Chromatin reconstituted from tandemly repeated cloned DNA fragments and core histones: a model system for study of higher order structure. Cell. 42, 799‒808.
Lowary P.T., Widom J. (1998) New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol. 276, 19‒42.
Schwarz P.M., Hansen J.C. (1994) Formation and stability of higher order chromatin structures. Contributions of the histone octamer. J. Biol. Chem. 269, 16284‒16289.
Correll S.J., Schubert M.H., Grigoryev S.A. (2012) Short nucleosome repeats impose rotational modulations on chromatin fibre folding. EMBO J. 31, 2416‒2426.
Bass M.V., Nikitina T., Norouzi D., Zhurkin V.B., Grigoryev S.A. (2019) Nucleosome spacing periodically modulates nucleosome chain folding and DNA topology in circular nucleosome arrays. J. Biol. Chem. 294, 4233‒4246.
Hansen J.C. (2002) Conformational dynamics of the chromatin fiber in solution: determinants, mechanisms, and functions. Annu. Rev. Biophys. Biomol. Struct. 31, 361‒392.
Pepenella S., Murphy K.J., Hayes J.J. (2014) Intra- and inter-nucleosome interactions of the core histone tail domains in higher-order chromatin structure. Chromosoma. 123, 3‒13.
Gao M., Nadaud P.S., Bernier M.W., North J.A., Hammel P.C., Poirier M.G., Jaroniec C.P. (2013) Histone H3 and H4 N-terminal tails in nucleosome arrays at cellular concentrations probed by magic angle spinning NMR spectroscopy. J. Am. Chem. Soc. 135, 15278‒15281.
Luger K., Hansen J.C. (2005) Nucleosome and chromatin fiber dynamics. Curr. Opin. Struct. Biol. 15, 188‒196.
Schwarz P.M., Felthauser A., Fletcher T.M., Hansen J.C. (1996) Reversible oligonucleosome self-association: dependence on divalent cations and core histone tail domains. Biochemistry. 35, 4009‒4015.
Carruthers L.M., Hansen J.C. (2000) The core pistone N termini function independently of linker histones during chromatin condensation. J. Biol. Chem. 275, 37285‒37290.
Tse C., Hansen J.C. (1997) Hybrid trypsinized nucleosomal arrays: identification of multiple functional roles of the H2A/H2B and H3/H4 N-termini in chromatin fiber compaction. Biochemistry. 36, 11381‒11388.
Maeshima K., Rogge R., Tamura S., Joti Y., Hikima T., Szerlong H., Krause C., Herman J., Seidel E., DeLuca J., Ishikawa T., Hansen J.C. (2016) Nucleosomal arrays self-assemble into supramolecular globular structures lacking 30-nm fibers. EMBO J. 35, 1115‒1132.
Kan P.Y., Lu X., Hansen J.C., Hayes J.J. (2007) The H3 tail domain participates in multiple interactions during folding and self-association of nucleosome arrays. Mol. Cell Biol. 27, 2084‒2091.
Kan P.Y., Caterino T.L., Hayes J.J. (2009) The H4 tail domain participates in intra- and internucleosome interactions with protein and DNA during folding and oligomerization of nucleosome arrays. Mol. Cell Biol. 29, 538‒546.
Sinha D., Shogren-Knaak M.A. (2010) Role of direct interactions between the histone H4 tail and the H2A core in long range nucleosome contacts. J. Biol. Chem. 285, 16572‒16581.
Zhou J., Fan J.Y., Rangasamy D., Tremethick D.J. (2007) The nucleosome surface regulates chromatin compaction and couples it with transcriptional repression. Nat. Struct. Mol. Biol. 14, 1070‒1076.
Chodaparambil J.V., Barbera A.J., Lu X., Kaye K.M., Hansen J.C., Luger K. (2007) A charged and contoured surface on the nucleosome regulates chromatin compaction. Nat. Struct. Mol. Biol. 14, 1105‒1107.
Arya G., Schlick T. (2009) A tale of tails: how histone tails mediate chromatin compaction in different salt and linker histone environments. J. Phys. Chem. A. 113, 4045‒4059.
Grigoryev S.A., Arya G., Correll S., Woodcock C.L., Schlick T. (2009) Evidence for heteromorphic chromatin fibers from analysis of nucleosome interactions. Proc. Natl. Acad. Sci. USA. 106, 13317‒13322.
Collepardo-Guevara R., Portella G., Vendruscolo M., Frenkel D., Schlick T., Orozco M. (2015) Chromatin unfolding by epigenetic modifications explained by dramatic Impairment of internucleosome interactions: A multiscale computational study. J. Am. Chem. Soc. 137, 10205‒10215.
Soshnev A.A., Josefowicz S.Z., Allis C.D. (2016) Greater than the sum of parts: complexity of the dynamic epigenome. Mol. Cell. 62, 681‒694.
Li G., Reinberg D. (2011) Chromatin higher-order structures and gene regulation. Curr. Opin. Genet. Dev. 21, 175‒186.
Tse C., Sera T., Wolffe A.P., Hansen J.C. (1998) Disruption of higher-order folding by core histone acetylation dramatically enhances transcription of nucleosomal arrays by RNA polymerase III. Mol. Cell. Biol. 18, 4629‒4638.
Shogren-Knaak M., Ishii H., Sun J.M., Pazin M.J., Davie J.R., Peterson C.L. (2006) Histone H4-K16 acetylation controls chromatin structure and protein interactions. Science. 311, 844‒847.
Chen Q., Yang R., Korolev N., Liu C.F., Nordenskiold L. (2017) Regulation of nucleosome stacking and chromatin compaction by the histone H4 N-terminal tail-H2A acidic patch interaction. J. Mol. Biol. 429, 2075‒2092.
Turner A.L., Watson M., Wilkins O.G., Cato L., Travers A., Thomas J.O., Stott K. (2018) Highly disordered histone H1-DNA model complexes and their condensates. Proc. Natl. Acad. Sci. USA. 115, 11964‒11969.
Gibson B.A., Doolittle L.K., Jensen L.E., Gamarra N., Redding S., Rosen M.K. (2019) Organization and regulation of chromatin by liquid-liquid phase separation. bioRxiv. https://doi.org/10.1101/523662
Евреинова Т.Н. (1966) Концентрирование веществ и действие ферментов в коацерватах. Отв. ред. Опарин А.И. Москва: Наука. 228 с.
Ernst J., Kheradpour P., Mikkelsen T.S., Shoresh N., Ward L.D., Epstein C.B., Zhang X., Wang L., Issner R., Coyne M., Ku M., Durham T., Kellis M., Bernstein B.E. (2011) Mapping and analysis of chromatin state dynamics in nine human cell types. Nature. 473, 43‒49.
Weintraub H., Groudine M. (1976) Chromosomal subunits in active genes have an altered conformation. Science. 193, 848‒856.
Weintraub H. (1984) Histone-H1-dependent chromatin superstructures and the suppression of gene activity. Cell. 38, 17‒27.
Weintraub H. (1985) Assembly and propagation of repressed and depressed chromosomal states. Cell. 42, 705‒711.
Kamakaka R.T., Thomas J.O. (1990) Chromatin structure of transcriptionally competent and repressed genes. EMBO J. 9, 3997‒4006.
Verreault A., Thomas J.O. (1993) Chromatin structure of the beta-globin chromosomal domain in adult chicken erythrocytes. Cold Spring Harb. Symp. Quant. Biol. 58, 15‒24.
Felsenfeld G., Burgess-Beusse B., Farrell C., Gasz-ner M., Ghirlando R., Huang S., Jin C., Litt M., Magdinier F., Mutskov V., Nakatani Y., Tagami H., West A., Yusufzai T. (2004) Chromatin boundaries and chromatin domains. Cold Spring Harb. Symp. Quant. Biol. 69, 245‒250.
Ridsdale J.A., Davie J.R. (1987) Chicken erythrocyte polynucleosomes which are soluble at physiological ionic strength and contain linker histones are highly enriched in beta-globin gene sequences. Nucl. Acids Res. 15, 1081‒1096.
Heitz E. (1928) Das heterochromatin der moose. I. Jb. Wisensch. Bot. 69, 762‒818.
Richards E.J., Elgin S.C. (2002) Epigenetic codes for heterochromatin formation and silencing: rounding up the usual suspects. Cell. 108, 489‒500.
Allshire R.C., Madhani H.D. (2018) Ten principles of heterochromatin formation and function. Nat. Rev. Mol. Cell. Biol. 19, 229‒244.
Eissenberg J.C., Elgin S.C., Paro R. (2000) Epigenetic regulation in Drosophila: unravelling the conspiracy of silence. in Chromatin Structure and Gene Expression. Eds Elgin S.C.A., Workman J.L. Oxford, UK: Oxford University Press, 229‒251.
Schotta G., Lachner M., Sarma K., Ebert A., Sengupta R., Reuter G., Reinberg D., Jenuwein T. (2004) A silencing pathway to induce H3-K9 and H4-K20 trimethylation at constitutive heterochromatin. Genes Dev. 18, 1251‒1262.
Plath K., Mlynarczyk-Evans S., Nusinow D.A., Panning B. (2002) Xist RNA and the mechanism of X chromosome inactivation. Annu. Rev. Genet. 36, 233‒278.
Fadloun A., Eid A., Torres-Padilla M.E. (2013) Mechanisms and dynamics of heterochromatin formation during mammalian development: closed paths and open questions. Curr. Topics Dev. Biol. 104, 1‒45.
Popova E.Y., Krauss S.W., Short S.A., Lee G., Villalobos J., Etzell J., Koury M.J., Ney P.A., Chasis J.A., Grigoryev S.A. (2009) Chromatin condensation in terminally differentiating mouse erythroblasts does not involve special architectural proteins but depends on histone deacetylation. Chromosome Res. 17, 47‒64.
Popova E.Y., Grigoryev S.A., Fan Y., Skoultchi A.I., Zhang S.S., Barnstable C.J. (2013) Developmentally regulated linker histone H1c promotes heterochromatin condensation and mediates structural integrity of rod photoreceptors in mouse retina. J. Biol. Chem. 288, 17895‒17907.
Malik J., Lillis J.A., Couch T., Getman M., Steiner L.A. (2017) The methyltransferase Setd8 is essential for erythroblast survival and maturation. Cell Rep. 21, 2376‒2383.
Weintraub H. (1978) The nucleosome repeat length increases during erythropoiesis in the chick. Nucl. Acids Res. 5, 1179‒1188.
Bates D.L., Thomas J.O. (1981) Histones H1 and H5: one or two molecules per nucleosome? Nucl. Acids Res. 9, 5883‒5894.
Scheffer M.P., Eltsov M., Frangakis A.S. (2011) Evidence for short-range helical order in the 30-nm chromatin fibers of erythrocyte nuclei. Proc. Natl. Acad. Sci. USA. 108, 16992‒16997.
Kizilyaprak C., Spehner D., Devys D., Schultz P. (2010) In vivo chromatin organization of mouse rod photoreceptors correlates with histone modifications. PLoS One. 5, e11039.
Grigoryev S.A., Nikitina T., Pehrson J.R., Singh P.B., Woodcock C.L. (2004) Dynamic relocation of epigenetic chromatin markers reveals an active role of constitutive heterochromatin in the transition from proliferation to quiescence. J. Cell Sci. 117, 6153‒6162.
Brown K.E., Baxter J., Graf D., Merkenschlager M., Fisher A. (1999) Dynamic repositioning of genes in the nucleus of lymphocytes preparing for cell division. Mol. Cell. 3, 207‒218.
Schubeler D., Francastel C., Cimbora D.M., Reik A., Martin D.I., Groudine M. (2000) Nuclear localization and histone acetylation: a pathway for chromatin opening and transcriptional activation of the human beta-globin locus. Genes Dev. 14, 940‒950.
Bartova E., Kozubek S., Jirsova P., Kozubek M., Gajova H., Lukasova E., Skalnikova M., Ganova A., Koutna I., Hausmann M. (2002) Nuclear structure and gene activity in human differentiated cells. J. Struct. Biol. 139, 76‒89.
Su R.C., Brown K.E., Saaber S., Fisher A.G., Merkenschlager M., Smale S.T. (2004) Dynamic assembly of silent chromatin during thymocyte maturation. Nat. Genet. 36, 502‒506.
Sinclair P., Bian Q., Plutz M., Heard E., Belmont A.S. (2010) Dynamic plasticity of large-scale chromatin structure revealed by self-assembly of engineered chromosome regions. J. Cell Biol. 190, 761‒776.
Popova E.Y., Claxton D.F., Lukasova E., Bird P.I., Grigoryev S.A. (2006) Epigenetic heterochromatin markers distinguish terminally differentiated leukocytes from incompletely differentiated leukemia cells in human blood. Exp. Hematol. 34, 453‒462.
Lukasova E., Koristek Z., Klabusay M., Ondrej V., Grigoryev S., Bacikova A., Rezacova M., Falk M., Vavrova J., Kohutova V., Kozubek S. (2013) Granulocyte maturation determines ability to release chromatin NETs and loss of DNA damage response; these properties are absent in immature AML granulocytes. Biochim. Biophys. Acta. 1833, 767‒779.
Solovei I., Kreysing M., Lanctot C., Kosem S., Peichl L., Cremer T., Guck J., Joffe B. (2009) Nuclear architecture of rod photoreceptor cells adapts to vision in mammalian evolution. Cell. 137, 356‒368.
Meehan R.R., Kao C.F., Pennings S. (2003) HP1 binding to native chromatin in vitro is determined by the hinge region and not by the chromodomain. EMBO J. 22, 3164‒3174.
Ryan D.P., Tremethick D.J. (2018) The interplay between H2A.Z and H3K9 methylation in regulating HP1alpha binding to linker histone-containing chromatin. Nucl. Acids Res. 46, 9353‒9366.
Thiru A., Nietlispach D., Mott H.R., Okuwaki M., Lyon D., Nielsen P.R., Hirshberg M., Verreault A., Murzina N.V., Laue E.D. (2004) Structural basis of HP1/PXVXL motif peptide interactions and HP1 localisation to heterochromatin. EMBO J. 23, 489‒499.
Canzio D., Liao M., Naber N., Pate E., Larson A., Wu S., Marina D.B., Garcia J.F., Madhani H.D., Cooke R., Schuck P., Cheng Y., Narlikar G.J. (2013) A conformational switch in HP1 releases auto-inhibition to drive heterochromatin assembly. Nature. 496, 377‒381.
Azzaz A.M., Vitalini M.W., Thomas A.S., Price J., Blacketer M.J., Cryderman D.E., Zirbel L.N., Woodcock C.L., Elcock A.H., Wallrath L.L., Shogren-Knaak M.A. (2014) Human heterochromatin protein 1alpha promotes nucleosome associations that drive chromatin condensation. J. Biol. Chem. 289, 6850‒6861.
Hiragami-Hamada K., Soeroes S., Nikolov M., Wilkins B., Kreuz S., Chen C., De La Rosa-Velazquez I.A., Zenn H.M., Kost N., Pohl W., Chernev A., Schwarzer D., Jenuwein T., Lorincz M., Zimmermann B., Walla P.J., Neumann H., Baubec T., Urlaub H., Fischle W. (2016) Dynamic and flexible H3K9me3 bridging via HP1beta dimerization establishes a plastic state of condensed chromatin. Nat. Commun. 7, 11310.
Machida S., Takizawa Y., Ishimaru M., Sugita Y., Sekine S., Nakayama J.I., Wolf M., Kurumizaka H. (2018) Structural basis of heterochromatin formation by human HP1. Mol. Cell. 69, 385‒397. e8.
Verschure P.J., van der Kraan I., de Leeuw W., van der Vlag J., Carpenter A.E., Belmont A.S., van Driel R. (2005) In vivo HP1 targeting causes large-scale chromatin condensation and enhanced histone lysine methylation. Mol. Cell Biol. 25, 4552‒4564.
Istomina N.E., Shushanov S.S., Springhetti E.M., Karpov V.L., Krasheninnikov I.A., Stevens K., Zaret K.S., Singh P.B., Grigoryev S.A. (2003) Insulation of the chicken beta-globin chromosomal domain from a chromatin-condensing protein, MENT. Mol. Cell Biol. 23, 6455‒6468.
Gilbert N., Boyle S., Sutherland H., de Las Heras J., Allan J., Jenuwein T., Bickmore W.A. (2003) Formation of facultative heterochromatin in the absence of HP1. EMBO J. 22, 5540‒5550.
Vakoc C.R., Mandat S.A., Olenchock B.A., Blobel G.A. (2005) Histone H3 lysine 9 methylation and HP1gamma are associated with transcription elongation through mammalian chromatin. Mol. Cell. 19, 381‒391.
Zhou B.R., Jiang J., Feng H., Ghirlando R., Xiao T.S., Bai Y. (2015) Structural mechanisms of nucleosome recognition by linker histones. Mol. Cell. 59, 628‒638.
Bednar J., Garcia-Saez I., Boopathi R., Cutter A.R., Papai G., Reymer A., Syed S.H., Lone I.N., Tonc-hev O., Crucifix C., Menoni H., Papin C., Skoufias D.A., Kurumizaka H., Lavery R., Hamiche A., Hayes J.J., Schultz P., Angelov D., Petosa C., Dimitrov S. (2017) Structure and dynamics of a 197 bp nucleosome in complex with linker histone H1. Mol. Cell. 66, 384‒397. e8.
Woodcock C.L., Skoultchi A.I., Fan Y. (2006) Role of linker histone in chromatin structure and function: H1 stoichiometry and nucleosome repeat length. Chromosome Res. 14, 17‒25.
Grigoryev S.A., Woodcock C.L. (1993) Stage-specific expression and localization of MENT, a nuclear protein associated with chromatin condensation in terminally differentiating avian erythroid cells. Exp. Cell Res. 206, 335‒343.
Springhetti E.M., Istomina N.E., Whisstock J.C., Nikitina T.V., Woodcock C.L., Grigoryev S.A. (2003) Role of the M-loop and reactive center loop domains in the folding and bridging of nucleosome arrays by MENT. J. Biol. Chem. 278, 43384‒43393.
McGowan S., Buckle A.M., Irving J.A., Ong P.C., Bashtannyk-Puhalovich T.A., Kan W.T., Henderson K.N., Bulynko Y.A., Popova E.Y., Smith A.I., Bottomley S.P., Rossjohn J., Grigoryev S.A., Pike R.N., Whisstock J.C. (2006) X-ray crystal structure of MENT: evidence for functional loop-sheet polymers in chromatin condensation. EMBO J. 25, 3144‒3155.
Arumugam P., Malik P. (2010) Genetic therapy for beta-thalassemia: from the bench to the bedside. Hematol. Am. Soc. Hematol. Edu. Program. 2010, 445‒450.
Becker J.S., McCarthy R.L., Sidoli S., Donahue G., Kaeding K.E., He Z., Lin S., Garcia B.A., Zaret K.S. (2017) Genomic and proteomic resolution of heterochromatin and its restriction of alternate fate genes. Mol. Cell. 68, 1023‒1037.
Brero A., Easwaran H.P., Nowak D., Grunewald I., Cremer T., Leonhardt H., Cardoso M.C. (2005) Methyl CpG-binding proteins induce large-scale chromatin reorganization during terminal differentiation. J. Cell Biol. 169, 733‒743.
Agarwal N., Hardt T., Brero A., Nowak D., Rothbauer U., Becker A., Leonhardt H., Cardoso M.C. (2007) MeCP2 interacts with HP1 and modulates its heterochromatin association during myogenic differentiation. Nucl. Acids Res. 35, 5402‒5408.
Rawlings J.S., Gatzka M., Thomas P.G., Ihle J.N. (2011) Chromatin condensation via the condensin II complex is required for peripheral T-cell quiescence. EMBO J. 30, 263–276.
Terranova R., Sauer S., Merkenschlager M., Fisher A.G. (2005) The reorganisation of constitutive heterochromatin in differentiating muscle requires HDAC activity. Exp. Cell Res. 20, 20.
Pinheiro I., Margueron R., Shukeir N., Eisold M., Fritzsch C., Richter F.M., Mittler G., Genoud C., Goyama S., Kurokawa M., Son J., Reinberg D., Lachner M., Jenuwein T. (2012) Prdm3 and Prdm16 are H3K9me1 methyltransferases required for mammalian heterochromatin integrity. Cell. 150, 948‒960.
Adriaens C., Serebryannyy L.A., Feric M., Schibler A., Meaburn K.J., Kubben N., Trzaskoma P., Shachar S., Vidak S., Finn E.H., Sood V., Pegoraro G., Misteli T. (2018) Blank spots on the map: some current questions on nuclear organization and genome architecture. Histochem. Cell Biol. 150, 579‒592.
Shin Y., Brangwynne C.P. (2017) Liquid phase condensation in cell physiology and disease. Science. 357, 1253.
Wei M.T., Elbaum-Garfinkle S., Holehouse A.S., Chen C.C., Feric M., Arnold C.B., Priestley R.D., Pappu R.V., Brangwynne C.P. (2017) Phase behaviour of disordered proteins underlying low density and high permeability of liquid organelles. Nat. Chem. 9, 1118‒1125.
Kroschwald S., Maharana S., Mateju D., Malinovska L., Nuske E., Poser I., Richter D., Alberti S. (2015) Promiscuous interactions and protein disaggregases determine the material state of stress-inducible RNP granules. Elife. 4, e06807.
Welsh T.J., Shen Y., Levin A., Knowles T.P.J. (2018) Mechanobiology of protein droplets: force arises from disorder. Cell. 175, 1457‒1459.
Erdel F., Rippe K. (2018) Formation of chromatin subcompartments by phase separation. Biophys. J. 114, 2262‒2270.
Tatavosian R., Kent S., Brown K., Yao T., Duc H.N., Huynh T.N., Zhen C.Y., Ma B., Wang H., Ren X. (2019) Nuclear condensates of the Polycomb protein chromobox 2 (CBX2) assemble through phase separation. J. Biol. Chem. 294, 1451‒1463.
Chong S., Dugast-Darzacq C., Liu Z., Dong P., Dailey G.M., Cattoglio C., Heckert A., Banala S., Lavis L., Darzacq X., Tjian R. (2018) Imaging dynamic and selective low-complexity domain interactions that control gene transcription. Science. 361, 378.
Wang R., Li Q., Helfer C.M., Jiao J., You J. (2012) The bromodomain protein Brd4 associated with acetylated chromatin is important for maintenance of higher-order chromatin structure. J. Biol. Chem. 287, 10738‒10752.
Sabari B.R., Dall’Agnese A., Boija A., Klein I.A., Coffey E.L., Shrinivas K., Abraham B.J., Hannett N.M., Zamudio A.V., Manteiga J.C., Li C.H., Guo Y.E., Day D.S., Schuijers J., Vasile E., Malik S., Hnisz D., Lee T.I., Cisse, II, Roeder R.G., Sharp P.A., Chakraborty A.K., Young R.A. (2018) Coactivator condensation at super-enhancers links phase separation and gene control. Science. 361, 412.
Larson A.G., Narlikar G.J. (2018) The role of phase separation in heterochromatin formation, function, and regulation. Biochemistry. 57, 2540‒2548.
Hirano T. (1999) SMC-mediated chromosome mechanics: a conserved scheme from bacteria to vertebrates? Genes. Dev. 13, 11‒19.
Holmes V.F., Cozzarelli N.R. (2000) Closing the ring: links between SMC proteins and chromosome partitioning, condensation, and supercoiling. Proc. Natl. Acad. Sci. USA. 97, 1322‒1324.
Gruber S., Haering C.H., Nasmyth K. (2003) Chromosomal cohesin forms a ring. Cell. 112, 765‒777.
Terakawa T., Bisht S., Eeftens J.M., Dekker C., Haering C.H., Greene E.C. (2017) The condensin complex is a mechanochemical motor that translocates along DNA. Science. 358, 672‒676.
Ganji M., Shaltiel I.A., Bisht S., Kim E., Kalichava A., Haering C.H., Dekker C. (2018) Real-time imaging of DNA loop extrusion by condensin. Science. 360, 102‒105.
Fudenberg G., Imakaev M., Lu C., Goloborodko A., Abdennur N., Mirny L.A. (2016) Formation of chromosomal domains by loop extrusion. Cell Rep. 15, 2038‒2049.
Vian L., Pekowska A., Rao S.S.P., Kieffer-Kwon K.R., Jung S., Baranello L., Huang S.C., El Khattabi L., Dose M., Pruett N., Sanborn A.L., Canela A., Maman Y., Oksanen A., Resch W., Li X., Lee B., Kovalchuk A.L., Tang Z., Nelson S., Di Pierro M., Cheng R.R., Machol I., St Hilaire B.G., Durand N.C., Shamim M.S., Stamenova E.K., Onuchic J.N., Ruan Y., Nussenzweig A., Levens D., Aiden E.L., Casellas R. (2018) The energetics and physiological impact of cohesin extrusion. Cell. 173, 1165‒1178. e20.
Consortium E.P. (2012) An integrated encyclopedia of DNA elements in the human genome. Nature. 489, 57‒74.
Lieberman-Aiden E., van Berkum N.L., Williams L., Imakaev M., Ragoczy T., Telling A., Amit I., Lajoie B.R., Sabo P.J., Dorschner M.O., Sandstrom R., Bernstein B., Bender M.A., Groudine M., Gnirke A., Stamatoyannopoulos J., Mirny L.A., Lander E.S., Dekker J. (2009) Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289‒293.
Gibcus J.H., Dekker J. (2013) The hierarchy of the 3D genome. Mol. Cell. 49, 773‒782.
Разин С.В., Гаврилов А.А. (2018) Структурно-функциональные домены эукариотического генома. Биохимия. 83, 440‒451.
Ulianov S.V., Khrameeva E.E., Gavrilov A.A., Flyamer I.M., Kos P., Mikhaleva E.A., Penin A.A., Logacheva M.D., Imakaev M.V., Chertovich A., Gelfand M.S., Shevelyov Y.Y., Razin S.V. (2016) Active chromatin and transcription play a key role in chromosome partitioning into topologically associating domains. Genome Res. 26, 70‒84.
Gavrilov A.A., Shevelyov Y.Y., Ulianov S.V., Khrameeva E.E., Kos P., Chertovich A., Razin S.V. (2016) Unraveling the mechanisms of chromatin fibril packaging. Nucleus. 7, 319‒324.
Nora E.P., Goloborodko A., Valton A.L., Gibcus J.H., Uebersohn A., Abdennur N., Dekker J., Mirny L.A., Bruneau B.G. (2017) Targeted degradation of CTCF decouples local insulation of chromosome domains from genomic compartmentalization. Cell. 169, 930‒944. e22.
Schwarzer W., Abdennur N., Goloborodko A., Pekowska A., Fudenberg G., Loe-Mie Y., Fonseca N.A., Huber W., Haering C.H., Mirny L., Spitz F. (2017) Two independent modes of chromatin organization revealed by cohesin removal. Nature. 551, 51‒56.
Rao S.S.P., Huang S.C., Glenn St Hilaire B., Engreitz J.M., Perez E.M., Kieffer-Kwon K.R., Sanborn A.L., Johnstone S.E., Bascom G.D., Bochkov I.D., Huang X., Shamim M.S., Shin J., Turner D., Ye Z., Omer A.D., Robinson J.T., Schlick T., Bernstein B.E., Casellas R., Lander E.S., Aiden E.L. (2017) Cohesin loss eliminates all loop domains. Cell. 171, 305‒320. e24.
Solovei I., Wang A.S., Thanisch K., Schmidt C.S., Krebs S., Zwerger M., Cohen T.V., Devys D., Fois-ner R., Peichl L., Herrmann H., Blum H., Engelkamp D., Stewart C.L., Leonhardt H., Joffe B. (2013) LBR and Lamin A/C sequentially tether peripheral heterochromatin and inversely regulate differentiation. Cell. 152, 584‒598.
Falk M., Feodorova Y., Naumova N., Imakaev M., Lajoie B.R., Leonhardt H., Joffe B., Dekker J., Fudenberg G., Solovei I., Mirny L.A. (2019) Heterochromatin drives compartmentalization of inverted and conventional nuclei. Nature. 570(7761), 395–399. https://doi.org/10.1038/s41586-019-1275-3
Bascom G.D., Myers C.G., Schlick T. (2019) Mesoscale modeling reveals formation of an epigenetically driven HOXC gene hub. Proc. Natl. Acad. Sci. USA. 116, 4955‒4962.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


