Молекулярная биология, 2020, T. 54, № 2, стр. 204-211
Функциональное гиперметилирование генов ALDH1L1, PLCL2 и PPP2R3A при раке толстой кишки
А. А. Дмитриев a, *, А. Д. Бениаминов a, Н. В. Мельникова a, Е. Н. Пушкова a, А. В. Геращенко b, А. В. Кудрявцева a, В. И. Кашуба b, c
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Институт молекулярной биологии и генетики Национальной академии наук Украины
03680 Киев, Украина
c MTC, Biomedicum, Karolinska Institutet
17165 Stockholm, Sweden
* E-mail: alex_245@mail.ru
Поступила в редакцию 30.04.2019
После доработки 28.06.2019
Принята к публикации 28.06.2019
Аннотация
Гиперметилирование и мутации ДНК считаются ключевыми механизмами подавления экспрессии генов-супрессоров опухолевого роста. Использование NotI-микрочипов позволило нам выявить гиперметилирование и/или делеции в 180 NotI-сайтах, ассоциированных со 188 генами хромосомы 3 человека, в 24 парных (опухоль/норма) образцах толстой кишки. Наиболее частые нарушения (более чем в 20% опухолевых образцов) обнаружены в промоторных областях 20 генов. Экспрессию этих генов и метилирование их промоторов анализировали на данных проекта The Cancer Genome Atlas для парных образцов толстой кишки. Обнаружены отрицательная корреляция между уровнем мРНК и гиперметилированием промотора и более чем двукратное среднее снижение экспрессии трех генов – ALDH1L1, PLCL2, PPP2R3A. Экспрессию этих трех генов затем оцененивали в 30 парных образцах толстой кишки методом количественной ПЦР. Выявлено частое (более чем в 60% случаев) и значительное (в среднем в 5‒9 раз) снижение уровня мРНК каждого из генов в опухолевых образцах. Получены результаты, указывающие на супрессорную роль генов ALDH1L1, PLCL2 и PPP2R3A при раке толстой кишки, а также на функциональное значение гиперметилирования в подавлении экспрессии этих генов.
ВВЕДЕНИЕ
Рак толстой кишки (РТК) ‒ одно из наиболее распространенных злокачественных новообразований. Опухоль в толстой или прямой кишке развивается к 70 годам почти у половины людей, и в 10% случаев эта опухоль перерождается в злокачественную [1]. Перерождение нормальной клетки в опухолевую сопровождается многочисленными генетическими и эпигенетическими изменениями. Как и любые злокачественные опухоли, новообразования толстой кишки чрезвычайно гетерогенны. В последние годы с развитием полногеномных высокопроизводительных методов исследования сделано несколько попыток классифицировать злокачественные опухоли толстой кишки на основе профилей экспрессии мРНК или белков, уровня мутаций или нарушений биохимических путей [2, 3], а также связать молекулярно-генетические изменения с гистологическим типом РТК [4]. Традиционно же при описании РТК выделяют два типа генетических и эпигенетических нарушений. Хромосомная нестабильность (CIN) – наиболее распространенная (85%) форма генетических изменений при РТК, определяется по множественным хромосомным изменениям или структурным аберрациям кариотипа. Микросателлитная нестабильность (MSI) наблюдается в 15% злокачественных опухолей толстой кишки, которым свойственна инактивация генов репарации ДНК и высокий уровень мутаций в геноме. РТК этого типа часто ассоциирован с так называемым метиляторным фенотипом (CIMP), который характеризуется плотным гиперметилированием промоторных областей различных групп генов. При этом наблюдается и гипометилирование непромоторных участков генома, метилированных в норме. Многочисленные исследования позволили выявить несколько генов и сигнальных путей (WNT, TGF-β, RAS-MAPK, PI3K), нарушения в которых чаще всего приводят к развитию РТК [3, 5, 6].
Целью данного исследования был поиск генов хромосомы 3 человека с высокой частотой эпигенетических и/или генетических нарушений в опухолях толстой кишки. Для выявления таких генов использовано несколько независимых выборок парных образцов толстой кишки и несколько подходов: NotI-микрочипы для обнаружения гиперметилирования и делеций, количественная ПЦР и данные RNA-seq проекта The Cancer Genome Atlas (TCGA) для оценки уровня экспрессии генов. Выявлены новые потенциальные гены-супрессоры опухолевого роста, а также общие и специфичные нарушения в опухолях с различными патоморфологическими характеристиками, что может быть использовано для дифференциальной диагностики РТК.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Образцы опухолей. Образцы ткани РТК различных стадий (I‒IV) и парные к ним образцы гистологически нормальной ткани (норма) получали сразу после операции от пациентов Национального медицинского исследовательского центра онкологии им. Н.Н. Блохина, не получавших до этого лучевой терапии или химиотерапии, и хранили в жидком азоте. Диагноз подтверждали гистологически и отбирали образцы, содержащие не менее 70–80% опухолевых клеток. Образцы прилежащей к опухоли (парной) нормальной ткани также контролировали гистологическим исследованием. Опухоли были охарактеризованы в соответствии с Международной системой классификации, основанной на дифференциации по трем параметрам опухоль/лимфоузел/метастазирование (TNM) [7]. Все образцы тканей собраны с разрешения этического комитета НМИЦ онкологии им. Н.Н. Блохина.
Выделение и очистка ДНК и РНК. Геномную ДНК и суммарную клеточную РНК выделяли с использованием наборов “Qiagen” (Германия) согласно протоколам производителя. Чистоту и целостность ДНК и РНК контролировали электрофоретически, а также с использованием спектрофотометра NanoDrop 1000 (“NanoDrop Technologies”, США). Качество образцов РНК оценивали на приборе Bioanalyzer 2100 (“Agilent Technologies”, США). Концентрацию образцов ДНК и РНК определяли на спектрофотометре NanoDrop 1000 и флуориметре Qubit 2.0 (“Invitrogen”, США).
NotI-микрочипы. Технология изготовления NotI-микрочипов разработана в лаборатории Е. Забаровского и подробно описана ранее [8]. После обработки геномной ДНК из образцов толстой кишки рестриктазами NotI и Sau3AI, лигирования адаптеров и амплификации в присутствии dCTP-Cy5 (опухоль) или dCTP-Cy3 (норма) ДНК гибридизовали на микрочипе при 42°С в течение 15 ч. Микрочип сканировали на приборе GenePix 4000A (“Axon Instruments”, США) на двух длинах волн ‒ 670 и 570 нм. Данные экспортировали в разработанное нами ранее приложение NIMAN [9] для расчета относительного уровня сигнала.
Количественная ПЦР. Для определения уровня экспрессии генов проводили количественную ПЦР с использованием коммерческих наборов праймеров и TaqMan-проб: Hs01003842_m1 для ALDH1L1, Hs00392897_m1 для PLCL2, Hs00160407_m1 для PPP2R3A (“Thermo Fisher Scientific”, США). Каждую реакцию проводили в трех повторностях. Для нормирования использовали контрольные гены GUSB (Hs00939627_m1) и RPN1 (Hs00161446_m1) [10, 11]. Эффективность ПЦР (Е) c каждой парой праймеров и относительный уровень мРНК в образцах рассчитывали с помощью оригинального программного приложения ATG [12]. Логарифм изменения уровня мРНК в опухолевом образце относительно нормы вычисляли по формуле:
Значимыми считали изменения уровня мРНК в образцах РТК в два и более раз.
Оценка экспрессии генов методом количественной ПЦР выполнена на оборудовании ЦКП “Геном” ИМБ РАН (http://www.eimb.ru/rus/ckp/ ccu_genome_c.php).
Бисульфитное секвенирование. Бисульфитное секвенирование фрагментов промоторов генов LRRC3B, ITGA9, CTDSPL, содержащих NotI-сайты, проводили согласно [13, 14]. Для бисульфитной конверсии неметилированных остатков цитозина в геномной ДНК использовали набор EZ DNA Methylation Kit (“Zymo Research”, США) согласно инструкции производителя. После амплификации ДНК-фрагментов и ТА-клонирования продуктов ПЦР отдельные клоны анализировали на автоматическом секвенаторе ABI Prism 3100-Avant Genetic Analyzer (“Applied Biosystems”, США).
Статистический анализ данных. Статистическую значимость изменений уровня мРНК в образцах РТК оценивали с использованием непараметрического критерия Уилкоксона. Точный критерий Фишера применяли для анализа изменений уровня метилирования ДНК в группах образцов РТК разных клинических стадий. Корреляции между уровнями экспрессии и метилирования рассчитывали с использованием коэффициента корреляции Спирмена. Поправку на множественное сравнение осуществляли методом Бенджамини‒Хохберга. Результаты со значениями р меньше 0.05 считали статистически значимыми. При анализе и статистической обработке данных использовали программы: ATG [12], NIMAN [9], BioStat [15], CrossHub [16].
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Выявление гиперметилирования и/или делеций хромосомы 3 с помощью NotI-микрочипов
Генетические и эпигенетические нарушения хромосомы 3 при РТК анализировали с использованием технологии NotI-микрочипов, которая позволяет одновременно выявлять в геноме гиперметилирование промоторных CpG-островков и делеции – две основные причины подавления экспрессии генов-супрессоров [8, 13]. Анализ образцов геномной ДНК по этой методике проводят с помощью рестриктазы NotI, чувствительной к метилированию любого CpG-динуклеотида в восьминуклеотидной последовательности 5'-GCGGCCGC-3'. NotI-микрочип, использованный нами, содержал 180 ДНК-зондов, соответствующих 180 различным сайтам NotI-рестрикции хромосомы 3. Положение использованных в чипе NotI-сайтов хромосомы 3 определяли по версии hg19 генома человека. В непосредственной близости к NotI-сайтам выявили 188 генов. Определенный в результате этого исследования характер нарушений 180 NotI-сайтов в 24 образцах РТК представлен на рис. 1.
Рис. 1.
Характер эпигенетических и генетических нарушений хромосомы 3 в образцах рака толстой кишки. По вертикали последовательно расположены 180 NotI-сайтов хромосомы 3 (от 3p26.2 до 3p11.1 – левая панель; от 3q11.2 до 3q29 – правая панель). По горизонтали в порядке увеличения клинической стадии заболевания (от I к IV) расположены 24 парных образца рака толстой кишки. Зеленые квадраты обозначают гиперметилирование и/или делеции ДНК в опухолевой ткани по сравнению с нормой, красные – амплификацию и/или гипометилирование, желтые – отсутствие изменений, белые – неинформативные случаи.
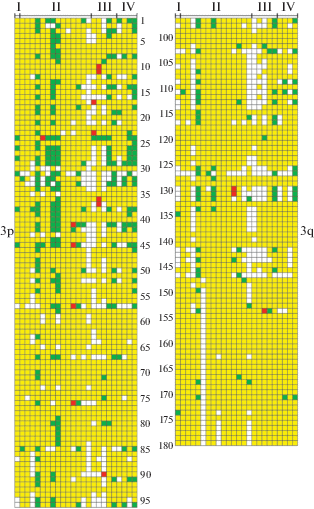
Из рис. 1 видно, что основной вариант нарушений ‒ гиперметилирование и/или делеции, а амплификации и/или гипометилирование представлены единичными случаями и не учитывались при дальнейшем анализе. Отдельные образцы характеризуются гиперметилированием и/или делециями одновременно многих Not-сайтов вдоль хромосомы 3. К ним можно отнести образцы РТК под номерами 5, 8, 9, 19, 20 и 24, нарушения в которых выявлены более чем в 20% NotI-сайтов. Естественно связать множественные нарушения в этих образцах с фенотипом CIMP [17]. Также видно (рис. 1), что фенотип CIMP не коррелирует со стадией РТК – встречается на II, III и IV стадиях заболевания.
Для определения генов, нарушения в которых могут быть ассоциированы с патоморфологическими характеристиками опухоли, сравнили частоты нарушений в образцах I‒II и III‒IV стадий РТК. В результате анализа найдены четыре NotI-сайта и пять ассоциированных с ними генов со статистически значимым различием в частоте гиперметилирования и/или делеций между стадиями I‒II и III‒IV РТК (табл. 1). Этот результат потенциально может быть использован для определения степени прогрессии заболевания.
Таблица 1.
Гены, частота гиперметилирования и/или делеций которых статистически значимо различаются при раке толстой кишки стадий I‒II и III‒IV
| Ген* | Номер NotI-сайта, рис. 1 |
Частота нарушений в образцах РТК I‒II стадий |
Частота нарушений в образцах РТК III‒IV стадий |
|---|---|---|---|
| ANKRD28 | 24 | 26.7 | 66.7 |
| IQSEC1 | 14 | 20.0 | 44.4 |
| NKIRAS1/RPL15 | 29 | 33.3 | 55.6 |
| ITGA9 | 41 | 33.3 | 55.6 |
Для поиска потенциальных генов-супрессоров опухолевого роста при РТК отбирали NotI-сайты, нарушения в которых выявлены в значительной части образцов РТК. Нарушения в 26 NotI-сайтах найдены более чем в 20% случаев. Из этих 26 NotI-сайтов отобраны те, которые попадали в промоторную область какого-либо гена хромосомы 3, т.е. локализовались в непосредственной близости от старта транскрипции гена (–1000…1000). В результате 18 таких NotI-сайтов найдены в промоторах 20 генов хромосомы 3 человека (два сайта были общими для пары транскрибируемых в противоположных направлениях генов: NKIRAS1/RPL15 и GORASP1/TTC21A). Список этих 20 генов, ранжированный по частоте гиперметилирования и/или делеций в соответствующем NotI-сайте в 24 образцах РТК, приведен в табл. 2. Указан номер NotI-сайта для каждого гена/пары генов и название ДНК-зонда на чипе, а также локализация NotI-сайта на хромосоме и расположение на соответствующем ему гене.
Таблица 2.
Гены с частотой гиперметилирования и/или делеций более 20% при раке толстой кишки
| Ген* | Номер NotI- сайта, рис. 1 |
NotI-зонд, название | Локализация NotI-сайта на хромосоме | Положение NotI-сайта относительно гена** | Частота нарушений |
|---|---|---|---|---|---|
| ANKRD28 | 24 | NL1G261R (D) | 3p25.1 | До СТ | 41.7 |
| NKIRAS1/RPL15 | 29 | NL1-CJ4R (C) | 3p24.2 | Экзон (оба гена) | 41.7 |
| ITGA9 | 41 | NL1A401R (D) | 3p21.3 | Интрон | 41.7 |
| CMTM6 | 38 | NRL063R (D) | 3p22.3 | Экзон | 37.5 |
| GORASP1/TTC21A | 45 | NL3003R (U) | 3p22–p21.33 | Экзон/интрон | 37.5 |
| LRRN1 | 1 | NL6-FJ5R (C) | 3p26.2 | Интрон | 33.3 |
| PLCL2 | 26 | NL4-AP18R (C) | 3p24.3 | До СТ | 33.3 |
| UBE2E2 | 28 | NR1-WF18R (C) | 3p24.2 | До СТ | 33.3 |
| LRRC3B | 33 | NL3-CA11RS | 3p24 | Экзон | 33.3 |
| IQSEC1 | 14 | NR1-XM13C | 3p25.2 | До СТ | 29.2 |
| HMGB1L5 | 27 | NL1-GC10C | 3p24 | Экзон | 29.2 |
| CTDSPL | 42 | NLJ-003RD | 3p21.3 | Интрон | 29.2 |
| NBEAL2 | 57 | NL3A006R (D) | 3p21.31 | Экзон | 25.0 |
| CGGBP1 | 96 | NR1-WE11RS | 3p12–p11.1 | Экзон | 25.0 |
| SNRK | 50 | NL3A009R (D) | 3p22.1 | Экзон | 25.0 |
| FOXP1 | 91 | NL1-BA6R | 3p14.1 | Интрон | 25.0 |
| ALDH1L1 | 111 | NL4-BC8R (C) | 3q21.3 | До СТ | 20.8 |
| PPP2R3A | 132 | NL1-FK10R (C) | 3q22.1 | Интрон | 20.8 |
Причиной нарушений, наблюдаемых на NotI-микрочипе, может быть либо метилирование соответствующего NotI-сайта, либо его мутация в опухолевом образце. Для проверки этого положения методом бисульфитного секвенирования нами протестированы два опухолевых образца. Короткие фрагменты промоторов генов LRRC3B, ITGA9 и CTDSPL (содержащие NotI-сайты с номерами 33, 41 и 42 соответственно) амплифицированы после бисульфитной конверсии геномной ДНК и клонированы в вектор. Секвенирование 10 клонов каждого гена – LRRC3B, ITGA9 и CTDSPL – выявило в двух опухолевых образцах уровень метилирования NotI-сайтов от 30 до 70%, в то время как уровень метилирования тех же NotI-сайтов в нормальных образцах не превышал 20%. При этом ни в одном из клонов не найдено мутаций (делеций или замен, кроме С на Т) в NotI-сайтах.
Гены, приведенные в табл. 2, отбирали как потенциальные гены-супрессоры опухолевого роста при РТК, действительно, среди них можно видеть такие хорошо известные гены-супрессоры, как CTDSPL и ITGA9 [18, 19]. Однако встречаются и гены, вовлеченность которых в канцерогенез толстой кишки ранее не была показана, например LRRN1, NBEAL2 и UBE2E2. В списке отобранных генов (табл. 2) выявляли наиболее вероятные кандидаты в гены-супрессоры, оценивая уровень их экспрессии при РТК, а также привлекая транскриптомные данные проекта TCGA.
Анализ экспрессии генов с высокой частотой нарушений NotI-сайтов при РТК
База данных проекта TCGA включает транскриптомные данные (секвенатор HiSeq, “Illumina”) тысяч опухолевых образцов более 30 видов рака. Для получения надежных результатов мы использовали только парные (опухоль/норма) образцы РТК. Анализ экспрессии 20 генов (из табл. 2), отобранных на NotI-микрочипах, провели с использованием приложения CrossHub и транскриптомных данных 26 парных образцов РТК, представленных в TCGA. Статистически значимое снижение в 2 и более раз показано только для трех генов: ALDH1L1 (медиана снижения – 3.4 раза), PLCL2 (4.3 раза), PPP2R3A (4.6 раза).
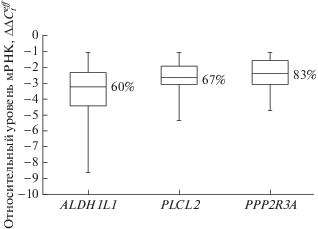
Подтверждение этого результата и его уточнение для генов ALDH1L1, PLCL2 и PPP2R3A выполнили методом количественной ПЦР на представительной выборке из 30 парных образцов РТК. Обнаружено значительное и частое снижение экспрессии каждого из этих генов: экспрессия ALDH1L1 снижалась в среднем в 9 раз в 60% (18/30) случаев, экспрессия PLCL2 – в среднем в 6 раз в 67% (20/30) случаев, а PPP2R3A – в среднем в 5 раз в 83% (25/30) случаев (рис. 2). Снижение уровня мРНК гена ALDH1L1 наблюдали реже, чем двух других генов, однако оно было наиболее значительным.
Рис. 2.
Изменение уровня мРНК генов ALDH1L1, PLCL2 и PPP2R3A при раке толстой кишки. Результаты количественной ПЦР для 30 парных (опухоль/норма) образцов толстой кишки. Прямоугольники соответствуют интервалу, содержащему 50% значений (между 25-м и 75-м процентилями); горизонтальная линия внутри прямоугольника ‒ медиана (50-й процентиль); “усы” – максимальное и минимальное значение ∆∆$C_{t}^{{{\text{eff}}}}$. Рядом с медианой указан процент опухолевых образцов, в которых уровень мРНК гена снижен в 2 и более раз (∆∆$C_{t}^{{{\text{eff}}}}$ < –1) по сравнению с нормой.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Использование NotI-микрочипов позволило определить профили эпигенетических и генетических нарушений хромосомы 3 человека в 24 парных образцах толстой кишки. Среди полученных профилей выделены образцы с одновременными множественными нарушениями вдоль хромосомы 3, свойственные фенотипу CIMP. Доля таких образцов в нашей выборке составляет примерно 20%, что соответствует встречаемости фенотипа CIMP при РТК [2, 6].
Выявлены частые нарушения в “промоторных” NotI-сайтах 20 генов хромосомы 3. Однако экспрессия только трех генов – ALDH1L1, PLCL2 и PPP2R3A – была значимо снижена в выборке из 26 парных образцов РТК проекта TCGA (данные RNA-seq). Этот результат методом количественной ПЦР подтвержден на нашей выборке из 30 парных образцов РТК. Таким образом, три отобранных гена представляют собой новые потенциальные гены-супрессоры опухолевого роста при РТК.
Продукт гена ALDH1L1, 10-формилтетрагидрофолатдегидрогеназа ‒ это высокоэкспрессируемый цитоплазматический фермент, участвующий в метаболизме фолата. Хотя супрессорная роль гена ALDH1L1 при раке не доказана, подавление его экспрессии наблюдали ранее во многих видах рака [20]. Кроме того, при раке молочной железы [21], гепатоцеллюлярной карциноме и аденокарциноме легкого [22] наблюдается гиперметилирование промотора ALDH1L1 и сильная корреляция между метилированием и подавлением экспрессии гена. Частое снижение экспрессии PLCL2 при раке почки [23] и шейки матки [13] показано ранее, однако отсутствуют данные о прямом участии гена PLCL2 или его продукта, белка, гомологичного фосфолипазе С, в процессах образования злокачественных опухолей. Продукт гена PPP2R3A – это регуляторная субъединица фосфатазы PP2А, гетеротримерного белка, состоящего из структурной, каталитической и регуляторной субъединицы. Изоформы регуляторной субъединицы комплекса разнообразны и определяют локализацию и специфическую активность холофермента. PP2A принимает участие в регуляции многочисленных процессов, включая апоптоз, пролиферацию клеток, ответ на повреждение ДНК, а инактивация фосфатазы РР2A обуславливает один из путей злокачественного перерождения клетки [24]. Данные о специфицеской роли изоформы PPP2R3A регуляторной субъединицы в канцерогенезе ограничены. Полученные нами результаты свидетельствуют о возможной роли изоформы PPP2R3A в злокачественной трансформации клеток толстой кишки.
Снижение экспрессии генов ALDH1L1, PLCL2 и PPP2R3A при РТК с одновременными частыми нарушениями NotI-сайтов в промоторах этих генов заставляют предположить, что основной механизм их инактивации связан с CpG-гиперметилированием промоторов. Частичную информацию о метилировании отдельных CpG-сайтов, включенных в микрочипы Infinium HumanMethylation450 BeadChip (“Illumina”), можно извлечь из результатов исследования проекта TCGA. На этих микрочипах представлено 25 CpG-сайтов, ассоциированных с геном ALDH1L1, 23 – с геном PLCL2 и 14 – с геном PPP2R3A. Проект TCGA содержит 16 парных образцов РТК, в которых одновременно определена экспрессия генов и метилирование ДНК. На этой выборке парных образцов мы рассчитали корреляцию между изменением экспрессии каждого из трех генов и изменением уровня метилирования каждого ассоциированного с ними CpG-сайта, представленного на микрочипе Illumina, с использованием приложения CrossHub.
Анализ данных TCGA выявил в промоторе каждого из генов – ALDH1L1, PLCL2, PPP2R3A – CpG-сайты, гиперметилирование которых сильно и статистически значимо коррелировало с экспрессией соответствующего гена (табл. 3). В гене PLCL2 единственный CpG-сайт с высоким коэффициентом корреляции локализован в начале первого интрона. Такие CpG-сайты найдены в промоторных областях генов ALDH1L1 и PPP2R3A в CpG-островках перед стартом транскрипции. Подробное изучение ассоциации метилирования CpG-динуклеотидов с экспрессией ALDH1L1 при раке молочной железы выявило наиболее значимые CpG-сайты в первом интроне [21], что указывает на отсутствие универсальных сценариев процесса гиперметилирования в различных видах злокачественных опухолей.
Таблица 3.
CpG-сайты, проявившие сильную корреляцию между уровнем метилирования и экспрессией генов ALDH1L1, PLCL2 и PPP2R3A при РТК
| Ген | CpG, номер | Положение CpG-сайтов относительно старта транскрипции | Коэффициент корреляции | p |
|---|---|---|---|---|
| ALDH1L1 | cg21601837 cg08481491 |
–100…0 п.н. | –0.67 –0.66 |
0.004 0.005 |
| PLCL2 | cg25223095 | 550 п.н. (интрон) | –0.65 | 0.006 |
| PPP2R3A | cg04907151 cg07038400 |
–400…–350 п.н. | –0.74 –0.70 |
0.001 0.002 |
Таким образом, совокупность полученных нами данных свидетельствует о супрессорной роли генов ALDH1L1, PLCL2 и PPP2R3A при РТК, а гиперметилирование промоторов этих трех генов можно рассматривать как основной механизм подавления их экспрессии при этом виде рака.
Работа выполнена при финансовой поддержке Российского научного фонда (грант № 17-74-20064).
Все процедуры, выполненные в данной работе, соответствуют этическим стандартам институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Kuipers E.J., Grady W.M., Lieberman D., Seufferlein T., Sung J.J., Boelens P.G., van de Velde C.J., Watanabe T. (2015) Colorectal cancer. Nat. Rev. Dis. Primers. 1, 15065.
The Cancer Genome Atlas Network (2012) Comprehensive molecular characterization of human colon and rectal cancer. Nature. 487, 330‒337.
Vogelstein B., Papadopoulos N., Velculescu V.E., Zhou S., Diaz L.A., Jr., Kinzler K.W. (2013) Cancer genome landscapes. Science. 339, 1546‒1558.
Alexander J., Watanabe T., Wu T.T., Rashid A., Li S., Hamilton S.R. (2001) Histopathological identification of colon cancer with microsatellite instability. Am. J. Pathol. 158, 527‒535.
Fearon E.R. (2011) Molecular genetics of colorectal cancer. Annu. Rev. Pathol. 6, 479‒507.
Kudryavtseva A.V., Lipatova A.V., Zaretsky A.R., Moskalev A.A., Fedorova M.S., Rasskazova A.S., Shibukhova G.A., Snezhkina A.V., Kaprin A.D., Alekseev B.Y., Dmitriev A.A., Krasnov G.S. (2016) Important molecular genetic markers of colorectal cancer. Oncotarget. 7, 53959‒53983.
Webber C., Gospodarowicz M., Sobin L.H., Wittekind C., Greene F.L., Mason M.D., Compton C., Brierley J., Groome P.A. (2014) Improving the TNM classification: findings from a 10-year continuous literature review. Int. J. Cancer. 135, 371‒378.
Li J., Protopopov A., Wang F., Senchenko V., Petushkov V., Vorontsova O., Petrenko L., Zabarovska V., Muravenko O., Braga E., Kisselev L., Lerman M.I., Kashuba V., Klein G., Ernberg I., Wahlestedt C., Zabarovsky E.R. (2002) NotI subtraction and NotI-specific microarrays to detect copy number and methylation changes in whole genomes. Proc. Natl. Acad. Sci. USA. 99, 10724‒10729.
Dmitriev A.A., Kashuba V.I., Haraldson K., Senchenko V.N., Pavlova T.V., Kudryavtseva A.V., Anedchenko E.A., Krasnov G.S., Pronina I.V., Loginov V.I., Kondratieva T.T., Kazubskaya T.P., Braga E.A., Yenamandra S.P., Ignatjev I., Ernberg I., Klein G., Lerman M.I., Zabarovsky E.R. (2012) Genetic and epigenetic analysis of non-small cell lung cancer with NotI-microarrays. Epigenetics. 7, 502‒513.
Krasnov G.S., Kudryavtseva A.V., Snezhkina A.V., Lakunina V.A., Beniaminov A.D., Melnikova N.V., Dmitriev A.A. (2019) Pan-cancer analysis of TCGA data revealed promising reference genes for qPCR normalization. Front. Genet. 10, 97.
Краснов Г.С., Опарина Н.Ю., Дмитриев А.А., Кудрявцева А.В., Анедченко Е.А., Кондратьева Т.Т., Забаровский Е.Р., Сенченко В.Н. (2011) Новый контрольный ген RPN1 для нормирования количественных данных при раке легкого и почки. Молекуляр. биология. 45, 238‒248.
Melnikova N.V., Dmitriev A.A., Belenikin M.S., Koroban N.V., Speranskaya A.S., Krinitsina A.A., Kra-snov G.S., Lakunina V.A., Snezhkina A.V., Sadritdinova A.F., Kishlyan N.V., Rozhmina T.A., Klimina K.M., Amosova A.V., Zelenin A.V., Muravenko O.V., Bolsheva N.L., Kudryavtseva A.V. (2016) Identification, expression analysis, and target prediction of flax genotroph microRNAs under normal and nutrient stress conditions. Front. Plant Sci. 7, 399.
Senchenko V.N., Kisseljova N.P., Ivanova T.A., Dmitriev A.A., Krasnov G.S., Kudryavtseva A.V., Panasenko G.V., Tsitrin E.B., Lerman M.I., Kisseljov F.L., Kashuba V.I., Zabarovsky E.R. (2013) Novel tumor suppressor candidates on chromosome 3 revealed by NotI-microarrays in cervical cancer. Epigenetics. 8, 409‒420.
Haraldson K., Kashuba V.I., Dmitriev A.A., Senchenko V.N., Kudryavtseva A.V., Pavlova T.V., Braga E.A., Pronina I.V., Kondratov A.G., Rynditch A.V., Lerman M.I., Zabarovsky E.R. (2012) LRRC3B gene is frequently epigenetically inactivated in several epithelial malignancies and inhibits cell growth and replication. Biochimie. 94, 1151‒1157.
Glantz S.A. (2005) Primer of Biostatistics. New York, USA: McGraw-Hill.
Krasnov G.S., Dmitriev A.A., Melnikova N.V., Zaretsky A.R., Nasedkina T.V., Zasedatelev A.S., Senc-henko V.N., Kudryavtseva A.V. (2016) CrossHub: a tool for multi-way analysis of The Cancer Genome Atlas (TCGA) in the context of gene expression regulation mechanisms. Nucl. Acids Res. 44, e62.
Toyota M., Ahuja N., Ohe-Toyota M., Herman J.G., Baylin S.B., Issa J.P. (1999) CpG island methylator phenotype in colorectal cancer. Proc. Natl. Acad. Sci. USA. 96, 8681‒8686.
Ghosh A., Ghosh S., Maiti G.P., Sabbir M.G., Zabarovsky E.R., Roy A., Roychoudhury S., Panda C.K. (2010) Frequent alterations of the candidate genes hMLH1, ITGA9 and RBSP3 in early dysplastic lesions of head and neck: clinical and prognostic significance. Cancer Sci. 101, 1511‒1520.
Бениаминов А.Д., Краснов Г.С., Дмитриев А.А., Пузанов Г.А., Снопок Б.А., Сенченко В.Н., Кашуба В.И. (2016) Взаимодействие двух супрессоров опухолевого роста – фосфатазы CTDSPL и белка Rb. Молекуляр. биология. 50, 504‒508.
Krupenko S.A., Krupenko N.I. (2018) ALDH1L1 and ALDH1L2 folate regulatory enzymes in cancer. Adv. Exp. Med. Biol. 1032, 127‒143.
Beniaminov A.D., Puzanov G.A., Krasnov G.S., Kaluzhny D.N., Kazubskaya T.P., Braga E.A., Kudryavtseva A.V., Melnikova N.V., Dmitriev A.A. (2018) Deep sequencing revealed a CpG methylation pattern associated with ALDH1L1 suppression in breast cancer. Front. Genet. 9, 169.
Oleinik N.V., Krupenko N.I., Krupenko S.A. (2011) Epigenetic silencing of ALDH1L1, a metabolic regulator of cellular proliferation, in cancers. Genes Cancer. 2, 130‒139.
Dmitriev A.A., Rudenko E.E., Kudryavtseva A.V., Krasnov G.S., Gordiyuk V.V., Melnikova N.V., Stakhovsky E.O., Kononenko O.A., Pavlova L.S., Kondratieva T.T., Alekseev B.Y., Braga E.A., Senchenko V.N., Kashuba V.I. (2014) Epigenetic alterations of chromosome 3 revealed by NotI-microarrays in clear cell renal cell carcinoma. BioMed. Res. Internat. 2014, 735292.
Mazhar S., Taylor S.E., Sangodkar J., Narla G. (2019) Targeting PP2A in cancer: Combination therapies. Biochim. Biophys. Acta. Mol. Cell Res. 1866, 51‒63.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


