Молекулярная биология, 2020, T. 54, № 2, стр. 224-232
Генетическая предрасположенность к инфаркту миокарда в разных возрастных группах
И. А. Гончарова a, b, *, М. С. Назаренко a, b, c, Н. П. Бабушкина a, А. В. Марков a, Т. Б. Печерина b, В. В. Кашталап b, Н. В. Тарасенко a, c, А. В. Понасенко b, О. Л. Барабараш b, В. П. Пузырёв a, c
a Научно-исследовательский институт медицинской генетики Томского национального исследовательского медицинского центра
634050 Томск, Россия
b Научно-исследовательский институт комплексных проблем сердечно-сосудистых заболеваний
650002 Кемерово, Россия
c Сибирский государственный медицинский университет
634050 Томск, Россия
* E-mail: irina.goncharova@medgenetics.ru
Поступила в редакцию 09.10.2019
После доработки 09.11.2019
Принята к публикации 14.11.2019
Аннотация
Цель исследования заключалась в выявлении особенностей генетической структуры подверженности инфаркту миокарда (ИМ) в зависимости от возраста пациентов. Всего обследовано 355 пациентов (n = 121, ранний ИМ ‒ индивиды, у которых первый ИМ зарегистрирован в возрасте до 60 лет; и n = 234 c первым ИМ в возрасте старше 60 лет) и 285 жителей Сибирского региона (контрольная группа). Генотипирование 58 однонуклеотидных вариантов (SNP) выполнено с помощью MALDI-TOF масс-спектрометрии. Регуляторный потенциал SNP оценивали с помощью онлайн сервисов (http://rsnp.psych.ac.cn/; http://www.gtexportal.org/; https://genenetwork.nl/bloodeqtlbrowser/). Генотипы GG rs1143674 гена ITGA4, СС rs1333049 гена CDKN2B-AS1 и СС rs3739998 гена KIAA1462 ассоциированы с ИМ в общей группе больных. Генотипы АА гена ADAMDEC1 (OR = 2.03; 95% CI 1.23‒3.33; p = 0.004) и GG rs2878771 гена AQP2 (OR = 2.24; 95% CI 1.23‒4.09; p = 0.006) связаны с развитием ИМ в раннем возрасте, а генотип ТТ гена TAS2R38 (OR = 1.82; 95% CI 1.11‒2.89; p = 0.009) был фактором риска первого ИМ, произошедшего после 60 лет. Генетические варианты, ассоциированные с ИМ, относятся к регуляторным (rSNP): влияют на аффинность связывания ДНК с транскрипционными факторами, вовлечены в посттранскрипционный контроль генной активности и модулируют уровень экспрессии генов в различных тканях. Ранний и поздний ИМ имеют в своей основе как общие генетические варианты генов ITGA4, CDKN2B-AS1, KIAA1462, так и специфичные: ADAMDEC1 и AQP2 для раннего ИМ и TAS2R38 для ИМ, произошедшего после 60 лет.
ВВЕДЕНИЕ
Несмотря на значительный прогресс в разработке стратегий, направленных на снижение смертности от сердечно-сосудистых заболеваний, частота первого инфаркта миокарда (ИМ), возникшего в раннем возрасте, увеличивается. Группу повышенного риска развития раннего ИМ составляют, прежде всего, индивиды с наследственной предрасположенностью. Используемые в практике генетические панели в основном выявляют редкие мутации в генах LDLR, APOB и PCSK9, приводящие к развитию семейной гиперхолестеринемии и повышающие риск сердечно-сосудистых заболеваний и ИМ в 3‒18 раз [1].
Многолетний опыт проведения широкогеномных ассоциативных исследований (GWAS) продемонстрировал, что полигенное наследование распространенных генетических вариантов с небольшим эффектом составляет существенную часть риска развития многофакторных заболеваний. На основании данных GWAS разработана полигенная шкала, которая позволяет рассчитать риск развития воспалительных заболеваний кишечника, рака молочной железы, раннего ИМ, фибрилляции предсердий и сахарного диабета 2 типа [2]. Показано, что в группе больных с ранним ИМ высокий полигенный индекс (наследование многих генетических вариантов с небольшим эффектом) встречается в 10 раз чаще, чем моногенные формы семейной гиперхолестеринемии [3].
Исследования GWAS выявили около 50 локусов, ассоциированных с ишемической болезнью сердца (ИБС) и ИМ [4, 5]. Однако работ, посвященных изучению генетической подверженности ИМ как отдельного фенотипа, проведено значительно меньше [6]. В исследованиях GWAS связь с ранним ИМ выявлена для 9 локусов: 1p32 (PCSK9), 1p13 (CELSR2-PSRC1-SORT1), 2q33 (WDR12), 6p24 (PHACTR1), 9p21 (CDKN2B, CDKN2A, MTAP, ANRIL), 10q11 (CXCL12), 1q41 (MIA3), 19p13 (LDLR) и 21q22 (MRPS6-SLC5A3-KCNE2) [7 ] . В России анализ ассоциаций в отношении первого ИМ, возникшего в раннем возрасте, выполнен лишь в нескольких работах с использованием кандидатного подхода [8, 9].
Таким образом, выявление особенностей генетической структуры подверженности ИМ в зависимости от возраста остается актуальным, что и стало целью нашего исследования. Особенность работы заключалась в анализе ассоциации генетического полиморфизма с ИМ в общей группе больных без разделения по возрасту, а также в отдельных подгруппах: ранний ИМ (индивиды, у которых первый ИМ зарегистрирован в возрасте до 60 лет) и поздний ИМ (пациенты с первым ИМ после 60 лет).
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Группа пациентов с ИМ (n = 355, 221 мужчина и 134 женщины, возраст 58.9 ± 8.8 лет) сформирована из индивидов, поступивших в Кемеровский кардиологический диспансер по поводу острого коронарного синдрома. Эту группу разделили на 2 подгруппы: “ранний ИМ” (первый ИМ в возрасте до 60 лет; n = 121, 67 мужчин и 54 женщины) и “поздний ИМ” (первый ИМ в возрасте после 60 лет; n = 234, 154 мужчины и 80 женщин). В качестве контрольной группы использована выборка, состоящая из жителей Сибирского региона (n = 285, 154 мужчины и 131 женщина, возраст 56.7 ± 10.1 лет). По этнической принадлежности все обследованные индивиды были русскими. Все индивиды подписали информированное согласие на добровольное участие в исследовании. Протокол исследования был одобрен Этическими комитетами участвующих в исследовании учреждений.
В работе проанализировано 58 однонуклеотидных полиморфных вариантов (SNP), локализованных в генах, белковые продукты которых вовлечены в различные биохимические пути, в том числе в процессы фиброгенеза. Для генотипирования выбраны известные SNP, которые ассоциированы, согласно данным литературы, с атеросклерозом и нестабильностью атеросклеротической бляшки, эндотелиальной дисфункцией, а также полиморфизм генов, для которых выявлены изменения уровней экспрессии при заболеваниях, связанных с фиброзом различных органов [10].
Генотипирование выполняли методом масс-спектрометрии на приборе Sequenom MassARRAY® (США). Полиморфные варианты, распределение частот генотипов которых не соответствовало ожидаемому при равновесии Харди–Вайнберга (rs17181457, rs9325154, rs12979860), были исключены из дальнейшего анализа.
Для статистического анализа данных применяли следующее программное обеспечение: Statistica 8.0 (“StatSoft Inc.”, США), а также пакеты Stats и Genetics в программной среде R (The R Foundation). Анализ различий по частотам аллелей и генотипов проводили с использованием критерия χ2 или точного теста Фишера между популяционным контролем и группой больных с ИМ, а также подгрупп с ранним ИМ и поздним ИМ. Статистические гипотезы при сравнительном анализе данных проверяли на 5%-ном уровне значимости.
Для оценки генетических различий между сравниваемыми группами проведен расчет генетических расстояний по Nei на основании различий в частотах аллелей генетических вариантов. Для визуализации генетических различий проведено преобразование матрицы генетических расстояний с помощью метода главных координат РСоА (надстройка GenAIEx 6.503 к MS Office Exсel) [11].
Регуляторный потенциал изученных SNP оценивали с помощью онлайн сервиса rSNPBase (http://rsnp.psych.ac.cn/). Связь полиморфизма с количественными изменениями в профиле экспрессии генов (eQTL) в различных тканях выявляли с помощью данных проекта Genotype-Tissue Expression (GTEx, http://www.gtexportal.org/) и онлайн сервиса Blood eQTL (https://genenetwork.nl/bloodeqtlbrowser/).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
С риском развития ИМ ассоциированы генотипы GG гена ITGA4 (rs1143674), СС гена CDKN2B-AS1 (rs1333049), СС гена KIAA1462 (rs3739998), AA гена ADAMDEC1 (rs3765124), GG гена AQP2 (rs2878771) и TT гена TAS2R38 (rs1726866) (табл. 1). Варианты генов ITGA4, CDKN2B-AS1, KIAA1462 и ADAMDEC1 ассоциированы с ИМ в общей группе больных. При разделении пациентов на подгруппы в зависимости от возраста первого ИМ выявлено, что с ранним ИМ связаны варианты генов ITGA4, CDKN2B-AS1, KIAA1462, ADAMDEC1 и AQP2. В то же время с ИМ, возникшем в возрасте старше 60 лет, ассоциирован полиморфизм генов ITGA4, CDKN2B-AS1, KIAA1462 и TAS2R38, причем для гена CDKN2B-AS1 показана ассоциация аллеля С, тогда как частоты генотипа СС не достигли статистически значимых различий между сравниваемыми подгруппами.
Таблица 1.
Ассоциация полиморфных вариантов генов ITGA4, CDKN2B-AS1, KIAA1462, ADAMDEC1, AQP2 и TAS2R38 с инфарктом миокарда
| Ген (SNP ID) |
MAF европейцы (gnomAD ncbi)a | Генотип, ассоцииро-ванный с ИМ | Частота генотипа в группахb | ||
|---|---|---|---|---|---|
| контроль/ИМ (%) | контроль/ранний ИМ (%) | контроль/поздний ИМ (%) | |||
| ITGA4 (rs1143674: A>G) синонимичный |
0.44 A | GG | 22.2/36.9 2.05 (1.39‒3.01); 0.0001 |
22.2/42,7 2.60 (1.45‒4.67); 0.008 |
22.2/35.3 1.91 (1.19‒3.07); 0.006 |
| CDKN2B-AS1 (rs1333049: G>C) |
0.47 C | СС | 17.3/27.8 1.85 (1.24‒2.75); 0.002 |
17.3/33.3 2.40 (1.34‒4.11); 0.001 |
44.8/51.1с 1.32 (1.01‒1.73); 0.039 |
| KIAA1462 (rs3739998: G>C) миссенс |
0.44 G | СС | 15.9/25.8 1.83 (1.23‒2.74); 0.002 |
15.9/28.7 2.13 (1.22‒3.70); 0.006 |
15.9/27.6 1.96 (1.23‒3.14); 0.003 |
| ADAMDEC1 (rs3765124: A>G) миссенс |
0.43 G | АА | 24.3/34.4 1.63 (1.15‒2.31); 0.005 |
24.3/39.5 2.03 (1.23‒3.33); 0.004 |
– |
| AQP2 (rs2878771: G>C) 3'UTR AQP2 интрон LOC101927318 |
0.18 C | GG | – | 61.6/77.45 2.24 (1.23‒4.09); 0.006 |
– |
| TAS2R38 (rs1726866: T>C) миссенс |
0.44 C | TT | – | – | 28.1/41.5 1.82 (1.11‒2.89); 0.009 |
ahttps://gnomad.broadinstitute.org/. bУказаны по порядку: отношение шансов (95%-ный доверительный интервал); уровень значимости. cЧастота аллеля C.
Таким образом, гены ITGA4, CDKN2B-AS1 и KIAA1462 можно назвать общими относительно риска развития ИМ (независимо от возраста возникновения первого события). Гены ADAMDEC1, AQP2 и TAS2R38 можно условно отнести к “специфическим”, поскольку для них получены возрастзависимые ассоциации: гены ADAMDEC1 и AQP2 связаны с риском ИМ в раннем возрасте, а ген TAS2R38 ассоциирован с развитием ИМ в возрасте 60 лет и старше (табл. 1).
В результате анализа генетических взаимоотношений между изученными выборками по 58 SNP показано, что данный массив разделяется на три группы в соответствии со статусом заболевания: ранний ИМ, поздний ИМ, популяционный контроль (рис. 1). Первые две основные оси дифференциации (координата 1 – различия между больными и контролем; координата 2 – различия между больными с ИМ, возникшем в разном возрасте) объясняют 91.55 и 8.45% от общей генетической вариабельности по изученным SNP соответственно. При поочередном включении в анализ сначала всех SNP, ассоциированных с ИМ, затем трех “общих” и затем трех “специфичных”, показано, что первая ось дифференциации объясняет соответственно 83.3, 99.6 и 57.6% от общей вариабельности; вторая ось дифференциации ‒ 16.7, 0.4 и 42.4% соответственно.
Рис. 1.
Межгрупповая генетическая дифференциация для выборок (ранний ИМ; поздний ИМ; популяционный контроль) по 58 SNP. В скобках указан процент объясняемой главными координатами генетической вариабельности по данным SNP.

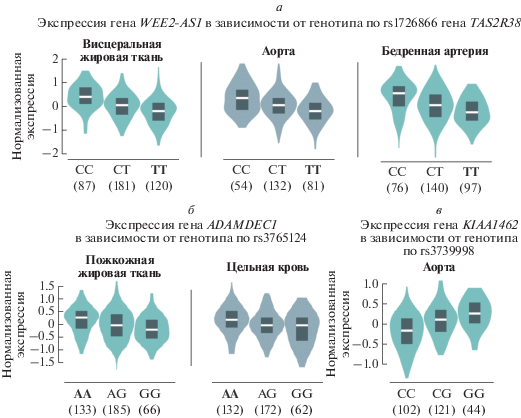
Варианты генов ITGA4, CDKN2B-AS1, KIAA1462, ADAMDEC1 и AQP2 относятся к регуляторным SNP (rSNP) и сцеплены с другими регуляторными полиморфными вариантами (табл. 2). Связь SNP c экспрессией генов в разных тканях показана для полиморфизмов генов KIAA1462, ADAMDEC1, AQP2 и TAS2R38. Более того, в большеберцовой артерии, в аорте и в висцеральной жировой ткани (сальнике) генотип TT гена TAS2R38 (rs1726866) ассоциирован со снижением экспрессии гена WEE2-AS1, в аорте генотип СС гена KIAA1462 (rs3739998) связан со снижением экспрессии собственного гена, а в крови и в висцеральной жировой ткани (сальнике) генотип AA гена ADAMDEC1 (rs3765124) ассоциирован с увеличением экспрессии собственного гена (рис. 2). При анализе связи SNP с количественными изменениями в экспрессионном профиле генов в клетках крови показано, что rs3765124 (ADAMDEC1) оказывает влияние на экспрессию генов ADAMDEC1 и ADAM28, rs2878771 (AQP2) – на LASS5, а rs1726866 (TAS2R38) – на TAS2R5 и CLEC5A.
Таблица 2.
Потенциальная функциональная значимость полиморфных вариантов генов ITGA4, CDKN2B-AS1, KIAA1462, ADAMDEC1, AQP2 и TAS2R38
| Ген (SNP ID) | Регуляторный SNP | eQTLe | |||
|---|---|---|---|---|---|
| собственно регуляторный или сцеплен с другим rSNPa | проксимальная регуляцияb | дистальная регуляцияc | регуляция посредством РНК-связывающих белковd | ||
| ITGA4 (rs1143674) |
+ | – | – | + | – |
| CDKN2B-AS1 (rs1333049) |
+ | – | + | – | – |
| KIAA1462 (rs3739998) |
+ | – | – | + | + |
| ADAMDEC1 (rs3765124) | + | + | – | + | + |
| AQP2 (rs2878771) | + | + | + | – | + |
| TAS2R38 (rs1726866) |
– | – | – | – | + |
a Локусы находятся в неравновесии по сцеплению с другими регуляторными локусами (коэффициент корреляции Пирсона r2 > 0.8). bSNP локализуются в сайте связывания транскрипционных факторов, когда ген экспрессируется с проксимального элемента промотора. cSNP локализуются в сайте связывания транскрипционных факторов, когда ген экспрессируется с дистального элемента промотора. dSNP локализуются в сайте связывания белков ELAVL1 и ABPC1, осуществляющих посттранскрипционный контроль экспрессии генов, с мРНК. eSNP влияют на экспрессию генов в разных тканях.
Рис. 2.
Влияние изученных eQTL-локусов на экспрессию генов в различных тканях. Связь полиморфизма TAS2R38 (rs1726866) с экспрессией гена WEE2-AS1 в большеберцовой артерии, аорте и висцеральной жировой ткани (сальнике) (а); связь полиморфизма ADAMDEC1 (rs3765124) с экспрессией собственного гена в крови и подкожной жировой ткани (б); связь полиморфизма гена KIAA1462 (rs3739998) с экспрессией собственного гена в аорте (в). Данные получены из портала проекта Genotype-Tissue Expression (GTEx, http://www.gtexportal.org/) и представлены в виде нормализованных значений уровня экспрессии (методы оценки и нормализации уровня экспрессии генов описаны в разделе “Документация” на сайте портала). Цифры в скобках ‒ число носителей различных генотипов.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Расчет генетических расстояний ‒ один из способов доказательства того, что выбранные для анализа генетические варианты связаны с развитием ИМ и могут дифференцировать ИМ, как по статусу заболевания, так и по возрасту возникновения первого ИМ. По статусу заболевания группы лучше всего дифференцируются на основании анализа “общих” ассоциированных полиморфизмов: гены ITGA4 (rs1143674), CDKN2B-AS1 (rs1333049), KIAA1462 (rs3739998), ‒ так как первая ось дифференциации (различия между больными и здоровыми) объясняет 99.6% от общей генетической вариабельности. Максимальные генетические различия между больными с ранним и поздним ИМ выявляют на основании анализа специфичных SNP: гены ADAMDEC1 (rs3765124), AQP2 (rs2878771), TAS2R38 (rs1726866), ‒ которые объясняют 42.4% от общей генетической вариабельности.
Результаты исследования показали, что с ИМ (независимо от возраста возникновения первого события) ассоциированы генотипы GG гена ITGA4 (rs1143674), СС гена CDKN2B-AS1 (rs1333049) и СС гена KIAA1462 (rs3739998).
Связь с ИМ синонимичного варианта rs1143674 гена ITGA4 ранее не была показана. В исследованиях GWAS полиморфизм этого гена ассоциирован с отношением “моноциты/лимфоциты”, количеством циркулирующих в крови моноцитов, а также с аутоиммунными заболеваниями [12]. Ген ITGA4 кодирует мембранный белок – субъединицу альфа-4 интегрина VLA-4 (CD49d), который экспрессируется в моноцитах, лимфоцитах, эндотелиальных клетках, эритроцитах и выполняет роль рецептора адгезии для VCAM-1 и фибронектина. Белок VLA-4 участвует в гематопоэзе, развитии сердечно-сосудистой системы, а также в процессе воспаления при атеросклерозе [13].
Генотип СС полиморфизма rs1333049, предрасполагающий к ИМ по результатам нашего исследования, связан с ИБС или ИМ во многих популяциях мира, в том числе в России [4, 14]. Известно, что генотип CC также ассоциирован с ранней манифестацией ИБС [15]. Полиморфизм rs1333049 располагается в межгенном регионе CDKN2B, CDKN2A, MTAP и ANRIL (CDKN2B-AS1) в локусе 9p21.3. Генетические варианты этого локуса связаны с изменением экспрессии длинной некодирующей РНК (ANRIL) в клетках крови и атеросклеротических бляшек. Предполагается, что при развитии атеросклероза дифференциальная экспрессия ANRIL через регуляцию активности многих генов, в том числе близлежащих генов клеточного цикла, изменяет пролиферацию и апоптоз гладкомышечных клеток сосудов, ремоделирование внеклеточного матрикса, ответ на воспаление [16]. Связь rs1333049 с риском ИМ может быть обусловлена не разрывом атеросклеротической бляшки/тромбозом, а ее прогрессией с поражением нескольких коронарных артерий, нарастанием степени тяжести атеросклероза и собственно ИБС [6].
Коморбидность пациента, по-видимому, может модулировать ассоциацию между SNP и фенотипами сердечно-сосудистых заболеваний. Ранее нами показано [17], что аллель C/генотип СС, наоборот, ассоциирован с протективным действием в отношении сочетания ИБС с артериальной гипертензией, сахарным диабетом 2 типа и гиперхолестеринемией (заболевания сердечно-сосудистого континуума). Генетические варианты локуса 9p21.3, действительно, могут быть связаны с сердечно-сосудистыми заболеваниями опосредованно ‒ через метаболические нарушения. Показано, что полиморфизм этого локуса ассоциирован со снижением экспрессии гена CDKN2B в подкожной жировой ткани, ингибированием адипогенеза и нарушением способности жировой ткани увеличивать объем, что приводит к ее эктопической аккумуляции в органах [18].
SNP в локусе 9p21.3 тесно сцеплены друг с другом, поэтому наиболее информативным считается анализ не одного варианта, а нескольких, образующих гаплоблок. Генетические варианты в разных группах сцепления, реализуясь в контекстзависимой и тканеспецифичной манере, могут участвовать в разных молекулярных процессах (проатерогенный, или протромботический с увеличением реактивности тромбоцитов, или дисбаланс формирования эндотелиальной трубки, неоваскуляризация) и соответственно быть связаны с разными клиническими фенотипами сердечно-сосудистых патологий (возраст манифестации и тяжесть ИСБ/ИМ) [6, 19].
Белок межклеточных взаимодействий, ассоциированный с кадгерином 5, – KIAA1462 ‒ участвует в формировании цитоскелета эндотелиальных клеток, регулирует целостность и проницаемость эндотелия, что, в свою очередь, влияет на скорость миграции моноцитов, которые трансформируются в макрофаги при развитии атеросклеротических бляшек. В экспериментальных исследованиях выявлена связь белка KIAA1462 с ИБС, после чего он был переименован в JCAD (junctional protein associated with coronary artery disease) [20]. Ассоциация rs3739998 (аллель C) гена KIAA1462 с ИМ, выявленная нами, ранее показана в исследовании GWAS [21]. Согласно базе данных GTEx, генотип СС по сравнению с другими генотипами ассоциирован со снижением экспрессии гена KIAA1462 в аорте. Не исключено, что этот вариант имеет функциональную значимость в отношении ИМ, косвенным подтверждением чего служит участие JCAD в патологическом ангиогенезе, приводящем к нестабильности атеросклеротической бляшки [22].
Нами выявлено, что генотипы AA гена ADAMDEC1 (rs3765124) и GG гена AQP2 (rs2878771) связаны с риском ИМ у пациентов в возрасте до 60 лет. Ген ADAMDEC1 входит в состав кластера генов ADAM7 и ADAM28 в локусе 8p12. В висцеральной жировой ткани генотип AA rs3765124 гена ADAMDEC1 ассоциирован с увеличением экспрессии собственного гена, а в клетках крови rs3765124 связан с экспрессией не только собственного гена, но и ADAM28. Экспрессия гена ADAMDEC1 значительно повышается в нестабильных регионах атеросклеротической бляшки по сравнению со стабильными областями [23]. Кроме того, вариант rs2878771 входит в состав гаплотипа, который ассоциирован с повышением уровня FVIII при венозной тромбоэмболии [24]. Однако высокий уровень FVIII может быть связан также с артериальным тромбозом при ИБС и инсульте [25].
Функциональная значимость обнаруженной ассоциации полиморфизма rs2878771 гена AQP2 с патологией неясна. Этот SNP относится к cis-eQTL-локусам и связан с изменением уровня экспрессии гена LASS5 (CERS5) в клетках крови. Продукт гена LASS5 изменяет синтез церамидов в эндотелиальных клетках через путь AMP-активируемой протеинкиназы-α1, что может быть важным звеном патогенеза атеросклероза [26]. Учитывая важную роль нарушения метаболизма липидов в формировании предрасположенности к более ранней манифестации ИБС в виде ИМ, логично предположить, что выявленная ассоциация неслучайна.
Среди генов, определяющих подверженность к ИМ у индивидов старше 60 лет, находится ген TAS2R38, который кодирует рецептор восприятия горечи. Нами установлено, что генотип TT rs1726866 связан с патологическим фенотипом. Более того, в предыдущем исследовании нашего коллектива аллель T/генотип TT увеличивал риск сочетания ИБС с артериальной гипертензией, сахарным диабетом 2 типа и гиперхолестеринемией (заболевания сердечно-сосудистого континуума) [17].
Вовлеченность вкусовых рецепторов в патогенез ИМ может быть связана со специфическим вкусовым восприятием, формирующим несбалансированную диету (употребление алкоголя, курение, пища с избытком углеводов и жиров), которая выступает как фактор риска развития заболеваний сердечно-сосудистого континуума. Накапливаются данные о том, что вкусовые рецепторы регулируют высвобождение нейротрансмиттеров и гормонов, участвующих в метаболизме жиров, что подтверждает связь между восприятием вкуса и ожирением [27, 28].
Кроме того, показано, что в большеберцовой артерии, аорте и висцеральной жировой ткани генотип TT гена TAS2R38 (rs1726866) ассоциирован со снижением экспрессии гена некодирующей РНК (WEE2-AS1), а в клетках крови выявлена связь данного варианта с экспрессией генов TAS2R5 и CLEC5A. Участие в атеросклерозе и его осложнениях наиболее хорошо установлено для рецептора лектина C-типа MDL1 (CLEC5A), который экспрессируется в моноцитах и служит сигнальным рецептором для освобождения провоспалительных цитокинов. Экспрессия гена CLEC5A выше в макрофагах пациентов с ИМ, которые были носителями рискового гаплотипа по локусу 9p21.3, включающему аллель С (rs1333049) [29]. Белок MDL-1 также обнаружен в макрофагах (особенно M1-типа) атеросклеротических бляшек. Изменение экспрессии MDL-1 в этих клетках сопровождалось изменениями содержания макрофагов в бляшке, а увеличение экспрессии MDL-1 коррелировало со снижением апоптоза макрофагов при стимуляции высокими дозами окисленных липопротеинов низкой плотности [30].
ИМ относится к сложным многофакторным заболеваниям, генетическая подверженность к нему определяется многими генетическими вариантами. В результате анализа генетических взаимоотношений между исследованными группами на основании изученных 58 SNP показано, что группы больных с ранним ИМ, более поздним ИМ и контроля характеризуются различной генетической структурой. Генетические варианты rs1143674 гена ITGA4, rs1333049 гена CDKN2B-AS1 и rs3739998 гена KIAA1462 ассоциированы с ИМ в общей группе больных, а rs3765124 гена ADAMDEC1, rs1726866 гена TAS2R38 и rs2878771 гена AQP2 связаны с ИМ в зависимости от возраста пациентов (до 60 лет и старше 60 лет).
Полиморфизм в генах CDKN2B-AS1 и KIAA1462 ассоциирован с ИМ в исследованиях GWAS, проведенных ранее. Генетические варианты, формирующие предрасположенность к ИМ, относятся к регуляторным или сцеплены с другими регуляторными SNP, а также связаны с изменениями в профилях экспрессии генов в различных тканях. Из этого можно сделать вывод об их функциональной значимости для формирования патологического фенотипа. Однако учитывая, что все пациенты c ИМ в нашем исследовании были с ИБС и другими коморбидными заболеваниями сердечно-сосудистого континуума, в основе выявленной ассоциации SNP и фенотипа может быть не собственно нестабильность атеросклеротической бляшки или тромбоз, а опосредованное влияние через патогенетические звенья атеросклероза и его факторов риска.
Работа проведена в рамках выполнения Государственного задания Министерства науки и высшего образования № 075-00603-19-00 (Федеральное государственное бюджетное научное учреждение “Томский национальный исследовательский медицинский центр Российской академии наук”).
Все процедуры, выполненные в данной работе, соответствуют этическим стандартам институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Bruikman C.S., Hovingh G.K., Kastelein J.J.P. (2017) Molecular basis of familial hypercholesterolemia. Curr. Opin. Cardiol. 32(3), 262–266. https://doi.org/10.1097/HCO.0000000000000385
Khera A.V., Chaffin M., Aragam K.G., Haas M.E., Roselli C., Choi S.H., Natarajan P., Lander E.S., Lubitz S.A., Ellinor P.T., Kathiresan S. (2018) Genome-wide polygenic scores for common diseases identify individuals with risk equivalent to monogenic mutations. Nat. Genet. 50, 1219–1224.
Khera A.V., Chaffin M., Zekavat S.M., Collins R.L., Roselli C., Natarajan P., Lichtman J.H., D’Onofrio G., Mattera J., Dreyer R., Spertus J.A., Taylor K.D., Psaty B.M., Rich S.S., Post W., Gupta N., Gabriel S., Lander E., Chen Y-D.I., Talkowski M.E., Rotter J.I., Krumholz H.M., Kathiresan S. (2019) Whole genome sequencing to characterize monogenic and polygenic contributions in patients hospitalized with early-onset myocardial infarction. Circulation. 139, 1593–1602.
Dehghan A., Bis J.C., White C.C., Smith A.V., Morrison A.C., Cupples L.A., Trompet S., Chasman D.I., Lumley T., Völker U., Buckley B.M., Ding J., Jen-sen M.K., Folsom A.R., Kritchevsky S.B., Girman C.J., Ford I., Dörr M., Salomaa V., Uitterlinden A.G., Eiriksdottir G., Vasan R.S., Franceschini N., Carty C.L., Virtamo J., Demissie S., Amouyel P., Arveiler D., Heckbert S.R., Ferrières J., Ducimetière P., Smith N.L., Wang Y.A., Siscovick D.S., Rice K.M., Wiklund P.G., Taylor K.D., Evans A., Kee F., Rotter J.I., Karvanen J., Kuulasmaa K., Heiss G., Kraft P., Launer L.J., Hofman A., Markus M.R., Rose L.M., Silander K., Wagner P., Benjamin E.J., Lohman K., Stott D.J., Rivadeneira F., Harris T.B., Levy D., Liu Y., Rimm E.B., Jukema J.W., Völzke H., Ridker P.M., Blankenberg S., Franco O.H., Gudnason V., Psaty B.M., Boerwinkle E., O’Donnell C.J. (2016) Genome-wide association study for incident myocardial infarction and coronary heart disease in prospective cohort studies: the CHARGE Consortium. PLoS One. 11, e0144997.
McPherson R., Tybjaerg-Hansen A. (2016) Genetics of coronary artery disease. Circ. Res. 118, 564–578.
Fan M., Dandona S., McPherson R., Allayee H., Hazen S.L., Wells G.A., Roberts R., Stewart A.F. (2013) Two chromosome 9p21 haplotype blocks distinguish between coronary artery disease and myocardial infarction risk. Circ. Cardiovasc. Genet. 6, 372–380.
Myocardial Infarction Genetics Consortium, Kathiresan S., Voight B.F., Purcell S., Musunuru K., Ardissino D., Mannucci P.M., Anand S., Engert J.C., Samani N.J., Schunkert H., Erdmann J., Reilly M.P., Rader D.J., Morgan T., Spertus J.A., Stoll M., Girelli D., McKeown P.P., Patterson C.C., Siscovick D.S., O’Donnell C.J., Elosua R., Peltonen L., Salomaa V., Schwartz S.M., Melander O., Altshuler D., Ardissino D., Merlini P.A., Berzuini C., Bernardinelli L., Peyvandi F., Tubaro M., Celli P., Ferrario M., Fetiveau R., Marziliano N., Casari G., Galli M., Ribichini F., Rossi M., Bernardi F., Zonzin P., Piazza A., Mannucci P.M., Schwartz S.M., Siscovick D.S., Yee J., Friedlander Y., Elosua R., Marrugat J., Lucas G., Subirana I., Sala J., Ramos R., Kathiresan S., Meigs J.B., Williams G., Nathan D.M., MacRae C.A., O’Donnell C.J., Salomaa V., Havulinna A.S., Peltonen L., Melander O., Berglund G., Voight B.F., Kathiresan S., Hirschhorn J.N., Asselta R., Duga S., Spreafico M., Musunuru K., Daly M.J., Purcell S., Voight B.F., Purcell S., Nemesh J., Korn J.M., McCarroll S.A., Schwartz S.M., Yee J., Kathiresan S., Lucas G., Subirana I., Elosua R., Surti A., Guiducci C., Gianniny L., Mirel D., Parkin M., Burtt N., Gabriel S.B., Samani N.J., Thompson J.R., Braund P.S., Wright B.J., Balmforth A.J., Ball S.G., Hall A., Wellcome Trust Case Control Consortium, Schunkert H., Erdmann J., Linsel-Nitschke P., Lieb W., Ziegler A., König I., Hengstenberg C., Fischer M., Stark K., Grosshennig A., Preuss M., Wichmann H.E., Schreiber S., Schunkert H., Samani N.J., Erdmann J., Ouwehand W., Hengstenberg C., Deloukas P., Scholz M., Cambien F., Reilly M.P., Li M., Chen Z., Wilensky R., Matthai W., Qasim A., Hakonarson H.H., Devaney J., Burnett M.S., Pichard A.D., Kent K.M., Satler L., Lindsay J.M., Waksman R., Knouff C.W., Waterworth D.M., Walker M.C., Mooser V., Epstein S.E., Rader D.J., Scheffold T., Berger K., Stoll M., Huge A., Girelli D., Martinelli N., Olivieri O., Corrocher R., Morgan T., Spertus J.A., McKeown P., Patterson C.C., Schunkert H., Erdmann E., Linsel-Nitschke P., Lieb W., Ziegler A., König I.R., Hengstenberg C., Fischer M., Stark K., Grosshennig A., Preuss M., Wichmann H.E., Schreiber S., Hólm H., Thorleifsson G., Thorsteinsdottir U., Stefansson K., Engert J.C., Do R., Xie C., Anand S., Kathiresan S., Ardissino D., Mannucci P.M., Siscovick D., O’Donnell C.J., Samani N.J., Melander O., Elosua R., Pelto-nen L., Salomaa V., Schwartz S.M., Altshuler D. (2009) Genome-wide association of early-onset myocardial infarction with single nucleotide polymorphisms and copy number variants. Nat. Genet. 41, 334–341.
Шестерня П.А., Шульман В.А., Никулина С.Ю. (2012) Генетические аспекты инфаркта миокарда: проблемы и перспективы. Российский кардиологический журнал. 93, 4–9.
Titov B.V., Osmak G.J., Matveeva N.A., Kukava N.G., Shakhnovich R.M., Favorov A.V., Ruda M.Y., Favorova O.O. (2017) Genetic risk factors for myocardial infarction more clearly manifest for early age of first onset. Mol. Biol. Rep. 44, 315–321.
Гончарова И.А., Макеева О.А., Голубенко М.В., Марков А.В., Тарасенко Н.В., Слепцов А.А., Пузырев В.П. (2016) Гены фиброгенеза в детерминации предрасположенности к инфаркту миокарда. Молекуляр. биология. 50, 94–105.
Peakall R., Smouse P.E. (2012) GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research–an update. Bioinformatics. 28, 2537–2539.
Lin B.D., Willemsen G., Fedko I.O., Jansen R., Penninx B., de Geus E., Kluft C., Hottenga J., Boomsma D.I. (2017) Heritability and GWAS studies for monocyte-lymphocyte ratio. Twin Res. Hum. Genet. 20, 97–107.
Liu S., Rose D.M., Han J., Ginsberg M.H. (2000) Alpha4 integrins in cardiovascular development and diseases. Trends Cardiovasc. Med. 10, 253–257.
Kukava N.G., Titov B.V., Osmak G.J., Matveeva N.A., Kulakova O.G., Favorov A.V., Shakhnovich R.M., Ruda M.Y., Favorova O.O. (2017) Multilocus analysis of genetic susceptibility to myocardial infarction in Russians: replication study. Acta Naturae. 9, 74–83.
Dandona S., Stewart A.F., Chen L., Williams K., So D., O’Brien E., Glover C., Lemay M., Assogba O., Vo L., Wang Y.Q., Labinaz M., Wells G.A., McPherson R., Roberts R. (2010) Gene dosage of the common variant 9p21 predicts severity of coronary artery disease. J. Am. Coll. Cardiol. 56, 479–486.
Cunnington M.S., Santibanez Koref M., Mayosi B.M., Burn J., Keavney B. (2010) Chromosome 9p21 SNPs associated with multiple disease phenotypes correlate with ANRIL expression. PLoS Genet. 6, e1000899.
Makeeva O.A., Sleptsov A.A., Kulish E.V., Barbarash O.L., Mazur A.M., Prokhorchuk E.B., Che-kanov N.N., Stepanov V.A., Puzyrev V.P. (2015) Genomic study of cardiovascular continuum comorbidity. Acta Naturae. 7, 89–99.
Hannou S.A., Wouters K., Paumelle R., Staels B. (2015) Functional genomics of the CDKN2A/B locus in cardiovascular and metabolic disease: what have we learned from GWASs? Trends Endocrinol. Metab. 26, 176–184.
Tajbakhsh A., Khorrami M.S., Hassanian S.M., Aghasizade M., Pasdar A., Maftouh M., Tabatabai E., Pariza-deh S.M., Fazeli M., Ferns G.A., Ghayour-Mobarhan M., Avan A. (2016) The 9p21 locus and its potential role in atherosclerosis susceptibility; molecular mechanisms and clinical implications. Curr. Pharm. Des. 22, 5730–5737.
Akashi M., Higashi T., Masuda S., Komori T., Furuse M. (2011) Coronary artery disease-associated gene pro-duct, JCAD/KIAA1462, is a novel component of endothelial cell-cell junctions. Biochem. Biophys. Res. Commun. 413, 224–229.
Erdmann J., Willenborg C., Nahrstaedt J., Preuss M., König I.R., Baumert J., Linsel-Nitschke P., Gieger C., Tennstedt S., Belcredi P., Aherrahrou Z., Klopp N., Loley C., Stark K., Hengstenberg C., Bruse P., Freyer J., Wagner A.K., Medack A., Lieb W., Grosshennig A., Sager H.B., Reinhardt A., Schäfer A., Schreiber S., El Mokhtari N.E., Raaz-Schrauder D., Illig T., Garlichs C.D., Ekici A.B., Reis A., Schrezenmeir J., Rubin D., Ziegler A., Wichmann H.E., Doering A., Meisinger C., Meitinger T., Peters A., Schunkert H. (2011) Genome-wide association study identifies a new locus for coronary artery disease on chromosome 10p11.23. Eur. Heart J. 32, 158–168.
Hara T., Monguchi T., Iwamoto N., Akashi M., Mori K., Oshita T., Okano M., Toh R., Irino Y., Shinohara M., Yamashita Y., Shioi G., Furuse M., Ishida T., Hirata K.I. (2017) Targeted disruption of JCAD (junctional protein associated with coronary artery disease)/KIAA1462, a coronary artery disease-associated gene product, inhi-bits angiogenic processes in vitro and in vivo. Arterioscler. Thromb. Vasc. Biol. 37, 1667–1673.
Papaspyridonos M., Smith A., Burnand K.G., Taylor P., Padayachee S., Suckling K.E., James C.H., Greaves D.R., Patel L. (2006) Novel candidate genes in unstable areas of human atherosclerotic plaques. Arterioscler. Thromb. Vasc. Biol. 26, 1837–1844.
Berger M., Moscatelli H., Kulle B., Luxembourg B., Blouin K., Spannagl M., Lindhoff-Last E., Schambeck C.M. (2008) Association of ADAMDEC1 haplotype with high factor VIII levels in venous thromboembolism. Thromb. Haemost. 99, 905–908.
Kamphuisen P.W., Eikenboom J.C., Bertina R.M. (2001) Elevated factor VIII levels and the risk of thrombosis. Arterioscler. Thromb. Vasc. Biol. 21, 731–738.
Çoban N., Güçlü Geyik F., Yıldırım Ö., Erginel Ünaltuna N. (2017) [Investigating the role of ceramide metabolism-associated CERS5 (LASS5) gene in atherosclerosis pathogenesis in endothelial cells]. Turk. Kardiyol. Dern. Ars. 45, 118–125.
Barragán R., Coltell O., Portolés O., Asensio E.M., Sorlí J.V., Ortega-Azorín C., González J.I., Sáiz C., Fernández-Carrión R., Ordovas J.M., Corella D. (2018) Bitter, sweet, salty, sour and umami taste perception decreases with age: sex-specific analysis, mo-dulation by genetic variants and taste-preference associations in 18 to 80 year-old subjects. Nutrients. 10, pii: E1539.
Rawal S., Huedo-Medina T.B., Hoffman H.J., Swede H., Duffy V.B. (2017) Structural equation modeling of associations among taste-related risk factors, taste functioning, and adiposity. Obesity (Silver Spring). 25, 781–787.
Zollbrecht C., Grassl M., Fenk S., Höcherl R., Hubauer U., Reinhard W., Esslinger U.B., Ebert S., Langmann T., Stark K., Hengstenberg C. (2013) Expression pattern in human macrophages dependent on 9p21.3 coronary artery disease risk locus. Atherosclerosis. 227, 244–249.
Xiong W., Wang H., Lu L., Xi R., Wang. F, Gu G., Tao R. (2017) The macrophage C-type lectin receptor CLEC5A (MDL-1) expression is associated with early plaque progression and promotes macrophage survival. J. Transl. Med. 15, 234.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


