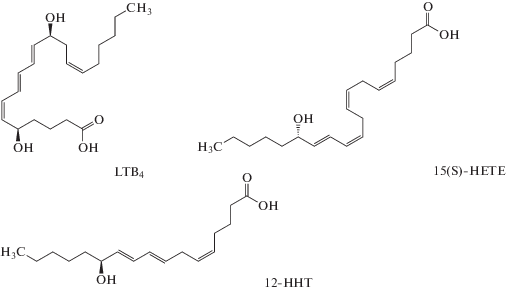
Молекулярная биология, 2020, T. 54, № 2, стр. 341-352
Сравнительное компьютерное моделирование связывания агонистов с лейкотриеновыми рецепторами BLT1 и BLT2
Г. Ф. Куракин a, *, Н. П. Лопина a, Г. Е. Бордина a
a Тверской государственный медицинский университет Министерства здравоохранения России
170000 Тверь, Россия
* E-mail: Phyzyk@mail.ru
Поступила в редакцию 25.02.2019
После доработки 16.09.2019
Принята к публикации 17.09.2019
Аннотация
Рецепторы лейкотриена B4 – BLT1 и BLT2 – считаются перспективными мишенями в разработке новых лекарственных средств, эффективных при аллергических и воспалительных заболеваниях. Однако отсутствуют рабочие модели связывания лигандов с этими рецепторами. Исходя из предположения, что гомологичные рецепторы связывают лиганд сходным образом, проведено компьютерное моделирование связывания агонистов с BLT1 и BLT2 с применением полностью гибкого докинга в веб-сервисе Galaxy 7TM. В моделях обоих рецепторов карбоксильная группа лиганда образует солевой мостик с остатком аргинина, а гидроксигруппа у его “хвоста” образует группу водородных связей с тремя аминокислотными остатками. Различная специфичность лигандов к BLT1 и BLT2 обусловлена заменой остатка гистидина на тирозин. В BLT1 остаток гистидина связывается с 5-OH-группой лиганда, тогда как в BLT2 остаток тирозина отталкивает ее. Полученные модели лучше согласуются с экспериментальными данными, нежели предложенные ранее, и могут быть использованы в разработке новых лекарственных средств, действующих на BLT1 и BLT2.
ВВЕДЕНИЕ
Лейкотриен B4 (LTB4) – сильный провоспалительный медиатор, синтезируемый из арахидоновой кислоты [1], участвующий в осуществлении таких важных звеньев воспалительной реакции, как адгезия гранулоцитов, их дегрануляция, трансмиграция и респираторный взрыв [2].
Действие LTB4 опосредовано двумя типами рецепторов: BLT1 и BLT2. Оба они принадлежат к семейству рецепторов, сопряженных с G-белком (GPCR), гомологичны друг другу (делят 45% идентичности, рис. 1), а их гены находятся в геноме в непосредственной близости [2, 3].
Рис. 1.
Выравнивание последовательностей BLT1 и BLT2 человека с помощью программы UniProt Align [4]. Видна гомология последовательностей (* – совпадающие остатки, : – остатки со значительным физико-химическим сходством, . – остатки с незначительными физико-химическим сходством). Цветом выделены аминокислотные остатки, предположительно необходимые для связывания (табл. 1).

Рецепторы BLT1 и BLT2 различаются профилем экспрессии и фармакологическими свойствами (рис. 2). BLT1 экспрессируется преимущественно на лейкоцитах, а BLT2 во всех тканях [2]. Естественным агонистом BLT1 является LTB4, с меньшей аффинностью с ним связываются гидроксиэйкозатетраеновые кислоты (HETE) [5–7]. BLT2, наоборот, имеет низкую аффинность к LTB4 [2, 3] и более сильно связывает 12(S)-гидроксигептадекатриеновую кислоту (12(S)-HHT), 12(S)-HETE и 15(S)-HETE [8, 9], а его основным естественным лигандом является 12-HHT [8].
Агонисты BLT1 и BLT2 имеют две структурные особенности: содержат карбоксильную группу и гидроксигруппу у “хвоста” молекулы (в положении 12–15). Эти два элемента представлены во всех лигандах BLT, депонированных в базу данных GuideToPharmacology [5–7], тогда как 5-OH-группа у ряда лигандов отсутствует и считается факультативной. Биологическая важность “хвостовой” гидроксигруппы подтверждается тем, что замена 12-OH-группы LTB4 на метильную устраняет его хемоаттрактантное действие по отношению к нейтрофилам [10]. Эти данные могут оказаться полезными при дальнейшем моделировании.
В последнее время BLT1 и BLT2 рассматривают в качестве мишеней для новых лекарственных препаратов, что связано с важной ролью лейкотриеновой сигнализации в развитии воспалительных заболеваний. Повышенная продукция LTB4 способствует развитию псориаза, бронхиальной астмы, ревматоидного артрита, воспалительных заболеваний кишечника и ишемического повреждения почек [2]. Предполагается, что блокирование BLT1 может стать новым направлением терапии бронхиальной астмы и ревматоидного артрита. Свою эффективность оно показало на мышиных моделях атопического дерматита и псориаза [1]. Блокирование BLT1 и BLT2 может предотвращать развитие хронического бронхита и острой дыхательной недостаточности [11].
Кроме того, оба рецептора играют роль в ноцицепции: активация BLT1 обеспечивает болевую сенситизацию, а активация BLT2 – антиноцицептивный эффект. Это открывает возможности для разработки новых обезболивающих средств на основе агонистов BLT2 и антагонистов BLT1 [12].
Актуальной остается проблема разработки лекарственных средств, действующих на лейкотриеновые рецепторы, так как специфические антагонисты BLT2 до сих пор не найдены [1]. Для успешного поиска лекарственных средств, влияющих на рецепторы LTB4, необходима компьютерная модель связывания этих рецепторов с их лигандами.
Попытки моделирования BLT1 были предприняты тремя группами исследователей. Sabirsh A. и соавт. [13] провели компьютерное моделирование с валидацией сайт-специфичным мутагенезом. Они предложили модель, в которой LTB4 располагается вдоль оси рецептора, Arg178 и Glu185 образуют с лигандом водородные связи, а Val105 и Ile108 обрамляют связывающий участок.
Basu S. и соавт. [14] также провели компьютерное моделирование с валидацией мутагенезом. Они показали, что описанная Sabirsh A. и соавт. [13] идентификация Arg178 как связывающего остатка, обусловлена ошибкой, допущенной при проведении сайт-специфичного мутагенеза.
Взамен Basu и соавт. [14] предложили свою модель, в которой карбоксильная группа лейкотриена связывается с Arg156, наиболее важным для связывания лиганда. Оказалось, что мутация Arg156 полностью устраняла связывание. Glu185, Tyr102, His94 в данной модели образовывали водородную связь с 5-гидроксигруппой, Asn241 – c 12-гидроксигруппой. Значимость перечисленных аминокислотных остатков для связывания подтверждена сайт-специфичным мутагенезом.
Ishino Y. и Harada T. [15] разработали новый метод моделирования GPCR в комплексе с лигандами. В качестве апробации метода они провели моделирование BLT1. В полученной модели карбоксильная группа агониста (использовали 12-кето-LTB4) образует водородную связь с Asn241, а карбонильная – с главной цепью (-N(H)- группой пептидной связи) остатка Gly246. Косвенная валидация модели проведена по величинам оценивающей функции докинга набора лигандов. Таким образом, в двух публикациях [13, 14] предложены практически диаметрально противоположные модели: в одной Asn241 связывается с “хвостом” агониста, в другой – с “головой” (карбоксильной группой), т.е. отсутствует консенсусная модель BLT1 в комплексе с LTB4.
Cведения о компьютерных моделях связывания BLT2 с лигандами до недавнего времени отсутствовали. Первая попытка сравнительного моделирования связывания агонистов и антагонистов с BLT1 и BLT2 предпринята Ibrahim M.A. и соавт. [16]. Показано, что лиганды связываются с сайтом, описанным ранее [14, 15], а величины расчетной энергии связывания антагонистов хорошо согласуются с экспериментально определенными аффинностями. Однако не приведены детальные данные о связывании лигандов с BLT и визуализации связей. Отмечено образование агонистами водородных связей с Asn241 и Arg267 в BLT1. При этом отсутствуют данные минимизации энергии агонистов и сравнения экспериментальной и расчетной аффинности в отличие от антагонистов. Кроме того, не описаны связи лигандов с BLT2, не рассмотрен механизм различий в аффинности лигандов к BLT1 и BLT2, что затрудняет использование этой модели для дальнейших исследований и ее сравнение с другими моделями.
Учитывая все это, целью нашего исследования стало построение молекулярных моделей связывания агонистов как с BLT1, так и с BLT2, а также сравнение лиганд-рецепторных взаимодействий в данных моделях. Ожидалось, что такое моделирование предоставит дополнительную информацию о способе связывания агонистов с BLT.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Структуры лигандов (рис. 2) получены из баз данных PubChem (https://pubchem.ncbi.nlm.nih.gov/) [17] и ZINC (http://zinc.docking.org/) [18], первичные структуры рецепторов – из базы данных UniProt (https://www.uniprot.org/) [19].
В качестве основной методики моделирования мы использовали онлайн-сервисы, предназначенные специально для моделирования GPCR и включающие в себя элементы молекулярной динамики. В частности, модели по гомологии загружали из базы данных GPCR-HGMod v.3 (https://zhanglab. ccmb.med.umich.edu/GPCR-HGmod/). Эта база содержит структуры, смоделированные с помощью сервера GPCR-I-TASSER, представляющего собой версию I-TASSER, адаптированную для моделирования GPCR [20].
Докинг проводили с использованием сервера Galaxy7TM (http://galaxy.seoklab.org/cgi-bin/submit.cgi?type=7TM) [21], предназначенного для полностью гибкого докинга лигандов в GPCR. Проводили прямой докинг, сайт связывания задавали номерами аминокислотных остатков: His94, Tyr102, Glu185, Asn241 в BLT1 (в [14–16] отмечены как связывающие) и Tyr129, Tyr137, Glu216, Asn275 (гомологи перечисленных остатков) в BLT2.
В тех случаях, когда необходимо было исключить гибкость рецептора и фиксировать его координаты при вычислениях, мы использовали сервер Swiss-Model (https://swissmodel.expasy.org/) [22, 23] для моделирования по гомологии и программу AutoDock Vina [24] для докинга с подготовкой и анализом в AutoDock Tools [25].
Для первичного анализа лиганд-рецепторных взаимодействий использовали программу LIGPLOT [26], интегрированную в Galaxy7TM [21]. Углубленный анализ взаимодействий выполняли с помощью веб-сервиса PLIP (http://plip.biotec.tu-dresden.de/plip-web/plip/index) [27], доступной онлайн среды ezCADD (http://dxulab.pharmacy.isu.edu/ curry/ezCADD/main.html) и приложения ezLigPlot в ее составе [28].
Для генерирования и анализа изображений использовали программы LigPlot+ [29], UCSF Chimera [30] и ezLigPlot [28].
Аминокислотные последовательности сравнивали с использованием сервиса Align (https:// www.uniprot.org/align/) в составе портала UniProt [4], поиск консервативных аминокислотных остатков проводили с помощью сервера ConSurf (http://consurf.tau.ac.il/2016/) [31].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Модель рецептора BLT1 загружена из базы данных CPCR-HGMod (UniProt ID Q15722, HG ID HG0452). В результате докинга LTB4 в эту структуру получены 10 решений докинга. В большинстве из них связь с карбоксильной группой лиганда образовывал Arg178 вместе с Arg267, Arg156 или Tyr172 (рис. 3). Большинство водородных связей в этих моделях концентрировались у карбоксильной группы лиганда, при этом в большинстве вариантов 12-OH-группа не образовывала водородной связи с рецептором или один аминокислотный остаток перекрестно связывал 12-OH- и 5-OH-группу (рис. 3), или 12-OH-группу и карбоксильную группу.
Рис. 3.
Результат докинга LTB4 в структуру BLT1, полученную из GPCR-HGMod. Пунктирные линии с числами – водородные связи и их длины, дуги со спицами – гидрофобные контакты. Обращает на себя внимание участие Arg178 в связывании карбоксильной группы, что противоречит данным сайт-специфичного мутагенеза [14].
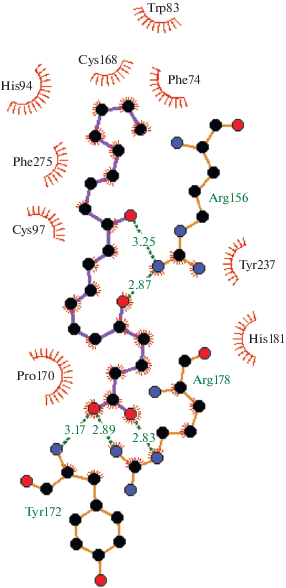
Эта особенность выглядит биологически неправдоподобной. Участие остатка Arg178 в связывании карбоксильной группы не согласуется с тем фактом, что его замена не влияет на связывание лиганда [14]. Возможно, эти модели в чем-то повторяют ошибки моделирования Sabirsh и соавт. [13]. В некоторых решениях докинга водородные связи Arg178 c лигандом отсутствовали, но при этом 12-OH-группа не образовывала водородной связи с рецептором. Такие модели тоже кажутся сомнительными, учитывая биологическую значимость 12-OH-группы.
Перечисленные попытки прямого моделирования BLT1 не привели к построению модели, согласующейся с экспериментальными данными [5‒7, 10, 14]. Мы рассмотрели гомологию между BLT1 и BLT2 и предприняли попытку построить модель BLT1 косвенно, используя модель BLT2.
Все аминокислотные остатки BLT1, упомянутые как образующие водородную связь с лигандом [14–16] или идентифицированные в этом качестве в предыдущих моделях BLT1, оказались высококонсервативными. В большинстве своем они сохранены и в BLT2. Однако в BLT2 два из них подвергнуты замене His <=> Tyr (табл. 1).
Таблица 1.
Гомологичные консервативные остатки BLT1 и BLT2, предположительно необходимые для связывания лиганда
| BLT1 | BLT2 |
|---|---|
| His94 | Tyr129 |
| Tyr102 | Tyr137 |
| Arg156 | Arg191 |
| Tyr172 | His203 |
| His181 | His212 |
| Glu185 | Glu216 |
| Tyr237 | Tyr271 |
| Asn241 | Asn275 |
Примечание. Эти же остатки отмечены цветом на рис. 1.
Гомология BLT1 и BLT2 и совпадение аминокислотных остатков, предположительно вовлеченных в связывание лиганда, свидетельствуют о том, что связывание лиганда в BLT2 и BLT1 должно быть сходным.
Модель BLT2 также загружена из базы данных GPCR-HGMod (HG ID HG0781, UniProt ID Q9NPC1). Так как LTB4 обладает низкой аффинностью к BLT2, для начального моделирования мы выбрали лиганды с большей аффинностью – 15(S)-HETE и 12-HHT.
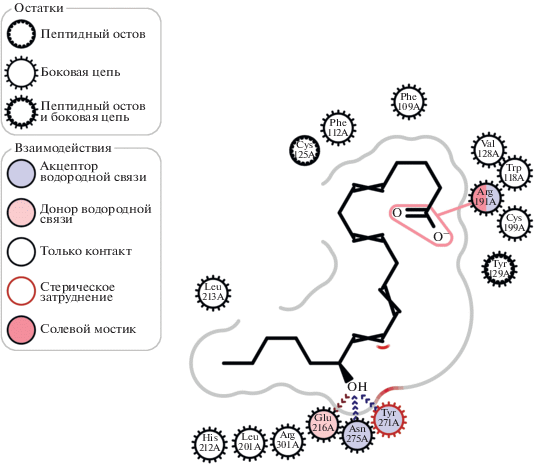
В результате докинга 15(S)-HETE в данную модель мы также получили 10 решений. Среди них по оценивающей функции Galaxy7TM лидировало решение, изображенное на рис. 4 (Приложение S1 , файл в формате *.pdb). При таком расположении карбоксильная группа лиганда образует солевой мостик с остатком Arg191 – гомологом Arg156 BLT1, а 15-OH-группа – водородные связи с Asn275 (гомологом Asn241 BLT1 и Tyr271 (рис. 4). Помимо этого, согласно анализу взаимодействий Galaxy7TM/LIGPLOT, карбоксильная группа образует также водородную связь с остатком Cys199, консервативным в BLT2 по данным ConSurf.
Рис. 4.
Результат докинга 15(S)-HETE в модель BLT2. Пунктирная линия с числом – водородная связь и ее длина, дуга со спицами – гидрофобный контакт.
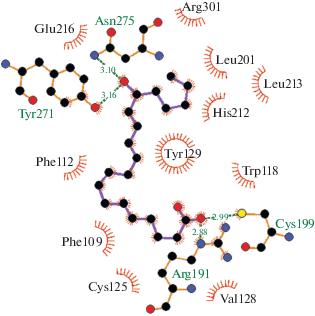
Такой способ связывания практически гомологичен способу, описанному Basu S. и соавт. [14] для BLT1. Повторный анализ лиганд-рецепторных взаимодействий в среде ezCADD позволил уточнить, что 15-OH-группа образует водородную связь не только с Asn275 и Tyr271, но и с Glu216 (гомологом Glu185 BLT1, рис. 5).
Гидрофобные взаимодействия лиганда с рецептором обеспечивались Phe109, Phe112, Trp118, Val128, Tyr129, Leu201, Leu213.
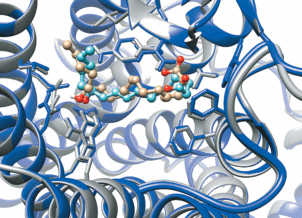
Модель с аналогичными взаимодействиями и конформацией лиганда получена с помощью Galaxy7TM для связывания 12-HHT (основного естественного лиганда) с BLT2 (Приложение S2 , файл в формате *.pdb). Среди результатов докинга эта модель занимала четвертое место из десяти. При пространственном наложении последние две структуры почти полностью соответствуют друг другу, вплоть до положения боковых цепей аминокислот связывающего участка (рис. 6).
Рис. 6.
Наложение результата гибкого докинга 12-HHT в модель BLT2 (синий и голубой цвет) на результат гибкого докинга 15(S)-HETE в модель BLT2 (серый и бежевый цвет). Лиганды (шаростержневые модели) занимают сходную позицию, положение остова (лента) и боковых цепей рецептора (стержневые модели) почти одинаково.
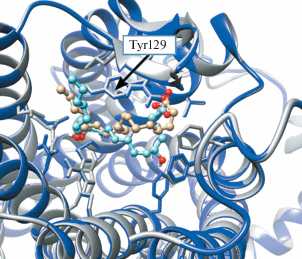
Далее мы провели гибкий докинг LTB4 в ту же самую структуру BLT2. Второе место из 10 результатов занимало решение докинга, аналогичное двум предыдущим моделям (Приложение S3 , файл в формате *.pdb). Аминокислотные остатки рецептора, карбоксильная группа и “хвостовая” часть LTB4 занимали такое же положение, как в случае с 15(S)-HETE и 12-HHT. Однако конформация лиганда в данном случае отличалась от предыдущих моделей резким изгибом в области 5-OH-группы. В таком положении 5-OH-группа была максимально отдалена от остатка Tyr129 (рис. 7).
Рис. 7.
Наложение результата гибкого докинга LTB4 в модель BLT2 (синий и голубой цвет) на результат гибкого докинга 15(S)-HETE в модель BLT2 (серый и бежевый цвет). LTB4 делает резкий изгиб в области 5-OH-группы, что отличает его конформацию от поз 12-HHT и 15(S)-HETE.
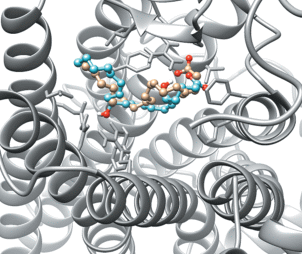
Для выяснения причины этих конформационных различий необходимо получить модель комплекса LTB4–BLT2, в которой LTB4 занимал такое же положение, как 15(S)-HETE и 12-HHT. С этой целью мы провели повторный докинг LTB4 в структуру BLT2 из комплекса 15(S)-HETE–BLT2, используя не гибкий докинг в Galaxy7TM, а полугибкий в AutoDock Vina. При этом ожидалось, что жесткий рецептор обеспечит пространственные ограничения для лиганда, заставляя его принять конформацию, аналогичную конформации 15(S)-HETE.
Такое решение было получено (рис. 8; Приложение S4 , файл в формате *.pdb), оно занимало второе место в списке из 9 результатов, имея расчетную энергию связывания –5.7 ккал/моль (энергия связывания в лучшем решении –6.0 ккал/моль). Полученная конформация LTB4 более точно совмещалась с 15(S)-HETE, а 5-OH-группа обращена в сторону Tyr129.
Рис. 8.
Наложение LTB4 (голубая шаростержневая модель) после повторного докинга в AutoDock Vina на 15(S)-HETE (бежевая шаростержневая модель) в связывающем участке BLT2 (серая ленточно-стержневая модель). LTB4 занимает такую же позицию, как 15(S)-HETE.
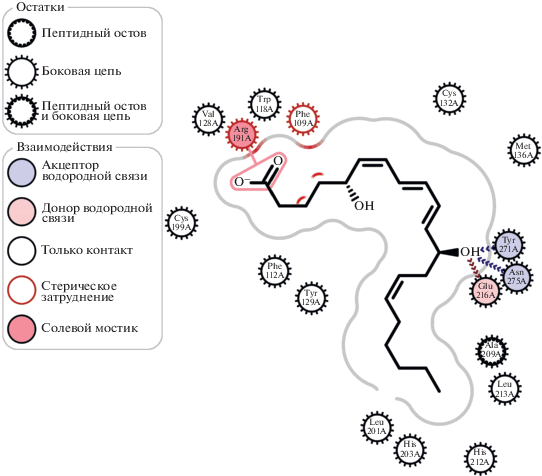
Взаимодействия LTB4 в такой конформации c BLT2 показаны на рис. 9. и аналогичны взаимодействиям, полученным ранее для 15(S)-HETE.
Далее мы использовали эту структуру как отправную точку для косвенного моделирования комплекса LTB4–BLT1. Мы предположили, что конформация, принимаемая лигандом в комплексах 12-HHT–BLT2, 15(S)-HETE–BLT2 и LTB4–BLT2 после повторного докинга, является нативной как в BLT1, так и в BLT2.
Чтобы получить комплекс LTB4–BLT1 с такой конформацией лиганда, мы заменили BLT2 на BLT1 с сохранением координат аминокислотных остатков. Мы воспользовались онлайн-программой моделирования по гомологии Swiss-Model. Координаты и конформация лиганда полностью заимствованы из комплекса LTB4–BLT2 после повторного докинга. Далее мы сравнили лиганд-рецепторные взаимодействия в полученной структуре (Приложение S5 , файл в формате *.pdb) и в комплексе LTB4–BLT2 после повторного докинга.
Большей частью связывание LTB4 c BLT1 было гомологичным связыванию с BLT2: Arg156 (гомолог Arg191 BLT2) образовывал солевой мостик с карбоксильной группой лиганда, Asn241 (гомолог Asn275 в BLT2), Glu185 (гомолог Glu216 в BLT2) и Tyr237 (гомолог Tyr271 в BLT2) образовывали водородные связи с 12-OH-группой LTB4. Однако к указанным взаимодействиям прибавилась водородная связь между His94 и 5-OH-группой LTB4. Гидрофобные контакты обеспечивались остатками Phe77, Pro170, Tyr172, Arg178, Leu182 (рис. 10).
Рис. 10.
Взаимодействия LTB4 со связывающим участком BLT1, смоделированные косвенным образом на основе гомологии между BLT1 и BLT2.
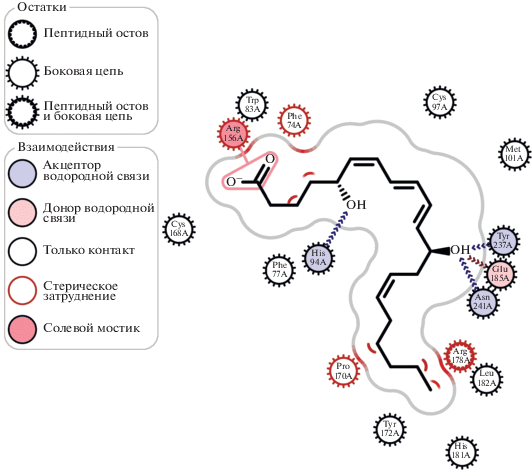
При сохранении тех же координат и остальных водородных связей комплекс LTB4–BLT1 отличался от комплекса LTB4–BLT2 лишь наличием образуемой 5-OH-группой водородной связи с рецептором. Наложение указанных структур (рис. 11) позволяет понять причину этого и объяснить различную специфичность лигандов к BLT1 и BLT2.
Рис. 11.
Пространственное наложение остатков BLT1 (бежевые) и BLT2 (голубые), образующих водородные связи с лигандом, в комплексе с LTB4 (фиолетовый).
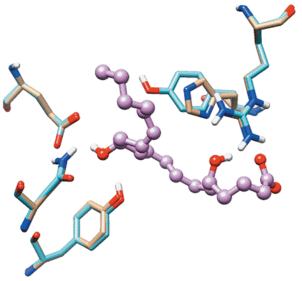
Аминокислотные остатки, образующие водородные связи с карбоксильной и 12-OH-группой LTB4, в обоих рецепторах расположены идентично. Различия касаются только His94 BLT1/Tyr129 BLT2.
Гидроксильная группа тирозина расположена слишком далеко от 5-OH-группы LTB4, поэтому этот остаток не образует с ней водородных связей. Наоборот, в связывающем участке BLT2 полярная 5-OH-группа упирается в массивное гидрофобное кольцо тирозина, что приводит к отталкиванию. Вероятно, это отталкивание обнаружено Galaxy7TM, вследствие чего LTB4 занял изогнутую позицию в модели комплекса BLT2–LTB4. Этим же объясняется низкая аффинность LTB4 к реальному рецептору BLT1.
15(S)-HETE и 12-HHT из-за отсутствия гидроксигруппы в аналогичной позиции не подвергаются отталкиванию. Наоборот, их алифатические цепи образуют гидрофобные контакты с Tyr129, что увеличивает аффинность к BLT2. В результате 15(S)-HETE и 12-HHT в модели располагаются более прямо, а их аффинность к BLT2 выше, чем у LTB4.
При расположении LTB4 в аналогичной позиции в BLT1 5-OH-группа лиганда направлена к дельта-атому азота His94, с которым образует дополнительную водородную связь, за счет чего аффинность LTB4 к BLT1 повышается. HETE не могут образовать такую связь с His94 BLT1, поэтому их аффинность к BLT1 ниже, чем у LTB4.
Весьма вероятно, что единственная замена His <=> Tyr в ходе эволюции привела к изменению специфичности одного из подтипов BLT к лиганду. Результаты моделирования связывания лиганда с BLT2 [14] указывают на влияние замены His94–Tyr129 на специфичность связывания. Наши результаты подтверждают это предположение.
Первое отличие нашей модели от модели [14] – концентрация основной массы водородных связей у 12-OH-группы лиганда (в случае LTB4), тогда как в модели Basu и соавт. [14] большую часть водородных связей образует 5-OH-группа. Наша модель может прояснить причину большей важности “хвостовой” гидроксигруппы лиганда для активации рецептора (ее потеря потенциально может привести к потере трех водородных связей), чем гидроксигруппы в положении С-5, потеря которой приведет к выпадению лишь одной водородной связи. Эта связь, согласно нашей модели, необходима для обеспечения различной специфичности лигандов к BLT1 и BLT2. Второе отличие заключается в расположении лиганда. В модели Basu и соавт. [14] оно вертикальное (вдоль оси рецептора), а в нашей модели – горизонтальное (поперек оси).
При этом набор связывающих остатков в нашей модели и в [14] в основном совпадает. Наша модель согласуется с результатами сайт-специфичного мутагенеза [14]. Так, в нашей модели Glu185 BLT1 (Glu216 в BLT2) образует водородную связь не с 5-OH-группой LTB4, а с его 12-OH-группой, однако остается важным для связывания, что соответствует результатам, полученным методом сайт-специфичного мутагенеза [14].
Полученная нами модель распространяется на оба типа BLT и объясняет, почему LTB4 имеет максимальную аффинность к BLT1, большую, чем у HETE [5–7], и низкую к BLT2, уступая лигандам, не содержащим 5-OH-группы [2, 3, 8, 9]. Она объясняет также биологическую значимость гидроксигруппы у “хвоста” лиганда и ее присутствие во всех агонистах [5–7, 10]. Таким образом, наша модель охватывает практически всю имеющуюся на сегодняшний день экспериментальную информацию о связывании агонистов с BLT1 и BLT2, т.е. лучше согласуется с эмпирическими данными, нежели предшествующие модели.
Нами не получены результаты, соответствующие модели [15]. Модель, полученная косвенным методом, более соответствует модели [14], но с некоторыми уточнениями. Сравнить наши модели с моделями [16] затруднительно из-за отсутствия детальных описаний связей.
Интересно, что попытки прямого докинга LTB4 в BLT1 как в Galaxy7TM, так и в AutoDock Vina (в модель BLT1 на основе комплекса BLT2–15(S)-HETE) приводят к противоречивым результатам, не согласующимся с экспериментальными данными. Чтобы частично прояснить механизм данного эффекта, мы с помощью UCSF Chimera заменили Tyr129 на гистидин в модели BLT2, полученной из GPCR-HGMod, и провели докинг в эту модель в Galaxy7TM. Одновременно мы заменили Tyr129 на гистидин в модели BLT2 из комплекса 15(S)-HETE–BLT2 и провели повторный докинг LTB4 в эту модель в AutoDock Vina.
В обоих случаях в результатах докинга не представлены описанные выше конформации, где карбоксильная группа LTB4 образует солевой мостик с Arg191, а 12-OH-группа – водородную связь c Glu216, Tyr271, Asn275. Лиганд-рецепторные взаимодействия в решениях докинга аналогичны полученным при гибком докинге LTB4 в BLT1. Мы предположили, что такой эффект наблюдается вследствие большого числа вращаемых связей в лиганде, который вдобавок представляет собой почти линейную молекулу.
В случае с BLT2 стабильность системы лиганд–рецептор при гибком докинге выше в силу существования только двух точек фиксации лиганда к рецептору водородными связями – в области связывания карбоксильной группы (первая точка) и гидроксигруппы у “хвоста” молекулы (вторая точка). В случае с BLT1 присутствует дополнительная точка образования водородной связи – связь His94 c 5-OH-группой лиганда, что увеличивает число возможных вариантов связывания. Положение лиганда с большим количеством вращаемых связей в таком связывающем участке будет ожидаемо нестабильным, что может приводить к отсутствию в результатах докинга “лиганд–BLT1” постулируемого нами способа связывания. Это предположение косвенно подтверждают результаты компьютерного эксперимента с заменой Tyr129 на гистидин.
Противоречивость данных моделирования BLT1, по-видимому, можно объяснить неустойчивостью системы “лиганд–BLT1” в компьютерной модели.
ЗАКЛЮЧЕНИЕ И ВЫВОДЫ
На основе гомологии и модели рецептора BLT2 косвенным образом удалось построить модели, описывающие различия в строении связывающего участка в BLT1и BLT2. В этих моделях карбоксильная группа LTB4 образует солевой мостик с остатком Arg156 BLT1 (Arg191 BLT2), а Glu185, Tyr237, Asn241 BLT1 (соответственно Glu216, Tyr271, Asn275 BLT2) образуют водородные связи с гидроксигруппой у “хвоста” лиганда (12-OH-группа LTB4, 15-OH-группа 15(S)-HETE).
Согласно полученным моделям, различия в специфичности лигандов к BLT1 и BLT2 обусловлены заменой одного из аминокислотных остатков. His94 BLT1 образует водородную связь с 5-OH-группой LTB4, обеспечивая тем самым высокую аффинность к LTB4. В соответствующей позиции BLT2 находится остаток Tyr129, который не образует водородную связь с 5-OH-группой LTB4 и отталкивает эту группу за счет гидрофобного взаимодействия. Это объясняет низкую аффинность BLT2 к LTB4 и высокую – к HHT и HETE, не содержащим 5-OH-группы.
Полученные нами модели лучше согласуются с опубликованными экспериментальными данными, чем предыдущие модели. Это позволяет нам предположить, что они будут полезны для разработки новых лекарственных препаратов, влияющих на BLT1 и BLT2.
ПРИМЕЧАНИЯ (CORRIGENDUM)
В феврале 2019 года была обновлена аминокислотная последовательность BLT2, представленная в UniProt (Q9NPC1), укороченная на 31 N-концевой остаток.
Модели BLT2, доступные в GPCR-HGMod и использованные нами, основаны на старой версии. Это же касается обновленных моделей, доступных в GPCR-HGMod v. 4 и GPCR-db. Учитывая это, мы использовали в работе старую нумерацию.
Номер аминокислотного остатка в новой версии последовательности можно получить, уменьшив “старый номер” на 31.
Мы приводим перечень основных соответствий (табл. 2):
Таблица 2.
Соответствия номеров ключевых аминокислотных остатков в старой и новой нумерации BLT2
| Старая нумерация | Новая нумерация |
|---|---|
| Tyr129 | Tyr98 |
| Arg191 | Arg160 |
| Glu216 | Glu185 |
| Asn275 | Asn244 |
| Tyr271 | Tyr240 |
| Phe109 | Phe78 |
| Phe112 | Phe81 |
Авторам хотелось бы поблагодарить Хайруллину Веронику Радиевну (кафедра физической химии и химической экологии Башкирского государственного университета, Уфа) за консультации в процессе исследования и работы над статьей.
Написание настоящей статьи не потребовало специального финансирования.
Работа не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Yokomizo T. (2014) Two distinct leukotriene B4 receptors, BLT1 and BLT2. J. Biochemistry. 157(2), 65–71.
Yokomizo T., Kato K., Terawaki K., Izumi T., Shimizu T. (2000) A second leukotriene B4 receptor, Blt2. J. Exp. Med. 192(3), 421–432.
Tager A.M., Luster A.D. (2003) BLT1 and BLT2: the leukotriene B4 receptors. Prostaglandins Leukot. Essent. Fatty Acids. 69(2–3), 123–134.
Pundir S., Martin M.J., O’Donovan C., The UniProt Consortium (2016) UniProt tools. Curr. Protoc. Bioinformatics. 53(1), 1.29.1–1.29.15.
Alexander S.P., Christopoulos A., Davenport A.P., Kelly E., Marrion N.V., Peters J.A., Faccenda E., Har-ding S.D., Pawson A.J., Sharman J.L., Southan C., Davies J.A.; CGTP Collaborators. (2017) The concise guide to pharmacology 2017/18: G protein-coupled receptors. Br. J. Pharmacol. 174, S17–S129.
Bäck M., Brink C., Chiang N., Dahlén S.E., Dent G., Drazen J., Evans J.F., Hay D.W.P., Nakamura M., Powell W., Rokach J., Rovati G.E., Serhan C.N., Shimizu T., Yokomizo T. (2019) Leukotriene receptors (version 2019.4) in the IUPHAR/BPS Guide to Pharmacology Database. IUPHAR/BPS Guide to Pharmacology CITE. 2019(4). Available from: https://doi.org/https://doi.org/10.2218/gtopdb/F35/2019.4
Yokomizo T., Izumi T., Chang K., Takuwa Y., Shimizu T. (1997) A G-protein-coupled receptor for leukotriene B4 that mediates chemotaxis. Nature. 387(6633), 620–624.
Okuno T., Iizuka Y., Okazaki H., Yokomizo T., Taguchi R., Shimizu T. (2008) 12(S)-hydroxyheptadeca-5Z, 8E, 10E–trienoic acid is a natural ligand for leukotriene B4 receptor 2. J. Exp. Med. 205(4), 759–766.
Yokomizo T., Kato K., Hagiya H., Izumi T., Shimizu T. (2001) Hydroxyeicosanoids bind to and activate the low affinity leukotriene B4 receptor, BLT2. J. Biol. Chem. 276(15), 12454–12459.
Djuric S.W., Miyashiro J.M., Penning T.D., Keith R.H., Villani-Price D., Tsai B.-S. (1991) Enantioselective total synthesis and pharmacologic profile of 12-deoxy-12(S)-methyl leukotriene B4. Bioorg. Med. Chem. Lett. 1(3), 179–182.
Хайруллина В.Р., Таипов И.А., Герчиков А.Я., Васильев М.Н., Зарудий Ф.С., Бегель Х. (2014) Структурный анализ антагонистов рецепторов лейкотриенов ЛТB4 – BLT1и BLT2. Химико-фарм. журн. 48(5), 18–22.
Zinn S., Sisignano M., Kern K., Pierre S., Tunaru S., Jordan H., Suo J., Treutlein E.M., Angioni C., Ferreiros N., Leffler A., DeBruin N., Offermanns S., Geisslinger G., Scholich, K. (2017) The leukotriene B4 receptors BLT1 and BLT2 form an antagonistic sensitizing system in peripheral sensory neurons. J. Biol. Chem. 292(15), 6123–6134.
Sabirsh A., Bywater R.P., Bristulf J., Owman C., Haeggström J.Z. (2006) Residues from transmembrane helices 3 and 5 participate in leukotriene B4 binding to BLT1†. Biochemistry. 45(18), 5733–5744.
Basu S., Jala V.R., Mathis S., Rajagopal S.T., Del Prete A., Maturu P., Trent J.O., Haribabu B. (2007) Critical role for polar residues in coupling leukotriene B4 binding to signal transduction in BLT1. J. Biol. Chem. 282(13), 10005–10017.
Ishino Y., Harada T. (2012) Novel computational methodologies for structural modeling of spacious ligand binding sites of G-protein-coupled receptors: development and application to human leukotriene B4 receptor. Sci. World J. 2012, 1–11.
Ibrahim M.A., Hassan A.M. (2018) Comparative modeling and evaluation of leukotriene B4 receptors for selective drug discovery towards the treatment of inflammatory diseases. Protein J. 37(6), 518–530.
Kim S., Thiessen P.A., Bolton E.E., Chen J., Fu G., Gindulyte A., Han L., He J., He S., Shoemaker B.A., Wang J., Yu B., Zhang J., Bryant S.H. (2015) PubChem Substance and Compound databases. Nucl. Acids Res. 44(D1), D1202–D1213.
Irwin J.J., Sterling T., Mysinger M.M., Bolstad E.S., Coleman R.G. (2012) ZINC: a free tool to discover chemistry for biology. J. Chem. Inf. Modeling. 52(7), 1757–1768.
UniProt Consortium (2017) UniProt: the universal protein knowledge base. Nucl. Acids Res. 45(D1), D158–D169.
Zhang J., Yang J., Jang R., Zhang Y. (2015) GPCR-I-TASSER: a hybrid approach to G protein-coupled receptor structure modeling and the application to the human genome. Structure. 23(8), 1538‒1549.
Lee G.R., Seok C. (2016) Galaxy7TM: flexible GPCR–ligand docking by structure refinement. Nucl. Acids Res. 44(W1), W502–W506.
Waterhouse A., Bertoni M., Bienert S., Studer G., Tauriello G., Gumienny R., Heer F.T., de Beer T.A.P., Rempfer C., Bordoli L., Lepore R., Schwede T. (2018) SWISS-MODEL: homology modelling of protein structures and complexes. Nucl. Acids Res. 46(W1), W296–W303.
Guex N., Peitsch M.C., Schwede T. (2009) Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer: A historical perspective. Electrophoresis. 30(S1), S162–S173.
Trott O., Olson A.J. (2009) AutoDockVina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading. J. Comput. Chem. 31(2), 455–461.
Sanner M.F. (1999) Python: a programming language for software integration and development. J. Mol. Graph. Model. 17(1), 57‒61.
Wallace A.C., Laskowski R.A., Thornton J.M. (1995) LIGPLOT: a program to generate schematic diagrams of protein-ligand interactions. Protein Eng. Design Select. 8(2), 127–134.
Salentin S., Schreiber S., Haupt V.J., Adasme M.F., Schroeder M. (2015) PLIP: fully automated protein–ligand interaction profiler. Nucl. Acids Res. 43(W1), W443–W447.
Tao A., Huang Y., Shinohara Y., Caylor M.L., Pashikanti S., Xu D. (2018) ezCADD: A rapid 2D/3D visualization-enabled Web modeling environment for democratizing computer-aided drug design. J. Chem. Inf. Modeling. 59(1), 18–24.
Laskowski R.A., Swindells M.B. (2011) LigPlot+: multiple ligand–protein interaction diagrams for drug discovery. J. Chem. Inf. Modeling. 51(10), 2778–2786.
Pettersen E.F., Goddard T.D., Huang C.C., Couch G.S., Greenblatt D.M., Meng E.C., Ferrin T.E. (2004) UCSF Chimera: A visualization system for exploratory research and analysis. J. Comput. Chem. 25(13), 1605–1612.
Ashkenazy H., Abadi S., Martz E., Chay O., Mayrose I., Pupko T., Ben-Tal N. (2016) ConSurf 2016: an improved methodology to estimate and visualize evolutionary conservation in macromolecules. Nucl. Acids Res. 44(W1), W344–W350.
Дополнительные материалы
- скачать ESM_1.pdb
- Дополнительные материалы 1
- скачать ESM_2.pdb
- Дополнительные материалы 2
- скачать ESM_3.pdb
- Дополнительные материалы 3
- скачать ESM_4.pdb
- Дополнительные материалы 4
- скачать ESM_5.pdb
- Дополнительные материалы 5
Инструменты
Молекулярная биология