Молекулярная биология, 2020, T. 54, № 3, стр. 426-434
Сравнительный структурный анализ трех кластеров piРНК в геноме Drosophila melanogaster
А. П. Котнова a, *, Ю. В. Ильин a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: alina_kotnova@mail.ru
Поступила в редакцию 17.12.2019
После доработки 27.01.2020
Принята к публикации 27.01.2020
Аннотация
В статье предпринята попытка ретроспективного воссоздания последовательности событий, приведших к образованию трех регуляторных piРНК-кластеров у Drosophila melanogaster. Исследованы piРНК-кластеры 20А, 38С и flamenco. Построены схемы их последовательного формирования и выявлены структурные особенности. В двух из трех исследованных кластеров, 38С и flamenco, обнаружены инвертированные последовательности, потенциально способные приводить к образованию двухцепочечных шпилек в РНК-транскрипте. Приведены доводы в пользу известной гипотезы о piРНК-кластерах как о “ловушках транспозонов”. Согласно этой модели, присутствие в геноме только одной копии транспозона свидетельствует о том, что его экспрессия была подавлена механизмами РНК-интерференции сразу после попадания мобильного элемента в piРНК-кластер. Приведены доказательства высокой структурной изменчивости piРНК-кластеров дрозофилы и событий горизонтального переноса мобильных элементов между родственными видами.
ВВЕДЕНИЕ
Геном Drosophila melanogaster населяет множество мобильных элементов, составляющих до 22% от всего генома [1]. Интактные, потенциально активные, копии мобильных элементов локализуются как в эухроматине, так и в прицентромерном и теломерном гетерохроматине. Кроме них в геноме дрозофилы присутствуют “молекулярные ископаемые” ‒ дефектные копии, давно утратившие способность к перемещениям. По последним данным, до 77% гетерохроматина дрозофилы представлено мобильными элементами. Бóльшая их часть разрушена инсерциями других транспозонов. Они не способны автономно перемещаться и сконцентрированы в прицентромерных областях хромосом. Эти регионы, первоначально рассматриваемые как кладбища мобильных элементов, вовлечены в систему защиты генома от нестабильности и исполняют роль регуляторов транспозиций [2, 3]. Такие кластеры транскрибируются, после чего транскрипты подвергаются процессингу и образуют короткие РНК, взаимодействующие с белком Piwi (piРНК), и эндогенные малые интерферирующие РНК (endo-siРНК). Короткие РНК в свою очередь приводят к блокировке перемещений мобильных элементов в гонадах и соматических тканях. Защитные механизмы, основанные на РНК-сайленсинге и направленные против распространения в геноме мобильных элементов, очень консервативны у эукариот [4].
На сегодняшний день крайне мало известно о судьбе деградировавших копий мобильных элементов, несмотря на их важную роль в формировании кластеров, принимающих участие в РНК-интерференции. В данной статье мы попытались воссоздать последовательность образования таких piРНК-кластеров.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
В работе использована пятая сборка генома D. melanogaster, доступная в базе данных FlyBase (ftp://ftp.flybase.net/genomes/Drosophila_melano-gaster/dmel_r5.49_FB2013_01/), и “канонический” набор последовательностей мобильных элементов (https://flybase.org/static_pages/downloads/ FB2015_02/transposons/transposon_sequence_set. embl.txt.gz). Для выравнивания нуклеотидных последовательностей использовали программы BLAST+ и MEGA5.
Выравнивание протяженных последовательностей piРНК-кластеров против всего набора мобильных элементов с помощью BLAST+ позволяет легко найти интактные копии мобильных элементов, а также их крупные фрагменты. В случае сильного повреждения копии мобильного элемента в результате множества делеций и вставок алгоритм BLAST способен найти и выровнять с канонической последовательностью только короткие ее фрагменты, которые получают небольшой скоринг и оказываются беспорядочно разбросанными в огромном массиве данных. С целью найти такие дефектные копии мы сначала сортировали результаты работы BLAST+ не по скорингу, а по положению внутри последовательности piРНК-кластера, а затем обрабатывали эти данные с помощью нескольких простейших скриптов и вручную.
В каноническом наборе есть мобильные элементы, очень близкие друг другу. Их последовательности схожи, поэтому при сравнении набора с какой-то третьей последовательностью с помощью алгоритма BLAST можно обнаружить в ней участки, схожие не с одним, а сразу с несколькими мобильными элементами. В первую очередь мы отфильтровывали такие перекрывающиеся результаты BLAST+, оставив в наборе данных варианты, имеющие наибольший скоринг и протяженность. Те области piRNA кластеров, в которых несколько фрагментов одного мобильного элемента были расположены близко друг к другу, анализировали вручную. Если эти фрагменты оказывались однонаправленными и следовали в геноме в точности в той же последовательности, что и в каноническом варианте мобильного элемента, или с небольшими перестройками, этот участок идентифицировали как дефектную копию мобильного элемента.
Предположения об очередности встраивания мобильных элементов в геном строили на основании анализа последовательностей, фланкирующих вставку мобильного элемента. Если элемент А был фланкирован в геноме справа и слева последовательностями, похожими на элемент Б, причем точка разрыва элемента Б совпадала слева и справа (с учетом дупликации сайта-мишени), то мы предполагали, что элемент А встроился в элемент Б.
Предположения о времени встраивания строили только для некоторых ретротранспозонов, имеющих длинные концевые повторы (LTR). Механизм их перемещения таков, что LTR у новой копии ретротранспозона в геноме всегда будут идентичными. Наличие внутри копии перестроек, к сожалению, не позволяет судить о времени ее перемещения, так как известны случаи перемещения дефектных копий ретротранспозонов с нарушенной кодирующей частью за счет активности белков, синтезированных на других, полноценных, копиях этого элемента.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
В 2007 году Brennecke с соавт. [2] продемонстрировали, что последовательности, с которых транскрибируются piРНК в геноме дрозофилы, не разбросаны по геному, а кластеризуются в небольшом числе дискретных локусов. Они определили 142 места в геноме, активно генерирующих короткие РНК. Эти сайты продуцируют 81% всех уникальных piРНК. Среди указанных 142 кластеров piРНК только 7 расположены в потенциально эухроматических областях, а остальные локализуются в прицентромерном и теломерном гетерохроматине. Размеры этих кластеров у D. melanogaster существенно различаются. Наибольшую известность среди них приобрел локус flamenco, охарактеризованный как регулятор транспозиций мобильных элементов задолго до открытия механизма РНК-интерференции.
В конце 1980-х–начале 1990-х было получено несколько генетически нестабильных линий D. melanogaster, в яичниках которых активно перемещался ретротранспозон gypsy. Оказалось, что нестабильность в этих линиях была вызвана комбинацией двух факторов: 1) присутствием транспозиционно активной копии gypsy и 2) мутацией, регулирующей перемещения этого мобильного элемента. Генетическими методами эта мутация была картирована в позиции 65,9 Х-хромосомы (секция 20A1‒А3) ‒ в непосредственной близости от β-гетерохроматина, накапливающего множество деградировавших мобильных элементов [5]. Локус получил название flamenco, поскольку “заставлял цыгана (gypsy) танцевать”. Примерно в этой же области Х-хромосомы в 1997 году картировали мутацию, контролирующую перемещения двух других ретротранспозонов: ZAM и Idefix. В связи с тем, что в мутантной линии с активно перемещающимися ZAM и Idefix не было обнаружено активных перемещений gypsy, Goriaux и др. [5] считали, что имеют дело с двумя различными генными локусами, расположенными по соседству: flamenco и COM. В последующие годы в лабораториях разных стран проводились интенсивные, но не увенчавшиеся успехом исследования, направленные на клонирование гена flamenco. Только после открытия явления РНК-интерференции появились предположения, что flamenco может оказаться не классическим геном-репрессором, а источником коротких регуляторных РНК. Но настоящий прорыв в понимании структуры локуса flamenco/COM произошел только в 2007 году, когда Brennecke с соавт. [2] охарактеризовали его как один из значимых piРНК-кластеров.
Нами предпринята попытка исследовать формирование трех таких кластеров и определить в них последовательности инсерций различных мобильных элементов.
Формирование piРНК-кластера flamenco
Локус flamenco, также известный как COM-локус, – главный регулятор перемещений gypsy, а также некоторых других ретротранспозонов, в частности ZAM и Idefix. Flamenco имеет длину примерно 180 т.п.н. и расположен в прицентромерном гетерохроматине в области 20А1‒А3 Х-хромосомы. Его последовательность большей частью составлена из фрагментов мобильных элементов, принадлежащих главным образом к семейству LTR-ретротранспозонов. В яичниках дрозофилы локус flamenco служит источником piРНК, а в соматических тканях – источником endo-siРНК. Роль flamenco заключается в предотвращении экспрессии gypsy и других мобильных элементов, фрагменты которых расположены в этом локусе. По этой причине он считается компонентом “адаптивного иммунитета” против ретротранспозонов [4].
Изучение локуса flamenco сыграло важную роль в понимании того, как малые некодирующие РНК репрессируют транспозиции мобильных элементов. От других piРНК-локусов flamenco отличается по некоторым параметрам. Во-первых, 94% всех уникальных РНК, кодируемых этим локусом, оказались партнерами белка Piwi. Во-вторых, большинство копий ретротранспозонов встроились во flamenco в одной и той же ориентации, поэтому локус продуцирует piРНК с заметной ассиметрией цепей [5].
Следует сказать, что flamenco относится не только к самым изученным, но и к самым интригующим piРНК-кластерам, поэтому мы проанализировали его в первую очередь. На рис. 1 приведена схема, иллюстрирующая последовательность встраивания мобильных элементов при формировании локуса flamenco.
Рис. 1.
Схема строения локуса flamenco. Светло-серым цветом обозначены деградировавшие фрагменты мобильных элементов (“молекулярные ископаемые”), темно-серым ‒ копии, имеющие значительное сходство с каноническими. Не закрашены делетированные области. Треугольниками обозначены длинные концевые повторы (LTR). Рамкой выделена область, потенциально способная формировать участки двухцепочечной РНК.
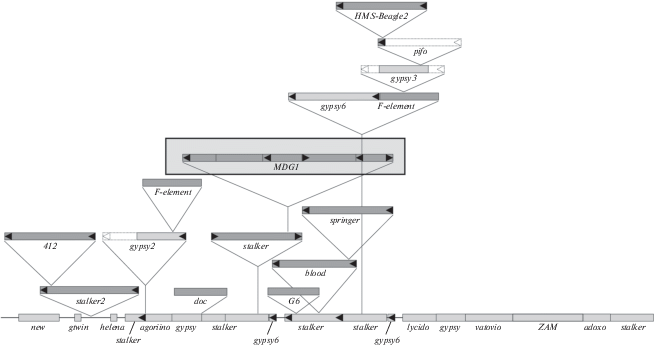
Мы предполагаем, что основой для образования локуса flamenco стала дуплицированная несколько раз последовательность, содержащая дивергировавшую копию мобильного элемента stalker или его предка. В данном случае речь идет не о нескольких независимых инсерциях этого мобильного элемента в близлежащие участки хромосомы, а о рекомбинационных событиях в геноме. Это подтверждается тем фактом, что внутри дуплицированного фрагмента обнаружены одни и те же перестройки и мутации.
На начальном этапе формирования кластера в эту последовательность встроились ретротранспозоны ZAM, gypsy, gypsy2, gypsy3, gypsy6, а также недавно описанные agoriino, lycido, vatovio и adoxo. Все эти элементы представлены в составе flamenco сильно деградировавшими фрагментами, что свидетельствует о давности произошедших событий.
Относительно недавно в локус flamenco попали мобильные элементы doc, G6, F-element, ретротранспозоны 412, blood, springer, HMS-Beagle2 и MDG1. Кроме того, нами обнаружены довольно свежие копии stalker и stalker2. По-видимому, эти события произошли в более поздний период, так как указанные мобильные элементы накопили мало замен по сравнению с образцом из канонического набора, а в LTR-ретротранспозонах присутствуют идентичные LTR. Известно, что в процессе обратной транскрипции таких элементов у новообразовавшейся копии формируются полностью идентичные друг другу концевые повторы [6, 7], поэтому по числу накопленных в LTR мутаций можно очень приблизительно оценить давность вставки.
Мобильный элемент pifo представлен одиночным LTR, что свидетельствует о вставке этого элемента и последующем его вырезании в результате гомологичной рекомбинации по LTR. Позднее внутрь оставшегося во flamenco LTR pifo встроилась копия ретротранспозона HMS-Beagle2.
Довольно интересна, с нашей точки зрения, вставка в локус flamenco мобильного элемента MDG1, содержащая сложную перестройку, представляющую сочетание инверсий и дупликаций (выделена рамкой на рис. 1). При транскрипции такой последовательности в РНК могут образовываться двухцепочечные участки, потенциально способные запускать механизм РНК-интерференции.
В 2013 году Zanni и др. [8] уже исследовали локус flamenco, однако их внимание было сосредоточено не на происхождении этого локуса, а на его функции как регулятора экспрессии мобильных элементов. Zanni с соавт. сравнивали “работу” локуса flamenco в трех различных линиях дрозофилы, характеризующихся разным уровнем экспрессии ретротранспозонов ZAM и Idefix (линии wIR6, Iso1A и Rev). Авторы определили, что структурные отличия локуса между линиями довольно велики: многочисленные случаи гомологичной и негомологичной рекомбинации, делеции, сегментальные дупликации, утеря и новые инсерции мобильных элементов. Построенная нами карта во многом сходна с опубликованной в статье Zanni и др. [8]. Так, по мнению авторов, основой образования локуса flamenco послужила дуплицированная несколько раз последовательность элемента stalker4, в которую встраивались в разное время мобильные элементы gypsy, MDG1, 412, F, doc, ZAM и другие. Но есть и немало отличий между предложенной нами картой и опубликованной Zanni и др. Например, в области flamenco нами выявлены ретротранспозоны gtwin, helena, agoriino, springer, lycido, vatovio и другие, не представленные на схеме Zanni с соавт. Кроме того, нами обнаружен еще один, ранее неизвестный мобильный элемент, пока не получивший собственного названия и обозначенный на схеме как “new”. Мы не выявили эухроматических копий этого элемента в геноме D. melanogaster, но обнаружили их у родственных видов: D. erecta и D. yakuba. По всей видимости, мы имеем дело со случаем горизонтального переноса ретроэлемента new в геном D. melanogaster.
Формирование piРНК-кластера 20А
В силу того, что исследовать структуру первого, самого протяженного piРНК-кластера, 42AB, вышеописанным способом оказалось невозможным именно по причине его протяженности, после flamenco мы изучили строение кластера 20А (аннотирован под номером 2 у Brennecke с соавт. [2]). Он расположен в секции 20А Х-хромосомы, его длина составляет примерно 40 т.п.н. Как и в случае flamenco, кластер 20А следует отнести к однонаправленным: почти все детектируемые внутри него piРНК считываются с плюс-цепи (78.4% против 0.2% по минус цепи) [2]. Схема строения кластера 20А представлена на рис. 2.
Рис. 2.
Схема строения piРНК-кластера 20А. Светло-серым цветом обозначены деградировавшие фрагменты мобильных элементов (“молекулярные ископаемые”), темно-серым – копии, имеющие значительное сходство с каноническими. Треугольниками обозначены длинные концевые повторы (LTR).
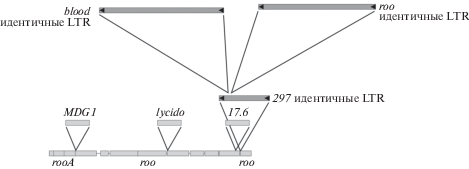
Этот кластер сформировался на базе деградировавшей копии ретротранспозона roo, содержащей вставки фрагментов других мобильных элементов: MDG1, lycido, rooA и 17.6, ‒ возраст которых определить не представляется возможным. Относительно недавно в этот кластер встроился мобильный элемент 297, в который, в свою очередь, внедрились ретротранспозоны blood и roo. Хотя внутри тела этих ретротранспозонов присутствуют делеции и вставки, мы полагаем, что инсерции произошли сравнительно недавно, так как LTR этих ретротранспозонов полностью идентичны друг другу. Кроме того, ретротранспозоны с внутренними делециями в некодирующих областях иногда способны перемещаться посредством ретротранспозиции за счет активности белков, синтезированных на других, полноценных копиях этого элемента.
Формирование piРНК-кластера 38С
Кластер 38С piРНК (аннотирован под номером 5 у Brennecke с соавт. [2]) заинтересовал нас как потенциальный регулятор ретротранспозиций элемента gtwin, который был впервые клонирован и много лет изучался в нашей лаборатории [9‒14].
Этот локус протяженностью свыше 79 т.п.н. локализован в секции 38С в левом плече хромосомы 3. Свыше 63% piРНК считываются по минус-цепи ДНК, в то время как по плюс-цепи ‒ 23.4% [2]. Схема строения piРНК-кластера 38С приведена на рис. 3.
Рис. 3.
Схема строения piРНК-кластера 38С. Светло-серым цветом обозначены деградировавшие фрагменты мобильных элементов (“молекулярные ископаемые”), темно-серым ‒ копии, имеющие значительное сходство с каноническими. Рамкой выделена область, потенциально способная формировать участки двухцепочечной РНК.
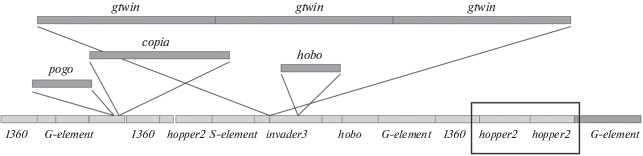
Несколько деградировавших копий мобильных элементов, подвергшихся неоднократным перестройкам, стали основой для формирования piРНК-кластера 38С. Одна из перестроек привела к дупликации значительного участка кластера, содержащего мобильные элементы G-element, 1360 и hopper2. Другая перестройка, по всей видимости, сопровождалась дупликацией и инверсией одной из копий элемента hopper2, что привело к образованию структуры, на которой, предположительно, может формироваться двухцепочечная РНК (выделена рамкой на рис. 3). О рекомбинационном происхождении этих фрагментов генома свидетельствуют идентичные перестройки в одних и тех же местах мобильных элементов.
Этот локус содержит несколько относительно недавно появившихся вставок мобильных элементов, принадлежащих к различным группам (процент их гомологии с последовательностями из канонического набора очень велик, а LTR идентичны). Вероятно, последней из них была вставка трех копий ретротранспозона gtwin, соединенных друг с другом общими LTR.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Не вызывает сомнения тот факт, что piРНК-кластеры могут быть устроены по-разному, что наводит на мысль о существовании различных механизмов их образования. Еще до открытия механизмов РНК-интерференции, при изучении случаев гибридного дисгенеза, исследователи наблюдали целый спектр негативных последствий вспышек транспозиционной активности для организма-хозяина, но нестабильность всегда оказывалась временным событием. Организм хозяина успешно брал “захватчика” под свой контроль [15‒18]. Для некоторых мобильных элементов – например, P-элемента – удалось показать существование молекулы-репрессора, кодируемой самим транспозоном. Для других транспозонов – в частности gypsy – были картированы генные локусы, контролирующие их перемещения, однако механизм такого репрессирования долгое время оставался неизвестным. Даже для P-элемента, авторегуляция которого за счет репрессора уже была детально изучена, обнаружили, что инсерция копии или ее фрагмента в определенные места генома может приводить к стабилизации дисгенной линии [19, 20]. С открытием РНК-интерференции стало очевидным, что любая из описанных выше моделей устойчивости к транспозициям может быть напрямую связана с воздействием разных видов коротких РНК.
В представленной работе мы попытались воссоздать ретроспективную последовательность образования трех piРНК-кластеров, контролирующих перемещения мобильных элементов в геноме D. melanogaster, изучить их детальное строение и сравнить между собой их внутреннее устройство.
Ранее показано, что при определенных условиях даже единичные копии мобильных элементов могут стать основой для формирования piРНК-кластеров [21]. Так, Оловников с соавт. [22] показали, что попадание в геном конструкции, содержащей транскрибируемый фрагмент I-элемента, приводит к образованию нового piРНК- и endo-siРНК-продуцирующего локуса. Короткие РНК обеих полярностей генерировались как с введенного в геном участка, так и с фланкирующих геномных последовательностей; при этом трансгенные транскрипты распознавались белком Piwi наравне с эндогенными I-специфичными piРНК. Подобное образование кластера piРНК, зависимое от сайта встраивания, наблюдается для всех классов мобильных элементов и добавляет новый уровень к piРНК-обеспечиваемой защите от экспансии мобильных элементов. Однако наши коллеги (Шпиз и др. [21] и Оловников и др. [22]) обнаружили, что не все копии мобильных элементов или инсерции трансгенных конструкций индуцируют образование мощных piРНК-кластеров. По всей видимости, формирование мастер-локуса de novo зависит от транскрипционного и хроматинового статуса мишени [23]. Можно предположить, что исследованные нами piРНК-кластеры формировались именно по такому пути: древняя инсерция мобильного элемента в определенный участок гетерохроматина по какой-то причине вызвала образование коротких РНК, после чего все последующие встроившиеся туда мобильные элементы оказывались “в ловушке”.
На сегодняшний день не удалось получить четкого представления о том, какой именно механизм играет роль пускового крючка для запуска экспрессии коротких РНК на определенных участках генома, что служит сигналом к их образованию и как регулируются эти процессы. Так, в 2016 году Guida и соавт. [4] показали, что способность локуса flamenco контролировать транспозиции мобильных элементов довольно изменчива и, по всей видимости, тесно связана с его хромосомным окружением, а также с “потерями” (делециями) и “приобретениями” (инсерциями) мобильных элементов внутри самого локуса. Они описали механизм косупрессии, вызванной инсерцией в эухроматин (не в локус flamenco!) новой копии gypsy, в линии D. melanogaster, до этого не подавляющей перемещения этого ретротранспозона. Авторы обнаружили, что “умолкание” gypsy сопровождалось “умолканием” других мобильных элементов, перемещения которых также регулирует flamenco. Этот механизм косупрессии зависел от посттранскрипционной регуляции с вовлечением путей как endo-siРНК, так и piРНК. Предполагается, что новая инсерция gypsy вызвала посттранскрипционный сайленсинг как смысловых, так и антисмысловых последовательностей gypsy, что в итоге привело к изменениям в активности локуса flamenco [4].
В структуре piРНК-кластеров 20А и flamenco нами обнаружены инвертированные последовательности, которые могут облегчать формирование двухцепочечных участков РНК-транскрипта. Подобные структуры довольно часто можно встретить в гетерохроматине. Иногда инвертируются сравнительно небольшие участки ДНК, но в некоторых случаях речь идет о тысячах пар нуклеотидов [24]. Выполняемая ими функция на данный момент остается неясной, однако их консервативность рассматривается как косвенное свидетельство важности выполняемых ими функций. Они могут участвовать в пока неизвестных механизмах репрессии транспозиций мобильных элементов или/и принимать участие в формировании устойчивых эпигенетических связей.
В 2006 году Bergman с соавт. [25] предположили, что “гнезда” подвергшихся перестройкам транспозонов, расположенные в β-гетерохроматине, служат ловушкой для новых инсерций мобильных элементов, таким образом играя роль компонента “адаптивного иммунитета” хозяйского генома. Разные линии дрозофилы заметно отличаются друг от друга по составу мобильных элементов, попавших в кластеры piРНК, что может быть причиной наблюдаемых различий в их способности подавлять разные семейства мобильных элементов. Присутствие в piРНК-кластерах, в том числе и в локусе flamenco, только одной копии транспозона свидетельствует о том, что это семейство мобильных элементов было подавлено механизмами РНК-интерференции сразу после попадания в локус, что предотвратило образование новых копий [25]. В 2013 году коллектив авторов из Франции проверил гипотезу Bergman и др. на разных линиях дрозофилы. Zanni и др. [8] обнаружили, что среди 52 различных мобильных элементов, присутствующих в локусе flamenco, подавляющее большинство (49) представлено в виде уникальной копии [8]. Это наблюдение согласуется с выдвинутой Bergman и др. [25] гипотезой о функционировании piРНК-кластеров как “ловушек транспозонов”. Результаты проведенного нами исследования также подтверждают эту гипотезу. В случае кластера 20А, как и в случае flamenco, только один элемент – roo – встречается более одного раза. Остальные инсерции уникальны. Возможно, древняя копия ретротранспозона roo, послужившая родоначальником кластера 20А, стала слишком сильно отличаться от современного варианта, поэтому в него смогла встроиться еще одна, “свежая”, копия этого мобильного элемента.
Основа кластера 38С была сформирована за счет дупликаций и инверсий участков генома, содержащих G-element, 1360 и hopper2 (здесь речь идет о рекомбинационных процессах, а не о многократном встраивании в локус одного и того же транспозона). А три копии gtwin могли попасть в локус вместе, в виде своеобразного тримера, либо дуплицироваться во время репликации. Ранее нами показано, что формирование таких структур характерно для gtwin [13].
Во многих исследованиях локуса flamenco выявлена его высокая структурная изменчивость. Так, между разными линиями D. melanogaster выявлено множество различий, возникающих в результате делеций, инсерций или дупликаций. Кроме того, при анализе последовательностей мобильных элементов, содержащихся в локусе flamenco, обнаружено, что многие из них, по всей видимости, только недавно встроились в этот локус. В работе Zanni и др. [8] в составе flamenco выявлено 12 новых мобильных элементов. Интересно, что 8 из них оказались близки к мобильным элементам D. simulans, D. sechellia, D. yakuba или D. erecta, что, по всей видимости, свидетельствует о случаях недавнего горизонтального переноса между видами Drosophila, принадлежащими к подгруппе melanogaster (см. [8, 12, 26] и элемент new в этой статье). Полученные нами результаты хорошо согласуются с этими данными: свежие инсерции мобильных элементов обнаружены и в локусе 20А (297, blood, roo), и в локусе 38С (pogo, copia, hobo, gtwin), и во flamenco (doc, G6, F-element, ретротранспозоны 412, blood, springer, HMS-Beagle2, MDG1 и др.). Следовательно, можно говорить о том, что piРНК-кластеры представляют собой довольно динамичные структуры.
Таким образом, ранее описанная гипотеза о “приручении чужака” [12, 27] подтверждена и результатами проведенного нами исследования. Для мобильного элемента выгоднее всего попасть в “девственный” геном, не содержащий гомологичных ему последовательностей. Наилучший способ найти такой геном – это заразить животных близкого вида посредством горизонтального переноса. Попав в новый для себя организм, мобильный элемент не подвергается никаким репрессирующим механизмам и его копии успешно “расселяются” по геному (период нестабильности). В этом случае вставки новых копий в какой-либо мастер-локус регуляции довольно вероятны, так как, по всей видимости, в этот процесс вовлечены неизвестные пока механизмы, делающие эти локусы ловушками для новых инсерций. Попадание нового элемента в piРНК-кластер приводит к образованию соответствующих коротких РНК, репрессирующих его дальнейшую экспрессию. Наступает период стабильности. В пользу этой гипотезы свидетельствуют данные, что в большинстве случаев в мастер-локусе регуляции присутствует только одна копия подавляемого локусом элемента. Последующие делеционные перестройки локуса могут привести к потере одной или нескольких копий мобильного элемента; при этом присутствующие в геноме копии элемента снова приобретают способность размножаться и начинается новый виток нестабильности. Таким образом, транспозиционные всплески, периоды стабильности/нестабильности напрямую связаны с мутационными событиями, затрагивающими piРНК-кластеры, подобные flamenco.
Раунды высокой транспозиционной активности могут вызывать генетическую нестабильность и сопряженные с нею вредные мутации, но нет никаких сомнений, что они также играют важную роль в эволюции видов. Геном дрозофилы можно рассматривать как некий отпечаток, или даже исторический срез, таких транспозиционных всплесков для большинства семейств мобильных элементов, так как в нем присутствуют и древние, и недавно встроившиеся копии. К ним относятся и идентифицированные нами blood, stalker, roo; тогда как древних копий pifo в геноме D. melanogaster не обнаружено, из чего можно сделать вывод о недавно произошедшем случае его горизонтального переноса.
Таким образом, вопросы о начальных этапах формирования piРНК-кластеров, последующих перестройках и детальных механизмах их функционирования до сих пор остаются открытыми, и для ответа на них необходимы дальнейшие исследования.
Работа поддержана грантом Программы фундаментальных исследований Президиума РАН “Постгеномные технологии и перспективные решения в биомедицине” по теме: “Сборка РНП-частиц некодирующих транскриптов и транскриптов с отложенной трансляцией”.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Kapitonov V.V., Jurka J. (2003) Molecular paleontology of transposable elements in the Drosophila melanogaster genome. Proc. Natl. Acad. Sci. USA. 100, 6569–6574.
Brennecke J., Aravin A.A., Stark A., Dus M., Kellis M., Sachidanandam R., Hannon G.J. (2007) Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell. 128, 1089–1103.
Saito K., Nishida K.M., Mori T., Kawamura Y., Miyoshi K., Nagami T., Siomi H., Siomi M.C. (2006) Specific association of Piwi with rasiRNAs derived from retrotransposon and heterochromatic regions in the Drosophila genome. Genes Dev. 20, 2214–2222.
Guida V., Cernilogar F.M., Filograna A., De Gregorio R., Ishizu H., Siomi M.C., Schotta G., Bellenchi G.C., Andrenacci D. (2016) Production of small noncoding RNAs from the flamenco locus is regulated by the gypsy retrotransposon of Drosophila melanogaster. Genetics. 204(2), 631–644.
Goriaux C., Théron E., Brasset E., Vaury C. (2014) History of the discovery of a master locus producing piRNAs: the flamenco/COM locus in Drosophila melanogaster. Front. Genet. 5, 257. https://doi.org/10.3389/fgene.2014.00257
Gilboa E., Mitra S., Goff S., Baltimore D. (1979) A detailed model of reverse transcription and a test of crucial aspects. Cell. 18(1), 93‒100.
Luciw P., Leung N. (1992) Mechanisms of retroviral replication. In: The Retroviridae. Ed. Levy J.A. New York: Plenum Press, 159‒298.
Zanni V., Eymery A., Coiffet M., Zytnicki M., Luyten I., Quesneville H., Vaury C., Jensen S. (2013) Distribution, evolution, and diversity of retrotransposons at the flamenco locus reflect the regulatory properties of piRNA clusters. Proc. Natl. Acad. Sci. USA. 110(49), 19842‒19847.
Котнова А.П., Карпова Н.Н., Феоктистова М.А., Любомирская Н.В., Ким А.И., Ильин Ю.В. (2005) Ретротранспозон gtwin: структурный анализ и распределение в линиях дрозофилы. Генетика. 41(1), 23‒29.
Глухов И.А., Карпова Н.Н., Котнова А.П., Любомирская Н.В., Ильин Ю.В. (2004) Структурные особенности третьей открытой рамки считывания ретротранспозона gtwin в различных линиях Drosophila melanogaster. ДАН. 399(2), 257‒259.
Котнова А.П., Феоктистова М.А., Глухов И.А., Саленко В.Б., Любомирская Н.В., Ким А.И., Иль-ин Ю.В. (2006) GTWIN – ретротранспозон дрозофилы, специфичный для подгруппы Melanogaster. ДАН. 409(5), 691‒693.
Kotnova A.P., Glukhov I.A., Karpova N.N., Salenko V.B., Lyubomirskaya N.V., Ilyin Yu.V. (2007) Evidence for recent horizontal transfer of gypsy-homologous LTR-retrotransposon gtwin into Drosophila erecta followed by its amplification with multiple aberrations. Gene. 396(1), 39‒45.
Стефанов Ю.Э., Котнова А.П., Глухов И.А., Любомирская Н.В., Ильин Ю.В. (2009) Особенности ретротранспозиционной активности эндогенного ретровируса gtwin в линии Г32 Drosophila melanogaster. ДАН. 424(4), 555‒558.
Саленко В.Б., Котнова А.П., Глухов И.А., Стефанов Ю.Э., Сурков С.А., Любомирская Н.В., Иль-ин Ю.В. (2011) Редкое подсемейство ретротранспозона gtwin, характеризующееся мутацией в сайте связывания тРНК-затравки для синтеза минус-цепи кДНК, амплифицировано в линии Г-32 Drosophila melanogaster. ДАН. 436(4), 549–552.
Bingham P.M., Kidwell M.G., Rubin G.M. (1982) The molecular basis of P-M hybrid dysgenesis: the role of the P element, a P-strain-specific transposone family. Cell. 29, 995‒1004.
Engels W.R. (1989) P elements in Drosophila melanogaster. In: Mobile DNA. Eds Berg D.E., Howe M.M. Washington, DC: American Society for Microbiology, 437‒484.
Finnegan D.J. (1989) The I factor and I-R hybrid dysgenensis in Drosophila melanogaster. In: Mobile DNA. Eds Berg D.E., Howe M.M. Washington, DC: American Society for Microbiology, 503‒517.
Bucheton A. (1990) I transposable elements and I-R hybrid dysgenensis. Trends Genet. 6, 16‒19.
Marin L., Lehmann M., Nouaud D., Izaabel H., Anxolabehere D., Ronsseray S. (2000). P-element repression in Drosophila melanogaster by a naturally occurring defective telomeric P copy. Genetics. 155(4), 1841–1854.
Stuart J.R., Haley K.J., Swedzinski D., Lockner S., Kocian P.E., Merriman P.J., Simmons M.J. (2002) Telomeric P elements associated with cytotype regulation of the P transposon family in Drosophila melanogaster. Genetics. 162, 1641–1654.
Shpiz S., Ryazansky S., Olovnikov I, Abramov Yu., Kalmykova A. (2014) Euchromatic transposon insertions trigger production of novel Pi- and endo-siRNAs at the target sites in the Drosophila germline. PLoS Genet. 10(2), e1004138.https://doi.org/10.1371/journal.pgen.1004138
Olovnikov I., Ryazansky S., Shpiz S., Lavrov S., Abramov Y., Vaury C., Jensen S., Kalmykova A. (2013) De novo piRNA cluster formation in the Drosophila germ line triggered by transgenes containing a transcribed transposon fragment. Nucleic Acids Res. 41, 5757–5768.
Akulenko N., Ryazansky S., Morgunova V., Komarov P., Olovnikov I., Vaury C., Jensen S., Kalmykova A. (2018) Transcriptional and chromatin changes accompanying de novo formation of transgenic piRNA clusters. RNA. 24(4), 574‒584.
Котнова А.П., Саленко В.Б., Любомирская Н.В., Ильин Ю.В. (2009) Структурная организация гетерохроматина D. melanogaster: инвертированные повторы кластеров мобильных элементов. ДАН. 429(1), 120–122.
Bergman C.M., Quesneville H., Anxolabéhère D., Ashburner M. (2006) Recurrent insertion and duplication generate networks of transposable element sequences in the Drosophila melanogaster genome. Genome Biol. 7(11):R112.
Bartolomé C., Bello X., Maside X. (2009) Widespread evidence for horizontal transfer of transposable elements across Drosophila genomes. Genome Biol. 10(2), R22.
Salenko V.B., Kotnova A.P., Lyubomirskaya N.V., Ilyin Yu.V. (2008) Russian-European Workshop on DNA Repair and Epigenetic Regulation of Genome Stability, St. Petersburg, Russia. Abstract book, Abstract 23, p. 81.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


