Молекулярная биология, 2020, T. 54, № 4, стр. 562-579
Изменение экспрессии генов ферментов первичного метаболизма и пластидных транспортеров в ходе роста культуры Chlamydomonas reinhardtii
Р. К. Пузанский a, Д. А. Романюк a, А. А. Кирпичникова a, М. Ф. Шишова a, *
a Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: mshishova@mail.ru
Поступила в редакцию 24.11.2019
После доработки 23.01.2020
Принята к публикации 07.02.2020
Аннотация
В миксотрофной культуре Chlamydomonas reinhardtii проведен анализ интенсивности экспрессии генов, кодирующих ферменты первичного метаболизма и пластидные транспортеры. Уровень экспрессии примерно 70% исследованных генов изменяется в процессе роста культуры, при этом на завершающих этапах роста экспрессия снижается. Общий профиль экспрессии в экспоненциальной фазе меняется более заметно, чем в стационарной. В середине фазы экспоненциального роста изменения транскрипции приводят к существенной перестройке метаболизма, в том числе к запуску синтеза липидов, что сопровождается изменением баланса энергии и восстановительной способности между пластидой и цитозолем.
ВВЕДЕНИЕ
Зеленая микроводоросль Chlamydomonas reinhardtii – модельный объект, используемый генетиками, биохимиками и физиологами. Одним из широко распространенных способов выращивания C. reinhardtii является периодическое культивирование, при котором популяция клеток растет в неизменном и необновляемом объеме среды без дополнительного снабжения питательными веществами. Такие периодические культуры могут быть автотрофными, гетеротрофными или миксотрофными [1, 2]. В последнем случае в качестве источника углерода используют ацетат, что заметно ускоряет накопление клеточной массы C. reinhardtii [3].
К настоящему времени накоплены данные о строении и механизмах работы фотосинтетичес-кого аппарата [4, 5], а также об основах минерального питания [6‒8], устойчивости к стрессорам [9‒11] и процессах старения культуры хламидомонады [12]. Однако сведения о динамике метаболизма хламидомонады в процессе роста миксотрофной периодической культуры достаточно фрагментарны. Опубликованные данные либо описывают короткие периоды экспоненциального роста (в течение нескольких часов после пересадки), либо, напротив, направлены на изучение метаболических перестроек в стационарной фазе [13‒15]. Показано, что в ходе стационарной фазы увеличивается содержание насыщенных жирных и органических кислот, тогда как концентрация некоторых аминокислот снижается [15‒18]. Предполагается, что накопление жирных кислот направлено на депонирование метаболитов, характерное для завершения роста. Изменение профиля метаболитов может отражать также деструктивные процессы, обусловленные адаптацией клеток хламидомонады к новому физиологическому состоянию, соответствующему старению [19]. Напротив, начальный этап экспоненциального роста С. reinhardtii (в первые 22 ч после пересадки клеток) характеризовался нелинейными изменениями содержания таких метаболитов, как углеводы, жирные кислоты, а также ряда интермедиатов энергетических циклов [20].
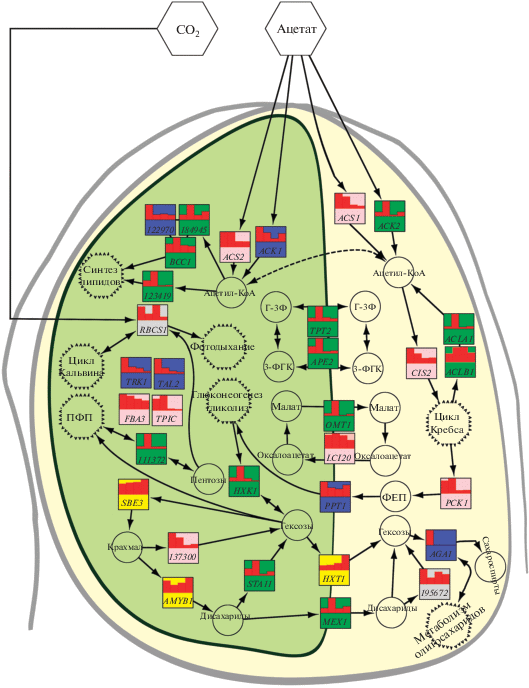
Настоящая статья является логическим продолжением метаболомного анализа клеток С. reinhardtii, выращенных на средах с добавлением ацетата [21]. Сравнение профилей метаболитов на разных стадиях развития культуры позволило выявить основные метаболические перестройки, особенно в ходе экспоненциального роста [21]. Анализ этих метаболомных данных позволил “отобрать” ключевые ферменты и транспортеры хлоропласта хламидомонады, которые могли быть наиболее значимыми в метаболических перестройках, опосредованных ацетатом. Анализ метаболических перестроек, происходящих во время экспоненциальной фазы роста миксотрофной культуры хламидомонады, на протяжении более длительного периода, охватывающего экспоненциальную и стационарную фазы (1–6 суток), показал, что динамичные перестройки метаболома тесно коррелируют с такими физиологическими процессами, как фотосинтез, дыхание, старение и др. [21]. Доказано существование различий в метаболитных профилях как в период экспоненциального роста (1–4-е сутки), так и после перехода к стационарной фазе (4–6-е сутки). Показано значительное и нелинейное изменение содержания жирных кислот и ацилглицеринов, а также сахаров, включая дисахариды, что может отражать разницу в уровне синтеза, катаболизма и депонирования различных соединений [21]. Тем самым установлено, что во время экспоненциального роста изменяются пути ассимиляции углерода и характер энергетического обмена в клетках водоросли. Оба процесса тесно связаны с механизмами включения ацетата в метаболизм хламидомонады при миксотрофном питании. Следовательно, можно ожидать изменения активности целого ряда ферментов, участвующих в гликолизе, циклах Кребса и Кальвина, синтезе жирных кислот и др. (рис. 1, по данным [21]). Учитывая тесное взаимодействие различных клеточных органелл как в катаболических, так и в анаболических процессах фотосинтезирующей, но растущей в миксотрофных условиях микроводоросли, большое значение приобретают транспортеры, локализованные на мембране хлоропласта. В связи с этим рассмотрим основные ферменты (и их гены) первичного метаболизма, в том числе ферменты, вовлеченные в метаболизм ацетата, а также транспортеры хлоропластов и гены, их кодирующие.
Рис. 1.
Схема, иллюстрирующая роль продуктов исследованных генов в метаболизме C. reinhardtii и их взаимосвязь.
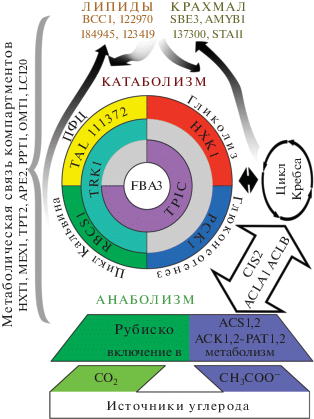
Метаболизм ацетата начинается с образования комплекса с КоА. Клетка хламидомонады способна превращать ацетат в ацетил-КоА двумя способами. При первом способе ацетил-КоА-синтаза превращает ацетат в ацетил-КоА. У хламидомонады этот фермент кодируют гены ACS1–4. Второй путь – двухэтапный. Сначала ацетат фосфорилируется ферментом ацетаткиназой, кодируемой генами ACK1 и 2. На втором этапе в результате работы фосфоацетилтрансферазы, кодируемой генами PAT1 и 2, образуется ацетил-КоА. Недавние молекулярно-биологические и протеомные исследования указывают на то, что хламидомонада обладает двумя ферментными системами АСК‒PAT. Установлено, что первая пара PAT1 и ACK2 локализуется в митохондриях, тогда как вторая ‒ PAT2 и ACK1, находится, предположительно, в хлоропластах [22‒24]. Установлено, что гены ACK1 и PAT2 локализованы на хромосоме 9, а ACK2 и PAT1 ‒ на хромосоме 17 [24].
Ацетил-КоА ‒ общее для различных биохимических путей соединение, регуляция усвоения которого определяет активность ряда важных биологических процессов. Ацетил-КоА включается в цикл трикарбоновых кислот цитратсинтазой, кодируемой генами CIS1 и 2. При этом CIS2 вовлечен преимущественно в глиоксилатный цикл, а CIS1 ‒ в цикл Кребса [25]. Ингибирование цитратсинтазы путем подавления экспрессии гена CIS с помощью интерференции приводило к росту уровня триацилглицерина (ТАГ). Напротив, сверхэкспрессия гена CIS приводит к снижению уровня ТАГ в 2 раза. Ввод ацетильных групп в цикл Кребса и их выход из него может осуществлять также цитратлиаза, субъединицы которой кодируются генами ACLA1 и ACLB1 хламидомонады [26].
Еще один механизм, обеспечивающий отток углерода из цикла Кребса и активацию глюконеогенеза, заключается в превращении оксалоацетата в фосфоенолпируват фосфоенолпируваткарбоксикиназой (PCK1) в условиях избытка АТР [27].
Реакция превращения ацетил-КоА в малонил-КоА, катализируемая ацетил-КоА-карбоксилазой (ACC), – ключевая в биосинтезе липидов [28]. У водорослей синтез липидов происходит преимущественно в пластидах. Хлоропластная форма ACC состоит из четырех субъединиц: биотин-карбоксил- переносящего белка (BCC1), биотинкарбоксилазы (CHLREDRAFT_122970) и двух субъединиц карбоксилтрансферазы (CHLREDRAFT_184945 – α-субъединица). Наряду с этим ацетильные группы могут участвовать в синтезе жирнокислотных цепей с помощью ацилтрансфераз, к числу которых относится FabH ‒ синтаза III 3-оксоацл-ацилпереносящий белка (KASIII), кодируемая у хламидомонады геном CHLREDRAFT_123419.
Изменение условий культивирования может существенно влиять на активность фиксации углерода и метаболизм сахаров. Поэтому особое регуляторное значение могут иметь гены, кодирующие ключевые ферменты цикла Кальвина, пентозофосфатного пути (ПФП), гликолиза и метаболизма сахаров: малую субъединицу Рубиско (RBCS1), фруктозобисфосфатальдолазу (FBA3), транскетолазу (TRK1), трансальдолазу (TAL2), PfkB-подобную карбогидраткиназу (CHLREDRAFT_111372), гексокиназу (HXK1), триозофосфатизомеразу (TPIC) и α-галактозидазу (AGA1). Изменения метаболизма сахаров могут влиять на баланс между расходованием и депонированием запасных веществ, в том числе крахмала. Ветвящий фермент SBE3 катализирует образование 1,6-α-гликозидных связей между цепями остатков глюкозы с образованием амилопектина ‒ основного компонента крахмала. Расщепление крахмала осуществляют фосфорилаза крахмала (CHLREDRAFT_137300, PHOB), изоамилазы и амилазы, в том числе β-амилаза (AMYB1). Образующаяся при этом мальтоза гидролизуется, в том числе 4-α-глюкантрансферазой STA11. Расщепление трегалозы катализирует α,α-трегалаза (CHLREDRAFT_195672).
Еще один ключевой регулятор энергетического взаимодействия пластид с цитозолем, участвующий в изменении интенсивности и направленности гликолиза и глюконеогенеза, ‒ пластидные транспортеры соединений С3 [27]. К ним, например, можно отнести триозофосфат/фосфатный антипортер TPT [29]. К семейству транспортеров TPT относится белок APE2, предположительно локализованный в зоне контакта внешней и внутренней мембран хлоропласта. В клетках C. reinhardtii описаны пластидный 2-оксоглутарат/малатный антипортер (OMT) [30], белок LCI20, ортолог 2-оксоглутарат/малатного обменника оболочки хлоропластов арабидопсиса [31, 32]. В геноме хламидомонады идентифицированы гены пластидных транспортеров гексоз (HXT1), мальтозы (MEX1), а также фосфат/фосфоенолпируватного антипортера PPT1.
Таким образом, изменения энергетического и пластического метаболизма, наблюдаемые в процессе развития миксотрофной периодической культуры хламидомонады, могут опосредоваться изменениями активности нескольких ферментов и транспортеров (рис. 1). Активность этих белков может регулироваться на разных уровнях, в том числе, за счет изменения транскрипции генов, их кодирующих. В данном исследовании проанализирован уровень транскрипции 32 генов (табл. 1), кодирующих ферменты ассимиляции ацетата, цикла Кальвина, гликолиза и др., а также хлоропластных транспортеров, активность которых может меняться в процессе экспоненциального роста культуры C. reinhardtii.
Таблица 1.
Проанализированные гены первичного метаболизма и хлоропластных транспортеров
| Локус tag | Ген | Продукт | Праймер |
|---|---|---|---|
| CHLREDRAFT_129982 | ACK1 | Ацетаткиназа | CCTGTCGGGTCGGCAGTG GTGGTCCTTGGCGGGAATCT |
| CHLREDRAFT_128476 | ACK2 | Ацетаткиназа | GCGTCTTGGTCCTGAAC ACATTCTCCTTGATGAAATCC |
| CHLREDRAFT_196544 | ACLA1 | Субъединица A АТP-цитратлиазы | TGCCTATCCAGGTGGTTCAGTTCA CGGGCGGTGGCGAAATCAAT |
| CHLREDRAFT_186611 | ACLB1 | Субъединица B АТP-цитратлиазы | CGAGACTCCATCTGTCGCTGCT CTGCGGAAGGAGGCGTAGTTGAT |
| CHLREDRAFT_194063 | ACS1 | Ацетил-КоА-синтетаза | CTGACTCTATTGTTGCGAAGG CGTTGATGTTGGACTGGC |
| CHLREDRAFT_139750 | ACS2 | Ацетил-КоА-синтетаза | GCAGGACAAGCAACAGTACGAGG TCCAGGCAGTTGTAGGCGATG |
| CHLREDRAFT_116873 | AGA1 | α-Галактозидаза | GATGACTGCTGGATGGAGAA TACACGCCGAACTTGAGG |
| CHLREDRAFT_183141 | AMYB1 | β-Амилаза | AGCCAGTGCGTTCTCAGA AAGCGGTCCTCGTCTATCATT |
| CHLREDRAFT_205633 | APE2 | Переносчик триозофосфатов | GCTCGTCCGTGCTGTTCTTCA CCTTCTTGTGCTTGTGCTTGCT |
| CHLREDRAFT_185478 | BCC1 | Биотин-карбоксил-переносящий белок (субъединица ацетил-КоА-карбоксилазы) | CCGAGGAGGTTGAGGCTGCTA CAGGCTGAAACGCTTGCTCTTC |
| CHLREDRAFT_105734* | CBLP, RACK1 | β-субъединица G-белка | GTCATCCACTGCCTGTGCTT CCTTCTTGCTGGTGATGTTG |
| CHLREDRAFT_111372 | CHLREDRAFT_111372 | Киназа сахаров типа PfkB | GCTTCCCGACCTTCTTTG CCGCCCACAATGATGATG |
| CHLREDRAFT_122970 | CHLREDRAFT_122970 | Биотин-карбоксилаза (субъединица ацетил-КоА–карбоксилазы) | GCGGCGAGATTGCTGTG GGGATGTTGAGGTAGGACT |
| CHLREDRAFT_123419 | CHLREDRAFT_123419 | Синтаза III 3-оксоацил-[ацил-переносящего белка] | GCCTCCTACGCCAACATCCACAT GCATCACCAGCCAGTCCACAGA |
| CHLREDRAFT_137300 | CHLREDRAFT_137300 | Фосфорилаза крахмала | GTTGTGGAGGGCGACGA TGTGGCAGCGGTGTTCT |
| CHLREDRAFT_184945 | CHLREDRAFT_184945 | α-Карбоксилтрансфераза (субъединица ацетил-КоА–карбоксилазы) | CGGTATCGGCAGCATCAAT ACATCTCACGCAGGTTCAC |
| CHLREDRAFT_195672 | CHLREDRAFT_195672 | α,α-Трегалаза | GACTCCTACTGGGTCATCCTGGG CAGGCTGAAACGCTTGCTCTTC |
| CHLREDRAFT_194915 | CIS2 | Цитратсинтаза | AACACCACCGCCGTCATC TCACATCAATCACCTCCGTCG |
| CHLREDRAFT_24459 | FBA3 | Фруктозо-1,6-дифосфат-альдолаза | GCCCTGATGATGAAGTCGTCGG CAACGGTGCCAGCGGTCTT |
| CHLREDRAFT_21582 | HXK1 | Гексокиназа | CACTCGGCATCCTCATCCTA CGTCGGCAGCATCATCAT |
| CHLREDRAFT_196325 | HXT1 | Транспортер гексоз | CTGATCGGCGTGCTGCTGTTC ACGGAGGATGAGAAGTAGACCAGGG |
| CHLREDRAFT_205915 | LCI20 | 2-Оксоглутарат/малат транслокатор | GCGGCGACCACCAATGAGAT GCGACTTGATGATGGGCACAAAGA |
| CHLREDRAFT_205893 | MEX1 | Экспортер мальтозы | GATGGAAGGGCGACGGACAAC CATTCTGGACGACTTGCGGAAGC |
| CHLREDRAFT_185488 | OMT1 | 2-Оксоглутарат/малат транслокатор | GGAGTGCCTGAACAACAAC AGAAGTAGTGCGAGTAGAAGT |
| CHLREDRAFT_19661 | PCK1 | Фосфоенолпируват-карбоксикиназа | TTATGAACAACGCCCGCATCC GCTCAGTCACGCCCATCTCAGT |
| CHLREDRAFT_144475 | PPT1 | Фосфат/фосфоенолпируват антипортер | GTCAAGTCGTGGCAGCAGTT GAAGAAGGGCGTCGTGGAG |
| CHLREDRAFT_82986 | RBCS1 | Малая субъединица Рубиско | CTGAAGCCCGCCGTCAAGG AGAGGAGGCAGGTAGGAGAAGGT |
| CHLREDRAFT_195590* | RPL19 | Рибосомный белок L19 | TCGGTTCTGAACTGCGGTCTG CGGGCTGCTTACGGAAGATGA |
| CHLREDRAFT_94079 | SBE3 | Ветвящий фермент | CCAACTGCGTCACCATCG GCTTCATCACCTCAATCCACTT |
| CHLREDRAFT_140452 | STA11 | 4-α-Глюканотрансфераза | GCTCATCAAGGACGGACTGC ACTGCCACCACGCCAACT |
| CHLREDRAFT_196431 | TAL2 | Трансальдолаза | CCGCCTTTCCACCATTGT GCTGTTGCTGTTCTTGCC |
| CHLREDRAFT_26265 | TPIC | Триозофосфатизомераза | ACGAGGTGGTGGCGAAGAAGACT GGCGTCCAGAACCTTGAACACAGA |
| CHLREDRAFT_182319 | TPT2 | Триозофосфат/фосфат антипортер | GCTCTGCAAAGGCACCAAGTTCAA GCCCTTATCGGACTCGCCATCT |
| CHLREDRAFT_141319 | TRK1 | Транскетолаза | CGACGCCATCAACAAGTCCAAGT TCGTAGCCAGTCAGGTGCATCAT |
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
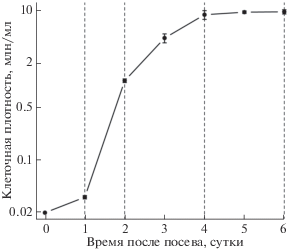
Объект и культивирование. Культура C. reinhardtii сс-124 получена из коллекции кафедры генетики и биотехнологии биологического факультета Санкт-Петербургского государственного университета. Культуру поддерживали аксенично на жидкой среде TAP (Tris Acetate Phosphate) [33] в конических стеклянных колбах объемом 500 мл, которые содержали 250 мл среды, при периодическом перемешивании, естественной концентрации CO2 и постоянном освещении люминесцентными лампами холодного света с интенсивностью 3000 лк. Плотность культуры определяли подсчетом клеток в камере Горяева. Отбор проб проводили 4 раза: в начале (1-е сутки), середине (2-е суток) и в конце (4-е сутки) экспоненциальной, а также в стационарной (6-е сутки) фазе развития культуры (рис. 2) в четырех биологических повторностях.
Рис. 2.
Рост клеточной плотности периодической миксотрофной культуры C. reinhardtii, и точки отбора проб (пунктирные линии), интервалы – SD.
Выделение РНК. Клетки хламидомонады (107 клеток) осаждали центрифугированием (5 мин, 3000 g), осадок ресуспендировали в 1 мл реактива Пурзол (PureZOL™, “Bio-Rad”, США) [34] и перемешивали. Пробы инкубировали при комнатной температуре в течение 5‒10 мин, гомогенат замораживали. После оттаивания пробы центрифугировали (10 мин, 12 000 g и 4°C), к гомогенату добавляли 0.2 мл хлороформа, интенсивно перемешивали в течение 15‒20 с и выдерживали при комнатной температуре (5 мин) с периодическим перемешиванием. Затем пробы центрифугировали (15 мин, 12500 g и 4°С), к верхней бесцветной водной фазе добавляли 0.5 мл изопропанола, инкубировали в течение 10 мин при комнатной температуре. Пробу центрифугировали (10 мин, 12 500 g, 4°С). Супернатант удаляли, осадок промывали, добавляя 1 мл 75%-ного этанола, перемешивали и центрифугировали (5 мин, 12 500 g и 4°С). Осадок подсушивали в ламинаре и растворяли в стерильной воде. Качество и количество выделенной РНК проверяли спектрофотометрически (спектрофотометр NanoDrop 1000, “Thermo Fisher Scientific”, США). Пробы хранили при –80°С.
Получение кДНК. Препарат РНК очищали от ДНК, используя обработку ДНКазой (DNase I, RNase-free, “Thermo Fisher Scientific”), согласно инструкциям производителя. кДНК cинтезировали с использованием Oligo(dT)-праймера (“Бигль”, Россия) и обратной транскриптазы M-MuLV (“Thermo Fisher Scientific”) согласно инструкциям производителя с применением ингибитора РНКаз (Thermo Scientific RiboLock RNase Inhibitor, США). Пробы хранили при –80°С. Качество кДНК оценивали по длине полученных фрагментов с помощью электрофореза в полиакрил- амидном геле.
Дизайн праймеров и ПЦР в реальном времени. Праймеры подбирали с использованием программы Beacon-Designer 8 (“Sigma Aldrich”, США). Длина праймеров составляла около 20 н. (табл. 1), длина ампликона 80–250 н., предсказанная температура плавления 60°С, доля GC не более 60%. Качество праймеров оценивали с помощью ПЦР в реальном времени (ПЦР-РВ) с последующим анализом кривых накопления продуктов реакции и кривых плавления, а также длины синтезированных фрагментов с помощью электрофореза в полиакриламидном геле. ПЦР-РВ проводили с использованием SYBR Green I (“Синтол”, Россия) на амплификаторе CFX96 (“BioRad”) в следующем режиме: 95°С ‒ 15 с, 60°С – 50 с, 45 циклов.
Математическая обработка. Интенсивность экспрессии определяли в трех биологических и трех аналитических провторностях. Основываясь на опубликованных данных [10, 35, 36], в качестве гена сравнения использовали ген CBLP (RACK1), кодирующий полипептид, сходный с β-субъединицей G-белка (G protein β subunit-like polypeptide) [37]. Был протестирован также ген RPL19, стабильность экспрессии которого показана ранее [38]. Относительный уровень экспрессии рассчитывали по формуле 2–ΔCt [39]. Изменения относительного уровня экспрессии, рассчитанные с использованием указанных выше генов сравнения, были сходными, поэтому в работе приведены значения, рассчитанные только с использованием CBLP.
Анализ проводили с помощью языка программирования R 3.3 [40]. Однофакторный дисперсионный анализ ‒ с помощью функции aov, переменные проверяли на нормальность распределения с помощью теста Шапиро‒Уилка (функция shapiro.test), а гомогенность дисперсии ‒ с помощью теста Флигера‒Киллена (функция fligner.test). Иерархический кластерный анализ проводили с использованием пакета pvclust [41]. В качестве дистанции использовали величину: 1-r, где r – коэффициент корреляции Пирсона. Для кластеризации использовали метод Варда. Кластеры выделяли на основе их статистической значимости по значению AU p (approximately unbiased p-value) [41]. Число кластеров, характеризующих динамику экспрессии, выбрано на основе анализа “графика локтя” (или “каменистой осыпи”, scree plot), учитывающего зависимость дистанций между кластерами и число кластеров (агломераций).
Анализ МГК выполнен с помощью пакета pcaMethods [42]; (О)ПЛС-ДА – пакета ropls [43]; МЕБА ‒ пакета timecourse [44]. Связь переменной со временем оценивали по значению T2 теста Хотеллинга.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Изменение уровня экспрессии генов в процессе развития культуры
Пробы С. reinhardtii отбирали 4 раза в процессе развития культуры: в начале, середине и конце экспоненциальной фазы, а также в стационарной фазе (рис. 2). Анализ накопления транскриптов 32 генов хламидомонады, которые кодируют ряд ключевых ферментов метаболизма и нескольких транспортеров метаболитов (рис. 1, табл. 1), выявил существенные изменения уровня их экспрессии в ходе развития культуры, выращенной в миксотрофных условиях (табл. 2, 3). Последующий дисперсионный анализ показал, что уровни экспрессии 22 из 32 генов статистически значимо (p < 0.05) изменялись на проверенных стадиях развития культуры. Данные апостериорного теста Тьюки приведены в табл. 3. Сравнение культуры в конце экспоненциальной фазы (4-е сутки) и в стационарной фазе (6-е сутки) показало, что изменение уровня экспрессии только одного гена ‒ HXT1, было значимым p< 0.05. При этом сравнение, проведенное на первые и вторые сутки, а также на вторые и четвертые, выявило 15 и 16 таких генов соответственно.
Таблица 2.
Относительный уровень экспрессии генов, кодирующих ферменты центрального метаболизма и пластидные транспортеры, на разных стадиях развития периодической миксотрофной культуры Chlamydomonas reinhardtii
| Ген | Относительный уровень экспрессии*, сутки после посева, ±SD | |||
|---|---|---|---|---|
| 1 | 2 | 4 | 6 | |
| ACLB1 | 4.07 ± 2.14 | 7.13 ± 0.67 | 7.41 ± 1.91 | 4.49 ± 1.32 |
| OMT1 | 92.59 ± 73.62 | 558.1 ± 151.58 | 23.32 ± 25.69 | 66.83 ± 55.39 |
| BCC1 | 5.99 ± 1.01 | 10.26 ± 0.6 | 5.03 ± 2.6 | 4.47 ± 3.71 |
| HXK1 | 0.17 ± 0.02 | 0.64 ± 0.16 | 0.12 ± 0.02 | 0.03 ± 0.01 |
| AMYB1 | 13.74 ± 5.34 | 8.72 ± 1.83 | 11.25 ± 1.22 | 17.51 ± 4.61 |
| SBE3 | 5.15 ± 1.66 | 5.68 ± 0.57 | 5.93 ± 1.04 | 7.37 ± 1.07 |
| TRK1 | 92.39 ± 12.57 | 36.42 ± 1.2 | 28.26 ± 8.22 | 10.12 ± 2.67 |
| ACS2 | 19.42 ± 6.89 | 18.83 ± 3.03 | 7.22 ± 2.2 | 7 ± 1.68 |
| ACK1 | 8.27 ± 3.69 | 5.03 ± 0.62 | 1.86 ± 0.26 | 4.59 ± 1.71 |
| RBCS1 | 149667.8 ± 40946.31 | 70174 ± 16065.76 | 169352.67 ± 108429.29 | 35939.86 ± 17346.31 |
| ACK2 | 21.43 ± 15.67 | 74.83 ± 8.74 | 7.02 ± 6.58 | 21.39 ± 18.89 |
| CHLREDRAFT_123419 | 3.79 ± 2.3 | 9.77 ± 1.73 | 0.62 ± 0.58 | 0.87 ± 0.18 |
| LCI20 | 0.8 ± 0.15 | 0.91 ± 0.17 | 0.78 ± 0.18 | 0.5 ± 0.18 |
| TPIC | 603.06 ± 220.58 | 789.43 ± 133.07 | 89.71 ± 30.41 | 63.85 ± 23.85 |
| APE2 | 21.22 ± 9.26 | 44.81 ± 6.78 | 6.28 ± 3.73 | 8.04 ± 3.04 |
| FBA3 | 827.45 ± 50.95 | 726.25 ± 53.77 | 563.91 ± 73.5 | 468.44 ± 235.13 |
| PCK1 | 220.01 ± 65.76 | 263.99 ± 8.27 | 53.01 ± 8.07 | 57.19 ± 27.85 |
| MEX1 | 1.45 ± 0.72 | 4.38 ± 0.2 | 1.37 ± 0.18 | 1.94 ± 1.32 |
| TAL2 | 11.15 ± 4.04 | 4.38 ± 0.93 | 2.12 ± 0.43 | 1.33 ± 0.28 |
| CHLREDRAFT_195672 | 0.09 ± 0.03 | 0.04 ± 0.01 | 0.07 ± NA | 0.06 ± 0 |
| CHLREDRAFT_184945 | 6.26 ± 2.1 | 18.26 ± 4.15 | 5.88 ± 2.74 | 10.19 ± 4.14 |
| STA11 | 7.92 ± 2.65 | 16.94 ± 3.22 | 1.89 ± 0.68 | 4.71 ± 3.08 |
| CHLREDRAFT_137300 | 57.56 ± 6.61 | 39.47 ± 7.54 | 10.75 ± 3.87 | 14.84 ± 6.26 |
| CHLREDRAFT_111372 | 0.37 ± 0.17 | 2.12 ± 0.42 | 0.3 ± 0.11 | 0.33 ± 0.09 |
| CIS2 | 102.4 ± 23.58 | 65.82 ± 3.85 | 34.82 ± 7.65 | 37.7 ± 10.25 |
| AGA1 | 35.19 ± 60.14 | 0.38 ± 0.03 | 0.27 ± 0.11 | 0.27 ± 0.01 |
| PPT1 | 5.09 ± 1.98 | 3.06 ± 0.07 | 2.67 ± 0.04 | 3.98 ± 1.27 |
| HXT1 | 1.67 ± 0.66 | 1.82 ± 0.28 | 2.47 ± 0.49 | 5.52 ± 1.27 |
| ACS1 | 115.61 ± 5.3 | 90 ± 14.08 | 27.54 ± 6.91 | 19.67 ± 11.55 |
| TPT2 | 1.53 ± 0.53 | 6.45 ± 4.46 | 1.72 ± 0.68 | 1.27 ± 0.47 |
| CHLREDRAFT_122970 | 7.21 ± 0.8 | 2.51 ± 0.71 | 2.79 ± 0.93 | 2.33 ± 1.3 |
| ACLA1 | 5.28 ± 2.16 | 31.72 ± 12.94 | 3.87 ± 3.57 | 5.56 ± 3.8 |
Таблица 3.
Статистический анализ изменений уровня экспрессии генов, кодирующих ферменты центрального метаболизма и пластидные транспортеры, в процессе развития периодической миксотрофной культуры Chlamydomonas reinhardtii
| Ген | T2 | ANOVA значение p* | FDR** | Тест Тьюки | |||||
|---|---|---|---|---|---|---|---|---|---|
| 2-1*** | 4-1 | 6-1 | 4-2 | 6-2 | 6-4 | ||||
| ACLB1 | 7.1 | 0.0701 | 0.091 | 0.171 | 0.128 | 0.988 | 0.996 | 0.263 | 0.198 |
| OMT1 | 268.9 | 0.000259 | 0.0008 | 0.00099 | 0.782 | 0.984 | 0.0004 | 0.00069 | 0.931 |
| BCC1 | 60.4 | 0.0589 | 0.082 | 0.193 | 0.955 | 0.855 | 0.0959 | 0.0637 | 0.991 |
| HXK1 | 290.4 | 0.0000641 | 0.0007 | 0.00041 | 0.907 | 0.26 | 0.0002 | 7.10E-05 | 0.562 |
| AMYB1 | 9.8 | 0.0901 | 0.107 | 0.401 | 0.841 | 0.615 | 0.837 | 0.0753 | 0.239 |
| SBE3 | 3.4 | 0.188 | 0.195 | 0.94 | 0.84 | 0.164 | 0.993 | 0.343 | 0.465 |
| TRK1 | 110.1 | 6.03E-06 | 0.0002 | 8.90E-05 | 3.30E-05 | 5.00E-06 | 0.584 | 0.0126 | 0.0764 |
| ACS2 | 54 | 0.0061 | 0.0103 | 0.998 | 0.0241 | 0.0221 | 0.0309 | 0.0283 | 1 |
| ACK1 | 42.6 | 0.0326 | 0.0496 | 0.291 | 0.0216 | 0.205 | 0.306 | 0.993 | 0.421 |
| RBCS1 | 25.8 | 0.0711 | 0.091 | 0.408 | 0.976 | 0.164 | 0.246 | 0.891 | 0.093 |
| ACK2 | 246.2 | 0.00124 | 0.0028 | 0.00544 | 0.58 | 1 | 0.0012 | 0.00542 | 0.582 |
| CHLREDRAFT_123419 | 210.3 | 0.000198 | 0.0008 | 0.00471 | 0.109 | 0.147 | 0.0003 | 0.00034 | 0.996 |
| LCI20 | 10.1 | 0.0825 | 0.102 | 0.862 | 0.997 | 0.203 | 0.769 | 0.0685 | 0.261 |
| TPIC | 348.5 | 0.000248 | 0.0008 | 0.36 | 0.00572 | 0.00425 | 0.0008 | 0.00061 | 0.995 |
| APE2 | 78.6 | 0.000219 | 0.0008 | 0.00719 | 0.0722 | 0.117 | 0.0003 | 0.00041 | 0.985 |
| FBA3 | 11.1 | 0.0357 | 0.052 | 0.773 | 0.133 | 0.0369 | 0.457 | 0.143 | 0.801 |
| PCK1 | 189.5 | 0.000146 | 0.0008 | 0.486 | 0.00214 | 0.00251 | 0.0005 | 0.00051 | 0.999 |
| MEX1 | 54.5 | 0.00388 | 0.0069 | 0.00662 | 0.999 | 0.86 | 0.0056 | 0.0184 | 0.798 |
| TAL2 | 122.5 | 0.00161 | 0.0032 | 0.0173 | 0.00321 | 0.00188 | 0.571 | 0.342 | 0.965 |
| CHLREDRAFT_195672 | 18.2 | 0.126 | 0.139 | 0.1 | 0.881 | 0.438 | 0.551 | 0.729 | 0.948 |
| CHLREDRAFT_184945 | 33.9 | 0.00702 | 0.0112 | 0.0109 | 0.999 | 0.524 | 0.0091 | 0.0761 | 0.453 |
| STA11 | 198 | 0.000562 | 0.0016 | 0.0123 | 0.0854 | 0.479 | 0.0005 | 0.00196 | 0.576 |
| CHLREDRAFT_137300 | 109.4 | 0.0000467 | 0.0007 | 0.0303 | 7.20E-05 | 0.00014 | 0.0021 | 0.00554 | 0.85 |
| CHLREDRAFT_111372 | 217 | 0.000126 | 0.0008 | 0.00028 | 0.989 | 0.999 | 0.0002 | 0.00051 | 0.999 |
| CIS2 | 32.1 | 0.00119 | 0.0028 | 0.0387 | 0.00147 | 0.00191 | 0.0491 | 0.0734 | 0.989 |
| AGA1 | 12.5 | 0.506 | 0.506 | 0.577 | 0.651 | 0.574 | 1 | 1 | 1 |
| PPT1 | 4.3 | 0.132 | 0.141 | 0.229 | 0.13 | 0.672 | 0.975 | 0.775 | 0.548 |
| HXT1 | 43.6 | 0.000874 | 0.0023 | 0.995 | 0.601 | 0.00125 | 0.734 | 0.00163 | 0.0055 |
| ACS1 | 151.7 | 0.000191 | 0.0008 | 0.152 | 0.00088 | 0.00033 | 0.0034 | 0.001 | 0.863 |
| TPT2 | 59.9 | 0.101 | 0.115 | 0.148 | 1 | 0.999 | 0.232 | 0.125 | 0.997 |
| CHLREDRAFT_122970 | 19.6 | 0.00129 | 0.0028 | 0.00243 | 0.00652 | 0.00194 | 0.988 | 0.995 | 0.95 |
| ACLA1 | 164.8 | 0.00341 | 0.0064 | 0.00772 | 0.994 | 1 | 0.0057 | 0.00821 | 0.991 |
Анализ методом главных компонент
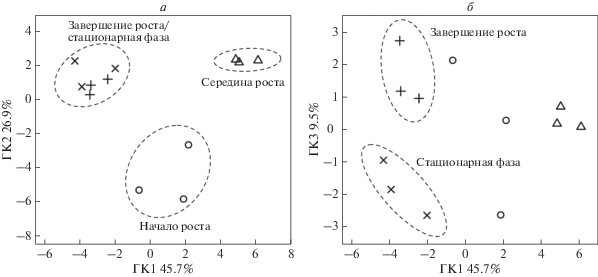
Для выявления различий между клетками хламидомонады на разных этапах развития культуры, их закономерностей и интерпретации использовали мультивариантный анализ. На первом этапе для установления сходства профилей транскрипции в разные временные точки использовали метод МГК. На рис. 3 представлены графики матрицы счетов первых трех ГК, которые объясняют 45.7, 26.9 и 9.5% дисперсии соответственно. Видно, что пробы группируются в зависимости от времени развития культуры (DBI = 0.29 для четырех кластеров, соответствующих времени, в пространстве трех ГК). Это свидетельствует о значительной связи развития культуры хламидомонады с динамикой экспрессии генов, кодирующих ферменты центрального метаболизма. Интересно, что различия сглаживались по мере приближения к стационарной фазе. Как видно из графика матрицы счетов, профили экспрессии растущих культур различаются между собой и отличаются от профилей в стационарной фазе в пространстве первых двух ГК (рис. 3а), а профили культур при завершении роста и в стационарной фазе различаются слабо лишь в пространстве ГК3 (рис. 3б).
Рис. 3.
Графики матрицы счетов, полученные в ходе анализа методом главных компонент профилей экспрессии генов, кодирующих ферменты первичного метаболизма и пластидные транспортеры, на разных временных этапах развития периодической миксотрофной культуры C. reinhardtii после посева. а – В пространстве ГК1 и ГК2; б – в пространстве ГК1 и ГК3.
Классификация методом ПЛС-ДА
Поскольку методом ГК показано, что пробы образуют три кластера (рис. 3а), которые соответствуют началу (1-е сутки), середине роста (2-е сутки), а также периоду, включающему завершение роста и стационарную фазу (4–6 суток), то различия между этими группами анализировали методом ПЛС-ДА. Полученная модель включала две ГК, объясняющие 46 и 27% дисперсии (R2Y = 0.94, Q2Y = 0.89). Гены, уровень экспрессии которых наиболее связан с различиями классов, отбирали по величинам параметра VIP, представленными на рис. 4 (ось абсцисс). Значения VIP у большинства проанализированных генов превышали 1. Это гены ACS1,2 и ACK2, кодирующие ферменты ассимиляции ацетата, CIS2 и ACLA1 – входа ацетильных групп в цикл Кребса, а также гены ферментов, вовлеченных в гликолиз и ПФП: TRK1, TAL2, PCK1, TPIC, HXK1, TPIC, CHLREDRAFT_111372, и гены таких ферментов метаболизма крахмала, как фосфорилаза крахмала (PHOB) – CHLREDRAFT_137300, α,α-трегалаза – CHLREDRAFT_195672, а также 4-α-глюкантрансфераза – STA11. Установлена выраженная связь гена CHLREDRAFT_122970 со сменой фаз роста. Этот ген кодирует биотинкарбоксилазу – домен ACC, включающей ацетильные группы в цепи жирных кислот. Кроме того, высокие значения VIP отмечены для генов трех пластидных транспортеров: OMT1, APE2, MEX1.
Рис. 4.
Рассеяние генов в пространстве значений VIP уровней экспрессии по результатам (О)ПЛС-ДА-моделирования динамики профиля экспрессии генов, кодирующих ферменты первичного метаболизма и пластидные транспортеры, в процессе развития миксотрофной культуры C. reinhardtii. По оси абсцисс – VIP ПЛС-ДА-модели с двумя ГК и тремя классами: начало роста (1-е сутки), середина роста (2-е сутки) и период, включающий завершение роста и стационарную фазу (4-е и 6-е сутки). По оси ординат – VIP предиктивной компоненты модели ОПЛС-ДА, включающей два класса: завершение роста (4-е сутки) и стационарная фаза (6-е сутки).
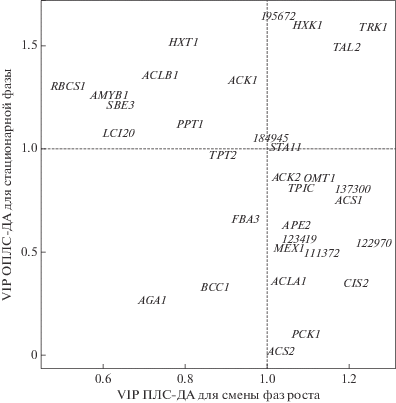
В предыдущей модели транскрипционные профили культур на 4-е и 6-е сутки развития были объединены в один класс, однако по данным, полученным методом МГК, между ними все-таки наблюдались слабые различия (рис. 3б), поэтому их рассматривали отдельно. Различия транскрипционных профилей культур, находящихся в конце фазы экспоненциального роста (4-е сутки) и в стационарной фазе (6-е сутки), анализировали методом ОПЛС-ДА. Предиктивная компонента описывала только 34% дисперсии. Таким образом, дисперсия, связанная со временем, оказалась сравнительно небольшой. На рис. 4 (ось ординат) представлены значения VIP предиктивной компоненты. Только около трети генов имели значения VIP более 1. Среди них три гена – HXT1, PPT1, LCI20, кодируют пластидные транспортеры. Связь со старением культуры характерна также для генов, кодирующих ферменты, вовлеченные в фиксацию углерода и метаболизм сахаров: RBCS1, HXK1, TRK1, TAL2; метаболизм крахмала – SBE3, AMYB1, STA11, и синтез жирных кислот (CHLREDRAFT_184945, кодирующий α-субъединицу карбоксилтрансферазы).
Cходство и различия в динамике транскрипционного профиля на протяжении роста культуры, перехода в стационарную фазу и в стационарной фазе анализировали, сравнивая значения VIP генов первой и второй моделей ПЛС-ДА. Как видно из рис. 4, на котором представлено рассеяние генов в пространстве их VIP, в описанных двух моделях не наблюдается никакой зависимости, что означает практически полное отсутствие связи между динамикой уровня транскрипции генов в стационарной фазе и в ходе роста культуры.
Мультивариантный эмпирический Байесовский анализ (МЕБА) динамики уровня экспрессии генов в процессе развития культуры
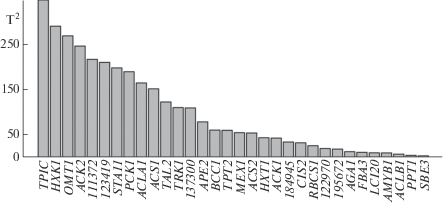
Особенностью МЕБА, одного из эффективных методов анализа динамики большого количества признаков во времени, является учет ковариации переменных у нескольких исследуемых объектов (в нашем случае это три независимые культуры хламидомонады). Это позволяет нивелировать различия между культурами и точнее определить влияние времени на анализируемые переменные. Связь признака со временем оценивали по величине T2 статистики Хотеллинга [45, 46]. На рис. 5 представлена диаграмма значений T2. Можно видеть, что наибольшие значения Т2 показывают гены TPIC, CHLREDRAFT_111372, HXK1, OMT1, ACK2, CHLREDRAFT_123419, STA11, ACLA1 и ACS1.
Динамика экспрессии генов
Для определения закономерностей динамики интенсивности экспрессии генов и выявления корегулируемых групп генов проведен корреляционный анализ с последующей иерархической кластеризацией с использованием в качестве дистанции коэффициента Пирсона. Для определения статистической значимости выделения кластеров в качестве критерия применена величина AU p, вычисляемая на основе многомасштабной перевыборки бутстрапом (multiscale bootstrap resampling) [41]. На основе анализа графика локтя (рис. 6а) и оценки статистической значимости кластеров (рис. 6б) выделены шесть кластеров с характерными паттернами динамики экспрессии генов интереса (рис. 6б, в).
Рис. 6.
Иерархическая кластеризация методом Варда генов ферментов первичного метаболизма и пластидных транспортеров C. reinhardtii. В качестве дистанций использовали коэффициент корреляции средних значений их экспрессии, измеренных в процессе развития миксотрофной культуры. a – График локтя, иллюстрирующий зависимость дистанций между объединяемыми кластерами и числом агломераций (и числом кластеров), пунктирные линии соответствуют выбранному числу кластеров и обрезке высоты дерева. б – Дендрограмма с выделеными статистически значимыми кластерами (AU p < 0.05). в – Графики стандартизированных значений (SD = 1, M = 0) уровней экспрессии генов (абсцисса), объединенных по паттернам динамики, ординаты – сутки после посева.
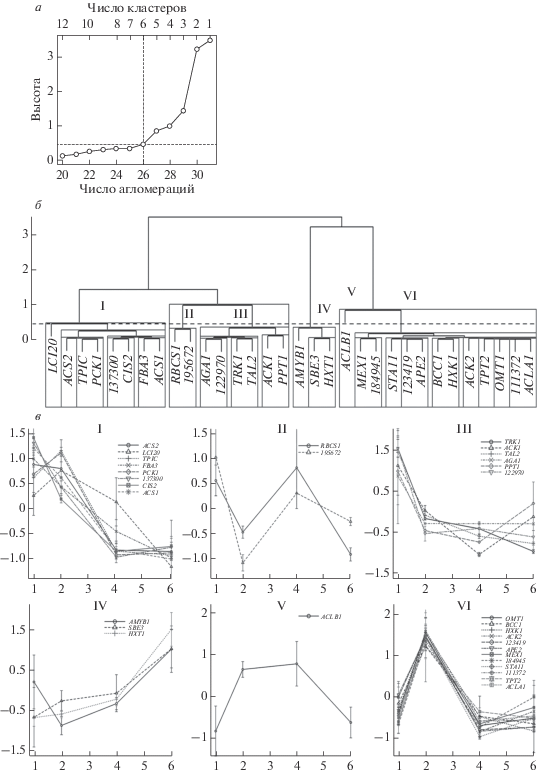
Гены группы I и III отличало снижение уровня экспрессии в процессе старения культуры. Первый паттерн отличался тем, что в период экспоненциального роста накопление продукта транскрипции оставалось на относительно высоком уровне, хотя могло умеренно снижаться. При завершении роста экспрессия генов данной группы резко падала и стабилизировалась. Такая динамика накопления транскриптов в процессе роста культуры присуща генам: PCK1, TPIC, ACS1,2, CIS2, FBA3, CHLREDRAFT_137300.
Для генов третьего кластера характерно резкое снижение экспрессии уже в середине экспоненциального роста, после чего уровень экспрессии оставался относительно стабильным (TAL2, CHLREDRAFT_122970), продолжал снижаться (TRK1) или наблюдался умеренный рост (ACK1).
Таким образом, тенденция к снижению экспрессии со временем наблюдалась у генов, кодирующих ферменты ассимиляции ацетата: ацетил-КоА-синтазу и пластидную ацетаткиназу ACK1. Экспрессия генов, кодирующих ферменты ПФП и цикла Кальвина – трансальдолазы, транскетолазы, также снижается с возрастом культуры. Эта же тенденция показана для генов фосфоенолпируваткарбоксикиназы, триозофосфатизомеразы и фруктозо-1,6-бисфосфатальдолазы, вовлеченных в гликолиз и глюконеогенез. Сходную картину можно наблюдать и для генов некоторых ферментов, участвующих в поступлении ацетильных групп в цикл Кребса, таких как цитратсинтаза. Таким образом, в процессе роста культуры происходит снижение активности генов, кодирующих ферменты основных энергетических и пластических путей клетки. При этом быстро снижается активность гена фосфорилазы крахмала.
Вторая группа близка к группе III и образует с ней большой кластер (рис. 6б). Группа II включает гены RBCS1 и CHLREDRAFT_195672. Динамика экспрессии этих генов не коррелирует с увеличением возраста культуры (рис. 6в). Кроме того, по данным дисперсионного анализа эти изменения не имеют статистической значимости, и согласно МЕБА имеют относительно низкие значения T2.
Устойчивый рост экспрессии с увеличением возраста культуры зарегистрирован лишь у небольшого числа генов (кластер IV) – это HXT1, AMYB1 и SBE3. Все три гена связаны с метаболизмом гексоз, тем не менее, если α-амилаза является одним из ключевых ферментов деградации крахмала, то SBE3, напротив, отвечает за его синтез. Лишь в случае HXT1 значимость изменения экспрессии подтверждается дисперсионным анализом (p = 0.0009). В случае гена AMYB1 можно обсуждать лишь наличие тенденции (p = 0.09).
Группы V и VI были близки и образовывали вместе большой кластер. Группа V включает лишь один ген – ACLB1, экспрессия которого возрастает на вторые сутки роста, поддерживается на этом уровне продолжительное время и снижается в значительно более поздней стационарной фазе. В этом случае статистическая значимость изменений относительно невысока (p = 0.07). Большую часть исследованных генов (OMT1, MEX1, CHLREDRAFT_184945, STA11, CHLREDRAFT_123419, APE2, HXK1, ACK2, CHLREDRAFT_111372, ACLA1, BCC1, TPT2) можно отнести к шестому паттерну – росту экспрессии в первой половине экспоненциального роста с последующим снижением при переходе в стационарную фазу. По данным дисперсионного анализа изменение экспрессии двух последних генов (BCC1 и TPT2) статистически слабо значимо (0.05 < p ≤ 0.1).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Хламидомонада обладает способностью усваивать экзогенный ацетат как дополнительный источник углерода при миксотрофном типе питания. За последние 2 года опубликованы результаты двух масштабных транскриптомных исследований, нацеленных на изучение изменений, происходящих в клетках хламидомонады при миксотрофном выращивании [47, 48]. В базе данных KEGG представлено 113 биологических процессов, в которые вовлечено 3145 генов. В этих статьях рассмотрены топ-10 путей и топ-100 генов, изменение которых соответствовало изменению характера роста культуры хламидомонады (на твердой или жидкой среде, на свету или в темноте, в присутствии ацетата в различной концентрации). Представлены данные о различиях в экспрессии генов в ходе экспоненциальной и стационарной фазы роста культуры. При выращивании хламидомонады при минимальном содержании ацетата и в жидкой среде (что максимально соответствует условиям нашего эксперимента) происходит накопление транскриптов генов белков фотосинтеза и деления на этапе экспоненциального роста, а в последующей стационарной фазе – белков катаболизма и деградации аминокислот, связанных со стрессом, шаперонов и др. Представленные данные, тем не менее, не ответили на один из важнейших вопросов – за счет каких метаболических путей, при участии каких ферментов происходит включение ацетата в метаболизм? Каким образом меняется экспрессия генов, кодирующих эти ферменты, особенно в фазу экспоненциального роста, когда метаболизация представляется наиболее интенсивной?
Наше исследование ставило своей целью выявить различия в экспрессии генов, кодирующих ряд ключевых ферментов метаболизма и транспорта метаболитов в ходе развития периодической культуры хламидомонады в миксотрофных условиях (табл. 1‒3, рис. 1). По данным базы KEGG лишь около 250 генов хламидомонады кодируют ферменты девяти основных биохимических путей центрального углеродного метаболизма. На основании проведенного нами метаболомного анализа [21] и ранее опубликованных данных [21] можно ожидать, что присутствие ацетата затрагивает, в первую очередь, пути его ассимиляции, цикл Кребса, синтез жирных кислот и фиксацию углерода. Поэтому были выбраны ключевые ферменты этих путей и найдены гены, их кодирующие. Нами проанализирована экспрессия 32 генов, список которых приведен в табл. 1, с указанием их продуктов, а также праймеров к ним. Снижение размерности с помощью МГК (рис. 3) выявила различия в профилях экспрессии генов в клетках хламидомонады на разных стадиях развития культуры, характеризующихся неодинаковым уровнем пролиферации и интенсивности основных физиологических процессов. В пространстве первых двух ГК, описывающих около 70% дисперсии, профили экспрессии формировали три группы, соответствующие началу экспоненциальной фазы, ее середине, а также группу, объединяющую завершение экспоненциального роста и стационарную фазу (рис. 3а). Различия этих трех периодов связаны прежде всего с ГК1 (46%), что может указывать на существенное сходство процессов, протекающих в течение всего развития культуры. Следует отметить своеобразие профиля экспрессии в начале экспоненциальной фазы, отражающее, по-видимому, изменение состояния клеток при выходе из лаг-фазы и переход к пролиферации.
Подчеркнем, что степень вариабельности профилей экспрессии снижается со временем. Об этом свидетельствуют результаты, полученные с помощью как МГК (рис. 3), так и теста Тьюки, показавшие, что уровни экспрессии большого числа генов статистически значимо меняются именно в фазе роста. Сходные тенденции наблюдали и ранее при анализе динамики профиля метаболитов в процессе развития миксотрофной культуры хламидомонады [21]. Таким образом, молекулярно-генетический статус клеток наиболее динамично меняется именно в ходе экспоненциального роста и стабилизируется после его завершения. Это может быть связано как с изменением экзогенных условий, параметры которых (например, состав среды) стабилизируются со временем, так и с физиологическим состоянием самих клеток, например, в результате остановки пролиферации. Анализ методами МЕБА и ПЛС-ДА показал, что с развитием культуры связаны, прежде всего, гены ферментов энергетического метаболизма: ассимиляции ацетата, цикла Кребса, гликолиза и ПФП (рис. 4, 5). Со старением культуры ассоциированы гены, кодирующие пластидные транспортеры и ферменты метаболизма крахмала и углеводов (рис. 4).
Нами выявлены шесть групп генов, динамика накопления транскриптов которых различается в ходе развития культуры (рис. 6, 7). Дифференциальная активность генов должна быть связана с их ролью в обеспечении клетки энергией и метаболитами на разных этапах роста культуры (рис. 1, 2). Показательно, что характер экспрессии генов кластера I сходен с паттерном изменения дыхания и фотосинтеза [21]. При этом снижение уровня дыхания и фотосинтеза во второй половине фазы роста соответствует снижению экспрессии генов кластеров III и VI. Таким образом, регуляция изменения активности физиологических процессов связана с регуляцией экспрессии генов этих кластеров.
Рис. 7.
Схема потоков углерода в миксотрофных клетках C. reinhardtii. Красные столбики – относительный уровень экспрессии генов в начале и середине экспоненциальной фазы, при переходе в стационарную фазу и в стационарной фазе. Цвет соответствует паттерну динамики уровня экспрессии: I – розовый, II – серый, III – синий, IV ‒ желтый, V и VI ‒ зеленый. ПФП – пентозофосфатный путь, ФЕП – фосфоенолпируват, Г-3Ф – глицеральдегид-3-фосфат, ФГК – 3-фосфоглицериновая кислота.
Важнейшим процессом, определяющим состояние клеток в миксотрофной культуре хламидомонады, является усвоение ацетата. Хламидомонада обладает пластидными и непластидными формами ферментов метаболизма ацетата (рис. 7). Возможно, цель такого разделения состоит в перераспределении ацетильных групп между процессами в различных компартментах клетки. К пластидному сегменту метаболизма, принимающему ацетильные группы, относится, прежде всего, синтез липидов, тогда как к цитозольному и митохондриальному ‒ цикл Кребса и его модификации. При этом гены, кодирующие как хлоропластную, так и непластидную ацетил-КоА-синтазу (рис. 6, 7), имеют одинаковый характер экспрессии (паттерн I), что свидетельствует о сходной временной регуляции поступления углерода в оба сегмента метаболизма. Показан высокий уровень экспрессии генов, кодирующих ACK, на первые и вторые сутки, когда скорость роста культуры, активность дыхания и фотосинтеза максимальны [21]. Известно, что хламидомонада быстрее поглощает ацетат на этапе интенсивного роста, чем при остановке пролиферации [25, 49]. С другой стороны, присутствие ацетата в среде культивирования приводило к повышению экспрессии гена ACS2 [50]. При переходе к стационарной фазе снижается уровень экспрессии генов, кодирующих ферменты ассимиляции ацетата, что, видимо, связано с исчерпанием ацетата в среде, поскольку известно, что хламидомонада потребляет почти весь ацетат уже в экспоненциальной фазе роста [18, 51, 52].
Изменения экспрессии генов CIS2 и PCK1 сходны с изменениями гена ACS1. Ассимиляция углерода ацетата связана с оттоком углерода из цикла Кребса (глиоксилатного пути), который может катализироваться фосфоенолпируваткарбоксикиназой. Известно, что активность изоцитратлиазы, ключевого фермента глиоксилатного цикла, максимальная в молодых миксотрофных культурах C. mundana, снижается на порядок при старении. Это происходит на фоне исчерпания ацетата и падения уровня дыхания [53]. О важной роли фосфоенолпируваткарбоксикиназы и цитратсинтазы в усвоении ацетата свидетельствует также повышение экспрессии генов этих ферментов в ответ на добавление ацетата в стареющие культуры C. reinhardtii [25]. Углерод в форме фосфоенолпирувата (ФЕП) перенаправляется в пластиду и включается в глюконеогенез, что согласуется с высоким уровнем экспрессии гена PPT1, кодирующего пластидный переносчик ФЕП, а также генов FBA3 и TPIC, белковые продукты которых вовлечены в глюконеогенез в начале и середине роста культуры. С другой стороны, соотношение активности фосфоенолпируваткарбоксикиназы и АСС, конкурирующих за углерод, может определять перераспределение углерода между синтезом липидов и глиоксилатным циклом [54, 55]. Поэтому высокий уровень экспрессии PCK1 на фоне относительно низкого уровня экспрессии генов двух субъединиц ACC в первые сутки позволяет предположить, что в начальный период роста культуры значение цитозольной части метаболизма, где ацетат через глиоксилатный цикл и с участием фосфоенолпируваткарбоксикиназы превращается в ФЕП, сравнительно велико, поскольку велика потребность в “строительном материале”.
В пластиде, напротив, на вторые сутки роста максимальна активность генов, связанных с синтезом липидов, в том числе генов субъединицы ацетил-КоА-карбоксилазы (BCC1, CHLREDRAFT_184945), а также KASIII (CHLREDRAFT_123419). Ацетил-КоА можно считать “точкой распределения” углерода. Ацетил-КоА-карбоксилаза направляет углерод на биосинтез жирных кислот и липидов [28]. В то же время, регуляция синтеза липидов не определяется исключительно активностью ферментов, синтезирующих углеродные цепи жирных кислот [15, 56–58]. Количество ацетильных групп, которые могут быть использованы в синтезе липидов, определяется, в том числе, уровнем их включения в цикл Кребса и выходом из него. Интересно, что в середине экспоненциальной фазы повышается уровень экспрессии генов, кодирующих субъединицы изоцитратлиазы, особенно ACLA1 (рис. 6). Этот фермент способен катализировать регенерацию ацетил-КоА, который может использоваться в синтезе жирных кислот [59]. Известно, что уровень ACL1 возрастает после перехода к синтезу липидов при азотном голодании [60]. Рост активности генов ферментов, направляющих ацетильные группы на синтез жирных кислот, согласуется со снижением экспрессии CIS2, “направляющего” их в цикл Кребса. Известно, что подавление экспрессии гена цитратсинтазы приводит к росту уровня ТАГ и его снижению при сверхэкспрессии этого гена [26]. Все это может отражать частичное перераспределение ацетильных групп из цикла Кребса/глиоксилатного цикла в пластидный путь синтеза жирнокислотных цепей.
В этот же период возрастает активность гена гексокиназы (HXK1), что может указывать на усиление гликолиза. О высокой активности гликолиза в этот период свидетельствует также сохранение высокого уровня экспрессии генов FBA3 и TPIC на вторые сутки роста на фоне снижения активности генов ПФП ‒ TRK и TAL2. Высокий уровень активности гликолиза можно связать с увеличением потребности в трехуглеродных молекулах для синтеза ТАГ.
Усиление гликолиза может привести к повышению синтеза АТР и восстановленных кофакторов в пластиде. А увеличенное использование ацетильных групп в синтезе липидов в середине экспоненциального роста – к снижению интенсивности окисления ацетильных групп в митохондриях. В результате будет наблюдаться переизбыток восстановителей и энергии в пластиде. Поддержание энергетического баланса в клетках хламидомонады осуществляется с помощью триозофосфатного шунта [27]. Поэтому не удивительно, что в этот же период достигает максимума активность генов, кодирующих белки-переносчики, что свидетельствует о восстановлении энергетического баланса между пластидой и цитозолем (рис. 6, 7). Увеличивается также накопление транскриптов гена OMT1, кодирующего оксоглутаратный/малатный антипортер, обеспечивающий выход восстановителей из пластиды.
Динамика экспрессии генов кластеров VI и V характеризуется первичным накоплением, а затем снижением содержания транскриптов, что согласуется с обнаруженным нами ранее изменением уровня многих метаболитов, увеличение содержания которых регистрировали в начале роста культуры, максимум – в середине, а затем, во второй половине фазы роста, наблюдали снижение [21]. К числу этих метаболитов можно отнести жирные кислоты, преимущественно с относительно короткими цепями. Такая же динамика была характерна и для интемедиатов энергетических и синтетических путей, в том числе гликолиза, циклов Кребса и Кальвина [21].
Уровень экспрессии почти всех исследованных генов снижается при завершении роста культуры. Сильное снижение уровня транскриптов большей части генов, кодирующих ферменты первичного метаболизма, в том числе связанные с дыханием и ассимиляцией углерода (рис. 6в, 7), к третьим суткам культивирования совпадает с резким падением уровня дыхания и фотосинтеза [21]. Можно предположить, что изменения экспрессии генов, связанных с циклами Кальвина и гликолизом, обусловлены постепенным исчерпанием ацетата и сменой трофических условий. Протеомные исследования указывают на то, что в условиях миксотрофии значительно возрастает содержание ряда ферментов цикла Кальвина, в том числе TPI, FBA3, TRK [61].
Несмотря на снижение экспрессии генов субъединиц ACC и KAS, в стационарной фазе возрастает содержание ряда свободных жирных кислот и ацилглицеринов [21]. Это подтверждает гипотезу, согласно которой в условиях стресса и в стареющих культурах пулы липофильных соединений пополняются в результате деструктивных процессов, связанных с аутофагией и деструкцией мембран [62]. Это согласуется с тем, что регуляция синтеза липидов не всегда определяется исключительно активностью ферментов, синтезирующих углеродные цепи жирных кислот, а активность ACC [56] или изменения экспрессии гена, его кодирующего, не соответствуют изменению накопления липидов [15, 57]. В ряде случаев повышенная экспрессия гена, кодирующего KASIII, также не приводит к росту синтеза жирных кислот в тканях растений [58]. Можно предположить, что участие перечисленных ферментов в регуляции синтеза жирных кислот более выражено при доступности большого числа ацетильных групп, при снижении доли таких групп снижается и роль этих ферментов в метаболизме углерода в клетках хламидомонады.
С возрастом культуры наблюдается заметный рост экспрессии гена HXT1, кодирующего белок экспорта гексоз из пластид в цитоплазму, что может отражать усиление роли потока углерода из пластид в условиях исчерпания ацетата. Источником этого углерода может быть не только непосредственно фотосинтез, но и деградация крахмала, на что указывает незначительный рост экспрессии гена AMYB1, кодирующего β-амилазу, поскольку баланс синтеза и деградации крахмала в клетках хламидомонады может зависеть от активности амилазы [58].
ЗАКЛЮЧЕНИЕ
Интенсивность экспрессии большинства проанализированных нами генов изменяется в процессе развития культуры хламидомонады. Завершение экспоненциальной фазы роста сопровождается снижением накопления транскриптов и интенсивности метаболизма. Наибольшие изменения профиля транскрипции выявлены в период, близкий к середине экспоненциального роста, когда происходит радикальная перестройка метаболизма, связанная с запуском синтеза липидов, что сопровождается изменением потоков углерода по биохимическим путям и смещением баланса энергии между пластидой и цитозолем.
Работа выполнена при частичной поддержке Российского фонда фундаментальных исследований (гранты № 15-04-03090 и 16-34-01122) и Санкт-Петербургского государственного университета (НИР 1.40.495.2017) с использованием оборудования Ресурсного центра СПбГУ “Развитие молекулярных и клеточных технологий”.
Настоящая статья не содержит исследований с использованием людей или животных.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Perez-Garcia O., Bashan Y. (2015) Microalgal heterotrophic and mixotrophic culturing for bio-refining: from metabolic routes to techno-economics. In: Algal Biorefineries. V. 2: Products and Refinery Design. Eds. Prokop A., Bajpai R.K., Zappi M.E., Springer, pp. 61‒131.
Fouchard S., Hemschemeier A., Caruana A., Pruvost J., Legrand J., Happe T., Peltier G., Cournac L. (2005) Autotrophic and mixotrophic hydrogen photoproduction in sulfur-deprived chlamydomonas cells. Appl. Environm. Microbiol. 71, 6199–6205.
Sager R., Granick S. (1953) Nutritional studies with Chlamydomanas reinhardtii. Ann. NY Acad. Sci. 56, 831‒838.
Rochaix J.D. (2002) Chlamydomonas, a model system for studying the assembly and dynamics of photosynthetic complexes. FEBS Lett. 529, 34‒38.
Grossman A., Lohr M., Im C. (2004) Chlamydomonas reinhardtii in the Landscape of Pigments. Annu. Rev. Genet. 38, 119‒173.
Grossman A. (2000) Acclimation of Chlamydomonas reinhardtii to its nutrient environment. Protist. 151, 201‒224.
Bolling C., Fiehn O. (2005) Metabolite profiling of Chlamydomonas reinhardtii under nutrient deprivation. Plant Physiol. 139, 1995‒2005.
Glaesener A., Merchant S., Blaby-Haas C. (2013) Iron economy in Chlamydomonas reinhardtii. Front. Plant Sci. 4, article 337.
Jamers A., Blust R., De Coen W., Griffin J., Jones O. (2013) An omics based assessment of cadmium toxicity in the green alga Chlamydomonas reinhardtii. Aquatic Toxicol. 126, 355‒364.
Schmollinger S., Schulz-Raffelt M., Strenkert D., Veyel D., Vallon O., Schroda M. (2013) Dissecting the heat stress response in Chlamydomonas by pharmaceutical and RNAi approaches reveals conserved and novel aspects. Mol. Plant. 6, 1795‒1813.
Valledor L., Furuhashi T., Hanak A., Weckwerth W. (2013) Systemic cold stress adaptation of Chlamydomonas reinhardtii. Mol. Cell. Proteomics. 12, 2032‒2047.
Moellering E., Benning C. (2009) RNA interference silencing of a major lipid droplet protein affects lipid droplet size in Chlamydomonas reinhardtii. Eukaryotic Cell. 9, 97‒106.
Lee D.Y., Park J.J., Barupa D.K., Fiehn O. (2012) System response of metabolic networks in Chlamydomonas reinhardtii to total available ammonium. Mol. Cell. Proteomics. 11, 973‒988.
Mettler T., Mühlhaus T., Hemme D., Schöttler MA., Rupprecht J., Idoine A., Veyel D., Pal SK., Yaneva-Roder L., Winck F.V., Sommer F., Vosloh D., Seiwert B., Erban A., Burgos A., Arvidsson S., Schönfelder S., Arnold A., Günther M., Krause U., Lohse M., Kopka J., Nikoloski Z., Mueller-Roeber B., Willmitzer L., Bock R., Schroda M., Stitt M. (2014) Systems analysis of the response of photosynthesis, metabolism, and growth to an increase in irradiance in the photosynthetic model organism Chlamydomonas reinhardtii. Plant Cell. 26, 2310‒2350.
Lv H., Qu G., Qi X., Lu L., Tian C., Ma Y. (2013) Transcriptome analysis of Chlamydomonas reinhardtii during the process of lipid accumulation. Genomics. 101, 229‒237.
Lee D.Y., Fiehn O. (2013) Metabolomic response of Chlamydomonas reinhardtii to the inhibition of target of rapamycin (TOR) by rapamycin. J. Microbiol. Biotechnol. 23, 923‒931.
Velmurugan N., Sung M., Yim S.S., Park M.S., Yang J.W., Jeong K.J. (2013) Evaluation of intracellular lipid bodies in Chlamydomonas reinhardtii strains by flow cytometry. Bioresour. Technol. 138, 30‒37.
Singh H., Shukla M.R., Chary K.V., Rao B.J. (2014) Acetate and bicarbonate assimilation and metabolite formation in Chlamydomonas reinhardtii: a 13C-NMR study. PLoS One. 9, e106457.
Humby P., Snyder E., Durnford D. (2013) Conditional senescence in Chlamydomonas reinhardtii (Chlorophyceae) J. Phycol. 49, 389‒400.
Lee D.Y., Fiehn O. (2008) High quality metabolomic data for Chlamydomonas reinhardtii. Plant Methods. 4, 7.
Puzanskiy R., Tarakhovskaya E., Shavarda A., Shishova M. (2018) Metabolomic and physiological changes of Chlamydomonas reinhardtii (Chlorophyceae, Chlorophyta) during batch culture development. J. Appl. Phycol. 30, 803‒818.
Terashima M., Specht M., Naumann B., Hippler M. (2010) Characterizing the anaerobic response of Chlamydomonas reinhardtii by quantitative proteomics. Mol. Cell. Proteomics. 9, 1514‒1532.
Atteia A., van Lis R., Tielens A.G., Martin W.F. (2013) Anaerobic energy metabolism in unicellular photosynthetic eukaryotes. Biochim. Biophys. Acta. 1827, 210‒223.
Yang W., Catalanotti C., D’Adamo S., Wittkopp T., Ingram-Smith C., Mackinder L. Miller T.E., Heuberger A.L., Peers G., Smith K.S., Jonikas M.C., Grossman A.R., Posewitz M.C. (2014) Alternative acetate production pathways in Chlamydomonas reinhardtii during dark anoxia and the dominant role of chloroplasts in fermentative acetate production. Plant Cell Online. 26, 4499‒4518.
Goodenough U., Blaby I., Casero D., Gallaher S., Goodson C., Johnson S., Lee J., Merchant, S.S., Pellegrini M., Roth R., Rusch J., Singh M., Umen J.G., Weiss T.L. Wulan T. (2014) The path to triacylglyceride obesity in the sta6 strain of Chlamydomonas reinhardtii. Eukaryotic Cell. 13, 591‒613.
Deng X., Cai J., Fei X. (2013) Effect of the expression and knockdown of citrate synthase gene on carbon flux during triacylglycerol biosynthesis by green algae Chlamydomonas reinhardtii. BMC Biochem. 14, 38.
Johnson X., Alric J. (2013) Central carbon metabolism and electron transport in Chlamydomonas reinhardtii: metabolic constraints for carbon partitioning between oil and starch. Eukaryotic Cell. 12, 776‒793.
Cronan J., Waldrop G. (2002) Multi-subunit acetyl-CoA carboxylases. Progress Lipid Res. 41, 407‒435.
Heldt H., Rapley L. (1970) Specific transport of inorganic phosphate, 3-phosphoglycerate and dihydroxyacetonephosphate, and of dicarboxylates across the inner membrane of spinach chloroplasts. FEBS Lett. 10, 143‒148.
Atteia A., Adrait A., Brugière S., Tardif M., van Lis R., Deusch O., Dagan T., Kuhn L., Gontero B., Martin W., Garin J., Joyard J., Rolland N. (2009) A proteomic survey of Chlamydomonas reinhardtii mitochondria sheds new light on the metabolic plasticity of the organelle and on the nature of the α-proteobacterial mitochondrial ancestor. Mol. Biol. Evol. 26, 1533–1548.
Weber A., Menzlaff E., Arbinger B., Gutensohn M., Eckerskorn C., Fluegge U. (1995) The 2-oxoglutarate/malate translocator of chloroplast envelope membranes: molecular cloning of a transporter containing a 12-helix motif and expression of the functional protein in yeast cells. Biochemistry. 34, 2621‒2627.
Terashima M., Specht M., Hipple M. (2011) The chloroplast proteome: a survey from the Chlamydomonas reinhardtii perspective with a focus on distinctive features. Curr. Genet. 57, 151‒168.
Gorman D., Levine R. (1965) Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proc. Natl. Acad. Sci. USA. 54, 1665‒1669.
Chomczynski P. (1987) Single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction. Analyt. Biochem. 162, 156‒159.
Pootakham W., Gonzalez-Ballester D., Grossman A. (2010) Identification and regulation of plasma membrane sulfate transporters in Chlamydomonas. Plant Physiol. 153, 1653‒1668.
Maikova A., Zalutskaya Z., Lapina T., Ermilova E. (2016) The HSP70 chaperone machines of Chlamydomonas are induced by cold stress. J. Plant Physiol. 204, 85‒91.
Schloss J. (1990) A Chlamydomonas gene encodes a G protein β-subunit-like polypeptide. Mol. Gen. Genet. 221, 443‒452.
Liu C., Wu G., Huang X., Liu S., Cong B. (2012) Validation of housekeeping genes for gene expression studies in an ice alga Chlamydomonas during freezing acclimation. Extremophiles. 16, 419‒425.
Zhang H., Wang W., Li Y., Yang W., Shen G. (2011) Mixotrophic cultivation of Botryococcus braunii. Biomass. Bioenergy. 35, 1710‒1715.
R Core Team (2016) R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. URL https://www.R-project.org/
Murtagh F., Legendre P. (2014) Ward’s hierarchical agglomerative clustering method: which algorithms implement Ward’s criterion? J. Classification. 31, 274‒295.
Stacklies W., Redestig H., Scholz M., Walther D., Selbig J. (2007) pcaMethods – a bioconductor package providing PCA methods for incomplete data. Bioinformatics. 23, 1164‒1167.
Thevenot E.A., Roux A., Xu Y., Ezan E., Junot C. (2015) Analysis of the human adult urinary metabolome variations with age, body mass index and gender by implementing a comprehensive workflow for univariate and OPLS statistical analyses. J. Proteome Res. 14, 3322‒3335.
Tai Y.C., Speed T.P. (2006) A multivariate empirical Bayes statistic for replicated microarray time course data. Ann. Statist. 34, 2387‒2412.
Xia J., Sinelnikov I., Han B., Wishart D.S. (2015) MetaboAnalyst 3.0 ‒ making metabolomics more meaningful. Nucl. Acids Res. 43(W1), W251‒257.
Xia J., Wishart D.S. (2016) Using MetaboAnalyst 3.0 for comprehensive metabolomics data analysis. Curr. Protocols Bioinformat. 55, 14.10.1‒14.10.91.
Bogaert K.A., Manoharan-Basil S.S., Perez E., Levi-ne R.D., Remacle F., Remacle C. (2018) Surprisal analysis of genome-wide transcript profiling identifies differentially expressed genes and pathways associated with four growth conditions in the microalga Chla-mydomonas. PLoS One. 13, e0195142.
Bogaert K.A., Perez E., Rumin J., Giltay A., Carone M., Coosemans N., Radoux M., Eppe G., Levine R.D., Remacle F., Remacle C. (2019) Metabolic, physiological, and transcriptomics analysis of batch cultures of the green microalga Chlamydomonas grown on different acetate concentrations. Cells. 8, 1367.
Work V., Radakovits R., Jinkerson R., Meuser J., Elliott L., Vinyard D., Laurens L., Dismukes C., Posewitz M. (2010) Increased lipid accumulation in the Chlamydomonas reinhardtii sta7-10 starchless isoamylase mutant and increased carbohydrate synthesis in complemented strains. Eukaryot. Cell. 9, 1251‒1261.
Ramanan R., Kim B., Cho D., Ko S., Oh H., Kim H. (2013) Lipid droplet synthesis is limited by acetate availability in starchless mutant of Chlamydomonas reinhardtii. FEBS Lett. 587, 370‒377.
Lalibertè G., de la Noüie J. (1993) Auto-, hetero-, and mixotrophic growth of Chlamydomonas humicola (Cmloroimiyckak) on acetate. J. Phycol. 29, 612‒620.
Therien J.B., Zadvornyy O.A., Posewitz M.C., Bryant D.A., Peters J.W. (2014) Growth of Chlamydomonas reinhardtii in acetate-free medium when co-cultured with alginate-encapsulated, acetate-producing strains of Synechococcus sp. PCC 7002. Biotechnol. Biofuels. 7, 154.
Eppley R., Gee R., Saltman P. (1963) Photometabolism of acetate by Chlamydomonas mundana. Physiologia Plantarum. 16, 777‒792.
Sugimoto T., Tanaka K., Monma M., Kawamura Y., Saio K. (1989) Phosphoenolpyruvate carboxylase level in soybean seed highly correlates to its contents of protein and lipid. Agricult. Biol. Chem. 53, 885‒887.
Chen J.Q., Lang C.X., Hu Z.H., Liu Z.H., Huang R.Z. (1999) Antisense PEP gene regulates to ratio of protein and lipid content in Brassica napus seeds. J. Agricult. Biotechnol. 7, 316‒320.
Leyva L., Bashan Y., Mendoza A., de-Bashan L. (2014) Accumulation fatty acids of in Chlorella vulgaris under heterotrophic conditions in relation to activity of acetyl-CoA carboxylase, temperature, and co-immobilization with Azospirillum brasilense. Naturwissenschaften. 101, 819‒830.
Radakovits R., Jinkerson R., Darzins A., Posewitz M. (2010) Genetic engineering of algae for enhanced biofuel production. Eukaryotic Cell. 9, 486‒501.
Mus F., Dubini A., Seibert M., Posewitz M., Grossman A. (2007) Anaerobic acclimation in Chlamydomonas reinhardtii: anoxic gene expression, hydrogenase induction, and metabolic pathways. J. Biol. Chem. 282, 25475‒25486.
Plancke C., Vigeolas H., Höhner R., Roberty S., Emonds-Alt B., Larosa V., Willamme R., Duby F., Onga D.D, Thonart P., Hiligsmann S., Franck F., Eppe G., Cardol P., Hippler M., Remacle C. (2014) Lack of isocitrate lyase in Chlamydomonas leads to changes in carbon metabolism and in the response to oxidative stress under mixotrophic growth. Plant J. 77, 404‒417.
Park J., Wang H., Gargouri M., Deshpande R., Skepper J., Holguin F., Juergens M.T., Shachar-Hill Y., Hicks L.M., Gang D.R. (2015) The response of Chlamydomonas reinhardtii to nitrogen deprivation: a systems biology analysis. Plant J. 81, 611‒624.
Wienkoop S., Weiß J., May P., Kempa S., Irgang S., Recuenco-Munoz L., Pietzke M., Schwemmer T., Rupprecht J., Egelhofer V., Weckwerth W. (2010) Targeted proteomics for Chlamydomonas reinhardtii combined with rapid subcellular protein fractionation, metabolomics and metabolic flux analyses. Mol. Biosystems. 6, 1018‒1031.
Davey M., Horst I., Duong G., Tomsett E., Litvinenko A., Howe C., Smith A. (2014) Triacylglyceride production and autophagous responses in Chlamydomonas reinhardtii depend on resource allocation and carbon source. Eukaryot. Cell. 13, 392‒400.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология