Молекулярная биология, 2020, T. 54, № 5, стр. 776-801
Длинные некодирующие РНК как конкурентные эндогенные РНК при остеосаркоме
Н. Е. Кушлинский a, М. В. Фридман b, Э. А. Брага c, d, *
a Национальный медицинский исследовательский центр онкологии им. Н.Н. Блохина Министерства здравоохранения России
115478 Москва, Россия
b Институт общей генетики им. Н.И. Вавилова Российской академии наук
117971 Москва, Россия
c Научно-исследовательский институт общей патологии и патофизиологии
125315 Москва, Россия
d Медико-генетический научный центр им. акад. Н.П. Бочкова
115478 Москва, Россия
* E-mail: eleonora10_45@mail.ru
Поступила в редакцию 29.04.2020
После доработки 17.05.2020
Принята к публикации 17.05.2020
Аннотация
Более двадцати лет назад микроРНК (miРНК) начали рассматривать и изучать как новый класс РНК, теперь приходит понимание их важной роли в регуляции генной экспрессии. Оказалось, что функция miРНК как “мастера-регулятора” может контролироваться другими не кодирующими белок РНК (ncРНК), в частности длинными ncРНК (lncРНК). Регуляторные функции lncРНК выявлены в опухолях разных локализаций, в том числе в остеосаркоме – самом распространенном и наиболее агрессивном злокачественном заболевании костей у детей в пубертатном периоде. В обзоре рассмотрены работы по изучению роли lncРНК в регуляции экспрессии генов по механизму конкурентных эндогенных РНК (ceРНК). Приведены данные, подтверждающие участие lncРНК в основных сигнальных путях: Notch, PI3K/AKT, Wnt/β-катенин, JNK, HIV/VEGF. Например, вовлеченность в сигнальные пути Notch и PI3K/AKT показана для 7 членов семейства SNHG (small nucleolar RNA host gene), причем почти для каждого из них выявлено по несколько регуляторных осей lncРНК/miРНК/мРНК. Рассмотрены функции и других многофункциональных онкогенных lncРНК, среди которых для TUG1, MALAT1 и XIST определено по 6‒10 таких осей. С применением баз данных Gene Cards, KEGG и Panther определены ключевые сигнальные пути для мишеней этих трех многофункциональных lncРНК. Изучение функций lncРНК ускорит разработку новых диагностических и прогностических маркеров при лечении больных остеосаркомой. На основании полученных данных можно утверждать, что взаимодействия между конкурентными эндогенными РНК: miРНК, мРНК и lncРНК ‒ это ранее неизвестный механизм регуляции экспрессии генов, который вовлечен в различные патофизиологические процессы, в том числе в онкогенез костей.
Эпигенетические механизмы регуляции генов вовлечены как в развитие организма, так и в разнообразные физиологические и патофизиологические процессы – от старения до образования и прогрессии опухолей. Эти “надгеномные” механизмы, т.е. не затрагивающие нуклеотидную последовательность генома, участвуют в регуляции экспрессии генов на разных уровнях [1]. В последние два десятилетия выявлена ключевая роль не кодирующих белок РНК (ncРНК) в регуляции экспрессии генов и сигнальных путей в клетке в норме и при патологии. Так, получены доказательства, что у эукариот, в том числе у человека, число генов ncРНК во много раз превышает число известных и предсказанных генов, кодирующих белки. С помощью анализа геномных баз данных и клонирования коротких РНК (длиной 20–200 нуклеотидов), присутствующих в суммарной клеточной РНК или ассоциированных с белками, обнаружены тысячи новых функциональных РНК. Входящие в состав общей массы РНК ncРНК могут быть условно классифицированы по длине: длинные (от ~200 до десятков тысяч нуклеотидов) и короткие (20–200 нуклеотидов). Группа коротких РНК включает в себя микроРНК (miРНК), тРНК, малые интерферирующие РНК (siРНК), транс-действующие siРНК (tasiРНК), РНК, ассоциированные с белками семейства Piwi (piРНК), и другие [2, 3].
Более двадцати лет назад miРНК начали рассматривать и изучать как новый класс РНК и выявили их регуляторную роль в генной экспрессии. Несмотря на то, что обычно регуляторное действие miРНК очень “тонкое” ‒ изменяет экспрессию целевого гена не более чем в два раза [4], консорциум ENCODE поместил miРНК в начало регуляторных сетей, возлагая на них роль “мастер-регуляторов” сигнальных каскадов в клетке [5]. Считается, что miРНК управляют экспрессией около 50% белоккодирующих генов, из чего следует, что они регулируют многие процессы жизнедеятельности клеток: пролиферацию, дифференцировку, апоптоз, адгезию, эпителиально-мезенхимальный переход (ЭМП) и метастазирование, ‒ что отражено в работах по изучению опухолей разного происхождения [6]. В последние годы актуальными стали вопросы эпигенетической регуляции самих miРНК. Это влияние метилирования промоторов на экспрессию генов miРНК [7], а также воздействие не так давно открытых длинных ncРНК (lncРНК) и кольцевых РНК.
Применение технологий секвенирования нового поколения позволило многое понять в механизмах реализации генетической информации. Так, большинство геномных последовательностей транскрибируется в виде lncРНК, число которых достигает 100 тыс. (см. базы данных http://www.noncode.org и http://www.lncrnadb.org/) и во много раз превышает число известных белоккодирующих генов и идентифицированных miРНК человека [8]. Молекулы lncРНК не кодируют белки, так как в них нет открытой рамки считывания достаточной длины (хотя описаны исключения, когда белок может кодироваться, но значимую роль играет и сама РНК [9]). Как правило, эволюционная консервативность всей последовательности lncРНК существенно меньше, чем у кодирующих белки генов, но выше, чем у нетранскрибируемых областей. Однако консервативность промоторных областей генов lncРНК такая же, как и у генов, кодирующих белки, а направляемая ими экспрессия lncРНК отличается высокой тканеспецифичностью [10].
Молекулы lncРНК имеют длину более 200 оснований, транскрибируются РНК-полимеразой II, кепируются и полиаденилируются по 5-' и 3′-концам соответственно [10]. Последовательности, кодирующие lncРНК, могут располагаться в межгенных областях, в интронах или частично перекрывать экзоны, локализуясь как на прямой, так и на обратной цепи [10]. В итоге их можно разделить на пять подклассов: смысловые, антисмысловые, двунаправленные, межгенные и интронные.
Молекулы lncРНК участвуют в разнообразных процессах: от модификации гистонов и влияния на ремоделирование хроматина до регуляции транскрипционных и посттранскрипционных процессов. Они могут быть энхансерами, каркасами, “губками”, конкурирующими за сайты связывания с другими РНК, а также предшественниками некоторых miРНК [8]. Аберрантная экспрессия lncРНК, в частности онкогенных lncРНК, приводит к нарушениям в сигнальных каскадах клетки, что может влиять на пролиферацию клеток и способствовать прогрессии и метастазированию опухолей [11].
В настоящее время наибольшее признание получила модель конкурентных эндогенных РНК (ceРНК), в которой lncРНК конкурирует с кодирующими белок мРНК за связывание с miРНК. Уже показано, что lncРНК взаимодействуют с теми сегментами miРНК, которые участвуют в связывании мРНК-мишеней. Так, еще в 2011 году Salmena и соавт. [12] высказали гипотезу, согласно которой транскрипты белоккодирующих генов, псевдогенов, miРНК и lncРНК вовлечены в сложную сеть взаимодействий за счет “ответных элементов miРНК” (miRNA response elements). Гораздо реже наблюдается взаимодействие lncРНК непосредственно с мРНК ‒ например, lncРНК WDR7-7 с мРНК гена GPR30 [13]. Таким образом, ceРНК формируют крупномасштабные регуляторные сети, в том числе и в опухолях [14‒16].
Остеосаркома (ОС) ‒ опухоль кости, наиболее распространенная у детей и подростков. ОС отличается высокой степенью злокачественности, крайне агрессивным течением, ранним гематогенным метастазированием и неблагоприятным прогнозом [17‒20]. Пик заболеваемости ОС отмечен в пубертатном периоде, причем в 2 раза чаще у мальчиков в возрастной группе 15‒18 лет ‒ когда начинают активно функционировать половые железы, продуцирующие тестостерон, который стимулирует рост остеобластов. Часто это сопровождается быстрым ростом длинных трубчатых костей нижних и верхних конечностей. Кости нижних конечностей поражаются в 5‒6 раз чаще, чем кости верхних конечностей, причем 80% всех опухолей нижних конечностей локализовано в области коленного сустава. Возможно, это связано с интенсивной пролиферацией остеобластов и повышенной частотой мутаций или эпигенетических модификаций в них. Среди детей пубертатного возраста 9% летальных исходов от онкологических заболеваний приходится на ОС. Следует сказать, что около 50% пациентов поступает на лечение в распространенной стадии заболевания с отдаленными метастазами в легких. Достигнуты значительные успехи в развитии терапевтических методов лечения ОС, включая адъювантную и неоадъювантную лучевую и химиотерапию. Однако показатель 5-летней выживаемости пациентов с метастатической ОС (менее 30%) не повысился за последнее десятилетие [21‒23]. У значительной части пациентов развивается резистентность опухоли к лекарственным препаратам, сохраняется высокий риск рецидива и развития метастазов после радикального хирургического удаления опухоли и курса химиотерапии.
Таким образом, остро стоит необходимость разработки новых чувствительных методов своевременной диагностики ОС и выявления мишеней для лекарственной терапии. Это позволит прогнозировать течение заболевания и оценивать эффективность проводимого лечения. Все это возможно при решении ключевого вопроса ‒ выяснении молекулярных механизмов регуляции генов и сигнальных путей в патогенезе ОС, в том числе роли ncРНК в этих процессах.
Ранее и нами [20], и другими авторами [24, 25] исследована возможная роль miРНК в процессах, вовлеченных в развитие ОС. Выявлено ингибирующее действие miРНК на трансляцию мРНК гена-мишени через их прямое связывание в составе комплекса RISC (RNA-induced silencing complex) для множества пар miРНК‒мРНК, задействованных в основных сигнальных путях, таких как Wnt (например, miR-342-5p/Wnt7b), Notch (mir-34a/Notch1) и PI3K/AKT (miR-196a/PTEN) [20, 26]. Вклад miРНК в патогенез ОС рассмотрен также в недавно опубликованных обзорах [27‒29].
За последние 5 лет выполнены сотни исследований, в которых была определена роль как известных, так и недавно открытых lncРНК в патогенезе ОС. В ряде обзорных статей отражены разнообразные функции lncРНК [30‒33] и продемонстрированы возможности их клинического применения как прогностических маркеров или мишеней для терапии [34‒36]. Однако накапливается все больше экспериментальных данных, согласно которым lncРНК участвует в патогенезе ОС по типу ceРНК c вовлечением в онкогенез miРНК и мРНК генов-мишеней.
В представленном обзоре проанализированы литературные данные по участию lncРНК в регуляции экспрессии ключевых генов, вовлеченных в патогенез ОС. При изучении взаимодействий эндогенных РНК разного типа, как правило, применяют комплекс методов. Так, уровни lncРНК, miРНК, мРНК in vivo и in vitro обычно оценивают методом количественной ПЦР с обратной транскрипцией или с использованием микрочипов. Для определения белков используют анализ вестерн-блот.
Для предсказания возможных взаимодействий между разными РНК применяют биоинформатику, а для экспериментального подтверждения “потенциальных взаимодействий” используют корреляционный анализ уровней lncРНК, miРНК и мРНК и двойной люциферазный тест. Кроме того, специально для выявления РНК, связывающихся с lncРНК, разработан метод РНК-pull-down. В связи с тем, что взаимодействия между разными ncРНК происходят в комплексе с белками семейства Argonaute, востребованы методы, подтверждающие прямое РНК-белковое взаимодействие, в частности анализ РНК-иммунопреципитации (RIP assay). При моделировании эффекта гиперэкспрессии той или иной РНК, как in vitro, так и in vivo (на ксенотрансплантатах), как правило, используют миметики или генно-инженерные конструкции. Снижения экспрессии целевого гена добиваются путем его нокдауна с помощью соответствующих siРНК или образующих шпильки коротких РНК (shРНК). Доказательством биологической значимости того или иного взаимодействия считается “отмена” эффекта гиперэкспрессии lncРНК (полностью или частично) при сверхэкспрессии miРНК-мишени и эффекта подавления экспрессии lncРНК (полностью или частично) при снижении экспрессии miРНК, с которой она связывается.
Изначально для поиска потенциально взаимодействующих РНК можно использовать методы биоинформатики, но стандартом современных исследований считается последующее подтверждение этого взаимодействия тем или иным биохимическим методом (см., например, работы [33, 37]). Нами обобщены данные в первую очередь тех исследований, в которых подтверждено прямое взаимодействие lncРНК и miРНК, приводящее к усилению трансляционной активности мРНК-мишеней этих miРНК.
СИГНАЛЬНЫЕ ПУТИ ПРИ ОСТЕОСАРКОМЕ
Важную роль в развитии ОС играет сигнальный путь Notch, регулирующий контроль дифференцировки остеобластов и сохранения стволовых клеток [38]. Notch-путь усиливает миграцию и инвазию клеток и стимулирует пролиферацию ОС, повышая при этом характеристики стволовости или стволовоподобия. Notch-путь повышает устойчивость к химиотерапии, а его ингибирование уменьшает размер опухолей и предотвращает метастазирование [39, 40].
Считается, что ОС возникает из остеобластов или еще более плюрипотентных предшественников в результате нарушения их дифференцировки, поэтому для развития опухоли важен Wnt-сигнальный путь, играющий важную роль в пролиферации и дифференцировке клеток. С ним связана активация стимулирующих пролиферацию онкогенов: c-Myc и CCND1 [41]. Супрессорным воздействием на этот путь обладает FOXO1, который способствует поддержанию и дифференцировке ранних предшественников линии остеобластов и подавляет пролиферацию более детерминированных их предшественников. FOXO1 ингибирует развитие остеосаркомы [42]. Путь FOXO, в свою очередь, подавляется путем PI3K (фосфоинозитид-3-киназа)/AKT (серин/треониновая протеинкиназа) [43]. Канонический каскад Wnt-β-катенин стимулирует прогрессию ОС, регуляция β‑катенина транскрипционным клеточным фактором TCF-1 и лимфоидным энхансерсвязывающим фактором LEF вносит важный вклад в этот путь [44].
Белок p53 и белок теплового шока-90 (HSP90) вовлечены в ингибирование TCF-1 и последующий апоптоз клеток ОС, играя, таким образом, антионкогенную роль [44]. Кроме того, p53 действует в качестве негативного регулятора остеобластогенеза за счет репрессии таких факторов транскрипции, как Osterix и Runx2 [45], которые требуются остеопрогениторным клеткам на начальной стадии остеогенеза [46, 47]. Вообще р53-путь, связанный с ответом на повреждения ДНК, арестом клеточного цикла, апоптозом и онкосупрессией, играет важнейшую роль в подавлении развития ОС. Мутации в р53 обнаруживают при ОС в 20‒50% наблюдений. MDM2 – негативный регулятор p53 – часто амплифицирован в ОС. В свою очередь, CDKN2A, ингибирующий MDM2 (что может быть одним из механизмов его действия), отсутствует примерно у 10% больных [48].
Mутации в гене Rb1 обнаруживают при ОС в 20‒40% случаев. Белок Rb ‒ онкосупрессор и негативный регулятор клеточного цикла. CDKN2A, опосредованно активирующий Rb, часто делетирован при ОС, а негативные регуляторы Rb – CDK4 и CCND1 – экспрессируются на повышенном уровне в ряде агрессивных ОС [48].
JNK – протеинкиназа, связанная с каскадным механизмом регуляции, “мастер-протеинкиназа”, играющая важную роль в пролиферации остеобластов, дифференцировке и апоптозе (см. обзор [49]). Ингибиторы соответствующего сигнального пути могут подавлять пролиферацию клеток ОС, а также метастазирование. Путь NF-κB (от англ. nuclear factor κB) изменяет транскрипционную активность многих генов в ответ на воздействия повреждающих агентов и цитокинов, поддерживает клеточную пролиферацию и защищает от апоптоза. Ингибирование этого пути за счет тех или иных механизмов обычно подавляет развитие ОС [50]. В развитие ОС вовлечена активация факторами роста (IGF, TGF, CTGF и др.) различных сигнальных путей (в частности PI3K/AKT и MAPK), влияющих на клеточный цикл и апоптоз [22]. С ответом на эти факторы связаны также васкуляризация и ангиогенез, существенные для роста опухоли. В этих процессах важную роль играет путь HIF-1 и активация, проангиогенного фактора VEGF, который наряду с путями ERK/NF-kB и PI3K/AKT, участвует также в активации антиапоптотического белка Bcl-2 и сурвивина. Для экспрессии VEGF важен активатор транскрипции STAT3, избыточная экспрессия которого в ОС ассоциирована с неблагоприятным прогнозом [22].
Недавно de Azevedo и соавт. [51] подробно описали сложившиеся на сегодняшний день представления по механизмам развития ОС. Они фокусируют внимание на роли различных эпигенетических факторов, влияющих на экспрессию значимых при ОС генов. Данные, полученные в последнее время методами биоинформатики, хорошо согласуются с этой картиной. Так, при поиске генов-драйверов ОС идентифицированы те, мутации в которых наблюдаются с высоким уровнем достоверности (“score”) предсказания. Из них выбраны гены, которые входят в базы данных Cancer Gene Census (СGC) и Osteosarcoma‑Gene Association (OGA). Это следующие пять генов: TP53, ЕGFR (вовлечены в сигнальные пути VEGF, ErbB, FOXO, MAPK, PD-L1, Ras, HIF-1 и PI3K/AKT), CREBBP (связан с ремоделированием хроматина, играет важную роль в эмбриональном развитии), SMAD4 (вовлечен в сигнальные пути TGFβ, FOXO и Wnt) и RB1 [52].
lncРНК В РЕГУЛЯЦИИ СИГНАЛЬНЫХ ПУТЕЙ ПРИ ОСТЕОСАРКОМЕ
Участники регуляторного процесса (оси) lncРНК/miРНК/мРНК вовлечены в основные сигнальные пути, задействованные в патогенезе ОС, такие как Notch, Wnt, JNK и PI3K/AKT. Роль ряда lncРНК в регуляции основных сигнальных путей, участвующих в патогенезе ОС, представлена на рис. 1.
Рис. 1.
Роль lncРНК в регуляции основных сигнальных путей в клетках остеосаркомы. Аббревиатуры/названия сигнальных путей даны на сером фоне. В овалах без фона даны регуляторные lncРНК.
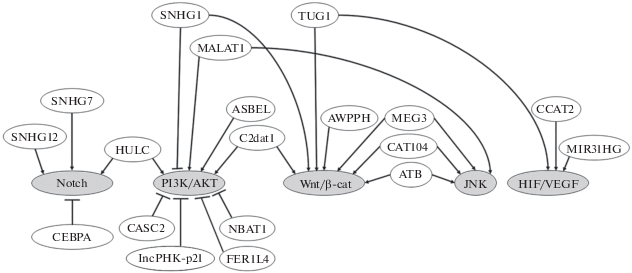
Так, сигнальный путь Notch играет важную роль в пролиферации клеток, дифференцировке, миграции, инвазии, метастазировании и активирован в клетках ОС [53]. При подавлении экспрессии lncРНК SNHG12 (small nucleolar RNA host gene 12) в клетках ОС повышается уровень miR-195-5p, что подавляет экспрессию Notch2, в результате чего блокируется клеточный цикл в фазе G0/G1, снижается скорость пролиферации и метастазирования опухоли [54]. Кроме того, повышение экспрессии других lncРНК семейства SNHG, связывающих и ингибирующих miРНК, ассоциировано с ростом и прогрессией ОС. Например, подавление SNHG7 в клетках ОС повышало экспрессию miR-34a, что индуцировало апоптоз, арест G1/S и сдерживало рост опухоли in vitro и in vivo, при этом была снижена экспрессия таких мишеней miR-34a, как Notch1, апоптозассоциированного белка BCL-2 (B-cell lymphoma 2 protein), Ki-67 и CDK6-киназы, участвующей в активации клеточного цикла [55].
Сигнальный путь PI3K/AKT активирован в клетках ОС и участвует в процессах повышения уровня пролиферации клеток, их выживаемости, миграции, инвазии и метастазирования [56, 57]. Так, lncРНК MALAT1 стимулирует пролиферацию и метастазирование клеток ОС, активируя путь PI3K/AKT, а также Rac1/JNK через связывание и ингибирование miR-509, а lncРНК AWPPH индуцирует прогрессию ОС, связывая и подавляя miR-93-3p, что приводит к активации ее мишени FZD7 и, как следствие, пути Wnt/β-катенин [58, 59]. Например, экзогенная экспрессия lncРНК CCAT1 (colon-cancer-associated transcript-1) в клетках ОС может повышать способность к пролиферации, миграции и инвазии in vitro посредством связывания miR-148a и, как следствие, повышения экспрессии онкогена, кодирующего PIK3IP1 (phosphatidyl inositol 3-kinase interacting protein 1) [60]. lncРНК DANCR индуцирует пролиферацию и инвазию ОС за счет конкурентного связывания и ингибирования miR-33a-5p и последующей активации рецепторной тирозинкиназы AXL, участвующей в сигнальном пути PI3K/AKT, что показано в экспериментах in vitro и на ксенотрансплантатах [61]. Нокдаун lncРНК HULC (highly upregulated in liver cancer) ингибирует пролиферацию, миграцию и инвазию клеток ОС in vitro за счет освобождения и активации miR-122 и ингибирования гена-мишени miR-122 ‒ HNF4G ‒ и подавления путей PI3K/AKT, JAK/STAT и Notch [62].
Сигнальные пути Wnt и JNK и их нарушения значимо связаны с патогенезом ОС [63, 64]. Нокдаун lncРНК CAT104 активирует апоптоз и ингибирует пролиферацию, миграцию и инвазию клеток ОС за счет освобождения и активации miR-381, подавления экспрессии гена-мишени miR-381 ‒ ZEB1 (zinc finger E box-binding homeobox 1) ‒ и снижения активности путей Wnt и JNK [65]. Нокдаун lncРНК MEG3 приводит к увеличению уровня апоптотических клеток in vitro, а также снижает уровень миграции, инвазии и метастазирования клеток ОС, что происходит за счет высвобождения miR-127 и подавления ее гена-мишени ZEB1 [66]. Эти данные соответствуют модели ceРНК (lncРНК MEG3 и мРНК ZEB1), конкурирующих за связывание miРНК miR-127. Показано, что miR-127 действует как репрессор путей Wnt и JNK, активных в клетках ОС, а ZEB1 – как их активатор. Следовательно, в эти сигнальные пути вовлечена также и lncРНК MEG3. Экзогенная экспрессия lncРНК ATB индуцировала пролиферацию, миграцию и инвазию клеток ОС, что было связано с подавлением экспрессии miR-200s и повышением экспрессии ее генов-мишеней ZEB1 и ZEB2 по механизму ceРНК [67]. Для lncРНК C2dat1 в экспериментах in vitro также показана онкогенная активность, повышение миграции и инвазии за счет связывания и ингибирования miR-34a-5p и повышения экспрессии Sirt1, что вносило вклад в активацию путей Wnt и p38/ERK/AKT [68]. Таким образом, для многих lncРНК определены сигнальные пути в клетках ОС и к ним относятся, в первую очередь, Notch, PI3K/AKT, Wnt/β- катенин и JNK, а также VEGF (см. рис. 1).
ОНКОГЕННЫЕ lncРНК, ДЕЙСТВУЮЩИЕ ПО МЕХАНИЗМУ ceРНК В КЛЕТКАХ ОСТЕОСАРКОМЫ
В опухолях, в том числе ОС, выявлено существенно больше lncРНК, проявляющих онкогенные свойства, чем онкосупрессорные.
Члены семейства SNHG (Small Nucleolar RNA Host Genes). В клетках ОС для многих членов семейства SNHG справедлива модель конкурентного взаимодействия между lncРНК и мРНК за связывание miРНК. Так, для lncРНК SNHG1 и в тканях, и в клеточных линиях ОС выявлена повышенная экспрессия одновременно с пониженной экспрессией miR-577, с геном-мишенью которой, WNT2B, lncРНК SNHG1 конкурирует за связывание miR-577. Это вовлекает lncРНК SNHG1 в сигнальный путь Wnt/β-катенин [69]. Ось SNHG1/miR-326/NOB1 также играет важную роль в прогрессии ОС, а lncРНК SNHG1 и мРНК NOB1 (nin one binding protein 1, предположительно играет роль в деградации мРНК) конкурируют за связывания miR-326 [70]. Аналогично lncРНК SNHG1 вовлечена также в ось SNHG1/miR-101-3p/ROCK1 (Rho‑associated coiled‑coil‑containing protein kinase 1) и участвует в активации ЭМП и инактивации пути PI3K/AKT [71].
lncРНК SNHG3 повышает экспрессию белка RAB22A (Ras-related protein Rab-22A) через цепочку SNHG3/miRNA-151a-3p/RAB22A, что может активировать миграцию и инвазию клеток ОС. Как и в большинстве цитированных работ, прямое связывание miRNA-151a-3p с lncРНК SNHG3 и с мишенью, мРНК RAB22A, подтверждено с применением комплекса методов, включая двойной люциферазный тест, анализ РНК-иммунопреципитации (RIP assay) и РНК-pull down [72]. Кроме того, повышенная в тканях ОС экспрессия lncРНК SNHG3 активирует синтез мРНК HOXC8 (Homeobox C8) через путь SNHG3/ miR-196a-5p/HOXC8, поэтому высокий уровень lncРНК SNHG3 может служить маркером плохого прогноза у больных ОС [73].
lncРНК SNHG5 индуцирует прогрессию и метастазирование клеток ОС за счет связывания miR-212-3p и последующей активации ее гена-мишени SGK3 (кодирует регулятор ионных каналов и мембранных транспортеров) через путь SNHG5/ miR-212-3p/SGK3 [74]. Аналогично, lncРНК SNHG5 действует как онкоген в клетках ОС через ось SNHG5/miR-26a/ROCK1 [75].
lncРНК SNHG7 стимулирует рост опухоли in vitro и in vivo и, связывая и снижая уровень miR-34a, повышает уровень экспрессии ряда мишеней miR-34a, включая ассоциированный с пролиферацией Notch1, апоптозассоциированный BCL-2 и ассоциированный с регуляцией клеточного цикла CDK6, а также ЭМП-ассоциированный SMAD4 [55].
lncРНК SNHG12 конкурирует с Notch2 за связывание miR-195-5p и вовлечена в миграцию и инвазию клеток ОС по оси SNHG12/miR-195-5p/Notch2 [54]. lncРНК SNHG12 может повышать пролиферацию и метастазирование ОС за счет активации другой мишени miR-195-5p ‒ мРНК IGF1R (receptor insulin-like growth factor 1) ‒ через путь SNHG12/miR-195-5p/IGF1R [76].
lncРНК SNHG16 индуцирует пролиферацию и прогрессию ОС, конкурируя с мРНК ZEB1 за связывание miR-205 и с мРНК BCL9 за связывание miR-1301, т.е. через две оси: SNHG16/miR-205/ZEB1 и SNHG16/miR-1301/BCL9 [77, 78].
lncРНК SNHG20 также играет роль онкогена при ОС и индуцирует пролиферацию и инвазию клеток через ось SNHG20/miR-139/RUNX2 (runt-related transcription factor 2). Нокдаун lncРНК SNHG20 активирует митохондриальный путь апоптоза [79].
Таким образом, прогрессию ОС стимулирует 7 членов семейства lncРНК SNHG: 1, 3, 5, 7, 12, 16 и 20; причем почти все они вовлечены в несколько сигнальных путей ‒ с разными “наборами” как miРНК, так и мРНК-мишеней.
lncРНК PVT1 (Plasmacytoma Variant Translocation 1) связывает miR-195 в клетках ОС in vitro, а снижение уровня miR-195 приводит к усилению экспрессии ее гена-мишени BCL2 и, как следствие, к ингибированию апоптоза. Увеличение содержания других мишеней miR-195 может индуцировать арест клеточного цикла через сигналинг miR-195/ CCND1 (Cyclin D1) и вызывать миграцию и инвазию через miR-195/FASN (fatty acid synthase) [80]. Валидирована также ось PVT1/miR-497/HK2 и роль PVT1 в активации гликолиза и прогрессии ОС [81]. Можно отметить, что, согласно базе starBase v2.0, на PVT1 имеются по одному, реже по два участка связывания для более чем 20 miРНК, включая miR-195-5p и miR-497-5p [80, 81]. Кроме того, экзосомы, секретируемые стволовыми клетками костного мозга, переносят и повышают содержание PVT1 в клетках ОС, что индуцирует метастазирование. В этом случае lncРНК PVT1 вовлечена в ось PVT1/miR-183-5p/ERG, которая ингибирует деградацию и убиквитинирование онкобелка ERG (v-ets avian erythroblastosis virus E26 oncogene homolog, относится к семейству транскрипционных факторов ETS) [82]. lncРНК PVT1 стимулирует онкогенез и прогрессию ОС по нескольким путям, включающим 3 miРНК (miR-195, miR-497, miR-183) и 5 их генов-мишеней (BCL2, CCND1, FASN, HK2, ERG).
Как показано на рис. 2, для lncРНК TUG1 (Taurine Upregulated 1) в клетках ОС идентифицировано 8 осей с конкурирующими мРНК при посредничестве miРНК (TUG1/miРНК/мРНК): TUG1/miR-9-5p/POU2F1 (POU class 2 homeobox 1) [83], TUG1/miRNA-144-3p/EZH2 (enhancer of zeste homolog 2), участвующего в пути Wnt/β-катенин [84], TUG1/miR-335-5p/ROCK1 [86], TUG1/ miR-132-3p/SOX4 [85], TUG1/miR-212-3p/ FOXA1 (forkhead box A1 ) [87], TUG1/miR-143-5p/HIF-1α [88], TUG1/miR-425-5p/CTNNB1 (CCND1, c-Myc) [89], TUG1/miR-140-5p/PFN2 (Profilin-2) [90].
Рис. 2.
Восемь регуляторных осей онкогенной lncРНК TUG1 в клетках остеосаркомы. Показано также соучастие lncРНК MALAT1 в ингибировании miR-425-5p, а также miR-425-5p – в ингибировании трех мРНК-мишеней. Обозначения lncРНК даны жирным шрифтом на сером фоне. В овалах без фона даны мишени lncРНК TUG1 и MALAT1 ‒ miРНК (средние ряды) ‒ и мишени miРНК ‒ мРНК белков (верхний и нижний ряды). Схема построена на основании данных, полученных в работах [83‒90].
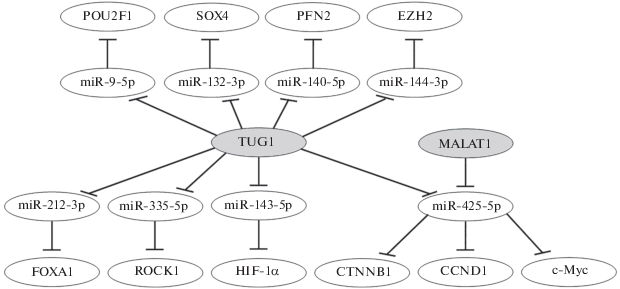
Эти данные раскрывают разнообразие механизмов влияния lncРНК TUG1 на прогрессию и метастазирование ОС. Интересным наблюдением стало участие двух lncРНК, TUG1 и MALAT1, в общих осях: TUG1 (MALAT1)/miR-425-5p/ CTNNB1 (CCND1, c-Myc), ‒ вовлеченных в сигнальный путь Wnt/β-катенин [89] (рис. 2). Можно предполагать, что изменения уровней lncРНК MALAT1 и TUG1 согласованно влияют на активность сигналинга Wnt/β-катенин.
Сообщено также о влиянии TUG1 на прогрессию ОС через воздействие непосредственно на мРНК гена RUNX2 (runt-related transcription factor 2), однако возможное посредническое участие какой-либо или каких-либо miРНК в этих регуляторных функциях пока не исследовано [91].
Характеристика функций белков, в регуляцию которых вовлечена lncРНК TUG1 в клетках ОС (рис. 2), и их участие в сигнальных путях по базам данным Gene Cards (https://www.genecards.org/), KEGG (https://www.genome.jp/kegg/pathway.html) и Panther (http://www.pantherdb.org/panther/globalSearch.jsp?) представлены в табл. 1.
Таблица 1.
Функции белков, связанных с регуляторными осями lncРНК TUG1
| Белок | Характеристика по GeneCards | Влияние на сигнальные пути и процессы в ОСа | Сигнальные пути по KEGG | Сигнальные пути по Panther |
|---|---|---|---|---|
| POU2F1 | Транскрипционный фактор, белок гомеобокса | Пролиферация, образование колоний, подавление апоптоза, переход от G0 к G1 [83] | С/п рецептора гонадотропин-релизинг-гормона | |
| SOX4 | Транскрипционный фактор, связан с путями апоптоза, ведущими к смерти клетки или онкогенезу, опосредует влияние паратиреоидного гормона на образование кости | Пролиферация и подавление апоптоза [86] | ||
| PFN2 | Регулирует полимеризацию актина в ответ на внеклеточные сигналы | Пролиферация, миграция и инвазия [90] | ||
| EZH2 | Принадлежит к группе Polycomb, репрессирует транскрипцию за счет метилирования гистонов | Миграция клеток и ЭМП, с/п Wnt/β-catenin [84] | ||
| FOXA1 | Активатор транскрипции, открывает конденсированный хроматин за счет взаимодействия с нуклеосомами | Пролиферация, подавление апоптоза [87] | ||
| ROCK1 | Серин/треониновая киназа, регулирующая фокальную адгезию и стрессовые волокна | Миграция и инвазия [85] | Направление аксонов, плотный контакт, трансэндотелиальная миграция лейко-цитов, фокальная адгезия, регуляция актинового цитоскелета; с/п: TGFβ, хемокинов | Регуляция цитоскелета RHO GTPазой; опосредованное хемокинами воспаление; с/п цитокинов |
| HIF1A | Мастер-регулятор ответа на гипоксию | Пролиферация, метастазирование, ангиогенез [88] | Дифференцировка Th17 клеток, митофагия (животные), аутофагия (животные), с/п: тиреоидных гормонов, HIF-1, PD-L1 экспрессии и PD-1 контрольной точки при раке | Ангиогенез; опосредованный активацией HIF ответ на гипоксию; с/п VEGF |
| CTNNB1 | Часть комплекса адгезионных контактов, связан с актиновым цитоскелетом и контактным торможением деления клеток | Пролиферация, миграция и инвазия [89] | Адгезионные контакты, фокальная адгезия трансэндотелиальная миграция лейкоцитов; с/п: Rap1, Wnt, Hippo, тиреоидных гормонов, регулирующие плюрипотентность стволовых клеток | Ангиогенез; с/п: кадгерина, Wnt, петли обратной связи-2 p53, рецептора гонадотропин-релизинг-гормона |
| cMYC | Ядерный фосфопротеин, играющий роль в прогрессии клеточного цикла и апоптозе, онкоген, образует гетеродимер с транскрипционным фактором MAX | Пролиферация, миграция и инвазия [89] | Центральный углеводный обмен при раке, нарушение регуляции транскрипции при раке, клеточное старение, клеточный цикл; с/п: MAPK, ErbB, PI3K/AKT, Wnt, TGFβ, Hippo, JAK/STAT, тиреоидных гормонов, p53 и регулирующие плюрипотентность стволовых клеток | Ответ на окислительный стресс; с/п: интерлейкинов, PDGF, петли обратной связи-2 p53 |
| CCND1 | Циклин, активность которого в комплексе с другими белками необходима для G1/S перехода | Пролиферация, миграция и инвазия [89] |
Клеточное старение, клеточный цикл, фокальная адгезия, плотные контакты, протеогликаны при раке; с/п: PI3K/AKT, Hedgehog, FoxO, p53, AMPK, Wnt, Hippo, JAK/STAT, тиреоидных гормонов | Клеточный цикл; с/п: PI3K, Wnt |
а Здесь и в табл. 2 и 3: с/п – сигнальный(е) путь(и).
Таблица 2.
Функции белков, связанных с регуляторными осями lncРНК XIST
| Белок | Характеристика по GeneCards |
Влияние на сигнальные пути и процессы в опухоли | Сигнальные пути по KEGG | Сигнальные пути по Panther |
|---|---|---|---|---|
| RAP2B | Предполагается роль в перестройке цитоскелета; GTP-связывающий белок, вовлечен в с/п EGFR и CHRM3 | Пролиферация и инвазия [105] | С/п интегринов | |
| mTOR | Киназа, опосредующая клеточный ответ на повреждение ДНК и другие стрессовые воздействия; связана с арестом клеточного цикла, часть с/п PI3K/AKT | Пролиферация и аутофагия, подавление апоптоза [108] | Клеточное старение, аутофагия (животные), устойчивость к ингибиторам EGFR тирозинкиназ, центральный углеводный обмен при раке, метаболизм холина при раке, дифференцировка Th17-клеток; синтез, секреция и действие гормона роста; с/п: mTOR, фосфолипазы D, инсулина, адипоцитокина, PI3K/AKT, ErbB, HIF-1, AMPK, тиреоидных гормонов, PD-L1 экспрессии и PD-1 контрольной точки при раке, JAK/STAT, MAPK, p53 | Ответ на гипоксию, опосредованный активацией HIF; с/п: интерлейкинов, PDGF, p53 и вызванный глюкозным голоданием |
| RSF1 | Часть комплекса ремоделирования хроматина, гистоновый шаперон | Активация с/п MAPK/ERK [106] | ||
| MMP2 | Расщепляет компоненты внеклеточного матрикса и молекулы, участвующие в передаче сигнала | Пролиферация и инвазия [109] | Протеогликаны при раке, трансэндотелиальная миграция лейкоцитов; с/п: эстрогена, рецептора гонадотропин-релизинг-гормона | |
| MMP9 | Играет роль в расщеплении внеклеточного матрикса и миграции лейкоцитов, в резорбции кости остеокластами | Пролиферация и инвазия [109] | Протеогликаны при раке, трансэндотелиальная миграция лейкоцитов; с/п: IL-17, TNF и эстрогена | |
| YAP1 | Транскрипционный регулятор (коактиватор и корепрессор), эффектор, действующий на сигнальный каскад ниже Hippo | Пролиферация инвазия, ЭМП [107] | С/п Hippo | |
| PDCD4 | Ингибирует инициацию трансляции, участвует в апоптозе, модулирует активацию JUN-киназы | Ингибирование пролиферации, миграции и инвазии [110] | Протеогликаны при раке |
Таблица 3.
Функции белков, связанных с регуляторными осями lncРНК MALAT1
| Белок | Характеристика по GeneCards |
Влияние на сигнальные пути и процессы в опухоли | Сигнальные пути по KEGG | Сигнальные пути по Panther |
|---|---|---|---|---|
| Rac1 | Мембранная GTPаза, регулирует секреторные процессы, поляризацию, миграцию и адгезию клеток | Пролиферация [58] | Направление аксонов, адгезионные контакты, плотные контакты, трансэндотелиальная миграция лейкоцитов, протеогликаны при раке, дифференцировка остеокластов, регуляция актинового цитоскелета, фокальная адгезия, опосредованная натуральными киллерами цитотоксичность, FcγR-опосредованный фагоцитоз; с/п: Wnt, Ras, Rap1, PI3K/AKT, Toll-подобных рецепторов, нейротрофинов, MAPK, хемокинов, сфинголипидов, VEGF, рецепторов В-клеток, FcεRI | Ангиогенез, наведение аксонов, активация В-клеток, регуляция цитоскелета GTPазой RHO, активация Т-клеток, активация сигналинга VEGF; с/п: опосредованное хемокинами и ци-токинами воспаление, EGF, Ras, рецептора гонадотропин-релизинг-гормона |
| RET | Тирозинкиназа ‒ трансмембранный рецептор, регулирует клеточную пролиферацию, миграцию и дифференцировку | Активирует с/п PI3K/AKT, увеличивает стволовость клеток [115] | Центральный углеводный обмен при раке | |
| HMGB1 | Ядерный негистоновый белок, влияющий на организацию ДНК, связан с репликацией, транскрипцией, ремоделированием хроматина, репарацией, рекомбинацией и стабильностью ДНК; ДНК-шаперон. Связан с иммунитетом, воспалением, клеточной дифференцировкой и миграцией | Пролиферация, подавление апоптоза [113] | Эксцизионная репарация оснований, аутофагия (животные), некроптоз | С/п p53 |
| CDK9 | Циклинзависимая протеинкиназа, регулирует прогрессию клеточного цикла, транскрипцию | Пролиферация [114] | Нарушения регуляции транскрипции при раке | |
| HDAC4 | Гистоновая деацетилаза, подавляет транскрипцию | Пролиферация, подавление апоптоза [116] | ||
| SMAD4 | Белок, активируемый трансмембранным рецептором ‒ серин/треониновой киназой ‒ в ответ на TGFβ. Регулятор транскрипции, антионкоген, подавляет ангиогенез и увеличивает сосудистую проницаемость; критическая роль в развитии кости | Пролиферация [111] | Дифференцировка клеток Th17, клеточный цикл, адгезионные контакты; с/п: TGFβ, Hippo, регулирующие плюрипотентность стволовых клеток, FoxO, Wnt | С/п: TGFβ, Wnt, активин β, рецептора гонадотропин-релизинг-гормона |
| SOX4 | Транскрипционный фактор, связан с путями апоптоза, ведущими к смерти клетки или онкогенезу, опосредует влияние паратиреоидного гормона на образование кости | Пролиферация, миграция и инвазия [117] | ||
| cМЕТ | Рецепторная тирозинкиназа, реагирующая на сигналы из внеклеточного матрикса, регулирует пролиферацию, морфогенез и выживание клеток, активирует с/п RAS/ERK и PI3K/AKT | Пролиферация, миграция и инвазия [117] | Устойчивость к ингибиторам EGFR тирозинкиназ, нарушение регуляции транскрипции при раке | |
| CCND1 | Циклин, активность которого в комплексе с другими белками необходима для перехода G1/S | Выживаемость, миграция и инвазия клеток [118] | Клеточное старение, клеточный цикл, фокальная адгезия, плотные контакты, протеогликаны при раке; с/п: PI3K/AKT, Hedgehog, FoxO, p53, AMPK, Wnt, Hippo, JAK/STAT, тиреоидных гормонов | Клеточный цикл; с/п: Wnt, киназы PI3K |
| ROCK1 | Серин/треониновая киназа, регулирующая фокальную адгезию и стрессовые волокна | Пролиферация, метастазирование [112] | Наведение аксонов, плотные контакты, трансэндотелиальная миграция лейкоцитов, фокальная адгезия, регуляция актинового цитоскелета, протеогликаны при раке; с/п: TGFβ и хемокинов | Регуляция цитоскелета GTPазой RHO, с/п воспаления, опосредованного хемокинами и цитокинами |
| ROCK2 | Серин/треониновая киназа, регулирующая фокальную адгезию и стрессовые волокна | Пролиферация, метастазирование [112] | Наведение аксонов, плотные контакты, трансэндотелиальная миграция лейкоцитов, фокальная адгезия, регуляция актинового цитоскелета, протеогликаны при раке; с/п: Wnt, хемокинов и сфинголипидов | Регуляция цитоскелета GTPазой RHO |
Как видно из табл. 1, среди белков, на экспрессию которых влияет lncРНК TUG1, много связанных с цитоскелетом и его преобразованиями, а также транскрипционных факторов.
lncРНК NEAT1 (Nuclear Enriched Abundant Transcript 1) также проявляет онкогенные свойства в тканях и клеточных линиях ОС. Для нее выявлены взаимодействия по осям NEAT1/miR-34c/BCL2 (CCND1) [92] и NEAT1/miR-339-5p/TGF-β1 (transforming growth factor β1) [93].
lncРНК APTR (Alu-mediated p21 Transcriptional Regulator) участвует в прогрессии ОС через ось APTR/miR-132-3p/YAP1 (Yes-associated protein 1) [94].
Онкогенная lncРНК DICER1-AS1 участвует в процессах пролиферации, миграции, аутофагии in vitro и in vivo через ось DICER1-AS1/miR-30b/ATG5 (autophagy related 5) [95].
Межгенная lncРНК LINC00858 участвует в патогенезе ОС через ось LINC00858/miR-139/CDK14 (cyclin-dependent kinase 14) [96].
Антисмысловая lncРНК HOXA11-AS (Homeobox A11 Antisense), локализованная в генном кластере HOXA11, участвует в развитии и прогрессии ОС посредством нескольких сигнальных осей, из которых определены две: HOXA11-AS-miR-124-3p-ROCK1 [97] и HOXA11-AS/ miR-125a-5p/Rab3D (Rab GTPase 3D, регулятор внутриклеточного транспорта везикул) [98].
Антисмысловая lncРНК FEZF1-AS1 (FEZF1 Antisense RNA 1, антисмысловая lncРНК гена цинковый палец-1 семейства FEZ) стимулирует пролиферацию, миграцию, инвазию ОС in vitro и in vivo через сигнальные взаимодействия FEZF1-AS1/ miR-4443/NUPR1 (nuclear protein 1, transcriptional regulator) [99].
Антисмысловая lncРНК TP73-AS1 (Tumour Protein P73 Antisense RNA 1), тоже проопухолевого типа, способствует прогрессии ОС через ось взаимодействий TP73-AS1/miR-142/Rac1 (RAS related C3 botulinum substrate 1) [100].
Нокдаун lncРНК ODRUL приводит к повышению уровня миграции клеток ОС, инвазии и роста опухоли in vitro и in vivo. Идентифицированы прямые последовательно ингибирующие взаимодействия по оси ODRUL/miR-3182/MMP2 (matrix metalloproteinase 2) [101].
lncРНК UCA1 (Urothelial Carcinoma Associated 1) активирует пролиферацию, миграцию и инвазию ОС. Выявлено прямое связывание UCA1 с miR-182 и miR-182 с iASPP (inhibitor of apoptosis-stimulating protein of p53), из чего следует, что в патогенез ОС вовлечена ось UCA1/miR-182/iASPP [102].
Межгенная lncРНК LINC00511 (Long Intergenic Non-protein Coding RNA 511) стимулирует прогрессию ОС, что связано с активацией протоонкогена MAEL (Maelstrom) через прямые взаимодействия вдоль оси LINC00511/miR-618/MAEL [37].
Уровень lncРНК OIP5-AS1 (Opa-Interacting Protein 5 Antisense Transcript 1) повышен при ОС, OIP5-AS1 связывает miR-200b-3p и повышает экспрессию CDK14 (циклинзависимой киназы 14) через ось OIP5-AS1/miR-200b-3p/CDK14, что ассоциировано с подавлением апоптоза и активацией пролиферации клеток ОС [103]. Активация другой мишени miR-200b-3p – мРНК FN1 (фибронектин 1) ‒ приводит к снижению чувствительности клеток ОС к доксорубицину in vitro и in vivo через ось OIP5-AS1/miR-200b-3p/FN1 [104].
Для онкогенной lncРНК XIST (X Inactive-Specific Transcript) выявлено 6 путей влияния на прогрессию ОС через прямые воздействия на несколько miРНК и их мишени (рис. 3). На рис. 3 отражены 5 осей проопухолевого влияния XIST: XIST/miR-320b/RAP2B (Ras-related protein 2B) [105], XIST/miR-193a-3p/RSF1 (remodeling and spacing factor 1) [106], XIST/miR-195-5p/YAP [107], XIST/miR-375-3p/mTOR [108] и XIST/miR-137/MMP2, MMP9 [109]. В противоречии с данными этих 5 статей находится работа [110], согласно которой lncРНК XIST негативно влияет на прогрессию и метастазирование ОС, повышая содержание онкосупрессора PDCD4 (programmed cell death 4) по оси XIST/miR-21-5p/ PDCD4) (рис. 3).
Рис. 3.
Регуляторные оси онкогенной lncРНК XIST в клетках остеосаркомы. Показано 5 онкогенных функций lncРНК XIST и одна супрессорная. Мишени lncРНК XIST – miРНК (средний ряд), мишени miРНК – мРНК белков (нижний ряд). Схема составлена на основании результатов, представленных в работах [105‒110].
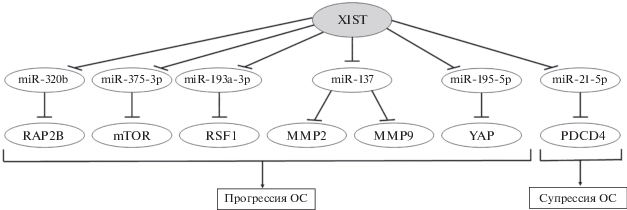
Среди белков, на экспрессию которых влияет lncРНК XIST, присутствуют связанные с перестройкой цитоскелета клетки, взаимодействием с внеклеточным матриксом, ремоделированием хроматина и регуляцией транскрипции. Особняком стоит PDCD4 (табл. 2). Возможно, при взаимодействии с разными мишенями функция lncРНК XIST может варьировать, чем можно объяснить “выпадение” результатов, полученных в работе [110]. В этой работе настораживает другое ‒ сниженный уровень этой lncРНК при ОС, в то время как по результатом других авторов он всегда повышен относительно нормального.
Для наиболее хорошо изученной онкогенной lncРНК MALAT1 (Metastasis-Associated Lung Adenocarcinoma Transcript 1) найдено наибольшее число (более 10) осей регуляции генов в клетках ОС по механизму ceРНК, а именно: MALAT1/miR-205/SMAD4 [111], MALAT1/miR-144-3p/ROCK1 (ROCK 2) [112], MALAT1/miR-142-3p/HMGB1 или MALAT1/miR-129-5p/HMGB1 [113], MALAT1/miR-206/CDK9 [114], MALAT1/ miR-129-5p/RET [115], MALAT1/miR-509/Rac1 [58], MALAT1/miR-140-5p/HDAC4 [116], MALAT1/ miR-34a/c-5p/c-Met, MALAT1/miR-34a/c-5p/ SOX4, MALAT1/miR-449a/b/c-Met, MALAT1/ miR-449a/b/SOX4 [117], MALAT1/miR‑34a/CCND1 [118]. Данные этих работ по участию MALAT1 в регуляции генов при ОС по механизму ceРНК суммированы на рис. 4. Соучастие lncРНК MALAT1 с lncРНК TUG1 в оси miR-425-5p/CTNNB1 (CCND1, c-Myc) [89] отражено выше на рис. 2.
Рис. 4.
Регуляторные оси множественной онкогенной lncРНК MALAT1 в клетках остеосаркомы. Мишени lncРНК MALAT1 – miРНК, мишени miРНК – мРНК белков. Показаны пересечения ряда осей в общих мРНК-мишенях (HMGB1, SOX4 и c-Met), наличие двух мишеней у miR-129-5p, miR-144-3p и miR-449a/b и трех мишеней у miR-34a/c-5p. Схема составлена на основании данных, представленных в работах [58, 111–118].
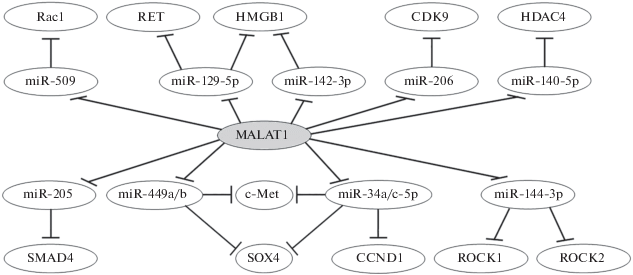
Стоит обратить внимание на пересечение осей, проходящих через miR-142-3p и miR-129-5p, в мРНК общего целевого белка HMGB1 (High mobility group box 1 ) (рис. 4) [113]. Через miR-129-5p lncРНК MALAT1 активирует и протоонкоген RET (REarranged during Transfection). Это повышает стволовоподобные свойства клеток ОС и активирует путь PI3K/AKT [115]. Множественность функций lncРНК отражает пересечение осей через miR-34a/c-5p и miR-449a/b в общих мишенях ‒ мРНК белков c-Met и SOX4 [117]. Через связывание miR-34a lncРНК MALAT1 активирует циклин D1 [118].
Среди белков, на экспрессию которых влияет lncРНК MALAT1, много участвующих в регуляции деления и клеточного цикла, состояния цитоскелета и взаимодействия клетки с внеклеточным матриксом, а также присутствуют факторы ремоделирования хроматина и транскрипции (табл. 3).
Обнаружение множества осей для ряда lncРНК через набор miРНК подтверждает наличие в их молекулах нескольких сайтов связывания, предназначенных для разных miРНК, а следовательно, и их мультифункциональность. В некоторых работах определены конкретные элементы ответа miРНК, за счет которых осуществляется регуляторное взаимодействие lncРНК с мРНК. Так, 7 нуклеотидов на 5'-конце miR-144-3p комплементарны идентичным между собой 7-нуклеотидным участкам 3'-нетранслируемой области (3'-UTR) MALAT1 и мРНК ROCK1 [111]. Элементы ответа miРНК выявлены также в miR-140-5p и в 3'-UTR lncРНК MALAT1 и мРНК HDAC4 (histone deacetylase 4) [116]. Отмечается чрезмерно высокий уровень экспрессии lncРНК MALAT1 в клетках ОС, который ассоциирован с усилением пролиферации, подвижности, инвазии, ЭМП и метастазирования, а также с активацией сигнальных путей PI3K/AKT и Rac1/JNK и повышенным проявлением стволовоподобных свойств. Во многих работах указано, что lncРНК MALAT1 может служить перспективной прогностической и терапевтической мишенью для пациентов с ОС [58, 89, 111–118]. Таким образом, эти данные подтверждают участие lncРНК MALAT1 в регуляции экспрессии генов пролиферации, клеточного цикла, подвижности, стволовости и метастазирования клеток ОС.
Недавно показано, что в прогрессию ОС вовлечен еще ряд онкогенных lncРНК по механизму ceРНК через оси, представленные на рис. 5.
Рис. 5.
Оси 9 онкогенных lncРНК в клетках остеосаркомы: SND1-IT1, DLEU1, CCAT2, KCNQ1OT1, HOTAIR, LINC00460, ILF3-AS1, LINC00689, MIR31HG. Мишени lncРНК – miРНК, мишени miРНК – мРНК белков (третий нижний ряд). Транскрипционный фактор SP1 активирует промоторы двух lncРНК: LINC00689 и ILF3-AS1. Схема составлена на основании данных, представленных в работах [119‒127].
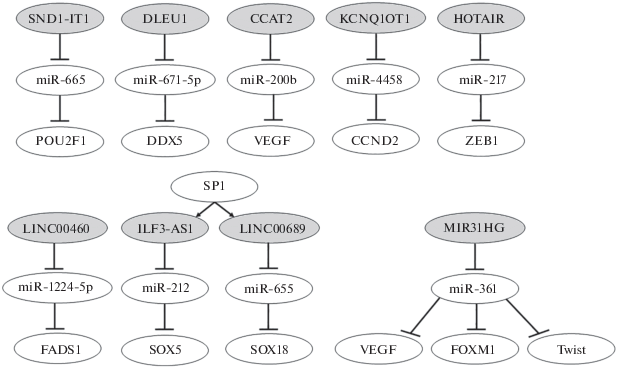
Так, lncРНК SND1-IT1 (SND1 Intronic Transcript 1) активирует ген POU2F1 (POU domain, class 2, transcription factor 1) через прямое связывание с miRNA-665 по оси SND1-IT1/miRNA-665/POU2F1, что усиливает пролиферацию и миграцию клеток ОС [119].
Антисмысловая lncРНК ILF3-AS1 (Interleukin Enhancer-Binding Factor 3, Antisense RNA 1), индуцированная фактором транскрипции SP1, активирует транскрипционный фактор SOX5 через связывание с miR-212 по оси SP1-ILF3-AS1/miR-212/SOX5, что стимулирует прогрессию ОС [120].
Межгенная lncРНК LINC00689, также индуцированная SP1, активирует транскрипционный фактор SOX18 по оси SP1-LINC00689/miR-655/SOX18 [121].
Антисмысловая lncРНК KCNQ1OT1 (KCNQ1 Opposite Strand/Antisense Transcript 1) активирует циклин D2 через miR-4458 по оси KCNQ1OT1/ miR-4458/CCND2, что также ассоциировано с прогрессией ОС [122].
lncРНК DLEU1 (Deleted in Lymphocytic Leukemia 1) широко экспрессирована в клетках и опухолевых тканях ОС и как онкоген активирует DDX5 (DEAD-box helicase 5, РНК-хеликаза) посредством прямых взаимодействий с miR-671-5p через ось DLEU1/miR-671-5p/DDX5 [123].
Межгенная антисмысловая lncРНК HOTAIR (HOX Transcript Antisense Intergenic RNA) через miR-217 вовлечена в активацию экспрессии белка ZEB1 (zinc finger transcription factor), стимулирующего переход ЭМП по оси HOTAIR/miR-217/ ZEB1 [124] (рис. 5).
В прогрессию ОС вовлечены также межгенная lncРНК LINC00460 по оси: LINC00460/miR-1224-5p/FADS1 (fatty acid desaturase 1) [125] и lncРНК CCAT2 (Colon Cancer-Associated Transcript 2), которая, активируя VEGF, стимулирует ангиогенез в опухоли по оси CCAT2/miR-200b/ VEGF [126].
lncРНК MIR31HG ((miR-31)-host gene) высоко экспрессирована в клеточных линиях ОС и в тканях опухолей пациентов с распространенной стадией ОС и отдаленными метастазами. Повышенная экспрессия MIR31HG in vitro и in vivo стимулирует рост клеток ОС посредством ингибирования miR-361 и усиления экспрессии VEGF, FOXM1 (активатор транскрипции, вовлеченный в клеточную пролиферацию) и Twist (транскрипционный фактор) по оси MIR31HG/miR-361/VEGF (FOXM1, Twist) [127].
Перечисленные выше 9 недавно открытых осей 9 онкогенных lncРНК, которые стимулируют развитие и прогрессию ОС по механизму сеРНК, приведены на рис. 5.
Таким образом, все рассмотренные выше (около 30) lncРНК действуют по типу ceРНК (по отношению к miРНК и мРНК) и проявляют, главным образом, онкогенные проопухолевые свойства в отношении развития и прогрессии ОС.
СУПРЕССОРНЫЕ lncРНК, ДЕЙСТВУЮЩИЕ ПО МЕХАНИЗМУ ceРНК В КЛЕТКАХ ОСТЕОСАРКОМЫ
Для ряда lncРНК выявлена супрессорная активность в отношении пролиферации, миграции и инвазии клеток ОС по механизму последовательного прямого связывания через ось lncРНК/miРНК/ мРНК. В этом случае, как правило, при низком содержании lncРНК повышен уровень соответствующей miРНК, что приводит к подавлению уровня мРНК гена-мишени.
Так, Ba и соавт. [128] обнаружили, что экспрессия lncРНК CASC2 снижена в клинических образцах и клеточных линиях ОС и это коррелировало с прогрессирующей стадией, в то время как эктопическая экспрессия lncРНК CASC2 ингибировала пролиферацию клеток ОС, образование колоний и инвазию. Авторы выяснили, что эктопическая экспрессия lncРНК CASC2 в клеточных линиях ОС подавляла экспрессию miR-181a и усиливала экспрессию RASSF6 (члена 6 семейства Ras-ассоциированных доменов), PTEN (phosphatase and tensin homolog deleted on chromosome ten; ингибитор PI3K/AKT пути) и Ser/Thr-протеинкиназы ATM ‒ важной контрольной точки клеточного цикла. Кроме того, мРНК RASSF6, PTEN и ATM идентифицированы как прямые мишени miR-181a. Подтверждением оси CASC2/ miR-181a/RASSF6 стало выявление прямой корреляции между уровнями lncРНК CASC2 и мРНК RASSF6 [128] (рис. 6).
Рис. 6.
Регуляторные оси 7 супрессорных lncРНК: CASC2, lncРНК-p21, NBAT1, FER1L4, CEBPA-AS1, LINC00588, GAS5 ‒ в клетках остеосаркомы. Мишени lncРНК – miРНК, мишени miРНК – мРНК белков (третий нижний ряд). Видны множественные мишени для пар CASC2/miR-181a и NBAT1/miR-21. Показаны две оси, регулируемые lncРНК GAS5, и регуляция самой GAS5 под действием транскрипционного фактора ZBTB7A и ингибирующей его miR-665a. Схема построена по данным, представленным в работах [128‒136].
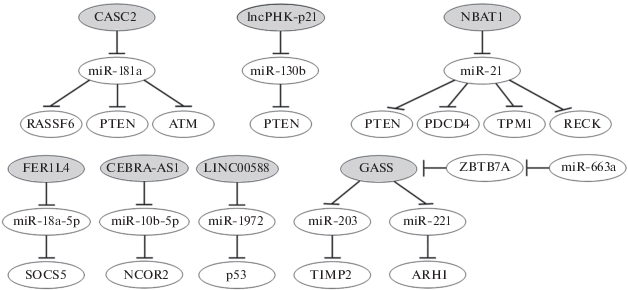
Экспрессия lncРНК-p21 снижена в ткани ОС, а in vitro показана способность этой lncРНК подавлять пролиферацию клеточных линий и повышать уровень PTEN через связывание miR-130b, т.е. по оси lncРНК-p21/miR-130b/PTEN [129] (рис. 6).
lncРНК NBAT1 (Neuroblastoma Associated Transcript 1) функционирует как опухолевый супрессор в разных видах злокачественных опухолей, в том числе и ОС. lncРНК NBAT1 подавляет рост и метастазирование in vitro и in vivo за счет ингибирования miR-21 путем прямого связывания, что приводит к повышению экспрессии мишеней последней ‒ мРНК, кодирующих PTEN, PDCD4 (programmed cell death protein 4), TPM1 (tropomyosin alpha-1 chain) и RECK (reversion inducing cysteine rich protein with kazal motifs). Все эти белки проявляют супрессорные свойства [130]. RECK, негативный регулятор MMP9, рассматривают как супрессор метастазирования. Эти взаимодействия отражает ось NBAT1/miR-21/PTEN (PDCD4, TPM1, RECK) (рис. 6).
lncРНК GAS5 (Growth Arrest-Specific 5) подавляет миграцию и инвазию клеток ОС через связывание miR-203a, тем самым ингибируя ее взаимодействие с мишенью ‒ мРНК TIMP2 (tissue inhibitor of metalloproteinases 2). Аналогично GAS5 влияет на путь miR-221/ARHI (aplysia ras homology member I, или GTP-binding protein Di-Ras3) [131, 132]. Интересно, что связывание транскрипционного фактора ZBTB7A (zinc finger and BTB domain-containing protein 7A) с промоторным районом гена lncРНК GAS5 ингибирует экспрессию этой lncРНК. В свою очередь экспрессию ZBTB7A может регулировать miR-663a, для которой мРНК этого белка служит мишенью [133]. Таким образом, суммируя данные по lncРНК GAS5 в клетках ОС [131‒133], можно предположить ее участие в двух регуляторных путях: 1) miR-663a/ZBTB7A/GAS5/miR-203/TIMP2 и 2) miR-663a/ZBTB7A/GAS5/miR-221/ARHI, ‒ сопряженность которых отражена на рис. 6. В работе Zhang и соавт. [133] показано, что белок ZBTB7A, ингибируя lncРНК GAS5, защищает клетки ОС от апоптоза, индуцированного стрессом эндоплазматического ретикулума. В целом, результаты, представленные в этих работах по lncРНК GAS5, отражают динамичность регуляторной функции lncРНК в опухолевых клетках, в частности ОС.
Супрессорная lncРНК FER1L4 (Fer-1-Like Protein 4) индуцирует апоптоз и ингибирует экспрессию ЭМП-маркеров и PI3K/AKT-сигналинг в результате прямого связывания и подавления miR-18a-5p и последующей активации SOCS5 (suppressor of cytokine signaling 5). Цепочка прямых ингибирующих взаимодействий FER1L4/ miR-18a-5p/SOCS5 (рис. 6) подтверждена с применением разных методов, включая люциферазный тест [134].
Антисмысловая онкосупрессорная lncРНК CEBPA-AS1 (CCAAT Enhancer-Binding Protein Alpha-AS1) при повышении экспрессии ингибирует сигнальный путь Notch в результате повышения содержания белка NCOR2 (nuclear receptor corepressor 2), что осуществляется через цепочку последовательных прямых ингибирующих взаимодействий: lncРНК CEBPA-AS1/miR-10b-5p/ NCOR2 (рис. 6). Показано, что эктопическая экспрессия CEBPA-AS1 ингибирует пролиферацию, миграцию и индуцирует апоптоз клеток через сигнальный путь Notch [135].
Межгенная lncРНК LINC00588, как показано Zhou и соавт. [136], ингибирует пролиферацию, миграцию, инвазию клеток и ЭМП, но не апоптоз. Установлено, что при этом LINC00588 выступает как сеРНК, ингибирующая miR-1972, что активирует мРНК p53 – прямую мишень miR-1972 ‒ по цепи LINC00588/miR-1972/p53 (рис. 6). Эти прямые взаимодействия подтверждены с применением комплекса методов, включая люциферазный тест [136]. Следует отметить неоднозначные свойства этой lncРНК: ее экспрессия снижена в ОС, но повышена в метастазах, выделенных из ткани легких [136]. Эти факты, по-видимому, отражают вариабельность влияния lncРНК на процессы в клетках ОС.
ПРИМЕРЫ АЛЬТЕРНАТИВНЫХ МЕХАНИЗМОВ
Итак, при рассмотрении опубликованных работ нами выявлено около 30 онкогенных и только 7 супрессорных lncРНК, регулирующих гены и сигнальные пути в клетках ОС по механизму сеРНК. Этот механизм действия lncРНК доказан во многих исследованиях, но обсуждаются и другие возможные механизмы действия lncРНК, например, активация miРНК под действием lncРНК.
Так, онкогенная lncРНК ASBEL (Anti-Sense Transcript of BTG3 (B cell translocation gene 3) locus) повышает экспрессию miR-21, которая, в свою очередь, подавляет свою мишень – мРНК PP2A (protein phosphatase 2A, связанная с негативным контролем клеточного роста и деления) по обычному сценарию для miРНК, взаимодействующих с мРНК гена-мишени [137]. Механизм участия ASBEL в активации miR-21 не выяснен, однако понятно, что lncРНК ASBEL и мРНК PP2A не конкурируют за связывание miR-21 по механизму ceРНК. Замечено, что lncРНК ASBEL, участвуя в подавлении экспрессии PP2A, стимулирует активацию онкогенных сигнальных путей PI3K/ AKT/GSK3β (phosphatidylinositol 3-kinase/protein kinase 3/glycogen synthase kinase-3β) и MEK/ERK (mitogen-activated protein kinase/extracellular regulated protein kinase) и может служить биомаркером и потенциальной терапевтической мишенью при лечении ОС [137].
Аналогично Shen и соавт. показали [138], что активация miR-361-5p происходит под действием lncРНК MEG3 (Maternally Expressed Gene 3), которая напрямую связывает miR-361-5p, не понижая, а повышая ее экспрессию. Молекулярный механизм пока не совсем понятен. Подтверждено (с использованием люциферазного теста) прямое связывание почти по всей длине miR-361-5p с сегментом, расположенным ближе к началу транскрипции lncРНК MEG3. Обнаружено, что изменение уровня MEG3 влияет на таковой для miR-361-5p, а изменение уровня miРНК MEG3 практически не отражается на lncРНК MEG3. Известно, что miR-361-5p ингибирует экспрессию гена FOXM1 (Forkhead box protein M1) ‒ как прямую мишень по классическому сценарию, ‒ связываясь с 3'-UTR транскрибируемой мРНК. Следует отметить, что данные литературы относительно свойств lncРНК MEG3 противоречивы. Так, авторы наблюдали пониженную экспрессию MEG3 и miR-361-5p, причем как в клеточных линиях, так и в 200 образцах ОС, что предполагает онкосупрессорные свойства MEG3, так как MEG3 и miR-361-5p, снижая содержание FOXM1, подавляют пролиферацию и инвазию клеток ОС. В то же время Wang & Kong [66], напротив, обнаружили повышенную экспрессию lncРНК MEG3 в культурах клеток и способность стимулировать прогрессию ОС, что указывает на возможные онкогенные свойства MEG3. По данным этих авторов, lncРНК MEG3 действует по типу ceРНК по оси lncРНК MEG3/miR-127/ZEB1. Вопрос о роли MEG3 как супрессора или онкогена при ОС требует дополнительных исследований. Расхождение данных разных авторов может быть связано с различным уровнем метилирования промоторного района гена, кодирующего lncРНК MEG3, что влияет на уровень ее экспрессии, ‒ как это показано Ding и соавт. [139].
КЛИНИЧЕСКИЙ ПОТЕНЦИАЛ lncРНК ПРИ ОСТЕОСАРКОМЕ
Известно, что молекулы lncРНК достаточно стабильны ‒ их детектируют в различных биологических жидкостях человека, ‒ что повышает их потенциал как перспективных биомаркеров опухолей [140]. Это относится и к диагностике ОС. Возможности применения циркулирующих lncРНК в диагностике ОС суммированы в обзоре Botti и соавт. [141]. Повышенный уровень в сыворотке или плазме lncРНК ATB [67], UCA1 [142], TUG1 [143], MALAT1 [144] позволяет с высокой точностью отличить больных ОС от здоровых. Оценка показателя AUC, характеризующего чувствительность и специфичность маркера, варьирует от 0.83 до 0.92, в то время как для щелочной фосфатазы, традиционно используемой в диагностике опухолей костей, AUC составляет 0.75. Сочетанное применение показателей lncРНК и щелочной фосфатазы позволяет еще больше повысить значение AUC, как по показаниям чувствительности, так и специфичности [144]. Таким образом, использование lncРНК в качестве маркеров позволит повысить эффективность неинвазивной диагностики ОС.
Актуально использование lncРНК и как прогностических маркеров выживаемости, риска осложнений и метастазирования ОС. В проведенном мета-анализе [145] обобщены результаты 20 исследований, охвативших 1 749 пациентов. Сделан вывод, что повышенный уровень тех или иных lncРНК достоверно коррелирует с общей выживаемостью, а также с наличием метастазов. Sun & Zhao [146] попытались идентифицировать наиболее значимые lncРНК, применение которых позволит разделить больных ОС с высоким и низким риском неблагоприятного прогноза. Среди 211 дифференциально экспрессирующихся lncРНК авторы выделили 9, сочетание данных по которым наиболее информативно для прогноза общей выживаемости. Это lncРНК CH17-360D5.2, LINC00987, LINC01526, RP11-15A1.3, RP11-213H15.1, RP11-218F4.1, RP11-242F11.2, RP11-411H5.1 и RP11-834C11.5. Коэффициент, вычисленный на основании их концентраций, дает AUC 0.93 на тестовой выборке и 0.90 при валидации.
lncРНК связаны с тонкой регуляцией биохимических путей в опухолях. Уже сейчас возможно их использование для предсказания чувствительности опухоли к тем или иным видам лечения и эффективности ответа на них. Li и соавт. [35] обнаружили, что уровень lncРНК ODRUL повышен в устойчивых к доксирубицину линиях ОС и у пациентов с плохим ответом на лечение. Нокдаун этой lncРНК снижал экспрессию ABCB1-кассеты и устойчивость клеток к доксирубицину. Терапия цисплатином усиливает экспрессию lncРНК LINC00161, что стимулирует апоптоз за счет ее взаимодействия с miR-645 [35]. Таким образом, увеличение экспрессии lncРНК LINC00161 может быть маркером эффективного ответа на лечение.
Прямое применение аналогов или антагонистов lncРНК в медицине пока что дальняя перспектива, однако в некоторых работах их эффект проверен не только на клеточных линиях, но и на ксенотрансплантатах. Так, в исследованиях in vitro и in vivo показано, что нокдаун онкогенной lncРНК NEAT1 повышает чувствительность к цисплатину [92]. Онкогенная lncРНК OIP5-AS1, связанная с фибронектином-1 через ось OIP5-AS1/miR-200b-3p/FN1, предложена в качестве терапевтической мишени для повышения чувствительности к доксорубицину у пациентов с ОС [104].
ЗАКЛЮЧЕНИЕ
Хотя miРНК связаны с тонкой регуляцией экспрессии генов, изменяя ее не более чем в два раза, консорциум ENCODE по результатам своих исследований поместил miРНК в начало регуляторных сетей, возлагая на них роль “мастер-регулятора” сигнальных каскадов в клетке. Считается, что miРНК способны управлять экспрессией до 50% белоккодирующих генов и регулируют многие процессы жизнедеятельности клеток, в частности раковых: пролиферацию, дифференцировку, апоптоз, адгезию, ЭМП и метастазирование. Однако еще более интригующим стало открытие порядка 100 тыс. молекул lncРНК, обладающих разнообразными, прежде всего регуляторными, функциями. Это приковало внимание исследователей к ним, особенно в последние пять лет.
Механизм действия lncРНК как эндогенных РНК, конкурирующих с мРНК белоккодирующих генов за связывание с miРНК, становится все более популярным и достаточно хорошо доказанным для опухолей разных локализаций, включая ОС. Этот механизм в клетках ОС реализуется не менее 40 lncРНК, среди которых около 30 онкогенных ‒ повышение их экспрессии приводит к активации белков с онкогенным потенциалом и прогрессии заболевания. Онкосупрессорных lncРНК, действующих при ОС по типу ceРНК, выявлено пока в разы меньше. Характерная ли это особенность lncРНК или отражает ориентацию исследователей на поиск молекулярных драйверов онкогенеза, пока непонятно.
В отличие от доказанных примеров участия lncРНК в патогенезе ОС по механизму ceРНК известны случаи, когда lncРНК участвуют в регуляции мРНК-мишеней не по классическому сценарию – при посредничестве miРНК, активируемой под действием lncРНК. Так, онкогенная lncРНК ASBEL активирует miR-21, что ведет к снижению мРНК белка PP2A и активирует онкогенные сигнальные пути PI3K/AKT/GSK3β и MEK/ERK. Еще один пример активирующего влияния lncРНК на miРНК ‒ прямое связывание lncРНК MEG3 с miR-361-5p и ее активация, что приводит к снижению содержания FOXM1 и подавлению пролиферации и инвазии клеток ОС. Разумеется, эти примеры нельзя отнести к классическому механизму действия ceРНК.
В некоторых работах вообще не рассматривается участие miРНК как посредника между lncРНК и мРНК. Например, показано влияние lncРНК TUG1 на прогрессию ОС через воздействие на ген RUNX2 (runt-related transcription factor 2), однако возможное посредническое участие каких-либо miРНК в этих регуляторных функциях не исследовано [91]. Аналогично показана роль lncРНК SNHG20 в активации ЭМП за счет изменения уровней экспрессии ЭМП-маркеров (ZEB1, ZEB2, E-кадгерин и т.п.), хотя прямое связывание lncРНК SNHG20 с мРНК этих белков не подтверждено [147].
Вызывают интерес широкие возможности влияния lncРНК на обратимость и динамику процессов в клетке по механизму конкурентных взаимодействий и с учетом числа (до 100 тыс.) уже открытых lncРНК. Неоднозначность действия miРНК общеизвестна ‒ в зависимости от клеточного контекста они могут проявлять и онкогенные, и супрессорные свойства [148]. Двойственность поведения обнаружена и для некоторых lncРНК, как в случае XIST, MEG3 и LINC00588, что расширяет наши представления о вариабельности и динамичности их влияния на клеточные процессы.
На примере ОС обнаружен целый ряд lncРНК (MALAT1, PVT1, члены семейства SNHG, TUG1, XIST, NEAT1, HOXA11-AS и др.) с множественными регуляторными осями, что подтверждает наличие на этих lncРНК множества участков связывания разных miРНК и раскрывает разнообразные функции lncРНК при ОС.
В регуляторные оси lncРНК/miРНК/мРНК, исследованные в клетках ОС, вовлечены мРНК многих транскрипционных факторов и белков, участвующих в ремоделировании хроматина, преобразованиях цитоскелета и взаимодействии клетки с внеклеточным матриксом, а также мастер-регуляторы различных процессов, в том числе клеточного цикла (согласно базам данных Gene Cards, KEGG и Panther [43, 149, 150]). Из этого можно сделать вывод, что эти lncРНК играют ключевую роль в патогенезе ОС.
Дифференциальная диагностика опухолей костей до сих пор недостаточно разработана. Во многих случаях диагноз установить не удается даже при привлечении ведущих морфологов. Кроме того, разработка новых методов диагностики остается важнейшей задачей детской онкологии. За последнее время созданы наборы lncРНК для неинвазивной “жидкостной” диагностики ОС, позволяющие отличить больных от здоровых с высокой точностью (AUC 0.9). Показан потенциал ряда lncРНК (ODRUL, LINC00161, NEAT1, OIP5-AS1) как маркеров лекарственной устойчивости – основной причины прогрессии и рецидивов у пациентов с ОС.
Таким образом, поиск и идентификация новых lncРНК и исследование их функций в регуляции генов позволит нам лучше понять механизмы развития и прогрессии ОС – самого распространенного и наиболее агрессивного злокачественного заболевания костей у детей в пубертатном периоде. На основании этих знаний уже предложены новые диагностические и прогностические биологические маркеры, потенциальные терапевтические мишени и маркеры ответа на химиотерапию.
Взаимодействия между разными видами эндогенных РНК, включая мРНК, miРНК и lncРНК, по механизму конкуренции, представляет собой новую форму регуляции генов, которая играет важную роль в патогенезе ОС.
Работа выполнена при поддержке Минобрнауки России на выполнение государственного задания учреждения и Минздрава РФ в 2020 году.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
ENCODE Consortium (2012) Landscape of transcription in human cells. Nature. 489(7414), 101–108. https://doi.org/10.1038/nature11233
Diederichs S., Bartsch L., Berkmann J.C., Fröse K., Heitmann J., Hoppe C., Iggena D., Jazmati D., Karschnia P., Linsenmeier M., Maulhardt T., Möhrmann L., Morstein J., Paffenholz S.V., Röpenack P., Rückert T., Sandig L., Schell M., Steinmann A., Voss G., Wasmuth J., Weinberger M.E., Wullenkord R. (2016) The dark matter of the cancer genome: aberrations in regulatory elements, untranslated regions, splice sites, non-coding RNA and synonymous mutations. EMBO Mol. Med. 8(5), 442–457. https://doi.org/10.15252/emmm.201506055
Yang D., Sun L., Li Z., Gao P. (2016) Noncoding RNAs in regulation of cancer metabolic reprogramming. Adv. Exp. Med. Biol. 927, 191–215. https://doi.org/10.1007/978-981-10-1498-7_7
Baek D., Villén J., Shin C., Camargo F.D., Gygi S.P., Bartel D.P. (2008) The impact of microRNAs on protein output. Nature. 455(7209), 64–71. https://doi.org/10.1038/nature07242
ENCODE Consortium (2012) Architecture of the human regulatory network derived from ENCODE data. Nature. 489(7414), 91–100. https://doi.org/10.1038/nature11245
Chan S.H., Wang L.H. (2015) Regulation of cancer metastasis by microRNAs. J. Biomed. Sci. 22, 9. https://doi.org/10.1186/s12929-015-0113-7
Логинов В.И., Рыков С.В., Фридман М.В., Брага Э.А. (2015) Метилирование генов микроРНК и онкогенез. Биохимия. 80(2), 184–203.
Sanchez Calle A., Kawamura Y., Yamamoto Y., Takeshita F., Ochiya T. (2018) Emerging roles of long non-co-ding RNA in cancer. Cancer Sci. 109(7), 2093–2100. https://doi.org/10.1111/cas.13642
Leygue E. (2007) Steroid receptor RNA activator (SRA1): unusual bifaceted gene products with suspected relevance to breast cancer. Nucl. Recept. Signal. 5, e006. https://doi.org/10.1621/nrs.05006
Quinn J.J., Chang H.Y. (2016) Unique features of long non-coding RNA biogenesis and function. Nat. Rev. Genet. 17(1), 47–62. https://doi.org/10.1038/nrg.2015.10
Буре И.В., Кузнецова Е.Б., Залетаев Д.В. (2018) Длинные некодирующие РНК и их роль в онкогенезе. Молекуляр. биология. 52(6), 907–920.
Salmena L., Poliseno L., Tay Y., Kats L., Pandolfi P.P. (2011) A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language? Cell. 146(3), 353–358. https://doi.org/10.1016/j.cell.2011.07.014
Tian J., Wang Y., Zhang X., Ren Q., Li R., Huang Y., Lu H., Chen J. (2017) Calycosin inhibits the in vitro and in vivo growth of breast cancer cells through WDR7-7-GPR30 signaling. J. Exp. Clin. Cancer Res. 36(1), 153. https://doi.org/10.1186/s13046-017-0625-y
Bhan A., Soleimani M., Mandal S.S. (2017) Long noncoding RNA and cancer: a new paradigm. Cancer Res. 77(15), 3965–3981. https://doi.org/10.1158/0008-5472.CAN-16-2634
Patop I.L., Kadener S. (2018) circRNAs in cancer. Curr. Opin. Genet. Dev. 48, 121–127. https://doi.org/10.1016/j.gde.2017.11.007
Chan J.J., Tay Y. (2018) Noncoding RNA: RNA regulatory networks in cancer. Int. J. Mol. Sci. 19(5), E1310. https://doi.org/10.3390/ijms19051310
Friebele J.C., Peck J., Pan X., Abdel-Rasoul M., Mayerson J.L. (2015) Osteosarcoma: a meta-analysis and review of the literature. Am. J. Orthop. (Belle Mead NJ). 44(12), 547–553.
Bahl A., George P., Bhattacharyya T., Ghoshal S., Bakshi J., Das A.J. (2015) Osteosarcoma of larynx: a rare case report with review of literature. Cancer Res. Ther. 11(4), 1038. https://doi.org/10.4103/0973-1482.139274
Siegel R.L., Miller K.D., Jemal A. (2016) Cancer statistics, 2016. CA Cancer J. Clin. 66(1), 7–30. https://doi.org/10.3322/caac.21332
Кушлинский Н.Е., Фридман М.В., Брага Э.А. (2016) Молекулярные механизмы и микроРНК в патогенезе остеосаркомы. Биохимия. 81(4), 448–464.
He X., Gao Z., Xu H., Zhang Z., Fu P. (2017) A meta-analysis of randomized control trials of surgical methods with osteosarcoma outcomes. J. Orthop. Surg. Res. 12(1), 5. https://doi.org/10.1186/s13018-016-0500-0
Zhu L., McManus M.M., Hughes D.P. (2013) Understanding the biology of bone sarcoma from early initiating events through late events in metastasis and disease progression. Front. Oncol. 3, 230. https://doi.org/10.3389/fonc.2013.00230
Lin Y.H., Jewell B.E., Gingold J., Lu L., Zhao R., Wang L.L., Lee D.F. (2017) Osteosarcoma: Molecular pathogenesis and iPSC modeling. Trends Mol. Med. 23, 737–755. https://doi.org/10.1016/j.molmed.2017.06.004
Palmini G., Marini F., Brandi M.L. (2017) What is new in the miRNA world regarding osteosarcoma and chondrosarcoma? Molecules. 22(3), 417. https://doi.org/10.3390/molecules22030417
Pan Y., Lu L., Chen J., Zhong Y., Dai Z. (2018) Identification of potential crucial genes and construction of microRNA-mRNA negative regulatory networks in osteosarcoma. Hereditas. 155, 21. https://doi.org/10.1186/s41065-018-0061-9
Liu Q., Wang Z., Zhou X., Tang M., Tan W., Sun T., Deng Y. (2019) miR-342-5p inhibits osteosarcoma cell growth, migration, invasion, and sensitivity to doxorubicin through targeting Wnt7b. Cell Cycle. 18(23), 3325–3336. https://doi.org/10.1080/15384101.2019.1676087
Wang J., Liu S., Shi J, Li J., Wang S., Liu H., Zhao S., Duan K., Pan X., Yi Z. (2019) The role of miRNA in the diagnosis, prognosis, and treatment of osteosarcoma. Cancer Biother. Radiopharm. 34(10), 605-613. https://doi.org/10.1089/cbr.2019.2939
Sasaki R., Osaki M., Okada F. (2019) MicroRNA-based diagnosis and treatment of metastatic human osteosarcoma. Cancers (Basel). 11(4), 553. https://doi.org/10.3390/cancers11040553
Viera G.M., Salomao K.B., de Sousa G.R., Baroni M., Delsin L.E.A., Pezuk J.A., Brassesco M.S. (2019) miRNA signatures in childhood sarcomas and their clinical implications. Clin. Transl. Oncol. 21(12), 1583–1623. https://doi.org/10.1007/s12094-019-02104-z
Li Z., Yu X., Shen J. (2016) Long non-coding RNAs: emerging players in osteosarcoma. Tumour Biol. 37(3), 2811–2816. https://doi.org/10.1007/s13277-015-4749-4
Chen R., Wang G., Zheng Y., Hua Y., Cai Z. (2017) Long non-coding RNAs in osteosarcoma. Oncotarget. 8(12), 20462–20475. https://doi.org/10.18632/oncotarget.14726
Smolle M.A., Pichler M. (2018) The role of long non-coding RNAs in osteosarcoma. Noncoding RNA. 4(1), 7. https://doi.org/10.3390/ncrna4010007
Wang J.Y., Yang Y., Ma Y., Wang F., Xue A., Zhu J., Yang H., Chen Q., Chen M., Ye L., Wu H., Zhang Q. (2020) Potential regulatory role of lncRNA-miRNA-mRNA axis in osteosarcoma. Biomed. Pharmacother. 121, 109627. https://doi.org/10.1016/j.biopha.2019.109627
Yang Z., Li X., Yang Y., He Z., Qu X., Zhang Y. (2016) Long noncoding RNAs in the progression, metastasis, and prognosis of osteosarcoma. Cell Death Dis. 7(9), 2389. https://doi.org/10.1038/cddis.2016.272
Li Z., Dou P., Liu T., He S. (2017) Application of long noncoding RNAs in osteosarcoma: biomarkers and therapeutic targets. Cell Physiol. Biochem. 42(4), 1407–1419. https://doi.org/10.1159/000479205
Wang C., Jing J., Cheng L. (2018) Emerging roles of non-coding RNAs in the pathogenesis, diagnosis and prognosis of osteosarcoma. Invest. New Drugs. 36(6), 1116–1132. https://doi.org/10.1007/s10637-018-0624-7
Guo W., Yu Q., Zhang M., Li F., Liu Y., Jiang W., Jiang H., Li H. (2019) Long intergenic non-protein coding RNA 511 promotes the progression of osteosarcoma cells through sponging microRNA 618 to upregulate the expression of maelstrom. Aging (Albany NY). 11(15), 5351–5367. https://doi.org/10.18632/aging.102109
McManus M.M., Weiss K.R., Hughes D.P. (2014) Understanding the role of Notch in osteosarcoma. Adv. Exp. Med. Biol. 804, 67–92. https://doi.org/10.1007/978-3-319-04843-7_4
Wang Y.M., Wang W., Qiu E.D. (2017) Osteosarcoma cells induce differentiation of mesenchymal stem cells into cancer associated fibroblasts through Notch and Akt signaling pathway. Int. J. Clin. Exp. Pathol. 10(8), 8479–8486.
Yu L., Xia K., Gao T., Chen J., Zhang Z., Sun X., Simões B.M., Eyre R., Fan Z., Guo W., Clarke R.B. (2019) The Notch pathway promotes osteosarcoma progression through activation of ephrin reverse signaling. Mol. Cancer Res. 17(12), 2383–2394. https://doi.org/10.1158/1541-7786
Danieau G., Morice S., Rédini F., Verrecchia F., Royer B.B. (2019) New insights about the Wnt/β-catenin signaling pathway in primary bone tumors and their microenvironment: a promising target to develop therapeutic strategies? Int. J. Mol. Sci. 20(15), 3751. https://doi.org/10.3390/ijms20153751
Guan H., Tan P., Xie L., Mi B., Fang Z., Li J., Yue J., Liao H., Li F. (2015) FOXO1 inhibits osteosarcoma oncogenesis via Wnt/β-catenin pathway suppression. Oncogenesis. 4, 166. https://doi.org/10.1038/oncsis.2015.25
Kanehisa M., Sato Y., Furumichi M., Morishima K., Tanabe M. (2019) New approach for understanding genome variations in KEGG. Nucleic Acids Res. 47(D1), 590-595. https://doi.org/10.1093/nar/gky962
Yang J., Li Y.H., He M.T., Qiao J.F., Sang Y., Cheang L.H., Gomes F.C., Hu Y., Li Z.Y., Liu N., Zhang H.T., Zha Z.G. (2020) HSP90 regulates osteosarcoma cell apoptosis by targeting the p53/TCF-1-mediated transcriptional network. J. Cell Physiol. 235(4), 3894–3904. https://doi.org/10.1002/jcp.29283
Velletri T., Xie N., Wang Y., Huang Y., Yang Q., Chen X., Chen Q., Shou P., Gan Y., Cao G., Melino G., Shi Y. (2016) P53 functional abnormality in mesenchymal stem cells promotes osteosarcoma development. Cell Death Dis. 7, 2015. https://doi.org/10.1038/cddis.2015.367
Han Y., Kim Y.M., Kim H.S., Lee K.Y. (2017) Melatonin promotes osteoblast differentiation by regulating Osterix protein stability and expression. Sci. Rep. 7, 5716. https://doi.org/10.1038/s41598-017-06304-x
Komori T. (2018) Runx2, an inducer of osteoblast and chondrocyte differentiation. Histochem. Cell Biol. 149, 313-323. https://doi.org/10.1007/s00418-018-1640-6
Zhou W., Hao M., Du X., Chen K., Wang G., Yang J. (2014) Advances in targeted therapy for osteosarcoma. Discov. Med. 17(96), 301–307.
Li Y.S., Deng Z.H., Zeng C., Lei G.H. (2016) JNK pathway in osteosarcoma: pathogenesis and therapeutics. J. Recept. Signal Transduct. Res. 36(5), 465–470. https://doi.org/10.3109/10799893.2015.1122045
Yu G.H., Li A.M., Li X., Yang Z., Peng H. (2017) Bispecific antibody suppresses osteosarcoma aggressiveness through regulation of NF-κB signaling pathway. Tumour Biol. 39(6), https://doi.org/10.1177/1010428317705572
de Azevedo J.W.V., de Medeiros Fernandes T.A.A., Fernandes J.V. Jr., de Azevedo J.C.V., Lanza D.C.F., Bezerra C.M., Andrade V.S., de Araújo J.M.G., Fernandes J.V. (2020) Biology and pathogenesis of human osteosarcoma. Oncol. Lett. 19(2), 1099–1116. https://doi.org/10.3892/ol.2019.11229
Si Z., Hu K. (2020) Identification of osteosarcoma driver genes using a network method. Oncol. Lett. 19(2), 1215–1222. https://doi.org/10.3892/ol.2019.11212
Pan B.L., Wu L., Pan L., Yang Y.X., Li H.H., Dai Y.J., He Z.Q., Tan L., Huang Y.G., Tong Z.W., Liao J.L. (2018) Up-regulation of microRNA-340 promotes osteosarcoma cell apoptosis while suppressing proliferation, migration, and invasion by inactivating the CTNNB1-mediated Notch signaling pathway. Biosci. Rep. 38(4), BSR20171615. https://doi.org/10.1042/BSR20171615
Zhou S., Yu L., Xiong M., Dai G. (2018) LncRNA SNHG12 promotes tumorigenesis and metastasis in osteosarcoma by upregulating Notch2 by sponging miR-195-5p. Biochem. Biophys. Res. Commun. 495(2), 1822–1832. https://doi.org/10.1016/j.bbrc.2017.12.047
Deng Y., Zhao F., Zhang Z., Sun F., Wang M. (2018) Long noncoding RNA SNHG7 promotes the tumor growth and epithelial-to-mesenchymal transition via regulation of miR-34a signals in osteosarcoma. Cancer Biother. Radiopharm. 33(9), 365–372. https://doi.org/10.1089/cbr.2018.2503
Mayer I.A., Arteaga C.L. (2016) The PI3K/AKT pathway as a target for cancer treatment. Annu. Rev. Med. 67, 11–28. https://doi.org/10.1146/annurev-med-062913-051343
Méndez-Pertuz M., Martínez P., Blanco-Aparicio C., Gómez-Casero E., Belen García A., Martínez-Torrecuadrada J., Palafox M., Cortés J., Serra V., Pastor J., Blasco M.A. (2017) Modulation of telomere protection by the PI3K/AKT pathway. Nat. Commun. 8(1), 1278. https://doi.org/10.1038/s41467-017-01329-2
Zhang Y., Dai Q., Zeng F., Liu H. (2018) MALAT1 promotes the proliferation and metastasis of osteosarcoma cells by activating the Rac1/JNK pathway via targeting miR-509. Oncol. Res. https://doi.org/10.3727/096504017X14957939026111
Li C., Wang F., Wei B., Wang L., Kong D. (2019) LncRNA AWPPH promotes osteosarcoma progression via activation of Wnt/beta-catenin pathway through modulating miR-93-3p/FZD7 axis. Biochem. Biophys. Res. Commun. 514(3), 1017–1022. https://doi.org/10.1016/j.bbrc.2019.04.203
Zhao J., Cheng L. (2017) Long non-coding RNA CCAT1/miR-148a axis promotes osteosarcoma proliferation and migration through regulating PIK3IP1. Acta Biochim. Biophys. Sin. (Shanghai). 49(6), 503–512. https://doi.org/10.1093/abbs/gmx041
Jiang N., Wang X., Xie X., Liao Y., Liu N., Liu J., Miao N., Shen J., Peng T. (2017) lncRNA DANCR promotes tumor progression and cancer stemness features in osteosarcoma by upregulating AXL via miR-33a-5p inhibition. J. Cell. Biochem. 405, 46–55. https://doi.org/10.1016/j.canlet.2017.06.009
Kong D., Wang Y. (2018) Knockdown of lncRNA HULC inhibits proliferation, migration, invasion, and promotes apoptosis by sponging miR-122 in osteosarcoma. J. Cell. Biochem. 119(1), 1050–1061. https://doi.org/10.1002/jcb.26273
Kumar A., Singh U. K., Kini S.G., Garg V., Agrawal S., Tomar P.K., Pathak P., Chaudhary A., Gupta P., Malik A. (2015) JNK pathway signaling: a novel and smarter therapeutic targets for various biological diseases. Future Med. Chem. 7(15), 2065–2086. https://doi.org/10.4155/fmc.15.132
Lin C.H., Ji T., Chen C.F., Hoang B.H. (2014) Wnt signaling in osteosarcoma. Adv. Exp. Med. Biol. 804, 33–45. https://doi.org/10.1007/978-3-319-04843-7_2
Xia B., Wang L., Feng L., Tian B., Tan Y., Du B. (2018) Knockdown of long non-coding RNA CAT104 inhibits the proliferation, migration and invasion of human osteosarcoma cells by regulating microRNA381. Oncol. Res. 27(1), 89–98. https://doi.org/10.3727/096504018X15199511344806
Wang Y., Kong D. (2018) Knockdown of lncRNA MEG3 inhibits viability, migration, and invasion and promotes apoptosis by sponging miR-127 in osteosarcoma cell. J. Cell. Biochem. 119(1), 669–679. https://doi.org/10.1002/jcb.26230
Han F., Wang C., Wang Y., Zhang L. (2017) Long noncoding RNA ATB promotes osteosarcoma cell proliferation, migration and invasion by suppressing miR-200s. Am. J. Cancer Res. 7(4), 770–783.
Jia D., Niu Y., Li D., Liu Z. (2018) lncRNA C2dat1 promotes cell proliferation, migration, and invasion by targeting miR-34a-5p in osteosarcoma cells. Oncol. Res. 26(5), 753–764. https://doi.org/10.3727/096504017X15024946480113
Jiang Z., Jiang C., Fang J. (2018) Up-regulated lnc-SNHG1 contributes to osteosarcoma progression through sequestration of miR-577 and activation of WNT2B/Wnt/β-catenin pathway. Biochem. Biophys. Res. Commun. 495(1), 238–245. https://doi.org/10.1016/j.bbrc.2017.11.012
Wang J., Cao L., Wu J., Wang Q. (2018) Long non-coding RNA SNHG1 regulates NOB1 expression by sponging miR-326 and promotes tumorigenesis in osteosarcoma. Int. J. Oncol. 52(1), 77–88. https://doi.org/10.3892/ijo.2017.4187
Deng R., Zhang J., Chen J. (2019) lncRNA SNHG1 negatively regulates miRNA‑101‑3p to enhance the expression of ROCK1 and promote cell proliferation, migration and invasion in osteosarcoma. Int. J. Mol. Med. 43(3), 1157–1166. https://doi.org/10.3892/ijmm.2018.4039
Zheng S., Jiang F., Ge D., Tang J., Chen H., Yang J., Yao Y., Yan J., Qiu J., Yin Z., Ni Y., Zhao L., Chen X., Li H., Yang L. (2019) LncRNA SNHG3/miRNA-151a-3p/RAB22A axis regulates invasion and migration of osteosarcoma. Biomed. Pharmacother. 112, 108695. https://doi.org/10.1016/j.biopha.2019.108695
Chen J., Wu Z., Zhang Y. (2019) LncRNA SNHG3 promotes cell growth by sponging miR-196a-5p and indicates the poor survival in osteosarcoma. Int. J. Immunopathol. Pharmacol. 33, 2058738418820743. https://doi.org/10.1177/2058738418820743
Ju C., Zhou R., Sun J., Zhang F., Tang X., Chen K.K., Zhao J., Lan X., Lin S., Zhang Z., Lv X.B. (2018) LncRNA SNHG5 promotes the progression of osteosarcoma by sponging the miR-212-3p/SGK3 axis. Cancer Cell Int. 18, 141. https://doi.org/10.1186/s12935-018-0641-9
Wang Z., Wang Z., Liu J., Yang H. (2018) Long non-coding RNA SNHG5 sponges miR-26a to promote the tumorigenesis of osteosarcoma by targeting ROCK1. Biomed. Pharmacother. 107, 598–605. https://doi.org/10.1016/j.biopha.2018.08.025
Xu N., Xu J., Zuo Z., Liu Y., Yan F., Han C. (2020) Downregulation of lncRNA SNHG12 reversed IGF1R-induced osteosarcoma metastasis and proliferation by targeting miR-195-5p. Gene. 726, 144145. https://doi.org/10.1016/j.gene.2019.144145
Zhu C., Cheng D., Qiu X., Zhuang M., Liu Z. (2018) Long noncoding RNA SNHG16 promotes cell proliferation by sponging microRNA-205 and upregulating ZEB1 expression in osteosarcoma. Cell Physiol. Biochem. 51(1), 429–440. https://doi.org/10.1159/000495239
Wang X., Hu K., Chao Y., Wang L. (2019) LncRNA SNHG16 promotes proliferation, migration and invasion of osteosarcoma cells by targeting miR-1301/BCL9 axis. Biomed. Pharmacother. 114, 108798. https://doi.org/10.1016/j.biopha.2019.108798
Wang W., Luo P., Guo W., Shi Y., Xu D., Zheng H., Jia L. (2018) LncRNA SNHG20 knockdown suppresses the osteosarcoma tumorigenesis through the mitochondrial a poptosis pathway by miR-139/RUNX2 axis. Biochem. Biophys. Res. Commun. 503(3), 1927–1933. https://doi.org/10.1016/j.bbrc.2018.07.137
Zhou Q., Chen F., Zhao J., Li B., Liang Y., Pan W., Zhang S., Wang X., Zheng D. (2016) Long non-co-ding RNA PVT1 promotes osteosarcoma development by acting as a molecular sponge to regulate miR-195. Oncotarget. 7(50), 82620–82633. https://doi.org/10.18632/oncotarget.13012
Song J., Wu X., Liu F., Li M., Sun Y., Wang Y., Wang C., Zhu K., Jia X., Wang B., Ma X. (2017) Long non-co-ding RNA PVT1 promotes glycolysis and tumor progression by regulating miR-497/HK2 axis in osteosarcoma. Biochem. Biophys. Res. Commun. 490(2), 217–224. https://doi.org/10.1016/j.bbrc.2017.06.024
Zhao W., Qin P., Zhang D., Cui X., Gao J., Yu Z., Chai Y., Wang J., Li J. (2019) Long non-coding RNA PVT1 encapsulated in bone marrow mesenchymal stem cell-derived exosomes promotes osteosarcoma growth and metastasis by stabilizing ERG and sponging miR-183-5p. Aging (Albany NY). 11(21), 9581–9596. https://doi.org/10.18632/aging.102406
Xie C.H., Cao Y.M., Huang Y., Shi Q.W., Guo J.H., Fan Z.W., Li J.G., Chen B.W., Wu B.Y. (2016) Long non-coding RNA TUG1 contributes to tumorigenesis of human osteosarcoma by sponging miR-9-5p and regulating POU2F1 expression. Tumour Biol. 37(11), 15031–15041. https://doi.org/10.1007/s13277-016-5391-5
Cao J., Han X., Qi X., Jin X., Li X. (2017) TUG1 promotes osteosarcoma tumorigenesis by upregulating EZH2 expression via miR-144-3p. Int. J. Oncol. 51(4), 1115–1123. https://doi.org/10.3892/ijo.2017.4110
Wang Y., Yang T., Zhang Z., Lu M., Zhao W., Zeng X., Zhang W. (2017) Long non-coding RNA TUG1 promotes migration and invasion by acting as a ceRNA of miR-335-5p in osteosarcoma cells. Cancer Sci. 108(5), 859–867. https://doi.org/10.1111/cas.13201
Li G., Liu K., Du X. (2018) Long non-coding RNA TUG1 promotes proliferation and inhibits apoptosis of osteosarcoma cells by sponging miR-132-3p and upregulating SOX4 expression. Yonsei Med. J. 59(2), 226–235. https://doi.org/10.3349/ymj.2018.59.2.226
Xie C., Chen B., Wu B., Guo J., Cao Y. (2018) LncRNA TUG1 promotes cell proliferation and suppresses apoptosis in osteosarcoma by regulating miR-212-3p/FOXA1 axis. Biomed. Pharmacother. 97, 1645–1653. https://doi.org/10.1016/j.biopha.2017.12.004
Yu X., Hu L., Li S., Shen J., Wang D., Xu R., Yang H. (2019) Long non-coding RNA Taurine upregulated gene 1 promotes osteosarcoma cell metastasis by mediating HIF-1α via miR-143-5p. Cell Death Dis. 10(4), 280. https://doi.org/10.1038/s41419-019-1509-1
Yang G., Zhang C., Wang N., Chen J. (2019) miR-425-5p decreases LncRNA MALAT1 and TUG1 expressions and suppresses tumorigenesis inosteosarcoma via Wnt/β-catenin signaling pathway. Int. J. Biochem. Cell Biol. 111, 42–51. https://doi.org/10.1016/j.biocel.2019.04.004
Zhao Z.Y., Zhao Y.C., Liu W. (2019) Long non-co-ding RNA TUG1 regulates the progression and metastasis of osteosarcoma cells via miR-140-5p/PFN2 axis. Eur. Rev. Med. Pharmacol. Sci. 23(22), 9781–9792. https://doi.org/10.26355/eurrev_201911_19541
Sheng K., Li Y. (2019) LncRNA TUG1 promotes the development of osteosarcoma through RUNX2. Exp. Ther. Med. 18(4), 3002–3008. https://doi.org/10.3892/etm.2019.7880
Hu Y., Yang Q., Wang L., Wang S., Sun F., Xu D., Jiang J. (2018) Knockdown of the oncogene lncRNA NEAT1 restores the availability of miR-34c and improves the sensitivity to cisplatin in osteosarcoma. Biosci. Rep. 38(3), BSR20180375. https://doi.org/10.1042/BSR20180375
Zhang L., Lu X.Q., Zhou X.Q., Liu Q.B., Chen L., Cai F. (2019) NEAT1 induces osteosarcoma development by modulating the miR-339-5p/TGF-β1 pathway. J. Cell Physiol. 234(4), 5097–5105. https://doi.org/10.1002/jcp.27313
Guan H., Shang G., Cui Y., Liu J., Sun X., Cao W., Wang Y., Li Y. (2019) Long noncoding RNA APTR contributes to osteosarcoma progression through repression of miR-132-3p and upregulation of yes-associated protein 1. J. Cell Physiol. 234(6), 8998–9007. https://doi.org/10.1002/jcp.27572
Gu Z., Hou Z., Zheng L., Wang X., Wu L., Zhang C. (2018) LncRNA DICER1-AS1 promotes the prolife-ration, invasion and autophagy of osteosarcoma cells via miR-30b/ATG5. Biomed. Pharmacother. 104, 110–118. https://doi.org/10.1016/j.biopha.2018.04.193
Gu Z., Hou Z., Zheng L., Wang X., Wu L., Zhang C. (2018) Long noncoding RNA LINC00858 promotes osteosarcoma through regulating miR-139-CDK14 axis. Biochem. Biophys. Res. Commun. 503(2), 1134–1140. https://doi.org/10.1016/j.bbrc.2018.06.131
Cui M., Wang J., Li Q., Zhang J., Jia J., Zhan X. (2017) Long non-coding RNA HOXA11-AS functions as a competing endogenous RNA to regulate ROCK1 expression by sponging miR-124-3p in osteosarcoma. Biomed. Pharmacother. 92, 437–444. https://doi.org/10.1016/j.biopha.2017.05.081
Cao K., Fang Y., Wang H., Jiang Z., Guo L., Hu Y. (2019) The lncRNA HOXA11-AS regulates Rab3D expression by sponging miR-125a-5p promoting metastasis of osteosarcoma. Cancer Manag. Res. 11, 4505–4518. https://doi.org/10.2147/CMAR.S196025
Zhou C., Xu J., Lin J., Lin R., Chen K., Kong J., Shui X. (2018) Long non-coding RNA FEZF1-AS1 promotes osteosarcoma progression by regulating miR-4443/NUPR1 axis. Oncol. Res. https://doi.org/10.3727/096504018X15188367859402
Yang G., Song R., Wang L., Wu X. (2018) Knockdown of long non-coding RNA TP73-AS1 inhibits osteosarcoma cell proliferation and invasion through sponging miR-142. Biomed. Pharmacother. 103, 1238–1245. https://doi.org/10.1016/j.biopha.2018.04.146
Zhu K.P., Ma X.L., Zhang C.L. (2017) LncRNA ODRUL contributes to osteosarcoma progression through the miR-3182/MMP2 axis. Mol. Ther. 25(10), 2383–2393. https://doi.org/10.1016/j.ymthe.2017.06.027
Li Q., Xing W., Gong X., Wang Y. (2018) Long non-coding RNA urothelial carcinoma associated 1 promotes proliferation, migration and invasion of osteosarcoma cells by regulating microRNA-182. Cell Physiol. Biochem. 51(3), 1149–1163. https://doi.org/10.1159/000495493
Dai J., Xu L., Hu X., Han G., Jiang H., Sun H., Zhu G., Tang X. (2018) Long noncoding RNA OIP5-AS1 accelerates CDK14 expression to promote osteosarcoma tumorigenesis via targeting miR-223. Biomed. Pharmacother. 106:1441–1447. https://doi.org/10.1016/j.biopha.2018.07.109
Kun-Peng Z., Chun-Lin Z., Xiao-Long M., Lei Z. (2019) Fibronectin-1 modulated by the long noncoding RNA OIP5-AS1/miR-200b-3p axis contributes to doxorubicin resistance of osteosarcoma cells. J. Cell Physiol. 234(5), 6927–6939. https://doi.org/10.1002/jcp.27435
Lv G.Y., Miao J., Zhang X.L. (2017) Long non-coding RNA XIST promotes osteosarcoma progression by targeting ras-related protein RAP2B via miR-320b. Oncol. Res. 26(6), 837–846. https://doi.org/10.3727/096504017X14920318811721
Wu D., Nie X., Ma C., Liu X., Liang X., An Y., Zhao B., Wu X. (2017) RSF1 functions as an oncogene in osteosarcoma and is regulated by XIST/miR-193a-3p axis. Biomed. Pharmacother. 95, 207–214. https://doi.org/10.1016/j.biopha.2017.08.068
Yang C., Wu K., Wang S., Wei G. (2018) Long non-coding RNA XIST promotes osteosarcoma progression by targeting YAP via miR-195-5p. J. Cell Biochem. 119(7), 5646–5656. https://doi.org/10.1002/jcb.26743
Sun X., Wei B., Peng Z.H., Fu Q.L., Wang C.J., Zheng J.C., Sun J.C. (2019) Knockdown of lncRNA XIST suppresses osteosarcoma progression by inactivating AKT/mTOR signaling pathway by sponging miR-375-3p. Int. J. Clin. Exp. Pathol. 12(5), 1507–1517.
Li H., Cui J., Xu B., He S., Yang H., Liu L. (2019) Long non-coding RNA XIST serves an oncogenic role in osteosarcoma by sponging miR-137. Exp. Ther. Med. 17(1), 730–738. https://doi.org/10.3892/etm.2018.7032
Zhang R., Xia T. (2017) Long non-coding RNA XIST regulates PDCD4 expression by interacting with miR-21-5p and inhibits osteosarcoma cell growth and metastasis. Int. J. Oncol. 51(5), 1460–1470. https://doi.org/10.3892/ijo.2017.4127
Li Q., Pan X., Wang X., Jiao X., Zheng J., Li Z., Huo Y. (2017) Long noncoding RNA MALAT1 promotes cell proliferation through suppressing miR-205 and promoting SMAD4 expression in osteosarcoma. Oncotarget. 8(63), 106648–106660. https://doi.org/10.18632/oncotarget.20678
Wang Y., Zhang Y., Yang T., Zhao W., Wang N., Li P., Zeng X., Zhang W. (2017) Long non-coding RNA MALAT1 for promoting metastasis and proliferation by acting as a ceRNA of miR-144-3p in osteosarcoma cells. Oncotarget. 8(35), 59417–59434. https://doi.org/10.18632/oncotarget.19727
Liu K., Huang J., Ni J., Song D., Ding M., Wang J., Huang X., Li W. (2017) MALAT1 promotes osteosarcoma development by regulation of HMGB1 via miR-142-3p and miR-129-5p. Cell Cycle. 16(6), 578–587. https://doi.org/10.1080/15384101.2017.1288324
Ren D., Zheng H., Fei S., Zhao J.L. (2018) MALAT1 induces osteosarcoma progression by targeting miR-206/CDK9 axis. J. Cell Physiol. 234(1), 950–957. https://doi.org/10.1002/jcp.26923
Chen Y., Huang W., Sun W., Zheng B., Wang C., Luo Z., Wang J., Yan W. (2018) LncRNA MALAT1 promotes cancer metastasis in osteosarcoma via activation of the PI3K-Akt signaling pathway. Cell Physiol. Biochem. 51(3), 1313–1326. https://doi.org/10.1159/000495550
Sun Y., Qin B. (2018) Long noncoding RNA MALAT1 regulates HDAC4-mediated proliferation and apoptosis via decoying of miR-140-5p in osteosarcoma cells. Cancer Med. 7(9), 4584–4597. https://doi.org/10.1002/cam4.1677
Sun Z., Zhang T., Chen B. (2019) Long non-coding RNA metastasis-associated lung adenocarcinoma transcript 1 (MALAT1) promotes proliferation and metastasis of osteosarcoma cells by targeting c-Met and SOX4 via miR-34a/c-5p and miR-449a/b. Med. Sci. Monit. 25, 1410-1422. https://doi.org/10.12659/MSM.912703
Duan G., Zhang C., Xu C., Xu C., Zhang L., Zhang Y. (2019) Knockdown of MALAT1 inhibits osteosarcoma progression via regulating the miR‑34a/cyclin D1 axis. Int. J. Oncol. 54(1), 17–28. https://doi.org/10.3892/ijo.2018.4600
Jin X.M., Xu B., Zhang Y., Liu S.Y., Shao J., Wu L., Tang J.A., Yin T., Fan X.B., Yang T.Y. (2019) LncRNA SND1-IT1 accelerates the proliferation and migration of osteosarcoma via sponging miRNA-665 to upregulate POU2F1. Eur. Rev. Med. Pharmacol. Sci. 23(22), 9772–9780. https://doi.org/10.26355/eurrev_201911_19540
Hu X.H., Dai J., Shang H.L., Zhao Z.X., Hao Y.D. (2019) SP1-mediated upregulation of lncRNA ILF3-AS1 functions a ceRNA for miR-212 to contribute to osteosarcoma progression via modulation of SOX5. Biochem. Biophys. Res. Commun. 511(3), 510–517. https://doi.org/10.1016/j.bbrc.2019.02.110
Xing W., Xu W.Y., Chang L., Zhang K., Wang S.R. (2020) SP1-induced lncRNA LINC00689 overexpression contributes to osteosarcoma progression via the miR-655/SOX18 axis. Eur. Rev. Med. Pharmacol. Sci. 24(5), 2205–2217. https://doi.org/10.26355/eurrev_202003_20486
Wang M., Wang Z., Zhu X., Guan S., Liu Z. (2019) LncRNA KCNQ1OT1 acting as a ceRNA for miR-4458 enhances osteosarcoma progression by regulating CCND2 expression. In Vitro Cell Dev. Biol. Anim. 55(9), 694–702. https://doi.org/10.1007/s11626-019-00386-9
Chen X., Zhang C., Wang X. (2019) Long noncoding RNA DLEU1 aggravates osteosarcoma carcinogenesis via regulating the miR-671-5p/DDX5 axis. Artif. Cells Nanomed. Biotechnol. 47(1), 3322–3328. https://doi.org/10.1080/21691401.2019.1648285
Wang B., Qu X.L., Liu J., Lu J., Zhou Z.Y. (2019) HOTAIR promotes osteosarcoma development by sponging miR-217 and targeting ZEB1. J. Cell Physiol. 234(5), 6173–6181. https://doi.org/10.1002/jcp.27394
Lian H., Xie P., Yin N., Zhang J., Zhang X., Li J., Zhang C. (2019) Linc00460 promotes osteosarcoma progression via miR-1224-5p/FADS1 axis. Life Sci. 233, 116757. https://doi.org/10.1016/j.lfs.2019.116757
Liu J., Kong D., Sun D., Li J. (2019) Long non-coding RNA CCAT2 acts as an oncogene in osteosarcoma through regulation of miR-200b/VEGF. Artif. Cells Nanomed. Biotechnol. 47(1), 2994–3003. https://doi.org/10.1080/21691401.2019.1640229
Sun Y., Jia X., Wang M., Deng Y. (2019) Long noncoding RNA MIR31HG abrogates the availability of tumor suppressor microRNA-361 for the growth of osteosarcoma. Cancer Manag. Res. 11, 8055–8064. https://doi.org/10.2147/CMAR.S214569
Ba Z., Gu L., Hao S., Wang X., Cheng Z., Nie G. (2018) Downregulation of lncRNA CASC2 facilitates osteosarcoma growth and invasion through miR-181a. Cell Prolif. 51(1), 12409. https://doi.org/10.1111/cpr.12409
Han W., Liu J. (2018) LncRNA-p21 inhibited the proliferation of osteosarcoma cells via the miR-130b/ PTEN/AKT signaling pathway. J. Biomed. Pharmacother. 97, 911–918. https://doi.org/10.1016/j.biopha.2017.11.014
Yang C., Wang G., Yang J., Wang L. (2017) Long noncoding RNA NBAT1 negatively modulates growth and metastasis of osteosarcoma cells through suppression of miR-21. Am. J. Cancer Res. 7(10), 2009–2019.
Wang Y., Kong D. (2018) LncRNA GAS5 represses osteosarcoma cells growth and metastasis via sponging miR-203a. Cell Physiol. Biochem. 45(2), 844–855. https://doi.org/10.1159/000487178
Ye K., Wang S., Zhang H., Han H., Ma B., Nan W. (2017) Long noncoding RNA GAS5 suppresses cell growth and epithelial-mesenchymal transition in osteosarcoma by regulating the miR-221/ARHI pathway. J. Cell Biochem. 118(12), 4772–4781. https://doi.org/10.1002/jcb.26145
Zhang L., Wang Y., Zhang L., Xia X., Chao Y., He R., Han C., Zhao W. (2019) ZBTB7A, a miR-663a target gene, protects osteosarcoma from endoplasmic reticulum stress-induced apoptosis by suppressing lncRNA GAS5 expression. Cancer Lett. 448, 105–116. https://doi.org/10.1016/j.canlet.2019.01.046
Ye F., Tian L., Zhou Q., Feng D. (2019) LncRNA FER1L4 induces apoptosis and suppresses EMT and the activation of PI3K/AKT pathway in osteosarcoma cells via inhibiting miR-18a-5p to promote SOCS5. Gene. 721, 144093. https://doi.org/10.1016/j.gene.2019.144093
Xia P., Gu R., Zhang W., Sun Y.F. (2020) lncRNA CEBPA-AS1 overexpression inhibits proliferation and migration and stimulates apoptosis of OS cells via Notch signaling. Mol. Ther. Nucleic Acids. 19, 1470–1481. https://doi.org/10.1016/j.omtn.2019.10.017
Zhou F.C., Zhang Y.H., Liu H.T., Song J., Shao J. (2020) LncRNA LINC00588 suppresses the progression of osteosarcoma by acting as a ceRNA for miRNA-1972. Front. Pharmacol. 11, 255. https://doi.org/10.3389/fphar.2020.00255
Zhao J., Zhang C., Gao Z., Wu H., Gu R., Jiang R. (2018) Long non-coding RNA ASBEL promotes osteosarcoma cell proliferation, migration, and invasion by regulating microRNA-21. J. Cell Biochem. 119(8), 6461–6469. https://doi.org/10.1002/jcb.26671
Shen B., Zhou N., Hu T., Zhao W., Wu D., Wang S. (2019) LncRNA MEG3 negatively modified osteosarcoma development through regulation of miR-361-5p and FoxM1. J. Cell Physiol. 234(8), 13464–13480. https://doi.org/10.1002/jcp.28026
Ding L., Tian Y., Wang L., Bi M., Teng D., Hong S. (2019) Hypermethylated long noncoding RNA MEG3 promotes the progression of gastric cancer. Aging (Albany NY). 11(19), 8139–8155. https://doi.org/10.18632/aging.102309
Pardini B., Sabo A.A., Birolo G., Calin G.A. (2019) Noncoding RNAs in extracellular fluids as cancer biomarkers: the new frontier of liquid biopsies. Cancer (Basel). 11(8), 1170. https://doi.org/10.3390/cancers11081170
Botti G., Giordano A., Feroce F., De Chiara A.R., Cantile M. (2019) Noncoding RNAs as circulating biomarkers in osteosarcoma patients. J. Cell Physiol. 234, 19249–19255. https://doi.org/10.1002/jcp.28744
Wen J.J., Ma Y.D., Yang G.S., Wang G.M. (2017) Analysis of circulating long non-coding RNA UCA1 as potential biomarkers for diagnosis and prognosis of osteosarcoma. Eur. Rev. Med. Pharmacol. Sci. 21(3), 498–503.
Ma B., Li M., Zhang L., Huang M., Lei J.B., Fu G.H., Liu C.X., Lai Q.W., Chen Q.Q., Wang Y.L. (2016) Upregulation of long non-coding RNA TUG1 correlates with poor prognosis and disease status in osteosarcoma. Tumour Biol. 37(4), 4445–4455. https://doi.org/10.1007/s13277-015-4301-6
Huo Y., Li Q., Wang X., Jiao X., Zheng J., Li Z., Pan X. (2017) MALAT1 predicts poor survival in osteosarcoma patients and promotes cell metastasis through associating with EZH2. Oncotarget. 8(29), 46993–47006. https://doi.org/10.18632/oncotarget.16551
Chen D., Wang H., Zhang M., Jiang S., Zhou C., Fang B., Chen P. (2018) Abnormally expressed long non-coding RNAs in prognosis of osteosarcoma: a systematic review and meta-analysis. J. Bone Oncol. 13, 76–90. https://doi.org/10.1016/j.jbo.2018.09.005
Sun K., Zhao J. (2019) A risk assessment model for the prognosis of osteosarcoma utilizing differentially expressed lncRNAs. Mol. Med. Rep. 19(2), 1128–1138. https://doi.org/10.3892/mmr.2018.9768
Zhang J., Ju C., Zhang W., Xie L. (2018) LncRNA SNHG20 is associated with clinical progression and enhances cell migration and invasion in osteosarcoma. IUBMB Life. 70(11), 1115–1121. https://doi.org/10.1002/iub.1922
Логинов В.И., Филиппова Е.А., Куревлев С.В., Фридман М.В., Бурденный А.М., Брага Э.А. (2018) Супрессорные и гиперметилируемые микроРНК в патогенезе рака молочной железы. Генетика. 54(7), 757–775. https://doi.org/10.1134/S0016675818070081
Stelzer G., Rosen R., Plaschkes I., Zimmerman S., Twik M., Fishilevich S., Iny Stein T., Nudel R., Lieder I., Mazor Y., Kaplan S., Dahary D., Warshawsky D., Guan-Golan Y., Kohn A., Rappaport N., Safran M., Lancet D. (2016) The GeneCards suite: from gene data mining to disease genome sequence analysis. Curr. Protoc. Bioinformatics. 54, 1.30.1‒1.30.33. doi 10.1002 / cpbi.5
Mi H., Thomas P. (2009) PANTHER pathway: an ontology-based pathway database coupled with data analysis tools. Methods Mol. Biol. 563, 123–140. https://doi.org/10.1007/978-1-60761-175-2_7
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


