Молекулярная биология, 2020, T. 54, № 5, стр. 837-848
Влияние А/G-состава 3'-контекстов стоп-кодонов на терминацию трансляции у эукариот
Е. Е. Соколова a, П. К. Власов b, Т. В. Егорова a, А. В. Шувалов a, Е. З. Алкалаева a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Institute of Science and Technology
3400 Klosterneuburg, Austria
* E-mail: alkalaeva@eimb.ru
Поступила в редакцию 27.02.2020
После доработки 20.03.2020
Принята к публикации 26.03.2020
Аннотация
Терминация трансляции – завершающий этап биосинтеза белка, в котором значительную роль играют не только белковые факторы терминации, но и ближайшее нуклеотидное окружение стоп-кодонов. Описано довольно много случаев сквозного прочтения стоп-кодонов, обусловленного специфическими нуклеотидными мотивами, следующими за этими стоп-кодонами. Тем не менее, имеющиеся данные разрознены и не объясняют механизм влияния нуклеотидного контекста на терминацию трансляции. Известно, что использование стоп-кодона UAA предпочтительно для А/Т-богатых генов, а UAG и UGA – для G/С-богатых, что, в свою очередь, связано с уровнем экспрессии этих генов. Нами изучена взаимосвязь между частотой встречаемости нуклеотидов в 3′-области стоп-кодонов в геноме человека и их влиянием на эффективность терминации трансляции. Обнаружено, что сходный нуклеотидный состав стоп-кодонов и их 3′-контекста стимулирует терминацию трансляции. В то же время, нуклеотидный состав 3′-контекста, отличный от последовательности стоп-кодона, снижает эффективность терминации трансляции.
ВВЕДЕНИЕ
Терминация трансляции осуществляется с помощью белковых факторов терминации классов I и II (RF1/RF2 и RF3 в клетках прокариот; eRF1 и eRF3 у эукариот) [1, 2]. Факторы класса I распознают стоп-кодоны на мРНК в А-сайте малой субъединицы рибосомы, а также запускают гидролиз эфирной связи пептидил-тРНК в пептидил-трансферазном центре большой рибосомной субъединицы. Факторы класса II являются GTPазами, способствующими работе факторов класса I. Известно, что нуклеотидные последовательности, окружающие стоп-кодоны, могут влиять на терминацию трансляции и контролировать таким образом синтез белка. Анализ областей мРНК вокруг стоп-кодонов позволяет предположить, что эффективность терминации может контролироваться не только стоп-кодоном, но и, дополнительно, окружающими его нуклеотидами.
Частоты использования стоп-кодонов существенно варьируют у разных групп организмов. Терминирующий кодон UGA наиболее часто используется у млекопитающих и однодольных растений, тогда как UAA предпочитают все остальные эукариоты, кроме Drosophila, у которой самым частым стоп-кодоном является UAG. В то же время, у всех остальных групп эукариот UAG-кодон используется реже [3–6]. Распространенность стоп-кодонов у прокариот и эукариот совпадает: самый частый кодон UAA, самый редкий – UGA [7].
Биоинформатический анализ частот встречаемости нуклеотидов, окружающих стоп-кодоны, показал, что у про- и эукариот есть определенные предпочтения, сильно выраженные для –1 положения 5′-контекста и +4 положения 3′-контекста. Результаты изучения влияния 3′-контекста у прокариот и эукариот позволили предположить, что сигналом терминации трансляции служит не триплет, а, по крайней мере, четыре или шесть нуклеотидов [8, 9]. Сравнение нескольких прокариотических геномов показало, что разные виды имеют разные предпочтения 3′-контекста [10]. У Escherichia coli наиболее часто встречаются U и G в положении +4, у Mycobacterium tuberculosis в этом положении находится С, а у Bacillus subtilis – А. У паразитических видов Rickettsia prowazekii и Chlamydophila pneumoniae не выявлено явных закономерностей в 5'- и 3'-области. Полученные результаты подтвердили и уточнили данные более ранней работы [7], в которой тоже отмечена повышенная встречаемость U в позиции +4 всех трех стоп-кодонов E. coli, в отличие от менее представленных A и C. Эта тенденция характерна для высокоэкспрессирующихся генов, но слабо выражена у низко экспрессирующихся, а также генов со стоп-кодоном UAG. Так, статистический анализ нуклеотидного окружения стоп-кодонов E. coli показал, что гены с высоким уровнем экспрессии имеют определенное распределение нуклеотидов в окрестностях стоп-кодонов [7]. У E. coli 92% генов с высоким уровнем экспрессии имеют стоп-кодон UAA. В 63% этих генов в положении +4 за стоп-кодоном находится U, в 32% – G, а А или С встречаются только в 5%. В то же время, стоп-кодон UAA встречается всего в 38% генов с низким уровнем экспрессии, и в положении +4 не преобладает U. Подобная закономерность наблюдалась и у Salmonella typhimurium, B. subtilis, некоторых фагов E. coli и метанобразующих архебактерий. Статистический анализ более широкой области 3′-контекста стоп-кодона UGA после 4160 генов E. coli показал, что встречаемость нуклеотидов до положения +10 включительно имеет неслучайный характер, а в более отдаленных положениях наблюдается случайное распределение нуклеотидов. Особенно часто в области от +4 до +10 находится А, а содержание С, напротив, снижено [9].
У эукариот в положении +4 наблюдается предпочтение пуринов по отношению к пиримидинам, что было неоднократно показано при изучении эукариотических геномов [4, 10]. У изученных генов представителей разных классов (D. melanogaster, H. sapiens, Caenorhabditis elegans, Arabidopsis thaliana, Saccharomyces cerevisiae) в положении +4 наиболее часто встречается A или G [10]. Более широкое исследование геномов различных групп эукариот (позвоночных, беспозвоночных, однодольных и двудольных растений, дрожжей и простейших) с применением статистического анализа показало, что между отдельными группами в зависимости от стоп-кодона есть незначительные различия в распространенности того или иного нуклеотида в положении +4, но с явным преимуществом пуринов в этой позиции [4]. У позвоночных в положении +4 чаще находится А, чем G, за исключением стоп-кодона UGA, в котором наиболее часто стоит G. Также для 3′-контекста стоп-кодона UGA характерно более высокое содержание U в положении +4 по сравнению с другими двумя стоп-кодонами. У беспозвоночных эукариот в положении +4 чаще всего стоит A и очень редко С. Интересно, что у вирусов позвоночных и у позвоночных наблюдается сходная тенденция, а именно, предпочтение А в положении +4, одинаковая, но меньшая частота G/U и намного более редкий С. В целом, у эукариот наиболее распространены стоп-сигналы UAA(A/G) и UGA(A/G). Например, в положении +4 у S. cerevisiae и D. melanogaster предпочтителен A/G, а их гены с высокой экспрессией имеют стоп-последовательность UAAG [3]. Исследования контекста стоп-кодонов генов растений [5] показывают, что он подчиняется общим закономерностям, характерным для эукариот, с небольшими различиями у однодольных и двудольных растений. У S. cerevisiae и, скорее всего, у всех эукариот нуклеотиды до положения +9 распределены неслучайным образом, тогда как не выявлено закономерностей в распределении остальных нуклеотидов вплоть до положения +65 в нетранслируемой области [3].
По эффективности терминации трансляции разные стоп-кодоны можно условно разделить на сильные и слабые. Кроме того, нуклеотидный контекст, окружающий стоп-кодоны, может и снижать, и повышать эффективность терминации трансляции [11]. По-видимому, у прокариот существуют определенные предпочтения стоп-сигналов, эффективность терминации на которых последовательно уменьшается в ряду: UAAU > UAAG > UAA(A/C). Сходное изменение эффективности терминации наблюдается у дрожжей, где предпочтителен стоп-сигнал UAA(A/G), а также у других организмов [3]. Эксперименты in vivo показали, что иерархия эффективности четвертого основания обоих стоп-кодонов UAA и UGA в терминации трансляции у E. coli выглядит как U > G > A (приблизительно равен С), а стоп-кодона UAG – G > U = A > C [12]. В другой своей работе [9], используя фотоактивируемые сшивки между мРНК и фактором терминации трансляции, эти же авторы показали, что нуклеотиды в положениях +4, +5 и +6 за стоп-кодоном также влияют на эффективность терминации трансляции, взаимодействуя с фактором терминации RF2. Варьирование нуклеотидов в этих позициях вело к изменению эффективности взаимодействия между первым основанием стоп-кодона UGA и фактором RF2. Эти данные предполагают, что прокариотический фактор терминации RF2 распознает последовательность по крайней мере из шести нуклеотидов, включающих стоп-кодон UGA [9]. В дальнейшем, исходя из предположения, что прокариотические факторы терминации чувствительны к контексту стоп-кодонов, другая группа исследователей показала влияние мутации Arg137Pro фактора RF1 на распознавание сильных (UAGU) и слабых (UAGA) стоп-сигналов [13]. Кроме того, показано, что увеличение концентраций факторов терминации RF в клетках прокариот и фактора eRF1 в трансляционной системе на основе лизата ретикулоцитов кролика приводит к повышению эффективности терминации на слабых стоп-сигналах [10]. Авторы этой работы предположили, что увеличение количества факторов терминации приводит к их более эффективной конкуренции с супрессорными тРНК за распознавание стоп-кодона.
Статистический анализ более 5000 генов млекопитающих и последующие эксперименты in vitro также показали, что эффективность терминации трансляции зависит от контекста, а иерархия оснований в положении +4 по силе стоп-сигнала выглядит как A = G $ \gg $ U = C для всех трех стоп-кодонов [14]. Выявлены наиболее часто встречающиеся стоп-сигналы UAA(A > G), UGA(G > A) и UAG(G > A). Самыми редкими оказались сигналы UAG(C/U) и UGA(C/U) [14]. Однако оценка эффективности супресcии каждого стоп-кодона в репортерной системе в S. cerevisiae привела к несколько иным результатам [15]. Оказалось, что уровень сквозного прочтения стоп-кодонов, обратный эффективности терминации, увеличивался в ряду UAA(G > A > U> C), UGA(G > U > > A> C) и UAG(A > U > C > G) [15].
Анализ уровня сквозного чтения стоп-кодонов дрожжей [8] показал, что нуклеотиды в положениях +4, +5, +6 и +8 имеют наибольшее значение для прочитывания стоп-кодона как значащего, в то время как замены нуклеотида в положении +7 практически не имели эффекта. Установлено также, что в клетках дрожжей уровень сквозного чтения стоп-кодонов зависит от нуклеотидов в позициях +4, +5, +6, +8 (или +9) в 3'-области стоп-кодона [16]. Определение уровня сквозного прочтения в клетках млекопитающих подтвердило значительное влияние нуклеотидов +4 и +8, следующих за стоп-кодоном [17]. Показано также, что присутствие G или U в положении +4 за стоп-кодоном UGA или UAG приводит к ослаблению терминации трансляции. Следующие за стоп-кодоном UAA нуклеотиды +4С и +4G также снижали уровень терминации, хотя их влияние менее выражено, чем у стоп-кодонов UGA или UAG. Нуклеотиды в положениях +5 и +6 вызывали повышение либо понижение уровня сквозного прочтения в зависимости от определенного стоп-кодона и нуклеотида +4, что предполагает кооперативный эффект соседствующих нуклеотидов. В то же время нуклеотиды в положениях в +7 и +9 не влияли существенно на терминацию трансляции. Присутствие С в положении +8 снижало эффективность терминации, +8U давал значительно более сильный сигнал терминации, в то время как эффекты +8А и +8G были умеренными.
Таким образом, на сегодняшний день накоплено много информации о частотах стоп-кодонов и нуклеотидов, их окружающих, а также об эффективности стоп-сигналов у разных организмов. Однако нет единого мнения о возможной связи частот встречаемости стоп-сигналов и их эффективности в терминации трансляции. В попытке определить фундаментальные основы влияния нуклеотидного контекста стоп-кодонов на терминацию трансляции мы предположили, что распространенность 3′-контекстов стоп-кодонов может коррелировать с эффективностью терминации трансляции. Для проверки этой гипотезы определены частоты встречаемости гексануклеотидных 3'-последовательностей стоп-кодонов в геноме человека, а затем на выбранных мотивах оценена эффективность терминации в реконструированной системе трансляции.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Биоинформатический анализ 3'-контекстов стоп-кодонов в геноме человека. Частоты 3′-контекстов стоп-кодонов анализировали с использованием данных 38-й версии генома человека, опубликованных в NCBI (ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/001/405/GCA_000001405.15_GRCh38). Были проанализированы все аннотированные гены прямой и обратной цепей на всех 24 хромосомах (включая X и Y), чтобы определить первые 6 нуклеотидов, следующих за стоп-кодонами. Конечные данные включали 20412 генов и 3′-контексты их стоп-кодонов. Далее была определена частота встречаемости каждого варианта контекста. Анализ последовательностей и статистический анализ проведен с помощью языка Perl.
Получение модельных мРНК. Конструкции, кодирующие модельные MVHL-стоп мРНК, содержащие один из трех стоп-кодонов (UAA, UAG или UGA), описаны ранее [18, 19]. В качестве матрицы для получения плазмид с различными 3′‑контекстами стоп-кодонов использовали плазмиду pET28a(+)-MVHL-stop-HindIII, полученную в нашей лаборатории на основе вектора MVHL-STOP/C-less. Чтобы получить мРНК с желаемыми 3′-контекстами стоп-кодонов, проведен сайт-направленный мутагенез имеющихся конструкций. Последовательности праймеров для каждой конструкции приведены в табл. 1. Перед транскрипцией все плазмиды линеаризовали с помощью рестриктазы XhoI. Модельные мРНК получали с помощью Т7 РНК-полимеразы в реакции run-off транскрипции.
Таблица 1.
Матрицы и праймеры, использованные в клонировании
| Конструкция | Матрица | Праймер | Эндонуклеаза рестрикции |
|---|---|---|---|
| UAA-A-rich | pET28a(+)-MVHL-stop-HindIII | UAA-A-rich, Bgl | BglII, HindIII |
| UAG-A-rich | pET28a(+)-MVHL-stop-HindIII | UAG-A-rich, Bgl | BglII, HindIII |
| UGA-A-rich | pET28a(+)-MVHL-stop-HindIII | UGA-A-rich, Bgl | BglII, HindIII |
| UAA-G-rich | pET28a(+)-MVHL-stop-HindIII | UAA-G-rich, Bgl | BglII, HindIII |
| UAG-G-rich | pET28a(+)-MVHL-stop-HindIII | UAG-G-rich, Bgl | BglII, HindIII |
| UGA-G-rich | pET28a(+)-MVHL-stop-HindIII | UGA-G-rich, Bgl | BglII, HindIII |
| UAA-Standard | pET28a(+)-MVHL-stop-HindIII | UAA-Standard, Bgl | BglII, HindIII |
| UAG-Standard | pET28a(+)-MVHL-stop-HindIII | UAG-Standard, Bgl | BglII, HindIII |
| UGA-Standard | pET28a(+)-MVHL-UAA-HindIII | UGA-Standard, Bgl | BglII, HindIII |
| UAA-Rare | pET28a(+)-MVHL-stop-HindIII | UAA-Rare, Bgl | BglII, HindIII |
| UAG-Rare | pET28a(+)-MVHL-stop-HindIII | UAA-Rare, Bgl | BglII, HindIII |
| UGA-Rare | pET28a(+)-MVHL-stop-HindIII | UAA-Rare, Bgl | BglII, HindIII |
Получение субъединиц рибосом и факторов трансляции. Рибосомные субъединицы 40S и 60S и эукариотические факторы трансляции eIF2, eIF3, eEF1 и eEF2 очищали из лизата клеток HeLa, как описано ранее [18]. Рекомбинантные факторы трансляции человека, несущие гистидиновый таг: eIF1, eIF1A, eIF4A, eIF4B, ∆eIF4G, ∆eIF5B, eIF5, eRF1, мутантный фактор eRF1(AGQ), а также фактор eRF3c, укороченный с N-конца на 138 аминокислотных остатков, нарабатывали в штамме BL21 E. coli, затем белки выделяли с помощью аффинной хроматографии на Ni-NTA-агарозе с последующей ионообменной хроматографией [18].
Сборка претерминационных комплексов (преТК) и анализ эффективности терминации трансляции. Формирование преТК в препаративных количествах проводили в 500 мкл реакционной смеси и собирали, как описано ранее [18, 20]. Эффективность терминации трансляции определяли по количеству высвободившегося меченого синтезированного пептида [21]. С этой целью 37 пмоль мРНК инкубировали в течение 30 мин при 37°C в буфере А (20 мM Tрис-ацетат, pH 7.5, 100 мM KAc, 2.5 мM MgCl2, 2 мM дитиотреитол) с добавлением 400 ед. акт. ингибитора РНКаз, 1 мM ATP, 0.25 мM спермидина, 0.2 мM GTP, 75 мкг суммарной тРНК (ацилированной всеми или отдельными аминокислотами и [35S]Met), 75 пмоль очищенных 40S и 60S рибосомных субъединиц, 125 пмоль каждого из факторов eIF2, eIF3, eIF4A, ∆eIF4G, eIF4B, eIF1, eIF1A, eIF5, ∆eIF5B, 200 пмоль eEF1 и 50 пмоль eEF2. Затем смесь центрифугировали в роторе Beckman SW55 в течение 95 мин при 4°C и 50 000 об/мин. в 10–30% (w/w) градиенте плотности сахарозы (SDG), приготовленном с использованием буфера А с 5 мM MgCl2. Фракции, содержащие преТК, в соответствии с присутствием в них [35S]Met, объединяли, разводили в 3 раза буфером А с 1.25 мM MgCl2 (до конечной концентрации 2.5 мM Mg2+) и анализировали с помощью гидролиза пептида. Для анализа высвобождения синтезированного пептида аликвоты (30 мкл), содержащие 0.09 пмоль преТК инкубировали в течение 15 мин при 37°C с 5 пмоль фактора eRF1 или 3 мин с 0.625 пмоль факторов eRF1, eRF3c и 0.2 мM GTP с добавлением 0.2 мM Mg2+. Рибосомные комплексы после гидролиза пептида осаждали холодной 5%-ной трихлоруксусной кислотой и центрифугировали при 14 000 g и 4°C. Количество высвободившегося пептида, содержащего [35S], в супернатанте определяли с помощью жидкостного сцинтилляционного спектрометра Intertechnique SL-30.
Анализ рибосомных комплексов методом toe-printing. Для анализа методом toe-printing к аликвотам объемом 10 мкл, содержащим 0.03 пмоль преТК, добавляли меченный 5'-FAM праймер (5'-FAM-GCATTTGCAGAGGACAGG-3'), комплементарный участку мРНК β-глобина (197–214 н.), и обратную транскриптазу AMV. Фрагменты кДНК разделяли с помощью капиллярного электрофореза с использованием ABI Prism® Genetic Analyser 3100 (“Applera”, США).
Определение эффективности терминации трансляции. Эффективность терминации трансляции определяли подсчетом количества радиоактивно меченного пептида MVHL, высвободившегося из рибосомных комплексов в присутствии факторов терминации eRF1 или eRF1 + eRF3c GTP. Значения kcat/KM вычисляли из значений kobs. В условиях реакции первого порядка при концентрации субстрата намного ниже ожидаемых значений KM уравнение Михаэлиса–Ментен v = (VmS)/(KM + S) приобретает вид v = (VmS)/KM, так как S $ \ll $ KM или v = (Vm/KM)S = kobsS = –S/dt, при интегрировании которого получаем lnS = –kobst + lnSo, где So – начальная концентрация субстрата, а S – концентрация субстрата в конкретный момент времени. Регистрация значений гидролиза в течение некоторого промежутка времени позволяет определить kobs из графика зависимости lnS от времени. Скорость пропорциональна концентрации субстрата. kobs= Vm/KM = (kcatEt)/KM, так как Vm = kcatEt, следовательно, kcat/KM = kobs/Et.
Статистическая обработка данных. Статистический анализ проводили с использованием Microsoft Office Excel (разработчик “Microsoft,” США) и пакета программ Statistica версии 7.0 (разработчик “StatSoft, Ins,” CША). Статистически значимые отличия выявляли с использованием t-критерия Стьюдента. Для проверки гипотез с помощью p-значения мы приняли р < 0.01 и р < 0.05. Средние значения и погрешности вычисляли по результатам не менее трех измерений. Планки погрешности показывают значения SD (стандартного отклонения).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Поиск распространенных и редких 3′-контекстов стоп-кодонов в геноме человека
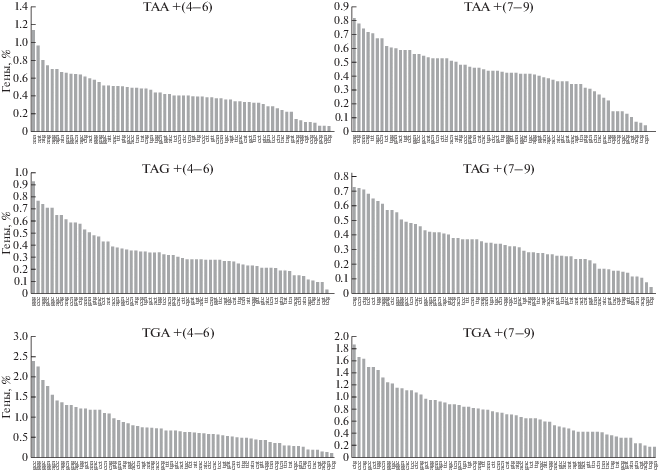
Мы провели биоинформатический анализ частот встречаемости первых шести нуклеотидов, следующих за стоп-кодонами, в геноме человека. Встречаемость всех гексануклеотидных последовательностей была примерно одинаковой, т.е. выявить определенные часто и редко встречающиеся варианты оказалось невозможным. Поэтому были проанализированы частоты встречаемости 3′-контекстов стоп-кодонов, разделенных на группы из двух последовательных триплетов, трех последовательных пар динуклеотидов и шести однонуклеотидных позиций (рис. 1–3). Данные для каждого стоп-кодона анализировали отдельно. Проведенный анализ позволил выявить наиболее редко и часто встречающиеся три-, ди- и мононуклеотиды в 3′-контекстах стоп-кодонов. Примечательно, что чем ближе нуклеотиды находились к стоп-кодону, тем более выраженным было предпочтение к специфическому нуклеотиду.
Рис. 1.
Встречаемость мононуклеотидов в 3′-области стоп-кодонов. По оси абсцисс – положение нуклеотидов после стоп-кодона (1–6).
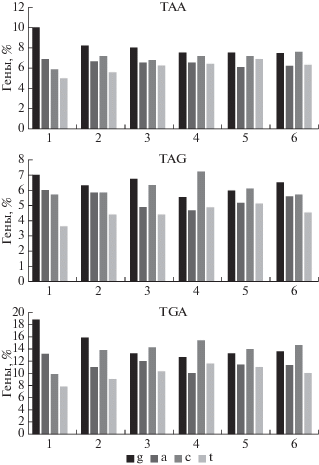
Рис. 2.
Встречаемость динуклеотидов в 3′-области стоп-кодонов. Номерами в заголовках диаграмм указаны положения нуклеотидов после стоп-кодона.
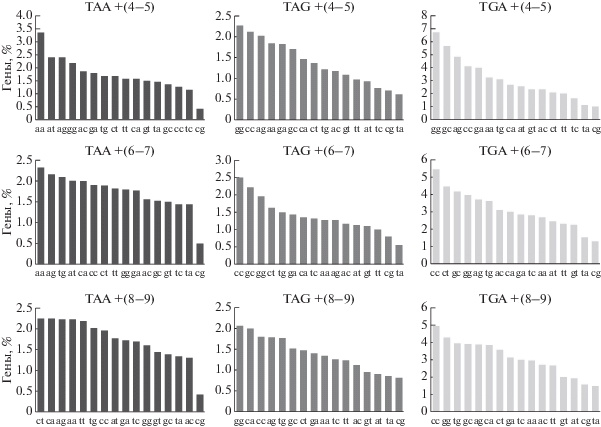
Рис. 3.
Встречаемость тринуклеотидов в 3′-области стоп-кодонов. В заголовках диаграмм указано положение нуклеотидов после стоп-кодона.
Для проверки гипотезы о корреляции между частотой встречаемости 3′-контекста стоп-кодона и эффективностью терминации трансляции с помощью биоинформатического анализа были выбраны модельные 3′-контексты стоп-кодонов. Последовательности выбранных 3′-контекстов и частоты их встречаемости в геноме человека отображены в табл. 2. В качестве часто встречающегося первого триплета для стоп-кодонов UAG и UGA выбран мотив GGG. Для UAA как наиболее распространенный был выбран триплет AAA. В качестве второго триплета в составе модельных распространенных контекстов для всех трех стоп-кодонов мы выбрали CUG. Мотив UCG UCG выбран как модельный редко встречающийся 3′-контекст всех трех стоп-кодонов. Последовательность UGU CGU, входящая в состав 3′-контекста исходной мРНК, в которую вносили новые контексты, названа “стандартной”. Эту последовательность мы использовали ранее в экспериментах в реконструированной системе трансляции млекопитающих [19–22]. Частоты встречаемости триплетов из данного контекста близки к расчетным частотам случайного триплета NNN 1/64 = 1.56% (табл. 2), поэтому мы использовали “стандартную” последовательность как контроль. Контексты, выбранные как частые, встречались в 2–2.5 раза чаще, чем среднестатистические; редкие контексты имели частоту в 10 раз меньшую, чем среднестатистические (табл. 2).
Таблица 2.
Частоты встречаемости три-, ди- и мононуклеотидов в 3′-контексте стоп-кодонов в геноме человека (%)
| Стоп-кодон | Распространенный контекст | Редкий контекст | Стандартный контекст |
|---|---|---|---|
| UAA | AAA CUG 4 2.8 |
UCG UCG 0.2 0.2 |
UGU GUG 1.5 1 |
| AA AC UG 12 5.6 7.2 | UC GU CG 4.2 5.4 1.5 |
UG UG UG 6 7.5 7.2 |
|
| A A A 36 30 30 |
U C G 21 20 24 |
U G U 21 24 24 |
|
| UAG | GGG CUG 4 3 | UCG UCG 0.15 0.15 |
UGU GUG 1.3 1.8 |
| GG GC UG 10 10 8 | UC GU CG 3.5 4.9 3.7 |
UG UG UG 5.5 7 8 |
|
| G G G 31 28 30 |
U C G 16 26 30 |
U G U 16 28 20 |
|
| UGA | GGG CUG 4.5 3.8 |
UCG UCG 0.2 0.4 |
UGU GUG 1 1.5 |
| GG GC UG 14 8 8 | UC GU CG 3.4 4.6 3 |
UG UG UG 6 7 8 |
|
| G G G 38 32 27 |
U C G 16 28 27 |
U G U 16 32 21 |
Частоты встречаемости специфических триплетов внутри 3′-контекстов не коррелируют с эффективностью терминации трансляции in vitro
Выбранные 3′-контексты стоп-кодонов (табл. 3) помещали в конструкции для получения модельных мРНК. Чтобы оценить эффективность терминации трансляции, мы проводили гидролиз пептидил-тРНК, добавляя факторы терминации в реконструированную систему трансляции млекопитающих, и определяли эффективность высвобождения синтезированного пептида. С этой целью построены кинетические кривые (рис. 4). Скорости реакции в присутствии фактора eRF3с были значительно выше, поэтому количество факторов терминации и время инкубации в эксперименте с eRF3с были уменьшены. На основе полученных данных вычисляли значения эффективностей терминации трансляции (kcat/KM) для всех трех контекстов на каждом стоп-кодоне в присутствии и в отсутствие eRF3-GTP (рис. 5).
Таблица 3.
3'-Контексты стоп-кодонов, выбранные для экспериментального анализа эффективности терминации трансляции
| Название | Стоп-кодон | 3'-контекст |
|---|---|---|
| Стандартный | UAA UAG UGA | UGU GUG |
| А-богатый (распространенный для UAA) | UAA UAG UGA | AAA CUG |
| G-богатый (распространенный для UAG/UGA) | UAA UAG UGA | GGG CUG |
| Редкий | UAA UAG UGA | UCG UCG |
Рис. 4.
Примеры кинетических кривых гидролиза пептидил-тРНК, индуцируемого факторами терминации человека, на стандартном контексте с добавлением eRF1 (а), eRF1 + eRF3 (б), на распространенном контексте с добавлением eRF1 (в), eRF1 + eRF3 (г), на редком контексте с добавлением eRF1 (д), eRF1+eRF3 (е). Доля меченного [35S] тетрапептида (MVHL), выщепляющегося из комплексов терминации, показана как функция от времени. Значение, равное единице, соответствует максимальному значению гидролиза пептидил-тРНК, индуцируемого факторами терминации.
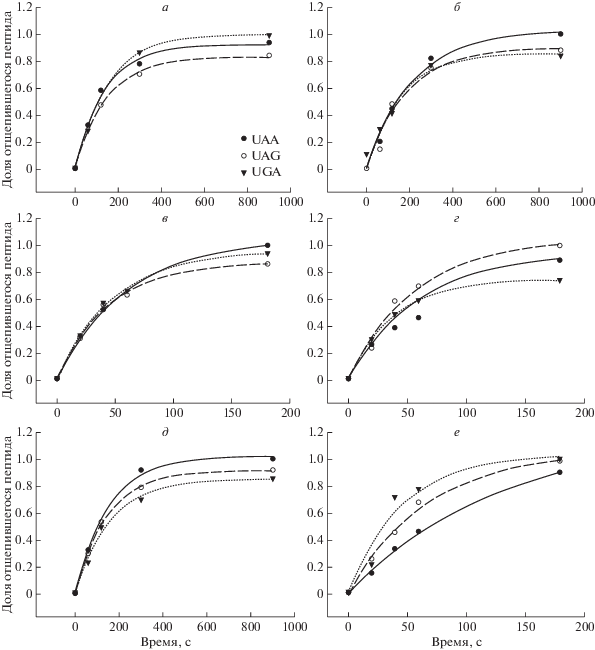
Рис. 5.
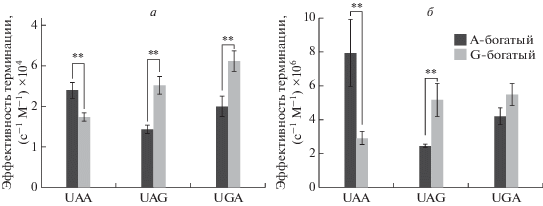
Влияние 3′-контекстов стоп-кодонов на эффективность гидролиза пептидил-тРНК. Эффект частоты встречаемости нуклеотидных контекстов на высвобождение пептида, индуцированное фактором eRF1 (а) и комплексом eRF1+eRF3c (б). Звездочками (**) обозначена статистически значимая разница относительно контроля p < 0.01 (n = 3).
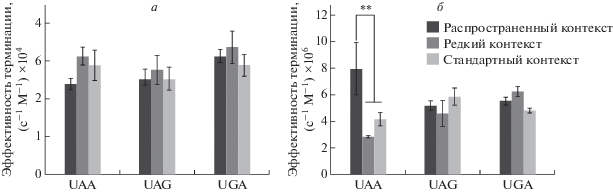
Оказалось, что эффективность терминации трансляции в присутствии фактора eRF1 на всех трех 3′-контекстах (распространенном, редком и стандартном) примерно одинакова на всех стоп-кодонах (рис. 5а). Однако при добавлении фактора eRF3c в присутствии GTP наблюдался рост эффективности терминации трансляции на стоп-кодоне UAA с распространенным контекстом AAA CUG (рис. 5б). Эффективность гидролиза пептидил-тРНК на распространенном контексте была в 3 раза выше, чем на редком, тогда как эффективность терминации трансляции на стандартном контексте принимала среднее значение. На стоп-кодонах UAG и UGA эффективность терминации трансляции была примерно одинаковой в присутствии всех трех 3′-контекстов. Таким образом, нам не удалось увидеть однозначной корреляции между эффективностью терминации трансляции и частотой встречаемости нуклеотидов в составе 3′-контекста, за исключением последовательности AAA CUG, следующей за стоп-кодоном UAA. По-видимому, частота использования нуклеотидов в 3'-области стоп-кодона определяется некими другими факторами.
A/G-состав 3 '-контекстов стоп-кодонов влияет на эффективность терминации трансляции in vitro
AT- и GC-состав генов определяет использование стоп-кодонов UAA и UAG/UGA соответственно [14]. Мы предположили, что AT- и GC-состав генов определяет также репертуар нуклеотидов в 3′-контексте стоп-кодонов. В таком случае распространенный контекст для стоп-кодона UAA должен быть А-богатым и G-богатым для UAG/UGA (табл. 3). Чтобы проверить эту гипотезу, мы поместили А- и G-богатый контексты в соответствующие плазмиды, получили набор мРНК и определили влияние контекстов стоп-кодонов на эффективность терминации трансляции в реконструированной системе эукариотической трансляции. Мы обнаружили, что эффективность терминации трансляции на G-богатом контексте, следующим за стоп-кодоном UAA (UAA GGG CUG), а также на А-богатом контексте за стоп-кодонами UAG/UGA (UAG/UGA AAA CUG) значительно снижалась в сравнении с распространенными контекстами этих стоп-кодонов (рис. 6а). Этот эффект сохранялся как при терминации трансляции одним фактором eRF1, так и при добавлении смеси факторов eRF1 + eRF3c (рис. 6б). Таким образом, нам удалось подтвердить важность A/G-состава в 3'-области стоп-кодонов для эффективного гидролиза пептидил-тРНК в ходе терминации трансляции.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Мы использовали сочетание биоинформатического анализа частот встречаемости нуклеотидов в 3′-области стоп-кодонов в геноме человека и экспериментальную проверку выбранных последовательностей в эукариотической системе трансляции in vitro. На основании данных о частотах встречаемости нуклеотидов (рис. 1–3) для экспериментальной проверки влияния контекстов на эффективность терминации трансляции отобрали распространенные, редкие и среднестатистические контексты стоп-кодонов (табл. 3). Эксперименты с модельными контекстами не выявили связи между частотами встречаемости 3′-контекстов и их влиянием на эффективность терминации трансляции. Однако в ходе работы обнаружено, что А/G-состав контекстов модулирует эффективность терминации трансляции в зависимости от стоп-кодона. Так, А-богатый контекст показал повышенную эффективность терминации на UAA-кодоне и пониженную – на UAG и UGA, а G-богатый наоборот – повышенную эффективность на UAG/UGA и пониженную на UAA (рис. 6). Судя по всему, влияние 3′-контекста определяется не точной последовательностью нуклеотидов, а предпочтением А-богатого контекста для UAA и G-богатого для UAG- и UGA-стоп-кодонов, что в целом подтверждается частотами встречаемости различных нуклеотидов в 3′-области стоп-кодонов (рис. 1–3). Этот результат объясняет отсутствие значительной разницы в эффективностях терминации на UAG- и UGA-стоп-кодонах с использованием контекстов с различными частотами встречаемости в геноме (рис. 5).
Интересно, что в одной из недавних работ по определению 3′-контекстов стоп-кодонов, влияющих на эффективность терминации трансляции и уровень сквозного чтения в клетках эукариот [17], проанализированы последовательности, схожие с выявленными нами распространенными и редкими контекстами. По полученным данным [17], последовательности стоп-сигналов UAA AAA, UAG GGG и UGA GGG в клетках млекопитающих показали уровень терминации трансляции, близкий к норме. На сигналах UAA GGG, UAG AAA и UGA AAA уровень сквозного чтения несколько повышался, хотя изменения нельзя назвать драматическими. Это подтверждает наши данные о снижении эффективности терминации при смене контекстов на А- и G-богатые в чистой системе эукариотической трансляции (рис. 6а, б). Примечательно, что эффект от смены контекста для разных стоп-кодонов [17] в клетках был не столь выражен, как в нашей системе in vitro, что, по-видимому, указывает на имеющийся в клетке механизм “спасения” в ситуации снижения эффективности терминации трансляции. Также показано [17], что сигналы UAA UCG, UAG UCG и UGA UCG, которые содержат после стоп-кодона первый триплет из выявленного нами редкого 3′-контекста, определяли либо нормальный уровень терминации (UAG UCG), либо очень слабое сквозное чтение (UAA UCG и UGA UCG), что также соответствует нашим результатам. Мотив UGU, соответствующий первому триплету из нашего стандартного контекста, также рассматривавшийся в этом исследовании, практически не менял эффективность терминации на стоп-кодоне UAA, однако слабо увеличивал сквозное чтение стоп-кодонов UAG и UGA в клетках. В нашей работе существенное изменение эффективности терминации трансляции в зависимости от контекста на стоп-кодоне UAA (почти в 3 раза) наблюдалось лишь в присутствии фактора терминации класса 2 eRF3. В отсутствие eRF3 разница в эффективности терминации в комплексах с разными контекстами не превышает 30%. Таким образом, влияние 3′-контекста проявляется в большей степени при совместной работе факторов терминации трансляции. Мы предполагаем, что этот эффект связан с направленным изменением конформации фактора eRF1 при взаимодействии с eRF3 и уменьшением угла между его С- и N-доменами, которое приводит к сближению C-домена eRF1 с 3'-областью стоп-кодонов [23]. Возможно, это способствует распознаванию фактором терминации eRF1 области мРНК за стоп-кодоном, что, в конечном итоге, изменяет эффективность узнавания стоп-кодона и осуществления гидролиза пептидил-тРНК.
Недавно в культуре клеток человека с использованием рибосомного профайлинга также был проведен анализ последовательностей вокруг стоп-кодонов, способствующих сквозному чтению после обработки аминогликозидными антибиотиками [24]. По результатам этой работы были сделаны несколько иные выводы: контексты, содержащие большое количество А и Т, способствуют повышению сквозного чтения стоп-кодонов, тогда как G- и C-богатые мотивы, напротив, обеспечивают эффективную терминацию трансляции. Однако здесь нет явного противоречия с нашими данными, так как нуклеотидный состав контекстов анализировали без привязки к определенному типу стоп-кодона, таким образом усредняя имеющиеся эффекты [24]. Кроме того, стоит отметить, что сквозное чтение стоп-кодонов, наблюдаемое в клетках, – это сложный процесс с участием аминоацил-тРНК, которые конкурируют с фактором терминации eRF1 за распознавание стоп-кодонов. В нашей же чистой in vitro системе на стадии терминации трансляции не использовались конкурирующие тРНК, поэтому мы имеем возможность судить о непосредственном влиянии выявленных контекстов на эффективность терминации.
Мы хотим поблагодарить сотрудников ЦКП “Геном” за секвенирование, Татьяну Пестову, Кристофера Хелен и Людмилу Юрьевну Фролову за предоставленные плазмиды, а также сотрудников лаборатории за плодотворное обсуждение результатов. Также благодарим за большой вклад в представленную работу бывших сотрудников лаборатории Юлию Владимировну Бочарову и Полину Николаевну Крючкову.
Работа выполнена при поддержке Российского научного фонда (грант № 19-74-10078).
В работе не использовали животных в качестве объектов исследования
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Zhouravleva G., Frolova L., Le Goff X., Le Guellec R., Inge-Vechtomov S., Kisselev L., Philippe M. (1995) Termination of translation in eukaryotes is governed by two interacting polypeptide chain release factors, eRF1 and eRF3. EMBO J. 14, 4065–4072.
Stansfield I., Jones K.M., Ter-Avanesyan M.D., Tuite M.F. (1995) The products of the SUP45 (eRF1) and SUP35 genes interact to mediate translation termination in Saccharomyces cerevisiae. EMBO J. 14, 4365–4373.
Brown C.M., Stockwell P.A., Trotman C.N., Tate W.P. (1990) Sequence analysis suggests that tetra-nucleotides signal the termination of protein synthesis in eukaryotes. Nucl. Acids Res. 18, 6339–6345.
Cavener D.R., Ray S.C. (1991) Eukaryotic start and stop translation sites. Nucl. Acids Res. 19, 3185–3192.
Angenon G., Van Montagu M., Depicker A. (1990) Analysis of the stop codon context in plant nuclear genes. FEBS Lett. 271, 144–146.
Sun J., Chen M., Xu J., Luo J. (2005) Relationships among stop codon usage bias, its context, isochores, and gene expression level in various eukaryotes. J. Mol. Evol. 61, 437–444.
Brown C.M., Stockwell P.A., Trotman C.N.A., Tate W.P. (1990) The signal for the termination of protein synthesis in procaryotes. Nucl. Acids Res. 18(8), 2079–2086. https://doi.org/10.1093/nar/18.8.2079
Namy O., Hatin I., Rousset J.P. (2001) Impact of the six nucleotides downstream of the stop codon on translation termination. EMBO Rep. 2, 787–793.
Poole E.S., Major L.L., Mannering S.A., Tate W.P. (1998) Translational termination in Escherichia coli: Three bases following the stop codon crosslink to release factor 2 and affect the decoding efficiency of UGA-containing signals. Nucl. Acids Res. 26(4), 954–960. https://doi.org/10.1093/nar/26.4.954
Cridge A.G., Major L.L., Mahagaonkar A.A., Poole E.S., Isaksson L.A., Tate W.P. (2006) Comparison of characteristics and function of translation termination signals between and within prokaryotic and eukaryotic organisms. Nucl. Acids Res. 34, 1959–1973.
Bertram G., Innes S., Minella O., Richardson J.P., Stansfield I. (2001) Endless possibilities: translation termination and stop codon recognition. Microbiology. 147, 255–269.
Poole E.S., Brown C.M., Tate W.P. (1995) The identity of the base following the stop codon determines the efficiency of in vivo translational termination in Escherichia coli. EMBO J. 14(1), 151–158. https://doi.org/10.1002/j.1460-2075.1995.tb06985.x
Zhang S., Rydén-Aulin M., Isaksson L.A. (1999) Interaction between a mutant release factor one and P-site peptidyl-tRNA is influenced by the identity of the two bases downstream of the stop codon UAG. FEBS Lett. 455(3), 355–358. https://doi.org/10.1016/S0014-5793(99)00912-6
Mccaughan K.K., Brown C.M., Dalphin M.E., Berry M.J., Tate W.P. (1995) Translational termination efficiency in mammals is influenced by the base following the stop codon. Proc. Natl. Acad. Sci. USA. 92(12), 5431–5435. https://doi.org/10.1073/pnas.92.12.5431
Bonetti B., Fu L., Moon J., Bedwell D.M. (1995) The efficiency of translation termination is determined by a synergistic interplay between upstream and downstream sequences in Saccharomyces cerevisiae. J. Mol. Biol. 251, 334–345.
Williams I., Richardson J., Starkey A., Stansfield I. (2004) Genome-wide prediction of stop codon readthrough during translation in the yeast Saccharomyces cerevisiae. Nucl. Acids Res. 32(22), 6605–6616. https://doi.org/10.1093/nar/gkh1004
Cridge A.G., Crowe-Mcauliffe C., Mathew S.F., Tate W.P. (2018) Eukaryotic translational termination efficiency is influenced by the 3 nucleotides within the ribosomal mRNA channel. Nucl. Acids Res. 6(4), 1927–1944. https://doi.org/10.1093/nar/gkx1315
Alkalaeva E.Z., Pisarev A.V., Frolova L.Y., Kisselev L.L., Pestova T.V. (2006) In vitro reconstitution of eukaryotic translation reveals cooperativity between release factors eRF1 and eRF3. Cell. 125, 1125–1136.
Alkalaeva E., Eliseev B., Ambrogelly A., Vlasov P., Kondrashov F.A., Gundllapalli S., Frolova L., Söll D., Kisselev L. (2009) Translation termination in pyrrolysine-utilizing archaea. FEBS Lett. 583, 3455–3460.
Susorov D., Mikhailova T., Ivanov A., Sokolova E., Alkalaeva E. (2015) Stabilization of eukaryotic ribosomal termination complexes by deacylated tRNA. Nucl. Acids Res. 43, 3332–3343.
Kryuchkova P., Grishin A., Eliseev B., Karyagina A., Frolova L., Alkalaeva E. (2013) Two-step model of stop codon recognition by eukaryotic release factor eRF1. Nucl. Acids Res. 41, 4573–4586.
Ivanov A., Mikhailova T., Eliseev B., Yeramala L., Sokolova E., Susorov D., Shuvalov A., Schaffitzel C., Alkalaeva E. (2016) PABP enhances release factor recruitment and stop codon recognition during translation termination. Nucl. Acids Res. 44(16), 7766–7776. https://doi.org/10.1093/nar/gkw635
Song H., Mugnier P., Das A.K., Webb H.M., Evans D.R., Tuite M.F., Hemmings B.A., Barford D. (2000) The crystal structure of human eukaryotic release factor eRF1—mechanism of stop codon recognition and peptidyl-tRNA hydrolysis. Cell. 100, 311–321.
Wangen J.R., Green R. (2020) Stop codon context influences genome-wide stimulation of termination codon readthrough by aminoglycosides. Elife. pii: e52611. doi: 7554/elife.52611
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология