Молекулярная биология, 2020, T. 54, № 6, стр. 997-1005
Филогеография и митохондриальный полиморфизм кижуча Азиатских стад
Д. А. Зеленина a, *, В. А. Сошнина a, b, А. А. Сергеев a
a Всероссийский научно-исследовательский институт рыбного хозяйства и океанографии
107140 Москва, Россия
b Кафедра ихтиологии биологического факультета Московского государственного университета
им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: zelenina@vniro.ru
Поступила в редакцию 06.07.2020
После доработки 20.07.2020
Принята к публикации 21.07.2020
Аннотация
Оледенения плейстоценовой эпохи существенным образом повлияли на генофонд многих видов, населяющих Северную Пацифику ‒ область земного шара с богатой гляциальной историей. Представленная работа посвящена изучению внутривидового полиморфизма кижуча и путей его расселения в пределах азиатской части ареала. Подобные задачи традиционно решают путем сравнения участков митохондриального генома. Мы провели исследование двух фрагментов митохондриальной ДНК (мтДНК): контрольного региона (D-loop) и гена цитохрома b (cytb). Показано, что заселению кижучем азиатского побережья Тихого океана предшествовала цепь последовательных миграционных событий из рефугиума, располагавшегося на североамериканском континенте к югу от ледникового щита, покрывавшего территорию современной Канады и юга Аляски. Низкий уровень генетического полиморфизма в азиатских популяциях кижуча объясним присутствием выраженного эффекта основателя, а не характерен для вида в целом.
ВВЕДЕНИЕ
Кижуч (Oncorhynchus kisutch Walbaum, 1792) ‒ представитель тихоокеанских лососей рода Oncorhynchus, группы ценных промысловых близкородственных видов, ареал которых охватывает северную часть Тихого океана и оба его побережья. На американском континенте кижуч обитает от Аляски до Калифорнии, на азиатском – от Чукотки до северной части Хабаровского края, острове Сахалин и северных островах Курильской гряды [1]. Среди тихоокеанских лососей кижуч считается одним из наиболее ценных видов, но работ по его изучению известно мало. К настоящему времени не проведено полномасштабных генетических исследований кижуча по всему ареалу обитания, в то время как знание генетической структуры вида необходимо для его рациональной эксплуатации и сохранения природного биоразнообразия.
Первые данные по популяционной генетике кижуча получены в конце прошлого века: в 1995 году был исследован полиморфизм аллозимных локусов в пяти азиатских популяциях кижуча [2], а годом позже – минисателлитных локусов в ряде популяций Британской Колумбии, Камчатки и Западной Аляски [3]. Дальнейшие исследования затрагивали, за единичным исключением, только американскую часть ареала, причем подавляющее большинство из них было сосредоточено на отдельных регионах североамериканского континента: Аляске, Орегоне, Калифорнии и Британской Колумбии [4‒9]. На азиатской части ареала в ряде рек магаданского побережья, Камчатки и Чукотки изучен полиморфизм митохондриального гена цитохрома b (cytb) [10], хотя популяции кижуча были представлены фрагментарно.
Сопоставление данных анализа полиморфизма контрольного региона митохондриальной ДНК (мтДНК) и микросателлитных локусов по всему североамериканскому ареалу вида с гляциальной историей Северной Пацифики позволило сделать заключение о едином происхождении всех современных популяций кижуча [11]. Во время ледникового максимума в эпоху плейстоцена этот вид сохранился к югу от ледникового щита, покрывавшего территорию современной Канады и юга Аляски, и в периоды отступления ледника последовательно заселял северные территории. Более подробно демографическая история вида охарактеризована по результатам масштабного геномного проекта [12]: помимо подтверждения роли южного Каскадийского рефугиума как источника значительной доли современного генетического разнообразия кижуча, были обозначены временные рамки поэтапного распространения вида и показано существование вторичных микрорефугиумов в северных районах.
Для выяснения происхождения азиатских популяций кижуча и оценки их генетического разнообразия мы исследовали митохондриальный полиморфизм в пределах всей азиатской части ареала вида. Сравнение гаплотипического состава азиатских и американских выборок при изучении контрольного региона позволило сделать предположение о путях расселения кижуча на евразийский континент [13]. Развернутое исследование полиморфизма гена cytb, наряду с дополнительным анализом ранее опубликованных данных, дает нам возможность предложить картину демографических процессов, сформировавших современную популяционную структуру кижуча азиатских стад.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материалы. Материал для проведения исследования собран в период с 2002 по 2019 гг. сотрудниками КамчатНИРО, МагаданНИРО, ХфТИНРО, ВНИРО, ТИНРО, ИПЭЭ РАН и кафедры ихтиологии МГУ им. М.В. Ломоносова во время нерестового хода в реках Дальнего Востока России, а также в ходе международной экспедиции на НИС “Профессор Кагановский” в заливе Аляска. Фрагменты тканей от живых особей фиксировали индивидуально в 96%-ном растворе этилового спирта. Характеристика материала представлена в табл. 1.
Таблица 1.
Характеристика исследованного материала
| Регион | Выборка | Число проанализированных образцов | |||
|---|---|---|---|---|---|
| Место сбора | Обозначение | Год | D-loop | cytb | |
| Восточная Камчатка | р. Апука | APU | 2007 | 35 | 39 |
| р. Камчатка | KAM | 2010 | 47 | 24 | |
| р. Кроноцкая | KRON | 2014 | 21 | 47 | |
| р. Авача | AVA | 2015 | 39 | 39 | |
| Западная Камчатка | р. Палана | PAL | 2015 | 45 | 40 |
| р. Утхолока | UTH | 2007 | 29 | 47 | |
| рр. Хайрюзова-Белоголовая | XABE | 2015 | 15 | 24 | |
| р. Крутогорова | KRUT | 2010 | 22 | 32 | |
| р. Кольа | KOL | 2007 | 27 | 44 | |
| р. Пымта | PYM | 2006 | 20 | 48 | |
| р. Большая | BOL | 2002 | – | 21 | |
| Магаданское побережье | р. Ола | OLA | 2018 | 48 | 47 |
| р. Тауй | TAUI | 2018 | 45 | 46 | |
| р. Охота | OKH | 2016 | 42 | 43 | |
| Сахалин | р. Лангери | LANG | 2017 | 42 | 46 |
| Американское побережье | Залив Аляска | AMER | 2019 | – | 48 |
Методы. Выделение и очистку ДНК из фрагментов плавников или мышечной ткани исследуемых особей кижуча проводили методом абсорбции на микроколонках АcroPrep™ 96 filter plаte, 1 mL ‒ 1.0 μm, glаss fiber mediа (“PALL”, США) по методике, описанной ранее [14].
Амплификацию и секвенирование контрольного региона (D-loop) и амплификацию гена cytb проводили в соответствии с ранее опубликованной методикой [15]. Для секвенирования гена cytb разработали внутренние праймеры: F15832 (5'-ATYACAAACCTCCTYTCYGC-3') и R16045 (5'-GGAGRTCTTTGTAYGAGAAGT-3'), ‒ использование которых позволяет получить полные нуклеотидные последовательности этого гена не только для кижуча, но и для других видов лососей рода Oncorhynchus. Секвенирование проводили на приборе ABI Prism Genetic Analyzer 3 500 (“Applied Biosystems”, США).
Для обработки результатов секвенирования и множественного выравнивания последовательностей использовали пакет программ Geneous 6.0.5 [16]. Для представления филогенетических отношений между гаплотипами применили метод максимальной экономии [17], реализованный в программе TCS [18]. Генетическое разнообразие оценивали в программе Arlequin v.3.5.1.3 [19].
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
В результате секвенирования двух участков мтДНК: полного гена cytb 587 особей и фрагмента контрольного региона (D-loop) 477 особей ‒ у азиатского кижуча выявлен низкий уровень митохондриального полиморфизма: 20 и 8 гаплотипов соответственно. Ввиду того, что для американской части ареала имелись данные только по контрольному региону [11], мы сочли целесообразным провести независимый анализ каждого из фрагментов.
Полиморфизм контрольного региона мтДНК
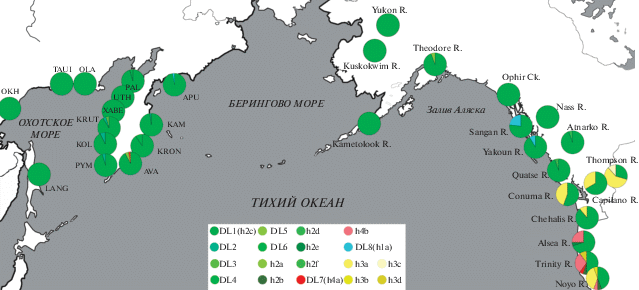
Исследуемый участок длиной 553 п.о. соответствовал позициям 15658–16660 полного митохондриального генома кижуча (GenBank #MH003640) [20] и позициям 7‒559 контрольного региона. У кижуча азиатских стад в этом фрагменте выявлено 7 полиморфных сайтов (табл. 2): подавляющее большинство особей несли гаплотип OKI-DL1 и только 7 гаплотипов отличались от массового одной заменой. Второй по частоте встречаемости гаплотип ‒ OKI-DL2 ‒ обнаружен в 3 популяциях западного побережья Камчатки и в выборке из бассейна реки Камчатка – крупнейшей реки полуострова, берущей начало в горах центральной его части, протекающей по Центральнокамчатской низменности и впадающей в Тихий океан (рис. 1).
Таблица 2.
Нуклеотидные замены в последовательностях контрольного региона (D-loop) азиатского кижуча
| Гаплотип | Genebank Acc. No. | Позиции нуклеотидова | ||||||
|---|---|---|---|---|---|---|---|---|
| 19 | 135 | 208 | 239 | 300 | 378 | 541 | ||
| OKI-DL1 | MK888691 | A | A | C | C | A | A | T |
| OKI-DL2 | MK888692 | . | T | . | . | . | . | . |
| OKI-DL3 | MK888689 | . | . | . | . | G | . | . |
| OKI-DL4 | MK888690 | . | . | . | . | . | . | C |
| OKI-DL5 | MK888688 | . | . | . | . | . | G | . |
| OKI-DL6 | MK888687 | G | . | . | . | . | . | . |
| OKI-DL7 | MK888685 | . | . | A | . | . | . | . |
| OKI-DL8 | MK888686 | . | . | . | T | . | . | . |
Согласно данным Smith и соавт. [11], на американском побережье также преобладает гаплотип OKI-DL1 (h2c), что наиболее выражено в северной части ареала; при этом авторы выделяют четыре гаплогруппы, две из которых (h3 и h4) присутствуют только в южной части ареала, а третья (h1) – на островах у побережья Британской Колумбии (рис. 1). На азиатском побережье доминирует гаплогруппа h2, однако на северо-востоке Камчатки единично встречается гаплотип OKI-DL8 (h1a), а на юго-востоке – OKI-DL7 (h4a). Наиболее высокий уровень нуклеотидного и гаплотипического разнообразия выявлен в выборках из южной части американского побережья (Калифорния, Орегон, Вашингтон и юг Британской Колумбии), а по мере продвижения на север эти показатели снижаются ‒ вплоть нулевых значений в выборках с Аляски (табл. 2). На азиатской части ареала определенный генетический полиморфизм выявлен в подавляющем большинстве популяций полуострова Камчатка; при этом выборки из других дальневосточных регионов оказались мономорфными (табл. 3).
Таблица 3.
Нуклеотидный (π) и гаплотипический (h) полиморфизм контрольного региона в выборках кижуча
| Регион | Выборка | Na | Hb | π × 100с | h (SE)d |
|---|---|---|---|---|---|
| Восточная Камчатка | APU | 35 | 2 | 0.0103 | 0.057 (0.053) |
| KAM | 47 | 2 | 0.0077 | 0.043 (0.040) | |
| KRON | 21 | 2 | 0.0327 | 0.181 (0.104) | |
| AVA | 39 | 3 | 0.0273 | 0.148 (0.075) | |
| Западная Камчатка | PAL | 45 | 2 | 0.0157 | 0.087 (0.056) |
| UTH | 29 | 1 | 0.000 | 0.000 (0.000) | |
| XABE | 15 | 1 | 0.000 | 0.000 (0.000) | |
| KRUT | 22 | 3 | 0.0329 | 0.178 (0.106) | |
| KOL | 27 | 2 | 0.0258 | 0.143 (0.086) | |
| PYM | 20 | 2 | 0.0181 | 0.100 (0.088) | |
| Магаданское побережье | OLA | 48 | 1 | 0.000 | 0.000 (0.000) |
| TAUI | 45 | 1 | 0.000 | 0.000 (0.000) | |
| OKH | 42 | 1 | 0.000 | 0.000 (0.000) | |
| Сахалин | LANG | 42 | 1 | 0.000 | 0.000 (0.000) |
| Аляскаe | Yukon R. | 19 | 1 | 0.000 | 0.000 (0.000) |
| Kuskokwim R. | 20 | 1 | 0.000 | 0.000 (0.000) | |
| Kametolook R. | 19 | 1 | 0.000 | 0.000 (0.000) | |
| Theodore R. | 18 | 2 | 0.021 | 0.111 (0.096) | |
| Ophir Ck. | 16 | 1 | 0.000 | 0.000 (0.000) | |
| Британская Колумбия (север)e | Nass R. | 19 | 1 | 0.000 | 0.000 (0.000) |
| Atnarko R. | 17 | 2 | 0.022 | 0.118 (0.101) | |
| Sangan R. | 13 | 2 | 0.073 | 0.385 (0.132) | |
| Yakoun R. | 23 | 2 | 0.032 | 0.166 (0.098) | |
| Quatse R. | 18 | 2 | 0.021 | 0.111 (0.096) | |
| Британская Колумбия (юг)e | Conuma R. | 18 | 2 | 0.099 | 0.523 (0.048) |
| Capilano R. | 18 | 2 | 0.089 | 0.471 (0.082) | |
| Thompson R. | 17 | 3 | 0.126 | 0.588 (0.093) | |
| Калифорния, Орегон, Вашингтонe | Chehalis R. | 18 | 2 | 0.040 | 0.209 (0.116) |
| Alsea R. | 20 | 3 | 0.188 | 0.468 (0.104) | |
| Trinity R. | 20 | 5 | 0.266 | 0.679 (0.080) | |
| Noyo R. | 18 | 5 | 0.181 | 0.680 (0.074) |
a Объем выборки. bЧисло гаплотипов в выборке. cНуклеотидное разнообразие. dГаплотипическое разнообразие, в скобках указана стандартная ошибка (SE). eДанные по выборкам с американского побережья приведены из работы [11].
Полиморфизм гена cytb
Исследуемый участок мтДНК – полноразмерная последовательность гена cytb длиной 1141 п.о. – соответствовал позициям 14 376‒15 516 полного митохондриального генома кижуча (GenBank #MH003640) [20]. В пределах азиатской части ареала кижуча найдено 20 гаплотипов; в дополнение к этому 5 гаплотипов обнаружено в выборке из залива Аляска. Сравнительный анализ последовательностей 25 гаплотипов выявил 27 полиморфных сайтов, в 9 из которых нуклеотидные замены были несинонимичными (табл. 4), а также существование не менее двух гаплогрупп, различающихся на 5 нуклеотидных замен.
Таблица 4.
Нуклеотидные замены в последовательностях гена cytb кижуча
| Гаплотип | Genebank Acc. No. | Позиции нуклеотидова | ||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 1 | 1 | 1 | 1 | ||||||||||||||||||||||||
| 1 | 1 | 1 | 2 | 2 | 3 | 3 | 4 | 4 | 4 | 5 | 5 | 5 | 6 | 6 | 7 | 7 | 9 | 9 | 9 | 9 | 0 | 0 | 0 | 0 | 0 | |||
| 7 | 5 | 6 | 9 | 2 | 5 | 3 | 5 | 5 | 8 | 9 | 0 | 5 | 6 | 0 | 2 | 8 | 9 | 0 | 0 | 7 | 8 | 1 | 2 | 5 | 6 | 7 | ||
| 4 | 9 | 4 | 6 | 2 | 2 | 6 | 1 | 6 | 0 | 8 | 8 | 8 | 2 | 6 | 7 | 6 | 8 | 5 | 7 | 9 | 4 | 7 | 9 | 1 | 5 | 8 | ||
| OKI-A1 | MT648531 | C | G | A | G | T | C | C | G | T | G | C | G | C | G | A | G | G | T | T | G | G | G | G | G | G | T | G |
| OKI-A2 | MT648532 | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A3 | MT648533 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . |
| OKI-A4 | MT648534 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A5 | MT648535 | . | . | . | . | . | . | . | . | . | . | . | A* | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A6 | MT648536 | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A7 | MT648537 | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A8 | MT648538 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . |
| OKI-A9 | MT648539 | T* | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A10 | MT648540 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A* | C | . |
| OKI-A11 | MT648541 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . |
| OKI-A12 | MT648542 | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A13 | MT648543 | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-A14 | MT648544 | . | . | . | . | . | . | . | . | . | . | . | . | . | A* | . | . | . | . | . | . | . | . | . | . | . | . | . |
| OKI-B1 | MT648545 | . | A | . | . | . | . | . | A | . | A | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | A* |
| OKI-B2 | MT648546 | . | A | . | . | . | . | . | A | . | A | . | . | . | . | G | . | A | . | . | . | . | . | . | . | . | . | A* |
| OKI-B3 | MT648547 | . | A | . | . | . | . | . | A | . | A | . | . | . | . | G | . | A | . | . | A* | . | . | . | . | . | . | A* |
| OKI-B4 | MT648548 | . | A | . | A* | . | . | . | A | . | A | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | A* |
| OKI-B5 | MT648549 | . | A | . | . | . | . | . | A | . | A | . | . | . | . | G | . | . | . | . | . | A* | . | . | . | . | . | A* |
| OKI-B6 | MT648550 | . | A | T* | . | . | . | . | A | . | A | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | A* |
| OKI-B7 | MT648551 | . | A | . | . | C | . | . | A | . | A | . | . | . | . | G | . | A | . | . | . | . | . | . | . | . | . | A* |
| OKI-B8 | MT648552 | . | A | . | . | . | . | . | A | . | A | . | . | T | . | G | . | . | . | . | . | . | . | . | . | . | . | A* |
| OKI-B9 | MT648553 | . | A | . | . | . | . | . | A | . | A | . | . | . | . | G | . | . | . | . | . | . | A | . | . | . | . | A* |
| OKI-C1 | MT648554 | . | . | . | . | . | . | . | A | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A* |
| OKI-C2 | MT648555 | . | . | . | . | . | . | . | A | C | A | . | . | . | . | . | . | A | . | . | . | . | . | . | A | . | . | A* |
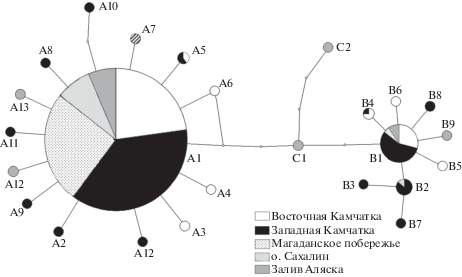
В сети гаплотипов, построенной на основании TCS-алгоритма (рис. 2), выявлено присутствие трех гаплогрупп, при этом расстояние между центральными гаплотипами двух наиболее генетически удаленных групп А и В составляет 5 нуклеотидных замен. В обеих гаплогруппах обнаружены редкие и уникальные гаплотипы, отличающиеся от центрального на одну‒две нуклеотидные замены.
Рис. 2.
Филогенетическая сеть гаплотипов cytb кижуча. Для каждого гаплотипа показана доля особей из различных регионов.
Сравнительный анализ частот гаплотипов выявил их неравномерное распределение на азиатской части ареала кижуча (рис. 3). В большинстве популяций полуострова Камчатка (за исключением двух выборок: APU и KRON) доля гаплогруппы В составила от 16% (KRUT) до 33% (КАМ); в популяциях острова Сахалин и материкового побережья Охотского моря эта гаплогруппа присутствовала лишь у 1‒2 особей. Третий по частоте встречаемости гаплотип OKI-B2 выявлен во всех выборках с западного побережья Камчатки и в популяции острова Сахалин. В выборке из смешанного морского скопления из залива Аляска обнаружена гаплогруппа С, занимающая промежуточное положение между группами А и В и отличающаяся от них на 3 и 2 замены соответственно (рис. 2 и 3).
Рис. 3.
Распределение галотипов cytb кижуча на ареале. Названия выборок приведены в соответствие с табл. 1. Римскими цифрами обозначены последовательности из GenBank: I – KU761856, II – KU761857, III – MH003640, IV – JX960809, V – JX960810, VI – KU872712, VII – MF621749, VIII – MF621751.
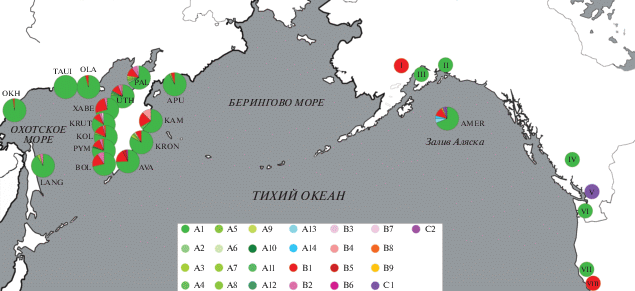
В попытке компенсировать отсутствие выборок с американского побережья мы локализовали на карте единичные образцы из GenBank, для которых известна полная последовательность гена cytb (I‒VIII на рис. 3). Значения гаплотипического и нуклеотидного разнообразия в выборках кижуча приведены в табл. 5.
Таблица 5.
Генетический полиморфизм азиатского кижуча на основании анализа гена cytb
| Выборка | Na | Hb | Sc | π (SE) × 100d | h (SE)e |
|---|---|---|---|---|---|
| APU | 39 | 3 | 6 | 0.0483 (0.0451) | 0.1484 (0.0753) |
| KAM | 24 | 4 | 7 | 0.2305 (0.1422) | 0.5725( 0.0954) |
| KRON | 47 | 5 | 7 | 0.0842 (0.0649) | 0.3080 (0.0849) |
| AVA | 39 | 4 | 7 | 0.1847 (0.1170) | 0.4224 (0.0860) |
| PAL | 40 | 8 | 11 | 0.1948 (0.1219) | 0.5333 (0.0899) |
| UTH | 47 | 7 | 10 | 0.1487 (0.0983) | 0.4070 (0.0872) |
| XABE | 24 | 3 | 6 | 0.1962 (0.1248) | 0.4529 (0.0948) |
| KRUT | 32 | 4 | 7 | 0.1354 (0.0925) | 0.2863 (0.1000) |
| KOL | 44 | 5 | 8 | 0.1454 (0.0968) | 0.3552 (0.0855) |
| BOL | 21 | 3 | 6 | 0.2037 (0.1294) | 0.4667 (0.1126) |
| PYM | 48 | 5 | 9 | 0.1545 (0.1012) | 0.3626 (0.0831) |
| OLA | 47 | 2 | 5 | 0.0365 (0.0377) | 0.0833 (0.0539) |
| TAUI | 46 | 1 | 0 | 0.0000 (0.0000) | 0.0000 (0.0000) |
| OKH | 43 | 2 | 5 | 0.0204 (0.0268) | 0.0465 (0.0439) |
| LANG | 47 | 3 | 6 | 0.0504 (0.0461) | 0.1628 (0.0711) |
| AMER | 48 | 7 | 11 | 0.1654 (0.1067) | 0.5142 (0.0825) |
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В результате сравнения результатов анализа полиморфизма D-loop в азиатских популяциях кижуча с данными Smith с соавт. [11] показано, что заселение азиатского побережья происходило на финальном этапе цепочки поэтапного распространения кижуча из Каскадийского рефугиума. Из данных, приведенных на рис. 1 и в табл. 3, можно сделать вывод о постепенном снижение гаплотипического разнообразия как следствия прохождения вида через “бутылочные горлышки”, преодолеть которые смогла единственная гаплогруппа h2. Мы не нашли однозначного объяснения обнаружению двух особей с гаплотипами из групп, не присутствующих в северных частях ареала: OKI-DL8 (h1a) в реке Апука и OKI-DL7 (h4a) в реке Авача. Однако, учитывая то, что обе реки расположены на восточном побережье полуострова Камчатка, можно предположить их появление как результат стреинга, то есть случайного возвращения на нерест не в родную реку, или же как следствие независимо произошедших мутационных событий.
Анализ нуклеотидных последовательностей гена cytb в азиатских популяциях кижуча выявил присутствие двух гаплогрупп, расстояние между центральными гаплотипами которых составляет 5 нуклеотидных замен, а редкие и уникальные гаплотипы отличаются от своих предшественников в большинстве случаев на одну замену (табл. 4). Такая звездчатая топология сети гаплотипов указывает на эффект основателя и характерна для молодых видов или видов, прошедших в некоторой части своего ареала через “бутылочное горлышко”. Таким образом, мы предполагаем, что в заселении азиатского побережья принимало участие ограниченное число производителей, несущих преимущественно массовые гаплотипы OKI-A1 и OKI-B1, а все локальное генетическое разнообразие было сформировано впоследствии в результате мутаций. В подтверждение этому можно отметить, что ни один из редких гаплотипов, присутствовавших на азиатской части ареала, не встречался в заливе Аляска.
Обнаружение в заливе Аляска гаплотипа OKI-C1, занимающего промежуточное положение между двумя основными гаплогруппами, дополнительно подтверждает концепцию последовательного расселения вида из Каскадийского рефугиума, тем более что ранее этот гаплотип регистрировали в южной части Британской Колумбии (рис. 3), расположенной в начале маршрута постледниковых миграций вида. Учитывая значительно более высокий уровень генетического полиморфизма в южных регионах североамериканского побережья, можно объяснить обнаруженный нами низкий внутривидовой полиморфизм кижуча эффектом основателя при формировании азиатских популяций, а не особенностью вида в целом.
Как ранее показали Rougemont и соавт. [12], расселение кижуча в северном направлении произошло в момент временного отступления ледника 55–25 тыс. лет назад, в результате чего сформировались локальные микрорефугиумы, в которых происходили свои мутационные процессы. Существование ряда редких и уникальных гаплотипов, характерных только для азиатской части ареала, позволяет предположить существование таких рефугиумов и в Азии. Возможно, локальный рефугиум существовал в центральной части полуострова Камчатка, где и образовалось большинство гаплотипов, впоследствии распространившихся по азиатской части ареала. Наиболее массовый из них гаплотип OKI-B2 присутствует во всех популяциях западной Камчатки и на Сахалине, из чего можно сделать вывод о заселении Сахалина кижучем западнокамчатского побережья. Низкий уровень генетического разнообразия в популяциях кижуча острова Сахалин и материкового побережья Охотского моря может быть следствием прохождение ими “бутылочного горлышка” на этапе формирования. Более детальное представление о демографических процессах в популяциях азиатского кижуча будет получено в ближайшее время на основании данных геномного генотипирования.
Авторы признательны сотрудникам ФГБНУ “ВНИРО” и его Камчатского, Магаданского и Хабаровского филиалов, принимавших участие в сборе материала на Камчатке и материковом побережье Охотского моря; К.В. Кузищину (каф. ихтиологии МГУ) за предоставленный материал из рек Коль и Утхолок; Е.А. Кирилловой (Институт проблем экологии и эволюции им. А.Н. Северцова РАН) за сбор образцов на острове Сахалин; А.А. Сомову (Тихоокеанский филиал ВНИРО) за сбор материала в заливе Аляска. Авторы благодарят А.А. Волкова (отдел молекулярной генетики ФГБНУ “ВНИРО”) за разработку праймеров для секвенирования гена cytb.
Работа выполнена при финансовой поддержке Российского фонда фундаментальных исследований (проекты 18-04-01375-a и 20-04-00572-a).
Все процедуры, проведенные с участием животных, соответствовали этическим стандартам учреждений или принятой практике таких исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Зорбиди Ж.Х. (2010) Кижуч азиатских стад. Петропавловск-Камчатский: КамчатНИРО.
Пустовойт С.П. (1995) Особенности генетической структуры кижуча Oncorhynchus kisutch (Walbaum). Генетика. 31, 709–714.
Beacham T.D., Miller K.M., Withler R.E. (1996) Minisatellite DNA variation and stock identification of coho salmon. J. Fish Biol. 49, 411–429.
Beacham T.D., Candy J.R., Supernault K.J., Ming T., Deagle B., Schultz A., Tuck D., Kaukinen K., Irvine J.R., Miller K.M., Withler R.E. (2001) Evaluation and application of microsatellite and major histocompatibility complex variation for stock identification of coho salmon in British Columbia. Trans. Am. Fish. Soc. 130, 1116–1155.
Beacham T.D., Wetklo M., Deng L., MacConnachie C. (2011) Coho salmon population structure in North America determined from microsatellites. Trans. Am. Fish. Soc. 140, 253–270.
Beacham T.D., Candy J.R., Wallace C., Wetklo M., Deng L., MacConnachie C. (2012) Microsatellite mixed-stock identification of coho salmon in British Columbia. Marine and Coastal Fisheries: Dynamics, Management, Ecosystem Sci. 4, 85–100.
Gharrett A.J., Gray A.K., Brykov V. (2001) Phylogeographic analysis of mitochondrial DNA variation in Alaskan coho salmon, Oncorhynchus kisutch. Fish. Bull. 99, 528–544.
Olsen J.B., Miller S.J., Spearman W.J., Wenburg J.K. (2003) Patterns of intra- and inter-population genetic diversity in Alaskan coho salmon: implications for conservation. Conserv. Genet. 4, 557–569.
Gilbert-Horvath E.A., Pipal K.A., Spence B.C., Williams T.H., Garza J.C. (2016) Hierarchical phylogeographic structure of coho salmon in California. Trans. Am. Fish. Soc. 145, 1122–1138.
Бачевская Л.Т., Иванова Г.Д., Переверзева В.В., Агапова Г.А. (2017) Генетическая структура кижуча Onchorhynchus kisutch из рек северо-востока России по данным о полиморфизме гена цитохрома b митохондриальной ДНК. Известия РАН. Серия биологическая. 44, 593–599.
Smith C.T., Nelson R.J., Wood C.C., Koop B.F. (2001) Glacial biogeography of North American coho salmon (Oncorhynchus kisutch). Mol. Ecol. 10, 2775–2785.
Rougemont Q., Moore J.-S., Leroy T., Normandeau E., Rondeau E.B., Withler R E., Van Doornik D.M., Crane P.A., Naish K.A., Garza J.C., Beacham T.D., Koop B.F., Bernatchez L. (2020) Demographic history shaped geographical patterns of deleterious mutation load in a broadly distributed Pacific salmon. bioRxiv. 732750. https://doi.org/10.1101/732750
Soshnina V.A., Sergeev A.A. and Zelenina D.A. (2019) The phylogeography of coho salmon based on mtDNA control region variation: Asian populations descend from the Beringian Refugium. In: XVI European Congress of Ichthyology, Conference Abstracts. Front. Mar. Sci. https://doi.org/10.3389/conf.fmars.2019.07.00119
Ivanova N.V., deWaard J., Hebert P.D.N. (2006) An inexpensive, automation-friendly protocol for recove-ring high-quality DNA. Mol. Ecol. Notes. 6, 998–1002.
Сошнина В.А., Павлов С.Д., Зеленина Д.А. (2016) Генетическое разнообразие гольцов Командорских островов по результатам исследования митохондриальной ДНК. Генетика. 52, 1336–1341.
Kearse M., Moir R., Wilson A., Stones-Havas S., Cheung M., Sturrock S., Buxton S., Cooper A., Markowitz, S., Duran C., Thierer T., Ashton B., Meintjes P., Drummond A. (2012) Geneious Basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data. Bioinformatics. 28, 1647‒1649.
Templeton A.R., Crandall K.A., Sing C.F. (1992) A cladistic analysis of phenotypic associations with haplotypes inferred from restriction endonuclease mapping and DNA sequence data. III. Cladogram estimation. Genetics. 132, 619–633.
Clement M., Posada D., Crandall K.A. (2000) TCS: a computer program to estimate gene genealogies. Mol. Ecol. 9, 1657–1659.
Excoffier L., Lischer H.E.L. (2010) Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows. Mol. Ecol. Res. 10, 564–567.
de Flamingh A., Mallott E.K., Roca A.L., Boraas A.S., Malhi R.S. (2018) Species identification and mitochondrial genomes of ancient fish bones from the Ri-verine Kachemak tradition of the Kenai Peninsula, Alaska. Mitochondrial DNA Part B. 3, 409‒411.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология