Молекулярная биология, 2021, T. 55, № 1, стр. 54-63
Отделы центральной нервной системы мыши отличаются по количеству транскриптов протеасомных генов
С. Ю. Фуников a, Д. С. Спасская a, А. В. Буров a, b, Е. В. Тетерина c, А. А. Устюгов c, В. Л. Карпов a, А. В. Морозов a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
c Институт физиологически активных веществ Российской академии наук
142432 Московская область, Черноголовка, Россия
* E-mail: Runkel@inbox.ru
Поступила в редакцию 18.06.2020
После доработки 29.06.2020
Принята к публикации 30.06.2020
Аннотация
Протеасома ‒ мультисубъединичный белковый комплекс, осуществляющий деградацию большинства внутриклеточных белков. Из 14 субъединиц 20S протеасом три (β1, β2 и β5) обладают каталитической активностью и осуществляют гидролиз пептидной связи после кислых, основных и гидрофобных аминокислот соответственно. В составе протеасом конститутивные каталитические субъединицы β1, β2 и β5 могут быть заменены так называемыми иммунными субъединицами β1i, β2i и β5i. Однако не всегда в составе протеасом присутствуют сразу все иммунные субъединицы; известны протеасомы, содержащие как иммунные, так и конститутивные субъединицы. Встраивание иммунных субъединиц меняет профиль пептидов, производимых протеасомами, что имеет большое значение в процессах презентации антигенов и клеточном ответе на стресс и, по-видимому, в целом ряде внутриклеточных сигнальных путей. Нами разработана система для количественного определения абсолютных уровней экспрессии генов конститутивных и иммунных субъединиц протеасом мыши, основанная на ПЦР в реальном времени. С помощью полученной системы оценены уровни экспрессии генов субъединиц протеасом в тканях центральной нервной системы (ЦНС) мыши. Выявлено, что количество транскриптов каталитических субъединиц протеасом в разных отделах ЦНС существенно отличается. Приведенные результаты позволяют подразделить исследованные отделы мозга на две категории: условно с “высокими” (кора головного мозга и спинной мозг) и “низкими” (гиппокамп и мозжечок) уровнями экспрессии генов субъединиц протеасом. Кроме того, можно выделить отделы с близкими и существенно различающимися профилями экспрессии генов каталитических субъединиц протеасом. Так, профили экспрессии генов в коре, спинном мозге и мозжечке похожи друг на друга, однако отличаются от профиля экспрессии в гиппокампе. На основании полученных данных можно сделать заключение о вероятных различиях в пуле протеасом, а также в функциональной нагрузке на убиквитин-протеасомную систему в разных отделах ЦНС.
ВВЕДЕНИЕ
20S протеасома представляет собой центральный элемент убиквитин-протеасомной системы (УПС), обеспечивающий деградацию большинства внутриклеточных белков. Протеасома состоит из 28 белков, организованных в четыре гептамерных кольца, уложенных друг на друга. Четырнадцать из этих белков относятся к альфа-, а другие 14 ‒ к бета-субъединицам; при этом каждая субъединица присутствует в составе протеасомы в двух копиях [1]. Таким образом, в составе 20S протеасомы присутствует 7 различных альфа- и 7 различных бета-субъединиц. Альфа-субъединицы кодируются генами PSMA1-7, а бета-субъединицы – группой генов PSMB1-7. Три из семи бета-субъединиц обладают каталитической активностью и, находясь в составе протеасом, гидролизуют пептидные связи после кислых (β1, кодируется геном PSMB6), основных (β2, кодируется геном PSMB7) и гидрофобных аминокислотных остатков (β5, кодируется геном PSMB5) [2]. Осуществляя деградацию внутриклеточных белков, УПС участвует в регуляции практически всех фундаментальных клеточных процессов, включая контроль клеточного цикла, регуляцию транскрипции и трансляции, ответ на биотические и абиотические стрессовые факторы [3]. Кроме того, УПС играет важную роль в патофизиологии опухолевого роста, аутоиммунных и нейродегенеративных заболеваний [3‒5]. УПС обладает пластичностью, что выражается в наличии различных форм протеасом, а также в тонкой регуляции активности протеасом для решения специфических задач, стоящих перед клеткой. Так, протеасомы могут взаимодействовать с белками-кофакторами, а субъединицы протеасом могут подвергаться посттрансляционным модификациям, влияющим на активность и субстратную специфичность комплекса [6]. Еще один важный механизм адаптации протеасом для выполнения специфических задач связан с заменой конститутивных каталитических субъединиц β1, β2 и β5 так называемыми иммунными субъединицами β1i, β2i и β5i (кодируются генами PSMB9, PSMB10 и PSMB8 соответственно). Это повышает эффективность деградации окисленных и поврежденных белков [7], а также отражается на наборе пептидов, производимых протеасомами. Иммунные протеасомы обладают повышенной химотрипсинподобной активностью и поэтому более эффективно производят пептиды с гидрофобными аминокислотами на С-конце, что необходимо для их эффективного связывания с молекулами главного комплекса гистосовместимости первого класса (MHC-I) и, как следствие, презентации антигенов [8]. По-видимому, это одна из причин большого количества таких протеасом в клетках иммунной системы. Этим же можно объяснить и стимуляцию синтеза иммунных субъединиц в соматических клетках при развитии воспалительных процессов. Однако, как оказалось, в клетках различных органов, таких как печень, почки, различные отделы кишечника, в норме присутствует большое количество промежуточных протеасом, содержащих как иммунные, так и конститутивные каталитические субъединицы [9]. Роль таких протеасом до конца не ясна, однако, учитывая, что при встраивании иммунных субъединиц происходят изменения в наборе производимых протеасомами пептидов, а также вероятную биологическую активность последних, можно предположить, что они выполняют регуляторные тканеспецифические функции [10]. Таким образом, экспрессия различных каталитических субъединиц протеасом ‒ важный параметр, отражающий не только физиологическое состояние органа и ткани, наличие воспаления или стресса, но, скорее всего, и пока до конца неохарактеризованные регуляторные механизмы, поддерживающие тканеспецифические функции в клетках. В этой связи количественная оценка и сравнительный анализ уровней экспрессии протеасомных генов представляет особый интерес.
Ранее, на основе метода ПЦР в реальном времени, нами была разработана система, позволяющая количественно оценивать и сравнивать между собой уровни экспрессии протеасомных генов человека [11]. Теперь нами разработана новая система для проведения ПЦР в реальном времени и измерения абсолютных уровней экспрессии генов протеасом мыши ‒ основного модельного объекта для изучения особенностей патогенеза различных заболеваний, включая нейродегенеративные патологии человека. Используя данную систему, мы провели эксперименты по сравнительному анализу уровней экспрессии протеасомных субъединиц в образцах различных отделов ЦНС мышей линии C57Bl.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Экспериментальные животные и диссекция отделов ЦНС. Мышей линии C57Bl содержали в SPF (Specific Pathogen Free) виварии не более 5 особей в клетке в условиях искусственно регулируемого светового дня (12 ч темного и 12 ч светлого времени) при температуре 22‒26°C и свободном доступе к корму и воде. Работы с животными проводили в соответствии с “Правилами лабораторной практики в Российской Федерации” от 1.04.2016 № 199н.
Для экспериментов были отобраны только 60-дневные самцы. Эвтаназию животных проводили методом цервикальной дислокации, извлекали головной мозг и отделы спинного мозга и препарировали анализируемые ткани на закрытой чашке Петри со льдом. Диссектированные отделы ЦНС замораживали в жидком азоте и помещали на хранение при ‒80°C.
Выделение тотальной РНК и синтез кДНК. Экстракцию тотальной РНК проводили с помощью реактива Extract RNA (“Евроген”, Россия), следуя инструкции производителя. Спиртовое осаждение РНК проводили в присутствии 40 мкг гликогена (“Thermo Fisher Scientific”, США). Концентрацию РНК определяли на спектрофотометре NanoDrop 1000 (“Thermo Fisher Scientific”). Качество РНК оценивали с помощью капиллярного электрофореза на приборе Agilent Bioanalyzer 2100 и набора Agilent RNA 6000 Nano kit (“Agilent Technologies”, США). Число целостности РНК (RNA integrity number; RIN) для выделенной тотальной РНК составляло не менее 8 единиц.
Далее тотальную РНК обрабатывали Turbo DNase из набора Turbo DNA-free kit (“Ambion”, США) и проводили реакцию обратной транскрипции, используя 1 мкг тотальной РНК, олиго(dT)-праймер и обратную транскриптазу MMLV из набора MMLV RT kit (“Евроген”, Россия). Реакцию проводили при 42°C в течение 80 мин, после чего образцы инкубировали 10 мин при 70°С для инактивации ревертазы.
Подбор праймеров, амплификация фрагментов целевых генов, клонирование и выделение плазмидной ДНК. Последовательности транскриптов протеасомных субъединиц мыши взяты из GenBank. Подбор геноспецифичных праймеров проводили с помощью программы NCBI Primer Blast (PMID: 22708584) так, чтобы прямой и обратный праймеры соответствовали фрагментам из разных экзонов мРНК, длина ампликона составляла 90‒200 п.н., температура плавления (Tm) ‒ 60‒62°C. Праймеры, использованные в работе, представлены в табл. 1.
Таблица 1.
Олигонуклеотиды, использованные в работе
| Праймер | Нуклеотидная последовательность 5′ → 3′ | Длина, п.н. | Положение по референсной мРНК | Размер ампликона, п.н. | Термодинамические параметрыа | ||
|---|---|---|---|---|---|---|---|
| Tm, °C | Гомодимеры
и шпильки, ΔG не менее, ккал/моль |
Гетеродимеры, ΔG не менее, ккал/моль |
|||||
| PSMB5 for | TAAGGAACGCATCTCGGTCG | 20 | NM_011186.1 | 148Экзон–экзон | 63.6 | ‒3.61 | ‒7.93 |
| п.н. 391‒410 | |||||||
| PSMB5 rev | GTTCCCCTCGCTGTCTACG | 19 | NM_011186.1 | 63.6 | ‒3.61 | ||
| п.н. 538–520 | |||||||
| PSMB6 for | ACTGCCAATGCTCTCGCTTT | 20 | NM_008946.4 | 197Экзон–экзон | 64.8 | ‒3.61 | ‒5.02 |
| п.н. 605–624 | |||||||
| PSMB6 rev | CGTCGGTATGGACCATCCTT | 20 | NM_008946.4 | 63.3 | ‒6.5 | ||
| п.н. 801–782 | |||||||
| PSMB7 for | CATGGGTTCTGGCTCCTTGG | 20 | NM_011187.1 | 105Экзон–экзон | 64.6 | ‒5.38 | ‒7.81 |
| п.н. 511–530 | |||||||
| PSMB7 rev | GCTGCAATAGCCTCACTCAC | 20 | NM_011187.1 | 62.8 | ‒7.05 | ||
| п.н. 615–596 | |||||||
| PSMB8 for | TGCCTATGGGGTGATGGACA | 20 | NM_010724.2 | 187Экзон–экзон | 65.2 | ‒3.14 | ‒4.64 |
| п.н. 199–218 | |||||||
| PSMB8 rev | CGTCTTCCTTCATGTGGTAC | 20 | NM_010724.2 | 60.0 | ‒5.38 | ||
| п.н. 385–366 | |||||||
| PSMB9 for | ACCGTGAGGACTTGTTAGCG | 20 | NM_013585.2 | 156Экзон–экзон | 63.9 | ‒3.61 | ‒5.99 |
| п.н. 350–369 | |||||||
| PSMB9 rev | AAGCTGCGTCCACATAACCA | 20 | NM_013585.2 | 64.3 | ‒6.34 | ||
| п.н. 505–486 | |||||||
| PSMB10 for | GGCTTGTGTTCCGAGATGG | 20 | NM_013640.3 | 94Экзон–экзон | 62.6 | ‒3.61 | ‒7.81 |
| п.н. 803–822 | |||||||
| PSMB10 rev | CAGCCCCACAGCAGTAGATT | 19 | NM_013640.3 | 63.9 | ‒3.14 | ||
| п.н. 896‒878 | |||||||
а Термодинамические параметры праймеров оценивали с помощью приложения IDT OligoAnalizer Tool (https://eu.idtdna.com/pages) при содержании солей [Na+] = = 50 мМ, [Mg2+] = 3 мМ, [dNTP] = 0.8 мМ.
Амплификацию фрагментов мРНК генов субъединиц протеасом проводили с матрицы кДНК, приготовленной из тотальной РНК. Тотальную РНК выделяли из клеток образца печени мыши. Амплификацию проводили с помощью смеси термостабильных ДНК-полимераз из набора Encyclo Plus PCR kit (“Евроген”), обладающих высокопроцессивной 5′ → 3′ ДНК-полимеразной активностью, а также корректирующей 3′ → 5′ экзонуклеазной активностью, на амплификаторе MJ Mini (“Bio-Rad”, США). Число циклов амплификации рассчитывали эмпирически в соответствии с уровнем экспрессии гена. Далее полученные ампликоны очищали от компонентов реакции ПЦР с помощью QIAquick Gel Extraction Kit (“QIAGEN”, Германия) и клонировали в вектор pAL2-T (“Евроген”). Для трансформации использовали компетентные клетки Escherichia сoli, штамм DH5α. Плазмидную ДНК выделяли с помощью набора Plasmid Miniprep (“QIAGEN”). Наличие вставки фрагментов мРНК протеасомных субъединиц проверяли с помощью секвенирования по Сэнгеру.
Определение абсолютного количества транскриптов субъединиц протеасом с помощью ПЦР с использованием стандартной кривой. ПЦР в реальном времени проводили на приборе ABI PRISM® 7500 (“Applied Biosystems”), используя реакционную смесь qPCRmix-HS LowROX (“Евроген”). В каждую реакцию (V = 15 мкл) добавляли по 3 мкл 5× реакционной смеси, по 0.15 мкл каждого праймера в концентрации 10 мкМ и по 1 мкл кДНК (1/20 часть от общего объема синтезированной кДНК). Детекцию продуктов амплификации осуществляли с помощью интеркалирующего красителя Eva Green (“Biotium”, США). Число копий мРНК определяли в абсолютных значениях, использую стандартные разведения плазмидной ДНК. Число копий плазмидной ДНК рассчитывали по формуле:
(1)
$N = {{(m \times 6.022 \times {{{10}}^{{23}}})} \mathord{\left/ {\vphantom {{(m \times 6.022 \times {{{10}}^{{23}}})} {(l \times {{{10}}^{9}} \times 650)}}} \right. \kern-0em} {(l \times {{{10}}^{9}} \times 650)}},$Разведение плазмидной ДНК проводили в буфере TN (10 мМ Трис-HCl, pH 7.6, 10 мМ NaCl), содержащем ДНК из спермы лосося (“Sigma”, США) в концентрации 100 нг/мкл. Эффективность амплификации определяли по наклону стандартной кривой в серии последовательных разведений плазмидной ДНК по формуле:
где E ‒ эффективность амплификации (%), tgα ‒ tg угла наклона кривой.Эффективность амплификации составляла не менее 90%. Исходя из того, что на одну ПЦР использовали 1/20 часть кДНК, синтезированной из 1 мкг тотальной РНК, итоговое число копий протеасомных субъединиц рассчитывали умножением полученного значения на 20. Для оценки значимости наблюдаемых изменений использовали однофакторный дисперсионный анализ (ANOVA) с применением критерия Тьюки в качестве поправки на множественные сравнения.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЯ
Разработка системы ПЦР в реальном времени
Для анализа уровней экспрессии генов PSMB5, PSMB6, PSMB7, PSMB8, PSMB9 и PSMB10 каталитических субъединиц протеасом были подобраны специфические олигонуклеотиды, удовлетворяющие условиям: длина ампликона ‒ 90‒200 п.н., Tm ‒ 60‒62°C (табл. 1). Кроме того, оценивали термодинамические параметры: свободную энергию Гиббса образования гомодимеров, шпилек и гетеродимеров праймеров (табл. 1). Показано, что значения ΔG образования шпилек и гомодимеров для отдельных олигонуклеотидов, а также гетеродимеров праймеров для амплификации фрагмента одного гена было не ниже ‒8 ккал/моль. На следующем этапе работы проводили оценку специфичности ПЦР-амплификации участков целевых генов. В качестве матрицы использовали кДНК, синтезированную на тотальной РНК из клеток печени мыши. Продукты ПЦР анализировали в 2%-ном агарозном геле, после чего их выделяли и проводили двунаправленное секвенирование. Показана высокая специфичность разработанного набора праймеров (рис. 1).
Рис. 1.
Оценка специфичности отжига праймеров на последовательности протеасомных генов мыши. Электрофорез в 2%-ном агарозном геле продуктов амплификации при использовании праймеров к протеасомным субъединицам мыши. В качестве матрицы использована кДНК, синтезированная на тотальной РНК из клеток печени мыши. Tm = 62°С, количественное определение продуктов реакции проводили после 30 циклов амплификации. М – маркеры длин ДНК (п.н.)

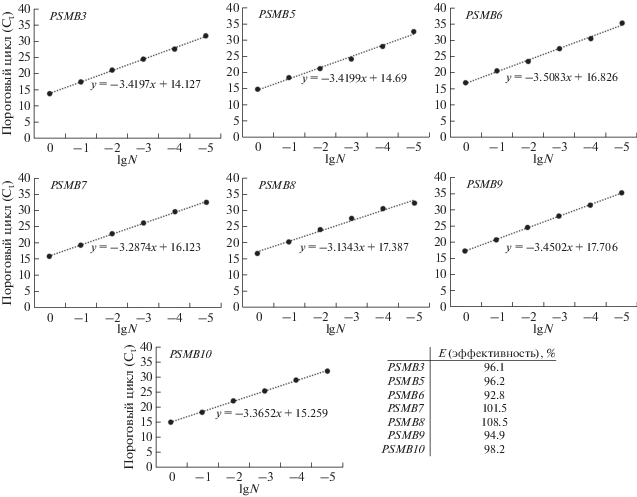
Полученные фрагменты клонировали в вектор pAL2-T. Плазмиды нарабатывали в клетках E. coli, после чего выделяли, очищали и определяли концентрацию. Затем с помощью онлайн-ресурса EndMemo (http://www.endmemo.com/bio/dnacopynum.php) определяли число копий вектора в единице объема. Далее проводили подбор оптимальных параметров проведения реакции и оценку эффективности амплификации целевых фрагментов методом ПЦР в реальном времени. В качестве матрицы использовали серии последовательных разведений полученных плазмид (от 100 копий до 10 млн копий), содержащих последовательности транскриптов PSMB5, PSMB6, PSMB7, PSMB8, PSMB9 и PSMB10 мыши. В ходе экспериментов определены оптимальные параметры проведения реакции, эффективность и чувствительность, подтверждена специфичность (по анализу кривых плавления) системы (рис. 2). Средняя эффективность амплификации праймеров, рассчитанная по формуле (2), составила 98.3%; с минимальной (92.8%) для гена PSMB6 (рис. 2). Таким образом, получена и откалибрована система, позволяющая проводить точное измерение числа копий транскриптов протеасомных субъединиц в тканях мышей.
Рис. 2.
Оценка эффективности амплификации праймеров к генам протеасом мыши. Зависимость порогового цикла амплификации (Ct) от числа копий матрицы (N) представлена для каждой исследованной пары праймеров. Анализ проведен в серии последовательных разведений плазмиды (от 100 копий до 10 млн копий), содержащей вставку последовательности транскриптов PSMB3, PSMB5, PSMB6, PSMB7, PSMB8, PSMB9 или PSMB10 мыши. Эффективность амплификации праймеров рассчитана по формуле (2).
Количественный анализ экспрессии генов субъединиц протеасом в различных отделах ЦНС мыши
Используя полученную систему, мы провели сравнительный анализ экспрессии генов протеолитических субъединиц протеасом в различных отделах ЦНС. Изучена экспрессия генов субъединиц протеасом в коре, спинном мозге, гиппокампе и мозжечке самцов мышей линии C57Bl в возрасте 60 дней.
Во всех исследованных отделах ЦНС наиболее высокие уровни экспрессии выявлены для генов, кодирующих конститутивные каталитические субъединицы протеасом: PSMB6 (медиана от 7.5 до 20.3 млн копий на 1 мкг тотальной РНК) и PSMB7 (медиана от 9.5 до 17.8 млн копий на 1 мкг тотальной РНК). Наименьший уровень экспрессии среди конститутивных субъединиц выявлен для гена PSMB5 (медиана от 4 до 9.5 млн копий на 1 мкг тотальной РНК). На основании этих данных можно предположить, что в исследованных отделах ЦНС среди каталитических субъединиц β5 ограничивает скорость сборки протеасом. Эти результаты хорошо согласуются с данными о критически важной роли субъединицы β5 и ее пропептида, выполняющих роль молекулярного шаперона в процессе сборки протеасом [12]. Уровни экспрессии генов иммунных субъединиц PSMB8 и PSMB9 были в среднем в 10‒70 раз ниже уровней экспрессии конститутивных субъединиц и составляли от 0.2 до 0.6 млн копий и от 0.1 до 0.3 млн копий (медиана значений) на 1 мкг РНК соответственно (рис. 3). В тоже время среди иммунных субъединиц следует выделить ген PSMB10, кодирующий субъединицу β2i. В сравнении с генами PSMB8 и PSMB9 уровень экспрессии PSMB10 был существенно выше и достигал значений, сравнимых с конститутивными субъединицами: от 1 до ~5 млн копий (медиана значений) на 1 мкг тотальной РНК (рис. 3). На основании полученных результатов можно говорить о преобладании конститутивных протеасом в различных отделах ЦНС, а различные уровни экспрессии генов иммунных субъединиц могут отражать присутствие как иммунных, так и промежуточных протеасом (протеасом, содержащих как конститутивные, так и иммунные каталитические субъединицы).
Рис. 3.
Число копий транскриптов генов протеасомных субъединиц в коре головного мозга, гиппокампе, мозжечке и спинном мозге мыши. Число копий указано для 1 мкг тотальной РНК. Точки на диаграмме отражают число транскриптов протеасомных субъединиц у индивидуальных особей. Выборка для анализа в гиппокампе, мозжечке и спинном мозге составила 5 особей, в коре головного мозга ‒ 6 особей.
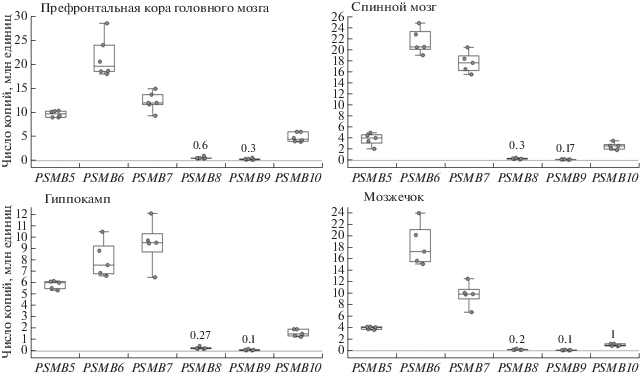
При сравнении количественных данных по общим уровням экспрессии генов межу отделами ЦНС последние удалось разделить на две категории: с условно “высокими” (кора и спинной мозг) и “низкими” (гиппокамп и мозжечок) уровнями экспрессии генов субъединиц протеасом. Так, различия между отделами с высокими и низкими уровнями экспрессии достигали 2‒2.5 раза для генов PSMB6 и PSMB7, от 1.7 до 3 раз для PSMB8 и PSMB9 и от 1.5 до 5 раз для PSMB10 (рис. 3). Таким образом, в разных отделах ЦНС необходимость в количестве протеасом в клетках может сильно различаться. В тоже время при анализе соотношений уровней экспрессии генов между собой можно выделить отделы ЦНС, имеющие близкие профили экспрессии генов субъединиц. Так, несмотря на существенные отличия по числу транскриптов, можно отметить близкие паттерны в коре головного мозга и мозжечке, несколько отличающийся профиль экспрессии генов в спинном мозге и существенно отличающийся в гиппокампе (рис. 3). На основании этих данных можно предполагать, что в отделах ЦНС с близкими профилями экспрессии генов протеасом имеется близкий набор различных форм протеасом, хотя их общее содержание может сильно отличаться.
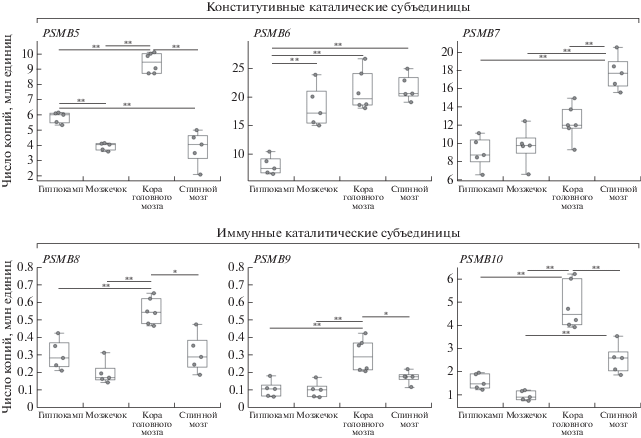
Сравнивая уровни экспрессии конкретных генов в разных отделах ЦНС, можно также выявить некоторые закономерности. Так, соотношения уровней экспрессии генов укладываются в два основных паттерна. Один представлен генами PSMB6 и PSMB7, где наименьшие уровни выявлены в гиппокампе, более высокие уровни – в мозжечке, далее в коре и, наконец, самые высокие в образцах спинного мозга (рис. 4). Примечательно, что уровень экспрессии PSMB6 в гиппокампе был существенно ниже (2‒2.5 раза, p < 0.01, ANOVA, критерий Тьюки), чем в других отделах ЦНС, ‒ в них различия в экспрессии были гораздо меньше (рис. 4). Для гена PSMB7 уровни экспрессии в гиппокампе и мозжечке отличались незначительно, однако в спинном мозге были в 1.5‒2 раза выше по сравнению с другими отделами ЦНС (p < 0.01, ANOVA, критерий Тьюки) (рис. 4). Для генов PSMB5, PSMB8, PSMB9 и PSMB10 выявлены другие особенности. Для этой группы генов самый низкий уровень экспрессии детектировали в мозжечке, затем по возрастающей: гиппокамп → спинной мозг → кора (рис. 4). В случае PSMB5 уровни экспрессии в мозжечке и спинном мозге были соизмеримы, в то время как отличия между остальными исследованными отделами ЦНС составляли 1.5‒2 раза (p < 0.01, ANOVA, критерий Тьюки). Для генов, кодирующих иммунные субъединицы протеасом, уровень экспрессии PSMB8 был выше приблизительно в 2 раза, PSMB9 в 2.5‒3 и PSMB10 в 2‒4 раза (p < 0.01, ANOVA, критерий Тьюки) в коре головного мозга по сравнению с остальными исследованными отделами ЦНС (рис. 4). Наряду с этим уровни экспрессии PSMB8 в гиппокампе и спинном мозге, а также PSMB9 в гиппокампе и мозжечке существенно не отличались. Интересно наличие как генов, кодирующих конститутивные (PSMB5), так и иммунные каталитические субъединицы во второй группе. По-видимому, это свидетельствует о наличии общих механизмов регуляции экспрессии каждой из выделенных групп генов в различных отделах ЦНС.
Рис. 4.
Сравнительный анализ экспрессии генов конститутивных (PSMB5, PSMB6 и PSMB7) и иммунных (PSMB8, PSMB9 и PSMB10) субъединиц протеасом в тканях ЦНС мыши. Число копий указано для 1 мкг тотальной РНК. Выборка для анализа в гиппокампе, мозжечке и спинном мозге составила 5 особей, в коре головного мозга ‒ 6 особей. Точки на диаграмме отражают число транскриптов протеасомных субъединиц у индивидуальных особей. *p < 0.05; **p < 0.01 (ANOVA, критерий Тьюки).
В заключение мы провели сравнительный анализ опубликованных данных транскриптомов образцов спинного мозга, коры головного мозга, мозжечка и гиппокампа мышей [13‒15] и выяснили, что полученные нами результаты хорошо согласуются с данными транскриптомных исследований (данные не приведены).
В ЦНС наряду с деградацией белка протеасомы выполняют еще несколько важных функций. Как уже отмечалось выше, протеасомы поставляют пептиды для презентации комплексов с молекулами MHC-I. Презентация таких комплексов на мембране клеток имеет принципиальное значение для клеточного иммунитета, однако, как выяснилось, в ЦНС это также необходимо для регуляции многих процессов развития мозга, включая рост нейритов, образование и функционирование синапсов, долгосрочную и гомеостатическую пластичность, а также зависящую от активности реорганизацию синапсов [16, 17]. Кроме того, недавно выявлена важная роль протеасом в межнейронных коммуникациях. Показано, что в мембраны нейронов встроены 20S протеасомы, которые разрушают специфические полипептиды с образованием биологически активных пептидов, регулирующих активацию соседних нейронов [18, 19]. Ramachandran и соавт. [18] обращают внимание на наличие в мембранах нейронов как протеасом с конститутивными, так и с иммунными бета-субъединицами. Orre и соавт. [20] показали, что при активации клеток микроглии вследствие развития воспаления или нейродегенерации увеличивается экспрессия иммунных субъединиц протеасом. Результаты этих исследований свидетельствуют о значимости баланса уровней экспрессии генов каталитических субъединиц протеасом в ЦНС, что имеет не только фундаментальное научное значение, но и прикладное ‒ например в диагностике нейродегенеративных заболеваний.
На основании полученных нами результатов можно сделать несколько важных выводов.
Во-первых, в отделах ЦНС мышей линии C57Bl преобладают конститутивные протеасомы, в тоже время в существенно меньших количествах, по-видимому, присутствуют промежуточные и иммунные протеасомы.
Во-вторых, по соотношению уровней экспрессии генов протеасом в целом различные отделы ЦНС можно разделить на отделы с условно “высоким” и “низким” уровнями экспрессии. Можно предполагать, что в отделах, составляющих одну группу, функциональная нагрузка на протеасомы сопоставима. Это может включать общие соотношения определенных субстратов протеасом внутри клеток, необходимость в производстве соизмеримого количества пептидов для обеспечения межнейронных взаимодействий [19], а также соответствующих пептидов, вовлеченных в пептидзависимые механизмы регуляции процессов внутри клеток [10].
В-третьих, при анализе соотношений уровней экспрессии генов между собой можно выделить отделы ЦНС, имеющие близкие профили экспрессии генов субъединиц; при этом число копий транскриптов для каждого конкретного гена в этих отделах может сильно различаться. Это значит, что в отделах с близкими профилями экспрессии генов протеасом, вероятно, имеется близкий набор различных форм протеасом; при этом их общее содержание может сильно отличаться. Однако важно обратить внимание на то, что экспрессия генов протеасом на транскрипционном уровне (мРНК) далеко не всегда коррелирует с посттрансляционным, то есть с экспрессией соответствующих белков, что было показано для иммунных субъединиц протеасом [21].
В-четвертых, можно выделить группы генов, экспрессия которых обладает общим паттерном изменений в разных отделах ЦНС, что указывает на вероятное наличие общих механизмов регуляции для каждой из выделенных групп генов в этих отделах.
ЗАКЛЮЧЕНИЕ
В последнее время анализ транскриптома с использованием технологии секвенирования нового поколения становится основным методом для характеристики уровней экспрессии генов в клетках и тканях. Однако для более быстрого и точного количественного анализа метод ПЦР в реальном времени по-прежнему остается “золотым стандартом” в исследовании уровней экспрессии генов. Нами разработана и протестирована система для количественного определения абсолютного содержания транскриптов конститутивных и иммунных каталитических субъединиц протеасом мыши. Используя эту систему, мы исследовали экспрессию генов субъединиц протеасом в образцах различных отделов ЦНС мыши. Обнаружены существенные различия по уровням экспрессии генов протеасом между разными отделами ЦНС. Их биологическую значимость предстоит выяснить в дальнейших исследованиях.
Работа выполнена при поддержке гранта РНФ №18-74-10095. Содержание животных обеспечено программой поддержки биоресурсных коллекций ИФАВ РАН (ФАНО №0090-2017-0016).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Groll M., Ditzel L., Löwe J., Stock D., Bochtler M., Bartunik H.D., Huber R. (1997) Structure of 20S proteasome from yeast at 2.4 A resolution. Nature. 386(6624), 463–471.
Heinemeyer W., Fischer M., Krimmer T., Stachon U., Wolf D.H. (1997) The active sites of the eukaryotic 20 S proteasome and their involvement in subunit precursor processing. J. Biol. Chem. 272, 25200–25209.
Rousseau A., Bertolotti A. (2018) Regulation of proteasome assembly and activity in health and disease. Nat. Rev. Mol. Cell Biol. 19(11), 697‒712.
Dantuma N.P., Bott L.C. (2014) The ubiquitin-proteasome system in neurodegenerative diseases: precipita-ting factor, yet part of the solution. Front. Mol. Neurosci. 7, 70.
Morozov A.V., Karpov V.L. (2019) Proteasomes and several aspects of their heterogeneity relevant to cancer. Front. Oncol. 9, 761.
Kors S., Geijtenbeek K., Reits E., Schipper-Krom S. (2019) Regulation of proteasome activity by (post-) transcriptional mechanisms. Front. Mol. Biosci. 6, 48.
Pickering A.M., Koop A.L., Teoh C.Y., Ermak G., Grune T., Davies K.J. (2010) The immunoproteasome, the 20S proteasome and the PA28αβ proteasome regulator are oxidative-stress-adaptive proteolytic complexes. Biochem. J. 432, 585–594.
Ferrington D.A., Gregerson D.S. (2012) Immunoproteasomes: structure, function, and antigen presentation. Prog. Mol. Biol. Transl. Sci. 109, 75–112.
Guillaume B., Chapiro J., Stroobant V., Colau D., Van Holle B., Parvizi G., Bousquet-Dubouch M.-P., Théate I., Parmentier N., Van den Eynde B. J. (2010) Two abundant proteasome subtypes that uniquely process some antigens presented by HLA class I molecules. Proc. Natl. Acad. Sci. USA. 107, 18599–18604.
Morozov A.V., Karpov V.L. (2018) Biological consequences of structural and functional proteasome diversity. Heliyon. 4(10), e00894.
Морозов А.В., Буров Б.А., Карпов В.Л. (2019) Динамика функциональной активности и экспрессии субъединиц протеасом в условиях адаптации клетки к тепловому шоку. Молекуляр. биология. 53(4), 638‒647.
Kunjappu M.J., Hochstrasser M. (2014) Assembly of the 20S proteasome. Biochim. Biophys. Acta. 1843(1), 2‒12.
Cembrowski M.S., Wang L., Sugino K., Shields B.C., Spruston N. (2016) Hipposeq: a comprehensive RNA-seq database of gene expression in hippocampal principal neurons. eLife. 5, e14997.
Funikov S.Y., Rezvykh A.P., Mazin P.V., Morozov A.V., Maltsev A.V., Chicheva M.M., Vikhareva E.A., Evgen′ev M.B., Ustyugov A.A. (2018) FUS(1‒359) transgenic mice as a model of ALS: pathophysiological and molecular aspects of the proteinopathy. Neurogenetics. 19(3), 189‒204.
Liu Q., Huang S., Yin P., Yang S., Zhang J., Jing L., Cheng S., Tang B., Li X-J., Pan Y., Li S. (2020) Cerebellum-enriched protein INPP5A contributes to selective neuropathology in mouse model of spinocerebellar ataxias type 17. Nat. Commun. 11(1), 1101.
Corriveau R.A., Huh G.S., Shatz C.J. (1998) Regulation of class I MHC gene expression in the developing and mature CNS by neural activity. Neuron. 21(3), 505‒520.
McAllister A.K. (2014) Major histocompatibility complex I in brain development and schizophrenia. Biol. Psychiatry. 75(4), 262–268.
Ramachandran K.V., Margolis S.S. (2017) A mammalian nervous-system-specific plasma membrane proteasome complex that modulates neuronal function. Nat. Struct. Mol. Biol. 24(4), 419–430.
Ramachandran K.V., Fu J.M., Schaffer T.B., Na C.H., Delannoy M., Margolis S.S. (2018) Activity-dependent degradation of the nascentome by the neuronal membrane proteasome. Mol. Cell. 71(1), 169–177.e6.
Orre M., Kamphuis W., Dooves S., Kooijman L., Chan E.T., Kirk C.J., Dimayuga Smith V., Koot S., Mamber C., Jansen A.H., Ovaa H., Hol E.M. (2013) Reactive glia show increased immunoproteasome activity in Alzheimer′s disease. Brain. 136(5), 1415‒1431.
Frentzel S., Kuhn-Hartmann I., Gernold M., Gött P., Seelig A., Kloetzel P.M. (1993) The major-histocompatibility-complex-encoded beta-type proteasome subunits LMP2 and LMP7. Evidence that LMP2 and LMP7 are synthesized as proproteins and that cellular levels of both mRNA and LMP-containing 20S proteasomes are differentially regulated. Eur. J. Biochem. 216(1), 119‒126.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


