Молекулярная биология, 2021, T. 55, № 1, стр. 126-138
Обработка стромальных слоев митомицином С стимулирует поддержание кроветворения в системе сокультивирования in vitro
О. Ф. Кандараков a, Ю. В. Кравацкий a, Н. С. Полякова a, А. В. Брутер a, Е. Г. Гордеева a, А. В. Белявский a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: abelyavs@yahoo.com
Поступила в редакцию 12.12.2019
После доработки 06.08.2020
Принята к публикации 13.08.2020
Аннотация
Изучено влияние обработки митомицином С (МитС) стромальных слоев клеток NIH 3T3, экспрессирующих лиганд рецептора Notch Jagged1, на размножение гемопоэтических Lin(–) клеток костного мозга мышей в системе сокультивирования. Обработка стромальных клеток МитС существенно повышает число гемопоэтических клеток и частоту колониеобразующих клеток при культивировании на строме. Анализ транскриптомов контрольного и обработанного МитС препаратов стромы с помощью дифференциального РНК-секвенирования показал, что гены, экспрессия которых подавляется при обработке, преимущественно связаны с контролем пролиферации, клеточным циклом, сегрегацией хромосом и метаболизмом ДНК. Транскриптомный анализ, однако, не выявил индукции ключевых гемопоэтических цитокинов при действии МитС, что позволяет исключить такую индукцию в качестве основной причины стимуляции кроветворения на обработанной строме. В то же время список наиболее индуцируемых МитС генов содержит большое количество генов цитокинов/ростовых факторов и белков клеточной поверхности, которые, возможно, стимулируют поддержание кроветворения стромой, обработанной МитС. Продукты некоторых из этих генов, как показано ранее, непосредственно участвуют в экспансии гемопоэтических стволовых/прогениторных клеток in vitro или in vivo.
ВВЕДЕНИЕ
Подготовка стромальных слоев к культивированию гемопоэтических стволовых и прогениторных клеток (ГСПК) зачастую включает этап обработки клеток стромы факторами, блокирующими их пролиферацию [1]. С этой целью применяют физические или химические методы подавления пролиферации клеток, к примеру, гамма-излучение или митомицин С (МитС) [2, 3]. Тем не менее, остается неясным, насколько важен этот этап для культивирования стволовых клеток на стромальном слое. В большинстве случаев культивирование ГСПК на стромальном слое проводят с использованием блокирования пролиферации [1]. С другой стороны, есть предположения, что блокировка пролиферации фидерных клеток может приводить к апоптозу и снижению продукции цитокинов и факторов роста клетками стромального слоя [4].
Согласно опубликованным результатам сравнительного культивирования эмбриональных стволовых клеток человека с фидерными эмбриональными фибробластами мыши (MEF), как контрольные, так и обработанные МитС MEF способны поддерживать продолжительную пролиферацию эмбриональных стволовых клеток, однако есть ограничения по плотности клеток фидерного слоя, не обработанных МитС [5]. Подобные сравнения проводили и с кератиноцитами, которые сокультивировали с аутологичными фибробластами и облученными клетками 3Т3 мыши. Показано, что необлученные аутологичные фибробласты лучше поддерживают рост кератиноцитов, чем облученные клетки 3Т3 [6].
Очевидно, что исходные и обработанные МитС клетки стромального слоя могут различаться экспрессией генов, что повлияет на поддержание ГСПК при культивировании. Следует также иметь в виду, что отсутствие блокировки деления клеток стромального слоя может приводить к постепенному увеличению плотности слоя и, как следствие, к истощению среды, в которой культивируют ГСПК, что может негативно сказаться на параметрах их культивирования.
Поскольку отсутствует общепринятое мнение о влиянии блокирования пролиферации клеток стромы в системах сокультивирования, мы поставили задачу оценить параметры культивирования гемопоэтических клеток на интактных и обработанных МитС стромальных слоях и, при обнаружении значительных различий в результатах культивирования, провести сравнительный транскриптомный анализ контрольных и блокированных МитС стром.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Культивирование клеток. Lin(–) клетки костного мозга мыши получали из бедренных костей мышей линии C57Bl/6 при помощи набора Lineage depletion kit (“Miltenyi Biotec”, Германия) по протоколу производителя.
Клетки GP293, NIH3T3 и 3T3-J/L культивировали в среде DMEM с 4.5 г/л глюкозы, 10% эмбриональной сыворотки крупного рогатого скота, 2 мМ глутамина, 100 мкг/мл стрептомицина и 100 МЕ/мл пенициллина при 37°С и 5% СО2 (далее DMEM). Клетки снимали раствором TrypLE Express (“Gibco”, США). Собранные клетки центрифугировали при 150 g, cупернатант удаляли и добавляли среду DMEM без сыворотки. Клетки обрабатывали 2.5 мкг/мл МитС в течение 2 ч в среде без сыворотки при 37°С и 5% СО2. Далее среду удаляли, клеточный монослой однократно промывали фосфатным буфером, затем добавляли полную среду DMEM и культивировали в течение 24 ч до внесения Lin(–) клеток. Перед внесением Lin(–) клеток на стромальный слой среду меняли на свежую. Клетки, не обработанные МитС, высевали в лунки шестилуночного планшета за сутки до внесения Lin(–) клеток и также меняли среду перед внесением Lin(–) клеток на стромальный слой. Начальная плотность клеток стромального слоя в исходном и обработанном МитС варианте составляла 105 кл./см2. Плотность посадки Lin(–) клеток – 5 × 103 кл./см2. Смену среды проводили через 2–3 сут. Исходные и обработанные МитС клетки стромального слоя для транскриптомного анализа культивировали в аналогичных условиях, но без внесения Lin(–) клеток. По окончании культивирования клетки лизировали Тризолом (Trizol Reagent, “Invitrogen”, США). Клетки подсчитывали с помощью гемоцитометра с использованием не менее трех независимых подсчетов для каждого образца. Клетки фотографировали камерой инвертированного микроскопа Olympus CKX41 с использованием фазового контраста.
Трансдукция клеток NIH 3T3-Jagged-LNGFR. Вирусный супернатант нарабатывали клетками упаковочной линии GP293 после кальций-фосфатной трансфекции клонированной бицистронной ретровирусной плазмидой, кодирующей Jagged и LNGFR. Для проведения трансдукции клетки NIH3T3 культивировали в течение 72 ч в среде, смешанной с вирусным супернатантом в соотношении 1 : 1, с добавлением полибрена (16 мкг/мл). Селекцию трансдуцированных клеток NIH 3T3-Jagged-LNGFR (3T3-J/L) проводили по поверхностному маркеру LNGFR с применением MACSelect™ LNGFR MicroBeads (“Miltenyi Biotec”), следуя инструкциям фирмы-производителя.
Селекция гемопоэтических клеток. Разделение клеток стромального слоя 3T3-J/L и гемопоэтических клеток проводили на колонке LS (“Miltenyi Biotec”), установленной на MACS магните (“Miltenyi Biotec”) после инкубации с 80 мкл MACSelect™ LNGFR MicroBeads (“Miltenyi Biotec”), следуя инструкциям фирмы-производителя. Выделенные гемопоэтические клетки культивировали в метилцеллюлозе MethoCult GF M3434 cat# 03434 (“StemCell Technologies”, США) при 37°С и 5% СО2 в течение 7–9 дней.
Подсчет колоний в метилцеллюлозе. Частоту колониеобразующих клеток в клеточных препаратах определяли в лунках 12-луночного планшета с 1 мл метилцеллюлозы, содержащей гемопоэтические факторы роста (MethoCult™ GF M3434, “StemCell Technologies”) и очищенные от магнитной стромы гемопоэтические клетки (104). Анализ проводили в трех повторах для каждого варианта. Клетки культивировали в метилцеллюлозе в течение 9–10 дней при 37°С и 5% СО2.
Транскриптомный анализ с помощью высокопроизводительного РНК-секвенирования. Транскриптомный анализ клеток стромального слоя проведен компанией “Геноаналитика” (Россия). Транскриптом каждого варианта анализировали в двух биологических повторностях, которые обрабатывали независимо. Суммарную РНК для транскриптомного анализа выделяли из клеток при помощи реагента Trizol (“Thermo Fisher Scientific”, США) согласно протоколу производителя. Качество и количество выделенной суммарной РНК проверяли на приборе BioAnalyser с использованием набора RNA 6000 Nano Kit (“Agilent”, США). Далее выделяли поли(А)-фракцию РНК при помощи олигоТ-магнитных шариков с использованием набора Dynabeads® mRNA Purification Kit (“Ambion”, ФРГ) согласно инструкции изготовителя. Далее из поли(А)-РНК готовили библиотеки для массового параллельного секвенирования при помощи набора NEBNext® Ultra™ II RNA Library Prep (“NEB”, США) согласно инструкции к набору. Концентрацию библиотек определяли при помощи набора Qubit dsDNA HS Assay Kit (“Thermo Fisher Scientific”) на приборе Qubit 2.0. Распределение длин фрагментов библиотеки оценивали при помощи набора Agilent High Sensitivity DNA Kit (“Agilent”). Секвенирование проводили на приборе HiSeq1500(“Illumina”, США) с генерацией не менее 10 млн коротких чтений длиной 50 нуклеотидов на реплику.
Биоинформатический анализ. Исходные чтения выравнивали на геном GRCm38 с помощью программы STAR (версия 2.5.3). Определение числа чтений, приходящихся на каждый ген (аннотация Ensembl v. 99), проводили с помощью программы htseq-count (версия 0.9.1). Количество чтений подсчитывали двумя методами: а) с использованием htseq-count по умолчанию; б) подсчет только чтений, полностью (100%) совпадающих с референсным геномом. Биоинформатический дифференциальный RNA-Seq-анализ проводили с помощью R-модуля DESeq2 [7]. Анализ генной онтологии (Gene Ontology, GO) проводили с использованием веб-сервера GOrilla [8].
Цитометрический анализ. Стромальные клетки 3T3-J/L окрашивали антителами anti-CD271-FITC (“BioLegend” (США) cat#345104, разведение 1 : 100), клетки 3T3-J/L перед окрашиванием anti-Jagged1-Alexa Fluor 647 (D4Y1R) (“Cell Signaling Technologies” cat#54340, разведение 1 : 50) фиксировали метанолом, следуя инструкции производителя антител. Выделенные гемопоэтические клетки красили антителами anti-CD45-APC (“BioLegend “cat#103112, разведение 1 : 200). Цитометрию проводили с помощью прибора BD LSRFortessa (“BD”, США).
Статистистический анализ. Эксперименты c культивированием Lin(–) клеток (pис. 1–3) проводили не менее 3 раз, на рисунках показаны репрезентативные примеры. При проведении статистического анализа (вычисление стандартных отклонений и статистической значимости отличий по t-критерию Стьюдента) использовали статистические функции программы Excel (“Microsoft”, США).
РЕЗУЛЬТАТЫ ИСCЛЕДОВАНИЯ
Экспрессия LNGFR и Jagged1 трансдуцированными клетками стромального слоя NIH 3T3
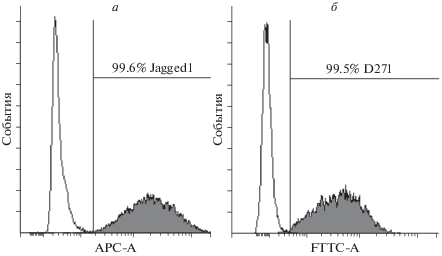
Гемопоэтические клетки мыши, обогащенные ГСПК (фракция Lin– клеток костного мозга), культивировали с использованием стромальных клеток NIH 3T3, экспрессирующих белок Jagged1 (трансмембранный лиганд рецептора Notch). Такие клетки значительно лучше поддерживают кроветворение in vitro, чем исходные, за счет активации сигнального пути Notch, что тормозит дифференцировку ГСПК [9]. Культивирование гемопоэтических клеток на стромальных слоях всегда сопряжено с задачей их последующего разделения. Ранее c этой целью мы использовали меченные магнитными наночастицами стромальные слои [10], что позволяло удалять из смеси клеток стромальный компонент. Однако последующие исследования показали, что стромальные клетки при культивировании теряют часть магнитной метки, что может ухудшать чистоту выделяемых гемопоэтических клеток [11]. Следовательно, необходимо было разработать эффективную систему отделения гемопоэтических клеток от клеток стромального слоя. Для выполнения этой задачи клетки NIH 3T3 трансдуцировали бицистронным ретровирусным вектором, кодирующим гены Jagged1 и LNGFR (low affinity nerve growth factor receptor), где Jagged1 стимулировал пролиферацию гемопоэтических клеток, а LNGFR использовали для магнитной селекции клеток стромального слоя, как показано ранее [12, 13]. Доля клеток 3T3-J/L, экспрессирующих белки Jagged1 и LNGFR после магнитной селекции с применением MACSelect™ LNGFR MicroBeads, превышала 99% (рис. 1), что доказывает высокую эффективность магнитной селекции трансдуцированных клеток по LNGFR с применением данного подхода. Инкубация смеси стромальных 3T3-J/L и гемопоэтических клеток с магнитно-мечеными антителами с последующим разделением на колонке LS Miltenyi Biotec позволяет отделять стромальные клетки, экспрессирующие LNGFR, от гемопоэтических клеток.
Культивирование гемопоэтических клеток на строме 3T3-J/L
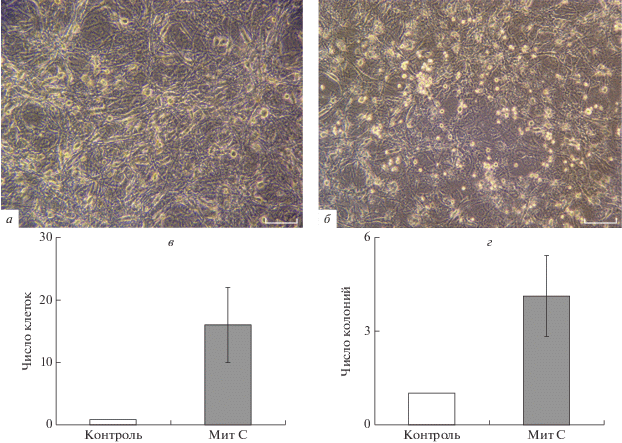
Гемопоэтические клетки культивировали на подготовленных стромальных слоях 3T3-J/L. Оказалось, что гемопоэтические клетки формируют колонии только на стромальном слое, обработанном МитС, в то время как в контрольном варианте такие колонии практически не встречались (рис. 2а, б). Однако различия касались не только колоний гемопоэтических клеток на стромальном слое, но и числа клеток с этих стромальных слоев. Наибольшее количество клеток выделено со стромального слоя, обработанного МитС (pис. 2в). Различие между гемопоэтическими клетками, выделенными с разных стромальных слоев, коснулось и числа колониеобразующих клеток в метилцеллюлозе. Число колоний, которые сформировали гемопоэтические клетки, снятые с обработанного МитС слоя, было значительно больше, чем с контрольного варианта (рис. 2г).
Рис. 2.
Рост Lin(–) клеток на исходном (а) и обработанном МитС (б) стромальном слое 3T3-J/L, число гемопоэтических клеток, выделенных на 7-е сутки культивирования (в), и число колоний, сформированных в метилцеллюлозе (г). На диаграммах показаны средние значения и стандартные отклонения нормированнных значений двух экспериментов. Число клеток и колоний нормировано относительно контрольных (не обработанных МитС) вариантов, принятых во всех экспериментах за единицу. Контроль и МитС – исходный и обработанный МитС стромальный слой соответственно. Клетки фотографировали с использованием фазового контраста. Планки на фотографиях соответствуют 100 мкм. На рис. 2в и 2г p < 0.05 (*) по сравнению с контролем (по t-критерию Стьюдента).
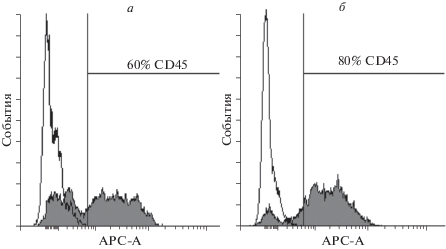
Мы также охарактеризовали цитометрически снятые со стромальных слоев клетки по пан-гемопоэтическому маркеру CD45. В популяции выделенных Lin(–) клеток доля CD45-позитивных клеток составляла 85%. При культивировании Lin(–) клеток на обработанном МитС стромальном слое доля CD45-позитивных клеток осталась высокой и составила 80%, при этом в популяции клеток, снятых с необработанного МитС стромального слоя, эта доля была несколько ниже (около 60%, рис. 3), однако значимость различий между контрольными и обработанными МитС культурами остается неясной. В целом, полученные данные свидетельствуют о том, что в клеточных популяциях, снятых со стромальных слоев, большинство составляют CD45-позитивные гемопоэтические клетки.
Анализ транскриптома стромальных клеток
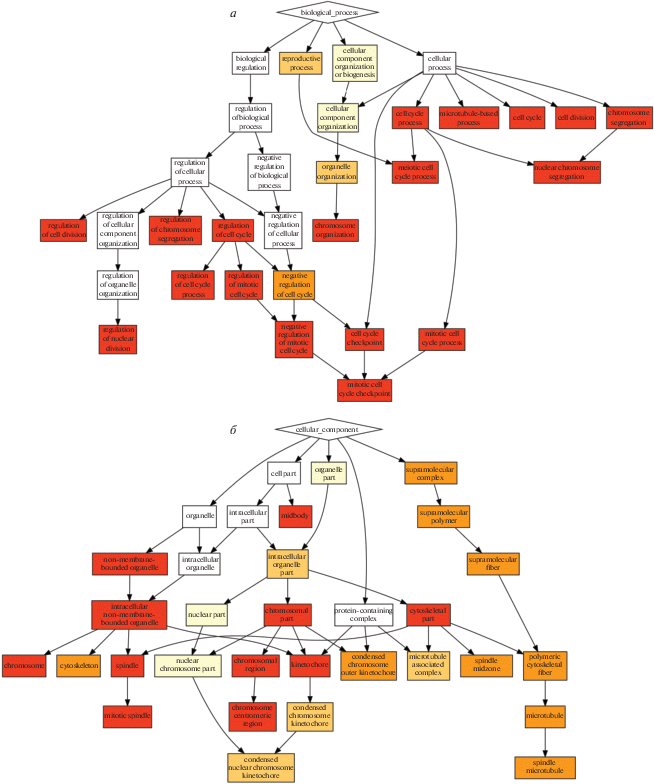
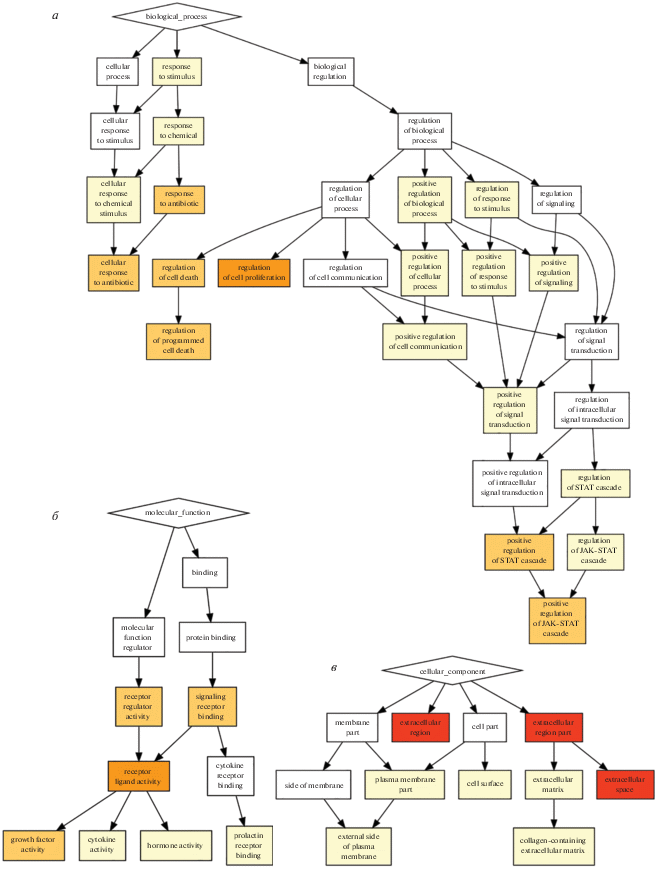
Поскольку стромальные слои, обработанные МитС, обладают существенно более высокой способностью поддерживать кроветворение, чем контрольные слои, мы предположили, что обработка вызывает в стромальных клетках стресс-ответ, включающий, в том числе, активацию экспрессии генов гемопоэтических цитокинов, таких, как Kitl или Thpo. Для проверки этого предположения, а также для идентификации других возможных изменений в паттерне генной экспрессии, проведен транскриптомный анализ клеток стромального слоя до и после обработки МитС. С этой целью применили метод глубокого секвенирования RNA-Seq с использованием более 10 млн чтений на реплику. Анализ качества чтений реплик и их согласованности показал высокое качество чтений (Q > 30 для всех нуклеотидов в чтениях во всех репликах) и высокую согласованность реплик (коэффициент корреляции Пирсона для каждой пары реплик, вычисленный программой deepTools, r = 0.99). Возможную роль дифференциально экспрессирующихся генов исследовали с использованием GO-анализа c помощью веб-сервера Gorilla [8]. Результаты GO-анализа генов, экспрессия которых подавляется при обработке стромальных клеток МитС, приведены на рис. 4. Среди генов с высокой степенью подавления экспрессии широко представлены гены, продукты которых участвуют в пролиферации, регуляции клеточного цикла и сегрегации хромосом. Среди этих генов также широко представлены гены, участвующие в метаболизме ДНК, которые не вошли в cхему, представленную на рис. 4б, из-за высокого порога отсечения (10–11).
Рис. 4.
Иерархическая блок-схема терминов GO (генной онтологии) для генов с наиболее высоким уровнем подавления экспрессии после обработки стромальных клеток МитС. а – Схема класса GO “процесс”; б – схема класса GO “компонент”. При построении схем использовали пороги отсечения 10–11 и 10–6 – а и б соответственно. Гены с низким уровнем экспрессии из анализа исключены.
Что касается генов, экспрессия которых индуцируется при обработке МитС, то, вопреки первоначальной гипотезе, не обнаружено заметной индукции генов, кодирующих известные гемопоэтические цитокины – как ранние (в частности, KitL, Tpo, Flt3L), так и поздние (Csf1, Csf2, Csf3). Единственное исключение представляет интерлейкин-6, экспрессия которого индуцируется в 3.8 раза при обработке МитС, однако уровень его экспрессии даже после индукции, если судить по числу чтений (18–20 на 10 млн), остается относительно низким. Анализ GO класса “процесс” (рис. 5а) выявил некоторые группы, с высокой степенью значимости обогащенные в списке индуцируемых генов, однако их связь с поддержанием кроветворения представляется неясной. Тем не менее, GO-анализ классов “функция” (рис. 5б) и “компонент” (рис. 5в) выявил обогащение генами, связанными с взаимодействиями лиганд–рецептор, с компонентами клеточной мембраны и внеклеточного пространства соответственно.
Рис. 5.
Иерархическая блок-схема терминов GO генов с наиболее высоким уровнем индукции экспрессии после обработки стромальных клеток МитС. а – Cхема класса GO “процесс”; б – схема класса GO “функция”; в – схема класса GO “компонент”. При построении схем использованы пороги отсечения 10–6, 10–4 и 10–3 (части а, б и в соответственно). Гены с низким уровнем экспрессии из анализа исключены.
Проведен анализ 40 генов с наиболее высокой степенью индукции (табл. 1), направленный на исследование их возможной связи с кроветворением и поддержанием ГСПК. При этом в анализ были включены только гены со средним и высоким уровнем экспрессии, поскольку функциональная значимость генов с низким уровнем экспрессии в наблюдаемых эффектах стромы представляется сомнительной. Приведенные в табл. 1 данные показывают, что среди проанализированных генов высокая доля (более 50%) принадлежит генам, кодирующим белки, либо локализованные на клеточной поверхности, либо секретируемые в среду, что согласуется с результатами GO-анализа класса “компонент”. Белки этой группы могут непосредственно участвовать в передаче сигналов от стромы к ГСПК и, вероятно, некоторые из них могут активировать пролиферацию ГСПК после обработки стромы МитС. Как отмечено в табл. 1, продукты некоторых из исследованных генов способствуют экспансии ГСПК in vitro и in vivo. Более подробно влияние этих генов на кроветворение рассмотрено в разделе “Обсуждение результатов”.
Таблица 1.
Гены с наиболее сильной индукцией экспрессии после обработки стромальных клеток митомицином С
| Офици-альное название | Полное название | Степень индукции экспрессии, log2 | Уровень экспрессии, TPM* | p | Поверхнос-тный/ секре-тируемый белок? | Комментарий* |
|---|---|---|---|---|---|---|
| Gm10800 | 3.53 | 2472.6 | 2.38E-08 | н.п. | Предположительно, некодирующая РНК, функция неизвестна | |
| Gm10801 | 3.53 | 654.6 | 2.07E-16 | н.п. | Предположительно, некодирующая РНК, функция неизвестна | |
| Agt | Angiotensinogen (serpin peptidase inhibitor, clade A, member 8) | 3.37 | 16.3 | 1.09E-14 | Да | |
| Ccl8 | Chemokine (C-C motif) ligand 8 | 2.96 | 14.6 | 0.0002 | То же | Хемокин, участвует в стимуляции хоуминга ГСПК к стромальным клеткам |
| Serpina3i | Serine (or cysteine) peptidase inhibitor, clade A, member 3I | 2.95 | 19.80 | 0.0004 | » | Способствует экспансии ГСПК |
| CD80 | CD80 antigen | 2.70 | 64.7 | 5.37E-48 | » | |
| Klra2 | Killer cell lectin-like receptor, subfamily A, member 2 | 2.57 | 15.3 | 7.68E-08 | ||
| Prl2c3 | Prolactin family 2, subfamily c, member 3 | 2.52 | 484.6 | 1.85E-25 | » | Способствует экспансии ГСПК |
| Ccn5 | Cellular communication network factor 5 | 2.47 | 28.2 | 4.36E-12 | » | Цитокин/фактор роста |
| Id2 | Inhibitor of DNA binding 2 | 2.24 | 54.5 | 4.32E-19 | Нет | Способствует экспансии ГСПК |
| Tnnt2 | Troponin T2, cardiac | 2.14 | 17.9 | 3.38E-06 | То же | |
| Id3 | Inhibitor of DNA binding 3 | 2.09 | 108.6 | 7.41E-27 | » | Способствует экспансии ГСПК |
| Aldh3a1 | Нет | 1.98 | 289.0 | 1.17E-76 | » | Роль в регуляции числа ГСПК |
| Lcn2 | Lipocalin 2 | 1.93 | 30.8 | 5.36E-08 | Да | Роль в регуляции числа ГСПК |
| Des | Desmin | 1.85 | 15.8 | 4.83E-07 | Нет | |
| Kazald1 | Kazal-type serine peptidase inhibitor domain 1 | 1.83 | 26.0 | 9.72E-10 | Да | |
| Serpina3h | Serine (or cysteine) peptidase inhibitor, clade A, member 3h | 1.83 | 20.8 | 0.0004 | Да | Способствует экспансии ГСПК |
| Thbs1 | Thrombospondin 1 | 1.77 | 42.9 | 4.35E-42 | Да | |
| Trp53inp1 | Transformation related protein 53 inducible nuclear protein 1 | 1.75 | 62.3 | 6.66E-55 | Нет | |
| Nqo1 | NAD(P)H dehydrogenase, quinone 1 | 1.74 | 131.0 | 4.24E-39 | То же | |
| Snora2b | Small nucleolar RNA, H/ACA box 2B | 1.65 | 105.3 | 0.0003 | » | |
| H2bc4 | H2B clustered histone 4 | 1.60 | 16.7 | 0.003 | » | |
| C4b | Сomplement component 4B | 1.62 | 105.9 | 1.09E-48 | Да | |
| Dusp4 | Dual specificity phosphatase 4 | 1.54 | 24.6 | 3.49E-11 | Нет | |
| Prl2c2 | Prolactin family 2, subfamily c, member 2 | 1.48 | 350.7 | 1.39E-07 | Да | Вероятно, способствует экспансии ГСПК |
| Nrp1 | Neuropilin 1 | 1.47 | 37.7 | 1.46E-21 | Да | |
| Id1 | Inhibitor of DNA binding 1 | 1.46 | 141.5 | 2.23E-20 | Нет | |
| Sdc1 | Syndecan 1 | 1.43 | 184.4 | 1.69E-47 | Да | |
| Gm2115 | 1.42 | 18.5 | 1.94E-15 | н.п. | ||
| Gm5886 | 1.40 | 390.9 | 7.73E-28 | н.п. | ||
| Btg2 | BTG anti-proliferation factor 2 | 1.38 | 44.7 | 1.54E-17 | Нет | |
| Tmem268 | Transmembrane protein 268 | 1.36 | 21.1 | 4.80E-09 | Да | |
| Eda2r | Ectodysplasin A2 receptor | 1.35 | 35.5 | 5.47E-18 | Да | |
| Ahr | Aryl-hydrocarbon receptor | 1.33 | 28.0 | 2.39E-14 | Нет | Роль в функционировании ГСПК |
| Lgi4 | Leucine-rich repeat LGI family, member 4 | 1.33 | 33.6 | 9.68E-16 | Да | |
| Cyp1b1 | Cytochrome P450, family 1, subfamily b, polypeptide 1 | 1.32 | 21.3 | 1.64E-13 | Нет | |
| Serpinb9b | Serine (or cysteine) peptidase inhibitor, clade B, member 9b | 1.32 | 22.8 | 2.63E-07 | Да | |
| Crlf1 | Cytokine receptor-like factor 1 | 1.31 | 306.3 | 8.49E-38 | Да | |
| Spag1 | Sperm associated antigen 1 | 1.29 | 20.2 | 1.53E-07 | Нет | |
| Slpi | Secretory leukocyte peptidase inhibitor | 1.27 | 648.8 | 1.17E-40 | Да | Способствует экспансии ГСПК |
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Сравнение результатов культивирования Lin(–) клеток на стромальных слоях 3T3-J/L, обработанных и не обработанных МитС, выявило значительные различия в числе гемопоэтических клеток, частоте колониеобразующих клеток и уровне экспрессии поверхностного маркера CD45. Известно, что разные стромальные слои способны по-разному поддерживать рост стволовых клеток, и причина этого различия, по всей вероятности, лежит в различиях паттернов экспрессии генов [14]. С другой стороны, обработка клеток МитС также меняет спектр и уровень экспрессии генов [15–17]. По данным транскриптомного анализа, проведенного в настоящей работе, обработка клеток стромального слоя МитС также вызывает значительные изменения генной экспрессии. Подобные изменения паттерна экспрессии являются наиболее вероятным механизмом увеличения поддержания кроветворения in vitro стромальными слоями после такой обработки. Однако на данный момент нельзя полностью исключить и альтернативный механизм, связанный с тем, что, по нашим наблюдениям, клетки NIH 3T3 не полностью подвержены контактному торможению в условиях монослоя и продолжают рост при достижении полного монослоя. В отсутствие обработки МитС это приводит к мало контролируемой пролиферации стромальных клеток и к ускоренному поглощению стромой питательных веществ в среде, что, в свою очередь, может ограничивать питание сокультивируемых кроветворных клеток и замедление их пролиферации. С таким объяснением, однако, не согласуется визуальный анализ процесса сокультивирования на строме, согласно которому различия в продукции кроветворных клеток на контрольной строме и строме, обработанной МитС, начинают проявляться уже в первые дни культивирования, когда плотность стромального слоя в контроле и при обработке МитС не сильно различается.
Следует отметить, что наборы генов, экспрессиия которых подавляется или индуцируется обработкой МитС, функционально сильно различаются. По данным GO-анализа для первой категории характерно доминирование генов, которые позитивно регулируют клеточный цикл, в том числе и расхождение хромосом. Очевидно, клетка снижает экспрессию тех генов, функция которых становится ненужной при блокировании пролиферации вследствие обработки МитС. Сходные изменения в экспрессии генов, связанных с пролиферацией, клеточным циклом и хромосомной сегрегацией, обнаружены ранее при обработке МитС нейральных стволовых клеток [17].
Весьма отчетливо функциональные различия между генами, экспрессия которых подавляется или, наоборот, индуцируется при обработке стромы МитС, выявляются при GO-анализе категории “компонент”. Если для первых характерно обогащение генами, связанными с хромосомами, кинетохорами и митотическим веретеном, то для последних характерно обогащение генами, связанными с клеточной мембраной и внеклеточным пространством, в том числе c внеклеточным матриксом.
Транскриптомный анализ показал, что, с одной стороны, не подтвердилась первоначальная гипотеза об индукции генов известных гемопоэтических цитокинов при обработке стромы МитС. Единственное исключение здесь составляет интерлейкин-6, уровень которого в обработанной МитС строме возрастает в 3.8 раза, однако низкий уровень этого цитокина даже после индукции заставляет сомневаться в его существенной роли в эффектах стромы, обработанной МитС.
Тем не менее, список генов со средним и высоким уровнем экспрессии, которые наиболее сильно индуцируются МитС, обогащен генами белков, локализованных на клеточной поверхности, и секретируемых цитокинов/ростовых факторов. Детальный анализ доступных опубликованных данных показал, что некоторые из этих белков играют существенную роль в кроветворении и экспансии ГСПК, т.е. они могут отвечать за стимуляцию кроветворения на строме при обработке МитС. В частности, высокая экспрессия и высокий уровень индукции (в 5.7 раза) отмечены у гена Prl2c3 (пролиферин-2), продукт которого, как показано ранее, стимулирует экспансию ГСПК ex vivo [18]. Высокий уровень экспрессии и значительная степень индукции обнаружены также у родственного гена Prl2c2 (пролиферин), который, судя по высокой идентичности на уровне белка с Prl2c3, должен обладать сходной функцией. Еще более высокая степень индукции (в 7.8 раза), хотя и при умеренном уровне транскрипции, обнаружена у гена хемокина Ccl8. Продукт этого гена вместе с хемокином Cxcl12 участвует в стимуляции хоуминга ГСПК к стромальным клеткам [19]. Кроме того, высокая степень индукции (в 5.5 раза) при умеренном уровне транскрипции характерна для гена Ccn5 (WISP2), принадлежащего к семейству факторов роста Ccn (cellular communication network factors). Следует отметить, что гены еще двух членов семейства Ccn, а именно Ccn2 и Ccn3, не вошедшие в исследуемый список, индуцируются не менее чем в 2 раза при обработке стромальных клеток МитС. Продукты этих генов, как показано ранее, участвуют непосредственно в экспансии ГСПК [20, 21]. Возможно, подобной функцией обладает и ген Ccn5. Показано, что сверхэкспрессия в костной ткани высоко индуцируемого гена липокалина 2 (Lcn2) вызывает увеличение числа ГСПК in vivo [22]. Обращает также на себя внимание, что среди 40 генов с наиболее высокой степенью индукции присутствуют четыре гена, кодирующих ингибиторы протеаз (Serpina3i, Serpina3h, Slpi и Serpinb9b). Первые три гена способствуют экспансии кроветворных клеток за счет ингибирования протеолиза гемопоэтических цитокинов согласно [23, 24].
Хотя рассмотренные гены кодируют секретируемые белки, которые, функционируя как факторы роста, могут непосредственно стимулировать пролиферацию ГСПК, в число генов с высокой степенью индукции входит также ряд генов, кодирующих цитоплазматические/ядерные белки, в частности факторы транскрипции. За последние годы надежно установлено, что клетки могут оказывать значительное воздействие друг на друга путем обмена секретируемыми внеклеточными везикулами – экзосомами и родственными образованиями. При этом, в отличие от классического взаимодействия клеток с использованием секретируемых факторов, внеклеточные везикулы содержат цитоплазматические белки, а также РНК, в том числе и матричные, которые могут передаваться клеткам-реципиентам. В этой связи важно упомянуть, что из четырех генов семейства факторов транскрипции Id (inhibitor of DNA binding) три (Id1, Id2 и Id3) показали средний или высокий уровень экспрессии и высокую степень индукции при обработке МитС. Вполне возможно, что это генное семейство играет значительную роль в выполнении программы индукции и/или подавлении экспрессии генов МитС. В контексте этой работы весьма важны данные, показывающие, что сверхэкспрессия Id2 в ГСПК способствует их экспансии in vitro примерно в 10 раз [25], а при сверхэкспрессии Id3 экспансия ГСПК происходит в еще более значительной степени [26]. Таким образом, можно предположить, что обработанные МитС стромальные клетки могут способствовать размножению ГСПК, в том числе и за счет экспорта РНК и/или белков семейства Id посредством экзосом. Ранее было показано, что ген фактора транскрипции Ahr (aryl-hydrocarbon receptor), существенно индуцируемый МитС, играет важную роль в ГСПК [27]. Более того, экспрессия Ahr в стромальных клетках также важна для функционирования ГСПК [28]. Интересно, что альдегиддегидрогеназа 3а1 (Aldh3a1), уровень экспрессии который повышается при индукции в 4 раза, участвует в регуляции числа ГСПК, предположительно, за счет регуляции образования активных форм кислорода и реактивных альдегидов [29].
Обращает на себя внимание, что в списке высоко экспрессируемых индуцируемых генов два первых места по уровню индукции занимают гены, получившие в базе ENSEMBL названия Gm10800 и Gm10801. В базе ENSEMBL они представлены как гены, кодирующие трансмембранные белки с пятью гидрофобными доменами, однако это чисто теоретическая классификация, которая не имеет экспериментального подтверждения. Анализ предсказанных РНК этих генов выявляет ряд свойств, не характерных для классических белоккодирующих РНК, в частности, у них отсутствуют 5′- и 3′-некодирующие области и даже терминирующий кодон, они также имеют необычно короткие (вплоть до одного основания) некоторые интроны. Мы предполагаем, что представленные в базе ENSEMBL последовательности РНК Gm10800 и Gm10801 могут быть результатом некорректного предсказания экзон-интронной структуры этих генов, а соответствующие им РНК на самом деле являются некодирующими, поскольку с использованием метода ОТ-ПЦР мы не смогли обнаружить в стромальных клетках РНК с предсказанной последовательностью (данные не приведены). При этом в геноме мыши найден целый ряд участков с родственными последовательностями (не менее 13 в общей сложности). По всей вероятности, транскрипция некоторых из этих участков происходит в стромальных клетках, индуцируется при обработке МитС и, возможно, имеет функциональное значение, однако точная последовательность соответствующих РНК и, тем более, их гипотетическая функция не установлены.
Авторы благодарят компанию “Геноаналитика” (Москва) за профессионально проведенный транскриптомный анализ и первичную биоинформатическую обработку данных.
Работа выполнена при поддержке Российского научного фонда (проект № 18-14-00300).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Llames S., García-Pérez E., Meana Á., Larcher F., del Río M. (2015) Feeder layer cell actions and applications Tissue Eng. Part B Rev. 21, 345–353.
Roy A., Krzykwa E., Lemieux R., Néron S. (2001) Increased efficiency of gamma-irradiated versus mitomycin C-treated feeder cells for the expansion of normal human cells in long-term cultures. J. Hematother. Stem Cell Res. 10, 873–880.
Chugh R.M., Chaturvedi M., Yerneni L.K. (2015) An evaluation of the choice of feeder cell growth arrest for the production of cultured epidermis. Burns. 41, 1788–1795.
Glettig D.L., Kaplan D.L. (2013) Extending human hematopoietic stem cell survival in vitro with adipocytes. Biores. Open Access. 2, 179–185.
Xie C.Q., Lin G., Yuan D., Wang J., Liu T.C., Lu G.X. (2005) Proliferative feeder cells support prolonged expansion of human embryonic stem cells. Cell Biol. Int. 29, 623–628.
Jubin K., Martin Y., Lawrence-Watt D.J., Sharpe J.R. (2011) A fully autologous co-culture system utilising nonirradiated autologous fibroblasts to support the expansion of human keratinocytes for clinical use. Cytotechnology. 63, 655–662.
Love M.I., Huber W., Anders S. (2014) Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15, 550.
Eden E., Navon R., Steinfeld I., Lipson D., Yakhini Z. (2009) GOrilla: a tool for discovery and visualization of enriched GO terms in ranked gene lists. BMC Bioinformatics. 10, 48.
Раевская А.А., Савватеева М.В., Бухинник С.С., Кандараков О.Ф., Бутылин П.А., Жук С.В., Демин А.М., Краснов В.П., Зарицкий А.Ю., Белявский А.В. (2017) Культивирование кроветворных предшественников мыши и человека на стромальных подслоях, экспрессирующих лиганды Notch. Молекуляр. биология. 51(2), 356–366.
Savvateeva M.V., Demin A.M., Krasnov V.P., Belyavsky A.V. (2016) Magnetic stromal layers for enhanced and unbiased recovery of co-cultured hematopoietic cells. Anal. Biochem. 509, 146–155.
Кандараков О.Ф., Дёмин А.М., Попенко В.И., Леонова О.Г., Копанцева Е.Е., Краснов В.П., Белявский А.В. (2020) Факторы, влияющие на мечение клеток NIH 3Т3 магнитными наночастицами. Молекуляр. биология. 54(1), 114–127.
Fehse B., Uhde A., Fehse N., Eckert H.G., Clausen J., Rüger R., Koch S., Ostertag W., Zander A.R., Stockschläder M. (1997) Selective immunoaffinity-based enrichment of CD34+ cells transduced with retroviral vectors containing an intracytoplasmatically truncated version of the human low-affinity nerve growth factor receptor (deltaLNGFR) gene. Hum. Gene Ther. 8(15), 1815–1824.
Hildinger M., Schilz A., Eckert H.G., Bohn W., Fehse B., Zander A., Ostertag W., Baum C. (1999) Bicistronic retroviral vectors for combining myeloprotection with cell-surface marking. Gene Ther. 6, 1222–1230.
Kueh J., Richards M., Ng S.W., Chan W.K., Bongso A. (2006) The search for factors in human feeders that support the derivation and propagation of human embryonic stem cells: preliminary studies using transcriptome profiling by serial analysis of gene expression. Fertil. Steril. 85, 1843–1846.
Stepp M.A., Pal-Ghosh S., Tadvalkar G., Li L., Brooks S.R., Morasso M.I. (2018) Molecular basis of mitomycin C enhanced corneal sensory nerve repair after debridement wounding. Sci. Rep. 8, 16960.
Sato N., Haga J., Anazawa T., Kenjo A., Kimura T., Wada I., Mori T., Marubashi S., Gotoh M. (2017) Ex vivo pretreatment of islets with mitomycin c: reduction in immunogenic potential of islets by suppressing secretion of multiple chemotactic factors. Cell Transplant. 26, 1392–1404.
Felfly H., Xue J., Zambon A.C., Muotri A., Zhou D., Haddad G.G. (2011) Identification of a neuronal gene expression signature: role of cell cycle arrest in murine neuronal differentiation in vitro. Am. J. Physiol. Regul. Integr. Comp. Physiol. 301, R727–R745.
Choong M.L., Tan A.C., Luo B., Lodish H.F. (2003) A novel role for proliferin-2 in the ex vivo expansion of hematopoietic stem cells. FEBS Lett. 550, 155–162.
Nguyen Hoang A.T., Liu H., Juaréz J., Aziz N., Kaye P.M., Svensson M. (2010) Stromal cell-derived CXCL12 and CCL8 cooperate to support increased development of regulatory dendritic cells following Leishmania infection. J. Immunol. 185, 2360–2371.
Istvánffy R., Vilne B., Schreck C., Ruf F., Pagel C., Grziwok S., Henkel L., Prazeres da Costa O., Berndt J., Stümpflen V., Götze K.S., Schiemann M., Peschel C., Mewes H.W., Oostendorp R.A.J. (2015) Stroma-derived connective tissue growth factor maintains cell cycle progression and repopulation activity of hematopoietic stem cells in vitro. Stem Cell Repts. 5, 702–715.
Gupta R., Hong D., Iborra F., Sarno S., Enver T. (2007) NOV (CCN3) functions as a regulator of human hematopoietic stem or progenitor cells. Science. 316, 590–593.
Costa D., Principi E., Lazzarini E., Descalzi F., Cancedda R., Castagnola P., Tavella S. (2017) LCN2 overexpression in bone enhances the hematopoietic compartment via modulation of the bone marrow microenvironment. J. Cell. Physiol. 232, 3077–3087.
Winkler I.G., Hendy J., Coughlin P., Horvath A., Lévesque J.P. (2005) Serine protease inhibitors serpina1 and serpina3 are down-regulated in bone marrow during hematopoietic progenitor mobilization. J. Exp Med. 201, 1077–1088.
Goselink H.M., Hiemstra P.S., van Noort P., Barge R.M., Willemze R., Falkenburg J.H. (2006) Cytokine-dependent proliferation of human CD34+ progenitor cells in the absence of serum is suppressed by their progeny’s production of serine proteinases. Stem Cells. 24, 299–306.
van Galen P., Kreso A., Wienholds E., Laurenti E., Eppert K., Lechman E.R., Mbong N., Hermans K., Dobson S., April C., Fan J.B., Dick J.E. (2014) Reduced lymphoid lineage priming promotes human hematopoietic stem cell expansion. Cell Stem Cell. 14, 94–106.
Ikawa T., Masuda K., Huijskens M.J.A.J., Satoh R., Kakugawa K., Agata Y., Miyai T., Germeraad W.T.V., Katsura Y., Kawamoto H. (2015) Induced developmental arrest of early hematopoietic progenitors leads to the generation of leukocyte stem cells. Stem Cell Repts. 5, 716–727.
Lindsey S., Papoutsakis E.T. (2012) The evolving role of the aryl hydrocarbon receptor (AHR) in the normophysiology of hematopoiesis. Stem Cell Rev. Rep. 8, 1223–1235.
Bennett J.A., Singh K.P., Welle S.L., Boule L.A., Lawrence B.P., Gasiewicz T.A. (2018) Conditional deletion of Ahr alters gene expression profiles in hematopoietic stem cells. PLoS One. 13, e0206407.
Gasparetto M., Sekulovic S., Brocker C., Tang P., Zakaryan A., Xiang P., Kuchenbauer F., Wen M., Kasaian K., Witty M.F., Rosten P., Chen Y., Imren S., Duester G., Thompson D.C., Humphries R.K., Vasi-liou V., Smith C. (2012) Aldehyde dehydrogenases are regulators of hematopoietic stem cell numbers and B-cell development. Exp. Hematol. 40, 318–329.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология