Молекулярная биология, 2021, T. 55, № 2, стр. 258-268
Изучение свойств рекомбинантной эндонуклеазы IV Mycobacterium tuberculosis
М. А. Дымова a, А. В. Ендуткин a, В. В. Полуновский b, А. И. Закабунин a, Е. А. Храпов a, Н. А. Торгашева a, А. В. Юдкина a, Г. В. Мечетин a, М. Л. Филипенко a, Д. О. Жарков a, b, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
b Новосибирский государственный университет
630090 Новосибирск, Россия
* E-mail: dzharkov@niboch.nsc.ru
Поступила в редакцию 04.09.2020
После доработки 19.10.2020
Принята к публикации 20.10.2020
Аннотация
В клетках Mycobacterium tuberculosis присутствуют две апурин-апиримидиновых (АP-) эндонуклеазы: эндонуклеаза IV (MtbEnd) и экзонуклеаза III (MtbXthA), ‒ причем MtbEnd играет основную роль в защите микобактериальной ДНК в условиях окислительного стресса. Эндонуклеаза IV микобактерий значительно отличается от гомологов из Escherichia coli и других протеобактерий по ряду консервативных позиций, образующих структурные элементы связывания ДНК и узнавания AP-сайта. В работе клонирован ген end M. tuberculosis, а также выделен и исследован рекомбинантный белок MtbEnd. Белок эффективно гидролизовал ДНК по натуральному AP-сайту и его 1′-дезокси-аналогу в присутствии двухвалентных катионов, особенно Ca2+, Mn2+ и Co2+. Экзонуклеазная активность в препаратах фермента отсутствовала; оптимум pH приходился на 7.0–8.0, а оптимум ионной силы ‒ на ~50 мМ NaCl. Ферментативная активность MtbEnd была низкой по отношению к AP-сайтам, модифицированным химиотерапевтическим агентом метоксиамином. Результаты исследования позволяют рассматривать MtbEnd в качестве возможной мишени при разработке новых противотуберкулезных средств.
Патогенный микроорганизм Mycobacterium tuberculosis проводит значительную часть своего жизненного цикла внутри макрофагов организма-хозяина и постоянно подвергается действию производимых ими активных форм кислорода и азота [1]. Для выживания клетки в таких условиях особенно важно функционирование систем репарации ДНК [2]. Значимую роль в защите генома бактерий в этой ситуации играет система эксцизионной репарации оснований (ЭРО), так как большинство повреждений в условиях окислительного стресса представлено окисленными основаниями и апурин-апиримидиновыми (AP-) сайтами, исправляемыми именно по пути ЭРО [3]. Определение нуклеотидной последовательности генома M. tuberculosis и сравнение его с другими бактериальными геномами позволило выявить основные ферменты, участвующие в этой системе [4, 5].
В число главных элементов системы ЭРО входят AP-эндонуклеазы ‒ ферменты, гидролизующие фосфодиэфирную связь с 5′-стороны от AP-сайта с образованием 5′-фосфата и 3′-гидроксильной группы [6‒8]. Отсутствие этой активности приводит к резкому накоплению мутаций или к гибели клетки. В клетках Escherichia coli присутствуют две AP-эндонуклеазы: конститутивная экзонуклеаза III, кодируемая геном xthA, и индуцируемая окислительным стрессом эндонуклеаза IV (EcoNfo), кодируемая геном nfo. У M. tuberculosis есть гомологи обоих этих белков: экзонуклеазе III соответствует белок MtbXthA, кодируемый геном xthA, а эндонуклеазе IV ‒ белок MtbEnd, кодируемый геном end. Заметим, что в отличие от E. coli, где фермент Nfo выполняет вспомогательную функцию, основную роль в защите микобактериальной ДНК в условиях окислительного стресса играет именно эндонуклеаза IV [9]. В мышиной модели ген end входит в число необходимых для успешной инфекции M. tuberculosis [10], хотя в исследованиях с использованием морских свинок не обнаружено разницы между эффективностью инфекции штаммом дикого типа и штаммом с инактивированным геном end [11]. Однако активность MtbEnd может быть более важна в условиях лекарственной терапии. Ген end индуцируется при стрессе, вызванном обработкой M. tuberculosis ингибиторами синтеза микобактериальной клеточной стенки ‒ изониазидом и тиокарлидом [12]. При исследовании чувствительности библиотеки транспозонных мутантов M. tuberculosis к антибиотикам ген end оказался в числе 5% генов, инактивация которых приводит к повышенной чувствительности к β-лактамному антибиотику меропенему (одно из средств “последней линии” при туберкулезе с множественной лекарственной устойчивостью) при росте in vitro [13]. В стационарной же фазе и в инфицированных макрофагах токсичность многих антибиотиков с разным механизмом действия (рифампицина, ципрофлоксацина, стрептомицина) для M. tuberculosis усиливается за счет окисления пула dNTP, если система репарации ДНК не успевает удалять включаемые поврежденные дезоксинуклеотиды [14]. Совокупность полученных на сегодняшний день данных позволяет рассматривать MtbEnd в качестве возможной мишени для новых противотуберкулезных препаратов, сенсибилизирующих бактерии к действию антибиотиков.
Ферментативные свойства MtbEnd частично охарактеризованы [9, 15], однако различия в свойствах, описанных в этих публикациях, и значительные отличия от EcoNfo в отношении специфичности к двухвалентным катионам и оптимальных значений pH и ионной силы не позволяют считать характеристику MtbEnd полной и однозначной. Разница в описанных свойствах MtbEnd может быть обусловлена разными последовательностями пептидов для аффинной хроматографии, введенных в рекомбинантный белок (пептид Strep-tag в работе [9] или два олигогистидиновых пептида на обоих концах белка в работе [15]), и разными процедурами выделения. Сведения об ингибировании MtbEnd потенциальными фармакологически активными молекулами в литературе отсутствуют.
Цель работы заключалась в изучении свойств рекомбинантной эндонуклеазы MtbEnd при сведении к минимуму модификации последовательности белка и продолжительности процедуры выделения. Для этого ген end штамма M. tuberculosis H37Rv клонировали в вектор для суперпродукции белка с олигогистидиновым пептидом на C-конце. Оптимизированы условия индукции синтеза рекомбинантного белка MtbEnd в трансформированных клетках E. coli, процедуры очистки рекомбинантного белка и проведения ферментативной реакции. Наконец, исследована чувствительность MtbEnd к проходящему клинические испытания ингибитору AP-эндонуклеаз ‒ метоксиамину.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материалы. В работе использовали штаммы E. coli XL10 Gold Δ(mcrA)183 Δ(mcrCB-hsdSMR-mrr)173 endA1 glnV44 thi-1 recA1 gyrA96 relA1 lac Hte F′[proAB lacIqZΔM15 Tn10(Tetr Amy Cmr)] (“Agilent Technologies”, США) и SoluBL21(DE3) F–ompT hsdSB (${\text{r}}_{{\text{B}}}^{ - }{\text{m}}_{{\text{B}}}^{ - }$) gal dcm (DE3) (“Genlantis”, США), геномную ДНК штамма M. tuberculosis H37Rv (ФГБУ “Новосибирский научно-исследовательский институт туберкулеза”, Россия), плазмиду pET-23b (“Merck Millipore”, США). Работу проводили с рестриктазами FauNDI, Sfr274I, ДНК-лигазой бактериофага T4 (“Сибэнзим”, Россия), полинуклеотидкиназой бактериофага T4, ДНК-полимеразой Taq и Pfu (“Биосан”, Россия) и урацил-ДНК-гликозилазой E. coli (“New England Biolabs”, США). Рекомбинантный белок EcoNfo был любезно предоставлен А.А. Ищенко из Университета Париж-Саклэ (Université Paris-Saclay, Франция). Олигодезоксирибонуклеотиды, в том числе модифицированные, синтезированы в лаборатории биомедицинской химии ИХБФМ СО РАН. Структура олигонуклеотидов представлена в табл. 1. Мечение олигонуклеотидов 32P по 5′‑концу проводили полинуклеотидкиназой бактериофага T4 в условиях, рекомендованных производителем фермента. Двухцепочечные субстраты получали отжигом меченого олигонуклеотида 23X с 1.3‑кратным избытком комплементарного олигонуклеотида 23comp. Для получения субстратов, содержащих натуральный (альдегидный) AP-сайт, дуплекс U:C обрабатывали урацил-ДНК-гликозилазой (1 U) в течение 30 мин согласно инструкции производителя.
Таблица 1.
Последовательности олигодезоксирибонуклеотидов, использованных в работе
| Название | Последовательность (5′ → 3′)a |
|---|---|
| end1 | TGAACATATGCTCATTGGTTCGCATGTC |
| end2 | GAACTCGAGGCTGCCGGTTCTTTCCC |
| 23X (X = THF, U, G, oxoG)b | CTCTCCCTTCXCTCCTTTCCTCT |
| 23comp | AGAGGAAAGGAGCGAAGGGAGAG |
Клонирование гена end M. tuberculosis. Амплификацию фрагмента размером 772 п.о. проводили в 20 мкл реакционной смеси, содержащей 65 мМ Трис-HCl (рН 8.9), 16 мМ (NH4)2SO4, 1.8 мМ MgCl2, 0.05% Tween 20, 0.2 мМ dNTP, 1 мкM праймеры end1 и end2 (табл. 1), 1 U ДНК-полимеразы Taq, 1 U ДНК-полимеразы Pfu и 1–10 нг геномной ДНК M. tuberculosis. Процесс амплификации состоял из начальной денатурации при 95°C (3 мин), 33 циклов, каждый из которых включал денатурацию при 96°C (10 с), отжиг при 60°C (10 с) и элонгацию при 72°C (40 с), и завершался элонгацией при 72°C (3 мин). Амплифицированный ген end клонировали в вектор pET-23b по сайтам NdeI и XhoI. Полученная плазмида pET-23b-MtbEnd кодировала белок MtbEnd, несущий на C-конце пептид His6, под промотором РНК-полимеразы бактериофага T7. Нуклеотидную последовательность вставки подтверждали секвенированием по Сэнгеру.
Выделение белка MtbEnd. Клетки E. coli SoluBL21(DE3), трансформированные плазмидой pET-23b-MtbEnd, выращивали при 37°C на среде LB до оптической плотности (OD600) 0.3. Затем 40 мл полученной культуры вносили в 4 л питательной среды LB и растили при 37°C до OD600 0.6. Экспрессию индуцировали добавлением изопропил-β-D-1-тиогалактопиранозида до концентрации 1 мМ. После культивирования в течение 3 ч при 25°C клетки осаждали центрифугированием при 14 000 × g. Все дальнейшие процедуры по выделению фермента проводили на льду или при 4°C. Биомассу суспендировали в буфере, содержащем 50 мМ Трис-HCl (pH 7.5) и 0.5 M NaCl (буфер A). Клетки разрушали ультразвуком, суспензию центрифугировали при 14 000 × g. Осветленный лизат прогревали при 60°C в течение 15 мин, денатурированные белки отделяли центрифугированием при 14 000 × g в течение 15 мин. Супернатант наносили на металл-хелатный сорбент Profinity IMAC Resin (“Bio-Rad Laboratories” США), насыщенный ионами Ni2+, промывали сорбент буфером A и вели элюцию буфером A, содержащим 50 и 500 мМ имидазол. Фракции, содержащие белок нужной молекулярной массы, обессоливали гель-фильтрацией на сорбенте Bio-Gel P6 (“Bio-Rad Laboratories”), предварительно уравновешенном в буфере, содержащем 50 мМ Трис-HCl (pH 7.5) и 2 мМ дитиотреитол. К раствору белка добавляли равный объем холодного глицерина и хранили при −20°C. Концентрацию белка определяли по методу Брэдфорда.
Определение активности AP-эндонуклеаз. Анализ проводили в 10 мкл реакционной смеси, содержащей 50 мМ Трис-HCl (pH 7.8), 50 мМ NaCl и 0.5 мМ MnCl2 или хлориды других двухвалентных металлов, в присутствии 50 нМ двухцепочечного олигонуклеотидного субстрата. Для исследования pH-зависимости реакции Трис-HCl заменяли на 20 мМ Na-фосфатный буфер с нужным значением pH. Реакцию проводили при температуре 37°C. После остановки реакции добавлением равного объема раствора, содержащего 80%-ный формамид, 20 мМ ЭДТА (pH 8.0), 0.1%-ный ксиленцианол и 0.1%-ный бромфеноловый синий, с последующим прогреванием в течение 2 мин при 95°C продукты реакции разделяли при помощи гель-электрофореза в 20%-ном полиакриламидном геле в присутствии 8 М мочевины при 45°C. Гели визуализировали радиолюминесцентным сканированием на установке Typhoon FLA 9500 (“GE Healthcare”, США). Относительное содержание продукта в смеси определяли с использованием программы Gel-Pro Analyzer v4.0 (“Media Cybernetics”, США). Результаты представлены как среднее и стандартное отклонение по трем независимым экспериментам.
Анализ консервативности в семействе Nfo. Биоинформатический анализ проводили на таксономически сбалансированной выборке из 93 полипептидных последовательностей, из которых 48 происходили из типа Actinobacteria, а 45 – из типа Proteobacteria. Для составления выборки вели поиск при помощи алгоритма blastp [16] в базе данных RefSeq Национального центра биотехнологической информации США [17], используя в качестве запроса последовательность MtbEnd для актинобактерий и EcoNfo для протеобактерий. В выборке оставляли не более 1 последовательности на таксономическое семейство. Последовательности выравнивали при помощи программы Clustal Omega [18]. Консервативность физико-химических свойств в позициях анализировали при помощи программы AMAS [19] с использованием стандартного набора Тейлора для физико-химических свойств аминокислот [20], допуская наличие не более 10% атипичных остатков. Для визуализации дерева последовательностей использовали программу iTOL [21]. Расчет значений pKa остатков, образующих сайты связывания ионов двухвалентных металлов (M2+), проводили в программе DelPhiPKa, реализующей решение уравнения Пуассона–Больцмана [22, 23], при концентрации соли 0.15 М в силовом поле AMBER для структур 1QTW для EcoNfo [24] и 5ZHZ для MtbEnd [25].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Анализ консервативности в семействе эндонуклеазы IV
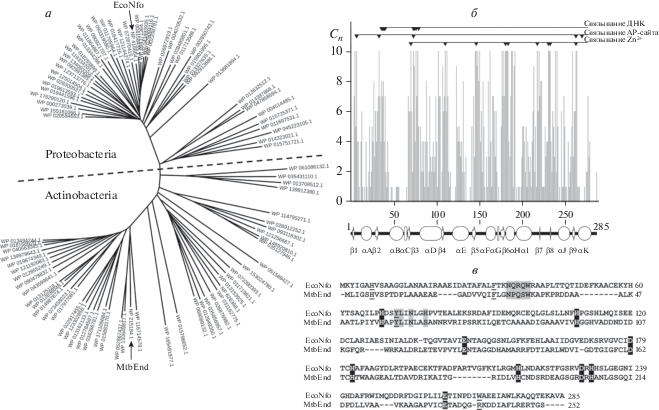
Последовательности, гомологичные эндонуклеазе IV, широко распространены среди бактерий. Анализ 93 последовательностей из типов Actinobacteria и Proteobacteria (рис. 1а, б) показал достаточно высокую консервативность физико-химических свойств аминокислот, в особенности в областях, лежащих в петлях между элементами вторичной структуры и несущих функционально важные остатки, принимающие участие в связывании ДНК, узнавании AP-сайта и координации ионов металлов. Однако некоторые остатки, идентифицированные как важные по результатам рентгеноструктурного анализа EcoNfo [24, 26], оказались менее консервативными. В частности, это относится к одной из петель, связывающей ДНК в малой бороздке с 3′-стороны от AP-сайта (35NQRQW39 в EcoNfo), а также к остатку Trp268 (в EcoNfo), который образует стенку кармана, узнающего AP-сайт (рис. 1б, в). В обоих случаях упомянутые позиции абсолютно или высококонсервативны в последовательностях из протеобактерий, но либо не консервативны среди актинобактерий, либо консервативны со значительным изменением физико-химических свойств. Аналогичная картина выявлена для ряда остатков, сближенных в пространственной структуре с петлей 35NQRQW39 и остатком Trp268. Таким образом, структурные элементы, отвечающие за узнавание поврежденной ДНК, частично реорганизованы в белке MtbEnd по сравнению с EcoNfo, и можно ожидать, что ферментативные свойства этих белков тоже не совсем идентичны.
Рис. 1.
Консервативность последовательности белков Nfo. а ‒ Неукорененная дендрограмма последовательностей гомологов эндонуклеазы IV из протеобактерий и актинобактерий. б ‒ График зависимости параметра консервативности Cn от положения в последовательности (0 ≤ Cn ≤ 10, где Cn = 0 при полном отсутствии консервативности физико-химических свойств аминокислотных остатков и Cn = 10 при полной консервативности во всем выравнивании). График построен для последовательности EcoNfo, элементы вторичной структуры приведены согласно пространственной структуре комплекса EcoNfo с ДНК (1QUM [24]). в ‒ Выравнивание последовательностей EcoNfo и MtbEnd. Черным цветом отмечены остатки, принимающие участие в координации ионов металлов, серым — связывающие ДНК, подчеркнуты остатки, принимающие участие в узнавании AP-сайта.
Клонирование гена end и выделение рекомбинантной эндонуклеазы IV M. tuberculosis
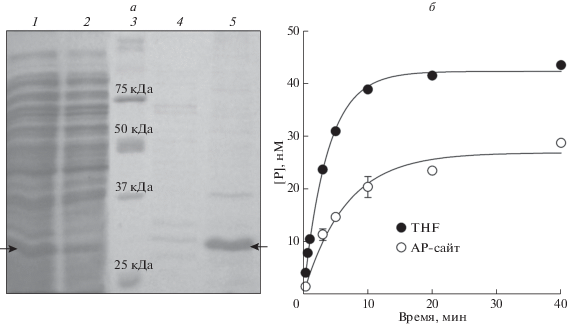
Нуклеотидная последовательность амплифицированного фрагмента, содержащего ген end, совпала с последовательностью из штамма M. tuberculosis H37Rv в базе данных GenBank (геномный идентификатор NC_000962.3), за исключением запланированной замены G→A в первом положении инициирующего кодона. Индуцированный синтез MtbEnd в клетках E. coli сопровождался образованием телец включения. Содержание фермента в растворимой фракции увеличивалось при понижении температуры культивирования с 37°C до 25°C, поэтому при получении биомассы культивирование проводили при пониженной температуре. Одной из задач исследования была разработка протокола быстрого выделения MtbEnd с минимальным числом стадий, так как хроматографические процедуры и продолжительный диализ могут приводить к утере двухвалентных катионов из активного центра белка. В связи с тем, что гомологичный белок Nfo из E. coli отличается повышенной термостабильностью [27], в качестве первого шага выделения использовали тепловую денатурацию осветленного лизата, что позволило избавиться от большей части примесных белков и, в частности, от экзонуклеазной активности в препарате, которую наблюдали ранее при работе с высокими концентрациями белка [9, 15]. После металл-хелатной хроматографии и обессоливания целевой белок с молекулярной массой ~28 кДа (расчетная молекулярная масса 27 910 Да) имел гомогенность >90% (рис. 2а) и не содержал неспецифичной нуклеазной активности. Фермент активно расщеплял 23-звенные олигонуклеотидные дуплексы, содержащие натуральный AP-сайт или его синтетический 1′-дезокси-аналог (3-гидрокситетрагидрофуран-2-ил)метилфосфат (THF; рис. 2б), и не проявлял активности при наличии в этих позициях нормального 2′-дезоксигуанозина или его окисленного производного 8-оксо-2′-дезок-сигуанозина. Гидролиз THF-субстрата был более эффективен, чем гидролиз натурального AP-сайта. Расчетные значения наблюдаемой константы скорости реакции в условиях избытка фермента (рис. 2б) составили 0.27 ± 0.01 мин−1 для субстрата THF:C и 0.14 ± 0.02 мин−1 для субстрата AP:C. По литературным данным, MtbEnd, в отличие от EcoNfo, проявляет повышенную специфичность к C и сниженную специфичность к T напротив продукта апуринизации (которой более всего подвержены нуклеозиды dG) [9], что ожидаемо для микобактерий и других организмов с высоким содержанием GC-пар в ДНК.
Рис. 2.
Выделение и ферментативная активность белка MtbEnd. а ‒ Электрофоретический анализ фракций в ходе выделения рекомбинантного белка MtbEnd: экстракт после прогревания (дорожка 1); белок, не связавшийся с металлхелатным сорбентом (дорожка 2); маркеры подвижности (дорожка 3); фракция, элюированная 50 мМ имидазолом (дорожка 4); фракция, элюированная 500 мМ имидазолом (дорожка 5). Стрелками отмечена полоса, соответствующая по подвижности белку MtbEnd. б ‒ Кинетика накопления продукта реакции гидролиза ферментом MtbEnd субстратов, содержащих натуральный AP-сайт и его аналог THF. Реакцию проводили в присутствии 2.4 мкМ MtbEnd, 50 нМ двухцепочечного субстрата и 0.5 мМ MnCl2.
Влияние ионов металлов на ферментативную активность MtbEnd
Белок EcoNfo представляет собой металлофермент, нуждающийся в ионах двухвалентных металлов (M2+) для поляризации связей P–O гидролизуемого фосфата и активации реагирующей молекулы воды [26, 28–30]. Однако требования EcoNfo к природе М2+ до конца не выяснены. Нативный белок, выделяемый из клеток E. coli без гетерологичной экспрессии, содержит 2.4–2.5 ио-нов Zn2+, 0.6–0.8 Mn2+, 0.1–0.4 Fe2+ и ~0.2 Cu2+ на молекулу, прочие ионы M2+ присутствуют в количестве <0.1 на молекулу белка [29]. Показано, что ферментативная активность Nfo сохраняется в отсутствие ионов M2+ в реакционной смеси, но подавляется в присутствии хелатирующих агентов [28, 29]. При реактивации ионами M2+ после хелатирования активность EcoNfo выше в присутствии Mn2+ и Co2+, чем в присутствии Zn2+ [29]. В кристаллах Nfo из разных видов бактерий три металлсвязывающих центра белка могут быть заняты в разных комбинациях ионами Zn2+, Mn2+, Fe2+ и Cd2+, включенными из кристаллизационного раствора [24‒26, 31‒33]. По данным структурных исследований в одном из металлсвязывающих центров обмен иона M2+ с раствором облегчен [26, 33]. В целом, EcoNfo присущи свойства металлзависимого фермента с умеренным сродством к каталитическим ионам M2+ и оптимальной композицией ионов 2Zn2+/1Mn2+.
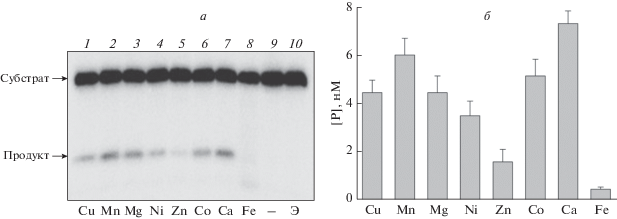
Аминокислотные остатки, координирующие ионы металлов, идентичны в белках EcoNfo и MtbEnd. Учитывая использование металлхелатной хроматографии для выделения MtbEnd, мы не можем исключить, что часть каталитических ионов была утеряна в ходе выделения и может быть замещена ионами из реакционной смеси. В связи с этим нами проанализирована активность MtbEnd в присутствии разных ионов M2+. На рис. 3 представлены данные о влиянии ионов Zn2+, Сa2+, Ni2+, Cu2+, Mn2+, Mg2+, Co2+ и Fe2+ на гидролиз дуплекса THF:C. Показано, что в присутствии 5 мМ ЭДТА ферментативная активность MtbEnd полностью исчезает (рис. 3а, дорожка 10). Наибольшую активность фермент проявлял в присутствии Ca2+, несколько меньшую ‒ при наличии в растворе ионов Mn2+ и Co2+, что для этих двух ионов согласуется с литературными данными по EcoNfo [26, 29] (рис. 3б). Как и в случае EcoNfo, в присутствии одних только ионов Zn2+ AP-эндонуклеазная активность MtbEnd была низкой. Фермент был умеренно активен при наличии в реакционной смеси ионов Cu2+ и Ni2+, а в присутствии Fe2+ регистирировали низкую степень специфического расщепления, сопровождавшегося дополнительной деградацией продукта (рис. 3а). В присутствии других ионов M2+ такой деградации не наблюдалось, в отличие от ранних сообщений о наличии 3′ → 5′-экзонуклеазной активности в препаратах MtbEnd [9]. Возможно, стадия тепловой денатурации в процессе выделения белка позволила избавиться от примесной экзонуклеазной активности.
Рис. 3.
Ферментативная активность MtbEnd в присутствии ионов двухвалентных металлов. а ‒ Электрофоретический анализ продуктов реакции гидролиза дуплекса THF:C в присутствии ионов двухвалентных металлов (дорожки 1–8). Дорожка 9 – субстрат в отсутствие фермента, дорожка 10 – реакция в присутствии ЭДТА (Э). б ‒ Выход продукта реакции в присутствии ионов двухвалентных металлов. Реакцию проводили в присутствии 0.5 мМ хлорида соответствующего двухвалентного металла, 100 нM MtbEnd и 50 нМ субстрата THF:C в течение 10 мин.
Интересно, что по литературным данным ионы Mg2+ и Ca2+, в присутствии которых MtbEnd проявляла высокую гидролитическую активность, не поддерживают активность EcoNfo [29]. Для MtbEnd ранее приводили следующие ряды “предпочтений”: Mg2+ > Ca2+ ~ Mn2+ > Co2+$ \gg $ Zn2+, Fe2+, Ni2+ [15] и Mg2+ ~ Ca2+ > Mn2+ > Zn2+, Co2+ [9]. Некоторые различия в результатах связаны, по всей видимости, с разным расположением пептидов для аффинной хроматографии и с разными протоколами выделения и хранения рекомбинантного белка, что приводит к разной остаточной насыщенности сайтов связывания металлов эндогенными ионами M2+. Для объяснения возможных причин разной металлспецифичности EcoNfo и MtbEnd был проведен расчет значений pKa остатков, образующих сайты связывания ионов M2+ в структурах этих белков (табл. 2). Основные различия обнаружены в области связывания иона 1 ‒ одного из двух ионов, поляризующих связи P=O в фосфатной группе AP-сайта и активирующих молекулу воды для нуклеофильной атаки по атому фосфора [26, 30]. Значения pKa остатков His56 и His96 в MtbEnd были соответственно на 0.37 и 0.24 единицы меньше, чем для гомологичных им остатков His69 и His109 в EcoNfo. Более выраженное депротонирование остатков His при нейтральном pH может приводить к более эффективному связыванию ионов M2+ в сайте 1 белка MtbEnd по сравнению с EcoNfo.
Таблица 2.
Расчетные значения pKa остатков, образующих сайты связывания ионов M2+ в структурах EcoNfo и MtbEnd
| Номер M2+-связывающего сайта | EcoNfo | MtbEnd | ||
|---|---|---|---|---|
| а.о.а | pKa | а.о. | pKa | |
| 1 | His69 | 7.14 | His56 | 6.77 |
| 1 | His109 | 6.74 | His96 | 6.50 |
| 1, 3 | Glu145 | 1.60 | Glu129 | 1.56 |
| 3 | Asp179 | 2.32 | Asp162 | 1.82 |
| 2 | His182 | 7.63 | His165 | 7.60 |
| 3 | His216 | 7.34 | His191 | 7.43 |
| 2 | Asp229 | 1.94 | Asp204 | 1.89 |
| 2 | His231 | 7.26 | His206 | 7.28 |
| 3 | Glu261 | 2.11 | Glu233 | 2.30 |
Как видно из данных, приведенных на рис. 3, присутствие в реакционной смеси ионов Mn2+ ассоциировано с достаточно высокой гидролитической активностью как MtbEnd, так и гомологичного белка EcoNfo, поэтому в дальнейших экспериментах использовали буфер, содержащий 0.5 мМ MnCl2.
Влияние pH и ионной силы на ферментативную активность MtbEnd
Изменение концентрации ионов водорода оказывает выраженное влияние на скорость многих ферментативных реакций за счет изменения состояния ионизации боковых радикалов аминокислот в активном центре фермента. Таким образом, значение рH среды влияет на способность боковых радикалов служить донорами или акцепторами протонов и координировать ионы металлов. В частности, значения pKa металлкоординирующих остатков His EcoNfo лежат в диапазоне 5.4–8.2 для белка в свободном состоянии и могут изменяться на 1–2 единицы pH при связывании ДНК [15]. Для EcoNfo pH-оптимум ферментативной активности гидролиза AP-сайта лежит в области 8.0–8.5 [34]. Для установления зависимости ферментативной активности MtbEnd от рH в отношении субстрата THF:C, проводили реакции в растворах, содержащих 25 мМ Na-фосфатный буфер в диапазоне рH 4.0–9.0 (рис. 4а). MtbEnd гидролизовала субстрат при всех значениях pH, при этом максимальные различия составляли около 3 раз. Зависимость активности от pH имела максимум в области pH 7–8, что согласуется с наличием как минимум двух критических для катализа ионизируемых аминокислотных остатков в активном центре фермента. Очевидными кандидатами на роль в возрастании активности с переходом в области pH 6 служат остатки His в позициях 56, 96, 165, 191 и 206, так как они координируют ионы M2+; а также остаток His6, принимающий участие в узнавании AP-сайта (рис. 1в). Падение активности фермента при дальнейшем повышении pH может быть связано либо с общей дестабилизацией укладки полипептидной цепи, либо с ионизацией остатка Tyr59, который в норме заполняет полость в ДНК после выворачивания AP-сайта [24].
Рис. 4.
Зависимость ферментативной активности MtbEnd от pH (а) и концентрации NaCl (б). Реакцию проводили в присутствии 0.5 нМ MtbEnd и 50 нМ субстрата THF:C в течение 30 мин.
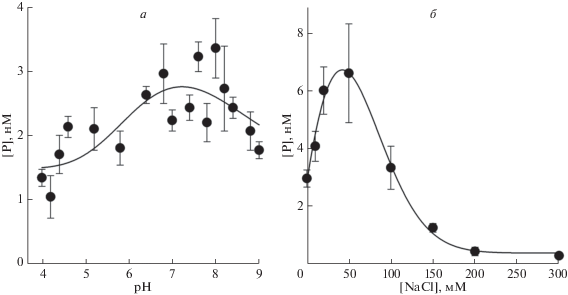
Влияние ионной силы раствора на гидролиз дуплекса (при pH 8.0) представлено на рис. 4б. Ферментативная активность MtbEnd имела выраженный оптимум при концентрации NaCl ~ 50 мМ, что сравнимо с оптимумом 50‒100 мМ для EcoNfo [28]. Следует отметить, что области белка, контактирующие с ДНК, у MtbEnd содержат меньше положительно заряженных аминокислотных остатков, чем у EcoNfo (рис. 1в), и этим может быть обусловлен некоторый сдвиг оптимума в область более низких значений ионной силы.
Гидролитическая активность MtbEnd по AP-сайтам, модифицированным метоксиамином
Метоксиамин представляет собой небольшую молекулу, которая реагирует с альдегидной группой в составе AP-сайтов, что предотвращает их расщепление AP-эндонуклеазой человека APEX1 [35, 36]. Вследствие этого метоксиамин усиливает цитотоксичность широкого спектра терапевтических агентов, повреждающих ДНК, и в настоящее время проходит клинические испытания как сенситизатор опухолевых клеток [37–40]. Для белка EcoNfo сообщалось как об умеренном подавлении активности при реакции AP-сайтов с метоксиамином [41], так и об отсутствии эффекта [42, 43]; для MtbEnd такие данные отсутствуют. Для сравнительной оценки влияния такой модификации на активность ферментов MtbEnd и EcoNfo нами проанализировано расщепление субстратов AP:C и THF:C в присутствии 0.01–10 мМ метоксиамина. Учитывая, что метоксиамин не ингибирует ферменты непосредственно, а модифицирует нативный AP-сайт, мы ожидали, что активность AP-эндонуклеаз снизится в отношении субстрата AP:C, но не THF:C. Результаты эксперимента полностью подтвердили предполагаемый механизм: наблюдалось зависимое от концентрации метоксиамина снижение количества продукта гидролиза AP:C, в то время как при любой концентрации метоксиамина оба фермента эффективно гидролизовали THF:C (рис. 5а, б). Заметим, что в сравнении с EcoNfo для MtbEnd выявлена тенденция к повышенной чувствительности к метоксиамину (рис. 5а).
Рис. 5.
Зависимость ферментативной активности MtbEnd и EcoNfo от концентрации метоксиамина при использовании в качестве субстратов AP:C (а) и THF:C (б).
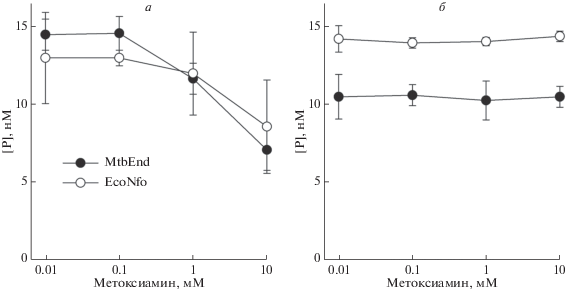
Таким образом, нами показано, что рекомбинантный белок MtbEnd проявляет высокую ферментативную активность в присутствии ионов Ca2+, Mn2+, Со2+ и в несколько меньшей степени ‒ Mg2+ или Cu2+, что сильно отличает его от эндонуклеазы IV из E. coli. Максимальная ферментативная активность MtbEnd наблюдалась при концентрации NaCl ~50 мМ и рH 7.0‒8.0, что свидетельствует о смещении оптимума в сторону пониженной ионной силы и оснóвности раствора по сравнению с ферментом из E. coli. Модификация AP-сайтов метоксиамином ‒ потенциальным противоопухолевым терапевтическим агентом ‒ подавляла ферментативную активность MtbEnd на натуральном субстрате. Зная, что MtbEnd представляет собой главную AP-эндонуклеазу M. tuberculosis, можно рассматривать метоксиамин и другие соединения алкоксиаминового ряда как потенциальные лекарственные препараты для терапии микобактериальных инфекций.
Работа частично поддержана проектами базового бюджетного финансирования ПФНИ ГАН № АААА-А17-117020210027-9 (в части клонирования и выделения MtbEnd), АААА-А17-117020210023-1 (в части характеристики MtbEnd) и FSUS-2020-0035 (в части биоинформатического анализа и молекулярного моделирования белков Nfo). Секвенирование выполнено в ЦКП “Геномика” СО РАН.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Saviola B., Bishai W. (2006) The genus Mycobacterium ‒ medical. In: The Prokaryotes. Ed. Dworkin M. New York: Springer, p. 919‒933.
Fang F.C. (2004) Antimicrobial reactive oxygen and nitrogen species: concepts and controversies. Nat. Rev. Microbiol. 2, 820‒832.
David S.S., O’Shea V.L., Kundu S. (2007) Base-excision repair of oxidative DNA damage. Nature. 447, 941‒950.
Mizrahi V., Andersen S.J. (1998) DNA repair in Mycobacterium tuberculosis. What have we learnt from the genome sequence? Mol. Microbiol. 29, 1331‒1339.
Kurthkoti K., Varshney U. (2011) Base excision and nucleotide excision repair pathways in mycobacteria. Tuberculosis. 91, 533‒543.
Demple B., Harrison L. (1994) Repair of oxidative damage to DNA: enzymology and biology. Annu. Rev. Biochem. 63, 915‒948.
Mol C.D., Hosfield D.J., Tainer J.A. (2000) Abasic site recognition by two apurinic/apyrimidinic endonuclease families in DNA base excision repair: the 3′ ends justify the means. Mutat. Res. 460, 211‒229.
Королев В.Г. (2005) Эксцизионная репарация поврежденных оснований ДНК: АП-эндонуклеазы и ДНК-полимеразы. Генетика. 41(10), 1301‒1309.
Puri R.V., Singh N., Gupta R.K., Tyagi A.K. (2013) Endonuclease IV is the major apurinic/apyrimidinic endonuclease in Mycobacterium tuberculosis and is important for protection against oxidative damage. PLoS One. 8, e71535.
Sassetti C.M., Rubin E.J. (2003) Genetic requirements for mycobacterial survival during infection. Proc. Natl. Acad. Sci. USA. 100, 12989‒12994.
Puri R.V., Reddy P.V., Tyagi A.K. (2014) Apurinic/ apyrimidinic endonucleases of Mycobacterium tuberculosis protect against DNA damage but are dispensable for the growth of the pathogen in guinea pigs. PLoS One. 9, e92035.
Waddell S.J., Stabler R.A., Laing K., Kremer L., Rey-nolds R.C., Besra G.S. (2004) The use of microarray analysis to determine the gene expression profiles of Mycobacterium tuberculosis in response to anti-bacterial compounds. Tuberculosis. 84, 263‒274.
Xu W., DeJesus M.A., Rücker N., Engelhart C.A., Wright M.G., Healy C., Lin K., Wang R., Park S.W., Ioerger T.R., Schnappinger D., Ehrt S. (2017) Chemical genetic interaction profiling reveals determinants of intrinsic antibiotic resistance in Mycobacterium tuberculosis. Antimicrob. Agents Chemother. 61, e01334-17.
Fan X.-Y., Tang B.-K., Xu Y.-Y., Han A.-X., Shi K.-X., Wu Y.-K., Ye Y., Wei M.-l., Niu C., Wong K.-W., Zhao G.-P., Lyu L.-D. (2018) Oxidation of dCTP contributes to antibiotic lethality in stationary-phase mycobacteria. Proc. Natl. Acad. Sci. USA. 115, 2210‒2215.
Abeldenov S., Talhaoui I., Zharkov D.O., Ishchenko A.A., Ramanculov E., Saparbaev M., Khassenov B. (2015) Characterization of DNA substrate specificities of apurinic/apyrimidinic endonucleases from Mycobacterium tuberculosis. DNA Repair. 33, 1‒16.
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res. 25, 3389‒3402.
O'Leary N.A., Wright M.W., Brister J.R., Ciufo S., Haddad D., McVeigh R., Rajput B., Robbertse B., Smith-White B., Ako-Adjei D., Astashyn A., Badretdin A., Bao Y., Blinkova O., Brover V., Chetvernin V., Choi J., Cox E., Ermolaeva O., Farrell C.M., Goldfarb T., Gupta T., Haft D., Hatcher E., Hlavina W., Joardar V.S., Kodali V.K., Li W., Maglott D., Masterson P., McGarvey K.M., Murphy M.R., O’Neill K., Pujar S., Rangwala S.H., Rausch D., Riddick L.D., Schoch C., Shkeda A., Storz S.S., Sun H., Thibaud-Nissen F., Tolstoy I., Tully R.E., Vatsan A.R., Wallin C., Webb D., Wu W., Landrum M.J., Kimchi A., Tatusova T., DiCuccio M., Kitts P., Murphy T.D., Pruitt K.D. (2016) Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation. Nucleic Acids Res. 44, D733‒D745.
Sievers F., Wilm A., Dineen D., Gibson T.J., Karplus K., Li W., Lopez R., McWilliam H., Remmert M., Söding J., Thompson J.D., Higgins D.G. (2011) Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Mol. Syst. Biol. 7, 539.
Livingstone C.D., Barton G.J. (1993) Protein sequence alignments: a strategy for the hierarchical analysis of residue conservation. Comput. Appl. Biosci. 9, 745‒756.
Taylor W.R. (1986) The classification of amino acid conservation. J. Theor. Biol. 119, 205‒218.
Letunic I., Bork P. (2016) Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic Acids Res. 44, W242‒W245.
Wang L., Li L., Alexov E. (2015) pKa predictions for proteins, RNAs, and DNAs with the Gaussian dielectric function using DelPhi pKa. Proteins. 83, 2186‒2197.
Pahari S., Sun L., Basu S., Alexov E. (2018) DelPhiPKa: including salt in the calculations and enabling polar residues to titrate. Proteins. 86, 1277‒1283.
Hosfield D.J., Guan Y., Haas B.J., Cunningham R.P., Tainer J.A. (1999) Structure of the DNA repair enzyme endonuclease IV and its DNA complex: double-nucleotide flipping at abasic sites and three-metal-ion cata-lysis. Cell. 98, 397‒408.
Zhang W., Xu Y., Yan M., Li S., Wang H., Yang H., Zhou W., Rao Z. (2018) Crystal structure of the apuri-nic/apyrimidinic endonuclease IV from Mycobacterium tuberculosis. Biochem. Biophys. Res. Commun. 498, 111‒118.
Garcin E.D., Hosfield D.J., Desai S.A., Haas B.J., Björas M., Cunningham R.P., Tainer J.A. (2008) DNA apurinic-apyrimidinic site binding and excision by endonuclease IV. Nat. Struct. Mol. Biol. 15, 515‒522.
Ljungquist S. (1980) Endonuclease IV from Escherichia coli. Methods Enzymol. 65, 212‒216.
Levin J.D., Johnson A.W., Demple B. (1988) Homogeneous Escherichia coli endonuclease IV: characterization of an enzyme that recognizes oxidative damage in DNA. J. Biol. Chem. 263, 8066‒8071.
Levin J.D., Shapiro R., Demple B. (1991) Metalloenzymes in DNA repair: Escherichia coli endonuclease IV and Saccharomyces cerevisiae Apn1. J. Biol. Chem. 266, 22893‒22898.
Ivanov I., Tainer J.A., McCammon J.A. (2007) Unra-veling the three-metal-ion catalytic mechanism of the DNA repair enzyme endonuclease IV. Proc. Natl. Acad. Sci. USA. 104, 1465‒1470.
Tomanicek S.J., Hughes R.C., Ng J.D., Coates L. (2010) Structure of the endonuclease IV homologue from Thermotoga maritima in the presence of active-site divalent metal ions. Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun. 66, 1003‒1012.
Asano R., Ishikawa H., Nakane S., Nakagawa N., Kuramitsu S., Masui R. (2011) An additional C-terminal loop in endonuclease IV, an apurinic/apyrimidinic endonuclease, controls binding affinity to DNA. Acta Crystallogr. D Biol. Crystallogr. 67, 149‒155.
Tsutakawa S.E., Shin D.S., Mol C.D., Izumi T., Arvai A.S., Mantha A.K., Szczesny B., Ivanov I.N., Hosfield D.J., Maiti B., Pique M.E., Frankel K.A., Hitomi K., Cunningham R.P., Mitra S., Tainer J.A. (2013) Conserved structural chemistry for incision activity in structurally non-homologous apurinic/apyrimidinic endonuclease APE1 and endonuclease IV DNA repair enzymes. J. Biol. Chem. 288, 8445‒8455.
Ljungquist S. (1977) A new endonuclease from Escherichia coli acting at apurinic sites in DNA. J. Biol. Chem. 252, 2808‒2814.
Liuzzi M., Talpaert-Borlé M. (1985) A new approach to the study of the base-excision repair pathway using methoxyamine. J. Biol. Chem. 260, 5252‒5258.
Wei S., Shalhout S., Ahn Y.-H., Bhagwat A.S. (2015) A versatile new tool to quantify abasic sites in DNA and inhibit base excision repair. DNA Repair. 27, 9‒18.
Liu L., Gerson S.R. (2004) Therapeutic impact of methoxyamine: blocking repair of abasic sites in the base excision repair pathway. Curr. Opin. Investig. Drugs. 5, 623‒627.
van Midwoud P.M., Sturla S.J. (2013) Improved efficacy of acylfulvene in colon cancer cells when combined with a nuclear excision repair inhibitor. Chem. Res. Toxicol. 26, 1674‒1682.
Oleinick N.L., Biswas T., Patel R., Tao M., Patel R., Weeks L., Sharma N., Dowlati A., Gerson S.L., Fu P., Zhang J., Machtay M. (2016) Radiosensitization of non-small-cell lung cancer cells and xenografts by the interactive effects of pemetrexed and methoxyamine. Radiother. Oncol. 121, 335‒341.
Caimi P.F., Cooper B.W., William B.M., Dowlati A., Barr P.M., Fu P., Pink J., Xu Y., Lazarus H.M., de Lima M., Gerson S.L. (2017) Phase I clinical trial of the base excision repair inhibitor methoxyamine in combination with fludarabine for patients with advanced hematologic malignancies. Oncotarget. 8, 79864‒79875.
Bailly V., Verly W.G. (1989) The multiple activities of Escherichia coli endonuclease IV and the extreme labi-lity of 5′-terminal base-free deoxyribose 5-phosphates. Biochem. J. 259, 761‒768.
Cunningham R.P., Saporito S.M., Spitzer S.G., Weiss B. (1986) Endonuclease IV (nfo) mutant of Escherichia coli. J. Bacteriol. 168, 1120‒1127.
Rosa S., Fortini P., Karran P., Bignami M., Dogliotti E. (1991) Processing in vitro of an abasic site reacted with methoxyamine: a new assay for the detection of abasic sites formed in vivo. Nucleic Acids Res. 19, 5569‒5574.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


