Молекулярная биология, 2021, T. 55, № 2, стр. 269-276
5'‑Дезоксирибозофосфатлиазная активность апуриновой/апиримидиновой эндонуклеазы 1
Е. С. Ильина a, b, О. И. Лаврик a, b, С. Н. Ходырева a, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
b Новосибирский национальный исследовательский государственный университет
630090 Новосибирск, Россия
* E-mail: svetakh@niboch.nsc.ru
Поступила в редакцию 27.08.2020
После доработки 16.10.2020
Принята к публикации 22.10.2020
Аннотация
Одно из наиболее часто встречающихся повреждений ДНК ‒ появление апуриновых/апиримидиновых (АР-) сайтов. Основной путь репарации АР-сайтов инициирует апуриновая/апиримидиновая эндонуклеаза 1 (АРЕ1). При гидролизе этим ферментом фосфодиэфирной связи с 5′-стороны от АР-сайта образуется однонуклеотидная брешь, фланкированная 3′-гидроксильной и 5′-дезоксирибозофосфатной (5′-dRP) группой. Известно, что после гидролиза АР-сайта АРЕ1 некоторое время остается связанной с продуктом. Этот этап работы АРЕ1 стал предметом проведенного нами исследования, в результате которого обнаружена способность АРЕ1 формировать продукт ковалентного присоединения к цепи ДНК, содержащей брешь с 5′-dRP-группой, которая обращена в сторону бреши. Кроме того, установлено, что, оставаясь в комплексе с продуктом гидролиза АР-сайта, АРЕ1 проявляет 5'-dRP-лиазную активность, катализируя отщепление остатка 5′-dRP. Ранее в литературе не было сообщений на эту тему. Наличие у АРЕ1 лиазной активности может быть важным моментом при репарации АР-сайтов в случае дефицита или мутаций ДНК-полимеразы β ‒ основного фермента, удаляющего 5′-dRP в процессе репарации поврежденных оснований.
Апуриновые/апиримидиновые (АР-) сайты – один из наиболее часто встречающихся типов повреждений ДНК. Основной путь их метаболизма в клетке – это гидролиз апуриновой/апиримидиновой эндонуклеазой 1 (АРЕ1) [1]. APE1 расщепляет сахарофосфатный остов c 5′-стороны от АР-сайта в ходе реакции гидролиза с образованием остатка дезоксирибозы на 5′-конце олигонуклеотида, фланкирующего брешь. Ранее нами была обнаружена способность АРЕ1 образовывать основание Шиффа с АР-сайтами [2]. Также известно, что АРЕ1 после гидролиза АР-сайта образует достаточно стабильный комплекс с продуктом реакции [1, 2], предположительно в основном за счет взаимодействия с остатком дезоксирибозы на 5′-конце бреши.
Учитывая это обстоятельство, мы предположили возможность формирования АРЕ1 основания Шиффа с дезоксирибозофосфатным остатком на 5′-конце олигонуклеотида, фланкирующего брешь. Для детекции таких продуктов необходимо вводить радиоактивную метку, например, на 3′-конец олигонуклеотида, содержащего 5′-дезоксирибозофосфат (5′-dRP). Варьируя положение радиоактивной метки на 3′- или 5′-конце олигонуклеотидов, формирующих дуплекс ДНК, нам удалось с помощью электрофореза в ПААГ в денатурирующих условиях зарегистрировать меченые АРЕ1-продукты, которые могли быть образованы в результате ковалентного присоединения АРЕ1 к ДНК, содержащей брешь, фланкированную 5′-dRP-группой. Время жизни продуктов ковалентного присоединения АРЕ1 к таким ДНК не превышало 15 мин. Кроме того, установлено, что, оставаясь в комплексе с продуктом гидролиза АР-сайта, АРЕ1 проявляет 5ʹ-dRP-лиазную активность, отщепляя 5′-dRP-остаток и оставаясь связанной с ним, по крайней мере, в течение 30 мин.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материалы. Синтетические олигонуклеотиды синтезированы в Лаборатории биомедицинской химии (ИХБФМ СО РАН). В работе использованы [γ-32P]ATP и [α-32P]dАTP (>3000 Ки/ммоль) – продукция Лаборатории биотехнологии ИХБФМ СО РАН; Т4-полинуклеотидкиназа, урацил-ДНК-гликозилаза Escherichia coli (UDG) (“Биосан”, Россия), маркеры молекулярных масс белков (“Thermo Fisher Scientific”, США). Плазмиды, содержащие кДНК APE1 человека и ДНК-полимеразы β (Polβ) крысы, любезно предоставлены Dr. S. Wilson (National Institute of Environmental Health Sciences, National Institutes of Health, США. Рекомбинантные белки APE1 и Polβ были выделены по ранее описанным методикам ([3] и [4] соответственно).
Получение AP- и 5'-dRP-ДНК. Последовательности использованных ДНК-дуплексов приведены в табл. 1, где D – остаток диэтиленгликоля, синтетический аналог АР-сайта [5].
Формирование ДНК-дуплексов, введение [32P]-метки на 5′-конец олигонуклеотидов, обработку UDG для получения АР- и 5′‑dRP-ДНК проводили по ранее опубликованной методике [6]. Так, введение 5′-[32P]-метки осуществляли с использованием Т4-полинуклеотидкиназы. Для получения ДНК-дуплексов соответствующие олигодезоксирибонуклеотиды смешивали в эквимолярном соотношении в буфере, содержащем 10 мM Трис-HCl (pH 8.0) и 1 мM EDTA, реакционную смесь нагревали в течение 5 мин при 90°C, а затем медленно снижали температуру до комнатной. UDG-обработку ДНК, содержащей dUMP, проводили непосредственно перед экспериментом: 1 пмоль ДНК инкубировали с 0.1 U UDG 30 мин при 37°C.
Введение радиоактивной метки на 3′-конец олигонуклеотида проводили с использованием [α-32P]dАTP и Polβ, как описано ранее [7]. Реакционную смесь (200 мкл), содержащую 50 мM Трис-HCl (pH 8.0), 50 мM NaCl, 10 мM MgCl2, 0.5 мкM ДНК, 5 МБк [α-32P]dАTP, 1 мкM dАTP и 0.2 мкM Polβ, инкубировали 90 мин при 37°C. Polβ инактивировали нагреванием при 90°C в течение 5 мин. Радиоактивно меченный олигонуклеотид выделяли, как описано ранее [6].
Ковалентное присоединение белков APE1 и Polβ к АР-/dRP-ДНК. Реакционную смесь (10 мкл), содержащую 50 мM Трис-HCl (pH 8.0), 50 мM NaCl, 10 мM EDTA, 0.1 мкM [32P]-dRP-ДНК и APE1 (0.3, 0.6, 1.2 мкМ) и/или Polβ (0.3 мкМ или 35 нМ), смешивали во льду и инкубировали при 37°C. Затем для стабилизации основания Шиффа в аддуктах [32P]-dRP-ДНК и белков в реакционную смесь добавляли NaBH4 до концентрации 20 мМ и инкубировали 30 мин во льду. После этого продукты анализировали электрофорезом в 12.5%-ном SDS-ПААГ [8]. Гели высушивали и анализировали с использованием Biomolecular Imager Typhoon FLA 9 500 (“GE Healthcare Life Sciences”, США) и программного обеспечения Quantity One (“Bio-Rad”, США).
Анализ 5'-dRP-лиазной активности АРЕ1. Реакционная смесь (10 мкл) содержала 0.1 мкМ ДНК-дуплекс с радиоактивной меткой на 3ʹ-конце 5′‑dRP-олигонуклеотида, 0.3 мкМ АРЕ1 (где указано) и следующие стандартные компоненты: 50 мM Трис-HCl (pH 8.0), 50 мM NaCl, 10 мM EDTA, 1 мM дитиотреитол, 0.05 мг/мл BSA. Пробы инкубировали при 37°C и отбирали аликвоты через 2, 5, 10, 15 и 20 мин. Реакцию останавливали добавлением метоксиамина до его конечной концентрации 30 мМ с последующей инкубацией при 0°C в течение 30 мин, после чего анализировали продукты электрофорезом в 20%-ном ПААГ в присутствии 7 М мочевины [9]. Далее гели обрабатывали, как описано в предыдущем разделе.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Взаимодействие АРЕ1 и Polβ с ДНК, содержащими 5′-дезоксирибозофосфатную группу
Для изучения взаимодействия белков с 5′‑dRP-группой использовали различные ДНК-структуры, содержащие упомянутую группу. Все они представлены в табл. 1. Во всех случаях ДНК с 5′-dRP-группами и АР-сайтами получали обработкой UDG соответствующих dUMP-содержащих ДНК непосредственно перед экспериментом. Для выяснения механизма образования продуктов ковалентного присоединения АРЕ1 к ДНК использовали ДНК-дуплекс, имитирующий продукт гидролиза АР-сайта АРЕ1 ‒ ДНК7, в котором однонуклеотидная брешь в цепи фланкирована с 5′-стороны дезоксирибозофосфатным остатком, содержащим введенную [32Р]-радиоактивную метку. Эта структура ДНК имитирует продукт гидролиза АР-сайта в реакции, катализируемой APE1. В этом случае радиоактивная метка содержится в 5'-dRP-остатке, непосредственно участвующем в образовании основания Шиффа с аминогруппой белка, которая вовлечена в удаление 5'-dRP-остатка. Такая локализация радиоактивной метки позволяет регистрировать ранний интермедиат ‒ белок с присоединенным к нему олигонуклеотидом, ‒ а также продукт β-элиминирования, из которого уже удален остаток олигонуклеотида, но дезоксирибозофосфат еще остается связанным с белком. Субстрат, имеющий такую локализацию радиоактивной метки, ранее был нами использован для поиска в клеточных экстрактах дезоксирибозофосфатлиаз, то есть белков, которые способны образовывать основания Шиффа с подобной структурой ДНК и осуществлять удаление остатка 5′-дезоксирибозы [10]. АРЕ1 инкубировали с ДНК-дуплексом, имитирующим продукт реакции гидролиза АР-сайта под действием APE1, в течение различных промежутков времени. В этой ДНК 5′-dRP-группа, обращенная в сторону бреши, содержала [32Р]. Для стабилизации ковалентных аддуктов АРЕ1-ДНК аликвоты обрабатывали боргидридом натрия и анализировали электорофоретическую подвижность продуктов в ПААГ (рис. 1). Образующиеся радиоактивно меченные продукты на основании их электрофоретической подвижности могут быть отнесены к АРЕ1, ковалентно присоединенной к ДНК, содержащей 5'‑dRP-остаток. Таким образом, результаты анализа согласуются с предположением о формировании АРЕ1 ковалентных аддуктов с продуктом катализируемой реакции гидролиза АР-сайта.
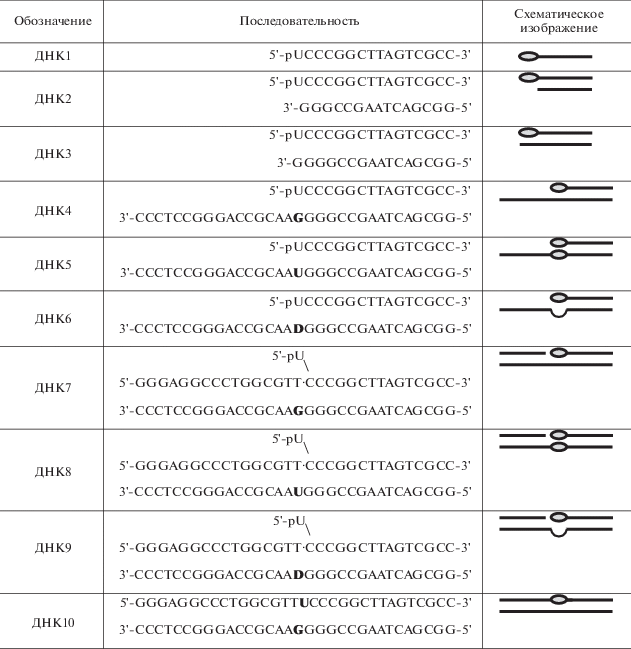
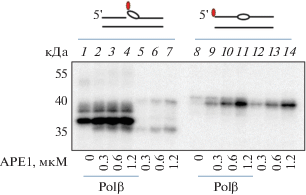
Рис. 1.
Схема формирования и анализа продуктов сшивки 5′-dRP-ДНК с АРЕ1. а ‒ ДНК, содержащие на 5′-конце олигонуклеотида остаток [32Р]-dUМР, обрабатывали UDG. б ‒ 100 нМ 5′-dRP-ДНК (ДНК7) инкубировали с 300 нМ АРЕ1, через указанные промежутки времени аликвоты реакционной смеси обрабатывали боргидридом натрия и анализировали электрофорезом в 12.5%-ном SDS-ПААГ. Дорожки 1‒4 соответствуют продуктам реакции за указанное время, дорожка 5 – контрольный образец, не содержащий АРЕ1.
По-видимому, за счет β-элиминирования происходит отщепление олигонуклеотида, а дезоксирибозофосфатный остаток остается связанным с белком, по крайней мере, в течение 30 мин. На такой сценарий указывает набор образованных меченых продуктов и последующая кинетика их распада.
Для анализа справедливости предположения о способности АРЕ1 проявлять АР/5'-dRP-лиазную активность и исследования специфичности этого процесса в отношении различных структур ДНК на основе 5′-[32Р]-меченого dUMP-содержащего олигонуклеотида (ДНК1) были созданы ДНК, имитирующие субстраты разных путей репарации, включая эксцизионную репарацию оснований (BER) (ДНК7–ДНК10) и негомологичное соединение концов (ДНК2‒ДНК6) (схематическое изображение ДНК см. в табл. 1). Основание Шиффа представляет собой интермедиат реакции β- и β,δ-элиминирования, катализируемого АР/5'-dRP-лиазами. Однако в литературе описаны примеры формирования основания Шиффа, не приводящего к последующему расщеплению остова ДНК [6, 11, 12]. Для слежения за удалением 5'-dRP остатка из ДНК, моделирующих субстраты репарационных процессов, использовали набор ДНК, в которых один из олигонуклеотидов содержал на 5'-конце радиоактивно меченную dRP-группу (см. табл. 1). В этих ДНК радиоактивная метка содержится в 5'-dRP-остатке, непосредственно участвующем в образовании основания Шиффа при катализе лиазной реакции. Целевые белки инкубировали с 5'-dRP-ДНК в течение определенных промежутков времени, затем восстанавливали основание Шиффа с помощью боргидрида натрия и продукты этих превращений подвергали электрофоретическому разделению в денатурирующих условиях. Проведен сравнительный анализ меченых продуктов сшивки ДНК‒белок для АРЕ1, а также полимеразы Polβ ‒ классической 5'-dRP-лиазы процесса BER [13]. Из данных, представленных на рис. 2, видно, что для обоих белков, с учетом их молекулярной массы (39 кДа Polβ и 35.5 кДа АРЕ1), в пределах одного типа ДНК наборы меченых продуктов схожи. В каждом случае наблюдаются минорные продукты со сниженной электрофоретической подвижностью и мажорные ‒ с электрофоретической подвижностью, практически совпадающей с таковой у исходных белков. Первый тип продуктов, вероятно, соответствует белку, содержащему ковалентно присоединенный олигонуклеотид (до расщепления связи), причем сам этот олигонуклеотид может находиться в составе ДНК-дуплекса или диссоциировать из него при плавлении дуплекса во время подготовки проб. Косвенно об этом можно судить по электрофоретической подвижности продуктов в зависимости от молекулярной массы ДНК-дуплекса, хотя присоединяемый к белку олигонуклеотид везде одинаков. Анализ продуктов сшивки белков с одноцепочечным 5'-dRP-содержащим олигонуклеотидом подтверждает это предположение. При подготовке образца для анализа по методу Леммли [8] в пробу не вносятся соединения, обеспечивающие разрушение водородных связей между основаниями ДНК, поэтому не всегда происходит количественное плавление дуплекса.
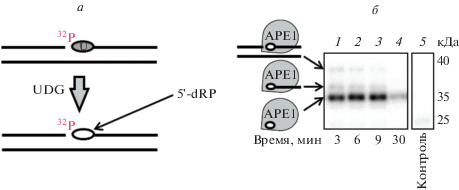
Рис. 2.
Кинетика образования и распада продуктов сшивки dRP-ДНК c Polβ (а) и АРЕ1 (б). Образец ДНК (100 нМ), содержащей 5′-[32Р]-dRP-остаток (ДНК1‒ДНК9, указано над рисунком), инкубировали с 300 нМ Polβ или 300 нМ АРЕ1 и через указанные промежутки времени отбирали аликвоты, обрабатывали боргидридом натрия и анализировали электрофорезом в 12.5%-ном SDS-ПААГ.
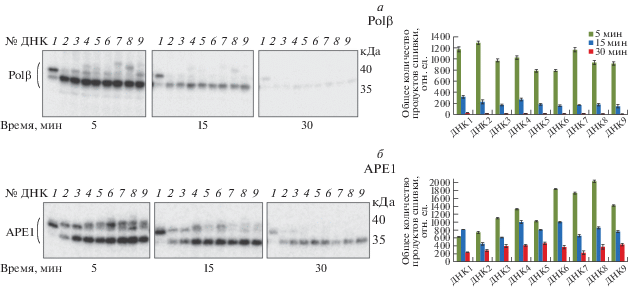
Для обоих белков с течением времени сначала исчезали продукты с более низкой электрофоретической подвижностью, которые относятся к белку с ковалентно присоединенной ДНК. В процессе реакции β-элиминирования олигонуклеотид высвобождается из ковалентного комплекса с белком, а дезоксирибозофосфат, несущий радиоактивную метку, продолжает оставаться связанным с белком. Этому продукту на радиоавтографе соответствуют полосы с максимальной электрофоретической подвижностью, так как присоединенный 5′-dRP-остаток практически не изменяет электрофоретическую подвижность белка (см. схему на рис. 1б). Правильность отнесения этого продукта была подтверждена сопоставлением положения в геле полосы белка (по данным окрашивания Кумасси) (рис. 3, левая панель) и соответствующей полосы на радиоавтографе (рис. 3, правая панель). Сравнительный анализ спектров продуктов пришивки различных 5′-dRP-ДНК к АРЕ1 и Polβ и кинетика их распада свидетельствуют, что и в случае АРЕ1 также происходит β‑элиминирование, хотя и c меньшей эффективностью, чем для Polβ (рис. 2). Следует отметить, что оба белка проявляют лиазную активность на ДНК различной структуры, а не только той, которая имитирует интермедиаты процесса BER, что, несомненно, тоже представляет интерес.
Рис. 3.
Анализ продуктов модификации АРЕ1 на основании их электрофоретической подвижности. 100 нМ 5′-dRP-ДНК (ДНК7) инкубировали с 300 нМ АРЕ1 в течение 3 мин, обрабатывали боргидридом натрия и анализировали электрофорезом в 12.5%-ном SDS-ПААГ. Левая панель – окрашивание геля Кумасси, правая панель – радиоавтограф этого же геля.
Для выяснения взаимовлияния АРЕ1 и Polβ при их взаимодействии с ДНК-интермедиатами BER проведены эксперименты по пришивке этих белков к ДНК7 (структура с расщепленным АР-сайтом) и ДНК10 (структура с интактным АР‑сайтом) при одновременном присутствии обоих белков в нескольких концентрациях. В качестве примера на рис. 4 приведен анализ продуктов, образовавшихся за 1 мин в присутствии 35 нМ Polβ и различных концентраций АРЕ1 (0.3, 0.6 и 1.2 мкМ). В целом, в выбранных условиях эффективность пришивки Polβ и АРЕ1 к ДНК с интактными АР-сайтами сопоставима. При взаимодействии Polβ со своим субстратом, ДНК-дуплексом с 5'-dRP-группой на краю бреши (рис. 4), сшивки образуются очень эффективно, при этом даже 40-кратный избыток АРЕ1 не влияет на выход продуктов, относящихся к Polβ. В то же время, несмотря на эффективное взаимодействие Polβ с ДНК, для АРЕ1 сохраняется возможность связываться с ДНК и осуществлять β-элиминирование, о чем свидетельствует образование продуктов АРЕ1-dRP (рис. 4, продукт с самой высокой электрофоретической подвижностью на дорожках 2‒4). Следует отметить, что в клетках количество АРЕ1 может варьировать в широком диапазоне, но концентрация APE1 всегда выше, чем Polβ [14, 15]; именно поэтому использованные нами в экспериментах соотношения этих белков вполне могут отражать таковые в клетке. Кроме того, по имеющимся в литературе данным, известно об эффективном взаимодействии АРЕ1 с продуктом реакции гидролиза АР-сайта [1, 16, 17], из чего можно сделать вывод о присущей этому ферменту лиазной активности и возможности ее реализации in situ.
Рис. 4.
Разделение продуктов ковалентного присоединения Polβ и АРЕ1 к ДНК, содержащей интактный (ДНК10) или расщепленный (ДНК7) АР-сайты. Положение радиоактивной метки обозначено красным овалом, АР-сайта (интактного или расщепленного) – незаполненным овалом. Образец ДНК (100 нМ), содержащей остаток dUМР, обрабатывали UDG, инкубировали с АРЕ1 (концентрация указана внизу рисунка) и/или 35 нМ Polβ (дорожки 1–4, 8–11) в течение 1 мин, обрабатывали боргидридом натрия и анализировали электрофорезом в 12.5%-ном SDS-ПААГ.
5'-dRP-лиазная активность АРЕ1
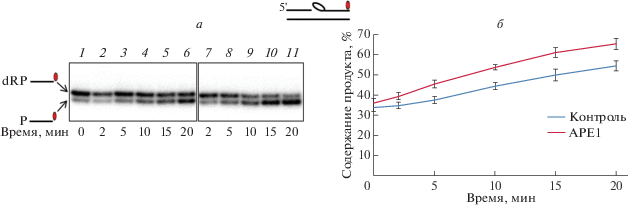
Для прямого доказательства способности АРЕ1 удалять 5'-dRP-остаток ДНК-интермедиат BER (ДНК7) с радиоактивной меткой на 3ʹ-конце dRP-содержащего олигонуклеотида инкубировали в присутствии белка АРЕ1 (300 нМ) или без него в течение указанных промежутков времени, затем реакционные смеси обрабатывали метоксиамином и анализировали электрофоретическую подвижность продуктов в ПААГ в присутствии 7 М мочевины (рис. 5). Метоксиамин способен формировать аддукты с альдегидными группами ДНК, что повышает устойчивость последних в процессе пробоподготовки и последующего электрофореза. В то же время метоксиамин не восстанавливает основание Шиффа, поэтому ДНК не фиксируется на белке и, следовательно, учитывается при анализе. В результате показано, что, действительно, при инкубации 5′-dRP-ДНК в присутствии или в отсутствие АРЕ1 накопление продукта с увеличенной электрофоретической подвижностью происходит быстрее в образцах с АРЕ1. В то же время сопоставимые уровни превращения 5'-dRP-субстрата в продукт под действием фермента Polβ достигаются при его концентрациях в 10‒20 раз меньше таковых для АРЕ1 [18]. Учитывая временные и концентрационные характеристики удаления 5'-dRP-группы ферментами Polβ и АРЕ1, можно сделать вывод, что в нормальных условиях в клетке вклад АРЕ1 в удаление 5'-dRP невелик, но значимость этой активности АРЕ1 может существенно возрастать при дефиците 5'-dRP-лиазной активности, например обусловленной низкой экспрессией Polβ либо ее мутациями. Кроме того, как отмечено выше, реализацию этой активности АРЕ1 нельзя исключить и для других механизмов репарации, включающих процессинг 5'‑dRP-ДНК.
Рис. 5.
Удаление остатка 5'-dRP под действием лиазной активности АРЕ1. Структура использованной ДНК (ДНК7) представлена вверху рисунка. Положение радиоактивной метки обозначено красным овалом, 5'-dRP-остатка – незаполненным овалом. Образец ДНК (100 нМ), содержащей остаток 5'-dUМР, обрабатывали UDG, инкубировали в отсутствие АРЕ1 (контроль, дорожки 1–6) или в присуствии 300 нМ АРЕ1 (АРЕ1, дорожки 7–11) в течение 0, 2, 5, 10, 15 и 20 мин. Через указанные промежутки времени реакцию останавливали добавлением метоксиамина. а ‒ Анализ продуктов реакции электрофорезом в 20%-ном ПААГ в присутствии 7 М мочевины. б ‒ Кинетические кривые, построенные на основании результатов, представленных на панели (а). По оси ординат указано процентное содержание продукта 5'-dRP-лиазной реакции в общем пуле меченых компонентов реакционной смеси.
Интересно отметить, что 5'-dRP-лиазная активность уже ранее обнаружена нами у некоторых белков, участвующих в BER: PARP1, PARP2 и HMGB1 [9, 18, 19]. Следовательно, в случае недостаточной активности Polβ эти белки могут быть вовлечены в регуляцию эффективности BER, участвуя в стадии, лимитирующей скорость процесса репарации оснований в целом [20, 21]. В то же время АРЕ1 может катализировать выщепление 5'-dRP-группы без диссоциации комплекса с продуктом своей основной реакции. Такой механизм представляется тем более вероятным, что эксперименты, проведенные в клетках, показали, что диссоциация АРЕ1 из комплекса с ДНК модулируется Polβ, и при отсутствии полимеразы скорость может снижаться [22]. Таким образом, лиазная активность АРЕ1 может быть необходимой для репарации АР-сайтов в случае дефицита или мутаций Polβ ‒ основного фермента, участвующего в удалении 5'-dRP-группы в ходе репарации поврежденных оснований [23].
Работа по изучению взаимодействия белков с ДНК выполнена при поддержке Российского фонда фундаментальных исследований (проект № 17-00-00097), а этапы наработки и характеристики белков для проведения исследований ‒ при поддержке Российского научного фонда (грант № 19-14-00204).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Дырхеева Н.С., Лебедева Н.А., Лаврик О.И. (2016) АР-эндонуклеаза 1 – ключевой фермент репарации апуриновых/апиримидиновых сайтов. Биохимия. 81, 1198–1216.
Ilina E.S., Khodyreva S.N., Lavrik O.I. (2018) Unusual interaction of human apurinic/apyrimidinic endonuclease 1 (APE1) with abasic sites via the Schiff-base-dependent mechanism. Biochimie. 150, 88–99.
Lebedeva N.A., Khodyreva S.N., Favre A., Lavrik O.I. (2003) AP endonuclease 1 has no biologically significant 3'→5'-exonuclease activity. Biochem. Biophys. Res. Commun. 300, 182–187.
Драчкова И.А., Петрусева И.О., Сафронов И.В., Захаренко А.Л., Шишкин Г.В., Лаврик О.И., Ходырева С.Н. (2001) Реагенты для модификации белково-нуклеиновых комплексов. II. Сайт-специфическая фотомодификация комплексов ДНК-полимеразы β праймерами, элонгированными экзо-N-замещенными арилазидными производными dCTP. Биоорган. химия. 27, 197–204.
Кутузов М.М., Ильина Е.С., Суханова М.В., Пышная И.А., Пышный Д.В., Лаврик О.И., Ходырева С.Н. (2011) Взаимодействие поли(ADP-рибозо)полимеразы-1 с апуриновыми/апиримидиновыми сайтами в составе кластерных повреждений ДНК. Биохимия. 76, 176–187.
Ilina E.S., Lavrik O.I., Khodyreva S.N. (2008) Ku antigen interacts with abasic sites. Biochim. Biophys. Acta. 1784, 1777–1785.
Белоусова Е.А., Кутузов М.М., Иванкина П.А., Ищенко А.А., Лаврик О.И. (2018) Новый путь репарации разрывов ДНК с участием PARP3 и белков эксцизионной репарации оснований. Доклады Академии наук. 482, 96–100.
Laemmli U.K. (1970) Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 277, 680–685.
Мазин А.В., Кузнеделов, Краев А.С. и др. (1990) Методы Молекулярной Генетики и Генной Инженерии, под. ред. Салганика Р.И. Новосибирск: “Наука”. Сибирское отделение, 238–239.
Prasad R., Liu Y., Deterding L.J., Poltoratsky V.P., Kedar P.S., Horton J.K., Kanno S.-I., Asagoshi K., Hou E.W., Khodyreva S.N., Lavrik O.I., Tomer K.B., Yasui A., Wilson S.H. (2007) HMGB1 is a cofactor in mammalian base excision repair. Mol. Cell. 27, 829–841.
Zharkov D.O., Grollman A.P. (1998) MutY DNA glycosylase: base release and intermediate complex formation. Biochemistry. 37, 12384–12394.
Kosova A.A., Khodyreva S.N., Lavrik O.I. (2015) Glyceraldehyde-3-phosphate dehydrogenase (GAPDH) interacts with apurinic/apyrimidinic sites in DNA. Mutat. Res. 779, 46–57.
Idriss H.T., Al-Assar O., Wilson S.H. (2002) DNA polymerase beta. Int. J. Biochem. Cell. Biol. 34, 321–324.
Mol C.D., Hosfield D.J., Tainer J.A. (2000) Abasic site recognition by two apurinic/apyrimidinic endonuclease families in DNA base excision repair: the 3' ends justify the means. Mutat. Res. 460, 211–229.
Horton J.K., Srivastava D.K., Zmudzka B.Z., Wilson S.H. (1995) Strategic down-regulation of DNA polymerase beta by antisense RNA sensitizes mammalian cells to specific DNA damaging agents. Nucleic Acids Res. 23, 3810–3815.
Freudenthal B.D., Beard W.A., Cuneo M.J., Dyrkheeva N.S., Wilson S.H. (2015) Capturing snapshots of APE1 processing DNA damage. Nat. Struct. Mol. Biol. 22, 924–931.
Cistulli C., Lavrik O.I., Prasad R., Hou E., Wilson S.H. (2004) AP endonuclease and poly(ADP-ribose) polymerase-1 interact with the same base excision repair intermediate. DNA Repair (Amst.). 3, 581–591.
Khodyreva S.N., Prasad R., Ilina E.S., Sukhanova M.V., Kutuzov M.M., Liu Y., Hou E.W., Wilson S.H., Lavrik O.I. (2010) Apurinic/apyrimidinic (AP) site recognition by the 5'-dRP/AP lyase in poly(ADP-ribose) polymerase-1 (PARP-1). Proc. Natl. Acad. Sci. USA. 107, 22090–22095.
Kutuzov M.M., Khodyreva S.N., Ilina E.S., Sukhanova M.V., Amé J.C., Lavrik O.I. (2015) Interaction of PARP-2 with AP site containing DNA. Biochimie. 112, 10–19.
Horton J.K., Prasad R., Hou E., Wilson S.H. (2000) Protection against methylation-induced cytotoxicity by DNA polymerase beta-dependent long patch base excision repair. J. Biol. Chem. 275, 2211–2218.
Srivastava D.K., Berg B.J., Prasad R., Molina J.T., Beard W.A., Tomkinson A.E., Wilson S.H. (1998) Mammalian abasic site base excision repair. Identification of the reaction sequence and rate-determining steps. J. Biol. Chem. 273, 21203–21209.
Janoshazi A.K., Horton J.K., Zhao M.-L., Prasad R., Scappini E.L., Tucker C.J., Wilson S.H. (2020) Shining light on the response to repair intermediates in DNA of living cells. DNA Repair (Amst.). 85, 102749.
Sobol R.W., Prasad R., Evenski A., Baker A., Yang X.P., Horton J.K., Wilson S.H. (2000) The lyase activity of the DNA repair protein beta-polymerase protects from DNA-damage-induced cytotoxicity. Nature. 405, 807–810.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология