Молекулярная биология, 2021, T. 55, № 3, стр. 500-509
Деубиквитинирующий (DUB) модуль SAGA участвует в Pol III-зависимой транскрипции
Ю. В. Николенко a, Ю. А. Вдовина a, Е. И. Фефелова a, А. А. Глухова a, Е. Н. Набирочкина a, Д. В. Копытова a, *
a Институт биологии гена Российской академии наук
119334 Москва, Россия
* E-mail: d_dmitrieva@mail.ru
Поступила в редакцию 23.09.2020
После доработки 16.10.2020
Принята к публикации 21.10.2020
Аннотация
Мультибелковый комплекс SAGA, отвечающий за ацетилирование N-концевых последовательностей гистонов и участвующий в активации транскрипции широкого спектра генов эукариот, содержит также белковый модуль, отвечающий за деубиквитинирование гистона H2B (DUB-модуль). Свойства и механизм работы DUB-модуля изучены недостаточно. В настоящей работе нами показано, что DUB-модуль может существовать в клетке независимо от SAGA. С промоторами генов, транскрибируемых РНК-полимеразой III (Pol III), в отличие от генов, транскрибируемых РНК-полимеразой II (Pol II), DUB-модуль взаимодействует независимо от SAGA. DUB-модуль необходим для привлечения транскрипционного фактора Brf1 (субъединицы комплекса TFIIIB), отвечающего за взаимодействие Pol III с промоторами Pol III-зависимых генов. DUB-модуль взаимодействует с Pol III in vivo. Он участвует в привлечении в промоторную область Pol III-зависимых генов типа III не только комплекса TFIIIB, но также комплекса PBP.
ВВЕДЕНИЕ
Доступность ДНК-матрицы для транскрипционных факторов контролируется уровнем компактизации хроматина, который в свою очередь регулируется АТP-зависимым ремоделированием нуклеосом и внесением или снятием ковалентных модификаций гистонов. Эти изменения хроматина приводят к формированию структуры молекулы ДНК, с которой связываются транскрипционные факторы. За эти процессы отвечают коактиваторные комплексы – эволюционно консервативные мультибелковые комплексы, участвующие в активации транскрипции.
Коактиваторные комплексы чаще всего имеют несколько ферментативных активностей, которые также регулируются другими белками. Фактически, большие коактиваторные комплексы состоят из нескольких модулей, которые играют определенную функциональную роль. SAGA – эволюционно консервативный эукариотический комплекс, способный ацетилировать N-концевые последовательности гистонов, участвует в активации транскрипции широкого спектра генов. В состав комплекса SAGA входят 18–20 субъединиц [1]. Генетические и биохимические исследования показали, что SAGA состоит из нескольких функциональных модулей: структурного основания, гистон-ацетилтрансферазного модуля (HAT), деубиквитинирующего модуля (DUB) и модуля связывания с активатором [2, 3]. SAGA многоклеточных организмов содержит также модуль сплайсинга.
В состав ацетил трансферазного модуля входит фермент ацетил-трансфераза Gcn5. Этот модуль активирует транскрипцию за счет ацетилирования остатков лизина в молекулах гистонов. Ацетилирование активирует экспрессию генов, создавая открытую структуру хроматина, что позволяет связаться с хроматином и ДНК другим транскрипционным факторам [4–6].
DUB-модуль SAGA деубиквитинирует гистон H2B [7–11]. В состав DUB-модуля у Drosophila melanogaster и Saccharomyces cerevisiae входят белки: Nonstop (yUbp8), Sgf11, ENY2 (ySus1) и ATXN7 (ySgf73) (табл. 1) [12, 13].
Таблица 1.
Ортологи субъединиц DUB-модуля комплекса SAGA и TFIIIB у различных организмов
| Комплекс | Ортологи субъединиц DUB-модуля комплекса SAGA и TFIIIB | ||
|---|---|---|---|
| D. melanogaster | H. sapiens | S. cerevisiae | |
| DUB | Nonstop, dNonstop | USP22, hUSP22 | Ubp8, yUbp8 |
| Sgf11, dSgf11 | ATXN7L3, hATXN7L3 | Sgf11, ySgf11 | |
| ENY2, dENY2 | ENY2, hENY2 | Sus1, ySus1 | |
| ATXN7, dATXN7 | ATXN7, hATXN7 | Sgf73, ySgf73 | |
| TFIIIB | TBP, dTBP | TBP, hTBP | TBP, yTBP |
| Bdp1, dBdp1 | BDP1, hBDP1 | BDP1, Brf1, yBrf1 | |
| Brf1, dBrf1 | BRF1, hBRF1 | BRF1, Brf1, yBrf1 | |
Изучение структуры DUB-модуля дрожжей показало, что он соединяется с остальным комплексом SAGA с помощью C-концевого участка белка Sgf73 (dATXN7) [14, 15]. Белок yUbp8 (dNonstop) является каталитической субъединицей, а Sgf11 необходим для взаимодействия yUbp8 с субстратом и поддержания его активной конформации [12, 13, 16].
Ранее мы показали, что белок Sgf11 участвует в формировании и экспорте мРНП-частицы независимо от DUB-модуля [17].
Недавно обнаружили, что SAGA участвует в транскрипции генов малых ядерных РНК (мяРНК) как Pol II-, так и Pol III-зависимых [18]. Pol III транскрибирует набор некодирующих РНК, включая тРНК, 5S РНК и U6 мяРНК [19]. Важную роль в Pol III-зависимой транскрипции играет транскрипционный фактор IIIB (TFIIIB). Этот фактор состоит из трех субъединиц и отвечает за рекрутирование Pol III на промоторы ее генов-мишеней. Каждая из субъединиц комплекса TFIIIB, (TBP (TATA-связывающий белок), BDP1 – большой SANT-доменсодержащий белок и родственные TFIIB факторы: BRF1 или BRF2), необходима для его правильного функционирования [20, 21].
Большинство транскрибируемых Pol III генов, таких как гены тРНК, имеют внутренние промоторы, содержащие два блока промоторных последовательностей (А и В), расположенных внутри области транскрипции. С этими блоками связывается транскрипционный фактор IIIC (TFIIIC) [22], который рекрутирует TFIIIB. Меньшая часть Pol III-транскрибируемых генов (гены мяРНК), имеют промотор, расположенный “выше” сайта старта транскрипции. Эти промоторы содержат TATA-box, который взаимодействует с TBP, PSE-элемент, связывающий фактор PBP, необходимый для привлечения TFIIIB, и DSE – элемент ДНК, обладающий свойствами энхансера [19, 23].
В транскрипции генов человека с промоторным элементом, расположенным внутри области транскрипции, участвует TFIIIB, содержащий белок BRF1, а в транскрипции с внешних промоторов – BRF2. У дрозофилы известен только Brf1, поэтому этот белок участвует в транскрипции и с внешних, и с внутренних промоторов.
С DSE взаимодействуют такие транскрипционные факторы, как Oct1, Sp1, NF1, Staf [23, 24]. Показано, что в генах U1, U2, 7SK и U6 мяРНК между элементами DSE и PSE находится нуклеосома, которая сближает эти две области в пространстве и облегчает взаимодействие между Oct1 и PBP [25–28]. Описано вовлечение в регуляцию Pol III-зависимой транскрипции фактора c-Myc, который рекрутирует кофакторы TRRAP и GCN5 к генам тРНК и 5S рРНК и стимулирует ацетилирование гистона Н3 в этих локусах [29].
Хотя влияние SAGA на транскрипцию генов изучено достаточно хорошо, нерешенным остается ряд вопросов, касающихся DUB-модуля SAGA. В частности, не ясно, существует ли DUB-модуль в клетке независимо от остального комплекса и как он взаимодействует с другими транскрипционными факторами. Мы показываем, что DUB-модуль может существовать в клетке независимо от SAGA, исследуем взаимодействие DUB-модуля с транскрипционным аппаратом Pol III, а также показываем, что механизмы взаимодействия DUB-модуля c транскрипционным аппаратом Pol II- и Pol III-зависимых генов различаются между собой.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Линии D. melanogaster. В работе использованы следующие линии D. melanogaster (табл. 2).
Таблица 2.
Линии D. melanogaster, использованные для генетического анализа и в экспериментах по иммунопреципитации хроматина
| Мутация в гене, кодирующем белок | Генотип | Источник |
|---|---|---|
| ENY2 | y[2]w [1]e(y)2[u1]/FM4 | Получена ранее, [30] |
| Sgf11 | w[1118]; PBac{RB} [10]Sgf11[e01308]/TM6B,Tb [1] | BDSC, Stock # 7941 |
| Nonstop | P{ry[+t7.2] = PZ}not[02069] ry[506]/TM6B, ry[CB] Tb[+] | BDSC, Stock # 11553 |
| Rpc2 | y[1] w[*]; Mi{y[+mDint2] = MIC}RpIII128[MI15114]/SM6a | BDSC, Stock # 59742 |
| Rpc31 | w[1118]; PBac{w[+mC] = PB}CG33051[c02392] | BDSC, Stock # 10908 |
| Rpc82 | w[1118]; PBac{w[+mC] = WH}CG12267[f05999]/TM6B, Tb [1] | BDSC, Stock # 18929 |
| Rpc53 | y[1] w[67c23]; P{w[+mC] y[+mDint2] = = EPgy2}CG5147[EY22749]/TM3, Sb [1] Ser [1] | BDSC, Stock # 22580 |
| Rpc37 | y[1] w[*]; P{w[+mC] = EP}Sin[G5366]/TM3, Sb [1] Ser [1] | BDSC, Stock # 27952 |
| Brf1 | w[1118]; PBac{w[+mC] = PB}Brf[c07161]/TM6B, Tb [1] | BDSC, Stock # 17804 |
| w[*]; TM3, Sb [1] Ser [1]/TM6B, Tb [1] | BDSC, Stock # 2537 |
Примечание. BDSC – эти линии получены из коллекции центра Bloomington Stock Center (http://flystocks.bio.indiana.edu/). Самцы и самки с генотипом TM6B,Tb [1]/+ (в тексте обозначены “TM6B/+”), использованные в качестве контроля в опытах по иммунопреципитации хроматина, получены путем скрещивания линий w[*]; TM3, Sb [1] Ser [1]/TM6B, Tb [1] и y[1]w [1]. Самцы с генотипом y[2]w [1]e(y)2[u1] (в тексте обозначены “e(y)2;+”), использованные в качестве контроля в анализе генетических взаимодействий, получены путем скрещивания линий y[2]w [1]e(y)2[u1]/FM4 и y[1]w [1].
Антитела. В работе использованы поликлональные антитела к белкам: Sgf11, ENY2, Gcn5, Brf1, Pbp45, полученные ранее в нашей лаборатории [17, 18, 30, 31].
Фракционирование Sgf11-содержащего комплекса из экстракта ядер эмбрионов дрозофилы. Экстракт из ядер эмбрионов получен согласно процедуре, описанной ранее [30], из 0–12-часовых эмбрионов. Детальная схема фракционирования показана на рис. 1а. Колонки были уравновешены в HEMG-буфере (25 мМ HEPES-KOH, pH 7.6, 12.5 мМ MgCl2, 0.1 мМ EDТА, 10% глицерина, 1 мМ дитиотреитол), содержащем 150 мМ NaCl. Иммуноаффинную очистку выполняли на колонке с иммобилизованными антителами к Sgf11, сшитыми с протеин-А-сефарозой. Не связавшиеся белки отмывали буфером HEMG 500, содержащим 0.1% NP-40. Детали процедуры очистки и MALDI-TOF MS описаны ранее [32, 33].
Рис. 1.
Очистка DUB-модуля комплекса SAGA из экстракта ядер эмбрионов. а – Схема очистки комплекса. ИП – иммунопреципитация антителами к Sgf11, сшитыми с носителем. б – Белки DUB-модуля комплекса SAGA, выявленные в ходе MALDI-TOF MS. Указаны ID белков, их названия, молекулярная масса, score –достоверность определения, количество выявленных пептидов.

Культура клеток S2 дрозофилы и эксперименты по нокдауну белков в S2-клетках. Клетки S2 культивировали при 25°C в среде Schneider’s insect medium (“Sigma”, США), содержащей 10% фетальной сыворотки крупного рогатого скота (“HyClone”, США). Нокдаун белков проводили с помощью РНК-интерференции согласно протоколу [34]. В эксперименте использовали 5–7 мкг дцРНК на 1 × 106 клеток. дцРНК синтезировали с помощью TranscriptAid T7 HighYield TranscriptionKit (“ThermoScientific”, США). Уровень экспрессии генов-мишеней измеряли с использованием количественной ПЦР и Вестерн-блот-анализа. дцРНК, соответствующую фрагменту GFP, использовали в качестве отрицательного контроля. дцРНК синтезировали с использованием следующих праймеров: ATXN7 – 5′-CGACTCACTATAGGGAGATCCATGCTGGAGCAGAAGGT-3′
и 5′-CGACTCACTATAGGGAGAGCAGTTCATTCAAACGCTTGGC-3′;
GPF – 5′-CGACTCACTATAGGGAGACGTAAACGGCCACAAGTTCAGC-3′
и 5′-CGACTCACTATAGGGAGAGATGCCGTT-CTTCTGCTTGTCG-3′.
Иммунопреципитация хроматина (ChIP). В одной реакции иммунопреципитации использовали хроматин, выделенный из 3 × 106 клеток S2 или из 50 имаго дрозофилы. “Сшивку” проводили, добавляя в реакционную среду формальдегид до концентрации 0.5% и инкубируя в течение 10 мин при комнатной температуре. Реакцию останавливали добавлением 2.5 М глицина до концентрации 0.125 М (1/20 от общего объема). После отмывки тремя объемами холодного раствора 1× PBS, хроматин ресуспендировали в SDS-содержащем буфере (50 мМ HEPES KOH, pH 7.9, 140 мМ NaCl, 1 мМ EDТА, 1% тритона X-100, 0.1% дезоксихолата Na, 0.1% SDS, 1× PIC (protease inhibitors cocktail (“Roche”, Швейцария)) и дробили до фрагментов размером ~500 нуклеотидов с помощью ультразвука. Обработанный ультразвуком хроматин центрифугировали дважды при 16000 g в течение 20 мин, а затем использовали в экспериментах по иммунопреципитации. В один эксперимент брали около 10 мкг антител и 15 мкл протеин-А-сефарозы (“Sigma”). В реакцию добавляли также ДНК спермы лосося и бычий сывороточный альбумин до конечной концентрации 1 мг/мл. Осажденный хроматин последовательно отмывали SDS-содержащим буфером, SDS-содержащим буфером с 0.5 М NaCl, LiCl-содержащим буфером (20 мМ Трис-HCl, pH 8.0, 1 мМ EDТА, 250 мМ LiCl, 0.5% NP-40, 0.5% дезоксихолат Na) и TE (20 мМ Трис-HCl, pH 8.0, 1 мМ EDТА). Комплексы хроматина инкубировали в буфере для элюции (50 мМ Трис-HCl, pH 8.0, 1 мМ EDТА, 1% SDS) в течение 30 мин при комнатной температуре. В раствор добавляли 5 М NaCl (16 мкл на 500 мкл раствора) и инкубировали при 65°С в течение 16 ч в термошейкере для “расшивки” хроматина. Затем хроматин обрабатывали протеазой (3 мкл протеиназы К, 5 мкл 0.5 М EDТА на 500 мкл раствора) в течение 4 ч при 55°С в термошейкере. ДНК экстрагировали смесью фенол–хлороформ и осаждали изопропанолом. Преципитат растворяли в буфере TE. Выделенную ДНК анализировали с помощью количественной ПЦР. Результаты иммунопреципитации оценивали по соотношению (%) осажденной/исходной фракции. Каждое измерение выполняли как минимум в трех повторах и вычисляли среднее значение.
Статистический анализ. Сравнение экспериментальных групп проводили с помощью t-критерия Стьюдента. Все данные представлены в виде среднего значения ± стандартное отклонение (шкала ошибок), по крайней мере, по трем независимым экспериментам. Звездочки указывают на статистическую значимость: данных *P < 0.05 или **P < 0.01.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Очистка деубиквитинирующего (DUB) модуля SAGA из экстракта ядер эмбрионов
Ранее из белкового экстракта ядер эмбрионов дрозофилы мы выделили и очистили Sgf11-содержащий комплекс массой 100 кДа, включающий CAP-связывающий белок (Cbp80), необходимый для формирования и экспорта мРНП-частицы [17].
В данной работе поставлена задача выделить DUB-модуль комплекса SAGA. Экстракт из ядер эмбрионов дрозофилы пошагово фракционировали с помощью ионообменной хроматографии и гель-фильтрации (рис. 1а). Содержание Sgf11 во фракциях после фракционирования детектировали с помощью Вестерн-блот-анализа. Фракции с максимальным содержанием Sgf11 собирали и использовали в дальнейшей работе.
При последнем фракционировании на гель-фильтрационной колонке Superose 6 HR Sgf11 мигрировал несколькими пиками. В дальнейшей работе использовали Sgf11-содержащие фракции, в которых по результатам Вестерн-блот-анализа выявлены и другие компоненты комплекса SAGA (масса ~1.3 МДа). Эти фракции содержали и другие компоненты DUB-модуля.
Фракции инкубировали с антителами к Sgf11, сшитыми с протеин-А-сефарозой, Связавшийся материал анализировали с помощью MALDI-TOF MS. В результате выявили белки DUB-модуля: Sgf11, ENY2, Nonstop (рис. 1б). С DUB-модулем соосаждались также (в гораздо меньшем количестве) более 100 белков, участвующих в транскрипции и формировании мРНП-частицы. Однако компонентов SAGA-комплекса среди них, несмотря на мягкие условия очистки, не обнаружено. Интересно, что в более жестких условиях (при включении дополнительных стадий очистки) выделены только три белка: Sgf11, Nonstop и убиквитин. Таким образом, наши данные показывают, что DUB-модуль может существовать в ядре как отдельный структурный компонент, не связанный с SAGA.
Анализ влияния белков комплекса SAGA на привлечение DUB-модуля в промоторную область SAGA-зависимых генов
Поскольку полученные данные показывают, что DUB-модуль может существовать в клетке независимо от SAGA, мы исследовали необходимость комплекса SAGA для привлечения DUB-модуля на промоторы генов. Присутствие DUB-модуля на промоторе изучено методом иммунопреципитации хроматина. Анализировали влияние нокдауна субъединицы ATXN7 комплекса SAGA, через которую, как показано ранее, DUB-модуль взаимодействует с SAGA. Нокдаун ATXN7 должен был показать, влияет ли SAGA на привлечение DUB-модуля на промотор.
Хроматин выделяли из клеток S2, в которых при помощи РНК-интерференции был проведен нокдаун ATXN7. В качестве контроля использовали клетки S2, инкубированные с дцРНК, соответствующей фрагменту GFP. В реакциях иммунопреципитации хроматина использовали антитела к Sgf11, ENY2 и Gcn5, имеющиеся в нашей лаборатории. Поскольку ранее было показано, что SAGA и DUB-модуль присутствуют на промоторах как Pol II-, так и Pol III-зависимых генов, мы оценили присутствие факторов на промоторе гена hsp70 (Pol II-зависимый ген) при тепловом шоке и на мишенях Pol III, присутствие SAGA на которых показано ранее [18, 35].
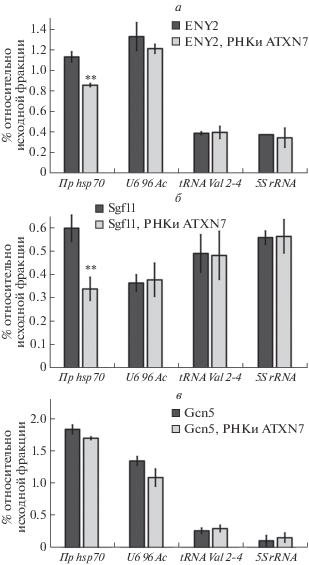
Нокдаун ATXN7 привел к уменьшению связывания субъединиц DUB-модуля Sgf11 и ENY2 с промотором hsp70, но не повлиял на их привлечение на гены, транскрибируемые Pol III (рис. 2). В качестве контроля изучено влияние нокдауна ATXN7 на уровень основной ацетилтрансферазной каталитической субъединицы SAGA, Gcn5. Как и ожидалось, нокдаун ATXN7 не повлиял существенно на связывание с промоторами Gcn5, так как ATXN7 не участвует во взаимодействии SAGA с промотором. Эти данные показывают, что механизм привлечения DUB-модуля на некоторые промоторы может быть SAGA-независимым и отличается на промоторах Pol II- и Pol III-зависимых генов.
Рис. 2.
Анализ влияния нокдауна ATXN7 на привлечение DUB-модуля на промоторы SAGA-зависимых генов. а, б, в – Анализ влияния нокдауна ATXN7 на привлечение ENY2, Sgf11 и Gcn5 в промоторную область генов. Влияние нокдауна ATXN7 на привлечение обозначенных белков на промоторы: hsp70 при тепловом шоке, U6 96 Ac, tRNA Val 2-4, 5S rRNA изучено в реакциях иммунопреципитации хроматина (ChIP). Результаты представлены в процентах от исходной фракции.
Взаимодействие DUB-модуля с аппаратом транскрипции Pol III in vivo
Далее изучали роль DUB-модуля в регуляции транскрипции Pol III-зависимых генов. На первом этапе взаимодействие DUB-модуля и компонентов аппарата транскрипции Pol III изучали генетическими методами на уровне целого организма. Из компонентов DUB-модуля для генетического анализа наиболее удобен ENY2, поскольку он кодируется геном e(y)2, который расположен на Х-хромосоме, что позволяет исследовать его в гемизиготном состоянии у самцов. В нашем распоряжении была линия D. melanogaster с мутацией e(y)2[u1] – слабой гипоморфной мутацией, очень удобной для анализа, так как она обладает плейотропным эффектом, а ее фенотип подробно охарактеризован ранее [30, 36]. Одно из проявлений мутации e(y)2[u1] – фенотип “широко расставленные крылья”, наблюдается у ~5% гемизиготных самцов.
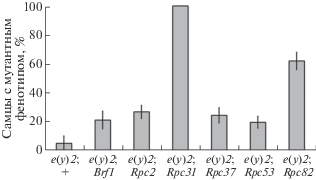
Линии дрозофилы с мутациями, затрагивающими компоненты аппарата транскрипции Pol III, получены из банка мутаций в Блумингтоне. Все эти мутации локализованы на аутосомах, летальны в гомозиготном состоянии, а их фенотипические проявления в гетерозиготном состоянии не описаны. Поэтому мы анализировали влияние этих мутаций на фенотипические проявления мутации e(y)2[u1]. С этой целью в результате скрещивания получили самцов, гемизиготных по мутации e(y)2[u1] и гетерозиготных по мутациям в генах компонентов аппарата транскрипции Pol III. Частота проявления (пенетрантность) мутантного фенотипа e(y)2[u1] у таких самцов значительно возрастала по сравнению с исходной линией e(y)2[u1] и с контрольным скрещиванием (рис. 3). Таким образом мы показали, что DUB-модуль и аппарат транскрипции Pol III взаимодействуют in vivo.
Рис. 3.
Взаимодействие DUB-модуля и аппарата транскрипции Pol III in vivo. Частота проявления фенотипа “широко расставленные крылья” у самцов с мутацией в гене, кодирующем ENY2 (обозначена как e(y)2;+), значительно возрастает на фоне мутаций в генах Brf1, Rpc2, Rpc31, Rpc37, Rpc53, Rpc82. Проанализировано не менее 200 самцов каждого генотипа.
Участие DUB-модуля в привлечении аппарата транскрипции Pol III на промоторы Pol III-зависимых генов мяРНК
Влияние DUB-модуля на экспрессию генов, транскрибируемых Pol III, изучали на линиях дрозофилы с мутациями по субъединицам DUB-модуля Sgf11 и Nonstop, полученным из банка мутаций. Поскольку эти мутации летальны в гомозиготном состоянии, мы использовали гетерозиготных особей Sgf11[e01308]/TM6B и not[02069]/TM6B, у которых уровень экспрессии генов Sgf11 и Nonstop был значительно ниже, чем у мух дикого типа [18]. В качестве контроля использовали особей TM6B/+.
Влияние DUB-модуля на Pol III-зависимые гены изучали с помощью реакции иммунопреципитации хроматина, полученного из дрозофил с мутацией Sgf11[e01308], антителами к Brf1.
Присутствие компонентов транскрипционного аппарата Pol III определено на трех типах Pol III-зависимых промоторов, описанных выше и представленных на рис. 4а. Оказалось, что наибольшее влияние мутации субъединиц модуля DUB оказывают на привлечение Brf1 в промоторную область TATA-box-содержащих Pol III-зависимых генов (рис. 4а, б). Мы предположили, что это происходит за счет влияния DUB-модуля на привлечение комплекса PBP, который, в свою очередь, необходим для привлечения TFIIIB. И на фоне нокдауна субъединиц DUB-модуля PBP-комплекс хуже взаимодействует с последовательностью PSE.
Рис. 4.
Анализ участия DUB-модуля в привлечении аппарата транскрипции Pol III на промоторы Pol III-зависимых генов мяРНК. а – Три типа промоторов Pol III-зависимых генов. На схеме указаны ДНК-элементы и белковые факторы. Стрелка показывает начало транскрипции и направление. ТТТТ – последовательность терминации транскрипции Pol III. Белки Brf1, Bdp1, TBP образуют комплекс TFIIIB. Позиции Brf1 и Bdp1 показаны согласно результатам ChIP-seq в клетках человека. Белок-белковые взаимодействия между TFIIIC, TFIIIA и TFIIIB также показаны на схеме [37]. б – Влияние снижения количества Sgf11 в мутантной линии Sgf11[e01308]/TM6B на привлечение Brf1 в промоторную область Pol III-зависимых генов. в – Влияние снижения количества Nonstop в мутантной линии not[02069]/TM6B на привлечение Pbp45 в промоторную область Pol III-зависимых генов мяРНК. Результаты получены методом иммунопреципитации хроматина (ChIP) и представлены в процентах относительно исходной фракции. В качестве контроля использовали мух с генотипом TM6B/+.
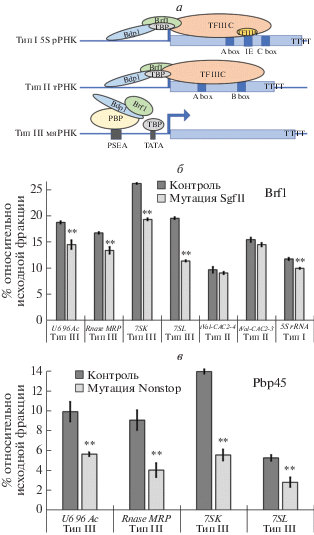
Для подтверждения этой гипотезы изучено влияние мутации Nonstop (деубиквитиназы DUB-модуля) на привлечение в промоторную область мяРНК белка Pbp45, субъединицы комплекса PBP. Из дрозофил с мутацией not[0206] был экстрагирован хроматин и проведены реакции иммунопреципитации хроматина с использованием антител к белку Pbp45 (рис. 4в). Оказалось, что мутация Nonstop вызывает более чем двукратное уменьшение присутствия Pbp45 на промоторе.
Таким образом, нокдаун субъединиц DUB-модуля приводит к уменьшению связывания PBP-комплекса с последовательностью PSE и менее эффективному привлечению фактора TFIIIB на промоторы TATA-box-содержащих Pol III-зависимых генов.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В настоящей работе мы очистили деубиквитинирующий (DUB) модуль SAGA из экстракта ядер эмбрионов дрозофилы и проанализировали его возможное участие в транскрипции независимо от остальных компонентов комплекса SAGA. Модульная структура комплекса SAGA установлена давно, однако способность отдельных модулей SAGA действовать независимо от других не исследована.
Ранее было показано, что белок ATXN7 в составе DUB-модуля необходим для его взаимодействия с остальным комплексом SAGA [14, 15]. Нокдаун ATXN7 должен был привести к отделению остального SAGA от DUB-модуля и нарушить таким образом привлечение DUB-модуля на SAGA-зависимые гены. Однако при нокдауне ATXN7 привлечение Sgf11 и ENY2, компонентов DUB-модуля, уменьшалось только на промоторе Pol II-зависимого гена hsp70 при тепловом шоке. Количество Sgf11 и ENY2 на Pol III-зависимых промоторах не менялось. Таким образом, мы показали, что структура комплекса SAGA на промоторах Pol II- и Pol III-зависимых генов может отличаться или привлечение модулей происходит по-разному.
Для привлечения Pol III на гены-мишени необходим фактор TFIIIB. Как показано ранее, мутации и компонентов DUB-модуля, и HAT-модуля приводят к уменьшению привлечения Brf1, одного из основных белков TFIIIB, на промоторы генов мяРНК [18]. Поскольку в нашей работе показано, что DUB-модуль может существовать независимо, и его привлечение на Pol III-зависимые промоторы происходит независимо от других модулей SAGA, далее мы изучили участие DUB-модуля в транскрипции Pol III-зависимых генов. Это исследование выполнено in vivo на уровне целого организма. Анализ взаимного влияния мутаций показал, что ENY2, субъединица DUB-модуля, взаимодействует не только с Brf1, но также с субъединицами Pol III.
Фактор TFIIIB, необходимый для транскрипции Pol III, состоит из трех белков (BDP1, TBP и BRF1 или BRF2). Состав этого фактора у человека и дрозофилы несколько отличается. У человека в состав TFIIIB, участвующего в транскрипции генов с внутренними промоторами (типы I и II), входит BRF2, а BRF1 присутствует на промоторах типа III [20, 21]. У дрозофилы один и тот же белок Brf1 входит в TFIIIB на промоторах всех трех типов. Мы оценили влияние нокдауна Sgf11 на привлечение Brf1 на все типы промоторов Pol III-зависимых генов дрозофилы. Влияние Sgf11 на привлечение Brf1 оказалось значительным только на промоторах типа III, включающих TATA-box и последовательность PSE. Мы предположили, что DUB-модуль необходим для привлечения на последовательность PSE PBP-комплекса, который отвечает за привлечение TFIIIB на промоторы генов мяРНК. Для проверки этой гипотезы мы исследовали как привлекается Pbp45, компонент PBP-комплекса, на PSE при нокдауне деубиквитиназы DUB-модуля Nonstop. При нокдауне Nonstop количество Pbp45 на промоторах типа III уменьшалось более чем в 2 раза. Мы предполагаем, что столь значительное влияние на привлечение PBP связано с убиквитинированием нуклеосомы, находящейся между PSE и DSE, выполняющим энхансерную функцию. Эта нуклеосома физически сближает PSE и DSE и дает возможность стабилизировать привлечение PBP-комплекса, необходимого для привлечения TFIIIB [25–28]. Ранее мы показали, что DUB-модуль участвует в активации транскрипции Pol III-зависимых генов типа III [18]. В совокупности полученные данные указывают на то, что основным комплексом, осуществляющим деубиквитинирование в этом процессе, может быть DUB-модуль комплекса SAGA.
Работа выполнена с использованием оборудования ЦКП ИБГ РАН. Авторы благодарны Зиганшину Р.Х. за проведение MALDI-TOF MS-анализа, выполненного на оборудовании Института биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова РАН.
Работа поддержана грантами Российского фонда фундаментальных исследований (№ 18-04-00514 А, Д.К., Е.Н., А.Г) и Российского научного фонда (№ 20-14-00269, Ю.Н., Ю.В.).
Все применимые международные, национальные и институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Spedale G., Timmers H., Pijnappel W. (2012) ATAC-king the complexity of SAGA during evolution. Genes Dev. 26, 527–541.
Lee K.K., Sardiu M.E., Swanson S.K., Gilmore J.M., Torok M., Grant P.A., Florens L., Workman J.L., Washburn M.P. (2011) Combinatorial depletion analysis to assemble the network architecture of the SAGA and ADA chromatin remodeling complexes. Mol. Systems Biol. 7, 503.
Helmlinger D., Marguerat S., Villén J., Swaney D.L., Gygi S.P., Bähler J., Winston F. (2011) Tra1 has specific regulatory roles, rather than global functions, within the SAGA co-activator complex. EMBO J. 30, 2843–2852.
Nagy Z., Tora L. (2007) Distinct GCN5/PCAF-containing complexes function as co-activators and are involved in transcription factor and global histone acetylation. Oncogene. 26, 5341–5357.
Hong L., Schroth G.P., Matthews H.R., Yau P., Bradbury E.M. (1993) Studies of the DNA binding properties of histone H4 amino terminus. Thermal denaturation studies reveal that acetylation markedly reduces the binding constant of the H4 ‘tail’ to DNA. J. Biol. Chem. 268, 305–314.
Kouzarides T. (2007) Chromatin modifications and their function. Cell. 128, 693–705.
Henry K.W., Wyce A., Lo W.-S., Duggan L.J., Emre N.C.T., Kao C.-F., Pillus L., Shilatifard A., Osley M.A., Berger S.L. (2003) Transcriptional activation via sequential histone H2B ubiquitylation and deubiquitylation, mediated by SAGA-associated Ubp8. Genes Dev. 17, 2648–2663.
Köhler A., Pascual-García P., Llopis A., Zapater M., Posas F., Hurt E., Rodríguez-Navarro S. (2006) The mRNA export factor Sus1 is involved in Spt/Ada/Gcn5 acetyltransferase-mediated H2B deubiquitinylation through its interaction with Ubp8 and Sgf11. Mol. Biol. Cell. 17, 4228–4236.
Zhang X.-Y., Varthi M., Sykes S.M., Phillips C., Warzecha C., Zhu W., Wyce A., Thorne A.W., Berger S.L., McMahon S.B. (2008) The putative cancer stem cell marker USP22 is a subunit of the human SAGA complex required for activated transcription and cell-cycle progression. Mol. Cell. 29, 102–111.
Lang G., Bonnet J., Umlauf D., Karmodiya K., Koffler J., Stierle M., Devys D., Tora L. (2011) The tightly controlled deubiquitination activity of the human SAGA complex differentially modifies distinct gene regulatory elements. Mol. Cell. Biol. 31, 3734–3744.
Zhao Y., Lang G., Ito S., Bonnet J., Metzger E., Sawatsubashi S., Suzuki E., Le Guezennec X., Stunnenberg H.G., Krasnov A., Georgieva S.G., Schule R., Takeyama K.-I., Kato S., Tora L., Devys D. (2008) A TFTC/STAGA module mediates histone H2A and H2B deubiquitination, coactivates nuclear receptors, and counteracts heterochromatin silencing. Mol. Cell. 29, 92–101.
Kohler A., Zimmerman E., Schneider M., Hurt E., Zheng N. (2010) Structural basis for assembly and activation of the heterotetrameric SAGA histone H2B deubiquitinase module. Cell. 141, 606–617.
Samara N.L., Datta A.B., Berndsen C.E., Zhang X., Yao T., Cohen R.E., Wolberger C. (2010) Structural insights into the assembly and function of the SAGA deubiquitinating module. Science (N.Y.). 328, 1025–1029.
Han Y., Luo J., Ranish J., Hahn S. (2014) Architecture of the Saccharomyces cerevisiae SAGA transcription coactivator complex. EMBO J. 33, 2534–2546.
Lee K.K., Swanson S.K., Florens L., Washburn M.P., Workman J.L. (2009) Yeast Sgf73/ataxin-7 serves to anchor the deubiquitination module into both SAGA and Slik(SALSA) HAT complexes. Epigenet. Chromatin. 2, 2.
Morgan M.T., Haj-Yahya M., Ringel A.E., Bandi P., Brik A., Wolberger C. (2016) Structural basis for histone H2B deubiquitination by the SAGA DUB module. Science (N.Y.). 351, 725–728.
Gurskiy D., Orlova A., Vorobyeva N., Nabirochkina E., Krasnov A., Shidlovskii Y., Georgieva S., Kopytova D. (2012) The DUBm subunit Sgf11 is required for mRNA export and interacts with Cbp80 in Drosophila. Nucl. Acids Res. 40, 86–96.
Popova V.V., Orlova A.V., Kurshakova M.M., Nikolenko J.V., Nabirochkina E.N., Georgieva S.G., Kopytova D.V. (2018) The role of SAGA coactivator complex in snRNA transcription. Cell Cycle. 17, 1859–1870.
Hung K.-H., Stumph W.E. (2011) Regulation of snRNA gene expression by the Drosophila melanogaster small nuclear RNA activating protein complex (DmSNAPc). Crit. Rev. Biochem. Mol. Biol. 46, 11–26.
Schramm L., Hernandez N. (2002) Recruitment of RNA polymerase III to its target promoters. Genes Dev. 16, 2593–2620.
Schramm L., Pendergrast P.S., Sun Y., Hernandez N. (2000) Different human TFIIIB activities direct RNA polymerase III transcription from TATA-containing and TATA-less promoters. Genes Dev. 14, 2650–2663.
Leśniewska E., Boguta M. (2017) Novel layers of RNA polymerase III control affecting tRNA gene transcription in eukaryotes. Open Biol. 7, 2.
Jawdekar G.W., Henry R.W. (2008) Transcriptional regulation of human small nuclear RNA genes. Biochim. Biophys. Acta. 1779, 295–305.
Janson L., Weller P., Pettersson U. (1989) Nuclear factor I can functionally replace transcription factor Sp1 in a U2 small nuclear RNA gene enhancer. J. Mol. Biol. 205, 387–396.
Boyd D.C., Greger I.H., Murphy S. (2000) In vivo footprinting studies suggest a role for chromatin in transcription of the human 7SK gene. Gene. 247, 33–44.
Boyd D.C., Pombo A., Murphy S. (2003) Interaction of proteins with promoter elements of the human U2 snRNA genes in vivo. Gene. 315, 103–112.
Stünkel W., Kober I., Seifart K.H. (1997) A nucleosome positioned in the distal promoter region activates transcription of the human U6 gene. Mol. Cell. Biol. 17, 4397–4405.
Zhao X., Pendergrast P.S., Hernandez N. (2001) A positioned nucleosome on the human U6 promoter allows recruitment of SNAPc by the Oct-1 POU domain. Mol. Cell. 7, 539–549.
Kenneth N.S., Ramsbottom B.A., Gomez-Roman N., Marshall L., Cole P.A., White R.J. (2007) TRRAP and GCN5 are used by c-Myc to activate RNA polymerase III transcription. Proc. Natl. Acad. Sci. USA. 104, 14917–14922.
Georgieva S., Nabirochkina E., Dilworth F.J., Eickhoff H., Becker P., Tora L., Georgiev P., Soldatov A. (2001) The novel transcription factor e(y)2 interacts with TAF(II)40 and potentiates transcription activation on chromatin templates. Mol. Cell. Biol. 21, 5223–5231.
Kurshakova M.M., Krasnov A.N., Kopytova D.V., Shidlovskii Y.V., Nikolenko J.V., Nabirochkina E.N., Spehner D., Schultz P., Tora L., Georgieva S.G. (2007) SAGA and a novel Drosophila export complex anchor efficient transcription and mRNA export to NPC. EMBO J. 26, 4956–4965.
Boehm A.K., Saunders A., Werner J., Lis J.T. (2003) Transcription factor and polymerase recruitment, modification, and movement on dhsp70 in vivo in the minutes following heat shock. Mol. Cell Biol. 23, 7628–7637.
Vorobyeva N.E., Soshnikova N.V., Nikolenko J.V., Kuzmina J.L., Nabirochkina E.N., Georgieva S.G., Shidlovskii Y.V. (2009) Transcription coactivator SAYP combines chromatin remodeler Brahma and transcription initiation factor TFIID into a single supercomplex. Proc. Natl. Acad. Sci. USA. 106, 11049–11054.
Clemens J.C., Worby C.A., Simonson-Leff. N., Muda M., Maehama T., Hemmings B.A., Dixon J.E. (2000) Use of double-stranded RNA interference in Drosophila cell lines to dissect signal transduction pathways. Proc. Natl. Acad. Sci. USA. 97, 6499–6503.
Li X., Seidel C.W., Szerszen L.T., Lange J.J., Workman J.L., Abmayr S.M. (2017) Enzymatic modules of the SAGA chromatin-modifying complex play distinct roles in Drosophila gene expression and development. Genes Dev. 31, 1588–1600.
Kopytova D.V., Orlova A.V., Krasnov A.N., Gurskiy D.Y., Nikolenko J.V., Nabirochkina E.N., Shidlovskii Y.V., Georgieva S.G. (2010) Multifunctional factor ENY2 is associated with the THO complex and promotes its recruitment onto nascent mRNA. Genes Dev. 24, 86–96.
Park J.-L., Lee Y.-S., Kunkeaw N., Kim S.-Y., Kim I.-H., Lee Y.S. (2017) Epigenetic regulation of noncoding RNA transcription by mammalian RNA polymerase III. Epigenomics. 9, 171–187.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


