Молекулярная биология, 2021, T. 55, № 3, стр. 441-459
Роль естественного отбора в формировании генетической компоненты преэклампсии
В. Н. Сереброва a, *, Е. А. Трифонова a, В. А. Степанов a
a Научно-исследовательский институт медицинской генетики Томского национального исследовательского медицинского центра Российской академии наук
634050 Томск, Россия
* E-mail: vika.serebrova@medgenetics.ru
Поступила в редакцию 25.09.2020
После доработки 28.11.2020
Принята к публикации 01.12.2020
Аннотация
Преэклампсия – тяжелая гипертензивная патология, затрагивающая в мировом масштабе от 2 до 8% беременностей. Плохо изученный этиопатогенез, отсутствие предсказательных биомаркеров и эффективных методов лечения данного осложнения беременности определяют высокую частоту материнской и перинатальной заболеваемости и смертности. Существование расовых и этнических различий в частоте развития преэклампсии представляет интерес с точки зрения эволюционной медицины, поскольку такую вариабельность можно рассматривать в качестве побочного эффекта адаптивных изменений генетической структуры современных популяций, происходящих с момента расселения Homo sapiens из Африки. Нами впервые изучено генетическое разнообразие популяций Северной Евразии и мировых популяций проекта “1000 геномов” по 10 ассоциированным с преэклампсией регуляторным полиморфным вариантам, а также оценена роль естественного отбора на микроэволюционном уровне в его формировании. Показан высокий уровень межпопуляционного разнообразия, наибольший вклад в который внесли различия по частотам аллелей rs3802252 гена NDRG1 (FST = 0.157). Выявлено действие естественного отбора для rs10423795 гена LHB, rs2167270 гена LEP, rs2227262 и rs3802252 гена NDRG1, rs56153523 и rs8109071 гена SYDE1, rs72959687 гена INHA. Полученные результаты соответствуют представлениям двух эволюционных гипотез возникновения преэклампсии: гипотезе “предковой предрасположенности” и гипотезе “генетического конфликта интересов”.
ВВЕДЕНИЕ
Преэклампсия (ПЭ) – тяжелая гипертензивная патология, частота возникновения которой варьирует в диапазоне от 2 до 8% беременностей во всем мире [1–3]. Плохо изученный этиопатогенез, отсутствие предсказательных биомаркеров и эффективных методов лечения ПЭ определяют высокую частоту материнской и перинатальной заболеваемости и смертности [1–6]. Несмотря на большое количество исследований, посвященных генетической архитектуре данной патологии, и использование различных подходов, главные гены подверженности к ПЭ не выявлены [7–9].
Изучение ПЭ в аспекте эволюционной медицины является одним из перспективных направлений поиска новых генетических маркеров, которые согласно эволюционным гипотезам могут быть вовлечены в этиопатогенез этого осложнения беременности. В этом контексте особый интерес представляют расовые и этнические различия в частотах развития ПЭ в современных популяциях человека [2, 6, 10, 11]. Так, минимальная частота данной патологии наблюдается среди монголоидов (1.2–1.9%), тогда как ее максимальные значения отмечаются у европеоидов и негроидов (2–5%) [10–12]. Примечательно, что такие различия можно рассматривать в качестве побочного результата адаптивной эволюции, действующей с момента расселения Homo sapiens из Африки на протяжении формирования генофондов данных популяций. Результаты ряда исследований свидетельствуют о том, что вариабельность частоты развития ПЭ в современных популяциях человека может зависеть от ряда факторов: количества потребляемой соли, характерного для традиционной диеты [11, 13, 14], адаптации к гипоксическому воздействию при проживании в высокогорных регионах [14, 15], различий в размерах малого таза у представительниц разных этносов [11, 16]. Таким образом, анализ роли естественного отбора на микроэволюционном уровне в формировании наследственной подверженности к ПЭ наряду с изучением средовых особенностей и традиционного образа жизни, вероятно, будет способствовать лучшему пониманию этиопатогенеза, а также причин происхождения и распространения данной патологии беременности в современных популяциях человека.
Представленная работа посвящена изучению вклада адаптивной эволюции в формирование наследственной подверженности к развитию ПЭ по системе регуляторных полиморфных вариантов (rSNP) дифференциально экспрессирующихся генов (ДЭГ), выявленных благодаря анализу транскриптома плацентарной ткани. В рамках данного исследования впервые охарактеризовано генетическое разнообразие популяций Северной Евразии и мировых популяций из проекта “1000 геномов” по ассоциированным с ПЭ 10 rSNP восьми ДЭГ (rs10985257 гена CORO2A, rs72959687 гена INHA, rs2167270 гена LEP, rs10423795 гена LHB, rs2227262, rs3802252 гена NDRG1, rs1671215 гена RDH13, rs34845949 гена SASH1, rs56153523 и rs8109071 гена SYDE1), а также оценена роль естественного отбора на микроэволюционном уровне в его формировании.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Материал для исследования представлен 1880 образцами ДНК из биологической коллекции “Биобанк населения Северной Евразии” НИИ медицинской генетики Томского НИМЦ (табл. 1). Первая группа образцов ДНК охватывает этнические выборки из 10 популяций Северной Евразии: русские, удмурты, цезы, ханты, хакасы, киргизы, якуты, буряты, калмыки, нивхи. Численность этой группы составила 955 неродственных индивидов, не разделенных по полу, возрасту и с отсутствием метисации более чем в трех поколениях. Этническая принадлежность и родословная каждого индивида установлена в соответствии с результатами индивидуального анкетирования. Вторая группа включает 925 образцов ДНК женщин из русской (N = 498) и якутской (N = 427) популяций, которые согласно течению и исходам беременности были разделены на группу больных ПЭ (195 русских и 217 якуток) и контрольную группу с физиологическим течением беременности (303 русские и 210 якуток). Эта выборка включена в исследование на этапе проведения ассоциативного анализа 46 rSNP 21 ДЭГ с развитием ПЭ у русских и якутов [17]. В состав выборки мировых популяций вошли 1986 индивидов, представителей 20 популяций из проекта “1000 геномов” (табл. 1): ишаны, гамбийцы, лухья, менде, йоруба, финны, британцы, иберийцы, тосканцы, индийцы, индийцы телугу, пенджабцы, ланкийские тамилы, бенгальцы, китайцы (разделены в соответствии с регионом проживания на три группы), японцы, вьеты, перуанцы [18]. Суммарный объем используемых выборок составил 3866 индивидов.
Таблица 1.
Характеристика обследуемых популяционных выборок
| Регион | Этнос | Локализация | N | |
|---|---|---|---|---|
| Африка | ESN | Ишаны | Нигерия | 99 |
| GWD | Гамбийцы | Гамбия | 113 | |
| LWK | Лухья | г. Уэбуай, Кения | 99 | |
| MSL | Менде | Сьерра-Леоне | 85 | |
| YRI | Йоруба | г. Ибадан, Нигерия | 108 | |
| Западная Европа | FIN | Финны | Финляндия | 99 |
| GBR | Британцы | Англия, Шотландия | 91 | |
| IBS | Иберийцы | Испания | 107 | |
| TSI | Тосканцы | Италия | 107 | |
| Кавказ | DAG | Цезы | с. Бежта, с. Кидеро, респ. Дагестан, РФ | 96 |
| Восточная Европа | UDB | Удмурты | пос. Балезино, Удмуртская респ., РФ | 91 |
| RUS | Русские | г. Кемерово, Кемеровская обл., РФ | 96 | |
| RUSCON* | Русские | г. Томск, Томская обл., РФ | 303 | |
| RUSPE* | Русские | г. Томск, Томская обл., РФ | 195 | |
| Индостан | GIH | Индийцы | Выходцы из штата Гуджарат, проживающие в г. Хьюстон, США | 103 |
| ITU | Индийцы телугу | Выходцы, проживающие в Великобритании | 102 | |
| PJL | Пенджабцы | г. Лахор, Пакистан | 96 | |
| STU | Ланкийские тамилы | Выходцы, проживающие в Великобритании | 102 | |
| BEB | Бенгальцы | Бангладеш | 86 | |
| Центральная Азия | KAL | Калмыки | г. Элиста, респ. Калмыкия, РФ | 96 |
| Средняя Азия | KGS | Киргизы | г. Ош, Киргизия | 96 |
| Сибирь | BUR | Буряты | пгт Агинское, Забайкальский край, РФ | 96 |
| KHA | Хакасы | Аскизский р-н, респ. Хакасия, РФ | 96 | |
| HAR | Ханты | д. Русскинская, ХМАО, РФ | 96 | |
| YAK | Якуты | с. Бяди, респ. Саха (Якутия), РФ | 96 | |
| YAKCON* | Якуты | г. Якутск, респ. Саха (Якутия), РФ | 210 | |
| YAKPE* | Якуты | г. Якутск, респ. Саха (Якутия), РФ | 217 | |
| Восточная Азия | CDX | Китайцы | Сишуанбаньна-Дайский АО, Китай | 93 |
| CHB | Китайцы | г. Пекин, Китай | 103 | |
| CHS | Китайцы | Южный Китай | 105 | |
| JPT | Японцы | г. Токио, Япония | 104 | |
| KHV | Вьеты | г. Хошимин, Вьетнам | 99 | |
| Дальний Восток | NIN | Нивхи | Охинский р-н, Сахалинская обл., РФ | 96 |
| Америка | PEL | Перуанцы | г. Лима, Перу | 85 |
Примечание. N – численность исследованной выборки (чел.), АО – автономный округ, ХМАО – Ханты-Мансийский автономный округ. Полужирным выделены популяции Северной Евразии. *Выборка, включенная в исследование на этапе проведения анализа методом случай–контроль [17]. CON – контрольная группа, PE – группа больных ПЭ.
Характеристику генетического разнообразия 10 популяций Северной Евразии по 10 rSNP восьми ДЭГ (rs10985257 гена CORO2A, rs72959687 гена INHA, rs2167270 гена LEP, rs10423795 гена LHB, rs56153523 и rs8109071 гена SYDE1, rs1671215 гена RDH13, rs34845949 гена SASH1, rs2227262 и rs3802252 гена NDRG1), ассоциированным с развитием ПЭ в нашем предыдущем исследовании [17, 19–22], проводили методом ПЦР в режиме реального времени с использованием TaqMan-зондов от “Applied Biosystems” (CША) и “ДНК-синтез” (Россия). Нуклеотидные последовательности праймеров доступны по запросу.
Статистическую обработку результатов проводили с помощью пакетов программ Statistica 7.0 и Arlequin 3.1. Соответствие распределения частот аллелей и генотипов равновесию Харди‒Вайнберга (РХВ) оценивали с помощью критерия χ2. Уровень генетического разнообразия и межпопуляционной дифференциации вычисляли методом анализа молекулярной вариабельности (AMOVA). Анализ генетических взаимоотношений между 30 популяциями проводили методом главных компонент. Селективную нейтральность изученных генетических маркеров оценивали с помощью теста Юинса–Ваттерсона [23]. Роль давления отбора в формировании генетического разнообразия в группах негроидов, европеоидов и монголоидов по ассоциированным с ПЭ 10 rSNP оценивали с использованием тестов нейтральности Таджимы и Фу [24, 25].
Проведение исследования одобрено Комитетом по биомедицинской этике НИИ медицинской генетики Томского НИМЦ. Экспериментальные исследования выполнены на базе Центра коллективного пользования научно-исследовательским оборудованием и экспериментальным биологическим материалом “Медицинская геномика” НИИ медицинской генетики Томского НИМЦ.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Характеристика генетического разнообразия исследуемых популяций по 10 rSNP, ассоциированным с развитием ПЭ
Распределения частот предковых аллелей (ПА) исследованных 10 rSNP в популяциях Северной Евразии и мировых популяциях из проекта “1000 геномов” приведены в табл. 2. Отклонение в соответствии распределения частот генотипов РХВ выявлено среди популяций Северной Евразии в 8% случаев, при этом накоплений по отдельным маркерам или популяциям не наблюдалось. С учетом поправки Бонферрони только три из восьми отклонений от РХВ достигли порогового уровня значимости (р = 0.005): rs56153523 гена SYDE1 (в популяции удмуртов) и rs3802252 гена NDRG1 (в популяциях цезов и нивхов). Такие результаты могут отражать как специфику популяционно-генетических процессов, происходящих в данных популяциях, так и вероятное действие естественного отбора [26]. В целом, частоты аллелей и генотипов исследуемых rSNP в популяциях Северной Евразии находятся в диапазоне, наблюдаемом в мировых популяциях проекта “1000 геномов” [18].
Таблица 2.
Характеристика частот ПА (%) 10 rSNP в изученных популяциях
| Регион | Этнос | rSNP (ген), ассоциированный с ПЭ* | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| rs10423795 (LHB) | rs10985257 (CORO2A) | rs1671215 (RDH13) | rs2167270 (LEP) | rs2227262 (NDRG1) | rs34845949 (SASH1) | rs3802252 (NDRG1) | rs56153523 (SYDE1) | rs72959687 (INHA) | rs8109071 (SYDE1) | ||
| C | A | A | G | C | T | T | C | A | C | ||
| Африка | ESN | 60.6 | 79.8 | 60.6 | 55.6 | 100 | 94.9 | 70.2 | 99.5 | 92.9 | 74.7 |
| GWD | 53.5 | 80.1 | 59.3 | 57.1 | 98.2 | 96.0 | 64.2 | 91.6 | 94.2 | 77.0 | |
| LWK | 47.0 | 70.2 | 65.7 | 50.0 | 99.0 | 90.4 | 76.3 | 87.9 | 91.9 | 67.7 | |
| MSL | 54.7 | 75.3 | 63.5 | 53.5 | 100 | 93.5 | 72.9 | 94.7 | 94.1 | 74.7 | |
| YRI | 53.2 | 79.2 | 63.0 | 53.2 | 100 | 95.4 | 69.4 | 98.1 | 90.3 | 79.6 | |
| Западная Европа | FIN | 38.4 | 72.2 | 77.8 | 60.1 | 85.4 | 74.7 | 42.9 | 70.7 | 79.3 | 69.7 |
| GBR | 42.9 | 75.3 | 70.3 | 61.0 | 80.8 | 69.8 | 44.5 | 81.3 | 80.8 | 79.7 | |
| IBS | 40.7 | 79.0 | 69.2 | 64.5 | 90.7 | 71.0 | 47.7 | 85.5 | 82.2 | 78.5 | |
| TSI | 43.9 | 70.6 | 62.1 | 66.4 | 84.6 | 70.6 | 40.7 | 84.1 | 80.8 | 80.4 | |
| Кавказ | DAG | 31.6 | 69.9 | 80.5 | 37.0 | 85.8 | 86.5 | 87.2 | 92.7 | 78.9 | 87.2 |
| Восточная Европа | UDB | 41.5 | 78.8 | 80.2 | 57.7 | 90.7 | 79.4 | 80.1 | 62.2 | 84.4 | 58.3 |
| RUS | 31.8 | 81.1 | 79.2 | 63.5 | 86.5 | 68.8 | 50.0 | 75.5 | 82.3 | 74.0 | |
| RUSCON | 41.0 | 92.0 | 74.0 | 69.1 | 84.0 | 70.0 | 45.0 | 59.0 | 82.0 | 75.0 | |
| RUSPE | 49.0 | 83.0 | 79.0 | 64.9 | 86.0 | 68.0 | 44.0 | 71.0 | 76.0 | 60.0 | |
| Индостан | GIH | 46.1 | 87.4 | 61.7 | 68.9 | 83.0 | 93.2 | 35.4 | 60.2 | 94.2 | 51.0 |
| ITU | 48.5 | 80.4 | 64.2 | 70.6 | 80.4 | 88.7 | 36.3 | 61.8 | 91.2 | 53.4 | |
| PJL | 47.9 | 81.8 | 65.6 | 77.6 | 77.1 | 87.0 | 32.8 | 69.8 | 92.7 | 59.9 | |
| STU | 41.7 | 85.3 | 60.8 | 73.0 | 78.9 | 89.2 | 34.3 | 60.8 | 92.2 | 52.0 | |
| BEB | 56.4 | 89.5 | 65.7 | 72.7 | 82.0 | 86.0 | 32.6 | 63.4 | 88.4 | 56.4 | |
| Центральная Азия | KAL | 55.5 | 88.0 | 70.3 | 83.7 | 93.1 | 66.3 | 88.7 | 67.4 | 83.7 | 58.9 |
| Средняя Азия | KGS | 74.7 | 87.7 | 71.9 | 72.9 | 86.3 | 67.7 | 70.3 | 70.9 | 83 | 61.1 |
| Сибирь | BUR | 44.7 | 85.8 | 61.7 | 82.8 | 89.9 | 60.9 | 50.0 | 61.5 | 79.7 | 54.7 |
| KHA | 76.7 | 89.5 | 58.9 | 76.6 | 84.4 | 72.9 | 83.5 | 69.8 | 68.9 | 62.6 | |
| HAR | 58.9 | 76.6 | 77.1 | 67.7 | 82.8 | 76.0 | 76.1 | 41.7 | 91.7 | 41.7 | |
| YAK | 64.4 | 76.4 | 70.8 | 71.4 | 90.4 | 63.5 | 58.0 | 53.7 | 93.2 | 47.9 | |
| YAKCON | 59.0 | 96.0 | 67.0 | 73.8 | 84.0 | 78.0 | 52.0 | 48.0 | 88.0 | 41.0 | |
| YAKPE | 60.0 | 92.0 | 71.0 | 78.2 | 89.0 | 75.0 | 60.0 | 44.0 | 88.0 | 41.0 | |
| Восточная Азия | CDX | 69.9 | 93.5 | 80.6 | 87.1 | 94.6 | 61.8 | 47.3 | 72.6 | 83.9 | 64.5 |
| CHB | 64.6 | 88.8 | 81.6 | 78.6 | 87.9 | 65.0 | 41.3 | 80.6 | 80.1 | 66.5 | |
| CHS | 66.2 | 84.3 | 82.4 | 78.1 | 89.0 | 61.4 | 34.3 | 63.3 | 78.1 | 48.1 | |
| JPT | 65.4 | 90.4 | 83.7 | 81.7 | 84.1 | 58.2 | 35.6 | 63.0 | 83.2 | 49.0 | |
| KHV | 66.7 | 88.9 | 75.8 | 76.3 | 91.4 | 59.6 | 44.9 | 65.7 | 82.8 | 49.5 | |
| Дальний Восток | NIN | 69.0 | 81.0 | 64.9 | 68.1 | 87.2 | 77.4 | 94.0 | 76.3 | 86.6 | 61.4 |
| Америка | PEL | 31.8 | 91.8 | 59.4 | 48.8 | 94.7 | 88.8 | 20.6 | 55.3 | 74.7 | 51.2 |
* Cогласно данным, полученным нами ранее [17, 19–22].
Примечание. Полужирным выделены ПА. ESN – ишаны; GWD – гамбийцы; LWK – лухья; MSL – менде; YRI – йоруба; FIN – финны; GBR – британцы; IBS – иберийцы; TSI – тосканцы; DAG – цезы; UDB – удмурты; RUS – русские (CON – контрольная группа, PE – группа больных ПЭ); GIH – индийцы; ITU – индийцы телугу; PJL – пенджабцы; STU – ланкийские тамилы; BEB – бенгальцы; KAL – калмыки; KGS – киргизы; BUR – буряты; KHA – хакасы; HAR – ханты; YAK – якуты (CON – контрольная группа, PE – группа больных ПЭ); CDX, CHB, CHS – китайцы; JPT – японцы; KHV – вьеты; NIN – нивхи; PEL – перуанцы.
Все изученные rSNP полиморфны (за исключением rs2227262 гена NDRG1) в трех популяциях Африки (ишаны, менде, йоруба). Примечательно, что для данного rSNP характерна высокая частота ПА, при этом его минимальные значения наблюдаются среди популяций Индостана (от 77.1% у пенджабцев до 83% у индийцев). Наряду с этим наименьшая вариабельность частоты ПА в 30 популяционных выборках показана для трех rSNP: rs10985257 гена CORO2A (от 69.9% у цезов до 93.5% у китайцев), rs1671215 гена RDH13 (от 58.9% у хакасов до 83.7% у японцев) и rs72959687 гена INHA (от 68.9% у хакасов до 94.2% у гамбийцев).
Наибольшая вариабельность частот ПА выявлена у шести rSNP: rs10423795, rs2167270, rs34845949, rs3802252, rs56153523 и rs8109071. Так, максимальная частота ПА полиморфного варианта rs10423795 гена LHB обнаружена у киргизов (74.7%) и хакасов (76.7%), тогда как минимальная – в популяциях финнов (38.4%), цезов (31.6%), русских (31.8%) и перуанцев (31.8%). Наибольшие значения частоты ПА rs2167270 гена LEP отмечены в популяциях калмыков (83.7%), бурятов (82%), китайцев (87.1%) и японцев (81.7%), минимальная частота показана у цезов (37%) и перуанцев (48.8%). Высокая частота ПА второго полиморфного варианта гена NDRG1 – rs3802252, наблюдается в некоторых популяциях Северной Евразии (87.2% у цезов, 80.1% у удмуртов, 88.7% у калмыков, 83.5% у хакасов, 94% у нивхов), при этом его наименьшая частота характерна для популяций Индостана (от 32.6% у бенгальцев до 36.3% у индийцев телугу), Восточной Азии (34.3% у китайцев и 35.6% у японцев) и Южной Америки (20.6% у перуанцев). Максимальная частота ПА среди представителей негроидной расы отмечается у двух rSNP: rs34845949 гена SASH1 (от 90.4% у лухья до 96% у гамбийцев) и rs56153523 гена SYDE1 (от 87.9% у лухья до 99.5% у ишанов). Наряду с популяциями Африки высокая частота ПА наблюдается среди популяций: Кавказа (86.5% у цезов), Индостана (от 86% у бенгальцев до 93.2% у индийцев) и Южной Америки (88.8% у перуанцев) для rs34845949; Западной Европы (81.3% у британцев, 85.5% у иберийцев, 84.1% у тосканцев), Кавказа (92.7% у цезов) для rs56153523. Низкая частота ПА показана в популяциях Восточной Азии (от 59.6% у вьетов до 65% у китайцев) и популяциях Сибири (63.5% у якутов, 60.9% у бурятов), а также в популяциях Сибири (41.7% у хантов, 53.7% у якутов) и Америки (55.3% у перуанцев) для rs34845949 и rs56153523 соответственно. Кроме того, высокая частота ПА полиморфного варианта rs8109071 гена SYDE отмечена в популяциях Африки (от 67.7% у лухья до 79.6% у йоруба), Кавказа (87.2% у цезов) и Западной Европы (от 69.7% у финнов до 80.4% у тосканцев), тогда как минимальная частота встречается среди популяций Сибири (41.7% у хантов и 47.9% у якутов).
Генетическая дифференциация исследуемых популяций и их взаимоотношения
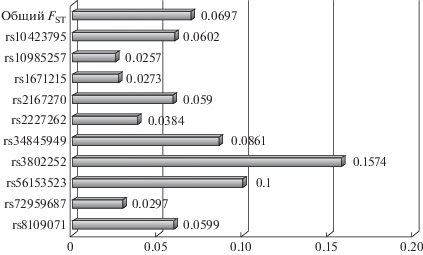
Характеристика степени генетической дифференциации (FST) суммарной выборки из 30 популяций по каждому из 10 rSNP представлена на рис. 1. Все изученные rSNP показали статистически значимую дифференциацию. Согласно полученным результатам, наибольший вклад в формирование межпопуляционного разнообразия вносит различие по частотам аллелей rs3802252 гена NDRG1 (0.157), тогда как наименьшая степень генной дифференциации популяций характерна для: rs10985257 гена CORO2A (0.0257), rs1671215 гена RDH13 (0.0273) и rs72959687 гена INHA (0.0297). Общий коэффициент генетической дифференциации суммарной выборки по изученным rSNP составил 0.0697 (p < 0.001).
Рис. 1.
Генетическая дифференциация суммарной выборки 30 исследуемых популяций по 10 rSNP, ассоциированным с развитием ПЭ.
Анализ межпопуляционных генетических взаимоотношений 10 популяций Северной Евразии, 20 мировых популяций проекта “1000 геномов” и четырех выборок русских и якутских женщин, разделенных на группу контроля и группу больных ПЭ, показал, что две первые главные компоненты объясняют 55% вариабельности частот аллелей исследуемых 10 rSNP (рис. 2). Наблюдается распределение изученных популяций по регионам: Африка (слева в области отрицательного значения первого и положительного значения второго факторов), Кавказ (слева в области отрицательных значений первого и второго факторов), Европа (слева в области отрицательных значений первого и второго факторов), Индостан (справа в области положительных значений первого и второго факторов). Отмечается выделение отдельного кластера монголоидов (справа, преимущественно в области отрицательных значений первого и второго факторов), при этом данный кластер подразделяется согласно территориальному расположению на популяции Сибири, Средней и Центральной Азии и популяции Восточной Азии. Примечательно, что выборки нивхов и перуанцев могут быть объединены в отдельный кластер (слева, в области отрицательных значений первого и положительных значений второго факторов), что согласуется с результатами исследований, в которых показана значимая роль популяции нивхов и популяций Восточной Азии в происхождении популяций Америки [27]. Наряду с кластеризацией исследуемых популяций наблюдается кластеризация группы больных ПЭ и группы контроля у русских (справа в области положительных значений первого и отрицательных значений второго факторов) и якутов (справа в области положительных значений первого и второго факторов), что может указывать на популяционную специфичность подверженности к ПЭ.
Рис. 2.
Пространство главных компонент 34 популяционных выборок по частотам аллелей 10 rSNP, ассоциированных с развитием ПЭ. ESN – ишаны; GWD – гамбийцы; LWK – лухья; MSL – менде; YRI – йоруба; FIN – финны; GBR – британцы; IBS – иберийцы; TSI – тосканцы; DAG – цезы; UDB – удмурты; RUS – русские (CON – контрольная группа, PE – группа больных ПЭ); GIH – индийцы; ITU – индийцы телугу; PJL – пенджабцы; STU – ланкийские тамилы; BEB – бенгальцы; KAL – калмыки; KGS – киргизы; BUR – буряты; KHA – хакасы; HAR – ханты; YAK – якуты (CON – контрольная группа, PE – группа больных ПЭ); CDX, CHB, CHS – китайцы; JPT – японцы; KHV – вьеты; NIN – нивхи; PEL – перуанцы.
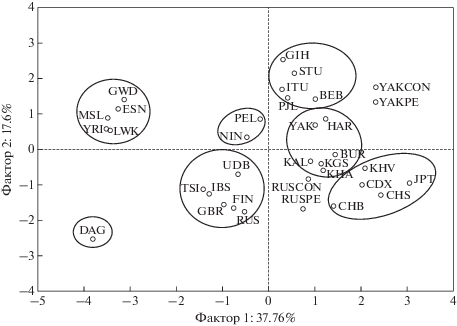
Полученная в пространстве главных компонент картина не отражает географическое расположение данных популяций, что отличается от ожиданий нейтральной эволюции генетического разнообразия за счет миграций и генетического дрейфа в ходе расселения современного человека из Африки. Можно предположить, что такие результаты выявляют участие естественного отбора в ходе адаптации популяций к новым средовым условиям в процессе заселения новых территорий за пределами Африки.
Вклад естественного отбора в формирование генетического разнообразия популяций по изученным маркерам ПЭ
Анализ селективной нейтральности 10 rSNP по 20 мировым популяциям из проекта “1000 геномов” и 10 популяциям Северной Евразии проводили с помощью теста Юинса–Ваттерсона (табл. 3). Отклонение от селективной нейтральности выявлено у пяти rSNP: rs10423795 гена LHB, rs2167270 гена LEP, rs3802252 гена NDRG1, rs56153523 и rs8109071 гена SYDE1. Важно отметить систематическое отклонение от нейтральности rs10423795 и rs8109071 в большинстве исследуемых популяций. Кроме того, в популяциях перуанцев и бурятов показано отклонение от гипотезы селективной нейтральности максимального числа rSNP: rs2167270, rs56153523, rs8109071 и rs10423795, rs3802252, rs8109071 соответственно.
Таблица 3.
Анализ селективной нейтральности 10 rSNP, ассоциированных с развитием преэклампсии
| Регион | Попу-ляции | Значение Fo/Fe | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| rs10423795 (LHB) | rs10985257 (CORO2A) | rs1671215 (RDH13) | rs2167270 (LEP) | rs2227262 (NDRG1) | rs34845949 (SASH1) | rs3802252 (NDRG1) | rs56153523 (SYDE1) | rs72959687 (INHA) | rs8109071 (SYDE1) | ||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| Африка | ESN | 0.57/0.83 | 0.91/0.83 | 0.68/0.83 | 0.99/0.83 | – | 0.91/0.83 | 0.79/0.83 | 0.58/0.83 | 0.99/0.83 | 0.54/0.83 |
| GWD | 0.99/0.83 | 0.74/0.83 | 0.9/0.83 | 0.84/0.83 | 0.94/0.83 | 0.71/0.84 | 0.91/0.84 | 0.88/0.83 | 0.94/0.83 | 0.99/0.83 | |
| LWK | 0.99/0.83 | 0.63/0.83 | 0.92/0.83 | 0.97/0.83 | 0.99/0.83 | 0.72/0.83 | 0.91/0.83 | 0.98/0.83 | 0.54/0.83 | 0.98/0.83 | |
| MSL | 0.99/0.83 | 0.99/0.83 | 0.99/0.83 | 0.98/0.83 | – | 0.7/0.83 | 0.93/0.82 | 0.91/0.83 | 0.83/0.83 | 0.98/0.82 | |
| YRI | 0.78/0.83 | 0.9/0.83 | 0.99/0.84 | 0.69/0.84 | – | 0.51/0.83 | 0.92/0.83 | 0.94/0.83 | 0.98/0.83 | 0.66/0.83 | |
| Западная Европа | FIN | 0.51/0.83 | 0.97/0.83 | 0.99/0.83 | 0.93/0.83 | 0.99/0.83 | 0.99/0.83 | 0.64/0.83 | 0.99/0.83 | 0.99/0.83 | 0.74/0.83 |
| GBR | 0.99/0.83 | 0.99/0.82 | 0.54/0.83 | 0.67/0.83 | 0.99/0.83 | 0.99/0.83 | 0.94/0.83 | 0.99/0.83 | 0.85/0.83 | 0.73/0.83 | |
| IBS | 0.51/0.83 | 0.98/0.83 | 0.81/0.83 | 0.91/0.83 | 0.71/0.83 | 0.5/0.83 | 0.95/0.83 | 0.54/0.83 | 0.52/0.83 | 0.98/0.83 | |
| TSI | 0.99/0.84 | 0.9/0.83 | 0.99/0.83 | 0.98/0.83 | 0.52/0.83 | 0.53/0.83 | 0.99/0.83 | 0.61/0.83 | 0.99/0.83 | 0.55/0.83 | |
| Кавказ | DAG | 0.62/0.83 | 0.87/0.82 | 0.96/0.83 | 0.99/0.83 | 0.63/0.83 | 0.54/0.83 | 0.76/0.82 | 0.98/0.83 | 0.64/0.83 | 0.95/0.83 |
| Восточная Европа | RUS | 0.87/0.83 | 0.5/0.82 | 0.63/0.83 | 0.92/0.83 | 0.99/0.83 | 0.85/0.83 | 0.81/0.83 | 0.54/0.83 | 0.57/0.83 | 0.96/0.83 |
| UDB | 0.97/0.83 | 0.99/0.82 | 0.99/0.83 | 0.9/0.83 | 0.56/0.83 | 0.98/0.83 | 0.99/0.82 | 0.94/0.82 | 0.82/0.83 | 0.69/0.83 | |
| Индостан | BEB | 0.99/0.82 | 0.71/0.83 | 0.91/0.83 | 0.98/0.83 | 0.77/0.83 | 0.6/0.83 | 0.57/0.82 | 0.85/0.83 | 0.99/0.83 | 0.94/0.83 |
| GIH | 0.99/0.83 | 0.72/0.83 | 0.6/0.83 | 0.85/0.83 | 0.81/0.83 | 0.53/0.83 | 0.68/0.83 | 0.51/0.83 | 0.55/0.83 | 0.99/0.83 | |
| ITU | 0.89/0.83 | 0.94/0.83 | 0.99/0.83 | 0.99/0.83 | 0.96/0.83 | 0.67/0.83 | 0.67/0.83 | 0.74/0.83 | 0.83/0.83 | 0.99/0.83 | |
| PJL | 0.98/0.83 | 0.55/0.83 | 0.99/0.83 | 0.96/0.83 | 0.99/0.83 | 0.69/0.83 | 0.97/0.83 | 0.98/0.83 | 0.84/0.83 | 0.61/0.83 | |
| STU | 0.99/0.83 | 0.77/0.83 | 0.77/0.83 | 0.93/0.83 | 0.9/0.83 | 0.94/0.83 | 0.96/0.83 | 0.76/0.83 | 0.99/0.83 | 0.82/0.83 | |
| Центральная Азия | KGS | 0.65/0.83 | 0.87/0.82 | 0.97/0.83 | 0.77/0.83 | 0.79/0.83 | 0.52/0.83 | 0.88/0.83 | 0.85/0.83 | 0.8/0.83 | 0.9/0.82 |
| Сибирь | HAR | 0.92/0.83 | 0.94/0.83 | 0.99/0.83 | 0.98/0.83 | 0.75/0.83 | 0.98/0.83 | 0.98/0.83 | 0.61/0.83 | 0.99/0.83 | 0.95/0.83 |
| YAB | 0.77/0.83 | 0.98/0.83 | 0.99/0.83 | 0.92/0.83 | 0.93/0.83 | 0.99/0.83 | 0.75/0.82 | 0.99/0.83 | 0.99/0.83 | 0.81/0.83 | |
| BUR | 0.75/0.83 | 0.87/0.83 | 0.61/0.83 | 0.96/0.83 | 0.79/0.83 | 0.98/0.83 | 0.98/0.83 | 0.78/0.83 | 0.86/0.83 | 0.78/0.83 | |
| KHA | 0.99/0.83 | 0.53/0.82 | 0.97/0.83 | 0.98/0.83 | 0.81/0.83 | 0.84/0.83 | 0.96/0.82 | 0.59/0.83 | 0.6/0.83 | 0.99/0.83 | |
| Средняя Азия | KAL | 0.83/0.83 | 0.98/0.83 | 0.64/0.83 | 0.76/0.83 | 0.99/0.83 | 0.94/0.83 | 0.59/0.82 | 0.82/0.83 | 0.99/0.83 | 0.52/0.83 |
| Восточная Азия | CDX | 0.99/0.83 | 0.77/0.83 | 0.96/0.83 | 0.87/0.83 | 0.96/0.83 | 0.57/0.83 | 0.5/0.83 | 0.93/0.83 | 0.77/0.83 | 0.99/0.83 |
| CHB | 0.97/0.83 | 0.62/0.83 | 0.97/0.83 | 0.52/0.83 | 0.99/0.83 | 0.98/0.83 | 0.99/0.84 | 0.93/0.83 | 0.95/0.83 | 0.97/0.83 | |
| CHS | 0.99/0.83 | 0.55/0.83 | 0.99/0.83 | 0.99/0.83 | 0.51/0.83 | 0.94/0.83 | 0.93/0.83 | 0.72/0.83 | 0.99/0.83 | 0.65/0.83 | |
| JPT | 0.99/0.83 | 0.85/0.83 | 0.82/0.83 | 0.92/0.83 | 0.75/0.83 | 0.61/0.83 | 0.97/0.83 | 0.73/0.83 | 0.93/0.83 | 0.97/0.83 | |
| KHV | 0.87/0.83 | 0.98/0.83 | 0.5/0.83 | 0.99/0.83 | 0.93/0.83 | 0.77/0.83 | 0.89/0.83 | 0.55/0.83 | 0.97/0.83 | 0.87/0.83 | |
| Дальний Восток | NIN | 0.51/0.83 | 0.94/0.82 | 0.93/0.83 | 0.99/0.83 | 0.98/0.83 | 0.54/0.83 | 0.55/0.83 | 0.53/0.83 | 0.88/0.83 | 0.52/0.83 |
| Америка | PEL | 0.72/0.82 | 0.63/0.83 | 0.83/0.82 | 0.66/0.83 | 0.74/0.83 | 0.55/0.83 | 0.92/0.83 | 0.95/0.82 | 0.5/0.82 | 0.52/0.83 |
Примечание. Fo – наблюдаемая гомозиготность, Fe – ожидаемая гомозиготность. Серым выделены значения Fo/Fe, для которых показано статистически значимое отклонение от селективной нейтральности (p < 0.05). Темно-серым отмечены значения, свидетельствующие о действии направленного отбора (при Fo˃ Fe), тогда как светло-серым цветом показано возможное действие балансирующего отбора (при Fo <Fe). ESN – ишаны; GWD – гамбийцы; LWK – лухья; MSL – менде; YRI – йоруба; FIN – финны; GBR – британцы; IBS – иберийцы; TSI – тосканцы; DAG – цезы; UDB – удмурты; RUS – русские (CON – контрольная группа, PE – группа больных ПЭ); GIH – индийцы; ITU – индийцы телугу; PJL – пенджабцы; STU – ланкийские тамилы; BEB – бенгальцы; KAL – калмыки; KGS – киргизы; BUR – буряты; KHA – хакасы; HAR – ханты; YAK – якуты (CON – контрольная группа, PE – группа больных ПЭ); CDX, CHB, CHS – китайцы; JPT – японцы; KHV – вьеты; NIN – нивхи; PEL – перуанцы.
Отклонение от селективной нейтральности свидетельствует о действии направленного отбора в случае статистически значимого увеличения наблюдаемой гомозиготности (Fo) по отношению к ожидаемой (Fe), тогда как значимое снижение Fo относительно Fe характерно для балансирующего отбора [26]. Таким образом, регуляторный полиморфный вариант rs10423795 гена LHB находится под действием направленного отбора в 10 популяциях (гамбийцы, лухья, менде, британцы, тосканцы, индийцы, индийцы телугу, пенджабцы, бенгальцы, калмыки), тогда как сигналы балансирующего отбора выявлены только в двух популяциях (йоруба, буряты). Действие естественного отбора характерно для rs2167270 гена LEP в популяциях Африки (направленный отбор у ишанов, лухья, менде; балансирующий отбор у йоруба) и Америки (балансирующий отбор у перуанцев). В случае гена NDRG1 действие естественного отбора выявлено только для rs3802252. Так, в популяциях британцев, иберийцев, бурятов и вьетов обнаружено действие направленного отбора, тогда как у финнов, русских и китайцев показано действие балансирующего отбора. Под действием направленного отбора также находятся два rSNP гена SYDE1: rs56153523 у якутов, перуанцев и rs8109071 у индийцев, индийцев телугу, бенгальцев, японцев, вьетов. Примечательно, что действие балансирующего отбора наблюдается только для rs8109071 у ланкийских тамилов, бурятов, якутов, китайцев и перуанцев.
Оценку роли естественного отбора в формировании уровня генетического разнообразия популяций, разделенных согласно расовой принадлежности на группы негроидов, европеоидов и монголоидов, проводили с использованием тестов нейтральности Таджимы и Фу (табл. 4) [24, 25]. Для трех исследуемых групп положительные и отрицательные значения критерия D Таджимы статистически значимо не отличались. Значения критерия Fs теста Фу показали статистически значимые отличия для пяти rSNP: rs72959687 гена INHA, rs2227262 гена NDRG1, rs56153523 и rs8109071 гена SYDE1 в группе негроидов и rs10423795 гена LHB в группе европеоидов. Все значения критерия Fs имеют отрицательное значение, что может свидетельствовать как о действии очищающего отбора, так и о популяционной экспансии и миграции.
Таблица 4.
Значение тестов нейтральности Таджимы и Фу
| rSNP | Тест Таджимы | Тест Фу | ||||
|---|---|---|---|---|---|---|
| Критерий D (rank score) | Критерий Fs (rank score) | |||||
| YRI | CEU | CHB | YRI | CEU | CHB | |
| rs10423795 | 0.2 (0.1) | 1.2 (0.1) | 0.7 (0.1) | –149.4 (1.0) | –130.0 (1.4) | –124.2 (1.1) |
| rs10985257 | –0.2 (0.2) | 0.3 (0.3) | 1.3 (0.3) | –159.9 (1.1) | –111.2 (1.2) | –93.6 (0.9) |
| rs1671215 | 0.1 (0.1) | 0.8 (0.1) | 0.7 (0.1) | –114.8 (0.6) | –62.3 (0.7) | –79.1 (0.7) |
| rs2167270 | –0.8 (0.6) | –0.5 (0.6) | –0.5 (0.6) | –107.8 (0.6) | –45.4 (0.5) | –23.1 (0.2) |
| rs2227262 | –0.7 (0.5) | 0.6 (0.2) | 0.2 (0.2) | –207.6 (2.2) | –113.4 (1.2) | –104.6 (0.9) |
| rs34845949 | –0.1 (0.1) | 1.0 (0.1) | 0.9 (0.1) | –69.8 (0.3) | –60.6 (0.7) | –59.2 (0.6) |
| rs3802252 | –0.5 (0.3) | –0.2 (0.5) | 1.1 (0.5) | –143.7 (0.9) | –54.9 (0.6) | –62.6 (0.6) |
| rs56153523 | –0.4 (0.3) | 0.6 (0.2) | 1.2 (0.2) | –188.2 (1.7) | –84.1 (0.9) | –113.8 (1.0) |
| rs72959687 | –0.9 (0.7) | 0.5 (0.2) | 1.0 (0.2) | –176.5 (1.4) | –76.7 (0.8) | –123.5 (1.1) |
| rs8109071 | –0.3 (0.2) | 1.5 (0.0) | 1.9 (0.0) | –180.8 (1.5) | –86.1 (0.9) | –97.7 (0.9) |
Примечание. Изученные группы разделены согласно расовой принадлежности, где CEU – европеоиды, CHB – монголоиды, YRI – негроиды. Критерий rank score – ранговое значение, равное –lg(p), где p – уровень значимости. Статистически значимые отличия выявлены при значении критерия rank score > 1.3 (выделены полужирным), что соответствует уровню значимости p < 0.05.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Полученные нами ранее результаты [17, 19–22] свидетельствуют о важной роли 10 rSNP восьми ДЭГ в формировании наследственной подверженности к развитию ПЭ у русских и якутов. В рамках настоящей работы по данным маркерам проведен поиск сигналов естественного отбора на микроэволюционном уровне, который выявил значимый вклад адаптивной эволюции в генетическую архитектуру ПЭ по системе семи rSNP пяти ДЭГ (рис. 3). Так, действие очищающего отбора выявлено с помощью теста нейтральности Фу для rs2227262 гена NDRG1 и rs72959687 гена INHA. При использовании теста Юинса–Ваттерсона отклонение от селективной нейтральности показали два rSNP: rs2167270 гена LEP и rs3802252 гена NDRG1. Кроме того, по совокупности результатов тестов Фу и Юинса–Ваттерсона действие естественного отбора обнаружено для rs10423795 гена LHB и rs56153523, rs8109071 гена SYDE1.
Рис. 3.
Вклад естественного отбора на микроэволюционном уровне в формирование наследственной подверженности к преэклампсии (ПЭ).

Продукты ДЭГ, биологические процессы и белок-белковые взаимодействия с их участием. Предполагаемая роль выявленных факторов транскрипции в этиопатогенезе ПЭ
Гены, rSNP которых находятся под действием естественного отбора, это новые гены-кандидаты ПЭ, выявленные благодаря анализу транскриптома плацентарной ткани [28–35]. Несмотря на то что функции большинства ДЭГ изучены недостаточно, результаты ряда исследований позволяют сделать выводы об их возможном вкладе в развитие ПЭ, основанные на характеристике молекулярных механизмов и функций продуктов данных генов (табл. 5).
Таблица 5.
Характеристика ДЭГ, rSNP которых находятся под действием естественного отбора на микроэволюционном уровне
| Ген (продукт гена) | Основные функции и их возможная роль в этиопатогенезе ПЭ | Cсылка |
|---|---|---|
| INHA (α-субъединица белковых комплексов ингибина А и В) | Ингибины являются факторами роста и дифференцировки. При ПЭ наблюдается увеличение уровня ингибина А в плаценте и сыворотке, происходящее преимущественно за счет клеток трофобласта. Высокий уровень ингибина А может играть компенсаторную роль в восстановлении функций плаценты | [31–34, 36, 37] |
| LEP (лептин) | Лептин обладает провоспалительными свойствами, в высоких концентрациях вызывает развитие гипертонии. Экспрессия плацентарного лептина поддерживает имплантацию, рост плаценты, защищает клетки от апоптоза. При ПЭ наблюдается гиперлептинемия и сверхэкспрессия данного гена в плаценте | [29–35, 38–41] |
| LHB (β-полипептид лютеинизирующего гормона) | Способствует сперматогенезу и овуляции, стимулируя семенники и яичники к синтезу стероидов. При ПЭ наблюдается повышение β-полипептида лютеинизирующего гормона в плаценте и сыворотке, что может быть следствием изменений в физиологии трофобласта | [28, 32, 35] |
| NDRG1 (цитоплазматический белок суперсемейства гидролаз) | Белок NDRG1 обладает цитопротективными свойствами. Сверхкспрессия гена NDRG1 в условиях гипоксии обеспечивает дифференцировку и уменьшение степени повреждения клеток трофобласта. Для ПЭ характерно повышение уровня экспрессии данного гена | [30–32, 42, 43] |
| SYDE1 (белок, активирующий GTPазу, гомолог 1) | Белок SYDE1 участвует в клеточной миграции, инвазии и ремоделировании цитоскелета, способен стимулировать миграцию клеток трофобласта. Ген SYDE1 сверхэкспрессируется в плаценте при ПЭ | [30, 44] |
Функциональная аннотация пяти ДЭГ проведена с помощью онлайн ресурса “WebGestalt”. Выявлено 10 категорий Gene Ontology (включающих различные биологические процессы и молекулярные функции), в которые вовлечены два и более исследуемых ДЭГ. Гены INHA и LEP представлены в шести категориях биологических процессов, связанных с секрецией фолликулостимулирующего гормона (ФСГ, GO:0046884, 1.3 × 10–6), регуляцией секреции ФСГ (GO:0046880, 9 × 10–7), положительной регуляцией секреции ФСГ (GO:0046881, 9 × 10–7), секрецией гонадотропинов (GO:0032274, 6.3 × 10–6), регуляцией секреции гонадотропинов (GO:0032276, 3.3 × 10–6), положительной регуляцией секреции гонадотропинов (GO:0032278, 1.3 × 10–6). Для трех ДЭГ (INHA, LEP, LHB) выявлены три категории биологических процессов, связанных с дифференциацией пола (GO:0007548, 3 × 10–5), развитием первичных половых признаков (GO:0045137, 1.7 × 10–5), развитием гонад (GO:0008406, 1.6 × 10–5), и одна категория молекулярных функций, связанная с гормональной активностью (GO:0005179, 2.5 × × 10–6). Наряду с этим, два ДЭГ (NDRG1, SYDE1) не обладают статистически значимой общностью биологических функций.
Взаимоотношения продуктов пяти ДЭГ охарактеризованы с помощью онлайн ресурса “STRING” [45]. Транскрипционные факторы (ТФ), взаимодействующие с семью rSNP, ассоциированными с развитием ПЭ, идентифицировали с использованием ресурса “RegulomeDB” [46]. Согласно полученным данным, cемь rSNP пяти ДЭГ располагаются в сайтах связывания с 87 ТФ, из которых 21 ТФ общий для двух и более изученных генов. В дальнейший анализ включены семь общих наиболее значимых ТФ: MYC, SP1, POLR2A, CEBPB, CEBPG, RAD21, BHLHE40. В качестве критериев выбора использовали: количество исследуемых rSNP, локализованных в сайтах связывания с каждым ТФ; функциональную значимость выявленных ТФ; степень и эффекты взаимодействия ТФ с продуктами пяти ДЭГ и между собой. На рис. 4 представлена характеристика белок-белковых взаимодействий пяти ДЭГ и семи ТФ.
Рис. 4.
Характеристика белок-белковых взаимодействий продуктов пяти ДЭГ и семи ТФ. Белки изображены в виде кругов, подсвечиванием выделены продукты исследуемых пяти ДЭГ. Толщина прямых линий отражает степень доказательности функциональной связи между белками. Знаки на концах линий характеризуют эффекты белок-белковых взаимодействий: стрелка – положительный, перпендикулярная линия – негативный, круг – неспецифичный. Пунктирной линией соединены ТФ и белки, rSNP в генах которых расположены в сайтах связывания с данными ТФ.
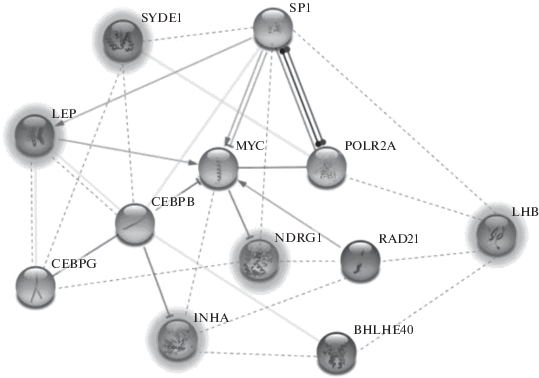
Согласно полученным данным, включенные в исследование ТФ могут играть значимую роль в этиопатогенезе ПЭ. Так, модулятор окислительного стресса CEBPB (CCAAT/Enhancer Binding Protein Beta) участвует в пролиферации и дифференцировке клеток, иммунных и воспалительных ответах [47, 48]. ТФ CEBPB контролирует процесс децидуализации у мышей, тогда как у человека он играет значимую роль в имплантации эмбриона и поддержании беременности [48, 49]. Показано, что сверхэкспрессия CEBPB, которая возникает в условиях гипоксии, приводит к нарушению инвазии вневорсинчатого трофобласта, поверхностной плацентации и развитию ПЭ [50, 51]. В сайтах связывания с данным ТФ расположены rs2167270 гена LEP и rs8109071 гена SYDE1. Поскольку эти гены участвуют в продукции провоспалительных медиаторов, способствуют клеточной миграции и формированию воспалительного ответа [44, 52, 53], можно предположить, что посредством ТФ CEBPB они могут вносить существенный вклад в этиопатогенез ПЭ на стадии формирования плаценты.
Еще одна важная функция ТФ CEBPB заключается в подавлении транскрипции гена INHA, кодирующего α-субъединицу белковых комплексов ингибина А и В (рис. 4). Показано, что ингибины принадлежат к суперсемейству TGF-β и являются факторами роста и дифференцировки, участвуют в регуляции апоптоза, иммунного ответа и секреции гормонов. Следует отметить, что исследуемый в данной работе rs72959687 гена INHA находится в сайтах связывания с ТФ MYC (MYC Proto-Oncogene, BHLH Transcription Factor). Примечательно, что ТФ CEBPB проявляет репрессорную активность в отношении ТФ MYC [54] (рис. 4). Такое взаимодействие приводит к смещению дифференцировки Т-клеток в сторону Т-хелперов второго типа, которые играют важную роль в успешном протекании беременности путем регулирования иммунного ответа на плод [3]. На сегодняшний день известно, что ТФ MYC контролирует клеточную пролиферацию, дифференцировку и апоптоз [55], способен ингибировать транскрипцию гена NDRG1 (рис. 4), продукт которого обладает цитопротективными свойствами [42, 43, 56]. Кроме того, в клетках цитотрофо-бласта наблюдается повышение уровня экспрессии ТФ MYC, который участвует в пролиферации данных клеток [57]. Также отмечено, что сверхэкспрессия ТФ MYC в цитотрофобласте препятствует его дифференцировке в синцитиотрофобласт [57, 58]. Интересно, что цитостатическую активность ТФ CEBPB ингибирует образование гетеродимера с ТФ CEBPG (CCAAT/Enhancer Binding Protein Gamma), который способствует пролиферации клеток [59] (рис. 4). ТФ CEBPG экспрессируется во всех клетках млекопитающих, однако его регуляторные функции не были охарактеризованы. Показано, что в сайтах связывания с данным ТФ расположены три rSNP: rs2167270 гена LEP, rs3802252 гена NDRG1, rs8109071 гена SYDE1.
Следующий выявленный ТФ BHLHE40 (Basic Helix-Loop-Helix Family Member E40) – белок E40 с основным доменом спираль–петля–спираль, модулирующий хондрогенез в рамках сАМР-сигнального пути. Этот ТФ обладает репрессорной активностью, участвует в различных биологических процессах, в том числе в контроле циркадного ритма и клеточной дифференцировке. Повышение его экспрессии регулируется многочисленными стимулами, включая ретиноевую кислоту, TGFβ, сАМР, цитокины, инсулин и гипоксию [60]. Важно отметить, что в плацентарной ткани при ПЭ наблюдается сверхэкспрессия ТФ BHLHE40 [30–32, 60], в сайтах связывания которого располагаются rs10423795 гена LHB и rs72959687 гена INHA.
ТФ RAD21 (RAD21 Cohesin Complex Component) участвует в когезии сестринских хроматид, правильной сегрегации хромосом, пострепликативной репарации ДНК. Примечательно, что в интерфазе когезины могут контролировать экспрессию генов. Кроме того, ТФ RAD21 может влиять на риск развития ПЭ посредством регуляции клеточного цикла [61]. Согласно полученным данным, в сайтах связывания с ТФ находятся четыре rSNP (rs10423795 гена LHB, rs72959687 гена INHA, rs2227262 и rs3802252 гена NDRG1), также показана функциональная связь между ТФ RAD21 и ТФ MYC (рис. 4).
Интерес представляет ТФ SP1 (Specificity Protein 1), который может активировать или подавлять транскрипцию большого количества генов в ответ на физиологические и патологические стимулы [57]. Данный ТФ активируется в условиях гипоксии, участвует в воспалительном ответе и различных биологических процессах, таких как ангиогенез, рост и дифференцировка клеток [62, 63]. В плаценте ТФ SP1 участвует в дифференцировке цитотрофобласта и регуляции экспрессии нескольких белков [64], а также может играть важную роль в дифференцировке и развитии клеток эмбриона, децидуализации эндометрия [63]. Для ПЭ характерен низкий уровень экспрессии SP1 в плацентарной ткани [63, 65, 66]. Примечательно, что в сайтах связывания ТФ SP1 расположены rs10423795 гена LHB, rs2227262 и rs3802252 гена NDRG1, rs8109071 гена SYDE1. Кроме того, выявлены белок-белковые взаимодействия ТФ SP1 и MYC, а также образование связи между SP1 и ТФ POLR2A (рис. 4).
Последний общий ТФ – POLR2A (RNA Polymerase II Subunit A), крупнейший каталитический компонент РНК-полимеразы II, который взаимодействует с тремя rSNP: rs10423795 гена LHB, rs2227262 и rs3802252 гена NDRG1. Можно предположить, что такое взаимодействие приводит к активации транскрипции генов LHB и NDRG1, необходимой для защиты клеток трофобласта в условиях гипоксии [43, 67], тогда как недостаточный уровень их экспрессии способствует развитию патологического процесса.
Таким образом, полученные результаты свидетельствуют о важной роли пяти ДЭГ, rSNP которых находятся под действием естественного отбора, в развитии ПЭ на ранних этапах формирования плаценты. Кроме того, семь исследованных rSNP располагаются в сайтах связывания общих ТФ, что, вероятно, указывает на их вовлеченность в общие молекулярно-генетические механизмы этиопатогенеза ПЭ.
Соответствие полученных результатов эволюционным гипотезам, объясняющим происхождение ПЭ
Сформулировано большое количество эволюционных гипотез, объясняющих причины возникновения ПЭ [11, 13–15, 68–74]. Однако до настоящего времени ни одна из гипотез не была подтверждена, поскольку существовали лишь косвенные свидетельства возможного вклада естественного отбора в формирование данной патологии [4, 75–79]. Результаты нашего предыдущего исследования впервые показали значимую роль слабого очищающего отбора на макроэволюционном уровне в ряду представителей эволюционной линии парвотряда Catarrhini (макак-резус, орангутан, шимпанзе, человек) в формировании наследственной предрасположенности к ПЭ по четырем rSNP: rs2227262 гена NDRG1 [17, 20], rs10985257 гена CORO2A [17, 21], rs72959687 гена INHA и rs34845949 гена SASH1 [17]. Следует отметить, что полученные результаты согласуются с гипотезой M.G. Elliot о “случайном закреплении” неблагоприятного фенотипа для rs2227262 гена NDRG1 [68] и гипотезой “генетического конфликта интересов” D. Haig [69] для rs10985257 гена CORO2A, rs72959687 гена INHA и rs34845949 гена SASH1.
В настоящей работе впервые выявлен вклад естественного отбора на микроэволюционном уровне в формирование генетической архитектуры ПЭ по семи rSNP пяти ДЭГ (рис. 3 и 5). Примечательно, что с развитием ПЭ у русских (rs10423795 гена LHB, rs56153523 гена SYDE1) и якутов (rs2227262 и rs3802252 гена NDRG1) ассоциирован ПА (рис. 5б), что, вероятно, соответствует гипотезе “предковой предрасположенности” [80]. Согласно этой гипотезе, адаптивные для древних популяций человека ПА могут оказывать вредное воздействие в случае изменения условий окружающей среды и образа жизни, которые происходили с момента расселения современного человека из Африки. Важное значение также имеют ассоциации производных аллелей трех rSNP (rs2167270 гена LEP, rs72959687 гена INHA, rs8109071 гена SYDE1) с ПЭ у русских (рис. 5в), поскольку такие результаты можно рассматривать с точки зрения гипотезы “генетического конфликта интересов” [69]. Основной причиной возникновения ПЭ может быть действие отбора как на гены плода, которые повышают поступление питательных веществ, так и на гены матери, направленные на сохранение общих ресурсов организма посредством ограничения потребностей плода. Следует отметить, что гипотеза “генетического конфликта интересов” является частным случаем гипотезы “Черной королевы” среди представителей одного вида [70]. Так, ПЭ могла сформироваться в процессе непрерывной “гонки вооружений” между матерью и плодом, наследующим материнские и отцовские гены, как побочный результат адаптивной эволюции [70].
Рис. 5.
Частоты предковых аллелей (ПА) 10 rSNP в изученных популяциях. Частоты ПА трех rSNP, действие естественного отбора на которые не выявлено (а). Частоты ПА четырех rSNP, находящихся под действием естественного отбора, с развитием ПЭ ассоциирован ПА (б). Частоты ПА трех rSNP, находящихся под действием естественного отбора, с развитием ПЭ ассоциирован производный аллель (в).
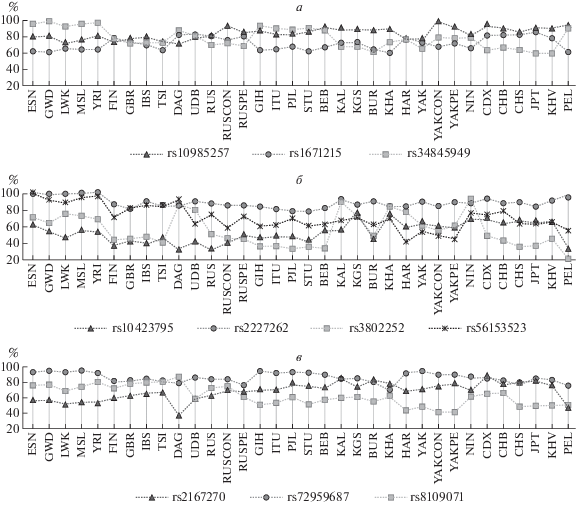
Существование расовых и этнических различий в частотах развития ПЭ в современных популяциях человека представляет большой интерес для исследователей, поскольку такие отличия могут быть результатом адаптивных изменений, возникающих на протяжении формирования генофондов данных популяций [2, 6, 10, 11]. Примечательно, что из 10 включенных в исследование rSNP восьми ДЭГ с развитием ПЭ в этнической выборке русских ассоциированы семь rSNP шести ДЭГ (rs10985257 гена CORO2A, rs72959687 гена INHA; rs2167270 гена LEP; rs10423795 гена LHB; rs1671215 гена RDH13; rs56153523 и rs8109071 гена SYDE1), тогда как у якутов показана ассоциация трех rSNP двух ДЭГ (rs34845949 гена SASH1, rs2227262 и rs3802252 гена NDRG1). Согласно результатам анализа генетической дифференциации, различие по частотам аллелей rs3802252 гена NDRG1, ассоциированного с развитием ПЭ у якутов, вносит наибольший вклад в формирование межпопуляционного разнообразия (FST = 0.157) (рис. 1 и 5, табл. 3). В то же время наименьшая степень генной дифференциации исследованных популяций наблюдается по трем rSNP, ассоциированным с развитием ПЭ у русских: rs10985257 гена CORO2A (FST = 0.0257), rs1671215 гена RDH13 (FST = 0.0273) и rs72959687 гена INHA (FST = 0.0297), что может свидетельствовать о популяционно-специфичном характере наследственной подверженности к ПЭ (рис. 1 и 5, табл. 3).
Таким образом, результаты настоящего исследования выявили значимый вклад естественного отбора на микроэволюционном уровне в формирование генетической подверженности и популяционной специфичности к развитию ПЭ. Биоинформатический анализ позволил установить важную роль пяти ДЭГ, rSNP которых находятся под действием естественного отбора, в развитии данной патологии на ранних этапах формирования плаценты. Наряду с этим обнаружены общие ТФ, показывающие вовлеченность семи исследованных rSNP в общие молекулярно-генетические механизмы этиопатогенеза ПЭ. Кроме того, полученные результаты соответствуют представлениям гипотезы “предковой предрасположенности” (rs10423795 гена LHB, rs56153523 гена SYDE1, rs2227262 и rs3802252 гена NDRG1) и гипотезы “генетического конфликта интересов” (rs2167270 гена LEP, rs72959687 гена INHA, rs8109071 гена SYDE1), которые рассматривают причины возникновения ПЭ с точки зрения адаптивной эволюции. В целом показана применимость эволюционного подхода к анализу генетической компоненты ПЭ и возможная перспектива его дальнейшего использования для изучения данного осложнения беременности.
Работа выполнена при поддержке Российского фонда фундаментальных исследований (грант № 18-29-13045).
Проведение исследования одобрено Комитетом по биомедицинской этике НИИ медицинской генетики Томского НИМЦ. У всех пациентов получено письменное информированное согласие.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Phipps E.A., Thadhani R., Benzing T., Karumanchi S.A. (2019) Pre-eclampsia: pathogenesis, novel diagnostics and therapies. Nat. Rev. Nephrol. 15(5), 275–289. https://doi.org/10.1038/s41581-019-0119-6
Tannetta D., Sargent I. (2013) Placental disease and the maternal syndrome of preeclampsia: missing links? Curr. Hypertens. Rep. 15(6), 590–599. https://doi.org/10.1007/s11906-013-0395-7
Gathiram P., Moodley J. (2016) Pre-eclampsia: its pathogenesis and pathophysiolgy. Cardiovasc. J. Afr. 27(2), 71–78. https://doi.org/10.5830/CVJA-2016-009
Robillard P.Y., Dekker G., Chaouat G., Scioscia M., Iacobelli S., Hulsey T.C. (2017) Historical evolution of ideas on eclampsia/preeclampsia: A proposed optimistic view of preeclampsia. J. Reprod. Immunol. 123, 72–77. https://doi.org/10.1016/j.jri.2017.09.006
Адамян Л.В., Артымук Н.В., Башмакова Н.В., Белокринницкая Т.Е., Беломестнов С.Р., Братищев И.В., Вученович Ю.Д., Краснопольский В.И., Куликов А.В., Левит А.Л., Никитина Н.А., Петрухин В.А., Пырегов А.В., Серов В.Н., Сидорова И.С., Филиппов О.С., Ходжаева З.С., Холин А.М., Шешко Е.Л., Шифман Е.М., Шмаков Р.Г. (2016) Клинические рекомендации “Гипертензивные расстройства во время беременности, в родах и послеродовом периоде. Преэклампсия. Эклампсия”. Москва, 72 с.
Burton G.J., Redman C.W., Roberts J.M., Moffett A. (2019) Pre-eclampsia: pathophysiology and clinical implications. BMJ. 366, l2381. https://doi.org/10.1136/bmj.l2381
Трифонова Е.А., Сваровская М.Г., Сереброва В.Н., Куценко И.Г., Агаркова Л.А., Степанов И.А., Жилякова О.В., Габидулина Т.В., Ижойкина Е.В., Степанов В.А. (2020) Геномные и постгеномные технологии в генетике преэклампсии. Генетика. 56(5), 495–513. https://doi.org/10.31857/S0016675820050136
Zhang G., Zhao J., Yi J., Luan Y., Wang Q. (2016) Association between gene polymorphisms on chromosome 1 and susceptibility to pre-eclampsia: an updated meta-analysis. Med. Sci. Monit. 22, 2202–2214. https://doi.org/10.12659/msm.896552
Zhou L., Cheng L., He Y., Gu Y., Wang Y., Wang C. (2016) Association of gene polymorphisms of FV, FII, MTHFR, SERPINE1, CTLA4, IL10, and TNFalpha with pre-eclampsia in Chinese women. Inflamm. Res. 65(9), 717–724. https://doi.org/10.1007/s00011-016-0953-y
Gong J., Savitz D.A., Stein C.R., Engel S.M. (2012) Maternal ethnicity and pre-eclampsia in New York City, 1995-2003. Paediatr. Perinat. Epidemiol. 26(1), 45–52. https://doi.org/10.1111/j.1365-3016.2011.01222.x
Nakimuli A., Chazara O., Byamugisha J., Elliott A.M., Kaleebu P., Mirembe F., Moffett A. (2014) Pregnancy, parturition and preeclampsia in women of African ancestry. Am. J. Obstet. Gynecol. 210(6), 510–520. e1. https://doi.org/10.1016/j.ajog.2013.10.879
Xiao J., Shen F., Xue Q., Chen G., Zeng K., Stone P., Zhao M., Chen Q. (2014) Is ethnicity a risk factor for developing preeclampsia? An analysis of the prevalence of preeclampsia in China. J. Hum. Hypertens. 28(11), 694–698. https://doi.org/10.1038/jhh.2013.148
Brown I.J., Tzoulaki I., Candeias V., Elliott P. (2009) Salt intakes around the world: implications for public health. Int. J. Epidemiol. 38(3), 791–813. https://doi.org/10.1093/ije/dyp139
Brown E.A., Ruvolo M., Sabeti P.C. (2013) Many ways to die, one way to arrive: how selection acts through pregnancy. Trends. Genet. 29(10), 585–592. https://doi.org/10.1016/j.tig.2013.03.001
Ahmed S., Ibrahim M.E., Khalil E. (2017) High altitude and pre-eclampsia: adaptation or protection. Med. Hypotheses. 104, 128–132. https://doi.org/10.1016/j.mehy.2017.05.007
Handa V.L., Lockhart M.E., Fielding J.R., Bradley C.S., Brubaker L., Cundiff G.W., Ye W., Richter H.E., Pelvic Floor Disorders Network (2008) Racial differences in pelvic anatomy by magnetic resonance imaging. Obstet. Gynecol. 111(4), 914–920. https://doi.org/10.1097/AOG.0b013e318169ce03
Сереброва В.Н., Трифонова Е.А., Степанов В.А. Международный конгресс “VII Съезд Вавиловского общества генетиков и селекционеров, посвященный 100-летию кафедры генетики СПбГУ, и ассоциированные симпозиумы”. (2019) Санкт-Петербург, Россия. Сборник тезисов. С. 673.
1000 Genomes Project Consortium, Auton A., Brooks L.D., Durbin R.M., Garrison E.P., Kang H.M., Korbel J.O., Marchini J.L., McCarthy S., McVean G.A., Abecasis G.R. (2015) A global reference for human genetic variation. Nature. 526(7571), 68–74. https://doi.org/10.1038/nature15393
Сереброва В.Н., Трифонова Е.А., Габидулина Т.В., Бухарина И.Ю., Агаркова Т.А., Евтушенко И.Д., Максимова Н.Р., Степанов В.А. (2016) Выявление новых маркеров предрасположенности к преэклампсии путем анализа регуляторных участков генов, демонстрирующих дифференциальную экспрессию в плацентарной ткани. Молекуляр. биология. 50(5), 870–879. https://doi.org/10.7868/S0026898416050165
Сереброва В.Н., Трифонова Е.А., Степанов В.А. (2018) Эволюционно-генетический анализ роли регуляторных участков гена NDRG1 в формировании структуры наследственной предрасположенности к преэклампсии в популяциях различного этнического происхождения. Мед. генетика. 17(1), 32–36.
Сереброва В.Н., Трифонова Е.А., Степанов В.А. (2018) Эволюционно-генетический анализ роли регуляторных участков гена CORO2A в формировании наследственной предрасположенности к преэклампсии у русских и якутов. Научные результаты биомедицинских исследований. 4(3), 38–48. https://doi.org/10.18413/2313-8955-2018-4-3-0-4
Сереброва В.Н., Трифонова Е.А., Ворожищева А.Ю., Степанов В.А. (2019) Роль регуляторных участков гена SYDE1 в формировании наследственной предрасположенности к преэклампсии. Мед. генетика. 18(1), 35–38.
Watterson G.A. (1975) On the number of segregating sites in genetical models without recombination. Theor. Popul. Biol. 7(2), 256–276. https://doi.org/10.1016/0040-5809(75)90020-9
Tajima F. (1989) Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics. 123(3), 585–595.
Fu Y.X. (1997) Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection. Genetics. 147 (2), 915–925.
Хедрик Ф. (2003) Генетика популяций. Москва: Техносфера. 592 с.
Raghavan M., Steinrücken M., Harris K., Schiffels S., Rasmussen S., DeGiorgio M., Albrechtsen A., Valdiosera C., Ávila-Arcos M.C., Malaspinas A.S., Eriksson A., Moltke I., Metspalu M., Homburger J.R., Wall J., Cornejo O.E., Moreno-Mayar J.V., Korneliussen T.S., Pierre T., Rasmussen M., Campos P.F., de Barros Damgaard P., Allentoft M.E., Lindo J., Metspalu E., Rodríguez-Varela R., Mansilla J., Henrickson C., Seguin-Orlando A., Malmström H., Stafford T., Jr., Shringarpure S.S., Moreno-Estrada A., Karmin M., Tambets K., Bergström A., Xue Y., Warmuth V., Friend A.D., Singarayer J., Valdes P., Balloux F., Leboreiro I., Vera J.L., Rangel-Villalobos H., Pettener D., Luiselli D., Davis L.G., Heyer E., Zollikofer C.P.E., Ponce de León M.S., Smith C.I., Grimes V., Pike K.A., Deal M., Fuller B.T., Arriaza B., Standen V., Luz M.F., Ricaut F., Guidon N., Osipova L., Voevoda M.I., Posukh O.L., Balanovsky O., Lavryashina M., Bogunov Y., Khusnutdinova E., Gubina M., Balanovska E., Fedorova S., Litvinov S., Malyarchuk B., Derenko M., Mosher M.J., Archer D., Cybulski J., Petzelt B., Mitchell J., Worl R., Norman P.J., Parham P., Kemp B.M., Kivisild T., Tyler-Smith C., Sandhu M.S., Crawford M., Villems R., Smith D.G., Waters M.R., Goebel T., Johnson J.R., Malhi R.S., Jakobsson M., Meltzer D.J., Manica A., Durbin R., Bustamante C.D., Song Y.S., Nielsen R., Willerslev E. (2015) Population genetics. Genomic evidence for the Pleistocene and recent population history of Native Americans. Science. 349(6250), aab3884. https://doi.org/10.1126/science.aab3884
Lapaire O., Grill S., Lalevee S., Kolla V., Hösli I., Hahn S. (2012) Microarray screening for novel preeclampsia biomarker candidates. Fetal. Diagn. Ther. 31(3), 147–153. https://doi.org/10.1159/000337325
Meng T., Chen H., Sun M., Wang H., Zhao G., Wang X. (2012) Identification of differential gene expression profiles in placentas from preeclamptic pregnancies versus normal pregnancies by DNA microarrays. OMICS. 16(6), 301–311. https://doi.org/10.1089/omi.2011.0066
Трифонова Е.А., Габидулина Т.В., Ершов Н.И., Сереброва В.Н., Ворожищева А.Ю., Степанов В.А. (2014) Характеристика транскриптома плацентарной ткани у женщин с физиологической беременностью и преэклампсией. Acta Naturae. 6(2), 77–90.
Nishizawa H., Ota S., Suzuki M., Kato T., Sekiya T., Kurahashi H., Udagawa Y. (2011) Comparative gene expression profiling of placentas from patients with severe pre-eclampsia and unexplained fetal growth restriction. Reprod. Biol. Endocrinol. 9, 107. https://doi.org/10.1186/1477-7827-9-107
Sitras V., Paulssen R.H., Grønaas H., Leirvik J., Hanssen, T.A., Vårtun A., Acharya G. (2009) Differential placental gene expression in severe preeclampsia. Placenta. 30(5), 424–433. https://doi.org/10.1016/j.placenta.2009.01.012
Enquobahrie D.A., Meller M., Rice K., Psaty B.M., Siscovick, D.S., Williams M.A. (2008) Differential placental gene expression in preeclampsia. Am. J. Obstet. Gynecol. 199 (5), 566.e1–566.11. https://doi.org/10.1016/j.ajog.2008.04.020
Reimer T., Koczan D., Gerber B., Richter D., Thie-sen H.J., Friese K. (2002) Microarray analysis of differentially expressed genes in placental tissue of pre-eclampsia: up-regulation of obesity-related genes. Mol. Hum. Reprod. 8 (7), 674–680. https://doi.org/10.1093/molehr/8.7.674
Várkonyi T., Nagy B., Füle T., Tarca A.L., Karászi K., Schönléber J., Hupuczi P., Mihalik N., Kovalszky I., Rigó J., Jr Meiri H., Papp Z., Romero R., Than N.G. (2011) Microarray profiling reveals that placental transcriptomes of early-onset HELLP syndrome and preeclampsia are similar. Placenta. 32. Suppl., S21–S29. https://doi.org/10.1016/j.placenta.2010.04.014
Paiwattananupant K., Phupong V. (2008) Serum inhibin A level in preeclampsia and normotensive pregnancy. Hypertens. Pregnancy. 27(4), 337–343. https://doi.org/10.1080/10641950802020545
Louwen F., Muschol-Steinmetz C., Reinhard J., Reitter A., Yuan J. (2012) A lesson for cancer research: placental microarray gene analysis in preeclampsia. Oncotarget. 3(8), 759–773. https://doi.org/10.18632/oncotarget.595
Matarese G., Moschos S., Mantzoros C.S. (2005) Leptin in immunology. J. Immunol. 174(6), 3137–3142. https://doi.org/10.4049/jimmunol.174.6.3137
Haram K., Mortensen J.H., Nagy B. (2014) Genetic aspects of preeclampsia and the HELLP syndrome. J. Pregnancy. 2014, 910751. https://doi.org/10.1155/2014/910751
Taylor B.D., Ness R.B., Olsen J., Hougaard D.M., Skogstrand K., Roberts J.M., Haggerty C.L. (2015) Serum leptin measured in early pregnancy is higher in women with preeclampsia compared with normotensive pregnant women. Hypertension. 65(3), 594–599. https://doi.org/10.1161/HYPERTENSIONAHA.114.03979
Kalinderis M., Papanikolaou A., Kalinderi K., Vyzantiadis T.A., Ioakimidou A., Tarlatzis B.C. (2015) Serum levels of leptin and IP-10 in preeclampsia compared to controls. Arch. Gynecol. Obstet. 292(2), 343–347. https://doi.org/10.1007/s00404-015-3659-4
Ellen T.P., Ke Q., Zhang P., Costa M. (2008) NDRG1, a growth and cancer related gene: regulation of gene expression and function in normal and disease states. Carcinogenesis. 29(1), 2–8. https://doi.org/10.1093/carcin/bgm200
Chen B., Nelson D.M., Sadovsky Y. (2006) N-myc down-regulated gene 1 modulates the response of term human trophoblasts to hypoxic injury. J. Biol. Chem. 281(5), 2764–2772. https://doi.org/10.1074/jbc.M507330200
Lo H.F., Tsai C.Y., Chen C.P., Wang L.J., Lee Y.S., Chen C.Y., Liang C.T., Cheong M.L., Chen H. (2017) Association of dysfunctional synapse defective 1 (SYDE1) with restricted fetal growth – SYDE1 regulates placental cell migration and invasion. J. Pathol. 241(3), 324–336. https://doi.org/10.1002/path.4835
Szklarczyk D., Morris J.H., Cook H., Kuhn M, Wyder S., Simonovic M., SantosA., Doncheva N.T., Roth A., Bork P., Jensen L.J., von Mering C. (2017) The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible. Nucl. Acids. Res. 45, D362–D368. https://doi.org/10.1093/nar/gkw937
Boyle A.P., Hong E.L., Hariharan M., Cheng Y., Schaub M.A., Kasowski M., Karczewski K.J., Park J., Hitz B.C., Weng S., Cherry J.M., Snyder M. (2012) Annotation of functional variation in personal genomes using RegulomeDB. Genome Res. 22(9), 1790–1797. https://doi.org/10.1101/gr.137323.112
Kim E.S., Cha Y., Ham M., Jung J., Kim S.G., Hwang S., Kleemann R., Moon A. (2014) Inflammatory lipid sphingosine-1-phosphate upregulates C-reactive protein via C/EBPβ and potentiates breast cancer progression. Oncogene. 33(27), 3583–3593. https://doi.org/10.1038/onc.2013.319
Wang W., Taylor R.N., Bagchi I.C., Bagchi M.K. (2012) Regulation of human endometrial stromal proliferation and differentiation by C/EBPβ involves cyclin E-cdk2 and STAT3. Mol. Endocrinol. 26(12), 2016–2030. https://doi.org/10.1210/me.2012-1169
Ramathal C., Wang W., Hunt E., Bagchi I.C., Bagchi M.K. (2011) Transcription factor CCAAT enhancer-binding protein beta (C/EBPbeta) regulates the formation of a unique extracellular matrix that controls uterine stromal differentiation and embryo implantation. J. Biol. Chem. 286(22), 19860–19871. https://doi.org/10.1074/jbc.M110.191759
Radde J., Löning T., Bamberger A.M. (2004) Expression pattern of the CCAAT/enhancer-binding protein C/EBP-beta in gestational trophoblastic disease. Int. J. Gynecol. Pathol. 23(4), 373–377. https://doi.org/10.1097/01.pgp.0000139645.32997.e1
Zhuang B., Luo X., Rao H., Li Q., Shan N., Liu X., Qi H. (2015) Oxidative stress-induced C/EBPβ inhibits β‑catenin signaling molecule involving in the pathology of preeclampsia. Placenta. 36(8), 839–846. https://doi.org/10.1016/j.placenta.2015.06.016
Cassano S., Pucino V., La Rocca C., Procaccini C., De Rosa V., Marone G., Matarese G. (2014) Leptin modulates autophagy in human CD4+CD25- conventional T cells. Metabolism. 63(10), 1272–1279. https://doi.org/10.1016/j.metabol.2014.06.010
Weedon-Fekjær M.S., Sheng Y., Sugulle M., John-sen G.M., Herse F., Redman C.W., Lyle R., Dechend R., Staff A.C. (2014) Placental miR-1301 is dysregulated in early-onset preeclampsia and inversely correlated with maternal circulating leptin. Placenta. 35(9), 709–717. https://doi.org/10.1016/j.placenta.2014.07.002
Tinel M., Berson A., Elkahwaji J., Cresteil T., Beaune P., Pessayre D. (2003) Downregulation of cytochromes P450 in growth-stimulated rat hepatocytes: role of c-Myc induction and impaired C/EBP binding to DNA. J. Hepatol. 39(2), 171–178. https://doi.org/10.1016/s0168-8278(03)00238-1
Wu K.J., Polack A., Dalla-Favera R. (1999) Coordinated regulation of iron-controlling genes, H-ferritin and IRP2, by c-MYC. Science. 283(5402), 676–679. https://doi.org/10.1126/science.283.5402.676
Shimono A., Okuda T., Kondoh H. (1999) N-myc-dependent repression of ndr1, a gene identified by direct subtraction of whole mouse embryo cDNAs between wild type and N-myc mutant. Mech. Dev. 83(1–2), 39–52. https://doi.org/10.1016/s0925-4773(99)00025-8
Zhang M., Muralimanoharan S., Wortman A.C., Mendelson C.R. (2016) Primate-specific miR-515 family members inhibit key genes in human trophoblast differentiation and are upregulated in preeclampsia. Proc. Natl. Acad. Sci. USA. 113(45), E7069–E7076. https://doi.org/10.1073/pnas.1607849113
Kumar P., Luo Y., Tudela C., Alexander J.M., Mendelson C.R. (2013) The c-Myc-regulated microRNA-17~92 (miR-17~92) and miR-106a~363 clusters target hCYP19A1 and hGCM1 to inhibit human trophoblast differentiation. Mol. Cell. Biol. 33(9), 1782–1796. https://doi.org/10.1128/MCB.01228-12
Huggins C.J., Malik R., Lee S., Salotti J., Thomas S., Martin N., Quiñones O.A., Alvord W.G., Olanich M.E., Keller J.R., Johnsona P.F. (2013) C/EBPγ suppresses senescence and inflammatory gene expression by heterodimerizing with C/EBPβ. Mol. Cell. Biol. 33(16), 3242–3258. https://doi.org/10.1128/MCB.01674-12
Vaiman D., Miralles F. (2016) An integrative analysis of preeclampsia based on the construction of an extended composite network featuring protein-protein physical interactions and transcriptional relationships. PLoS One. 11(11), e0165849. https://doi.org/10.1371/journal.pone.0165849
Wu Y., Fu X., Wang L. (2016) Identification of novel biomarkers for preeclampsia on the basis of differential expression network analysis. Exp. Ther. Med. 12(1), 201–207. https://doi.org/10.3892/etm.2016.3261
Liu N., Ding D., Hao W., Yang F., Wu X., Wang M., Xu X., Ju Z., Liu J.P., Song Z., Shay J.W., Guo Y., Cong Y.S. (2016) hTERT promotes tumor angiogenesis by activating VEGF via interactions with the Sp1 transcription factor. Nucl. Acids Res. 44(18), 8693–8703. https://doi.org/10.1093/nar/gkw549
Zhang Y, Yang J, Lv S, Zhao D.Q., Chen Z.J., Li W.P., Zhang C. (2018) Downregulation of decidual SP1 and P300 is associated with severe preeclampsia. J. Mol. Endocrinol. 60(2), 133–143. https://doi.org/10.1530/JME-17-0180
Vaiman D., Calicchio R., Miralles F. (2013) Landscape of transcriptional deregulations in the preeclamptic placenta. PLoS One. 8(6), e65498. https://doi.org/10.1371/journal.pone.0065498
He P., Chen Z., Sun Q., Li Y., Gu H., Ni X. (2014) Reduced expression of 11β-hydroxysteroid dehydrogenase type 2 in preeclamptic placentas is associated with decreased PPARγ but increased PPARα expression. Endocrinology. 155(1), 299–309. https://doi.org/10.1210/en.2013-1350
Lu W., Ma Y.Y., Shao Q.Q., Liang J., Qi T.T., Huang Y., Wang Q.J. (2020) ROS/p53/miR‑335‑5p/Sp1 axis modulates the migration and epithelial to mesenchymal transition of JEG‑3 cells. Mol. Med. Rep. 21(3), 1208–1216. https://doi.org/10.3892/mmr.2019.10901
Gormley M., Ona K., Kapidzic M., Garrido-Gomez T., Zdravkovic T., Fisher S.J. (2017) Preeclampsia: novel insights from global RNA profiling of trophoblast subpopulations. Am. J. Obstet. Gynecol. 217(2), 200.e1–200.e17. https://doi.org/10.1016/j.ajog.2017.03.017
Elliot M.G. (2016) Oxidative stress and the evolutionary origins of preeclampsia. J. Reprod. Immunol. 114, 75–80. https://doi.org/10.1016/j.jri.2016.02.003
Haig D. (1993) Genetic conflicts in human pregnancy. Q. Rev. Biol. 68(4), 495–532. https://doi.org/10.1086/418300
Pijnenborg R., Vercruysse L., Hanssens M. (2008) Fetal-maternal conflict, trophoblast invasion, preeclampsia, and the red queen. Hypertens. Pregnancy. 27(2), 183–196. https://doi.org/10.1080/10641950701826711
Abrams E.T., Rutherford J.N. (2011) Framing postpartum hemorrhage as a consequence of human placental biology: an evolutionary and comparative perspective. Am. Anthropol. 113(3), 417–430. https://doi.org/10.1111/j.1548-1433.2011.01351.x
Cole L.A., Khanlian S.A., Kohorn E.I. (2008) Evolution of the human brain, chorionic gonadotropin and hemochorial implantation of the placenta: insights into origins of pregnancy failures, preeclampsia and choriocarcinoma. J. Reprod. Med. 53(8), 549–557.
Chaline J. (2003) Increased cranial capacity in hominid evolution and preeclampsia. J. Reprod. Immunol. 59(2), 137–152. https://doi.org/10.1016/s0165-0378(03)00043-3
Robillard P.Y., Dekker G.A., Hulsey T.C. (2002) Evolutionary adaptations to pre-eclampsia/eclampsia in humans: low fecundability rate, loss of oestrus, prohibitions of incest and systematic polyandry. Am. J. Reprod. Immunol. 47(2), 104–111. https://doi.org/10.1034/j.1600-0897.2002.1o043.x
Crosley E.J., Elliot M.G., Christians J.K., Crespi B.J. (2013) Placental invasion, preeclampsia risk and adaptive molecular evolution at the origin of the great apes: evidence from genome-wide analyses. Placenta. 34(2), 127–132. https://doi.org/10.1016/j.placenta.2012.12.001
Elliot M.G., Crespi B.J. (2015) Genetic recapitulation of human pre-eclampsia risk during convergent evolution of reduced placental invasiveness in eutherian mammals. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 370(1663), 20140069. https://doi.org/10.1098/rstb.2014.0069
Than N.G., Romero R., Xu Y., Erez O., Xu Z., Bhatti G., Leavitt R., Chung T.H., El-Azzamy H., LaJeunesse C., Wang B., Balogh A., Szalai G., Land S., Dong Z., Hassan S.S., Chaiworapongsa T., Krispin M., Kim C.J., Tarca A.L., Bohn H. (2014) Evolutionary origins of the placental expression of chromosome 19 cluster galectins and their complex dysregulation in preeclampsia. Placenta. 35(11), 855–865. https://doi.org/10.1016/j.placenta.2014.07.015
Zadora J., Singh M., Herse F., Przybyl L., Haase N., Golic M., Yung H.W., Huppertz B., Cartwright J.E., Whitley G., Johnsen G.M., Levi G., Isbruch A., Schulz H., Luft F.C., Müller D.N., Staff A.C., Hurst L.D., Dechend R., Izsvák Z. (2017) Disturbed placental imprinting in preeclampsia leads to altered expression of DLX5, a human-specific early trophoblast marker. Circulation. 136(19), 1824–1839. https://doi.org/10.1161/CIRCULATIONAHA.117.028110
Kaartokallio T., Wang J., Heinonen S., Kajantie E., Kivinen K., Pouta A., Gerdhem P., Jiao H., Kere J., Laivuori H. (2016) Exome sequencing in pooled DNA samples to identify maternal pre-eclampsia risk variants. Sci. Rep. 6, 29085. https://doi.org/10.1038/srep29085
Di Rienzo A., Hudson R.R. (2005) An evolutionary framework for common diseases: the ancestral-susceptibility model. Trends Genet. 21(11), 596–601. https://doi.org/10.1016/j.tig.2005.08.007
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


