Молекулярная биология, 2021, T. 55, № 4, стр. 562-577
Применение олигонуклеотидов, полученных с помощью микрочиповых синтезаторов ДНК, для синтеза генетических конструкций
А. Н. Синяков a, *, В. А. Рябинин a, Е. В. Костина a
a Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
* E-mail: sinyakov@niboch.nsc.ru
Поступила в редакцию 19.11.2020
После доработки 08.02.2021
Принята к публикации 13.02.2021
Аннотация
В обзоре описано использование олигонуклеотидов, полученных с помощью синтезаторов ДНК нового поколения (микрочиповых синтезаторов ДНК), в биологических исследованиях. В настоящее время олигонуклеотиды, полученные на таких синтезаторах, используются преимущественно для конструирования больших библиотек вариантов нуклеотидных последовательностей. Эти библиотеки применяют при изучении сплайсинга, белково-нуклеиновых взаимодействий, транскрипции, трансляции и других процессов в клетках млекопитающих, бактерий и дрожжей. Сочетание методов конструирования библиотек генов из олигонуклеотидов, полученных с помощью микрочиповых синтезаторов ДНК, с развитыми способами комбинирования ДНК-дуплексов позволит получить сложные генетические конструкции, используемые в синтетической биологии.
ВВЕДЕНИЕ
Эффективную, но трудоемкую технологию рекомбинантных ДНК, развитую в 70-х годах предыдущего столетия, используют в биологических и медицинских исследованиях и в настоящее время. Эта технология, основанная на комбинировании in vitro фрагментов ДНК (небольших искусственных ДНК-дуплексов, ПЦР-фрагментов и природных генетических элементов, таких как плазмидная и геномная ДНК), позволяет осуществлять перенос генетического материала (ДНК) из одного организма в другой. Фактически технология рекомбинантных ДНК только в небольшой степени модифицирует уже существующую ДНК, как правило, бактериальные плазмиды или вирусные геномы. Технология имеет значительные ограничения, поскольку позволяет ввести только определенное число изменений в исходные генетические элементы.
Альтернативу технологии рекомбинантных ДНК при получении генетического материала in vitro представляет de novo химический синтез ДНК. При этом весь генетический материал получают из относительно простых и доступных реактивов. Химический синтез олигонуклеотидов с последующей их сборкой в требуемые ДНК-дуплексы значительно упрощает получение различных протяженных ДНК по сравнению с модификацией уже существующих природных фрагментов ДНК.
В 1970 годах с помощью химического синтеза можно было получать относительно короткие фрагменты ДНК–олигонуклеотиды длиной менее 20 нуклеотидов. В 1970–1990 годы совершенствовались методы синтеза олигонуклеотидов и их сборки в генетические конструкции, что позволило собирать искусственные гены. Следующим этапом стал синтез протяженных фрагментов ДНК (до 5000 нуклеотидов). Сейчас синтез олигонуклеотидов с последующей их сборкой в гены, а в ряде случаев в вирусные геномы, рядовое событие, поскольку автоматические синтезаторы ДНК стали коммерчески доступными.
Хотя разработано множество вариантов автоматических синтезаторов ДНК, однако прогресс в этой области не останавливается, что свидетельствует о важности этого направления. В конечном итоге именно успехи в олигонуклеотидном синтезе и привели к появлению новой инженерной науки – синтетической биологии.
Основным отличием синтетической биологии от генетической инженерии является то, что целевые генетические конструкции, получаемые в рамках этого метода, состоят исключительно из синтетических олигонуклеотидов. Синтетическая биология – новейшее направление науки на стыке информатики, электроники, химии и биологии, которое объединяет передовые области исследований с целью проектирования и синтеза новых живых систем, в том числе никогда не существовавших в природе. Эта наука представляет собой инженерный инструментарий для проектирования функциональных и управляемых живых систем с заданными свойствами [1, 2]. Для реализации проектов, связанных с синтетической биологией, необходим очень большой массив синтетических олигонуклеотидов.
Прогресс в синтетической биологии, по-видимому, будет связан с олигонуклеотидными микрочиповыми синтезаторами, значительно более производительными, чем традиционные колоночные или планшетные.
АВТОМАТИЧЕСКИЕ СИНТЕЗАТОРЫ ДНК
Работа автоматических синтезаторов ДНК основана на твердофазном олигонуклеотидном синтезе. Нерастворимый носитель с присоединенным первым звеном олигонуклеотидной цепи помещают в колонку или лунку планшета синтезатора и последовательно обрабатывают реагентами и растворителями. На первой стадии с помощью кислоты удаляют временную 4,4′-диметокситритильную защитную группу с первого звена, на следующей – присоединяют второй нуклеотид, затем блокируют непрореагировавшие гидроксильные группы и окисляют трехвалентный фосфор в пятивалентный. Цикл наращивания цепи повторяют до тех пор, пока целевой олигонуклеотид не будет синтезирован. Такая схема синтеза реализована во множестве различных синтезаторов ДНК (традиционные синтезаторы ДНК). В традиционных синтезаторах ДНК необходимо подводить реагенты по выделенным каналам к колонкам или лункам планшета, в которых на поверхности твердого носителя происходит синтез индивидуальных олигонуклеотидов. В таких синтезаторах каждый олигонуклеотид синтезируется и выделяется индивидуально. Чем больше олигонуклеотидов синтезируется одновременно, тем сложнее схема коммуникаций синтезатора. Поэтому, как правило, в работе используются 8–16-колоночные синтезаторы или 96–384-луночные планшетные синтезаторы ДНК. Для задач синтетической биологии такая производительность мала. Так, полный геном человека 96-луночный планшетный ДНК-синтезатор сможет синтезировать, работая каждый день в три смены, только за ~1100 лет.
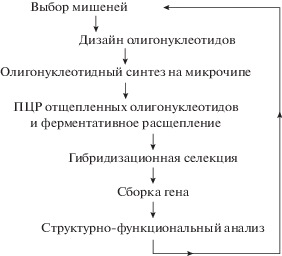
В начале 90-х годов прошлого века фирма “Affymetrix” разработала твердофазный метод синтеза олигонуклеотидов с использованием фотолабильных соединений (рис. 1), что в конечном итоге привело к созданию индустрии ДНК-микрочипов [3, 4].
Рис. 1.
Схема олигонуклеотидного синтеза на микрочипе с использованием фотолабильных защитных групп.
На поверхность слайда ковалентно присоединяют спейсер, несущий фотолабильную группу. Через физическую маску (фотошаблон, изготовленный из пластины с прорезями для засвечивания слайда УФ-излучением) происходит засветка участков поверхности – спотов, на которых синтезируются олигонуклеотиды одной структуры. В этих местах происходит удаление фотолабильной защитной группы. Затем всю поверхность слайда обрабатывают соответствующим амидофосфитом, несущим фотолабильную группу. Присоединение амидофосфита происходит только в местах деблокирования фотолабильной группы. На следующих стадиях происходит облучение других спотов и присоединение в этих местах другого амидофосфита (рис. 1). Циклы наращивания цепи повторяются до тех пор, пока не будет завершен синтез последнего олигонуклеотида. Фактически при синтезе олигонуклеотидов используется традиционный амидофосфитный метод [5] с заменой кислотолабильной 4,4′-диметокситритильной группы на фотолабильную защитную.
Вначале в “Affymetrix” использовали фотолитографию с физическими масками для деблокирования временных фотолабильных защитных групп. В настоящее время физические фотомаски в приборах заменены на виртуальные, значительно упрощающие процесс синтеза [6, 7]. Фотошаблоны в этом случае создаются с помощью платы управляемых микрозеркал.
На одном слайде “Affymetrix” можно синтезировать до 1.3 млн спотов, содержащих 25-членные олигонуклеотиды [8]. Громадное увеличение производительности синтеза произошло из-за отказа от индивидуального синтеза олигонуклеотидов и, как следствие, от множества коммуникаций. Фактически в чипе есть только вход и выход, по которым подаются и выводятся реактивы. Управление синтезом олигонуклеотидов происходит с помощью УФ-излучения.
К настоящему времени появились и другие фирмы, сконструировавшие микрочиповые синтезаторы ДНК.
Микрочиповый реактор фирмы “Febit Gmbh” состоял из восьми независимых фрагментов, на каждом из которых можно было проводить одновременный синтез до 15 000 разных олигонуклеотидов (всего 120 000 олигонуклеотидов). Способ проведения олигонуклеотидного синтеза был аналогичен способу фирмы “Affymetrix” за одним исключением, олигонуклеотидный синтез проводили в капиллярах, а не на поверхности плоского слайда [9]. В течение суток на одном реакторе можно было получить до 480000 олигонуклеотидов. В настоящее время “Febit Gmbh” отсутствует на рынке.
Указанные фирмы используют в своих синтезаторах дорогие амидофосфиты, содержащие фотолабильную группу.
С целью снижения операционных расходов созданы микрочиповые синтезаторы, использующие стандартные амидофосфиты с 4,4′-диметокситритильной группой. В этих синтезаторах деблокирование осуществляется с помощью кислоты, получаемой путем электролиза либо генерируемой из фотокислот УФ-облучением. В обоих типах таких синтезаторов поверхность слайда пришлось разбивать на ячейки для синтеза индивидуальных олигонуклеотидов. Они необходимы для удержания кислоты от диффузии на участки слайда, где не должно проходить деблокирование. Из-за необходимости создания ячеек на слайде число одновременно синтезируемых олигонуклеотидов в этих синтезаторах значительно меньше, чем в синтезаторах, использующих фотолабильные амидиты.
Биочиповый реактор microfluidic µParaflo, разработанный сотрудниками фирмы “LC Sciences” [7], использует стандартные реагенты для олигонуклеотидного синтеза и позволяет одновременно синтезировать 4000–8000 разных олигонуклеотидов. Синтез осуществляется в индивидуальных ячейках, отделенных друг от друга. Деблокирование кислотолабильной 4,4′-диметокситритильной группы происходит при разложении фотокислоты под действием УФ-излучения. Фотокислота закачивается одновременно во все ячейки, но освещается только та их часть, в которой должно проходить наращивание олигонуклеотидной цепи. Для проведения синтеза используются фотокислоты, строение которых неизвестно.
“CombiMatrix Corporation” разработан микрочиповый синтезатор, в котором используется поле управляемых электродов [10]. Поверхность чипа разбита на ячейки, в которых осуществляется синтез и находится пара электродов. Для олигонуклеотидного синтеза используется стандартная схема и реагенты амидофосфитного олигонуклеотидного синтеза. Кислота, необходимая для деблокирования временной диметокситритильной группы, генерируется в микроячейке с помощью электролиза. Кассеты для синтеза содержат 8, 12 или 90 тысяч ячеек.
Синтезатор фирмы “Agilent Technologies” способен синтезировать до 1 млн олигонуклеотидов на одном слайде [11, 12]. Синтезатор использует стандартные амидофосфиты и традиционную схему олигонуклеотидного синтеза с временной кислотолабильной 4,4′-диметокситритильной защитной группой. Основное отличие этого синтезатора состоит в использовании для доставки реагентов в споты микрочипа устройства, применяемого в струйных принтерах. Длина синтезируемых олигонуклеотидов достигает 200 нуклеотидных звеньев.
Олигонуклеотиды, полученные с помощью микрочиповых синтезаторов, на несколько порядков дешевле, чем олигонуклеотиды, полученные на традиционных синтезаторах (цена за нуклеотид варьирует в зависимости от длины, масштаба и используемого синтезатора). Микрочиповые синтезаторы позволяют получать олигонуклеотиды в очень широких пределах: от десятков до миллионов олигонуклеотидов с одного микрочипа.
Чрезвычайно широкий ассортимент олигонуклеотидов, получаемых на микрочиповых синтезаторах ДНК, наряду с дешевизной, побудили применить их в генно-инженерных работах.
Ниже рассмотрены примеры применения таких олигонуклеотидов для создания библиотек генетических элементов и методы, позволяющие использовать их смеси для конструирования генов и более протяженных фрагментов геномной ДНК.
КОНСТРУИРОВАНИЕ БИБЛИОТЕК ИЗ ОЛИГОНУКЛЕОТИДОВ, ПОЛУЧЕННЫХ С ПОМОЩЬЮ МИКРОЧИПОВЫХ СИНТЕЗАТОРОВ ДНК
В настоящее время одним из основных направлений использования олигонуклеотидов, полученных на микрочиповых синтезаторах, является конструирование больших библиотек регуляторных элементов для оптимизации экспрессии генов. Изучению структурных и функциональных характеристик тысяч цис-регуляторных элементов, отвечающих за транскрипционные, трансляционные и другие процессы в клетках млекопитающих, бактерий и дрожжей, посвящено большое количество работ.
С помощью пула олигонуклеотидов, синтезированных на микрочиповом синтезаторе, получена библиотека векторов, экспрессирующих короткие РНК, образующие шпильки (shРНК), предшественники малых интерферирующих РНК (siРНК) [13]. Всего проанализировано 32 000 генов мыши и человека и выбрано 382 982 последовательности, гомологичных исследуемым генам.
Синтезированные на микрочипе олигонуклеотиды отделяли от слайда, деблокировали и амплифицировали. Полученные ампликоны клонировали в целевом векторе. С помощью этого подхода получена библиотека, продуцирующая 53 478 различных shРНК. Выход целевых векторов, экспрессирующих shРНК, составил ~14%, что связано, по-видимому, с отсутствием стадии очистки олигонуклеотидов от мутированных последовательностей. Во многих работах по генетическому конструированию эта стадия стала обязательной.
Сконструирована библиотека векторов, экспрессирующих shРНК к 14 500 генам человека и 12 700 генам мыши [14].
Для проектирования искусственных генных сетей очень важно иметь хорошо охарактеризованные регуляторные элементы, функционирующие в различных микроорганизмах.
Получена библиотека, содержащая 29 249 регуляторных элементов (промотор, сайт связывания рибосомы, сайт старта транскрипции) длиной в среднем 165 п.н., относящихся к 184 прокариотическим геномам. Эту библиотеку использовали для изучения уровня транскрипции и трансляции генов в различных бактериях [15].
Подобную работу провели Church и сотрудники [16], которые получили библиотеку из 114 промоторов и 111 сайтов связывания рибосомы Escherichia coli, содержащую 12 563 элемента, и использовали ее для оценки уровней транскрипции и экспрессии генов.
С целью изучения структурно-функциональной организации бактериальных промоторов фагов T3, T7 и SP6 из олигонуклеотидов длиной 200 п.н. синтезировали 12 972 варианта этих промоторов [17].
При изучении влияния структуры сайтов связывания факторов транскрипции на силу синтетических промоторов использовали набор из 52 429 олигонуклеотидов длиной 100 нуклеотидных звеньев [18].
В лаборатории Kosuri [19] создана библиотека, содержавшая 10 898 промоторов σ70, которую использовали для изучения влияния структуры –10, –35, UP и спейсерных элементов на эффективность экспрессии генов.
Для изучения структурно-функциональных свойств промоторов из набора 150-членных олигонуклеотидов была сконструирована библиотека, включающая 6500 различных промоторов [20].
Библиотеку из ~27 000 элементов, полученную из пула 142-членных олигонуклеотидов, использовали для изучения транскрипционных регуляторных элементов в клетках человека [21]. Библиотека была получена как комбинация вариантов двух энхансеров. В развитие предложенного метода изучения энхансеров генов человека из олигонуклеотидов, полученных с помощью микрочипового синтезатора, сконструирована библиотека, содержащая 54 000 элементов потенциальных энхансеров [22].
С помощью 150-членных олигонуклеотидов в работе [23] получено 1040 энхансеров гена родопсина мыши. Для создания библиотеки из 1300 энхансеров использовали 150-членные олигонуклеотиды [24].
Библиотека из ~5000 последовательностей, содержащих различные сайты связывания 12 транскрипционных факторов, получена с помощью 200-членных олигонуклеотидов [25].
Из олигонуклеотидов, полученных с помощью микрочипового синтезатора ДНК, синтезировали библиотеку для транскрипции 127 рибопереключателей [26].
С помощью синтетических библиотек, полученных из микрочиповых олигонуклеотидов, можно упростить и интенсифицировать проведение работ в области эпигенетики, сплайсинга, мутагенеза, оптимизации гетерологичной экспрессии генов и изучения белково-нуклеиновых взаимодействий.
Библиотеку из 27 733 вариантов 2198 экзонов использовали для изучения механизма сплайсинга [27].
В экспериментах по определению специфичности нуклеиново-белковых взаимодействий в нуклеосомах использовали 40 000 ДНК-дуплексов, полученных из 150-членных олигонуклеотидов [28].
Использование библиотеки, содержащей ~10 000 181-членных олигонуклеотидов, полученных путем микрочипового олигонуклеотидного синтеза, позволило ввести с помощью технологии CRISPR/Cas9 множественные мутации в состав 1034 генов Saccharomyces cerevisiae с целью выявления генов, влияющих на жизнеспособность этого микроорганизма [29].
С помощью олигонуклеотидов, полученных на микрочиповых синтезаторах, создана библиотека клонов (более 18 000), экспрессирующих гидовые РНК (gРНК) системы CRISPR/Cas9, для 1117 генов дрожжей [30].
Библиотеку из 13 000 130-членных олигонуклеотидов использовали для проведения множественного мутагенеза в бактериальных геномах [31].
С помощью олигонуклеотидов, полученных на микрочиповых синтезаторах ДНК, создана библиотека, содержащая 7800 из 7828 возможных мутантных форм гена β2-адренергического рецептора (ADRB2) [32].
Сконструирована библиотека фрагментов ДНК, содержащая 14 234 различные комбинации промоторов, сайты связывания рибосомы и 11 N-концевых кодонов 5′-области гена [33]. Использование редких N-концевых кодонов позволило значительно повысить уровень его экспрессии (до 14 раз). Предложенный подход позволяет оптимизировать экспрессию генов в штаммах-продуцентах [34].
С помощью олигонуклеотидов, синтезированных на микрочиповых синтезаторах ДНК, получена библиотека [35] из 17 406 цис-регуляторных элементов, изучено влияние их структуры на уровень экспрессии генов.
ГЕНЕТИЧЕСКОЕ КОНСТРУИРОВАНИЕ ИЗ ОЛИГОНУКЛЕОТИДОВ, ПОЛУЧЕННЫХ НА ТРАДИЦИОННЫХ КОЛОНОЧНЫХ ИЛИ ПЛАНШЕТНЫХ СИНТЕЗАТОРАХ
Как отмечено выше, олигонуклеотиды, полученные на традиционных синтезаторах, выделяют индивидуально. Каждый из них можно очистить с помощью ВЭЖХ или гель-электрофореза. Количество каждого олигонуклеотида, получаемого при синтезе, на несколько порядков превосходит потребности генетического конструирования. При этом всегда существует возможность замены части олигонуклеотидов, если встретятся проблемы при их сборке в целевую конструкцию, путем дополнительного синтеза.
Однако число олигонуклеотидов в наборах для конструирования генов, полученных с помощью традиционных синтезаторов, ограничено и варьирует от 5 до 50, а длина синтезируемых ДНК-фрагментов колеблется от 200 до 3000 п.н.
Первые искусственные гены были получены группой Gobind Khorana [36, 37] из коротких фосфорилированных по 5′-концу олигонуклеотидов (6–8 нуклеотидов).
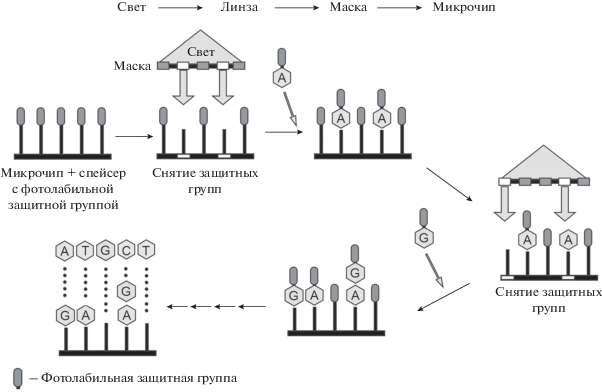
Олигонуклеотиды отжигали в реакционной смеси, формировали дуплекс, в котором происходила сшивка смысловой и антисмысловой цепей (рис. 2а). Процедуру сшивки верхних и нижних цепей повторяли с другими дуплексами до полного завершения синтеза целевого гена.
Рис. 2.
Получение фрагментов ДНК методом Khorana (а) и с помощью полимеразной циклической сборки (б).
С появлением термостабильных ДНК-лигаз и ДНК-полимераз, развитием олигонуклеотидного синтеза, позволяющего получать протяженные олигонуклеотиды (~40 п.н.), формирующие более стабильные ДНК-дуплексы, стало возможным собирать гены в один прием [38, 39]. С этой целью применяют, в частности, полимеразную циклическую сборку (PCA, Polymerase cycling assembly) (рис. 2б), которая используется для удлинения перекрывающихся олигонуклеотидов в двухцепочечный фрагмент при термоциклировании [40]. На финальной стадии в методах, использующих лигирование и PCA, для получения полноразмерных продуктов из частично собранных конструкций применяют ПЦР. Недавно Gibson с сотрудниками разработали как in vivo [41, 42], так и in vitro [43, 44] одностадийный протокол для сборки и клонирования ДНК-дуплексов непосредственно в плазмиду. Все эти протоколы, лежащие в основе различных вариантов синтеза генов, многократно улучшались (обзоры [45–48]). Поскольку синтез и сборка не происходят безошибочно, гены клонируют и подтверждают их структуру секвенированием.
У каждого подхода есть свои достоинства и недостатки. Методы, основанные на лигировании дуплексов, приводят к пониженному числу ошибок, поскольку неправильные дуплексы “сшиваются” с меньшей эффективностью. Но в этом случае больше времени требуется для синтеза олигонуклеотидов (необходимо синтезировать как смысловую, так и антисмысловую цепи, фосфорилировать 5′-концы олигонуклеотидов, в результате чего повысится стоимость исходных олигонуклеотидов). В подходах, основанных на использовании PCA, используют частичное перекрывание олигонуклеотидов (15–25 п.н.), что снижает необходимый объем синтеза, однако метод страдает от повышенного числа ошибок, связанного с отсутствием отбора неправильных дуплексов на части целевой последовательности. В процессе PCA-конструирования формируются одноцепочечные районы, поэтому такие районы можно использовать для получения измененных форм целевой последовательности. Поскольку на финальной стадии используется амплификация частично комплементарных фрагментов, длина генетических структур, полученных этими методами, обычно не превышает 5000 п.н.
Следует отметить, что получение генетических структур запланированной последовательности сильно зависит от чистоты исходных олигонуклеотидов. По статистике при конструировании генов из 50-членных очищенных олигонуклеотидов, полученных с выходом 99.9% на стадию, только одна из 10 последовательностей длиной 1000 п.н. имеет правильную структуру, остальные содержат различные “мутации”. Число ошибок растет экспоненциально с ростом длины конструируемой последовательности, поэтому одновременная сборка большого количества олигонуклеотидов непрактична. Гетерогенность продуктов сборки олигонуклеотидов приводит к необходимости клонирования, выделения и секвенирования ДНК отдельных клонов при поиске структуры, не содержащей ошибок [49]. Это значительно удорожает и удлиняет процедуру генетического конструирования. A priori дополнительная очистка исходных олигонуклеотидов должна упростить и удешевить процесс генетического конструирования. Ниже приведены примеры конструирования протяженных ДНК из олигонуклеотидов традиционных синтезаторов.
Синтез генетических конструкций начался с работ Gobind Khorana [36, 37] по конструированию искусственных генов аланиновой тРНК (72 п.н.) и супрессорной тирозиновой тРНК (126 п.н.). Олигонуклеотиды синтезировали без применения автоматических синтезаторов. Работа по получению каждого из генов заняла ~ 8 лет.
С появлением автоматических синтезаторов работы по конструированию генов значительно интенсифицировались, и исследователи смогли перейти к синтезу in vitro полных геномов микроорганизмов. Первым получили геном полиовируса в виде его кДНК-копии (7500 п.н.) [50]. Работа по конструированию заняла 3 года. Ventor и сотрудники синтезировали геном бактериофага φX174 (5386 п.н.) [51], а Evans и сотрудники – геном вируса оспы лошадей (212 633 п.н.) [52].
К настоящему времени синтезировано несколько бактериальных геномов. Группа Ventor потратила ~15 лет и 40 млн долларов на создание первых искусственных геномов бактерий Mycoplasma genitalium (582970 п.н.) и M. mycoides (1 080 000 п.н.) [53, 54].
Консорциум, состоящий из 21 института, осуществил in vitro синтез генома пресноводной бактерии Caulobacter ethensis-2.0 (78 5701 п.н) [55].
Как видно из приведенных выше примеров, конструирование протяженных геномов занимает много времени. Поэтому получение геномов, более сложных, чем бактериальные, например генома человека, требует среди прочего более производительных ДНК-синтезаторов. Микрочиповые синтезаторы представляются вполне пригодными для этой цели. Однако для их успешного применения необходима модификация методов очистки олигонуклеотидов, полученных на этих синтезаторах, и методов их сборки в целевые последовательности ДНК.
СИНТЕЗ ГЕНЕТИЧЕСКИХ СТРУКТУР ИЗ ПРЕДВАРИТЕЛЬНО СЕКВЕНИРОВАННЫХ ОЛИГОНУКЛЕОТИДОВ
Микрочиповые синтезаторы способны очень быстро синтезировать десятки тысяч и даже миллионы олигонуклеотидов заданной структуры. В отличие от традиционных синтезаторов все олигонуклеотиды, полученные с помощью микрочиповых синтезаторов, находятся в одной реакционной смеси (олигонуклеотидном пуле). Кроме того, концентрация олигонуклеотидов пула, как правило, недостаточна для проведения генно-инженерных работ, а их качество ниже из-за большего процента ошибок в структуре ДНК. Как отмечено выше, успех генетического конструирования очень сильно зависит от чистоты олигонуклеотидов. Очистить олигонуклеотиды, полученные на микрочипе, традиционными методами невозможно.
Методы высокопроизводительного секвенирования (NGS) позволяют отбирать из олигонуклеотидного пула последовательности, не содержащие ошибок в своей структуре. В процессе NGS-секвенирования происходит увеличение количества целевых олигонуклеотидов вследствие их амплификации. Таким образом, применение процедуры NGS-секвенирования позволяет получить олигонуклеотиды правильной структуры, пригодные для конструирования протяженных ДНК, в количествах, необходимых для проведения генно-инженерных работ.
Секвенатор GS FLX Roche 454 использовали для NGS-секвенирования олигонуклеотидов, полученных на микрочиповых или традиционных синтезаторах [56–58]. Микрошарики, содержащие на свой поверхности олигонуклеотиды целевой структуры, доставали из платы секвенатора с помощью роботизированного микроманипулятора. Олигонуклеотиды отщепляли от поверхности микрошариков, амплифицировали, с помощью эндонуклеаз рестрикции LguI/SapI удаляли адапторы для секвенирования и использовали для генетического конструирования. Предполагается, что такой подход позволяет снизить уровень ошибок в целевых конструкциях в 500 раз.
Метод, в котором применяли специально сконструированную систему с использованием пульсирующего лазера для отбора микрошариков с олигонуклеотидами правильной структуры с секвенатора NGS GS Junior, получил название “снайперское клонирование”. Этот метод позволяет получить за неделю 5188 олигонуклеотидов целевой структуры из одного олигонуклеотидного пула. [59, 60]
В рассмотренных методах применяют механические способы отбора клонов целевых олигонуклеотидов. В качестве альтернативы создан метод отбора совершенных олигонуклеотидов с помощью баркодированных праймеров.
Для фракционирования олигонуклеотидного пула используют клонирование в дрожжевом векторе с последующим баркодированием и NGS-секвенированием на секвенаторе Illumina [61].
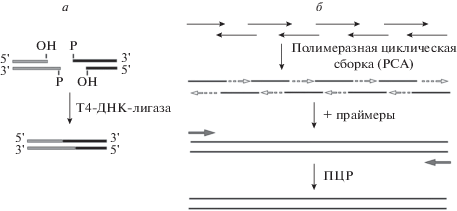
Разработан метод “дробовика”, использующего баркодированные олигонуклеотиды [62, 63] (рис. 3).
Рис. 3.
Общая схема синтеза ДНК с помощью метода “дробовика”. Синтез олигонуклеотидов на микрочипе (а), частичное расщепление рестриктазой типа IIS (б), присоединение баркодов и селективная ПЦР (с), сборка кластеров генов (д).
В работе использовали 228 олигонуклеотидов длиной 150 нуклеотидов, которые содержали две фланкирующие последовательности (2 × 25 нуклеотидов), включающие EarI- или BtsI-сайты и конструкционную часть (100 нуклеотидов). Олигонуклеотиды были синтезированы на микрочипе, отщеплены от поверхности и амплифицированы (рис. 3а). Реакционную смесь обрабатывали рестриктазами EarI и BtsI, частично отщепляя праймеры для амплификации. На следующей стадии осуществляли попарную сборку фрагментов, получая конструкции с 200 п.н., и амплификацию продуктов с помощью оставшихся праймеров (см. рис. 3б). Затем к ДНК-дуплексам присоединяли баркодированные праймеры, используемые для секвенирования и амплификации (рис. 3в). Не содержащие ошибок фрагменты после амплификации с баркодированными праймерами и выделения конструкционной части успешно использовали для получения 11 т.п.н. последовательности кластера генов биосинтетического пенициллина. Уровень ошибок при получении фрагментов кластера составлял 1/4600 п.н.
Подобный метод, за исключением промежуточной сборки 200-членных нуклеотидных дуплексов, использовали для конструирования 15 генов E. coli K12 [64].
СИНТЕЗ ГЕНЕТИЧЕСКИХ СТРУКТУР ИЗ ПУЛОВ ОЛИГОНУКЛЕОТИДОВ, ПОЛУЧЕННЫХ НА МИКРОЧИПОВЫХ СИНТЕЗАТОРАХ ДНК
При применении олигонуклеотидного пула для генетического конструирования необходимо преодолеть ряд затруднений, связанных с тем что:
1. уровень ошибок в олигонуклеотидах, полученных на микрочипе, выше, чем у олигонуклеотидов, синтезируемых традиционным методом;
2. сборка фрагментов генов становится затруднительной в смесях, содержащих значительное количество “посторонних” олигонуклеотидов;
3. возможность кросс-гибридизации накладывает строгие ограничения на структуру синтезируемых на микрочипе олигонуклеотидов;
4. мольные количества олигонуклеотидов, находящихся в пуле, недостаточны для проведения ферментативных реакций.
Синтез на микрочипе идет на очень малой поверхности (1–1.5 см2), поэтому обычно весь пул олигонуклеотидов, полученный с одного микрочипа, содержит 1–20 пмоль смеси олигонуклеотидов. Это означает, что содержание каждого отдельного олигонуклеотида колеблется в пределах от нескольких амоль до нескольких фмоль, что недостаточно для проведения ферментативных реакций в стандартных условиях (1 пмоль).
“Combimatrix Corporation”, по-видимому, первой попыталась использовать олигонуклеотиды, полученные с помощью микрочиповых синтезаторов для сборки генов [65].
Первыми трудности, связанные с использованием олигонуклеотидных пулов при генетическом конструировании протяженных последовательностей ДНК, были успешно преодолены Tian и сотрудниками [66–68]. Для повышения концентрации целевых олигонуклеотидов они предложили амплифицировать синтезированные олигонуклеотиды с 105 (или 109 в случае микрочипов низкой плотности) молекул до 109 (или 1012 в случае микрочипов низкой плотности), что позволяет провести стадии отбора олигонуклеотидов правильной структуры и их сборки в целевую последовательность.
Общая схема генетического конструирования приведена на рис. 4. Олигонуклеотиды синтезировали с использованием микрочипового синтезатора Atactic/Xeotron 4K, содержащего 3968 реакционных ячеек. Выход целевого олигонуклеотида с каждой ячейки не превышал 5 фмоль. Каждый олигонуклеотид содержал последовательности сайтов для амплификации и сайты эндонуклеаз рестрикции типа IIS для удаления амплификационных праймеров. Схожая стратегия конструирования структур олигонуклеотидов, содержащих сайты MluI-рестриктазы, использована в [69, 70]. В результате амплификации количество каждого олигонуклеотида увеличивалось в миллион раз.
Как уже сказано, в структуре синтезируемых олигонуклеотидов в процессе их получения неизбежно возникают ошибки, поэтому очень важно провести предварительное выделение олигонуклеотидов правильной структуры. Это значительно упростит поиск целевых генетических структур после сборки олигонуклеотидов. Для очистки олигонуклеотидов использовали гибридизацию с короткими олигонуклеотидами [66–68]. Олигонуклеотиды после амплификации обрабатывали рестриктазами IIS-типа, отщепляющими праймерные участки, полученные смеси последовательно гибридизовали с двумя олигонуклеотидными зондами. Целевой олигонуклеотид, необходимый для генетического конструирования, очищали на двух олигонуклеотидах, комплементарных каждой его половине.
Олигонуклеотиды, предназначенные для гибридизации, также синтезировали на микрочипе, амплифицировали и иммобилизовывали на магнитные частицы. При сборке генов использовали одностадийную полимеразную сборку. Оценивали эффективность проведенной очистки олигонуклеотидов.
Как видно из табл. 1, очистка олигонуклеотидов путем гибридизации перед “сшивкой” снижает уровень ошибок в целевой конструкции в 8.7 раза по сравнению с неочищенными олигонуклеотидами. Всего из олигонуклеотидного пула [66–68] получен 21 ген, кодирующий варианты малой рибосомной субъединицы E. coli. В работе проведен компьютерный расчет структуры целевых генов с целью элиминирования неправильного спаривания олигонуклеотидов.
Таблица 1.
Уровень ошибок при конструировании генов из олигонуклеотидных пулов
| Метод очистки | Всего, п.н. | Замены | Трансверсии | Делеции | Вставки | П.н./одна ошибка |
|---|---|---|---|---|---|---|
| Гибридизация | 23 641 | 7 | 3 | 5 | 2 | 1394 |
| ПААГ | 24 546 | 28 | 12 | 11 | 3 | 455 |
| Без очистки (РАМ) | 9243 | 25 | 13 | 19 | 1 | 159 |
| Без очистки (лигирование) | 6093 | 6 | 6 | 22 | 4 | 160 |
Аналогичный подход к синтезу генов из олигонуклеотидных пулов использован в работах [71, 72]. Как правило, одномоментно собирали фрагменты ДНК протяженностью 300–700 п.н. (в редких случаях 1–2 т.п.н.). Для упрощения синтеза протяженных генов предложено разбивать такие последовательности на фрагменты длиной не более 500 п.н. [73]. Таким способом были успешно синтезированы гены ДНК-полимераз Dpo4 (1056 п.н.) и Pfu (2325 п.н.).
Очистка олигонуклеотидов пула с помощью гибридизации существенным образом сказалась на снижении уровня ошибок в структуре целевого гена. При этом гибридизационная селекция олигонуклеотидов, не содержащих ошибок в последовательности, требовала проведения отдельной стадии.
Боровков и соавт. [74] интегрировали процесс отбора олигонуклеотидов правильной структуры в саму стадию сборки целевой последовательности. Они применяли лигазную сборку с использованием термостабильной Taq-ДНК-лигазы (LCR, ligase chain reaction, лигазная цепная реакция). Это позволило упростить и удешевить процесс конструирования генетического материала, поскольку в этом случае использовали неочищенные пулы олигонуклеотидов. Лигирование при повышенной температуре позволяет “отсеять” олигонуклеотиды с ошибками в структуре, которые не образуют стабильных дуплексов при этой температуре, и тем самым значительно снизить число ошибок в целевой конструкции.
Метод включал следующие стадии.
1. Все гены набора объединяли в виртуальную последовательность, которую разбивали на нормализованные по температуре олигонуклеотиды (55 ± 1°C). Олигонуклеотиды выбирали с помощью специальной компьютерной программы, упрощающей процесс сборки путем разрушения A-T- и G-C-богатых районов последовательности, прямых и обратных повторов, микросателлитов и других нежелательных ДНК-фрагментов.
2. Синтезировали олигонуклеотиды с фосфатной группой на 5′-конце и выделяли в виде олигонуклеотидного пула.
3. Олигонуклеотидный пул превращали в смесь частично перекрывающихся блоков унифицированного размера. Каждый блок состоял из фрагмента гена и фланкирующих участков (UPR), содержащих сайт для расщепления рестриктазой MlyI. Блоки собирали лигированием в тщательно контролируемых условиях, предотвращающих внедрение в их состав олигонуклеотидов с ошибками. Присутствие в лигазной смеси PEG6000 значительно ускоряло процесс сборки в условиях очень низкой концентрации олигонуклеотидов. Собранные блоки амплифицировали (10 циклов PCA), чтобы получить достаточное количество материала для ПЦР всех запланированных индивидуальных генов.
4. После удаления фланкированных районов частично перекрывающиеся блоки собирали в целевые гены с помощью ПЦР, каждый ген со своей парой специфических праймеров.
Экспериментально установлено, что эффективную сборку генов можно проводить при общем количестве олигонуклеотидов в пуле не более 4000 и размере блока 270 п.н. Уровень ошибок составлял 2.7 на 1 т.п.н.
Использование LCR снижало уровень ошибок в целевых конструкциях в 4 раза (с 6.3/1 до 1.6/1 т.п.н.) [71]. Таким образом, по уровню ошибок неочищенные олигонуклеотиды, полученные с помощью микрочиповых синтезаторов, в LCR сравнимы с олигонуклеотидами, синтезированными на традиционных синтезаторах, в PCA.
С помощью метода, предложенного Боровковым и соавт. [74], в котором используется термостабильная лигаза, получены 85 вариантов гена GFP, длиной 800 п.н. каждый [75], и библиотека для транскрипции 127 рибопереключателей (суммарная длина синтезированной последовательности – 40 т.п.н.) [26].
СИНТЕЗ ГЕНЕТИЧЕСКИХ СТРУКТУР ИЗ ПОДПУЛОВ ОЛИГОНУКЛЕОТИДОВ, ПОЛУЧЕННЫХ НА МИКРОЧИПОВЫХ СИНТЕЗАТОРАХ ДНК
В упомянутых выше методах протяженные фрагменты ДНК конструировали с использованием от нескольких десятков до нескольких тысяч олигонуклеотидов. Получение отдельных генов с использованием пулов, содержащих большое количество олигонуклеотидов (больше 1000), становится трудной задачей в основном из-за кросс-гибридизации [76].
Решить эту проблему можно с помощью двух подходов, основанных на выделении подпулов, необходимых для получения только одной генетической конструкции.
В первом подходе использовали баркоды, позволяющие проводить ПЦР олигонуклеотидов, относящихся только к одной сборке, с последующим отщеплением этих баркодов и сборкой целевой конструкции стандартными методами [77–79]. Эту схему тестировали с использованием двух олигонуклеотидных пулов, содержащих около 13 000 130- или 200-членных олигонуклеотидов, соответственно. На рис. 5. приведена схема использования этих пулов для олигонуклеотидного синтеза.
Рис. 5.
Схема синтеза генов с использованием олигонуклеотидного пула. Предварительно рассчитанные олигонуклеотиды синтезированы на микрочипе, затем отщеплены от поверхности с образованием олигонуклеотидного пула. В структуру заложены последовательности праймеров, позволяющие амплифицировать подпулы. Последовательности праймеров затем отщепляли и проводили сборку целевых генов.
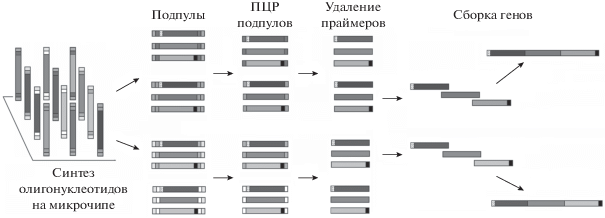
Для облегчения разделения исходного олигонуклеотидного пула на подпулы выбрали наборы 20-членных праймеров с малой способностью к кросс-гибридизации (“ортогональные праймеры”). Эти праймеры были созданы на основе набора из 244 000 25-членных ортогональных последовательностей, разработанных для баркодирования [80]. Из-за разных требований к дальнейшему использованию исходных олигонуклеотидных пулов были сконструированы два отдельных набора ортогональных праймеров к ним.
Олигонуклеотиды пулов 1 и 2 были спроектированы для тестирования эффекта длины перекрывания при синтезе. Гены, получаемые с помощью олигонуклеотидов пула 1, образовывали совершенные дуплексы. Олигонуклеотиды пула 2 формировали ДНК-фрагменты с частичным перекрыванием (около 20 п.н.) и превращались в полные дуплексы после проведения ПЦР.
Сконструирован набор генов флуоресцентных белков (GFP), предназначенных для проверки эффективности метода синтеза генов с помощью обоих олигонуклеотидных пулов. Гены выделяли и клонировали в экспрессионном векторе. При этом уровень ошибок в 130-членных олигонуклеотидах был достаточно низким, поскольку более 50% клонов содержали правильную структуру клонированного фрагмента. В этом случае уровень ошибок в генах, полученных из олигонуклеотидов, синтезированных на микрочиповых синтезаторах, был таким же, как в генах, получаемых с помощью олигонуклеотидов, синтезированных на традиционных синтезаторах.
При конструировании генов из пула, содержащего 200-членные олигонуклеотиды, получено только 6% правильных структур, уровень ошибок составил 1/250 п.н., что значительно выше, чем в случае 130-членных олигонуклеотидов (1/1000). Это не удивительно, поскольку уровень апуринизации синтезируемого олигонуклеотида зависит от числа деблокирований, т.е. от его длины.
Для снижения уровня ошибок в генах, синтезируемых с использованием обоих олигонуклеотидных пулов, применяли набор ErrASE – коммерческую смесь ферментов, обнаруживающих и гидролизующих дуплексы, содержащие неспаренные пары оснований. При этом уровень ошибок в генах, полученных из 200-членных олигонуклеотидов, удалось понизить в 4–8 раз – до 1/(1000–2000) п.н. В генах, полученных из 130‑членных олигонуклеотидов, уровень ошибок составил 1/(5000–7000) п.н.
С целью проверки разработанной технологии с использованием 200-членных олигонуклеотидов осуществлен синтез 42 генов, кодирующих вариабельные районы фрагментов одноцепочечных антител (scFv). Средний уровень ошибок в секвенированных генах составил 1/315 п.н., что также значительно выше, чем при сборке GFP из 130-членных олигонуклеотидов, но оставался приемлемым для конструирования генов этого размера (правильную структуру должны иметь в среднем 10% клонов).
Исходя из схемы, предложенной в [77], разработан высокопроизводительный метод синтеза ДНК, включающий одновременную амплификацию всех подпулов, исправление ошибок и сборку фрагментов [81]. Расчет структуры использованных олигонуклеотидов проводили с помощью программы DNAworks. 36 Подпулов (479 олигонуклеотидов) амплифицировали в одной реакции с ортогональными праймерами. Использовали очень эффективный высокопроизводительный метод коррекции ошибок после полной амплификациии олигонуклеотидного пула. На этой стадии несовершенные дуплексы удаляли путем аффинной хроматографии на колонках, содержащих белки системы репарации E. coli. Все олигонуклеотиды пула были успешно амплифицированы и очищены. Олигонуклеотидный пул, обогащенный структурами, не содержащими ошибок, выделяли и одновременно амплифицировали в 36 целевых фрагментов ДНК, используя LCR. При этом из 36 целевых последовательностей получены 24 структуры. Оставшиеся 12 фрагментов ДНК собирали в другом мультиплексном эксперименте.
Метод обладает достаточно высокой точностью вследствие эффективного удаления несовершенных дуплексов. Точность сборки целевых фрагментов ДНК увеличилась в 6.48 раза (с 14.25 до 2.20 ошибок на 1000 п.н.), в 12.85 раза увеличилась и доля конструкций, не содержащих ошибок (с 3.23 до 41.51%), по сравнению с нефракционированными олигонуклеотидами.
Применимость упрощенного метода сборки генетических конструкций из олигонуклеотидных пулов оценили путем успешного de novo синтеза генов метаболического пути каротиноида ликопена. Синтезированы 10 генов с оптимизированными кодонами (891–2412 п.н. всего 11.8 т.п.н.).
С использованием струйного синтезатора получены наборы олигонуклеотидов в отдельных лунках, составляющие генетическую конструкцию длиной 0.5–1 т.п.н. [82–84]. На чипе такие наборы физически отделены друг от друга, что исключает необходимость их разделения с помощью амплификации. Синтез олигонуклеотидов ведется на силикагельных спотах размером в 150 мкм. На стандартном слайде размером 25 × 25 мм размещали 30 лунок, содержащих 361 спот, что позволяло синтезировать 10830 различных олигонуклеотидов на всем слайде.
Синтезируемые 85-членные олигонуклеотиды включают 60-звенную конструкционную часть и 25-членный адаптор, содержащий сайт расщепления никирующей эндонуклеазы. После окончания синтеза и удаления защитных групп олигонуклеотиды остаются иммобилизованными в своих лунках. Затем эти ячейки заполняют реакционной смесью для амплификации и сборки гена. Большой фрагмент Bst-ДНК-полимеразы (указан шестиугольником на рис. 6), используют для постройки комплементарной цепи с вытеснением ранее синтезированной последовательности. Никирующая эндонуклеаза (указана овалом на рис. 6) отщепляет конструкционную часть олигонуклеотида универсального праймера (отмечен штриховкой на рис. 6). После завершения амплификации проводят сборку гена в ячейке с помощью PCA. Для гидролиза несовершенных дуплексов используют Surveyor-нуклеазу. Обработка нуклеазой снижает уровень ошибок в гене на порядок – с 1.9 до 0.19 на 1000 п.н.
Рис. 6.
Схема синтеза генов на микрочипе с физически разделенными лунками. Шестиугольником выделен большой фрагмент Bst-ДНК-полимеразы, овалом указана никирующая эндонуклеаза.
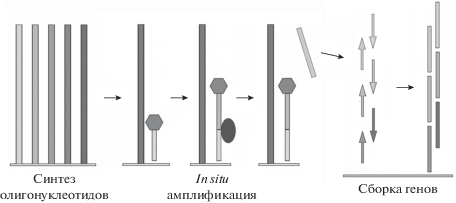
С помощью этого метода с целью повышения уровня экспрессии гена lacZα синтезирован набор вариантов этого гена, различающихся использованием синонимичных кодонов. Синтезированы также 74 версии генов антигенов дрозофилы, содержащие различные варианты кодонов. Продукция белков, кодируемых этими генами, изменялась в пределах 0–60% от массы общих клеточных белков.
С целью выделения олигонуклеотидов, требуемых для сборки конкретного гена, предложено фракционировать олигонуклеотидный пул с использованием водно-масляных эмульсий и парамагнитных шариков [85, 86]. Пул олигонуклеотидов превращали в ДНК-дуплексы и формировали одноцепочечные выступающие 5′-концы, одинаковые для олигонуклеотидов одного гена. Каждый шарик содержит одноцепочечный баркод для выделения олигонуклеотидов, подлежащих сборке в целевой ген. Олигонуклеотиды амплифицировали в эмульсии на поверхности шариков, из них ферментативно выделяли фрагменты для сборки целевых генов и с помощью PСA собирали гены. Этим методом синтезирована библиотека из 5775 гомологов гена дигидрофолатредуктазы и 1152 гомологов гена фосфопантетеин-аденилилтрансферазы. Себестоимость каждого из генов (450–675 п.н.), полученных данным методом, составляет 1–2 $.
ЗАКЛЮЧЕНИЕ
Одно из основных направлений использования олигонуклеотидов, полученных на микрочиповых синтезаторах – создание больших библиотек вариантов нуклеотидных последовательностей. Такие библиотеки используют при изучении структурных и функциональных характеристик тысяч цис-регуляторных элементов, отвечающих за транскрипционные, трансляционные и другие процессы в клетках млекопитающих, бактерий и дрожжей. Синтетические библиотеки, полученные из олигонуклеотидов, могут быть полезными при изучении сплайсинга, эпигенетических механизмов и в направленном мутагенезе [13–35].
Чрезвычайно эффективно использование этих олигонуклеотидов для получения библиотек генов для нужд белковой инженерии. Стоимость синтеза одного гена из такой библиотеки примерно на два порядка меньше, чем при получении гена с использованием олигонуклеотидов, полученных на традиционных синтезаторах. Целенаправленное варьирование кодонов позволяет получать штаммы с высокой экспрессией нужного гена [82–84].
Разработка методов конструирования библиотек генов из олигонуклеотидов, полученных с помощью микрочиповых синтезаторов [65–68, 74, 77–79, 81–86], в сочетании с развитыми способами комбинирования ДНК-дуплексов [42–45] дает основу для получения сложных генетических конструкций в интересах синтетической биологии.
На сегодняшний день микрочиповые синтезаторы, по-видимому, являются единственными эффективными инструментами конструирования элементов реестра стандарных блоков (Registry of Standard Biological Parts) [87] для нужд синтетической биологии.
Примеры конструирования полных геномов на основе олигонуклеотидных пулов еще не опубликованы, но, несомненно, это дело недалекого будущего.
Развитие высокопроизводительных методов получения генетического материала в России сдерживалось отсутствием отечественных микрочиповых синтезаторов, поскольку экспорт зарубежных синтезаторов в нашу страну запрещен. Учитывая важность развития нового направления (синтетической биологии) для нужд современной фундаментальной и прикладной науки в Сибирском отделении РАН создан консорциум из нескольких институтов (ИХБФМ СО РАН, ИФП СО РАН, НИОХ СО РАН, ИАиЭ СО РАН) для создания макета микрочипового синтезатора олигонуклеотидов. В настоящее время эта работа успешно завершена [88]. Сконструированный макет открывает возможность развития синтетической биологии в нашей стране. Он позволяет синтезировать до 12000 олигонуклеотидов в одном эксперименте, может использовать как фотолабильные амидиты, так и фотокислоты.
Написание обзора не потребовало специального финансирования.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Marchisio M.A. (2018) Introduction in Synthetic Biology. Springer.
Smolke C., Lee S.Y., Nielsen J., Stephanopoulos G. (2018) Synthetic Biology: Parts, Devices and Applications. Germany: Wiley-VCH.
Fodor S.P., Read J.L., Pirrung M.C., Stryer L., Lu A.T., Solas D. (1991) Light-directed, spatially addressable parallel chemical synthesis. Science. 251, 767–773.
Pease A.C., Solas D., Sullivan E.J., Cronin M.T., Holmes C.P., Fodor S.P. (1994) Light-generated oligonucleotide arrays for rapid DNA sequence analysis. Proc. Natl. Acad. Sci. USA. 91, 5022–5026.
Beaucage S.L., PIyer R. (1992) Advances in the synthesis of oligonucleotides by the phosphoramidite approach. Tetrahedron. 48(12), 2223–2311.
Singh-Gasson S., Green R.D., Yue Y., Nelson C., Blattner F., Sussman M.R., Cerrina F. (1999) Maskless fabrication of light-directed oligonucleotide microarrays using a digital micromirror array. Nat. Biotechnol. 17, 974–978.
Gao X., LeProust E., Zhang H., Srivannavit O., Gulari E., Yu P., Nishiguchi C., Xiang Q., Zhou X. (2001) A flexible light-directed DNA chip synthesis gated by deprotection using solution photogenerated acids. Nucl. Acids Res. 29, 4744.
http://www.affymetrix.com/support/technical/datasheets/nimble_programs_datasheet.pdf.
Baum M., Bielau S., Rittner N., Schmid K., Eggelbusch K., Dahms M., Schlauersbach A., Tahedl H., Eeier M., Guimil R., Scheffler M., Hermann C., Funk J., Wixmerten A., Rebscher H., Hönig M., Andreae C., Büchner D., Moschel E., Glathe A., Jäger E., Thom M., Greil A., Bestvater F., Obermeier F., Burgmaier J., Thome K., Weichert S., Hein S., Binnewies T., Foit-zik V., Müller M.J., Stähler C.F., Stähler P.F. (2003) Validation of a novel, fully integrated and flexible microarray benchtop facility for gene expression profiling. Nucl. Acids Res. 31, e151.
Maurer K., Cooper J., Caraballo M., Crye J., Suciu D., Ghindilis A., Leonetti J.A., Wang W., Rossi F.M., Stӧver A.G., Larson C., Gao H., Dill K., McShea A. (2006) Electrochemically generated acid and its containment to 100 micron reaction areas for the production of DNA microarrays. PLoS One. 1, e34.
Blanchard A.P., Kaiser R.J., Hood L.E. (1996) High-density oligonucleotide arrays. Biosens. Bioelectron. 11, 687–690.
Hughes T.R., Mao M., Jones A.R., Burchard J., Marton M.J., Shannon K.W., Lefkowitz S.M., Ziman M., Schelter J.M., Meyer M.R., Kobayashi S., Davis C., Dai H., He Y.D., Stephaniants S.B., Cavet G., Walker W.L., West A., Coffey E., Shoemaker D.D., Stoughton R., Blanchard A.P., Friend S.H., Linsley P.S. (2001) Expression profiling using microarrays fabricated by an ink-jet oligonucleotide synthesizer. Nat. Biotechnol. 19, 342–347.
Cleary M.A., Kilian K., Wang Y., Bradshaw J., Cavet G., Ge W., Kulkarni A., Paddison P.J., Chang K., Sheth N., Leproust E., Coffey E.M., Burchard J., McCombie W.R., Linsley P., Hannon G.J. (2004) Production of complex nucleic acid libraries using highly parallel in situ oligonucleotide synthesis. Nat. Meth. 1(3), 241–248.
Root D.E., Hacohen N., Hahn W.C., Lander E.S., Sabatini D.M. (2006) Genome-scale loss-of-function screening with a lentiviral RNAi library. Nat. Meth. 3(9), 715–719.
Johns N.I., Gomes A.L.C., Yim S.S., Yang A., Blazejewski T., Smillie C.S., Smith M.B., Alm E.J., Kosuri S., Wang H.H. (2018) Metagenomic mining of regulatory elements enables programmable species-selective gene expression. Nat. Methods. 15(5), 323–329.
Kosuri S., Goodman D.B., Cambray G., Mutalik V.K., Gao Y., Arkin A.P., Endy D., Church G.M. (2013) Composability of regulatory sequences controlling transcription and translation in Escherichia coli. Proc. Natl. Acad. Sci. USA. 110, 14024–14029.
Patwardhan R.P., Lee C., Litvin O., Young D.L., Pe’er D., Shendure J. (2009) High-resolution analysis of DNA regulatory elements by synthetic saturation mutagenesis. Nat. Biotechnol. 27(12), 1173–1175.
Schlabach M.R., Hu J.K., Li M., Elledge S.J. (2010) Synthetic design of strong promoters. Proc. Natl. Acad. Sci. USA. 107, 2538–2543.
Urtecho G., Tripp A.D., Insigne K.D., Kim H., Kosuri S. (2019) Systematic dissection of sequence elements controlling σ70 promoters using a genomically-encoded multiplexed repor. Biochemistry. 58(11), 1539–1551.
Sharon E., Kalma E., Sharp A., Raveh-Sadka T., Levo M., Zeevi D., Keren L., Yakhini Z., Weinberg A., Segal E. (2012) Inferring gene regulatory logic from high-throughput measurements of thousands of systematically designed promoters. Nat. Biotechnol. 30, 521–530.
Melnikov A., Murugan A., Zhang X., Tesileanu T., Wang L., Rogov P., Feizi S., Gnirke A, Callan C.G. Jr, Kinney J.B., Kellis M., Lander E.S., Mikkelsen T.S. (2012) Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat. Biotechnol. 30, 271–277.
Kheradpour P., Ernst J., Melnikov A., Rogov P., Wang L., Zhang X., Alston J., Mikkelsen T.S., Kellis M. (2013) Systematic dissection of regulatory motifs in 2000 predicted human enhancers using a massively parallel reporter assay. Genome Res. 23, 800–811.
Kwasnieski J.C., Mogno I., Myers C.A., Corbo J.C., Cohen B.A. (2012) Complex effects of nucleotide variants in a mammalian cis-regulatory element. Proc. Natl. Acad. Sci. USA. 109, 19498–19503.
White M.A., Myers C.A., Corbo J.C., Cohen B.A. (2013) Massively parallel in vivo enhancer assay reveals that highly local features determine the cis-regulatory function of ChIP-seq peaks. Proc. Natl. Acad. Sci. USA. 110, 11952–11957.
Smith R.P., Taher L., Patwardhan R.P., Kim M.J., Inoue F., Shendure J., Ovcharenko I., Ahituv N. (2013) Massively parallel decoding of mammalian regulatory sequences supports a flexible organizational model. Nat. Genet. 45(9), 1021–1028.
Sun H.H., Zhu C., Wu Y., Guo J.-F. (2009) De novo synthesis and assembly of multiplex riboswitches in vitro. Biotechnol. Prog. 25(5), 1228–1235.
Cheung R., Insigne K.D., Yao D., Burghard C.P., Wang J., Hsiao Y.-H.E., Jones E.M., Goodman D.B., Xiao X., Kosuri S. (2019) A multiplexed assay for exon recognition reveals that an unappreciated fraction of rare genetic variants cause large-effect splicing disruptions. Mol. Cell. 73(1), 183–194.e8.
Kaplan N., Moore I.K., Fondufe-Mittendorf Y., Gossett AJ., Tillo D., Field Y., LeProust E.M., Hughes T.R., Lieb J.D., Widom J., Segal E. (2009) The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362–366.
Sadhu M.J., Bloom J.S., Day L., Siegel J.J., Kosuri S., Kruglyak L. (2018) Highly parallel genome variant engineering with CRISPR/Cas9. Nat. Genet. 50(4), 510–514.
Smith J.D., Schlecht U., Xu W., Suresh S., Horecka J., Proctor M.J., Aiyar R.S., Bennett R.A., Chu A., Li Y.F., Roy K., Davis R.W., Steinmetz L.M., Hyman R.W., Levy S.F., Onge R.P. (2017) A method for high-throughput production of sequence-verified DNA libraries and strain collections. Mol. Systems Biol. 13(913), 1–15.
Bonde M.T., Kosuri S., Genee H.J., Sarup-Lytzen K., George M. G.M., Sommer M.O.A, Wang H.H. (2015) Direct mutagenesis of thousands of genomic targets using microarray-derived oligonucleotides. Synth. Biol. 4, 17−22.
Jones E.M., Lubock N.B., Venkatakrishnan A.J., Wang J., Tseng A.M., Paggi J.M., Latorraca N.R., Cancilla D., Satyadi M., Davis J.E., Babu M.M., Dror R.O., Kosuri S. (2020) Structural and functional characterization of G protein-coupled receptors with deep mutational scanning. eLife. 9, e54895.
Goodman D.B., Church G.M., Kosuri S. (2013) Causes and effects of N-terminal codon bias in bacterial genes. Science. 342, 475–479.
Quan J., Saaem I., Tang N., Ma S., Negre N., Gong H., White K.P., Tian J. (2011) Parallel on-chip gene synthesis and application to optimization of protein expression. Nat. Biotechnol. 29, 449–452.
Davis J.E., Insigne K.D., Jones E.M., Hastings Q.A., Boldridge W.C., Kosuri S. (2020) Dissection of c-AMP response element architecture by using genomic and episomal massively parallel reporter assays. Cell Systems. 11(1), 75–85, e7
Agarwal K.L., Büchi H., Caruthers M.H., Gupta N., Khorana H.G., Kleppe K., Kumar A., Ohtsuka E., Rajbhandary U.L., Van de Sande J.H., Sgaramella V., Weber H., Yamada T. (1970) Total synthesis of the gene for an alanine transfer ribonucleic acid from yeast. Nature. 227, 27–34.
Sekiya T., Takeya T., Brown E.L., Belagaje R., Contreras R., Fritz H.J., Gait M.J., Lees R.G., Ryan M.J., Khorana H.G., Norris K.E. (1979) Total synthesis of a tyrosine suppressor transfer RNA gene. XVI. Enzymatic joinings to form the total 207-base pair-long DNA. J. Biol. Chem. 254, 5787–5801.
Au L.C., Yang F.Y., Yang W.J., Lo S.H., Kao C.F. (1998) Gene synthesis by a LCR-based approach: high-level production of leptin-L54 using synthetic gene in Escherichia coli. Biochem. Biophys. Res. Commun. 248, 200–203.
Bang D., Church G.M. (2008) Gene synthesis by circular assembly amplification. Nat. Methods. 5, 37–39.
Stemmer W.P., Crameri A., Ha K.D., Brennan T.M., Heyneker H.L. (1995) Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides. Gene. 164, 49–53.
Gibson D.G. (2009) Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984–6990.
Gibson D.G. (2012) Oligonucleotide assembly in yeast to produce synthetic DNA fragments. Methods Mol. Biol. 852, 11–21.
Dormitzer P.R., Suphaphiphat P., Gibson D.G., Wentworth D.E., Stockwell T.B., Algire M.A., Alperovich N., Barro M., Brown D.M., Craig S., Datti-lo B.M., Denisova E.A., De Souza I., Eickmann M., Dugan V.G., Ferrari A., Gomila R.C., Han L., Judge C., Mane S., Matrosovich M., Merryman C., Palladino G., Palmer G.A., Spencer T., Strecker T., Trusheim H., Uhlendorff J., Wen Y., Yee A.C., Zaveri J., Zhou B., Becker S., Donabedian A., Mason P.W., Glass J.I., Rappuoli R., Venter J.C. (2013) Synthetic generation of influenza vaccine viruses for rapid response to pandemics. Sci. Transl. Med. 5, 185ra168.
Gibson D.G., Smith H.O., Hutchison C.A. III, Venter J.C., Merryman C. (2010) Chemical synthesis of the mouse mitochondrial genome. Nat. Methods. 7, 901–903.
Tang N., Ma S., Tian J. (2013) New tools for cost-effective DNA synthesis. In: Synthetic Biology. Ed. Zhao H. Acad. Press, 3–21.
Carr P.A., Church G.M. (2009) Genome engineering. Nat. Biotechnol. 27, 1151–1162.
Czar M.J., Anderson J.C., Bader J.S., Peccoud J. (2009) Gene synthesis demystified. Trends Biotechnol. 27, 63–72.
Xiong A.S., Peng R.H., Zhuang J., Gao F., Li Y., Cheng Z.M., Yao Q.H. (2008) Chemical gene synthesis: strategies, softwares, error corrections, and applications. FEMS Microbiol. Rev. 32, 522–540.
Larsen L.S.Z., Wassman C.D., Hatfield G.W., Lathrop R.H. (2008) Computationally optimised DNA assembly of synthetic genes. Int. J. Bioinform. Res. Appl. 4, 324–336.
Cello J., Paul A.V., Wimmer E. (2002) Chemical synthesis of poliovirus cDNA: generation of infectious virus in the absence of natural template. Science. 297, 1016–1018.
Smith H.O., Hutchison C.A. 3rd, Pfannkoch C., Venter J.C. (2003) Generating a synthetic genome by whole genome assembly: φX174 bacteriophage from synthetic oligonucleotides. Proc. Natl. Acad. Sci. USA. 100(26), 15440–15445.
Noyce R.S., Lederman S., Evans D.H. (2018) Construction of an infectious horsepox virus vaccine from chemically synthesized DNA fragments. PLoS One. 19(13), e0188453.
Gibson D.G., Benders G.A., Andrews-Pfannkoch C., Denisova E.A., Baden-Tillson H., Zaveri J., Stockwell T.B., Brownley A., Thomas D.W., Algire M.A., Merryman C., Young L., Noskov V.N., Glass J.I., Venter J.C. Hutchison C.A. 3rd, Smith H.O. (2008) Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome. Science. 319, 1215–1220.
Gibson D.G., Glass J.I., Lartigue C., Noskov V.N., Chuang R.Y., Algire M.A., Benders G.A., Montague M.G., Ma L., Moodie M.M., Merryman C., Vashee S., Krishnakumar R., Assad-Garcia N., Andrews-Pfannkoch C., Hutchison C.A. 3rd, Smith H.O. (2010) Creation of a bacterial cell controlled by a chemically synthesized genome. Science. 329, 52–56.
Venetz J.E., Medico L.D., Wölfle A., Schächle P., Bucher Y., Appert D., Tschan F., Flores-Tinoco C.E., van Kooten M., Guennoun R., Deutsch S., Christen M., Christen B. (2019) Chemical synthesis rewriting of a bacterial genome to achieve design flexibility and biological functionality. Proc. Natl. Acad. Sci. USA. 116(16), 8070–8079.
Matzas M., Stähler P.F., Kefer N., Siebelt N., Boisguérin V., Leonard J.T., Keller A., Stähler C.F., Häberle P., Gharizadeh B., Babrzadeh F., Church G.M. (2010) High-fidelity gene synthesis by retrieval of sequence-verified DNA identified using high-throughput pyrosequencing. Nat. Biotechnol. 28(12), 1291–1294.
Stähler P.F., Carapito R., Stähler C.F., Malzas M., Leonard J.T., Jäger J., Beier M. (2010) Patent WO No. 2010/094772 Al. Geneva, Switzerland World Intellectual Property Organization International Bureau.
Stähler P.F., Carapito R., Stähler C.F., Malzas M., Leonard J.T., Jäger J., Beier M. (2018) Patent US No. US 2017/0267999 A1. Washington, DC: U.S. Patent and Trademark Office.
Lee H., Kim H., Kim S., Ryu T., Kim H., Kwon D.B.S. (2015) A high-throughput optomechanical retrieval method for sequence-verified clonal DNA from the NGS platform. Nat. Commun. 6, 6073.
Bang D., Kim H., Cho N., Lim H., Park S., Han H. (2020) Patent US No. 10 526 640 B2. Washington, DC: U.S. Patent and Trademark Office.
Smith J.D., Schlecht U., Xu W., Suresh S., Horecka J., Proctor M.J., Aiyar R.S. (2010) A method for high throughput production of sequence verified DNA libraries and strain collections. Mol. Systems Biol. 13(2), 913.
Kim H., Han H., Ahn J., Lee J., Cho N., Jang H., Kim H., Kwon S., Bang D. (2012) Shotgun DNA synthesis for the high-throughput construction of large DNA molecules. Nucl. Acids Res. 40(18), e140.
Bang D., Kim H., Han H. (2019) Patent US No. 10 358 642 B2. Washington, DC: U.S. Patent and Trademark Office.
Schwartz J.J., Lee C., Shendure J. (2012) Accurate gene synthesis with tag-directed retrieval of sequence-verified DNA molecules. Nat. Methods. 9(9), 913–915.
Oleinikov A.V. (2004) Patent WO No. 2004/024886 A2. Geneva, Switzerland World Intellectual Property Organization International Bureau.
Tian J., Gong H., Sheng N., Zhou X., Gulari E., Gao X., Church G. (2004) Accurate multiplex gene synthesis from programmable DNA microchips. Nature. 432, 1050–1054.
Church G.M., Jingdong Tian J. (2005) Patent WO No. 2005/089110 A2. Geneva, Switzerland World Intellectual Property Organization International Bureau.
Church G.M., Jingdong T.J. (2006) Patent US No. 2006/0127920 A1. Washington, DC: U.S. Patent and Trademark Office.
Richmond K.E., Li M.-H., Rodesch M.J., Patel M., Lowe A.M., Kim C., Chu L.L., Venkataramaian N., Flickinger S.F., Kaysen J., Belshaw P.J., Sussman M.R., Cerrina F. (2004) Amplification and assembly of chip-eluted DNA (AACED): a method for high-throughput gene synthesis. Nucl. Acids Res. 32, 5011–5018.
Kim C., Kaysen J., Richmond K., Rodesch M., Binkowski B., Chu L., Li M., Heinrich K., Blair S., Belshaw P., Sussman M., Cerrina F. (2006) Progress in gene assembly from a MAS-driven DNA microarray. Microelectronic Engineering. 83, 1613–1616.
Zhou X., Cai S., Hong A., You Q., Yu P., Sheng N., Srivannavit O., Muranjan S., Rouillard J.M., Xia Y., Zhang X., Xiang Q., Ganesh R., Zhu Q., Matejko A., Gulari E., Gao X. (2004) Microfluidic PicoArray synthesis of oligodeoxynucleotides and simultaneous assembling of multiple DNA sequences. Nucl. Acids Res. 32, 5409–5417.
Ma Q., Li S. (2017) US Patent No. US 9 695 417 B2. Washington, DC: U.S. Patent and Trademark Office.
Kim H., Jeong J., Bang D. (2011) Hierarchical gene synthesis using DNA microchip oligonucleotides. J. Biotechnol. 151, 319–324.
Borovkov A.Y., Loskutov A.V., Robida M.D., Day K.M., Cano J.A., Olson T.L., Patel H., Brown K, Hunter P.D., Sykes K.F. (2010) High-quality gene assembly directly from unpurified mixtures of microarray-synthesized oligonucleotides. Nucl. Acids Res. 38(19), e180.
Zhou X., Cai S., Hong A., You Q., Yu P., Sheng N., Srivannavit O., Muranjan S., Rouillard J.M., Xia Y., Zhang X., Xiang Q., Ganesh R., Zhu Q., Matejko A., Gulari E., Gao X. (2004) Microfluidic PicoArray synthesis of oligodeoxynucleotides and simultaneous assembling of multiple DNA sequences. Nucl. Acids Res. 32, 5409–5417.
Kosuri S., Church G.M. (2014) Large-scale de novo DNA synthesis: technologies and applications. Nat. Meth. 11(5), 499–507.
Kosuri S., Eroshenko N., LeProust E., Super M., Way J., Li J.B., George M., Church G.M. (2010) Scalable gene synthesis by selective amplification of DNA pools from high-fidelity microchips. Nat. Biotechnol. 28, 1295–1299.
Church G.M., Kosuri S., Eroshenko N. (2014) Patent US No. US 2014/0045728A1. Washington, DC: U.S. Patent and Trademark Office.
Church G.M., Kosuri S., Eroshenko N. (2012) Patent WO No. 2012/154201 Al. Geneva, Switzerland World Intellectual Property Organization International Bureau.
Xu Q., Schlabach M.R., Hannon G.J., Elledge S.J. (2009) Design of 240000 orthogonal 25mer DNA barcode probes. Proc. Natl. Acad. Sci. USA. 106, 2289–2294.
Wan W., Lu M., Wang D., Gao X., Hong J. (2017) High-fidelity de novo synthesis of pathways using microchip synthesized oligonucleotides and general molecular biology equipment. Sci. Rep. 7(1), 6119.
Tian J. (2018) Patent US No. US 2018 / 0080069 A1. Washington, DC: U.S. Patent and Trademark Office.
Tian J. (2013) Patent EP 2 826 862 A1. Munich, Germany European Patent Office.
Quan J., Saaem I., Tang N., Ma S., Negre N., Gong H., White K.P., Tian J. (2011) Parallel on-chip gene synthesis and application to optimization of protein expression. Nat. Biotechnol. 29, 449–452.
Plesa C., Sidore A.M., Lubock N.B., Zhang D., Kosuri S. (2018) Multiplexed gene synthesis in emulsions for exploring protein functional landscapes. Science. 359(6373), 343–347.
Sidore A.M., Plesa C., Samson J.A., Lubock N.B., Kosuri S. (2020) DropSynth 2.0: high-fidelity multiplexed gene synthesis in emulsions. Nucl. Acids Res. 48(16), e95.
Canton B., Labno A., Endy D. (2008) Refinement and standardization of synthetic biological parts and devices. Nat. Biotechnol, 26, 787–793.
Синяков А.Н., Бессмельцев В.П., Камаев Г.Н. (2018) Микрочиповый синтезатор ДНК. II. Всероссийская научно-практическая конференция “Научное приборостроение – современное состояние и перспективы развития”. Казань 4–7 июня. Сб. материалов конференции. с. 280–282.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология