Молекулярная биология, 2022, T. 56, № 1, стр. 157-167
Изучение множественного ферментативного встраивания модифицированных нуклеотидов пуриновой и пиримидиновой природы в растущую цепь ДНК
С. А. Лапа a, *, О. С. Волкова a, В. Е. Кузнецова a, А. С. Заседателев a, А. В. Чудинов a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: lapa@biochip.ru
Поступила в редакцию 27.04.2021
После доработки 27.04.2021
Принята к публикации 21.05.2021
- EDN: KKAFYM
- DOI: 10.31857/S0026898422010050
Аннотация
Изучены субстратные свойства модифицированных по азотистому основанию производных пуриновых и пиримидиновых дезоксинуклеозидтрифосфатов при их одновременном попарном встраивании в растущую цепь ДНК. Модифицированные нуклеотиды вводили с помощью полимеразной цепной реакции в режиме реального времени и реакции удлинения праймера; в одной реакции использовали производные как с различными, так и с аналогичными функциональными заместителями. В качестве матриц применяли геномную бактериальную ДНК, специальным образом сконструированные синтетические фрагменты ДНК, а также библиотеки для SELEX. Реакции проводили с помощью ДНК-полимераз, не имеющих 3'-5'-корректирующей экзонуклеазной активности: Taq, Vent (exo-), DeepVent (exo-) и KOD XL. Показано, что на субстратную эффективность дезоксинуклеозидтрифосфата влияет как размер группы-заместителя, так и его химическая природа. Субстратная эффективность значительно зависит также от используемой полимеразы. Наиболее эффективными субстратами оказались пиримидиновые дезоксинуклеозидтрифосфаты в сочетании с ДНК-полимеразой Vent (exo-). Получены ДНК, модифицированные парами разноименных нуклеотидов (dU + dC, dU + dA, dC + dA) с аналогичными и различными функциональными заместителями.
ВВЕДЕНИЕ
Нуклеотиды, модифицированные по азотистому основанию различными химическими группами, используются преимущественно для создания аптамеров (одноцепочечных фрагментов нуклеиновых кислот, проявляющих аффинность к определенным молекулярным мишеням) с расширенными свойствами. Присутствие в составе аптамера функциональных групп, не характерных для природной ДНК (РНК), позволяет увеличить его сродство к молекулярным мишеням за счет расширения спектра физико-химических взаимодействий [1–3].
Модифицированные ДНК получают чаще всего ферментативным способом, а наиболее распространенным модифицированным субстратом для ДНК-полимераз служит дезоксиуридинтрифосфат (dUTP) с различными функциональными группами, введенными по 5-положению пиримидинового цикла. Модифицированные dCTP применяют в ферментативных реакциях амплификации ДНК [4, 5], при этом субстратные свойства пуриновых нуклеотидов изучены в гораздо меньшей степени и в настоящее время не находят широкого применения при создании модифицированных аптамеров в силу сложностей их синтеза.
Следующим шагом в создании модифицированных аптамеров может стать одновременное введение в одну цепь ДНК разноименных нуклеотидов (например, dU и dC, dU и dA) с различными модификациями. Теоретически такой подход способен резко расширить спектр физико-химических взаимодействий аптамера с мишенями и, следовательно, расширить спектр мишеней для создания высокоспецифичных аптамеров.
Этот подход использовали для получения модифицированных аптамеров к белковой мишени [6], однако он не получил широкого распространения.
Одним из наиболее важных аспектов создания аптамеров нового поколения становится изучение совместимости разноименных модифицированных нуклеотидов с полимеразами, используемыми в SELEX.
В настоящей работе изучена субстратная эффективность различных модифицированных пуриновых и пиримидиновых dNTP при их одновременном ферментативном встраивании в растущую цепь ДНК.
Субстратное поведение модифицированных производных dNTP анализировали, используя генетические бактериальные мишени и специальным образом сконструированные матричные олигонуклеотидные последовательности. С применением полимеразной цепной реакции в режиме реального времени (ПЦР-РВ) и реакции удлинения праймера (РЕХ) проанализирована кинетика и определен выход продуктов ферментативных реакций в условиях последовательных множественных замен природных нуклеотидов на модифицированные пуриновые и пиримидиновые модифицированные аналоги (поли-dA-, поли-dG- и поли-dT-матрицы).
Осуществлен выбор наиболее эффективных ДНК-полимераз с отсутствующей 3'-5'-экзонуклеазной активностью для реакций амплификации при одновременном введении в растущую цепь ДНК разноименных модифицированных производных dNTP.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
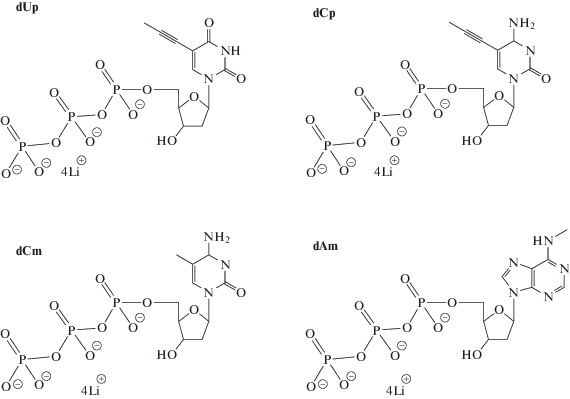
Модифицированные dNTP. Все соединения, использованные в работе, произведены фирмой “Trilink Biotechnologies” (США): 5-пропинил-2'-дезоксиуридин-5'-трифосфат (N-2016), 5-пропинил-2'-дезоксицитидин-5'-трифосфат (N-2017), 5-метил-2'-дезоксицитидин-5'-трифосфат (N-2025), N6-метил-2'-дезоксиаденозин-5'-трифосфат (N-2026).
Структуры использованных соединений приведены на рис. 1.
Рис. 1.
dUTP и dATP, модифицированные по азотистому основанию. dUp и dCp – производные, содержащие 5-пропинил; dCm и dAm – производные, содержащие метил в качестве модифицирующей функциональной группы.
Матрицы. В работе использованы бактериальные и синтетические матрицы. ПЦР-РВ проводили на полногеномных бактериальных матрицах (Mycobacterium tuberculosis, Staphylococcus aureus, Legionella pneumophila), что позволило получить ПЦР-продукты различающиеся длиной и GC-составом.
В качестве синтетической ПЦР-матрицы использовали комбинаторную ДНК-библиотеку для SELEX и соответствующие праймеры [7].
При изучении множественного последовательного встраивания модифицированных нуклеотидов с помощью PEX применяли специальным образом сконструированные матрицы. Последовательности специализированных матриц приведены на рис. 2. Использовали праймер, полностью комплементарный указанной на рис. 2 праймерной части матрицы.
Рис. 2.
Матричные олигонуклеотиды для изучения индивидуального и попарного множественного последовательного встраивания модифицированных производных dU и dC методом PEX. Курсивом на каждой последовательности выделена праймерная часть. Полужирным показаны олигонуклеотиды, комплементарные модифицированным производным dU, dC и dA. а – Олигонуклеотиды, использованные в экспериментах с множественным встраиванием модифицированных dU и dC [8]; б – олигонуклеотиды, сконструированные для определения одновременного множественного встраивания пар модифицированных пиримидиновых и пуриновых нуклеотидов (dU + dA и dС + dA).

Твердофазный синтез матричных олигонуклеотидов. Твердофазный синтез матричных олигонуклеотидов проводили с помощью автоматического синтезатора ABI 394 DNA/RNA (“Applied Biosystems”, США) по стандартному регламенту с использованием коммерческих растворителей и реагентов. 3'-Амидофосфиты природных 2'-дезоксирибонуклеозидов (dA-CE “Phosphoramidite”, кат. номер 10-1000-5S, dmf-dG-CE Phosphorami-dite, кат. номер 10-1029-5S, dT-CE Phosphoramidite, кат. номер 10-1030-5S, Ac-dC-CE Phosphoramidite, кат. номер 10-1015-5S) использовали в виде 0.1 М растворов в абсолютном ацетонитриле. В качестве твердой фазы использовали высокопористое стекло (CPG-1000), содержащее иммобилизованный нуклеозид и применяемое для синтеза олигонуклеотидов длиной примерно 40 н.
Хроматографическая очистка матричных олигонуклеотидов. Очистку олигонуклеотидов проводили с использованием хроматографической колонки (“Thermo Scientific” BDS Hypersil C18 размером 250 × 4.6 мм, размер частиц 5 мкм) в системe элюентов: буфер А – 0.1 М TEAA, буфер Б – 50% ацетонитрила в буфере А. Использовали “градиент для олигонуклеотидов с DMT группой”, или “TR-on”, который наилучшим образом соответствовал характеру очищаемого продукта. Оба буфера готовили с использованием воды качества milliQ и ацетонитрила для ВЭЖХ (ChromAR® HPLC, “Macron Fine Chem.”, США), фильтровали через фильтр ZAPCAP-CR Nylon 0.22 мкм 47 мм (“Sigma-Aldrich”, США). Олигонуклеотиды разделяли при комнатной температуре (скорость подачи элюента – 1 мл/мин) и детектировали на двух длинах волн: λ1 = 270 нм и λ2 = = 295 нм.
ДНК-полимеразы. В работе использовали Taq-полимеразу (“ThermoScientific”, США), Vent (exo-), Deep Vent (exo-) (“New England Biolabs”, США), KOD XL (“Novataq”, США) в реакционных буферах и концентрациях, рекомендованных производителями.
Реакция удлинения праймера (PEX). Реакционная смесь содержала природные dCTP и dGTP каждый в концентрации 0.2 мМ, а также различные сочетания модифицированных dUTP и/или dATP (при тестировании dCTP природный трифосфат также заменяли искусственным модифицированным аналогом); полимеразу (из списка тестируемых) в количестве, рекомендуемом производителем (реакционный буфер соответствовал полимеразе); праймер для PEX; одну из синтетических матриц. Реакцию проводили на ДНК-амплификаторе MiniCycler (“MJ Research”, США) по следующей программе: 5 мин при 95°C; далее 30 с при 65°C и 40 мин при 72°C.
Полимеразная цепная реакция (ПЦР). Реакционная смесь для ПЦР была аналогична смеси для PEX. В качестве матрицы использовали фрагменты бактериальных генов различной длины либо комбинаторную библиотеку и соответствующие праймеры. Температурно-временной профиль ПЦР состоял из предварительной денатурации при 95°С в течение 3 мин, за которой следовал 31 цикл: 95°С, 30 с; 65°С, 30 с; 72°С, 40 с, затем завершающая инкубация при 72°С в течение 5 мин.
ПЦР в режиме реального времени (ПЦР-РВ). Для визуализации процесса в реакционную смесь добавляли краситель EvaGreen (“Biotium”, Россия). Амплификацию проводили на приборе IQ5 (“Bio-Rad”, США).
Определение выхода продуктов амплификации. Полученные ПЦР-продукты разделяли в 4%-ном агарозном геле, окрашивали бромидом этидия. Количество продукта определяли по оптической плотности полос в дорожках геля с использованием программы ImageJ (NIH, США).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Ранее мы изучили попарное множественное встраивание модифицированных производных dUTP и dCTP. Анализ методом ПЦР-РВ показал, что производные с менее объемными функциональными группами (dUp и dCp, содержащие пропинильный заместитель в 5-положении цикла) характеризуются высокой скоростью накопления продукта, в то время как у массивных (dUi и dCi, содержащие индольный заместитель в 5-положении цикла) наблюдается либо пологая кривая накопления сигнала, либо задержка порогового цикла (Ct) в зависимости от использованной комбинации dU + dC. По-видимому, гораздо более короткие временные интервалы элонгации праймеров в ПЦР по сравнению с PEX были причиной более сильного проявления ингибирующего влияния модифицированных производных в ПЦР [8].
В настоящей работе нами определены такие показатели субстратной эффективности модифицированных dNTP, как величина “Е” и выход целевого продукта при использовании различных матриц, ферментативных реакций и ДНК-полимераз. В соответствии с опубликованными данными [8], из рассмотрения были исключены производные с массивными заместителями. Основная цель нашей работы состояла в изучении одновременного встраивания нуклеотидов пиримидиновой (dU и dC) и пуриновой (dA) природы.
ПЦР-РВ с применением полногеномных бактериальных матриц
Один из важных показателей пригодности модифицированного субстрата для полимераз в ПЦР – эффективность амплификации “Е”, определяемая математически [9, 10].
Кинетику встраивания изучали методом ПЦР-РВ на термоциклере (амплификаторе) BioRad IQ5 с использованием ДНК-полимеразы семейства B – Vent (exo-), ранее показавшей наибольшую эффективность в реакциях с модифицированными dNTP [8, 11].
ПЦР-продукты разной длины получали с использованием генетических бактериальных мишеней различного GC-состава и сконструированных ранее праймеров [11]. Использовали следующие бактерии, фрагменты генов для амплификации и последовательности праймеров:
1. Mycobacterium tuberculosis, ген rpoB, праймеры f1272 (5'-CGCCGCGATCAAGGAGTTCT-3') и r1398 (5'-TCACGTGACAGACCGCCGGG-3'). Длина ПЦР-продукта – 126 п.н.;
2. Staphylococcus aureus, ген ebpS, праймеры ebpS-f1 (5'-ACTCGACTGAGGATAAAGCGTCT-3') и ebps-r1 (5'-CCTCCAAATATCGCTAATGCACC-3'). Длина ПЦР-продукта – 283 п.н.;
3. Legionella pneumophila, ген sidA, праймеры ebpS-f1 (5'-TTCCACTGGTGGGTGGGTTTTG-3') и ebps-r1 (5'-TCATGTTGGAGTTCTATGGCACG-3'). Длина ПЦР-продукта – 370 п.н.
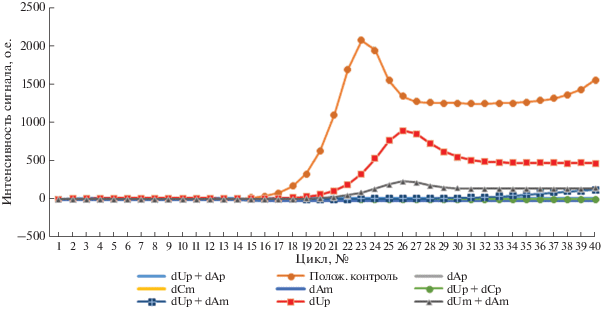
Субстратное поведение модифицированных dNTP определяли с использованием фрагментов бактериального генома различной длины и GC-состава, что позволило выявить закономерности влияния различных циклических структур в модификациях для производных с функциональными группами (потенциально применимыми в mod-SELEX) на выход реакции и ее эффективность в зависимости от длины ПЦР-продукта (рис. 3, табл. 1).
Рис. 3.
Встраивание модифицированных dNTP с использованием в качестве матицы геномной ДНК L. pneumophila (ПЦР-продукт 370 п.н.). Полимераза Vent (exo-).

Таблица 1.
Эффективность амплификации на матрицах с фиксированной последовательностью (полимераза Vent (exo-)
| Положительный контроль | dUp | dCp | dCm | dAm | dUp + dCp* | dUp + dAm | dCp + dAm | dCm + dAm |
|---|---|---|---|---|---|---|---|---|
| Mycobacterium tuberculosis (126 п.н.) | ||||||||
| Е =1.82 | 1.70 | 1.64 | 1.45 | 1.73 | 1.54 | 1.63 | 1.48 | 1.48 |
| Staphylococcus aureus (283 п.н.) | ||||||||
| Е = 1.85 | 1.76 | 1.62 | 1.42 | 1.22 | 1.64 | 1.50 | 1.14 | 1.11 |
| Legionella pneumophila (370 п.н.) | ||||||||
| Е = 1.83 | 1.71 | 1.45 | 1.34 | 1.27 | 1.30 | 1.29 | 1.00** | 1.00** |
ПЦР-РВ с применением комбинаторных ДНК-библиотек в качестве матриц
Изучение возможности одновременного применения различных трифосфатов для введения модификаций в комбинаторные библиотеки крайне важно, поскольку амплификация библиотек является одной из наиболее важных составляющих SELEX. Введение разноименных модифицированных нуклеотидов в процессе обогащения библиотек может быть востребовано для получения аптамеров с расширенными свойствами.
В отличие от матриц с фиксированной последовательностью, при амплификации комбинаторных библиотек наблюдается более сложный характер накопления сигнала ПЦР. На рис. 4 приведен типичный пример кривых амплификации ДНК-библиотек. Подобный характер кривых накопления сигнала (с одним или более максимумами) описан нами ранее [7, 12, 13] и определяется поведением библиотек при их амплификации.
Кинетику встраивания модифицированных нуклеотидов изучали методом ПЦР-РВ на термоциклере (амплификаторе) BioRad IQ5 с использованием ДНК-полимераз Taq (семейство А), Vent (exo-) и DeepVent (exo-) – (семейство B) и полимеразы KOD XL. У всех этих полимераз отсутствует 3'-5'-корректирующая экзонуклеазная активность, что позволяет использовать их для получения ДНК с включением модифицированных нуклеотидов.
Эффективность амплификации “E” рассчитывали по углу наклона прямого участка S-образной кривой накопления сигнала [9, 10]. С учетом характера амплификации ДНК-библиотек расчеты проводили до достижения кривой первого максимума. Из рис. 4 и табл. 2 видно, что все исследованные модифицированные dNTP как пуриновой, так и пиримидиновой природы в различной степени ингибируют ПЦР. Этот результат хорошо коррелирует с полученными ранее данными [11].
Таблица 2.
Сводная таблица эффективности в ПЦР с комбинаторной ДНК-библиотекой и различными сочетаниями модифицированных dNTP при полном замещении природных dNTP
| Полимераза | Положительный контроль | dUp | dCp | dCm | dAm | dUp + dCp* | dUp + dAm | dCp + dAm | dCm + dAm |
|---|---|---|---|---|---|---|---|---|---|
| Vent (exo-) | 1.87 | 1.82 | 1.57 | 1.40 | 1.01 | 1.83 | 1.21 | 1.41 | 1.67 |
| Deep Vent (exo-) | 1.95 | 1.88 | 1.80 | 1.77 | 1.49 | 1.68 | 1.73 | 1.70 | 1.59 |
| Taq | 1.91 | 1.25 | 1.15 | 1.16 | 1.08 | 1.11 | 1.70** | 1.05 | 1.11 |
| KOD XL | 1.71 | 1.82 | 1.01 | 1.00 | 1.00 | 1.02 | 1.32 | 1.72 | 1.61 |
* Расчеты произведены для контроля корреляции с данными [8]. ** Высокое значение “Е” обусловлено образованием неполноразмерных продуктов.
Определение выхода полноразмерных продуктов ПЦР
Выход модифицированных продуктов определяли электрофоретически в режиме как индивидуального встраивания, так и одновременного параллельного встраивания разноименных модифицированных нуклеотидов. Матрицей служила комбинаторная ДНК-библиотека Matrix_1f-а 5'‑CTGTCAGCTCCATACTGGTAGCC-(N)40-GCGTTCGAATCTAGACGGTACGA-3' и праймеры для ее амплификации:
Forw_1-06 5'-CTGTCAGCTCCATACTGGTAGCC-3' 23 н.
и
Rev_1-06 5'-TCGTACCGTCTAGATTCGAACGC-3' 23 н.
Выход продукта определяли по оптической плотности полосы геля при электрофоретическом разделении продуктов ПЦР. Следует заметить, что в ряде случаев наблюдается низкая корреляция эффективности амплификации и выхода продукта. Это связано с тем, что кривая наклона может иметь крутой всплеск, но быстро выходить на плато, т.е. в таких случаях, согласно данным расчета по тангенусу угла наклона прямого участка S-образной кривой накопления сигнала, наблюдается интенсивная, но быстро затухающая динамика процесса накопления целевого продукта.
Электрофореграммы продуктов ПЦР, полученных с различными полимеразами, приведены на рис. 5. Видно, что производное dAm служит плохим субстратом для всех изученных полимераз, не обладающих 3'-5'-корректирующей экзонуклеазной активностью. Однако при использовании dAm в сочетании с модифицированными производными dCT в ряде случаев удавалось получить полноразмерный продукт. Сочетания соединений в одной реакции описаны в разделе, посвященном множественному параллельному встраиванию.
Рис. 5.
ПЦР с использованием комбинаторной ДНК-библиотеки в качестве матрицы. Под каждой электрофореграммой приведено название полимеразы, не обладающей 3'-5'-корректирующей экзонуклеазной активностью. 1 – Маркер длин фрагментов ДНК (ladder 50 bp), 2 – положительный контроль (только природные dNTP), 3 – dUp, 4 – dCp, 5 – dCm, 6 – dAm, 7 – ladder 50 bp, 8 – dUp + dCp, 9 – dUp + dAm, 10 – dCp + dAm, 11 – dCm + dAm, 12 – отрицательный контроль.
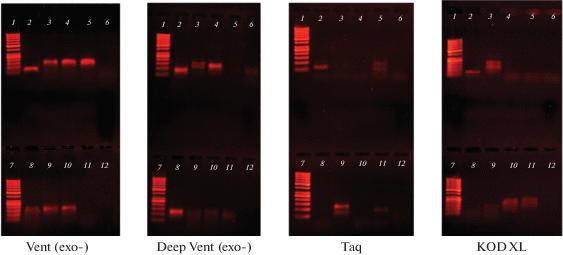
Наибольшую эффективность в реакции с модифицированными субстратами показали полимеразы семейства В: Vent (exo-) и DeepVent (exo-). При анализе эффективности “Е” полученные данные важно дополнять результатами электрофоретического анализа, так как само значение “Е”, будучи кинетической характеристикой, не дает информации о получаемом продукте. Так, в ряде случаев при хороших значениях эффективности этого показателя дальнейший анализ длин продуктов с помощью электрофореза позволил выявить большую долю неполноразмерных продуктов (это характерно для полимераз DeepVent (exo-) и KOD XL). Taq-полимераза показала низкую способность воспринимать модифицированные dNTP в качестве субстратов при полном замещении природных нуклеотидов, необходимом, в частности, для проведения SELEX. В то же время ранее мы показали, что Taq-полимераза пригодна для включения пиримидиновых нуклеотидов (dU), модифицированных флуорофорами, при частичной замене природных dT [11].
Результаты ПЦР с различными полимеразами показаны на рис. 5.
Изучение субстратных свойств модифицированных dNTP в реакции удлинения праймера (PEX)
С целью изучения субстратных свойств исследуемых соединений c использованием PEX сконструированы и синтезированы специализированные матричные олигонуклеотиды. Эти синтетические матрицы сконструированы таким образом, чтобы изучать последовательное встраивание: одного, двух, трех и четырех модифицированных нуклеотидов.
Последовательности матриц приведены на рис. 2.
Матрицы сконструированы с учетом опыта изучения множественного последовательного встраивания модифицированных нуклеотидов в растущую цепь ДНК [8, 14].
Матрица М1A предназначена для изучения последовательного множественного встраивания производных dA в индивидуальном режиме. Встраивание происходит последовательно 1, 2, 3 и 4 раза в процессе элонгации праймера по матричной цепи. Это позволяет оценить эффективность множественного последовательного встраивания mod-dA. После праймерной части последовательность матрицы такова, что, кроме комплементарного T, не содержит других нуклеотидов, комплементарных модифицированным dA. Видно, что спейсерные участки матрицы не содержат комплементарных аденину нуклеотидов (T). Кроме того, для сравнения с результатами попарного встраивания с dU (на матрицах М2UA и M2AU) спейсерные участки также не содержат dA.
Матрицы М2UA и M2AU предназначены для изучения множественного попарного встраивания dA и dU в различных последовательностях, что отражено в названиях матричных олигонуклеотидов.
Аналогичная матрица для dU уже синтезирована и описана ранее [8], как и матрицы для dC и (dU + dC), рис. 2а. Применение этих матриц позволяет в параллельном режиме (каждый разноименный нуклеотид на своей матрице) изучать особенности встраивания только определенных пар, например A и U (C и U, C и A).
Так, для изучения одновременного встраивания модифицированных производных dU и dA сконструированы и синтезированы матрицы M2AU и M2UA, последовательности которых приведены на рис. 2б. Эти матрицы позволяют изучать последовательное встраивание нескольких производных в последовательности A-U и U-A в растущую цепь ДНК.
Таким образом, нами использованы синтетические матрицы M1U, M1А, М2UA и M2AU.
На рис. 6 показаны результаты получения модифицированных ДНК с производными пуриновой природы dAm и dAp как отдельно, так и в сочетании с dUp, методом PEX. Применены специализированные синтетические матрицы.
Рис. 6.
PEX с применением производных пуриновой и пиримидиновой природы. а – Схема расположения продуктов PEX в лунках геля. б – Cхема маркера длин двухцепочечной ДНК GeneRuler 50 bp (“Thermo”, США). в–е – Результаты PEX с различными полимеразами. 1 – Маркер длин GeneRuler 50 bp (“Thermo”); 2 – положительный контроль; 3 – dUp; 4 – dAm; 5 – dUp + dAm, далее как для других матриц (указаны на схеме а).
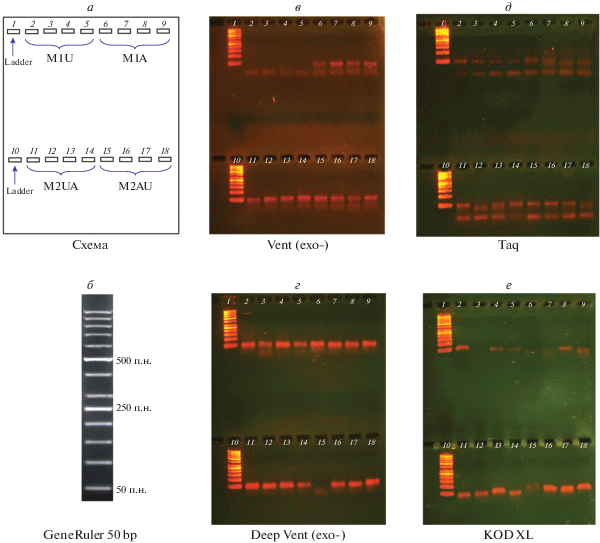
Из электрофореграмм видно, что для получения ДНК с модифицированными дезоксиаденозинтрифосфатами наиболее подходят полимеразы семейства В. Получить полноразмерные продукты удалось во всех случаях, но с разными выходами целевого продукта. Видны полосы непрореагировавших праймеров, более заметные в случае Taq-полимеразы, которая характеризуется наименьшим выходом целевого продукта.
PEX обеспечивает более подходящие условия для встраивания модифицированного пуринового производного, в том числе последовательного множественного встраивания. Применение ферментов семейства В – Vent (exo-) и DeepVent (exo-) – позволило получить полноразмерные модифицированные продукты как при индивидуальном, так и при одновременном параллельном применении модифицированных производных пиримидиновой и пуриновой природы. Taq-полимеразу отличают низкие выходы продукта и образование неполноразмерных продуктов. KOD XL может использоваться для множественного встраивания, однако она не отличается высокой воспроизводимостью результатов, а на электрофореграммах видны побочные продукты (by-products).
Таким образом, для ферментативного получения ДНК с множественными модификациями по совокупности характеристик (воспроизводимость и выход целевого полноразмерного продукта) выбрана Vent (exo-)-ДНК-полимераза. Получены продукты элонгации праймера, содержащие множественные модификации, в том числе одновременно на пуриновых и пиримидиновых нуклеотидах.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Полученные ранее данные как основа настоящих исследований
Ранее мы изучили закономерности одновременного встраивания пиримидиновых нуклеотидов в ДНК [8]. С использованием ДНК-полимераз Taq, Vent (exo-), KOD XL детально изучено субстратное поведение dC и dU в PEX и ПЦР, рассчитаны выход и эффективность амплификации. Осуществлено попарное введение разноименных модифицированных dNTP (mod-dU + + mod-dC) как в ПЦР, так и в PEX. Получены ДНК, содержащие несколько модификаций в одной цепи.
Определено, что модифицированные нуклеотиды с менее массивными заместителями являются более эффективными субстратами для полимераз семейства В при полной замене соответствующих природных dNTP. Тестирование модифицированных dU и dC показало, что для ферментативного получения модифицированных ДНК предпочтительно использовать полимеразы семейства В, не обладающие 3'-5'-экзонуклеазной активностью [8, 11], а именно Vent (exo-) и DeepVent (exo-). При этом DeepVent (exo-) чувствительна к количеству матрицы и склонна к образованию побочных продуктов реакции. Сходным образом проявляет себя и полимераза KOD XL.
Выбор ДНК-полимераз, наиболее эффективных в реакциях амплификации, в случае одновременного введения в растущую цепь ДНК модифицированных производных dU, dC и dA
Из рис. 5 видно, что все протестированные нами полимеразы встраивают использованное модифицированное пуриновое производное менее эффективно, чем производные пиримидиновой природы. Причины этого могут быть связаны как с модифицирующей группой, так и с плохим восприятием модификаций по позиции 6 пуринового цикла полимеразами [15–17].
Тем не менее, в комбинации с разноименными трифосфатами пиримидинового ряда удалось получить продукт для dAm. Наиболее воспроизводимые результаты получены при использовании полимеразы Vent (exo-), принадлежащей к семейству В. В целом, результаты, полученные при применении большинства ферментов (исключая Vent (exo-)) на комбинаторных ДНК-библиотеках, недостаточно воспроизводимы, что может быть связано с флуктуацией состава библиотеки, содержащей сотни тысяч последовательностей, при добавлении аликвоты такой разнородной смеси в каждую из реакционных пробирок.
Несколько большая эффективность амплификации библиотек по сравнению с матрицами с фиксированной последовательностью обусловлена, предположительно, избирательной (конкурентной) амплификацией, при которой амплифицируются преимущественно только матрицы, “удобные” для полимеразы. Падение эффективности амплификации было более заметным в случае фиксированных матриц, содержащих лишь одну последовательность, и низкой субстратной совместимости.
Сопоставляя эффективность амплификации с применением ПЦР-РВ и данные электрофоретического разделения ПЦР-продуктов, следует заключить, что Vent (exo-)-полимераза в условиях ПЦР способна воспринимать в качестве субстратов все исследованные производные dNTP. Производное пуринового ряда оказалось более сложным субстратом по сравнению с производными пиримидинового ряда.
PEX характеризуется длинным циклом элонгации праймера, что в условиях медленной кинетики встраивания модифицированных нуклеотидов оказывается несомненным преимуществом (рис. 6). С другой стороны, при этом способе амплификации наблюдается арифметическая прогрессия накопления продукта и, соответственно, небольшой выход. ПЦР более чувствительна к модифицированным субстратам, однако при подходящем сочетании субстрата-полимеразы выход целевого продукта будет гораздо больше, чем в PEX.
Таким образом, нами получены ДНК с полной заменой одного и нескольких природных нуклеотидов на модифицированные аналоги как пуриновой, так и пиримидиновой природы.
ВЫВОДЫ
По результатам тестирования различных полимераз в реакциях ПЦР и PEX в режимах полной замены одного пуринового нуклеотида (dAp или dAm), а также при различных комбинациях mod-dA + mod-dU и mod-dС + mod-dU наибольшую воспроизводимость результатов и больший выход полноразмерных продуктов обеспечивала полимераза Vent (exo-), которую часто выбирают для получения модифицированных аптамеров [3].
Нами показано, что полимеразами, пригодными для ферментативного получения ДНК, содержащих модифицированные основания (как пуриновые, так и пиримидиновые), следует считать ДНК-полимеразы семейства В, а также KOD XL. При этом более высокий выход полноразмерных продуктов как в ПЦР, так и в PEX, а также относительно высокие значения эффективности амплификации “Е” в ПЦР указывают на предпочтительность использования полимеразы Vent (exo-).
Расширение набора применяемых dNTP новыми производными дезоксицитидина и дезоксиаденозина позволит увеличить спектр физико-химических свойств модифицированных фрагментов ДНК и, как следствие, расширить сферу их применения в молекулярно-биологических исследованиях и медицинской диагностике.
Исследование поддержано Российским фондом фундаментальных исследований (грант № 19-04-01217).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследования.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Gold L., Ayers D., Bertino J., Bock C., Bock A., Brody E.N., Carter J., Dalby A.B., Eaton B.E., Fitzwater T., Flather D., Forbes A., Foreman T., Fowler C., Gawande B., Goss M., Gunn M., Gupta S., Halladay D., Heil J., Heilig J., Hicke B., Husar G., Janjic N., Jarvis T., Jennings S., Katilius E., Keeney T.R., Kim N., Koch T.H., Kraemer S., Kroiss L., Le N., Levine D., Lindsey W., Lollo B., Mayfield W., Mehan M., Mehler R., Nelson S.K., Nelson M., Nieuwlandt D., Nikrad M., Ochsner U., Ostroff R.M., Otis M., Parker T., Pietrasiewicz S., Resnicow D.I., Rohloff J., Sanders G., Sattin S., Schneider D., Singer B., Stanton M., Sterkel A., Stewart A., Stratford S., Vaught J.D., Vrkljan M., Walker J.J., Watrobka M., Waugh S., Weiss A., Wilcox S.K., Wolfson A., Wolk S.K., Zhang C., Zichi D. (2010) Aptamer-based multiplexed proteomic technology for biomarker discovery. PLoS One. 5, e15004.
Hollenstein M. (2012) Nucleoside triphosphates-building blocks for the modification of nucleic acids. Molecules. 17, 13569–13591.
Lapa S.A., Chudinov A.V., Timofeev E.N. (2016) The toolbox for modified aptamers. Mol. Biotechnol. 58, 79–92.
Sandin P., Stengel G., Ljungdahl T., Borjesson K., Macao B., Wilhelmsson L.M. (2009) Highly efficient incorporation of the fluorescent nucleotide analogs tC and tCO by Klenow fragment. Nucl. Acids Res. 37, 3924–3933.
Kielkowski P., Cahova H., Pohl R., Hocek M. (2016) Flexible double-headed cytosine-linked 2'-deoxycytidine nucleotides. Synthesis, polymerase incorporation to DNA and interaction with DNA methyltransferases. Bioorg. Med. Chem. 24, 1268–1276.
Gawande B.N., Rohloff J.C., Carter J.D., von Carlowitz I., Zhang C., Schneider D.J., Janjic N. (2017) Selection of DNA aptamers with two modified bases. Proc. Natl. Acad. Sci. USA. 114, 2898–2903.
Лапа С.А., Ромашова К.С., Спицын М.А., Шершов В.Е., Кузнецова В.Е., Гусейнов Т.О., Заседателева О.А., Радько С.П., Тимофеев Э.Н., Лисица А.В., Чудинов А.В. (2018) Получение модифицированных комбинаторных ДНК-библиотек методом ПЦР в обратной эмульсии с последующим разделением цепей. Молекуляр. биология. 52, 984–996.
Чудинов А.В., Шершов В.Е., Павлов А.С., Волкова О.С., Кузнецова В.Е., Заседателев А.С., Лапа С.А. (2020) Одновременное встраивание модифицированных производных dU и dC в растущую цепь ДНК в реакции удлинения праймера и ПЦР. Биоорган. химия. 46, 546–549.
Ramakers C., Ruijter J.M., Deprez R.H., Moorman A.F. (2003) Assumption-free analysis of quantitative realtime polymerase chain reaction (PCR) data. Neurosci. Lett. 339, 62–66.
Peirson S.N., Butler J.N., Foster R.G. (2003) Experimental validation of novel and conventional approaches to quantitative real-time PCR data analysis. Nucl. Acids Res. 31, e73.
Лапа С.А., Гусейнов Т.О., Павлов А.С., Шершов В.Е., Кузнецова В.Е., Заседателев А.С., Чудинов А.В. (2020) Одновременное применение Cy5-модифицированных производных дезоксиуридина и дезоксицитидина в ПЦР. Биоорган. химия. 46, 418–424.
Лапа С.А., Павлов А.С., Кузнецова В.Е., Шершов В.Е., Спицын М.А., Гусейнов Т.О., Радько С.П., Заседателев А.С., Лисица А.В., Чудинов А.В. (2019) Ферментативное получение модифицированных ДНК: изучение кинетики ПЦР в режиме реального времени. Молекуляр. биология. 53, 513–523.
Радько С.П., Лапа С.А., Чудинов А.В., Хмелёва С.А., Маннанова М.М., Курбатов Л.К., Киселёва Я.Ю., Заседателев А.С., Лисица А.В. (2019) Оценка разнообразия комбинаторных ДНК-библиотек на основе анализа формы амплификационных кривых для мониторинга эффективности селекции аптамеров. Биомед. химия. 65, 477–484.
Василисков В.А., Шершов В.Е., Мифтахов Р.А., Кузнецова В.Е., Радько С.П., Лисица А.В., Лапа С.А., Суржиков С.А., Тимофеев Э.Н., Заседателев А.С., Чудинов А.В. (2020) Эффект проскальзывания в реакции элонгации праймера при использовании модифицированных 2'‑дезоксиуридинтрифосфатов. Биоорган. химия. 46, 270–272.
Porter K.W., Tomasz J., Huang F., Sood A., Shaw B.R. (1995) N7-cyanoborane-2'-deoxyguanosine 5'-triphosphate is a good substrate for DNA polymerase. Biochemistry. 34, 11963–11969.
Rohloff J.C., Gelinas A.D., Jarvis T.C., Ochsner U.A., Schneider D.J., Gold L., Janjic N. (2014) Nucleic acid ligands with protein-like side chains: modified aptamers and their use as diagnostic and therapeutic agents. Mol. Ther. Nucl. Acids. 3, e201.
Giller G., Tasara T., Angerer B., Mühlegger K., Amacker M., Winter H. (2003) Incorporation of reporter molecule-labeled nucleotides by DNA polymerases. I. Chemical synthesis of various reporter group-labeled 2'-deoxyribonucleoside-5'-triphosphates. Nucl. Acids Res. 31, 2630–2635.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология