Молекулярная биология, 2022, T. 56, № 2, стр. 206-226
Роль белков, взаимодействующих с факторами терминации трансляции eRF1 и eRF3, в регуляции трансляции и прионизации
Г. А. Журавлева a, b, *, С. А. Бондарев a, b, О. М. Землянко a, b, С. Е. Москаленко a, c
a Кафедра генетики и биотехнологии Санкт-Петербургского государственного университета
199034 Санкт-Петербург, Россия
b Лаборатория биологии амилоидов Санкт-Петербургского государственного университета
199034 Санкт-Петербург, Россия
c Санкт-Петербургский филиал Института общей генетики им. Н.И. Вавилова Российской академии наук
199034 Санкт-Петербург, Россия
* E-mail: g.zhuravleva@spbu.ru
Поступила в редакцию 21.07.2021
После доработки 12.08.2021
Принята к публикации 15.08.2021
- EDN: ZOTXUR
- DOI: 10.31857/S002689842201013X
Аннотация
В обзоре рассмотрена роль белков, взаимодействующих с факторами терминации трансляции eRF1 и eRF3, в контроле синтеза белка и прионизации. Эти белки взаимодействуют не только друг с другом, но и со многими другими белками, которые контролируют эффективность терминации трансляции, а также осуществляют связь этого процесса с другими клеточными процессами. Терминация трансляции сопряжена не только с реинициацией трансляции и рециклингом рибосом, но также со стабильностью мРНК и с клеточной системой контроля качества белков. Эту связь обеспечивает взаимодействие eRF1 и eRF3 с белками, участвующими в различных метаболических процессах, таких как транспорт мРНК из ядра в цитоплазму (белки Dbp5/DDX19 и Gle1), рециклинг рибосом (белок Rli1/ABCE1), деградация мРНК (белки Upf), инициация трансляции (белок Pab1/PABP). Существует не только генетический, но и эпигенетический контроль терминации трансляции. Он связан с прионной полимеризацией белка Sup35, приводящей к образованию приона [PSI+]. Для поддержания фактора [PSI+], как и других дрожжевых прионов, необходима работа системы молекулярных шаперонов и факторов сортировки белков. В нашем обзоре подробно обсуждается взаимодействие факторов терминации трансляции с белками, участвующими в различных клеточных процессах.
Биосинтез белка – один из ключевых процессов живой клетки. Как и все матричные процессы, он включает три основные стадии: инициацию, элонгацию и терминацию. Основными участниками терминации трансляции у эукариот являются белки eRF1 и eRF3, кодируемые у дрожжей Saccharomyces cerevisiae генами SUP45 и SUP35 соответственно. Поэтому у дрожжей часто используют оба обозначения этих белков: Sup45, или eRF1 и Sup35, или eRF3 (см. обзор [1]). Сохранению названия Sup35 способствует то, что агрегация белка Sup35 в клетках дрожжей приводит к образованию приона [PSI+]. В клетках с прионом этот белок формирует амилоидные агрегаты [2–5]. Это приводит к снижению уровня мономерного eRF3, способного образовывать комплекс с eRF1. Следом за этим падает эффективность терминации трансляции, а вероятность прочтения преждевременного стоп-кодона как значащего (эффективность нонсенс‑супрессии) увеличивается (см. обзор [6]).
Белки eRF1/Sup45 и eRF3/Sup35 в комплексе осуществляют узнавание стоп-кодона и GTP-зависимое высвобождение вновь синтезированного полипептида. Нарушение точности процесса терминации приводит к “прочтению” преждевременных стоп-кодонов как значащих, что в простых генетических системах может регистрироваться в виде омнипотентной супрессии. Помимо омнипотентной нонсенс-супрессии, мутации в дрожжевых генах SUP35 и SUP45 характеризуются многочисленными плейотропными эффектами, в том числе летальностью при повышенной или пониженной температуре, полной или частичной неспособностью к дыханию, чувствительностью к повышенному осмотическому давлению и аминогликозидным антибиотикам (см. обзор [1]). Возможно, что часть этих эффектов связана с участием белков eRF1 и eRF3 в других процессах. Терминация трансляции прямо связана не только с реинициацией трансляции и рециклингом рибосомы, но и со стабильностью мРНК, а также с клеточной системой контроля качества белков. Эту связь обеспечивает взаимодействие белков eRF1 и eRF3 с белками, участвующими в различных метаболических процессах, таких как транспорт мРНК из ядра в цитоплазму (Dbp5/DDX19 и Gle1), рециклинг рибосом (Rli1/ABCE1), деградация мРНК (белки Upf), инициация трансляции (Pab1/PABP) и другие. Обсуждению этой темы, а также белкам, взаимодействующим с прионом [PSI+], и посвящен данный обзор.
ТЕРМИНАЦИЯ ТРАНСЛЯЦИИ, БЕЛКИ eRF1 И eRF3
Терминация трансляции в клетках эукариот осуществляется с помощью основных факторов терминации классов I и II – eRF1 и eRF3, способных образовывать прочный комплекс, что показано в экспериментах in vivo и in vitro [7, 8]. У белков eRF1 и eRF3 обнаружены паралоги Dom34 (дрожжи)/Pelota (млекопитающие) и Hbs1 соответственно, которые не участвуют в терминации, но играют важную роль в процессах, происходящих при нарушении биосинтеза белка [9]. Реконструкция структуры с помощью криоэлектронной микроскопии выявила сходство комплексов eRF1–eRF3 и Dom34–Hbs1 с элонгационным комплексом между аа-тРНК и eEF1A [10–12].
Эукариотический фактор eRF1 (фактор терминации класса I) распознает все три известных стоп-кодона: UAA, UAG, UGA [13], и запускает гидролиз эфирной связи пептидил-тРНК в пептидилтрансферазном центре рибосомы.
С помощью рентгеноструктурного анализа определена пространственная структура eRF1 человека, который имеет Y-образную форму и делится на три домена: N (1–142), M (143–275) и C (276–437) [14] (рис. 1а). Согласно модели молекулярной мимикрии [15], положение этих доменов в пространстве соответствует антикодоновой петле, акцепторному стеблю и Т-петле тРНК. Выявлены консервативные аминокислотные остатки, расположенные в N-концевом домене белка и важные для узнавания стоп-кодонов (см. обзор [16]). Методом ядерного магнитного резонанса в С-домене выделен также подвижный минидомен (ак 329–372), который существует в двух возможных конформациях и, вероятно, может сближаться с распознающим стоп-кодоны участком N-домена eRF1 [17]. Предполагается, что этот участок может влиять на эффективность терминации трансляции. В М-домене белка eRF1 находится мотив GGQ (ак 183–185 белка человека), участвующий в гидролизе пептидил-тРНК [18]. Также показано взаимодействие между М-доменом eRF1 и фактором eRF3.
Рис. 1.
Взаимодействие факторов терминации трансляции eRF1 и eRF3. а –Структура белка eRF1/Sup45: домены N, М и C. Наверху приведена консенсусная последовательность eRF1 [10], участвующая во взаимодействии с eRF3. Аминокислотные остатки eRF1, непосредственно вовлеченные во взаимодействие, отмечены звездочками: F288 (291), I291 (294), Y298 (301), F300 (303), Q396 (397), Q400 (401), F405 (406) у S. pombe (в скобках показаны номера соответствующих аминокислотных остатков у H. sapiens). б – Структура белка eRF3/Sup35. У eRF3 S. pombe и H. sapiens (прионизация которых не обнаружена) домены N и М объединены в один N-концевой домен (обозначен фигурной скобкой). С-концевой домен подразделяют на субдомены: G (или GTP-связывающий домен), 2 и 3. Внизу показана консенсусная последовательность eRF3, участвующая во взаимодействии с eRF1 [10]. Аминокислотные остатки eRF3, прямо связывающие eRF1, отмечены звездочками – S571 (542), I572 (543), Y577 (548), F612 (584), D647 (619) у S. pombe (в скобках показаны номера соответствующих аминокислотных остатков в белке H. sapiens). Здесь и на рис. 2–4 основные взаимодействующие участки показаны сплошными линиями, дополнительные – пунктиром. Номерами указаны положения аминокислот в белках соответствующих видов. Домены показаны не в масштабе.
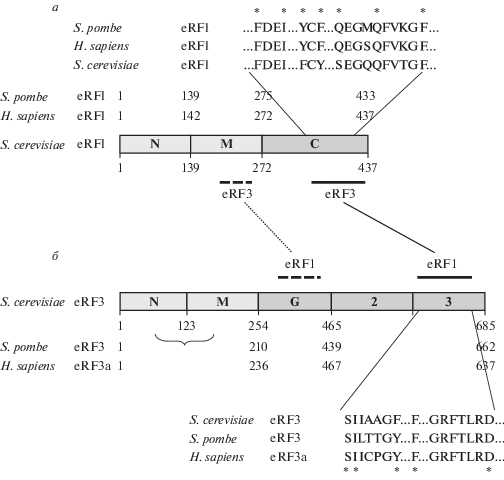
С-концевой домен eRF1 отвечает за связывание фактора eRF1 с С-концевым доменом eRF3 [7, 8, 19–21]. С использованием делеционного анализа в eRF1 дрожжей S. cerevisiae выявлен участок, взаимодействующий с eRF3 (ак 411–418), показано также, что делеция 32 С-концевых остатков eRF1 летальна для клетки, в то время как делеция 19 аминокислот приводит к нарушению эффективности терминации трансляции in vivo [22]. В состав С-домена eRF1 входят также участки взаимодействия с рибосомой [22]. В исследованиях на дрожжах S. pombe установлена важная роль М- и С-доменов eRF1 [19]. Для взаимодействия eRF1 человека с eRF3 необходимы два участка, локализованные в С-концевом домене [20]. Аминокислотные остатки, расположенные между этими участками С-домена eRF1 человека (ак 338–381), взаимодействуют с каталитической субъединицей фосфатазы PP2A [23], однако значение данного взаимодействия не до конца понятно. Предполагается, что взаимодействие eRF1–PP2A может играть важную роль в процессе NMD (Nonsense-Mediated mRNA Decay) [10]. Сам С‑домен eRF1 не стимулирует GTPазную активность eRF3, тогда как при добавлении М-домена происходит гидролиз GTP [24].
Белок eRF3 является GTPазой, стимулирующей работу фактора eRF1. Белок eRF3 связывается с GTP только в присутствии eRF1 и Mg2+, поэтому комплекс eRF1–eRF3–GTP–Mg2+ попадает в A-сайт рибосомы, что приводит к формированию претерминационного комплекса [25–27]. Для GTPазной активности eRF3 требуются eRF1 и рибосомы [28, 29].
У млекопитающих, в отличие от дрожжей, есть два подсемейства eRF3: eRF3a и eRF3b, которые кодируются генами GSPT1 и GSPT2 соответственно [30, 31].
Белок Sup35 S. cerevisiae состоит из трех доменов: N, M и C, за начало которых принято считать три остатка метионина в положениях 1, 124 и 254 соответственно (рис. 1б) [32]. Функции N-концевого домена (кроме роли в прионизации у дрожжей, см. далее) ясны не до конца, но известно, что он специфически взаимодействует с поли(А)-связывающим белком Pab1/PABP [33]. Также NM-домен Sup35 взаимодействует с РНК-связывающим белком Pub1 (Tia1 – его гомолог у человека), участвующим в формировании стресс-гранул [34].
С-домен eRF3 гомологичен фактору элонгации трансляции eEF1A, он высоко консервативен у эукариот, в то время как N- и M-домены очень вариабельны (см. обзор [1]). С помощью делеционного анализа показано, что С-домен eRF3 взаимодействует с С-концевым районом eRF1 [8, 22, 35]. Благодаря рентгеноструктурному анализу удалось установить трехмерную структуру С-концевого домена eRF3 из S. pombe и подразделить его на три субдомена: G (или GTP-связывающий), 2 и 3 (рис. 1б). Во взаимодействии с eRF1 участвует мотив GRFTLRD, локализованный в субдомене 3. Интересно, что аминокислотные остатки 215–236 в М-домене eRF3 S. pombe ингибируют взаимодействие между факторами [36].
Детальное понимание процессов, которые происходят в ходе взаимодействия факторов терминации трансляции, пришло в результате решения кристаллических структур комплекса eRF1 с субдоменами 2 и 3 eRF3 S. pombe [10]. Показано, что во взаимодействии eRF1–eRF3 участвуют гидрофобные аминокислотные остатки eRF1 (F288, I291, Y298, F300, F405) и eRF3 (I572, F612) (рис. 1). Кроме того, белки контактируют между собой за счет ван-дер-ваальсового взаимодействия между Q396 и Q400, в eRF1, I572 и D647 в eRF3. В результате этого взаимодействия изменяется положение М-домена eRF1, который перемещается в пептидилтрансферазный центр таким образом, что расстояние между GGQ-мотивом и участком eRF1, узнающим стоп-кодон, становится равным расстоянию между антикодоном и последовательностью ССА в тРНК (переход из “открытого” в “закрытое” состояние). Таким образом, во взаимодействии факторов eRF1 и eRF3 принимают участие преимущественно С-концевые домены белков, контактируют также M-домен eRF1 и G-домен eRF3, причем в этом взаимодействии важная роль принадлежит GTP.
К настоящему времени методами криоэлектронной микроскопии получены структуры комплексов факторов терминации с рибосомой [37, 38]. Согласно предложенной модели терминации трансляции, на первом этапе eRF1 связывается с eRF3 или eRF3–GDP за счет их С-доменов. Далее происходит обмен GDP на GTP, так как в клетке его концентрация выше, и комплекс eRF1–eRF3 имеет большее сродство к GTP. Образование первичного комплекса приводит к сближению М-домена eRF1 и G-домена eRF3. Это, в свою очередь, стабилизирует неструктурированные участки в G-домене eRF3. Связывание комплекса eRF1–eRF3–GTP с рибосомой приводит к попаданию N‑домена eRF1 в район декодирующего центра рибосомы, а GGQ-мотива eRF1 в пептидилтрансферазный центр. Далее в результате значительных конформационных изменений eRF1 вместе с GTP-активирующим центром рибосомы стимулирует GTPазную активность eRF3. После этого GGQ-мотив располагается таким образом, что возможным становится гидролиз пептидил-тРНК. В рибосоме факторы терминации взаимодействуют не только друг с другом, но также с рРНК и рибосомными белками. Так eRF1, кроме образования водородных связей со стоп-кодоном, взаимодействует с 40S и 60S рРНК, а также с белками rpS30e, rpS31e, rpS23e, rpL23e и rpL12e, а eRF3 связывается с SRL-петлей 60S рРНК и белками rpL23e, rpL40e, rpL9e [38].
БЕЛКИ, ВЗАИМОДЕЙСТВУЮЩИЕ С eRF1 И/ИЛИ eRF3
Белки UPF
Белки Upf (UP-frameshift) участвуют в деградации транскриптов, содержащих преждевременные стоп-кодоны. Этот процесс, названный NMD, обнаружен у различных эукариот. В настоящее время не существует единой модели NMD, описывающей его прохождение у разных организмов. У S. cerevisiae идентифицированы три гена UPF, необходимых для NMD [39–41], в то время как у Caenorhabditis elegans в этом процессе участвуют семь генов smg (Suppressors with Morphological defects on Genitalia) [42, 43]. Гены smg2, smg3 и smg4 являются ортологами генов UPF1, UPF2 и UPF3 S. cerevisiae соответственно [44, 45]. Основное отличие дрожжей от многих эукариотических организмов – отсутствие гена SMG1, кодирующего киназу, участвующую в образовании комплекса SURF (от SMG, UPF1 и eRF) на первом этапе NMD [46]. Предполагается, что ген SMG1 был потерян в линии грибов уже после их дивергенции от линии животных [47]. Согласно данным филогенетического анализа, большинство компонентов комплекса SURF консервативны у основных групп эукариот, что позволило предположить возможность присутствия SURF и у последнего общего предка эукариот (LECA, Last Eukaryotic Common Ancestor) [48]. Некоторые авторы выделяют четыре основных типа NMD [49]. По этой классификации дрожжи S. cerevisiae обладают NMD третьего типа (древний SMG1-независимый путь NMD). Несмотря на различия между типами NMD, у разных организмов в этом процессе участвуют три основных белка (Upf1, Upf2 и Upf3), которые взаимодействуют друг с другом (см. обзор [50]), а также с полисомами [51].
Upf1
У разных организмов ген UPF1 может иметь разные названия: NORF1 (NOnsense mRNA Reducing Factor 1), RENT1 (Regulator of Nonsense Transcripts 1), или SMG2. У дрожжей ген UPF1 не относится к жизненно важным [41]. Белок Upf1 содержит два основных домена: в N-концевой части находится СН-домен (Cysteine- and Histidine-rich), в С-концевой части расположен хеликазный (ATPase/helicase) домен [41, 52, 53] (рис. 2). Upf1 – это мультифункциональный белок, участвующий как в контроле стабильности мРНК, так и в трансляции. Делеция UPF1 приводит к супрессии нонсенс-мутаций [41]. Впоследствии предположили, что нонсенс-супрессорный фенотип клеток upf1Δ может быть связан не только с дефектами терминации трансляции, но и с регуляцией внутриклеточного содержания ионов магния из-за нарушения экспрессии гена ALR1 вследствие сбоев в NMD [54]. Уже в первых работах, в которых изучали белки, участвующие в NMD, обнаружили, что белок Upf1 взаимодействует с белком Nmd2 (Upf2) [55], который может обеспечивать взаимодействие Upf1 с Upf3 [56]. С помощью мутационного анализа удалось отделить участки Upf1, необходимые для NMD, от участков, влияющих на терминацию трансляции [53, 57]. Мутации в участке UPF1, кодирующем хеликазный домен, блокировали участие Upf1 в NMD, но не влияли на нонсенс-супрессию [53], в то время как изменения в СН-домене приводили к супрессии нонсенс-мутаций [57]. Эти результаты позволили предположить прямое участие Upf1 в терминации трансляции, что подтверждено с помощью различных методов. С использованием аффинной хроматографии выявлено взаимодействие Upf1 с факторами терминации eRF1/Sup45 и eRF3/Sup35; белок Upf1 (наряду с Sup45) также присутствует в агрегатах Sup35, формирующихся в клетках [PSI+] [58]. Уменьшение содержания Upf1 приводило к дефектам терминации трансляции в клетках человека [59].
Рис. 2.
Взаимодействие факторов терминации трансляции eRF1 и eRF3 с белком Upf1. а – Структура белка eRF1/Sup45: N, М и C – домены. б – Структура белка Upf1. Расположение мутаций (или концов делеций) показано над схемой белка. Домен CH (Сysteine/Нistidine) участвует во взаимодействии с eRF3 [59], Upf2 [56, 65], Pab1 [66], Rps26 [67]. В нем локализованы мутации, приводящие к супрессии нонсенс-мутаций, в том числе и мутация C122S [57]. С eRF1 взаимодействует участок 289–555 Upf1 [60]. Мутация K436A нарушала взаимодействие Upf1 с eRF1 [41]. Мутация UPF1-D4 влияла на NMD [41], но не на взаимодействие с полисомами [51]. в – Структура белка eRF3/Sup35. C-концевая часть eRF3 взаимодействует с Upf1 (ак 254–465), а также с eRF1, Upf2 и Upf3 (ак 465–685) [61]. Обозначения как на рис. 1.
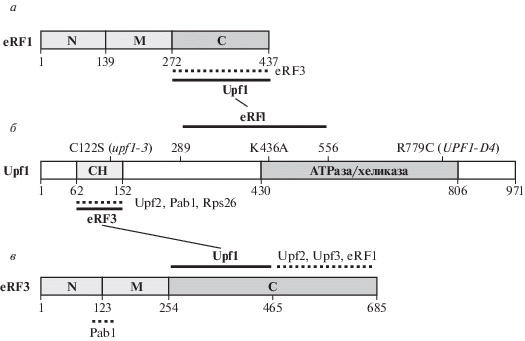
Во взаимодействии с Upf1 участвует С-концевой домен eRF1 (ак 270–437 в белке человека) [59] (рис. 2). Этот домен, по-видимому, связывается с АТРазным доменом Upf1: взаимодействующий участок Upf1 локализован в районе 289–555 ак [60], мутация K436A нарушала взаимодействие Upf1pK436A с eRF1 [58] (рис. 2). Во взаимодействии eRF3 с Upf1 участвуют CH-домен Upf1 [59] и С‑концевой домен eRF3, при этом сайт взаимодействия находится в начале GTPазного домена и не перекрывается с участками взаимодействия eRF3 с eRF1, Upf2 и Upf3 [61] (рис. 2). Показано, что и у дрожжей, и у человека с Upf1 связывается С-концевой участок eRF3 (ак 200–637) [59], а для ассоциации комплекса Upf1–РНК с eRF3 необходима АТР [58]. Мутация в UPF1, которая нарушала связывание АТР (K498Q в белке Upf1 человека), приводила к нарушению взаимодействия Upf1 с eRF3 [46]. Гидролиз АТР вызывал быструю диссоциацию комплекса, состоящего из рибосом, РНК и белков Upf, что приводило к эффективной терминации трансляции [62].
Таким образом, исследования, проведенные с использованием различных систем in vivo, показали, что Upf1 прямо связывается с факторами терминации eRF1 и eRF3. При этом результаты, полученные in vitro, предполагают, что Upf1 человека не участвует в элонгации и терминации трансляции, а также в рециклинге рибосом [63]. Подобные различия можно объяснить тем, что в системе in vitro отсутствуют дополнительные факторы. Высказано предположение, что белок Upf1 человека связывается с факторами терминации трансляции не напрямую, а за счет белков-посредников, например, Upf3b [64]. Противоречия между данными, согласно которым Upf1 дрожжей прямо участвует в терминации трансляции и освобождении рибосом на преждевременных стоп-кодонах [62], и результатами, полученными на реконструированной системе in vitro [64], по-видимому, можно объяснить различиями в системах NMD у низших и высших эукариот.
Upf2
Ген UPF2 (он же RENT2, или SMG3 у разных видов) в клетках дрожжей, где его чаще называют NMD2, не является жизненно важным [55, 68]. Белок Upf2 содержит несколько доменов MIF4G (Middle domain of translation Initiation Factor 4G), точное число которых трудно определить из-за вырожденности последовательности. Например, в белке Upf2 S. cerevisiae одни авторы идентифицируют два таких домена [69], другие – три [70]. Во взаимодействии Upf2 с Upf3 участвует один из доменов MIF4G (рис. 3) [56], в то время как с Upf1 связывается С-концевой домен Upf2 [71]. С фактором терминации eRF3 взаимодействуют как Upf2 дрожжей (установлено с использованием очищенных белков в системе in vitro) [61], так и человека [72]. В дрожжевой системе белки Upf2, Upf3 и eRF1 конкурируют друг с другом за связывание с eRF3 [61]. Взаимодействие между Upf2 и Upf3 человека является более сильным, чем Upf2 и eRF3. Так как в этих взаимодействиях принимают участие перекрывающиеся участки Upf2, то предполагается, что в ходе NMD белок Upf2 может связываться с различными компонентами этого процесса, включая Upf1, Upf3 и eRF3 [72].
Upf3
Рис. 3.
Взаимодействие между фактором терминации трансляции eRF3, белками Upf2 и Upf3. Границы доменов Upf2 и Upf3 изображены согласно https://www.yeastgenome.org. а – Структура белка Upf2. Границы доменов MIF4G: 35–241; 377–557; 579–817, С-концевого домена (С-терм) 961–1085. Участки взаимодействия Upf2 с Upf1 (933–1061 ак) соответствуют данным [71]; Upf2 с Upf3 (564–923 Upf2) показаны по [56]; Upf2 с eRF3 (ак 886–938) по [61]. б – Структура белка eRF3/Sup35. C-концевая часть eRF3, или GTPазный домен, взаимодействует с Upf3 (ак 465–685), а также с eRF1, Upf1 и Upf2 [61]. в – Структура белка Upf3. Границы RRM-домена 87–189. Участки взаимодействия Upf3 с Upf2 (ак 78–278) соответствуют данным [56], UPF3b человека связывается с eRF3 посредством RRM-домена [72]. Обозначения как на рис. 1.
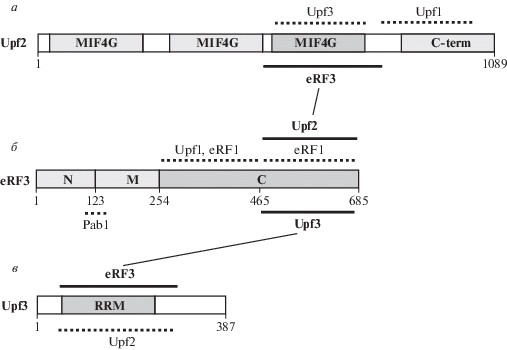
Ген UPF3 у разных видов может называться также RENT3 или SMG4. В клетках человека представлены две изоформы белка Upf3: Upf3a и Upf3b [73]. У дрожжей ген UPF3 не относится к жизненно важным [41]. Белок Upf3 содержит сигналы ядерной локализации (Nuclear Localization Signals, NLS) и ядерного экспорта (Nuclear Export Signals, NES), что позволяет ему перемещаться между ядром и цитоплазмой [74, 75]. Белок Upf3 дрожжей прямо связывается с Upf2, тогда как взаимодействие с Upf1 обусловлено, по-видимому, белком Upf2 [56]. Этот вывод подтверждается данными о том, что делеция гена NMD2 (UPF3) полностью нарушала взаимодействие между Upf1 и Upf3. В то же время показано прямое связывание белков Upf3b и Upf1 человека [64]. Белок Upf3b также взаимодействует c eRF3a, при этом участки связывания Upf3 с Upf2 и eRF3a перекрывались и соответствовали домену RRM [72] (рис. 3). Во взаимодействии с Upf3 участвует C‑концевая часть eRF3 дрожжей (ак 465–685) [61], что согласуется с данными [72], согласно которым Upf3b связывается с обоими вариантами eRF3 (eRF3a и eRF3b млекопитающих). Белки eRF3a и eRF3b характеризуются высокой степенью сходства С-концевых доменов и полностью различаются своими N-доменами [30]. Поскольку продуцируемый в клетках человека белок eRF3a агрегировал, поэтому в работе [72] использовали eRF3b. При этом показано взаимодействие полноразмерного eRF3a, но не его варианта, делетированного с N-конца, с Upf3 [64]. В этом случае eRF3a синтезировали в бакуловирусной системе экспрессии. Таким образом, существующие противоречивые данные не позволяют однозначно определить роль Upf3 в терминации трансляции.
Согласно нашим данным, проявления нонсенс-мутаций в генах SUP45 и SUP35 на фоне делеции гена UPF3 отличаются от проявлений на фоне делеций генов UPF1 или UPF2. Как правило, мутанты sup45-n и sup35-n характеризуются пониженной жизнеспособностью за счет уменьшения содержания факторов терминации трансляции eRF1 или eRF3, кодируемых генами SUP45 или SUP35 соответственно [21, 76]. Делеция гена UPF1 или UPF2 приводила к повышению жизнеспособности таких мутантов в отличие от клеток с делецией гена UPF3 [60, 77, 78]. Известно, что белки eRF1 и eRF3 ингибируют АТРазную активность Upf1 [58], в то время как комплекс Upf1–Upf2 эту активность стимулирует [79]. По-видимому, снижение содержания факторов терминации в клетке приводит к стимуляции АТРазной активности Upf1 и препятствует терминации трансляции. В отсутствие Upf1 (или его активатора Upf2) происходит более эффективное образование комплекса терминации трансляции и, как следствие, повышается жизнеспособность клеток с мутациями в генах SUP45 или SUP35. Это предположение подтверждается данными о повышении устойчивости мутантов sup45-n к паромомицину [77].
PABP
К особенностям большинства эукариотических мРНК относится присутствие поли(A)-последовательностей на 3'-конце молекулы. Консервативные белки, способные связываться с поли(A)-участками, получили название РАВР (poly(A) binding proteins). Впервые цитоплазматический РАВРС (C – Cytoplasmic), играющий важную роль в трансляции и деградации мРНК, описали в 1973 г. [80], а вскоре соответствующие гены обнаружили у различных эукариот, в том числе у S. cerevisiae [81, 82]. Как правило, одноклеточные эукариоты имеют один ген, кодирующий РАВР (Pab1 у S. cerevisiae). Клетки многоклеточных животных и растений содержат несколько генов РАВР (см. обзоры [83, 84]). В клетках млекопитающих идентифицированы три группы генов PABP: один ядерный (PABPN1), три цитоплазматических (PABPC1, PABPC3 и iPABP), в отдельную группу выделяют PABPC5, локализованный в Х-хромосоме; кроме того, известно четыре псевдогена.
Ген PAB1 является жизненно важным, но делеция участка, кодирующего С-концевой домен белка Pab1, не нарушает жизнеспособность клеток дрожжей [82, 85]. В N-концевой части белка Pab1 находятся четыре тандемных повтора RRM, которые соединяются короткими линкерными последовательностями. Высоко консервативный С-концевой домен (также называемый MLLE, см. далее) белка Pab1 человека состоит из пяти α‑спиралей, в то время как в РАВРС дрожжей только четыре α-спирали [86]. Известно, что этот домен РАВР участвует в присоединении различных белков, регулирующих полиаденилирование, деаденилирование, инициацию и терминацию трансляции.
Размер поли(A)-участка мРНК у млекопитающих и дрожжей составляет 200–250 и 70–90 н. соответственно. В полиаденилировании мРНК участвует большой белковый комплекс размером 500–1000 кДа, включающий поли(А)-полимеразу РАР (Poly(A) Polymerase) и множество дополнительных факторов, которые регулируют процесс полиаденилирования и определяют длину поли(A)-участка (см. обзор [83]).
После попадания мРНК в цитоплазму PABP, связанный с поли(A)-участком, стимулирует контакт между 5'- и 3'-концами мРНК за счет взаимодействия с eIF4G [87, 88]. Существование регуляторной связи между PABP, eIF4G и eIF4E согласуется с нарушением трансляции в штаммах дрожжей со сниженным содержанием Pab1 [89], а также с тем, что присутствие кепа и поли(А)-участка приводит к синергической стимуляции трансляции [90–92]. При этом создается возможность реинициации трансляции и рециклирования рибосом на одной и той же мРНК за счет взаимодействия ее 3'- и 5'-концов. Исследования, проведенные на клетках дрожжей и человека, показали, что для взаимодействия Pab1 с eIF4G необходимы домены RRM1, RRM2 Pab1 и N-концевой домен eIF4G [92–96].
Взаимодействие Pab1 с фактором eRF3 указывает на его прямое участие в терминации трансляции. Это взаимодействие консервативно в эволюции, оно выявлено у самых разных организмов – от одноклеточных эукариот [33] до многоклеточных организмов [97–100]. Сверхпродукция Pab1 приводит к увеличению эффективности терминации трансляции в клетках дрожжей [33], в то время как уменьшение содержания PABPC1 способствует прочтению стоп-кодона UGA в клетках человека [59]. Предполагается, что PABP увеличивает точность распознавания стоп-кодона за счет участия в правильной ориентации eRF3a в рибосоме [101].
Во взаимодействии белков eRF3 и Pab1 участвуют NМ-домены eRF3 и С-домен Pab1 (рис. 4). В то время как белки семейства eRF3 многоклеточных эукариот содержат консервативную последовательность PAM2 (PABP-interacting Motif 2) [102], NM-домен eRF3 дрожжей значительно отличается от своих гомологов, и идентифицировать последовательность PAM2 у него достаточно сложно. Согласно данным делеционного анализа, во взаимодействии с Pab1 участвует последовательность 69–239 ак белка eRF3 дрожжей [33]. Выравнивание последовательностей гомологов eRF3 с последующим анализом их вторичной структуры позволяет считать участок 110–121 ак белка eRF3 дрожжей потенциальным сайтом PAM2 (рис. 4а). Интересно, что аллели гена GSPT1 человека, кодирующие варианты eRF3a, различающиеся числом остатков глицина в N-концевом домене, характеризуются различной эффективностью взаимодействия с PABP. При этом сила взаимодействия варианта eRF3a “12-GGC”, ранее ассоциированного с развитием опухолей человека [103–105], была значительно ниже, чем у других вариантов [106]. Это достаточно неожиданные данные, так как глициновые повторы находятся вне участка взаимодействия eRF3a с PABP.
Рис. 4.
Взаимодействие между фактором терминации трансляции eRF3 и белком Pab1. Выравнивание аминокислотных последовательностей получено с использованием программы MULTALIN (http://npsa-pbil.ibcp.fr). Под аминокислотными последовательностями приведена вторичная структура, предсказанная алгоритмом SOPM [118]: h – участки альфа-спиралей; c – random coil; e – extended strand; t – beta turn. а – Структура белка eRF3 (Sup35) человека. Подчеркнут консенсусный сайт РАМ2 (LNVNAKPFVP), участвующий во взаимодействии с PABP [107], F76 выделен более крупным шрифтом, стрелкой показан сайт процессинга в eRF3а [112]. б – Структура белка Pab1. Во взаимодействиях с Upf1 и eIF4G участвуют RRM1 и RRM2 [96]. MLLE, или С-концевой домен, взаимодействует с eRF3 и рядом других белков (см. обзор [108]). Остальные обозначения как на рис.1.
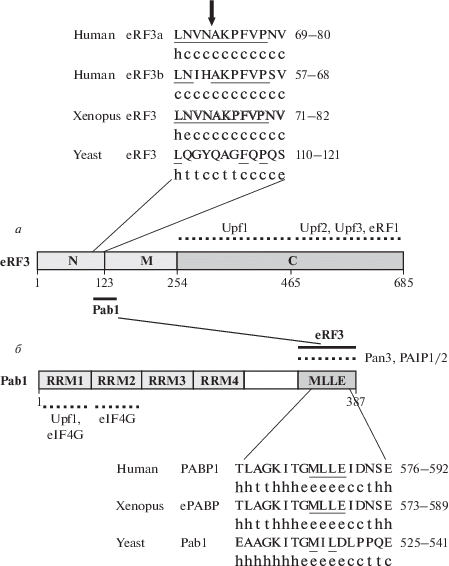
С-концевая часть белка РАВР содержит консервативный участок, который принято называть доменом MLLE (Mademoiselle) из-за четырех представленных в нем консервативных остатков MLLE [107] (рис. 4б). Показано, что этот пептид участвует в узнавании PAM2, найденного во многих эукариотических белках (см. обзор [108]). Особенность eRF3 высших эукариот – две перекрывающиеся последовательности PAM2 (PAM2-N и PAM2-C), способные связываться с доменом MLLE белка PABPC1 [109, 110]. По данным рентгеноструктурного анализа пептиды PAM2-C и PAM2-N eRF3 человека содержат общий остаток F76, необходимый для связывания каждой из этих последовательностей с доменом MLLE [111].
Белок eRF3а человека может подвергаться процессингу, в результате которого отщепляется N-концевой фрагмент размером 72 ак, что приводит к экспозиции консервативного сайта, участвующего во взаимодействии с белками-ингибиторами апоптоза (рис. 4а) [112]. Предполагается, что частично нарушенный при этом сайт РАМ2 будет обеспечивать взаимодействие с PABPC1, хотя и с меньшей эффективностью [111].
Консервативные последовательности PAM2 обнаружены более чем у 20 белков человека (см. обзор [108]). Участие eRF3 в различных процессах, в том числе в деаденилировании мРНК, можно, по-видимому, объяснить конкуренцией за связывание с РАВР1 [113–115]. У дрожжей Pab1 взаимодействует с нуклеазой Pan3 [116], в то время как у высших эукариот он связывается и с другими белками, участвующими в деаденилировании мРНК (см. обзор [108]). Предполагается, что комплексы деаденилаз конкурируют с eRF3 за связывание с PABPC1, при этом перекрывающиеся мотивы РАМ2 в eRF3 обеспечивают значительно более эффективное взаимодействие с доменом MLLE по сравнению с другими белками [111]. К конкурентам связывания между eRF3 и PABPC1 могут относиться также белки PAIP1 и PAIP2 (PABP Interacting Proteins), содержащие консервативные участки, взаимодействующие с PABPC1 – РАМ1 и РАМ2 [108, 117].
Rli1/ABCE1
Освобождение посттерминационной рибосомы с мРНК и ее возврат в новый раунд трансляции называется рециклингом. В первом этапе этого процесса после освобождения синтезированного белка участвуют белки eRF1 и Rli1 (ABCE1 у человека). В результате происходит диссоциация рибосомы на субъединицы. Несмотря на то, что факторы терминации трансляции сами способны запускать рециклинг in vitro, добавление Rli1 значительно увеличивает скорость реакции за счет гидролиза АТP [119, 120]. Белки Rli1/ABCE1 это цитоплазматические АТPазы семейства ABC, которые содержат два нуклеотидсвязывающих домена и N-концевой железо-серный домен, необходимый для функционирования белка [121]. Rli1 взаимодействует с факторами терминации трансляции как in vitro, так и in vivo [119, 122]. Считается, что белок ABCE1 связывается с С-доменом eRF1 в рибосоме и стабилизирует его “открытую” конформацию, необходимую для позиционирования центрального домена в пептидилтрансферазном центре рибосомы. Далее следует высвобождение синтезированного пептида и стадия АТP-зависимой диссоциации рибосомы на субчастицы, опосредованной белком ABCE1 [123].
Dbp5/DDX19 и Gle1-IP6
Белок Dbp5 дрожжей (DEAD-box protein 5), также известный как Rat8 (Ribonucleic acid-trafficking protein 8) и его гомолог DDX19 у человека, консервативные у эукариот, принадлежат к семейству АТР-зависимых РНК-xеликаз, содержащих мотив DEAD [124, 125]. Белок Dbp5 имеет N‑концевой домен с неизвестной функцией и два RecA подобных домена, в которых расположены участки взаимодействия с РНК и АТР, что характерно для многих представителей семейства белков DEAD-бокс (см. обзор [126]).
Белок Dbp5 и его гомологи участвуют в экспорте мРНК из ядра в цитоплазму. Считается, что Dpb5 взаимодействует с мРНK-белковым комплексом уже во время транскрипции, так как он связывается с комплексом TFIIH [127]. В околоядерном пространстве белок Dbp5/DDX19 взаимодействует с цитоплазматическими филаментами комплекса ядерных пор через N-концевой домен нуклеопорина Nup159 у дрожжей или Nup214 у человека [128].
В 2007 году установили роль белка Dbp5 в терминации трансляции в клетках дрожжей [129]. Оказалось, что белок Dbp5 находится во фракции полисом, где идет активный синтез белка. Мутанты по гену Dbp5 (rat8-2, rat8-3) чувствительны к ингибиторам трансляции паромомицину, гигромицину В и циклогексимиду. Сверхпродукция белка Dbp5 также приводит к нонсенс-супрессии у мутантов по генам SUP45 и SUP35. Методом иммунопреципитации доказано физическое взаимодействие между eRF1 и Dbp5, и этот комплекс не разрушался при обработке РНКазой. На основании полученных данных предположили, что белок Dbp5 ремоделирует мРНК-белковый комплекс и позиционирует белок eRF1 в районе декодирующего центра. Согласно предложенной модели, белок eRF3 связывается с eRF1 в рибосоме после диссоциации Dbp5 [129].
Белок Gle1, как и Dbp5, представляет собой фактор экспорта поли(А)+-мРНК из ядра в цитоплазму. Этот белок впервые обнаружили у дрожжей S. cerevisiae [130], позднее его гомологи нашли у других эукариот, в том числе белок hGle1 человека [131]. Gle1 дрожжей активирует белок Dbp5 во время экспорта мРНК [132]. Он, как и Dbp5, связывается на цитоплазматической стороне ядерной поры путем взаимодействия с нуклеопорином Nup42. Белок DDX19 человека связывается с нуклеопоринами Nup155 и Nup42 [133]. Для эффективной работы Gle1 в ядерном комплексе требуется молекула инозитол-1,2,3,4,5,6-гексакисфосфата (IP6). Таким образом, Gle1-IP6 вместе с Dbp5 ремоделирует мРНК-белковый комплекс для его направленного экспорта через ядерную пору. Третичная структура комплекса между Dbp5 и Gle1 очень похожа на структуру других белков, участвующих в инициации трансляции (eIF4A и eIF4G), а также белков NMD (Upf1 и Upf2) [134].
Показано также участие Gle1 в терминации трансляции в клетках дрожжей [135]. В присутствии ингибиторов трансляции паромомицина и циклогексимида у мутантов gle1 (gle1-2 и gle1-4), как и у мутантов dbp5, снижалась скорость роста. Небольшое количество белка Gle1 обнаружено во фракции 80S рибосом. Выявлено также генетическое и физическое взаимодействие белков Gle1 и Sup45. У мутанта gle1-4 (эта мутация таким образом нарушает взаимодействие между Gle1 и Dbp5, что Gle1 не может стимулировать ATРазную активность белка Dbp5) фактор терминации eRF1 и Dbp5 связаны с полисомной фракцией рибосом, в которой отсутствовал eRF3. Таким образом, на первом этапе формирования комплекса терминации у дрожжей требуются белки Dbp5 и Gle1–IP6 [126]. Белок Gle1 также участвует в инициации трансляции, поскольку взаимодействует с субъединицами фактора eIF3, а у мутантов gle1 нарушена инициация трансляции [135].
eIF3
Фактор eIF3 – мультисубъединичный белковый комплекс, участвующий в инициации трансляции, состоит у дрожжей из шести субъединиц. Белок Hcr1 (eIF3j у млекопитающих), входящий в состав eIF3, стимулирует образование 43S-преинициаторного комплекса, который связывается с молекулой мРНК и сканирует ее в целях обнаружения инициирующего кодона [136]. Как и ряд других белков (ABCE1/Rli1, Gle1 и др.), eIF3 принимает участие и в инициации, и в терминации трансляции. Так, eIF3 стимулирует диссоциацию субчастиц рибосом после терминации трансляции [137]. Делеция гена HСR1 приводит к нонсенс-супрессии и увеличению количества eRF3 во фракции 80S [138]. Кроме того, белок Hcr1 вместе с eRF3 найден во фракции полисом 80S только в присутствии eRF1. Показано взаимодействие двух субъединиц eIF3 (g/TIF35 и i/TIF34) с N- и NM-доменами eRF1. Таким образом, предложена модель, согласно которой eIF3, во-первых, связывается с претерминационным комплексом, влияя на эффективность прочитывания стоп-кодона, и, во-вторых, при участии Hcr1 способствует освобождению eRF3-GDP из рибосомы и связыванию факторов рециклинга Rli1/ABCE1.
СВЯЗЬ БЕЛКОВ eRF1 И eRF3 С ЦИТОСКЕЛЕТОМ
Цитоскелет обеспечивает нормальную морфологию клеток, их полярность, деление и внутриклеточный транспорт. Факты, указывающие на связь факторов терминации трансляции с компонентами цитоскелета, появились давно. Еще до того, как установили основные функции белков Sup45 и Sup35, в гене SUP35 обнаружили мутацию, приводящую к остановке клеточного деления на переходе от стадии G1 к S [139]. К числу многочисленных плейотропных эффектов мутаций в генах SUP35 и SUP45 относится чувствительность к беномилу – веществу, нарушающему веретено деления [140]. Показано, что у мутанта dsup35(63D) (гомолог Sup35 дрозофилы) нарушена сборка веретена деления и расхождение хромосом в сперматогенезе [141]. Мутанты sup35 и sup45, полученные на гаплоидном штамме дрожжей с дисомией по хромосоме III, характеризовались повышенной нестабильностью хромосом, что позволило утверждать, что факторы терминации трансляции участвуют в контроле сегрегации хромосом [142]. При этом предполагалось как прямое взаимодействие факторов терминации и компонентов цитоскелета, так и косвенное влияние нарушения трансляции на клеточные пути, контролирующие сегрегацию хромосом [142].
Дальнейшие исследования показали, что мутации как sup35, так и sup45 приводят к нарушению морфологии клеток, почкообразования и распределения актина в клетках дрожжей. При этом наблюдали различия в проявлении эффектов мутаций. Так, снижение уровня eRF3 вызывало нарушение морфологии клеток и распределения актина, а уменьшение eRF1 останавливало почкование и приводило к накоплению ДНК. Таким образом, факторы терминации имеют дополнительные функции и вовлечены в другие клеточные процессы [143].
Позже показали, что Sup45 взаимодействует с Mlc1 – жизненно важным белком легкой цепи миозина, связывающим Myo1 и Myo2 и координирующим формирование кольца актомиозина и его связь с мембраной при делении клетки [144]. Сверхэкспрессия MLC1 компенсирует температурочувствительные мутации sup45, а мутации sup45 приводят к нарушению распределения Mlc1 внутри клетки. Стоит отметить, что связывание Sup45 c Mlc1 препятствует его взаимодействию с Sup35. Таким образом, конкурентное взаимодействие с партнерами свидетельствует о том, что именно eRF1 вовлечен в цитокинез и контроль клеточного цикла [145].
С использованием двухгибридной системы показано взаимодействие прионогенного N-домена Sup35 с С-доменом Sla1, компонентом актинового цитоскелета. Sla1 связывает актин с клатрином, присутствующем в актиновом кортикальном патче (пятне) при формировании почки. Мутанты sla1 жизнеспособны, но в сочетании с некоторыми другими мутациями по генам аппарата актинового цитоскелета оказываются летальными. Оказалось, что Sla1 участвует в формировании и распространении [PSI+], а мутации “[PSI+] no more” (sup35Δ22-69 и SUP35-PNM2 (G58D)) нарушают взаимодействие Sup35 и Sla1 [146].
Белок Sla1 входит в состав мультибелкового комплекса, который регулирует сборку и разборку актиновых филаментов, а также вовлечен в формирование эндоцитозных везикул. В этом процессе участвует целый ряд белков: Sla2, End3, Arp2 и Arp3, взаимодействие которых с прионогенным N-доменом Sup35 выявлено в двухгибридной системе. С помощью флуоресцентной микроскопии показано, что агрегаты Sup35 колокализуются по крайней мере с некоторыми компонентами актинового кортикального цитоскелета, вовлеченными в путь эндоцитоза. Изучение влияния делеций отдельных генов актинового аппарата на фактор [PSI+] показало, что нарушение актинового цитоскелета не влияет на поддержание [PSI+], а индукция [PSI+] de novo снижается в клетках с дефектным актиновым цитоскелетом. Очевидно, что активную роль в формировании прионных агрегатов Sup35 и их транспортировке играют различные компоненты актинового цитоскелета: сначала агрегаты собираются у актиновых патчей, затем перемещаются на периферию вакуоли [147].
Идентифицированы две группы генов, влияющих на индукцию [PSI+]. К первой группе относятся гены bug1D, bem1D, arf1D, hog1D, делеция которых снижала эффективность этого процесса. Однако эти делеции не влияли на агрегацию Sup35NM при его сверхпродукции: этот белок образовывал агрегаты кольцевой формы – “предшественники” [PSI+]. Делеции генов второй группы (las17D, vps5D, sac6D) подавляли как индукцию [PSI+], так и способность формировать “кольца”. Таким образом, белки актинового комплекса участвуют в возникновении и “созревании” прионов [148].
Асимметричное распределение белковых агрегатов в клетке также регулируется компонентами актинового цитоскелета. Показано как непосредственное взаимодействие Sup35 с некоторыми из них (Sla1 и Sla2), так и влияние мутаций и делеций генов, кодирующих отдельные белки этого комплекса (Lsb, Las17, Act1, End3), на возникновение и передачу [PSI+]. Возможно, локализация агрегатов в компартменте IPOD (Insoluble Protein Deposit) способствует дальнейшей передаче агрегатов при клеточном делении за счет фрагментации и зависит от нормальной сегрегации вакуоли в почку. В этом процессе участвует ряд белков: Vsp5, Bem1, Arf1, Bug1, делеции генов которых нарушают сегрегацию вакуоли, поляризацию клеток и приводят к нарушению индукции [PSI+] (см. обзор [149]).
Чаще всего связь [PSI+] и компонентов цитоскелета выявляется при изменении условий окружающей среды – при термическом или осмотическом шоке. Это может быть связано с адаптивной функцией белковых агрегатов. Показано, что в условиях окислительного стресса [PSI+] может формироваться без сверхэкспрессии прионогенного белка [150]. На фоне мутантных пероксидаз (tsa1, tsa2) идентифицирован ряд компонентов кортикального актинового цитоскелета (Act1, Sac6, Crn1, Abp1, Arc40, Arp2, Arp3, Arc35), вовлеченных в индукцию [PSI+] при стрессе. Эти белки не участвовали в спонтанной индукции [PSI+], или в его индукции при сверхпродукции Sup35. Актиновый цитоскелет, вероятнее всего, определяет расположение агрегированных белков в специализированных компартментах клетки (IPOD или JUNQ (JUxta Nuclear Quality control compartment)) [151].
Показано, что белок Lsb2, участвующий в сборке актиновых филаментов, выступает в роли стресс-зависимого индуктора [PSI+]. Важно отметить, что высокий уровень продукции этого белка может быть достигнут при повышении температуры. Lsb2 взаимодействует с N-доменом Sup35 и колокализуется с агрегатами Sup35. При сверхэкспрессии LSB2 белок может переходить в прионную форму и образовывать нестабильные цитоплазматические агрегаты, которые сохраняются после прекращения сверхэкспрессии. Внесение мутации в участок убиквитинирования белка Lsb2 или его связывания с убиквитин-лигазой увеличивает митотическую стабильность [LSB+]. Мутация, нарушающая связывание Lsb2 с Las17 (белок актинового цитоскелета), приводит к нарушению формирования [LSB+] и, следовательно, [PSI+] [152, 153].
Паралог Lsb2 – Lsb1, также участвует в контроле агрегации белков при термическом стрессе. Lsb1 циркулирует между актиновыми патчами и эндоплазматическим ретикулумом, взаимодействует с Lsb2, Las17, а также с Sup35. Таким образом, регулируя и направляя агрегацию неправильно сложенных белков, оба белка (Lsb1 и Lsb2) могут участвовать в защите клетки при термическом шоке [154].
Pub1/Tia1 – Q/N-богатый белок, способный образовывать прионные агрегаты в клетках дрожжей. Белок Pub1 может вместе с Sup35NM формировать агрегаты, которые визуализируются как линейные структуры, ориентированные по направлению к почке вдоль тубулинового цитоскелета. При этом присутствующие в клетке точечные агрегаты Tia1 препятствуют формированию линейных структур Tia–Sup35. Методом коиммунопреципитации показано, что оба белка связываются с тубулином как при совместной экспрессии, так и по отдельности. Установлено также, что в состав тубулинового комплекса входят агрегаты Sup35. Оказалось, что именно прионогенный домен обеспечивает взаимодействие Sup35 c микротрубочками, тогда как прионогенный домен Pub1 не столь важен для взаимодействия с цитоскелетом. Предполагается, что комплексы между Sup35 и Pub1 участвуют в генезисе тубулинового цитоскелета. Однако нельзя исключать и возможность использования микротрубочек как направляющей опоры для белковых агрегатов [155].
Таким образом, многочисленные исследования указывают на связь прионов и цитоскелета. В первую очередь, актиновый цитоскелет определяет эндоцитоз и цитокинез и, следовательно, влияет на формирование и передачу прионов, в том числе и [PSI+]. Однако существует функциональная связь между белками Sup35 и Sup45 и отдельными компонентами актинового и тубулинового цитоскелета, что говорит о сложной регуляции и взаимосвязи различных клеточных процессов.
ФАКТОРЫ, НЕОБХОДИМЫЕ ДЛЯ ПРИОНИЗАЦИИ Sup35
Для поддержания [PSI+] наиболее важен N-домен белка Sup35, в отсутствие которого прион не может существовать (см. обзоры [1, 6, 174]). Стоит отметить необычный аминокислотный состав этого домена: он обогащен остатками аспарагина и глутамина (более 55%) (регион NQ) [32], что характерно и для других прионов (см. обзор [6]). N‑концевой участок содержит один неполный и пять полных несовершенных повторов, состоящих из 9 аминокислотных остатков (QGGYQQYNP) [32].
Ключевое условие появления прионной изоформы Sup35 de novo – присутствие в клетке дополнительных факторов, получивших общее название Pin (от inducible to [PSI+]) [156]. Первым из них был описан прион [PIN+] (изоформа белка Rnq1, которая также приводит к его агрегации) [156–159]. Согласно современным представлениям, прион [PIN+] играет роль затравки для образования [PSI+] [158]. Эта гипотеза опирается на следующие факты: при индукции [PSI+] агрегаты Sup35 колокализуются с Rnq1 (белок, формирующий [PIN+] [157, 158]), а фибриллы Rnq1 ускоряют агрегацию Sup35 in vitro [160]. По некоторым представлениям взаимодействие агрегатов Sup35 и Rnq1 ограничивается только стадией появления [PSI+] [161], однако существуют данные о том, что белок Rnq1 также влияет на поддержание сформированного приона [162].
В качестве фактора Pin может выступать увеличение концентрации некоторых белков либо их участков: Ure2, Swi1, Cyc8, New1, Yck1, Ste18, Nup116, Pin4 и Lsm4 [158, 163]. Стоит отметить, что первые четыре белка из списка способны формировать другие дрожжевые прионы [URE3+] (Ure2) [164], [SWI+] (Swi1) [165, 166], [OCT+] (Cyc8) [167], [NU+] [163]. Молекулярные механизмы, которые обеспечивают возможность индукции приона [PSI+] de novo, в перечисленных примерах не так очевидны, как в случае [PIN+].
Интересно, что сверхпродукция белков, которые могут действовать как факторы Pin, приводит к дестабилизации уже сформированного приона, например, Lsm4 [168], Pin4, Cyc8, Ste18, Yck1, Pin3, New1 [169]. Предложены различные гипотезы, объясняющие этот феномен, одна из которых связана с конкуренцией за шапероны [169, 170].
Шапероны
С уже сформированными прионными агрегатами Sup35 физически взаимодействуют многие шапероны (Hsp104, Sse1, Sis1, Ydj1, Ssb1, Ssb2, Ssa1, Ssa2) [171, 172]. За счет этих белков осуществляется фрагментация агрегатов Sup35, что обеспечивает появление новых “зерен” приона и, как результат, стабильное его сохранение в череде клеточных поколений (см. обзоры [6, 173–175]). Один из ключевых белков этой системы – Hsp104, отсутствие которого приводит к исчезновению [PSI+] [176]. Этот белок разрезает прионные агрегаты Sup35, увеличивая таким образом их количество, благодаря чему прион может передаваться в дочерние клетки (см. обзоры [6, 173–175]). В отсутствие Hsp104 или при блокировании его активности прион теряется в череде клеточных поколений [177].
Кофактор Hsp104 – белок Sis1 (Hsp40), который также вовлечен в поддержание [PSI+] (см. обзоры [6, 173, 175]): при уменьшении или увеличении его количества прион теряется [178, 179]. На поддержание приона влияет не только изменение количества шаперонов, но и их локализация в клетке. Сверхпродукция белка Cur1 вызывает перемещение Sis1 в ядро, что приводит к усилению фенотипа [PSI+] независимо от варианта приона [180]. Согласно одной из точек зрения, Sis1 необходим для взаимодействия Hsp104 и Hsp70 с прионными агрегатами Sup35, хотя некоторые данные противоречат этой гипотезе (см. обзоры [6, 173, 175]). Аналогичную функцию может выполнять и белок Sgt2. Предполагается, что он участвует в привлечении Ssa1 (Hsp70) и Hsp104 к амилоидным агрегатам Sup35 [181].
Hsp104 также участвует в распределении агрегатов между клетками при делении [182]. Судя по всему, в условиях, когда соотношение Hsp104 и Ssa1 сильно смещается в сторону Hsp104, нарушается процесс распределения агрегатов Sup35. Большая их часть задерживается в материнской клетке и не попадает в почку (см. обзоры [6, 173, 175]). С этой гипотезой хорошо согласуются данные о потере приона при краткосрочном тепловом шоке. Вероятно, это вызвано тем, что при повышении температуры количество Hsp104 возрастает гораздо быстрее, чем Ssa1 [183].
Представители семейства Hsp70 – белки Ssa и Ssb, по-разному влияют на стабильность приона. Повышение количества Ssa1 в клетке препятствует потере [PSI+], вызванной сверхпродукцией Hsp104 [184]. Делеция SSA2 приводит к элиминации некоторых вариантов приона [183]. При этом увеличение количества белка Ssb1 или Ssb2 вызывает усиление изгнания [PSI+] при сверхпродукции Hsp104 [185]. Подобные различия в эффектах связаны с пептидсвязывающим доменом белков семейства Hsp70 [186]. Согласно предложенной модели, Ssa и Ssb могут конкурировать за неправильно уложенный белок Sup35. При этом взаимодействие с Ssa способствует последующей агрегации, а с Ssb – повторной укладке Sup35 в нативную конформацию или его деградации [186].
Отсутствие в клетке шаперонов, ассоциированных с рибосомой (Zuo1 и Ssz1), приводит к изменению локализации Ssb (нарушается его взаимодействие с рибосомами), что, в свою очередь, влияет на эффективность излечивания приона при сверхпродукции Hsp104 [187]. Увеличение количества белков Sfl1, Ssn8 и Rpp0 в клетке может способствовать исчезновению [PSI+], но они действуют не напрямую. Судя по всему, изменение уровня этих белков в клетке влечет за собой повышение экспрессии HSP104 и SSA4. Избыток шаперонов Apj1p и Sti1p также дестабилизирует [PSI+] [179].
Антиприонные системы и токсичность [PSI+]
В клетках S. cerevisiae выделяют в общей сложности до восьми различных антиприонных систем (см. обзор [188]), часть из которых рассмотрена выше. Кроме этих систем в поддержании приона [PSI+] участвует инозитол-фосфатаза Siw14, вовлеченная в изгнание некоторых вариантов фактора [PSI+] даже при базовом уровне их продукции [189]. Аналогичные результаты получены для белков Upf1 и Upf3 [190]. Делеция гена ARG82 приводит к потере большинства вариантов [PSI+] [189]. Этот эффект, по-видимому, связан с отсутствием киназной активности Arg82, он может быть обусловлен и другими нарушениями в пути биосинтеза инозитолпирофосфата [189].
Сверхпродукция Sup35NM в клетках с прионом приводит к их гибели (см. обзор [6]). Известен ряд белков, которые могут модифицировать этот процесс, например, Abf1, Fkh2, Reb1, Sfp1, Gln3 и Mot3 [191, 192]. Делеции некоторых генов, в частности PUB1 и UPF1, могут вызывать гибель клеток с сильными вариантами приона [PSI+] даже без сверхпродукции Sup35NM [193].
Кроме перечисленных групп белков, с агрегатами Sup35 взаимодействует ряд белков, участвующих в различных клеточных процессах: метаболизме глюкозы, цикле трикарбоновых кислот, ответе на различные виды стресса, трансляции и многих других [172]. Недавно показали, что агрегаты белка клеточной стенки Toh1 физически взаимодействуют с агрегатами Sup35NM [194]. Однако роль этих взаимодействий в поддержании приона [PSI+] в клетке остается неясной.
ЗАКЛЮЧЕНИЕ
Согласно базе данных генома дрожжей, ген SUP35 взаимодействует с 883 другими генами, в то время как количество выявленных взаимодействий SUP45 значительно меньше – всего 130 уникальных генов (https://www.yeastgenome.org). Следует отметить, что большинство этих взаимодействий обнаружено в массовых скринингах и детально не изучено. В предлагаемом обзоре мы сконцентрировались на наиболее изученных взаимодействиях. За рамками обзора остались белки, взаимодействие которых с факторами терминации трансляции описано в единичных работах. Будем надеяться на выявление их роли в терминации трансляции и прионизации.
Исследование выполнено при финансовой поддержке Российского фонда фундаментальных исследований в рамках научного проекта № 20-14-50271. (Funding: The reported study was funded by RFBR, project number 20-14-50271.)
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Inge-Vechtomov S., Zhouravleva G., Philippe M. (2003) Eukaryotic release factors (eRFs) history. Biol. Cell. 95, 195–209.
Patino M.M., Liu J.J., Glover J.R., Lindquist S. (1996) Support for the prion hypothesis for inheritance of a phenotypic trait in yeast. Science. 273, 622–626.
Paushkin S.V., Kushnirov V.V., Smirnov V.N., Ter-Avanesyan M.D. (1996) Propagation of the yeast prion-like [PSI+] determinant is mediated by oligomerization of the SUP35-encoded polypeptide chain release factor. EMBO J. 15, 3127–3134.
Glover J.R., Kowal A.S., Schirmer E.C., Patino M.M., Liu J.J., Lindquist S. (1997) Self-seeded fibers formed by Sup35, the protein determinant of [PSI+], a heritable prion-like factor of S. cerevisiae. Cell. 89, 811–819.
King C.Y., Tittmann P., Gross H., Gebert R., Aebi M., Wüthrich K. (1997) Prion-inducing domain 2-114 of yeast Sup35 protein transforms in vitro into amyloid-like filaments. Proc. Natl. Acad. Sci. USA. 94, 6618–6622.
Liebman S.W., Chernoff Y.O. (2012) Prions in yeast. Genetics. 191, 1041–1072.
Stansfield I., Jones K.M., Kushnirov V.V., Dagkesamanskaya A.R., Poznyakovski A.I., Paushkin S.V., Nierras C.R., Cox B.S., Ter-Avanesyan M.D., Tuite M.F. (1995) The products of the SUP45 (eRF1) and SUP35 genes interact to mediate translation termination in Saccharomyces cerevisiae. EMBO J. 14, 4365–4373.
Zhouravleva G., Frolova L., Le Goff X., Le Guellec R., Inge-Vechtomov S., Kisselev L., Philippe M. (1995) Termination of translation in eukaryotes is governed by two interacting polypeptide chain release factors, eRF1 and eRF3. EMBO J. 14, 4065–4072.
Doma M.K., Parker R. (2006) Endonucleolytic cleavage of eukaryotic mRNAs with stalls in translation elongation. Nature. 440, 561–564.
Cheng Z., Saito K., Pisarev A.V., Wada M., Pisareva V.P., Pestova T.V., Gajda M., Round A., Kong C., Lim M., Nakamura Y., Svergun D.I., Ito K., Song H. (2009) Structural insights into eRF3 and stop codon recognition by eRF1. Genes Dev. 23, 1106–18.
Chen L., Muhlrad D., Hauryliuk V., Cheng Z., Lim M.K., Shyp V., Parker R., Song H. (2010) Structure of the Dom34-Hbs1 complex and implications for no-go decay. Nat. Struct. Mol. Biol. 17, 1233–1240.
Becker T., Armache J.-P., Jarasch A., Anger A.M., Villa E., Sieber H., Motaal B.A., Mielke T., Berninghausen O., Beckmann R. (2011) Structure of the no-go mRNA decay complex Dom34-Hbs1 bound to a stalled 80S ribosome. Nat. Struct. Mol. Biol. 18, 715–720.
Frolova L., Le Goff X., Rasmussen H.H., Cheperegin S., Drugeon G., Kress M., Arman I., Haenni A.L., Celis J.E., Philippe M. (1994) A highly conserved eukaryotic protein family possessing properties of polypeptide chain release factor. Nature. 372, 701–703.
Song H., Mugnier P., Das A.K., Webb H.M., Evans D.R., Tuite M.F., Hemmings B.A., Barford D. (2000) The crystal structure of human eukaryotic release factor eRF1-mechanism of stop codon recognition and peptidyl-tRNA hydrolysis. Cell. 100, 311–321.
Ito K. (1996) Conserved motifs in prokaryotic and eukaryotic polypeptide release factors: tRNA-protein mimicry hypothesis. Proc. Natl. Acad. Sci. USA. 93, 5443–5448.
Hellen C.U.T. (2018) Translation termination and ribosome recycling in eukaryotes. Cold Spring Harb. Perspect. Biol. 10, a032656.
Mantsyzov A.B., Ivanova E.V., Birdsall B., Kolosov P.M., Kisselev L.L., Polshakov V.I. (2007) NMR assignments of the C-terminal domain of human polypeptide release factor eRF1. Biomol. NMR Assign. 1, 183–185.
Frolova L.Y., Tsivkovskii R.Y., Sivolobova G.F., Oparina N.Y., Serpinsky O.I., Blinov V.M., Tatkov S.I., Kisselev L.L. (1999) Mutations in the highly conserved GGQ motif of class 1 polypeptide release factors abolish ability of human eRF1 to trigger peptidyl-tRNA hydrolysis. RNA. 5, 1014–1020.
Ito K., Ebihara K., Nakamura Y. (1998) The stretch of C-terminal acidic amino acids of translational release factor eRF1 is a primary binding site for eRF3 of fission yeast. RNA. 4, 958–972.
Merkulova T.I., Frolova L.Y., Lazar M., Camonis J., Kisselev L.L. (1999) C-terminal domains of human translation termination factors eRF1 and eRF3 mediate their in vivo interaction. FEBS Lett. 443, 41–47.
Moskalenko S.E., Chabelskaya S.V., Inge-Vechtomov S.G., Philippe M., Zhouravleva G.A. (2003) Viable nonsense mutants for the essential gene SUP45 of Saccharomyces cerevisiae. BMC Mol. Biol. 4, 2.
Eurwilaichitr L., Graves F.M., Stansfield I., Tuite M.F. (1999) The C-terminus of eRF1 defines a functionally important domain for translation termination in Saccharomyces cerevisiae. Mol. Microbiol. 32, 485–496.
Andjelković N., Zolnierowicz S., Van Hoof C., Goris J., Hemmings B.A. (1996) The catalytic subunit of protein phosphatase 2A associates with the translation termination factor eRF1. EMBO J. 15, 7156–7167.
Kononenko A.V., Mitkevich V.A., Dubovaya V.I., Kolosov P.M., Makarov A.A., Kisselev L.L. (2008) Role of the individual domains of translation termination factor eRF1 in GTP binding to eRF3. Proteins. 70, 388–393.
Mitkevich V.A., Kononenko A.V., Petrushanko I.Y., Yanvarev D.V., Makarov A.A., Kisselev L.L. (2006) Termination of translation in eukaryotes is mediated by the quaternary eRF1*eRF3*GTP*Mg2+ complex. The biological roles of eRF3 and prokaryotic RF3 are profoundly distinct. Nucl. Acids Res. 34, 3947–3954.
Alkalaeva E.Z., Pisarev A.V., Frolova L.Y., Kisselev L.L., Pestova T.V. (2006) In vitro reconstitution of eukaryotic translation reveals cooperativity between release factors eRF1 and eRF3. Cell. 125, 1125–1136.
Hauryliuk V., Zavialov A., Kisselev L., Ehrenberg M. (2006) Class-1 release factor eRF1 promotes GTP binding by class-2 release factor eRF3. Biochimie. 88, 747–757.
Frolova L., Le Goff X., Zhouravleva G., Davydova E., Philippe M., Kisselev L. (1996) Eukaryotic polypeptide chain release factor eRF3 is an eRF1- and ribosome-dependent guanosine triphosphatase. RNA. 2, 334–341.
Pisareva V.P., Pisarev A.V., Hellen C.U.T., Rodnina M.V., Pestova T.V. (2006) Kinetic analysis of interaction of eukaryotic release factor 3 with guanine nucleotides. J. Biol. Chem. 281, 40224–40235.
Hoshino S., Imai M., Mizutani M., Kikuchi Y., Hanaoka F., Ui M., Katada T. (1998) Molecular cloning of a novel member of the eukaryotic polypeptide chain-releasing factors (eRF). J. Biol. Chem. 273, 22254–22259.
Zhouravleva G., Schepachev V., Petrova A., Tarasov O., Inge-Vechtomov S. (2006) Evolution of translation termination factor eRF3: is GSPT2 generated by retrotransposition of GSPT1’s mRNA? IUBMB Life. 58, 199–202.
Kushnirov V.V., Ter-Avanesyan M.D., Telckov M.V., Surguchov A.P., Smirnov V.N., Inge-Vechtomov S.G. (1988) Nucleotide sequence of the SUP2 (SUP35) gene of Saccharomyces cerevisiae. Gene. 66, 45–54.
Cosson B., Couturier A., Chabelskaya S., Kiktev D., Inge-Vechtomov S., Philippe M., Zhouravleva G. (2002) Poly(A)-binding protein acts in translation termination via eukaryotic release factor 3 interaction and does not influence [PSI+] propagation. Mol. Cell. Biol. 22, 3301–3315.
Urakov V.N., Mitkevich O.V., Safenkova I.V., Ter-Avanesyan M.D. (2017) Ribosome-bound Pub1 modulates stop codon decoding during translation termination in yeast. FEBS J. 284, 1914–1930.
Paushkin S.V., Kushnirov V.V., Smirnov V.N., Ter-Avanesyan M.D. (1997) Interaction between yeast Sup45p (eRF1) and Sup35p (eRF3) polypeptide chain release factors: implications for prion-dependent regulation. Mol. Cell. Biol. 17, 2798–2805.
Kong C., Ito K., Walsh M.A., Wada M., Liu Y., Kumar S., Barford D., Nakamura Y., Song H. (2004) Crystal structure and functional analysis of the eukaryotic class II release factor eRF3 from S. pombe. Mol. Cell. 14, 233–234.
des Georges A., Hashem Y., Unbehaun A., Grassucci R.A., Taylor D., Hellen C.U.T., Pestova T.V., Frank J. (2014) Structure of the mammalian ribosomal pre-termination complex associated with eRF1.eRF3.GDPNP. Nucl. Acids Res. 42, 3409–3418.
Taylor D., Unbehaun A., Li W., Das S., Lei J., Liao H.Y., Grassucci R.A., Pestova T.V., Frank J. (2012) Cryo-EM structure of the mammalian eukaryotic release factor eRF1-eRF3-associated termination complex. Proc. Natl. Acad. Sci. USA. 109, 18413–18418.
Culbertson M.R., Underbrink K.M., Fink G.R. (1980) Frameshift suppression Saccharomyces cerevisiae. II. Genetic properties of group II suppressors. Genetics. 95, 833–853.
Leeds P., Peltz S.W., Jacobson A., Culbertson M.R. (1991) The product of the yeast UPF1 gene is required for rapid turnover of mRNAs containing a premature translational termination codon. Genes Dev. 5, 2303–2314.
Leeds P., Wood J.M., Lee B.S., Culbertson M.R. (1992) Gene products that promote mRNA turnover in Saccharomyces cerevisiae. Mol. Cell. Biol. 12, 2165–2177.
Pulak R., Anderson P. (1993) mRNA surveillance by the Caenorhabditis elegans smg genes. Genes Dev. 7, 1885–1897.
Cali B.M., Anderson P. (1998) mRNA surveillance mitigates genetic dominance in Caenorhabditis elegans. Mol. Gen. Genet. 260, 176–184.
Page M.F., Carr B., Anders K.R., Grimson A., Anderson P. (1999) SMG-2 is a phosphorylated protein required for mRNA surveillance in Caenorhabditis elegans and related to Upf1p of yeast. Mol. Cell. Biol. 19, 5943–5951.
Aronoff R., Baran R., Hodgkin J. (2001) Molecular identification of smg-4, required for mRNA surveillance in C. elegans. Gene. 268, 153–164.
Kashima I., Yamashita A., Izumi N., Kataoka N., Morishita R., Hoshino S., Ohno M., Dreyfuss G., Ohno S. (2006) Binding of a novel SMG-1-Upf1-eRF1-eRF3 complex (SURF) to the exon junction complex triggers Upf1 phosphorylation and nonsense-mediated mRNA decay. Genes Dev. 20, 355–367.
Lloyd J.P.B., Davies B. (2013) SMG1 is an ancient nonsense-mediated mRNA decay effector. Plant J. 76, 800–810.
Causier B., Li Z., De Smet R., Lloyd J.P.B., Van de Peer Y., Davies B. (2017) Conservation of nonsense-mediated mRNA decay complex components throughout eukaryotic evolution. Sci. Rep. 7, 16692.
Lloyd J.P.B. (2018) The evolution and diversity of the nonsense-mediated mRNA decay pathway. F1000Research. 7, 1299.
Culbertson M.R., Leeds P.F. (2003) Looking at mRNA decay pathways through the window of molecular evolution. Curr. Opin. Genet. Dev. 13, 207–214.
Atkin A.L., Schenkman L.R., Eastham M., Dahlseid J.N., Lelivelt M.J., Culbertson M.R. (1997) Relationship between yeast polyribosomes and Upf proteins required for nonsense mRNA decay. J. Biol. Chem. 272, 22163–22172.
Altamura N., Groudinsky O., Dujardin G., Slonim-ski P.P. (1992) NAM7 nuclear gene encodes a novel member of a family of helicases with a Zn-ligand motif and is involved in mitochondrial functions in Saccharomyces cerevisiae. J. Mol. Biol. 224, 575–587.
Weng Y., Czaplinski K., Peltz S.W. (1996) Genetic and biochemical characterization of mutations in the ATPase and helicase regions of the Upf1 protein. Mol. Cell. Biol. 16, 5477–5490.
Johansson M.J.O., Jacobson A. (2010) Nonsense-mediated mRNA decay maintains translational fidelity by limiting magnesium uptake. Genes Dev. 24, 1491–1495.
He F., Jacobson A. (1995) Identification of a novel component of the nonsense-mediated mRNA decay pathway by use of an interacting protein screen. Genes Dev. 9, 437–454.
He F., Brown A.H., Jacobson A. (1997) Upf1p, Nmd2p, and Upf3p are interacting components of the yeast nonsense-mediated mRNA decay pathway. Mol. Cell. Biol. 17, 1580–1594.
Weng Y., Czaplinski K., Peltz S.W. (1996) Identification and characterization of mutations in the UPF1 gene that affect nonsense suppression and the formation of the Upf protein complex but not mRNA turnover. Mol. Cell. Biol. 16, 5491–5506.
Czaplinski K., Ruiz-Echevarria M.J., Paushkin S. V, Han X., Weng Y., Perlick H.A., Dietz H.C., Ter-Avanesyan M.D., Peltz S.W. (1998) The surveillance complex interacts with the translation release factors to enhance termination and degrade aberrant mRNAs. Genes Dev. 12, 1665–1677.
Ivanov P.V., Gehring N.H., Kunz J.B., Hentze M.W., Kulozik A.E. (2008) Interactions between UPF1, eRFs, PABP and the exon junction complex suggest an integrated model for mammalian NMD pathways. EMBO J. 27, 736–747.
Журавлева Г. А., Грызина В.А. (2012) Влияние генов UPF на проявление мутаций в гене SUP45. Молекуляр. биология. 46, 285–297.
Wang W., Czaplinski K., Rao Y., Peltz S.W. (2001) The role of Upf proteins in modulating the translation read-through of nonsense-containing transcripts. EMBO J. 20, 880–890.
Serdar L.D., Whiteside D.L., Baker K.E. (2016) ATP hydrolysis by UPF1 is required for efficient translation termination at premature stop codons. Nat. Commun. 7, 14021.
Schuller A.P., Zinshteyn B., Enam S.U., Green R. (2018) Directed hydroxyl radical probing reveals Upf1 binding to the 80S ribosomal E site rRNA at the L1 stalk. Nucl. Acids Res. 46, 2060–2073.
Neu-Yilik G., Raimondeau E., Eliseev B., Yeramala L., Amthor B., Deniaud A., Huard K., Kerschgens K., Hentze M.W., Schaffitzel C., Kulozik A.E. (2017) Dual function of UPF3B in early and late translation termination. EMBO J. 36, 2968–2986.
Kadlec J., Guilligay D., Ravelli R.B., Cusack S. (2006) Crystal structure of the UPF2-interacting domain of nonsense-mediated mRNA decay factor UPF1. RNA. 12, 1817–1824.
Kim Y.K., Maquat L.E. (2019) UPFront and center in RNA decay: UPF1 in nonsense-mediated mRNA decay and beyond. RNA. 25, 407–422.
Min E.E., Roy B., Amrani N., He F., Jacobson A. (2013) Yeast Upf1 CH domain interacts with Rps26 of the 40S ribosomal subunit. RNA. 19, 1105–1115.
Cui Y., Hagan K.W., Zhang S., Peltz S.W. (1995) Identification and characterization of genes that are required for the accelerated degradation of mRNAs containing a premature translational termination codon. Genes Dev. 9, 423–436.
Mendell J.T., Medghalchi S.M., Lake R.G., Noensie E.N., Dietz H.C. (2000) Novel Upf2p orthologues suggest a functional link between translation initiation and nonsense surveillance complexes. Mol. Cell. Biol. 20, 8944–8957.
Fourati Z., Roy B., Millan C., Coureux P.-D., Kervestin S., van Tilbeurgh H., He F., Usón I., Jacobson A., Graille M. (2014) A highly conserved region essential for NMD in the Upf2 N-terminal domain. J. Mol. Biol. 426, 3689–3702.
He F., Brown A.H., Jacobson A. (1996) Interaction between Nmd2p and Upf1p is required for activity but not for dominant-negative inhibition of the nonsense-mediated mRNA decay pathway in yeast. RNA. 2, 153–170.
López-Perrote A., Castaño R., Melero R., Zamarro T., Kurosawa H., Ohnishi T., Uchiyama A., Aoyagi K., Buchwald G., Kataoka N., Yamashita A., Llorca O. (2016) Human nonsense-mediated mRNA decay factor UPF2 interacts directly with eRF3 and the SURF complex. Nucl. Acids Res. 44, 1909–1923.
Lykke-Andersen J., Shu M.D., Steitz J.A. (2000) Human Upf proteins target an mRNA for nonsense-mediated decay when bound downstream of a termination codon. Cell. 103, 1121–1131.
Shirley R.L., Lelivelt M.J., Schenkman L.R., Dahlseid J.N., Culbertson M.R. (1998) A factor required for nonsense-mediated mRNA decay in yeast is exported from the nucleus to the cytoplasm by a nuclear export signal sequence. J. Cell Sci. 111, 3129–3143.
Shirley R.L., Ford A.S., Richards M.R., Albertini M., Culbertson M.R. (2002) Nuclear import of Upf3p is mediated by importin-alpha/-beta and export to the cytoplasm is required for a functional nonsense-mediated mRNA decay pathway in yeast. Genetics. 161, 1465–1482.
Chabelskaya S., Kiktev D., Inge-Vechtomov S., Philippe M., Zhouravleva G. (2004) Nonsense mutations in the essential gene SUP35 of Saccharomyces cerevisiae are non-lethal. Mol. Genet. Genomics. 272, 297–307.
Chabelskaya S., Gryzina V., Moskalenko S., Le Goff C., Zhouravleva G. (2007) Inactivation of NMD increases viability of sup45 nonsense mutants in Saccharomyces cerevisiae. BMC Mol. Biol. 8, 71.
Шабельская С.В., Журавлева Г.А. (2010) Мутации в гене SUP35 нарушают процесс деградации мРНК, содержащих преждевременные стоп-кодоны. Молекуляр. биология. 44, 51–59.
Chamieh H., Ballut L., Bonneau F., Le Hir H. (2008) NMD factors UPF2 and UPF3 bridge UPF1 to the exon junction complex and stimulate its RNA helicase activity. Nat. Struct. Mol. Biol. 15, 85–93.
Blobel G. (1973) A protein of molecular weight 78,000 bound to the polyadenylate region of eukaryotic messenger RNAs. Proc. Natl. Acad. Sci. USA. 70, 924–928.
Adam S.A., Nakagawa T., Swanson M.S., Woodruff T.K., Dreyfuss G. (1986) mRNA polyadenylate-binding protein: gene isolation and sequencing and identification of a ribonucleoprotein consensus sequence. Mol. Cell. Biol. 6, 2932–2943.
Sachs A.B., Bond M.W., Kornberg R.D. (1986) A single gene from yeast for both nuclear and cytoplasmic polyadenylate-binding proteins: domain structure and expression. Cell. 45, 827–835.
Mangus D.A., Evans M.C., Jacobson A. (2003) Poly(A)-binding proteins: multifunctional scaffolds for the post-transcriptional control of gene expression. Genome Biol. 4, 223.
Kühn U., Wahle E. (2004) Structure and function of poly(A) binding proteins. Biochim. Biophys. Acta. 1678, 67–84.
Sachs A.B., Davis R.W., Kornberg R.D. (1987) A single domain of yeast poly(A)-binding protein is necessary and sufficient for RNA binding and cell viability. Mol. Cell. Biol. 7, 3268–3276.
Kozlov G., Siddiqui N., Coillet-Matillon S., Trempe J.-F., Ekiel I., Sprules T., Gehring K. (2002) Solution structure of the orphan PABC domain from Saccharomyces cerevisiae poly(A)-binding protein. J. Biol. Chem. 277, 22822–22828.
Tarun S.Z., Sachs A.B. (1996) Association of the yeast poly(A) tail binding protein with translation initiation factor eIF-4G. EMBO J. 15, 7168–7177.
Wells S.E., Hillner P.E., Vale R.D., Sachs A.B. (1998) Circularization of mRNA by eukaryotic translation initiation factors. Mol. Cell. 2, 135–140.
Sachs A.B., Davis R.W. (1989) The poly(A) binding protein is required for poly(A) shortening and 60S ribosomal subunit-dependent translation initiation. Cell. 58, 857–867.
Munroe D., Jacobson A. (1990) mRNA poly(A) tail, a 3′-enhancer of translational initiation. Mol. Cell. Biol. 10, 3441–3455.
Gallie D.R. (1991) The cap and poly(A) tail function synergistically to regulate mRNA translational efficiency. Genes Dev. 5, 2108–2116.
Tarun S.Z., Wells S.E., Deardorff J.A., Sachs A.B. (1997) Translation initiation factor eIF4G mediates in vitro poly(A) tail-dependent translation. Proc. Natl. Acad. Sci. USA. 94, 9046–9051.
Imataka H., Gradi A., Sonenberg N. (1998) A newly identified N-terminal amino acid sequence of human eIF4G binds poly(A)-binding protein and functions in poly(A)-dependent translation. EMBO J. 17, 7480–7489.
Kessler S.H., Sachs A.B. (1998) RNA recognition motif 2 of yeast Pab1p is required for its functional interaction with eukaryotic translation initiation factor 4G. Mol. Cell. Biol. 18, 51–57.
Otero L.J., Ashe M.P., Sachs A.B. (1999) The yeast poly(A)-binding protein Pab1p stimulates in vitro poly(A)-dependent and cap-dependent translation by distinct mechanisms. EMBO J. 18, 3153–3163.
Richardson R., Denis C.L., Zhang C., Nielsen M.E.O., Chiang Y.-C., Kierkegaard M., Wang X., Lee D.J., Andersen J.S., Yao G. (2012) Mass spectrometric identification of proteins that interact through specific domains of the poly(A) binding protein. Mol. Genet. Genomics. 287, 711–730.
Hoshino S., Imai M., Kobayashi T., Uchida N., Katada T. (1999) The eukaryotic polypeptide chain releasing factor (eRF3/GSPT) carrying the translation termination signal to the 3’-Poly(A) tail of mRNA. Direct association of erf3/GSPT with polyadenylate-binding protein. J. Biol. Chem. 274, 16677–16680.
Cosson B., Couturier A., Le Guellec R., Moreau J., Chabelskaya S., Zhouravleva G., Philippe M. (2002) Characterization of the poly(A) binding proteins expressed during oogenesis and early development of Xenopus laevis. Biol. Cell. 94, 217–231.
Cosson B., Berkova N., Couturier A., Chabelskaya S., Philippe M., Zhouravleva G. (2002) Poly(A)-binding protein and eRF3 are associated in vivo in human and Xenopus cells. Biol. Cell. 94, 205–216.
Uchida N., Hoshino S.-I., Imataka H., Sonenberg N., Katada T. (2002) A novel role of the mammalian GSPT/eRF3 associating with poly(A)-binding protein in Cap/Poly(A)-dependent translation. J. Biol. Chem. 277, 50286–50292.
Ivanov A., Mikhailova T., Eliseev B., Yeramala L., Sokolova E., Susorov D., Shuvalov A., Schaffitzel C., Alkalaeva. E. (2016) PABP enhances release factor recruitment and stop codon recognition during translation termination. Nucl. Acids Res. 44, 7766–7776.
Albrecht M., Lengauer T. (2004) Survey on the PABC recognition motif PAM2. Biochem. Biophys. Res. Commun. 316, 129–138.
Brito M., Malta-Vacas J., Carmona B., Aires C., Costa P., Martins A.P., Ramos S., Conde A.R., Monteiro C. (2005) Polyglycine expansions in eRF3/GSPT1 are associated with gastric cancer susceptibility. Carcinogenesis. 26, 2046–2049.
Malta-Vacas J., Chauvin C., Gonçalves L., Nazaré A., Carvalho C., Monteiro C., Bagrel D., Jean-Jean O., Brito M. (2009) eRF3a/GSPT1 12-GGC allele increases the susceptibility for breast cancer development. Oncol. Rep. 21, 1551–1558.
Miri M., Hemati S., Safari F., Tavassoli M. (2012) GGCn polymorphism of eRF3a/GSPT1 gene and breast cancer susceptibility. Med. Oncol. 29, 1581–1585.
Jerbi S., Jolles B., Bouceba T., Jean-Jean O. (2016) Studies on human eRF3-PABP interaction reveal the influence of eRF3a N-terminal glycin repeat on eRF3-PABP binding affinity and the lower affinity of eRF3a 12-GGC allele involved in cancer susceptibility. RNA Biol. 13, 306–315.
Kozlov G., Trempe J.F., Khaleghpour K., Kahvejian A., Ekiel I., Gehring K. (2001) Structure and function of the C-terminal PABC domain of human poly(A)-binding protein. Proc. Natl. Acad. Sci. USA. 98, 4409–4413.
Xie J., Kozlov G., Gehring K. (2014) The “tale” of poly(A) binding protein: the MLLE domain and PAM2-containing proteins. Biochim. Biophys. Acta. 1839, 1062–1068.
Kozlov G., De Crescenzo G., Lim N.S., Siddiqui N., Fantus D., Kahvejian A., Trempe J-F., Elias D., Ekiel I., Sonenberg N., O’Connor-McCourt M., Gehring K. (2004) Structural basis of ligand recognition by PABC, a highly specific peptide-binding domain found in poly(A)-binding protein and a HECT ubiquitin ligase. EMBO J. 23, 272–281.
Kononenko A.V., Mitkevich V.A., Atkinson G.C., Tenson T., Dubovaya V.I., Frolova L.Y., Makarov A.A., Hauryliuk V. (2010) GTP-dependent structural rearrangement of the eRF1:eRF3 complex and eRF3 sequence motifs essential for PABP binding. Nucl. Acids Res. 38, 548–558.
Kozlov G., Gehring K. (2010) Molecular basis of eRF3 recognition by the MLLE domain of poly(A)-binding protein. PLoS One. 5, e10169.
Hegde R., Srinivasula S.M., Datta P., Madesh M., Wassell R., Zhang Z., Cheong N., Nejmeh J., Fernandes-Alnemri T.,Hoshino S., Alnemri E.S. (2003) The polypeptide chain-releasing factor GSPT1/eRF3 is proteolytically processed into an IAP-binding protein. J. Biol. Chem. 278, 38699–38706.
Hosoda N., Kobayashi T., Uchida N., Funakoshi Y., Kikuchi Y., Hoshino S., Katada T. (2003) Translation termination factor eRF3 mediates mRNA decay through the regulation of deadenylation. J. Biol. Chem. 278, 38287–38291.
Funakoshi Y., Doi Y., Hosoda N., Uchida N., Osawa M., Shimada I., Tsujimoto M., Suzuki T., Katada T., Hoshino S. (2007) Mechanism of mRNA deadenylation: evidence for a molecular interplay between translation termination factor eRF3 and mRNA deadenylases. Genes Dev. 21, 3135–3148.
Osawa M., Hosoda N., Nakanishi T., Uchida N., Kimura T., Imai S., Machiyama A., Katada T., Hoshino S., Shimada I. (2012) Biological role of the two overlapping poly(A)-binding protein interacting motifs 2 (PAM2) of eukaryotic releasing factor eRF3 in mRNA decay. RNA. 18, 1957–1967.
Mangus D.A., Evans M.C., Agrin N.S., Smith M., Gongidi P., Jacobson A. (2004) Positive and negative regulation of poly(A) nuclease. Mol. Cell. Biol. 24, 5521–5533.
Ivanov A., Shuvalova E., Egorova T., Shuvalov A., Sokolova E., Bizyaev N., Shatsky I., Terenin I., Alkalaeva E. (2019) Polyadenylate-binding protein-interacting proteins PAIP1 and PAIP2 affect translation termination. J. Biol. Chem. 294, 8630–8639.
Geourjon C., Deléage G. (1994) SOPM: a self-optimized method for protein secondary structure prediction. Protein Eng. 7, 157–164.
Pisarev A.V., Skabkin M.A., Pisareva V.P., Skabkina O.V., Rakotondrafara A.M., Hentze M.W., Hellen C.U.T., Pestova T.V. (2010) The role of ABCE1 in eukaryotic posttermination ribosomal recycling. Mol. Cell. 37, 196–210.
Shoemaker C.J., Green R. (2011) Kinetic analysis reveals the ordered coupling of translation termination and ribosome recycling in yeast. Proc. Natl. Acad. Sci. USA. 108, E1392–E1398.
Barthelme D., Scheele U., Dinkelaker S., Janoschka A., Macmillan F., Albers S.-V., Driessen A.J.M., Stagni M.S., Bill E., Meyer-Klaucke W., Schünemann V., Tampé R. (2007) Structural organization of essential iron-sulfur clusters in the evolutionarily highly conserved ATP-binding cassette protein ABCE1. J. Biol. Chem. 282, 14598–14607.
Khoshnevis S., Gross T., Rotte C., Baierlein C., Ficner R., Krebber H. (2010) The iron-sulphur protein RNase L inhibitor functions in translation termination. EMBO Rep. 11, 214–219.
Becker T., Franckenberg S., Wickles S., Shoemaker C.J., Anger A.M., Armache J.-P., Sieber H., Ungewickell C., Berninghausen O., Daberkow I., Karcher A., Thomm M., Hopfner K-P., Green R., Beckmann R. (2012) Structural basis of highly conserved ribosome recycling in eukaryotes and archaea. Nature. 482, 501–506.
Snay-Hodge C.A., Colot H.V., Goldstein A.L., Cole C.N. (1998) Dbp5p/Rat8p is a yeast nuclear pore-associated DEAD-box protein essential for RNA export. EMBO J. 17, 2663–2676.
Tseng S.S., Weaver P.L., Liu Y., Hitomi M., Tartakoff A.M., Chang T.H. (1998) Dbp5p, a cytosolic RNA helicase, is required for poly(A)+ RNA export. EMBO J. 17, 2651–2662.
Tieg B., Krebber H. (2013) Dbp5 – from nuclear export to translation. Biochim. Biophys. Acta. 1829, 791–798.
Estruch F., Hodge C., Gómez-Navarro N., Peiró-Chova L., Heath C.V., Cole C.N. (2012) Insights into mRNP biogenesis provided by new genetic interactions among export and transcription factors. BMC Genet. 13, 80.
Schmitt C., von Kobbe C., Bachi A., Panté N., Rodrigues J.P., Boscheron C., Rigaut G., Wilm M., Séraphin B., Carmo-Fonseca M., Izaurralde E. (1999) Dbp5, a DEAD-box protein required for mRNA export, is recruited to the cytoplasmic fibrils of nuclear pore complex via a conserved interaction with CAN/Nup159p. EMBO J. 18, 4332–4347.
Gross T., Siepmann A., Sturm D., Windgassen M., Scarcelli J.J., Seedorf M., Cole C.N., Krebber H. (2007) The DEAD-box RNA helicase Dbp5 functions in translation termination. Science. 315, 646–649.
Murphy R., Wente S.R. (1996) An RNA-export mediator with an essential nuclear export signal. Nature. 383, 357–360.
Watkins J.L., Murphy R., Emtage J.L., Wente S.R. (1998) The human homologue of Saccharomyces cerevisiae Gle1p is required for poly(A)+ RNA export. Proc. Natl. Acad. Sci. USA. 95, 6779–6784.
Alcázar-Román A.R., Tran E.J., Guo S., Wente S.R. (2006) Inositol hexakisphosphate and Gle1 activate the DEAD-box protein Dbp5 for nuclear mRNA export. Nat. Cell Biol. 8, 711–716.
Adams R.L., Mason A.C., Glass L., Aditi, Wente S.R. (2017) Nup42 and IP6 coordinate Gle1 stimulation of Dbp5/DDX19B for mRNA export in yeast and human cells. Traffic. 18, 776–790.
Montpetit B., Thomsen N.D., Helmke K.J., Seeliger M.A., Berger J.M., Weis K. (2011) A conserved mechanism of DEAD-box ATPase activation by nucleoporins and InsP6 in mRNA export. Nature. 472, 238–242.
Bolger T.A., Folkmann A.W., Tran E.J., Wente S.R. (2008) The mRNA export factor Gle1 and inositol hexakisphosphate regulate distinct stages of translation. Cell. 134, 624–633.
Hinnebusch A.G. (2006) eIF3: a versatile scaffold for translation initiation complexes. Trends Biochem. Sci. 31, 553–562.
Pisarev A.V., Hellen C.U.T., Pestova T.V. (2007) Recycling of eukaryotic posttermination ribosomal complexes. Cell. 131, 286–299.
Beznosková P., Cuchalová L., Wagner S., Shoemaker C.J., Gunišová S., von der Haar T., Valášek L.S. (2013) Translation initiation factors eIF3 and HCR1 control translation termination and stop codon read-through in yeast cells. PLoS Genet. 9, e1003962.
Kikuchi Y., Shimatake H., Kikuchi A. (1988) A yeast gene required for the G1-to-S transition encodes a protein containing an A-kinase target site and GTPase domain. EMBO J. 7, 1175–1182.
Tikhomirova V.L., Inge-Vechtomov S.G. (1996) Sensitivity of sup35 and sup45 suppressor mutants in Saccharomyces cerevisiae to the anti-microtubule drug benomyl. Curr. Genet. 30, 44–49.
Basu J., Williams B.C., Li Z., Williams E.V., Goldberg M.L. (1998) Depletion of a Drosophila homolog of yeast Sup35p disrupts spindle assembly, chromosome segregation, and cytokinesis during male meiosis. Cell Motil. Cytoskeleton. 39, 286–302.
Borchsenius A.S., Tchourikova A.A., Inge-Vechtomov S.G. (2000) Recessive mutations in SUP35 and SUP45 genes coding for translation release factors affect chromosome stability in Saccharomyces cerevisiae. Curr. Genet. 37, 285–291.
Valouev I.A., Kushnirov V.V., Ter-Avanesyan M.D. (2002) Yeast polypeptide chain release factors eRF1 and eRF3 are involved in cytoskeleton organization and cell cycle regulation. Cell Motil. Cytoskeleton. 52, 161–173.
Stevens R.C., Davis T.N. (1998) Mlc1p is a light chain for the unconventional myosin Myo2p in Saccharomyces cerevisiae. J. Cell Biol. 142, 711–722.
Valouev I.A., Urakov V.N., Kochneva-Pervukhova N.V., Smirnov V.N., Ter-Avanesyan M.D. (2004) Translation termination factors function outside of translation: yeast eRF1 interacts with myosin light chain, Mlc1p, to effect cytokinesis. Mol. Microbiol. 53, 687–696.
Bailleul P.A., Newnam G.P., Steenbergen J.N., Chernoff Y.O. (1999) Genetic study of interactions between the cytoskeletal assembly protein Sla1 and prion-forming domain of the release factor Sup35 (eRF3) in Saccharomyces cerevisiae. Genetics. 153, 81–94.
Ganusova E.E., Ozolins L.N., Bhagat S., Newnam G.P., Wegrzyn R.D., Sherman M.Y., Chernoff Y.O. (2006) Modulation of prion formation, aggregation, and toxicity by the actin cytoskeleton in yeast. Mol. Cell. Biol. 26, 617–629.
Manogaran A.L., Hong J.Y., Hufana J., Tyedmers J., Lindquist S., Liebman S.W. (2011) Prion formation and polyglutamine aggregation are controlled by two classes of genes. PLoS Genet. 7, e1001386.
Moosavi B., Mousavi B., Yang G.-F. (2016) Actin, membrane trafficking and the control of prion induction, propagation and transmission in yeast. Traffic. 17, 5–20.
Tyedmers J., Madariaga M.L., Lindquist S. (2008) Prion switching in response to environmental stress. PLoS Biol. 6, e294.
Speldewinde S.H., Doronina V.A., Tuite M.F., Grant C.M. (2017) Disrupting the cortical actin cytoskeleton points to two distinct mechanisms of yeast [PSI+] prion formation. PLoS Genet. 13, e1006708.
Chernova T.A., Romanyuk A.V., Karpova T.S., Shanks J.R., Ali M., Moffatt N., Howie R.L., O’Dell A., McNally J.G., Liebman S.W., Chernoff Y.O., Wilkinson K.D. (2011) Prion induction by the short-lived, stress-induced protein Lsb2 is regulated by ubiquitination and association with the actin cytoskeleton. Mol. Cell. 43, 242–252.
Chernova T.A., Kiktev D.A., Romanyuk A.V., Shanks J.R., Laur O., Ali M., Ghosh A., Kim D., Yang Z., Mang M., Chernoff Y.O., Wilkinson K.D. (2017) Yeast short-lived actin-associated protein forms a metastable prion in response to thermal stress. Cell Rep. 18, 751–761.
Ali M., Chernova T.A., Newnam G.P., Yin L., Shanks J., Karpova T.S., Lee A., Laur O., Subramanian S., Kim D., McNally J.G., Seyfried N.T., Chernoff Y.O., Wilkinson K.D. (2014) Stress-dependent proteolytic processing of the actin assembly protein Lsb1 modulates a yeast prion. J. Biol. Chem. 289, 27625–27639.
Li X., Rayman J.B., Kandel E.R., Derkatch I.L. (2014) Functional role of Tia1/Pub1 and Sup35 prion domains: directing protein synthesis machinery to the tubulin cytoskeleton. Mol. Cell. 55, 305–318.
Derkatch I.L., Bradley M.E., Zhou P., Chernoff Y.O., Liebman S.W. (1997) Genetic and environmental factors affecting the de novo appearance of the [PSI+] prion in Saccharomyces cerevisiae. Genetics. 147, 507–519.
Sondheimer N., Lindquist S. (2000) Rnq1: an epigenetic modifier of protein function in yeast. Mol. Cell. 5, 163–172.
Derkatch I.L., Bradley M.E., Hong J.Y., Liebman S.W. (2001) Prions affect the appearance of other prions: the story of [PIN+]. Cell. 106, 171–182.
Derkatch I.L., Bradley M.E., Masse S.V., Zadorsky S.P., Polozkov G.V., Inge-Vechtomov S.G., Liebman S.W. (2000) Dependence and independence of [PSI+] and [PIN+]: a two-prion system in yeast? EMBO J. 19, 1942–1952.
Derkatch I.L., Uptain S.M., Outeiro T.F., Krishnan R., Lindquist S.L., Liebman S.W. (2004) Effects of Q/N-rich, polyQ, and non-polyQ amyloids on the de novo formation of the [PSI+] prion in yeast and aggregation of Sup35 in vitro. Proc. Natl. Acad. Sci. USA. 101, 12934–12939.
Bagriantsev S., Liebman S.W. (2004) Specificity of prion assembly in vivo: [PSI+] and [PIN+] form separate structures in yeast. J. Biol. Chem. 279, 51042–51048.
Бондарев С.А., Лихолетова Д.В., Белоусов М.В., Журавлева Г.А. (2017) Белок Rnq1 защищает прион [PSI+] от эффекта PNM мутации. Молекуляр. биология. 51, 367–371.
Osherovich L.Z., Weissman J.S. (2001) Multiple Gln/Asn-rich prion domains confer susceptibility to induction of the yeast [PSI+] prion. Cell. 106, 183–194.
Wickner R. (1994) [URE3] as an altered URE2 protein: evidence for a prion analog in Saccharomyces cerevisiae. Science. 264, 566–569.
Du Z., Park K.-W., Yu H., Fan Q., Li L. (2008) Newly identified prion linked to the chromatin-remodeling factor Swi1 in Saccharomyces cerevisiae. Nat. Genet. 40, 460–465.
Du Z., Li L. (2014) Investigating the interactions of yeast prions: [SWI+], [PSI+], and [PIN+]. Genetics. 197, 685–700.
Patel B.K., Gavin-Smyth J., Liebman S.W. (2009) The yeast global transcriptional co-repressor protein Cyc8 can propagate as a prion. Nat. Cell Biol. 11, 344–349.
Kurahashi H., Oishi K., Nakamura Y. (2011) A bipolar personality of yeast prion proteins. Prion. 5, 305–310.
Yang Z., Hong J.Y., Derkatch I.L., Liebman S.W. (2013) Heterologous Gln/Asn-rich proteins impede the propagation of yeast prions by altering chaperone availability. PLoS Genet. 9, e1003236.
Derkatch I.L., Liebman S.W. (2013) The story of stolen chaperones: how overexpression of Q/N proteins cures yeast prions. Prion. 7, 294–300.
Bagriantsev S.N., Gracheva E.O., Richmond J.E., Liebman S.W. (2008) Variant-specific [PSI+] infection is transmitted by Sup35 polymers within [PSI+] aggregates with heterogeneous protein composition. Mol. Biol. Cell. 19, 2433–2443.
Nevzglyadova O.V., Artemov A.V., Mittenberg A.G., Solovyov K.V., Kostyleva E.I., Mikhailova E.V., Kuznetsova I.M., Turoverov K.K., Soidla T.R. (2009) Prion-associated proteins in yeast: comparative analysis of isogenic [PSI+] and [psi-] strains. Yeast. 26, 611–631.
Chernova T.A., Wilkinson K.D., Chernoff Y.O. (2014) Physiological and environmental control of yeast prions. FEMS Microbiol. Rev. 38, 326–344.
Kushnirov V.V., Ter-Avanesyan M.D. (1998) Structure and replication of yeast prions. Cell. 94, 13–16.
Matveenko A.G., Barbitoff Y.A., Jay-Garcia L.M., Chernoff Y.O., Zhouravleva G.A. (2018) Differential effects of chaperones on yeast prions: CURrent view. Curr. Genet. 64, 317–325.
Chernoff Y., Lindquist S., Ono B., Inge-Vechtomov S., Liebman S. (1995) Role of the chaperone protein Hsp104 in propagation of the yeast prion-like factor [PSI+]. Science. 268, 880–884.
Ferreira P.C., Ness F., Edwards S.R., Cox B.S., Tuite M.F. (2001) The elimination of the yeast [PSI+] prion by guanidine hydrochloride is the result of Hsp104 inactivation. Mol. Microbiol. 40, 1357–1369.
Higurashi T., Hines J.K., Sahi C., Aron R., Craig E.A. (2008) Specificity of the J-protein Sis1 in the propagation of 3 yeast prions. Proc. Natl. Acad. Sci. USA. 105, 16596–16601.
Kryndushkin D.S., Smirnov V.N., Ter-Avanesyan M.D., Kushnirov V.V. (2002) Increased expression of Hsp40 chaperones, transcriptional factors, and ribosomal protein Rpp0 can cure yeast prions. J. Biol. Chem. 277, 23702–23708.
Barbitoff Y.A., Matveenko A.G., Moskalenko S.E., Zemlyanko O.M., Newnam G.P., Patel A., Chernova T.A., Chernoff Y.O., Zhouravleva G.A. (2017) To CURe or not to CURe? Differential effects of the chaperone sorting factor Cur1 on yeast prions are mediated by the chaperone Sis1. Mol. Microbiol. 105, 242–257.
Kiktev D.A., Patterson J.C., Muller S., Bariar B., Pan T., Chernoff Y.O. (2012) Regulation of chaperone effects on a yeast prion by cochaperone Sgt2. Mol. Cell. Biol. 32, 4960–4970.
Liu B., Larsson L., Caballero A., Hao X., Oling D., Grantham J., Nyström T. (2010) The polarisome is required for segregation and retrograde transport of protein aggregates. Cell. 140, 257–267.
Newnam G.P., Birchmore J.L., Chernoff Y.O. (2011) Destabilization and recovery of a yeast prion after mild heat shock. J. Mol. Biol. 408, 432–448.
Newnam G.P., Wegrzyn R.D., Lindquist S.L., Chernoff Y.O. (1999) Antagonistic interactions between yeast chaperones Hsp104 and Hsp70 in prion curing. Mol. Cell. Biol. 19, 1325–1333.
Chernoff Y.O., Newnam G.P., Kumar J., Allen K., Zink A.D. (1999) Evidence for a protein mutator in yeast: role of the Hsp70-related chaperone ssb in formation, stability, and toxicity of the [PSI+] prion. Mol. Cell. Biol. 19, 8103–8112.
Allen K.D., Wegrzyn R.D., Chernova T.A., Müller S., Newnam G.P., Winslett P.A., Wittich, K.B., Wilkinson K.D., Chernoff Y.O. (2005) Hsp70 chaperones as modulators of prion life cycle: novel effects of Ssa and Ssb on the Saccharomyces cerevisiae prion [PSI+]. Genetics. 169, 1227–1242.
Kiktev D.A., Melomed M.M., Lu C.D., Newnam G.P., Chernoff Y.O. (2015) Feedback control of prion formation and propagation by the ribosome-associated chaperone complex. Mol. Microbiol. 96, 621–632.
Wickner R.B., Edskes H.K., Son M., Wu S., Niznikiewicz M. (2020) How do yeast cells contend with prions? Int. J. Mol. Sci. 21, 4742.
Wickner R.B., Kelly A.C., Bezsonov E.E., Edskes H.K. (2017) [PSI+] prion propagation is controlled by inositol polyphosphates. Proc. Natl. Acad. Sci. USA. 114, E8402–E8410.
Son M., Wickner R.B. (2018) Nonsense-mediated mRNA decay factors cure most [PSI+] prion variants. Proc. Natl. Acad. Sci. USA. 115, E1184–E1193.
Матвеенко А.Г., Белоусов М.В., Бондарев С.А., Москаленко С.Е., Журавлева Г.А. (2016) Идентификация новых генов, влияющих на токсичность приона [PSI+] у дрожжей Saccharomyces cerevisiae. Молекуляр. биология. 50, 803–813.
Matveenko A.G., Drozdova P.B., Belousov M.V, Moskalenko S.E., Bondarev S.A., Barbitoff Y.A., Nizhnikov A.A., Zhouravleva G.A. (2016) SFP1-mediated prion-dependent lethality is caused by increased Sup35 aggregation and alleviated by Sis1. Genes Cells. 21, 1290–1308.
Urakov V.N., Mitkevich O.V., Dergalev A.A., Ter-Avanesyan M.D. (2018) The Pub1 and Upf1 proteins act in concert to protect yeast from toxicity of the [PSI+]. Int. J. Mol. Sci. 19, 3663.
Sergeeva A.V., Sopova J.V., Belashova T.A., Siniukova V.A., Chirinskaite A.V., Galkin A.P., Zadorsky S.P. (2019) Amyloid properties of the yeast cell wall protein Toh1 and its interaction with prion proteins Rnq1 and Sup35. Prion. 13, 21–32.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


