Молекулярная биология, 2022, T. 56, № 2, стр. 325-333
Протокол полнотранскриптомного анализа децидуальных клеток плаценты
А. А. Бабовская a, *, Е. А. Трифонова a, b, В. Н. Сереброва a, М. Г. Сваровская a, b, А. А. Зарубин a, О. В. Жилякова b, Т. В. Габидулина b, А. А. Полтанова b, Л. В. Рычкова c, В. А. Степанов a
a Научно-исследовательский институт медицинской генетики Томского национального исследовательского медицинского центра
634050 Томск, Россия
b Сибирский государственный медицинский университет
634050 Томск, Россия
c Научный центр проблем здоровья семьи и репродукции человека
664003 Иркутск, Россия
* E-mail: anastasia.babovskaya@medgenetics.ru
Поступила в редакцию 02.07.2021
После доработки 29.09.2021
Принята к публикации 13.10.2021
- EDN: ZYYCEV
- DOI: 10.31857/S0026898422020045
Аннотация
Появление технологий высокопроизводительного секвенирования позволило расширить наше представление о биологической значимости некодирующих участков генома человека. В последние годы появляется все больше исследований, посвященных изучению роли некодирующих РНК в развитии болезней, а также их участия в различных клеточных процессах. До настоящего времени все полнотранскриптомные исследования нативной плацентарной ткани с описанием области некодирующей РНК проводили без выделения отдельных клеточных популяций. Такой подход, ввиду высокой клеточной гетерогенности плацентарной ткани, существенно усложняет возможность определения молекулярно-биологических функций отдельных клеток и их роли в молекулярном патогенезе репродуктивных нарушений. В представленной работе предложена методика получения тотальной РНК из единичных децидуальных клеток замороженной плацентарной ткани, полученных с помощью технологии лазерной микродиссекции, для последующего секвенирования полного транскриптома, включая кластер некодирующих РНК. Данная методика может быть успешно использована при изучении полногеномного профиля экспрессии в других клеточных популяциях плаценты. Высокая точность результатов транскриптомного профилирования децидуальных клеток, полученных с помощью разработанной методики, дополнительно подтверждается интегративным анализом с данными эксперимента, выполненного на платформе 10х Genomics.
Плацента человека давно стала предметом научного интереса, так как играет ключевую роль в установлении равновесия и толерантности на уровне системы мать‒плод. Это динамичный и неоднородный орган со множеством выполняемых функций, критически важных для успешного протекания беременности. По строению плацента представляет собой гетерогенное образование, состоящее из клеток материнского и эмбрионального происхождения, организованных в виде многослойных ворсинчатых единиц [1]. Вариабельность экспрессии генов в плацентарной ткани на уровне транскриптома исследуют с использованием различных подходов, как при анализе цельной ткани, так и с выделением отдельных клеток. Значительные отличия по клеточному составу и профилю экспрессии среди плацентарных компартментов свидетельствуют о необходимости рассмотрения ландшафта экспрессии на уровне отдельных клеточных субпопуляций [2–7]. Одним из решающих событий для успешной имплантации эмбриона и развития плаценты считается гормонально контролируемая дифференцировка стромальных клеток эндометрия в децидуальные клетки и формирование децидуальной оболочки. В литературе особенно подчеркивается важный вклад децидуальных стромальных клеток и их микроокружения в иммунный контроль над инвазией трофобласта, процессы ремоделирования тканей и нормального развития плода [8, 9]. Таким образом, децидуальные стромальные клетки служат основным функциональным клеточным компонентом в децидуальной ткани плаценты, выполняя трофическую, гормональную, защитную, иммунорегуляторную и гемопоэтическую функции, в связи с чем анализ их транскрипционной активности с использованием новых молекулярно-генетических методов актуален, как с теоретической точки зрения, так и с позиции предиктивной медицины.
Современные технологии высокопроизводительного секвенирования относятся к мощным инструментам для исследования молекулярных механизмов реализации генетической информации, дифференцировки клеток, межклеточного сигналинга, а также для идентификации активных биологических процессов [10]. Фокус исследований в последние годы все больше смещается к анализу генома, транскриптома и эпигенома на уровне отдельных клеток. Однако для получения полных и достоверных данных о характере экспрессии в том или ином эксперименте необходимо владеть методикой получения единичных клеток и/или клеточных субпопуляций и преодолеть проблему деградации РНК в процессе работы с тканью. Благодаря появлению технологии 10х охарактеризован транскриптомный профиль большинства структурных клеток плаценты, однако возможности этого метода ограничены лишь белоккодирующей частью генома. Наряду с этим появляется все больше данных, на основании которых становится очевидной важная роль некодирующих РНК в механизмах эпигенетической регуляции экспрессии генов и связь абберации нетранслируемых участков генома с развитием заболеваний у человека [11–14], включая нарушение процессов имплантации эмбриона и патологическое течение беременности [15–17].
В представленном исследовании разработан протокол полнотранскриптомного анализа RNA-seq единичных децидуальных клеток свежезамороженной ткани плаценты, полученных с помощью технологии лазерной микродиссекции.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Реактивы и оборудование. Для приготовления всех фиксирующих и красящих растворов в работе использована вода без РНКаз, не обработанная диэтилпирокарбонатом (DEPC), и чистый перегнанный спирт; для получения осушенного этанола использовали молекулярные сита с диаметром пор 3 Å (15 г гранул молекулярного сита на 500 мл этанола); набор Single Cell RNA Purification kit (“Norgen Biotek”, Канада) для выделения РНК из клеток; ингибитор РНКаз RiboLock (“Thermo Fisher Scientific”, США); набор SMARTer Stranded Total RNA-Seq kit v2 (“Takara BIO”, Япония) для синтеза полнотранскриптомных библиотек. Для получения тонких срезов из замороженных тканей использовали криостат Thermo Microm HM525 (“Thermo Fisher Scientific”) и предметные стекла с мембраной из полиэтиленнафталата (PEN), свободные от РНКаз/ДНКаз (“Carl Zeiss”, Германия). Лазерную микродиссекцию проводили на оборудовании PALM MicroBeam UV (“Carl Zeiss”) с использованием пробирок с адгезивной крышкой той же фирмы. Для оценки качества и количества выделенной РНК использовали набор High Sensitivity RNA ScreenTape kit и прибор TapeStation 4150 (“Agilent Technologies”, США). Размер полученных библиотек также оценивали с использованием прибора TapeStation 4150 и набора High Sensitivity D1000 ScreenTape kit. Концентрацию библиотек измеряли на флуориметре Qubit 3.0 (“Thermo Fisher Scientific”).
Биобанк тканей: сбор и хранение. Образцы, используемые в работе, были получены при биопсии плацентарной ткани в макроскопически нормальных областях, исключая участки кровоизлияний, сразу после отделения плаценты согласно протоколу, описанному S. Robson и соавт. [18]. Биоптаты ткани вырезали из материнской части плаценты вблизи пуповины размером 5 × 5 мм, глубиной около 3 мм. Полученный материал немедленно промывали физиологическим раствором и помещали в криопробирки, которые хранили в жидком азоте, чтобы свести к минимуму деградацию РНК и изменение экспрессии некоторых генов в связи с некрозом/ишемией ткани. Все собранные ткани длительно хранили в резервуарах с жидким азотом.
Криодиссекция. Из замороженной плацентарной ткани получали срезы толщиной 7 мкм и помещали их на предварительно охлажденные стекла с мембраной из PEN. Готовые стекла со срезами замораживали и оставляли в криостате до начала окрашивания.
На этом этапе были соблюдены все необходимые условия проведения эксперимента: 1) непосредственно перед процедурой получения срезов спиртосодержащими детергентами тщательно промывали столик криостата, лезвие ножа, патроны для тканей и пинцеты; 2) патроны и пинцеты дополнительно обрабатывали раствором RNaseZap (“Thermo Fisher Scientific”) и ультрафиолетом для деконтаминации рабочих поверхностей; 3) пинцеты, предметные стекла, фиксирующие и окрашивающие растворы предварительно охлаждали до ‒30…‒45°С (для предотвращения оттаивания срезов и деградации РНК в процессе работы с тканью).
Окрашивание. Для визуализации ткани использовали модифицированную методику окраски гематоксилином-эозином. Готовые замороженные стекла вынимали из криостата и помещали на охлажденный до –30°С элемент, где срезы немедленно фиксировали в 70%-ном этаноле в течение 90 с. Далее стекло однократно промывали в 1 мл деионизованной воды, свободной от РНКаз, ДНКаз и пирогенов, и наносили водный раствор гематоксилина на 90 с, после чего повторяли процедуру промывки. После удаления гематоксилина на срезы наносили водный раствор эозина на 10‒12 с, затем стекло быстро промывали этанолом в возрастающей концентрации: 70 → 95 → 100%.
Окрашенные стекла быстро и тщательно просушивали для предотвращения образования конденсата в процессе их транспортировки и хранения. РНК в зафиксированных таким образом клетках стабильна в течение 24 ч. Для длительного хранения (до 1 мес.) готовые стекла помещали в специальные боксы с селикагелем и хранили при температуре не выше –70°С [19].
В качестве альтернативного красителя в протоколах подготовки срезов для лазерной микродиссекции нередко используют крезил фиолетовый (Cresyl violet) [19–21]. При работе с этим красителем отпадает необходимость в этапе регидратации тканей, что относится к несомненным плюсам, так как эндогенные РНКазы остаются неактивными на протяжении всего времени окрашивания. В работе M. Cummings и соавт. [19] приведен протокол успешного использования крезила в экспериментах с тканями эндометрия. Однако при воспроизведении данной методики было установлено, что для визуализации децидуальных клеток плаценты этот краситель оказался мало пригоден ‒ на окрашенных таким образом препаратах четко видны клетки трофобласта, имеющие большое ядро и мало цитоплазмы, тогда как контуры децидуальных клеток почти неразличимы из-за слабо прокрашенной цитоплазмы (рис. 1).
Рис. 1.
Децидуальная ткань плаценты человека (срез). Микрофотографии окрашенных Cresyl violet ядер децидуальных клеток (а) и окрашенных гематоксилином-эозином скоплений децидуальных клеток. Увеличение 150×.

Лазерная микродиссекция. Процедуру лазерной микродиссекции проводили на приборе PALM MicroBeam UV, оснащенном инвертированным микроскопом Axio Observer Z1 и камерой Аxiocam MRm (“Carl Zeiss”). В ходе эксперимента проводили диссекцию 1000 морфологически однородных децидуальных клеток (рис. 1б) в специальных пробирках (см. “Реактивы и оборудование”), предварительно обработанных ультрафиолетом в течение 15 мин, после чего полученный материал лизировали в 100 мкл свежеприготовленного RL-буфера (“Norgen Biotek”), содержащего 1 мкл β-меркаптоэтанола. Клеточный лизат тщательно перемешивали и приступали к выделению РНК. Дополнительно стоит отметить, что при диссекции интересующих областей соблюдали рекомендованное [20, 22] ограничение по времени ‒ не более 2 ч на стекло в целях сохранения РНК максимального качества.
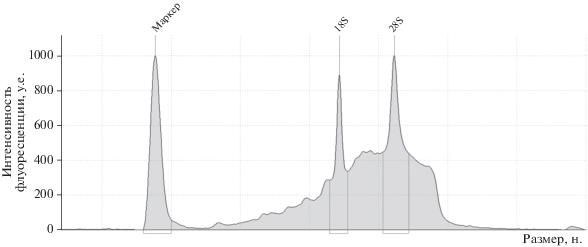
Выделение и контроль качества РНК. Для выделения тотальной РНК использовали набор Single Cell RNA Purification kit (“Norgen Biotek”). Вкратце методика выделения РНК, предлагаемая при использовании этого набора, состоит в следующем. Растворенный клеточный лизат, после предварительной инкубации и осаждения 70%-ным этанолом, помещали на колонки. В результате нескольких циклов центрифугирования и отмывки Wash-буфером (входит в набор) на мембране колонки сорбируются нуклеиновые кислоты. Для удаления примесей геномной ДНК через колонку пропускали раствор ДНКазы I, который затем вновь наносили на фильтр колонки и инкубировали, после чего колонку промывали Wash-буфером и элюировали с нее очищенную РНК. Все этапы работы проводили согласно протоколу и рекомендациям производителя с небольшой модификацией: перед процедурой элюирования РНК с колонки на дно пробирки вносили ингибитор РНКаз RiboLock (“Thermo Fisher Scientific”) в концентрации 20 U в 14 мкл элюирующего буфера. Для оценки качества (RIN) и количества выделенной РНК двухмикроллитровую аликвоту полученного элюата анализировали с помощью системы автоматического гель-электрофореза на приборе TapeStation 4150 (рис. 2).
Рис. 2.
Электрофореграмма тотальной РНК, выделенной из децидуальных клеток плаценты. Концентрация РНК в образце ‒ 1.3 нг/мкл, RIN ‒6.7. Указаны позиции 18S и 28S рибосомных РНК.
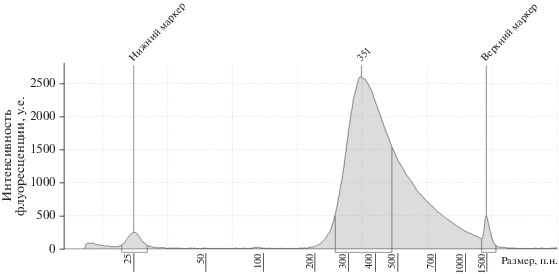
Подготовка библиотек, оценка размерности, сек-венирование. Синтез библиотек для полнотранскриптомного анализа проводился набором SMARTer Stranded Total RNA-Seq kit v2 (“Takara BIO”), адаптированного для работы с низкими входными концентрациями. Сразу после получения экстрагированной РНК, не более чем через 1 ч, ее фрагментировали при 82°С в течение 3.5 мин и проводили реакцию обратной транскрипции. Условия фрагментации были подобраны экспериментально, исходя из величины показателя RIN (неопубликованные данные). Синтезированную кДНК хранили при ‒20°С в течение двух месяцев. Дальнейшие этапы подготовки библиотек выполняли строго по протоколу производителя с выбором 15 циклов конечной амплификации. Продукты ПЦР очищали, используя технологию магнитных частиц AMPure XP (“Beckman Coulter Inc.”, США). Вкратце так. Реакционную смесь инкубировали с магнитными частицами ‒ при этом с поверхностью частиц связываются фрагменты ДНК размером более 100 п.н. Пробирки с магнитными частицами помещали на магнитный штатив и дважды промывали свежеприготовленным 80%-ным этанолом. Очищенную библиотеку элюировали с магнитных частиц 15 мкл ТЕ-буфера (входит в комплект набора SMARTer Stranded Total RNA-Seq kit v2), после чего одномикролитровую аликвоту полученного раствора использовали для количественного определения концентрации ДНК с помощью флуориметра Qubit 3.0 (“Thermo Fisher Scientific”). Для оценки размера фрагментов библиотеки системой автоматического гель-электрофореза использовали набор High Sensitivity kit для прибора TapeStation 4150 (рис. 3).
Рис. 3.
Оценка размера библиотеки системой капиллярного гель-электрофореза на приборе TapeStation 4150 с реагентами High Sensitivity D1000 ScreenTapes kit. Приведенные значения интенсивности флуоресценции нормализованы по стандарту для High Sensitivity D1000 ScreenTapes kit. Готовые библиотеки объединяли в пул и выполняли одноконцевое прочтение 75 циклов на платформе Illumina Next-seq 500 (“Illumina, Inc.”, США).
При моделировании эксперимента для подготовки библиотек секвенирования мы также использовали альтернативный набор ‒ CORALL Total RNA-Seq Library Prep kit (“Lexogen”, Австрия) ‒ с дополнительным комплектом реагентов для истощения рибосомной РНК ‒ RiboCop rRNA Depletion kit (“Lexogen”). В связи с тем, что дизайн представленной работы предполагал последовательное и непрерывное проведение этапов получения окрашенных срезов, диссекции клеток, выделения РНК и синтеза стабильной кДНК, минуя этап хранения РНК, наиболее удобным оказалось использование набора SMARTer Stranded Total RNA-Seq kit v2. Согласно протоколу SMARTer, время получения кДНК составляет 2.5 ч, после чего образцы можно поместить на ‒20°С на срок до одного месяца, в то время как при использовании набора фирмы “Lexogen” время до получения кДНК и возможности безопасного прерывания протокола составляет более 4 ч (так как вначале необходимо провести очистку тотальной РНК от рибосомных компонентов), что существенно усложняет работу.
Для оценки производительности наборов было синтезировано две библиотеки из одного и того же образца плацентарной ткани, с одинаковым количеством входного материала (500 пг). Ниже представлено сравнение полученных библиотек, просеквенированных в одном запуске, по ряду параметров (табл. 1). Безусловно, мы не претендуем на точность оценки эффективности этих двух наборов на основании полученных данных ‒ это задача специальных исследований.
Таблица 1.
Сравнение библиотек, полученных при использовании CORALL Total RNA-Seq Library Prep kit (“Lexogen”) и SMARTer Stranded Total RNA-Seq kit v2 (“Takara BIO”)
| Параметры | CORALL Total RNA-Seq Library Prep kit | SMARTer Stranded Total RNA-Seq kit v2 |
|---|---|---|
| Входное количество РНК, нг | 0.5 | 0.5 |
| Концентрация полученной библиотеки, нг/мкл | 11.4 | 12.4 |
| Размер библиотеки, п.н. | 238.0 | 336.0 |
| Число выровненных прочтений, млн | 11.5 | 12.8 |
| Содержание дубликатов, % | 21.8 | 35.1 |
| Содержание GC, % | 53.0 | 52.0 |
Как видно из приведенных данных, при использовании набора фирмы “Takara BIO” выход кДНК выше, чем для набора от “Lexogen”, при равном исходном количестве материала. Дополнительно хочется отметить, что набор SMARTer Stranded Total RNA-Seq kit v2 (“Takara BIO”) имеет минимальный порог в 250 пг для концентрации РНК на входе, тогда как для набора CORALL Total RNA-Seq Library Prep Kit установлен порог в 1 нг.
Биоинформатическая обработка данных. Ниже приведено описание данных секвенирования трех библиотек, подготовленных набором SMARTer Stranded Total RNA-Seq Kit v2. Каждая библиотека получена из индивидуального биоптата плаценты и представляет транскриптом пула 1000 морфологически однородных децидуальных клеток. Расчетное число прочтений на образец составляло не менее 20 млн. Полученные данные РНК-секвенирования выравнивали на hg38-сборку человеческого генома в программе STAR (https://sites.google.com/site/spiketrainanalysiswithr), после чего проводили подсчет прочтений, приходящихся на отдельные гены с использованием пакета HTSeq согласно базе данных GENCODE V19 (https://www.gencodegenes.org/human/). Дальнейшую обработку проводили в программной среде R с использованием пакета edgeR (https://bioconductor.org/packages/release/bioc/html/edgeR.html).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В проведенный нами анализ было включено усредненное число транскриптов по трем образцам (16 844), для которых число прочтений на миллион (CPM) составляло не менее 1 (CPM ≥ 1). Показатель CPM в данном случае использовали для компенсации смещения между образцами при вычислении уровня экспрессии генов. Как видно из данных, представленных в табл. 2, большинство активных областей транскрипции соответствует белоккодирующим участкам генома человека (13 683). В то же время около 20% от всей совокупности идентифицированных в децидуальных клетках транскриптов приходится на нетранслируемые регионы. В этом кластере преобладают антисмысловые и длинные некодирующие РНК и присутствуют последовательности транскрибируемых с псевдогенов РНК, микроРНК, малых ядрышковых РНК и других транскриптов.
Таблица 2.
Аннотация полученных в работе транскриптов
| Идентифицированные транскриптыa | Число транскриптовb |
|---|---|
| Антисмысловая РНК (antisense RNA) | 685 |
| Длинная некодирующая РНК (lncRNA) | 689 |
| МикроРНК (miRNA) | 40 |
| Неидентифицированная РНК (misc_RNA) | 131 |
| Митохондриальная рРНК (Mt_rRNA) | 2 |
| Митохондриальная тРНК (Mt_tRNA) | 14 |
| Полиморфный псевдоген (polymorphic_pseudogene) | 3 |
| Процессированный псевдоген (processed_pseudogene) | 470 |
| Процессированный транскрипт (processed_transcript) | 134 |
| Белоккодирующий транскрипт (protein_coding) | 13 683 |
| рРНК (rRNA) | 55 |
| Интронные последовательности (sense_intronic) | 89 |
| Перекрывающиеся транскрипты (sense_overlapping) | 27 |
| Малая ядрышковая РНК (snoRNA) | 109 |
| Малая ядерная РНК (snRNA) | 48 |
| Транскрибируемый процессированный псевдоген (transcribed_processed_pseudogene) | 74 |
| Транскрибируемый непроцессированный псевдоген (transcribed_unprocessed_pseudogene) | 177 |
| Непроцессированный псевдоген (unprocessed_pseudogene) | 89 |
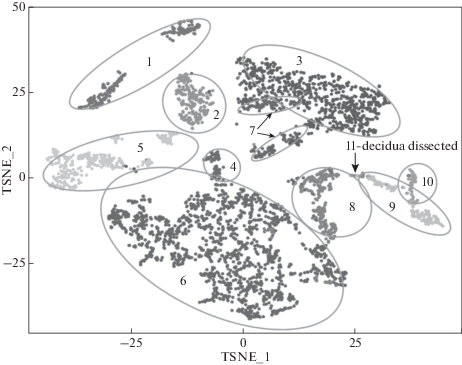
Для сравнения полученных в этом эксперименте данных были привлечены результаты сек-венирования плацентарной ткани с помощью технологии Chromium (SRS3815606) фирмы “10x Genomics” (США; https://panglaodb.se/view_ data.php?sra=SRA782908&srs=SRS3815606). Проведен интегративный анализ, который включал максимальное число клеток плацентарной ткани, исследованное с применением технологии секвенирования единичных клеток 10x. С целью объединить полученные нами данные и результаты 10x chromium секвенирования клеток плаценты мы нормализовали число прочтений в исследованных нами образцах на медианное число прочтений, приходящихся на одну клетку, во внешнем эксперименте. После этого нормализованные данные добавляли к опубликованным для 10x chromium как отдельные клетки с присвоением новых уникальных индексов и вновь обрабатывали с использованием библиотеки Seurat (https://cran.r-project.org/web/packages/Seurat/index.html). По результатам кластеризации объединенного набора данных полученные нами библиотеки локализуются в кластере децидуальных и эритроидных клеток (рис. 4).
Рис. 4.
Кластеризация образцов, включенных в интегративный анализ данных, полученных нами и во внешнем эксперименте секвенирования единичных клеток плацентарной ткани (https://panglaodb.se/view_data.php?sra= SRA782908&srs=SRS3815606) с помощью технологии 10х. Кластер 1 – клетки эндотелия, 2 – Т-лимфоциты, 3 – естественные киллерные клетки, 4 – звездчатые клетки поджелудочной железы, 5 – дендритные клетки, 6 – фибробласты, 7 – γδ T-клетки, 8 – эритроидные клетки и их предшественники, 9 – децидуальные клетки, 10 – холангиоциты, 11 – образцы клеток, полученных нами методом микродиссекции (decidua dissected), указаны стрелкой.
На основании представленных результатов можно говорить о возможности применения лазерной микродиссекции для получения дискретных клеточных популяций из гетерогенных тканей и последующего анализа транскриптома. Ее эффективность сравнима с передовыми методами транскриптомики: секвенированием единичных клеток (scRNA-seq) и секвенированием отдельных ядер (snRNA-seq). Эти две технологии, безусловно, относятся к самым высокопроизводительным на современном этапе, но при реализации прикладных задач уступают лазерной микродиссекции в экономическом аспекте.
К ограничениям лазерной микродиссекции стоит отнести методологически сложную технологию и главную проблему ‒ поддержание молекулярной целостности РНК. На деградацию РНК влияет много факторов: способ хранения тканей, метод окраски, скорость диссекции и др. Несоблюдение температурного режима на любом из этапов также может привести к существенной потере РНК. Кроме того, необходимо учитывать, что для разных типов тканей характерно различное содержание РНКаз и их ингибиторов. Ввиду перечисленных причин уже имеющиеся протоколы для получения клеток с помощью микродиссекции [20‒24 и др.] приходится адаптировать для каждого эксперимента с учетом всех возможных факторов.
Цель проведенного нами исследования заключалась в разработке практичного и воспроизводимого протокола для выделения децидуальных клеток и оптимизации условий выделения целостной РНК. Впервые описана методика получения полного транскриптома единичных децидуальных клеток из замороженных тканей плаценты с помощью лазерной микродиссекции. Разработанный метод может быть использован в экспериментах и с другими тканями для изучения транскриптома отдельных клеток или субпопуляций клеток.
Работа проведена при финансовой поддержке Российского фонда фундаментальных исследований (гранты № 20-34-90128 и № 18-29-13045).
Настоящее исследование прошло экспертизу комитета по биомедицинской этике Научно-исследовательского института медицинской генетики Федерального государственного бюджетного научного учреждения “Томский национальный исследовательский медицинский центр” (регистрационный номер 136, дата регистрации 11.11.2019). Все процедуры, выполненные в исследовании с участием людей, соответствуют этическими стандартами Институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики. От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Tsang J., Vong J., Liona L., Poon C., Jiang P., Lui K., Ni Y., To K., Cheng Y., Chiu R., Ming Y., Lo D. (2017) Integrative single-cell and cell-free plasma RNA transcriptomics elucidates placental cellular dynamics. Proc. Natl. Acad. Sci. USA. 114(37), 7786–7795.
Vinketova K., Mourdjeva M., Oreshkova T. (2016) Human decidual stromal cells as a component of the implantation niche and a modulator of maternal immunity. J. Pregnancy. 2016, 8689436.
Pavlicev M., Wagner G., Chavan A., Owens K., Maziarz J., Dunn-Fletcher C., Kallapur S., Muglia L., Jones H. (2017) Single-cell transcriptomics of the human placenta: inferring the cell communication network of the maternal-fetal interface. Genome Res. 27, 349–361.
Liu Y., Fan X., Wang R., Lu X., Dang Y., Wang H., Lin H., Zhu C., Ge H., Cross J., Wang H. (2018) Single-cell RNA-seq reveals the diversity of trophoblast subtypes and patterns of differentiation in the human placenta. Cell Res. 28, 819–832.
Suryawanshi H., Morozov P., Straus A., Sahasrabudhe N., Max K., Garzia A., Kustagi M., Tuschl T., Williams Z. (2018) A single-cell survey of the human first-trimester placenta and decidua. Sci. Adv. 4, 4788.
Pique-Regi R., Romero R., Tarca A., Sendler E., Xu Y., Garcia-Flores V., Leng Y., Luca F., Hassan S., Gomez-Lopez N. (2019) Single cell transcriptional signatures of the human placenta in term and preterm parturition. eLife. 8, e52004.
Pique-Regi R., Romero R., Tarca A., Luca F., Xu Y., Alazizi A., Leng Y., Hsu C., Gomez-Lopez N. (2020) Does the human placenta express the canonical cell entry mediators for SARS-CoV-2? eLife. 9, e58716.
Brosens J., Pijnenborg R., Brosens I. (2002) The myometrial junctional zone spiral arteries in normal and abnormal pregnancies: a review of the literature. Am. J. Obstet. Gynecol. 187(5), 1416–1423.
Conrad K., Maria Belen Rabaglino, Uiterweer E. (2017) Emerging role for dysregulated decidualization in the genesis of preeclampsia. Placenta. 60, 119–129.
Fasterius E., Uhlen M., C. Szigyarto (2019) Single-cell RNA-seq variant analysis for exploration of genetic heterogeneity in cancer. Sci. Rep. 9, 9524.
Yoffe L., Gilam A., Yaron O., Polsky A., Farberov L., Syngelaki A., Nicolaides K., Hod M., Shomron N. (2018) Early detection of preeclampsia using circulating small non-coding RNA. Sci. Rep. 8, 3401.
Klohonatz K., Coleman S., Islas-Trejo A., Medrano J., Hess A., Kalbfleisch T., Thomas M., Bouma G., Bru-emmer J. (2019) Coding RNA sequencing of equine endometrium during maternal recognition of pregnancy. Genes. 10, 749.
Aznaourova M., Janga H., Sefried S., Kaufmann A., Dorna J., Volkers S., Georg P., Lechner M., Hoppe J., Dokel S., Schmerer N., Gruber A., Linne U., Bauer S., Sander L., Schmeck B., Schulte L. (2020) Noncoding RNA MaIL1 is an integral component of the TLR4-TRIF pathway. Proc. Natl. Acad. Sci. USA. 117(16), 9042–9053.
Statello L., Guo C., Chen L., Huarte M. (2020) Gene regulation by long non-coding RNAs and its biological functions. Nat. Rev. Mol. Cell Biol. 22, 96–118.
Liang J., Wang S., Wang Z. (2017) Role of microRNAs in embryo implantation. Reprod. Biol. Endocrinol. 15, 90.
Skalis G., Katsi V., Miliou A., Georgiopoulos G., Papazachou O., Vamvakou G., Nihoyannopoulos P., Tousoulis D., Makris T. (2019) MicroRNAs in preeclampsia. MicroRNA. 8(1), 28–35.
Apicella C., Ruano C., Mehats C., Miralles F., Vaiman D. (2019) The role of epigenetics in placental development and the etiology of preeclampsia. Int. J. Mol. Sci. 20, 2837.
Robson S., Simpson H., Ball E., Lyall F., Bulmer J. (2002) Punch biopsy of the human placental bed. Am. J. Obstet. Gynecol. 187(5), 1349–1355.
Cummings M., Mappa G., Orsi N.M. (2018) Laser capture microdissection and isolation of high-quality RNA from frozen endometrial tissue. Methods Mol. Biol. 1723, 155‒166. https://doi.org/10.1007/978-1-4939-7558-7_8
Bevilacqua C., Makhzami S., Helbling J. C., Defrenaix P., Martin P. (2010) Maintaining RNA integrity in a homogeneous population of mammary epithelial cells isolated by laser capture microdissection. BMC Cell Biol. 11, 95.
Nichterwitz S., Benitez J.A, Hoogstraaten R., Deng Q., Hedlund E. (2018) LCM-Seq: a method for spatial transcriptomic profiling using laser capture microdissection coupled with polya-based RNA sequencing. Methods Mol. Biol. 1649, 95–110.
Barwinska D., Ferkowicz M.J., Cheng Y.H., Winfree S., Dunn K. W., Kelly K.J., Sutton T.A., Rovin B.H., Parikh S.V., Phillips C.L., Dagher P.C., El-Achkar T.M., Eadon M.T., Kidney Precision Medicine Project. (2020) Application of laser microdissection to uncover regional transcriptomics in human kidney tissue. J. Vis. Exp. 160, https://doi.org/10.3791/61371. 10.3791/61371
Ong C.J., Tan Q.X., Lim H.J., Shannon N.B., Lim W.K., Hendrikson J., Ng W.H., Tan J., Koh K., Wasudevan S.D., Ng C., Rajasegaran V., Lim T., Ong C.K., Kon O.L., Teh B.T., Tan G., Chia C.S., Soo K.C., Teo M. (2020) An optimised protocol harnessing laser capture microdissection for transcriptomic analysis on matched primary and metastatic colorectal tumours. Sci. Rep. 10(1), 682.
Pan D., Xu M., Chang X., Xia M., Fang Y., Fu Y., Shen W., Wang Y., Sun X. (2020) Laser capture microdissection-based RNA microsequencing reveals optic nerve crush-related early mRNA alterations in retinal ganglion cell layer. Transl. Vis. Sci. Technol. 9(11), 30.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


