Молекулярная биология, 2022, T. 56, № 2, стр. 259-274
Белки SFPQ, NONO и длинная некодирующая РНК NEAT1: функции в клетке и в жизненном цикле ВИЧ-1
О. А. Шадрина a, b, Т. Ф. Кихай a, Ю. Ю. Агапкина a, b, М. Б. Готтих a, b, *
a Химический факультет Московского государственного университета им. М.В. Ломоносова
119991 Москва, Россия
b Научно-исследовательский институт физико-химической биологии имени А.Н. Белозерского Московского государственного университета им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: gottikh@belozersky.msu.ru
Поступила в редакцию 06.09.2021
После доработки 06.10.2021
Принята к публикации 06.10.2021
- EDN: TBXYOW
- DOI: 10.31857/S0026898422020161
Аннотация
Около 20 лет назад в ядрах клеток обнаружили большие РНК-белковые комплексы, названные параспеклями, основными компонентами которых являются длинная некодирующая РНК NEAT1 и белки SFPQ и NONO. Позднее эти белки нашли в свободном состоянии в ядре и даже в цитоплазме. Функции NEAT1 и белков параспеклей достаточно разнообразны, в их число входят удержание в ядре РНК, подвергшихся множественному редактированию аденозина в инозин, ответ на повреждения ДНК, а также регуляция транскрипции, контроль стабильности мРНК, регуляция сплайсинга, участие в ответе клетки на вирусную инфекцию. Так, получены достаточно многочисленные, хотя и противоречивые, данные об участии NEAT1, SFPQ и NONO в репликативном цикле ВИЧ-1 на разных его этапах. В настоящем обзоре мы постарались кратко рассмотреть основные клеточные функции РНК NEAT1 и белков SFPQ и NONO, а также суммировать и по возможности систематизировать данные об их роли в жизненном цикле ВИЧ-1.
ВВЕДЕНИЕ
В жизненном цикле вируса иммунодефицита человека первого типа (ВИЧ-1) участвуют многие клеточные белки, которые могут выступать и как отрицательные, и как положительные факторы. Благодаря развитию технологий высокопроизводительного скрининга, становятся известны все новые клеточные белки, взаимодействующие с компонентами вируса и влияющие на эффективность его репликации. Однако роль далеко не всех этих новых белков в жизненном цикле вируса определена однозначно. Детальное исследование роли клеточных белков в развитии вирусной инфекции важно как для понимания механизмов взаимодействия вируса с клеткой, так и для разработки новых подходов к ингибированию репликации вируса. Известно, что одной из основных проблем в борьбе с ВИЧ-инфекцией является быстрое возникновение устойчивости к лекарственным средствам на основе ингибиторов вирусных ферментов. В связи с этим достаточно перспективным представляется подход к ингибированию ВИЧ-1, основанный на подавлении взаимодействия вирусных белков с клеточными партнерами, необходимыми для успешной репликации вируса.
Опубликован ряд работ, посвященных влиянию основных компонентов ядерных телец параспеклей – белков SFPQ и NONO, а также некодирующей РНК NEAT1, на репликацию ВИЧ-1, однако отсутствует полная картина их участия в репликации. В этой связи настоящий обзор направлен в основном на поиск и систематизацию данных о роли SFPQ, NONO и NEAT1 в жизненном цикле ВИЧ-1. Для более корректного понимания возможностей их участия в репликации вируса мы сочли необходимым кратко рассмотреть их основные функции в клетках человека.
ФУНКЦИИ SFPQ, NONO И РНК NEAT1 В КЛЕТКЕ
SFPQ, NONO и NEAT1 – основные компоненты клеточных параспеклей
В 2002 году были впервые описаны новые ядерные тельца – большие РНК-белковые комплексы, названные параспеклями [1]. Эти комплексы обнаружены как локальные скопления различных РНК-связывающих белков, расположенные недалеко от ядерных структур спеклей. Параспекли присутствуют практически во всех клетках за исключением стволовых [2]. В качестве первых белковых компонентов параспеклей идентифицированы PSPС1 (paraspeckle protein 1), PSPС2 (paraspeckle protein 2), p54 (ядерный РНК-связывающий белок 54 кДа, известный как NONO (Non-POU domain-containing octamer-binding protein), SFPQ (splicing factor proline glutamine rich)/ PSF (polypyrimidine tract-binding protein-associated splicing factor), Fus (fused in sarcoma), CFIm68 (cleavage factor Im 68 kDa, CPSF6) [1]. Позднее нашли еще примерно 30 белков, колокализующихся с параспеклями [3]. Среди обнаруженных белков особенно важными для формирования параспеклей оказались NONO и SFPQ, нокдаун которых в клетках приводил к дезинтеграции телец [4].
SFPQ, NONO и PSPС1 входят в семейство белков DBHS (Drosophila behavior/human splicing), объединенных по наличию в них центрального участка, состоящего из четырех доменов: двух РНК-связывающих – RRM1 и RRM2, а также NOPS (NonA/paraspeckle) и Сoiled-coil (рис. 1а) [5]. Высоко консервативный участок белков DBHS состоит примерно из 300 аминокислотных остатков. Так, аминокислотные последовательности этого участка в белках SFPQ (Acc. no. P23246 в базе UniProt) и NONO (Acc. no. Q15233 в базе UniProt) человека имеют 72% идентичности согласно выравниванию с помощью сервиса Blast [6, 7]. Центральный участок DBHS-белков образует кор белка, в то время как N- и С-концевые участки не структурированы и значительно различаются внутри семейства, придавая каждому члену индивидуальные функции. Например, из всех членов семейства DBHS только SFPQ обладает ДНК-связывающим доменом, расположенным ближе к N-концу относительно центрального консервативного участка. Два РНК-связывающих домена в составе этих белков отвечают за взаимодействие с РНК, а домен Сoiled-сoil – за белок-белковые взаимодействия [5]. Все белки семейства DBHS содержат сигнал ядерной локализации на C-конце и в норме находятся в ядре, хотя появляются данные о возможных функциях этих белков в цитоплазме нервных клеток и на поверхности определенных опухолевых клеток [8, 9].
Рис. 1.
Структура белков семейства DBHS. а – Схематическое представление доменной организации белков семейства DBHS человека. Центральный консервативный регион показан одинаковыми цветами: РНК-связывающие домены RRM1 и RRM2, NOPS и домены Coiled-coil. N- и С-концевые участки белков являются регионами низкой сложности. ДНК-связывающий домен в составе SFPQ обозначен как DBD. NLS – сигнал ядерной локализации. б – Структура гомодимера SFPQ (276–598 а.к.), PDB код: 4WIJ. Домены обеих субъединиц выделены цветом. Регионы, отвечающие за димеризацию и полимеризацию димеров белка, обозначены в соответствии с [5, 10, лицензия Creative Commons CC-BY-NC].
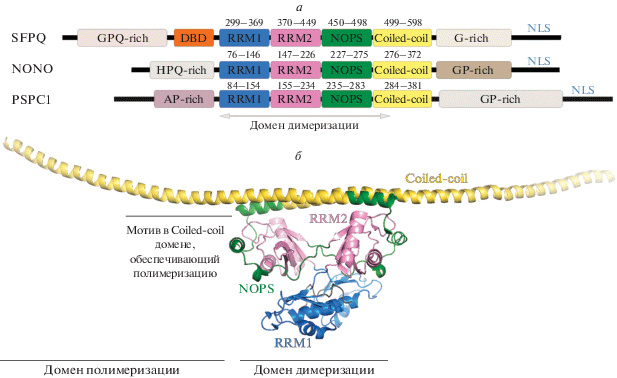
Белки семейства DBHS функционируют в виде димеров, при этом они способны образовывать как гомодимеры, так и гетеродимеры [5]. С помощью рентгеноструктурного анализа гетеродимера NONO/PSPC1 и гомодимера SFPQ установлено, что за димеризацию отвечают домены RRM2 и NOPS, взаимодействующие друг с другом внутри субъединиц (рис. 1б) [10, 11]. Мутации в этих доменах, мешающие димеризации, приводят к потере белком своих функций [10]. В зависимости от типа клеток и биологического процесса различные димеры могут быть как функционально взаимозаменяемыми, так и обладать уникальными функциями. Так, например, нокаут NONO может быть компенсирован повышением уровня PSPС1 для формирования гетеродимера с SFPQ при репарации ДНК [12]. Домен Сoiled-coil располагается вне кора в виде торчащей α-спирали, которая формирует поверхность для олигомеризации [10]. В этом домене находится консервативный мотив, отвечающий за обратимую олигомеризацию [11]. Показано, что способность SFPQ к олигомеризации важна для кооперативного связывания с нуклеиновыми кислотами, что также влияет на привлечение этого белка в параспекли [11].
Обнаружение чувствительности параспеклей к обработке РНКазами позволило предположить, что в качестве структурной основы они содержат некую РНК [13, 14]. В 2009 году четыре независимые исследовательские группы показали, что с параспеклями колокализуется длинная некодирующая РНК NEAT1, играющая важную структурную роль в организации этих телец [2, 4, 15, 16]. Оказалось, что снижение уровня NEAT1 приводит к дезорганизации параспеклей, снижению их количества и распределению белковых компонентов в нуклеоплазме. Именно путем взаимодействия с NEAT1 белки NONO, SFPQ и PSPС1 привлекаются в параспекли [15]. Интересно, что уровень РНК NEAT1 снижается при нокдауне белков SFPQ и NONO, что может также объяснить дезорганизацию параспеклей [4]. Таким образом, РНК NEAT1 – это ключевой компонент в образовании параспеклей: их формирование начинается в месте транскрипции NEAT1, когда ее концентрация достигает локального максимума [17]. Недавно обнаружили, что NEAT1 обогащена G-квадруплексными мотивами, а NONO специфично распознает именно эти мотивы в составе NEAT1 и за счет этого привлекается в параспекли [18].
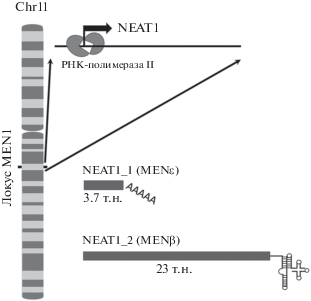
РНК NEAT1 транскрибируется РНК-полимеразой II с локуса MENI на хромосоме 11 человека, поэтому ее часто называют MENβ или MENε. Она имеет две изоформы: короткую, длиной порядка 3.7 т.н. (NEAT1_1, или MENε), и длинную порядка 23 т.н. (NEAT1_2, или MENβ). Оба транскрипта синтезируются с одного промотора (рис. 2), в них обоих отсутствуют интроны, но терминация транскрипции происходит по-разному. Изоформа NEAT1_1 полиаденилируется на 3'-конце классическим для транскриптов РНК-полимеразы II путем, а длинная изоформа NEAT1_2 процессируется неканонически [19]. На 3'-конце NEAT1_2 РНКаза Р узнает структуру в виде клеверного листа, похожую на структуру тРНК, и разрезает транскрипт после нее [16]. Эта структура важна для поддержания стабильности неполиаденилированной изоформы NEAT1 [20]. Обе изоформы NEAT1 из ядра не экспортируются, однако не предложен механизм их удержания в ядре. Показано, что белки SFPQ и NONO стабилизируют в первую очередь длинный транскрипт NEAT1_2, их нокдаун снижает уровень NEAT1_2 и практически не влияет на уровень короткого транскрипта NEAT1_1 [4]. Кроме того, уровень именно NEAT1_2 в клетке влияет на организацию параспеклей и их de novo формирование [3, 21], хотя в некоторых работах отмечено, что сверхэкспрессия короткой формы NEAT1_1 также способна несколько увеличивать количество параспеклей в клетке и инициировать их формирование [15, 22]. Вместе с тем, на клеточной линии U2OS показано, что короткая изоформа NEAT1_1 не важна для формирования параспеклей, более того NEAT1_1 локализуется в отличных от параспеклей участках, названных микроспеклями, функции и состав которых пока неясны [23].
Рис. 2.
Положение NEAT1 в геноме человека. Отмечены позиции локуса MEN I на хромосоме 11. Стрелка указывает направление транскрипции. Транскрибируемая РНК-полимеразой II РНК NEAT1 имеет две изоформы – NEAT1_1 и NEAT1_2, терминация которых происходит по-разному.
Параспекли обычно представляют собой вытянутые тельца. Оказалось, что диаметр поперечного сечения постоянен, его максимальный размер в клетках человека достигает в среднем 350 нM и не зависит от длины тельца [24]. Методом электронной микроскопии показано, что 5'- и 3'-концы длинной изоформы NEAT1_2 и 5'-конец короткой изоформы располагаются на периферии параспеклей, а центральный регион NEAT1_2 формирует внутренний кор, что также указывает на важность длинной изоформы для образования параспеклей [24]. Параспекли формируются в виде структур, похожих на сосиски (“sausage-like structures”), составленные из сфероидов, которые содержат центральный кор, включения и оболочку [25]. Кор состоит из белков семейства DBHS (SFPQ, NONO, PSPС1) и центрального региона РНК NEAT1_2. Оболочка параспеклей образована 5'- и 3'-концами NEAT1 и белком Tardbp (TAR DNA-binding protein 43), ограничивающими кор от нуклеоплазмы.
Основные функции параспеклей
Одна из наиболее изученных функций параспеклей – удержание в ядре РНК, подвергшихся множественному редактированию аденозина в инозин [2, 14, 26]. Белки SFPQ и NONO связывают гиперредактированные РНК и удерживают их в параспеклях, мешая экспорту из ядра [27]. Этот механизм предотвращает трансляцию редактированных РНК и используется для регуляции экспрессии некоторых генов [14]. В эмбриональных стволовых клетках редактирование A→I происходит особенно активно, но модифицированные РНК не удерживаются в ядре, что согласуется с отсутствием в эмбриональных стволовых клетках РНК NEAT1 и, соответственно, параспеклей. Однако после дифференцировки NEAT1 начинает экспрессироваться и регулировать удержание РНК в ядре [2].
Параспекли могут также влиять на протекание различных клеточных процессов, контролируя количество свободных форм белков, принимающих в них участие. Так, NEAT1 опосредованно участвует в регуляции транскрипции через секвестирование факторов транскрипции за счет их включения в параспекли [28, 29].
Интересно, что за счет своей способности регулировать количество белков в клетке параспекли могут быть вовлечены и в ответ клетки на вирусную инфекцию. Установлено, что SFPQ действует как репрессор промотора гена интерлейкина 8 (IL8) [29]. В ответ на введение поли-I/поли-C в клетках происходит активация клеточного иммунитета через узнавание двухцепочечных участков РНК Tоll-подобным рецептором 3 (TLR3). Это приводит к повышению уровня РНК NEAT1_2. Повышение уровня NEAT1 в свою очередь приводит к увеличению количества параспеклей и секвестированию в них SFPQ с промотора гена IL8. В результате этого активируется транскрипция IL8, а также других антивирусных генов. Уровень NEAT1 повышается и в ответ на инфицирование вирусом гриппа и вирусом простого герпеса, активирующих клеточный ответ через TLR3 [29]. В то же время, у пациентов с хроническим гепатитом В в стадии обострения уровень экспрессии NEAT1 существенно понижен, а уровень TLR3, наоборот, повышен [30].
Показано также участие NEAT1 и белков параспеклей в регуляции врожденного иммунного ответа клетки на присутствие чужеродной ДНК посредством активации пути cGAS–STING–IRF3 [31]. Интересно, что в этом случае NEAT1 служит основой для формирования большого рибонуклеопротеинового комплекса, в состав которого, помимо SFPQ и NONO, входят компоненты ДНК-зависимой протеинкиназы (Ku70, Ku80, DNA-PKcs) и белок HEXIM1, участвующий в регуляции элонгации транскрипции РНК-полимерозой II [32]. Описано также участие белков параспеклей в репликации вируса энцефаломиокардита (EMCV) [33]. Оказалось, что снижение внутриклеточного уровня SFPQ нарушало продукцию EMCV, в то время как его сверхэкспрессия способствовала репликации вируса.
Опубликованы данные о повышении уровня NEAT1 в ответ на заражение ВИЧ-1 [34, 35], хотя единые представления о том, как запускается внутриклеточный антивирусный ответ, отсутствуют. Так показано, что в мононуклеарных клетках периферической крови (PBMC) ВИЧ-1-инфицированных пациентов, не получающих антиретровирусную терапию, уровень РНК NEAT1 (в первую очередь, длинной изоформы NEAT1_2) в 3–5 раз выше, чем у здоровых доноров [34]. Уровень NEAT1 в клетках ВИЧ-положительных пациентов, применяющих терапию, не отличался значительно от его уровня у здоровых людей. Инфицирование клеток линий Jurkat, MT4 и THP1 вирусом также приводило к повышению уровня NEAT1_2 в 5–8 раз [35]. Следует также упомянуть роль NONO в cGAS-опосредованной активации иммунитета при ВИЧ-инфекции в дендритных клетках и макрофагах [36]. Так, NONO может взаимодействовать с cGAS и уровень cGAS в ядре коррелирует с уровнем NONO. Установлено также, что NONO непосредственно связывается с капсидным белком ВИЧ, а когда ассоциированный с вирусной ДНК капсид достигает ядра дендритных клеток, он взаимодействует в нем с NONO, который, в свою очередь, требуется для присутствия cGAS в ядре. В результате NONO выступает посредником в опосредованном cGAS сенсинге ДНК ВИЧ, запуская антивирусный иммунный ответ.
Более подробно участие NEAT1 и белков параспеклей в репликации ВИЧ-1 будет рассмотрено ниже.
Клеточные функции SFPQ и NONO, не связанные с параспеклями
Участие белков DBHS в формировании параспеклей считается отличительным свойством этого семейства. Тем не менее, обладая доменами для взаимодействия как с белками, так и с нуклеиновыми кислотами, белки DBHS могут служить посредниками в регуляции клеточных процессов, соединяя вместе разные компоненты в пространстве. Этим объясняется участие DBHS в широком спектре клеточных процессов и невозможность выделить одну основную их функцию.
Белки SFPQ и NONO участвуют в ответе на повреждения ДНК, в регуляции транскрипции, контроле стабильности мРНК, регуляции сплайсинга [5]. Показано, что SFPQ связывается с промоторами и может привлекать эпигенетические регуляторы, например гистондеацетилазы, для подавления экспрессии генов [37]. В другом механизме регуляции транскрипции белки SFPQ/NONO в форме гомо- или гетеродимеров связываются с целым рядом ядерных рецепторов гормонов, модулируя их транскрипционную активность [37–41]. Например, связавшись с андрогеновым рецептором, димер SFPQ/NONO мешает его взаимодействию с ДНК в регулируемых генах [39]. Кроме того, SFPQ и NONO способны одновременно взаимодействовать с С-концевым доменом РНК-полимеразы II и с транскрибирующейся РНК [42]. NONO участвует в активации транскрипции генов циркадного ритма, способствуя привлечению РНК-полимеразы II к сАМР-зависимым промоторам [43–45]. SFPQ и NONO участвуют также в привлечении 5'–3'-экзорибонуклеазы 2 (XRN2) к месту терминации транскрипции [46] и могут быть вовлечены в регуляцию стабильности РНК, например РНК NEAT1 [4].
Белки SFPQ/NONO неоднократно находили в составе сплайсосомы, однако они не являются важной ее частью. Предполагается, что эти белки участвуют в регуляции альтернативного сплайсинга некоторых мРНК [5].
Установлено, что белки SFPQ/NONO участвуют в репарации двухцепочечных разрывов ДНК по пути негомологичного объединения концов (NHEJ), стимулируя процесс соединения концов ДНК [47, 48]. Репарация двухцепочечных разрывов протекала медленнее в клетках с нокдауном NONO, в них накапливалось больше хромосомных аберраций, индуцированных ионизирующим излучением, по сравнению с клетками дикого типа [49]. Показана ассоциация SFPQ и NONO с белками системы NHEJ: гетеродимером Ku70/Ku80 и лигазой Lig4. Кроме того, обнаружено рекрутирование SFPQ и NONO к сайтам двухцепочечных повреждений в ДНК, а нокдаун SFPQ приводил к задержке репарации [50]. Показано также, что SFPQ взаимодействует с белками, участвующими в репарации по пути гомологичной рекомбинации – RAD51 [51, 52] и TopBP1 [53], стимулирует образование синаптического комплекса [54] и миграцию оцДНК поврежденной хроматиды вдоль сестринской хроматиды [51, 52]. Недавно установлено, что SFPQ и NONO могут участвовать в репарации повреждений ДНК, возникающих при химиотерапии трижды негативного рака молочной железы по PARP-зависимому пути [55]. Они взаимодействуют с белком-3, связывающим инсулиноподобный фактор роста (IGFBP-3), в результате чего повышается резистентность опухоли к терапии. Ранее обнаружили, что взаимодействие NONO с PARP1 в ходе репарации двухцепочечных разрывов ДНК стимулирует путь NHEJ и подавляет гомологичную репарацию [56].
Очень интересная функция гетеродимера SFPQ/NONO – участие в регуляции работы теломер [57]. Известно, что РНК TERRA с последовательностью (UUAGGG)n может образовывать РНК/ДНК-гибриды с С-богатой цепью теломерной ДНК, при этом формируется так называемая R-петля [58]. Эта петля вовлечена в регуляцию экспрессии некоторых генов, может изменять структуру хроматина, способствует рекомбинации. Кроме того, R-петля участвует в репликативном стрессе, повышает хрупкость хромосом, а также может активировать репарацию ДНК путем рекомбинации, что вызывает перестройку хромосом [59]. Предполагается, что гетеродимер SFPQ/NONO связывается с РНК TERRA, стабилизирует ее структуру и препятствует формированию ДНК/РНК-гибридов [57]. В результате подавляется образование R-петли, рекомбинация теломер, а также повышается стабильность хромосомы.
В заключение рассказа о клеточных функциях белков параспеклей, не связанных с РНК NEAT1 и самими параспеклями, необходимо отметить участие SFPQ в нейрогенезе и развитии нейродегенеративных заболеваний [60, 61]. Установлено, что в развивающемся мозге мыши SFPQ играет важную роль в поддержания элонгации транскрипции длинных генов [60], поскольку в развивающемся мозге некоторые гены экспрессируются с образованием гораздо более длинных транскриптов, чем в мозге взрослой мыши [62]. Фенотипически нарушение SFPQ или уменьшение его уровня вызывает апоптоз нейронов в развивающемся мозге мышей [60]. Многие из генов, экспрессирующих длинные РНК, связаны с нейродегенеративными и психическими заболеваниями, они важны для управления аксонами, миграции нейронов и образования синапсов, что указывает на важную нейропротекторную роль SFPQ во время развития и выживания нейронов [60].
Хотя SFPQ считается ядерным белком, обнаружен он был также в цитоплазме аксонов [63]. Установлено, что цитоплазматический SFPQ критически важен для развития аксонов у полосатого данио (Danio rerio). Особи, у которых отсутствовала цитоплазматическая форма SFPQ, умирали на 4 день своего развития.
Однако слишком высокая концентрация SFPQ в цитоплазме может иметь обратный эффект. Так, описано ядерное истощение и цитоплазматическая агрегация SFPQ при нейродегенеративных заболеваниях, таких как боковой амиотрофический склероз (БАС) [64], болезнь Альцгеймера (БА) [65] и лобно-височная деменция [66]. Причины подобных явлений не установлены, но существует предположение о возможном влиянии увеличения концентрации цинка в цитоплазме [67]. Цинк-индуцированная полимеризация SFPQ вызывает значительные конформационные изменения в домене Сoiled-сoil, что, вероятно, предотвращает функциональную полимеризацию SFPQ, опосредованную данным доменом. И, действительно, увеличение концентрации цинка в цитоплазме нейронов приводит к накоплению и агрегации SFPQ [67]. Более подробно участие SFPQ в развитии нейродегеративных заболеваниях рассмотрено в обзоре [68].
SFPQ, NONO и NEAT1 В РЕПЛИКАЦИИ ВИЧ-1
Как отмечено выше, благодаря своей многофункциональности, компоненты параспеклей SFPQ, NONO и NEAT1 вовлечены в различные клеточные процессы, а также в ответ клетки на инфицирование различными вирусами. В этой связи не вызывают удивления данные об их участии в репликативном цикле ВИЧ-1 на разных его этапах.
Жизненный цикл ВИЧ-1
ВИЧ-1 принадлежит к роду лентивирусов (Lentivirus) семейства ретровирусов (Retroviridae). Этот вирус поражает иммунную систему человека и вызывает синдром приобретенного иммунодефицита (СПИД). Геном ВИЧ-1, как и других лентивирусов, представлен одноцепочечной (+)РНК, на основе которой вирусная обратная транскриптаза синтезирует ДНК-копию геномной РНК вируса. Эта дцДНК в составе прединтеграционного комплекса (ПИК) транспортируется в ядро, где встраивается в геном зараженной клетки благодаря каталитической активности вирусного фермента интегразы. Далее начинается регулируемая экспрессия интегрированного провирусного генома. После инициации транскрипции синтезируются короткие абортивные транскрипты длиной около 60–80 н., которые образуют на 5′-конце стабильную шпильку, называемую TAR (trans-activation response element) РНК. Главным вирусным регулятором на стадии элонгации выступает вирусный белок Tat (trans-activator of transcription), который на порядки увеличивает эффективность синтеза вирусной РНК: только в присутствии Tat образуется 99% полноразмерных транскриптов [69]. Эти транскрипты в дальнейшем подвергаются альтернативному сплайсингу, в результате которого образуются мРНК, кодирующие все вирусные белки. Полностью сплайсированные вирусные мРНК, кодирующие белки Tat, Rev, Nef, экспортируются в цитоплазму, как и обычные клеточные мРНК. Несплайсированная и не полностью сплайсированные мРНК содержат интроны, и для экспорта таких РНК из ядра вирус использует свой регуляторный белок Rev [70–73]. Rev взаимодействует со структурированной областью в РНК, названной элементом, отвечающим на Rev (Rev-responsive element, RRE), расположенной в гене env. В комплексе с РНК Rev взаимодействует с экспортином 1 (Crm1) и обеспечивает таким образом экспорт вирусной РНК через ядерную пору. В отсутствие Rev несплайсированная и не полностью сплайсированные вирусные мРНК удерживаются в ядре и деградируют [73, 74]. Они содержат cis-действующие репрессирующие последовательности (CRS) и регионы нестабильности (INS), расположенные в генах gag/pol и env [74–77]. В цитоплазме начинается синтез вирусных белков, необходимых для сборки новых вирионов, и поздние этапы репликативного цикла завершаются сборкой и отпочковыванием вирусных частиц [70–72].
Влияние NONO и SFPQ на обратную транскрипцию и интеграцию
В ряде исследований, посвященных поиску клеточных белков, взаимодействующих с вирусными, обнаружены белки NONO и SFPQ [78–81]. Так, NONO идентифицирован в составе каталитически активного ПИК, выделенного из цитоплазматической фракции инфицированных CD4+ Т-клеток [78]. Кроме того, NONO найден среди белков комплекса обратной транскрипции и/или ПИК в T-клеточной линии, инфицированной ВИЧ-1 [80]. В инфицированной клеточной линии HeLa NONO обнаружен в составе интерактома вирусного белка Rev, при этом ассоциация NONO и Rev увеличивалась в присутствии вирусной мРНК, содержащей последовательность RRE [79]. В число белков-партнеров интегразы ВИЧ-1, найденных в ядерных лизатах клеток HeLa, входят NONO и SFPQ [81]. Кроме того, очищенные рекомбинантные интеграза и SFPQ могут взаимодействовать друг с другом. Методами докинга и молекулярной динамики предсказаны сайты связывания этих белков: во взаимодействии задействованы остатки N-концевого и каталитического доменов интегразы, а также остатки доменов RRM1, RRM2 и NOPS SFPQ [81], очень консервативные в белках DBHS. По данным молекулярной динамики со стороны интегразы в сайт связывания входит подвижная петля (аминокислотные остатки 140–149), которая располагается рядом с каталитическим центром интегразы и важна для позиционирования вирусной ДНК в каталитическом центре [81]. Однако в опытах с рекомбинантными белками SFPQ не влиял на реакции 3′-процессинга и переноса цепи, катализируемые интегразой [81]. Это достаточно странно, если предположить, что SFPQ действительно связывается c интегразой в непосредственной близости от ее каталитического центра.
При заражении клеток линии Jurkat и первичных CD4+ T-клеток репликативно-некомпетентным VSV-G-псевдотипированным лентивирусным вектором со встроенным геном люциферазы нокдаун NONO приводил к 2–3-кратному увеличению экспрессии люциферазы и вирусного белка p24, а также к повышению уровня мРНК, кодирующей p24 [82]. Нокдаун SFPQ также приводил к повышению в 3 раза уровня люциферазы в клетках TZM-bl, зараженных репликативно-некомпетентным VSV-G-псевдотипированным лентивирусным вектором [79]. Геном клеток линии TZM-bl, происходящей от клеток HeLa, содержит репортерный ген под контролем промотора ВИЧ-1. Эти клетки используются в качестве системы для тестирования эффективности репликации вируса. В клетках TZM-b1 вирус проходит все стадии жизненного цикла, включая сборку вирионов и заражение новых клеток, при этом вирусный белок Tat активирует транскрипцию интегрированного репортера, поэтому по уровню репортерного белка можно судить об эффективности репликации вируса. Таким образом, согласно [81, 82], SFPQ и NONO являются негативными регуляторами репликации ВИЧ-1.
С помощью ПЦР в реальном времени можно детектировать различные формы вирусной ДНК в клетке: общее количество обратно-транскрибированной, интегрированной и неинтегрированной форм, формирующих циклы 2-LTR и 1-LTR, и таким образом следить за эффективностью обратной транскрипции и интеграции. Оказалось, что нокдаун как NONO, так и SFPQ приводит к увеличению количества вирусной ДНК в клетке, влияя на стадию обратной транскрипции [81, 82], что могло быть одной из причин наблюдаемого в этих исследованиях повышения экспрессии вирусных и репортерных генов. Стоит, однако, отметить, что авторы не проверяли влияния SFPQ и NONO на транскрипцию с промотора ВИЧ-1, которое также могло бы объяснить отрицательный эффект SFPQ и NONO на экспрессию репортеров под контролем этого промотора. Отметим также, что NONO и SFPQ найдены в составе цитоплазматических комплексов, сформированных обратной транскриптазой и интегразой ВИЧ-1, и влияли на обратную транскрипцию, протекающую в цитоплазме клетки [78–82]. Механизм их присутствия в этих комплексах и участия в обратной транскрипции остается неизученным, но вызывает особый интерес, учитывая, что NONO и SFPQ это, как уже упоминалось, ядерные белки, хотя они и были найдены в составе рибонуклеопротеиновых гранул в цитоплазме ганглионарных нервных клеток сетчатки [9] и в цитоплазме аксонов [63].
Влияние NONO и SFPQ на уровень вирусных мРНК
Влияние SFPQ и NONO на экспрессию генов ВИЧ-1 изучали также с использованием трансфекции клеток репортерными плазмидами, кодирующими полный вирусный геном или его части. Обнаружено, что SFPQ и NONO соосаждаются с мРНК ВИЧ-1 [83–85], причем взаимодействие SFPQ и вирусной мРНК в клетке зависит от присутствия регионов нестабильности (INS) в гене gag [83]. Как уже упоминалось, РНК, кодирующие полипептид Gag и имеющие внутри открытой рамки считывания INS-последовательности, не подвергаются сплайсингу и экспортируются из ядра Rev-зависимым образом [74]. Показано также, что за взаимодействие SFPQ с вирусной несплайсированной мРНК отвечают консервативные последовательности в мРНК, расположенные вблизи 5′-концевого сайта сплайсинга недалеко от гена gag [85]. Кроме того, обнаружена РНК-зависимая ассоциация SFPQ с вирусным белком Rev [84], а также взаимодействие NONO с Rev, которое усиливалось в присутствии элемента RRE в составе вирусной мРНК [79]. Однако изучение функциональной значимости ассоциации SFPQ и NONO с вирусной мРНК и белком Rev не привело к получению однозначного результата.
Нокдаун SFPQ приводил к снижению уровня несплайсированных, содержащих INS мРНК и кодируемых ими белков, причем уровень этих мРНК менялся и в ядре, и в цитоплазме клеточной линии HEK293T [84]. При этом нокдаун SFPQ не влиял существенно на количество Rev-независимых сплайсированных форм мРНК, а также на уровень белков, транслирующихся с них. Этот эффект наблюдался только в присутствии вирусного белка Rev, когда несплайсированная вирусная РНК не подвергается сплайсингу полностью, может накапливаться в ядре и экспортироваться из него, а также легко детектируется методом нозерн-блотинга. Предполагается, что SFPQ оказывает позитивное влияние на стабильность несплайсированной вирусной РНК в ядре [84]. На клеточной линии HEK293T, наоборот, показано, что сверхэкспрессия SFPQ, меченного GFP, приводила к снижению уровня всех транскриптов ВИЧ-1 в присутствии белка Rev [83]. При этом значительно более сильным было влияние на несплайсированные и частично сплайсированные формы, содержащие INS и RRE. При использовании вектора, кодирующего геном ВИЧ-1 без гена белка Rev, все транскрипты полностью подвергались сплайсингу, и уровень сплайсированных форм отражал уровень/стабильность синтезированного полноразмерного предшественника. Сверхэкспрессия GFP-SFPQ значительно снижала количество полностью сплайсированных мРНК, при этом SFPQ не влиял на стадию транскрипции с LTR-промотора. Предполагается, что SFPQ негативно влияет на стабильность несплайсированной пре-мРНК ВИЧ-1 [83]. Итак, в обеих работах [83, 84] показано, что SFPQ участвует в поддержании уровня несплайсированной и частично сплайсированной вирусной мРНК, содержащей регионы нестабильности и RRE и экспортирующейся из ядра Rev-зависимым образом. Неясным все же остается механизм этого процесса, а также то, каким регулятором является SFPQ – позитивным или негативным.
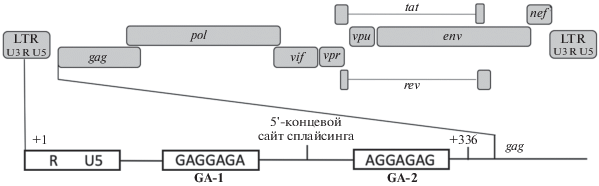
Позднее в мРНК ретровирусов идентифицировали два консервативных участка, окружающих 5′-сайт сплайсинга [85] (рис. 3). Эти участки состоят из семи рибонуклеотидов и содержат только G и A, поэтому они названы мотивами GA-1 и GA-2. При этом оба мотива содержатся только в несплайсированной мРНК, а при сплайсинге GA-2 вырезается. Мутации в этих мотивах приводят к снижению уровня несплайсированной мРНК и повышению уровня частично и полностью сплайсированных. Таким образом, в отсутствие GA-1 и GA-2 активируется альтернативный сплайсинг вирусной пре-мРНК, поэтому возможно, что, взаимодействуя с клеточными белками, привлекаемыми во время транскрипции, эти регионы поддерживают уровень несплайсированной вирусной мРНК, защищая ее от альтернативного сплайсинга.
В экспериментах in vitro с выделенным рекомбинантным белком и на культурах клеток показано, что SFPQ последовательность-специфичным образом связывает регионы GA-1 и GA-2 [85]. В этой же работе впервые с помощью метода ОТ-ПЦР в реальном времени проведен анализ уровня разных форм вирусной мРНК при сверхэкспрессии SFPQ или его нокдауне, тогда как в работах [83, 84] использовали только нозерн-блотинг и только одни условия. Установлено, что при эктопической сверхэкспрессии SFPQ снижались уровни несплайсированной и частично сплайсированной вирусной РНК, которая содержит INS и RRE, что соответствует данным [83]. Помимо этого, значительно снижался и уровень белка Gag, транслируемого с несплайсированной формы мРНК. Одновременно с этим нокдаун SFPQ также приводил к падению уровня белка Gag и уровня несплайсированной мРНК, хотя количество частично сплайсированной формы в этом исследовании увеличивалось. Этот результат согласуется с данными [84]. При этом стоит отметить, что влияние нокдауна SFPQ на уровни форм вирусной мРНК совпадало с эффектом замен в регионах GA-1 и GA-2, с которыми он взаимодействует. В то же время, нокдаун SFPQ не влиял на уровни форм вирусной мРНК, содержащих мутации в этих регионах. Учитывая, что GA-1 и GA-2, возможно, защищают несплайсированную вирусную мРНК от сплайсинга, предположили, что SFPQ участвует в этом процессе, а при его недостатке в клетке альтернативному сплайсингу подвергается большая часть вирусной мРНК. Вместе с тем, экспрессия экзогенного SFPQ не влияет на этот процесс, вероятно, из-за высокого содержания SFPQ в клетке. Отрицательный эффект сверхэкспрессии SFPQ на несплайсированные и частично сплайсированные РНК связывают с влиянием на поддержание уровня форм вирусной РНК после сплайсинга. Интересно, что в работе [85] влияние сверхэкспрессии SFPQ на уровни форм вирусной РНК, содержащей замены в регионах GA-1 и GA-2, не изучали. Результаты исследований, посвященных влиянию SFPQ на уровень различных форм вирусных мРНК и синтезируемых с них белков, суммированы в табл. 1.
Таблица 1.
Влияние SFPQ на уровень вирусных мРНК
| Эксперимент | Влияние на форму мРНК и/или белок, с нее синтезируемый | Вывод | Ссылка | ||
|---|---|---|---|---|---|
| несплайсированная, US | частично сплайсированная, SS | полностью сплайсированная, MS | |||
| Сверхэкспрессия GFP-SFPQ в HEK293, нозерн-блотинг | В присутствии Rev уровень US РНК снижается намного сильнее, чем других форм | В присутствии Rev снижение уровня SS РНК | В присутствии Rev снижение уровня MS РНК | Вызывает INS-зависимую деградацию пре-мРНК до сплайсинга и Rev-зависимого экспорта | [83] |
| Нокдаун SFPQ в HEK293T, нозерн-блотинг |
Значительное снижение количества РНК
в ядре и в цитоплазме только в присутствии Rev – Cнижение уровня Gag |
Н/Д | Нет изменения уровня репортера, кодируемого MS РНК | Поддержание стабильности несплайсированной пре-мРНК в ядре после ее транскрипции | [84] |
| Сверхэкспрессия Flag-SFPQ в HEK293T, ОТ-ПЦР в реальном времени |
Снижение уровня US РНК – Снижение уровня белка Gag | Снижение уровня SS РНК | Небольшое увеличение уровня MS РНК | Недостаток белка SFPQ в клетке стимулирует процессинг пре-мРНК, это влияние зависит
от наличия GA-мотивов около 5′-концевого сайта сплай-синга. В то же время, избыток экзогенного SFPQ, вероятно, негативно регулирует стабильность несплайсированной РНК уже после сплайсинга |
[85] |
| Нокдаун SFPQ в HEK 293T, ОТ-ПЦР в реальном времени |
Снижение US РНК – Снижение уровня белка Gag |
Увеличение уровня SS РНК | Небольшое увеличение уровня MS РНК | ||
Влияние NONO на стабильность вирусных мРНК изучено только в одной работе [83], в которой идентифицирована ассоциация NONO с INS-содержащей мРНК ВИЧ-1. Тем не менее сверхэкспрессия NONO, в отличие от SFPQ, никак не влияла на стабильность вирусной мРНК и на экспрессию вирусных генов. Это достаточно удивительный результат, учитывая, что NONO с SFPQ обладают высокой степенью идентичности коровых структур и в клетке часто формируют гетеродимер.
Влияние уровня NEAT1 и параспеклей в целом на репликацию ВИЧ-1
В клетке SFPQ и NONO могут как распределяться по нуклеоплазме в свободной форме, так и входить в состав параспеклей. В рассмотренных нами работах не уточнено, в какой именно форме белок вовлечен в регуляцию репликации ВИЧ-1 и какова роль непосредственно параспеклей в регуляции разных этапов жизненного цикла вируса. Как уже упоминалось, нокдаун NEAT1 ведет к дезорганизации параспеклей и переходу DBHS-белков в свободную форму в нуклеоплазме [2, 4, 15, 16]. Изучение влияния снижения уровня NEAT1, а следовательно, и снижения количества параспеклей на репликацию ВИЧ-1 показало, что при инфицировании клеток Jurkat и HeLa со сниженным содержанием NEAT1 эффективность репликации вируса повышается: увеличивается уровень белков-продуктов генов gag (p55 и p24) и env (gp160/120) [35, 86]. Это вирусные белки, экспрессирующиеся с несплайсированной или с частично сплайсированной мРНК, содержащей RRE и INS-элементы. Однако влияние нокдауна NEAT1 на ранние стадии репликации вируса (обратная транскрипция и интеграция) в [35] не отслеживали, а влияние NEAT1 на транскрипцию с LTR-промотора не было выявлено. Тем не менее, установлено, что усиление репликации вируса связано с повышением уровня несплайсированных INS-содержащих вирусных транскриптов только в цитоплазматической фракции, тогда как уровни сплайсированных форм не изменялись ни в одной из фракций. Нокаут NEAT1 в клетках Jurkat, зараженных вирусом, также приводит к повышению уровней белков p55 и p24 (продукты гена gag) и gp160/120 (продукты гена env), а также белка Nef, поскольку повышается уровень всех форм мРНК вируса в цитоплазме [86]. На основании всех этих данных можно заключить, что NEAT1 РНК и параспекли в целом каким-то образом участвуют в регуляции уровня и/или экспорта несплайсированной вирусной мРНК.
Известно, что одна из описанных функций параспеклей – удержание A→I гиперредактированной мРНК, содержащей двухцепочечные участки [2, 14, 26]. У высших эукариот такое редактирование дцРНК осуществляется в ядре ферментом дцРНК-зависимой аденозиндезаминазой (ADAR). Опубликован ряд наблюдений о редактировании двухцепочечных участков мРНК ВИЧ-1. Так, описано Tat-зависимое редактирование TAR РНК в ооцитах лягушки Xenopus [87], но редактирование этого региона в клетках человека не обнаружено [84, 88, 89]. Показана важность редактирующей активности дцРНК-зависимых ADAR1 и ADAR2 для посттранскрипционной регуляции ВИЧ-1 [88]. Обнаружено, что сверхэкспрессия каталитически активного ADAR1 в клетках HEK293T, трансфицированных вектором pNL4-3, кодирующим вирусный геном, повышает уровень белка p24, а нокдаун ADAR1 приводит, соответственно, к его снижению. Оказалось, что это связано с изменением уровня несплайсированной вирусной мРНК в цитоплазме, с которой p24 транслируется: при нокдауне ADAR1 он снижался. Показано, что регион несплайсированной мРНК около RRE-элемента и сам RRE-элемент подвергаются A→I редактированию. Рибосома при трансляции воспринимает инозин как гуанозин, поэтому в сам вектор pNL4-3 были внесены замены A→G в позициях, подвергающихся редактированию мРНК. Оказалось, что они приводят к повышению уровня экспрессии p24, аналогичному наблюдаемому при сверхэкспрессии ADAR1, что доказывает связь между редактированием A→I этих позиций и ADAR1-опосредованным повышением уровня p24 [88]. Однако в клетках HEK293T, трансфицированных вектором pNL4-3, при эндогенном уровне ферментов ADAR не обнаружили какого-либо значительного редактирования A→I в RRE-участке мРНК [84].
Показано также, что ADAR1 стимулирует инфекцию ВИЧ-1 [89]. При сверхэкспрессии и каталитически активной, и каталитически неактивной ADAR1 наблюдалось повышение уровня вирусных белков p24, gp120, Nef в клетках HEK293T, трансфицированных вектором pNL4-3. Однако только сверхэкспрессия каталитически активного белка стимулировала накопление вируса в среде, что определяли по уровню p24. Обнаружено, что ADAR1 связывается в клетке как с несплайсированной, так и со всеми сплайсированными формами вирусной мРНК. Кроме RRE-элемента, ADAR1-зависимому редактированию подвергается участок в 5'-нетранслируемой области, общей для всех форм мРНК [89]. Этот участок располагается в R-регионе вирусных LTR, он представляет собой шпильку с сигналом полиаденилирования AAUAAA, в 3'LTR он используется для терминации транскрипции.
Отметим, что удержание в ядре A→I гиперредактированной мРНК является важной функцией параспеклей. Однако гипотеза о том, что влияние параспеклей на репликацию ВИЧ-1 может быть опосредовано удержанием редактированной несплайсированной вирусной мРНК, содержащей RRE, не согласуется с приведенными выше данными. Во-первых, в работах [88, 89] редактирование последовательности RRE вирусной РНК при сверхэкспрессии ADAR1 приводило к активации экспрессии вирусных белков с этих РНК, что противоречит их удержанию в ядре в параспеклях. Во-вторых, негативное влияние параспеклей наблюдалось и при стандартном уровне ADAR [35, 86], при котором не обнаружено значительного редактирования RRE-участка [84].
Можно также отметить работу [90], в которой изучали образование крупных скоплений геномов ВИЧ-1 в ядрах инфицированных макрофагов. Эти скопления содержат как вирусную ДНК, так и геномную РНК, которая может подвергаться обратной транскрипции, приводящей к образованию транскрипционно-компетентной вирусной ДНК. Таким образом, наблюдаемая неинтегрированная ДНК [90] может быть связана с реактивацией ВИЧ в организме человека. Визуализация NEAT1 и NONO показала, что параспекли появлялись в непосредственной близости от кластеров вирусной ДНК, хотя и не перекрывались с ними. Интересно, что очаги вирусной ДНК/РНК были ассоциированы с белком CPSF6, входящим в состав параспеклей. Очевидно, непосредственная близость параспеклей и кластеров вирусной ДНК/РНК способствует перемещению CPSF6 из параспеклей в вирусные кластеры.
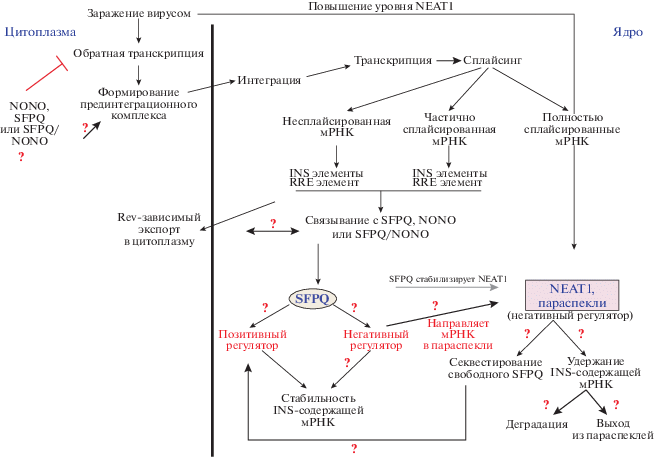
Исходя из полученных к настоящему времени данных, можно сделать вывод, что NEAT1 действует как негативный регулятор репликации вируса, и повышение уровня этой РНК в клетке в ответ на заражение ВИЧ-1 может рассматриваться как часть клеточного защитного механизма [34, 35]. Однако непонятно, как происходит регуляция уровня NEAT1 при инфицировании ВИЧ-1. Неясен и механизм опосредованного параспеклями подавления репликации. Таким механизмом может быть удержание несплайсированной или не полностью сплайсированных форм вирусных мРНК в параспеклях за счет взаимодействия с белками SFPQ и NONO. При этом, A→I редактирование вирусной РНК на этот процесс, по-видимому, не влияет. Секвестирование РНК в параспеклях могло бы мешать Rev-зависимому экспорту из ядра и/или каким-то образом регулировать деградацию этих РНК. Однако колокализацию вирусных транскриптов с SFPQ наблюдали только в нуклеоплазме, но не в параспеклях [84]. С другой стороны, возможно, что отрицательное влияние параспеклей заключается в секвестировании из нуклеоплазмы свободного SFPQ, который, в таком случае, мог бы там стабилизировать несплайсированные INS-содержащие вирусные мРНК. На рис. 4 суммированы варианты предполагаемого участия SFPQ, NONO и NEAT1 в репликации ВИЧ-1.
ЗАКЛЮЧЕНИЕ
Благодаря доменам для взаимодействия и с нуклеиновыми кислотами, и с белками, SFPQ и NONO вовлечены в различные клеточные процессы, но их отличает участие в формировании кора больших ядерных рибонуклеопротеиновых комплексов – параспеклей. Параспекли, состоящие из различных РНК-связывающих белков и длинной некодирующей РНК NEAT1, часто называют бесполезными для клетки, поскольку у них не найдено какой-то критической для жизни клетки функции. Считается, что они необходимы для удержания A→I гиперредактированных мРНК и секвестирования белков, входящих в состав параспеклей, из нуклеоплазмы. Последняя роль позволяет им опосредованно участвовать, например, в регуляции транскрипции, антивирусного ответа. И если описано участие параспеклей в активации врожденного иммунного ответа клетки на вирусную инфекцию, запускаемого через TLR3, и показан его механизм, то роль параспеклей в ответе на ВИЧ-инфекцию, очевидно, более сложная и пока неясная.
Анализ работ, в которых изучено участие белков параспеклей SFPQ и NONO, а также РНК NEAT1 в репликации ВИЧ-1, выявил несколько стадий, на которые эти факторы могут влиять: обратная транскрипция, интеграция, поддержание уровня несплайсированной вирусной мРНК. Однако полученные результаты противоречивы, не во всех работах проверено влияние этих белков на все этапы жизненного цикла вируса, что не позволяет составить полноценную картину участия в нем SFPQ, NONO и NEAT1 РНК. Мы находимся на этапе накопления данных о роли этих белков и РНК на разных стадиях репликации, однако уже сейчас можно проследить некоторые закономерности. По всей видимости, существует путь, приводящий к активации транскрипции NEAT1 в ответ на инфицирование клетки ВИЧ-1, как в случае вирусов гриппа и простого герпеса. Однако нет единого мнения о том, какие сигнальные каскады запускаются при заражении клеток разного типа ВИЧ-1, и неясно, как регулируется транскрипция NEAT1. Тем не менее, NEAT1, как опорный компонент параспеклей, подавляет репликацию ВИЧ-1. Прямое ли это влияние на вирусную мРНК или оно опосредовано регуляцией уровня белков, ассоциированных с параспеклями, непонятно. Кроме того, какую роль играют ядерные белки SFPQ и NONO в этой регуляции? Как они влияют на обратную транскрипцию вирусной РНК, происходящую в цитоплазме, действительно ли взаимодействуют с интегразой и какое функциональное значение имеют эти взаимодействия. В ряде работ показано влияние SFPQ на уровень вирусной несплайсированной мРНК. Во всех исследованиях использовали репортерный вектор, содержащий под контролем LTR-промотора ВИЧ-1 часть вирусного гена gag с самым изученным регионом INS-10 и участок RRE в 3′-нетранслируемой области этой мРНК, для обеспечения Rev-зависимого экспорта. Во всех исследованиях SFPQ влиял на экспрессию такой мРНК, но не ее мутанта с нарушенной последовательностью INS. Однако полноразмерная вирусная мРНК содержит и другие регионы нестабильности, важность которых для SFPQ-опосредованной регуляции пока не исследована.
Работа поддержана Российским фондом фундаментальных исследований (грант № 20-04-00437).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Fox A.H., Lam Y.W., Leung A.K.L., Lyon C.E., Andersen J., Mann M., Lamond A.I. (2002) Paraspeckles: a novel nuclear domain. Curr. Biol. 12, 13–25.
Chen L.-L., Carmichael G.G. (2009) Altered nuclear retention of mRNAs containing inverted repeats in human embryonic stem cells: functional role of a nuclear noncoding RNA. Mol. Cell. 35, 467–478.
Naganuma T., Nakagawa S., Tanigawa A., Sasaki Y.F., Goshima N., Hirose T. (2012) Alternative 3′-end processing of long noncoding RNA initiates construction of nuclear paraspeckles. EMBO J. 31, 4020–4034.
Sasaki Y.T.F., Ideue T., Sano M., Mituyama T., Hirose T. (2009) MENε/β noncoding RNAs are essential for structural integrity of nuclear paraspeckles. Proc. Natl. Acad. Sci. USA. 106, 2525–2530.
Knott G.J., Bond C.S., Fox A.H. (2016) The DBHS proteins SFPQ, NONO and PSPC1: a multipurpose molecular scaffold. Nucl. Acids Res. 44, 3989–4004.
Altschul S.F., Gish W., Miller W., Myers E.W., Lipman D.J. (1990) Basic local alignment search tool. J. Mol. Biol. 215, 403–410.
Blast [Internet]. Bethesda (MD): National Library of Medicine (US), National Center for Biotechnology Information; 2004. https://blast.ncbi.nlm.nih.gov/Blast.cgi
Ren S., She M., Li M., Zhou Q., Liu R., Lu H., Yang C., Xiong D. (2014) The RNA/DNA-binding protein PSF relocates to cell membrane and contributes cells’ sensitivity to antitumor drug, doxorubicin. Cytometry A. 85, 231–241.
Furukawa M.T., Sakamoto H., Inoue K. (2015) Interaction and colocalization of HERMES/RBPMS with NonO, PSF, and G3BP1 in neuronal cytoplasmic RNP granules in mouse retinal line cells. Genes Cells. 20, 257–266.
Passon D.M., Lee M., Rackham O., Stanley W.A., Sadowska A., Filipovska A., Fox A.H., Bond C.S. (2012) Structure of the heterodimer of human NONO and paraspeckle protein component 1 and analysis of its role in subnuclear body formation. Proc. Natl. Acad. Sci. USA. 109, 4846–4850.
Lee M., Sadowska A., Bekere I., Ho D., Gully B.S., Lu Y., Iyer K.S., Trewhella J., Fox A.H., Bond C.S. (2015) The structure of human SFPQ reveals a coiled-coil mediated polymer essential for functional aggregation in gene regulation. Nucl. Acids Res. 43, 3826–3840.
Li S., Li Z., Shu F.-J., Xiong H., Phillips A.C., Dynan W.S. (2014) Double-strand break repair deficiency in NONO knockout murine embryonic fibroblasts and compensation by spontaneous upregulation of the PSPC1 paralog. Nucl. Acids Res. 42, 9771–9780.
Fox A.H., Bond C.S., Lamond A.I. (2005) P54nrb forms a heterodimer with PSP1 that localizes to paraspeckles in an RNA-dependent manner. Mol. Biol. Cell. 16, 5304–5315.
Prasanth K.V., Prasanth S.G., Xuan Z., Hearn S., Freier S.M., Bennett C.F., Zhang M.Q., Spector D.L. (2005) Regulating gene expression through RNA nuclear retention. Cell. 123, 249–263.
Clemson C.M., Hutchinson J.N., Sara S.A., Ensminger A.W., Fox A.H., Chess A., Lawrence J.B. (2009) An architectural role for a nuclear noncoding RNA: NEAT1 RNA is essential for the structure of paraspeckles. Mol. Cell. 33, 717–726.
Sunwoo H., Dinger M.E., Wilusz J.E., Amaral. PP., Mattick J.S., Spector D.L. (2008) MEN/nuclear-retained non-coding RNAs are up-regulated upon muscle differentiation and are essential components of paraspeckles. Genome Res. 19, 347–359.
Carmo-Fonseca M., Rino J. (2011) RNA seeds nuclear bodies. Nat. Cell. Biol. 13, 110–112.
Simko E.A.J., Liu H., Zhang T., Velasquez A., Teli S., Haeusler A.R., Wang J. (2020) G-quadruplexes offer a conserved structural motif for NONO recruitment to NEAT1 architectural lncRNA. Nucl. Acids Res. 48, 7421–7438.
Naganuma T., Hirose T. (2013) Paraspeckle formation during the biogenesis of long non-coding RNAs. RNA Biol. 10, 456–461.
Wilusz J.E., JnBaptiste C.K., Lu L.Y., Kuhn C.-D., Joshua-Tor L., Sharp P.A. (2012) A triple helix stabilizes the 3' ends of long noncoding RNAs that lack poly(A) tails. Genes Dev. 26, 2392–2407.
Nakagawa S., Naganuma T., Shioi G., Hirose T. (2011) Paraspeckles are subpopulation-specific nuclear bodies that are not essential in mice. J. Cell. Biol. 193, 31–39.
Shevtsov S.P., Dundr M. (2011) Nucleation of nuclear bodies by RNA. Nat. Cell. Biol. 13, 167–173.
Li R., Harvey A.R., Hodgetts S.I., Fox A.H. (2017) Functional dissection of NEAT1 using genome editing reveals substantial localization of the NEAT1_1 isoform outside paraspeckles. RNA. 23, 872–881.
Souquere S., Beauclair G., Harper F., Fox A., Pierron G. (2010) Highly ordered spatial organization of the structural long noncoding NEAT1 RNAs within paraspeckle nuclear bodies. Mol. Biol. Cell. 21, 4020–4027.
West J.A., Mito M., Kurosaka S., Takumi T., Tanegashima C., Chujo T., Yanaka K., Kingston R.E., Hirose T., Bond C., Fox A., Nakagawa S. (2016) Structural, super-resolution microscopy analysis of paraspeckle nuclear body organization. J. Cell. Biol. 214, 817–830.
Chen L.-L., DeCerbo J.N., Carmichael G.G. (2008) Alu element-mediated gene silencing. EMBO J. 27, 1694–1705.
Zhang Z., Carmichael G.G. (2001) The fate of dsRNA in the nucleus. Cell. 106, 465–476.
Hirose T., Virnicchi G., Tanigawa A., Naganuma T., Li R., Kimura H., Yokoi T., Nakagawa S., Bénard M., Fox A.H., Pierron G. (2014) NEAT1 long noncoding RNA regulates transcription via protein sequestration within subnuclear bodies. Mol. Biol. Cell. 25, 169–183.
Imamura K., Imamachi N., Akizuki G., Kumakura M., Kawaguchi A., Nagata K., Kato A., Kawaguchi Y., Sato H., Yoneda M., Kai C., Yada T., Suzuki Y., Yamada T., Ozawa T., Kaneki K., Inoue T., Kobayashi M., Kodama T., Wada Y., Sekimizu K., Akimitsu N. (2014) Long noncoding RNA NEAT1-dependent SFPQ relocation from promoter region to paraspeckle mediates IL8 expression upon immune stimuli. Mol. Cell. 53, 393–406.
Zeng Y., Wu W., Fu Y., Chen S., Chen T., Yang B., Ou Q. (2019) Toll-like receptors, long non-coding RNA NEAT1, and RIG-I expression are associated with HBeAg-positive chronic hepatitis B patients in the active phase. J. Clin. Lab. Anal. 33, e22886.
Morchikh M., Cribier A., Raffel R., Amraoui S., Cau J., Severac D., Dubois E., Schwartz O., Bennasser Y., Benkirane M. (2017) HEXIM1 and NEAT1 long non-coding RNA form a multi-subunit complex that regulates DNA-mediated innate immune response. Mol. Cell. 67, 387–399.e5.
Li Q., Cooper J.J., Altwerger G.H., Feldkamp M.D., Shea M.A., Price D.H. (2007) HEXIM1 is a promiscuous double-stranded RNA-binding protein and interacts with RNAs in addition to 7SK in cultured cells. Nucl. Acids Res. 35, 2503–2512.
Zhou B., Wu F., Han J., Qi F., Ni T., Qian F. (2019) Exploitation of nuclear protein SFPQ by the encephalomyocarditis virus to facilitate its replication. Biochem. Biophys. Res. Commun. 510, 65–71.
Jin C., Peng X., Xie T., Lu X., Liu F., Wu H., Yang Z., Wang J., Cheng L., Wu N. (2016) Detection of the long noncoding RNAs nuclear-enriched autosomal transcript 1 (NEAT1) and metastasis associated lung adenocarcinoma transcript 1 in the peripheral blood of HIV-1-infected patients. HIV Med. 17, 68–72.
Zhang Q., Chen C.-Y., Yedavalli V.S.R.K., Jeang K.-T. (2013) NEAT1 long noncoding RNA and paraspeckle bodies modulate HIV-1 posttranscriptional expression. MBio. 4, e00596–e00612.
Lahaye X., Gentili M., Silvin A., Conrad C., Picard L., Jouve M., Zueva E., Maurin M., Nadalin F., Knott G.J., Zhao B., Du F., Rio M., Amiel J., Fox A. H., Li P., Etienne L., Bond C.S., Colleaux L., Manel N. (2018) NONO detects the nuclear HIVcapsid to promote cGAS-mediated innate immune activation. Cell. 175, 488–501.e22.
Mathur M., Tucker P.W., Samuels H.H. (2001) PSF is a novel corepressor that mediates its effect through Sin3A and the DNA binding domain of nuclear hormone receptors. Mo.l Cell. Biol. 21, 2298–2311.
Dong X., Shylnova O., Challis J.R.G., Lye S.J. (2005) Identification and characterization of the protein-associated splicing factor as a negative co-regulator of the progesterone receptor. J. Biol. Chem. 280, 13329–13340.
Dong X., Sweet J., Challis J.R.G., Brown T., Lye S.J. (2007) Transcriptional activity of androgen receptor is modulated by two RNA splicing factors, PSF and p54nrb. Mol. Cell. Biol. 27, 4863–4875.
Dong X., Yu C., Shynlova O., Challis J.R.G., Rennie P.S., Lye S.J. (2009) p54nrb is a transcriptional corepressor of the progesterone receptor that modulates transcription of the labor-associated gene, connexin 43 (Gja1). Mol. Endocrinol. 23, 1147–1160.
Ishitani K., Yoshida T., Kitagawa H., Ohta H., Nozawa S., Kato S. (2003) p54nrb acts as a transcriptional coactivator for activation function 1 of the human androgen receptor. Biochem. Biophys. Res. Commun. 306, 660–665.
Emili A., Shales M., McCracken S., Xie W., Tucker P.W., Kobayashi R., Blencowe B.J., Ingles C.J. (2002) Splicing and transcription-associated proteins PSF and p54nrb/nonO bind to the RNA polymerase II CTD. RNA. 8, 1102–1111.
Amelio A.L., Miraglia L.J., Conkright J.J., Mercer B.A., Batalov S., Cavett V., Orth A.P., Busby J., Hogenesch J.B., Conkright M.D. (2007) A coactivator trap identifies NONO (p54nrb) as a component of the cAMP-signaling pathway. Proc. Natl. Acad. Sci. USA. 104, 20314–20319.
Ong S.A., Tan J.J., Tew W.L., Chen K.-S. (2011) Rasd1 modulates the coactivator function of NonO in the cyclic AMP pathway. PLoS One. 6, e24401.
Duong H.A., Robles M.S., Knutti D., Weitz C.J. (2011) A molecular mechanism for circadian clock negative feedback. Science. 332(6036), 1436–1439.
Kaneko S., Rozenblatt-Rosen O., Meyerson M., Manley J.L. (2007) The multifunctional protein p54nrb/ PSF recruits the exonuclease XRN2 to facilitate pre-mRNA 3’ processing and transcription termination. Genes Dev. 21, 1779–1789.
Bladen C.L., Udayakumar D., Takeda Y., Dynan W.S. (2005) Identification of the polypyrimidine tract binding protein-associated splicing factor·p54(nrb) complex as a candidate DNA double-strand break rejoining factor. J. Biol. Chem. 280, 5205–5210.
Udayakumar D., Dynan W.S. (2015) Characterization of DNA binding and pairing activities associated with the native SFPQ·NONO DNA repair protein complex. Biochem. Biophys. Res. Commun. 463, 473–478.
Li S., Kuhne W.W., Kulharya A., Hudson F.Z., Ha K., Cao Z., Dynan W.S. (2009) Involvement of p54(nrb), a PSF partner protein, in DNA double-strand break repair and radioresistance. Nucl. Acids Res. 37, 6746–6753.
Salton M., Lerenthal Y., Wang S.-Y., Chen D.J., Shiloh Y. (2010) Involvement of matrin 3 and SFPQ/NONO in the DNA damage response. Cell Cycle. 9, 1568–1576.
Morozumi Y., Takizawa Y., Takaku M., Kurumizaka H. (2009) Human PSF binds to RAD51 and modulates its homologous-pairing and strand-exchange activities. Nucl. Acids Res. 37, 4296–4307.
Rajesh C., Baker D.K., Pierce A.J., Pittman D.L. (2011) The splicing-factor related protein SFPQ/PSF interacts with RAD51D and is necessary for homology-directed repair and sister chromatid cohesion. Nucl. Acids Res. 39, 132–145.
Kuhnert A., Schmidt U., Monajembashi S., Franke C., Schlott B., Grosse F., Greulich K.O., Saluz H.-P., Hänel F. (2011) Proteomic identification of PSF and p54(nrb) as TopBP1-interacting proteins. J. Cell. Biochem. 113, 1744–1753.
Morozumi Y., Ino R., Takaku M., Hosokawa M., Chuma S., Kurumizaka H. (2012) Human PSF concentrates DNA and stimulates duplex capture in DMC1-mediated homologous pairing. Nucl. Acids Res. 40, 3031–3041.
de Silva H., Lin M., Phillips L., Martin L., Baxter R. (2019) IGFBP-3 interacts with NONO and SFPQ in PARP-dependent DNA damage repair in triple-negative breast cancer. Cell. Mol. Life Sci. 76, 2015–2030.
Krietsch J., Caron M.-C., Gagné J.-P., Ethier C., Vignard J., Vincent M., Rouleau M., Hendzel M.J., Poirier G.G., Masson J.-Y. (2012) PARP activation regulates the RNA-binding protein NONO in the DNA damage response to DNA double-strand breaks. Nucl. Acids Res. 40, 10287–10301.
Petti E., Buemi V., Zappone A., Schillaci O., Broccia P.V., Dinami R., Matteoni S., Benetti R., Schoeftner S. (2019) SFPQ and NONO suppress RNA:DNA-hybrid-related telomere instability. Nat. Commun. 10, 1–14.
Santos-Pereira J.M., Aguilera A. (2015) R loops: new modulators of genome dynamics and function. Nat. Rev. Genet. 16, 583–597.
Skourti-Stathaki K., Proudfoot N. (2014) A double-edged sword: R loops as threats to genome integrity and powerful regulators of gene expression. Genes Dev. 28, 1384–1396.
Takeuchi A., Iida K., Tsubota T., Hosokawa M., Denawa M., Brown J.B., Ninomiya K., Ito M., Kimura H., Abe T., Kiyonari H., Ohno K., Hagiwara M. (2018) Loss of Sfpq causes long-gene transcriptopathy in the brain. Cell Rep. 23, 1326–1341.
Iida K., Hagiwara M., Takeuchi A. (2020) Multilateral bioinformatics analyses reveal the function-oriented target specificities and recognition of the RNA-binding protein SFPQ. iScience. 23, 101325.
Gabel H.W., Kinde B., Stroud H., Gilbert C.S., Harmin D.A., Kastan N.R., Hemberg M., Ebert D.H., Greenberg M.E. (2015) Disruption of DNA-methylation-dependent long gene repression in Rett syndrome. Nat. 522, 89–93.
Thomas-Jinu S., Gordon P.M., Fielding T., Taylor R., Smith B.N., Snowden V., Blanc E., Vance C., Topp S., Wong C.H., Bielen H., Williams K.L., McCann E.P., Nicholson G.A., Pan-Vazquez A., Fox A.H., Bond C.S., Talbot W.S., Blair I.P., Shaw C.E., Houart C. (2017) Non-nuclear pool of splicing factor SFPQ regulates axonal transcripts required for normal motor development. Neuron. 94, 322–336.e5.
Luisier R., Tyzack G.E., Hall C.E., Mitchell J.S., Devine H., Taha D.M., Malik B., Meyer I., Green-smith L., Newcombe J., Ule J., Luscombe N.M., Patani R. (2018) Intron retention and nuclear loss of SFPQ are molecular hallmarks of ALS. Nat. Commun. 9, 2010.
Younas N., Zafar S., Shafiq M., Noor A., Siegert A., Arora A., Galkin A., Zafar A., Schmitz M., Stadelmann C., Andreoletti O., Ferrer I., Zerr I. (2020) SFPQ and Tau: critical factors contributing to rapid progression of Alzheimer’s disease. Acta Neuropathol. 140, 317–339.
Ishigaki S., Riku Y., Fujioka Y., Endo K., Iwade N., Kawai K., Ishibashi M., Yokoi S., Katsuno M., Watanabe H., Mori K., Akagi A., Yokota O., Terada S., Kawakami I., Suzuki N., Warita H., Aoki M., Yoshida M., Sobue G. (2020) Aberrant interaction between FUS and SFPQ in neurons in a wide range of FTLD spectrum diseases. Brain. 143, 2398–2405.
Huang J., Ringuet M., Whitten A., Caria S., Lim Y., Badhan R., Anggono V., Lee M. (2020) Structural basis of the zinc-induced cytoplasmic aggregation of the RNA-binding protein SFPQ. Nucl. Acids Res. 48, 3356–3365.
Lim Y., James D., Huang J., Lee M. (2020) The emerging role of the RNA-binding protein SFPQ in neuronal function and neurodegeneration. Int. J. Mol. Sci. 21, 1–16.
Ruelas D.S., Greene W.C. (2013) An integrated overview of HIV-1 latency. Cell. 155, 519–529.
Yedavalli V.S.R.K., Jeang K.-T. (2011) Rev-ing up post-transcriptional HIV-1 RNA expression. RNA Biol. 8, 195–199.
Dayton A.I. (2004) Within you, without you: HIV-1 Rev and RNA export. Retrovirology. 1, 35.
LeBlanc J., Weil J., Beemon K. (2013) Posttranscriptional regulation of retroviral gene expression: primary RNA transcripts play three roles as pre-mRNA, mRNA, and genomic RNA. WIREs RNA. 4, 567–580.
Karn J., Stoltzfus C.M. (2012) Transcriptional and posttranscriptional regulation of HIV-1 gene expression. Cold Spring Harb Perspect Med. 2, a006916–a006916.
Toro-Ascuy D., Rojas-Araya B., Valiente-Echeverría F., Soto-Rifo R. (2016) Interactions between the HIV-1 unspliced mRNA and host mRNA decay machineries. Viruses. 8, 320.
Schwartz S., Campbell M., Nasioulas G., Harrison J., Felber B.K., Pavlakis G.N. (1992) Mutational inactivation of an inhibitory sequence in human immunodeficiency virus type 1 results in Rev-independent gag expression. J. Virol. 66, 7176–7182.
Schwartz S., Felber B.K., Pavlakis G.N. (1992) Distinct RNA sequences in the gag region of human immunodeficiency virus type 1 decrease RNA stability and inhibit expression in the absence of Rev protein. J. Virol. 66, 150–159.
Schneider R., Campbell M., Nasioulas G., Felber B.K., Pavlakis G.N. (1997) Inactivation of the human immunodeficiency virus type 1 inhibitory elements allows Rev-independent expression of Gag and Gag/protease and particle formation. J. Virol. 71, 4892–4903.
Raghavendra N.K., Shkriabai N., Graham R.L., Hess S., Kvaratskhelia M., Wu L. (2010) Identification of host proteins associated with HIV-1 preintegration complexes isolated from infected CD4+ cells. Retrovirology. 7, 66.
Naji S., Ambrus G., Cimermančič P., Reyes J.R., Johnson J.R., Filbrandt R., Huber M.D., Vesely P., Krogan N.J., Yates J.R., Saphire A.C., Gerace L. (2012) Host cell interactome of HIV-1 Rev includes RNA helicases involved in multiple facets of virus production. Mol. Cell. Proteomics. 11, M111.015313.
Schweitzer C.J., Jagadish T., Haverland N., Ciboro-wski P., Belshan M. (2013) Proteomic analysis of early HIV-1 nucleoprotein complexes. J. Proteome Res. 12, 559–572.
Yadav P., Sur S., Desai D., Kulkarni S., Sharma V., Tandon V. (2019) Interaction of HIV-1 integrase with polypyrimidine tract binding protein and associated splicing factor (PSF) and its impact on HIV-1 replication. Retrovirology. 16, 1–18.
St. Gelais C., Roger J., Wu L. (2015) Non-POU domain-containing octamer-binding protein negatively regulates HIV-1 infection in CD4+ T cells. AIDS Res. Hum. Retroviruses. 31, 806–816.
Zolotukhin A.S., Michalowski D., Bear J., Smulevitch S.V., Traish A.M., Peng R., Patton J., Shatsky I.N., Felber B.K. (2003) PSF acts through the human immunodeficiency virus type 1 mRNA instability elements to regulate virus expression. Mol. Cell Biol. 23, 6618–6630.
Kula A., Gharu L., Marcello A. (2013) HIV-1 pre-mRNA commitment to Rev mediated export through PSF and Matrin 3. Virology. 435, 329–340.
Singh G., Rife B.D., Seufzer B., Salemi M., Rendahl A., Boris-Lawrie K. (2018) Identification of conserved, primary sequence motifs that direct retrovirus RNA fate. Nucl. Acids Res. 46, 7366–7378.
Liu H., Hu P.-W., Couturier J., Lewis D.E., Rice A.P. (2018) HIV-1 replication in CD4+ T cells exploits the down-regulation of antiviral NEAT1 long non-coding RNAs following T cell activation. Virology. 522, 193–198.
Sharmeen L., Bass B., Sonenberg N., Weintraub H., Groudine M. (1991) Tat-dependent adenosine-to-inosine modification of wild-type transactivation response RNA. Proc. Natl. Acad. Sci. USA. 88, 8096–8100.
Phuphuakrat A., Kraiwong R., Boonarkart C., Lauhakirti D., Lee T.-H., Auewarakul P. (2008) Double-stranded RNA adenosine deaminases enhance expression of human immunodeficiency virus type 1 proteins. J. Virol. 82, 10864–10872.
Doria M., Neri F., Gallo A., Farace M.G., Michienzi A. (2009) Editing of HIV-1 RNA by the double-stranded RNA deaminase ADAR1 stimulates viral infection. Nucl. Acids Res. 37, 5848–5858.
Rensen E., Mueller F., Scoca V., Parmar J.J., Souque P., Zimmer C., Nunzio F.Di. (2021) Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40, e105247.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология