Молекулярная биология, 2022, T. 56, № 3, стр. 476-490
Распространенность, разнообразие и эволюция ДНК-транспозонов L18 (DD37E) в геномах стрекающих (Cnidaria)
М. В. Пузаков a, *, Л. В. Пузакова a
a Федеральный исследовательский центр “Институт биологии южных морей им. А.О. Ковалевского”
Российской академии наук
299011 Севастополь, Россия
* E-mail: puzakov@ngs.ru
Поступила в редакцию 02.10.2021
После доработки 02.11.2021
Принята к публикации 03.11.2021
- EDN: BFJKIE
- DOI: 10.31857/S0026898422030120
Аннотация
Мобильные генетические элементы оказывают значительное влияние на структуру и функционирование геномов многоклеточных, а также служат источником новых генов. Изучение разнообразия и эволюции мобильных генетических элементов в различных таксонах необходимо для понимания их роли в геномах. Мобильные элементы Tc1/mariner образуют одну из наиболее распространенных и разнообразных групп ДНК-транспозонов. Нами впервые изучены структура, распространение, разнообразие и эволюция элементов L18 (DD37E) в геномах стрекающих (Cnidaria). Установлено, что группа L18 является самостоятельным семейством (а не подсемейством семейства TLE, как считалось ранее), входящим в суперсемейство Tc1/mariner. Потенциально функциональные копии найдены только у четырех элементов из 51. Предполагается, что L18-транспозоны имеют древнее происхождение, а элементы, обнаруженные в геномах представителей классов Anthozoa и Hydrozoa, не имеют общего предка внутри типа Cnidaria. У представителей класса Hydrozoa транспозоны L18 появились в результате горизонтального переноса в более поздний период времени. Внутривидовое сравнение разнообразия элементов L18 выявляет высокую гомогенность по “старым” транспозонам, которые уже утратили свою активность. В то же время отдаленные популяции, как в случае Hydra viridissima, различаются представленностью ДНК-транспозонов и количеством их копий. Полученные данные дополняют знания о разнообразии и эволюции транспозонов Tc1/mariner и будут способствовать изучению влияния мобильных генетических элементов на эволюцию многоклеточных.
ВВЕДЕНИЕ
Распространение и разнообразие мобильных генетических элементов (МГЭ) активно изучается в различных группах организмов, от бактерий до человека. Влияние МГЭ на модификацию и регуляцию генов изучают с момента их открытия (уже более 60 лет). МГЭ, будучи основными детерминантами размера генома [1–4], также вносят значительный вклад в процессы изменчивости, адаптации и эволюции в целом [2, 5, 6]. Известно, что МГЭ – это не только фрагменты ДНК, способные перемещаться в геноме хозяина и увеличивать свою копийность. Внедряясь в геном хозяина, МГЭ могут прицельно встраиваться в наиболее предпочтительные для них геномные локусы [7], проходить определенные этапы своего жизненного цикла [8], взаимодействовать друг с другом по принципу паразитизм/сотрудничество/конкуренция [9, 10]. Геном же, в свою очередь, способен регулировать активность МГЭ [11–13] и, более того, “использовать” внедрившийся генетический материал в своих целях. Есть данные, свидетельствующие о том, что МГЭ одомашниваются, превращаясь в гены, и участвуют в формировании теломерных концов [14–16], в связывании с центромерами [17, 18], а также в процессах адаптации и стрессового ответа, в формировании устойчивости к вирусным инфекциям и химическим веществам [6, 19].
МГЭ эукариот делят на ретротранспозоны и ДНК-транспозоны, опираясь на механизмы их перемещения. Ретротранспозоны перемещаются по принципу “копирование–вставка”, создавая РНК-посредник, который транскрибируется в кДНК с помощью фермента обратной транскриптазы и интегрируется в геном [20–22]. ДНК-транспозоны перемещаются по принципу “вырезание–вставка”, не создавая посредников, а непосредственно вырезаясь из прежнего места нахождения в геноме и встраиваясь в новое с помощью фермента транспозазы [20–22].
ДНК-транспозоны образуют очень разнообразную и многочисленную группу МГЭ, включающую три подкласса и не менее 17 суперсемейств [21, 23]. Одной из широко распространенных групп ДНК-транспозонов у эукариот является Tc1/mariner [24].
Транспозоны Tc1/mariner обнаружены у самых различных организмов, таких как коловратки, грибы, растения, рыбы и млекопитающие [23, 25]. Транспозоны Tc1/mariner несут, как правило, одну открытую рамку считывания, которая кодирует фермент транспозазу протяженностью от 300 до 400 аминокислотных остатков. С обоих концов от гена транспозазы расположены концевые инвертированные повторы (КИП) длиной в среднем 20–30 п.н. Длина Tc1/mariner-транспозонов составляет примерно 1.3–2.4 т.п.н. [26–28].
Транспозаза Tc1/mariner содержит ДНК-связывающий (PAIRED) и каталитический (DDE/D) домены [26]. PAIRED-домен расположен в первой половине аминокислотной последовательности транспозазы (N-концевой части) и состоит из шести α-спиралей – первые три – это PAI-субдомен, вторые три – RED-субдомен. Между этими субдоменами располагается GRPR-подобный мотив. Считается, что этот мотив опосредует связывание домена PAIRED с ДНК-мишенью [29]. Вторая половина транспозазы (С-концевая часть) содержит DDE/D-домен, который обладает эндонуклеазной и лигирующей активностью, необходимой для вырезания и вставки МГЭ. Также транспозаза Tc1/mariner содержит сигнал ядерной локализации (NLS-сигнал), который, предположительно, способствует транспорту транспозазы через ядерную мембрану [30, 31].
Классификация суперсемейства Tc1/mariner с каждым годом меняется и дополняется. На данный момент группа Tc1/mariner объединяет несколько семейств: TLE (DD34-46E), MLE (DD34D), maT (DD37D), Guest (DD39D), Visitor (DD41D) и mosquito (DD37E) [26, 27, 32–34].
Группа транспозонов Tc1/mariner, названная L18 и имеющая каталитический домен DD37E, найдена при изучении МГЭ тихоокеанской устрицы Crassostrea gigas [35]. Эта группа включена в семейство Tc1-подобных элементов [28, 35]. Транспозазы L18 обнаружены в геномах некоторых двустворчатых, ракообразных, насекомых, клещей, рыб, стрекающих и иглокожих. В настоящий момент структура, распространение, разнообразие и эволюция L18-транспозонов детально не изучены, поскольку проведен только филогенетический анализ последовательностей транспозаз.
Нами изучены L18-транспозоны стрекающих. Стрекающие, или Cnidaria – тип многоклеточных животных, обитающих в водной среде и имеющих стрекательные клетки. Тип Cnidaria появился около 741 миллионов лет назад (м.л.н.) (http://www.timetree.org), он включает шесть классов – Anthozoa, Cubozoa, Hydrozoa, Myxozoa, Scyphozoa, Staurozoa, а также две клады, филогенетические отношения которых не прояснены, и носящих временные названия (https://www.marinespecies.org). Помимо изучения распространения, разнообразия и эволюции L18-транспозонов в геномах стрекающих, наша работа дает новое видение их филогенетического положения и, следовательно, классификации.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Поиск L18-транспозонов. Поиск L18-транспозонов (DD37E) мы проводили с помощью tBLASTn со стандартными настройками (https://blast.ncbi. nlm.nih.gov/Blast.cgi). В качестве образца использовали аминокислотную последовательность транспозазы элемента Mariner-18_CGi устрицы C. gigas [35].
Полногеномные нуклеотидные последовательности представителей типа Cnidaria получены из базы данных NCBI Assembly (https://www.ncbi. nlm.nih.gov/assembly/). Полный список проанализированных сборок геномов представлен в табл. 1.
Таблица 1.
Сборки геномов представителей типа Cnidaria, использованные в работе
| Вид | Идентификатор GenBank | Вид | Идентификатор GenBank |
|---|---|---|---|
| Acropora acuminata | GCA_014633975.1 | A. awi | GCA_014634005.1 |
| A. cytherea | GCA_014634045.1 | A. digitifera | GCA_000222465.2 |
| A. digitifera | GCA_014634065.1 | A. echinata | GCA_014634105.1 |
| A. florida | GCA_014634605.1 | A. gemmifera | GCA_014634125.1 |
| A. hyacinthus | GCA_020536085.1 | A. hyacinthus | GCA_014634145.1 |
| A. intermedia | GCA_014634585.1 | A. microphthalma | GCA_014634165.1 |
| A. millepora | GCA_013753865.1 | A. millepora | GCA_004143615.1 |
| A. muricata | GCA_014634545.1 | A. nasuta | GCA_014634205.1 |
| A. selago | GCA_014634525.1 | A. tenuis | GCA_014633955.1 |
| A. yongei | GCA_014634225.1 | Ac. equina | GCA_011057435.1 |
| Actinia tenebrosa | GCA_009602425.1 | Alatina alata | GCA_008930755.1 |
| Alatinidae sp. | GCA_010016025.1 | Anemonia viridis | GCA_900234385.1 |
| Anthopleura sola | GCA_016068315.1 | Astreopora myriophthalma | GCA_014634185.1 |
| Aurelia aurita | GCA_004194415.1 | Au. aurita complex sp. | GCA_004194395.1 |
| Au. coerulea | GCA_011634815.1 | Calvadosia cruxmelitensis | GCA_900245855.1 |
| Carybdea marsupialis | GCA_010016065.1 | C. andromeda | GCA_018155075.1 |
| Cassiopea xamachana | GCA_900291935.1 | Ch. achlyos | GCA_015164055.1 |
| Chrysaora chesapeakei | GCA_011763395.1 | Ch. fuscescens | GCA_009936425.1 |
| Ch. quinquecirrha | GCA_014526335.1 | Ch. quinquecirrha | GCA_012295145.1 |
| Clytia hemisphaerica | GCA_902728285.1 | Craspedacusta sowerbii | GCA_003687565.1 |
| Dendronephthya gigantea | GCA_004324835.1 | Enteromyxum leei | GCA_001455295.2 |
| Exaiptasia diaphana | GCA_001417965.1 | Haliclystus octoradiatus | GCA_916610825.1 |
| Heteractis crispa | GCA_015164035.1 | Henneguya salminicola | GCA_009887335.1 |
| Hydra oligactis | GCA_004118135.1 | He. magnifica | GCA_011763375.1 |
| H. viridissima | GCA_004118115.1 | H. viridissima | GCA_014706445.1 |
| H. vulgaris | GCA_000219015.1 | H. vulgaris | GCA_000004095.1 |
| Kudoa iwatai | GCA_001407335.1 | K. iwatai | GCA_001407235.2 |
| Montipora capitata | GCA_006542545.1 | M. cactus | GCA_014634245.1 |
| Morbakka virulenta | GCA_003991215.1 | M. efflorescens | GCA_014634505.1 |
| Nematostella vectensis | GCA_000209225.1 | Myxobolus squamalis | GCA_010108815.1 |
| Orbicella faveolata | GCA_002042975.1 | Nemopilema nomurai | GCA_003864495.1 |
| Pachyseris speciosa | GCA_016490735.1 | O. faveolata | GCA_001896105.1 |
| Platygyra sinensis | GCA_019787425.1 | Phymanthus crucifer | GCA_009858155.1 |
| Pocillopora verrucosa | GCA_014529365.1 | P. damicornis | GCA_003704095.1 |
| Renilla reniformis | GCA_900177555.1 | Porites rus | GCA_900290455.1 |
| Sanderia malayensis | GCA_013076295.1 | Rhopilema esculentum | GCA_013076305.1 |
| Stichodactyla helianthus | GCA_015163945.1 | Sphaeromyxa zaharoni | GCA_001455285.1 |
| Stylophora pistillata | GCA_002571385.1 | St. mertensii | GCA_011800005.1 |
| Trachythela sp. | GCA_016169945.1 | Thelohanellus kitauei | GCA_000827895.1 |
Для поиска КИП и выявления полноразмерных элементов последовательности, гомологичные транспозазе Mariner-18_CGi, были взяты из соответствующих скаффолдов вместе с фланкирующими участками длиной 3000 п.н. Нуклеотидные последовательности ДНК полноразмерных элементов (или наиболее протяженных фрагментов) далее использовали для поиска и подсчета числа копий L18-транспозонов, присутствующих в геноме. При подсчете общего числа копий учитывали гомологичные фрагменты длиной 10–100% от длины полноразмерного элемента. Фрагменты длиной менее 10% не учитывали. Кроме того, отдельно оценивали количество потенциально функциональных копий. Полноразмерными мы считали элементы длиной более 95% от длины интактного элемента, которые содержали оба КИП и транспозазу, состоящую не менее чем из 300 аминокислотных остатков.
Анализ структуры последовательностей. КИП выявляли с помощью BLASTn-анализа нуклеотидных последовательностей [36]. Границы гипотетических ОРС определяли с помощью ORF Finder (https://www.ncbi.nlm.nih.gov/orffinder/) и уточняли визуально. Локализацию GRPR-подобного мотива и маркерных аминокислотных остатков: аспартат (D), аспартат (D) и глутамат (E) каталитического домена идентифицировали визуально по гомологии. Для выявления ДНК-связывающего домена PAIRED анализировали вторичную структуру транспозазы, предсказанную с помощью PSIPRED v4.0 [37]. Последовательность сигнала ядерной локализации (NLS) определяли с помощью программы PSORT (https://www.genscript.com/psort.html).
Филогенетический анализ. Филогенетический анализ проводили с использованием аминокислотных последовательностей транспозаз, относящихся к разным группам транспозонов Tc1/mariner, транспозаз, обнаруженных у Cnidaria, и IS630-транспозазы в качестве внешней группы. Множественное выравнивание аминокислотных последовательностей выполнено с помощью MUSCLE [38] и стандартных настроек. Филогенетический анализ проводили с использованием пакета программ MEGA X [38]. Для построения дендрограммы применяли метод максимального правдоподобия. Достоверность топологии оценивали с использованием бутстреп-теста (1000 репликаций).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Семейство L18-транспозонов у Cnidaria
При поиске транспозонов L18 Cnidaria в качестве образца мы использовали аминокислотную последовательность транспозазы элемента Mariner-18_CGi устрицы C. gigas, первого обнаруженного представителя группы L18 [35]. При анализе результатов, полученных с помощью tBLASTn, мы выбирали гомологичные последовательности с наилучшим сочетанием длины покрытия с процентом идентичности и расстоянием между вторым (аспартат) и третьим (глутамат) маркерными аминокислотными остатками каталитического домена DDE, равным 37 а.о. (DD37E). В некоторых случаях в анализ брали последовательности с высокой идентичностью к транспозазе Mariner-18_CGi, но с паттерном каталитического домена, отличным от DD37E. Встречались транспозазы, у которых в DDE-домене вместо глутамата в третьей позиции находился глутамин (Q), аспартат (D), аргинин (R) или лизин (K) (табл. 2).
Таблица 2.
ДНК-транспозоны L18 Cnidaria
| Таксон | Организм | Транспозон | Длина элемента, п.н. | Длина КИП, п.н. | Транс-позаза, а.о. | Всего копий | Полнораз-мерные копии | Потенциаль-но функцио-нальные копии | DDE/D-домен |
|---|---|---|---|---|---|---|---|---|---|
| Cnidaria/ Hydrozoa | H. viridissima | L18-1_HVir/GCA_014706445.1 | 1778 | 30/30 | 356 | 20 | 2 | 1 | D93D35E |
| L18-1_HVir/GCA_004118115.1 | 525 | – | 174 | 8 | 0 | 0 | D64D37E | ||
| H. vulgaris | L18-1_HVul/GCA_000004095.1 | 1993 | 163/163 | 374 | 47 | 7 | 1 | D102D37E | |
| L18-1_HVul/GCA_000219015.1 | 1723 | 28/28 | 374 | 137 | 46 | 2 | D102D37E | ||
| Cnidaria/ Anthozoa |
Trachythela sp | L18-1_TSp | 1360 | 31/31 | 230 | 2 | 0 | 0 | D103D37E |
| Pachyseris speciosa | L18-1_PSpe | 1453 | 31/31 | 357 | 56 | 35 | 0 | D103D37E | |
| A. acuminata | L18-1_AAcu | 543 | – | 181 | 5 | 0 | 0 | A99D37Q | |
| L18-2_AAcu | 615 | – | 204 | 8 | 0 | 0 | 104D37E | ||
| A. awi | L18-1_AAwi | 2951 | 23/23 | 96 | 4 | 0 | 0 | ?E37E | |
| L18-2_AAwi | 642 | – | 213 | 7 | 0 | 0 | D96D37E | ||
| A. cytherea | L18-1_ACyt | 577 | – | 176 | 5 | 0 | 0 | D84E37E | |
| L18-2_ACyt | 1006 | 24/24 | 226 | 7 | 0 | 0 | G102E37E | ||
| A. digitifera | L18-1_ADig/GCA_014634065.1 | 723 | 29/29 | 131 | 6 | 0 | 0 | ?E37D | |
| L18-1_ADig/GCA_000222465.2 | 321 | – | 106 | 8 | 0 | 0 | ?E37E | ||
| L18-2_ADig/GCA_014634065.1 | 1002 | 23/23 | 225 | 6 | 0 | 0 | G101D32R | ||
| L18-2_ADig/GCA_000222465.2 | 582 | – | 193 | 5 | 0 | 0 | D97D32R | ||
| A. echinata | L18-1_AEch | 1470 | 36/36 | 288 | 6 | 0 | 0 | D96E37E | |
| A. florida | L18-1_AFlo | 321 | – | 106 | 6 | 0 | 0 | ?E37E | |
| L18-2_AFlo | 1006 | 24/24 | 235 | 11 | 0 | 0 | G102E37E | ||
| A. gemmifera | L18-1_AGem | 759 | – | 252 | 81 | 0 | 0 | D84D37E | |
| L18-2_AGem | 516 | – | 171 | 5 | 0 | 0 | ?D37E | ||
| A. hyacinthus | L18-1_AHya | 1959 | 44/45 | 106 | 257 | 0 | 0 | ?E37E | |
| A. intermedia | L18-1_AInt | 2198 | 38/39 | 106 | 406 | 0 | 0 | ?E37E | |
| L18-2_AInt | 516 | – | 171 | 6 | 0 | 0 | ?D37E | ||
| A. microphthalma | L18-1_AMic | 1534 | 31/31 | 56 | 4 | 0 | 0 | ?D37E | |
| L18-2_AMic | 480 | – | 160 | 3 | 0 | 0 | ?D37E | ||
| A. muricata | L18-1_AMur | 321 | – | 106 | 6 | 0 | 0 | ?E37E | |
| L18-2_AMur | 597 | – | 199 | 5 | 0 | 0 | *104D37E | ||
| Cnidaria/ Anthozoa |
Astreopora myriophthalma | L18-1_AMyr | 1465 | 31/31 | 387 | 47 | 22 | 1 | D103E37E |
| A. nasuta | L18-1_ANas | 1478 | 36/36 | 292 | 7 | 0 | 0 | D99E37E | |
| L18-2_ANas | 408 | – | 136 | 5 | 0 | 0 | ?D32K | ||
| A. selago | L18-1_ASel | 1980 | 35/36 | 106 | 358 | 0 | 0 | ?E37E | |
| L18-2_ASel | 1005 | 22/22 | 241 | 8 | 0 | 0 | G103E37E | ||
| A. tenuis | L18-1_ATen | 480 | – | 160 | 6 | 0 | 0 | D103D37E | |
| L18-2_ATen | 996 | 31/32 | 167 | 8 | 0 | 0 | G102D? | ||
| A. yongei | L18-1_AYon | 1520 | 56/60 | 211 | 11 | 0 | 0 | D102D37E | |
| L18-2_AYon | 477 | – | 159 | 8 | 0 | 0 | ?D37E | ||
| Montipora cactus | L18-1_MCac | 1458 | 32/36 | 329 | 19 | 4 | 0 | D105D37E | |
| M. efflorescens | L18-1_MEff | 1443 | 29/33 | 327 | 17 | 3 | 0 | D103D37E | |
| A. millepora | L18-1_AMil/ GCA_013753865.1 | 168 | – | 56 | 7 | 0 | 0 | ?E37E | |
| L18-1_AMil/GCA_004143615.1 | 1818 | 36/36 | 180 | 5 | 0 | 0 | D96E37E | ||
| L18-2_AMil/GCA_004143615.1 | 1019 | 29/30 | 210 | 9 | 0 | 0 | D102D38E | ||
| L18-2_AMil/GCA_013753865.1 | 648 | – | 215 | 9 | 0 | 0 | E104D38E | ||
| Porites rus | L18-1_PRus | 1467 | 29/29 | 353 | 177 | 6 | 0 | D139D37E | |
| Orbicella faveolata | L18-1_OFav/GCA_001896105.1 | 1167 | 29/29 | 295 | 8 | 0 | 0 | D103D37E | |
| L18-1_OFav/GCA_002042975.1 | 1167 | 29/29 | 294 | 8 | 0 | 0 | D103D37E | ||
| Montipora capitata | L18-1_MCap | 1402 | 33/33 | 334 | 63 | 1 | 0 | Q104D37E | |
| Pocillopora damicornis | L18-1_PDam | 210 | – | 70 | 2 | 0 | 0 | н/о | |
| Stylophora pistillata | L18-1_SPis | 123 | – | 41 | 1 | 0 | 0 | н/о | |
| P. verrucosa | L18-1_PVer | 1664 | 20/21 | 70 | 3 | 0 | 0 | н/о | |
| Nematostella vectensis | L18-1_NVec | 1341 | 34/34 | 315 | 18 | 2 | 0 | D102D37E |
В результате анализа 78 полногеномных последовательностей 35 организмов (30 видов) нами идентифицирован 51 элемент L18 при этом полноразмерные копии обнаружены только у 10 из них. В качестве образцов для дополнительного поиска гомологичных МГЭ использовали транспозазы полноразмерных элементов.
Структурные особенности L18-транспозонов Cnidaria
Длина обнаруженных полноразмерных элементов L18 колебалась от 1341 до 1993 п.н. Длина транспозаз составляла 315–387 а.о. КИП найдены у 30 элементов, их длина колебалась от 20 до 60 п.н. и лишь у одного элемента – L18-1_HVul/GCA_ 000004095.1 – достигала 163 п.н. Субконцевые инвертированные повторы (СИП) не обнаружены (табл. 2).
Если сравнить L18 и родственные группы элементов MLE и TLE, длина которых не превышает 1300–1400 п.н., длина КИП равна примерно 30 п.н., а длина транспозазы около 340–360 а.о. [39, 40], то заметны некоторые различия. Общая длина элементов L18 и длина транспозазы соответствуют длинам в семействах MLE и TLE, лишь у отдельных элементов эти цифры незначительно отличаются (табл. 2). Длина КИП у большинства элементов L18 соответствует длине КИП MLE и TLE, однако у отдельных элементов, например, у L18-1_HVul/GCA_000004095.1, L18-1_AYon и L18-1_AHya, встречаются значительно более длинные КИП (табл. 2). Длина элементов L18, имеющих делеции, варьировала от 123 до 2951 п.н. (табл. 2). Подобное увеличение общей длины – 2198 и 2951 п.н. у поврежденных элементов L18-1_AIn и L18-1_AAwi, объясняется тем, что они содержат, по всей видимости, вставки в некодирующие части, в то время как их ОРС укорочена.
Наличие и целостность PAIRED-домена, GRPR-подобного мотива, NLS и каталитического домена DDE могут свидетельствовать о сохранении функциональности транспозазы. Мы изучали структурные особенности транспозазы у всех полноразмерных элементов, так как нас интересовала их потенциальная функциональность. В анализ мы включили также полноразмерные элементы, имеющие стоп-кодоны в ОРС, чтобы иметь больше последовательностей для сравнения, хотя их потенциальная функциональность в данном случае исключалась.
PAIRED-домен в полном или урезанном виде представлен во всех изученных нами транспозазах. Первые три альфа-спирали отсутствовали только в транспозазе элемента L18-1_NVec, в которой эта часть белка оказалась делетированной. Третья альфа-спираль была фрагментирована в L18-1_HVul/GCA_000004095.1, L18-1_HVul/GCA_000219015.1 и L18-1_MCaс. Вторые три альфа-спирали имеют четкие границы в транспозазах элементов L18-1_HVul/GCA_000004095.1, L18-1_HVul/GCA_000219015.1 и L18-1_MCap. В остальных случаях альфа-спирали домена RED не имеют четкого рисунка, они фрагментированы, укорочены, слиты или отсутствуют (рис. 1). GRPR-подобный мотив в семи транспозазах из восьми имел последовательность GRPT/S, в транспозазе элемента L18-1_PRus он был малоузнаваемым, а в белках элементов L18-1_MCaс и L18-1_NVec не обнаружен вовсе. Расположение мотива было типичным – между первой и второй триадами альфа-спиралей (рис. 1). NLS-последовательность обнаружена в транспозазах пяти элементов L18, в двух из них найдено по два NLS, но наиболее типичным было расположение NLS между PAIRED-доменом и каталитическим доменом DDE (рис. 1).
Рис. 1.
Множественное выравнивание аминокислотных последовательностей L18-транспозаз. Серым выделены шесть α-спиралей, формирующих PAIRED-домен; жирным обозначен гипотетический сигнал ядерной локализации (NLS); выделен жирным и подчеркнут GRPR-подобный мотив; черным показаны маркерные локусы DD37E-домена.

Каталитический домен DD37E обнаружен у всех исследованных полноразмерных элементов L18 (рис. 1), но в аминокислотной последовательности транспозазы элемента L18-1_MCap первый аспартат в домене DDE заменен на глутамин (Q), а расстояние между D и E в каталитическом домене элемента L18-1_HVir/GCA_014706445.1 равно 35 а.о. (табл. 2).
Анализ структуры белка и строения элементов L18 позволяет заключить, что функциональной активностью потенциально обладают только четыре элемента – L18-1_HVul/GCA_000004095.1, L18-1_HVul/GCA_000219015.1, L18-1_HVir/GCA_ 014706445.1 и L18-1_AMyr, при этом один из них – L18-1_HVul/GCA_000219015.1 – представлен двумя копиями, остальные – только одной (табл. 2). Столь малое число потенциально функциональных элементов L18, обнаруженных у Cnidaria, вполне согласуется с данными других исследований, которые также показывают, что наряду со значительным общим пулом элементов, имеющих делеции, потенциально функциональные элементы встречаются в единичном количестве [41–44].
Филогенетический анализ L18-транспозонов Cnidaria
Филогенетический анализ, в который вошли транспозазы всех обнаруженных нами L18-транспозонов стрекающих и некоторые L18-транспозазы иглокожих, моллюсков и рыб [35], а также транспозазы, представляющие семейство TLE (Intruder, Incomer, Traveler, TLEWI, Tc1, Zvezda, TRT, ctmTLE) и семейство MLE (в качестве внешней группы) [40, 45–49], показал, что группа элементов L18 не является подсемейством группы TLE, как считалось ранее [35] (рис. 2). L18-транспозоны с достаточно высокой значимостью (значение бутстреп-теста 67%) формируют единую кладу, тогда как их объединение с другими группами семейства TLE недостоверно (значение бутстреп-теста менее 50%). Таким образом, мы полагаем, что L18-транспозоны – это отдельное монофилетическое семейство внутри суперсемейства Tc1/mariner.
Рис. 2.
Эволюционные взаимоотношения транспозонов L18 и TLE. Черными кругами обозначены ДНК-транспозоны, обнаруженные в этой работе. Филогенетический анализ выполнен в программе MEGA X c помощью метода максимального правдоподобия. Используемая модель WAG+G. Достоверность топологии оценивали с использованием бутстреп-теста (1000 репликаций). Значения коэффициентов поддержки бутстреп-теста менее 50% не показаны.
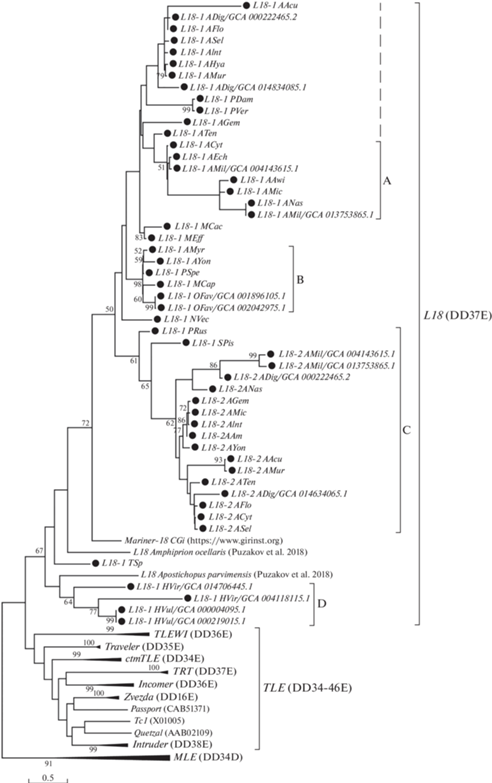
Филогенетическое исследование L18-транспозонов позволило выделить внутри семейства четыре кластера (A, B, C, D) (рис. 2). В кластеры мы объединили элементы, которые формируют группы, включающие более двух L18-транспозонов и имеющие значение бутстреп-теста более 50%. В кластеры A, B и C вошли транспозоны, обнаруженные у представителей класса Anthozoa. При этом в кластер С объединяют преимущественно элементы, обозначенные нами как L18-2. Эти элементы обнаружены также у представителей класса Anthozoa в качестве второго, альтернативного элемента L18. Кластер D содержал только транспозоны L18 класса Hydrozoa.
Внутривидовое разнообразие
Поскольку на момент исследования в NCBI были депонированы по две сборки геномов пяти видов Cnidaria: H. viridissima, H. vulgaris, A. digitifera, A. millepora и Orbicella faveolata (табл. 3), мы получили возможность изучить внутривидовое разнообразие МГЭ.
Таблица 3.
Внутривидовое разнообразие L18-транспозонов у Cnidaria
| Организм | Место сбора | Время сбора | Элемент/Сборка | Перекрытие/
идентичность гена транс-позазы, % |
Перекрытие/ идентичность транспозазы, % |
|---|---|---|---|---|---|
| H. viridissima | Австралия | 2014-04-15 | L18-1_HVir/ GCA_014706445.1 | 4/76.60 | 75/32.51 |
| Лабораторная линия Европа | Нет данных | L18-1_HVir/ GCA_004118115.1 |
|||
| H. vulgaris | Лабораторная линия № 105 | Нет данных | L18-1_HVul/ GCA_000004095.1 | 100/100 | 100/100 |
| Лабораторная линия № 105 | Нет данных | L18-1_HVul/ GCA_000219015.1 |
|||
| A. digitifera | Япония | 2015-05 | L18-1_ADig/ GCA_014634065.1 |
80/89.72 | 74/79.59 |
| Кунигами, Окинава, Япония | 2008 | L18-1_ADig/ GCA_000222465.2 |
|||
| Япония | 2015-05 | L18-2_ADig/ GCA_014634065.1 |
87/88.11 | 74/75.60 | |
| Кунигами, Окинава, Япония | 2008 | L18-2_ADig/ GCA_000222465.2 |
|||
| A. millepora | Индонезия | 2017 | L18-1_AMil/ GCA_013753865.1 |
30/97.62 | 31/96.43 |
| Магнетик-Айленд, Австралия | 1999-10-28 | L18-1_AMil/ GCA_004143615.1 |
|||
| Индонезия | 2017 | L18-2_AMil/ GCA_004143615.1 |
95/93.36 | 100/86.05 | |
| Магнетик-Айленд, Австралия | 1999-10-28 | L18-2_AMil/ GCA_013753865.1 |
|||
| Orbicella faveolata | Флорида, США | 2011-08 | L18-1_OFav/ GCA_001896105.1 |
100/95.73 | 99/95.24 |
| Панама | 2013-08 | L18-1_OFav/ GCA_002042975.1 |
ДНК H. viridissima была выделена из образцов, представляющих различные популяции – из лабораторной линии (Европа) и природной популяции (Австралия). Элементы L18-1_HVir/GCA_014706445.1 и L18-1_HVir/GCA_ 004118115.1 существенно различаются, перекрывание нуклеотидных последовательностей этих двух транспозонов равно 4%, идентичность транспозаз этих элементов также была низкий – 32.51% (табл. 3). Такой уровень сходства белков транспозазы характерен для отдаленных видов. Элемент L18-1_HVir/GCA_014706445.1 считается потенциально активным, тогда как L18-1_HVir/GCA_ 004118115.1 сильно укорочен и содержит стоп-кодоны, а длина транспозазы равна 174 а.о. По всей видимости, эволюция элемента L18 в этих двух популяциях из-за их географической удаленности проходила по-разному – в лабораторной линии элемент подвергся деградации, а в природной популяции сохранил свою активность.
Обе сборки генома H. vulgaris получены из лабораторной линии 105. Обнаруженные в них транспозоны L18 идентичны, что, вероятно, обусловлено высокой гомогенностью особей внутри лабораторной линии (табл. 3).
Секвенированные геномы A. digitifera принадлежали особям, выловленным на побережье Японии с интервалом 7 лет (табл. 3). В обеих сборках обнаружены по два элемента L18 (табл. 2). Сходство между первой и второй парой элементов довольно высокое как на уровне нуклеотидной (88–89%), так и аминокислотной (75–79%) последовательности. Транспозоны относятся к достаточно быстро мутирующим элементам генома, поскольку на них не действует стабилизирующий (очищающий) отбор. Поэтому идентичность нуклеотидных последовательностей различных копий одного и того же ДНК-транспозона внутри одного генома может варьировать от 80 до 100%, а в некоторых случаях достигать 70% [35]. По всей вероятности, столь высокую идентичность транспозонов можно объяснить тем, что образцы принадлежат к одной и той же популяции.
Две особи A. millepora собраны в Индонезии и Австралии с интервалом 18 лет. В каждой сборке генома этого вида также обнаружено по два элемента L18. Выявлена очень высокая идентичность обоих элементов на уровне нуклеотидной и аминокислотной последовательностей (табл. 3). Обращает на себя внимание низкая перекрываемость (%) элементов L18-1, но это объясняется тем, что самая протяженная копия (L18-1_AMil/ GCA_013753865.1) кодирует фрагмент транспозазы длиной всего 56 а.о. Высокая идентичность элементов может объясняться географической близостью Индонезии и Австралии и высокой вероятностью того, что образцы принадлежат к одной популяции. Сходная ситуация наблюдается и у образцов O. faveolata (табл. 3), собранных во Флориде (США) и государстве Панама. Очень высокая идентичность нуклеотидных последовательностей этих элементов (95%) также может объясняться географической близостью Флориды и Панамы.
В общем, можно сделать вывод о довольно высокой гомогенности одной и той же популяции по “старым” транспозонам, которые уже утратили свою активность. В разобщенных популяциях, как в случае H. viridissima, транспозоны сильно различаются.
Жизненный цикл L18-транспозонов
Степень активности элементов L18 в геномах Cnidaria, а также давность их вторжения мы попытались оценить по их копийности в геномах стрекающих. У пяти элементов общее число копий было очень высоким – от 137 до 406. Еще у пяти элементов общее число копий оказалось достаточно высоким – 47–81. У оставшегося большинства элементов количество копий не превышало 20. Число полноразмерных копий 10 элементов L18 распределялось таким образом: семь имели не более 10 копий и только у трех элементов количество полноразмерных копий было умеренно высоким – 22, 35 и 46 (табл. 2). Подобное распределение позволяет предположить, что элементы L18-1_HVul/GCA_000219015.1 и L18-1_ AMyr могут быть активными в геномах данных организмов в настоящее время, поскольку они имеют много полноразмерных копий (46 и 22), в том числе и одну–две потенциально функциональных. Элементы L18-1_PSpe могли быть активными в недавнем прошлом, поскольку на фоне довольно высокого общего числа копий (56) обнаружено много (35) их полноразмерных копий, но ни одной потенциально функциональной. Общее высокое число копий и отсутствие полноразмерных копий, как, например, у элементов L18-1_AHya, L18-1_AInt, L18-1_ASel, может свидетельствовать о высокой активности этих элементов в прошлом, что привело к их амплификации, но в настоящее время наблюдается элиминация этих элементов из геномов хозяина. Низкое общее число копий и отсутствие полноразмерных копий, которое встречается в большинстве случаев (табл. 2), может свидетельствовать о том, что эти элементы завершают свой жизненный цикл в геноме данных хозяев и большая их часть уже элиминирована. Это согласуется с данными, приведенными в работе [50], в которой выдвинута гипотеза о том, что активное заселение видов МГЭ осталось в далеком прошлом, сегодня же мы наблюдаем лишь завершающую стадию жизненного цикла подавляющего большинства элементов.
Эволюция элементов L18
Элементы семейства L18 обнаружены только в двух из шести классов стрекающих (полные геномы которых представлены в NCBI): Hydrozoa и Anthozoa (рис. 3). Наибольшее их число (в 29 из 30 геномов) выявлено у организмов отряда Scle-ractinia (подкласс Hexacoralia; класс Anthozoa), при этом у 16 представителей подотряда Astrocoeniina найдены по два различных МГЭ группы L18 (рис. 2, табл. 1). В другом отряде (Actiniaria) того же подкласса в исследованных 9 геномах L18-транспозон обнаружен только в одном семействе (Edwardsiidae). В подклассе Octocorallia у трех организмов также обнаружен только один L18-транспозон (подотряд Stolonifera; отряд Alcyonacea). МГЭ группы L18 идентифицированы также в четырех геномах (из семи) представителей класса Hydrozoa (подотряд Aplanulata). Элементы семейства L18 представителей Hydrozoa входят в кластер D и имеют большую идентичность с L18-транспозоном иглокожих, тогда как элементы представителей Anthozoa с достаточно высокой значимостью (бутстреп 50%) формируют отдельную группу, включающую кластеры A, B и C (рис. 2). Сопоставление результатов филогенетического анализа и распространенности L18 среди представителей Cnidaria дает основания полагать, что имели место несколько инвазий L18-транспозонов в геномы древних стрекающих. Горизонтальный перенос Тc1/mariner-элементов встречается относительно часто [51–53].
Рис. 3.
Представленность элементов L18 у представителей стрекающих. На таксономическом дереве приведены только те группы организмов, геномы представителей которых секвенированы. В столбцах (в серых прямоугольниках) указано количество доступных для анализа полногеномных последовательностей и число геномов, в которых обнаружены соответствующие группы ДНК-транспозонов.
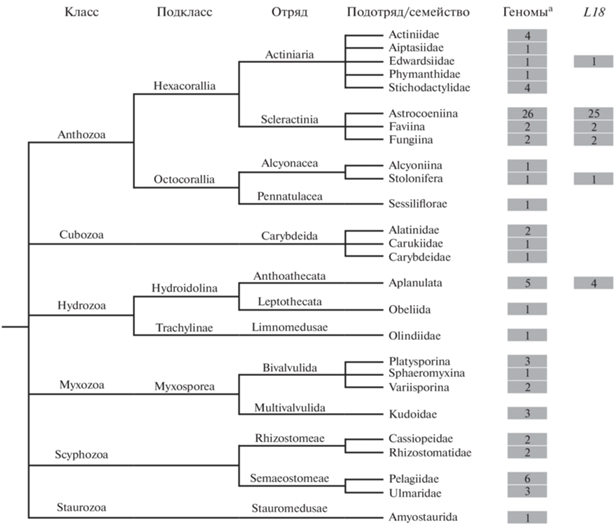
Согласно данным TimeTree (http://www.timetree.org/), дивергенция представителей класса Anthozoa и представителей классов Hydrozoa, Cubozoa и Scyphozoa произошла в диапазоне от 540 до 667 м.л.н. Поскольку L18-транспозоны обнаружены в обоих подклассах, то инвазия предкового транспозона могла произойти в этот период. С другой стороны, в подклассе Octocorallia обнаружен только один L18-транспозон (L18-1_TSp) – в кладе Stolonifera (рис. 3). При этом на филогенетическом дереве он занял обособленное место, не войдя ни в один из кластеров (рис. 2) других Anthozoa. Кластеры A, B и C оказались ближе к элементу моллюсков (Mariner-18_CGi), чем к L18-1_TSp. Данные о времени возникновения клады Stolonifera отсутствуют, тогда как отряд Alcyonacea (включающий эту кладу) дивергировал 368 м.л.н. (TimeTree, http://www.timetree.org/). По-видимому, предок L18-1_TSp вторгся в геном организма позднее 368 м.л.н. Эти временные оценки согласуются с тем, что данный элемент представлен всего двумя достаточно сильно поврежденными копиями. Нельзя исключить как более ранней инвазии элемента и полной элиминации из геномов других представителей Octocorallia, так и более поздней инвазии, слабой транспозиционной активности и быстрой деградации. Более точные заключения можно будет сделать с появлением новых данных.
L18-транспозоны подкласса Hexacorallia представлены тремя кластерами A, B и C. Если эти элементы являются потомками одного транспозона, то предположительное время инвазии – 540–667 м.л.н. С другой стороны, каждый кластер может быть результатом независимых событий горизонтального переноса. Но поскольку в кластерах присутствуют элементы различных таксонов подкласса Hexacorallia, то вероятно, что горизонтальные переносы происходили в рамках этой группы животных, тогда время этих событий оценить затруднительно.
В классе Hydrozoa L18-транспозоны выявлены только в отряде Anthoathecata, который обособился 440–459 м.л.н. (TimeTree, http://www.timetree.org/). Виды H. viridissima и H. vulgaris дивергировали около 193 м.л.н. (TimeTree, http:// www.timetree.org/). Таким образом, мы предполагаем, что L18-транспозоны появились у организмов этой группы в результате горизонтального переноса в промежутке от 193 до 459 м.л.н. Однако, опираясь на то, что у обоих видов гидр сохранились потенциально функциональные копии L18, и они, соответственно, являются относительно молодыми, мы полагаем время инвазии ближе к 193 м.л.н. Более точные оценки можно будет дать после того, как будут секвенированы и станут доступными для анализа геномы представителей других подотрядов Anthoathecata.
ЗАКЛЮЧЕНИЕ
В данной работе впервые описаны структура, распространение, разнообразие и эволюция элементов L18 (DD37E) в геномах стрекающих. В результате филогенетического анализа установлено, что группа L18 – это самостоятельное семейство (а не подсемейство группы TLE, как считалось ранее), входящее в суперсемейство Tc1/mariner. Среди 44 представителей Anthozoa обнаружен только один МГЭ, имеющий потенциально функциональную копию (L18-1_AMyr), тогда как среди семи представителей Hydrozoa выявлены четыре потенциально функциональные копии трех элементов: L18-1_HVul/GCA_000004095.1, L18-1_HVul/GCA_000219015.1, L18-1_HVir/GCA_ 014706445.1. В связи с этим предполагается, что элементы L18 имеют древнее происхождение, а у L18-транспозонов, обнаруженных в геномах организмов классов Anthozoa и Hydrozoa, нет общего предка внутри типа Cnidaria. Возможно, в геномах организмов подотряда Aplanulata (класс Hydrozoa) L18-транспозоны появились в результате горизонтального переноса в более поздний период времени. Сравнение внутривидового разнообразия элементов L18 показывает высокую гомогенность по “старым” транспозонам, которые уже утратили свою активность. В то же время, отдаленные популяции, как в случае H. viridissima, могут различаться представленностью ДНК-транспозонов и количеством их копий. Полученные данные дополняют знания о разнообразии и эволюции Tc1/mariner-транспозонов и будут способствовать изучению влияния МГЭ на эволюцию многоклеточных.
Данное исследование проведено в рамках государственного задания ФИЦ ИнБЮМ “Функциональные, метаболические и токсикологические аспекты существования гидробионтов и их популяций в биотопах с различным физико-химическим режимом”, номер гос. регистрации 121041400077-1.
Настоящая статья не содержит исследований с использованием животных в качестве объектов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Arkhipova I.R., Yushenova I.A. (2019) Giant transposons in eukaryotes: is bigger better? Genome Biol. Evol. 11, 906–918. https://doi.org/10.1093/gbe/evz041
Bourque G., Burns K.H., Gehring M., Gorbunova V., Seluanov A., Hammell M., Imbeault M., Izsvak Z., Levin H.L., Macfarlan T.S., Mager D.L., Feschotte C. (2018) Ten things you should know about transposable elements. Genome Biol. 19, 199. https://doi.org/10.1186/s13059-018-1577-z
Gao B., Shen D., Xue S., Chen C., Cui H., Song C. (2016) The contribution of transposable elements to size variations between four teleost genomes. Mob. DNA. 7, 1–16. https://doi.org/10.1186/s13100-016-0059-7
Petrov D.A. (2001) Evolution of genome size: new approaches to an old problem. Trends Genet. 17, 23–28. https://doi.org/10.1016/S0168-9525(00)02157-0
Sotero-Caio C.G., Platt R.N., Suh A., Ray D.A. (2017) Evolution and diversity of transposable elements in vertebrate genomes. Genome Biol. Evol. 9, 161–177. https://doi.org/10.1093/gbe/evw264
Casacuberta E., González J. (2013) The impact of transposable elements in environmental adaptation. Mol. Ecol. 22, 1503–1517. https://doi.org/10.1111/mec.12170
Sultana T., Zamborlini A., Cristofari G., Lesage P. (2017) Integration site selection by retroviruses and transposable elements in eukaryotes. Nat. Rev. Genet. 18, 292–308.
Blumenstiel J.P. (2019) Birth, school, work, death, and resurrection: the life stages and dynamics of transposable element proliferation. Genes (Basel). 10, 336. https://doi.org/10.3390/genes10050336
Venner S., Feschotte C., Biémont C. (2009) Dynamics of transposable elements: towards a community ecology of the genome. Trends Genet. 25, 317–323.
Robillard É., Rouzic A.L., Zhang Z., Capy P., Hua-Van A. (2016) Experimental evolution reveals hyperparasitic interactions among transposable elements. Proc. Natl. Acad. Sci. USA. 113, 14763–14768.
Sienski G., Dönertas D., Brennecke J. (2012) Transcriptional silencing of transposons by Piwi and maelstrom and its impact on chromatin state and gene expression. Cell. 151, 964–980. https://doi.org/10.1016/j.cell.2012.10.040
Brennecke J., Aravin A.A., Stark A., Dus M., Kellis M., Sachidanandam R., Hannon G.J. (2007) Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell. 128, 1089–1103. https://doi.org/10.1016/j.cell.2007.01.043
Teixeira F.K., Okuniewska M., Malone C.D., Coux R.X., Rio D.C., Lehmann R. (2017) piRNA-mediated regulation of transposon alternative splicing in the soma and germ line. Nature. 552, 268–272. https://doi.org/10.1038/nature25018
Casacuberta E. (2017) Drosophila: retrotransposons making up telomeres. Viruses. 9, 192. https://doi.org/10.3390/v9070192
Belfort M., Curcio M.J., Lue N.F. (2011) Telomerase and retrotransposons: reverse transcriptases that shaped genomes. Proc. Natl. Acad. Sci. USA. 108, 20304–20310.
Fulcher N., Derboven E., Valuchova S., Riha K. (2014) If the cap fits, wear it: an overview of telomeric structures over evolution. Cell. Mol. Life Sci. 71, 847–865.
Casola C., Hucks D., Feschotte C. (2007) Convergent domestication of pogo-like transposases into centromere-binding proteins in fission yeast and mammals. Mol. Biol. Evol. 25, 29–41.
Kursel L.E., Malik H.S. (2016) Centromeres. Curr. Biol. 26, 487–490.
Чересиз С.В., Юрченко Н.Н., Иванников А.В., Захаров И.К. (2008) Мобильные элементы и стресс. Информ. вестник ВОГиС. 12, 217–242.
Kojima K.K. (2020) Structural and sequence diversity of eukaryotic transposable elements. Genes Genet. Syst. 94, 233–252. https://doi.org/10.1266/ggs.18-00024
Kapitonov V.V., Jurka J. (2008) A universal classification of eukaryotic transposable elements implemented in Repbase. Nat. Rev. Genet. 9, 411–412. https://doi.org/10.1038/nrg2165-c1
Wicker T., Sabot F., Hua-Van A., Bennetzen J.L., Capy P., Chalhoub B., Flavell A., Leroy P., Morgante M., Panaud O., Paux E., SanMiguel P., Schulman A.H. (2007) A unified classification system for eukaryotic transposable elements. Nat. Rev. Genet. 8, 973–982. https://doi.org/10.1038/nrg2165
Yuan Y.W., Wessler S.R. (2011) The catalytic domain of all eukaryotic cut-and-paste transposase superfamilies. Proc. Natl. Acad. Sci. USA. 108, 7884–7889.
Feschotte C., Pritham E.J. (2007) DNA transposons and the evolution of eukaryotic genomes. Annu. Rev. Genet. 41, 331–368.
Muñoz-López M., García-Pérez J.L. (2010) DNA transposons: nature and applications in genomics. Curr. Genomics. 11, 115–128. https://doi.org/10.2174/138920210790886871
Tellier M., Bouuaert C.C., Chalmers R. (2015) Mariner and the ITm superfamily of transposons. Microbiol. Spectr. 3, MDNA3-0033-2014. https://doi.org/10.1128/microbiolspec.MDNA3-0033-2014
Wang S., Diaby M., Puzakov M., Ullah N., Wang Y., Danley P., Chen C., Wang X., Gao B., Song C. (2021) Divergent evolution profiles of DD37D and DD39D families of Tc1/mariner transposons in eukaryotes. Mol. Phylogenet. Evol. 161, 107143. https://doi.org/10.1016/j.ympev.2021.107143
Dupeyron M., Baril T., Bass C., Hayward A. (2020) Phylogenetic analysis of the Tc1/mariner superfamily reveals the unexplored diversity of pogo-like elements. Mob. DNA. 11, 21. https://doi.org/10.1186/s13100-020-00212-0
Ivics Z., Izsvák Z. (2015) Sleeping Beauty transposition. Microbiol. Spectr. 3, MDNA3-0042-2014. https://doi.org/10.1128/microbiolspec.MDNA3-0042-2014
Ivics Z., Hackett P.B., Plasterk R.H., Izsvak Z. (1997) Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells Cell. 91, 501–510. https://doi.org/10.1016/S0092-8674(00)80436-5
Plasterk R.H., Izsvak Z., Ivics Z. (1999) Resident aliens: the Tc1/mariner superfamily of transposable elements. Trends Genet. 15, 326–332. https://doi.org/10.1016/S0168-9525(99)01777-1
Shao H., Tu Z. (2001) Expanding the diversity of the IS630-Tc1-mariner superfamily: discovery of a unique DD37E transposon and reclassification of the DD37D and DD39D transposons. Genetics. 159, 1103–1115.
Zhang H.H., Shen Y.H., Xiong X.M., Han M.J., Zhang X.G. (2016) Identification and evolutionary history of the DD41D transposons in insect. Genes Genom. 38, 109–117.
Shen D., Gao B., Miskey C., Chen C., Sang Y., Zong W., Wang S., Wang Y., Wang X., Ivics Z., Song C. (2020) Multiple invasions of Visitor, a DD41D family of Tc1/mariner transposons, throughout the evolution of vertebrates. Genome Biol. Evol. 12, 1060–1073. https://doi.org/10.1093/gbe/evaa135
Puzakov M.V., Puzakova L.V., Cheresiz S.V. (2018) An analysis of IS630/Tc1/mariner transposons in the genome of a pacific oyster, Crassostrea gigas. J. Mol. Evol. 86, 566–580. https://doi.org/10.1007/s00239-018-9868-2
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z., Miller W., Lipman D.J. (1997) Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucl. Acids Res. 25, 3389–3402. https://doi.org/10.1093/nar/25.17.3389
Buchan D.W.A., Jones D.T. (2019) The PSIPRED protein analysis workbench: 20 years on. Nucl. Acids Res. 47, 402–407. https://doi.org/10.1093/nar/gkz297
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. (2018) MEGA X: molecular evolutionary genetics analysis across computing platforms. Mol. Biol. Evol. 35, 1547–1549.
Jacobson J.W., Medhora M.M., Hartl D.L. (1986) Molecular structure of a somatically unstable transposable element in Drosophila. Proc. Natl. Acad. Sci. USA. 83, 8684–8688.
Puzakov M.V., Puzakova L.V., Cheresiz S.V., Sang Y. (2021) The IS630/Tc1/mariner transposons in three Ctenophore genomes. Mol. Phylogenet. Evol. 163, 107231. https://doi.org/10.1016/j.ympev.2021.107231
Langin T., Capy P., Daboussi M.J. (1995) The transposable element impala, a fungal member of the Tc1-mariner superfamily. Mol. Gen. Genet. 246, 19–28.
Clark K.J., Carlson D.F., Leaver M.J., Foster L.K., Fahrenkrug S.C. (2009) Passport, anative Tc1 transposon from flatfish, is functionally active in vertebrate cells. Nucl. Acids Res. 37, 1239–1247.
Emmons S.W., Yesner L., Ruan K., Katzenberg D. (1983) Evidence for a transposon in Caenorhabditis elegans. Cell. 32, 55–65.
Franz G., Savakis C. (1991) Minos, a new transposable element from Drosophila hydei, is a member of the Tc1-like family of transposons. Nucl. Acids Res. 19, 6646.
Puzakov M.V., Puzakova L.V., Cheresiz S.V. (2020) The Tc1-like elements with the spliceosomal introns in mollusk genomes. Mol. Genet. Genomics. 295, 621–633.
Zong W., Gao B., Diaby M., Shen D., Wang S., Wang Y., Sang Y., Chen C., Wang X., Song C. (2020) Traveler, a new DD35E family of Tc1/mariner transposons, invaded vertebrates very recently. Genome Biol. Evol. 12, 66–76.
Sang Y., Gao B., Diaby M., Zong W., Chen C., Shen D., Wang S., Wang Y., Ivics Z., Song C. (2019) Incomer, a DD36E family of Tc1/mariner transposons newly discovered in animals. Mobile DNA. 10, 45.
Gao B., Zong W., Miskey C., Ullah N., Diaby M., Chen C., Wang X., Ivics Z., Song C. (2020) Intruder (DD38E), a recently evolved sibling family of DD34E/Tc1 transposons in animals. Mobile DNA. 11, 32.
Zhang H.H., Li G.Y., Xiong X.M., Han M.J., Zhang X.G., Dai F.Y. (2016) TRT, a vertebrate and protozoan Tc1-like transposon: current activity and horizontal transfer. Genome Biol. Evol. 8, 2994–3005.
Schaack S., Gilbert C., Feschotte C. (2010) Promiscuous DNA: horizontal transfer of transposable elements and why it matters for eukaryotic evolution. Trends Ecol. Evol. 25, 537–546.
Filee J., Rouault J.-D., Harry M., Hua-Van A. (2015) Mariner transposons are sailing in the genome of the blood-sucking bug Rhodnius prolixus. BMC Genomics. 16, 1061. https://doi.org/10.1186/s12864-015-2060-9
Sanllorente O., Vela J., Mora P., Ruiz-Mena A., Tor-res M.I., Lorite P., Palomeque T., (2020) Complex evolutionary history of mboumar, a mariner element widely represented in ant genomes. Sci. Rept. 10, 2610. https://doi.org/10.1038/s41598-020-59422-4
Maruyama K., Hartl D.L. (1991) Evidence for interspecific transfer of the transposable element mariner between Drosophila and Zaprionus. J. Mol. Evol. 33, 514–524. https://doi.org/10.1007/BF02102804
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


