Молекулярная биология, 2022, T. 56, № 3, стр. 491-497
Изменение уровней мРНК генов ДНК-метилтрансфераз в эндотелиальных клетках, обработанных митомицином
М. Ю. Синицкий a, *, А. В. Синицкая a, Д. К. Шишкова a, А. Г. Кутихин a, В. И. Минина b, А. В. Понасенко a
a Научно-исследовательский институт комплексных проблем сердечно-сосудистых заболеваний
650002 Кемерово, Россия
b Кемеровский государственный университет
650000 Кемерово, Россия
* E-mail: max-sinitsky@rambler.ru
Поступила в редакцию 27.08.2021
После доработки 07.10.2021
Принята к публикации 18.10.2021
- EDN: BTQXHG
- DOI: 10.31857/S0026898422030156
Аннотация
ДНК-метилтрансферазы катализируют метилирование остатков цитозина в CpG-сайтах ДНК, что играет важную роль в регуляции транскрипции, стабильности хромосом и других процессах. Показано, что уровень метилирования ДНК изменяется под действием генотоксического стресса, вызванного различными кластогенными агентами. Генотоксический стресс, в свою очередь, является одним из триггеров эндотелиальной дисфункции. С помощью количественной полимеразной цепной реакции мы оценили изменения уровней мРНК генов DNMT1, DNMT3A и DNMT3B в линиях эндотелиальных клеток коронарной (HCAEC) и внутренней грудной (HITAEC) артерий человека, экспонированных алкилирующим мутагеном – митомицином С. Установлено, что непосредственно после мутагенного воздействия уровень мРНК гена DNMT1 в культурах клеток HCAEC повышается в 1.7 раза по сравнению с контролем. После удаления мутагена из культуральной среды и последующего культивирования клеток HCAEC в “чистой” среде в течение 1 суток уровень мРНК гена DNMT3B в культурах, обработанных митомицином С, возрастал в 2 раза по сравнению с контролем. Одновременно с этим не обнаружено изменения уровней мРНК генов изученных ДНК-метилтрансфераз в экспонированных мутагеном культурах HITAEC. По-видимому, изменения уровней мРНК генов ДНК-метилтрансфераз могут вносить вклад в формирование эндотелиальной дисфункции в ответ на мутагенную нагрузку в эксперименте in vitro.
ВВЕДЕНИЕ
ДНК-метилтрансферазы, катализирующие перенос метильных групп с S-аденозилметионина на остатки цитозина в ДНК, играют важную роль в развитии млекопитающих [1]. Семейство ДНК-метилтрансфераз человека состоит из четырех членов – DNMT1, DNMT3A, DNMT3B и DNMT3L, из которых только последний не обладает выраженной ферментативной активностью [2]. DNMT1, DNMT3A и DNMT3B играют важную роль в поддержании стабильности генетического аппарата, нарушение которой приводит к повреждению хромосом, генотоксическому стрессу и канцерогенезу. Роль генов семейства DNMT в развитии различных онкопатологий изучена достаточно хорошо в экспериментах in vivo, в то время как возможные изменения их экспрессии в клеточных культурах (в том числе в эндотелиоцитах), обработанных различными генотоксическими и канцерогенными агентами, не изучены [3]. Известно также, что метилирование промоторных участков генов, катализируемое ДНК-метилтрансферазами, приводит к нарушению транскрипции и вносит вклад в развитие атеросклероза [4–6].
На сегодняшний день установлено, что генотоксический стресс (как и классические факторы риска – курение, сахарный диабет, дислипидемия и другие) служит триггером эндотелиальной дисфункции, считающейся первым этапом атерогенеза [7–9]. Недавно обнаружены изменения экспрессии генов-маркеров эндотелиальной дисфункции, вовлеченных в атерогенез, в эндотелиальных клетках человека, обработанных мутагеном алкилирующего механизма действия – митомицином С (ММС) [10, 11]. Вместе с тем, механизмы развития эндотелиальной дисфункции, индуцированной мутагенным воздействием, остаются неизученными. Учитывая высокий уровень заболеваемости и смертности от сердечно-сосудистых заболеваний (в частности, от атеросклероза) [12], понимание молекулярных механизмов развития эндотелиальной дисфункции в ответ на генотоксический стресс представляется чрезвычайно важным как для фундаментальной, так и прикладной науки.
В нашей работе проведен анализ изменений уровней мРНК генов DNMT1, DNMT3A и DNMT3B в первичных эндотелиальных клетках человека, обработанных алкилирующим мутагеном ММС.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
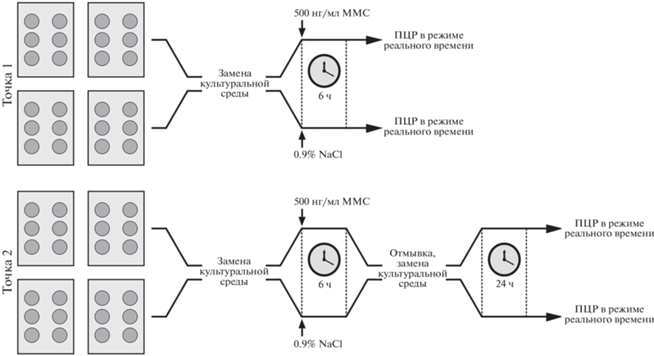
Культивирование эндотелиальных клеток. Исследование выполнено на коммерческих культурах эндотелиальных клеток коронарной (Human Coronary Artery Endothelial Cells, HCAEC) и внутренней грудной (Human Internal Thoracic Artery Endothelial Cells, HITAEC) артерий человека (“Cell Applications Inc.”, США).
Все работы с клеточными культурами проводили в асептических условиях. Клетки культивировали в культуральных флаконах Т-75, содержащих 15 мл среды Human MesoEndo Cell Growth Medium (“Cell Applications Inc.”), при температуре 37°C, 5% CO2 и повышенной влажности до достижения 90% конфлюентности. После четырех пассажей клетки снимали смесью трипсин/EDТА (“Cell Applications Inc.”), 2 × 105 клеток пересевали в 6-луночные планшеты (восемь планшетов на каждую клеточную культуру HCAEC и HITAEC), в каждую лунку добавляли 2 мл среды Human MesoEndo Cell Growth Medium и культивировали планшеты в стандартных условиях в течение еще 24 ч. По истечении этого времени старую среду удаляли, в каждую лунку планшета приливали 2 мл свежей среды, содержащей 500 нг/мл ММС (“AppliChem”, Испания) (экспериментальная группа), либо 0.9%-ный раствор NaCl (контрольная группа). После 6 ч культивирования четыре планшета с клетками каждой линии (два экспериментальных и два контрольных) выводили из эксперимента (точка 1), в оставшихся четырех планшетах культуральную среду заменяли чистой средой (без добавления каких-либо агентов) и культивировали планшеты в течение еще 1 суток, после чего выводили из эксперимента (точка 2). Дизайн исследования представлен на рис. 1. Концентрацию ММС и схему культивирования выбирали с учетом рекомендаций по моделированию мутагенеза в экспериментах in vitro [13, 14] и полученных нами ранее результатов [10].
Выделение РНК. Из каждой лунки 6-луночного планшета после окончания культивирования удаляли культуральную среду, клетки отмывали 2 раза холодным фосфатно-солевым буфером и лизировали 1 мл регента QIAzol® Lysis Reagent (“Qiagen”, Германия). Выделение суммарной РНК из клеток и ее очистку от геномной ДНК осуществляли с помощью набора RNeasy® Plus Universal Mini Kit (“Qiagen”) согласно протоколу производителя. Выделенную РНК хранили при температуре –80°C. Все рабочие поверхности и оборудование, использованное для выделения РНК, обрабатывали ингибитором РНКаз RNaseZap™ RNase Decontamination Solution (“Invitrogen”, США). Целостность РНК оценивали по величине индекса RIQ (RNA Integrity and Quality), который определяли с использованием набора реагентов Qubit RNA IQ Assay Kit (“Invitrogen”) и флуориметра Qubit 4 (“Invitrogen”). Концентрацию и чистоту РНК определяли на спектрофотометре NanoDrop™ 2000 (“Thermo Scientific, США).
Синтез кДНК. кДНК синтезировали с помощью коммерческих наборов High Capacity cDNA Reverse Transcription Kit (“Applied Biosystems”, США), согласно рекомендациям производителя. В качестве матрицы использовали 10 мкл раствора суммарной РНК (100 нг/мкл). Полученную кДНК хранили при температуре –20°C.
Оценка изменений уровня мРНК генов DMNT. Уровень мРНК генов DNMT1, DNMT3A и DNMT3B определяли в образцах эндотелиальных клеток (точки 1 и 2) с помощью количественной ПЦР на амплификаторе ViiA7 (“Applied Biosystems) с использованием праймеров с флуоресцентным красителем SYBR Green (“Евроген”, Москва) (табл. 1). ПЦР проводили в 96-луночном планшете, содержащем 26 анализируемых образцов, пять стандартов (кДНК с известной концентрацией) с двукратным разведением и отрицательный контроль (реакционная смесь без кДНК). Каждый образец, стандарт и отрицательный контроль анализировали в трех технических повторностях. Для каждого образца готовили 10 мкл реакционной смеси, включающей 5 мкл мастер-микса PowerUp SYBR Green Master Mix (“Applied Biosystems”), по 500 нМ прямого и обратного праймеров (“Евроген”) и образец кДНК в конечной концентрации 10 нг/мкл. Амплификацию проводили по следующей схеме: 50°C, 2 мин; 95°C, 2 мин; затем 40 циклов – 95°C, 15 с и 60°C, 60 с. Результаты ПЦР нормировали на среднее геометрическое значение Ct трех референсных генов HPRT1, GAPDH и B2M (“Евроген”) в соответствии с имеющимися рекомендациями [15] (табл. 1).
Таблица 1.
Характеристика праймеров, использованных в эксперименте
| Ген | Праймер |
|---|---|
| DNMT1 | F:CTGGTCCGCATGGGCTATCA R:TCCGGGAACAGAGGGAGCTT |
| DNMT3A | F:ACCCTGTTTGCCTCCCTGAG R:ACAATCACCCAGCCCTCTCC |
| DNMT3B | F:AACTGGAGCCACGACGTAACA R:GGCATCCGTCATCTTTCAGC |
| HPRT1 | F:TTGCTTTCCTTGGTCAGGCA R:TCGTGGGGTCCTTTTCACCA |
| GAPDH | F:AGCCACATCGCTCAGACAC R:GCCCAATACGACCAAATCC |
| B2M | F:TCCATCCGACATTGAAGTTG R:CGGCAGGCATACTCATCTT |
Уровни мРНК генов DNMT1, DNMT3A и DNMT3B рассчитывали по методу ΔCt (Уровень транскрипции = 2Ct [референсные гены] –Ct [ген интереса]). Качество амплификации оценивали с использованием кривых амплификации и стандартных кривых в программе QuantStudio™ Real-Time PCR Software v.1.3 (“Applied Biosystems”). Амплификацию считали успешной при эффективности 90–110%, значении R 2 > 0.990 и отсутствии реакции в отрицательном контроле. Все работы по определению уровня транскрипции генов выполнены в соответствии с международными стандартами MIQE [16].
Статистический анализ. Статистическую обработку результатов проводили в программе GraphPad Prism 8. Рассчитывали медиану (m) и межквартильный размах (IQR) количественных показателей. Различия между группами оценивали с помощью рангового U-критерия Манна–Уитни и считали статистически значимыми при p < 0.05.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Для оценки уровней мРНК генов DNMT использовали препараты суммарной РНК с концентрацией 260.4–601.8 нг/мкл, достаточной чистотой (A260/280 и A260/230 составили 2.05–2.09 и 1.82–2.25 соответственно) и целостностью (индекс RIQ больше 93%), выделенные из эндотелиальных клеток. На основе выделенной РНК синтезировано от 1512.6 до 1861.2 нг/мкл кДНК.
В результате проведения количественной ПЦР установлено, что в точке 1 (непосредственно после обработки клеток ММС) в культурах HCAEC статистически значимо (p = 0.014) повышен уровень мРНК гена DNMT1 по сравнению с контролем (0.1384 ± 0.0542 и 0.0816 ± 0.0653 усл. ед. соотв.) (рис. 2). После удаления мутагена (точка 2) в экспонированных ММС образцах HCAEC выявлено статистически значимое (p = 0.021) двукратное увеличение уровня мРНК гена DNMT3B (0.0010 ± 0.0003 против 0.0005 ± 0.0002 усл. ед. в контрольной группе) (рис. 2). Кратность изменений уровней мРНК изученных генов в экспонированных мутагеном культурах по сравнению с неэкспонированным контролем приведена в табл. 2.
Рис. 2.
Уровни мРНК генов DNMT в эндотелиальных клетках (HCAEC – эндотелиальные клетки коронарной артерии; HITAEC – эндотелиальные клетки внутренней грудной артерии).
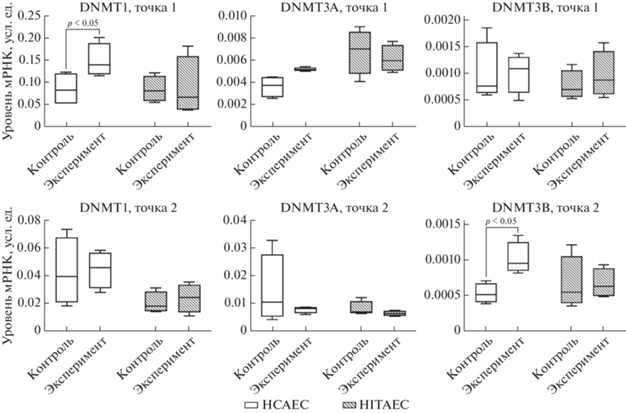
Таблица 2.
Относительные уровни мРНК генов DNMT в эндотелиальных клетках, экспонированных ММС, в сравнении с контролем (кратность изменения)
| Ген | Точка 1 | Точка 2 | ||
|---|---|---|---|---|
| HCAEC | HITAEC | HCAEC | HITAEC | |
| DNMT1 | 1.70* | 0.82 | 1.16 | 1.34 |
| DNMT3A | 1.37 | 0.85 | 0.78 | 0.93 |
| DNMT3B | 1.38 | 1.29 | 2.00* | 1.20 |
В клеточных культурах HITAEC не обнаружено статистически значимых изменений уровней мРНК ни одного из генов ДНК-метилтрансфераз (рис. 2).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Известно, что ДНК-метилтрансфераза DNMT1 наиболее активно метилирует полуметилированные CpG-сайты ДНК [17]. Фермент может проявлять аномальную метилирующую активность, в частности, метилировать CpG-пары в области петли оцДНК, уже содержащей метилированные сайты CpG. Известно, что инактивация гена DNMT1 приводит к значительному (до 70%) уменьшению метилирования генома, что делает этот ген одним из наиболее важных в регуляции уровня метилирования ДНК у млекопитающих [18]. Кроме того, DNMT1, благодаря своей способности рекрутироваться в область двойных разрывов ДНК, может участвовать в процессах репарации повреждений ДНК напрямую, посредством механизма, не связанного с метилированием (в частности, путем модуляции функции протеинкиназы CHK1 и поддержания активности сенсорных киназ ATM и ATR, вовлеченных в репарацию повреждений генетического аппарата) [19–21]. ДНК-метилтрансфераза DNMT3B метилирует полуметилированные и неметилированные CpG-сайты, в частности, сателлитные повторы в области центромерного линкера [22].
Показано, что метилирование ДНК и клеточный ответ на повреждения ДНК, вызванные как экзогенными, так и эндогенными агентами, связаны друг с другом. Это подтверждается, главным образом, гиперметилированием промоторов и подавлением экспрессии генов репарации ДНК при различных онкологических заболеваниях [23]. DNMT1 рекрутируется в области двойных разрывов ДНК (вызванных, в том числе, и действием ММС) посредством взаимодействия с ядерным антигеном пролиферирующих клеток (PCNA) [20]. Известно, что гиперметилирование промоторных CpG-сайтов вызывает значительное подавление транскрипции, что инактивирует клеточные пути, приводя к эффектам, сходным с эффектами генетических мутаций [23]. Так, гиперметилирование промоторов таких генов репарации ДНК, как MLH1, MSH2, MSH3, MSH6 (мисматч-репарация), MBD4, TDG, OGG1, XPC, XRCC1, RAD23B (эксцизионная репарация нуклеотидов), BRCA1, BRCA2, FANC (гомологичная рекомбинация) и ряда других приводит к снижению их экспрессии, уменьшению активности синтезируемых ферментов репарации ДНК и, как следствие, снижению эффективности репарации повреждений генетического аппарата [3]. Кроме подавления активности отдельных генов, гиперметилирование ДНК нарушает процесс активации сенсорных киназ ATM и ATR, распознающих повреждения ДНК [24], что приводит к снижению эффективности репарации двойных разрывов ДНК, образующихся, главным образом, в результате воздействия алкилирующих мутагенов (к числу которых относится и ММС, использованный нами). Однако следует отметить, что в подавляющем большинстве случаев связь метилирования ДНК с повреждением генетического материала изучали при различных онкологических заболеваниях, поэтому естественно, что основной интерес вызывала роль генов семейства DNMT в развитии рака [3].
Опубликованы данные о снижении уровня маркеров окислительного стресса в микрососудах сетчатки глаза крысы при ингибировании генов семейства Dnmt [25]; культивирование клеток эпителия легкого человека А549 в присутствии ингибиторов ДНК-метилтрансфераз существенно снижало уровень радиационно-индуцированных повреждений ДНК, радиочувствительности этих клеток и повышало уровень экспрессии генов репарации ДНК FANCA, BRCA1 и RAD51C [26]. При этом нам не удалось найти публикаций, в которых изучали ассоциацию экспрессии генов семейства DNMT со степенью проявления генотоксических эффектов и уровнем повреждения ДНК в эндотелиальных клетках, обработанных мутагеном. Полученные нами результаты свидетельствуют о том, что в клеточных культурах HCAEC, в которых повышен уровень транскрипции генов DNMT1 и DNMT3B, повышен также уровень повреждений ДНК [27], что может быть связано с усилением метилирования генов репарации.
Помимо участия метилирования ДНК в формировании ответа клеток на генотоксический стресс, этот эпигенетический механизм вовлечен в формирование эндотелиальной дисфункции и в атерогенез [4–6]. Так, известно, что осцилляторный режим эндотелиального напряжения сдвига (ЭНС), ассоциированный с повышенным риском развития атерогенных эффектов, может повышать уровень транскрипции генов DNMT1 и DNMT3, что, в свою очередь, увеличивает риск развития эндотелиальной дисфункции, воспаления и подавляет синтез оксида азота эндотелиальными клетками [6]. Одновременно с этим пульсирующий режим ЭНС не вызывает изменения уровня экспрессии генов ДНК-метилтрансфераз и характеризуется атеропротективным эффектом [28]. Полученные нами результаты полностью согласуются с опубликованными данными – повышенный уровень экспрессии генов ДНК-метилтрансфераз выявлен в эндотелиальных клетках коронарной артерии (осцилляторный режим ЭНС), характеризующихся более выраженным повышением экспрессии молекулярных маркеров эндотелиальной дисфункции в ответ на мутагенную нагрузку (по сравнению с клетками внутренней грудной артерии, крайне редко поражаемой атеросклерозом за счет своих гемодинамических и физиологических особенностей) [11].
Таким образом, можно предположить, что усиление транскрипции генов ДНК-метилтрансфераз может быть одним из молекулярных механизмов, лежащих в основе формирования эндотелиальной дисфункции в ответ на мутагенную нагрузку в эксперименте in vitro.
Исследование выполнено за счет гранта Российского научного фонда (№ 21-75-10052 “Молекулярные механизмы развития эндотелиальной дисфункции в ответ на генотоксический стресс”, https://rscf.ru/project/21-75-10052/).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Robertson K.D. (2005) DNA methylation and human disease. Nat. Rev. Genet. 6(8), 597–610.
Kareta M.S., Botello Z.M., Ennis J.J., Chou C., Chédin F. (2006) Reconstitution and mechanism of the stimulation of de novo methylation by human DNMT3L. J. Biol. Chem. 281(36), 25893–25902.
Jin B., Robertson K.D. (2013) DNA methyltransferases, DNA damage repair, and cancer. Adv. Exp. Med. Biol. 754, 3–29.
Dunn J., Thabet S., Jo H. (2015) Flow-dependent epigenetic DNA methylation in endothelial gene expression and atherosclerosis. Arterioscler. Thromb. Vasc. Biol. 35(7), 1562–1569.
Lyko F. (2018) The DNA methyltransferase family: a versatile toolkit for epigenetic regulation. Nat. Rev. Genet. 19(2), 81–92.
Lee D.Y., Chiu J.J. (2019) Atherosclerosis and flow: roles of epigenetic modulation in vascular endothelium. J. Biomed. Sci. 26(1), 56.
Gray K., Bennett M. (2011) Role of DNA damage in atherosclerosis – bystander or participant? Biochem. Pharmacol. 82(7), 693–700.
Shah N.R., Mahmoudi M. (2015) The role of DNA damage and repair in atherosclerosis: a review. J. Mol. Cell. Cardiol. 86, 147–157.
Кутихин А.Г., Синицкий М.Ю., Понасенко А.В. (2017) Роль мутагенеза в развитии атеросклероза. Комплексные проблемы сердечно-сосудистых заболеваний. 6, 92–101.
Sinitsky M.Y., Kutikhin A.G., Tsepokina A.V., Shishkova D.K., Asanov M.A., Yuzhalin A.E., Minina V.I., Ponasenko A.V. (2020) Mitomycin C induced genotoxic stress in endothelial cells is associated with differential expression of proinflammatory cytokines. Mutat. Res. Genet. Toxicol. Environ. Mutagen. 858–860, 503252.
Синицкий М.Ю., Цепокина А.В., Кутихин А.Г., Шишкова Д.К., Понасенко А.В. (2021) Профиль генной экспрессии в эндотелиальных клетках, культивируемых в присутствии митомицина С. Биомед. химия. 67(2), 130–136.
GBD 2017 Causes of Death Collaborators (2018) Global, regional, and national age-sex-specific mortality for 282 causes of death in 195 countries and territories, 1980–2017: a systematic analysis for the Global Burden of Disease Study 2017. Lancet. 392(10159), 1736–1788.
Lorge E., Thybaud V., Aardema M.J., Oliver J., Wa-kata A., Lorenzon G., Marzin D. (2006) SFTG international collaborative study on in vitro micronucleus test I. General conditions and overall conclusions of the study. Mutat. Res. 607(1), 13–36.
OECD, Guidelines for the Testing of Chemicals no. 487: In vitro Mammalian Cell Micronucleus Test (Mnvit), OECD, Paris, France, 2016.
Vandesompele J., De Preter K., Pattyn F., Poppe B., Van Roy N., De Paepe A., Speleman F. (2002) Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 3(7), RESEARCH0034.
Bustin S.A., Benes V., Garson J.A., Hellemans J., Huggett J., Kubista M., Mueller R., Nolan T., Pfaffl M.W., Shipley G.L., Vandesompele J., Wittwer C.T. (2009) The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin. Chem. 55(4), 611–622.
Bronner C., Alhosin M., Hamiche A., Mousli M. (2019). Coordinated dialogue between UHRF1 and DNMT1 to ensure faithful inheritance of methylated DNA patterns. Genes (Basel). 10(1), 65. https://doi.org/10.3390/genes10010065
Adam S., Anteneh H., Hornisch M., Wagner V., Lu, J., Radde N.E., Bashtrykov P., Song J., Jeltsch A. (2020) DNA sequence-dependent activity and base flipping mechanisms of DNMT1 regulate genome-wide DNA methylation. Nat. Commun. 11(1), 3723.
Unterberger A., Andrews S.D., Weaver I.C., Szyf M. (2006) DNA methyltransferase 1 knockdown activates a replication stress checkpoint. Mol. Cell. Biol. 26(20), 7575–7586.
Ha K., Lee G.E., Palii S.S., Brown K.D., Takeda Y., Liu K., Bhalla K.N., Robertson, K.D. (2011) Rapid and transient recruitment of DNMT1 to DNA double-strand breaks is mediated by its interaction with multiple components of the DNA damage response machinery. Hum. Mol. Genet. 20(1), 126–140.
Loughery J.E., Dunne P.D., O’Neill K.M., Meehan R.R., McDaid J.R., Walsh C.P. (2011) DNMT1 deficiency triggers mismatch repair defects in human cells through depletion of repair protein levels in a process involving the DNA damage response. Hum. Mol. Genet. 20(16), 3241–3255.
Gagliardi M., Strazzullo M., Matarazzo M.R. (2018) DNMT3B functions: novel insights from human disease. Front. Cell Dev. Biol. 6, 140.
Jacinto F.V., Esteller M. (2007) Mutator pathways unleashed by epigenetic silencing in human cancer. Mutagenesis. 22(4), 247–253.
Williams L.H., Choong D., Johnson S.A., Campbell I.G. (2006) Genetic and epigenetic analysis of CHEK2 in sporadic breast, colon, and ovarian cancers. Clin. Cancer Res. 12(23), 6967–6972.
Duraisamy A.J., Mishra M., Kowluru A., Kowluru R.A. (2018) Epigenetics and regulation of oxidative stress in diabetic retinopathy. Invest. Ophthalmol. Vis. Sci. 59(12), 4831–4840.
Kang H.C., Chie E.K., Kim H.J., Kim J.H., Kim I.H., Kim K., Shin B.S., Ma E. (2019) A phthalimidoalkanamide derived novel DNMT inhibitor enhanced radiosensitivity of A549 cells by inhibition of homologous recombination of DNA damage. Invest. New Drugs. 37(6), 1158–1165.
Синицкий М.Ю., Кутихин А.Г., Шишкова Д.К., Асанов М.А., Понасенко А.В. (2020) Оценка цитотоксических и генотоксических эффектов митомицина С в культурах эндотелиальных клеток человека. Гены и Клетки. 15(1), 45–49.
Zhang Y.P., Huang Y.T., Huang T.S., Pang W., Zhu J.J., Liu Y.F., Tang R.Z., Zhao C.R., Yao W.J., Li Y.S., Chien S., Zhou J. (2017) The mammalian target of rapamycin and DNA methyltransferase 1 axis mediates vascular endothelial dysfunction in response to disturbed flow. Sci. Rep. 7(1), 14996.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология