Молекулярная биология, 2022, T. 56, № 4, стр. 546-556
Фактор рестрикции ВИЧ APOBEC3G и перспективы его использования в генной терапии ВИЧ-инфекции
А. С. Тихонов a, *, Р. Р. Минтаев a, b, Д. В. Глазкова a, Е. В. Богословская a, Г. А. Шипулин a
a Центр стратегического планирования и управления медико-биологическими рисками здоровью
Федерального медико-биологического агентства
119833 Москва, Россия
b Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова
105064 Москва, Россия
* E-mail: andrey.tikhonov97@gmail.com
Поступила в редакцию 27.11.2021
После доработки 11.02.2022
Принята к публикации 17.02.2022
- EDN: DFNIOQ
- DOI: 10.31857/S0026898422040115
Аннотация
Механизмы защиты организма человека от вирусных или бактериальных агентов чрезвычайно разнообразны. В одном из таких механизмов важная роль принадлежит семейству цитидиндезаминаз APOBEC3, которые являются факторами врожденного иммунитета и защищают организм от многочисленных вирусных агентов. Один из белков этого семейства ‒ APOBEC3G ‒ способен защищать от вируса иммунодефицита человека типа 1 в отсутствие вирусного белка Vif. В свою очередь, Vif противостоит действию APOBEC3G, вызывая его полиубиквитинирование и деградацию в протеасоме. В обзоре описаны возможные пути увеличения анти-ВИЧ-активности APOBEC3G, придания ему устойчивости к вирусному белку Vif, а также потенциальные подходы к использованию модифицированного APOBEC3G в генотерапии ВИЧ-инфекции.
ВВЕДЕНИЕ
Течение ВИЧ-инфекции в настоящее время успешно контролируется при помощи высокоактивной антиретровирусной терапии (ВААРТ), которая подавляет репликацию вируса и снижает вирусную нагрузку, что существенно замедляет развитие СПИДа и препятствует дальнейшему распространению вируса [1]. Однако ВААРТ не способна полностью избавить организм от ВИЧ, поскольку остаются латентно инфицированные клетки, репликация вируса в которых возобновляется при отмене терапии. Длительный прием препаратов приводит к появлению побочных эффектов и развитию лекарственной резистентности, что делает необходимым изменение схемы лечения [2].
Очевидна необходимость в поиске подходов, которые позволят сдерживать репликацию ВИЧ при отсутствии постоянной антиретровирусной терапии или даже полностью элиминируют вирус из организма. Один из таких подходов ‒ модификация присутствующих в клетках-мишенях факторов рестрикции ВИЧ, направленная на придание им большей эффективности, таких как TRIM5a, SAMHD1, IFITM, TETHRIN, MARCH8, APOBEC3G и других [3‒8]. Довольно много исследований посвящено белку TRIM5α, механизмам рестрикции и способам повышения его активности в отношении ВИЧ [9‒11]. Ведутся работы по созданию на его основе генно-терапевтического препарата [12]. В настоящем обзоре рассмотрен другой перспективный фактор рестрикции ‒ белок семейства APOBEC3, участвующего во врожденном противовирусном иммунном ответе [13, 14]. Белки этого семейства способны дезаминировать вирусную одноцепочечную ДНК, приводя к гипермутациям вирусного генома и делая вирус нежизнеспособным [15‒18].
Настоящий обзор посвящен фактору рестрикции APOBEC3G, так как многочисленные исследования показывают, что из всего семейства APOBEC3 наибольший вклад в подавление репликации ВИЧ вносит именно белок APOBEC3G [19‒21].
МЕХАНИЗМ АНТИ-ВИЧ- АКТИВНОСТИ APOBEC3G
Попадая в организм человека, ВИЧ поражает в основном CD4+ лимфоциты, а также макрофаги, моноциты и некоторые другие популяции клеток. Жизненный цикл этого вируса состоит из нескольких ключевых стадий, блокирование которых может быть использовано в борьбе с ним. Для входа в клетку вирусные частицы, на поверхности которых находится белок оболочки Env (гликопротеин gp120), взаимодействуют с клеточным рецептором CD4 и одним из корецепторов CCR5 или CXCR4. После слияния мембран образуется предынтеграционный комплекс и начинается процесс обратной транскрипции вирусной РНК. Предынтеграционный комплекс транспортируется в ядро, где провирусная ДНК встраивается в геном клетки-хозяина. Провирусная ДНК служит матрицей для синтеза нескольких форм вирусных РНК, одни из которых транслируются в вирусные белки, а другие упаковываются в вирусные частицы. После упаковки полноразмерных вирусных РНК в капсид от мембраны клетки-хозяина отпочковываются новые вирионы, которые представляют собой капсиды в оболочке, сформированной из клеточной мембраны [22, 23].
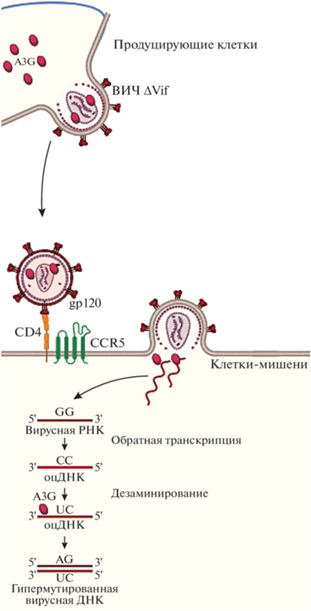
Понимание механизма действия APOBEC3G на ВИЧ пришло после обнаружения способности этого белка полностью ингибировать репликацию ВИЧ, дефектного по белку Vif [24]. Оказалось, что присутствующий в зараженной клетке APOBEC3G способен упаковываться в новые вирусные частицы, связываясь с РНК ВИЧ [25, 26], либо образуя комплекс с полипротеином Gag [27, 28]. Попадая в новую клетку-мишень в составе вирусной частицы, APOBEC3G во время обратной транскрипции модифицирует остатки цитозина до урацила в зарождающейся первой цепи вирусной кДНК [29, 30]. Это приводит к появлению множества мутаций в геноме вируса при синтезе второй цепи ДНК, в которой происходят замены гуанина на аденин (рис. 1) [31, 32], что может вызывать образование стоп-кодонов или аминокислотных замен в белках вируса и делать его нежизнеспособным. Необходимо подчеркнуть, что в присутствии функционального белка Vif противовирусное действие APOBEC3G полностью подавляется.
Рис. 1.
Ингибирование репликации ВИЧ в отсутствие функционального белка Vif. В зараженных клетках в отсутствие вирусного фактора Vif белок APOBEC3G упаковывается в вирион ВИЧ. Гликопротеин gp120 связывается с рецептором CD4 и корецептором CCR5, что индуцирует слияние клеточной и вирусной мембран. После проникновение вируса в клетку ревертаза ВИЧ синтезирует вирусную ДНК на матрице РНК. В это время APOBEC3G дезаминирует остатки цитозина в первой цепи ДНК вируса, что в дальнейшем приводит к множественным заменам гуанина на аденин.
Вирусный белок Vif защищает вирус от действия цитозиндезаминаз [33, 34]. Он вызывает убиквитинирование и деградацию APOBEC3G [34]. Vif связывает N-концевой домен APOBEC3G, после чего вовлекает белковые факторы Сul5, RBX2, EloC, EloB и CBF-β в формирование убиквитинлигазного комплекса (рис. 2). Основным компонентом этого комплекса является белок Cul5, связывающий убиквитинлигазу через белок RBX2 (RING-box protein 2) [36]. Факторы элонгации EloC, EloB и транскрипционный фактор CBF-β необходимы для стабилизации связи между Vif и Cul5 в формирующемся комплексе [36, 37].
Рис. 2.
Образование убиквитинлигазного комплекса и протеасомная деградация APOBEC3G. Вирусный белок Vif связывает APOBEC3G. Вокруг Vif-APOBEC3G формируется убиквитинлигазный комплекс, включающий факторы Сul5, RBX2, EloC, EloB, и CBF-β, индуцирующий полиубиквитинирование APOBEC3G и его протеасомную деградацию.
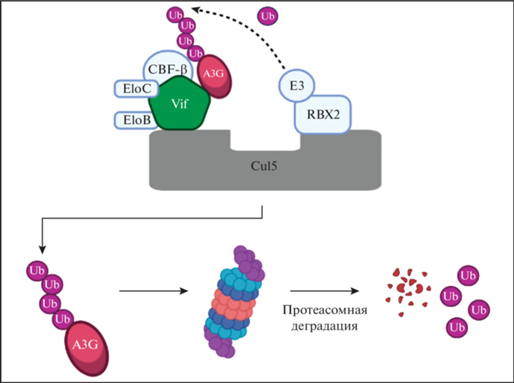
В образовавшемся комплексе убиквитинлигаза оказывается в непосредственной близости от APOBEC3G, в результате чего становится возможным множественное убиквитинирование его С-концевого домена по остаткам лизина. Убиквитинированный APOBEC3G подвергается протеасомной деградации, что приводит к уменьшению количества этого белка. Снижение концентрации APOBEC3G в клетке делает неэффективной его упаковку в вирусные частицы, а, следовательно, дезаминирование и последующую гипермутацию вирусных геномов. Низкая частота мутирования ВИЧ, вызываемая остаточной активностью APOBEC3G, приводит к увеличению его генетического разнообразия, что способствует уходу вируса от адаптивного иммунного ответа [38–41].
Таким образом, наиболее очевидным подходом к увеличению противовирусной активности APOBEC3G является ингибирование его связывания с белком Vif. Однако предложены и другие подходы, которые будут рассмотрены ниже.
ОНКОГЕННЫЙ ПОТЕНЦИАЛ APOBEC3G
Прежде чем переходить к способам увеличения активности APOBEC3G, стоит несколько слов сказать о тех последствиях, к которым это может привести. К сожалению, повышенная активность данного белка связана с потенциальной онкогенной активностью, что показано в ряде публикаций [42‒48].
Белок APOBEC3G в норме локализован в цитоплазме клетки и не может взаимодействовать с геномной ДНК [49]. Однако Nowarski и соавт. показали [42], что в клетках лимфомы с индуцированными двухцепочечными разрывами ДНК этот белок рекрутируется в ядро. Botvinnik и соавт. [43] установили, что APOBEC3G проникает в ядро и участвует в репарации ДНК, увеличивая генетическое разнообразие опухоли и повышая ее устойчивость к химио- и радиотерапии. Схожие результаты получены и на клетках множественной миеломы [44]. Ядерная локализация АРОВЕС3G показана также и в нормальных, не трансформированных клетках – активированных CD4+ лимфоцитах [50]. Причина изменения внутриклеточной локализации APOBEC3G в активированных CD4+ лимфоцитах ясна не до конца, но, по-видимому, он может участвовать в поддержании стабильности генома, защищая от активности ретротранспозонов [51].
Недавно обнаружили, что APOBEC3G может иметь онкогенную активность, не связанную с механизмами дезаминирования. Так, например, Ding и соавт. [45] установили, что частота метастазирования колоректального рака в печень связана с уровнем экспрессии гена APOBEC3G. Показано, что в основе механизма онкогенной трансформации лежит ингибирующее действие APOBEC3G на микроРНК-29, которая подавляет экспрессию гена ММР-2 [45]. Увеличение концентрации APOBEC3G приводит к повышению экспрессии MMP-2. В то же время, повышенную секрецию MMP-2 считают плохим прогностическим признаком при многих видах рака, в том числе колоректального [46].
Обнаружен и другой механизм онкогенной трансформации, связанный с APOBEC3G. Показано, что APOBEC3G способен связывать 3′‑нетранслируемую область мРНК белка KLF4, тем самым подавляя экспрессию его гена. Подавление экспрессии KLF4 приводит к повышению экспрессии генов SP1 и AATF. Они запускают процесс метаболического переключения клетки на опухолевый рост посредством ингибирования экспрессии гена p51 и активации ряда других генов [47, 48].
Строгой связи между уровнем APOBEC3G и онкогенной трансформацией клетки пока не обнаружено, но проблема безопасности использования APOBEC3G в качестве терапевтического средства остается крайне актуальной и требует дальнейшего изучения.
ПОДХОДЫ К УВЕЛИЧЕНИЮ АНТИ-ВИЧ-АКТИВНОСТИ APOBEC3G
Существующие подходы к увеличению активности APOBEC3G в основном направлены на блокирование одной из стадий его деградации, инициированной белком Vif: связывания APOBEC3G и Vif; образования убиквитинлигазного комплекса; убиквитинирования APOBEC3G; протеасомной деградации APOBEC3G (см. рис. 3).
Рис. 3.
Стадии деградации APOBEC3G под действием Vif и механизмы их блокирования. а ‒ Мутантный APOBEC3G не связывается с Vif. б – RUNX1 конкурентно блокирует образование убиквитинлигазного комплекса. в ‒ Деубиквитинирование A3G под действием USP49. г – Полиубиквитинированный слитый белок APOBEC3G-UBA2 не подвергается деградации в протеасоме.
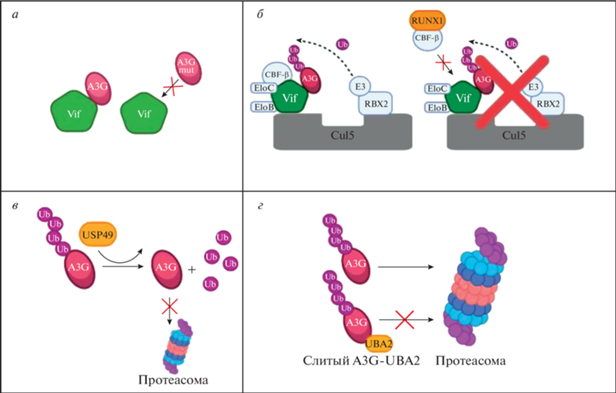
Нарушение связывания APOBEC с Vif
В настоящее время предложено несколько подходов к достижению устойчивости APOBEC3G к действию Vif. Один из них – внесение мутаций в сайт связывания APOBEC3G и Vif, что может предотвратить деградацию APOBEC3G и, как следствие, восстановить защиту от ВИЧ. Однако детали взаимодействия APOBEC3G и Vif до недавнего времени были недостаточно изучены. Известно лишь, что критически важной областью APOBEC3G является петля β4-α4, которая включает в себя аминокислотные остатки D128–D130. Недавно удалось точно установить область контакта APOBEC3G и Vif. В своем исследовании Letko и соавт. [57] показали, что во взаимодействии участвуют три аминокислотных остатка: D128, Y125 и D130 белка APOBEC3G. Каждый из этих остатков потенциально может быть мишенью для создания мутантного APOBEC3G, устойчивого к Vif.
Экспериментально показано, что замена остатка аспартата (К) в позиции 128 на лизин (D128K) позволяет предотвратить деградацию APOBEC3G в присутствии Vif. На базе линий CEM, PM1, C8166, стабильно экспрессирующих трансген APOBEC3G D128K, созданы модельные перевиваемые клеточные линии. Заражение таких клеточных линий ВИЧ показало их полную устойчивость к вирусу [58–60]. Защита от широкого спектра вариантов Vif ВИЧ наблюдалась при введении в APOBEC3G мутаций Y125R, D130R [57], а также P129A [61]. Однако из четырех мутаций (Y125R, D130R, P129A, D128K) наиболее широкую и полную защиту от ВИЧ обеспечивал только вариант D128K [57].
Использованию мутантного APOBEC3G (D128K) в генной терапии препятствует ряд проблем, затрудняющих его доставку в клетки-мишени. Помимо собственно доставки трансгена в клетки, необходимо обеспечивать его стабильную и долговременную экспрессию в них. Это необходимое условие для продолжительного и прогнозируемого сдерживания репликации вируса. На сегодняшний день наиболее эффективно доставку трансгенов в клетки-мишени для их стабильной экспрессии осуществляют при помощи лентивирусных векторов, сконструированных на основе ВИЧ. Они способны доставлять трансген в CD4+ Т-лимфоциты, обеспечивать его встраивание в геном, поддерживать постоянную экспрессию и возможность передачи дочерним клеткам в процессе пролиферации [62]. Но производство лентивирусного вектора, содержащего ген APOBEC3G, затрудняется противовирусной активностью белка APOBEC3G, направленной на этот вектор. В клетках-продуцентах с векторной ДНК активно синтезируется белок APOBEC3G, который включается в вирусные частицы. При попадании в целевые клетки во время обратной транскрипции белок APOBEC3G вносит мутации в вектор, в том числе в ген, кодирующий APOBEC3G, что препятствует интеграции функционального гена [63]. Предложены различные варианты решения данной проблемы.
Так, Wang и соавт. предложили дополнительно экспрессировать в клетках, продуцирующих вектор, вариант Vif, активный по отношению к APOBEC3G D128K [59]. В таком случае APOBEC3G D128K будет подвергаться протеасомной деградации и не сможет упаковываться в векторные частицы.
Вторым подходом, предложенным теми же авторами [59], стало использование индуцируемого доксициклином промотора для контроля экспрессии APOBEC3G D128K в клетках-продуцентах.
Лентивирусным вектором, полученным при совместном использовании этих подходов, трансдуцировали первичные CD4+ T-клетки и макрофаги человека. Эффективность трансдукции составила 35 и 40% соответственно. Установлено, что уровень репликации ВИЧ в этих клетках снижается в 2–3 раза относительно контрольных клеток, трансдуцированных вектором, несущим только маркерный ген [59].
Альтернативный подход предложен Delviks-Frankenberry и соавт. [58]. Суть этого подхода состоит в том, что во время наработки лентивирусного вектора ген APOBEC3G, который упаковывается в частицы, находится в инактивированном состоянии. Это достигается путем включения в ген повторяющейся последовательности, которая нарушает рамку считывания. Однако в ходе обратной транскрипции в целевых клетках повтор не включается в образующуюся ДНК, что обусловлено особенностью функционирования ревертазы ретровирусов. В результате ген APOBEC3G, интегрированный в геном, не имеет повтора и является полностью функциональным [64].
Авторами создан основанный на таком подходе лентивирусный вектор, который эффективно ингибировал ВИЧ в экспериментах на перевиваемых клеточных линиях. Однако эффективность трансдукции СD4+ лимфоцитов и CD34+ стволовых клеток составила лишь 15 и 30%, соответственно, поэтому подавления репликации ВИЧ не выявили [58].
Проблемы доставки APOBEC3G в составе лентивирусного вектора можно решить и с помощью других приемов. Например, использовать мобильные генетические элементы [65] или распределить кодирующую последовательность APOBEC3G на несколько вирусных векторов с их объединением в целевых клетках при помощи интеинов [66].
Существуют и другие методы интеграции трансгенов, такие как направленная интеграция с помощью программируемых нуклеаз (Cas9, TALEN и др.). В этом случае нуклеаза вносит разрыв в целевую область хромосомы, в которую затем встраивается донорная ДНК, несущая трансген. Для доставки донорной ДНК удобно использовать AAV-векторы, которые эффективно переносят трансген в ядро клетки, где он встраивается в область разрыва за счет системы репарации клеточной ДНК. Таким образом можно добиться длительной и стабильной экспрессии трансгенов.
Этот подход можно использовать для встраивания модифицированного гена APOBEC3G в клетки-мишени ВИЧ, причем доставка гена с помощью AAV является эффективной, так как известно, что APOBEC3G не влияет на продукцию AAV и трансдукцию клеток вектором, в отличие от лентивирусных векторов [67, 68].
Zhujun Ao и соавт. [61] показана принципиальная возможность использования AAV для доставки APOBEC3G-P129A в мононуклеарные клетки периферической крови (PBMC) и макрофаги человека. AAV-вектор, несущий APOBEC3G P129A, трансдуцировал PBMC и макрофаги человека с эффективностью 23 и 40% соответственно. В зараженных ВИЧ PBMC человека на третий день после трансдукции AAV наблюдалось 2‒3-кратное снижение накопления вирусного антигена p24 по сравнению с нетрансдуцированными клетками.
Блокирование связывания APOBEC3G и Vif возможно и при помощи альтернативных стратегий. Одна из них – использование низкомолекулярных веществ, связывающих Vif и блокирующих его взаимодействие с APOBEC3G. Успехи в разработке таких молекул (RN-18 и его аналоги), достигнутые за последние годы, убеждают в эффективности этого подхода [53‒56]. Но использование блокаторов связывания APOBEC3G и Vif – это лишь новое направление в химиотерапии ВИЧ-инфекции, которое не лишено таких недостатков АРТ, как пожизненный прием препаратов и их токсичность.
Повышение эффективности упаковки APOBEC3G в вирусную частицу
Как отмечено выше, упаковка APOBEC3G в вирусные частицы чрезвычайно важна для дезаминирования кДНК и способности подавлять репликацию ВИЧ. Для повышения эффективности упаковки предложено использовать пептид R14-88 ‒ фрагмент вирусного белка VPR, который отвечает за упаковку VPR в вирионы. Пептид R14-88 объединили с APOBEC3G в один слитый белок R14-88–APOBEC3G, а также создали клеточную линию C8166-APOBEC3G со стабильной экспрессией белка R14-88–APOBEC3G. Уровень репликации ВИЧ на этой клеточной линии снижается более чем в 10 раз относительно контрольной линии клеток [69]. Кроме того, ВИЧ, полученный после репликации в клетках C8166-APOBEC3G, использовали для заражения PBMC человека. Вирус, полученный из культуры клеток, экспрессирующей R14-88–APOBEC3G, оказался нежизнеспособным.
Добавление фрагмента вирусного белка VPR теоретически могло повлиять на локализацию белка APOBEC3G внутри клетки (так как VPR это ядерный белок) и сделать его небезопасным с учетом возможного онкогенного потенциала. Однако показано, что внутриклеточная локализация APOBEC3G, слитого с фрагментом R14-88 этого белка, не изменяется, он остается в цитоплазме [69].
Слитый белок R14-88–APOBEC3G для достижения наибольшего противовирусного эффекта целесообразно использовать в комбинации с другими подходами, например, совместно с внесением рассмотренных выше мутаций, блокирующих связывание APOBEC3G с Vif. Совместное применение двух подходов позволит наиболее полно преодолеть ограничения, накладываемые Vif, и обеспечить защиту от вируса [63, 69].
Другие подходы, описанные ниже, имеют скорее теоретическое значение, они освещают механизмы, вовлеченные в деградацию APOBEC, поскольку их применение существенно влияет на внутриклеточную концентрацию белка APOBEC3G, что сопряжено с риском онкогенной трансформации.
Блокирование образования убиквитинлигазного комплекса с помощью конкурентного ингибирования
Как показано недавно, деградацию APOBEC3G, опосредованную белком Vif, можно предотвратить с помощью одного из клеточных белков ‒ RUNX1 (Runt-related transcription factor 1) [70]. Установлено, что RUNX1 связывается с CBF-β и может конкурентно блокировать взаимодействие Vif с CBF-β, критически важное для образования комплекса Cul5–убиквитинлигаза (рис. 3б) [71]. При отсутствии взаимодействия между Vif и CBF-β убиквитинирование APOBE3G не происходит [71, 72]. Поэтому увеличение концентрации RUNX1 может предотвращать деградацию фактора APOBE3G.
Для проверки этого предположения в клетках HEK293T собирали вирусные частицы, содержащие функциональный Vif, в присутствии APOBEC3G и RUNX1 или только APOBEC3G. Затем определяли количество белка APOBEC3G в вируссодержащем супернатанте. За 100% принимали содержание APOBEC3G в вирусных частицах, дефектных по белку Vif. Относительное содержание APOBEC3G в вирусных частицах с функциональным Vif из клеток со сверхэкспрессией RUNX1, составляло около 60% от уровня в контрольных клетках, в то время как содержание APOBEC3G в вирусных частицах из клеток без сверхэкспрессии RUNX1 составляло 20% от значения в контрольных клетках [71].
Однако увеличение экспрессии RUNX1 небезопасно само по себе [73]. Белок RUNX1 – это транскрипционный фактор, который участвует в процессе дифференцировки гемопоэтических стволовых клеток. Известно, что белок RUNX1 связан с патогенезом нескольких видов лейкозов, а также рака молочной железы [74].
Блокирование убиквитинирования APOBEC3G
Как известно, сигналом убиквитинирования служит остаток лизина в аминокислотной последовательности APOBEC3G [75]. Белок APOBEC3G включает 20 таких аминокислотных остатков, 13 из них расположены в N-концевом домене белка и семь в С-концевом.
Установлено, что замена остатков лизина в С-концевом домене на остатки аргинина, блокирует протеасомную деградацию, тогда как модификация лизинов N-концевого домена к этому не приводит. Таким образом, белок с заменами в С-концевой области оказался более устойчивым к фактору вирулентности Vif. При этом APOBEC3G с заменами в С-концевом домене сохранял свою дезаминазную активность в отношении ВИЧ, упаковывался в вирионы и эффективно подавлял инфекцию в присутствии Vif, что показано на клеточной линии TZM-bl [76].
Снизить уровень убиквитинирования и предотвратить деградацию APOBEC3G вирусным белком Vif можно за счет механизма деубиквитинирования. Команда Pan и соавт. [77] выяснила, что белок USP49, клеточная деубиквитиназа, способна напрямую связываться с убиквитинированным APOBEC3G и деубиквитинировать остатки лизина, предотвращая тем самым деградацию белка. Установлено, что сверхэкспрессия USP49 действительно повышает устойчивость клеток к ВИЧ.
Нарушение протеасомной деградации
Недавно предложили еще один подход, позволяющий избежать деградации APOBEC3G в протеасоме. Этот подход основан на стабилизации APOBEC3G при помощи регуляторных элементов, расположенных в той же молекуле. В качестве примера такой стабилизации можно привести слитый белок APOBEC3G–UBA2, в котором APOBEC3G объединен с белковым доменом UBA2 (Ubiquitin associated domain 2) [78]. Белки, в состав которых входит домен UBA2, подвергаются убиквитинированию и взаимодействуют с протеасомой, но это не вызывает их деградации [78]. В своем исследовании Li и соавт. [78] сравнили снижение уровня белков APOBEC3G–UBA2 и APOBEC3G в клетках в присутствии Vif. Оказалось, что APOBEC3G дикого типа подвергается полной деградации, в то время как уровень слитого белка APOBEC3G–UBA2 снижается на 60% [78].
В целом, необходимо еще раз подчеркнуть, что действия, направленные на снижение убиквитинирования или протеасомой деградации APOBEC3G, нарушают естественный механизм элиминирования белка и увеличивают его концентрацию в клетке, что, принимая во внимание мутагенную активность APOBEC3G, потенциально опасно.
ЗАКЛЮЧЕНИЕ
Белки семейства APOBEC рассматриваются в качестве привлекательных мишеней для генной терапии ВИЧ-инфекции. На настоящий момент доказана высокая антиретровирусная активность APOBEC3G, разработаны несколько принципиально разных подходов к достижению устойчивости APOBEC3G к деградации вирусным белком Vif при ВИЧ-инфекции. Наибольший интерес представляет совместное использование устойчивого к вирусному белку Vif мутантного варианта APOBEC3G D128K и пептида R14-88, улучшающего упаковку APOBEC3G D128K в вирусные частицы. Показано, что и тот, и другой белок не увеличивают уровень APOBEC3G в клетке [58, 69], что может указывать на безопасность данного подхода, однако это необходимо подтвердить, проведя дополнительные исследования мутагенной активности варианта APOBEC3G с мутацией D128K.
Нерешенной остается проблема эффективной доставки модифицированного гена в клетки-мишени. Однако в настоящее время активно разрабатываются различные варианты доставки генов и белков в клетки-мишени как ex vivo, так и in vivo. Поэтому в ближайшее время можно ожидать появления препаратов против ВИЧ, основанных на модификации естественных факторов рестрикции, в том числе APOBEC3G.
Написание обзора не потребовало специального финансирования.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследования.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Antiretroviral Therapy Cohort Collaboration. (2008) Life expectancy of individuals on combination antiretroviral therapy in high-income countries: a collaborative analysis of 14 cohort studies. Lancet. 372, 293‒299.
Wang J., Holmes M.C. (2016) Engineering hematopoietic stem cells toward a functional cure of human immunodeficiency virus infection. Cytotherapy. 18(11), 1370–1381.
Arias J.F., Heyer L.N., von Bredow B., Weisgrau K.L., Moldt B., Burton D.R., Rakasz E.G., Evans D.T. (2014) Tetherin antagonism by Vpu protects HIV-infected cells from antibody-dependent cell-mediated cytotoxicity. Proc. Natl. Acad. Sci. USA. 111(17), 6425–6430.
Ayinde D., Bruel T., Cardinaud S., Porrot F., Prado J.G., Moris A., Schwartz O. (2015) SAMHD1 Limits HIV-1 antigen presentation by monocyte-derived dendritic cells. J. Virol. 89(14), 6994–7006.
Jimenez-Moyano E., Ruiz A., Kløverpris H.N., Rodriguez-Plata M.T., Peña R., Blondeau C., Selwood D.L., Izquierdo-Useros N., Moris A., Clotet B., Goulder P., Towers G.J., Prado J.G. (2016) Nonhuman TRIM5 variants enhance recognition of HIV-1-infected cells by CD8+ T cells. J. Virol. 90(19), 8552–8562.
Stopak K.S., Chiu Y.L., Kropp J., Grant R.M., Greene W.C. (2006) Distinct patterns of cytokine regulation of APOBEC3G expression and activity in primary lymphocytes, macrophages, and dendritic cells. J. Biol. Chem. 282(6), 3539–3546.
Lu J., Pan Q., Rong L., Liu S.L., Liang C., Liang C. (2011) The IFITM proteins inhibit HIV-1 infection. J. Virol. 85(5), 2126–2137.
Tada T., Zhang Y., Koyama T., Tobiume M., Tsunetsugu-Yokota Y., Yamaoka S., Fujita H., Tokunaga K. (2015) MARCH8 inhibits HIV-1 infection by reducing virion incorporation of envelope glycoproteins. Nat. Med. 21(12), 1502–1507.
Dufour C., Claudel A., Joubarne N., Merindol N., Maisonnet T., Masroori N., Plourde M. B., Berthoux, L. (2018) Editing of the human TRIM5 gene to introduce mutations with the potential to inhibit HIV-1. PLoS One. 13(1), e0191709.
Омельченко Д.О., Глазкова Д.В., Богословская Е.В., Урусов Ф.А., Жогина Ю.А., Цыганова Г.М., Шипулин Г.А. (2018) Защита лимфоцитов от ВИЧ c помощью лентивирусного вектора, несущего комбинацию генов TRIM5A-HRH и микроРНК против CCR5. Молекуляр. биология. 52(2), 294–305.
Jimenez-Guardeño J.M., Apolonia L., Betancor G., Malim M.H. (2019) Immunoproteasome activation enables human TRIM5α restriction of HIV-1. Nat. Microbiol. 4(6), 933‒940.
Uchida N., Hsieh M.M., Washington K.N., Tisdale J.F. (2013) Efficient transduction of human hematopoietic repopulating cells with a chimeric HIV1-based vector including SIV capsid. Exp. Hematol. 41(9), 779‒788.
Harris R.S., Liddament M.T. (2004) Retroviral restriction by APOBEC proteins. Nat. Rev. Immunol. 4(11), 868–877.
Conticello S.G. (2008) The AID/APOBEC family of nucleic acid mutators. Genome Biol. 9(6), 229.
Sheehy A.M., Gaddis N.C., Choi J.D., Malim M.H. (2002) Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral vif protein. Nature. 418(6898), 646‒650.
LaRue R.S., Jonsson S.R., Silverstein K.A., Lajoie M., Bertrand D., El Mabrouk N., Hotzel I., Andresdottir V., Smith T.P., Harris R.S. (2008) The artiodactyl APOBEC3 innate immune repertoire shows evidence for a multi-functional domain organization that existed in the ancestor of placental mammals. BMC Mol. Biol. 9, 104.
Vieira V.C., Soares M.A. (2013) The role of cytidine deaminases on innate immune responses against human viral infections. Biomed. Res. Int. 2013, 683095
Stavrou S., Ross S.R. (2015) APOBEC3 proteins in viral immunity. J. Immunol. 195(10), 4565‒4570.
Chaipan C., Smith J.L., Hu W.S., Pathak V.K. (2013) APOBEC3G restricts HIV-1 to a greater extent than APOBEC3F and APOBEC3DE in human primary CD4+ T cells and macrophages. J. Virol. 87(1), 444‒453.
Zennou V., Bieniasz P.D. (2006) Comparative analysis of the antiretroviral activity of APOBEC3G and APOBEC3F from primates. Virology. 349(1), 31‒40.
Mbisa J.L., Bu W., Pathak V.K. (2010) APOBEC3F and APOBEC3G inhibit HIV-1 DNA integration by different mechanisms. J. Virol. 84(10), 5250‒5259.
Chan D.C., Kim P.S. (1998) HIV entry and its inhibition. Cell. 93(5), 681‒684.
Li C., Burdick R.C., Nagashima K., Hu W.S., Pathak V.K. (2021) HIV-1 cores retain their integrity until minutes before uncoating in the nucleus. Proc. Natl. Acad. Sci. USA. 118(10), e2019467118.
Harris R.S., Dudley J.P. (2015) APOBECs and virus restriction. Virology. 479‒480, 131‒145.
Khan M.A., Kao S., Miyagi E., Takeuchi H., Goila-Gaur R., Opi S., Gipson C.L., Parslow T.G., Ly H., Strebel K. (2005) Viral RNA is required for the association of APOBEC3G with human immunodeficiency virus type 1 nucleoprotein complexes. J. Virol. 79(9), 5870‒5874.
Yang H., Ito F., Wolfe A.D., Li S., Mohammadzadeh N., Love R.P., Yan M., Zirkle B., Gaba A., Chelico L., Chen X.S. (2020) Understanding the structural basis of HIV-1 restriction by the full length double-domain APOBEC3G. Nat. Commun. 11(1), 632.
Schäfer A., Bogerd H.P., Cullen B.R. (2004) Specific packaging of APOBEC3G into HIV-1 virions is mediated by the nucleocapsid domain of the Gag polyprotein precursor. Virology. 328(2), 163‒168.
Cen S., Guo F., Niu M., Saadatmand J., Deflassieux J., Kleiman L. (2004) The interaction between HIV-1 Gag and APOBEC3G. J. Biol. Chem. 279(32), 33177–33184.
Salter J.D., Polevoda B., Bennett R.P., Smith H.C. (2019) Regulation of antiviral innate immunity through APOBEC ribonucleoprotein complexes. Subcell. Biochem. 93, 193‒219.
Morse M., Naufer M.N., Feng Y., Chelico L., Rouzina I., Williams M.C. (2019) HIV restriction factor APOBEC3G binds in multiple steps and conformations to search and deaminate single-stranded DNA. Elife. 8, e52649.
Browne E.P., Allers C., Landau N.R. (2009) Restriction of HIV-1 by APOBEC3G is cytidine deaminase-dependent. Virology. 387(2), 313‒321.
Yu Q., König R., Pillai S., Chiles K., Kearney M., Palmer S., Richman D., Coffin J.M., Landau N.R. (2004) Single-strand specificity of APOBEC3G accounts for minus-strand deamination of the HIV genome. Nat. Struct. Mol. Biol. 11(5), 435‒442.
Feng Y., Baig T.T., Love R.P., Chelico L. (2014) Suppression of APOBEC3-mediated restriction of HIV-1 by vif. Front. Microbiol. 5, 450.
Soliman M., Srikrishna G., Balagopal A. (2017) Mechanisms of HIV-1 control. Curr. HIV/AIDS Rep. 14(3), 101‒109.
Yu X., Yu Y., Liu B., Luo K., Kong W., Mao P., Yu X.F. (2003) Induction of APOBEC3G ubiquitination and degradation by an HIV-1 vif-Cul5-SCF complex. Science. 302(5647), 1056‒1060.
Guo Y., Dong L., Qiu X., Wang Y., Zhang B., Liu H., Yu Y., Zang Y., Yang M., Huang Z. (2014) Structural basis for hijacking CBF-β and CUL5 E3 ligase complex by HIV-1 vif. Nature. 505(7482), 229‒233.
Went M., Kinnersley B., Sud A., Johnson D.C., Weinhold N., Försti A., van Duin M., Orlando G., Mit-chell J.S., Kuiper R., Walker B.A., Gregory W.M., Hoffmann P., Jackson G.H., Nöthen M.M., da Silva Filho M.I., Thomsen H., Broyl A., Davies F.E., Thorsteinsdottir U., Hansson M., Kaiser M., Sonneveld P., Goldschmidt H., Stefansson K., Hemminki K., Nilsson B., Morgan G.J., Houlston R.S. (2019) Transcriptome-wide association study of multiple myeloma identifies candidate susceptibility genes. Hum. Genomics. 13(1), 37.
Kim E.Y., Lorenzo-Redondo R., Little S.J., Chung Y.S., Phalora P.K., Maljkovic Berry I., Archer J., Penugonda S., Fischer W., Richman D.D., Bhattacharya T., Malim M.H., Wolinsky S.M. (2014) Human APOBEC3 induced mutation of human immunodeficiency virus type-1 contributes to adaptation and evolution in natural infection. PLoS Pathol. 10(7), e1004281.
Kim E.Y., Bhattacharya T., Kunstman K., Swantek P., Koning F.A., Malim M.H., Wolinsky S.M. (2010) Human APOBEC3G-mediated editing can promote HIV-1 sequence diversification and accelerate adaptation to selective pressure. J. Virol. 84(19), 10402‒10405.
Venkatesan S., Rosenthal R., Kanu N., McGranahan N., Bartek J., Quezada S.A., Hare J., Harris R.S., Swanton C. (2018) Perspective: APOBEC mutagenesis in drug resistance and immune escape in HIV and cancer evolution. Ann. Oncol. 29(3), 563‒572.
Ikeda T., Yue Y., Shimizu R., Nasser H. (2021) Potential utilization of APOBEC3-mediated mutagenesis for an HIV-1 functional cure. Front. Microbiol. 12, 686357.
Nowarski R., Wilner O.I., Cheshin O., Shahar O.D., Kenig E., Baraz L., Britan-Rosich E., Nagler A., Harris R.S., Goldberg M., Willner I., Kotler M. (2012) APOBEC3G enhances lymphoma cell radioresistance by promoting cytidine deaminase-dependent DNA repair. Blood. 120(2), 366‒375.
Botvinnik A., Shivam P., Smith Y., Sharma G., Olshevsky U., Moshel O., Manevitch Z., Climent N., Oliva H., Britan-Rosich E., Kotler M. (2021) APOBEC3G rescues cells from the deleterious effects of DNA damage. FEBS J. 288(20), 6063‒6077.
Talluri S., Samur M.K., Buon L., Kumar S., Potluri L.B., Shi J., Prabhala R.H., Shammas M.A., Munshi N.C. (2021) Dysregulated APOBEC3G causes DNA damage and promotes genomic instability in multiple myeloma. Blood Cancer J. 11(10), 166.
Ding Q., Chang C.J., Xie X., Xia W., Yang J.Y., Wang S.C., Wang Y., Xia J., Chen L., Cai C., Li H., Yen C.J., Kuo H.P., Lee D.F., Lang J., Huo L., Cheng X., Chen Y.J., Li C.W., Jeng L.B., Hsu J.L., Li L.Y., Tan A., Curley S.A., Ellis L.M., Dubois R.N., Hung M.C. (2011) APOBEC3G promotes liver metastasis in an orthotopic mouse model of colorectal cancer and predicts human hepatic metastasis. J. Clin. Invest. 121(11), 4526‒4536.
Desimmie B.A., Delviks-Frankenberrry K.A., Burdick R.C., Qi D., Izumi T., Pathak V.K. (2014) Multiple APOBEC3 restriction factors for HIV-1 and one vif to rule them all. J. Mol. Biol. 426(6), 1220‒1245.
Garg A., Kaul D., Chauhan N. (2015) APOBEC3G governs to ensure cellular oncogenic transformation. Blood Cells Mol. Dis. 55(3), 248‒254.
Sharma S., Garg A., Dhanda R.S., Kaul D. (2016) APOBEC3G governs the generation of truncated AATF protein to ensure oncogenic transformation. Cell Biol. Int. 40(12), 1366‒1371.
Lackey L., Law E.K., Brown W.L., Harris R.S. (2013) Subcellular localization of the APOBEC3 proteins during mitosis and implications for genomic DNA deamination. Cell Cycle. 12, 762–772.
Oliva H., Pacheco R., Martinez-Navio J.M., Rodríguez-García M., Naranjo-Gómez M., Climent N., Prado C., Gil C., Plana M., García F., Miró J.M., Franco R., Borras F.E., Navaratnam N., Gatell J.M., Gallart T. (2016) Increased expression with differential subcellular location of cytidine deaminase APOBEC3G in human CD4(+) T-cell activation and dendritic cell maturation. Immunol. Cell Biol. 94(7), 689‒700.
Arias J.F., Koyama T., Kinomoto M., Tokunaga K. (2012) Retroelements versus APOBEC3 family members: no great escape from the magnificent seven. Front. Microbiol. 3, 275.
Chiang A.C., Massagué J. (2008) Molecular basis of metastasis. N. Engl. J. Med. 359(26), 2814‒2823.
Nathans R., Cao H., Sharova N., Ali A., Sharkey M., Stranska R., Stevenson M., Rana T.M. (2008) Small-molecule inhibition of HIV-1 Vif. Nat. Biotechnol. 26(10), 1187‒1192.
Mohammed I., Kummetha I.R., Singh G., Sharova N., Lichinchi G., Dang J., Stevenson M., Rana T.M. (2016) 1,2,3-Triazoles as amide bioisosteres: discovery of a new class of potent HIV-1 Vif antagonists. J. Med. Chem. 59(16), 7677‒7682.
Zhou M., Luo R.H., Hou X.Y., Wang R.R., Yan G.Y., Chen H., Zhang R.H., Shi J.Y., Zheng Y.T., Li R., Wei Y.Q. (2017) Synthesis, biological evaluation and molecular docking study of N-(2-methoxyphenyl)-6-((4-nitrophenyl)sulfonyl)benzamide derivatives as potent HIV-1 Vif antagonists. Eur. J. Med. Chem. 129, 310‒324.
Sharkey M., Sharova N., Mohammed I., Huff S.E., Kummetha I.R., Singh G., Rana T.M., Stevenson M. (2019) HIV-1 escape from small-molecule antagonism of Vif. mBio. 10(1), e00144-19.
Letko M., Booiman T., Kootstra N., Simon V., Ooms M. (2015) Identification of the HIV-1 vif and human APOBEC3G protein interface. Cell Rep. 13(9), 1789–1799.
Delviks-Frankenberry K.A., Ackerman D., Timberlake N.D., Hamscher M., Nikolaitchik O.A., Hu W.S., Torbett B.E., Pathak V.K. (2019) Development of lentiviral vectors for HIV-1 gene therapy with vif-resistant APOBEC3G. Mol. Ther. Nucl. Acids. 18, 1023–1038.
Wang X., Ao Z., Jayappa K.D., Shi B., Kobinger G., Yao X. (2014) R88-APOBEC3Gm inhibits the replication of both drug-resistant strains of HIV-1 and viruses produced from latently infected cells. Mol. Ther. Nucl. Acids. 3(3), e151.
Voit R.A., McMahon M.A., Sawyer S.L., Porteus M.H. (2013) Generation of an HIV resistant T-cell line by targeted “stacking” of restriction factors. Mol. Ther. 21(4), 786‒795.
Ao Z., Wang X., Bello A., Jayappa K.D., Yu Z., Fowke K., He X., Chen X., Li J., Kobinger G., Yao X. (2011) Characterization of anti-HIV activity mediated by R88-APOBEC3G mutant fusion proteins in CD4+ T cells, peripheral blood mononuclear cells, and macrophages. Hum. Gene. Ther. 22(10), 1225‒1237.
Зайкова Е.К., Левчук К.A., Поздняков Д.Ю., Дакс А.А., Зарицкий А.Ю., Петухов А.В. (2020) Эффективная трансдукция Т-лимфоцитов лентивирусными частицами в онкоиммунологических исследованиях. Клиническая онкогематология. 13(3), 295–306.
Delviks-Frankenberry K.A., Desimmie B.A., Pathak V.K. (2020) Structural insights into APOBEC3-mediated lentiviral restriction. Viruses. 12(6), 587.
Hu W.S., Pathak V.K. (2000) Design of retroviral vectors and helper cells for gene therapy. Pharmacol. Rev. 52(4), 493‒511.
Sandoval-Villegas N., Nurieva W., Amberger M., Ivics Z. (2021) Contemporary transposon tools: a review and guide through mechanisms and applications of sleeping beauty, piggyBac and Tol2 for genome engineering. Int. J. Mol. Sci. 22(10), 5084.
Jillette N., Du M., Zhu J.J., Cardoz P., Cheng A.W. (2019) Split selectable markers. Nat. Commun. 10(1), 4968.
Chen H., Lilley C.E., Yu Q., Lee D.V., Chou J., Narvaiza I., Landau N.R., Weitzman M.D. (2006) APOBEC3A is a potent inhibitor of adeno-associated virus and retrotransposons. Curr. Biol. 16(5), 480‒485.
Narvaiza I., Linfesty D.C., Greener B.N., Hakata Y., Pintel D.J., Logue E., Landau N.R., Weitzman M.D. (2009) Deaminase-independent inhibition of parvoviruses by the APOBEC3A cytidine deaminase. PLoS Pathog. 5(5), e1000439.
Ao Z., Yu Z., Wang L., Zheng Y., Yao X. (2008) Vpr14-88-APOBEC3G fusion protein is efficiently incorporated into vif-positive HIV-1 particles and inhibits viral infection. PLoS One. 3(4), e1995.
Miyagi E., Welbourn S., Sukegawa S., Fabryova H., Kao S., Strebel K. (2020) Inhibition of vif-mediated degradation of APOBEC3G through competitive binding of core-binding factor beta. J. Virol. 94(7), e01708-19.
Wang H., Liu B., Liu X., Li Z., Yu X.F., Zhang W. (2014) Identification of HIV-1 vif regions required for CBF-β interaction and APOBEC3 suppression. PLoS One. 9(5), e95738.
Miyagi E., Kao S., Yedavalli V., Strebel K. (2014) CBFβ enhances de novo protein biosynthesis of its binding partners HIV-1 Vif and RUNX1 and potentiates the Vif-induced degradation of APOBEC3G. J. Virol. 88(9), 4839–4852.
Blyth K., Cameron E.R., Neil J.C. (2005) The RUNX genes: gain or loss of function in cancer. Nat. Rev. Cancer. 5(5), 376‒387.
Asou N. (2003) The role of a Runt domain transcription factor AML1/RUNX1 in leukemogenesis and its clinical implications. Crit. Rev. Oncol. Hematol. 45(2), 129‒150.
Iwatani Y., Chan D.S., Liu L., Yoshii H., Shibata J., Yamamoto N., Levin J.G., Gronenborn A.M., Sugiura W. (2009) HIV-1 vif-mediated ubiquitination/degradation of APOBEC3G involves four critical lysine residues in its C-terminal domain. Proc. Natl. Acad. Sci. USA. 106(46), 19539‒19544.
Turner T., Shao Q., Wang W., Wang Y., Wang C., Kinlock B., Liu B. (2016) Differential contributions of ubiquitin-modified APOBEC3G lysine residues to HIV-1 vif-induced degradation. J. Mol. Biol. 428(17), 3529‒3539.
Pan T., Song Z., Wu L., Liu G., Ma X., Peng Z., Zhou M., Liang L., Liu B., Liu J., Zhang J., Zhang X., Huang R., Zhao J., Li Y., Ling X., Luo Y., Tang X., Cai W., Deng K., Li L., Zhang H. (2019) USP49 potently stabilizes APOBEC3G protein by removing ubiquitin and inhibits HIV-1 replication. Elife. 8, e48318.
Li L., Liang D., Li J.Y., Zhao R.Y. (2008) APOBEC3G-UBA2 fusion as a potential strategy for stable expression of APOBEC3G and inhibition of HIV-1 replication. Retrovirology. 5, 72. 1.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


