Молекулярная биология, 2023, T. 57, № 2, стр. 316-329
Разработка и апробация ДНК-зондов для определения активности ключевых ферментов пути эксцизионной репарации оснований ДНК в клетках человека
И. В. Алексеева a, А. А. Кузнецова a, О. А. Кладова a, В. О. Шендер b, П. В. Шнайдер b, О. С. Федорова a, Н. А. Кузнецов a, c, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
b Федеральный научно-клинический центр физико-химической медицины Федерального медико-биологического агентства России
119435 Москва, Россия
c Новосибирский государственный университет
630090 Новосибирск, Россия
* E-mail: nikita.kuznetsov@niboch.nsc.ru
Поступила в редакцию 10.08.2022
После доработки 09.09.2022
Принята к публикации 09.09.2022
- EDN: EELFGZ
- DOI: 10.31857/S0026898423020027
Аннотация
Исследования механизмов сохранения генетической информации, проводимые с момента открытия систем репарации ДНК, обусловлены ключевой ролью повреждений ДНК в возникновении различных заболеваний, включая онкологические. До сих пор остается актуальным вопрос о связи между накоплением повреждений ДНК, сбоями в работе путей репарации ДНК и повышенным риском развития некоторых заболеваний. За последние несколько лет предприняты значительные усилия, направленные на разработку методов анализа активности ферментов эксцизионной репарации оснований ДНК в клетках человека. Нами разработаны флуоресцентные ДНК-зонды, позволяющие определять в клеточных экстрактах уровень активности ключевых ферментов эксцизионной репарации оснований ДНК, а именно ДНК-гликозилаз UNG2, SMUG1, MBD4, TDG, AAG, NEIL1, NTHL1 и OGG1 и АР-эндонуклеазы APE1. Чувствительность ДНК-зондов определяли на очищенных препаратах ферментов. Определение активности ферментов репарации в экстрактах клеточных линий опухолей яичников человека TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21 позволило выявить значительную вариабельность уровня активности ферментов в этих линиях клеток. Полученные результаты могут стать основой для создания тест-системы определения активности ключевых ферментов эксцизионной репарации оснований ДНК в организме человека.
ВВЕДЕНИЕ
Репарация ДНК – важный процесс поддержания целостности генома и, как следствие, генетической стабильности и жизнеспособности клеток. Эксцизионная репарация оснований ДНК (base excision repair, BER) – один из путей репарации, отвечающий за удаление из ДНК необъемных повреждений, возникающих в таких процессах, как дезаминирование, алкилирование, окисление азотистых оснований, гидролиз N-гликозидных и фосфодиэфирных связей [1]. В исследованиях процесса BER in vitro показано существование двух альтернативных путей: “короткозаплаточного”, в котором замене подвергается только один нуклеотид, и “длиннозаплаточного”, где происходит достройка двух и более нуклеотидов [2–5]. В обоих случаях ДНК-гликозилаза, имеющая специфичность к определенному типу повреждений, удаляет поврежденное азотистое основание, после чего AР-эндонуклеаза APE1 гидролизует ДНК с образованием 3'-концевой OH-группы [6]. Репарация отдельных окисленных азотистых оснований, инициированная бифункциональными гликозилазами (например, OGG1 и NEIL1-3), может протекать по независимому от АРЕ1 пути, в котором эти гликозилазы расщепляют AР-сайты по механизму β- или β/δ-элиминирования [7, 8]. ДНК-полимераза β (Polβ) катализирует удаление 5'-дезоксирибозофосфатного фрагмента (5'-dRP) и присоединяет к 3'-концевой OH-группе неповрежденный нуклеотид, который должен быть лигирован на последней стадии BER для восстановления целостности сахарофосфатного остова [6].
Впервые результаты исследований, в которых предполагалось, что репарация ДНК играет важную роль в предотвращении развития онкологических заболеваний, были опубликованы в конце 1960-х годов [9, 10]. Однако до сих пор актуальным остается изучение полноценного функционирования путей репарации ДНК в клетках человека, а также связи между сбоями в этих механизмах и повышенным риском развития некоторых заболеваний [11].
Необходимо отметить, что первые предложенные методы анализа активности ферментов репарации ДНК, основанные на радиоактивном мечении [12, 13], гель-электрофорезе [14], хроматографии [15, 16] или на захвате парамагнитными шариками со стрептавидином [17], были весьма трудоемкими и обладали невысокой чувствительностью [14]. Кроме того, существенным недостатком этих подходов является необходимость использования специализированных реагентов [12, 13], оборудования [15] или выполнения сложных процедур [17]. Поэтому в последние годы все чаще применяются флуоресцентные методы, простые в использовании и обладающие высокой чувствительностью [18–20]. Одно из существенных преимуществ анализа ферментов репарации ДНК с помощью флуоресцентных зондов – возможность проведения анализа непосредственно в биологических средах, включая лизаты и интактные клетки [21–23]. Еще одним положительным фактором использования флуоресцентных зондов является их достаточно легкая адаптация к проведению параллельного анализа активности нескольких ферментов [24, 25]. При этом флуоресцентные зонды, способные проникнуть в клетку, можно использовать и для прямой регистрации активности целевых ферментов непосредственно в клетках [24, 26]. В последние годы были предприняты значительные усилия, направленные на разработку методов анализа активности ферментов репарации ДНК [27] в различных клеточных линиях и тканях с помощью флуориметрии [28, 29], флуоресцентной микроскопии [25] и проточной цитометрии [30].
Нами разработаны флуоресцентные ДНК-зонды, позволяющие определять в клеточных экстрактах уровень активности ключевых ферментов эксцизионной репарации ДНК, а именно ДНК-гликозилаз UNG2, SMUG1, MBD4, TDG, AAG, NEIL1, NTHL1 и OGG1, и АР-эндонуклеазы APE1. Чувствительность ДНК-зондов определяли с использованием очищенных препаратов ферментов. Определение активности ферментов репарации в клеточных экстрактах линий опухолей яичников человека TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21 позволило апробировать ДНК-зонды и выявило значительную вариабельность уровня активности ферментов в этих линиях. Результаты нашей работы могут стать основой для создания тест-системы анализа функционирования системы BER в клетках человека и проведения персонализированной терапии онкологических заболеваний.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Структура ДНК-зондов. Олигонуклеотидные ДНК-зонды (табл. 1) синтезированы на автоматическом ДНК/РНК-синтезаторе ASM-800 (“Биоссет”, Новосибирск, Россия) с использованием коммерческих амидофосфитов 2′-дезоксирибонуклеозидов и CPG-носителей (“GlenResearch”, США). Синтезированные ДНК-зонды очищали с помощью ВЭЖХ на ионообменной колонке (PRP-X500 Hamilton Company (12–30 мкм) 3.9 × × 300 мм) и последующей обращенно-фазовой хроматографии (Bondapak C18 (15–20 мкм) 3.9 × × 300 мм, “Waters”, Ирландия). Концентрацию олигонуклеотидов в исходном растворе определяли по закону Бугера–Ламберта–Бера, используя значение коэффициента молярного поглощения олигонуклеотидов при длине волны 260 нм, рассчитанное по данным [31].
Таблица 1.
Структура и специфичность использованных ДНК-зондов
| Фермент | Структура поврежденного нуклеотида | ДНК-зонд |
|---|---|---|
| APE1 | F | 5'-FAM-GCTCA(F)GTACAGAGCTGTTTTTCAGCTCTGTACGTGAGCps-BHQ1-3' |
| UNG2, SMUG1, MBD4, TDG | U | 5'-FAM-GCTCA(U)GTACAGAGCTGTTTTTCAGCTCTGTACGTGAGCps-BHQ1-3' |
| AAG | εA | 5'-FAM-GCTCA(ɛA)GTACAGAGCTGTTTTTCAGCTCTGTACGTGAGCps-BHQ1-3' |
| NEIL1, NTHL1 | Tg | 5′-FAM-GCTCA(Tg)GTACAGAGCTGTTTTTCAGCTCTGTACGTGAGCps-BHQ1-3′ |
| OGG1 | oxoG | 5'-FAM-GCTCA(oxoG)GTACAGAGCTGTTTTTCAGCTCTGTACGTGAGCps-BHQ1-3′ |
| Неспецифическое расщепление | C | 5'-FAM-GCTCA(C)GTACAGAGCTGTTTTTCAGCTCTGTACGTGAGCps-BHQ1-3′ |
Определение чувствительности ДНК-зондов на очищенных препаратах ферментов. Калибровочные кривые, отражающие зависимость начальной скорости расщепления ДНК-зонда от концентрации целевого фермента, получали для каждого из представленных зондов (кроме зонда, содержащего неповрежденное основание) с использованием рекомбинантных ферментов. Очищенные препараты ферментов получены по ранее опубликованным методикам: АР-эндонуклеаза APE1 согласно [32, 33], 8-оксогуанин-ДНК-гликозилаза OGG1 [34, 35], урацил-ДНК-гликозилаза SMUG1 [36], алкиладенин-ДНК-гликозилаза AAG [37] и эндонуклеаза VIII NEIL1 [38]. Кинетические кривые получали при взаимодействии 1 × 10–6 М ДНК-зонда и 1 × 10–6, 5 × 10–7, 1 × 10–7, 5 × 10–8, 1 × 10–8, 1 × 10–9 М целевого фермента, специфичного к данному зонду. В случае калибровочных кривых для монофункциональных ДНК-гликозилаз SMUG1 и AAG в реакционной смеси присутствовала АР-эндонуклеаза APE1 в концентрации 1 × 10–6 М, гидролизующая АР-сайты, образованные действием монофункциональных ДНК-гликозилаз. Кинетические кривые изменения интенсивности флуоресценции FAM регистрировали при температуре 37°C на спектрофлуориметре Infinite 200 Pro (“Tecan”, Швейцария). Каждая кинетическая кривая представляет собой результат усреднения минимум трех экспериментальных кривых. Эксперименты проводили в 50 мМ Tрис-HCl-буфере pH 7.5, содержащем 50 мМ KCl, 1 мМ EDТА, 5 мМ MgCl2, 1 мМ ДТТ, 7% глицерина. Возбуждение флуоресценции проводили на длине волны 485 нм, изменение интенсивности флуоресценции ДНК-зонда регистрировали на длине волны 520 нм в интервале времени 15–3600 с.
Начальную скорость расщепления ДНК-зондов при заданной концентрации фермента рассчитывали, используя начальные линейные участки кинетических кривых. Зависимость начальной скорости от концентрации ферментов имела линейный вид, что позволяет использовать полученные зависимости в качестве калибровочных кривых для оценки концентрации целевых ферментов в клеточном экстракте.
Клеточные линии. Для анализа активности ферментов репарации ДНК с помощью ДНК-зондов использовали линии клеток рака яичника человека TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21. Клетки культивировали в среде DMEM (“Gibco”, “ThermoFisher Scienific”, США) с 10%‑ной фетальной сывороткой крупного рогатого скота (“Gibco”, “ThermoFisher”), 100 мкг/мл стрептомицина и 100 ед./мл пенициллина в атмосфере 5% CO2 при 37°C. Клетки механически снимали с поверхности культурального флакона, осаждали центрифугированием (1000 об./мин, 2 мин), ресуспендировали в растворе PBS и центрифугировали повторно (1000 об./мин, 2 мин). Данную процедуру повторяли дважды.
Приготовление клеточного экстракта. Лизис клеток проводили в буфере: 10 мМ Tрис-HCl pH 7.5, 1 мМ MgCl2, 1 мМ EDTA, 0.5% CHAPS, 10% глицерин, 0.1 мМ PMSF, 0.5 мМ β-меркаптоэтанол. К клеточному осадку добавляли 150 мкл лизирующего буфера, выдерживали на льду в течение 30 мин., затем центрифугировали (14 500 об./мин, 10 мин). Общую концентрацию белка в полученном экстракте измеряли по методу Брэдфорда (концентрация белков, как правило, составляла 1–1.5 мкг/мкл). Клеточный экстракт готовили непосредственно перед проведением флуоресцентного анализа активности ферментов.
Определение оптимального общего количества белка в клеточном экстракте. Клетки линии TOV112 лизировали в буфере (10 мМ Tрис-HCl pH 7.5, 1 мМ MgCl2, 1 мМ EDTA, 0.5% CHAPS, 10% глицерин, 0.1 мМ PMSF, 0.5 мМ β-меркаптоэтанол). Общая концентрация белка, измеренная по методу Брэдфорда, составила 1.1 мкг/мкл. Регистрировали изменения интенсивности флуоресценции реакционных смесей, содержащих 1 × 10–6 М ДНК-зонд и клеточный экстракт в концентрации 50, 25, 10, 5 мкг/мкл по общему белку. Спектры флуоресценции регистрировали в 50 мМ Tрис-HCl-буфере pH 7.5, содержащем 50 мМ KCl, 1 мМ EDТА, 5 мМ MgCl2, 1 мМ DТТ, 7% глицерина при 37°C отдельно для каждого типа ДНК-зонда. Каждая кинетическая кривая, представленная на рисунках, получена усреднением не менее трех экспериментальных кривых.
Определение активности ферментов BER в клеточном экстракте. Пробы готовили в 100 мкл буферного раствора, содержащего 50 мМ Tрис-HCl pH 7.5, 50 мМ KCl, 1 мМ EDТА, 5 мМ MgCl2, 1 мМ DТТ, 7% глицерина и клеточный экстракт в концентрации 20 мкг/мкл по общему белку. ДНК-зонд, содержащий поврежденный нуклеотид определенного типа (табл. 1), добавляли к клеточному экстракту до конечной концентрации 1 × 10-6 М. Перемешивание (5 с, амплитуда кругового перемешивания 1 мм) и регистрацию FRET-сигнала проводили, используя спектрофлуориметр Infinite 200 Pro (“Tecan”) при 37°С. Длина волны возбуждения флуоресценции составляла λex = 485 нм, эмиссию регистрировали при λem = 520 нм. Максимальное время реакции 3600 с. Каждый эксперимент повторяли трижды.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Ранее мы предложили подход к определению активности ряда ферментов BER c использованием флуоресцентно меченных ДНК-дуплексов, содержащих поврежденный нуклеотид [39]. В качестве поврежденных нуклеотидов использовали 7,8-дигидро-8-оксогуанозин (oxoG), 5,6-дигидроуридин (DHU), уридин (U), 1,N6-этеноаденозин (εA) и апуриновый/апиримидиновый сайт (АР-сайт). Набор ДНК-зондов, содержащих эти повреждения, позволил охарактеризовать активность нескольких ферментов репарации человека: AP-эндонуклеазы APE1; 8-оксогуанин-ДНК-гликозилазы OGG1; алкиладенин-ДНК-гликозилазы AAG; ДНК-гликозилаз UNG2, SMUG1, MBD4 и TDG, специфически узнающих урацил в ДНК; ДНК-гликозилаз NEIL1 и NTHL1, узнающих 5,6‑дигидроуридин. Активность AP-эндонуклеазы APE1 определяли с использованием ДНК-зонда, содержащего химически стабильный аналог AP-сайта – остаток (2R,3S)-2-(гидроксиметил)-3-гидрокситетрагидрофурана (F-сайт).
Анализ активности ферментов репарации ДНК с помощью дуплексных ДНК-зондов в линиях опухолевых клеток человека различного гистологического происхождения, а именно A549, HeLa, MCF7, WT-7, HEK293T и HKC8, показал, что дуплексные ДНК-зонды, содержащие oxoG и εА, расщепляемые ферментами OGG1 и AAG, соответственно, не обладают чувствительностью, достаточной для регистрации активности [39]. Полученные данные свидетельствовали о том, что дуплексные ДНК-зонды не могут использоваться для определения активности в клеточном экстракте, полученном менее чем из 106 клеток. Кроме того, время анализа было ограничено 2000 с для предотвращения неспецифической деградации ДНК-зонда клеточными нуклеазами.
В связи с выявленными недостатками мы провели оптимизацию структуры ДНК-зондов, направленную на улучшение их устойчивости к неспецифической деградации в клеточных экстрактах. ДНК-зонды, используемые в данной работе (рис. 1), представляли собой самокомплементарные олигодезоксирибонуклеотиды, несущие FRET-пару FAM/BHQ1 на концах цепи, а также содержащие повреждение, специфичное для определенного фермента или группы ферментов (табл. 1). В качестве поврежденных нуклеотидов использовали 7,8-дигидро-8-оксогуанозин (oxoG), тимидингликоль (Tg), уридин (U), 1,N6-этеноаденозин (εA) и F-сайт. Для предотвращения 3'–5'-экзонуклеазной деградации ДНК-зондов в клеточном экстракте 3'-концевой межнуклеотидный фосфатный остаток заменили тиофосфатной группой (ps).
Рис. 1.
Схематичное изображение ДНК-зонда, используемого в работе, и пути генерирования FRET-сигнала в тест-системе анализа активности ферментов пути BER в клеточных экстрактах. Х – повреждение, специфичное для анализируемого фермента.
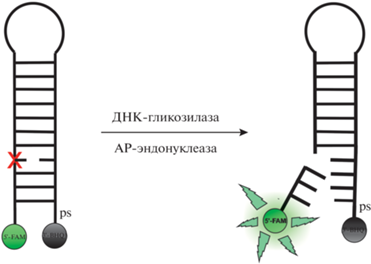
Специфическое узнавание поврежденного нуклеотида и его удаление ДНК-гликозилазой с последующим расщеплением сахарофосфатного остова сопровождается значительным ростом сигнала флуоресценции FAM. Необходимо отметить, что для расщепления АР-сайтов, образующихся под действием монофункциональных ДНК-гликозилаз, в реакционной смеси должна присутствовать АР-эндонуклеаза. Кроме того, известно, что АР-эндонуклеаза АPE1 стимулирует активность ДНК-гликозилаз человека [40, 41], поэтому при анализе активности всех ДНК-гликозилаз в реакционную смесь добавляли APE1 в одинаковой концентрации 1.0 мкМ.
На рис. 2 представлены кинетические кривые, характеризующие расщепление ДНК-зондов ДНК-гликозилазами OGG1, NEIL1, AAG, SMUG1 и АР-эндонуклеазой APE1. Концентрация ДНК-зонда во всех экспериментах составляла 1.0 мкМ. Кинетические кривые, характеризующие активность АР-эндонуклеазы человека APE1, получали с использованием очищенного препарата фермента с концентрацией 5 × 10–10–1 × 10–6 М. В случае кривых для ДНК-гликозилаз использовали также очищенные препараты фермента с концентрацией 1 × 10–9–1 × 10–6 М, но в реакционную смесь добавляли АР-эндонуклеазу человека APE1 в концентрации 1.0 мкМ.
Рис. 2.
Кинетические кривые, характеризующие расщепление ДНК-зондов, содержащих F (а), U (б), εA (в), Tg (г) и oxoG (д), при взаимодействии с очищенными препаратами ферментов APE1, SMUG1, AAG, NEIL1 и OGG1 соответственно.
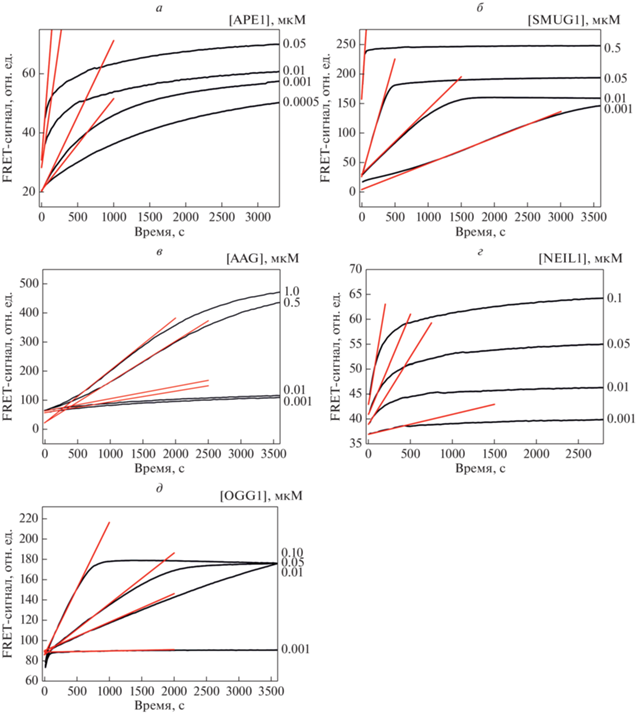
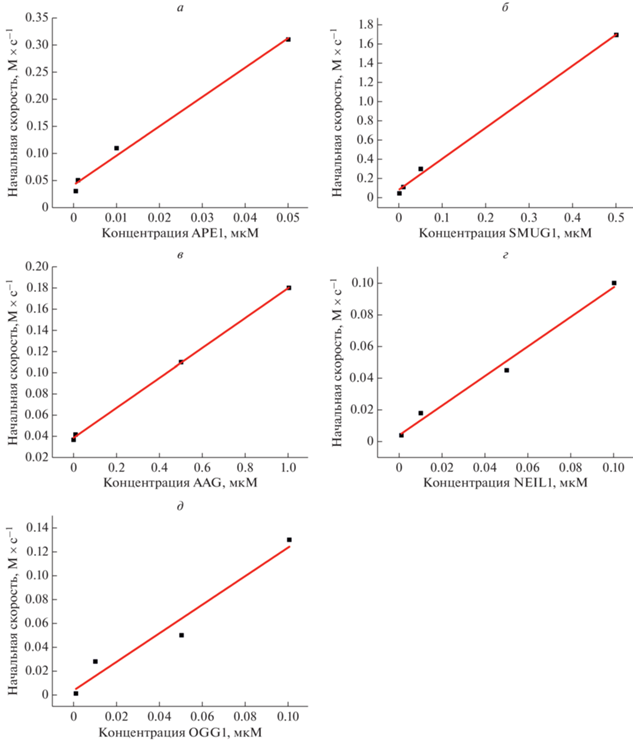
Начальную скорость расщепления ДНК-зондов оценивали путем линеаризации (или линейной аппроксимации) начального участка кинетических кривых (рис. 3). Зависимость начальной скорости от концентрации фермента имела линейный вид и позволила определить концентрацию целевого фермента в клеточном экстракте.
Рис. 3.
Зависимость начальной скорости накопления продукта от концентрации APE1 (а), SMUG1 (б), AAG (в), NEIL1 (г) и OGG1 (д).
Чтобы определить количество общего белка, оптимальное для анализа, сравнили относительную активность ферментов в экстрактах клеток линии TOV112, содержащих 5, 10, 25 и 50 мкг общего белка (рис. 4). Для всех типов ДНК-зондов получены кинетические кривые, свидетельствующие об увеличении FRET-сигнала после добавления клеточного экстракта. Начальные участки кинетических кривых аппроксимировали линейным уравнением, что позволило определить начальную скорость расщепления ДНК-зонда. Зависимость начальной скорости расщепления ДНК-зондов от концентрации общего белка в экстракте имела линейный вид в случае ДНК-зондов, содержащих F, U, Tg и oxoG (рис. 5). Однако в случае εA-зонда начальная скорость роста FRET-сигнала не коррелировала с концентрацией белков в клеточном экстракте. Следует отметить, что удаление εA, в отличие от F, U, Tg и oxoG, из ДНК может проходить по двум независимым механизмам: BER с помощью фермента AAG [37, 42, 43], приводящего к удалению поврежденного основания и генерации АР-сайта, и прямой репарации алкилированных нуклеотидов с помощью ДНК-диоксигеназы ABH2 [44, 45] за счет окисления алкильного заместителя и его последующей деградации, приводящей к восстановлению структуры неповрежденного аденозина в составе ДНК. Можно предположить, что совместное узнавание εA-зонда ферментами, входящими в независимые пути удаления повреждения из ДНК, приводит к усложнению интерпретации полученных данных.
Рис. 4.
Определение оптимального для анализа количества общего белка в клеточном экстракте на примере линии клеток TOV112. Кинетические кривые, характеризующие расщепление ДНК-зондов, содержащих F (а), U (б), εA (в), Tg (г) и oxoG (д), в клеточных экстрактах, содержащих 5, 10, 25 и 50 мкг общего белка.
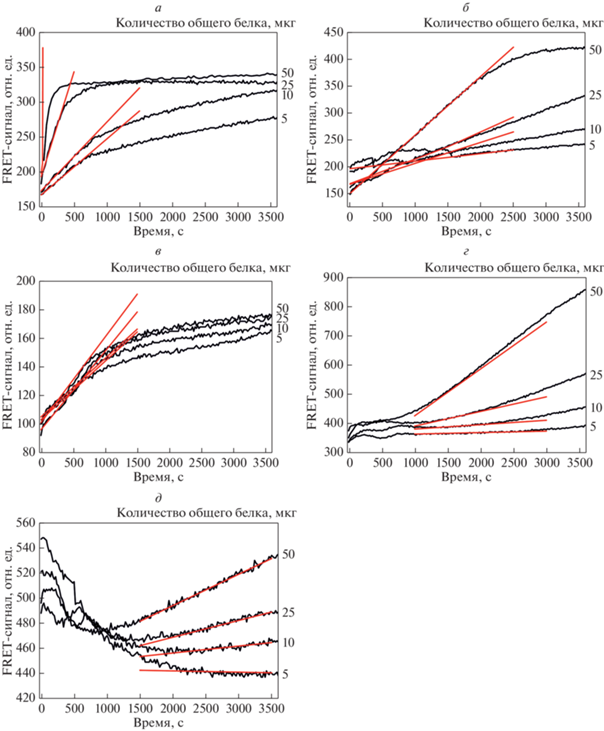
Рис. 5.
Зависимость начальной скорости расщепления ДНК-зондов, содержащих F (a), U (б), εA (в), Tg (г) и oxoG (д), при взаимодействии с клеточным экстрактом линии клеток TOV112 при различных концентрациях общего белка (5, 10, 25 и 50 мкг/мкл).
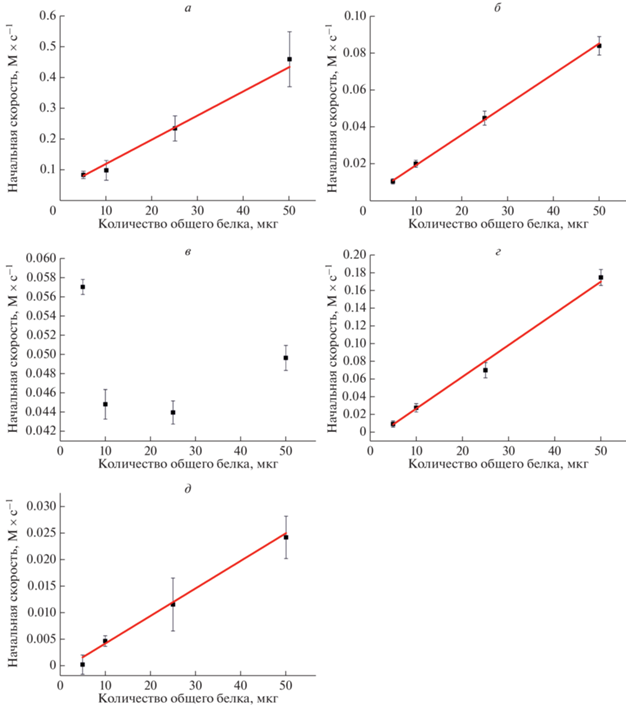
На основании полученных данных во всех дальнейших экспериментах по определению активности целевых ферментов использовали 25 мкг общего белка для ДНК-зонда каждого типа. Таким образом, для анализа активности с использованием пяти ДНК-зондов суммарно требуется не менее 125 мкг общего белка. Однако необходимо отметить, что 125 мкг белка можно получить при лизисе ~100 000 клеток (в зависимости от линии клеток), что свидетельствует о перспективности данного метода для определения активности ферментов репарации в относительно небольшом числе клеток, например, полученных из крови человека с целью определения репарационного статуса организма.
На следующем этапе с использованием линии SCOV3 сравнили активность ферментов пути BER в полном клеточном экстракте и в экстракте предварительно выделенной ядерной фракции. Интересно отметить, что кинетические кривые, характеризующие расщепление всех типов ДНК-зондов белками общего и ядерного экстрактов, имели сходный вид, что свидетельствует о возможности проводить анализ ферментов, используя общий клеточный экстракт (рис. 6).
Рис. 6.
Кинетические кривые, характеризующие расщепление ДНК-зондов, содержащих F (а), U (б), εA (в), Tg (г) и oxoG (д), при взаимодействии с экстрактом клеточной линии SCOV3 (черный цвет – ядерная фракция, красный – общий экстракт).

Затем на примере нескольких линий клеток опухоли яичника человека (TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21) провели сравнительный анализ активности тестируемых ферментов репарации в общих клеточных экстрактах. Для каждой клеточной линии получили кинетические кривые, характеризующие расщепление ДНК-зондов (рис. 7), и определили начальную скорость расщепления. На основании начальных скоростей расщепления ДНК-зондов и калибровочных кривых, полученных с использованием очищенных препаратов ферментов, оценили концентрации целевых ферментов в экстрактах (табл. 2, рис. 8).
Рис. 7.
Кинетические кривые, характеризующие расщепление ДНК-зондов, содержащих F (а), U (б), εA (в), Tg (г) и oxoG (д), при взаимодействии с экстрактами клеточных линий TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21.
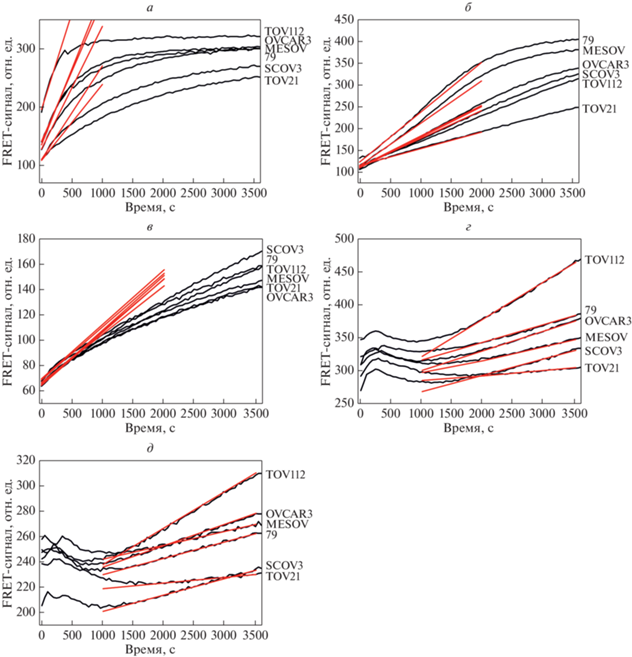
Таблица 2.
Концентрации целевых ферментов в клеточных экстрактах линий рака яичников человека TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21
| Линия клеток | Концентрация ферментов BER, нМ | ||||
|---|---|---|---|---|---|
| APE1 | UDG | AAG | NEIL1/NTH1 | OGG1 | |
| TOV112 | 47 ± 5 | 5.1 ± 0.9 | 10.3 ± 0.7 | 42 ± 3 | 12.6 ± 1.5 |
| TOV21 | 16 ± 3 | 2.2 ± 0.5 | 7.1 ± 0.9 | 3.3 ± 0.2 | 2.2 ± 0.5 |
| 79 | 26 ± 4 | 14.5 ± 4.3 | 7.7 ± 1.2 | 19 ± 5 | 5.8 ± 1.2 |
| SCOV3 | 20 ± 4 | 3.7 ± 0.9 | 8.0 ± 0.5 | 15 ± 3 | 6.0 ± 0.8 |
| MESOV | 33 ± 7 | 5.5 ± 1.4 | 7.3 ± 0.8 | 12 ± 5 | 4.8 ± 0.9 |
| OVCAR3 | 31 ± 8 | 4.1 ± 0.8 | 6.8 ± 0.6 | 21 ± 5 | 7.4 ± 1.7 |
Рис. 8.
Концентрация ферментов репарации в экстракте клеточных линий рака яичника человека TOV112, 79, OVCAR3, MESOV, SCOV3 и TOV21.
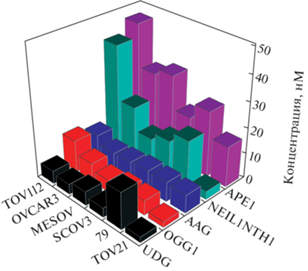
Сравнение полученных данных выявило значительную вариабельность оценочной концентрации ферментов репарации в различных клеточных линиях. Так, например, концентрация APE1 в линии TOV112 была более чем в 2 раза выше, чем в линиях TOV21 и SCOV3. Необходимо также отметить существенно более высокую оценочную концентрацию ферментов, отвечающих за удаление окисленных пиримидинов (Tg-зонд), в клетках линии TOV112. При этом эффективность удаления алкилированных нуклеотидов на примере εA-зонда была примерно одинаковой во всех использованных клеточных линиях. Однако, как отмечено ранее, эффективность репарации алкилированных азотистых оснований, включая ɛA, может проходить по двум независимым путям, что осложняет интерпретацию полученных данных. Обнаружено, что клетки линии TOV21 обладают пониженной репарационной активностью на ДНК-зондах всех типов.
ЗАКЛЮЧЕНИЕ
Несмотря на то, что понимание механизмов разных путей репарации ДНК значительно расширилось за последнее время, многие инструменты, позволяющие изучать функционирование отдельных участников этих процессов, все еще нуждаются в улучшении. Нами разработаны флуоресцентные ДНК-зонды, с помощью которых можно определять в клеточных экстрактах уровень активности ключевых ферментов BER – ДНК-гликозилаз и АР-эндонуклеазы АРЕ1. Определение активности ферментов репарации в клеточных экстрактах нескольких линий рака яичников человека позволило апробировать ДНК-зонды и выявило значительную вариабельность уровня активности ферментов в этих линиях клеток.
Полученные данные показывают перспективность предложенного метода, учитывая простоту применения и анализа полученных данных. Результаты нашей работы могут стать основой для создания тест-системы определения активности ключевых ферментов BER в живых организмах. Учитывая множество преимуществ флуоресцентных методов, существует большой потенциал для расширения этой области, предоставляющей инструменты, столь необходимые для определения индивидуального репарационного статуса организма пациента. Будущие персонализированные методы лечения, связанные с применением ДНК-модифицирующих химических препаратов, несомненно, должны быть взаимосвязаны с эффективностью работы ферментов, отвечающих за удаление ДНК-повреждений, вызываемых конкретными препаратами, у каждого конкретного пациента.
Работа выполнена при поддержке Российского научного фонда (грант № 21-14-00018) и при частичной поддержке бюджетного финансирования № 121031300041-4.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Krokan H.E., Bjørås M. (2013) Base excision repair. Cold Spring Harb. Perspect. Biol. 5, a012583.
Dianov G., Price A., Lindahl T. (1992) Generation of single-nucleotide repair patches following excision of uracil residues from DNA. Mol. Cell. Biol. 12, 1605–1612.
Matsumoto Y., Bogenhagen D.F. (1994) Proliferating cell nuclear antigen-dependent abasic site repair in Xenopus laevis oocytes: an alternative pathway of base excision DNA repair. Mol. Cell. Biol. 14, 6187–6197.
Frosina G., Fortini P., Rossi O., Carrozzino F., Raspaglio G., Cox L.S., Lane D.P., Abbondandolo A., Dogliotti E. (1996) Two pathways for base excision repair in mammalian cells. J. Biol. Chem. 271, 9573–9578.
Klungland A., Lindahl T. (1997) Second pathway for completion of human DNA base excision-repair: reconstitution with purified proteins and requirement for DNase IV (FEN1). EMBO J. 16, 3341–3348.
Kim Y.-J., Wilson D.M. III (2012) Overview of base excision repair biochemistry. Curr. Mol. Pharmacol. 5, 3–13.
Wiederhold L., Leppard J.B., Kedar P., Karimi-Busheri F., Rasouli-Nia A., Weinfeld M., Tomkinson A.E., Izumi T., Prasad R., Wilson S.H. (2004) AP endonuclease-independent DNA base excision repair in human cells. Mol. Cell. 15, 209–220.
Das A., Wiederhold L., Leppard J.B., Kedar P., Prasad R., Wang H., Boldogh I., Karimi-Busheri F., Weinfeld M., Tomkinson A.E., Wilson S.H., Mitra S. (2006) NEIL2-initiated, APE-independent repair of oxidized bases in DNA: evidence for a repair complex in human cells. DNA Repair (Amst). 5, 1439–1448.
Cleaver J.E. (1968) Defective repair replication of DNA in Xeroderma pigmentosum. DNA Repair (Amst). 3, 183–187.
Setlow R.B., Regan J.D., German J., Carrier W.L. (1969) Evidence that Xeroderma pigmentosum cells do not perform the first step in the repair of ultraviolet damage to their DNA. DNA Repair (Amst). 3, 188–195.
Helleday T., Eshtad S., Nik-Zainal S. (2014) Mechanisms underlying mutational signatures in human cancers. Nat. Rev. Genet. 15, 585–598.
Grin I.R., Konorovsky P.G., Nevinsky G.A., Zharkov D.O. (2009) Heavy metal ions affect the activity of DNA glycosylases of the Fpg family. Biochemistry. 74, 1253–1259.
Kreklau E.L., Limp-Foster M., Liu N., Xu Y., Kelley M.R., Erickson L.C. (2001) A novel fluorometric oligonucleotide assay to measure O 6-methylguanine DNA methyltransferase, methylpurine DNA glycosylase, 8-oxoguanine DNA glycosylase and abasic endonuclease activities: DNA repair status in human breast carcinoma cells overexpressing. Nucl. Acids Res. 29, 2558–2566.
Dianov G.L. (2003) Monitoring base excision repair by in vitro assays. Toxicology. 193, 35–41.
Weiss J.M., Goode E.L., Ladiges W.C., Ulrich C.M. (2005) Polymorphic variation in hOgg1 and risk of cancer: a review of the functional and epidemiologic literature. Mol. Carcinog. 42, 127–141.
Lee A.J., Hodges N.J., Chipman J.K. (2005) Interindividual variability in response to sodium dichromate-induced oxidative DNA damage: role of the Ser 326 Cys polymorphism in the DNA-repair protein of 8-oxo-7,8-dihydro-2v-deoxyguanosine DNA glycosylase 1. Cancer Epidemiol. Biomarkers Prev. 14, 497–505.
Xia L., O′Connor T.R. (2001) DNA glycosylase activity assay based on streptavidin paramagnetic bead substrate capture. Anal. Biochem. 298, 322–326.
Liu B., Yang X., Wang K., Tan W., Li H., Tang H. (2007) Real-time monitoring of uracil removal by uracil-DNA glycosylase using fluorescent resonance energy transfer probes. Anal. Biochem. 366, 237–243.
Wang X., Hou T., Lu T., Li F. (2014) Autonomous exonuclease iii-assisted isothermal cycling signal amplification: a facile and highly sensitive fluorescence DNA glycosylase activity assay. Anal. Chem. 86, 9626–9631.
Chen C., Zhou D., Tang H., Liang M., Jiang J. (2013) A sensitive, homogeneous fluorescence assay for detection of thymine DNA glycosylase activity based on exonuclease-mediated amplification. Chem. Commun. 49, 5874.
Cao X., Sun Y., LuP., Zhao M. (2020) Fluorescence imaging of intracellular nucleases – a review. Anal. Chim. Acta. 1137, 225–237.
Mirbahai L., Kershaw R.M., Green R.M., Hayden R.E., Meldrum R.A., Hodges N.J. (2010) Use of a molecular beacon to track the activity of base excision repair protein OGG1 in live cells. DNA Repair (Amst). 9, 144–152.
Belhadj S., Rentsch A., Schwede F., Paquet-Durand F. (2021) Fluorescent detection of PARP activity in unfixed tissue. PLoS One. 16, 1–13.
Torchinsky D., Michaeli Y., Gassman N.R., Ebenstein Y. (2019) Simultaneous detection of multiple DNA damage types by multi-colour fluorescent labelling. Chem. Commun. 55, 11414–11417.
Hu J., Liu M.-H., Li Y., Tang B., Zhang C.-Y. (2018) Simultaneous sensitive detection of multiple DNA glycosylases from lung cancer cells at the single-molecule level. Chem. Sci. 9, 712–720.
Maksimenko A., Ishchenko A.A., Sanz G., Laval J., Elder R.H., Saparbaev M.K. (2004) A molecular beacon assay for measuring base excision repair activities. Biochem. Biophys. Res. Commun. 319, 240–246.
Pearl L.H., Schierz A.C., Ward S.E., Al-Lazikani B., Pearl F.M.G. (2015) Therapeutic opportunities within the DNA damage response. Nat. Rev. Cancer. 15, 166–180.
Zhang Y., Li C.-C., Zhang X., Xu,Q., Zhang C.-Y. (2020) Development of oxidation damage base-based fluorescent probe for direct detection of DNA methylation. Anal. Chem. 92, 10223–10227.
Liu G., He W., Liu C. (2019) Sensitive detection of uracil-DNA glycosylase (UDG) activity based on terminal deoxynucleotidyl transferase-assisted formation of fluorescent copper nanoclusters (CuNCs). Talanta. 195, 320–326.
Suggitt M., Fearnley J., Swaine D., Volpato M., Phillips R., Bibby M., Loadman P., Anderson D., Anderson D. (2003) Comet assay and flow cytometry analysis of DNA repair in normal and cancer cells treated with known mutagens with different mechanisms of action. Teratog. Carcinog. Mutagen. 2, 13–29.
Fasman G.D. (1975) Handbook of Biochemistry and Molecular Biology. 3rd ed. Cleveland: CRC Press.
Мирошникова А.Д., Кузнецова А.А., Кузнецов Н.А., Федорова О.С. (2016) Термодинамика конформационных переходов АП-эндонуклеазы человека APE1 при взаимодействии с ДНК. Acta Naturae. 8, 103–110.
Kuznetsova A.A., Fedorova O.S., Kuznetsov N.A. (2018) Kinetic features of 3’-5’ exonuclease activity of human AP-endonuclease APE1. Molecules. 23, 2101.
Kuznetsova A.A., Kuznetsov N.A., Ishchenko A.A., Saparbaev M.K., Fedorova O.S. (2014) Step-by-step mechanism of DNA damage recognition by human 8‑oxoguanine DNA glycosylase. Biochim. Biophys. A-cta. 1840, 387–395.
Кузнецов Н.А., Коваль В.В., Федорова О.С. (2011) Механизмы ферментативного катализа и узнавания поврежденных участков ДНК 8-оксогуанан-ДНК-гликозилазой человека hOGG1. Биохимия. 76, 118–130.
Kuznetsova A.A., Iakovlev D.A., Misovets I.V., Ishchenko A.A., Saparbaev M.K., Kuznetsov N.A., Fedorova O.S. (2017) Pre-steady-state kinetic analysis of damage recognition by human single-strand selective monofunctional uracil-DNA glycosylase SMUG1. Mol. Biosyst. 13, 2638–2649.
Kuznetsov N.A., Kiryutin A.S., Kuznetsova A.A., Panov M.S., Barsukova M.O., Yurkovskaya A.V., Fedorova O.S. (2017) The formation of catalytically competent enzyme-substrate complex is not a bottleneck in lesion excision by human alkyladenine DNA glycosylase. J. Biomol. Struct. Dyn. 35, 950–967.
Kladova O.A., Grin I.R., Fedorova O.S., Kuznetsov N.A., Zharkov D.O. (2019) Conformational dynamics of damage processing by human DNA glycosylase NEIL1. J. Mol. Biol. 431, 1098–1112.
Кладова О.А., Яковлев Д.А., Гройсман Р., Ищенко А.А., Сапарбаев М.К., Федорова О.С., Кузнецов Н.А. (2020) Определение сравнительной активности ключевых ферментов эксцизионной репарации оснований в клеточных экстрактах. Биохимия. 8, 480–489.
Kladova O.A., Alekseeva I.V., Saparbaev M., Fedorova O.S., Kuznetsov N.A. (2020) Modulation of the apurinic/apyrimidinic endonuclease activity of human APE1 and of its natural polymorphic variants by base excision repair proteins. Int. J. Mol. Sci. 21, 7174.
Kladova O.A., Bazlekowa-Karaban M., Baconnais S., Piétrement O., Ishchenko A.A., Matkarimov B.T., Iakovlev D.A., Vasenko A., Fedorova O.S., Le Cam E. (2018) The role of the N-terminal domain of human apurinic/apyrimidinic endonuclease 1, APE1, in DNA glycosylase stimulation. DNA Repair (Amst). 64, 10–25.
Saparbaev M., Langouet S., Privezentzev C.V., Guengerich F.P., Cai H., Elder R.H., Laval J. (2002) 1,N(2)-ethenoguanine, a mutagenic DNA adduct, is a primary substrate of Escherichia coli mismatch-specific uracil-DNA glycosylase and human alkylpurine-DNA-N-glycosylase. J. Biol. Chem. 277, 26987–26993.
O’Brien P.J., Ellenberger T. (2004) Dissecting the broad substrate specificity of human 3-methyladenine-DNA glycosylase. J. Biol. Chem. 279, 9750–9757.
Ringvoll J., Moen M.N., Nordstrand L.M., Meira L.B., Pang B., Bekkelund A., Dedon P.C., Bjelland S., Samson L.D., Falnes P.Ø. (2008) AlkB homologue 2-mediated repair of ethenoadenine lesions in mammalian DNA. Cancer Res. 68, 4142–4149.
Kuznetsov N.A., Kanazhevskaya L.Y., Fedorova O.S. (2021) DNA demethylation in the processes of repair and epigenetic regulation performed by 2-ketoglutarate-dependent DNA dioxygenases. Int. J. Mol. Sci. 22, 10540.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


