Молекулярная биология, 2023, T. 57, № 2, стр. 185-196
Сравнительный анализ ДНК-полимераз семейства А как инструмент поиска ферментов с новыми свойствами
А. А. Булыгин a, b, А. А. Кузнецова a, О. С. Федорова a, Н. А. Кузнецов a, b, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
b Новосибирский национальный исследовательский государственный университет
630090 Новосибирск, Россия
* E-mail: nikita.kuznetsov@niboch.nsc.ru
Поступила в редакцию 21.07.2022
После доработки 07.09.2022
Принята к публикации 07.09.2022
- EDN: EGAVKM
- DOI: 10.31857/S0026898423020040
Аннотация
ДНК-полимеразы катализируют синтез ДНК при репликации, репарации и рекомбинации ДНК. Ряд ДНК-полимераз, например полимераза Taq из Thermus aquaticus, нашли применение в различных приложениях молекулярной биологии и биотехнологии, в частности, в качестве инструментов амплификации ДНК. Однако эффективность этих ферментов зависит от таких факторов, как происхождение ДНК, состав праймера, длина и GC-содержание матрицы, способность формировать стабильные вторичные структуры. Подобные ограничения делают актуальным поиск новых ферментов с улучшенными свойствами. В нашем обзоре рассмотрены основные структурные и молекулярно-кинетические особенности функционирования ДНК-полимераз, принадлежащих к структурному семейству А, включая Taq-полимеразу. Филогенетический анализ этих ферментов позволил установить высококонсервативный консенсусный “слепок”, содержащий 62 аминокислотных остатка, распределенных по структуре фермента. Сравнительный анализ этих аминокислотных остатков у малоизученных ДНК-полимераз позволил выявить семь ферментов, потенциально обладающих свойствами, необходимыми для их использования в амплификации ДНК.
ВВЕДЕНИЕ
ДНК-полимеразы играют ключевую роль в сохранении стабильности генома. Ферменты этого класса катализируют полимеризацию 2'-дезоксирибонуклеотидмонофосфатов вдоль цепи ДНК, которую фермент “читает” и использует в качестве матрицы. Тип нового нуклеотида определяется по принципу комплементарности с нуклеотидом, расположенным в матрице, с которой ведется считывание. Таким образом, вновь синтезируемая молекула полностью комплементарна матричной цепи и идентична одной из цепей двойной спирали ДНК.
ДНК-полимеразы играют важную роль не только в ряде процессов, необходимых для жизни клеток, например, в процессах репликации и репарации ДНК, но используются и в различных приложениях молекулярной биологии и биотехнологии, в частности, в методе полимеразной цепной реакции (ПЦР). ПЦР является быстрым, специфичным и чувствительным методом амплификации нуклеотидных последовательностей, который широко используется в научных исследованиях и клинической практике. Для проведения ПЦР необходимы термостабильные полимеразы, обладающие (в зависимости от решаемой задачи) определенным набором дополнительных свойств, таких как высокая точность, 5ꞌ–3ꞌ- и 3ꞌ–5ꞌ-экзонуклеазные активности, высокая скорость работы, создание тупых или липких концов у синтезируемых цепей ДНК [1].
Необходимо отметить, что в ПЦР используются в основном ДНК-полимеразы, принадлежащие к двум из семи известных структурных семейств, а именно, семействам А и Б. Наиболее известные представители этих семейств – полимеразы Taq из Thermus aquaticus и Pfu из Pyrococcus furiosus. Для получения ферментов с улучшенными каталитическими свойствами природные варианты этих ферментов и их гомологи модифицировали с использованием технологий белковой инженерии.
Наибольшие трудности возникают при попытках амплификации ДНК, находящихся в клеточных экстрактах, крови, образцах “древней ДНК” и других природных материалах. В подобных случаях возникают проблемы, обусловленные присутствием различных ингибиторов полимераз, большой долей поврежденной ДНК и малым количеством ДНК в целом. Кроме того, амплификация протяженных фрагментов ДНК сталкивается с ограничениями, связанными с процессивными свойствами конкретной ДНК-полимеразы. Более того, в ДНК могут встречаться особо сложные последовательности, например GC-богатые, или участки, формирующие прочные вторичные структуры, которые существенно снижают процессивность ДНК-полимераз. На данный момент не обнаружено и не создано “универсальной” ДНК-полимеразы, способной преодолевать все описанные препятствия при проведении ПЦР как в научных исследованиях, так и в диагностических целях. Выбор конкретной полимеразы зависит от нескольких факторов: происхождения ДНК, структуры праймеров, длины матрицы, содержания GC-богатых участков [2]. На работу полимераз влияют также концентрации солей, катионов металлов и 2'-дезоксирибонуклеотидтрифосфатов [3].
В нашем обзоре рассмотрены основные структурные и молекулярно-кинетические особенности функционирования ДНК-полимераз, принадлежащих к структурному семейству А. Филогенетический анализ этих ферментов позволил установить консенсусный “слепок”, включающий 62 высококонсервативных аминокислотных остатка, распределенных в структуре фермента. Проведение сравнительного анализа остатков, входящих в “функциональный слепок”, у малоизученных ДНК-полимераз позволило выявить семь ферментов, потенциально обладающих свойствами, необходимыми для использования в биотехнологии.
ФЕРМЕНТАТИВНЫЕ СВОЙСТВА ДНК-ПОЛИМЕРАЗ СЕМЕЙСТВА А
ДНК-полимеразы семейства А обладают репликативной и репарационной активностями. 3' → 5'-Экзонуклеазная активность выполняет корректирующую функцию, позволяя в случае ошибки удалять присоединенный некомплементарный нуклеотид, и таким образом повышает точность синтеза ДНК. При этом 5' → 3'-экзонуклеазная активность позволяет удалять РНК-праймеры. Полимеразы этого семейства обнаружены у эукариот (Pol γ, Pol θ, Pol v), бактерий (Pol I) и вирусов (T7 DNA pol) [4–6].
Первые термостабильные полимеразы семейства А были выделены из бактерий рода Thermus, оптимальная температура роста которых близка к 70°C, при этом некоторые виды могут выживать и при 80°C [7]. Кроме нуклеотидилтрансферазной активности, ДНК-полимеразы этих организмов обладают 5'–3'-экзонуклеазной активностью, однако у них нет 3'–5'-экзонуклеазной активности, значительно повышающей точность синтеза ДНК за счет удаления ошибочно присоединенных нуклеотидов [8–10]. Точность синтеза ДНК этими ферментами зависит от рН и концентрации ионов Mg2+. Кроме того, на ошибочность синтеза влияет концентрация dNTP, причем оптимальным является равенство концентраций различных нуклеозидтрифосфатов. Эти полимеразы добавляют также 3′-адениновый “хвост” к синтезированной цепи.
Наиболее известным и хорошо изученным представителем бактериальных полимераз семейства А является Taq-полимераза из T. aquaticus [11]. Это первая термостабильная ДНК-полимераза, использованная в ПЦР [12] и ставшая одним из самых важных инструментов молекулярной биологии. Оптимальная температура работы Taq находится в промежутке 75–80°C; для ее работы необходимы ионы Mg2+ [9]. Время полужизни полимеразы составляет около 40 мин при 95°C, что с момента открытия архейных термостабильных полимераз считается низким.
В настоящее время Taq в основном применяется, когда требуется амплифицировать короткие последовательности ДНК – до 1 т.п.н., так как уже при такой длине (и при 50%-ном GC-содержании) эффективность синтеза составляет около 80% и сильно падает при дальнейшем увеличении длины [13]. Taq легко ингибируется компонентами плазмы крови, включая гемоглобин, лактоферрин и Ca2+, поэтому при проведении клинических анализов в смесь добавляют бычий сывороточный альбумин, увеличивающий выход реакции [14]. В других случаях используют специальный буфер, повышающий точность синтеза вплоть до 3.3 × 10–6 ошибок на пару оснований, что считается приемлемым, но все еще уступает показателям ДНК-полимераз семейства Б [15–17].
Помимо Taq-полимеразы применяют и другие ферменты. Tfl из T. flavus является практически полной копией Taq по характеристикам [18], Tth из T. thermophilis [19] и Tfi из T. filiformis [8] имеют схожие с Taq характеристики кроме немного сниженной термостабильности. Кроме того, найдены более термостабильные варианты полимераз: Tbr из T. brockianus, Tca из Thermophilus caldophilus [10] и несколько полимераз из бактерий рода Thermotoga, живущих при температурах вплоть до 90°C [20].
МЕХАНИЗМ ДЕЙСТВИЯ ДНК-ПОЛИМЕРАЗ
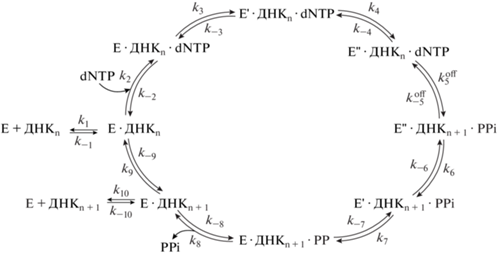
Молекулярный механизм добавления нуклеотидов ДНК-полимеразами был предложен по аналогии с практически идентичным механизмом 3'–5'-экзонуклеазной реакции, катализируемой ДНК-полимеразой I [21, 22], получившим название “two metal ion mechanism”. После связывания полимеразы на стыке дуплекса праймер–матрица входящий нуклеотид связывается и позиционируется в активном сайте с помощью Уотсон–Криковских взаимодействий с основанием матрицы, межмолекулярных контактов с аминокислотными остатками активного центра и координацией ионами двухвалентных металлов. Один ион металла связывается между концевым атомом O3' праймера и α-фосфатом входящего dNTP и часто упоминается как металл A (MA), или каталитический ион металла (рис. 1). Второй ион металла координируется входящим dNTP через немостиковые атомы кислорода α-, β- и γ-фосфатов и часто упоминается как металл B, или нуклеотидсвязывающий ион металла (MB) (pис. 1). Во время катализа MA служит кислотой Льюиса для снижения pKa 3'-OH-группы праймера для отщепления и последующей прямой нуклеофильной атаки на α-фосфат dNTP с образованием тригонально-бипирамидального пентакоординированного переходного состояния, которое стабилизируется за счет координации ионов металлов с кислородами β-γ-фосфатных групп. Этот этап приводит к инверсии стереоконфигурации ɑ-фосфата и согласованному высвобождению уходящего пирофосфата, координированного MB. Ион MB ориентирует трифосфатную часть связанного нуклеотида в пригодное для катализа состояние и дестабилизирует тройной комплекс полимеразы. После нуклеофильной атаки MB стабилизирует пентакоординированное переходное состояние и нейтрализует отрицательный заряд на уходящей группе PPi (pис. 1).
Рис. 1.
Предполагаемый механизм реакции полимеризации ДНК, катализируемой ДНК-полимеразой. Катализ опосредуется двумя ионами двухвалентных металлов, координированными в ДНК-полимеразе Pol I из T. aquaticus аминокислотными остатками Asp610, Asp785, Glu786.
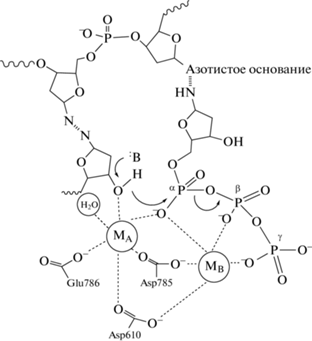
При взаимодействии с ферментом дуплекс матрица–праймер связывается в неглубокой щели между “большим пальцем” и 3'-экзодоменом. При этом значительная часть контактов с фосфатным остовом обеспечивается ɑ-спиралью “большого пальца” (рис. 2) [23, 24]. В бинарном комплексе полимераза-ДНК концевая пара оснований праймера примыкает к области, образованной субдоменом пальцев. Эта граница активного центра образована преимущественно длинной α-спиралью (O-спираль) и проходит по всей длине субдомена пальцев; она содержит на поверхности группу важных и высококонсервативных аминокислотных остатков, ориентированных внутрь щели. На С-конце О-спирали находится боковая цепь Tyr. Остаток тирозина находится в стекинге с концевой парой оснований. При связывании корректного dNTP образуется тройной комплекс полимераза-ДНК-dNTP, при этом полимераза претерпевает значительные конформационные изменения. При переходе от открытой (бинарный комплекс) к закрытой (тройной комплекс) конформации боковая цепь Tyr на С-конце О-спирали перемещается в активный центр. Конформационная динамика вносит важный вклад в высокую точность ДНК-полимераз, поскольку после начального распознавания канонической структуры входящего основания в структуре фермента происходят более значительные конформационные изменения.
Рис. 2.
Структура комплекса KlenTaq (фрагмент Кленова Taq-полимеразы) с ДНК (PDB ID: 3RTV). Домены большого пальца, пальцев и ладони располагаются в форме правой руки, типичной для большинства известных полимераз.
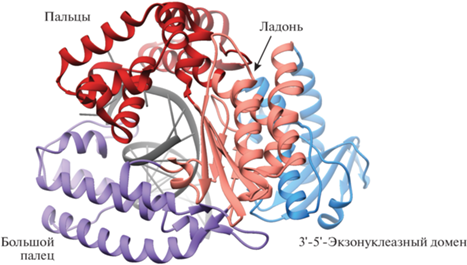
Кинетическая схема включения нуклеотидов в растущую цепь праймера ДНК под действием ДНК-полимераз протекает с участием иона двухвалентного металла и предполагает упорядоченное добавление субстратов (pис. 3). На первом этапе происходит связывание дуплекса праймер–матрица с образованием бинарного комплекса E·ДНКn. Правильное позиционирование 3'-конца праймера создает необходимое пространство для связывания входящего dNTP на следующем этапе. Этот этап включает также связывание иона металла в сайте связывания B (MB) и, возможно, в сайте связывания A (MA). После образования “открытого” тройного комплекса E·ДНКn·dNTP многие полимеразы претерпевают конформационные изменения, что приводит к образованию “закрытого” тройного комплекса E'⋅ДНКn⋅dNTP. Каталитически компетентное состояние E"⋅ДНКn⋅dNTP образуется после второго конформационного изменения в активном центре полимеразы. Реакционноспособные группы, включая ионы двухвалентных металлов, каталитические карбоксилатные остатки, 3'-ОН-цепь праймера и α-фосфат связанного нуклеотида выравниваются должным образом для последующего переноса нуклеотида. В результате протекает химическая стадия и происходит удлинение цепи праймера на один нуклеотид с образованием комплекса E″⋅ДНКn+ 1⋅PPi. После химической стадии фермент возвращается в исходное состояние и происходит высвобождение пирофосфата PPi из активного сайта полимеразы. Далее процесс может протекать по двум путям. Полимераза может транслоцироваться на одну пару оснований вдоль ДНК для осуществления последующего цикла включения нуклеотида (процессивный синтез ДНК), либо возможна диссоциация двойного комплекса (дистрибутивный синтез ДНК). Конкретные детали данной модели могут варьировать в зависимости от ДНК-полимеразы или системы. Включение и порядок элементарных стадий, представленных на рис. 3, подтверждаются кинетическими, структурными и/или биофизическими данными [6, 23, 26, 27].
АРХИТЕКТУРА БЕЛКОВОЙ ГЛОБУЛЫ И ИНЖЕНЕРИЯ ДНК-ПОЛИМЕРАЗ СЕМЕЙСТВА А
Первой изученной ДНК-полимеразой семейства А стала Pol I из Escherichia coli. Анализ кинетических и рентгеноструктурных данных изучения фрагмента Кленова и его гомологов из бактериофага Т7 и термофилов T. aquaticus и Bacillus stearothermophilus позволил обосновать механизм реакции, катализируемый ДНК-полимеразой, а также предложить “two metal ion mechanism” [21, 22, 28].
Во второй половине 1990-х гг. было получено большое количество структур полимераз семейства А, включая Taq [23]. Эти полимеразы имеют 5'–3'-экзонуклеазный, 3'–5'-экзонуклеазный и 5'–3'-полимеразный домены. Полимеразный домен, в свою очередь, подразделяется на домены большого пальца, пальцев и ладони. Полимераза без 5'–3'-экзонуклеазного домена называется фрагментом Кленова и именно в таком виде обычно используется в ПЦР, чтобы исключить деградацию праймеров. 3'–5'-Экзонуклеазный домен не работает в полимеразах некоторых организмов, включая род Thermus, поскольку не содержит специальные мотивы ExoI, ExoII и ExoIII.
За связывание с дуплексом матрица–праймер отвечает домен большого пальца. Он обхватывает ДНК со стороны малой бороздки и заставляет ее слегка изогнуться. В этом домене обнаружены несколько аминокислотных остатков, замены которых влияют на свойства полимеразы. В одной из работ показано, что замена Ser543Asn в молекуле Taq-полимеразы приводит к увеличению эффективности полимеразы на GC-богатых участках [29]. Кроме того, замены Arg660Thr, Arg487Val, Lys508Trp, Arg536Lys и Arg587Ile повышают точность синтеза ДНК-полимеразой [30]. При этом замены Arg487Val и Lys508Trp значительно снижали скорость работы фермента. Следует отметить, что двойные и тройные мутанты по точности синтеза не превосходили мутантную форму Arg660Thr, поэтому именно эта мутантная форма Taq признана наиболее перспективной [30]. Получена также мутантная форма [31], содержащая сразу восемь замен (Asn483Lys, Glu507Lys, Ser515Asn, Lys540Gly, Ala570Glu, Asp578Gly, Val586Gly, Ile614Met), которая отличается от фермента дикого типа более высокой точностью синтеза. Примечательно, что этот набор из восьми аминокислотных остатков не перекрывается с набором из пяти остатков, полученным в [30].
С одноцепочечным фрагментом ДНК-матрицы и dNTP связывается домен пальцев. В нем выделяют две α-спирали: первая (аминокислотные остатки 737–756) взаимодействует с нуклеотидами матрицы на границе дуплекса, вторая (656–671) взаимодействует с dNTP. Показано, что остатки Glu742 и Ala743 α-спирали 737–756 полимеразы Taq расположены в непосредственной близости от нуклеотида, идущего следом за стоящим напротив входящего dNTP, и сильно влияют на сродство полимеразы к ДНК. Установлено также, что замены Glu742Ala или Ala743Arg/Lys/His заметно увеличивают процессивность фермента [32]. Важную роль играет и Asn750 (нумерация согласно Taq), замена которого на Ala, наоборот, ухудшает свойства полимеразы из E. coli [33]. Интересный результат получен при изучении химерной Taq с дополнительными девятью остатками в петле после Arg736 – эта химерная полимераза работала быстрее и имела в целом бо́льшую производительность [34].
Для взаимодействия с dNTP важны высококонсервативные остатки Arg659, Lys663, Phe667 и Tyr671. Первые два связывают трифосфатную часть, Phe667 взаимодействует с азотистым основанием dNTP. До связывания dNTP в активном сайте фермента Tyr671 взаимодействует с основанием первого нуклеотида дуплексной части матрицы, а после связывания dNTP на место Tyr671 встает нуклеотид одноцепочечного фрагмента ДНК-матрицы, который до этого находился в вывернутом из спирали ДНК состоянии [35]. Замена этих аминокислотных остатков приводит к снижению точности синтеза [36]. Интересно, что замена Phe667Tyr приводит к лучшему связыванию ddNTP, что может быть полезным при секвенировании по методу Сэнгера.
Аминокислотные остатки, ответственные за катализ, располагаются в домене ладони. В Taq-полимеразе консервативные остатки Asp610, Asp785 и Glu786 связывают два иона металла. Кроме этих трех остатков один ион координирует 3'-ОН и α-фосфатную группы dNTP, и две молекулы воды, а второй ион – три фосфатных группы и карбонильный кислород Tyr611 [25]. Предполагаемый механизм реакции полимеризации ДНК, катализируемой ДНК-полимеразами, представлен на рис. 1.
За исправление ошибок синтеза отвечает 3'–5'-экзонуклеазный домен. Установлено, что работа этого домена максимально эффективна, когда его последовательность содержит три мотива: ExoI, ExoII и ExoIII [37]. Основу экзонуклеазного активного центра полимеразы E. coli составляют остатки Asp355, Glu357, Asp424 и Asp501. У полимераз из бактерий рода Thermus в соответствующих позициях нет ни одного остатка с боковой карбоксильной группой. Введение четырех аналогичных остатков в Taq-полимеразу (Gly308Asp, Val310Glu, Leu356Asp, Arg405Asp) приводит к восстановлению экзонуклеазной активности, но снижает частоту ошибок всего в 2 раза [38].
Еще один интересный пример удачной инженерии – химера Taq и Tth [39]. В данной работе решили объединить по два домена от каждого фермента: экзонуклеазный с большим пальцем из Tth-полимеразы (остатки до 600-го) и ладонь с пальцами из Taq-полимеразы (остатки после 600-го). В итоге получился фермент, обладающий объединенными плюсами полимераз Taq и Tth. Химера отличалась производительностью в 5 раз большей, чем у Taq-полимеразы и была в 6 раз более чувствительной к некомплементарности в праймере, чем Tth-полимераза, равняясь по этому показателю с полимеразой Taq. Также химерный фермент обладал повышенной способностью реплицировать длинные последовательности.
Таким образом, проведена значительная работа по улучшению свойств полимераз семейства А. Получены ферменты, в несколько раз превосходящие Taq-полимеразу по всем параметрам. Тем не менее, даже в таком виде полученные ферменты далеки от требований, предъявляемых современной биотехнологией, поэтому продолжается поиск новых полимераз с улучшенными свойствами. В данном обзоре под термином “новые ДНК-полимеразы” подразумеваются любые неохарактеризованные ДНК-полимеразы, последовательности которых значительно отличаются от последовательностей охарактеризованных ферментов.
ОТБОР МИКРООРГАНИЗМОВ С НОВЫМИ ДНК-ПОЛИМЕРАЗАМИ
Одно из ключевых свойств ДНК-полимеразы, применяемой в ПЦР, – термостабильность. На данный момент понимание причин природной термостабильности остается довольно скудным. Поэтому главный критерий, по которому отбирают организмы с надеждой найти новые термостабильные ферменты, это их термофильность, т.е. повышенная (обычно выше 60°C) оптимальная температура роста. Но, к сожалению, и этот критерий не гарантирует получения термостабильного фермента. Известны организмы, живущие при повышенных вплоть до 80°C температурах, полимеразы которых работают при таких температурах, но их стабильность значительно снижается при более высоких температурах, поэтому они не могут использоваться в ПЦР. С другой стороны, можно предположить, что фермент определенного организма может оставаться стабильным при температурах, превышающих оптимальную температуру роста данного организма.
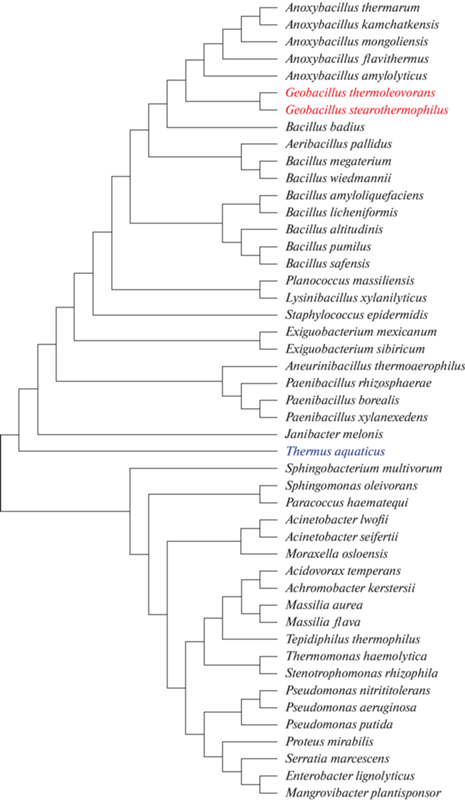
С целью апробации алгоритма поиска новых ДНК-полимераз были проанализированы последовательности полимераз всех микроорганизмов из Коллекции экстремофильных микроорганизмов и типовых культур (КЭМТК) ИХБФМ СО РАН (http://www.niboch.nsc.ru/doku.php/emtc_collection). Анализ более 4000 депонированных микроорганизмов позволил отобрать 47, обитающих при температуре выше 50°C. Сравнение нуклеотидных последовательностей генов ДНК-полимераз этих 47 микроорганизмов позволило построить филогенетическое дерево, отражающее сходство ферментов (рис. 4). Необходимо отметить, что три из этих 47 ДНК-полимераз, выделенные из представителей родов Thermus и Geobacillus, уже применяются на практике, поэтому далее были рассмотрены оставшиеся 44 фермента.
Рис. 4.
Филогенетичесое дерево ДНК-полимераз семейства А, отобранных из коллекции экстремофильных микроорганизмов, типовых культур и организмов КЭМТК. Цветом отмечены ДНК-полимеразы, которые применяются на практике: представители рода Thermus (выделен синим) и Geobacillus (выделены красным).
ДНК-полимеразы представителей рода Thermus являются основным источником биотехнологических ДНК-полимераз семейства А. На основании представленных в базе данных Uniprot последовательностей ДНК-полимераз 12 бактерий рода Thermus (с учетом аминокислотных замен S543N, R660T, A743R, оказывающих положительный эффект на ферментативные свойства) получена консенсусная последовательность этих ДНК-полимераз. Эту консенсусную последовательность использовали в качестве эталонной для сравнения с 44 ДНК-полимеразами семейства А микроорганизмов, отобранных из коллекции КЭМТК и не относящихся к Thermus и Geobacillus. Попарное сравнение полимеразных доменов 44 ферментов показало, что сходство всех ДНК-полимераз с консенсусной последовательностью составляет около 30–36%. Таким образом, на данном этапе был сформирован список ДНК-полимераз семейства А, которые отличаются от полимераз представителей рода Thermus, что может привести к различиям в таких важных свойствах данных ферментов, как, например, процессивность и точность. Однако это слишком общий критерий селекции новых ДНК-полимераз, поэтому требуются дополнительные ограничения.
С целью более детального сравнения были проанализированы структуры комплекса KlenTaq (фрагмент Кленова Taq-полимеразы) с ДНК [25], что позволило выделить 62 аминокислотных остатка, непосредственно контактирующих с субстратами и кофакторами – ДНК, dNTP и ионами металлов, т.е. либо имеющих водородные связи с ними, либо расположенных на расстоянии до 3 Å от них. Как оказалось, эти остатки обладают высокой степенью консервативности во всех вариантах ДНК-полимераз семейства А не только из рода Thermus, но среди отобранных 44 ферментов, что может отражать их функциональную значимость (рис. 5). Интересно отметить, что около половины этих остатков расположены в домене большого пальца и имеют прямой контакт с двухцепочечной частью ДНК (рис. 6). Еще около трети находятся в пальцах и связываются с одноцепочечной частью ДНК, включая пять dNTP-связывающих остатков. Наконец, оставшиеся семь остатков из домена ладони располагаются в различных местах активного центра.
Рис. 5.
Графическое представление относительных частот встречаемости различных остатков в последовательностях ДНК-полимераз из организмов, приведенных на рис. 4. Показан полимеразный домен, нумерация остатков соответствует ферменту Taq. Звездочками отмечены 62 аминокислотных остатка, взаимодействующих с субстратами и ионами металла.
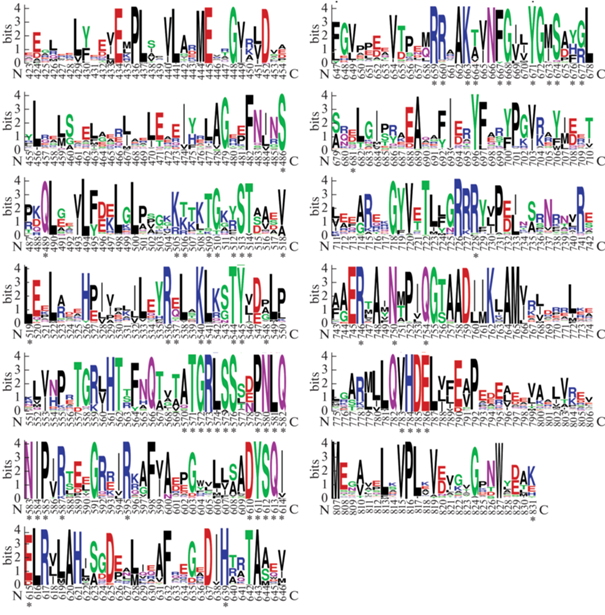
Рис. 6.
Схема расположения доменов в аминокислотной последовательности ДНК-полимеразы на примере Taq. Красными точками и полосами возле шкалы обозначены области, в которых находятся 62 субстрат- и кофактор-связывающих остатка.

В общем смысле “слепок” из ферментов Thermus, как набор функциональных аминокислотных остатков в определенных местах, может характеризовать все полимеразы семейства А, в том числе не обладающие термостабильностью. Например, в молекуле ДНК-полимеразы I E. coli аналоги описанных 62 остатков расположены в тех же местах и выполняют те же функции. Таким образом, набор этих консервативных остатков может выступать функциональным слепком, характеризующим ДНК-полимеразы семейства А в целом, а параметр термостабильности должен быть связан с дополнительными критериями отбора, например, с местом обитания микроорганизма.
Дальнейшее сравнение аминокислотного состава 44 ДНК-полимераз проводили по сходству остатков, входящих в данный функциональный слепок, при их попарном сравнении. Интересно отметить, что во всех 44 отобранных ферментах, принадлежащих к структурному семейству А, сходство этих 62 аминокислотных остатков с функциональным слепком было существенно выше и находилось в промежутке 71–85%, что свидетельствует о высокой функциональной важности входящих в него аминокислотных остатков (табл. 1). Тем не менее, не все эти остатки сохраняют консервативность в анализируемых ферментах. Это может свидетельствовать об изменении свойств отдельных ферментов и их отличии от свойств представителей полимераз Thermus.
Таблица 1.
Сходство функционального слепка ферментов бактерий рода Thermus и ДНК-полимераз других организмов
| Номер | Организм* | Сходство, % | UniProt ID |
|---|---|---|---|
| 1 | Exiguobacterium sibiricum | 84.4 | B1YKA1 |
| 2 | Exiguobacterium mexicanum | 84.4 | A0A099DI91 |
| 3 | Bacillus licheniformis | 84.3 | A0A1Q9FQ82 |
| 4 | Bacillus wiedmannii | 84.3 | A0A0G8C2W8 |
| 5 | Anoxybacillus kamchatkensis | 83.5 | A0A4S3L4B1 |
| 6 | Anoxybacillus mongoliensis | 83.5 | A0A7W8JGI3 |
| 7 | Anoxybacillus flavithermus | 83.5 | A0A178TIV8 |
| 8 | Anoxybacillus thermarum | 83.5 | A0A0D0RXS3 |
| 9 | Anoxybacillus amylolyticus | 83.5 | A0A167TTL3 |
| 10 | Acidovorax temperans | 82.9 | A0A543L9Q8 |
| 11 | Sphingobacterium multivorum | 82.0 | A0A2X2J883 |
| 12 | Acinetobacter lwoffii | 81.7 | A0A4Y3IZ28 |
| 13 | Pseudomonas aeruginosa | 80.8 | A0A069PX19 |
| 14 | Massilia aurea | 79.8 | A0A422QJZ7 |
| 15 | Achromobacter kerstersii | 78.6 | A0A6S6ZT87 |
| 16 | Bacillus altitudinis | 78.5 | A0A1K1VQ13 |
| 17 | Aneurinibacillus thermoaerophilus | 78.4 | A0A1G7WTW7 |
| 18 | Staphylococcus epidermidis | 78.4 | A0A5R1C3J3 |
| 19 | Paenibacillus borealis | 78.2 | A0A089LEG1 |
| 20 | Tepidiphilus thermophilus | 78.0 | A0A0K6IUJ5 |
| 21 | Acinetobacter seifertii | 77.8 | N8R043 |
| 22 | Lysinibacillus xylanilyticus | 77.5 | A0A0K9F7W4 |
| 23 | Proteus mirabilis | 77.4 | A0A5F0S8B1 |
| 24 | Serratia marcescens | 77.2 | A0A0P0Q8W4 |
| 25 | Enterobacter lignolyticus | 77.2 | E3G5L7 |
| 26 | Mangrovibacter plantisponsor | 77.2 | A0A317PXC7 |
| 27 | Moraxella osloensis | 77.1 | A0A378QCD4 |
| 28 | Paracoccus haematequi | 77.0 | A0A3S4GLG8 |
| 29 | Paenibacillus rhizosphaerae | 77.0 | A0A1R1ERS0 |
| 30 | Paenibacillus xylanexedens | 77.0 | A0A1L5LNH1 |
| 31 | Pseudomonas nitrititolerans | 76.9 | A0A7Y3RE40 |
| 32 | Janibacter melonis | 76.6 | A0A176QCH1 |
| 33 | Planococcus massiliensis | 76.4 | A0A098EN26 |
| 34 | Bacillus megaterium | 76.3 | A0A0L1MA76 |
| 35 | Bacillus pumilus | 76.3 | A0A0H1RZB5 |
| 36 | Bacillus amyloliquefaciens | 76.3 | A0A4V7TSC5 |
| 37 | Bacillus safensis | 76.3 | A0A1L6ZGT4 |
| 38 | Aeribacillus pallidus | 76.1 | A0A223EA52 |
| 39 | Bacillus badius | 76.0 | A0A0C2Y898 |
| 40 | Pseudomonas putida | 75.7 | A0A177SGM0 |
| 41 | Massilia flava | 75.0 | A0A562Q539 |
| 42 | Stenotrophomonas rhizophila | 74.1 | A0A023XYJ8 |
| 43 | Thermomonas haemolytica | 73.0 | A0A4R3N9Q2 |
| 44 | Sphingomonas oleivorans | 71.3 | A0A2T5FXB6 |
Необходимо отметить, что среди микроорганизмов, обладающих максимальным сходством (табл. 1), встречаются представители одного рода, отличия между которыми относительно невелики. Поэтому на основании проведенного анализа (при условии отбора по одному представителю одного рода) можно выделить семь ДНК-полимераз, выделенных из Exiguobacterium sibiricum, Bacillus licheniformis, Anoxybacillus kamchatkensis, Acidovorax temperans, Sphingobacterium multivorum, Acinetobacter lwoffii и Pseudomonas aeruginosa, аминокислотная последовательность которых обладала сходством более 80% с функциональным слепком ферментов Thermus. Можно предположить, что обнаруженные отличия состава функционально важных аминокислотных остатков ДНК-полимераз из этих организмов приведут к улучшению свойств ферментов, необходимых для их использования в качестве биотехнологических инструментов.
Интересно отметить, что лишь одна из ДНК-полимераз этих семи организмов изучена в настоящее время, а именно ДНК-полимераза B. licheniformis. В работе 1977 года показано, что полимераза этого умеренно термофильного организма может работать при температуре 55°С [40]. Кроме того, два представителя рода Anoxybacillus, входящие в список, могут существовать при температурах выше 60°С [41, 42], но их ДНК-полимеразы пока не охарактеризованы.
Таким образом, полученный функциональный слепок позволил сравнить представителей ДНК-полимераз семейства А с ферментами, которые применяются в биотехнологии и нуждаются в улучшении их ферментативных характеристик. Так как сравнение выполнено не по полной аминокислотной последовательности, а лишь по важным функциональным аминокислотным остаткам, можно предположить, что степень соответствия консенсусу будет коррелировать с характеристиками ДНК-полимераз семейства А, которые используются в настоящее время, и при этом позволит выявить ферменты, превосходящие Taq и другие ДНК-полимеразы Thermus по точности синтеза.
Работа выполнена при поддержке Министерства науки и высшего образования, соглашение № 075-15-2021-1085.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Nikoomanzar A., Chim N., Yik E.J., Chaput J.C. (2020) Engineering polymerases for applications in synthetic biology. Q. Rev. Biophys. 53, 1–31.
Wu D.A.N.Y., Ugozzoli L., Pal B.K., Qian J.I.N., Wallace R.B. (1991) The effect of temperature and oligonucleotide primer length on the specificity and efficiency of amplification by the polymerase chain reaction. DNA Cell Biol. 10, 233–238.
Owczarzy R., Moreira B.G., You Y., Behlke M.A., Walder J.A. (2008) Predicting stability of DNA duplexes in solutions containing magnesium and monovalent cations. Biochemistry. 47, 5336–5353.
Garcia-Diaz M., Bebenek K. (2007) Multiple functions of DNA polymerases. CRC. Crit. Rev. Plant Sci. 26, 105–122.
Alba M.M. (2001) Replicative DNA polymerases. Genome Biol. 2, 1–7.
Rothwell P.J., Waksman G. (2005) Structure and mechanism of DNA polymerases. Adv. Protein Chem. 71, 401–440.
Chien A., Edgar D.B., Trela J.M. (1976) Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus. J. Bacteriol. 127, 1550–1557.
Choi J.J., Jung S.E., Kim H.K., Kwon S.T. (1999) Purification and properties of Thermus filiformis DNA polymerase expressed in Escherichia coli. Biotechnol. Appl. Biochem. 30, 19–25.
Lawyer F.C., Stoffel S., Saiki R.K., Chang S.Y., Landre P.A., Abramson R.D., Gelfand D.H. (1993) High-level expression, purification, and enzymatic characterization of full-length Thermus aquaticus DNA polymerase and a truncated form deficient in 5′ to 3′ exonuclease activity. Genome Res. 2, 275–287.
Park J.H., Kim J.S., Kwon S.-T., Lee D.-S. (1993) Purification and characterization of Thermus caldophilus GK24 DNA polymerase. Eur. J. Biochem. 214, 135–140.
Каледин А.С., Слюсаренко А.Г., Городецкий С.И. (1980) Выделение и свойства ДНК-полимеразы из экстремально-термофильной бактерии Thermus aquaticus YT1. Биохимия. 45, 644–651.
Saiki R.K., Gelfand D.H., Stoffel S., Scharf S.J., Higuchi R., Horn G.T., Mullis K.B., Erlich H.A. (1988) Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science. 239, 487–491.
Arezi B., Xing W., Sorge J.A., Hogrefe H.H. (2003) Amplification efficiency of thermostable DNA polymerases. Anal. Biochem. 321, 226–235.
Al-Soud W.A., Rådström P. (2001) Purification and characterization of PCR-inhibitory components in blood cells. J. Clin. Microbiol. 39, 485–493.
Flaman J.-M., Frebourg T., Moreau V., Charbonnier F., Martin C., Ishioka C., Friend S.H., Iggo R. (1994) A rapid PCR fidelity assay. Nucl. Acids Res. 22, 3259–3260.
Ling L.L., Keohavong P., Dias C., Thilly W.G. (1991) Optimization of the polymerase chain reaction with regard to fidelity: modified T7, Taq, and Vent DNA polymerases. Genome Res. 1, 63–69.
Lee J.I., Kim Y.J., Bae H., Cho S.S., Lee J.-H., Kwon S.-T. (2010) Biochemical properties and PCR performance of a family B DNA polymerase from hyperthermophilic euryarchaeon Thermococcus peptonophilus. Appl. Biochem. Biotechnol. 160, 1585–1599.
Harrell R.A., Hart R.P. (1994) Rapid preparation of Thermus flavus DNA polymerase. Genome Res. 3, 372–375.
Carballeira N., Nazabal M., Brito J., Garcia O. (1990) Purification of a thermostable DNA polymerase from Thermus thermophilus HB8, useful in the polymerase chain reaction. Biotechniques. 9, 276–281.
Yang S.-W., Astatke M., Potter J., Chatterjee D.K. (2002) Mutant Thermotoga neapolitana DNA polymerase I: altered catalytic properties for non-templated nucleotide addition and incorporation of correct nucleotides. Nucl. Acids Res. 30, 4314–4320.
Steitz T.A. (1993) DNA- and RNA-dependent DNA polymerases. Curr. Opin. Struct. Biol. 3, 31–38.
Steitz T.A. (1998) A mechanism for all polymerases. Nature. 391, 231–2323.
Steitz T.A. (1999) DNA polymerases: structural diversity and common mechanisms. J. Biol. Chem. 274, 17395–17398.
Joyce C.M. (2013) DNA polymerase I, Bacterial. in Encyclopedia of Biological Chemistry, 2nd ed. 87–90. Elsevier Inc.
Betz K., Malyshev D.A., Lavergne T., Welte W., Diederichs K., Dwyer T.J., Ordoukhanian P., Romesberg F.E., Marx A. (2012) KlenTaq polymerase replicates unnatural base pairs by inducing a Watson-Crick geometry. Nat. Chem. Biol. 8, 612–614.
Raper A.T., Reed A.J., Suo Z. (2018) Kinetic mechanism of DNA polymerases: contributions of conformational dynamics and a third divalent metal ion. Chem. Rev. 118, 6000–6025.
Berdis A.J. (2009) Mechanisms of DNA polymerases. Chem. Rev. 109, 2862–2879.
Brautigam C.A., Steitz T.A. (1998) Structural and functional insights provided by crystal structures of DNA polymerases and their substrate complexes. Curr. Biol. 8, 54–63.
Ignatov K.B., Bashirova A.A., Miroshnikov A.I., Kramarov V.M. (1999) Mutation S543N in the thumb subdomain of the Taq DNA polymerase large fragment suppresses pausing associated with the template structure. FEBS Lett. 448, 145–148.
Drum M., Kranaster R., Ewald C., Blasczyk R., Marx A. (2014) Variants of a Thermus aquaticus DNA polymerase with increased selectivity for applications in allele- and methylation-specific amplification. PLoS One. 9, e96640.
Raghunathan G., Marx A. (2019) Identification of Thermus aquaticus DNA polymerase variants with increased mismatch discrimination and reverse transcriptase activity from a smart enzyme mutant library. Sci. Rep. 9, 590.
Yamagami T., Ishino S., Kawarabayasi Y., Ishino Y. (2014) Mutant Taq DNA polymerases with improved elongation ability as a useful reagent for genetic engineering. Front. Microbiol. 5. 461.
Minnick D.T., Bebenek K., Osheroff W.P., Turner R.M., Astatke M., Liu L., Kunkel T.A., Joyce C.M. (1999) Side chains that influence fidelity at the polymerase active site of Escherichia coli DNA polymerase I (Klenow fragment). J. Biol. Chem. 274, 3067–3075.
Yamagami T., Matsukawa H., Tsunekawa S., Kawarabayasi Y., Ishino S., Ishino Y. (2016) A longer finger-subdomain of family A DNA polymerases found by metagenomic analysis strengthens DNA binding and primer extension abilities. Gene. 576, 690–695.
Roberts R.J. (1995) On base flipping. Cell. 82, 9–12.
Suzuki M., Yoshida S., Adman E.T., Blank A., Loeb L.A. (2000) Thermus aquaticus DNA polymerase I mutants with altered fidelity. J. Biol. Chem. 275, 32728–32735.
Bernad A., Blanco L., Lázaro J., Martín G., Salas M. (1989) A conserved 3′→5′ exonuclease active site in prokaryotic and eukaryotic DNA polymerases. Cell. 59, 219–228.
Park Y., Choi H., Lee D.S., Kim Y. (1997) Improvement of the 3′-5′ exonuclease activity of Taq DNA polymerase by protein engineering in the active site. Mol. Cells. 7, 419–424.
Ignatov K., Kramarov V., Billingham S. (2009) Chimeric DNA polymerase. US20090209005A1, US
Stenesh J., McGowan G.R. (1977) DNA polymerase from mesophilic and thermophilic bacteria. Biochim. Biophys. Acta – Nucl. Acids Protein Synth. 475, 32–41.
Kevbrin V.V., Zengler K., Lysenko A., Wiegel J. (2005) Anoxybacillus kamchatkensis sp. nov., a novel thermophilic facultative aerobic bacterium with a broad pH optimum from the Geyser valley, Kamchatka. Extremophiles. 9, 391–398.
Namsaraev Z., Babasanova O., Dunaevsky Y., Akimov V., Barkhutova D., Gorlenko V.M., Namsaraev B. (2010) Anoxybacillus mongoliensis sp. nov., a novel thermophilic proteinase producing bacterium isolated from alkaline hot spring, central Mongolia. Mикpoбиoлoгия. 79, 516–523.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология