Молекулярная биология, 2023, T. 57, № 2, стр. 209-219
Современные подходы белковой инженерии к созданию ферментов с новыми каталитическими свойствами
Т. Е. Тюгашев a, О. С. Федорова a, Н. А. Кузнецов a, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
* E-mail: nikita.kuznetsov@niboch.nsc.ru
Поступила в редакцию 08.08.2022
После доработки 16.09.2022
Принята к публикации 16.09.2022
- EDN: EFXRBQ
- DOI: 10.31857/S0026898423020234
Аннотация
Аденин-ДНК-гликозилаза MutY – монофункциональный фермент, катализирующий гидролиз N-гликозидных связей с остатками аденина, расположенными в ДНК напротив остатков 8-оксогуанина. Проведен рациональный дизайн мутантных форм фермента с измененной каталитической активностью. Анализ структур мутантных форм MutY, рассчитанных методом молекулярной динамики, позволил сделать вывод, что некоторые мутантные формы MutY могут не только расщеплять N-гликозидную связь, но и обладают АР-лиазной активностью, как в случае бифункциональных ДНК-гликозилаз. Методом сайт-направленного мутагенеза получены мутантные формы MutY с заменами A120K или S124K и определена их каталитическая активность. Показано, что замена S120K приводит к появлению дополнительной АР-лиазной активности, в то время как замена A124K полностью инактивирует фермент.
ВВЕДЕНИЕ
Разработка ферментов с измененными свойствами – одно из важных приложений, направленных на использование ферментов в биотехнологических и медицинских целях. В настоящее время актуальны подходы, позволяющие найти фермент с измененными или улучшенными свойствами, такие как экспериментальный скрининг природных ферментов из различных источников; создание библиотек генов целевого фермента, имеющих случайно распределенные мутации, с последующим скринингом ферментативных свойств и рациональный дизайн ферментов с использованием молекулярного моделирования и прогнозирования свойств [1, 2]. Действительно, можно констатировать, что современные экспериментальные и вычислительные методы позволяют создавать улучшенные формы ферментов [3]. Например, достигнуты значительные успехи в улучшении точности, процессивности, термостабильности и других параметров таких широко используемых в диагностических целях ферментов, как ДНК-полимеразы [2].
Однако создание ферментов с измененными свойствами по-прежнему сталкивается со значительными проблемами, поскольку функциональные свойства белков определяются не только их относительно жесткой общей структурой, но, что еще более важно, их динамическими свойствами [4, 5]. В процессе фермент-субстратного взаимодействия в некоторых областях белковой глобулы могут происходить согласованные перемещения одних частей белковой молекулы относительно других, при этом некоторые участки очень динамичны, тогда как другие относительно статичны [6–8]. Такие сложные взаимодействия образуют динамическую кросс-корреляционную сеть, которая может иметь важное значение для аллостерических переходов [9, 10], а при формировании белок-белковых комплексов может выступать в качестве эффекторов каталитической активности [11, 12].
Последние 20 лет белковая инженерия активно развивалась за счет появления новых технологий, среди которых можно отметить направленную эволюцию, например, с помощью технологии фагового дисплея [13, 14], и рациональный дизайн, включающий сайт-направленный мутагенез и создание химерных ферментов [2, 15]. Стремительное развитие генетических технологий привело к тому, что актуальными объектами рационального дизайна стали ферменты, модифицирующие нуклеиновые кислоты, необходимые для осуществления основных процессов генетической инженерии.
Структурно-ориентированная инженерия остается одним из важнейших подходов к созданию ферментов с измененными свойствами. Действительно, высокая субстратная специфичность и/или определенный тип каталитической активности, характерные для большинства ферментов, могут препятствовать их использованию в практических приложениях. Поэтому важным представляется направленное изменение субстратной специфичности или каталитических свойств отдельных ферментов, процессирующих нуклеиновые кислоты.
Аденин-ДНК-гликозилаза MutY катализирует удаление аденина из неканонической пары с 7,8-дигидро-8-oксогуанином (oxoG) [16–20]. Помимо удаления аденин из пары oxoG:A MutY способна также удалять аденин из пары G:A [21], но со значительно меньшей скоростью [22]. Такая уникальная специфичность MutY должна обеспечиваться множественными контактами фермента с oxoG:А- и G:A-парами нуклеотидов. В настоящей работе нами проведен рациональный дизайн аминокислотных остатков активного центра фермента и создана мутантная форма аденин-ДНК-гликозилазы MutY с измененной каталитической активностью. Методом молекулярной динамики получены модели мутантных форм фермента MutY, которые не только гидролизуют N-гликозидную связь с аденином, но и потенциально могут обладать новым типом каталитической активности, а именно, катализировать разрыв 2'‑дезоксирибофосфатного остова ДНК в сайте узнавания ферментом. Методом сайт-направленного мутагенеза на основе данных моделирования получены мутантные формы MutY из E. coli, содержащие замены A120K или S124K, и определена их каталитическая активность. Показано, что замена S120K приводит к появлению дополнительной активности, в то время как A124K полностью инактивирует фермент.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Молекулярное моделирование. В молекулярно-динамических расчетах использовали структуру MutY из Geobacillus stearothermophilus в комплексе с нерасщепляемым одиннадцатизвенным ДНК-субстратом, содержащим 2'-фторо-2'-дезоксиаденозин (FLRC, PDB ID: 3G0Q) [23]. На основании этой структуры построены начальные модели фермента дикого типа и мутантных форм MutY из G. stearothermophilus , содержащих замены Y126K и A130K, соответствующие заменам S120K и A124K в MutY из E. coli. Были применены силовые поля AMBER ff99SB-ildn для описания белка [24–26] и bsc1 для ДНК [27, 28] с дополнительными параметрами для железо-серного кластера [4Fe-4S]2+ и 8-оксогуанина [29, 30], модель воды TIP3P и соответствующие параметры ионов [31, 32]. Протонирование аминокислотных остатков оценивали с использованием опубликованных данных и с помощью веб-сервера H++ [33]. Расчеты выполняли с помощью пакета ПО GROMACS [34, 35]. Комплекс помещали в додекаэдрическую периодическую ячейку, заполняемую молекулами воды и ионами, с минимальным расстоянием 1.1 нм до границы ячейки. Радиус обрезания дальних взаимодействий составлял 1 нм, электростатические взаимодействия рассчитывали методом PME [36], ковалентные связи с атомами водорода обрабатывали методом LINCS [37], шаг интегрирования составлял 2 фс. Релаксацию системы выполняли последовательно в ансамблях NVT и NPT с гармоническими ограничениями на подвижность тяжелых атомов фермент-субстратного комплекса, длительностью 0.5 нс для каждого этапа. После релаксации системы генерировали независимые молекулярно-динамические траектории длительностью 100 нс троекратно для каждого варианта комплекса. Использовали модифицированный термостат Берендсена с установленной температурой 300 K и баростат Паринелло–Рамана [38].
Олигодезоксирибонуклеотиды. Олигодезоксирибонуклеотиды синтезировали фосфитамидным методом на синтезаторе ASM-700 (“Biosset Ltd”, Россия) с использованием мономеров производства “Glen Research” (США). Олигодезоксирибонуклеотиды очищали с помощью ВЭЖХ на ионообменной колонке (PRP-X500 “Hamilton Company” 3.9 × 300 мм с размером частиц 12–30 мкм) и последующей обращенно-фазовой хроматографии (Nucleoprep 100-20 C18 10 × 250 мм, “Macherey-Nagel”, Германия). Чистоту олигонуклеотидов проверяли с помощью электрофореза в денатурирующем 20%-ном полиакриламидном геле (ПААГ). Концентрацию олигонуклеотидов измеряли по оптической плотности растворов на длине волны 260 нм в электронных спектрах поглощения и рассчитывали по закону Бугера–Ламберта–Бера, исходя из коэффициентов молярной экстинкции, определенных в приближении метода ближайших соседей [39]. В качестве ДНК-субстрата использовали олигодезоксирибонуклеотидный дуплекс с последовательностью CTCTC(oxoG)CCTTCC, содержащий аденин напротив 8-оксогуанина.
Ферменты. MutY выделен из клеток штамма BL21(DE3) E. coli, трансформированных плазмидой pET28c-MutY, как описано ранее [40]. Плазмида pET28c-MutY, содержащая ген MutY, любезно предоставлена М.К. Сапарбаевым (Groupe “Réparation de l’AND”, Université Paris-Sud XI, Institut Gustave Roussy, France).
Мутантные формы MutY с заменами S120K и A124K получены методом сайт-направленного мутагенеза. Правильность введения замен в ген MutY проверена секвенированием. Мутантные формы фермента очищали по следующей методике. Культуру клеток E. coli BL21(DE3) выращивали в среде LB (1 л), содержащей 25 мкг/мл канамицина, при температуре 37°С до оптической плотности 0.6–0.7 при длине волны 600 нм. После этого индуцировали транскрипцию добавлением изопропил-β-D-тиогалактопиранозида до 0.2 мМ и инкубировали клетки в течение 16 ч. Затем клетки осаждали центрифугированием (10 мин, 10 000 g) и готовили суспензию клеток в 30 мл буферного раствора 20 мМ HEPES-NaOH pH 7.8, 40 мМ NaCl, содержащего набор ингибиторов протеаз (“Complete”, ФРГ). Клетки лизировали при помощи френч-пресса. Все последующие процедуры проводили при 4°С. Лизат клеток центрифугировали (40 мин, 40000 g), фермент выделяли хроматографией. Супернатант наносили на колонку I (Q-Sepharose Fast Flow, “Amersham Biosciences”, Швеция) и промывали буферным раствором, содержащим 20 мМ HEPES-NaOH pH 7.8, 200 мМ NaCl. Фракции, содержащие белок MutY, собирали и наносили на колонку II (HiTrap- Chelating™, “Amersham Biosciences”) в буферном растворе (20 мМ HEPES-NaOH pH 7.8, 500 мМ NaCl, 20 мМ имидазол). Хроматографию проводили в линейном градиенте 20 → 500 мМ имидазола, оптическую плотность раствора регистрировали при длине волны 280 нм. Степень чистоты белка определяли с помощью гель-электрофореза. Фракции, содержащие мутантные формы MutY, диализовали в 20 мМ HEPES-NaOH-буфере pH 7.5, содержащем 1 мМ EDТА, 1 мМ дитиотреит, 250 мМ NaCl, 50%-ный глицерин, и хранили при –20°С. Концентрацию ферментов рассчитывали из значений оптической плотности белка при длине волны 280 нм и коэффициента молярной экстинкции 77328 М–1см–1 [41].
Электрофорез в ПААГ. В экспериментах по разделению продуктов реакции с помощью электрофореза в ПААГ использовали олигонуклеотиды, меченные 32P по 5'-концу. Все эксперименты выполняли при 25°С в буферном растворе 50 мМ Трис-HCl pH 7.5, 50 мМ KCl, 1 мМ EDTA, 1 мМ дитиотреит, 7%-ный глицерин. Зависимости степени превращения субстратов от времени получали с использованием следующей методики. К 10 мкл буферного раствора, содержащего 32P‑меченый ДНК-субстрат, добавляли 10 мкл 2.0–8.2 мкМ фермента в том же буферном растворе. Реакционную смесь быстро перемешивали и через определенные промежутки времени отбирали аликвоты объемом 2 мкл, которые переносили в пробирки, содержащие 2 мкл раствора 7 М мочевины, 0.1% бромфенолового синего и 0.1% ксиленцианола. Затем пробы делили пополам, к одной части добавляли 1 мкл 1 М NaOH и инкубировали при 56°C в течение 15 мин для гидролиза фосфодиэфирных связей в АР-сайтах. Раствор нейтрализовали 1 мкл 1 М HCl, наносили на ПААГ и проводили электрофорез при напряженности 50 В/см. Накопление продуктов реакции в ПААГ оценивали согласно [40].
Количество образующегося продукта определяли путем сканирования радиоавтографа на приборе Molecular Imager FX phosphorimager (“Bio-Rad”, США) и обработки данных в программном пакете Gel-Pro Analyzer 4.0 (“Media Cybernetics”, США). Степень накопления продуктов рассчитывали как отношение площадей пиков продуктов к сумме площадей пиков продуктов и пика исходного олигонуклеотида. Ошибка определения степени превращения не превышала, как правило, 20%.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Степень изгибания сахарофосфатного остова в составе фермент-субстратных комплексов
Известно несколько кристаллических структур MutY: определены структуры фрагмента фермента из E. coli в свободном виде [42, 43], мутантных форм MutY из G. stearothermophilus в комплексе с различными олигонуклеотидными дуплексами [23, 44–47], а также фрагментов MUTYH человека и мыши [48, 49]. Рентгеноструктурный анализ MutY в свободном состоянии и в комплексе с ДНК показал, что в каталитически активном комплексе ДНК-дуплекс значительно изогнут. Однако угол изгиба и степень деформации ДНК, связанной в активном центре фермента, различаются в структуре комплекса каталитически-активного MutY и 2'-F-dA ДНК-субстрата (рис. 1, FLRC, 3G0Q) и в структуре комплекса каталитически неактивной мутантной формы MutY D144N и ДНК-субстрата (рис. 1, LRC, 1RRQ). Возможной причиной таких отличий может быть как наличие у MutY механизма специфичного связывания с субстратом через перенос электрона и окисление кластера [4Fe-4S]2+ до [4Fe-4S]3+ поврежденной ДНК, так и эффект 2'-фтор-модификации нуклеотида в комплексе FLRC. Интересно отметить, что в процессе молекулярной динамики угол изгиба ДНК занимал среднее значение между данными структурами в комплексах FLRC и LRC соответственно (рис. 1).
Рис. 1.
Сравнение степени изгиба сахарофосфатного остова субстрата через расстояние между атомами P, фланкирующими вывернутое основание нуклеотидов: желтый – кристаллографическая структура комплекса каталитически активного MutY и 2′-F-dA ДНК-субстрата (FLRC, 3G0Q); розовый – каталитически неактивный MutY D144N и ДНК-субстрат (LRC, 1RRQ); голубой – репрезентативный кадр из траектории молекулярной динамики (MD) комплекса MutY и ДНК.

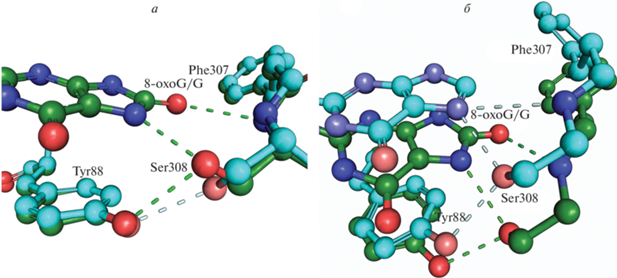
Cтруктура сайта узнавания oxoG
Сайт узнавания oxoG в MutY образован консервативной петлей, содержащей аминокислотные остатки Phe307-Ser308-His309. Остатки Gln48, Thr49, Leu86, Tyr88, Ser308 образуют прямые контакты с oxoG. При этом замена oxoG на G изменяет конформацию остатка Ser308, образующего две водородные связи с атомами N7 и С8 oxoG, и ни одной связи с гуанином (рис. 2). Показано, что замена S308A приводит к пропорциональному снижению сродства фермента и к oxoG:A-, и к G:A-субстратам с сохранением селективности, а для утраты специфичности к oxoG-содержащей ДНК требуется двойная замена F307A/S308A [47]. Однако по результатам молекулярной динамики (MD) в комплексе MutY с ДНК, содержащей G:A-пару, Ser308 также способен поддерживать водородную связь с атомом N7 гуанина за счет подвижности пептидного остова петли. При этом и у фермента дикого типа, и у мутантных форм изменяется конформация Phe307 с вращением боковой цепи на ~90° относительно положения, характерного для моделей с oxoG:A-парой или кристаллографических структур. Таким образом, молекулярно-динамические расчеты свидетельствуют об участии Phe307 в распознавании целевого субстрата.
Структура сайта узнавания 2′-дезоксиаденозина и механизм гидролиза N-гликозидной связи
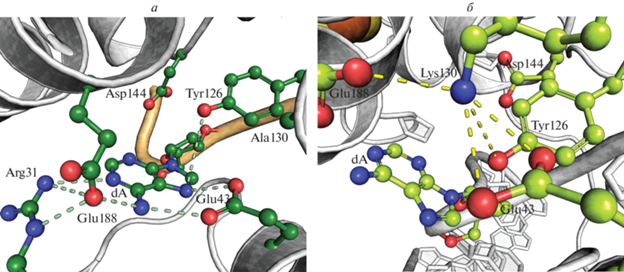
В составе фермент-субстратных комплексов 2′-дезоксиаденозин вывернут из двойной спирали и располагается в активном центре фермента, образованном Arg26, Leu28, Trp30, Arg31, Leu46, Glu43, Val51, Tyr126, Glu188, Ile191, Glu192, (номера аминокислотных остатков соответствуют MutY из G. stearothermophilus) (рис. 3а).
Рис. 3.
Структура сайта узнавания 2′-дезоксиаденозина (а) (FLRC, 3G0Q) и механизм гидролиза N-гликозидной связи (б) [53, Creative Commons Attribution License].

На основании рентгеноструктурных данных и квантовомеханических расчетов предложен каталитический механизм гидролиза N-гликозидной связи 2'-дезоксиаденозина [23, 47, 50, 51]. Предполагается [50], что в активном центре фермента аминокислотный остаток Glu43 находится в нейтральной форме (рис. 3б). Таким образом, опубликованные данные свидетельствуют о том, что в каталитическом комплексе основание Ade протонируется по N7 остатком Glu43, в результате чего происходит разрыв N-гликозидной связи по механизму SN1 [52, 53]. Существование этого механизма подтверждается данными о том, что аналог аденозина, в котором атом N7 заменен на группу C7-H [54], а также замена Glu43 [23, 42] приводят к полной потере каталитической активности MutY.
Выбор аминокислотных остатков активного центра MutY, необходимых для АР-лиазной активности
С целью расширения каталитических свойств фермента при сохранении высокой субстратной специфичности использовали структурную гомологию активного центра монофункционального MutY с бифункциональными ДНК-гликозилазами (рис. 4). Так, мутантную форму MutY S120K (Y126K в MutY G. stearothermophilus) предложили по аналогии с другими представителями структурного семейства спираль-шпилька-спираль (HhH), обладающими не только гликозилазной, но и АР-лиазной активностью – бифункциональными ДНК-гликозилазами hOGG1 и Nth из E. coli [56]. Действительно положение Ser120 в MutY из E. coli соответствует положению каталитических остатков Lys249 у hOGG1 и Lys120 в Nth.
Рис. 4.
Сравнение активных центров комплексов ферментов hOGG1 (зеленый, 3KTU) и MutY G. stearothermophilus (желтый, 3G0Q).

Исходя из стерических соображений предложена замена A124K (A130K в MutY G. stearothermophilus), в которой боковая цепь остатка лизина также расположена в каталитическом центре фермента. Необходимо отметить, что положение Ala130 у MutY G. stearothermophilus соответствует пространственному положению остатка Cys253 ДНК-гликозилазы hOGG1, который стабилизирует боковую цепь каталитического Lys249. При этом фермент с двойной заменой K249C/C253K, сохраняющей стабилизирующее взаимодействие между остатками лизина и цистеина в активном центре hOGG1, обладает частичной каталитической активностью [57].
Создание и анализ модельных структур MutY Y126K и MutY A130K
Для создания модельных структур комплексов мутантных форм MutY Y126K и MutY A130K с ДНК использовали кристаллическую структуру MutY G. stearothermophilus в комплексе с ДНК-субстратом, содержащим 2'-фторо-2'-дезоксиаденозин (PDB ID: 3G0Q) [23].
В полученных нами MD моделях фермента дикого типа сохраняется структура активного центра с координацией N7 атома аденина боковыми цепями аминокислотных остатков Tyr126 и Glu43, что хорошо согласуется с протеканием реакции согласно механизму, предложенному для MutY [50] (рис. 5а). Кроме того, оценка pKa аминокислотных остатков белка алгоритмом H++ также свидетельствует о том, что в составе комплекса фермента дикого типа и мутантной формы Y126K с ДНК Glu43 находится в протонированной форме, но не в случае мутантной формы A130K.
Модельная структура комплекса мутантной формы MutY A130K с ДНК свидетельствует об изменении ориентации ключевых аминокислотных остатков в структуре активного центра за счет создаваемых боковой цепью Lys130 водородных связей с остатками Glu43 и Glu188 (рис. 5б). Формирование таких контактов приводит к отдалению вывернутого аденина от каталитических аминокислотных остатков и должно сопровождаться уменьшением N-гликозилазной активности.
Интересно отметить, что в MD-траекториях комплекса мутантной формы MutY Y126K оптимальная для осуществления N-гликозилазной активности ориентация аминокислотных остатков выявлена лишь на относительно небольшом участке, ~5% от общего времени симуляции (рис. 6а). При этом большую часть симуляционного времени вывернутое основание аденозина находится на большом расстоянии от пары Lys126–Glu43 не позволяющем сформировать прямые контакты, необходимые для осуществления N-гликозилазной активности по механизму, предложенному для MutY [50]. Однако среди возможных состояний взаиморасположения можно выделить состояние, подобное комплексу ДНК-гликозилазы hOGG1 с поврежденной ДНК, где аминогруппа боковой цепи лизина ориентирована в положение, подходящее для протонирования N1 атома вывернутого азотистого основания, что позволяет предположить возможность протекания реакции гидролиза N-гликозидной связи в случае MutY Y126K по пути, описанному для hOGG1 [58] (рис. 6б).
Рис. 6.
Активный центр мутантной формы Y126K. а – Наложение конфигураций активного центра с оптимальным для катализа расположением аминокислотных остатков (зеленый, 5% времени MD траектории) и более устойчивым состоянием (желтый). б – Наложение структуры комплекса hOGG1 с вывернутым oxoG в активном центре (зеленый, 3KTU) и подобной по пространственной организации структуры комплекса MutY Y126K из МD-траектории (желтый).

Каталитические свойства мутантных форм фермента MutY из E. coli, содержащих замены S120K и A124K
С целью подтверждения предположений, полученных с помощью молекулярно-динамических расчетов, экспериментально изучена активность MutY дикого типа и мутантных форм по отношению к ДНК-субстрату, содержащему пару oxoG:A. При этом для регистрации продуктов N-гликозилазной реакции, содержащих АР-сайт, пробы реакционной смеси обрабатывали щелочным раствором, что приводит к расщеплению ДНК. Прямое же разделение реакционной смеси электрофорезом в ПААГ без дополнительной щелочной обработки позволяет зарегистрировать собственную АР-лиазную активность мутантных форм.
Полученные данные свидетельствуют о том, что замена A124K полностью инактивирует фермент. При этом MutY S120K приобрел свойства бифункциональной ДНК-гликозилазы, удаляя аденин и расщепляя полученный АР-сайт. На рис. 7 представлена типовая электрофореграмма и кинетические кривые накопления продуктов N-гликозилазной и АР-лиазной реакций при взаимодействии MutY S120K с ДНК-субстратом. Сравнение с ферментом дикого типа [40, 59] показало снижение N-гликозилазной активности фермента по отношению к паре oxoG:A. Такое уменьшение каталитической активности может объясняться тем, что замена S120K одновременно снижает pKa каталитического остатка Glu43, и аминокислотный остаток Lys120 более не может стабилизировать оксикарбениевый катион-интермедиат [53]. Также необходимо отметить, что в процессе молекулярной динамики исходная ориентация остатков активного центра, необходимая для протекания реакции по механизму, предложенному для MutY дикого типа, сохраняется лишь у ~5% траектории. При этом альтернативное и более устойчивое относительное положение боковой цепи остатка лизина и вывернутого азотистого основания может приводить к возможности разрыва N-гликозидной связи по пути, аналогичному механизму действия гликозилазы hOGG1, пусть и с меньшей эффективностью.
Рис. 7.
Накопление продукта реакции в процессе взаимодействия MutY и ДНК-субстратов, данные электрофореза в ПААГ. а – Гель-электрофореграмма, характеризующая расщепление oxoG:A-субстрата MutY S120K без обработки KOH, над дорожками указано время (с) отбора пробы. Кинетические кривые накопления продуктов расщепления oxoG:A-субстрата, с обработкой (б) и без обработки (в) щелочным раствором.

ЗАКЛЮЧЕНИЕ
Таким образом, проведенный молекулярно-динамический анализ позволил установить природу ключевых взаимодействий в активном центре, обеспечивающих специфическое узнавание повреждения и каталитическую реакцию гидролиза N-гликозидной связи ферментом MutY. Полученные данные позволили предложить замены аминокислотных остатков активного центра фермента, которые помимо гидролиза N-гликозидной связи с аденином, потенциально могут обеспечить появление нового типа каталитической активности, а именно, АР-лиазной и катализировать разрыв 2′-дезоксирибофосфатного остова ДНК в сайте узнавания ферментом. Это означает, что путем направленного мутагенеза монофункциональная ДНК-гликозилаза MutY может быть превращена в бифункциональную ДНК-гликозилазу.
Методом сайт-направленного мутагенеза получены мутантные формы MutY из E. coli, содержащие замены S120K или A124K, и определена их каталитическая активность. Показано, что замена A124K полностью инактивирует фермент за счет нарушения сети контактов в составе активного центра, приводящей к отдалению каталитических остатков от вывернутого азотистого основания. При этом мутантная форма MutY S120K приводит к появлению дополнительной АР-лиазной активности и проявляет свойства бифункциональной ДНК-гликозилазы, удаляя аденин и расщепляя полученный АР-сайт.
Работа выполнена при поддержке Министерства науки и высшего образования (соглашение № 075-15-2021-1085).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Vanella R., Kovacevic G., Doffini V., de Santaella J.F., Nash M.A. (2022) High-throughput screening, next generation sequencing and machine learning: advanced methods in enzyme engineering. Chem. Commun. 58, 2455.
Nikoomanzar A., Chim N., Yik E.J., Chaput J.C. (2020) Engineering polymerases for applications in synthetic biology. Q. Rev. Biophys. 53, 1–31.
Siedhoffa N.E., Schwaneberg U., Davari M.D. (2020) Machine learning-assisted enzyme engineering. in Methods in Enzymology. 643, 281–315.
Kuznetsov N.A., Fedorova O.S. (2020) Kinetic milestones of damage recognition by DNA glycosylases of the helix-hairpin-helix structural superfamily. Adv. Exp. Biol. Med. 1241, 1–18.
Kuznetsova A.A., Fedorova O.S., Kuznetsov N.A. (2022) Structural and molecular kinetic features of activities of DNA polymerases. Int. J. Mol. Sci. 23, 6373.
Yu H., Dalby P.A. (2020) A beginner’s guide to molecular dynamics simulations and the identification of cross-correlation networks for enzyme engineering. In: Methods in Enzymology. 643, 15–49.
Bulygin A.A., Kuznetsova A.A., Vorobjev Y.N., Fedorova O.S., Kuznetsov N.A. (2020) The role of active-site plasticity in damaged-nucleotide recognition by human apurinic/apyrimidinic endonuclease APE1. Molecules. 25, 3940.
Bulygin A.A., Fedorova O.S., Kuznetsov N.A. (2022) Insights into mechanisms of damage recognition and catalysis by APE1-like enzymes. Int. J. Mol. Sci. 23, 4361.
Bowerman S., Wereszczynski J. (2016) Detecting allosteric networks using molecular dynamics simulation. Methods in Enzymology. 578, 429–447.
Tekpinar M., Neron B., Delarue M. (2021) Extracting dynamical correlations and identifying key residues for allosteric communication in proteins by correlation plus. J. Chem. Inf. Model. 61, 4832–4838.
Kladova O.A., Bazlekowa-Karaban M., Baconnais S., Piétrement O., Ishchenko A.A., Matkarimov B.T., Iakovlev D.A., Vasenko A., Fedorova O.S., Le Cam E., Tudek B., Kuznetsov N.A., Saparbaev M. (2018) The role of the N-terminal domain of human apurinic/apyrimidinic endonuclease 1, APE1, in DNA glycosylase stimulation. DNA Repair (Amst). 64, 10–25.
Kladova O.A., Alekseeva I.V., Saparbaev M., Fedorova O.S., Kuznetsov N.A. (2020) Modulation of the apurinic/apyrimidinic endonuclease activity of human APE1 and of its natural polymorphic variants by base excision repair proteins. Int. J. Mol. Sci. 21, 7174.
Smith G.P., Petrenko V.A. (1997) Phage display. Chem. Rev. 97, 391–410.
Ghadessy F., Ong J., Holliger P. (2001) Directed evolution of polymerase function by compartmentalized self-replication. Proc. Natl. Acad. Sci. USA. 98, 4552–4557.
Choi J., Kim H.-S. (2020) Structure-guided rational design of the substrate specificity and catalytic activity of an enzyme. Methods in Enzymology. 643, 181–202.
Au K.G., Cabrera M., Miller J.H., Modrich P. (1988) Escherichia coli MutY gene-product is required for specific A-G-]C.G mismatch correction. Proc. Natl. Acad. Sci. USA. 85, 9163–9166.
Slupska M.M., Baikalov C., Luther W.M., Chiang J.-H., Wei Y.-F., Miller J.H. (1996) Cloning and sequencing a human homolog (hMYH) of the Escherichia coli mutY gene whose function is required for the repair of oxidative DNA damage. J. Bacteriol. 178, 3885–3892.
Back J.H., Park J.H., Chung J.H., Kim D.S.H.L., Han Y.S. (2006) A distinct TthMutY bifunctional glycosylase that hydrolyzes not only adenine but also thymine opposite 8-oxoguanine in the hyperthermophilic bacterium, Thermus thermophilus. DNA Repair. 5, 894–903.
Kunrath-Lima M., Repolês B.M., Alves C.L., Furtado C., Rajão M.A., Macedo A.M., Franco G.R., Pena S.D.J., Valenzuela L., Wisnovsky S., Kelley S.O., Galanti N., Cabrera G., Machado C.R. (2017) Characterization of Trypanosoma cruzi MutY DNA glycosylase ortholog and its role in oxidative stress response. Infect. Genet. Evol. 55, 332–342.
Li X., Lu A.L. (2001) Molecular cloning and functional analysis of the MutY homolog of Deinococcus radiodurans. J. Bacteriol. 183, 6151–6158.
Au K.G., Clark S., Miller J.H., Modrich P. (1989) Escherichia coli mutY gene encodes an adenine glycosylase active on G-A mispairs. Proc. Natl. Acad. Sci. USA. 86, 8877–8881.
Bulychev N.V, Varaprasad C.V, Dorman G., Miller J.H., Eisenberg M., Grollman A.P., Johnson F. (1996) Substrate specificity of Escherichia coli MutY protein. Biochemistry. 35, 13147–13156.
Lee S., Verdine G.L. (2009) Atomic substitution reveals the structural basis for substrate adenine recognition and removal by adenine DNA glycosylase. Proc. Natl. Acad. Sci. USA. 106, 18497–18502.
Cornell W.D., Cieplak P., Bayly C.I., Gould I.R., Merz K.M., Ferguson D.M., Spellmeyer D.C., Fox T., Caldwell J.W., Kollman P.A. (1995) A second generation force field for the simulation of proteins, nucleic acids, and organic molecules. J. Am. Chem. Soc. 117, 5179–5197.
Lindorff-Larsen K., Piana S., Palmo K., Maragakis P., Klepeis J.L., Dror R.O., Shaw D.E. (2010) Improved side-chain torsion potentials for the Amber ff99SB protein force field. Proteins. 78(8), 1950–1958. doi: 1002/prot.22711
Hornak V., Abel R., Okur A., Strockbine B., Roitberg A., Simmerling C. (2006) Comparison of multiple amber force fields and development of improved protein backbone parameters. Proteins. 65, 712–725.
Ivani I., Dans P.D., Noy A., Pérez A., Faustino I., Hospital A., Walther J., Andrio P., Goñi R., Balaceanu A., Portella G., Battistini F., Gelpi J.L., Gonzalez C., Vendruscolo M., Laughton C.A., Harris S.A., Case D.A., Orozco M. (2016) PARMBSC1: a refined force-field for DNA simulations. Nat. Methods. 13, 55–58.
Pérez A., Marchán I., Svozil D., Sponer J., Cheatham T.E., Laughton C.A., Orozco M. (2007) Refinement of the AMBER force field for nucleic acids: improving the description of α/γ conformers. Biophys. J. 92, 3817–3829.
Cheng X., Kelso C., Hornak V., De Los Santos C., Grollman A.P., Simmerling C. (2005) Dynamic behavior of DNA base pairs containing 8-oxoguanine. J. Am. Chem. Soc. 127(40), 13906‒13918. https://doi.org/10.1021/ja052542s
Smith D.M.A., Xiong Y., Straatsma T.P., Rosso K.M., Squier T.C. (2012) Force-field development and molecular dynamics of [NiFe] hydrogenase. J. Chem. Theory Comput. 8, 2103–2114.
Jorgensen W.L., Chandrasekhar J., Madura J.D., Impey R.W., Klein M.L. (1983) Comparison of simple potential functions for simulating liquid water. J. Chem. Phys. 79, 926.
Joung I.S., Cheatham T.E. (2008) Determination of alkali and halide monovalent ion parameters for use in explicitly solvated biomolecular simulations. J. Phys. Chem. B. 112, 9020–9041.
Anandakrishnan R., Aguilar B., Onufriev A.V. (2012) H++ 3.0: automating PK prediction and the preparation of biomolecular structures for atomistic molecular modeling and simulations. Nucl. Acids Res. 40, 537–541.
Abraham M.J., Murtola T., Schulz R., Páll S., Smith J.C., Hess B., Lindah E. (2015) Gromacs: high performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX.https://doi.org/10.1016/j.softx.2015.06.001
Berendsen H.J.C., Spoel D. van der, Drunen R. van (1995) GROMACS: a message-passing parallel molecular dynamics implementation. Comput. Phys. Commun. 91, 43–56.
Essmann U., Perera L., Berkowitz M.L., Darden T., Lee H., Pedersen L.G. (1995) A smooth particle mesh Ewald method. J. Chem. Phys. 103, 8577.
Hess B., Bekker H., Berendsen H.J.C., Fraaije J.G.E.M. (1997) LINCS: a linear constraint solver for molecular simulations. J. Comput. Chem. .https://doi.org/10.1002/(SICI)1096-987X(199709)18:12<1463::AID-JCC4>3.0.CO;2-H
Bussi G., Donadio D., Parrinello M. (2007) Canonical sampling through velocity rescaling. J. Chem. Phys. 126, 014101.
Fasman G.D. (1975) Handbook of Biochemistry and Molecular Biology / 3rd ed. Cleveland: CRC Press, p. 589.
Тюгашев Т.Е., Кузнецова А.А., Кузнецов Н.А., Федорова О.С. (2017) Особенности взаимодействия аденин-ДНК-гликозилазы MutY из E. coli с ДНК-субстратами. Биоорган. химия. 43, 18–28.
Gill S.C., von Hippel P.H. (1989) Calculation of protein extinction coefficients from amino acid sequence data. Anal. Biochem. 182, 319–326.
Guan Y., Manuel R.C., Arvai A.S., Parikh S.S., Mol C.D., Miller J.H., Lloyd R.S., Tainer J.A. (1998) MutY catalytic core, mutant and bound adenine structures define specificity for DNA repair enzyme superfamily. Nat. Struct. Biol. 5, 1058–1064.
Zharkov D.O., Gilboa R., Yagil I., Kycia J.H., Gerchman S.E., Shoham G., Grollman A.P. (2000) Role for lysine 142 in the excision of adenine from A:G mispairs by MutY DNA glycosylase of Escherichia coli. Biochemistry. 39, 14768–14778.
Fromme J.C., Banerjee A., Huang S.J., Verdine G.L. (2004) Structural basis for removal of adenine mispaired with 8-oxoguanine by MutY adenine DNA glycosylase. Nature. 427, 652–656.
Wang L., Lee S.J., Verdine G.L. (2015) Structural basis for avoidance of promutagenic DNA repair by MutY adenine DNA glycosylase. J. Biol. Chem. 290, 17096–17105.
Wang L., Chakravarthy S., Verdine G.L. (2017) Structural basis for the lesion-scanning mechanism of the MutY DNA glycosylase. J. Biol. Chem. 292(12), 5007‒5017. https://doi.org/10.1074/jbc.M116.757039
Russelburg P.L. O′Shea M., Valerie L. Demir M., Knutsen K.R., Sehgal S.L., Cao S., David S.S., Horvath M.P. (2020) Structural basis for finding OG lesions and avoiding undamaged G by the DNA glycosylase MutY. ACS Chem. Biol. 15, 93–102.
Luncsford P.J., Chang D.Y., Shi G., Bernstein J., Madabushi A., Patterson D.N., Lu A.L., Toth E. (2010) A structural hinge in eukaryotic MutY homologues mediates catalytic activity and Rad9-Rad1-Hus1 checkpoint complex interactions. J. Mol. Biol. 403, 351–370.
Nakamura T., Okabe K., Hirayama S., Chirifu M., Ikemizu S., Morioka H., Nakabeppu Y., Yamagata Y. (2021) Structure of the mammalian adenine DNA glycosylase MUTYH: insights into the base excision repair pathway and cancer. Nucl. Acids Res. 49, 7154–7163.
Kellie J.L., Wilson K.A., Wetmore S.D. (2013) Standard role for a conserved aspartate or more direct involvement in deglycosylation? An ONIOM and MD investigation of adenine–DNA glycosylase. Biochemistry. 52, 8753–8765.
Brunk E., Arey J.S., Rothlisberger U. (2012) Role of environment for catalysis of the DNA repair enzyme MutY. J. Am. Chem. Soc. 134, 8608–8616.
McCann J.A., Berti P.J. (2008) Transition-state analysis of the DNA repair enzyme MutY. J. Am. Chem. Soc. 130, 5789–5797.
Woods R.D., O′Shea V.L., Chu A., Cao S., Richards J.L., Horvath M.P., David S.S. (2016) Structure and stereochemistry of the base excision repair glycosylase MutY reveal a mechanism similar to retaining glycosidases. Nucl. Acids Res. 44, 801–810.
Porello S.L., Williams S.D., Kuhn H., Michaels M.L., David S.S. (1996) Specific recognition of substrate analogs by the DNA mismatch repair enzyme MutY. J. Am. Chem. Soc. 118, 10684–10692.
Kaur R., Nikkel D.J., Wetmore S.D. (2020) Computational studies of DNA repair: insights into the function of monofunctional DNA glycosylases in the base excision repair pathway. WIREs Comput. Mol. Sci. 10, e1471.
Ludwig D.L., MacInnes M.A., Takiguchi Y., Purtymun P.E., Henrie M., Flannery M., Meneses J., Pedersen R.A., Chen D.J. (1998) A murine AP-endonuclease gene-targeted deficiency with post-implantation embryonic progression and ionizing radiation sensitivity. Mutat. Res. 409(1), 17‒29. https://doi.org/10.1016/S0921-8777(98)00039-1
Dalhus B., Forsbring M., Helle I.H., Vik E.S., Forstrom R.J., Backe P.H., Alseth I., Bjoras M. (2011) Separation-of-function mutants unravel the dual-reaction mode of human 8-oxoguanine DNA glycosylase. Structure. 19, 117–127.
Sebera J., Hattori Y., Sato D., Reha D., Nencka R., Kohno T., Kojima C., Tanaka Y., Sychrovsky V. (2017) The mechanism of the glycosylase reaction with hOGG1 base-excision repair enzyme: concerted effect of Lys249 and Asp268 during excision of 8-oxoguanine. Nucl. Acid-s Res. 45(9), 5231‒5242. https://doi.org/10.1093/nar/gkx157
Williams S.D., David S.S. (2000) A single engineered point mutation in the adenine glycosylase MutY confers bifunctional glycosylase/AP lyase activity. Biochemistry. 39, 10098–10109.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология