Журнал общей биологии, 2021, T. 82, № 3, стр. 201-228
Рибосомная РНК мечниковеллид в транскриптомах грегарин и рДНК микроспоридий sensu lato в метагеномах
К. В. Михайлов 1, 2, Е. С. Насонова 3, 4, Е. А. Шишкин 3, Г. Г. Паскерова 3, Т. Г. Симдянов 5, В. А. Юдина 5, А. В. Смирнов 3, Я. Янушковец 6, В. В. Алешин 1, 2, *
1 Московский государственный университет им. М.В. Ломоносова, Научно-исследовательский институт
физико-химической биологии им. А.Н. Белозерского
119991 Москва, Ленинские горы, 1, Россия
2 Институт проблем передачи информации им. А.А. Харкевича РАН
127051 Москва, Большой Каретный пер., 19, стр. 1, Россия
3 Санкт-Петербургский государственный университет, биологический факультет, кафедра зоологии беспозвоночных
199034 Санкт-Петербург, Университетская наб., 7/9, Россия
4 Институт цитологии РАН
194064 Санкт-Петербург, Тихорецкий пр., 4, Россия
5 Московский государственный университет им. М.В. Ломоносова, биологический факультет,
кафедра зоологии беспозвоночных
119991 Москва, Ленинские горы, 1, Россия
6 Институт микробиологии Чешской академии наук, Центр Algatech, лаборатория фотосинтеза
37981 Тршебонь, Opatovický mlýn, Чешская Республика
* E-mail: aleshin@genebee.msu.su
Поступила в редакцию 04.11.2020
После доработки 10.02.2021
Принята к публикации 01.03.2021
Аннотация
В метагеномах, транскриптомах и библиотеках ампликонов, используемых для меташтрихкодирования, присутствуют многочисленные нуклеотидные последовательности микроспоридий sensu lato, главным образом относящихся к “Cryptomycota” (Rozellida, Rozellomycota, Rozellosporidia, рассматриваемых здесь как синонимы). В настоящем сообщении мы описываем найденные в транскриптомах одноклеточных из группы Apicomplexa (Alveolata) последовательности рДНК, принадлежащие гиперпаразитическим микроспоридиям-мечниковеллидам. Обнаружено, что транскриптом эугрегарины Polyrhabdina sp. (GenBank SRX6640468) загрязнен кДНК Metchnikovella incurvata, транскриптом архигрегарины Selenidium pygospionis (GenBank SRX6640459) загрязнен кДНК Metchnikovella dogieli, а в транскриптомe бластогрегарины Siedleckia cf. nematoides (GenBank SRX6640464) найдены кДНК, присходящие от вида из неописанного семейства мечниковеллид. С учетом ковариационных нуклеотидных замен смоделирована вторичная структура области “ITS2” идентифицированных и неидентифицированных мечниковеллид. На основе предсказанной вторичной структуры рРНК, картирования чтений из библиотек кДНК, отсутствия эндорибонуклеазы Las1 (PF04031) сделан вывод об отсутствии процессинга ITS2 и наличии ковалентной связи зрелых “5,8S”- и “28S”-подобной (LSU) рРНК мечниковеллид так же, как это известно в отношении других представителей группы типичных микроспоридий. Рассмотрено несколько предложенных ранее (Chytridiopsis typographi, BAQA065) и новых кандидатов на роль сестринской группы микроспоридий sensu stricto. Обсуждается вероятный вклад нейтральных и селективных процессов в эволюцию сверхмалых генов рРНК микроспоридий и сверхдлинных генов рРНК с интронами группы I паразитических и лишайниковых грибов.
Поток молекулярно-генетических данных об эукариотических микроорганизмах, интересных с точки зрения филогенетики, поступает по нескольким направлениям. Наиболее важные сведения доставляет геномика и транскриптомика при выведении новых штаммов в культуру и методом “single cell” при отборе микроманипулятором отдельных клеток некультивируемых видов (Seenivasan et al., 2013; Burki et al., 2013, 2016; Janouškovec et al., 2017; Gawryluk et al., 2019; Strassert et al., 2019; Tikhonenkov et al., 2020a, b, и др.). Но есть и другие источники данных. При исследовании маркерных генов в ДНК, выделенной из проб планктона, почвы, донных осадков и других субстратов (Семенов, 2019), встречаются нуклеотидные последовательности, которые не удается отнести к известным типам (царствам). Согласно эмпирическому правилу (Kim et al., 2016), такие филогенетически изолированные группы представлены видами, редкими в природе. Это доказывается малым числом прочтений в библиотеках и редкостью повторных находок в независимо сконструированных библиотеках. Как выглядят организмы, гены которых нельзя отнести к определенным типам (царствам), неизвестно, поэтому надежды на их скорое выведение в культуру или поимку микроманипулятором для применения “single cell” геномики не так велики. Источником сведений о них остаются данные метагеномики и меташтрихкодирования, а также анализ загрязнений геномов и транскриптомов симбионтами и паразитами (Borner, Burmester, 2017) – то есть тоже, по сути, метагеномов.
В библиотеках ампликонов, получаемых из природных проб, разнообразие нуклеотидных последовательностей превосходит разнообразие, видимое в микроскоп. В природных экосистемах присутствует как бы “темная материя жизни” – некультивируемые в лабораторных условиях микроорганизмы (Filée et al., 2005; Marcy et al., 2007). Значительная часть, иногда более 50% скрытого эукариотического разнообразия – это одноклеточные паразиты и паразитоиды (Lefèvre et al., 2007, 2008; Lepère et al., 2008; Mahé et al., 2017), если судить по немногочисленным идентифицированным родственникам. Так, в пикопланктоне Мирового океана, судя по преобладанию рДНК в ампликонах, от поверхности до абиссали доминируют группы паразитоидов (López-García et al., 2001; Moon-van der Staay et al., 2001; Christaki et al., 2017), названные Marine Alveolata (MALV) Groups I и II (Guillou et al., 2008), которые были идентифицированы как ихтиодиниевые и синдиниевые динофлагелляты, разнообразие и экологическая роль которых до того недооценивались. В библиотеках, полученных из пресноводных и почвенных проб, обычно преобладают гены рРНК грибов и их родственников – других Holomycota (Lepère et al., 2006; Monchy et al., 2011; Nakai et al., 2012; Ishida et al., 2015; Rojas-Jimenez et al., 2019). Изначально самая массовая группа родственников грибов была названа LKM11 (Hannen et al., 1999; Lara et al., 2010), а затем – “скрытыми грибами”, Cryptomycota (Jones et al., 2011a, b). Постепенно выяснилась принадлежность к LKM11 (“криптомикотам”) представителей известного с XIX в. рода Rozella Cornu 1872, включающего внутриклеточных паразитов грибов, оомицетов и одноклеточных водорослей (Gleason et al., 2014; Letcher, Powell, 2018), незадолго до этого на основании молекулярных данных выведенного из состава хитридиевых грибов и сближенного с микроспоридиями (James et al., 2006). Другие известные “криптомикоты” – внутриядерные и цитоплазматические паразиты лобозных амеб, водорослей, членистоногих (дафний, насекомых). Примечательно, что питающиеся клетки видов Rozella, находясь в цитоплазме хозяина, фагоцитируют ее как амебоидные организмы, тогда как их зооспоры похожи на зооспоры хитридиевых (тем, что не питаются, и тем, что несут единственный задний жгутик с прилежащей к базальному телу второй центриолью). К грибам относят мицелиальные и дрожжеподобные формы, питающиеся осмотрофно, и если включать в состав того же таксона представителей Rozella, питающихся фагоцитозом, то таксон “грибы” лишается любых немолекулярных диагностических признаков. Другая группа первоначально неидентифицированных последовательностей рДНК была отнесена к афелидам (Karpov et al., 2013; Letcher et al., 2013), также известным с XIX в. и также незадолго до этого перенесенных из хитридиевых грибов (в этом случае к “корненожкам”, “Rhizopoda”) с присвоением ранга класса (Громов, 2000). Афелиды – внутриклеточные паразитоиды, похожие на Rozella: с трофическими стадиями, питающимися путем фагоцитоза, и непитающимися одножгутиковыми или амебоидными зооспорами (Karpov, Paskerova, 2020). Сходный образ жизни и облик ближайших родственников грибов (афелид и розеллид) поднимает вопрос, насколько на них были похожи предки настоящих грибов, в том числе были ли они паразитами или паразитоидами наземных одноклеточных водорослей (Алешин и др., 2015).
В электронных базах данных за два десятилетия меташтрихкодирования собраны фрагменты маркерных генов (рДНК) тысяч неидентифицированных “криптомикот”, на порядок меньшего числа афелид, и еще на порядок меньшего числа Holomycota, таксономическая привязка которых на сегодня отсутствует (например, представителей клады GS01 (Tedersoo et al., 2017)). Зооспоровые грибы (хитридиевые, бластокладиевые и др.), “криптомикоты”, афелиды и другие микроскопические родственники грибов оказываются на сегодня не учитываемой “темной материей” в наземных сообществах (Grossart et al., 2016). Только для считанного числа “криптомикот”, помимо Rozella, молекулярные данные сочетаются с таксономическим описанием: для возрожденного рода Nucleophaga Dangeard 1895 (Corsaro et al., 2014a), новых родов Mitosporidium, Paramicrosporidium и Morellospora (Haag et al., 2014; Corsaro et al., 2014b, 2020). Все охарактеризованные “криптомикоты” – внутриклеточные паразиты.
Микроспоридии в традиционном понимании (Исси, Воронин, 2007; Исси, 2020; Wadi, Reinke, 2020) – облигатные внутриклеточные паразиты позвоночных и беспозвоночных животных и различных одноклеточных (инфузорий, грегарин, парамиксид). Эта богатая видами группа (описано 1500 видов, и это небольшая часть разнообразия), она, как раньше казалось, ясно выделяется по морфологическим и ультраструктурным признакам: утрата жгутиков и центриолей, канонических митохондрий, видоизменение аппарата Гольджи на цитологическом и биохимическом уровне (утрата везикулярного и клатринового траспорта), наличие спор, снабженных сложным аппаратом для заражения хозяина. После исключения из “споровиков” в старом понимании (объединявшем микроспоридий, миксоспоридий, гаплоспоридий и частично современных Apicomplexa) микроспоридий рассматривали как тип (Sprague, 1977; Weiser, 1977), о родственных связях которого выдвигали самые разные предположения. Высказанная на основе филогенетического анализа шести генов гипотеза о родстве типа микроспоридий и р. Rozella (James et al., 2006) в настоящее время получила убедительную поддержку. Более того, установлено, что многие “криптомикоты” филогенетически еще ближе к микроспоридиям, чем Rozella spp. Таким образом, “криптомикоты” парафилетичны относительно микроспоридий. Если исключить из рассмотрения афелид, то сестринской по отношению к грибам окажется клада, объединяющая микроспоридий с “криптомикотами”. После нескольких взаимоисключающих инициатив по таксономии, пытавшихся уравновесить “царство” грибов, “тип” микроспоридий, р. Rozella и другие роды “криптомикот”, было предложено расширить объем микроспоридий на всех “криптомикот”, за исключением р. Rozella и других филогенетически ранних линий (Bass et al., 2018). Тедерсоо и соавторы (Tedersoo et al., 2018) пошли дальше и в недавней ревизии системы грибов предложили общее название для группы, объединяющей микроспоридий и розеллид (подцарство Rozellomyceta). Проблема наименования подобных филогенетически единых, но структурно гетерогенных крупных групп требует отдельного рассмотрения. В частности, присоединение всех “криптомикот” к микроспоридиям дает общее имя большой монофилетической группе, но и одновременно издержку в виде утраты диагноза таксоном микроспоридий в новом понимании, поскольку он объединяет плезиоморфные жгутиковые и фаготрофные формы, переходные формы и высокоспециализированных типичных микроспоридий. Применение названия “Rozellomyceta” имеет издержку в виде утраты диагноза грибами. Общая проблема плезиоморфных и специализированных видов в одной кладе сформулирована Хеннигом (Hennig, 1966) в “правиле девиации” и В.С. Чуповым (2002) в концепции скрытородственных (криптаффинных) таксонов и неизбежна в рамках филогенетической системы. В рамках нашей работы мы рассматриваем группу, содержащую микроспоридий sensu lato вместе с Rozella, NCLC1 и кладой XI (Lazarus, James, 2015), а наименование “криптомикоты” будем использовать в качестве обиходного для нерангового парафилетического остатка после выделения монофилетической группы типичных микроспоридий.
Для описания истории специализации типичных микроспоридий (sensu stricto) нужно ранжировать скрытородственных (криптаффинных) им “криптомикот” по степени родства. В настоящее время кандидатом на место ближайшего родственника типичных микроспоридий считают Chytridiopsida (Corsaro et al., 2019). Раньше Chytridiopsida относили к типичным микроспоридиям, иногда в составе класса Metchnikovellidea (Weiser, 1977). Другим, дополнительным подходом является изучение наименее специализированных из типичных микроспоридий, к которым относят мечниковеллид (Исси, Воронин, 2007) – гиперпаразитов, известных из грегарин, паразитирующих в полихетах. Мечниковеллиды описаны в конце XIX в., но их принадлежность к микроспоридиям была доказана только с помощью электронной микроскопии (Vivier, 1965), и с тех пор они считаются примитивными представителями микроспоридий в ранге класса Rudimicrosporea (Sprague, 1977) или Metchnikovellidea (Weiser, 1977). От других типичных микроспоридий их отличают менее специализированные споры с короткой полярной трубкой, без поляропласта, якорного диска и задней вакуоли; их жизненный цикл включает, помимо спорогонии, ведущей к свободным спорам, также продукцию спор, лежащих в цистах (эти свойства они разделяют с Chytridiopsida). Гипотеза о начальном расхождении микроспоридий sensu stricto на мечниковеллид и всех остальных подтверждена геномными данными (Mikhailov et al., 2017; Galindo et al., 2018; Nasso-nova et al., 2021). Описаны, однако, и другие микроспоридии, считающиеся примитивными: Buxtehudea, Jiroveciana, Burkea, Hessea (Исси, Воронин, 2007; Larsson, 2014). По ним не имеется молекулярных данных, и далее мы их не обсуждаем, поскольку основной целью нашего обзора являются неидентифицированные и загрязняющие гены рРНК микроспоридий из разных источников.
МАТЕРИАЛЫ И МЕТОДЫ
Базы данных и процедура поиска. Нуклеотидные последовательности извлекали из баз данных NCBI (https://www.ncbi.nlm.nih.gov/). Помимо базы nr (non-redundant, GenBank; Benson et al., 2013), использовали оригинальные транскриптомные данные (Janouškovec et al., 2019), базы SRA (The Sequence Read Archive) (ERX2404075–ERX2404077), содержащие длинные контиги, собранные по результатам высокопроизводительного секвенирования ампликонов (Jamy et al., 2019), и метагеномные данные из wgs (Whole Genome Shotgun), содержащие длинные контиги (Mitchell et al., 2018; Sanchez, Cao, 2019; Cotto et al., 2020; Sharrar et al., 2020; Thornton et al., 2020). Для поиска целевых последовательностей использовали инструменты BLAST (Altschul et al., 1997), в виде входного запроса использовали последовательности 18S рРНК представителей основных групп “криптомикот”, выявленных предыдущими исследованиями.
Филогенетический анализ. Нуклеотидные последовательности рРНК добавляли к подготовленному ранее выравниванию как к профилю, используя MUSCLE (Edgar, 2004). Участки, выравнивание которых проблематично, перед построением дерева удаляли вручную. Байесовы деревья строили с помощью MrBayes 3.2.5 (Ronquist et al., 2012), используя модель нуклеотидных замен GTR (General Time-Reversible) (Tavaré, 1986) с учетом неравномерности сайтов по скорости эволюции согласно гамма-распределению, аппроксимируемому 10, 12 или 16 категориями, с использованием ковариационной модели эволюции или без таковой. Число независимых запусков, число Марковских цепей Монте-Карло (MCMC) в каждом запуске и число генераций варьировали. Деревья визуализировали с помощью FigTree (Rambaut, 2010) и MEGA (Kumar et al., 2016).
Структурный анализ. Картирование сырых чтений для измерения покрытия вдоль транскриптов мечниковеллид выполнено Bowtie 2 (Langmead, Salzberg, 2012) с параметрами “–end-to-end –no-mixed –no-discordant”. Данные покрытия извлечены из bam файлов выравниваний чтений при помощи BEDTools (Quinlan, Hall, 2010) с параметрами “-pc” для расчета общего покрытия по фрагментам (т.е. от прямого и до обратного чтения) и “–5 -strand +” для подсчета точек начала фрагментов. В контрольном опыте библиотеку SRA: SRR10440982 картировали на оперон рРНК Saccharomyces cerevisiae S288C (NC_001144.5: 451786-457732). В моделях вторичной структуры рРНК большой и малой субчастиц использовали известную номенклатуру (Wuyts et al., 2001); структуру спиралей 19–21 корректировали по модельным видам: инфузории Tetrahymena thermophila и дрожжам S. cerevisiae (Lee, Gutell, 2012). Неканонические пары, в том числе подтвержденные рентгено-структурным анализом и криоэлектронной микроскопией рибосом модельных видов, не учитывали; напротив, комплементарные пары на концах спиралей включали в предсказанные спирали, даже если такое расширение спиралей не характерно для большинства видов. Локальное сворачивание РНК предсказывали с помощью сервера Mfold (Zuker, 2003) с параметром учета субоптимальных структур (15%). Использовали как текущую версию, предсказывающую структуры при 37°C, так и версию 2.3 с возможностью выбора температуры (в этом случае выбирали 15°C). В предсказании структуры ITS2 использовали, насколько это было возможно, модель “четырех доменов” с характерными элементами в спиралях II и III (Joseph et al., 1999; Schultz et al., 2005; Coleman, 2007, 2015). В случае анализа региона, соответствующего редуцированному ITS2 у мечниковеллид, использовали программу RNAalifold (Bernhart et al., 2008) в составе ViennaRNA web-сервиса (Gruber et al., 2015), учитывающую информацию о выравнивании первичных структур. Визуализировали модели с помощью RnaViz 2 (Rijk et al., 2003).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Мечниковеллиды. При анализе результатов высокопроизводительного секвенирования кДНК эугрегарин, архигрегарин и бластогрегарин (Janouškovec et al., 2019) в трех библиотеках обнаружены последовательности, принадлежащие микроспоридиям – мечниковеллидам. Здесь мы описываем контиги рРНК, хотя многие белок-кодирующие транскрипты мечниковеллид также могут быть собраны из этих библиотек. Мечниковеллиды из эугрегарины Polyrhabdina sp. на основании сходства нуклеотидных последовательностей (GenBank QXFS01001040, QXFS01000707; Galindo et al., 2018) отнесены к виду Metchnikovella incurvata Caullery et Mesnil 1914, известному из этого хозяина (Sokolova et al., 2013; Rotari et al., 2015). Мечниковеллиды из архигрегарины Selenidium pygospionis на основании сходства нуклеотидных последовательностей отнесены к виду Metchnikovella dogieli Paskerova et al., 2016 – обычному паразиту S. pygospionis (GenBank MT969020; Paskerova et al., 2016, 2018; Nassonova et al., 2021). В бластогрегаринах мечниковеллид ранее не находили; нуклеотидные последовательности, найденные в библиотеке кДНК (GenBank GHVV01457926, GHVV01457913, GHVV01457924) – первая такая находка. РНК для синтеза кДНК была выделена из нескольких десятков клеток бластогрегарин, извлеченных из кишечника морских политет Scoloplos cf. armiger и формально определенных как Siedleckia nematoides. Однако полиморфизм белок-кодирующих транскриптов в библиотеке говорит в пользу того, что выборка клеток бластогрегарин представлена смесью видов-двойников. Если это так, то на сегодня хозяина правильнее обозначить Siedleckia cf. nematoides. Близкого сходства рРНК мечниковеллиды из S. cf. nematoides с какими-либо известными нуклеотидными последовательностями не обнаружено (ее принадлежность мечниковеллиде обосновывается построенным филогенетическим деревом). В библиотеках из природных субстратов не найдено последовательностей рРНК мечниковеллид (за исключением контаминанта, номер в GenBank KX214678). С другой стороны, в трех видах грегарин из пяти, исследованных методом высокопроизводительного секвенирования (Janouškovec et al., 2019), обнаружены мечниковеллиды. По-видимому, разнообразие и распространенность мечниковеллид недооцениваются, в том числе и при анализе ампликонов рДНК, получаемых из проб морских осадков.
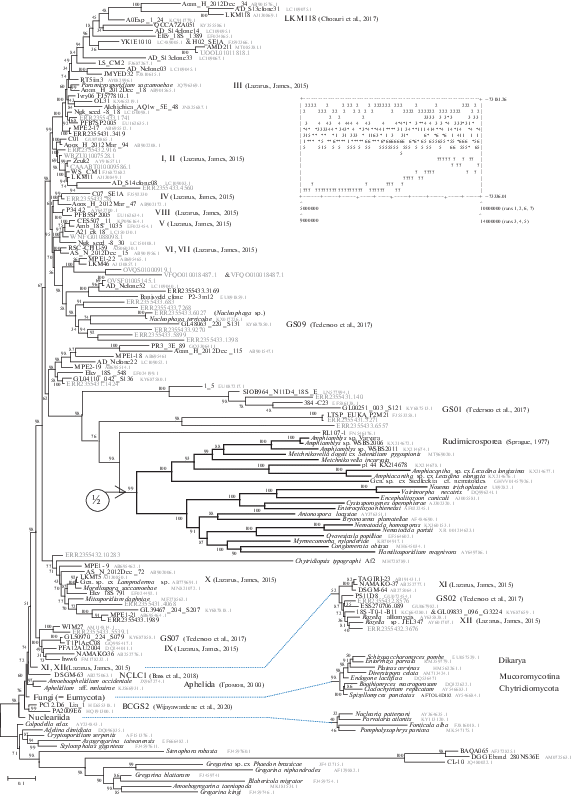
Мечниковеллиды образуют на филогенетическом дереве РНК малой субчастицы рибосом монофилетическую группу, сестринскую остальным типичным микроспоридиям (рис. 1). Этот результат повторяет ранее опубликованныe (Mikhailov et al., 2017; Bass et al., 2018; Galindo et al., 2018; Corsaro et al., 2019, 2020; Nassonova et al., 2021). Виды Amphiacantha обособлены от Metchnikovella spp. и Amphiamblys spp. (p-дистанци, вычисленные для выравниваемых участков рРНК, – около 0.3), на основании чего можно принять выделение для них двух отдельных семейств: Amphiacanthidae и Metchnikovellidae (Larsson, 2000, 2014). Паразит бластогрегарин образует на филогенетическом дереве третью ветвь мечниковеллид, обособленную от Amphiacanthidae и Metchnikovellidae.
Рис. 1.
Байесово дерево РНК малой субчастицы рибосом микроспоридий. Представлен результат лучшего запуска “run2” (ngen = 10 000 000, nchains = 8, nst = 6, ngammacat = 10, covarion = yes). На диаграмме рассеяния представлены значения логарифма правдоподобия в семи лучших запусках (из 14). Длина ветвей в поддереве типичных микроспоридий сокращена вдвое (широкие линии). Апостериорные вероятности даны в процентах. Сходимость топологий не достигнута, в частности, некоторые клады с высокой апостериорной вероятностью в независимых запусках отсутствуют в настоящем дереве, и наоборот. Вероятные артефакты положения клад BAQA065 и GS01 обсуждаются в тексте.
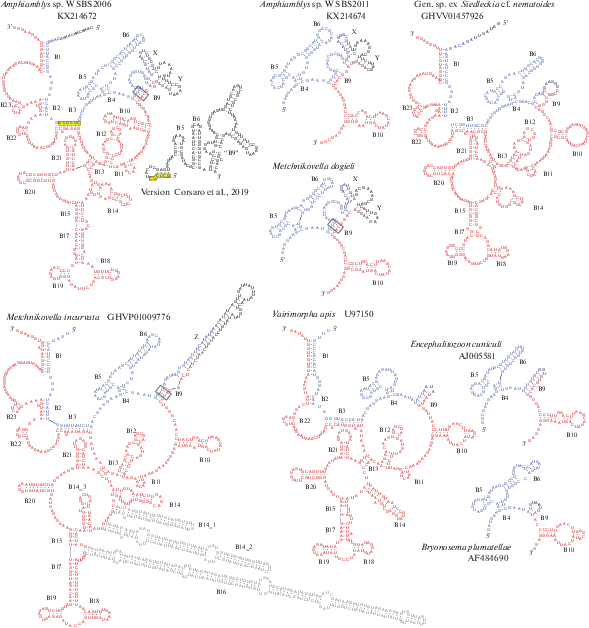
Одна из особенностей микроспоридий sensu stricto – соединение в единую молекулу зрелых 5,8S- и 28S-подобных рРНК (отсутствие самостоятельной 5,8S рРНК, так же как у прокариот) (Vossbrinck, Woese, 1986). У типичных микроспоридий нет консервативных элементов ITS2, считающихся важными для процессинга (Coleman, 2015). У других эукариот с ITS2 граничат шпильки B7 и B8 – консервативные элементы 5,8S рРНК. Шпильки B7 и B8 отсутствуют у типичных микроспоридий, а у некоторых отсутствует и шпилька B6 (рис. 2) (Rijk et al., 1998; Peyretaillade et al., 1998; Van de Peer et al., 2000). У мечниковеллид мы предсказываем шпильку B6 и не находим шпилек B7 и B8, тогда как Корсаро и соавторы (Corsaro et al., 2019, fig. 5) гомологизируют спирали B6 Amphiamblys sp. и B8 Chytridiopsis typographi, называя их “GC-богатым стеблем” (GC-rich stem). Возникает вопрос: каким реконструкциям больше доверять? Возможность альтернативных, при этом близких по значениям свободной энергии образования вариантов делают предсказания для отдельных видов сомнительными, а вариабельность рассматриваемой области затрудняет привлечение удаленных видов для выявления совпадающих элементов. Но для мечниковеллид в целом можно заметить три пары компенсаторных нуклеотидных замен в стебле B4 относительно Vairimorpha apis и две пары компенсаторных замен в стебле B9 у Metchnikovella incurvata относительно M. dogieli и Amphiamblys spp. (рис. 2, четырехугольник). Эти компенсаторные замены позволяют определить положение спиралей B4 и B9. У мечниковеллиды из бластогрегарин стебель B9 оканчивается простой петлей, так же как у других типичных микроспоридий. У остальных мечниковеллид это место “ITS2” занято специфическими структурными элементами, не вписывающимися, однако, в модель “четырех доменов”. Инструмент RNAalifold способен подогнать предсказанные структуры трех видов к общей для них модели в виде “огородного пугала” (рис. 2), а для M. incurvata можно предполагать экспансию в это место нуклеотидных последовательностей, как и во многие другие места рДНК этого вида. Не ясно, как модель, сгенерированная RNAalifold, соотносится с моделью “четырех доменов” и насколько она окажется устойчивой к новым данным.
Рис. 2.
Предсказанная вторичная структура слитных “5,8S”- и “28S”-подобной рРНК типичных микроспоридий. Участки последовательностей, гомологичные 5,8S рРНК, отмечены синим, 28S рРНК – красным; границы 5,8S и 28S рРНК приходятся на участок спирали B9, и неопределенность положения границ отмечена градиентом цвета. Специфические для мечниковеллид спирали обозначены x, y, z. Две пары компенсаторных замен в B9 между Metchnikovella incurvata, M. dogieli и Amphiamblys spp. выделены серым прямоугольником. Часть контакта спирали B3, отнесенная Корсаро и соавт. (Corsaro et al., 2019, fig. 5) к внутримолекулярной спирали в пределах 5,8S рРНК, выделена желтым; обозначения спиралей B5, B6 и “B9” на модели Корсаро и соавторов добавлены нами. Структура для Encephalitozoon cuniculi и Vairimorpha apis (родовое название согласно последней ревизии (Tokarev et al., 2020)) приведена по ранним моделям (Peyretaillade et al., 1998; Rijk et al., 1998; Van de Peer et al., 2000) с незначительными изменениями. Номенклатура спиралей рРНК по: Wuyts еt al., 2001.
Мечниковеллиды сохраняют в модифицированном виде консервативный мотив ggyryryygtttGAGtrtcr вблизи 3'-границы 5,8S рДНК (рис. 3). Обычно этот участок комплементарен 5'-концевой области 28S рРНК, и вместе они составляют основание спирали B9. Но у мечниковеллид ему просто нет комплементарных нуклеотидов в ближайшей окрестности. И в модели “огородного пугала”, и в модели Корсаро и соавторов (рис. 2) консервативная последовательность мечниковеллид, гомологичная 3'-концу 5,8S рРНК, предсказана не в составе спирали, как у других видов, а частично в виде одноцепочечной петли. Помимо мечниковеллид, рассматриваемый участок, как кажется, занимает нестандартное положение во вторичной структуре рРНК и некоторых других видов, например Chytridiopsis typographi. По-видимому, функция 3'-концевого участка 5,8S рРНК не ограничивается связыванием с 28S рРНК для образования спирали B9, но также предполагает связывание и с альтернативным (неизвестным) лигандом. Иначе парные компенсаторные замены постепенно разрушили бы консервативный мотив, оставив за счет компенсаторных замен только комплементарность нитей B9 подобно тому, как это наблюдается в вариабельных спиралях ITS2. У других типичных микроспоридий утрачивается и эта (неизвестная) функция вместе с соответствующей нуклеотидной последовательностью. Вероятно, двойственная природа свойственна многим спиралям рРНК, попеременно выступающим либо как двойная спираль, либо как специфическая последовательность нуклеотидов, что обеспечивает в эволюции не только компенсаторные замены, но и длительное сохранение первичных структур.
Рис. 3.
Выравнивание нуклеотидных последовательностей, образующих основание спирали B9. Пропущенные нуклеотиды ITS2 и дистальной части B9 заменены многоточием. Комплементарные нуклеотиды спирали B9 дрожжей и Amphiamblys sp. соединены пунктирными дугами. Невыравниваемая область дана на сером фоне.

Возникает вопрос, обеспечивают ли своеобразные элементы на месте соединения 5,8S и остальной части РНК большой субчастицы рибосом процессинг пре-рРНК у мечниковеллид. Область “ITS2” трех видов мечниковеллид, для которых доступны библиотеки кДНК, покрыта отдельными чтениями на уровне, близком к покрытию соседних структурных элементов, тогда как при процессинге ITS2 наблюдается снижение количества кДНК и числа чтений в этой области, как видно на примере контроля – библиотеки кДНК дрожжей (рис. 4). У мечниковеллид не видно признаков конца матриц в области “ITS2”, тогда как на месте стыка ITS1 и 5,8S такие признаки видны по резкому изменению уровня покрытия на границе (рис. 4) и по нуклеотидным последовательностям – адаптерам – в значительной доле чтений (рис. 5). У мечниковеллиды Amphiamblys sp. не обнаружен ген эндорибонуклеазы Las1 (PF04031), участвующей в процессинге ITS2. Этого гена нет в известных геномах микроспоридий sensu stricto, но он имеется в геномах “криптомикот” с протяженной областью ITS2 – Mitosporidium daphniae и Paramicrosporidium saccamoebae. Таким образом, совокупность косвенных данных свидетельствует об отсутствии процессинга “ITS2” у мечниковеллид и о том, что у них “5,8S”- и “28S”-подобная рРНК слиты в зрелом состоянии в единую молекулу подобно тому, как это имеет место у других микроспоридий sensu stricto.
Рис. 4.
Покрытие чтениями высокопроизводительного секвенирования кДНК области соединения “5,8S”- и “28S”-подобной рРНК микроспоридий-мечниковеллид и подвергаемых процессингу ITS1 и ITS2 дрожжей Saccharomyces cerevisiae. Верхние диаграммы для каждого организма показывают покрытие чтениями из транскриптомных библиотек; нижние диаграммы показывают отношение точек начала чтений к общему покрытию; предсказанные области генов 5,8S (синяя) и 28S (красная) рРНК показаны цветом ниже диаграмм.
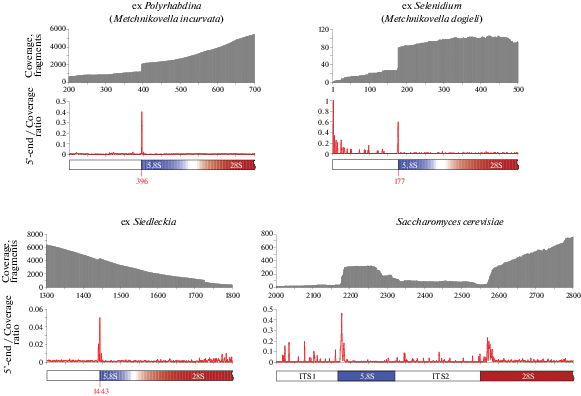
Рис. 5.
Адаптеры на 5'-конце 5,8S рРНК микроспоридии-мечниковеллиды Metchnikovella dogieli. Адаптеры свидетельствуют об обрыве матрицы на 5'-конце 5,8S рРНК многих матриц (в результате процессинга ITS1). Видна вариабельность 5'-конца (AA, либо AAA, либо AAAA). Аналогичных массовых обрывов в месте соединения “5,8S”- и “28S”-подобной рРНК мечниковеллид не наблюдается, что свидетельствует об отсутствии процессинга ITS2.

Проблема Chytridiopsis typographi. Род Chytridiopsis Schneider, 1884 известен с XIX в. и включает паразитов кишечного эпителия насекомых (жуков, ручейников). Традиционно его рассматривали в составе микроспоридий, а в некоторых системах сближали с мечниковеллидами (Weiser, 1977; Larsson, 1993). Как и мечниковеллиды, Chytridiopsida отличаются спорами с короткой полярной трубкой (однако более длинной, нежели у мечниковеллид, формирующей несколько витков и покрытой сотовидной структурой) и неразвитым поляропластом. В жизненном цикле два этапа спорогонии, приводящей либо к спорам, свободно лежащим в паразитофорной вакуоли, либо к спорам, заключенным в общую цисту.
Анализ нуклеотидных последовательностей 18S рРНК первого исследованного представителя рода, Chytridiopsis typographi (паразита жука-короеда Ips typographi), вывел его за пределы типичных микроспоридий (включая мечниковеллид), поставив сестринской группой (Corsaro et al., 2019). Однако по общей архитектуре 18S рРНК C. typographi больше похожа на типичную эукариотическую рРНК. Области V2, V3, V4, V7, V8, V9 в рРНК C. typographi не укорочены столь существенно, как у типичных микроспоридий (Barandun et al., 2019). У типичных микроспоридий даже в наименее редуцированном состоянии из области V4 утрачен участок комплементарности с областью V2 (спирали 23/e13 и 23/e14). В 18S рРНК C. typographi предсказывается (рис. 6) “эукариотический” контакт V2–V4 (Alkemar, Nygård, 2003). Как и у большинства эукариот, первичная структура области V4 C. typographi допускает миграцию ветвей между спиралями 23/e13 и 23/e14 (Wuyts et al., 2000). В зависимости от этой миграции, образование псевдоузла V2–V4 кажется скорее одной из возможностей (даже серией возможностей) “дышащей” структуры, нежели “замороженным” состоянием, которое можно предполагать из кристаллизованных рибосом или данных криоэлектронной микроскопии (Lee, Gutell, 2012; Petrov et al., 2014). Протяженная область несовершенной комплементарности между участками V2 и V4 (рис. 6, выделение шрифтом) свидетельствует скорее в пользу перехода in vivo между несколькими (более чем двумя) альтернативными состояниями, чем исключительно kissing-loops между небольшими терминальными петлями двух стабильных шпилек “21es6d” и “9” (рис. 6, версия 4).
Рис. 6.
Области V2 и V4 18S рРНК Chytridiopsis typographi и двух типичных микроспоридий. Вероятная зона контакта V2–V4 (различные версии) выделена шрифтом. * – пары, предсказанные из ковариации, но не подтвержденные структурами модельных видов; # – неканонические пары, введенные для согласования с моделью (Lee, Gutell, 2012).
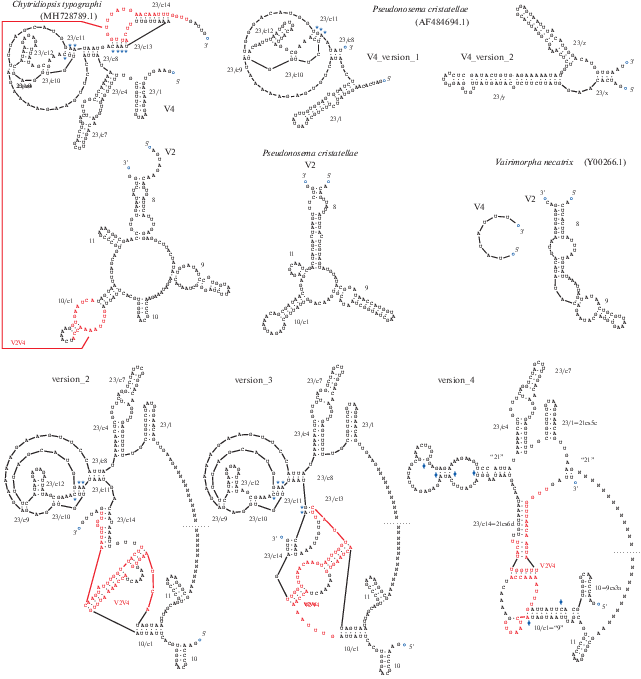
В отличие от типичных микроспоридий, у C. typographi имеются шпильки B7 и B8 в 3'-области 5,8S рРНК, а размер и структура ITS2 близки к типичным. Из спектра предсказанных структур ITS2, близких по свободной энергии образования, можно подобрать структуру из трех шпилек, из них вторая (по порядку и по длине) имеет внутреннюю U-U-петлю, как положено канонической шпильке II (разные версии см. на рис. 7 и у Корсаро и соавт. (Corsaro et al., 2019, fig. 5)). Шпилька IV отсутствует, так же как, по-видимому, у Paramicrosporidium. Отсутствие шпильки IV не мешает процессингу ITS2 у дрожжей S. cerevisiae (Coleman, 2015). Для верификации моделей вторичных структур ITS2 “криптомикот” необходимы сведения о близких видах и компенсаторных заменах в ITS2. В целом можно предполагать у C. typographi близкую к типичной структуру ITS2, процессинг и вследствие этого раздельные 5,8S и 28S рРНК. Плезиоморфии C. typographi в 18S рРНК, 5,8S рРНК и ITS2 не отвергают гипотезу о сестринских отношениях с микроспоридиями sensu stricto, для обоснования которой необходимы свидетельства (синапоморфии), которые пока не предъявлены.
Рис. 7.
Фрагмент пре-рРНК (5,8S–ITS2–B-домен 28S рРНК) двух видов “криптомикот”. Предсказанные шпильки I, II, III соответствуют спейсеру (ITS2).
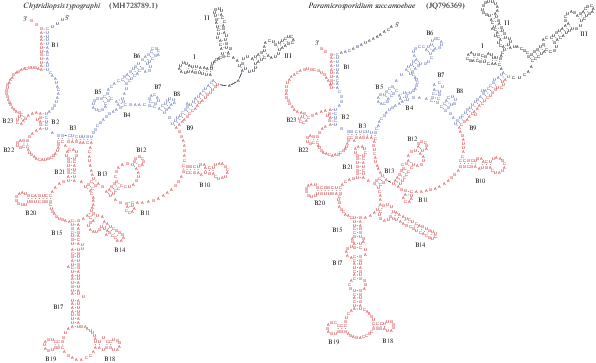
Не обнаружив в структуре рРНК на уровне петель и спиралей синапоморфий C. typographi с типичными микроспоридиями, мы отмечаем многочисленные аутапоморфии: делеции в областях спиралей 23, 24, 27, 28, 29 в 18S рРНК, частично указанные раньше (Corsaro et al., 2019). Часть из них затрагивает одну ветвь спирали (23, 24) при сохранении типичного размера и нуклеотидной последовательности другой ветви. Такие нарушения желательно подтвердить независимым исследованием других представителей Chytridiopsida. Помимо этого, в первичной структуре рРНК C. typographi фиксировано множество нуклеотидных замен, приводящих к тому, что ветвь C. typographi на дереве рРНК микроспоридий одна из длиннейших: следующая по длине после ветвей микроспоридий sensu stricto. В объединении длинных ветвей всегда можно подозревать артефакт притяжения длинных ветвей (Felsenstein, 1978; Hendy, Penny, 1989), и нужны веские основания, чтобы исключить такую возможность в отношении C. typographi и микроспоридий sensu stricto. Мы не смогли добиться схождения цепей и стабильной топологии дерева рРНК микроспоридий. Только часть элементов дерева обнаруживала стабильную поддержку, и объединение C. typographi с типичными микроспоридиями не входило в их число. В отдельных цепях (MCMC) и даже отдельных целых запусках при построении байесового дерева 18S рРНК мы наблюдали включение C. typographi в кладу, ранее обозначенную номером X (Lazarus, James, 2015). Аналогичные результаты получены нами при анализе последовательностей гена 28S рРНК. В эту же группу X входит Mitosporidium daphniae и Morellospora saccamoebae – другие “криптомикоты” с неподвижными спорами с полярной трубкой и без жгутиковых зооспор в жизненном цикле. Включения C. typographi в другие группы с неподвижными спорами – III (Paramicrosporidium) и Nucleopha-ga spp. – мы не наблюдали. Таким образом, мы не нашли подтверждения сестринских отношений C. typographi и типичных микроспоридий и считаем этот вопрос подлежащим дальнейшему исследованию. Обостряется также вопрос о месте на филогенетическом дереве других родов (Intexta, Nolleria), относимых в настоящее время к Chytridiopsida, но пока не изученных молекулярно-генетическими методами.
Вид Chytridiopsis typographi традиционно включали в состав микроспоридий, но филогенетически он входит в одну из клад грады “криптотомикот”, иллюстрируя проблематичность разграничения на уровне морфологических и ультраструктурных признаков микроспоридий sensu stricto и “криптомикот”.
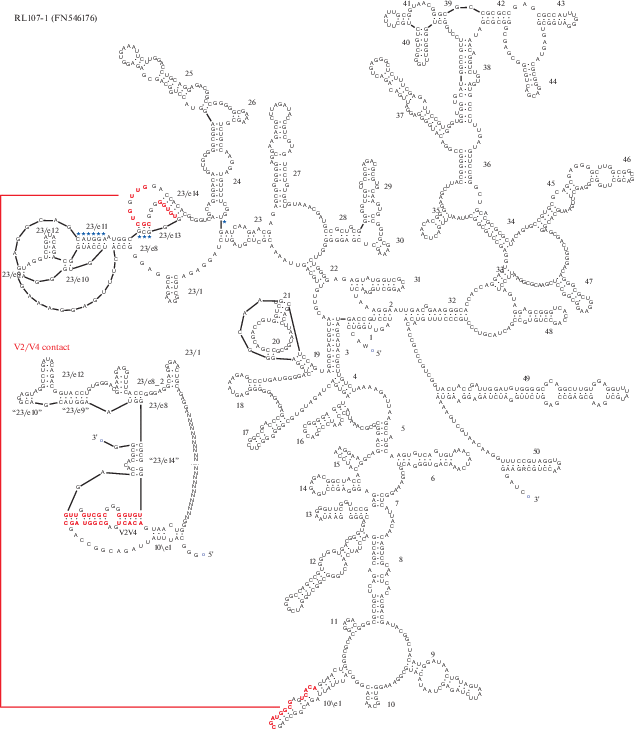
Проблема BAQA065 (GenBank AF372825). В одной из первых работ по меташтрихкодированию (Dawson, Pace, 2002) была амплифицирована последовательность BAQA065 из ДНК, выделенной из донного осадка мелководной солоноватоводной лагуны (Беркли, США). Согласно доступной на то время технике построения деревьев, BAQA065 на сконструированном дереве оказалась между эукариотической “кроной” и длинными ветвями микроспоридий, дипломонад и трихомонад (“Archezoa”). За прошедшие 19 лет 5'-конец BAQA065 верифицирован двумя независимыми находками похожих последовательностей: в глубоких слоях прибрежного осадка Северного моря и в содовом озере в Индии (фрагмент DGGE band 280NS36E, GenBank AM072563 (Wilms et al., 2006) и CL-10, GenBank JQ480022 (Antony et al., 2013)); 3'-конец к настоящему времени не верифицирован, что согласуется с редкостью в библиотеках ампликонов генов рРНК, не идентифицированных до типа (царства) (Kim et al., 2016) (и, наверное, редкостью в природе несущих их клеток). В более поздних работах клада BAQA065 представлялась сестринской типичным микроспоридиям (Mikhailov et al., 2017, fig. S1 ), более близкой, чем Chytridiopsis typographi (Corsaro et al., 2019, 2020; Nassonova et al., 2021). В отличие от C. typographi, рРНК малой субчастицы BAQA065 имеет размер, близкий к “прокариотическому”, с мозаикой предковых признаков (не свойственных типичным микроспоридиям) и признаков типичных микроспоридий. К первым относится сохранение двух длинных шпилек (17 и 18) в области V3 и шпильки 46 в области V8. Общие с типичными микроспоридиями признаки – резко уменьшенные области V2, V4, V7 (рис. 8). Область V4 сокращена примерно вдвое по сравнению с типичной для эукариот. При отсутствии сходных последовательностей с компенсаторными заменами, нам не удалось восстановить такие предсказанные элементы области V4, как псевдоузлы (Wuyts et al., 2000). Однако большое число альтернативных вариантов укладки свидетельствует в пользу того, что предсказанная для области V4 молекулярная динамика (Wuyts et al., 2000) сохраняется в каком-то виде у BAQA065. Например, “слабые” пары AU и GU составляют 50% на неоднозначно укладываемом участке спирали (рис. 8, варианты 1 и 2), тогда как на общем участке на “слабые” пары приходится 30%, а 70% – на “сильные” GC пары. Не исключено взаимодействие V2–V4, так как между ними имеется протяженный участок с частичной комплементарностью.
Рис. 8.
Предсказанная структура рРНК малой субчастицы клона BAQA065. Отсутствующий 5'-конец дополнен за счет клона DGGE band 280NS36E, таким образом, спирали 2–6 гибридные. Вероятная зона контакта V2–V4 выделена шрифтом и соединена линиями, а также дублирована на врезке. * – пары, предсказанные из ковариации, но не подтвержденные структурами модельных видов (Lee, Gutell, 2012).

По первичной структуре консервативного участка в основании спирали 49 клон BAQA065 похож не на Opisthokonta (Cavalier-Smith, Chao, 2003; Алешин и др., 2007), а на эугрегарин сем. Gregarinidae (рис. 9). Кроме Gregarinidae, такой же мотив имеется у представителя другого семейства эугрегарин, Ascogregarina taiwanensis (Actinocephalidae). Другие виды актиноцефалид не похожи на Gregarinidae. Этот пример показывает, что гомоплазия по рассматриваемой области – это явление хотя и редкое, но возможное. Таким образом, BAQA065 сочетает альтернативные сигналы: “микроспоридий” (в виде общей апоморфной архитектуры рРНК) и “эугрегарин сем. Gregarinidae” (в виде короткого специфического мотива). В таксономических наборах без эугрегарин программы построения деревьев ставят BAQA065 ближайшей к типичным микроспоридиям ветвью на сконструированных деревьях (Mikhailov et al., 2017; Corsaro et al., 2019, 2020; Nassonova et al., 2021). В присутствии Gregarinidae в независимых цепях BAQA065 занимает альтернативные положения. Из шести независимых запусков (из четырех Марковских цепей Монте-Карло каждый), в двух запусках клада BAQA065 группировалась с типичными микроспоридиями (апостериорная вероятность 0.8 и 0.94), а в четырех – с эугрегаринами сем. Gregarinidae (апостериорная вероятность от 0.91 до 1.0). Естественно, статистика независимых запусков не имеет значения для выбора истинного дерева, но она указывает на конфликт сигналов и ошибочную реконструкцию по крайней мере в некоторых запусках. В отсутствие BAQA065 родственные короткие последовательности CL-10 и DGGE band 280NS36E, лишенные проблемного 3'-конца, занимают место сестринской группы типичных микроспоридий с апостериорной вероятностью, близкой к 1.0 (дерево не показано), и не группируются с эугрегаринами. Несмотря на прогресс в технике филогенетического анализа рРНК, положение BAQA065 остается неопределенным. Нельзя исключить технической причины противоречий – химерности BAQA065 (Berney et al., 2004). Как отмечено выше, проблемный 3'-конец BAQA065 пока не верифицирован. Другая возможная причина – какой-то из вариантов молекулярной гомоплазии. Согласно одному варианту, эволюционно ранние микроспоридии sensu stricto – BAQA065 – наряду с множеством других аутапоморфий изменили консервативный мотив в спирали 49. Этот вариант согласуется с положением на дереве CL-10 и DGGE band 280NS36E в отсутствие BAQA065 вблизи микроспоридий s. s. и поднимает вопрос о причине эволюционной консервативности небольшого элемента в масштабе всех Opisthokonta и обстоятельствах прекращения этого состояния у отдельных видов (BAQA065). Согласно другому варианту, сходные сокращения тех же самых вариабельных областей рРНК (V2, V4, V7) произошли у филогенетически неродственных эукариот: Holomycota (типичные микроспоридии) и Gregarinidae (BAQA065); и тут поднимается вопрос о функциональных особенностях рибосом без этих элементов. Необходимо отметить, что сильное сокращение областей V2, V4, V7, а нередко также V3 и V9, свойственно не только микроспоридиям, но и неродственным паразитическим видам: дипломонадам, парабазалиям, асцетоспоридиям р. Mikrocytos, грибам р. Neozygites (рис. 10), а в рРНК некоторых других паразитических видов (в роде архигрегарин Selenidium, роде нематод Pelodera) эти области слегка сокращены. У наиболее специализированных паразитов – митохондрий – рРНК сокращена максимально – у многоклеточных животных, кинетопластид, споровиков в 2–3 раза относительно микроспоридий. Рибосомы с такими рРНК способны к синтезу белка, но разнообразие мРНК, которые им приходится транслировать, в тысячи раз меньше, чем разнообразие мРНК в цитоплазме.
Рис. 9.
Фрагмент выравнивания гена РНК малой субчастицы рибосом Opisthokonta (включая микроспоридий), споровиков (включая эугрегарин) и последовательностей BAQA065 и RL107-1. Специфическое для Opisthokonta состояние дано на сером фоне, специфическое для Gregarinidae – на черном фоне. Многоточие символизирует пропущенные нуклеотиды.

Рис. 10.
Предсказанная структура рРНК малой субчастицы Neozygites. Паразитические грибы Neozygitomycetes (Entomophthoromycotina) не родственны микроспоридиям (White et al., 2006), но у тех и других происходит сходное (хотя и не идентичное) сокращение вариабельных областей рРНК (Freimoser et al., 2000).
RL107-1 (GenBank: FN546176). Если не учитывать кладу BAQA065, то наиболее близкий родственник типичных микроспоридий – организм RL107-1 (рис. 1). Его ДНК добыта из сильно закисленной воды затопленного угольного карьера в Германии с очень бедным эукариотическим населением (Huss, Bauer, 2011). По размеру и другим признакам РНК малой субчастицы рибосом RL107-1 оказывается промежуточной между типичными эукариотическими 18S рРНК и рРНК микроспоридий “прокариотического” типа. Спирали 46 и 17 имеются (последняя укороченная). Спираль 43 укороченная, в области V4 полностью утрачена спираль 23/e4–23/e7, а спираль 23/1 сокращена до 4 п. н. Несмотря на сокращение областей V2 и V4, возможность контакта между ними за счет участка комплементарности РНК у RL107-1, в отличие от типичных микроспоридий, сохраняется (рис. 11). Такой контакт, скорее всего, реализуется не на постоянной основе, а как один из вариантов внутримолекулярных перестроек рРНК (Wuyts et al., 2000), так же как и у других эукариот. Таким образом, RL107-1 ближе и филогенетически, и по структурным признакам рРНК к типичным микроспоридиям, чем Chytridiopsis typographi.
Рис. 11.
Предсказанная структура рРНК малой субчастицы клона RL107-1. Вероятная зона контакта V2–V4 выделена шрифтом и соединена линиями, а также дублирована на врезке. * – пары, предсказанные из ковариации, но не подтвержденные структурами модельных видов (Lee, Gutell, 2012).
Не имеется никаких сведений о строении и образе жизни RL107-1, а также о том, получена их ДНК из вегетирующих в кислотном озере или покоящихся (занесенных с берега) клеток. Неизвестно, происходит последовательность RL107-1 от неописанного организма или принадлежит какому-то известному виду примитивных микроспоридий, для которого еще не определена нуклеотидная последовательность рДНК. В библиотеках из других природных субстратов не найдено рДНК, похожих на RL107-1.
Другие вероятные родственники типичных микроспоридий. Реконструкция филогенетического дерева микроспоридий sensu lato находится на самой начальной стадии. Эта группа сестринская грибам, а значит, по возрасту столь же древняя, как царство грибов, и столь же, если не более, дифференцированная на уровне первичных структур. По гетерогенности скоростей молекулярной эволюции она охватывает, кажется, весь известный для эукариотической жизни диапазон, поскольку включает наиболее быстро эволюционирующих типичных микроспоридий, рРНК которых столь видоизменена, что раньше ее выводили за пределы эукариотического типа (Vossbrinck et al., 1987). Однозначное выравнивание некоторых участков рРНК невозможно. Таксономическая выборка микроспоридий крайне разрежена, что приводит к недооценке генетических расстояний и дополнительно усложняет задачу реконструкции. Для многих групп микроспоридий высокого ранга известны только фрагменты генов рРНК – другие маркеры пока не доступны. Вряд ли генов рРНК будет достаточно для достоверной реконструкции всех узлов филогенетического дерева микроспоридий, особенно при таких осложняющих обстоятельствах.
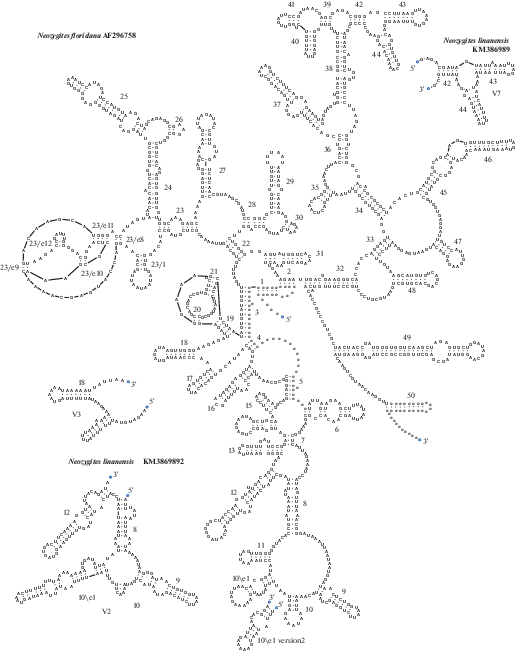
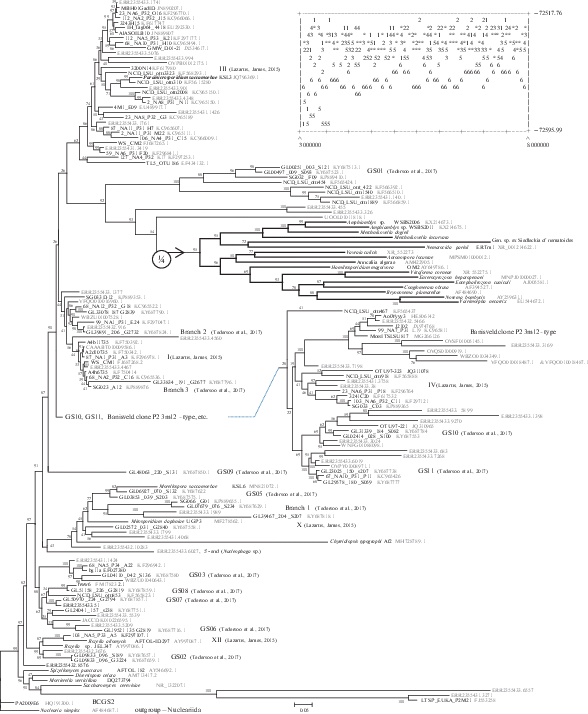
В составе “криптомикот” найдено до двух десятков клад (Lazarus, James, 2015; Chouari et al., 2017; Matsubayashi et al., 2017; Tedersoo et al., 2017), объединение которых в более крупные, за немногими исключениями, недостоверно. В частности, не известно, какие клады ближе к микроспоридиям sensu stricto. На сконструированных деревьях, как правило, такими оказываются, в разных комбинациях, наиболее длинные ветви: Chytridiopsis typographi, Nucleophaga spp., Paramicrosporidium spp., LKM118, LKM15, MPE1-21, клада VIII, клон Banisveld P2-3m12, GS03 (клон GL48063.220.S131) и др. (Corsaro et al., 2016, 2019, 2020; Grossart et al., 2016; Stentiford et al., 2017; Tedersoo et al., 2017; Bass et al., 2018). На сконструированных нами деревьях большинство ранее намеченных кандидатов заняли близкое к типичным микроспоридиям положение в отдельных запусках программы MrBayes. Однако в других, независимых запусках любой из кандидатов оказывался далеко от типичных микроспоридий, причем альтернативные положения получали высокие значения апостериорной вероятности в конкретных запусках. Таким образом, в наших реконструкциях филогении по генам рРНК не была достигнута сходимость топологий, несмотря на большое число генераций MCMC (более 10 млн). Мы использовали большое число категорий (10, 16) и наборы из более чем 150 операциональных таксономических единиц для учета гетерогенности сайтов рРНК по скорости эволюции. Большое число категорий создает опасность так называемой перепараметризации и плохой сходимости. Однако использование малого числа категорий приводило к низкому разрешению (низким значениям апостериорных вероятностей многих клад). Варьирование параметра “temp” (в диапазоне от 0.02 до 0.5) также не позволило достичь сходимости. По-видимому, для схождения топологии дерева 18S рРНК требуются многие десятки миллионов генераций MCMC.
В некоторых работах по меташтрихкодированию получены большие массивы данных (Mueller et al., 2014; Taylor et al., 2014; Timling et al., 2014) или единичные последовательности (Smith et al., 2007; Taylor et al., 2007, 2008; Eichorst, Kuske, 2012; McGuire et al., 2013; Lipson et al., 2014; Wurzbacher et al., 2014; Page, Flannery, 2018) ITS и фрагментов (менее 1 т. п. н.) гена 28S рРНК “криптомикот”. В байесовом дереве выбранных из них представителей (рис. 12) расположение крупных групп “криптомикот” во многом совпадает с таковым на дереве 18S рРНК, о чем можно судить по ампликонам, перекрывающим оба гена (Weber et al., 2009; Tedersoo et al., 2017; Jamy et al., 2019), или по длинным контигам из метагеномных сборок (Mitchell et al., 2018; Sanchez, Cao, 2019; Cotto et al., 2020; Sharrar et al., 2020; Thornton et al., 2020). При построении дерева 28S рРНК возникают проблемы со сходимостью топологии, подобно тому как и с 18S рРНК. Некоторые особенно длинные ветви не находят правильного положения. Например, одна из ранних клад Holomycota, GS01 (Tedersoo et al., 2017), на дереве 28S рРНК ответвляется вблизи корня типичных микроспоридий (рис. 12) вместо того, чтобы группироваться с BCGS2, как наблюдается в некоторых наших запусках и на опубликованных деревьях (Wijayawardene et al., 2020). Положение GS01 можно отнести за счет артефакта притяжения длинных ветвей, который проявляется на деревьях 18S рРНК и 28S рРНК, однако некоторые другие различия между деревьями 18S рРНК и 28S рРНК, возможно, обусловлены дополнительным филогенетическим сигналом, позволяющим лучше разрешить некоторые узлы по 18S рРНК, а другие – по 28S рРНК.
Рис. 12.
Байесово дерево конкатенированных 5,8S и 28S рРНК микроспоридий, включая “криптомикот” (консенсус из четырех лучших запусков, nst = 6, ngammacat = 10, rates = invgamma, temp = 0.1, ngen = 8 000 000). На диаграмме рассеяния значения логарифма правдоподобия в шести лучших запусках (из 12). Апостериорные вероятности выражены в процентах. Длины ветвей в поддереве типичных микроспоридий сокращены в 4 раза (широкие линии).
Адаптация методик высокопроизводительного секвенирования для получения протяженных контигов и сборка метагеномов открывают новые перспективы меташтрихкодирования и филогенетического анализа благодаря более полному учету разнообразия и привлечению связанных данных по генам 18S рРНК и 28S рРНК без выделения лабораторных культур. В единичном исследовании с применением высокопроизводительного секвенирования (Jamy et al., 2019) обнаружены десятки операциональных таксономических единиц “криптомикот”, представляющих большинство известных до того по рДНК клад, но также и некоторые новые, не встреченные прежде. Часть новых клад представлена на дереве 18S рРНК особо длинными ветвями, из которых ERR2355431.3271 и ERR2355433.6557 ближе к микроспоридиям sensu stricto, чем Chytridiopsis typographi и все другие ранее намеченные кандидаты (по результатам по крайней мере некоторых запусков программы MrBayes), но дальше, чем RL107-1 (рис. 1). Для одной из них, ERR2355431.3271, депонированы более ранние находки (номера в GenBank FJ553258.1, FJ553594.1) – внутренний транскрибируемый спейсер с небольшими флангами генов (Hartmann et al., 2009). По общим признакам рДНК (размер областей V2, V3, V4, V7 18S рРНК, шпильки B7 и B8 в 3'-области 5,8S рРНК, ITS2) эти организмы сохраняют плезиоморфное состояние. Они не разделяют аутапоморфий C. typographi в организации рДНК и не группируются с этим видом на дереве. Другой кандидат, представленный контигом UOOL01011818 из метагенома активного ила (Mitchell et al., 2018), проявляет большее тяготение к типичным микроспоридиям на дереве большой, нежели малой субчастицы (рис. 1, 12). Возможно, некоторые из нынешних кандидатов в самом деле близкие родственники микроспоридий sensu stricto, но необходима более придирчивая проверка этой гипотезы для каждого кандидата. Объединение данных по двум генам рРНК перспективно для филогенетики, но требует проверки депонированных контигов. Например, контиг ERR2355433.6027 на участке, соответствующем 18S рРНК, на 95% идентичен Nucleophaga terricolae KTt-1, и на 94% идентичен N. amoebae KTq-2, и доставляет, по-видимому, сведения о 5'-конце гена 28S рРНК Nucleophaga sp. (рис. 12). Однако примерно 1000 нуклеотидов гена 28S рРНК на 3'-конце контига происходят скорее всего от одного из видов тихоходок, с генами которых они сходны. Успешное выявление подобных химер ограничено недостатком сведений о нуклеотидных последовательностях гена 28S рРНК.
Проблема скоростей молекулярной эволюции. Рибосомы типичных микроспоридий сильно отличаются от рибосом других эукариот. Значительные отличия от “типичной” рРНК в первичной, а иногда и вторичной структуре обнаружены и в некоторых других таксонах (у многих или у всех исследованных представителей). Это экскаваты (Giardia lamblia, Trichomonas vaginalis и многие другие), асцетоспоридии (Mikrocytos mackini, Marteilia и др.), фораминиферы, некоторые Amoebozoa, гемоспоридии и некоторые другие споровики (Plasmodium, Trichotokara, Trollidium, Cephaloidophoroidea и др.), некоторые животные (миксоспоридии, дициемиды, ортонектиды, веерокрылые насекомые, многие нематоды, из которых выделяются роды Pelodera, Riouxgolvania) и др. (Leipe et al., 1993; Smothers et al., 1994; Pawlowski et al., 1994, 1996; Chalwatzis et al., 1995; Fitch et al., 1995; Katiyar et al., 1995; Whiting et al., 1997; Carnegie et al., 2003; Rueckert et al., 2011, 2013; Hasegawa et al., 2012; Wakeman, 2020). Не все, но многие из них – паразиты. Возникает вопрос, почему в этих группах сильно изменены рибосомы. Хотя рибосомы выполняют и некоторые специфические функции, все же синтез белка – их главная и универсальная функция, и трудно представить, чтобы естественный отбор сильно модифицировал рибосомы микроспоридий или других организмов для выполнения какой-то особой функции. Предположение об адаптивности массовых отличий в рРНК не вписывается в базовую концепцию о преобладающей роли нейтральных событий в молекулярной эволюции (Кимура, 1985). У видов с сильно измененными рРНК обычно сильно изменены и другие универсальные молекулярные машины – эволюционно консервативные структурные белки и ферменты (элементы цитоскелета, аппарата репликации ДНК, ферменты метаболизма и др.). Такая картина свидетельствует о повышенной скорости молекулярной эволюции не только рДНК, а большой части генома в некоторых группах родственных видов, и должна быть общая причина, ускоряющая их молекулярную эволюцию на протяжении длительного времени, которую хотелось бы узнать.
Внутриклеточные паразиты живут “на всем готовом”, забирают многие метаболиты у хозяина вместо самостоятельного их биосинтеза. Гены, ответственные за синтез этих веществ, освобождаются от отбора, под действием мутаций изменяются и в конце полностью утрачиваются из генома. Но это обстоятельство как будто не должно прямо сказаться на функции рибосом и скорость их эволюции. На паразитов действуют, однако, и другие общие факторы, например, снижающие эффективную численность (Ne): низкая численность, ограниченная численностью хозяина; периодические резкие спады численности согласно модели Лотки–Вольтерры; ограничение панмиксии из-за родственных скрещиваний в субпопуляциях в теле одного хозяина; распространенность бесполого размножения – адаптация паразита к низкой интенсивности инвазии. Нейтральный дрейф должен идти с одинаковой скоростью в малых и больших популяциях: быстрота фиксации в одних компенсируется пропорционально большим числом мутаций в других (Кимура, 1985). Но слабовредные (подверженные отбору) в больших популяциях мутации при сниженной эффективной численности (для коэффициента отбора s при достижении порога |s| ≤ 1/(2Ne)) становятся эффективно нейтральными, т.е. фиксируются с динамикой нейтральных (Кимура, 1985). Они увеличивают пул “нейтральных” мутаций в малых популяциях. Снижение частоты полового процесса за счет бесполого размножения у паразитов замедляет комбинирование, в том числе появление нескольких слабовредных мутаций у одного индивида, а значит, замедляет “очищающий” отбор против слабовредных мутаций и дополнительно повышает вероятность их фиксации. Микроспоридии представляются живым примером реализации “храповика Меллера”, постепенно и необратимо разрушающего их консервативные молекулярные структуры, в том числе рибосомы (Melnikov et al., 2018a), причем снижение эффективности трансляционного аппарата микроспоридий подтверждается экспериментально обнаруженным снижением точности трансляции (Melnikov et al., 2018b). Для исправления дефектных белков нужны дополнительные затраты на синтез новых молекул взамен или затраты АТФ на работу шаперонов. Хотя паразит черпает ресурсы из хозяина, кажется, что вместо того, чтобы исправлять за их счет ошибки трансляции, он мог бы направить эти ресурсы на приготовление нескольких дополнительных спор и повысить таким образом число потомков, т.е. повысить свою приспособленность и выиграть (при определенном соотношении s и Ne) у конкурентов с менее точным аппаратом трансляции.
Если причина высокой скорости эволюции микроспоридий в паразитизме, то поднимаются два других вопроса. Все известные микроспоридии sensu lato – внутриклеточные паразиты. Хотя бесспорная реконструкция образа жизни их ближайшего общего предка пока не выполнена, наиболее экономной (на основе имеющихся данных) кажется гипотеза, что он был внутриклеточным паразитом (Алешин и др., 2015). Но тогда почему за одно и то же время эволюции от общего предка “храповик Меллера” почти “разрушил” рибосомы типичных микроспоридий и мало повлиял на рибосомы Rozella и других “криптомикот”? На этот вопрос пока нет ответа. Второй вопрос: почему типичные микроспоридии с “оптимизированными”, почти разрушенными рибосомами находятся в состоянии биологического прогресса?
Сверхмалые и сверхдлинные гены рРНК. Типичные микроспоридии – обладатели сверхмалых рРНК. Среди дальних родственников микроспоридий – настоящих грибов – сильно укороченные и крайне дивергентные рРНК известны только у похожих на микроспоридий по образу жизни видов Neozygites (Entomophthoromycotina: Neozygitomycetes) – паразитов мелких членистоногих (рис. 10) (Freimoser et al., 2000; Delalibera et al., 2004; Zhou et al., 2017). Зато многие лишайниковые и паразитические грибы обладают сверхдлинными генами рРНК (DePriest, Been, 1992; Gargas et al., 1995). Удлинение генов происходит за счет самосплайсирующихся интронов группы I. Для интронов группы I достоверно не известно о кодируемых ими микроРНК или других “полезных” для хозяина генах или регуляторных элементах, как это свойственно сплайсосомным интронам в мРНК (обзор: Chorev, Carmel, 2012). Интроны группы I внедряются только в консервативные участки рДНК, удаляются при созревании рРНК, в рибосому не попадают, и первичные структуры таких рРНК не отличаются чрезмерной скоростью эволюции. Интроны группы I встречаются у разных эукариот (Rogers, 2019), в том числе и свободноживущих, но интенсивность и экстенсивность заселения ими генов рРНК не одинаковая. Например, они обычны в генах рРНК лишайниковых водорослей. В отличие от лишайниковых грибов, многие водоросли живут как в лишайниковой ассоциации, так и в свободном состоянии. Высказана гипотеза о переносе интронов – взаимном перезаражении интронами паразитов и хозяев в составе ассоциации, но она в целом не получила поддержи, так как в большинстве водорослевые интроны похожи на другие водорослевые, а грибные – на другие грибные (Bhattacharya et al., 1996, 2002; Karpov et al., 2019). На рРНК приходится 50–80% всей транскрипции в клетке млекопитающих и дрожжей (Paule, 1998; Warner, 1999; Moss, Stefanovsky, 2002); уровень синтеза рРНК влияет на физиологию клетки, скорость роста (продолжительность клеточного цикла), деление и дифференцировку (Warner, 1999; Russell, Zomerdijk, 2005; Noack Watt et al., 2016). При этом уровень синтеза рРНК, как было многократно показано, лимитируется копийностью генов рРНК (Stevenson, Schmidt, 2004; Rogers, 2019, и др.), следовательно, можно ожидать, что из того же числа генов рРНК, “нагруженных” интронами, в единицу времени получится меньше зрелой рРНК (для интронов группы I нет наблюдений, что их наличие повышает уровень экспрессии, как это неоднократно сообщалось для сплайсосомных интронов в мРНК). У отдельных видов лишайниковых и паразитических грибов на интроны группы I приходится до половины длины генов рРНК (Gargas et al., 1995; Karpov et al., 2017), и, значит, на транскрипцию интронов уходит до 20–30% от всех затрат на транскрипцию. Возникает вопрос, значимы ли эти энергетические затраты и можно ли экспансию интронов группы I списать на случайный дрейф нейтрального признака. Пусть роскошь содержания интронов группы I перекладывается на хозяина, но почему бы паразиту не избавиться от транскрипции бесполезных участков ДНК, повысив вместо этого продукцию функциональных молекул, и в конечном итоге числа спор, т.е. числа потомков, что равносильно повышению приспособленности? В дрожжевой клетке S. cerevisiae около 2 × 105 рибосом. При активном росте дрожжи делятся каждые полтора часа, таким образом, синтезируется не мене 33 копий рРНК в секунду на клетку (Warner, 1999), и эта величина заниженная, так как не учитывает распада рибосом. Принимая цену синтеза рибонуклеотидтрифосфата в 12 макроэргических связей для клетки, анаэробно сбраживающей глюкозу на минимальной среде (Wagner, 2005), получаем цену транскрипции среднего интрона группы I в 12 × 350 × 33 = 1.4 × × 105 макроэргов в секунду на клетку, или около 1.4 ×105/1.34 × 107 = 0.01 от общих энергетических затрат клетки дрожжей на транскрипцию (Wagner, 2005), что с точностю до порядка совпадает с оценкой, получаемой просто из соотношения длин одного интрона к пре-рРНК и вклада рРНК в общую траскрипцию. Необходимо отнести эти затраты к общим энергетическим затратам клетки. По имеющимся оценкам, затраты на синтез РНК составляют от 5 до 10% затрат на синтез белка (Wagner, 2005), а вместе на синтез РНК и белка приходится до 76.6% от общего расхода АТФ клетки (Förster et al., 2003). Таким образом, на транскрипцию одного интрона группы I расходуется около 3.8 × 10–4 от энергетичеких затрат клетки дрожжей. В случаях, когда скорость роста лимитируется доступной энергией (питательными веществами), это приведет к замедлению скорости роста (производства потомков) на ту же величину 3.8 × 10–4. Для диких видов Saccharomyces эффективная численность Ne оценивается в 1.36 × 107 (Wagner, 2005), следовательно, для гаплоидной популяции при превышении порогового значения коэффициента отбора |s| = 0.73 × 10–7 признак не может быть нейтральным, и его судьба в популяции будет определяться преимущественно отбором, а не случайным дрейфом. “Цена” даже одного интрона группы I превышает пороговое значeние s для дрожжей примерно на три порядка, следовательно, существование интронов в рДНК нельзя объяснить дрейфом нейтрального признака, по крайней мере для частоты более чем 0.1%. Применительно к другим видам можно ожидать иных значений коэффициента отбора s. Например, паразиты, импортирующие нуклеотиды или их предшественники из цитоплазмы хозяина, могут вместо затрат АТФ на полный синтез обходиться затратами на работу переносчиков, снизив таким образом расходы на трансляцию на порядок, а это уже способно объяснить выживание обладателя одного интрона в рРНК за счет случайности у 1% видов при том же значении Ne. И, что еще важнее, рост и размножение паразита, да и вообще любого вида, может лимитироваться не питательными веществами, а каким-то отдельным метаболитом (витамином, незаменимой аминокислотой, незаменимой жирной кислотой и т.п.) или конкретным минеральным веществом. В таком случае пропорциональность значений s и энергозатрат на размножение нарушится. Наконец, при низкой эффективной численности (Ne) меняются пороговые значения s, что способно сделать присутствие интронов не столь невероятным даже при лимитировании размножения пищей.
В отличие от гипотезы исходного морфологического многообразия (Мамкаев, 1968), интуитивно понятно превращение деградирующих органов или макромолекул в разнообразные “останцы” из-за разрушения и модификации несущественных участков. Например, у слепых видов наблюдаются самые разные дефекты глаз. Сверхмалые и сверхбольшие гены рРНК кажутся разными вариантами закрепления путем дрейфа слабовредных признаков (Melnikov et al., 2018b). Однако широкое распространение модификации генов рРНК у паразитов поднимает вопрос, не скрыта ли в этих модификациях адаптивная компонента. Ориентируясь на расчеты выше, мы можем предположить, что сокращение длины рРНК у микроспоридий дает им существенную экономию в транскрипции, которая с лихвой перекрывает затраты на снижение точности трансляции (тогда возникает вопрос, почему другие виды не “оптимизируют” рибосому). Паразитам обычно выгодна продукция большого числа яиц или спор. Но могут быть ситуации, когда единовременной массовой продукцией не в сезон не перекрыть убыль спор. И не всегда вероятность заражения привязана простым способом к сезону, времени года. Хозяева иногда, как 17-летние цикады, замедляют прохождение своего жизненного цикла для того, чтобы рассинхронизировать его с циклами паразитов. На примере умеренных бактериофагов известно, что лизогения может быть не менее эффективной стратегией паразита, чем продуктивная инфекция. Сверхмалые рибосомы микроспоридий по причине сниженной эффективности трансляции (Melnikov et al., 2018b), а сверхбольшие гены рРНК паразитических грибов по причине избыточной нагрузки на аппарат транскрипции, вероятно, замедляют развитие паразита и растягивают продукцию спор на более длительный срок. Мы наблюдали длительное (многолетнее) сосуществование в лабораторной пробирке нитчатой желто-зеленой водоросли Tribonema gayanum и убивающего ее клетки паразита (паразитоида) Sanchytrium tribonematis с сильно интронированными генами рРНК (до 6 интронов в гене 18S рРНК, до 9 интронов в гене 28S рРНК), хотя зависимость скорости развития от числа интронов группы I не изучали. Аргументом против связи экспансии интронов группы I с паразитическим образом жизни можно считать присутствие интронов в генах рРНК свободноживущих видов. И хотя интроны у них встречаются не часто (~10–3–10–2 “в среднем” на вид) и число интронов редко превышает 1–2 на ген рРНК, но по крайней мере в одной группе Amoebozoa – у миксомицетов – интронов не меньше, если не больше (и по доле видов с интронированными генами, и по числу интронов на ген), чем у паразитических грибов. Трудно придумать причину, по которой бы миксомицетам требовалось конститутивно замедлять жизненный цикл. Предполагаемые способы замедления развития за счет интронов или дефектов рибосом не регулируемы и не несут заметной выгоды при наличии тонких способов регуляции синтеза рРНК (Warner, 1999; Moss, Stefanovsky, 2002; Engel et al., 2013; Torreira et al., 2017; Fernández-Tornero, 2018), в том числе на отдельных стадиях роста или в рамках quorum sensing (Najmi, Schneider, 2021) и с точностью, направляющей клеточную дифференцировку в эмбриональном развитии (Noack Watt et al., 2016), регуляции биогенеза рибосом (Chaker-Margot, 2018) и регуляции работы готовых рибосом (Усачев и др., 2020), известной в том числе и у микроспоридий (Barandun et al., 2019). Наконец, длительная жизнеспособность спор может быть альтернативой или хорошим дополнением к растянутой по времени продукции спор. В метагеномах обнаруживается большое разнообразие генов рРНК “криптомикот”, но только немногие из них массовые в конкретных библиотеках. Не исключено, что разнообразные, но малочисленные виды генов рРНК происходят из покоящихся стадий, еще не погибших в ожидании своего хозяина. И все же идеальная придуманная организация не обязательно реализуется в природе. Реальное соотношение деструктивных процессов за счет “храповика Меллера” и адаптаций к паразитизму в генах рРНК микроспоридий требует дальнейшего изучения.
Мы очень мало знаем о реальной и эффективной численности микроорганизмов. Население “криптомикот” в наземных и морских биотопах, согласно меташтрихкодированию ампликонов рДНК, обильное и разнообразное (Lepère et al., 2006; Monchy et al., 2011; Nakai et al., 2012; Grossart et al., 2016; Rojas-Jimenez et al., 2017, 2019; Arroyo et al., 2018), тогда как типичные микроспоридии при использовании этой методики находятся гораздо реже. Насколько соотношение генов рРНК в библиотеках отражает реальное соотношение жизнеспособных клеток в природе и какая часть ампликонов происходит из вегетирующих клеток, спор и ДНК мертвых клеток? Вывод о низкой численности типичных микроспоридий в природе противоречит мнению об их биологическом прогрессе, но объяснил бы стремительную деградацию их геномов за счет “храповика Меллера”. Однако такой вывод был бы преждевременным, поскольку применяемые методики меташтрихкодирования способны приводить к недоучету типичных микроспоридий из-за селекции ампликонов по размеру (отличающему рДНК типичных микроспоридий, но не “криптомикот”, от типичных эукариот), а также из-за высокого уровня отличий нуклеотидных последовательностей, снижающего эффективность отжига “универсальных” праймеров и распознавания определенных последовательностей как рДНК. Адаптация методик меташтрихкодирования для типичных микроспоридий поможет их более адекватному учету (Trzebny et al., 2020). С другой стороны, аллельный полиморфизм зависит от эффективной численности (Кимура, 1985) и может оказаться простым и хорошим средством ее измерения в эпоху активного накопления геномных данных. Такие оценки необходимы для более предметного обсуждения причин разной скорости эволюции “криптомикот” и типичных микроспоридий.
Авторы благодарны д-ру Ф.М. Фреймозеру за любезные консультации, Российскому научному фонду за финансирование исследования библиотек кДНК эугрегарин, архигрегарин и бластогрегарин (грант № 18-14-00123) и Российскому фонду фундаментальных исследований за поддержку работ по прояснению разнообразия и филогении одноклеточных консументов одноклеточных (грант № 18-04-01210).
Список литературы
Алешин В.В., Константинова А.В., Михайлов К.В., Никитин М.А., Петров Н.Б., 2007. Нужно ли много генов для филогенетического дерева? // Биохимия. Т. 72. № 12. С. 1610–1623.
Алешин В.В., Михайлов К.В., Карпов С.А., 2015. О происхождении и ранней эволюции грибов и микроспоридий // Сборник научно-популярных статей и фотоматериалов – победителей конкурса РФФИ 2015 года. Вып. 18. М.: МОЛНЕТ. С. 215–223.
Громов Б.В., 2000. Паразиты водорослей из группы “Монад” Ценковского родов Aphelidium, Amoeboaphelidium и Pseudaphelidium как представители нового класса // Зоол. журн. Т. 79. № 5. С. 517–525.
Исси И.В., 2020. Развитие микроспоридиологии в России // Вестник защиты растений. Т. 103. № 3. С. 161–176.
Исси И.В., Воронин В.Н., 2007. Тип Microsporidia Balbiani, 1882 – Микроспоридии // Протисты: руководство по зоологии. Ч. 2 / Под ред. Алимова А.Ф. СПб.: Наука. С. 994–1045.
Кимура М., 1985. Молекулярная эволюция: теория нейтральности. М.: Мир. 394 с.
Мамкаев Ю.В., 1968. Сравнение морфологических различий в низших и высших группах одного филогенетического ствола // Жypн. oбщ. биoлогии. Т. 29. № 1. С. 48–56.
Семенов М.Е., 2019. Метабаркодинг и метагеномика в почвенно-экологических исследованиях: успехи, проблемы и возможности // Журн. общ. биологии. Т. 80. № 6. С. 403–417.
Усачев К.С., Юсупов М.М., Валидов Ш.З., 2020. Гибернация – стадия функционирования рибосом // Биохимия. Т. 85. № 11. С. 1690–1700.
Чупов В.С., 2002. Форма боковой филогенетической ветви у растений // Успехи соврем. биологии. Т. 122. № 3. С. 227–238.
Alkemar G., Nygård O., 2003. A possible tertiary rRNA interaction between expansion segments ES3 and ES6 in eukaryotic 40S ribosomal subunits // RNA. V. 9. № 1. P. 20–24.
Altschul S.F., Madden T.L., Schäffer A.A., Zhang J., Zhang Z. et al., 1997. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs // Nucleic Acids Res. V. 25. № 17. P. 3389–3402.
Antony C.P., Kumaresan D., Hunger S., Drake H.L., Murrell J.C., Shouche Y.S., 2013. Microbiology of Lonar Lake and other soda lakes // ISME J. V. 7. № 3. P. 468–476.
Arroyo A.S., López-Escardó D., Kim E., Ruiz-Trillo I., Najle S.R., 2018. Novel diversity of deeply branching Holomycota and unicellular holozoans revealed by metabarcoding in Middle Paraná River, Argentina // Front. Ecol. Evol. V. 6. Art. 99.
Barandun J., Hunziker M., Vossbrinck C.R., Klinge S., 2019. Evolutionary compaction and adaptation visualized by the structure of the dormant microsporidian ribosome // Nat. Microbiol. V. 4. № 11. P. 1798–1804.
Bass D., Czech L., Williams B.A.P., Berney C., Dunthorn M. et al., 2018. Clarifying the relationships between Microsporidia and Cryptomycota // J. Eukaryot. Microbiol. V. 65. № 6. P. 773–782.
Benson D.A., Cavanaugh M., Clark K., Karsch-Mizrachi I., Lipman D.J. et al., 2013. GenBank // Nucleic Acids Res. V. 41. № D1. P. D36–D42.
Berney C., Fahrni J., Pawlowski J., 2004. How many novel eukaryotic “kingdoms”? Pitfalls and limitations of environmental DNA surveys // BMC Biol. V. 2. Art. 13.
Bernhart S.H., Hofacker I.L., Will S., Gruber A.R., Stadler P.F., 2008. RNAalifold: Improved consensus structure prediction for RNA alignments // BMC Bioinformatics. V. 9. Art. 474.
Bhattacharya D., Friedl T., Damberger S., 1996. Nuclear-encoded rDNA group I introns: Origin and phylogenetic relationships of insertion site lineages in the green algae // Mol. Biol. Evol. V. 13. № 7. P. 978–989.
Bhattacharya D., Friedl T., Helms G., 2002. Vertical evolution and intragenic spread of lichen-fungal group I introns // J. Mol. Evol. V. 55. № 1. P. 74–84.
Borner J., Burmester T., 2017. Parasite infection of public databases: A data mining approach to identify apicomplexan contaminations in animal genome and transcriptome assemblies // BMC Genom. V. 18. № 1. Art. 100.
Burki F., Corradi N., Sierra R., Pawlowski J., Meyer G.R. et al., 2013. Phylogenomics of the intracellular parasite Mikrocytos mackini reveals evidence for a mitosome in Rhizaria // Curr. Biol. V. 23. № 16. P. 1541–1547.
Burki F., Kaplan M., Tikhonenkov D.V., Zlatogursky V., Minh B.Q. et al., 2016. Untangling the early diversification of eukaryotes: A phylogenomic study of the evolutionary origins of Centrohelida, Haptophyta and Cryptista // Proc. Biol. Sci. V. 283. № 1823. Art. 20152802.
Carnegie R.B., Meyer G.R., Blackbourn J., Cochennec-Laureau N., Berthe F.C., Bower S.M., 2003. Molecular detection of the oyster parasite Mikrocytos mackini, and a preliminary phylogenetic analysis // Dis. Aquat. Organ. V. 54. № 3. P. 219–227.
Cavalier-Smith T., Chao E.E., 2003. Phylogeny and classification of phylum Cercozoa (Protozoa) // Protist. V. 154. № 3–4. P. 341–358.
Chaker-Margot M., 2018. Assembly of the small ribosomal subunit in yeast: Mechanism and regulation // RNA. V. 24. № 7. P. 881–891.
Chalwatzis N., Baur A., Stetzer E., Kinzelbach R., Zimmermann F.K., 1995. Strongly expanded 18S rRNA genes correlated with a peculiar morphology in the insect order of Strepsiptera // Zoology. V. 98. № 2. P. 115–126.
Chorev M., Carmel L., 2012. The function of introns // Front. Genet. V. 3. Art. 55.
Chouari R., Leonard M., Bouali M., Guermazi S., Rahli N. et al., 2017. Eukaryotic molecular diversity at different steps of the wastewater treatment plant process reveals more phylogenetic novel lineages // World J. Microbiol. Biotechnol. V. 33. № 3. Art. 44.
Christaki U., Genitsaris S., Monchy S., Li L.L., Rachik S. et al., 2017. Parasitic eukaryotes in a meso-eutrophic coastal system with marked Phaeocystis globosa blooms // Front. Mar. Sci. V. 4. Art. 416.
Coleman A.W., 2007. Pan-eukaryote ITS2 homologies revealed by RNA secondary structure // Nucleic Acids Res. V. 35. № 10. P. 3322–3329.
Coleman A.W., 2015. Nuclear rRNA transcript processing versus internal transcribed spacer secondary structure // Trends Genet. V. 31. № 3. P. 157–163.
Corsaro D., Walochnik J., Venditti D., Hauröder B., Michel R., 2020. Solving an old enigma: Morellospora saccamoebae gen. nov., sp. nov. (Rozellomycota), a Sphaerita-like parasite of free-living amoebae // Parasitol. Res. V. 119. № 3. P. 925–934.
Corsaro D., Walochnik J., Venditti D., Müller K.-D., Hauröder B., Michel R., 2014a. Rediscovery of Nucleo-phaga amoebae, a novel member of the Rozellomycota // Parasitol. Res. V. 113. № 12. P. 4491–4498.
Corsaro D., Walochnik J., Venditti D., Steinmann J., Müller K.D., Michel R., 2014b. Microsporidia-like parasites of amoebae belong to the early fungal lineage Rozellomycota // Parasitol. Res. V. 113. № 5. P. 1909–1918.
Corsaro D., Michel R., Walochnik J., Venditti D., Müller K.D. et al., 2016. Molecular identification of Nucleophaga terricolae sp. nov. (Rozellomycota), and new insights on the origin of the microsporidia // Parasitol. Res. V. 115. № 8. P. 3003–3011.
Corsaro D., Wylezich C., Venditti D., Michel R., Walochnik J., Wegensteiner R., 2019. Filling gaps in the microsporidian tree: rDNA phylogeny of Chytridiopsis typographi (Microsporidia: Chytridiopsida) // Parasitol. Res. V. 118. № 1. P. 169–180.
Cotto I., Dai Z., Huo L., Anderson C.L., Vilardi K.J. et al., 2020. Long solids retention times and attached growth phase favor prevalence of comammox bacteria in nitrogen removal systems // Water Res. V. 169. Art. 115268.
Dawson S.C., Pace N.R., 2002. Novel kingdom-level eukaryotic diversity in anoxic environments // Proc. Natl. Acad. Sci. USA. V. 99. № 12. P. 8324–8329.
Delalibera I., Jr., Hajek A.E., Humber R.A., 2004. Neozygites tanajoae sp. nov., a pathogen of the cassava green mite // Mycologia. V. 96. № 5. P. 1002–1009.
DePriest P.T., Been M.D., 1992. Numerous group I introns with variable distributions in the ribosomal DNA of a lichen fungus // J. Mol. Biol. V. 228. № 2. P. 315–321.
Edgar R.C., 2004. MUSCLE: Multiple sequence alignment with high accuracy and high throughput // Nucleic Acids Res. V. 32. № 5. P. 1792–1797.
Eichorst S.A., Kuske C.R., 2012. Identification of cellulose-responsive bacterial and fungal communities in geographically and edaphically different soils by using stable isotope probing // Appl. Environ. Microbiol. V. 78. № 7. P. 2316–2327.
Engel C., Sainsbury S., Cheung A.C., Kostrewa D., Cramer P., 2013. RNA polymerase I structure and transcription regulation // Nature. V. 502. № 7473. P. 650–655.
Felsenstein J., 1978. Cases in which parsimony or compatibility methods will be positively misleading // Syst. Biol. V. 27. № 4. P. 401–410.
Fernández-Tornero C., 2018. RNA polymerase I activation and hibernation: Unique mechanisms for unique genes // Transcription. V. 9. № 4. P. 248–254.
Filée J., Tetart F., Suttle C.A., Krisch H.M., 2005. Marine T4-type bacteriophages, a ubiquitous component of the dark matter of the biosphere // Proc. Natl. Acad. Sci. USA. V. 102. № 35. P. 12471–12476.
Fitch D.H., Bugaj-Gaweda B., Emmons S.W., 1995. 18S ribosomal RNA gene phylogeny for some Rhabditidae related to Caenorhabditis // Mol. Biol. Evol. V. 12. № 2. P. 346–358.
Förster J., Famili I., Fu P., Palsson B.Ø., Nielsen J., 2003. Genome-scale reconstruction of the Saccharomyces cerevisiae metabolic network // Genome Res. V. 13. № 2. P. 244–253.
Freimoser F.M., Tuor U., Aebi M., 2000. Phylogenetic analysis of SSU rDNA sequences suggest a common origin of Neozygitaceae (Zoopagomycota; Entomophthorales), fungal pathogens of arthropods, and of microsporidia, obligate intracellulae parasite // Freimoser F.M., 2000. Cultivation, sporulation and phylogenetic analysis of Neozygites parvispora and Entomophthora thripidum, two fungal pathogens of thrips. Diss. ETH № 13869. Zürich: ETH Zürich. P. 29–43. https:// www.research-collection.ethz.ch/handle/20.500.11850/ 144847.
Galindo L.J., Torruella G., Moreira D., Timpano H., Paskerova G. et al., 2018. Evolutionary genomics of Metchnikovella incurvata (Metchnikovellidae): An early branching microsporidium // Genome Biol. Evol. V. 10. № 10. P. 2736–2748.
Gargas A., DePriest P.T., Taylor J.W., 1995. Positions of multiple insertions in SSU rDNA of lichen-forming fungi // Mol. Biol. Evol. V. 12. № 2. P. 208–218.
Gawryluk R.M.R., Tikhonenkov D.V., Hehenberger E., Husnik F., Mylnikov A.P., Keeling P.J., 2019. Non-photosynthetic predators are sister to red algae // Nature. V. 572. № 7768. P. 240–243.
Gleason F.H., Lilje O., Marano A.V., Sime-Ngando T., Sullivan B.K. et al., 2014. Ecological functions of zoosporic hyperparasites // Front. Microbiol. V. 5. Art. 244.
Grossart H.-P., Wurzbacher C., James T.Y., Kagami M., 2016. Discovery of dark matter fungi in aquatic ecosystems demands a reappraisal of the phylogeny and eco-logy of zoosporic fungi // Fungal Ecol. V. 19. № 1. P. 28–38.
Gruber A.R., Bernhart S.H., Lorenz R., 2015. The Vienna- RNA web services // Methods Mol. Biol. V. 1269. P. 307–326.
Guillou L., Viprey M., Chambouvet A., Welsh R.M., Kirkham A.R. et al., 2008. Widespread occurrence and genetic diversity of marine parasitoids belonging to Syndiniales (Alveolata) // Environ. Microbiol. V. 10. № 12. P. 3349–3365.
Haag K.L., James T.Y., Pombert J.F., Larsson R., Schaer T.M. et al., 2014. Evolution of a morphological novelty occurred before genome compaction in a lineage of extreme parasites // Proc. Natl. Acad. Sci. USA. V. 111. № 43. P. 15480–15485.
Hannen E.J., van, Mooij W., Agterveld M.P., van, Gons H.J., Laanbroek H.J., 1999. Detritus-dependent development of the microbial community in an experimental system: Qualitative analysis by denaturing gradient gel electrophoresis // Appl. Environ. Microbiol. V. 65. № 6. P. 2478–2484.
Hartmann M., Lee S., Hallam S.J., Mohn W.W., 2009. Bacterial, archaeal and eukaryal community structures throughout soil horizons of harvested and naturally disturbed forest stands // Environ. Microbiol. V. 11. № 12. P. 3045–3062.
Hasegawa H., Satô M., Maeda K., Murayama Y., 2012. Description of Riouxgolvania kapapkamui sp. n. (Nematoda: Muspiceoidea: Muspiceidae), a peculiar intradermal parasite of bats in Hokkaido, Japan // J. Parasitol. V. 98. № 5. P. 995–1000.
Hendy M.D., Penny D., 1989. A framework for the quantitative study of evolutionary trees // Syst. Zool. V. 38. № 4. P. 277–290.
Hennig W., 1966. Phylogenetic Systematics. Urbana: Univ. Illinois Press. 263 p.
Huss V.A.R., Bauer C., 2011. A highly divergent 18S rRNA sequence identified by environmental PCR from an extremely acidic mining lake (pH 2.3) in Lusatia (Germany). Deposited under Accession Number FN546176.1. Aug. 30, 2011. https://www.ncbi.nlm.nih.gov/nuccore/345107473.
Ishida S., Nozaki D., Grossart H.P., Kagami M., 2015. Novel basal, fungal lineages from freshwater phytoplankton and lake samples // Environ. Microbiol. Rep. V. 7. № 3. P. 435–441.
James T.Y., Kauff F., Schoch C.L., Matheny P.B., Hofstetter V. et al., 2006. Reconstructing the early evolution of Fungi using a six-gene phylogeny // Nature. V. 443. № 7113. P. 818–822.
Jamy M., Foster R., Barbera P., Czech L., Kozlov A. et al., 2019. Long-read metabarcoding of the eukaryotic rDNA operon to phylogenetically and taxonomically resolve environmental diversity // Mol. Ecol. Resour. V. 20. № 2. P. 429–443.
Janouškovec J., Tikhonenkov D.V., Burki F., Howe A.T., Rohwer F.L. et al., 2017. A new lineage of eukaryotes illuminates early mitochondrial genome reduction // Curr. Biol. V. 27. № 23. P. 3717–3724.
Janouškovec J., Paskerova G.G., Miroliubova T.S., Mikhailov K.V., Birley T. et al., 2019. Apicomplexan-like parasites are polyphyletic and widely but selectively dependent on cryptic plastid organelles // eLife. V. 8. Art. e49662.
Jones M.D.M., Richards T.A., Hawksworth D.J., Bass D., 2011a. Validation and justification of the phylum name Cryptomycota phyl. nov. // IMA Fungus. V. 2. № 2. P. 173–175.
Jones M.D.M., Forn I., Gadelha C., Egan M.J., Bass D. et al., 2011b. Discovery of novel intermediate forms redefines the fungal tree of life // Nature. V. 474. № 7350. P. 200–203.
Joseph N., Krauskopf E., Vera M.I., Michot B., 1999. Ribosomal internal transcribed spacer 2 (ITS2) exhibits a common core of secondary structure in vertebrates and yeast // Nucleic Acids Res. V. 27. № 23. P. 4533–4540.
Karpov S.A., Paskerova G.G., 2020. The aphelids, intracellular parasitoids of algae, consume the host cytoplasm “from the inside” // Protistology. V. 14. № 4. P. 258–263.
Karpov S.A., Mikhailov K.V., Mirzaeva G.S., Mirabdullaev I.M., Mamkaeva K.A. et al., 2013. Obligately phagotrophic aphelids turned out to branch with the earliest-diverging fungi // Protist. V. 164. № 2. P. 195–205.
Karpov S.A., Mamanazarova K.S., Popova O.V., Aleoshin V.V., James T.Y. et al., 2017. Monoblepharidomycetes diversity includes new parasitic and saprotrophic species with highly intronized rDNA // Fungal Biol. V. 121. № 8. P. 729–741.
Karpov S.A., Moreira D., Mamkaeva M.A., Popova O.V., Aleoshin V.V., López-García P., 2019. New member of Gromochytriales (Chytridiomycetes) – Apiochytrium granulosporum nov. gen. et sp. // J. Eukaryot. Microbiol. V. 66. № 4. P. 582–591.
Katiyar S.K., Visvesvara G.S., Edlind T.D., 1995. Comparisons of ribosomal RNA sequences from amitochondrial protozoa: Implications for processing, mRNA binding and paromomycin susceptibility // Gene. V. 152. № 1. P. 27–33.
Kim E., Sprung B., Duhamel S., Filardi C., Kyoon Shin M., 2016. Oligotrophic lagoons of the South Pacific Ocean are home to a surprising number of novel eukaryotic microorganisms // Environ. Microbiol. V. 18. № 12. P. 4549–4563.
Kumar S., Stecher G., Tamura K., 2016. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Mol. Biol. Evol. V. 33. № 7. P. 1870–1874.
Langmead B., Salzberg S.L., 2012. Fast gapped-read alignment with Bowtie 2 // Nat. Methods. V. 9. № 4. P. 357–359.
Lara E., Moreira D., López-García P., 2010. The environmental clade LKM11 and Rozella form the deepest branching clade of Fungi // Protist. V. 161. № 1. P. 116–121.
Larsson J.I.R., 1993. Description of Chytridiopsis trichopterae n. sp. (Microspora, Chytridiopsidae), a microsporidian parasite of the caddis fly Polycentropus flavomaculatus (Trichoptera, Polycentropodidae), with comments on relationships between the families Chytridiopsidae and Metchnikovellidae // J. Eukaryot. Microbiol. V. 40. № 1. P. 37–48.
Larsson J.I.R., 2000. The hyperparasitic microsporidium Amphiacantha longa Caullery et Mesnil, 1914 (Microspora : Metchnikovellidae) – description of the cytology, redescription of the species, emended diagnosis of the genus Amphiacantha and establishment of the new family Amphiacanthidae // Folia Parasitol. V. 47. № 4. P. 241–256.
Larsson J.I.R., 2014. The primitive Microsporidia // Microsporidia: Pathogens of Opportunity / Eds Weiss L.M., Becnel J.J. Hoboken: Wiley-Blackwell. P. 605–634.
Lazarus K.L., James T.Y., 2015. Surveying the biodiversity of the Cryptomycota using a targeted PCR approach // Fungal Ecol. V. 14. № 1. P. 62–70.
Lee J.C., Gutell R.R., 2012. A comparison of the crystal structures of the eukaryotic and bacterial SSU ribosomal RNAs reveals common structural features in the hypervariable regions // PLoS One. V. 7. № 5. Art. e38203.
Lefèvre E., Roussel B., Amblard C., Sime-Ngando T., 2008. The molecular diversity of freshwater picoeukaryotes reveals high occurrence of putative parasitoids in the plankton // PLoS One. V. 3. № 6. Art. e2324.
Lefèvre E., Bardot C., Noël C., Carrias J.F., Viscogliosi E. et al., 2007. Unveiling fungal zooflagellates as members of freshwater picoeukaryotes: Evidence from a molecular diversity study in a deep meromictic lake // Environ. Microbiol. V. 9. № 1. P. 61–71.
Leipe D.D., Gunderson J.H., Nerad T.A., Sogin M.L., 1993. Small subunit ribosomal RNA+ of Hexamita inflata and the quest for the first branch in the eukaryotic tree // Mol. Biochem. Parasitol. V. 59. № 1. P. 41–48.
Lepère C., Domaizon I., Debroas D., 2008. Unexpected importance of potential parasites in the composition of the freshwater small-eukaryote community // Appl. Environ. Microbiol. V. 74. № 10. P. 2940–2949.
Lepère C., Boucher D., Jardillier L., Domaizon I., Debroas D., 2006. Succession and regulation factors of small eukaryote community composition in a lacustrine ecosystem (Lake Pavin) // Appl. Environ. Microbiol. V. 72. № 4. P. 2971–2981.
Letcher P.M., Powell M.J., 2018. A taxonomic summary and revision of Rozella (Cryptomycota) // IMA Fungus. V. 9. P. 383–399.
Letcher P.M., Lopez S., Schmieder R., Lee P.A., Behnke C. et al., 2013. Characterization of Amoeboaphelidium protococcarum, an algal parasite new to the cryptomycota isolated from an outdoor algal pond used for the production of biofuel // PLoS One. V. 8. № 2. Art. e56232.
Lipson D.A., Kuske C.R., Gallegos-Graves L.V., Oechel W.C., 2014. Elevated atmospheric CO2 stimulates soil fungal diversity through increased fine root production in a semiarid shrubland ecosystem // Glob. Change Biol. V. 20. № 8. P. 2555–2565.
López-García P., Rodriguez-Valera F., Pedrós-Alió C., Moreira D., 2001. Unexpected diversity of small eukaryotes in deep-sea Antarctic plankton // Nature. V. 409. № 6820. P. 603–607.
Mahé F., Vargas C., de, Bass D., Czech L., Stamatakis A. et al., 2017. Parasites dominate hyperdiverse soil protist communities in Neotropical rainforests // Nat. Ecol. Evol. V. 1. № 4. Art. 91.
Marcy Y., Ouverney C., Bik E.M., Lösekann T., Ivanova N. et al., 2007. Dissecting biological “dark matter” with single-cell genetic analysis of rare and uncultivated TM7 microbes from the human mouth // Proc. Natl. Acad. Sci. USA. V. 104. № 29. P. 11889–11894.
Matsubayashi M., Shimada Y., Li Y.Y., Harada H., Kubota K., 2017. Phylogenetic diversity and in situ detection of eukaryotes in anaerobic sludge digesters // PLoS One. V. 12. № 3. Art. e0172888.
McGuire K.L., Allison S.D., Fierer N., Treseder K., 2013. Ectomycorrhizal-dominated boreal and tropical forests have distinct fungal communities, but analogous spatial patterns across soil horizons // PLoS One. V. 8. № 7. Art. e68278.
Melnikov S.V., Manakongtreecheep K., Rivera K.D., Makarenko A., Pappin D.J., Söll D., 2018a. Muller’s ratchet and ribosome degeneration in the obligate intracellular parasites Microsporidia // Int. J. Mol. Sci. V. 19. № 12. Art. 4125.
Melnikov S.V., Rivera K.D., Ostapenko D., Makarenko A., Sanscrainte N.D. et al., 2018b. Error-prone protein synthesis in parasites with the smallest eukaryotic genome // Proc. Natl. Acad. Sci. USA. V. 115. № 27. P. E6245–E6253.
Mikhailov K.V., Simdyanov T.G., Aleoshin V.V., 2017. Genomic survey of a hyperparasitic microsporidian Amphiamblys sp. (Metchnikovellidae) // Genome Biol. Evol. V. 9. № 3. P. 454–467.
Mitchell A.L., Scheremetjew M., Denise H., Potter S., Tarkowska A. et al., 2018. EBI Metagenomics in 2017: Enriching the analysis of microbial communities, from sequence reads to assemblies // Nucleic Acids Res. V. 46. № D1. P. D726–D735.
Monchy S., Sanciu G., Jobard M., Rasconi S., Gerphagnon M. et al., 2011. Exploring and quantifying fungal diversity in freshwater lake ecosystems using rDNA cloning/sequencing and SSU tag pyrosequencing // Environ. Microbiol. V. 13. № 6. P. 1433–1453.
Moon-van der Staay S.Y., Wachter R., de, Vaulot D., 2001. Oceanic 18S rDNA sequences from picoplankton reveal unsuspected eukaryotic diversity // Nature. V. 409. № 6820. P. 607–610.
Moss T., Stefanovsky V.Y., 2002. At the center of eukaryotic life // Cell. V. 109. № 5. P. 545–548.
Mueller R.C., Moya-Balasch M., Kuske C.R., 2014. Contrasting soil fungal community responses to experimental nitrogen addition using the large subunit rRNA taxonomic marker and cellobiohydrolase I functional marker // Mol. Ecol. V. 23. № 17. P. 4406–4417.
Najmi S.M., Schneider D.A., 2021. Quorum sensing regulates rRNA synthesis in Saccharomyces cerevisiae // Gene. V. 776. Art. 145442.
Nakai R., Abe T., Baba T., Imura S., Kagoshima H. et al., 2012. Eukaryotic phylotypes in aquatic moss pillars inhabiting a freshwater lake in East Antarctica, based on 18S rRNA gene analysis // Polar. Biol. V. 35. № 10. P. 1495–1504.
Nassonova E.S., Bondarenko N.I., Paskerova G.G., Kovacikova M., Frolova E.V., Smirnov A.V., 2021. Evolutionary relationships of Metchnikovella dogieli Paskerova et al., 2016 (Microsporidia: Metchnikovellidae) revealed by multigene phylogenetic analysis // Parasitol. Res. V. 120. № 2. P. 525–534.
Noack Watt K.E., Achilleos A., Neben C.L., Merrill A.E., Trainor P.A., 2016. The roles of RNA polymerase I and III subunits Polr1c and Polr1d in craniofacial development and in zebrafish models of Treacher Collins syndrome // PLoS Genet. V. 12. № 7. Art. e1006187.
Page K.A., Flannery M.K., 2018. Microbial epiphytes of deep-water moss in Crater Lake, Oregon // Northwest Sci. V. 92. № 4. P. 240–250.
Paskerova G.G., Frolova E.V., Kováčiková M., Panfilkina T.S., Mesentsev E.S. et al., 2016. Metchnikovella dogieli sp. n. (Microsporidia: Metchnikovellida), a parasite of archigregarines Selenidium sp. from polychaetes Pygospio elegans // Protistology. V. 10. № 4. P. 148–157.
Paskerova G.G., Miroliubova T.S., Diakin A., Kováčiková M., Valigurová A. et al., 2018. Fine structure and molecular phylogenetic position of two marine gregarines, Selenidium pygospionis sp. n. and S. pherusae sp. n., with notes on the phylogeny of Archigregarinida (Apicomplexa) // Protist. V. 169. № 6. P. 826–852.
Paule M.R., (ed.), 1998. Transcription of Ribosomal RNA Genes by Eukaryotic RNA Polymerase I. Berlin; L.; N.Y.: Springer-Verlag Inc. and R.G. Landes Co. 321 p.
Pawlowski J., Bolivar I., Guiard-Maffia J., Gouy M., 1994. Phylogenetic position of foraminifera inferred from LSU rRNA gene sequences // Mol. Biol. Evol. V. 11. № 6. P. 929–938.
Pawlowski J., Montoya-Burgos J.I., Fahrni J.F., Wüest J., Zaninetti L., 1996. Origin of the Mesozoa inferred from 18S rRNA gene sequences // Mol. Biol. Evol. V. 13. № 8. P. 1128–1132.
Petrov A.S., Bernier C.R., Gulen B., Waterbury C.C., Hershkovits E. et al., 2014. Secondary structures of rRNAs from all three domains of life // PLoS One. V. 9. № 2. Art. e88222.
Peyretaillade E., Biderre C., Peyret P., Duffieux F., Méténier G. et al., 1998. Microsporidian Encephalitozoon cuniculi, a unicellular eukaryote with an unusual chromosomal dispersion of ribosomal genes and a LSU rRNA reduced to the universal core // Nucleic Acids Res. V. 26. № 15. P. 3513–3520.
Quinlan A.R., Hall I.M., 2010. BEDTools: A flexible suite of utilities for comparing genomic features // Bioinformatics. V. 26. № 6. P. 841–842.
Rambaut A., 2010. FigTree v1.3.1. Edinburgh: Institute of Evolutionary Biology, University of Edinburgh. http://tree.bio.ed.ac.uk/software/figtree/
Rijk P., de, Gatehouse H.S., Wachter R., de, 1998. The secondary structure of Nosema apis large subunit ribosomal RNA // Biochim. Biophys. Acta. V. 1442. № 1–2. P. 326–328.
Rijk P., de, Wuyts J., Wachter R., de, 2003. RnaViz 2: An improved representation of RNA secondary structure // Bioinformatics. V. 19. № 2. P. 299–300.
Rogers S.O., 2019. Integrated evolution of ribosomal RNAs, introns, and intron nurseries // Genetica. V. 147. № 2. P. 103–119.
Rojas-Jimenez K., Wurzbacher C., Bourne E.C., Chiuchiolo A., Priscu J.C., Grossart H.P., 2017. Early diverging lineages within Cryptomycota and Chytridiomycota dominate the fungal communities in ice-covered lakes of the McMurdo Dry Valleys, Antarctica // Sci. Rep. V. 7. № 1. Art. 15348.
Rojas-Jimenez K., Rieck A., Wurzbacher C., Jürgens K., Labrenz M., Grossart H.P., 2019. A salinity threshold separating fungal communities in the Baltic Sea // Front. Microbiol. V. 10. Art. 680.
Ronquist F., Teslenko M., Mark P., van der, Ayres D.L., Darling A. et al., 2012. MrBayes 3.2: Efficient Bayesian phylogenetic inference and model selection across a large model space // Syst. Biol. V. 61. № 3. P. 539–542.
Rotari Y.M., Paskerova G.G., Sokolova Y.Y., 2015. Diversity of metchnikovellids (Metchnikovellidae, Rudimicrosporea), hyperparasites of bristle worms (Annelida, Polychaeta) from theWhite Sea // Protistology. V. 9. № 1. P. 50–59.
Rueckert S., Wakeman K.C., Leander B.S., 2013. Discovery of a diverse clade of gregarine apicomplexans (Apicomplexa: Eugregarinorida) from Pacific eunicid and onuphid polychaetes, including descriptions of Paralecudina n. gen., Trichotokara japonica n. sp., and T. eunicae n. sp. // J. Eukaryot. Microbiol. V. 60. № 2. P. 121–136.
Rueckert S., Simdyanov T.G., Aleoshin V.V., Leander B.S., 2011. Identification of a divergent environmental DNA sequence clade using the phylogeny of gregarine parasites (Apicomplexa) from crustacean hosts // PLoS One. V. 6. № 3. Art. e18163.
Russell J., Zomerdijk J.C.B.M., 2005. RNA-polymerase-I-directed rDNA transcription, life and works // Trends Biochem. Sci. V. 30. № 2. P. 87–96.
Sanchez L.R.S., Cao E.P., 2019. Metagenomic analysis reveals the presence of heavy metal response genes from cyanobacteria thriving in Balatoc Mines, Benguet Province, Philippines // Philippine J. Sci. V. 148. № S1. P. 71–82.
Schultz J., Maisel S., Gerlach D., Műller T., Wolf M., 2005. A common core of secondary structure of the internal transcribed spacer 2 (ITS2) throughout the eukaryote // RNA. V. 11. № 4. P. 361–364.
Seenivasan R., Sausen N., Medlin L.K., Melkonian M., 2013. Picomonas judraskeda gen. et sp. nov.: The first identified member of the Picozoa phylum nov., a widespread group of picoeukaryotes, formerly known as 'picobiliphytes' // PLoS One. V. 8. № 3. Art. e59565.
Sharrar A.M., Crits-Christoph A., Méheust R., Diamond S., Starr E.P., Banfield J.F., 2020. Bacterial secondary metabolite biosynthetic potential in soil varies with phylum, depth, and vegetation type // mBio. V. 11. № 3. Art. e00416-20.
Smith M.E., Douhan G.W., Rizzo D.M., 2007. Ectomycorrhizal community structure in a xeric Quercus woodland as inferred from rDNA sequence analysis of pooled EM roots and sporocarps // New Phytol. V. 174. № 4. P. 847–863.
Smothers J.F., Dohlen C.D., von, Smith L.H., Jr., Spall R.D., 1994. Molecular evidence that the myxozoan protists are metazoans // Science. V. 265. № 5179. P. 1719–1721.
Sokolova Y.Y., Paskerova G.G., Rotari Y.M., Nassonova E.S., Smirnov A.V., 2013. Fine structure of Metchnikovella incurvata Caullery and Mesnil 1914 (Microsporidia), a hyperparasite of gregarines Polyrhabdina sp. from the polychaete Pygospio elegans // Parasitology. V. 140. № 7. P. 855–867.
Sprague V., 1977. Classification and phylogeny of the microsporidia // Comparative Pathobiology. V. 2. Systematics of the Microsporidia / Eds Bulla L.A., Cheng T.C. N.Y.: Plenum Press. P. 1–30.
Stentiford G.D., Ramilo A., Abollo E., Kerr R., Bateman K.S. et al., 2017. Hyperspora aquatica n. gen., n. sp. (Microsporidia), hyperparasitic in Marteilia cochillia (Paramyxida), is closely related to crustacean-infecting microspordian taxa // Parasitology. V. 144. № 2. P. 186–199.
Stevenson B.S., Schmidt T.M., 2004. Life history implications of rRNA gene copy number in Escherichia coli // Appl. Environ. Microbiol. V. 70. № 11. P. 6670–6677.
Strassert J.F.H., Jamy M., Mylnikov A.P., Tikhonenkov D.V., Burki F., 2019. New phylogenomic analysis of the enigmatic phylum Telonemia further resolves the Eukaryote Tree of Life // Mol. Biol. Evol. V. 36. № 4. Art. 757765.
Tavaré S., 1986. Some probabilistic and statistical problems in the analysis of DNA sequences // Lectures Math. Life Sci. V. 17. P. 57–86.
Taylor D.L., Herriott I.C., Long J., O’Neill K., 2007. TOPO TA is A-OK: A test of phylogenetic bias in fungal environmental clone library construction // Environ. Microbiol. V. 9. № 5. P. 1329–1334.
Taylor D.L., Booth M.G., McFarland J.W., Herriott I.C., Lennon N.J. et al., 2008. Increasing ecological inference from high throughput sequencing of fungi in the environment through a tagging approach // Mol. Ecol. Resour. V. 8. № 4. P. 742–752.
Taylor D.L., Hollingsworth T.N., McFarland J.W., Lennon N.J., Nusbaum C., Ruess R.W., 2014. A first comprehensive census of fungi in soil reveals both hyperdiversity and fine-scale niche partitioning // Ecol. Monogr. V. 84. № 1. P. 3–20.
Tedersoo L., Bahram M., Puusepp R., Nilsson R.H., James T.Y., 2017. Novel soil-inhabiting clades fill gaps in the fungal tree of life // Microbiome. V. 5. № 1. Art. 42.
Tedersoo L., Sánchez-Ramírez S., Kõljalg U., Bahram M., Döring M. et al., 2018. High-level classification of the Fungi and a tool for evolutionary ecological analyses // Fungal Divers. V. 90. № 1. P. 135–159.
Thornton C.N., Tanner W.D., VanDerslice J.A., Brazelton W.J., 2020. Localized effect of treated wastewater effluent on the resistome of an urban watershed // Gigascience. V. 9. № 11. Art. giaa125.
Tikhonenkov D.V., Mikhailov K.V., Hehenberger E., Karpov S.A., Prokina K.I. et al., 2020a. New lineage of microbial predators adds complexity to reconstructing the evolutionary origin of animals // Curr. Biol. V. 30. № 22. P. 4500–4509.
Tikhonenkov D.V., Strassert J.F.H., Janouškovec J., Mylnikov A.P., Aleoshin V.V. et al., 2020b. Predatory colponemids are the sister group to all other alveolates // Mol. Phylogenet. Evol. V. 149. Art. 106839.
Timling I., Walker D.A., Nusbaum C., Lennon N.J., Taylor D.L., 2014. Rich and cold: Diversity, distribution and drivers of fungal communities in patterned-ground ecosystems of the North American Arctic // Mol. Ecol. V. 23. № 13. P. 3258–3272.
Tokarev Y.S., Huang W.F., Solter L.F., Malysh J.M., Becnel J.J., Vossbrinck C.R., 2020. A formal redefinition of the genera Nosema and Vairimorpha (Microsporidia: Nosematidae) and reassignment of species based on molecular phylogenetics // J. Invertebr. Pathol. V. 169. Art. 107279.
Torreira E., Louro J.A., Pazos I., González-Polo N., Gil-Carton D. et al., 2017. The dynamic assembly of distinct RNA polymerase I complexes modulates rDNA transcription // eLife. V. 6. Art. e20832.
Trzebny A., Slodkowicz-Kowalska A., Becnel J.J., Sanscrainte N., Dabert M., 2020. A new method of metabarcoding Microsporidia and their hosts reveals high levels of microsporidian infections in mosquitoes (Culicidae) // Mol. Ecol. Resour. V. 20. P. 1486–1504.
Van de Peer Y., Ben Ali A., Meyer A., 2000. Microsporidia: Accumulating molecular evidence that a group of amitochodriate and suspectedly primitive eukaryotes are just curious fungi // Gene. V. 246. № 1–2. P. 1–8.
Vivier E., 1965. Étude, au microscope électronique, de la spore de Metchnikovella hovassei n. sp.: Appartenance des Metchnikovellidae aux Microsporidies // Compt. Rend. Hebd. Séan. Soc. Biol. V. 260. № 26. P. 6982–6984.
Vossbrinck C.R., Woese C.R., 1986. Eukaryotic ribosomes that lack a 5.8S RNA // Nature. V. 320. № 6059. P. 287–288.
Vossbrinck C.R., Maddox J.V., Friedman S., Debrunner-Vossbrinck B.A., Woese C.R., 1987. Ribosomal RNA sequence suggests Microsporidia are extremely ancient eukaryotes // Nature. V. 326. № 6111. P. 411–414.
Wadi L., Reinke A.W., 2020. Evolution of microsporidia: An extremely successful group of eukaryotic intracellular parasites // PLoS Pathog. V. 16. № 2. Art. e1008276.
Wagner A., 2005. Energy constraints on the evolution of gene expression // Mol. Biol. Evol. V. 22. № 6. P. 1365–1374.
Wakeman K.C., 2020. Molecular phylogeny of marine gregarines (Apicomplexa) from the Sea of Japan and the Northwest Pacific including the description of three novel species of Selenidium and Trollidium akkeshiense n. gen. n. sp. // Protist. V. 171. № 1. Art. 125710.
Warner J.R., 1999. The economics of ribosome biosynthesis in yeast // Trends Biochem. Sci. V. 24. № 11. P. 437–440.
Weber S.D., Hofmann A., Wanner G., Pilhofer M., Agerer R. et al., 2009. The diversity of fungi in aerobic sewage granules assessed by 18S rRNA gene and ITS sequence analyses // FEMS Microbiol. Ecol. V. 68. № 2. P. 246–254.
Weiser J., 1977. Contribution to the classification of microsporidia // Vestn. Cesk. Spol Zool. V. 41. № 4. P. 308–321.
White M.M., James T.Y., O’Donnell K., Cafaro M.J., Tanabe Y., Sugiyama J., 2006. Phylogeny of the Zygomycota based on nuclear ribosomal sequence data // Mycologia. V. 98. № 6. P. 872–884.
Whiting M.F., Carpenter J.C., Wheeler Q.D., Wheeler W.C., 1997. The Strepsiptera problem: Phylogeny of the holometabolous insect orders inferred from 18S and 28S ribosomal DNA sequences and morphology // Syst. Biol. V. 46. № 1. P. 1–68.
Wijayawardene N.N., Hyde K.D., Al-Ani L.K.T., Tedersoo L., Haelewaters D. et al., 2020. Outline of Fungi and fungus-like taxa // Mycosphere. V. 11. № 1. P. 1060–1456.
Wilms R., Sass H., Köpke B., Köster J., Cypionka H., Engelen B., 2006. Specific bacterial, archaeal, and eukaryotic communities in tidal-flat sediments along a vertical profile of several meters // Appl. Environ. Microbiol. V. 72. № 4. P. 2756–2764.
Wurzbacher C., Rösel S., Rychła A., Grossart H.P., 2014. Importance of saprotrophic freshwater fungi for pollen degradation // PLoS One. V. 9. № 4. Art. e94643.
Wuyts J., Van de Peer Y., Wachter R., de, 2001. Distribution of substitution rates and location of insertion sites in the tertiary structure of ribosomal RNA // Nucleic Acids Res. V. 29. № 24. P. 5017–5028.
Wuyts J., Rijk P., de, Van de Peer Y., Pison G., Rousseeuw P., Wachter R., de, 2000. Comparative analysis of more than 3000 sequences reveals the existence of two pseudoknots in area V4 of eukaryotic small subunit ribosomal RNA // Nucleic Acids Res. V. 28. № 23. P. 4698–4708.
Zhou X., Montalva C., Arismendi N., 2017. Neozygites linanensis sp. nov., a fungal pathogen infecting bamboo aphids in southeast China // Mycotaxon. V. 132. № 2. P. 305–315.
Zuker M., 2003. Mfold web server for nucleic acid folding and hybridization prediction // Nucleic Acids Res. V. 31. № 13. P. 3406–3415.
Дополнительные материалы отсутствуют.
Инструменты
Журнал общей биологии


