Журнал общей биологии, 2022, T. 83, № 1, стр. 38-50
Характеристика клещей Varroa underwoodi (Acari: Varroidae) в популяции Apis cerana ussuriensis (Hymenoptera: Apidae) в Приморском крае, Россия
Р. А. Ильясов 1, 2, 3, *, Д. И. Такахаши 4, **, М. Л. Ли 2, М. Ю. Прощалыкин 5, А. С. Лелей 5, ***, Х. В. Квон 2, ****, В. Н. Даниленко 1, *****, А. Г. Николенко 3, ******
1 Институт общей генетики им. Н.И. Вавилова РАН
119991 Москва, ул. Губкина, 3, Россия
2 Инчхонский национальный университет, Отделение наук о жизни, специализация в области биологических наук, Центр исследований насекомых-переносчиков болезней
22012 Инчхон, Академи-ро 119, Ёнсу-гу, Сонгдо-донг, Республика Корея
3 Институт биохимии и генетики Уфимского федерального исследовательского центра РАН
450054 Уфа, Просп. Октября, 71, Россия
4 Университет Киото Сангё, факультет естественных наук
Камигамо Мотояма
603-8555 Киото, Кита Вард, Япония
5 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии, ДВО РАН
690022 Владивосток, Просп. 100-летия Владивостока, 159, Россия
* E-mail: apismell@hotmail.com
** E-mail: jit@cc.kyoto-su.ac.jp
*** E-mail: lelej@biosoil.ru
**** E-mail: hwkwon@inu.ac.kr
***** E-mail: valerid@vigg.ru
****** E-mail: a-nikolenko@yandex.ru
Поступила в редакцию 30.08.2021
После доработки 05.10.2021
Принята к публикации 19.10.2021
- EDN: ICHTVI
- DOI: 10.31857/S0044459622010055
Аннотация
Виды рода Varroa являются эктопаразитическими клещами медоносных пчел рода Apis. В отличие от хорошо известных видов клещей V. destructor и V. jacobsoni, V. underwoodi остается мало изученным. Современный ареал V. underwoodi в популяции A. cerana включает Непал, Южную Корею, Японию, Малайзию, Индию, Индонезию, Папуа-Новую Гвинею, Вьетнам и Китай. Недавно он был найден в России (Приморский край) на A. cerana ussuriensis. Поскольку V. destructor и V. jacobsoni обладают способностью легко переходить на другие виды медоносных пчел, есть большая вероятность того, что V. underwoodi может в дальнейшем перейти в массовом порядке с A. cerana на A. mellifera. Первый случай паразитирования V. underwoodi в семьях пчел A. mellifera зафиксирован в Папуа-Новой Гвинее. Varroa underwoodi требует тщательного изучения, поскольку является новым потенциальным паразитом A. mellifera и способен принести с собой новые виды и штаммы вирусов и бактерий, изменить состав микробиома кишечника пчел, нарушить защитные и адаптивные механизмы их организма. В статье представлены данные морфометрии V. underwoodi и полиморфизма его гена COX1 мтДНК. Проведено сравнение V. underwoodi с другими видами клещей V. destructor и V. jacobsoni. Средняя генетическая дивергенция и p-дистанция между V. underwoodi и другими видами Varroa составляли 9% и 0.09 соответственно, что согласуется со средним уровнем видовых различий у насекомых. Нуклеотидные последовательности гена COX1 мтДНК V. underwoodi из Приморского края (Россия) LC532104 и провинции Цзилинь (Китай) MH205176 оказались идентичными и отнесены к гаплотипу China 1 MH205176. Предполагается, что между популяциями A. cerana России и Китая происходит непрерывный обмен, который привел к появлению V. underwoodi в природной популяции A. cerana ussuriensis в Приморском крае. Современная северная граница ареала V. underwoodi проходит по территории Дальнего Востока России и, вероятно, совпадает с таковой A. cerana ussuriensis (45.06° с.ш.). Вероятно, селекция семей пчел A. mellifera по гигиеническому поведению против клещей V. destructor может оказаться эффективной также против V. underwoodi и позволит предотвратить возможный переход V. underwoodi с азиатских пчел A. cerana на европейских пчел A. mellifera.
Паразитические клещи рода Varroa Oudemans, 1904 относятся к семейству Varroidae инфраотряда Gamasina отряда Mesostigmata надотряда Parasitiformes. Клещи р. Varroa являются эктопаразитами пчел р. Apis и представлены четырьмя видами (Anderson, Trueman, 2000; Rosenkranz et al., 2010): 1) V. jacobsoni, описанный с A. cerana (о-в Ява, Индонезия) (Oudemans, 1904), позднее найден и на A. nigrocincta в Индонезии (Hadisoesilo, Otis, 1998; Anderson, Trueman, 2000) и A. mellifera в Папуа-Новой Гвинее (Roberts et al., 2015); 2) V. destructor (первоначально ошибочно идентифицированный как V. jacobsoni) описан с A. cerana (Китай, Япония, Южная Корея, Таиланд), позднее найден и на A. mellifera в Японии (Anderson, 2000; Anderson, Trueman, 2000); 3) V. rindereri с A. koschevnikovi (Сабах, Малайзия) (Guzman, Delfinado-Baker, 1996), на других видах пчел пока не обнаружен; 4) V. underwoodi, описанный c A. cerana (Непал) (Delfinado-Baker, Aggarwal, 1987), позднее найден на A. nigrocincta в Индонезии (Anderson et al., 1997; Кузнецов, 2005).
Медоносная пчела A. mellifera поражается во всем мире преимущественно V. destructor (Traynor et al., 2020), а в Папуа-Новой Гвинее – V. jacobsoni (Roberts et al., 2015). Описано несколько митохондриальных гаплотипов V. destructor, из которых два – K (корейский) и J (японский) – способны размножаться и паразитировать в семьях A. mellifera (Anderson, 2000; Anderson, Trueman, 2000; Muñoz et al., 2008). Клещи V. rindereri и V. underwoodi в наименьшей степени изучены (Oudemans, 1904; Delfinado-Baker, Aggarwal, 1987; Guzman, Delfinado-Baker, 1996; Anderson et al., 1997; Rath, 1999; Anderson, Trueman, 2000; Wang et al., 2019a).
Другой вид медоносной пчелы, A. cerana, поражается преимущественно клещом V. destructor и в меньшей степени V. underwoodi; последний встречается в меньшей численности (Wang et al., 2019a, b; Lin et al., 2021). Сравнительно недавно V. underwoodi выявлен и в России (Приморский край), описаны его морфометрия и полиморфизм гена COX1 мтДНК в природной популяции A. cerana (Кузнецов, 2005; Кузнецов, Лелей, 2005; Ilyasov et al., 2021). Данные работы посвящены первому обнаружению V. underwoodi в природной популяции A. cerana в России, поэтому поверхностно и недостаточно детально обсуждают особенности эволюции и генетической структуры популяции V. underwoodi, не приводят детальный анализ полиморфизма нуклеотидной последовательности гена COX1 мтДНК Varroa underwoodi из разных популяций, а также не характеризуют V. underwoodi как переносчика новых патогенов пчел – вирусов и бактерий. Существует необходимость в систематизации всех имеющихся знаний о клеще V. underwoodi, оценке возможных угроз V. underwoodi для других видов пчел р. Apis, на основе которых могут быть разработаны превентивные меры, направленные на предотвращение расширения ареала этого вида клеща в странах Азии и России.
Наряду с расширением ареала V. underwoodi растет число его видов-хозяев: A. cerana в Непале (Delfinado-Baker, Aggarwal, 1987), A. nuluensis в Малайзии (Delfinado-Baker, Aggarwal, 1987; Guzman et al., 1996; Anderson et al., 1997), A. nigrocincta в Индонезии (Anderson et al., 1997; Hadisoesilo, 1997), A. mellifera в Папуа-Новой Гвинее (Lee, 1995; Anderson et al., 1997; Guzman, Rinderer, 1999). Клещи V. underwoodi успешно размножаются в семьях пчел A. cerana, но обнаружение его в семьях других видов медоносных пчел позволяет предположить способность этих клещей к межвидовой смене хозяев. Особенно опасен он может быть для семей A. mellifera, содержащихся рядом с семьями A. cerana, как это принято в большинстве азиатских стран (Zheng et al., 2011, 2018; Chanta-wannakul et al., 2016; Wang et al., 2019a, b; Roberts et al., 2020).
Поскольку разные гаплотипы клещей Varroa имеют разную способность к паразитированию на разных видах пчел р. Apis (Anderson, 2000; Anderson, Trueman, 2000; Muñoz et al., 2008) (гаплотипы K и J из шести гаплотипов клещей V. destructor способны паразитировать на пчелах A. mellifera), то существует высокая вероятность того, что некоторые гаплотипы V. underwoodi смогут паразитировать на пчелах A. mellifera и затем, подобно V. destructor, распространиться по всему миру. V. underwoodi характеризуется высоким уровнем генетического разнообразия (Navajas et al., 2010; Roberts et al., 2015; Wang et al., 2019a). Высокий уровень генетического разнообразия этого вида позволяет быстро сформировать гаплотипы V. underwoodi, способные паразитировать на A. mellifera. Это подтверждается и законом гомологических рядов Н.И. Вавилова (1920), согласно которому у близких форм могут параллельно развиваться сходные признаки и, таким образом, способность видов V. destructor и V. jacobsoni к паразитированию на A. mellifera может также проявляться и у V. underwoodi. Следовательно, A. mellifera является потенциальным хозяином для V. underwoodi в ходе его дальнейшей эволюции (Anderson, 2000; Anderson, Trueman, 2000; Muñoz et al., 2008; Wang et al., 2019a, b; Ilyasov et al., 2021).
Переход V. underwoodi на нового хозяина A. mellifera может сопровождаться трансмиссией новых видов и штаммов вирусов и бактерий, провоцированием новых болезней, ведущих к нарушению микробиома, снижению выживаемости и иммунитета (Sandionigi et al., 2015; Hubert et al., 2017; Raymann et al., 2017; Diaz et al., 2019; Marche et al., 2019; Wang et al., 2019a, b; Bleau et al., 2020; Chen et al., 2021). Исследование особенностей V. underwoodi позволит заранее разработать методы борьбы с новым паразитом (Guzman, Rinderer, 1999; Kolar, Lodge, 2001; Woolhouse et al., 2005). Одним из таких методов является селекция семей пчел A. mellifera по гигиеническому поведению против клеща V. destructor, которая, вероятно, будет также работать и против клеща V. underwoodi (Mondragón et al., 2005; Allsopp, 2006; Locke, Fries, 2011; Çakmak, Fuchs, 2013; Locke, 2016; Conlon et al., 2018; McMullan, 2018; Alphen, Fernhout, 2020). В настоящей работе дана детальная характеристика клещей V. underwoodi в популяции A. cerana ussuriensis в Приморском крае на основе анализа морфометрии и полиморфизма гена COX1 мтДНК, систематизированы все имеющиеся знания о клеще V. underwoodi, проведена оценке возможных угроз V. underwoodi для других видов пчел р. Apis, в частности A. mellifera, предложены превентивные меры, направленные на предотвращение расширения ареала этого вида клеща в странах Азии и России.
МАТЕРИАЛЫ И МЕТОДЫ
Имаго клещей Varroa underwoodi собраны летом 2004 г. из выводковых ячеек двух семей № 2 и № 5 Apis cerana ussuriensis на пасеке в селе Ромашка Хасанского района Приморского края России (43.5° с.ш., 131.3° в.д.). Все собранные образцы клещей V. underwoodi были помещены в 70% этанол и хранились при –20°C.
Клещи V. underwoodi обнаружены только в семьях A. cerana ussuriensis, где уровень зараженности семей составлял 50%.
Для предварительного подтверждения видовой принадлежности морфометрические характеристики и размер взрослых самок клещей V. underwoodi (n = 10) сравнивали с литературными данными (Delfinado-Baker, Aggarwal, 1987; Woo, 1992; Anderson et al., 1997; Huang, 2004; Wang et al., 2019a). Перед оценкой морфометрии отобранные образцы клещей V. underwoodi высушивались при комнатной температуре в течение 1 мин. У клещей V. underwoodi для морфометрии использовался дорсальный щит с боковыми щетинками (Delfinado-Baker, Aggarwal, 1987; Woo, 1992; Anderson et al., 1997; Huang, 2004; Wang et al., 2019a). Измерение проводили с помощью цифрового микроскопа EOS Kiss X7 (Canon, Япония) с объективом MP-E 65mm f/2.8 1-5x Macro Photo (Canon, Япония) при увеличении ×150.
Тотальную ДНК экстрагировали из трех клещей на семью A. cerana ussuriensis с использованием набора Qiagen DNEasy для тканей животных с использованием колонок для связывания ДНК (Qiagen, Валенсия, Калифорния). Последовательность гена COX1 мтДНК использовали для идентификации вида клещей и определения гаплотипа мтДНК. ПЦР-амплификация гена COX1 мтДНК V. underwoodi проведена по методике Вана и соавторов (Wang et al., 2019a) с использованием пары праймеров (COX1_821_F: 5'-GGAGTAGGTACAGGTTGAACGG-3' и COX1_821_R: 5'-ACAACCCCAGCAATAATAGCAA-3') с продуктом 821 п.н. (Wang et al., 2019a).
Все продукты ПЦР были очищены с помощью набора QIAquick PCR Purification Kit (250) (QIAGEN, Хильден, Германия) в соответствии с инструкциями производителя. Нуклеотидные последовательности гена COX1 мтДНК образцов V. underwoodi определяли путем двухстороннего секвенирования продуктов ПЦР с использованием метода секвенирования Сэнгера (Sanger et al., 1977) и пары праймеров (F-V51: 5'-GTAATTTGTATACAAAGAGGG-3' и R-V1400: 5'-CAATATCAATAGAAGAATTAGC-3') (Warrit et al., 2004) на капиллярном секвенаторе ABI 3730xl (Applied Biosystems, Фостер-Сити, США) с помощью набора ABI PRISM BigDye Terminator v3.1 Cycle Sequencing Kit в соответствии с инструкциями производителя. Нуклеотидная последовательность гена COX1 мтДНК V. underwoodi размером 458 п.н. загружена в базу данных Генбанка DDBJ/GenBank под регистрационным номером LC532104.
Последовательности гена COX1 мтДНК V. underwoodi (MH205173 (Ханчжоу, Китай), MH205174 (Цзиньхуа, Китай), MH205175 (Наньчан, Китай), MH205176 (Цзилинь, Китай), MH205177 (Маомин, Китай)), V. destructor (KJ403739, KJ507740, KJ403742, KJ403744 (Эр-Рияд, Саудовская Аравия)) и V. jacobsoni (MF462134 (Порт-Морсби, Папуа-Новая Гвинея), AF010479 (Канберра, Австралия)) из Генбанка использованы для сравнительного анализа с V. underwoodi из Приморского края. Образцы клещей видов V. destructor и V. jacobsoni использованы также для сравнительного анализа в качестве внешних групп.
Уровень генетической дивергенции и p-дистанции между видами клещей V. underwoodi, V. destructor и V. jacobsoni оценивали на основе последовательностей гена COX1 мтДНК с применением метода выравнивания CLUSTALW в MEGA 10.0.5 (Kumar et al., 2018). Дендрограмма филогенетических отношений методом ближайшего соседа, основанная на p-дистанции последовательностей гена COX1 мтДНК построена с 2000 бутстрэп-репликациями в CLC Genomics Workbench 21 (Qiagen Inc., Миссиссога, Канада). Статистический анализ и анализ молекулярной дисперсии (AMOVA) выполняли с использованием ARLEQUIN 3.5.2 (Excoffier, Lischer, 2010), STATISTICA 8.0 (StatSoft, США) и EXCEL 2010 (Microsoft, США).
РЕЗУЛЬТАТЫ
Таксономическая принадлежность образцов клеща V. underwoodi определена с использованием данных морфометрии и полиморфизма гена COX1 мтДНК. Эллипсоидальное тело самок V. underwoodi каштаново-коричневое. Поверхность дорсального щита слегка бороздчатая, сетчатая, с густыми наклоненными щетинками примерно одинаковой длины и небольшими шипиками. Боковые щетинки постепенно удлиняются кзади, последние три укорачиваются. Длина тела взрослой самки V. underwoodi 767.5 ± 20.5 мкм (среднее значение ± стандартное отклонение), ширина 1300.5 ± 20.5 мкм (n = 10). Для сравнения, длина и ширина тела взрослых самок V. underwoodi в семьях A. cerana – 700–752 мкм × 1089–1157 мкм (n = 15); в семьях A. mellifera – 700–735 мкм × × 1090–1120 мкм (n = 6); в семьях A. cerana (провинция Западное Папуа, Индонезия) – 690–730 мкм × × 1050–1130 мкм (n = 5); в семьях A. cerana (Сулавеси, Ява, Индонезия) – 720–780 мкм × 1050–1080 мкм (n = 2); в семьях A. nigrocincta (Сулавеси) – 740–760 мкм × 1120–1220 мкм (n = 5) (Anderson et al., 1997); в семьях A. cerana (Непал) – 741–780 мкм × × 1151–1168 мкм (n = 2) (Delfinado-Baker, Aggarwal, 1987); в семьях A. cerana (Южная Корея) – 703–784 мкм × 1135–1324 мкм (n = 2) (Woo, 1992). Морфологические параметры наших экземпляров соответствуют ранее опубликованным данным по V. underwoodi (Delfinado-Baker, Aggarwal, 1987; Anderson et al., 1997; Huang, 2004; Wang et al., 2019a).
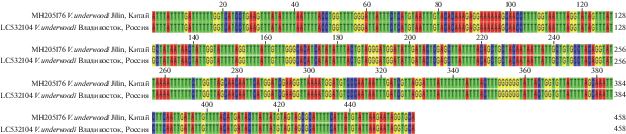
Был проведен сравнительный анализ нуклеотидной последовательности гена COX1 мтДНК видов клещей V. underwoodi, V. destructor и V. jacobsoni. Выровненные нуклеотидные последовательности гена COX1 мтДНК позволяют рассчитать различия нуклеотидов в соответствующих позициях у разных образцов клещей (рис. 1).
Рис. 1.
Сравнительный анализ нуклеотидной последовательности гена COX1 мтДНК клещей Varroa underwoodi, V. destructor и V. jacobsoni.

Все различия между V. underwoodi, V. destructor и V. jacobsoni рассчитаны на основе полиморфизма последовательностей гена COX1 мтДНК. В табл. 1 представлены усредненные попарные оценки числа нуклеотидных и аминокислотных замен, p‑дистанции и процент генетической дивергенции на основе последовательностей гена COX1 мтДНК клещей V. underwoodi (LC532104 (Приморский край, Россия), MH205173 (Ханчжоу, Китай), MH205174 (Цзиньхуа, Китай), MH205175 (Наньчан, Китай), MH205176 (Цзилинь, Китай), MH205177 (Маомин, Китай)), V. destructor (KJ403739, KJ507740, KJ403742, KJ403744 (Эр-Рияд, Саудовская Аравия)) и V. jacobsoni (MF462134 (Порт-Морсби, Папуа-Новая Гвинея), AF010479 (Канберра, Австралия)). Обнаружено, что нуклеотидные последовательности гена COX1 мтДНК образцов V. underwoodi LC532104 из Приморского края (Россия) идентичны MH205176 из провинции Цзилинь (Китай) (Wang et al., 2019a).
Таблица 1.
Оценка средних значений p-дистанции и генетической дивергенции (ниже диагонали) и замен нуклеотидов и аминокислот (выше диагонали) между видами клещей Varroa на основе последовательности гена COX1 мтДНК
| Виды Varroa | V. underwoodi | V. destructor | V. jacobsoni | |
|---|---|---|---|---|
| n = 6 | n = 4 | n = 2 | ||
| число нуклеотидных замен/ число аминокислотных замен | ||||
| V. underwoodi | p-дистанция/генетическая дивергенция, % | 45/36 | 44/34 | |
| V. destructor | * 0.099/10 | 33/26 | ||
| V. jacobsoni | * 0.097/10 | * 0.072/7 | ||
Последовательности гена COX1 мтДНК видов клещей V. underwoodi, V. destructor и V. jacobsoni различаются на статистически значимом уровне (p ≤ 0.05). Нуклеотидная последовательность гена COX1 мтДНК позволяет с 95%-й вероятностью различать виды клещей р. Varroa. Из трех видов клещей наиболее близки друг к другу V. destructor и V. jacobsoni со значением генетической дивергенции 7%. V. underwoodi равноудален от V. destructor и V. jacobsoni и отличается от них по значению генетической дивергенции 10%.
В табл. 2 представлены попарные генетические дистанции, генетическая дивергенция, число замен нуклеотидов и аминокислот между каждым образцом V. underwoodi (n = 6), V. destructor (n = 4) и V. jacobsoni (n = 2), рассчитанные на основе сравнения последовательностей гена COX1 мтДНК.
Таблица 2.
Попарные различия и генетическая дивергенция между видами Varroa, рассчитанные на основе полиморфизма последовательностей гена COX1
| Виды Varroa | 1. MH205176, V. underwoodi | 2. LC532104, V. underwoodi | 3. MH205175, V. underwoodi | 4. MH205174, V. underwoodi | 5. MH205173, V. underwoodi | 6. MH205177, V. underwoodi | 7. KJ403744, V. destructor | 8. KJ403742, V. destructor | 9. KJ507740, V. destructor | 10. KJ403739, V. destructor | 11. MF462134, V. jacobsoni | 12. AF010479, V. jacobsoni | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Число нуклеотидных замен / Число аминокислотных замен | |||||||||||||
| 1. MH205176, V. underwoodi, Цзилинь, Китай | p-дистанция/генетическая дивергенция % | 0/0 | 1/1 | 1/1 | 2/2 | 10/8 | 44/36 | 46/36 | 46/36 | 44/35 | 44/34 | 4 /34 | |
| 2. LC532104, V. underwoodi, Приморский край, Россия | 0.000/0 | 1/1 | 1/1 | 2/2 | 10/8 | 44/36 | 46/36 | 46/36 | 44/35 | 44/34 | 44/34 | ||
| 3. MH205175, V. underwoodi, Наньчан, Китай | 0.002/0 | 0.002/0 | 0/0 | 1/1 | 10/8 | 45/37 | 47/37 | 47/37 | 45/36 | 45/35 | 45/35 | ||
| 4. MH205174, V. underwoodi, Цзиньхуа, Китай | 0.002/0 | 0.002/0 | 0.000/0 | 1/1 | 10/8 | 45/37 | 47/37 | 47/37 | 45/6 | 45/35 | 45/35 | ||
| 5. MH205173, V. underwoodi, Ханчжоу, Китай | 0.004/0 | 0.004/0 | 0.002/0 | 0.002/0 | 9/7 | 45/37 | 47/37 | 47/37 | 45/36 | 45/35 | 45/35 | ||
| 6. MH205177, V. underwoodi, Маомин, Китай | 0.022/2 | 0.022/2 | 0.022/2 | 0.022/2 | 0.020/2 | 42/36 | 44/36 | 44/36 | 42/35 | 44/36 | 44/36 | ||
| 7. KJ403744, V. destructor, Саудовская Аравия | 0.096/10 | 0.096/10 | 0.098/10 | 0.098/10 | 0.098/10 | 0.092/9 | 4/2 | 4/2 | 2/1 | 32/27 | 32/27 | ||
| 8. KJ403742, V. destructor, Саудовская Аравия | 0.100/10 | 0.100/10 | 0.103/10 | 0.103/10 | 0.103/10 | 0.096/10 | 0.009/1 | 0/0 | 4/1 | 34/27 | 34/27 | ||
| 9. KJ507740, V. destructor, Саудовская Аравия | 0.100/10 | 0.100/10 | 0.103/ 0 | 0.103/10 | 0.103/10 | 0.096/10 | 0.009/1 | 0.000/0 | 4/1 | 34/27 | 34/27 | ||
| 10. KJ403739, V. destructor, Саудовская Аравия | 0.096/10 | 0.096/10 | 0.098/10 | 0.098/10 | 0.098/10 | 0.092/9 | 0.004/1 | 0.009/1 | 0.009/1 | 32/26 | 32/26 | ||
| 11. MF462134
V. jacobsoni, Папуа-Новая Гвинея |
0.096/10 | 0.096/10 | 0.098/10 | 0.098/10 | 0.098/10 | 0.096/10 | 0.070/7 | 0.074/7 | 0.074/7 | 0.070/7 | 0/0 | ||
| 12. AF010479, V. jacobsoni, Австралия | 0.096/10 | 0.096/10 | 0.098/10 | 0.098/10 | 0.098/10 | 0.096/10 | 0.070/7 | 0.074/7 | 0.074/7 | 0.070/7 | 0.000/0 | ||
Между образцами гена COX1 мтДНК V. underwoodi значения генетической дивергенции варьировали от 0 до 2%, p-дистанции – от 0.000 до 0.022, количества нуклеотидных замен – от 0 до 10, аминокислотных замен – от 0 до 8. Не наблюдалось генетических различий между образцами MH205176, V. underwoodi (Цзилинь, Китай) и LC532104, V. underwoodi (Приморский край, Россия), а также MH205175, V. underwoodi, (Наньчан, Китай) и MH205174, V. underwoodi (Цзиньхуа, Китай). Наименьшие генетические различия наблюдались между образцами MH205176, V. underwoodi (Цзилинь, Китай) и MH205175, V. underwoodi (Наньчан, Китай); LC532104, V. underwoodi (Приморский край, Россия) и MH205175, V. underwoodi (Наньчан, Китай); MH205176, V. underwoodi (Цзилинь, Китай) и MH205174, V. underwoodi (Цзиньхуа, Китай); LC532104, V. underwoodi (Приморский край, Россия) и MH205174, V. underwoodi (Цзиньхуа, Китай). Наибольшие генетические различия наблюдались между MH205177 (Маомин, Китай) и остальными образцами V. underwoodi. Выравнивание нуклеотидных последовательностей гена COX1 мтДНК MH205176 (Цзилинь, Китай) и LC532104 (Приморский край, Россия) показывает отсутствие нуклеотидных замен между ними. Эти последовательности гена COX1 мтДНК обозначены как гаплотип China 1 MH205176 (рис. 2) (Ilyasov et al., 2021).
Рис. 2.
Сравнительный анализ нуклеотидной последовательности гена COX1 мтДНК Varroa underwoodi из Китая (Цзилинь) и России (Приморский край) – гаплотип China 1 MH205176.
Между образцами V. destructor генетическая дивергенция варьировала от 0 до 1%, p-дистанция – от 0.000 до 0.022, количество нуклеотидных замен – от 0 до 4, а количество аминокислотных замен – от 0 до 1. Наименьшие генетические различия наблюдались между образцами V. destructor KJ403744 и KJ403739 из Саудовской Аравии (Эр-Рияд). Не обнаружено генетических различий между образцами V. destructor KJ403742 и KJ403740 из Саудовской Аравии (Эр-Рияд), а также между образцами V. jacobsoni MF462134 из Папуа-Новой Гвинеи (Порт-Морсби) и AF010479 из Австралии (Канберра).
Нами построена дендрограмма филогенетических отношений методом ближайшего соседа с 2000 бутстрэп репликациями на основе p-дистанций между последовательностями гена COX1 мтДНК трех видов клещей V. underwoodi, V. destructor, V. jacobsoni. Последовательность гена COX1 мтДНК LC532104 V. underwoodi из России (Приморский край) (гаплотип China 1 MH205176) объединена в один кластер со всеми последовательностями представителей V. underwoodi MH205173, MH205174, MH205176, MH205177 из Китая. Отдельными кластерами располагаются последовательности COX1 мтДНК представителей V. destructor KJ403739, KJ403742, KJ403742, KJ403742 (Эр-Рияд, Саудовская Аравия) и представителей V. jacobsoni MF462134 (Порт-Морсби, Папуа-Новая Гвинея), AF010479 (Канберра, Австралия) (рис. 3).
Рис. 3.
Дендрограмма филогенетических отношений методом ближайшего соседа с 2000 бутстрэп-репликациями на основе p-дистанций между последовательностями гена COX1 мтДНК трех видов Varroa. Цифры на каждой ветви указывают генетические дистанции.
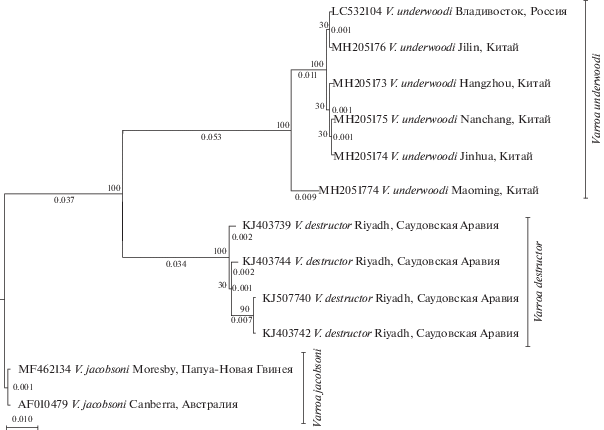
На дендрограмме филогенетических отношений в кластере V. underwoodi образец LC532104 (Приморский край, Россия) располагается наиболее близко к самому северному образцу MH205176 (Цзилинь, Китай), находящемуся на удалении 450 км, а наиболее удаленно – от самого южного образца MH205177 (Маомин, Китай), находящегося на удалении 3000 км. Кластер V. underwoodi генетически ближе к кластеру V. destructor, чем к кластеру V. jacobsoni. Южный образец MH205177 V. underwoodi (Маомин, Китай) наиболее удален от остальных образцов V. underwoodi и ближе к кластеру V. destructor (рис. 3).
ОБСУЖДЕНИЕ
Клещ Varroa underwoodi, вероятно, произошел из южных регионов Азии, а позже распространился на север в результате совместной миграции с пчелами Apis cerana. Предыдущие исследования, основанные на изучении морфологии и гена COX1 мтДНК, показали паразитирование клеща V. underwoodi в семьях пчел A. cerana в Непале (Delfinado-Baker, Aggarwal, 1987), Южной Корее (Woo, 1992; Кузнецов, 2005; Chantawannakul et al., 2016), Индонезии (Anderson et al., 1997; Chanta-wannakul et al., 2016), Папуа-Новой Гвинее (Lee, 1995; Anderson et al., 1997; Chantawannakul et al., 2016), Китае (Huang, 2004; Wang et al., 2019a), Вьетнаме и Японии (Guzman, Rinderer, 1999; Chantawannakul et al., 2016). Сравнительно недавно V. underwoodi выявлен и в России (Приморский край) (Кузнецов, 2005; Кузнецов, Лелей, 2005; Ilyasov et al., 2021). Нами показано распространение V. underwoodi на Дальнем Востоке России в семьях пчел A. cerana ussuriensis с использованием методов морфометрии и секвенирования митохондриального гена COX1 мтДНК (рис. 1, 2) (Ilyasov et al., 2021). Место обнаружения V. underwoodi в Приморье находится на удалении 450 км от места обнаружения этого вида в Северном Китае (Ilyasov et al., 2021). По результатам сравнительного анализа гена COX1 мтДНК (рис. 3) все образцы V. underwoodi объединяются в один кластер, кроме образца MH205177 (Маомин, Китай), который группируется отдельно (рис. 3). Вероятно, данный образец следует отнести к новому подвиду V. underwoodi или даже новому виду в р. Varroa, что может выясниться после дополнительного специального исследования.
Клещи V. underwoodi, вероятно, всегда встречались в небольшом количестве в природной популяции диких пчел A. cerana, обитающих в дуплах деревьев в Приморском крае в России. В ульях A. cerana условия для размножения V. underwoodi оказались более благоприятными, чем в бортях в дуплах деревьев, что привело к возрастанию численности клещей на пасеках. По данным В.Н. Кузнецова (2005), в 2002 г. в трутневом расплоде A. cerana на пасеке клещи V. underwoodi обнаружены только один раз, но в 2004 г. наблюдалось массовое размножение V. underwoodi. В 2004 г. расплод трутней на пасеке был заражен клещами V. underwoodi на 2.8% в июне, на 35% в июле и на 58% в августе, а рабочие пчелы были заражены только на 1%. Следует отметить, что отдельные трутневые ячейки A. cerana содержали до 5–6 имаго и 2–3 нимфы V. underwoodi. Имаго и нимфы V. underwoodi обнаружены на куколках трутней A. cerana и редко наблюдались в выводке рабочих пчел. Самки V. underwoodi в основном встречались на молодых трутнях и очень редко на молодых рабочих пчелах A. cerana. Летом 2004 г. до 16% молодых трутней были заражены клещами V. underwoodi, но осенью клещи у взрослых пчел отсутствовали. Возможно, что большое количество клещей на расплоде пчел способствовало активному роению семей A. cerana для очистки семьи пчел от клещей. В 2004 г. наблюдалось частое роение семей A. cerana на пасеке, сильно пораженной клещами V. underwoodi (Кузнецов, 2005). Сейчас в России пчелы A. cerana не разводятся в специальных ульях на пасеках для получения меда. Этот вид медоносной пчелы сохранился на Дальнем Востоке России в диком состоянии и обитает в лесах в дуплах деревьев.
Уровень заражения пчел клещами V. underwoodi значительно выше в северных провинциях Китая, чем в южных (Wang et al., 2019a). Это связано с тем, что массовое размножение клещей происходит в семьях пчел, выращивающих расплод в зимний безоблетный период. На более высокий уровень заражения V. underwoodi в Северной Азии влияет время выращивания расплода трутней, которое немного короче в холодном климате (Wang et al., 2019a). Идентичность последовательностей гена COX1 мтДНК V. underwoodi из Приморского края (Россия) LC532104 и провинции Цзилинь (Китай) MH205176 позволяет предположить, что гаплотип China 1 MH205176 V. underwoodi является адаптированным к паразитированию на пчелах A. cerana, обитающих в холодном климате Северной Азии. Возможно, что дальневосточный регион заселен единой популяцией V. underwoodi гаплотипа China 1 MH205176, распространенной на обширной территории Северной Азии в результате миграции семей A. cerana (Traynor et al., 2020; Ilyasov et al., 2021).
Клещи Varroa, собранные в Приморском крае (Россия), идентифицированы как V. underwoodi на основании их морфометрии, которая находится в диапазоне ранее описанных популяций этого вида (Delfinado-Baker, Aggarwal, 1987; Anderson et al., 1997; Huang, 2004; Wang et al., 2019a), а также последовательности гена COX1 мтДНК, который был идентичен с образцом MH205176 из провинции Цзилинь (Китай) (табл. 2, рис. 2) (Wang et al., 2019a; Ilyasov et al., 2021). Длина и ширина тела взрослых самок V. underwoodi из Приморского края немного больше, чем у представителей этого вида из южных популяций, что можно объяснить его северным распространением. Скорость молекулярной эволюции у видов клещей р. Varroa очень низкая, а их геномы более консервативны, чем геномы их видов-хозяев пчел р. Apis (Ilyasov et al., 2021). Эти генетические особенности представителей V. underwoodi из удаленных друг от друга регионов Дальнего Востока России и Северного Китая можно объяснить паразитическим образом жизни внутри семей A. cerana. Аналогичным образом можно объяснить отсутствие различий между образцами V. destructor из отдаленных регионов Саудовской Аравии, с одной стороны, и Папуа-Новой Гвинеи и Австралии – с другой.
Средний уровень генетической дивергенции между V. destructor и V. jacobsoni в данной работе (7%) очень близок к таковой (6%) между V. destructor из семи стран (Южная Корея, Франция, Вьетнам, Китай, Япония, Непал, Шри Ланка) и V. jacobsoni из четырех стран (Индонезия, Малайзия, Лаос, Папуа-Новая Гвинея) (табл. 1) (Techer et al., 2019). Средняя генетическая дивергенция между тремя видами клещей V. underwoodi, V. destructor и V. jacobsoni по нуклеотидной последовательности гена COX1 мтДНК варьирует в диапазоне от 7 до 10%, а p-дистанция – от 0.072 до 0.099. Это согласуется с диапазоном генетических различий между видами насекомых (8–17% и 0.100–0.200 соответственно) (Tan et al., 2007; Han et al., 2016; Eimanifar et al., 2017; Ilyasov et al., 2018, 2019).
Наличие клещей V. underwoodi в закрытых ячейках расплода рабочих в одной семье A. mellifera в Папуа-Новой Гвинее (Roberts et al., 2015) показывает, что может произойти межвидовая смена хозяина и переход V. underwoodi с A. cerana на A. mellifera. В настоящее время V. underwoodi не выявлен в массовом порядке на пасеках в семьях A. mellifera на Дальнем Востоке России. Однако миграция A. cerana между Россией и Китаем может привести к появлению на Дальнем Востоке России новых гаплотипов V. underwoodi из Китая, способных к паразитированию в семьях A. mellifera. Клещ V. underwoodi генетически близок к V. destructor, который является обычным паразитом как A. mellifera, так и A. cerana, поэтому весьма вероятен переход V. underwoodi на A. mellifera (Roberts et al., 2020). Надо быть готовым к появлению у пчел A. mellifera нового паразита V. underwoodi с разрушительными эффектами для популяции (Anderson, 2000; Anderson, Trueman, 2000; Muñoz et al., 2008; Rosenkranz et al., 2010; Roberts et al., 2015; Wang et al., 2019a; Ilyasov et al., 2021).
Разрушительные эффекты для пчел A. mellifera могут быть вызваны больше не самими клещами, а трансмиссией новых, не характерных для данного вида патогенов – вирусов и бактерий. Паразитические клещи V. destructor и V. underwoodi имеют различный видовой состав микробиома кишечника и могут быть переносчиками разных видов вирусов (восемь РНК-вирусов и один ДНК-вирус) (Sandionigi et al., 2015; Wang et al., 2019b; Sacca, Lodesani, 2020; Chen et al., 2021). В популяции A. cerana клещи V. destructor переносят вирусы DWV, IAPV, BQCV, KBV, CBPV, SBV и AmFV, а V. underwoodi переносят вирусы DWV, CBPV, AmFV, BQCV, IAPV и KBV (Wang et al., 2019b; Chen et al., 2021). Кроме того, один и тот же вирус имеет разное действие на разные виды пчел. Так, вирус мешотчатого расплода Korean Sacbrood Virus (kSBV) уничтожил 95% популяции A. cerana и не был вирулентен для популяции A. mellifera (Choi et al., 2010; Koetz, 2013; Vung et al., 2017; Wang et al., 2019b). В семьях пчел A. mellifera, инфицированных паразитическим клещом V. destructor, наблюдается повышенное количество бактерий Snod-grassella alvi и уменьшение количества бактерий сем. Lactobacillaceae в кишечнике рабочих особей (Hubert et al., 2017; Marche et al., 2019; Bleau et al., 2020). Микробиом пораженных клещом личинок становится сходным с микробиомом V. destructor, что свидетельствует об обмене микробиомом кишечника между пчелой и эктопаразитическим клещом (Sandionigi et al., 2015). Искусственное заражение пчел патогенной микроспоридией Nosema ceranae также изменяет состав микробиома кишечника и провоцирует рост численности бактерии Gilliamella apicola (Rubanov et al., 2019). Заражение паразитическим клещом V. destructor является более важным фактором нарушения и изменения состава микробиома кишечника взрослых пчел A. mellifera, чем заражение микроспоридиями N. ceranae, N. apis и трипаносомой Lotmaria passim (Hubert et al., 2017). Для борьбы с этими паразитами пчеловоды часто используют химические вещества, такие как фумагиллин (против Nosema spp.) и щавелевую кислоту (против V. destructor), которые, в свою очередь, уменьшают разнообразие и численность бактерий микробиома кишечника A. mellifera (Raymann et al., 2017; Diaz et al., 2019). Такие изменения микробиома кишечника пчел могут негативно влиять на физиологию, иммунитет, выживаемость и адаптацию к условиям окружающей среды (Bleau et al., 2020; Sacca, Lodesani, 2020; Ильясов и др., 2021).
Для борьбы с распространением V. destructor и предотвращения потенциального перехода V. underwoodi возможно применять селекции пчел по гигиеническому поведению, устойчивых к паразитированию клещами Varroa spp. Сейчас в мире существует девять устойчивых к клещу V. destructor популяций медоносной пчелы A. mellifera, которые были получены путем целенаправленной селекции: 1) популяция медоносных пчел северного графства Дублин в Ирландии; 2) популяция медоносных пчел подвида A. m. scutellata в Бразилии и Южной Африке; 3) популяция медоносных пчел в Тулузе во Франции; 4) популяция медоносных пчел на острове Фернандо де Норонья в Бразилии; 5) приморская популяция медоносных пчел в Приморском крае России; 6) популяция медоносных пчел Готланд в Швеции; 7) популяция медоносных пчел Авиньон во Франции; 8) популяция медоносных пчел Арнотского леса в Итаке в штате Нью-Йорк США; 9) популяция медоносных пчел на острове Мармара в Турции (Mondragón et al., 2005; Allsopp, 2006; Locke, Fries, 2011; Çakmak, Fuchs, 2013; Locke, 2016; Conlon et al., 2018; McMullan, 2018; Alphen, Fernhout, 2020). Селекция семей медоносных пчел по гигиеническому поведению на устойчивость к клещу V. destructor позволит использовать меньше акарицидов и предотвратить нарушения микробиома кишечника, обеспечивающего защитную функцию организма и играющего важную роль в иммунитете и адаптации пчел к условиям окружающей среды (Çakmak, Fuchs, 2013). Селекция семей пчел по гигиеническому поведению против клеща V. destructor может также оказаться эффективной против клеща V. underwoodi.
ЗАКЛЮЧЕНИЕ
До недавнего времени известный ареал клещей Varroa underwoodi охватывал практически все страны, где встречается медоносная пчела Apis cerana, включая и Дальний Восток России. Идентичность последовательностей гена COX1 у экземпляров V. underwoodi из Северного Китая и Приморского края России, удаленных между собой на 450 км, указывает на возможную свободную миграцию A. cerana и расселению паразита, а также на низкую скорость молекулярной эволюции генома V. underwoodi в результате паразитического образа жизни. Хотя северные границы ареала V. underwoodi еще не исследованы, но в 2020 г. образцы A. cerana ussuriensis собраны в районе села Терней Приморского края (45.06° с.ш., 136.61° в.д.), северной границе ареала данного вида пчел. Возможно, таким образом, что северная граница ареала клеща V. underwoodi будет совпадать с северной границей ареала A. cerana. В дальнейшем планируется охарактеризовать популяцию V. underwoodi с помощью дополнительных маркеров COX3, ATP6 и CYTB, которые позволят найти генетические различия между российскими и китайскими образцами V. underwoodi и выявить его биогеографические связи. Медоносная пчела A. mellifera может стать новым хозяином для паразитического клеща V. underwoodi, поскольку он широко встречается в семьях A. cerana, находящихся рядом с семьями A. mellifera. Возможный переход клеща V. underwoodi на нового хозяина A. mellifera может сопровождаться трансмиссией новых видов и штаммов вирусов и бактерий, изменением микробиома кишечника, подавлением иммунитета и адаптации к изменяющимся климатическим условиям. Селекция семей медоносных пчел по гигиеническому поведению на устойчивость к клещу V. destructor может стать эффективной защитой от перехода близкородственного клеща V. underwoodi с A. cerana на A. mellifera.
Список литературы
Вавилов Н.И., 1920. Закон гомологических рядов в наследственной изменчивости // Тр. 3-го Всеросс. съезда по селекции и семеноводству. Саратов: Губполиграфотдел, 3-е отд. С. 41–56.
Ильясов Р.А., Марсова М.В., Ковтун А.С., Ватлин А.А., Юнес Р.А. и др., 2021. Роль микробиома кишечника медоносных пчел // Пчеловодство. № 7. С. 18–20.
Кузнецов В.Н., 2005. Китайская восковая пчела Apis cerana cerana F. (Hymenoptera, Apidae) на Дальнем Востоке России. М.: Т-во науч. изд. КМК. 112 с.
Кузнецов В.Н., Лелей А.С., 2005. О паразитировании клещей рода Varroa Oudemans, 1904 (Acari: Varroidae) на китайской восковой пчеле Apis cerana cerana Fabricius, 1793 (Hymenoptera: Apidae) в Приморском крае // Чтения памяти А.И. Куренцова. № 16. С. 39–46.
Allsopp M.H., 2006. Analysis of Varroa destructor infestation of Southern African honeybee populations. MSc dissertation. Pretoria: Univ. Pretoria. 285 p.
Alphen J.J.M., van, Fernhout B.J., 2020. Natural selection, selective breeding, and the evolution of resistance of honeybees (Apis mellifera) against Varroa // Zool. Lett. V. 6. https://doi.org/10.1186/s40851-020-00158-4
Anderson D.L., 2000. Variation in the parasitic bee mite Varroa jacobsoni Oud. // Apidologie. V. 31. № 2. P. 281–292.
Anderson D.L., Trueman J.W., 2000. Varroa jacobsoni (Acari: Varroidae) is more than one species // Exp. Appl. Acarol. V. 24. № 3. P. 165–189.
Anderson D.L., Halliday R.B., Otis G.W., 1997. The occurrence of Varroa underwoodi (Acarina: Varroidae) in Papua New Guinea and Indonesia // Apidologie. V. 28. № 3–4. P. 143–147.
Bleau N., Bouslama S., Giovenazzo P., Derome N., 2020. Dynamics of the honeybee (Apis mellifera) gut microbiota throughout the overwintering period in Canada // Microorganisms. V. 8. № 8. https://doi.org/10.3390/microorganisms8081146
Çakmak I., Fuchs S., 2013. Exploring a treatment strategy for long-term increase of Varroa tolerance on Marmara Island, Turkey // J. Apic. Res. V. 52. № 5. P. 242–250.
Chantawannakul P., Guzman L.I., de, Li J., Williams G.R., 2016. Parasites, pathogens, and pests of honeybees in Asia // Apidologie. V. 47. P. 301–324.
Chen G., Wang S., Jia S., Feng Y., Hu F. et al., 2021. A new strain of virus discovered in China specific to the parasitic mite Varroa destructor poses a potential threat to honey bees // Viruses. V. 13. https://doi.org/10.3390/v13040679
Choi Y.S., Lee M.Y., Hong I.P., Kim N.S., Kim H.K., et al., 2010. Occurrence of sacbrood virus in Korean apiaries from Apis cerana (Hymenoptera: Apidae) // J. Apic. V. 25. P. 187–191.
Conlon B.H., Frey E., Rosenkranz P., Locke B., Moritz R.F.A., Routtu J., 2018. The role of epistatic interactions underpinning resistance to parasitic Varroa mites in haploid honey bee (Apis mellifera) drones // J. Evol. Biol. V. 31. № 6. P. 801–809.
Delfinado-Baker M., Aggarwal K., 1987. A new Varroa (Acari: Varroidae) from the nest of Apis cerana (Apidae) // Int. J. Acarology. V. 13. № 4. P. 233–237.
Diaz T., Del-Val E., Ayala R., Larsen J., 2019. Alterations in honey bee gut microorganisms caused by Nosema spp. and pest control methods // Pest Manag. Sci. V. 75. № 3. P. 835–843.
Eimanifar A., Kimball R.T., Braun E.L., Moustafa D.M., Haddad N. et al., 2017. The complete mitochondrial genome of the Egyptian honey bee, Apis mellifera lamarckii (Insecta: Hymenoptera: Apidae) // Mitochondrial DNA B Resour. V. 2. № 1. P. 270–272.
Excoffier L., Lischer H.E., 2010. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Resour. V. 10. № 3. P. 564–567.
Hadisoesilo S., 1997. A comparative study of two species of cavity-nesting honey bees of Sulawesi, Indonesia. PhD thesis. Guelph: The Univ. Guelph. 199 p.
Hadisoesilo S., Otis G.W., 1998. Differences in drone cappings of Apis cerana and Apis nigrocincta // J. Apic. Res. V. 37. № 1. P. 11–15.
Han T., Lee W., Lee S., Park I.G., Park H., 2016. Reassessment of species diversity of the subfamily Denticollinae (Coleoptera: Elateridae) through DNA barcoding // PLoS One. V. 11. № 2. https://doi.org/10.1371/journal.pone.0148602
Huang S., 2004. The main kinds of honeybee ectoparasitic mites and the first found of Varroa underwoodi in China // Apic. China. V. 55. P. 6. (In Chinese).
Hubert J., Bicianova M., Ledvinka O., Kamler M., Lester P.J. et al., 2017. Changes in the bacteriome of honey bees associated with the parasite Varroa destructor, and pathogens Nosema and Lotmaria passim // Microb. Ecol. V. 73. № 3. P. 685–698.
Guzman L.I., de, Delfinado-Baker M., 1996. A new species of Varroa (Acari: Varroidae) associated with Apis koschevnikovi (Apidae: Hymenoptera) in Borneo // Int. J. Acarology. V. 22. № 1. P. 23–27.
Guzman L.I., de, Rinderer T.E., 1999. Identification and comparison of Varroa species infesting honey bees // Apidologie. V. 30. № 2–3. P. 85–95.
Guzman L.I., de, Rinderer T.E., Delatte G.T., Macchiavelli R.E., 1996. Varroa jacobsoni Oudemans tolerance in selected stocks of Apis mellifera L. // Apidologie. V. 27. № 4. P. 193–210.
Ilyasov R.A., Park J., Takahashi J., Kwon H.W., 2018. Phylogenetic uniqueness of honeybee Apis cerana from the Korean peninsula inferred from the mitochondrial, nuclear, and morphological data // J. Apic. Sci. V. 62. № 2. P. 189–214.
Ilyasov R.A., Han G.Y., Lee M.-L., Kim K.W., Proshchalykin M.Y. et al., 2019. Phylogenetic relationships of Russian Far-East Apis cerana with other North Asian populations // J. Apic. Sci. V. 63. № 2. P. 289–314.
Ilyasov R.A., Takahashi J.I., Proshchalykin M.Y., Lelej A.S., Lee M.L. et al., 2021. First evidence of presence of Varroa underwoodi mites on native Apis cerana colonies in Primorsky Territory of Russia based on COX1 gene // J. Apic. Sci. V. 65. № 1. P. 177–186.
Koetz A.H., 2013. Ecology, behaviour and control of Apis cerana with a focus on relevance to the Australian incursion // Insects. V. 4. P. 558–592.
Kolar C.S., Lodge D.M., 2001. Predicting invaders: Response from Kolar and Lodge // Trends Ecol. Evol. V. 16. № 10. P. 546.
Kumar S., Stecher G., Li M., Knyaz C., Tamura K., 2018. MEGA X: Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evol. V. 35. № 6. P. 1547–1549.
Lee B., 1995. Mites, bees, and plagues that are and might be // Partners Res. Dev. № 8. P. 2–9.
Lin Z., Wang S., Neumann P., Chen G., Page P. et al., 2021. Population genetics and host specificity of Varroa destructor mites infesting eastern and western honeybees // J. Pest Sci. V. 94. № 4. P. 1487–1504.
Locke B., 2016. Natural Varroa mite-surviving Apis mellifera honeybee populations // Apidologie. V. 47. № 3. P. 467–482.
Locke B., Fries I., 2011. Characteristics of honey bee colonies (Apis mellifera) in Sweden surviving Varroa destructor infestation // Apidologie. V. 42. № 4. P. 533–542.
Marche M.G., Satta A., Floris I., Pusceddu M., Buffa F., Ruiu L., 2019. Quantitative variation in the core bacterial community associated with honey bees from Varroa-infested colonies // J. Apic. Res. V. 58. № 3. P. 444–454.
McMullan J., 2018. Adaptation in honey bee (Apis mellifera) colonies exhibiting tolerance to Varroa destructor in Ireland // Bee World. V. 95. № 2. P. 39–43.
Mondragón L., Spivak M., Vandame R., 2005. A multifactorial study of the resistance of honeybees Apis mellifera to the mite Varroa destructor over one year in Mexico // Apidologie. V. 36. № 3. P. 345–358.
Muñoz I., Dall’Olio R., Lodesani M., De La Rúa P., 2008. Sequence variation in the mitochondrial tRNAleu-COX2 intergenic region of African and African-derived honey bee populations // EurBee 3. 3rd European Conference of Apidology. Belfast: Queens Univ. P. 73.
Navajas M., Anderson D.L., Guzman L.I., de, Huang Z.Y., Clement J. et al., 2010. New Asian types of Varroa destructor: A potential new threat for world apiculture // Apidologie. V. 41. P. 181–193.
Oudemans A.C., 1904. On a new genus and species of parasitic Acari // Notes from the Leyden Museum. V. 24. P. 216–222.
Rath W., 1999. Co-adaptation of Apis cerana Fabr. and Varroa jacobsoni Oud. // Apidologie. V. 30. № 2–3. P. 97–110.
Raymann K., Shaffer Z., Moran N.A., 2017. Antibiotic exposure perturbs the gut microbiota and elevates mortality in honeybees // PLoS Biol. V. 15. № 3. https://doi.org/10.1371/journal.pbio.2001861
Roberts J.M., Anderson D.L., Tay W.T., 2015. Multiple host shifts by the emerging honeybee parasite, Varroa jacobsoni // Mol. Ecol. V. 24. № 10. P. 2379–2391.
Roberts J.M.K., Schouten C.N., Sengere R.W., Jave J., Lloyd D., 2020. Effectiveness of control strategies for Varroa jacobsoni and Tropilaelaps mercedesae in Papua New Guinea // Exp. Appl. Acarol. V. 80. № 3. P. 399–407.
Rosenkranz P., Aumeier P., Ziegelmann B., 2010. Biology and control of Varroa destructor // J. Invert. Pathol. V. 103. Suppl. 1. P. 96–119.
Rubanov A., Russell K.A., Rothman J.A., Nieh J.C., McFrederick Q.S., 2019. Intensity of Nosema ceranae infection is associated with specific honey bee gut bacteria and weakly associated with gut microbiome structure // Sci. Rep. V. 9. https://doi.org/10.1038/s41598-019-40347-6
Sacca M.L., Lodesani M., 2020. Isolation of bacterial microbiota associated to honey bees and evaluation of potential biocontrol agents of Varroa destructor // Benef. Microbes. V. 11. № 7. P. 641–654.
Sandionigi A., Vicario S., Prosdocimi E.M., Galimberti A., Ferri E. et al., 2015. Towards a better understanding of Apis mellifera and Varroa destructor microbiomes: Introducing 'phyloh' as a novel phylogenetic diversity analysis tool // Mol. Ecol. Resour. V. 15. № 4. P. 697–710.
Sanger F., Nicklen S., Coulson A.R., 1977. DNA sequencing with chain-terminating inhibitors // Proc. Natl. Acad. Sci. USA. V. 74. № 12. P. 5463–5467.
Tan K., Warrit N., Smith D.R., 2007. Mitochondrial DNA diversity of Chinese Apis cerana // Apidologie. V. 38. P. 238–246.
Techer M.A., Rane R.V., Grau M.L., Roberts J.M.K., Sullivan S.T. et al., 2019. Divergent evolutionary trajectories following speciation in two ectoparasitic honey bee mites // Commun. Biol. V. 2. https://doi.org/10.1038/s42003-019-0606-0
Traynor K.S., Mondet F., Miranda J.R., de, Techer M., Kowallik V. et al., 2020. Varroa destructor: A complex parasite, crippling honey bees worldwide // Trends Parasitol. V. 36. № 7. P. 592–606.
Vung N.N., Lee M.-L., Lee M.-Y., Kim H.K., Kang E.J. et al., 2017. Breeding and selection for resistance to Sacbrood Virus for Apis cerana // J. Apic. V. 32. № 4. P. 345–352.
Wang S., Chen G., Lin Z., Wu Y., Hu F. et al., 2019a. Occurrence of multiple honeybee viruses in the ectoparasitic mites Varroa spp. in Apis cerana colonies // J. Invertebr. Pathol. V. 166. https://doi.org/10.1016/j.jip.2019.107225
Wang S., Lin Z., Dietemann V., Neumann P., Wu Y. et al., 2019b. Ectoparasitic mites Varroa underwoodi (Acarina: Varroidae) in eastern honeybees, but not in western honeybees // J. Econom. Ent. V. 112. № 1. P. 25–32.
Warrit N., Hagen T.A.R., Smith D.R., Çakmak I., 2004. A survey of Varroa destructor strains on Apis mellifera in Turkey // J. Apic. Res. V. 43. № 4. P. 190–191.
Woo K.S., 1992. New honeybee mite Varroa underwoodi on Apis cerana in South Korea // Honeybee Sci. V. 13. P. 173–174. (in Japanese).
Woolhouse M.E., Haydon D.T., Antia R., 2005. Emerging pathogens: The epidemiology and evolution of species jumps // Trends Ecol. Evol. V. 20. № 5. P. 238–244.
Zheng H.-Q., Wei W.-T., Hu F.-L., 2011. Beekeeping industry in China // Bee World. V. 88. P. 41–44.
Zheng H., Cao L., Huang S., Neumann P., Hu F., 2018. Current status of the beekeeping industry in China // Asian Beekeeping in the 21st Century. Heidelberg: Springer-Verlag. P. 129–158.
Дополнительные материалы отсутствуют.
Инструменты
Журнал общей биологии


