Поверхность. Рентгеновские, синхротронные и нейтронные исследования, 2021, № 10, стр. 23-27
Влияние нарушения границы между мономерами в димере на структурно-динамические свойства HU-белка из Spiroplasma melliferum
А. С. Комолов a, Ю. К. Агапова a, *, В. И. Тимофеев a, b, Т. В. Ракитина a, c
a Национальный исследовательский центр “Курчатовский институт”
123182 Москва, Россия
b Институт кристаллографии им. А.В. Шубникова
ФНИЦ “Кристаллография и фотоника” РАН
119333 Москва, Россия
c Институт биоорганической химии им. академиков
М.М. Шемякина и Ю.А. Овчинникова РАН
117997 Москва, Россия
* E-mail: agapova.jk@gmail.com
Поступила в редакцию 26.12.2020
После доработки 22.01.2021
Принята к публикации 27.01.2021
Аннотация
Исследовано влияние замен аминокислотных остатков, формирующих границу между мономерами в димере HU-белка из Spiroplasma melliferum (HUSpm), на стабильность и конформационную динамику молекулы. Методом молекулярной динамики изучено поведение одиночных и множественных мутантов HUSpm. Моделирование осуществляли с использованием программного пакета GROMACS, в качестве силового поля использовали OPLS. Для каждого мутанта проведена полноатомная симуляция продолжительностью 50 нс. Полученные данные показали, что нарушения границы между мономерами в области альфа-спирального домена увеличивают подвижность молекулы в области ДНК-связывающего домена.
ВВЕДЕНИЕ
ДНК-связывающие гистоноподобные белки бактерий (HU-белки) относятся к семейству белков, ассоциированных с нуклеоидом (NAP – nucleoid-associated protein); они участвуют в поддержании суперскрученности и компактизации геномной ДНК прокариотической клетки, а также в регуляции основных ДНК-зависимых процессов [1] и поддержании транскриптомного и протеомного профиля бактериальной клетки [2]. Гены HU-белков найдены во всех известных на сегодня бактериальных геномах [3]. В подавляющем большинстве бактерий HU-белки представляют собой гомодимеры размером около 20 кДа, состоящие из относительно стабильного альфа-спирального домена и ДНК-связывающего домена, подвижного в отсутствие ДНК [4, 5]. HU-белки критически важны для жизнедеятельности микроорганизмов класса Mollicutes [6], а также ряда других болезнетворных бактерий [7] и, соответственно, являются перспективными мишенями для разработки антибактериальных препаратов [8–12].
На настоящий момент найдены низкомолекулярные ингибиторы HU-белков, действующие как на ДНК-связывающий домен [8, 9], так и на альфа-спиральный домен [10–12]. Их мишенью является граница между мономерами в димере, нарушение которого, предположительно, влияет на ДНК-связывающие свойства HU-белка и его способность выполнять свою биологическую функцию.
Целью работы было изучение влияния аминокислотных замен, нарушающих контакты между мономерами в димере HU-белка из микоплазмов Spiroplasma melliferum (HUSpm), на конформационную динамику молекулы. Было исследовано поведение одиночных и множественных мутантов HUSpm в процессе полноатомной молекулярно-динамической симуляции продолжительностью 50 нс. Полученные данные показали, что нарушения границы между мономерами в димере приводит к увеличению подвижности молекулы в области ДНК-связывающего домена. Аналогичное изменение конформационной динамики наблюдалось при моделировании связывания бисфенольных производных флуорена с альфа-спиральным доменом HUSpm [10].
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Выбор аминокислотных остатков для мутагенеза
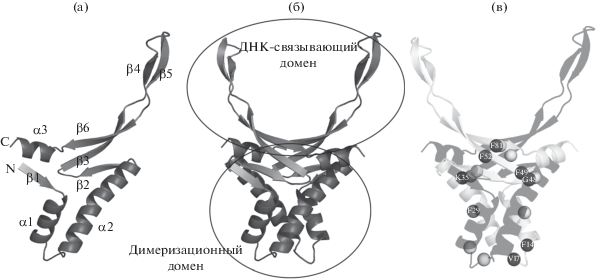
В подавляющем большинстве бактерий HU-белки функционируют в виде гомодимеров, которые состоят из стабильного α-спирального (димеризационного) домена и более подвижного β-ленточного ДНК-связывающего домена (рис. 1а, 1б). В ДНК-связывающем домене можно выделить седлообразную ДНК-связывающую платформу (β-слои 2, 3 и 6, и α-спираль 3) и подвижные в отсутствие ДНК “руки”, состоящие из β-слоев 4 и 5 и примыкающих к ним петель. Все α-спирали и петля между β-слоями 2 и 3 участвуют в формировании межмолекулярных контактов [4, 5]. Анализ границы между мономерами в димере HUSpm, проведенный в [13, 14], выявил в нем два центра межмолекулярных стэкинг-взаимодействий между боковыми радикалами остатков фенилаланина. Один центр формируется между петлей, соединяющей β-слои 2 и 3, и α-спиралями 2 и 3, второй – между С-концевой частью α-спирали 1 и N-концевой частью α-спирали 2 [13] (рис. 1в). Эти центры вносят основной вклад в стабилизацию димеров HU-белков: один известен как сигнал димеризации [15], аминокислотные остатки другого являются структурными детерминантами термостабильности HUSpm [4]. Поэтому для изучения влияния нарушения границы между мономерами в димере на конформационную динамику ДНК-связывающего домена были проведены единичные и множественные замены аминокислотных остатков (в первую очередь остатков фенилаланина), формирующих центры стэкинг-взаимодействий HUSpm: F49A, F52A + F81A и F29A, F29A + F14A + V17T, а также замена K35T, нарушающая межмолекулярную водородную связь K35-G48 и усиливающая чувствительность HUSpm к бисфенольным производным флуорена [10].
Рис. 1.
Пространственная структура мономера и димера HUSpm: а – элементы вторичной структуры (показаны на мономере HUSpm); б – доменная организация белка (показана на димере HUSpm); в – два ароматических центра стэкинг-взаимодействий, расположенные на границе между мономерами в димере HU-белка. Аминокислотные остатки, выбранные для мутагенеза, подписаны на одном мономере.
ИССЛЕДОВАНИЕ МУТАНТНЫХ БЕЛКОВ МЕТОДОМ МОЛЕКУЛЯРНОЙ ДИНАМИКИ
Модели мутантных белков были получены на основе кристаллической структуры HUSpm (PDB ID 5L8Z [16]) c помощью интерактивной графической программы COOT [17]. Для проведения молекулярно-динамических расчетов использовали программный пакет GROMACS [18]. В качестве силового поля было выбрано поле OPLS. Комплексы были помещены в кубическую ячейку, заполненную водой с добавлением ионов в количестве, необходимом для того, чтобы суммарный заряд систем оставался нулевым. В качестве модели молекулы воды была выбрана TIP3P. На первом этапе молекулярно-динамического эксперимента была проведена минимизация энергии комплекса для релаксации любых возможных искажений. Затем на подвижность атомов системы были наложены ограничения, и при помощи моделирования системы в баростате и термостате были установлены температура 300 К и давление 1 атм. После снятия всех ограничений был проведен молекулярно-динамический эксперимент, параметры которого представлены в табл. 1.
Таблица 1.
Протокол молекулярно-динамического эксперимента
| Система в термостате | |
|---|---|
| Термостат | Берендсена |
| Температура, К | 300 |
| Продолжительность NVT динамики, пс | 100 |
| Шаг временнóй сетки, пс | 0.001 |
| Система в баростате | |
| Баростат | Паринелло–Рахмана |
| Устанавливаемое давление, атм. | 1 |
| Температура, К | 300 |
| Продолжительность NPT динамики, пс | 100 |
| Шаг временнóй сетки, пс | 0.001 |
| Продуктивная молекулярная динамика | |
| Шаг временной сетки dt, пс | 0.002 |
| Количество шагов | 25 000 000 |
| Длина траектории, нс | 50 |
| Интегратор | Стохастический динамический алгоритм “Leap-frog” |
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Был проведен сравнительный анализ флуктуаций Сα-атомов основной цепи, наблюдаемых при молекулярно-динамических симуляциях в течение 50 нс HUSpm дикого типа, и мутантных вариантов белка, в которых аминокислотные замены приводили к нарушениям межмолекулярных взаимодействий в области границы между мономерами в димере (рис. 2). Мутациям подвергались аминокислотные остатки, играющие ключевую роль в стабилизации димера HUSpm [4, 13] (рис. 1в). Одна из исследуемых мутаций также усиливала ингибирующий эффект бисфенольных производных флуорена на ДНК-связывающую способность HUSpm [10].
Рис. 2.
Среднеквадратичная флуктуация (СКФ) Сα-атомов в течение 50 нс симуляции методом молекулярной динамики, вычисленная для свободного HUSpm дикого типа (серые столбики), и HUSpm с заменами аминокислотных остатков, формирующих два центра стекинг-взаимодействий на границе между мономерами в димере HUSpm (рис. 1в): а – мутация F29A (сплошная линия), F29A + F14A + V17T (пунктир); б – мутации F49A (штриховая линия), F52A + F81A (сплошная линия), K35T (пунктир). Вторичная структура HUSpm [13] показана сверху.
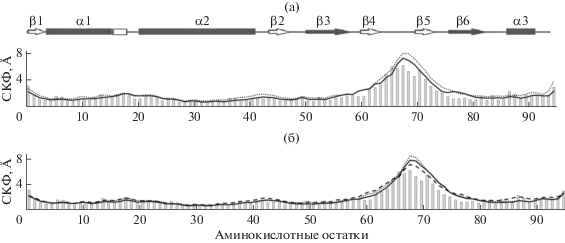
Рис. 2 иллюстрирует конформационную динамику HUSpm дикого типа, характерной особенностью которой является значительная подвижность β-ленточных рук ДНК-связывающего домена в отсутствие ДНК. Именно такое поведение HU-белков в растворе наблюдается при их исследованиях с помощью ЯМР-спектроскопии [5, 13]. Аланиновые замены остатков фенилаланина, образующих ароматические кластеры стэкинг-взаимодействий в α-спиральном домене и в области перехода α-спиралей в β-складчатую укладку, приводили к дополнительному усилению подвижности ДНК-связывающего домена, включая не только β-ленточные руки (β-слои 4 и 5 и петля между), но и относительно стабильную седловидную основу (β-слои 3 и 6), которая обеспечивают адаптацию ДНК-связывающего домена при переходе белка из свободного состояния в конформацию захвата ДНК, что, как полагают, обеспечивает высокую прочность ДНК-белковых комплексов, имеющих константы диссоциации в наномолярных областях [10]. Такой же эффект наблюдался при замене K35T, приводящей к нарушению межмолекулярного полярного контакта между сигналом димеризации (петля между β-слоями 2 и 3) одного мономера и С-концевой областью α-спирали 2 второго мономера. Данные результаты коррелируют с результатами молекулярно-динамических исследований комплексов HUSpm c ингибиторами, мишенями которых является граница между мономерами в димере: в [10, 12] показано, что нарушение межмолекулярных контактов, вызванное связыванием ингибитора, приводит к увеличению подвижности ДНК-связывающего домена. Таким образом, полученные результаты подтверждают постулат о том, что, воздействуя на границу между мономерами в димере, можно контролировать ДНК-связывающие свойства HU-белков и, как следствие, подавлять их биологические функции.
Список литературы
Dillon S.C., Dorman C.J. // Nature Rev. Microbiol. 2010. V. 8. P. 185. https://doi.org./10.1038/nrmicro2261
Камашев Д.Э., Ракитина Т.В., Матюшкина Д.С., Евсютина Д.В., Ванюшкина А.А., Агапова Ю.К., Анисимова В.Е., Дробышев А.Л., Бутенко И.О., Побегуц О.В., Фисунов Г.Ю. // Биоорг. химия. 2019. Т. 45. С. 524. https://doi.org./10.1134/S0132342319050026
Kamashev D.E., Agapova Y.K., Rastorguev S., Talyzina A.A., Boyko K.M., Korzhenevskiy D.A., Vlaskina A., Vasilov R., Timofeev V.I., Rakitina T.V. // PLoS One. 2017. V. 12(11). P. e188037. https://doi.org/10.1371/journal.pone.0188037
Boyko K.M., Gorbacheva M.A., Rakitina T.V., Korzhenevsky D.A., Dorovatovsky P.V., Lipkin A.V., Popov V.O. // Sci. Rep. 2016. V. 3. P. 36 366. https://doi.org./10.1134/S1607672914040012
Altukhov D.A., Talyzina A.A., Agapova Y.K., Vlaskina A.V., Korzhenevskiy D.A., Bocharov E.V., Rakitina T.V., Timofeev V.I., Popov V.O. // J. Biomol. Struct. Dyn. 2016. V. 36. P. 45. https://doi.org/10.1080/07391102.2016.1264893
Glass J.I., Assad-Garcia N., Alperovich N., Yooseph N., Lewis S., Maruf M.R., Hutchison M., Smith C.A., Venter H.O., Craig J. // Proc. Natl. Acad. Sci. USA. 2006. V. 103. P. 425. https://doi.org/10.1073/pnas.0510013103
Stojkova P., Spidlova P., Stulik J. // Front. Cell. Infect. Microbiol. 2019. V. 9. P. 159. https://doi.org./10.3389/fcimb.2019.00159
Bhowmick T., Ghosh S., Dixit K. // Nature Commun. 2014. V. 5. P. 4124. https://doi.org/10.1038/ncomms5124
Liu R., Sun Y., Chai Y., Li S., Li S., Wang L., Su J., Yu S., Yan J., Gao F., Zhang G., Qiu H., Gao G., Qi J., Wang H. // PNAS. 2020. V. 117. P. 11 000. https://doi.org/10.1073/pnas.1922523117
Agapova Y.K., Altukhov D.A., Timofeev V.I., Stroylov V.S., Mityanov V.S., Korzhenevskiy D.A., Vlaskina A.V., Smirnov E.V., Bocharov E.V., Rakitina T.V. // Sci. Rep. 2020. V. 10. P. 15128. https://doi.org./10.1038/s41598-020-72113-4
Raj R., Agarwal N., Raghavan S., Chakraborti T., Poluri K.M., Kumar D. // Int. J. Biol. Macromol. 2020. V. 161. P. 231. https://doi.org/10.1016/j.ijbiomac.2020.06.039
Агапова Ю.К., Алтухов Д.А., Камашев Д.Э., Тимофеев В.И., Смирнова Е.В., Ракитина Т.В. // Кристаллография. 2020. Т. 65 С. 900. https://doi.org./10.31857/S0023476120060041
Timofeev V.I., Altukhov D.A., Talyzina A.A., Agapova Y.K., Vlaskina A.V., Korzhenevskiy D.A., Kleymenov S.Y., Bocharov E.V., Rakitina T.V. // J. Biomol. Struct. Dyn. 2018. V. 36. P. 4392. https://doi.org./10.1080/07391102.2017.1417162
Агапова Ю.К., Талызина А.А., Алтухов Д.А., Лаврентьев А.Л., Тимофеев В.И., Ракитина Т.В. // Кристаллография. 2019. Т. 64. С. 602. https://doi.org./10.1134/S1063774519030027
Christodoulou E., Rypniewski W.R., Vorgias C.R. // Extremophiles. 2003. V. 7 P. 111. https://doi.org./10.1007/s00792-002-0302-7
Boyko K.G., Rakitina T.V., Korzhenevskiy D.A. Korzhenevskiy D., Vanyushkina A., Kamashev D., Lipkin A., Popov V. // Acta Crystallogr. F. 2015. V. 71. P. 24. https://doi.org./10.1107/S2053230X14025333
Emsley P., Lohkamp B., Scott W.G., Cowtan K. // Acta Crystallogr. D. 2010. V. 66. P. 486. https://doi.org./10.1107/S0907444910007493
Abraham R.G., Murtola T., Schulz R., Páll S., Smith G.S., Hess B., Lindahl E. // SoftwareX. 2015. V. 1–2. P. 19. https://doi.org/10.1016/j.softx.2015.06.001
Дополнительные материалы отсутствуют.
Инструменты
Поверхность. Рентгеновские, синхротронные и нейтронные исследования


