Поверхность. Рентгеновские, синхротронные и нейтронные исследования, 2022, № 10, стр. 42-47
Структурные особенности механически деформированных ядер HeLa, наблюдаемые методом атомно-силовой микроскопии
В. Ю. Байрамуков a, *, М. В. Филатов a, Р. А. Ковалев a, Р. А. Пантина a, С. В. Григорьев a, b, Е. Ю. Варфоломеева a
a Петербургский институт ядерной физики им. Б.П. Константинова,
Национальный исследовательский центр “Курчатовский институт”
188300 Гатчина, Россия
b Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: bayramukov_vy@pnpi.nrcki.ru
Поступила в редакцию 30.12.2021
После доработки 25.03.2022
Принята к публикации 28.03.2022
- EDN: ZBQLGS
- DOI: 10.31857/S1028096022100041
Аннотация
Методом атомно-силовой микроскопии исследован рельеф поверхности ядер HeLa после их механической деформации. Ядра выделяли из клеток линии HeLa, деформировали под действием центробежного ускорения и фиксировали глутаральдегидом. Показано, что наблюдаемый рельеф поверхности обусловлен главным образом высокой устойчивостью хроматина к деформации. Природа этой устойчивости коррелирует с суперспирализацией ДНК. Действие ингибиторов топоизомераз I и II приводило к снятию суперскрученности и значительному уплощению ядер. Действие ДНК-интеркалятора, наоборот, приводило к увеличению жесткости ДНК и, как следствие, устойчивости хроматина к механическому воздействию. Таким образом, наблюдаемые изменения морфологии отражают функциональные особенности клеточного ядра.
ВВЕДЕНИЕ
В клеточном ядре ДНК, РНК и белки расположены в макромолекулярных доменах, участвующих в различных этапах экспрессии генов, таких как транскрипция, репликация, репарация [1, 2]. Домены в ядрах клеток млекопитающих включают хроматин, ядерный матрикс, ядерную оболочку и ядерную ламину, поровые комплексы и ядрышки, которые можно наблюдать методами оптической и электронной микроскопии [3–6]. Использование дополнительных методов прямого наблюдения могло бы расширить возможности понимания принципов организации клеточных ядер.
Одним из технических достижений последних десятилетий является техника сканирующей зондовой микроскопии. В частности, атомно-силовая микроскопия (АСМ) используется для анализа морфологии поверхности с разрешением от десятков ангстрем вплоть до атомарного уровня [7]. Это позволяет визуализировать различные биологические объекты, такие как нуклеосомы, ДНК, которые можно выделить из биоматериала и исследовать, поместив на атомарно плоскую подложку [8–11].
Для выделенных клеточных ядер, фиксированных в суспензии и помещенных на подложку, зачастую характерна относительно гладкая форма без отличительных особенностей морфологии [12]. Очевидно, что в таком виде невозможно использовать АСМ для исследования структуры ядра. Показано, что фиксация ядер клеток Lacandonia schismatica, Ginkgo bilobaс и почки мыши с последующим получением тонких срезов и анализа материала методом АСМ выявляет различия в пространственном распределении хроматина [13]. В другом исследовании методом АСМ определили морфологию и уровни компактизации хроматина на масштабах от 15 до 110 нм в ядрах куриных эритроцитов, лизированных в гипотоническом растворе [14]. В [15] клетки HeLa подвергали действию детергентов и раствора высокой ионной силы, что приводило к удалению клеточной мембраны и возможности использования АСМ для анализа структуры клеточного ядра. Анализ морфологии позволил выявить базовые составляющие хроматина в ядре эукариотической клетки [16]. В [17] авторы исследовали изменение модуля Юнга ядер в зависимости от приложенной частоты колебания кантилевера методом микрореологии. Было показано, что для различных клеточных линий эластичность ядер увеличивается от центра к периферии, что авторы связывают с уменьшением степени сшивания в хроматине.
В настоящей работе естественная эластичность ядер позволила реализовать новый подход к исследованию организации ядерной структуры посредством АСМ. На примере ядер HeLa показано, что механическое воздействие (центробежное ускорение) преобразует особенности внутриядерной организации в изменение рельефа поверхности ядра. Экспериментально подтверждено, что наблюдаемая морфология коррелирует с функциональными особенностями ядра, обусловлена суперскручиванием ДНК и изменяется в ответ на действие веществ, подавляющих суперскручивание, либо, наоборот, увеличивающих жесткость ДНК и, как следствие, устойчивости хроматина.
МАТЕРИАЛЫ И МЕТОДЫ
Клеточные ядра выделены из культуры HeLa по стандартной методике, а именно путем воздействия неионного детергента TritonX-100 (0.15%), отмыты от остатков клеточной мембраны в процессе центрифугирования в 10 мл полифосфатного буфера.
В качестве химических соединений-ингибиторов топоизомераз были выбраны камптотецин (ингибитор топоизомеразы I) и этопозид (ингибитор топоизомеразы II), которые инкубировали с клеточной культурой в течение суток в концентрациях 40 и 80 мкг/мл соответственно. ДНК-интеркалятор, бромистый этидий (C21H20BrN3), добавляли сразу после выделения ядер в концентрации 10 мкг/мл и инкубировали в течение 5 мин.
Механическая деформация ядер включала следующие действия. В чашку Петри помещали подложку из предметного стекла, покрытого 0.001% полилизином. На подложку наносили суспензию (1 мкл) ядер и незамедлительно чашку Петри помещали в бакет-ротор, установленный в центрифуге UNION 5KR. Ядра откручивали при минимально возможном центробежном ускорении (60g) в течение 5 мин, после чего незамедлительно фиксировали с помощью 0.5% глутаральдегида. После 10 мин инкубации стекло промывали в потоке дистиллированной воды и оставляли для естественного высушивания.
АСМ-исследования проводили с помощью микроскопа Solver BIO (NT-MDT, Россия), оснащенного инвертированным оптическим микроскопом Olimpus. Использовали зонды NSG03 (NT-MDT, Россия) с радиусом закругления 10 нм в полуконтактном режиме сканирования с частотой 0.9 Гц и амплитудой 25 нм. Размер записываемого изображения составлял 29 × 29 мкм, число точек 512 × 512.
Для получения изображений конфокальной микроскопии фиксированные клеточные ядра окрашивали флуоресцентным красителем Hoehst 33 342 (10 мкг/мл) в фосфатном буфере. Использовали микроскоп Leica TCS SP5X SP5 (Leica, Германия), длина волны возбуждения составляла 405 нм. Обработку изображений проводили в программе Gwyddion 2.56.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Подходы оптической микроскопии, в частности конфокальной микроскопии, позволяют получить изображения ядра как в проходящем свете, так и при окрашивании ДНК. Разрешение таких изображений ограничено длиной волны света. Соответственно, оптически плотные ядрышки размером несколько микрометров хорошо различимы на изображениях ядра HeLa (на рис. 1а и далее ядрышки показаны стрелками), в то время как детальная структура хроматина неразличима (рис. 1б). Морфология при механической деформации, наблюдаемая на двумерном АСМ-изображении ядра (рис. 1в), схожа с морфологией, визуализируемой в случае оптической микроскопии, однако обусловлена совершенно иным физическим явлением – устойчивостью внутриядерных компонент к деформации. Так, значительно увеличивается латеральный размер ядра, и наиболее ярким внутриядерным объектом являются ядрышки размером ~1–5 мкм, возвышающиеся над базовой структурой деформированного ядра (~200 нм), что отчетливо видно на трехмерном изображении (рис. 1г) и соответствующем профиле рельефа поверхности.
Рис. 1.
Двумерные (а–в) и трехмерное (г) изображения ядер HeLa, полученные методами конфокальной микроскопии в проходящем свете и при окрашивании ядерной ДНК (а, б) и АСМ (в, г) при механической деформации ядер. Профиль сечения деформированного ядра приведен внизу рисунка.
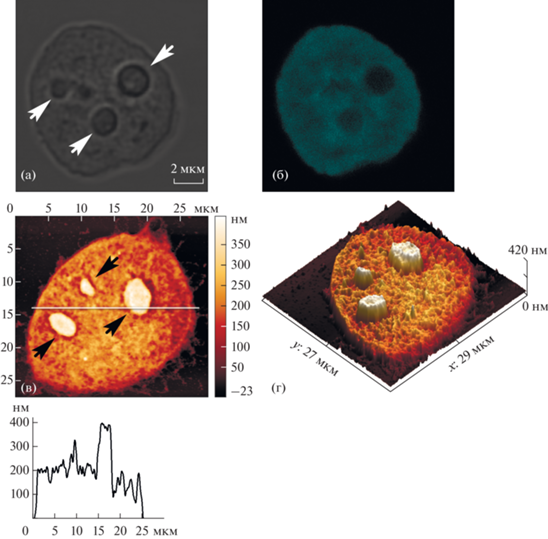
По-видимому, в случае ядер HeLa с высокой транскрипционной активностью [18], подвергнутых механической деформации, наблюдается тенденция образования плоской (~200 нм) рельефной базовой структуры механически устойчивого хроматина с сохранением ядрышковой структуры [19]. Напротив, в случае ядер куриных эритроцитов с минимальным уровнем транскрипции не наблюдались ярко выраженные особенности морфологии ядра при механической деформации [20].
В настоящей работе показано, что наблюдаемая морфология является следствием функциональных особенностей клеточного ядра. В [21] предложена модель транскрипции, обусловленной суперскручиванием ДНК. Так, при активной транскрипции экспрессия генов сильно коррелирует и жестко регулируется суперспирализацией. Модель предсказывает, что ферменты, изменяющие топологию, нарушают суперскручивание и, как следствие, подавляют транскрипцию. Данное утверждение проверено экспериментально на ядрах HeLa. В клеточном ядре присутствуют ферменты-топоизомеразы, поддерживающие суперскручивание ДНК, обеспечивая процессы репликации и транскрипции [22]. Очевидно, подавление (ингибирование) ферментов отразится на морфологии, наблюдаемой методом АСМ.
Так, клетки HeLa инкубировали с ингибиторами топоизомераз I и II (камптотецин и этопозид). АСМ-изображения механически деформированных ядер представлены на рис. 2. В обоих случаях воздействие ингибиторов на ядра HeLa ведет к значительному уменьшению высоты (от ~200 до 50 нм), что следует из приведенного профиля поверхности. Можно заключить, что воздействие ингибиторов привело к кардинальному уменьшению степени суперскучивания вследствие подавления транскрипции, что отразилось на рельефе поверхности деформированных ядер.
Рис. 2.
Двумерные (а, в) и трехмерные (б, г) АСМ-изображения механически деформированных ядер HeLa при действии ингибиторов топоизомеразы I (а, б) и топоизомеразы II (в, г). Профили сечений деформированных ядер приведены внизу соответствующих рисунков.
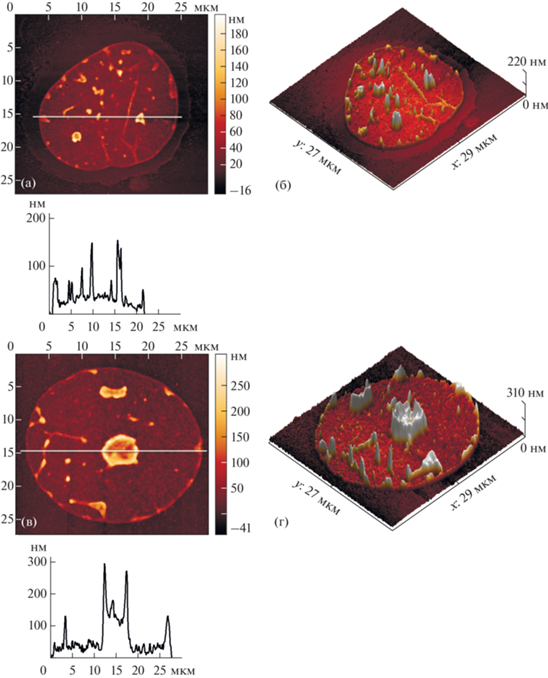
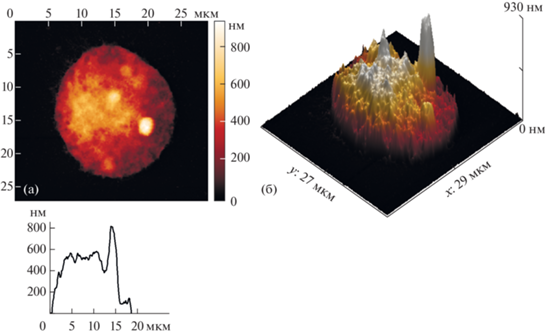
Обратная картина должна наблюдаться, если внести в укладку ДНК такие изменения, при которых торсионные напряжения, присущие суперскрученной ДНК, не будут скомпенсированы. Это приведет к увеличению жесткости ДНК, и, как следствие, к сопротивлению деформации. Бромистый этидий – широко применяемый ДНК-интеркалятор, нарушающий структуру хроматина [23]. Его добавление к ядрам с последующей механической деформацией действительно приводит к изменению наблюдаемого рельефа (рис. 3).
Рис. 3.
Двумерное (а) и трехмерное (б) АСМ-изображения ядер HeLa после воздействия бромистого этидия. Профиль сечения деформированного ядра приведен внизу рисунка.
Обобщенная картина вариации высоты профиля ядер HeLa при деформации в зависимости от типа воздействия на упаковку хроматина представлена на рис. 4. Так, в случае контрольного образца (кривая 1) базисная высота деформированного ядра составила ~200 нм, наблюдалось отличительное ядрышковое образование. Добавление ингибиторов топоизомераз к клеткам в обоих случаях приводило к снижению высоты до значений ~40 нм (кривые 2 и 3), на фоне которых выделялись одно или несколько ядрышковых образований. Использование ДНК-интеркалятора, напротив, значительно увеличивало высоту профиля при деформирмации ядра (~600 нм) из-за возрастания жесткости хроматина (кривая 4).
ЗАКЛЮЧЕНИЕ
В ядре клетки содержится чрезвычайно большая концентрация высокополимерных молекул: ДНК, РНК, белка. Концентрация каждого из этих компонентов так высока, что трудно, если вообще возможно, искусственно создать раствор соответствующей концентрации. По существующим оценкам, общая концентрация макромолекул в ядре может превышать 100 мг/мл [24, 25]. Любые небиологические образования, содержащие столь высокие концентрации макромолекул, обладали бы чрезвычайной вязкостью и представляли собой упругий гель, устойчивый к деформации. Нужно полагать, что организация материала ядер в значительной мере уникальна, что может быть связано со значительным снижением вязкости, так что большая часть макромолекул может диффундировать сквозь скопления других молекул без заметного трения. Природа этого необычного поведения нуклеоплазмы не может быть предметом рассмотрения настоящей работы. Однако следствием его является возможность реализовать предлагаемый метод визуализации внутриядерной организации с помощью АСМ. Суть метода заключается в том, чтобы преобразовать неоднородности внутриядерной организации в поверхностные явления посредствам их механической деформации. Результатом являются наблюдаемые изменения морфологии, которые отражают функциональные особенности клеточного ядра.
Список литературы
Spector D.L. // Annu. Rev. Cell Biol. 1993. V. 9. P. 265.https://doi.org/10.1146/annurev.cb.09.110193.001405
Lamond A.I., Earnshaw W.C. // Science. 1998. V. 280. P. 547.https://doi.org/10.1126/science.280.5363.547
Cohen M., Tzur Y.B., Neufeld E., Feinstein N., Delannoy M.R., Wilson K.L., Gruenbaum Y. // J. Struct. Biol. 2002. V. 140. P. 232. https://doi.org/10.1016/S1047-8477(02)00516-6
Wachtler F., Stahl A. // Micron. 1993. V. 24. P.473.https://doi.org/10.1016/0968-4328(93)90026-W
Tchélidzé P., Chatron-Colliet A., Thiry M., Lalun N., Bobichon H., Ploton D. // Crit. Rev. Oncol. Hematol. 2008 V. 69. P. 127. https://doi.org/10.1016/j.critrevonc.2008.07.022
Gulka M., Salehi H., Varga B. et al. // Sci. Rep. 2020. V. 10. P. 9791. https://doi.org/10.1038/s41598-020-66593-7
Binnig G., Quate C.F., Gerber Ch. // Phys. Rev. Lett. 1986. V. 56. P. 930. https://doi.org/10.1103/PhysRevLett.56.930
Lyubchenko Y.L., Oden P.I., Lampner D., Lindsay S.M., Dunker K.A. // Nucl. Acids Res. 1993. V. 21. P. 1117. https://doi.org/10.1093/nar/21.5.1117
Hansma H.G., Vesenka J., Siegerist C., Kelderman G., Morrett H., Sinsheimer R.L., Elings V., Bustamante C., Hansma P.K. // Science. 1992. V. 256. P. 1180. https://doi.org/10.1126/science.256.5060.1180
Lyubchenko Y.L., Gall A.A, Shlyakhtenko L.S., Harrington R.E., Jacobs B.L., Oden P.I., Lindsay S.M. // J. Biomol. Struct. Dyn. 1992. V. 10. P. 598. https://doi.org/10.1080/07391102.1992.10508670
Shlyakhtenko L.S., Lushnikov A.Y., Lyubchenko Y.L. // Biochem. 2009. V. 48. P. 7842. https://doi.org/10.1021/bi900977t
Isaev-Ivanov V.V., Lebedev D.V., Lauter H. et al. // Phys. Solid State. 2010. V. 52. P. 1063. https://doi.org/10.1134/S1063783410050379
Jiménez–García L.F., Fragoso-Soriano R. // J. Struct. Biol. 2000. V. 129. P. 218. https://doi.org/10.1006/jsbi.2000.4233
Qian R., Liu Z., Zhou M. et al. // Cell Res. 1997. V. 7. P. 143. https://doi.org/10.1038/cr.1997.15
Yoshimura S.H., Kim J., Takeyasu K. // J. Electron Microsc. (Tokyo) 2003. V. 52. P. 415. https://doi.org/10.1093/jmicro/52.4.415
Hirano Y., Takahashi H., Kumeta M. et al. // Pflugers Arch. – Eur. J. Physiol. 2008. V. 456. P. 139. https://doi.org/10.1007/s00424-007-0431-z
Lherbette M., dos Santos Á. // Sci. Rep. 2017. V. 7. P. 8116. https://doi.org/10.1038/s41598-017-08517-6
Rahbari R., Sheahan T., Modes V., Collier P., Macfarlane C., Badge R.M. // Biotech. 2009. V. 46. P. 277. https://doi.org/10.2144/000113089
Grigoriev S.V., Iashina E.G., Wu B., Pipich V., Lang Ch., Radulescu A., Bairamukov V.Yu., Filatov M.V., Pantina R.A., Varfolomeeva E.Yu. // Phys. Rev. E. 2021. V. 104. P. 044404.https://doi.org/10.1103/PhysRevE.104.044404
Grigoriev S.V., Iashina E.G., Bairamukov V.Yu., Pipich V., Radulescu A., Filatov M.V., Pantina R.A., Varfolomeeva E.Yu. // Phys. Rev. E. 2020. V. 102. P. 032415.https://doi.org/10.1103/PhysRevE.102.032415
Brackley C.A., Johnson J., Bentivoglio A., Corless S., Gilbert N., Gonnella G., Marenduzzo D. // Phys. Rev. Lett. 2016. V. 117. P. 018101 https://doi.org/10.1103/PhysRevLett.117.018101
Champoux J.J. // Annu. Rev. Biochem. 2001. V. 70. P. 369. https://doi.org/10.1146/annurev.biochem.70.1.369
Garbett N.C., Hammond N.B., Graves D.E. // Biophys. J. 2004. V. 87. P. 3974. https://doi.org/10.1529/biophysj.104.047415
Hancock R. // Biol. Cell. 2004. V. 96. P. 595. https://doi.org/10.1016/j.biolcel.2004.05.003
Hancock R. // J. Struct. Biol. 2004. V. 146. P. 281.https://doi.org/10.1016/j.jsb.2003.12.008
Дополнительные материалы отсутствуют.
Инструменты
Поверхность. Рентгеновские, синхротронные и нейтронные исследования



