Успехи современной биологии, 2019, T. 139, № 6, стр. 583-593
Гипотеза матричного синтеза апериодических полисахаридов
С. В. Чепур 1, *, Н. Н. Плужников 1, С. А. Сайганов 2, Л. С. Бакулина 3, О. В. Чубарь 1, М. А. Юдин 1, А. С. Никифоров 1
1 Государственный научно-исследовательский испытательный институт военной медицины Минобороны России
Санкт-Петербург, Россия
2 Северо-западный государственный медицинский университет им. И.И. Мечникова Минздрава России
Санкт-Петербург, Россия
3 Воронежский государственный медицинский университет им. Н.Н. Бурденко Минздрава России
Воронеж, Россия
* E-mail: gniiivm_2@mail.ru
Поступила в редакцию 30.04.2019
После доработки 12.06.2019
Принята к публикации 15.07.2019
Аннотация
Сформирована научная гипотеза, согласно которой роль матрицы выполняют белки, фиксирующие на себе гликозилтрансферазы и гликозидазы различного строения, обеспечивая синтез апериодических полисахаридов. Таким образом, за счет аминокислотного кода детерминируется синтез разноообразных полисахаридов, обеспечивающих реализацию индивидуально специфичных процессов при организации метаболических каскадов и регуляторных элементов макроорганизма, определяющего его связь с микробиомом и характеристики тканеспецифичности. Такие белки, таргетированные во внутриклеточные мембранные компартменты клеток и имеющие локусы ассоциации с доменами гликозилтрансфераз, вполне могут выполнять роль своеобразной матрицы в процессе биосинтеза полисахаридов, выстраивая ферменты синтеза гликанов в виде функциональных конвейерных линий.
ВВЕДЕНИЕ
Исследования участия гликанов в рецепции различных сигнальных молекул, детерминированность и постоянство групп крови, особенности формирования индивидуального воспроизводимого на протяжении всей жизни человека микробиотического пейзажа на поверхностях слизистых оболочек, определяемого факторами колонизации и взаимодействия, позволяют предполагать наличие устойчивых механизмов регуляции синтеза полисахаридов. Полагая генетическое кодирование ферментов синтеза и модификации структуры гликанов, вместе с тем, остаются не ясными вопросы структурной организации их сборки, определяющие порядок часто индивидуально специфичной полисахаридной последовательности. Эти обстоятельства позволили предположить наличие матриц для синтеза гликанов, определяющих структурную организацию ферментативных ансамблей, а, следовательно, и вариант последовательности присоединяемых олигосахаридов, рестрикции или разветвления цепи.
ЭНТРОПИЯ И ИНФОРМАЦИЯ
По причине сложной и многоуровневой организации биологических систем в процессе их жизнедеятельности неизбежно накопление “поломок” механизмов поддержания гомеостаза, что, в конечном итоге, приводит к гибели организма. В процессе биологической эволюции были выработаны механизмы, ограничивающие продолжительность жизни многоклеточных организмов (Skulachev, 2002; Gavrilov, Gavrilova, 2002; Prinzinger, 2005; Longo et al., 2005; Ljubuncic, Reznick, 2009; Martins, 2011; DiLoretto, Murphy, 2015; Nelson, Masel, 2017). Термодинамический и эволюционный запреты на бессмертие отдельно взятой биологической системы преодолеваются за счет размножения (копирования самих себя), когда вместо заведомо безнадежного сохранения сложных материальных структур организма следующему поколению передается исчерпывающая информация о нем. Поэтому биологическая эволюция с позиций информатики, в первую очередь, – это эволюция механизмов сохранения, воспроизведения и передачи генетической информации (Щербаков, 2005а,б).
В 1948 г. Шеннон использовал термин “информация” применительно к передаче кодов как к коммуникации, в процессе которой устраняется неопределенность (Shannon, 1948a,b). Одновременно с ним Н. Винер обосновал видение информации как фундаментальной характеристики явлений живой и неживой природы (Винер, 1983; Wiener, 1948). А почти через два десятилетия А.Н. Колмогоров предложил алгоритмическую теорию информации, в которой понятие энтропия трактуется как сложность объекта, равная сложности алгоритма, описывающего этот объект (Колмогоров, 1965). В свете постулатов современной информатики и статистической физики движение к большей структурной сложности – очевидная общая тенденция развития материального мира и эволюции биосферы. В свою очередь, возрастание структурной сложности биомолекул эквивалентно увеличению энтропии и пропорциональному возрастанию их информационной емкости (Волькенштейн, 1978; Силин, 1994; Бакулина и др. 2012).
БИОЛОГИЧЕСКИЕ КОДЫ
Известны три типа биологических апериодических полимеров: нуклеиновые кислоты, полипептиды и полисахариды, представляющие собой базисные элементы биологических организмов и составляющие в совокупности до 80–90% сухой массы клеток (Cooper, Hausman, 2016). Каждый из этих биополимеров несет закодированную биологическую информацию.
Генетический код. Каждая молекула ДНК состоит из нуклеотидов, представленных четырьмя фосфорными эфирами гликозилированных пуринов и пиримидинов: аденином, гуанином, тимином и цитозином, объединенных в триплеты (кодоны) – единицы генетического кода. Четыре азотистых основания могут создать всего 64 кодона ($A_{4}^{{'3}}~$ = 43 = 64), формирующих гены – структурно-функциональные единицы наследственной информации биологических систем, определяющие аминокислотную последовательность полипептидных цепей, либо нуклеотидную последовательность регуляторных РНК. Около 15% эволюционно высококонсервативного генома человека составляют кодоны с двойной функцией (duons), определяющие одновременно структуру матричных РНК и соответствующих им аминокислотных последовательностей полипептидов, а также сайтов связывания факторов транскрипции. Поэтому нарушения первичной структуры ДНК (мутации) могут проявляться не только в виде изменений последовательности матричных РНК и соответствующих им последовательностям аминокислот в пептидах, но и нарушать программы контроля экспрессии генов. Двойное кодирование информации, вероятно, – основная особенность генома эукариот, обеспечивающая предупреждение накопления “поломок” механизмов онтогенеза, уровень базальной экспрессии ответов на внутри и внеклеточные сигналы, а также контроль клеточного цикла. Относительно информационной емкости удвоенного генетического кода можно заметить, что теоретически полимер из шести нуклеотидов может быть представлен уже в 4096 вариантах (Griffiths et al., 2002, 2012; Stergachis et al., 2013).
Полипептидный код. Последовательность аминокислот полипептидной цепи, детерминирующая первичную структуру протеинов, предопределяет также вторичную и третичную структуру белков и их способность формировать функционально значимые мультимерные комплексы (Siltberg-Liberles et al., 2011; Hatton, Warr, 2015; Chi, Liberles, 2016; Gilson et al., 2017). Емкость полипептидного кода существенно превосходит емкость генетического кода, так как 20 протеиногенных аминокислот потенциально могут создать десятки миллионов шестимерных полипептидов ($A_{{20}}^{{'6}}~$ = 206 = = 64 × 106) (Landhuis, 2017).
Полное секвенирование ДНК выявило, что геном человека содержит около 20–25 тыс. белок-кодирующих генов (International …, 2004), что оказалось в 4–5 раз меньше их прогнозируемого количества. В связи с этим было сформулировано предположение о том, что относительно небольшое количество белок-кодирующих генов может быть компенсировано комбинаторным разнообразием протеинов (множественностью протеоформ), генерируемых посредством посттранскрипционного редактирования структуры пре-матричных рибонуклеиновых кислот (альтернативный сплайсинг пре‑мРНК) и посттрансляционного модифицирования структуры самих полипептидов (Venter et al., 2001; Knorre et al., 2009; Smith, Kelleher, 2013). Механизмы альтернативного сплайсинга значительно увеличивают разнообразие белков, в результате чего количество экспрессируемых в организме человека протеоформ в десятки раз превышает число белок-кодирующих генов (Pan et al., 2008; Nilsen, Graveley, 2010; Hu et al., 2015). В еще большей степени структурная палитра белков расширяется вследствие их посттрансляционной модификации, когда среди сотен известных типов модификации мембранных и секретируемых белков (Minguez et al., 2012; Beltrao et al., 2013) до 50% химических превращений осуществляется через процессы гликозилирования, которые остаются в то же время наиболее химически и биологически сложными (Aguilar et al., 2017; Vliegenthart, 2017; Audagnotto, Dal Peraro, 2017).
Углеводный код. Плазматическая мембрана клеток представляет собой экран для экспонирования и восприятия биологических сигналов, необходимых для адаптационно-регуляторного реагирования и межклеточной коммуникации (Cohen, Varki, 2010; Varki, 2011; Wasim et al., 2019). Реализация этих биологических функций обеспечивается высокой плотностью и разнообразным представительством экспонируемых сигнальных биомолекул. В роли таких сигнальных молекул часто выступают гликированные протеины, содержащие в своем составе моно- и олигосахариды, а также гликаны. Именно непревзойденное многообразие полимеров моносахаридов способно формировать высокую стереоспецифичность межклеточных коопераций и регуляцию метаболических процессов. Гликаны, в сравнении с нуклеиновыми кислотами и полипептидами, – безусловные лидеры критериального соответствия качествам эффективного носителя сигнальной информации (Gabius et al., 2011; Delbianco et al., 2016; Gabius, Roth, 2017; Latxague et al., 2018; Gabius, 2018). Теоретически шестизвенный, линейный и разветвленный олигосахарид может быть представлен более чем триллионом (1.05 × 1012) форм (Laine, 1994). Вероятно, именно поэтому на плазматической мембране каждой клетки млекопитающих экспонируется не менее 1 × 106 N- или O-связанных гликанов (Wang et al., 2013).
Внушительная информационная емкость гликанов определена их структурными особенностями (Laine, 1997; Rüdiger, Gabius, 2009; Gabius, Kayser, 2014):
– последовательностью остатков моносахаридов в линейных или разветвленных частях полимера;
– изомерной формой остатков моносахаридов (D- и L-формы);
– количеством атомов углерода в циклах моносахаридов (фуранозы, пиранозы);
– аномерной конфигурацией остатков моносахаридов (α- и β-формы);
– положением гликозидной связи (1 → 2, 1 → 3 и т.д.);
– позицией локуса разветвления;
– наличием редуцирующих или нередуцирующих концевых остатков моносахаридов.
По-видимому, информационная емкость гликанов предопределяет то, что:
– в составе апериодических олигосахаридов человека из множества моносахаридов выявляют, главным образом, 8–10 представителей: D-N-ацетилглюкозамин (31.8%), D-галактозу (24.8%), D-маннозу (18.9%), D‑N‑ацетилнейраминовую кислоту (18.9%), L-фукозу (7.2%), D-N-ацетилгалактозамин (4.8%), D-глюкозу (2.5%), D-глюкуроновую кислоту (0.3%), L-идуроновую кислоту (0.1%), D-ксилозу (0.1%) (Schnaar, 2016);
– в среднем олигосахарид состоит примерно из восьми моносахаридных блоков (Werz et al., 2007; Seeberger, 2011);
– гликан-распознающий домен лектинов связывается только с 2–7 моносахаридными звеньями полисахаридной цепи (Taylor, Drickamer, 2014).
БИОЛОГИЧЕСКИЕ ФУНКЦИИ ГЛИКАНОВ
Гликаны относят к числу наиболее сложных биомолекул живой природы (Varki et al., 2015). Считается, что углеводы, которым принадлежит незаменимая роль во всех типах биологических организмов, обеспечили молекулярный базис биологической эволюции (Stern, Jedrzejas, 2008; Lauc et al., 2014). У высших эукариот структура почти всех интегральных и секретируемых белков претерпевает посттрансляционное модифицирование посредством ковалентного присоединения олигосахаридных цепей (Zielinska et al., 2010), вследствие чего образуется новый молекулярный объект с определенной (зависимой от структуры гликана) биологической функцией (Shinkawa et al., 2003; Kaneko et al., 2006; Anthony et al., 2011; Ackerman et al., 2013; Mahan, 2016; de Haan et al., 2018). То есть, в отличие от других посттрансляционных модификаций, обычно функционирующих в качестве переключателей молекулярных эффектов (“все или ничего”), гликозилирование призвано обеспечивать тонкую настройку (тюнинг) функциональной активности генных продуктов (Varki, 2017) и затрагивает практически все внутриклеточные и межклеточные процессы жизнедеятельности эукариот (National Research …, 2012). Гликаны также универсальны по своей биологической природе, как нуклеиновые кислоты, белки и липиды (Marth, 2008) и также необходимы для существования любых известных биологических систем (Varki, 2011). Спектр биологических функций гликоконъюгатов включает множество структурно-функциональных, регуляторных и сигнально-модуляторных ролей (Varki, 2017). Подходы к оценке биологических функций углеводов более всего сравнимы с пониманием биологической значимости белков, в силу обширности их молекулярных, клеточных и биофизических ролей (Varki et al., 2009). Биологические эффекты конъюгатов олигосахаридов в значительной степени опосредованы взаимодействием с гликан-связывающими белками (лектинами), а специфичность этих взаимодействий предопределяется структурными особенностями апериодической полимерной цепи гликанов (Kletter et al., 2013; Kamili et al., 2016; Sood et al., 2018).
СОВРЕМЕННЫЕ ПРЕДСТАВЛЕНИЯ О БИОСИНТЕЗЕ АПЕРИОДИЧЕСКИХ ПОЛИСАХАРИДОВ
Уникальная химическая структура, определяющая информационную емкость апериодических олигосахаридов, ассоциирована со сложностью биохимической организации их синтеза. Гликаны в клетках эукариот синтезируются при участии изящно-специфичных гликозилтрансфераз, эпимераз, гликозидаз и гликан-модифицирующих трансфераз (Наумов, 2011; Allard et al., 2001; Lairson et al., 2008; Muthana et al., 2012; van Overtveldt et al., 2015; Moremen et al., 2018). Гликозилтрансферазы – основные энзимы, катализирующие синтез (образование гликозидных связей) разнообразных углеводсодержащих структур (McArthur, Chen, 2016) посредством переноса активированных производных моносахаридов (главным образом, нуклеозиддифосфат-сахаров) на нуклеофильные гликозилакцепторы (Lairson et al., 2008; Williams, Thorson, 2009). Предполагают, что 1–2% белок-кодирующих генов архей, про- и эукариот приходится на долю генов, кодирующих структуру гликозилтрансфераз (Mackenzie et al., 1997; Coutinho et al., 2003; Lairson et al., 2008). В геноме человека идентифицировано почти две с половиной сотни генов различных гликозилтрансфераз (Joud et al., 2018; Narimatsu et al., 2018). А в общей сложности в процессы гликозилирования может быть вовлечено более 10% генома (Nairn et al., 2008; Yamamoto-Hino et al., 2010; Lauc et al., 2013).
Гликозилтрансферазы представляют собой трансмембранные белки II типа с эволюционно- консервативной четырехдоменной структурой, включающей короткий N‑концевой цитозольный, трансмембранный, высоковариабельный стволовой и люминальный глобулярный С-концевой каталитический домены (Colley, 1997; Lairson et al., 2008).
Несмотря на то, что трансмембранный домен гликозилтрансфераз примерно на пять аминокислотных остатков короче, чем у интегральных белков плазматических мембран, он вносит определяющий вклад в их интеграцию в мембранах аппарата Гольджи и эндоплазматического ретикулума (Masibay et al., 1993; Gleeson et al., 1994; Munro, 1998; Rhee et al., 2005). Соотношение активности гликозилтрансфераз, локализованных на мембранах эндоплазматического ретикулума и аппарата Гольджи приблизительно 10 : 90 (Rhee et al., 2005). Пул трансфераз комплекса Гольджи постоянно пополняется за счет переноса ферментов из эндоплазматической сети в виде их гомомерных ассоциаций (Storrie et al., 1998). В аппарате Гольджи эти гомомерные комплексы гликозилтрансфераз распадаются (Hassinen, Kellokumpu, 2014) и далее формируют функционально активные гетеромерные ассоциации (конвейерные линии), обеспечивающие синтез олигосахаридов со строго детерминированной структурой путем последовательного проявления ферментативной активности (Jungmann, Munro, 1998; McCormick et al., 2000; de Graffenried, Bertozzi, 2004; Hassinen et al., 2011; Schoberer et al., 2013).
Таким образом, структура синтезируемых олигосахаридов зависит как от спектра экспрессируемых гликозилтрансфераз, так и от пространственной локализации энзимов на мембранах комплекса Гольджи (Nairn et al., 2012). Но как гликозилтрансферазы становятся резидентными белками аппарата Гольджи и как эти ферменты, многие из которых конкурируют за одни и те же акцепторы – нуклеозиддифосфат-сахара, могут формировать функциональные гетерокомплексы для синтеза олигосахаридов определенной структуры, во многом остается загадкой (Hassinen, Kellokumpu, 2014). В связи с этим сформулировано несколько дополняющих друг друга гипотез, призванных объяснить биохимические феномены, определяющие структуру олигосахаридов:
– длина трансмембранного домена гликозилтрансфераз, состоящего из 16–22 гидрофобных аминокислотных остатков, соответственно колебаниям толщины плазмалеммы, может предопределять локализацию на поверхности мембран цистерн аппарата Гольджи каталитического С-концевого домена и участка стволового домена (Teasdale et al., 1992; Munro, 1995). В процессе удержания данных трансфераз в составе мембран комплекса Гольджи, по-видимому, участвуют также цитозольные N-концевые домены ферментов (Quiroga et al., 2013; Becker et al., 2018). Фиксация фермента относительно мембранных структур во многом определяется формированием SS-связей и подвержена окислительно-восстановительной модификации;
– гликозилтрансферазы, катализирующие различные типы реакций, локализованы в различных цистернах комплекса Гольджи и не конкурируют между собой, вступая в реакции лишь на соответствующей стадии синтеза олигосахарида (Rabouille et al., 1995);
– замкнутый объем цистерн аппарата Гольджи предоставляет возможность поддерживать особый характер микросреды (значения величины рН, показатели концентрации ионов), оптимальный для активности ферментов в конкретной цистерне (Weisz, 2003; Paroutis et al., 2004; Rivinoja et al., 2009; Reynders et al., 2011).
Следует подчеркнуть, что ни одна из этих гипотез не имеет достаточного экспериментального подтверждения.
Схема расположения различных типов гликозилтрансфераз в мембране цистерн аппарата Гольджи и формирование их комплексов посредством взаимодействия со связывающими белками. Между фрагментами молекул фермента и интегрирующими белками формируются электростатические и SS-взаимодействия. В настоящее время допускают, что места фиксации связывающих белков и ферментов могут находиться как субмембранно (N-концевые домены), так и в просвете цистерн (вариабельные по размеру stem-домены).
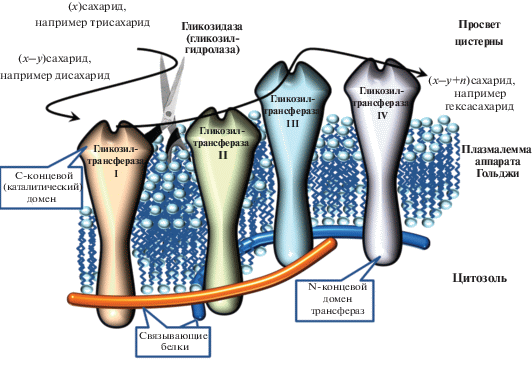
НЕОБХОДИМОСТЬ ВТОРИЧНОЙ МАТРИЦЫ ДЛЯ СИНТЕЗА ПОЛИСАХАРИДОВ
Среди специалистов-гликобиологов широко распространено мнение о том, что синтез олигосахаридов осуществляется без матрицы (Varki, 2011; Zhang et al., 2016; Aguilar et al., 2017). То есть, считается, что набор олигосахаридов в структуре гликанов закодирован в геноме не прямо, а опосредованно, при этом их строение не тождественно структурной организации ДНК (von der Lieth et al., 2004; Springer, Gagneux, 2013; Krasnova, Wong, 2016; Aguilar et al., 2017). Но, несмотря на отсутствие прямого генетического шаблона (матрицы), наследуемость структуры олигосахаридов весьма высока (Knežević et al., 2009; Pucić et al., 2011) и достигает 80% для некоторых гликанов, декорирующих иммуноглобулины (Menni et al., 2013; Lomax-Browne et al., 2017).
Учитывая некоторые особенности экспрессии апериодических полисахаридов, можно предположить облигатное участие вторичных матриц в их синтезе:
– профиль экспрессируемых гликанов может быстро (в течение 12 ч) изменяться при воспалении (Rombouts et al., 2016; Joenvaara et al., 2018);
– доказана обратимость аберрантного гликозилирования, ассоциированного с воспалением (Guipaud et al., 2007; Chaze et al., 2013; Collins et al., 2013; Horvat et al., 2013);
– олигосахариды репродуцируемы, а их способность высокоспецифично изменять эффекторные функции протеинов предполагает структурное соответствие тех или иных гликоформ белков структурам соответствующих лектинов, антигенов и рецепторов (Arnold et al., 2007; Taylor, Drickamer, 2014; Hayes et al., 2014; Subedi, Barb, 2015; Jennewein, Alter, 2017; Quast et al., 2017);
– полимерные цепочки олигосахаридов в процессе их синтеза претерпевают не только реакции присоединения активированных моносахаридов, но также могут подвергаться реконструированию – удалению части гликановых цепей и инициированию их разветвления; последовательное упорядочивание структуры гликановых цепей высокоселективно и иерархично (Kadirvelraj et al., 2018). В редактировании структуры гликанов наряду с гликозилтрансферазами активное участие принимают гликозил-гидролазы, формируя высокоспецифичный информационный код, обеспечивающий межклеточную кооперацию и взаимодействие с микробиомом;
– в процессе внутриклеточного синтеза гликанов (в отличие от синтеза in vitro) различные гликозилтрансферазы, катализирующие специфические типы реакций, локализованы в разных цистернах комплекса Гольджи, вступают в реакции лишь на соответствующей стадии формирования цепи апериодического олигосахарида (Rabouille et al., 1995) и не конкурируют между собой за акцептор, то есть финальная структура гликанов не является результатом конкурентных отношений активностей гликозилтрансфераз (Rabouille et al., 1995; Pucić et al., 2011).
В топологической (пространственной) организации трансмембранных ферментов синтеза гликанов, то есть в упорядочении их локализации на мембранах аппарата Гольджи, также могут принимать участие белки, не обладающие ферментативной активностью относительно формирования гликозидных связей. Такие протеины, определяя топологию гликозилтрансфераз и гликозидаз в составе мультиферментных ассоциаций, тем самым предопределяют структуру апериодических олигосахаридов. На роль топологических организаторов высокоспецифичных функциональных ассоциаций ферментов синтеза апериодических олигосахаридов могут претендовать некоторые ГТФазы семейства Rab. Известно около семидесяти представителей ГТФаз данного семейства, около трети из которых ассоциированы с мембранами секреторных органелл, способны формировать и поддерживать структуру белковых комплексов аппарата Гольджи (Goud et al., 2018). Такие белки, поступающие во внутриклеточные мембранные компартменты клеток и вступающие в связи с доменами гликозилтрансфераз (во многих случаях взаимное расположение ферментов синтеза гликанов в цистернах аппарата Гольджи зависит от N-концевых доменов энзимов (Sasai et al., 2001; Nagai et al., 2004; Tu, Banfield, 2010; Becker et al., 2018), вполне могут выполнять роль своеобразной матрицы для биосинтеза олигосахаридов, выстраивая ферменты синтеза гликанов в виде функциональных конвейерных линий. Представления об организации синтеза гликанов на мембранах аппарата Гольджи систематизированы на схеме (рисунок).
Естественно, структура таких матричных белковых молекул, а следовательно и их сродство к ферментам синтеза гликанов, могут быстро изменяться под влиянием динамики значений величины рН и показателя окислительно-восстановительного потенциала биосреды цистерн аппарата Гольджи, что косвенно подтверждается экспериментальными данными (Rivinoja et al., 2009; Maeda, Kinoshita, 2010; Hassinen et al., 2011; Hassinen, Kellokumpu, 2014; Taniguchi et al., 2016). Каждый из матричных протеинов, вероятно, может фиксировать в составе функционально активных комплексов всего лишь несколько энзимов в силу размеров их каталитических доменов. Поэтому в процессе синтеза 10-/20-звенных олигосахаридов растущая углеводная цепь последовательно передается от одного полиферментного комплекса к другому, локализованным как в пределах одной цистерны, так и в других компартментах (цис-, медиальных и транс-отделах) аппарата Гольджи. Учитывая многообразие и структурную сложность гликанов, множественность форм энзимов синтеза олигосахаридов, следует исходить из того, что синтез “правильных” апериодических олигосахаридов осуществляется при участии множества малоразмерных белков-матриц, формирующих функциональные полиферментные комплексы в цистернах аппарата Гольджи.
Уникальное структурное разнообразие апериодических олигосахаридов, по сути дела, представляет собой трудно преодолимый барьер как в изучении их структурно-функциональных характеристик гликанов (Hsu et al., 2018a,b), так и особенностей их синтеза (Wang, Amin, 2014; Pardo-Vargas et al., 2018). Поэтому гликаны во многом еще остаются “темной материей” биологической вселенной (Varki, 2013). Идентификация белков-матриц биосинтеза апериодических олигосахаридов может оказаться полезной в решении проблем биотехнологии синтеза гликанов и направленной коррекции их биологических свойств.
ЗАКЛЮЧЕНИЕ
Таким образом, формирование гипотезы о матричном синтезе апериодических полисахаридов позволяет объяснить постоянство определяемых гликанами на протяжении всей жизни характеристик структурной организации метаболических процессов, характеристик межклеточной кооперации в макроорганизме и его взаимоотношения с микробиомом. По-видимому, ансамблевая организация ферментов синтеза гликанов определяется различными белками, не ограниченными принадлежностью к семейству Raf. Наличие белкового (аминокислотного) кода, реализуемого в многообразии форм гликанов, определяет вариабельность форм структурной и метаболической организации в живых системах.
Список литературы
Бакулина Л.С., Константинов Д.П., Купрякова А.Ф. и др. Энтропия, кислород и биогенез // Оксидативный стресс и воспаление: патогенетическое партнерство / Ред. О.Г. Хурцилава, Н.Н. Плужников, А.Я. Накатис. СПб.: СЗГМУ, 2012. С. 6–21.
Винер Н. Кибернетика, или управление и связь в животном и машине. М.: Наука, 1983. 344 с.
Волькенштейн М.В. Общая биофизика. М.: Наука, 1978. 592 с. (С. 87–95).
Колмогоров А.Н. Три подхода к определению понятия “количество информации” // Пробл. передачи информ. 1965. Т. 1. Вып. 1. С. 3–11.
Наумов Д.Г. Иерархическая классификация гликозил-гидролаз // Биохимия. 2011. Т. 76. Вып. 6. С. 764–780.
Силин А.А. Энтропия, вероятность, информация // Вестн. РАН. 1994. Т. 64. № 6. С. 490–496.
Щербаков В.П. Эволюция как сопротивление энтропии. I. Механизмы видового гомеостаза // Журн. общ. биол. 2005а. Т. 66. № 3. С. 195–211.
Щербаков В.П. Эволюция как сопротивление энтропии. II. Консервативная роль полового размножения // Журн. общ. биол. 2005б. Т. 66. № 4. С. 300–309.
Ackerman M.E., Crispin M., Yu X. et al. Natural variation in Fc glycosylation of HIV-specific antibodies impacts antiviral activity // J. Clin. Invest. 2013. V. 123. № 5. P. 2183–2192.
Aguilar A.L., Briard J.G., Yang L. et al. Tools for studying glycans: recent advances in chemoenzymatic glycan labeling // ACS Chem. Biol. 2017. V. 12. № 3. P. 611–621.
Allard S.T., Giraud M.F., Naismith J.H. Epimerases: structure, function and mechanism // Cell. Mol. Life Sci. 2001. V. 58. № 11. P. 1650–1665.
Anthony R.M., Kobayashi T., Wermeling F., Ravetch J.V. Intravenous gammaglobulin suppresses inflammation through a novel TH2 pathway // Nature. 2011. V. 475. № 7354. P. 110–113.
Arnold J.N., Wormald M.R., Sim R.B. et al. The impact of glycosylation on the biological function and structure of human immuniglobulins // Annu. Rev. Immunol. 2007. V. 25. P. 21–50.
Audagnotto M., Dal Peraro M. Protein post-translational modifications: in silico prediction tools and molecular modeling // Comput. Struct. Biotechnol. J. 2017. V. 15. P. 307–319.
Becker J.L., Tran D.T., Tabak L.A. Members of the GalNAc-T family of enzymes utilize distinct Golgi localization mechanisms // Glycobiology. 2018. V. 28. № 11. P. 841–848.
Beltrao P., Bork P., Krogan N.J., van Noort V. Evolution and functional cross-talk of protein post-translational modifications // Mol. Syst. Biol. 2013. V. 9. Art. 714.
Chaze T., Slomianny M.C., Milliat F. et al. Alteration of the serum N-glycome of mice locally exposed to high doses of ionizing radiation // Mol. Cell. Proteom. 2013. V. 12. № 2. P. 283–301.
Chi P.B., Liberles D.A. Selection on protein structure, interaction, and sequence // Protein Sci. 2016. V. 25. № 7. P. 1168–1178.
Cohen M., Varki A. The sialome – far more than the sum of its parts // OMICS. 2010. V. 14. № 4. P. 455–464.
Colley K.J. Golgi localization of glycosyltransferases: more questions than answers // Glycobiology. 1997. V. 7. № 1. P. 1–13.
Collins E.S., Galligan M.C., Saldova R. et al. Glycosylation status of serum in inflammatory arthritis in response to anti-TNF treatment // Rheumatology. 2013. V. 52. № 9. P. 1572–1582.
Cooper G.M., Hausman R.E. The cell: a molecular approach. Sunderland: Sinauer Associates, 2016. 832 p.
Coutinho P.M., Deleury E., Davies G.J., Henrissat B. An evolving hierarchical family classification for glycosyltransferases // J. Mol. Biol. 2003. V. 328. № 2. P. 307–317.
de Graffenried C.L., Bertozzi C.R. The roles of enzyme localisation and complex formation in glycan assembly within the Golgi apparatus // Curr. Opin. Cell. Biol. 2004. V. 16. № 4. P. 356–363.
de Haan N., Boeddha N.P., Ekinci E. et al. Differences in IgG Fc glycosylation are associated with outcome of pediatric meningococcal sepsis // MBio. 2018. V. 9. № 3. Art. e00546-18.
Delbianco M., Bharate P., Varela-Aramburu S., Seeberger P.H. Carbohydrates in supramolecular chemistry // Chem. Rev. 2016. V. 116. № 4. P. 1693–1752.
DiLoretto R., Murphy C.T. The cell biology of aging // Mol. Biol. Cell. 2015. V. 26. № 25. P. 4524–4531.
International Human Genome Sequencing Consortium. Finishing the euchromatic sequence of the human genome // Nature. 2004. V. 431. № 7011. P. 931–945.
Gabius H.-J. The sugar code: why glycans are so important // Biosystems. 2018. V. 164. P. 102–111.
Gabius H.-J., Kayser K. Introduction to glycopathology: the concept, the tools and the perspectives // Diagn. Pathol. 2014. V. 9. Art. 4.
Gabius H.-J., Roth J. An introduction to the sugar code // Histochem. Cell Biol. 2017. V. 147. № 2. P. 111–117.
Gabius H.-J., Andre S., Jimenez-Barbero J. et al. From lectin structure to functional glycomics: principles of the sugar code // Trends Biochem. Sci. 2011. V. 36. № 6. P. 298–313.
Gavrilov L.A., Gavrilova N.S. Evolutionary theories of aging and longevity // Sci. World J. 2002. V. 2. P. 339–356.
Gilson A.I., Marshal-Christensen A., Choi J.M., Shakhnovich E.I. The role of evolutionary selection in the dynamics of protein structure evolution // Biophys. J. 2017. V. 112. № 7. P. 1350–1365.
Gleeson P.A., Teasdale R.D., Burke J. Targeting of proteins to the Golgi apparatus // Glycoconj. J. 1994. V. 11. № 5. P. 381–394.
Goud B., Liu S., Storrie B. Rab proteins as major determinants of the Golgi complex structure // Small GTPases. 2018. V. 9. № 1–2. P. 66–75.
Griffiths A.J.F., Gelbart W.M., Lewontin R.C., Miller J.H. Modern genetic analysis: integrating genes and genomes. N.Y.: W.H. Freeman, 2002. 736 p.
Griffiths A.J.F., Wessler S.R., Carroll S.B., Doebley J. An introduction to genetic analysis. N.Y.: W.H. Freeman, 2012. 862 p.
Guipaud O., Holler V., Buard V. et al. Time-course analysis of mouse serum proteome changes following exposure of the skin to ionizing radiation // Proteomics. 2007. V. 7. № 21. P. 3992–4002.
Hassinen A., Kellokumpu S. Organizational interplay of Golgi N-glycosyltransferases involves organelle microenvironment-dependent transition between enzyme homo- and heteromers // J. Biol. Chem. 2014. V. 289. № 39. P. 26937–26948.
Hassinen A., Pujol F.M., Kokkonen N. et al. Functional organization of Golgi N- and O‑glycosylation pathways involves pH-dependent complex formation that is impaired in cancer cells // J. Biol. Chem. 2011. V. 286. № 44. P. 38329–38340.
Hatton L., Warr G. Protein structure and evolution: are they constrained globally by a principle derived from information theory? // PLoS One. 2015. V. 10. № 5. Art. e0125663.
Hayes J.M., Cosgrave E.F., Struwe W.B. et al. Glycosylation and Fc receptors // Curr. Top. Microbiol. Immunol. 2014. V. 382. P. 165–199.
Horvat T., Dezeljin M., Redzic I. et al. Reversibility of membrane N-glycome of HeLa cells upon treatment with epigenetic inhibitors // PLoS One. 2013. V. 8. № 1. Art. e54672.
Hsu H.C., Liew C.Y., Huang S.P. et al. Simple method for de novo structural determination of underivatised glucose oligosaccharides // Sci. Rep. 2018a. V. 8. Art. 5562.
Hsu H.C., Liew C.Y., Huang S.P. et al. Simple approach for de novo structural identification of mannose trisaccharides // J. Am. Soc. Mass. Spectrom. 2018b. V. 29. № 3. P. 470–480.
Hu Z., Scott H., Qin G. et al. Revealing missing human protein isoforms based on ab initio prediction, RNA-seq and proteomics // Sci. Rep. 2015. V. 5. Art. 10940.
Jennewein M.F., Alter G. The immunoregulatory roles of antibody glycosylation // Trends Immunol. 2017. V. 38. № 5. P. 358–372.
Joenvaara S., Saraswat M., Kuusela P. et al. Quantitative N-glycoproteomics reveals altered glycosylation levels of various plasma proteins in bloodstream infected patients // PLoS One. 2018. V. 13. № 3. Art. e0195006.
Joud M., Moller M., Olsson M.L. Identification of human glycosyltransferase genes expressed in erythroid cells predicts potential carbohydrate blood group loci // Sci. Rep. 2018. V. 8. Art. 6400.
Jungmann J., Munro S. Multi-protein complexes in the cis Golgi of Saccharomyces cerevisiae with alpha-1,6-mannosyltransferase activity // EMBO J. 1998. V. 17. № 2. P. 423–434.
Kadirvelraj R., Yang J.Y., Sanders J.H. et al. Human N-acetylglucosaminyltransferase II substrate recognition uses a molecular architecture that includes a convergent exosite // PNAS USA. 2018. V. 115. № 18. P. 4637–4642.
Kamili N.A., Arthur C.M., Gerner-Smith C. et al. Key regulators of galectins-glycan interactions // Proteomics. 2016. V. 16. № 24. P. 3111–3125.
Kaneko Y., Nimmerjahn F., Ravetch J.V. Anti-inflammatory activity of immunoglobulin G resulting from Fc sialylation // Science. 2006. V. 313. № 5787. P. 670–673.
Kletter D., Singh S., Bern M., Haab B.B. Global comparison of lectin-glycan interactions using a database of analyzed glycan array data // Mol. Cell. Proteom. 2013. V. 12. № 4. P. 1026–1035.
Knežević A., Polašek O., Gornik O. et al. Variability, heritability and environmental determinants of human plasma N-glycome // J. Proteome Res. 2009. V. 8. № 2. P. 694–701.
Knorre D.G., Kudryashova N.V., Godovikova T.S. Chemical and functional aspects of posttranslational modification of proteins // Acta Nature. 2009. V. 1. № 3. P. 29–51.
Krasnova L., Wong C.H. Understanding the chemistry and biology of glycosylation with glycan synthesis // Annu. Rev. Biochem. 2016. V. 85. P. 599–630.
Laine R.A. A calculation of all possible oligosaccharide isomers both branched and linear yields 1.05 × 1012 structures for a redicing hexasaccharide: the isomer barrier to development of single-method saccharide sequencing or synthesis systems // Glycobiology. 1994. V. 4. № 6. P. 759–767.
Laine R.A. The information-storing potential of the sugar code // Glycosciences: status and perspectives / Eds H.‑J. Gabius, S. Gabius. London: Chapman and Hall, 1997. P. 1–14.
Lairson L.L., Henrissat B., Davies G.J., Withers S.G. Glycosyltransferases: structure, functions, and mechanisms // Annu. Rev. Biochem. 2008. V. 77. P. 521–555.
Landhuis E. Glycobiology: sweet success // Nature. 2017. V. 547. № 7661. P. 127–129.
Latxague L., Gaubert A., Barthelemy P. Recent advances in the chemistry of glycoconjugates amphiphiles // Molecules. 2018. V. 23. № 1. Art. 89.
Lauc G., Huffman J.E., Pucic M. et al. Loci associated with N-glycosylation of human immunoglobulin G show pleiotropy with autoimmune diseases and haematological cancers // PLoS Genet. 2013. V. 9. № 1. Art. e1003225.
Lauc G., Krištić J., Zoldoš V. Glycans – the third revolution in evolution // Front. Genet. 2014. V. 5. Art. 145.
Ljubuncic P., Reznick A.Z. The evolutionary theories of aging revisited – a mini-review // Gerontology. 2009. V. 55. № 2. P. 205–216.
Lomax-Browne H.J., Visconti A., Pusey C.D. et al. IgA1 glycosylation is heritable in healthy twins // J. Am. Soc. Nephrol. 2017. V. 28. № 1. P. 64–68.
Longo V.D., Mitteldorf J., Skulachev V.P. Programmed and altruistic ageing // Nat. Rev. Genet. 2005. V. 6. № 11. P. 866–872.
McArthur J.B., Chen X. Glycosyltransferase engineering for carbohydrate synthesis // Biochem. Soc. Trans. 2016. V. 44. № 1. P. 129–142.
McCormick C., Duncan G., Goutsos K.T., Tufaro F. The putative suppressors EXT1 and EXT2 form a stable complex that accumulates in the Golgi apparatus and catalyzes the synthesis of heparan sulfate // PNAS USA. 2000. V. 97. № 2. P. 668–673.
Mackenzie P.I., Owens I.S., Burchell B. et al. The UDP glycosyltransferase gene superfamily: recommended nomenclature update based on evolutionary divergence // Pharmacogenetics. 1997. V. 7. № 4. P. 255–269.
Maeda Y., Kinoshita T. The acidic environment of the Golgi is critical for glycosylation and transport // Methods Enzymol. 2010. V. 480. P. 495–510.
Mahan A.E., Jennewein M.F., Suscovich T. et al. Antigen-specific antibody glycosylation is regulated via vaccination // PLoS Pathog. 2016. V. 12. № 3. Art. e1005456.
Marth J.D. A unified vision of the building blocks of life // Nat. Cell. Biol. 2008. V. 10. № 9. P. 1015–1016.
Masibay A.S., Balaji P.V., Boeggman E.E., Qasba P.K. Mutational analysis of the Golgi retention signal of bovine beta-1,4-galactosyltransferase // J. Biol. Chem. 1993. V. 268. № 13. P. 9908–9916.
Martins A.C.R. Change and aging senescence as an adaptation // PLoS One. 2011. V. 6. № 9. Art. e24328.
Menni C., Keser T., Mangino M. et al. Glycosylation of immunoglobulin G: role of genetic and epigenetic influences // PLoS One. 2013. V. 8. № 12. Art. e82558.
Minguez P., Parca L., Diella F. et al. Deciphering a global network of functionally associated post-translational modifications // Mol. Syst. Biol. 2012. V. 8. № 1. Art. 599.
Moremen K.W., Ramiah A., Stuart M. et al. Expression system for structural and functional studies of human glycosylation enzymes // Nat. Chem. Biol. 2018. V. 14. № 2. P. 156–162.
Munro S. An investigation of the role of transmembrane domains in Golgi protein retention // EMBO J. 1995. V. 14. № 19. P. 4695–4704.
Munro S. Localization of proteins to the Golgi apparatus // Trends Cell Biol. 1998. V. 8. № 1. P. 11–15.
Muthana S.M., Campbell C., Gildersleeve J.C. Modifications of glycans: biological significance and therapeutic opportunities // ACS Chem. Biol. 2012. V. 7. № 1. P. 31–43.
Nagai N., Habuchi H., Esko J.D., Kimata K. Stem domains of heparan sulfate 6‑O‑sulfotransferase are required for Golgi localization, oligomer formation and enzyme activity // J. Cell Sci. 2004. V. 117. № 15. P. 3331–3341.
Nairn A.V., York W.S., Harris K. et al. Regulation of glycan structures in animal tissues: transcript profiling of glycan-related genes // J. Biol. Chem. 2008. V. 283. № 25. P. 17298–17313.
Nairn A.V., Aoki K., dela Rosa M. et al. Regulation of glycan structures in murine embryonic stem cells: combined transcript profiling of glycan-related genes and glycan structural analysis // J. Biol. Chem. 2012. V. 287. № 45. P. 37835–37856.
Narimatsu Y., Joshi H.J., Yang Z. et al. A validated gRNA library for CRISPR/Cas9 targeting of the human glycosyltransferase genome // Glycobiology. 2018. V. 28. № 5. P. 295–305.
National Research Council. Transforming glycoscience: a roadmap for the future. Washington, DC: National Academies Press, 2012. 208 p.
Nelson P., Masel J. Intercellular competition and the inevitability of multicellular aging // PNAS USA. 2017. V. 114. № 49. 12982–12987.
Nilsen T.W., Graveley B.R. Expansion of the eukaryotic proteome by alternative splicing // Nature. 2010. V. 463. № 7280. P. 457–463.
Pan Q., Shai O., Lee L.J. et al. Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing // Nat. Genet. 2008. V. 40. № 12. P. 1413–1415.
Pardo-Vargas A., Delbianco M., Seeberger P.H. Automated glycan assembly as an enabling technology // Curr. Opin. Chem. Biol. 2018. V. 46. P. 48–55.
Paroutis P., Touret N., Grinstein S. The pH of the secretory pathway: measurement, determinants, and regulation // Physiology. 2004. V. 19. № 4. P. 207–215.
Prinzinger R. Programmed ageing: the theory of maximal metabolic scope // EMBO Rep. 2005. V. 6 (Suppl). P. S14–S19.
Pucić M., Knežević A., Vidič J. et al. High throughput isolation and glycosylation analysis of IgG-variability and heritability of the IgG glycome in three isolated human populations // Mol. Cell. Proteom. 2011. V. 10. № 10. Art. M111.010090.
Quast I., Peschke B., Lunemann J.D. Regulation of antibody effector functions through IgG Fc N-glycosylation // Cell Mol. Life Sci. 2017. V. 74. № 5. P. 837–847.
Quiroga R., Trenchi A., Gonzalez Montoro A. et al. Short transmembrane domains with high-volume exoplasmic halves determine retention of type II membrane proteins in the Golgi complex // J. Cell. Sci. 2013. V. 126. № 23. P. 5344–5349.
Rabouille C., Hui N., Hunte F. et al. Mapping the distribution of Golgi enzymes involved in the construction of complex oligosaccharides // J. Cell. Sci. 1995. V. 108. № 4. P. 1617–1627.
Reynders E., Foulquier F., Annaert W., Matthijs G. How Golgi glycosylation meets and needs trafficking: the case of the COG complex // Glycobiology. 2011. V. 21. № 7. P. 853–863.
Rhee S.W., Starr T., Forsten-Williams K., Storrie B. The steady-state distribution of glycosyltransferases between the Golgi apparatus and the endoplasmic reticulum is aproximatelly 90:10 // Traffic. 2005. V. 6. № 11. P. 978–990.
Rivinoja A., Hassinen A., Kokkonen N. et al. Elevated Golgi pH impairs terminal N‑glycosylation by inducing mislocalization of Golgi glycosyltransferases // J. Cell. Physiol. 2009. V. 220. № 1. P. 144–154.
Rombouts Y., Jónasdóttir H., Hipgrave Ederveen A.L. et al. Acute phase inflammation characterized by rapid changes in plasma/peritoneal fluid N-glycosylation in mice // Glycoconj. J. 2016. V. 33. № 3. P. 457–470.
Rüdiger H., Gabius H.-J. The biochemical basis and coding capacity of sugar code // The sugar code: fundamentals of glycosciences / Ed. H.-J. Gabius. Weinheim: Wiley-Blackwell, 2009. P. 3–13.
Sasai K., Ikeda Y., Tsuda T. et al. The critical role of the stem region as a functional domain responsible for the oligomerization and Golgi localization of N-acetylglucosaminyltransferase V. The involvement of a domain hemophilic interaction // J. Biol. Chem. 2001. V. 276. № 1. P. 759–765.
Schnaar R.L. Glycobiology simplified: diverse roles of glycan recognition in inflammation // J. Leukoc. Biol. 2016. V. 99. № 6. P. 825–838.
Schoberer J., Liebminner E., Botchway S.W. et al. Time-resolved fluorescence imaging reveals differential interactions of N-glycan processing enzymes across the Golgi stack in planta // Plant Physiol. 2013. V. 161. № 4. P. 1737–1754.
Seeberger P.H. Bioinformatics – key to the future of chemical glycomics // Proceeding of the International Beilstein Symposium on Glyco-Bioinformatics: Bits`n`Bytes of Sugars / Eds M.G. Hicks, C. Kettner. Frankfurt: Logos Verlag Berlin, 2011. P. 25–36.
Shannon C.E. A mathematical theory of communication // Bell Syst. Tech. J. 1948. V. 27. № 3. P. 379–423.
Shannon C.E. A mathematical theory of communication // Bell Syst. Tech. J. 1948. V. 27. № 4. P. 623–656.
Shinkawa T., Nakamura K., Yamane N. et al. The absence of fucose but not the presence of galactose or bisecting N-acetylglucosamine of human IgG1 complex-type oligosaccharides shows the critical role of enhancing antibody-dependent cellular cytotoxicity // J. Biol. Chem. 2003. V. 278. № 5. P. 3466–3473.
Siltberg-Liberles J., Grahnen J.A., Liberles D.A. The evolution of protein structures and structural ensembles under functional constrains // Genes. 2011. V. 2. № 4. P. 748–762.
Skulachev V.P. Programmed death phenomena: from organelle to organism // Ann. N.Y. Acad. Sci. 2002. V. 959. № 1. P. 214–237.
Smith L.M., Kelleher N.L. Proteoform: a single term describing protein complexity // Nat. Methods. 2013. V. 10. № 3. P. 186–187.
Sood A., Gerlits O.O., Ji Y. et al. Defining the specificity of carbohydrate-protein interactions by quantifying functional group contributions // J. Chem. Inf. Model. 2018. V. 58. № 9. P. 1889–1901.
Springer S.A., Gagneux P. Glycan evolution in response to collaboration, conflict, and constraint // J. Biol. Chem. 2013. V. 288. № 10. P. 6904–6911.
Stergachis A.B., Haugen E., Shafer A. et al. Exonic transcription factor directs codon choice and affects protein evolution // Science. 2013. V. 342. № 6164. P. 1367–1372.
Stern R., Jedrzejas M.J. Carbohydrate polymers at the center of life`s origins: the importance of molecular processivity // Chem. Rev. 2008. V. 108. № 12. P. 5061–5085.
Storrie B., White J., Rottger S. et al. Recycling of Golgi-resident glycosyltransferases through the ER reveals a novel pathway and provides an explanation for nocodazole-induced Golgi scattering // J. Cell. Biol. 1998. V. 143. № 6. P. 1505–1521.
Subedi G.P., Barb A.W. The structural role of antibody N-glycosylation in receptor interactions // Structure. 2015. V. 23. № 9. P. 1573–1583.
Taniguchi N., Kizuka Y., Takamatsu S. et al. Glyco-redox, a link between oxidative stress and changes of glycans: lessons from research on glutathione, reactive oxygen and nitrogen species to glycobiology // Arch. Biochem. Biophys. 2016. V. 595. P. 72–80.
Taylor M.E., Drickamer K. Convergent and divergent mechanisms of sugar recognition across kingdoms // Curr. Opin. Struct. Biol. 2014. V. 28. P. 14–22.
Teasdale R.D., D’Agostaro G., Gleeson P.A. The signal for Golgi retention of bovine beta 1,4‑galactosyltransferase is in the transmembrane domain // J. Biol. Chem. 1992. V. 267. № 6. P. 4084–4096.
Tu L., Banfield D.K. Localization of Golgi-resident glycosyl-transferases // Cell. Mol. Life Sci. 2010. V. 67. № 1. P. 29–41.
van Overtveldt S., Verhaeghe T., Joosten H.J. et al. A structural classification of carbohydrate epimerases: from mechanistic insights to practical applications // Biotechnol. Adv. 2015. V. 33. № 8. P. 1814–1828.
Varki A. Evolutionary forces shaping the Golgi glycosylation machinery: why cell surface glycans are universal to living cells Cold Spring Harb // Perspect. Biol. 2011. V. 3. № 6. Art. a005462.
Varki A. Omics: account for the “dark matter” of biology // Nature. 2013. V. 497. № 7451. Art. 565.
Varki A. Biological roles of glycans // Glycobiology. 2017. V. 27. № 1. P. 3–49.
Varki A., Cummings R.D., Esko J.D. et al. Essentials of glycobiology. N.Y.: Cold Spring Harbor Laboratory Press, 2009. 784 p.
Varki A., Cummings R.D., Aebi M. et al. Symbol nomenclature for graphical representation of glycans // Glycobiology. 2015. V. 25. № 12. P. 1323–1324.
Venter J.C., Adams M.D., Meyers E.W. et al. The sequence of the human genome // Science. 2001. V. 291. № 5507. P. 1304–1351.
Vliegenthart J.F.G. The complexity of glycoprotein-derived glycans // Proc. Jpn. Acad. Ser. B Phys. Biol. Sci. 2017. V. 93. № 2. P. 64–86.
von der Lieth C.W., Bohne-Lang A., Lohmann K.K., Frank M. Bioinformatics for glycomics: status, methods, requirements and perspectives // Brief Bioinform. 2004. V. 5. № 2. P. 164–178.
Wang L.X., Amin M.N. Chemical and chemoenzymatic synthesis of glycoproteins for deciphering functions // Chem. Biol. 2014. V. 21. № 1. P. 51–66.
Wang Z., Chinoy Z.S., Ambre S.G. et al. A general strategy for the chemoenzymatic synthesis of asymmetrically branched N-glycans // Science. 2013. V. 341. № 6144. P. 379–383.
Wasim L., Buhardi F.H.M., Yoganathan M. et al. N-linked glycosylation regulates CD22 organization and function // Front. Immunol. 2019. V. 10. Art. 699.
Weisz O.A. Acidification and protein traffic // Int. Rev. Cytol. 2003. V. 226. P. 259–319.
Werz D.B., Ranzinger R., Herget S. et al. Exploring structural diversity of mammalian carbohydrates (“glycospace”) by statistical databank analysis // ACS Chem. Biol. 2007. V. 2. № 10. P. 685–691.
Wiener N. Cybernetics: or control and communication in the animal and the machine. Cambridge: The Technology, 1948. 194 p.
Williams G.J., Thorson J.S. Natural product glycosyltransferases: properties and applications // Adv. Enzymol. Relat. Areas Mol. Biol. 2009. V. 76. P. 55–119.
Yamamoto-Hino M., Kanie Y., Awano W. et al. Identification of genes required for neural-specific glycosylation using functional genomics // PLoS Genet. 2010. V. 6. № 12. P. e1001254.
Zhang P., Woen S., Wang T. et al. Challenges of glycosylation analysis and control: an integrated approach to producing optimal and consistent therapeutic drugs // Drug Discov. Today. 2016. V. 21. № 5. P. 740–765.
Zielinska D.F., Gnad F., Wisniewski J.R., Mann M. Precision mapping of an in vivo N‑glycoproteome reveals rigid topological and sequence constrains // Cell. 2010. V. 141. № 5. P. 897–907.
Дополнительные материалы отсутствуют.
Инструменты
Успехи современной биологии


