Успехи физиологических наук, 2021, T. 52, № 3, стр. 4-23
Редактирование генома и регуляция экспрессии генов с помощью технологий CRISPR/Сas в нейробиологии
Л. Н. Гринкевич *
Федеральное государственное бюджетное учреждение науки Институт физиологии им. И.П. Павлова РАН
199034 Санкт-Петербург, Россия
* E-mail: Larisa_Gr_SPB@mail.ru
Поступила в редакцию 02.02.2021
После доработки 10.03.2021
Принята к публикации 12.03.2021
Аннотация
CRISPR/Cas – это революционные технологии для направленного редактирования генома и эпигенома, позволяющие включать/выключать, а также модифицировать практически любой ген. Эти технологии обладают значительным потенциалом как для фундаментальных исследований, в том числе в области когнитивных наук, так и для терапии когнитивных расстройств. С помощью технологии CRISPR/Cas можно модифицировать гены человека, животных, растений и микроорганизмов и регулировать их экспрессию, что достаточно просто технически и не очень затратно. Данный обзор посвящен использованию CRISPR/Cas для исследования молекулярных механизмов функционирования центральной нервной системы (ЦНС) в норме и при патологиях. Основные трудности применения CRISPR/Cas для редактирования генома в ЦНС связаны со сложностью ее строения, неделимостью нейронов и наличием гематоэнцефалического барьера. Для изучения функций генов в ЦНС часто необходимо редактировать одновременно несколько генов, причем в разных структурах и клетках мозга. В обзоре обсуждены достижения последних лет, связанные с адаптацией технологий CRISPR/Cas для генетических модификаций и регуляции экспрессии генов в нейронах in vitro и in vivo. Последний раздел обзора будет посвящен возможностям применения этих технологий для коррекции когнитивных дисфункций на модельных животных и потенциальным терапевтическим подходам для лечения заболеваний, связанных с когнитивными нарушениями у людей.
Технологии направленного редактирования генома CRISPR/Cas позволяют модифицировать гены-мишени, удалять или вставлять новые. Позже эти технологии были адаптированы для регуляции экспрессии генов на уровне транскрипции и редактирования эпигенома, что открывает перспективы для включения или выключения экспрессии гена в строго определенные временные рамки и, что важно, эти изменения обратимы. С помощью технологии CRISPR/Cas можно модифицировать гены человека, животных, растений и микроорганизмов или регулировать их экспрессию, что достаточно просто технически и не очень затратно. За последние несколько лет достигнуты огромные успехи в редактировании генома с помощью CRISPR/Cas. Была значительно повышена эффективность, клеточная специфичность и целевая направленность CRISPR/Cas-опосредованного нокаута и генной коррекции [52, 60]. Произошел значительный прогресс от направленной модификации генов к трехмерной инженерии генома, что позволит лучше понять механизмы регуляции экспрессии генов при адаптивных перестройках [110]. Ученые надеются, что с использованием технологий CRISPR/Cas можно будет лечить тяжелые заболевания, в том числе моногенные генетические [107]. Кроме того, системы CRISPR/Cas обладают большим потенциалом для адресной доставки лекарств и их высвобождения, что важно для развития персонализированной медицины. Данный обзор посвящен перспективам использования CRISPR/Cas для исследования молекулярных механизмов функционирования ЦНС и потенциальным терапевтическим подходам для лечения заболеваний, связанных с когнитивными нарушениями. Основные трудности применения CRISPR/Cas для редактирования генома в ЦНС связаны со сложностью ее строения и неделимостью нейронов; кроме того, нервная система ограждена гематоэнцефалическим барьером и поверхностно недоступна. Для изучения функций генов в нервной системе часто необходимо редактировать одновременно несколько генов, причем в разных структурах и клетках мозга, что требует введения конструкций, обладающих высокой клеточной специфичностью. Данная область в настоящее время находится в стадии интенсивных разработок [88]. В настоящее время технологии CRISPR/Cas непрерывно совершенствуются [из различных видов бактерий выделяются CRISPR/Cas, обладающие различными новыми свойствами, которые, к тому же, путем генно-инженерных манипуляций наделяются нужными характеристиками], что позволяет в ближайшее время ожидать большой прогресс в этой области.
ИСТОРИЯ ОТКРЫТИЯ CRISPR/CAS
В CRISPR/Cas-технологиях используется система CRISPR, открытая Франсиско Мохика, которая лежит в основе адаптивного иммунитета у бактерий. Исследуя древние бактерии Археи, он обнаружил наличие у них повторяющихся, идентичных палиндромных последовательностей ДНК, разделенных уникальными последовательностями – спейсерами. Эти кластерные структуры были названы CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats). Дальнейшие исследования Мохика показали, что последовательности нуклеотидов многих спейсеров совпадают с последовательностями геномов вирусов, паразитирующих на данных бактериях, и предположил, а впоследствии и доказал, что CRISPR система – это основа адаптивного иммунитета бактерий. В 2005 году он с трудом опубликовал на эту тему первую публикацию, которая сначала была отклонена несколькими крупными научными журналами, как не представляющая интереса и была опубликована только спустя 2 года [72]. К 2002 году были обнаружены белки Cas (CRISPR-associated sequence), кодируемые генами, обычно расположенными рядом с CRISPR-кассетами [41]. Белки CAS являются нуклеазами, они способны разрезать ДНК и соответственно уничтожать бактериальных паразитов. Прокариотические CRISPR кодируют две РНК, одна из них tracrRNA (трансактивируемая CRISPR-RNA), обеспечивающая активацию белка Сas, а другая crRNA (CRISPR-RNA), кодируемая спейсером, узнает ген-мишень, и комплементарно с ним связывается. При попадании вируса в клетку, белки Cas в комплексе с crRNA узнают вирус, и, если в спейсерах есть информация о данном вирусе (последовательность РНК, комплементарная последовательности спейсера), то белки Cas активируются связанными с ними tracrRNA, разрезают вирусную ДНК и уничтожают ее. Впоследствии было показано, что CRISPR- кассета с последовательностями, кодирующими белки CAS (CRISPR/CAS), составляет систему противовирусной защиты большинства прокариот [73].
В 2012 году Дженнифер Дудна и Эммануэль Шарпантье из Университета Беркли показали, что бактериальный комплекс CRISPR/Cas9 пригоден для расщепления практически любой двухцепочечной ДНК (дцДНК) и тем самым может быть эффективным инструментом для редактирования геномов любых организмов [43]. Для удобства работы они объединили tracrRNA с crRNA в единый транскрипт, названный sgRNA, добавили последовательность, кодирующую Cas9, и получили конструкцию для направленного расщепления любой ДНК-мишени, комплементарной небольшому участку sgRNA. Система CRISPR/Cas9 оказалась значительно более точной для сайт-специфического расщепления ДНК по сравнению с технологиями, использовавшимися ранее, а также значительно менее трудоемкой и менее дорогостоящей. Впоследствии группа Джорджа Черча и Фенг Жанга из Института Броуда экспериментально показали, что CRISPR/Cas9 способны работать в клетках многих эукариотов, в том числе человека [19].
Комплексы CRISPR/Cas, состоящие из белка Cas и sgRNA, могут быть экспрессированы в клетках при помощи ДНК плазмид, ДНК-вирусов, а также с помощью молекул РНК. Кроме того, возможна прямая доставка очищенного белка Cas в комплексе с искусственно синтезированной gRNA в виде рибонуклеиновых комплексов (RNP) или наночастиц/наноклубков [97]. В первом случае по замыслу экспериментатора собирается конструкция, состоящая из последовательностей ДНК, кодирующих белок CAS и sgRNA (tracrRNA + crRNA), а также промоторов, обеспечивающих запуск транскрипции. В crRNA закодирован РНК-гид, комплементарный участку гена, который планируется модифицировать. Схема конструкции представлена на рис. 1а.
Рис. 1.
Система редактирования генома CRISPR/Cas. (a) Схема генетической конструкции, кодирующей элементы системы CRISPR/Cas. Cas9 – последовательность, кодирующая белок Cas9. sgRNA – единая химерная РНК, содержащая части crRNA и tracrRNA. В crRNA закодирован RNA гид. U6, CMV – промоторы необходимые для транскрипции. NLS – сигнал ядерной локализации, необходимый для транслокации конструкций в ядро. ПолиА – сигнал полиаденилирования. (б) В клетках происходит синтез белка Сas9 (выделен сиреневым цветом) и sgРНК (crRNA + tracrRNA). Комплекс Сas9-sgРНК распознает искомый участок ДНК клетки и Сas9 создает двухцепочечный разрез.
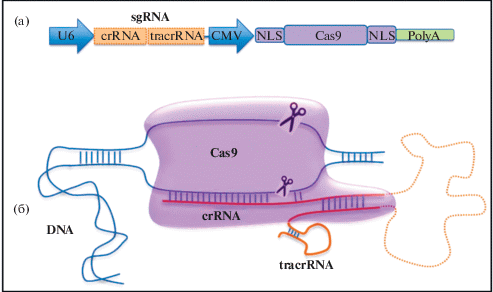
Далее конструкция CRISPR/Cas встраивается в плазмиду/упаковывается в вирусные частицы, и осуществляется трансфекция/инфекция клеток- мишеней. В клетках происходит синтез белка Сas9 и sgРНК. Комплекс sgРНК–Cas9 распознает искомый участок ДНК клетки, и Сas9 создает двухцепочечный разрез (DSB) в ДНК, если в ней содержится локус РАМ (последовательность нуклеотидов 5'-NGG (где N – любой нуклеотид)). (рис. 1б). После чего клетка запускает механизм репарации (зашивания) ДНК с негомологичным соединением концов (NHEJ), при котором часто возникают инсерционные и делеционные мутации (инделы). Если в данном месте происходит вставка или удаление нуклеотидов, не кратных трем, то возникает сдвиг рамки считывания, что часто ведет к нокауту (выключению) гена, или, если делеция кратна трем, происходит удаление одной или нескольких аминокислот [19, 43, 67]. С более низкой вероятностью может происходить гомологически направленная репарация (HDR) и, если в систему внесена донорская ДНК-матрица, то она может быть вставлена (скопирована) в область разрыва, что позволяет осуществить специфическую модификацию гена [19, 67]. К настоящему времени найдены белки CAS, которые, в отличие от Cas9, работают с иными локусами РАМ, что расширяет возможности редактирования.
Наиболее просто CRISPR/Cas9-зависимая модификация генов может быть произведена в легко доступных делящихся клетках и клетках в системе in vitro, поэтому практически сразу начались работы на эмбрионах, стволовых и раковых клетках. В 2013 году появились первые сообщения о редактировании с помощью CRISPR/Cas9 генома стволовых клеток человека, больного муковисцидозом [91]. В 2015 году были отредактированы геномы человеческих эмбрионов для исправления мутации в гене, который вызывает бета-талассемию, болезнь, затрудняющую продуцирование здоровых красных кровяных телец [55]. Технологии CRISPR/Cas9 были успешно применены для исправления патогенной мутации в стволовых клетках, полученных от пациента с дегенерацией фоторецепторов, что открывает возможности для лечения тяжелого пигментного ретинита [6]. К настоящему времени работ с применением CRISPR/Cas9 насчитывается сотни, и их количество растет в геометрической прогрессии. В 2020 году, в результате большой важности внедрения технологии редактирования генов CRISPR/Cas9 для получения фундаментальных знаний и биомедицинских и биотехнологических приложений, Дженнифер Дудне и Эммануэль Шарпантье была присуждена Нобелевская премия по химии. Ниже будут обсуждены технологии CRISPR/Cas для редактирования работы генома в ЦНС и потенциальные терапевтические подходы для лечения заболеваний, связанных с когнитивными нарушениями.
CОЗДАНИЕ ТРАНСГЕННЫХ И НОКАУТНЫХ ЖИВОТНЫХ
Одним из способов изучения функций нейрональных белков in vivo является нокаут/нокдаун (удаление/уменьшение экспрессии) кодирующих их генов. Ранее для этих целей использовались генетически модифицированные животные, для создания которых применялась сложная, трудоемкая и дорогостоящая генетическая инженерия. Кроме того, практически невозможным было создание животных с мультигенными изменениями. Технологии CRISPR/Cas оказались практически идеальными как для создания трансгенных животных через редактирование зародышевых линий [5, 65, 85], так и нокаута/нокдауна генов непосредственно в нейрональных клетках взрослых животных [99].
Cоздание трансгенных организмов с помощью CRISPR редактирования яйцеклеток позволяет достаточно быстро создавать не только трансгенных мышей, но и других животных, в том числе трансгенных крыс, которые, из-за способности к более стабильной длительной работе и меньшему влиянию отвлекающих некогнитивных факторов, широко применяются в качестве моделей в исследованиях поведенческой нейробиологии [28], а также приматов [106]. В 2017 году система CRISPR/Cas9 была применена для создания линий крыс Cre knockin, которые несут гены Cre в целевых генных локусах: доставка gRNAs/Cas9 и донорской ДНК осуществлялась инъекцией в оплодотворенные яйцеклетки, причем получение трансгенных крыс занимало всего несколько недель [65]. У этих животных активность рекомбиназы Cre можно контролировать в конкретном типе клеток или запускать внешними стимулами, такими, как химический сигнал или тепловой шок, что позволяет регулировать экспрессию в определенные временные интервалы. С использованием данной технологии в 2019 были получены три новые трансгенные линии Cre-зависимых крыс с очень высокой эффективностью редактирования генома в мозге – это линии Cre-Cas9, Cas9(D10A) и DAT-iCre, нацеленные на дофаминергические нейроны [5]. Данный подход может быть использован и для нацеливания на другие типы нейронов, в том числе ГАМКергические, что делает эти линии крыс перспективными для нейробиологических исследований. Также технологии CRISPR/Cas были применены для получения приматов с нокаутом генов Prrt2 [124] и Bmal1 [82]. Обезьяны макаки с нокаутом BMAL1 демонстрируют снижение сна и психические расстройства. Генетически модифицированные приматы являются очень важными моделями для фундаментальных и прикладных нейробиологических исследований.
Технологии CRISPR/Cas в настоящее время применяются и для нокаутов/нокдаунов генов в нейронах взрослых млекопитающих. Основными трудностями в данном случае являются обеспечение доставки конструкции CRISPR в мозг, специфичность и эффективность редактирования, а также осуществление одновременных нокаутов нескольких генов. Эти трудности в значительной мере удалось преодолеть через создание вирусных платформ, платформ на основе рибонуклеопротеиновых (RNP) комплексов и наноклубков [13, 20, 95, 99]. Так, в 2015 году была создана платформа, использующая аденовирус (AAV) для доставки и экспрессии CRISPR-spCas9 (spCAS9 – это CAS9, выделенный из Streptococcus pyogenes и наиболее часто применяемый в CRISPR-технологиях), в которой кассеты для экспрессии spCAS9 и sgRNA упаковывались в два отдельных вирусных вектора [99]. Эта система была нацелена на ген Х-хромосомы Mecp2, кодирующий метил CpG-связывающий белок 2 (MeCP2), дисфункция которого может вызывать когнитивные нарушения и аутистические заболевания [33, 69, 74]. Было показано, что нокдаун Mecp2 в культивированных нейронах изменяет морфологию дендритного дерева и плотность шипиков, а нокдаун в мозге взрослых животных in vivo снижает уровень белка MeCP2 более чем на 60%, при этом специфически нарушается контекстная память, без изменения других видов памяти (тестирование в открытом поле, распознавание новых объектов или приподнятый крестообразный лабиринт) [99]. Нацеливание этой системы на гены ДНК-метилтрансфераз (DNMT: Dnmt1, Dnmt3a и Dnmt3b) в мозге взрослой мыши in vivo приводит к нокдауну DNMT генов, и, как в случае с Mecp2, нарушается формирование контекстной памяти без воздействия на другие виды памяти. DNMT катализируют метилирование ДНК, тем самым регулируя экспрессию генов. Dnmt1 и Dnmt3a высоко экспрессируются в мозге взрослых и необходимы для синаптической пластичности и ДП, а Dnmt3b в основном экспрессируется во время развития нервной системы [29]. Таким образом, данные линии крыс являются удобными моделями для изучения когнитивных расстройств и отработки способов их исправления. Описанная выше система оказалась не совсем пригодной в условиях длительных терапий, так как пролонгированная экспрессия Cas9 снижает специфичность. Впоследствии специфичность системы была повышена в 25 раз за счет снижения активности Cas9 4-гидрокситамоксифеном, введенным после модификации целевого гена [20].
Для функционального тестирования синаптических белков в постмитотических нейронах с помощью CRISPR были делетированы кодирующие их множественные гены [40, 104]. В нейронах (культуры срезов гиппокампа) были инактивированы два синаптических белка, субъединицы GluN1 рецептора NMDA и GluA2 – рецептора AMPA, что приводило к полному подавлению синаптической передачи [40]. CRISPR/Cas9 были также успешно применены для функциональной оценки белков постсинаптической плотности в ингибиторных синапсах, в результате была показана важная роль белков InSyn1 и InSyn2 в механизмах синаптического ингибирования [104]. Таким образом, CRISPR/Cas9 оказывается чрезвычайно эффективным для редактирования геномов в нейронах и его использование открывает большие перспективы для понимания молекулярной основы синаптической пластичности. Возможность вводить CRISPR/Cas9 в конкретные клетки позволяет избежать проблемы с летальностью, часто имеющей место при глобальной инактивации генов у животных с нокаутом.
Другим подходом для доставки CRISPR/Cas для редактирования постмитотических нейронов в мозге взрослых мышей являются невирусные системы на основе рибонуклеопротеинов (RNP), состоящих из белка Cas9 и РНК-гида. RNP-комплексы позволяют десятикратно повысить эффективность редактирования нейронов и удобны для потенциальных терапевтических применений, так как не вызывают активации иммунного ответа [95]. Система CRISPR-Gold на основе Cas9 RNP позволяет редактировать гены в нейронах, астроцитах и микроглии [53]. Например, удаление гена метаботропного рецептора глутамата (mGluR5) с помощью CRISPR-Gold эффективно снижает уровни mGluR5 в полосатом теле и спасает мышей с расстройствами аутистического спектра (синдром ломкой Х-хромосомы (FXS)) от патологически повторяющегося поведения. Также разработаны нанокомплексы на основе слияния амфифильного пептида R7L10 с RNP Cas9, которые способны проходить через клеточные мембраны и поддерживать высокие уровни экспрессии более недели [77].
Как известно, сложное поведение животных, в особенности когнитивное, задействует множество нейрональных сетей с вовлечением значительного количества генов, и, соответственно, для их изучения часто требуется возможность одновременного редактирования нескольких генов. Из бактерий Prevotella и Franciscella была выделена нуклеаза Cas12a, которая, в отличие от Cas9, использует более короткую направляющую РНК, что делает CRISPR-Cas12а применимым для одновременного редактирования нескольких генов [98]. Так, используя CRISPR/Cas12a с добавлением стабилизатора третичной структуры РНК, были одновременно отредактированы сразу 20 генов [13]. На основе LbCas12a из бактерии Lachnospiraceae была создана платформа, с помощью которой было отредактировано несколько генов in vivo у Drosophila [80]. Разная степень активности LbCas12a в зависимости от температуры позволила авторам осуществить временное независимое редактирование генов, причем в различных тканях. Таким образом, редактирование генов с использованием Cas12a предоставляет мощную платформу для исследования и управления сложными генетическими программами, лежащими в основе сетевых взаимодействий.
МОДИФИКАЦИЯ ГЕНОВ В МОЗГЕ МЛЕКОПИТАЮЩИХСЯ
Кроме нокаутов генов, для исследований в нервной системе востребованы также подходы, позволяющие исправлять мутации в целевых генах или заменять гены новыми. Стратегии на основе CRISPR/Cas, применяемые для этих целей, суммированы на рис. 2. В основе стратегий vSLENDR и HMEJ, адаптированных для постмитотических нейронов, лежит вставка донорских последовательностей через гомологически направленную репарацию HDR. Через систему vSLENDR можно вставлять в гены как небольшие последовательности, например, кодирующие метку эпитопа (часть макромолекулы антигена, которая распознается известным антителом), или даже длинные – кодирующие флуоресцентный белок mEGFP [71, 76]. Эпитопная метка позволяет проследить локализацию и динамику белка, кодируемого данным геном, а через флуоресцентный белок возможно визуализировать эндогенные белки в реальном времени in vivo. Вирусная доставка донорской матрицы vSLENDR в мозг позволила успешно осуществить редактирование генов CaMKIIα, ERK2 и β актина в зрелых нейронах, а также в митотических нейронах и глии с эффективностью около 30%, что показывает полезность этого метода для фундаментальных нейробиологических исследований [76].
Рис. 2.
Стратегии CRISPR/Cas для генной коррекции. vSLENDR, HMEJ, HITI, ORANGE, HiUGE – стратегии включения донорских последовательностей на основе CRISPR/Cas.
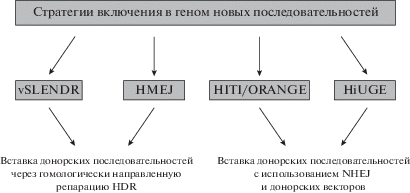
В основе стратегий HITI и HiUGE лежит вставка донорских последовательностей через репарацию NHEJ, и они демонстрируют более высокую частоту редактирования и точность репарации чем HDR [88]. Стратегия HITI была успешно применена для эндогенного мечения белков в постмитотических нейронах [115], а универсальные доноры HiUGE, использующие векторы AAV с инсерционными последовательностями, кодирующими различные функциональные модификации, позволяют быстро модифицировать белки in vitro/in vivo для их маркировки с целью динамической визуализации и осуществлять модификации белков, специфичных для нейронов [30]. На основе HITI были разработаны технологии ORANGE для мультигенного редактирования генома нейронов in vivo с целевой геномной интеграцией меток эпитопа, что позволяет визуализировать синтезированные белки и количественно оценить экспрессию, локализацию и динамику практически любого белка с разрешением на наноуровне [108]. Путем объединения ORANGE с микроскопией сверхвысокого разрешения живых клеток, удалось выявить динамическую организацию рецепторов нейротрансмиттеров и белков синаптического каркаса.
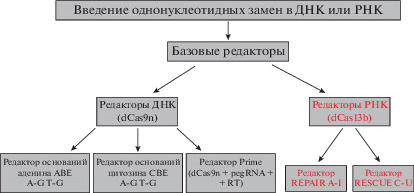
Одним из самых последних достижений в области редактирования генома является “базовое редактирование” – введение однонуклеотидных замен в ДНК или мРНК в живых клетках без использования двухцепочечных разрывов, что устраняет недостатки HDR, связанные с побочными мутациями. Учитывая тот факт, что около половины известных генетических заболеваний вызываются однонулеотидными полиморфизмами, эти технологии имеет большой потенциал для лечения болезней, в том числе заболеваний, связанных с ментальными нарушениями [8, 39, 84]. Ожидается что исправление точечных мутаций станет одним из основных направлений медицины будущего. Базовые редакторы основаны на слияниях dCas9 (каталитически неактивная Cas9) – с ферментами, которые могут преобразовывать один нуклеотид на другой [39, 84, 88]. К базовым редакторам ДНК относятся редакторы оснований аденина (ABE), цитозина (CBE) и редактор Prime (Рис. 3). Наиболее эффективным и многофункциональным является Prime. Он состоит из dCas, слитой с ферментом обратной транскриптазы (RT), и управляющей РНК редактирования (pegRNA), способной идентифицировать целевой сайт и предоставлять новую генетическую информацию для замены нуклеотидов в ДНК. С помощью Prime не только изменяют основания, но и осуществляют целевые вставки и делеции.
К базовым редакторам РНК относятся REPAIR и RESCUE, с помощью которых возможна замена аденина на инозин, а цитозина на урацил соответственно, что позволяет исправлять мутации на уровне матричной РНК (рис. 3). Современные редакторы могут достигать эффективности редактирования, близкой к 100% в культивируемых клетках млекопитающих и 70% в нейронах взрослых мышей in vivo [39].
При изучении функции генов часто возникает необходимость в системах, способных редактировать гены в строгих временных интервалах. С этой целью CRISPR/Cas9 был объединен с системами, которые можно регулировать генетически, оптически или с помощью небольших молекул [24, 48]. В первых индуцибельных системах CRISPR регуляция осуществлялась тетрациклином (Tet) или доксициклином (Dox) через Tet или Dox-зависимые промоторы соответственно [22, 24]. Dox-система была успешно применена для временной регуляции редактирования генома в мозге, причем редактирование инициировалось через корм, содержащий доксициклин, в течении всего одного дня [22]. Кроме того, на основе эндонуклеазы SaCas9 из Staphylococcus aureus и вектора AAV были разработаны Cre/Dox-индуцируемые системы CRISP, с помощью которых было осуществлено редактирование генома в нейронах мышей и человека in vitro и in vivo в нейронах мозга мыши [50]. Причем временная индукция редактирования осуществлялась с помощью Dox, а пространственная с помощью Cre-рекомбиназы. Однако, Tet- и Cre-индуцируемые системы работают относительно медленно, так как зависят от транскрипции. В связи с чем были разработаны системы, регулируемые посттрансляционно, через лиганд-связывающий домен рецептора эстрогена человека (ETR), слитый с Cas9 и запускаемые лигандом ETR 4-гидрокситамоксифеном (4-HT) [20, 58], а также система 4-HT-индуцибельного фермента iCas с носителем ETR2, которая способна осуществлять двунаправленное редактирование путем введения/удаления 4-HT [59].
В последние годы успешно разрабатываются системы редактирования геномов, индуцируемые оптически, что полезно для ограничения редактирования генов или регуляции транскрипции по времени и месту. Оптический контроль функций Cas9 достигается с помощью фотоуправляемых неприродных аминокислот или с помощью индуцированных светом взаимодействий белков. В частности, были созданы фотосопереключаемые белки ps-Cas9, регулируемые освещением, которые успешно опосредуют активацию транскрипции одного гена и редактирование другого в одних и тех же клетках, что позволяет обеспечивать общий метод управления множеством функций [123].
РЕГУЛЯЦИЯ ЭКСПРЕССИИ ГЕНОВ ПРИ ПОМОЩИ CRISPR/CAS
Значительный прорыв в использовании CRISPR/ CAS для изучения функции генов был достигнут благодаря созданию каталитически неактивного dCas9 [81]. Применение CRISPR-dCAS позволяет обратимо активировать или репрессировать любой ген по замыслу экспериментатора. Это особенно важно для изучения функционирования нервной системы, в частности, молекулярных механизмов когнитивных процессов и улучшения их работы в случае нарушения.
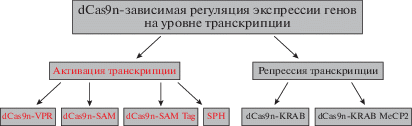
Регуляция экспрессии генов с помощью dCas9 на уровне транскрипции. В основе этой технологии регуляции экспрессии генов лежит неспособность каталитически неактивного dCas9 делать разрез ДНК, но способного связывать репрессоры или активаторы транскрипции и доставлять их к регуляторным элементам генов-мишеней. Точность доставки CRISPR-dCAS обеспечивает молекула РНК-гида. В первых системах для активации ген-специфической транскрипции (CRISPRa) использовали слияние dCas9 с активаторным доменом белка герпеса (VP64), или трансактивирующей субъединицей NF-kB p65 (p65), а для репрессии (CRISPRi) – бокс-домен (Krüppel associated box) of Kox1 (KRAB), которые направлялись на промоторные или энхансерные последовательности выбранных генов посредством разработанных РНК-гидов [32]. В дальнейшем поколение активаторов dCas9-VP64 и dCas9-P65 было улучшено (эффективность экспрессии была повышена на несколько порядков), системы получили название SunTag, SAM и VPR [88]. Системы, регулирующие экспрессию генов представлены на рис. 4. Кроме того, элементы систем SunTag и SAM были объединены в новом активаторе транскрипции SPH, что позволило с высокой эффективностью регулировать экспрессию нескольких целевых генов одновременно в мозге in vivo, используя трансгенных мышей, несущих Cre‑индуцируемую систему SPH, причем активность рекомбиназы Cre регулировалась пространственно-временным образом [121]. Таким образом, учитывая наличие полногеномных библиотек sgRNA, CRISPRa мыши SPH могут широко использоваться в исследованиях механизмов функционирования ЦНС. Система dCas9-VP64 была оптимизирована для мультигенной активации в нейронах in vivo/in vitro через доставку в лентивирусных (LV) частицах с раздельной экспрессией dCas9-VP64 и sgRNA под контролем нейрон-специфического промотора синапсина [89]. Эта система, нацеленная на уникальные промоторы нейротрофического фактора Bdnf, продемонстрировала способность к специфической индукции различных транскриптов BDNF in vivo в различных структурах мозга.
Рис. 4.
dCas9 – зависимая регуляция экспрессии генов на уровне транскрипции. Системы dCas9-VPR, dCas9-SAM, dCas9-SanTag и SPH применяются для активации транскрипции, системы dCas9-KRAB и dCas9-KRAB MeCP2 применяются для репрессии транскрипции.
Для подавления экспрессии генов в ЦНС на уровне транскрипции in vivo на основе dCas9 было разработано несколько репрессоров [116]. Наиболее высокоэффективным и специфичным оказался dCas9-KRAB-MeCP2, способный подавлять кодирующие и некодирующие гены и обеспечивать одновременную репрессию нескольких выбранных генов. Высокую эффективность этого репрессора авторы объясняют совместной работой двух доменов. Домен KRAB репрессирует транскрипцию посредством взаимодействия с KAP1, обеспечивающих каркас для набора корепрессоров, включая гетерохроматиновый белок 1, гистоновые деацетилазы и SETDB1. Домен репрессии транскрипции MeCP2 связывается с другим набором регуляторов транскрипции, включая ДНК-метилтрансферазу DNMT1 и корепрессорный комплекс SIN3A-гистондеацетилазы.
Параллельно была разработана система CRISPRi c использованием репрессора dCas9-KRAB и LV-вектора, способная с эффективностью порядка 90% осуществлять инактивацию генов, необходимых для высвобождения нейромедиаторов: синаптотагмина 1 (Syt1), связанного с везикулами мембранного белка 1 (Vamp1), синтаксина 1А (Stx1a) и Snap25 [120]. Далее эта система была использована для подавления экспрессии синаптотагмина (Syt1) в глутаматергических (CaMKIIα-dCas9-KRAB) и ГАМКергических (VGAT-dCas9-KRAB) нейронах гиппокампа мышей in vivo. Анализ показал, что экспрессия Syt1 полностью устраняется клеточно-специфическим образом, при этом практически полностью исчезают возбуждающие постсинаптические потенциалы в глутаматергических нейронах и тормозные – в ГАМКергических нейронах [120]. Ингибирование функционирования глутаматергических нейронов посредством CRISPRi вызывает ухудшение пространственной памяти по сравнению с животными, у которых ингибировали ГАМКергические нейроны, однако, у них лучше формировалась долговременная память, индуцируемая страхом [120]. Таким образом, избирательное нацеливание на Syt1 в данных типах нейронов позволяет изменять соотношение торможения и возбуждения в зубчатой извилине гиппокампа и влиять на формирование долговременной памяти. CRISPR-dCas9-KRAB был применен также для репрессии гена риска психических заболеваний анкирина 3 (ANK3). В результате, были раскрыты ранее неизвестные базовые клеточные и молекулярные механизмы, лежащие в основе специфического для мозга нарушения экспрессии ANK3, которые могут быть связаны с психическими заболеваниями [31].
Применение CRISPR-Cas13d и CRISPR-CasRx для регуляции экспрессии генов через манипуляции с матричной РНК. Возможность регуляции экспрессии генов через манипулирование с РНК очень перспективна для исследований, связанных с транскриптомом и терапевтическими разработками, направленными на борьбу с когнитивными нарушениями так как она позволяет избежать необратимые эффекты, вызванные непредвиденным мутагенезом в случае манипуляций с геномной ДНК. И здесь будущее в значительной мере принадлежит технологиям CRISPR/Cas [7, 49]. Для возможности манипуляций с РНК в последние годы разработаны CRISPR-системы, нацеленные на РНК-транскрипты, в том числе содержащие патогенные мутации, что стало возможным благодаря открытию CRISPR-Cas, способных идентифицировать и разрезать не ДНК, а РНК. Так, были найдены и усовершенствованы бактериальные белки Cas13d, CasRx и NmeCas9. CasRx отличается высокой компактностью и может быть упакован в аденовирус, что облегчает его доставку. CRISPR-NmeCas9 способен к распознаванию как ДНК, так и РНК-мишеней и расщеплению независимо от PAM, что делает перспективным его использование как для модификаций как ДНК, так и РНК [86]. Каталитически неактивные dCasRx, нацеленные на цис-элементы пре-мРНК, были успешно применены для регулирования соотношения изоформ белка тау в нейронной модели лобно-височной деменции [49], а нацеленные на подавление мРНК РНК-связывающего белка Ptbp1 позволили индуцировать нейроны с дофаминергическими свойствами в стриатуме (модель болезни Паркинсона) и тем самым улучшить двигательные дефекты у мышей [122]. Еще одним подходом к регуляции экспрессии генов на уровне РНК является манипуляции с метилированием РНК. Была создана фотоактивируемая CRISPR-Cas13-зависимая система редактирования метиладенозина (m6A), важной формы метилирования в мРНК, названная PAMECR [119]. Для этого светоиндуцируемые гетеродимеризующиеся белки CIBN и CRY2PHR были слиты с каталитически неактивными dCas13 и m6A соответственно, что обеспечило пространственно-временное управление редактированием m6A в ответ на синий свет одновременно в нескольких различных мРНК. Система позволяет осуществлять удаленное управление редактированием РНК и является многообещающей оптогенетической платформой для применения в эпитранскриптомной инженерии, визуализации и в будущих терапевтических разработках [119].
РЕГУЛЯЦИИ ЭКСПРЕССИИ ГЕНОВ ЧЕРЕЗ ЭПИГЕНЕТИЧЕСКИЕ МАНИПУЛЯЦИИ
Исследования последних двух десятилетий выявили многочисленные нарушения эпигенетической регуляции экспрессии генов, связанные с когнитивными дисфункциями. Эпигенетическая регуляция экспрессии генов осуществляется главным образом через метилирование ДНК и/или посттранскрипционные модификации гистонов, а также посредством изменения биогенеза микроРНК. Эпигенетические процессы широко вовлекаются в регуляцию онтогенеза, созревание нейронов мозга и различные адаптационные процессы, в том числе формирование долговременной памяти [1, 3, 21]. Через воздействие на эпигенетические надстройки возможно влиять на экспрессию генов и потенциально использовать в терапии различных заболеваний, что до недавнего времени являлось достаточно сложной задачей, главным образом связанной с трудностями обеспечения точечной регуляции определенных генов. В связи с развитием технологий CRISPR/ Cas начался бурный прогресс в этой области [61, 117]. Вышеописанный каталитически неактивный белок dCas9 был наделен способностью доставлять на определенный участок геномной ДНК белки регулирующие эпигенетические модификации ДНК и гистонов, что позволило регулировать экспрессию генов эпигенетическим путем.
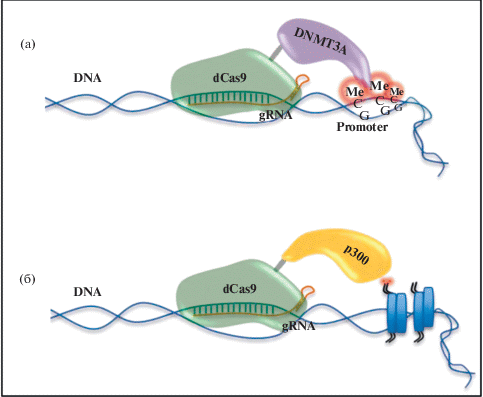
Применение CRISPR-dCas9 для изменения метилирования ДНК. Метилирование ДНК играет важную роль в регуляции экспрессии генов, в том числе в развитии нервной системы и регуляции адаптивных процессов, включающих реакцию на стресс и когнитивные функции [21]. Метилирование ДНК – это обратимая модификация ДНК, происходящая в результате присоединения метильной группы к цитозину в составе динуклеотида CpG. Процесс регулируется ДНК метилтрансферазами и деметилазами. Метилтрансферазы осуществляют метилирование ДНК, тем самым блокируя экспрессию генов. Деметилазы удаляют метильные группы, чем активируют экспрессию. Метилирование генов в промоторных областях позволяет моделировать сложные процессы в нейрональных сетях. Нарушение метилирования ДНК часто приводит к когнитивным нарушениям и различным патологиям работы ЦНС [21, 26].
Относительно недавно было показано, что на основе CRISPR-Cas технологий можно целенаправленно регулировать метилирование генов-мишеней. При этом dCas9, лишенный нуклеазной активности, доставляет на определенный участок геномной ДНК ферменты, модифицирующие метилирование ДНК, а молекула РНК-гида обеспечивает точность доставки к гену-мишени [45, 54, 62]. Таким образом, на уровне регулирования метилирования можно включать и выключать определенные гены для изучения их функций, либо выключать поврежденные гены с целью терапии заболеваний. Схема эпигенетической регуляции генома с помощью системы CRISPR-dCas9 на уровне метилирования ДНК представлена на рис. 5а.
Рис. 5.
Эпигенетическая регуляция генома с помощью системы CRISPR-dCas. Каталитически неактивный белок dCas9 обладает способностью связываться с ДНК, а также с доменами для целевого редактирования эпигенома. Точность доставки к гену-мишени обеспечивает молекула РНК-гида (gRNA = crRNA + tracrRNA). (а) dCas9 слитый с ДНК-метилтрансферазами (например, DNMT3А) способен влиять на экспрессию генов через метилирование промоторов. (б) dCas9 слитый с ацетилтрансферазами, в частности р300, способен ацетилировать гистоны, тем самым активируя экспрессию генов.
Для целевого редактирования метилирования ДНК в интроне 1 была разработана система CRISPR-dCas9-DMNT3A, основанная на слиянии dCas9 с каталитическим доменом ДНК-метилтрансферазы 3A (DNMT3A) [45]. В результате было индуцировано гиперметилирование ДНК в интроне 1 SNCA дофаминергических нейронов, полученных из индуцированных плюрипотентных стволовых клеток (ИПСК) пациента с БП, нарушенное при БП, что восстанавливало жизнеспособность клеток. В 2016 году эпигенетическое регулирование метилирования ДНК удалось осуществить у млекопитающих через слитие dCas9 с деметилазой TET1. Было показано, что LV система dCas9-TET1/sgRNA нацеленная на деметилирование промотора нейротрофического фактора BDNF в нейронах, увеличивает экспрессию BDNF в 6 раз [62].
Предприняты также попытки редактирования, нарушенного метилирования ДНК, для потенциального лечения распространенной генетической формы умственной отсталости у мужчин – синдрома ломкой Х-хромосомы (FXS). Это заболевание вызывается подавлением гена FMR1, связанным с многократным удвоением CGG островков (>200) в области 5 'UTR FMR1, сопровождаемым гиперметилированием [78]. Направленное деметилирование повторов CGG в патологическом локусе FMR1 с помощью dCas9-Tet1/sgRNA увеличивало ацетилирование гистонов H3K27 и триметилирование H3K4, уменьшало триметилирование H3K9 на промоторе FMR1, переключало статус гетерохроматина промотора FMR1 в активное состояние и восстанавливало экспрессию FMR1 в нейронах FXS, индуцированных из плюрипотентных стволовых клеток (ИПСК) пациентов FXS [63]. Полученные нейроны восстанавливали фенотип дикого типа и полноценные электрофизиологические характеристики. Экспрессия FMR1 в отредактированных нейронах длительно сохранялась in vivo после внедрения в мозг мыши. Наконец, деметилирование повторов CGG в постмитотических нейронах FXS также реактивировало FMR1 и обращало вспять спонтанную гиперактивность. Таким образом, авторы полагают, что деметилирование CGG через dCas9-Tet1/sgRNA может служить потенциальной стратегией для терапии FXS [63].
Применение CRISPR-dCas9 для эпигенетической модификации гистонов. Гистоны — это небольшие белки, необходимые для сборки и упаковки ДНК в компактные структуры нуклеосомы. Кроме того, гистоны играют важную роль в регуляции экспрессии генов через ремоделирование хроматина. В зависимости от эпигенетических модификаций гистонов, в частности, ацетилирования и метилирования, изменяется их сродство к ДНК, чем контролируется доступ регуляторных белков и РНК-полимераз к сайтам связывания ДНК и, соответственно, регулируется экспрессия генов. Показано, что ацетилирование и метилирование гистонов играют важную роль в формировании ДП, а их нарушение приводит к значительным когнитивным дисфункциям при различных заболеваниях [1]. Повышенный интерес к ацетилированию и метилированию вызван потенциальной возможностью через эти процессы влиять на формирование долговременной памяти в случае ее нарушения. Степень ацетилирования гистонов регулируется гистон-ацетилтрансферазами (HAT) и гистон-деацетилазами (HDAC). Высокая степень ацетилирования приводит к активации транскрипции, а деацетилирование вызывает ингибирование. Метилирование гистонов является более сложным процессом, контролируется гистон-метилазами и деметилазами и приводит как к индукции, так и репрессии транскрипции в зависимости от степени и сайта метилирования [1].
В последние годы на основе CRISPR-Cas9 были созданы технологии, позволяющие изменять гистоновые модификации, тем самым обратимо регулировать экспрессию целевых генов, а также изучать механизмы их регуляции. Так, для индукции ацетилирования гистонов была создана программируемая CRISPR/dCas9-зависимая ацетилтрансфераза (dCas9-p300), способная катализировать ацетилирование гистона H3 по лизину 27 в его сайтах-мишенях, для чего каталитически неактивная dCas9 была слита с каталитическим ядром ацетилтрансферазы человека p300 [35]. Точность доставки обеспечивает молекула РНК-гида. Было показано, что dCas9-p300 индуцирует устойчивую и высокоспецифичную транскрипционную активацию генов-мишеней, как от промоторов, так и энхансеров, тем самым позволяя не только активировать экспрессию генов, но и исследовать взаимосвязь между эпигеномом и контролем транскрипции.
На основе dCas9 была также создана конструкция (dCas9-HDAC8), позволяющая ингибировать экспрессию генов за счет деацетилирования гистонов путем слития с dCas9 c гистондеацетилазой HDAC8 [16]. dCas9-p300 и dCas9-HDAC8 были использованы для активации или блокирования ацетилирования гистонов локально на энхансерах нейронального гена Fos с целью изучения его регуляции [16]. Ген Fos относится к немедленным ранним генам, он кодирует один из основных транскрипционных факторов, участвующих в различных адаптивных процессах ЦНС, в том числе в когнитивных [1].
Несколько ранее Bohnsack с коллегами [9] показали, что усиление ацетилирования гистонов в промоторе гена Gabra1, кодирующего рецептор γ-аминомасляной кислоты (ГАМК), с использованием dCas9-P300 предотвращает вызванное этанолом снижение его экспрессии в культивируемых корковых нейронах. Рецепторы ГАМК опосредуют большую часть тормозной нейротрансмиссии в мозге взрослого человека. Таким образом, полагают, что данная технология может быть полезна для разработки терапевтических средств лечения алкогольной зависимости, эпилепсии и стресса, при которых экспрессия Gabra1 нарушена [9]. CRISPR технологии редактирования эпигенома были использованы для пространственного и временного сетевого контроля экспрессии генов. Так, нацеливание комплекса dCas9-p300 на дистальные нерегулирующие области генома перепрограммирует состояние хроматина этих областей в элементы, подобные энхансерам [51]. В частности, контролируя пространственное расстояние этих индуцированных энхансеров до промотора, можно жестко регулировать амплитуду экспрессии гена.
На основе dCas9 была также создана конструкция CRISPR/dCas9 SunTag-JARID1A, позволяющая ингибировать экспрессию генов за счет деметилирования гистона H3K4me3 путем слития dCas9 с каталитическим доменом деметилазы JARID1A. Конструкция успешно была применена для ингибирования экспрессии α-синуклеина (SNCA), повышение которого связано с болезнью Паркинсона [34]. Таким образом, манипулирование эпигеномом при помощи CRISPR/dCas9 становится многообещающей стратегией для персонализированной медицины, в том числе для терапии нейродегенеративных патологий [92].
Применение CRISPR-dCas9 для манипуляций с микроРНК. МикроРНК – это высоко консервативные, небольшие молекулы РНК, каждая из которых может регулировать экспрессию сотен генов через репрессию или деградацию их матричных РНК. Таким образом, нарушение регуляции даже одной микроРНК может оказывать значительный системный эффект. Наиболее высок и разнообразен уровень экспрессии микроРНК в ЦНС. В последние годы было показано, что микроРНК активно вовлекаются в формирование ДП, а нарушение их биогенеза может являться причиной когнитивных дисфункций при шизофрении, расстройствах аутистического спектра и нейродегенеративных патологиях [2, 57]. Потенциально, терапия этих заболеваний возможна через воздействие на метаболизм микроРНК [4]. Путем репрессии посредством CRISPRi или активации через CRISPRa возможен временный или постоянный контроль экспрессии miRNA. Регуляция транскрипции miRNA также может быть обеспечена dCas9, слитой с эпигенетическими модификаторами ДНК или гистонов, а также CasRx-опосредованном нокдауне РНК о которых сообщалось выше.
Так как микроРНК служат важнейшими биомаркерами множества заболеваний, они востребованы для клинической диагностики. Недавно был создан первый микрожидкостный электрохимический биосенсор с использованием CRISPR/Cas13a для локального обнаружения микроРНК, в котором количественная оценка микроРНК осуществляется без их амплификации. Биосенсор на основе CRISPR высокочувствителен и позволяет регистрировать микроРНК в объеме менее 0.6 мкл [11]. И более того, данный биосенсор был усовершенствован для параллельного обнаружения нескольких микроРНК [12].
На основе различия в специфичности экспрессии микроРНК в различных типах клеток, были разработаны системы CRISPR/Cas9, позволяющие проводить редактирование генома в конкретной клеточной популяции, не затрагивая другие органы и ткани [36, 38]. Так, Хиросава и его коллеги разработали miRNA-чувствительную систему CRISPR/Cas9 с использованием переключателей основе miR-21 или miR-302, которые посттранскрипционно ослабляли активность Cas9 только в клетках-мишенях и могли по-разному контролировать редактирование генома через эндогенные активности miRNA в гетерогенной популяции клеток. Эта система переключения miR-Cas9 обеспечивает основу для селективного редактирования генома на основе информации о внутриклеточных miRNA.
ПЕРСПЕКТИВЫ ТЕРАПИИ ЗАБОЛЕВАНИЙ, СВЯЗАННЫХ С КОГНИТИВНЫМИ НАРУШЕНИЯМИ
Исследования последних 2-х десятилетий выявили многочисленные нарушения регуляции экспрессии генов, связанные с когнитивными дисфункциями при психических и нейродегенеративных заболеваниях. Большие надежды на возможность терапии этих патологий связаны с технологиями CRISPR/Cas. В основе многих когнитивных нарушений лежит нейродегенерация. В первую очередь это относится к болезням Альцгеймера и Паркинсона, старческой деменции и инсультам. В 2020 году вышли две интересные статьи, посвященные восполнению потери нейронов и восстановлению синаптических связей с использованием CRISPR/Cas [75, 122]. Так, для пополнения нейронов, в нейроны (посредством CRISPR-CasRx нацеленной на РНК) были преобразованы глиальные клетки, а именно, глия Мюллера в ганглии сетчатки – клетки (RGC) [122]. Преобразование было осуществлено через подавление РНК-связывающего белка Ptbp1. В результате были облегчены симптомы заболевания, связанные с потерей этих нейронов. Кроме того, применение CRISPR-CasRx позволило индуцировать нейроны с дофаминергическими свойствами в стриатуме и тем самым улучшить двигательные дефекты у мышей (модель болезни Паркинсона). Авторы полагают, что преобразование глии в нейрон с помощью CasRx-опосредованного нокдауна Ptbp1 представляет собой многообещающий генетический подход in vivo для лечения различных заболеваний, вызванных потерей нейронов. Кроме того, при помощи CRISPR/dCas9-KRAB-зависимого эпигенетического подавления экспрессии белка PTEN, негативного регулятора сигнального пути PI3K/AKT/mTOR, участвующего в процессах регенерации, успешно проведено стимулирование регенерации аксонов и функциональное восстановления связей в ЦНС после травмы [75]. Подавление транскрипции PTEN посредством CRISPR/dCas9-KRAB было осуществлено в линиях клеток человека и в нервных клетках, полученных из индуцированных плюрипотентных стволовых клеток (ИПСК) человека, через индукцию метилирования и деацетилирования гистона H3 на промоторе PTEN.
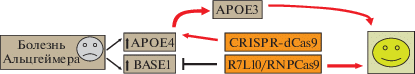
Болезнь Альцгеймера. Болезнь Альцгеймера является самым сложным нейродегенеративным заболеванием и характеризуется многофакторной патологией, что определяет незначительные успехи в лечении. Заболевание быстро прогрессирует с возрастом, приводит к значительным когнитивным нарушениям и завершается летальным исходом. Самым большим генетическим фактором риска спорадической болезни Альцгеймера (sAD) считается повышенный вариант аполипопротеина E4 (APOE4). Чтобы понять, как APOE4 влияет на клетки головного мозга человека, участвующие в патогенезе AD, были применены технологии CRISPR/Cas9 и клетки ИПСК [56]. В результате выявлены сотни дифференциально экспрессирующихся генов, при этом клетки, происходящие из APOE4 ИПСК, воспроизводили многие фенотипы, связанные с sAD. А именно, нейроны демонстрировали повышенное количество синапсов и повышенную секрецию Aβ, в астроцитах нарушалось поглощение Aβ и накоплялся холестерин, а в микроглиеподобных клетках нарушался иммунный ответ. Причем, преобразование APOE4 в APOE3 в клетках мозга, полученных из ИПСК от пациентов с болезнью Альцгеймера, было достаточным для ослабления множества патологий, связанных с этой патологией [56]. Технологии CRISPR/Cas9 для подавления экспрессии генов, ассоциирующихся с болезнью Альцгеймера обобщены на рис. 6. Кроме того, удалось снизить секрецию Aβ, уменьшить содержание бляшек и улучшить долговременную память у трансгенных мышей 5XFAD (модель AD) через введение в CA3 область гиппокампа нанокомплексов на основе слияния амфифильного пептида R7L10 c RNP Cas9, направленных на β-протеазу 1 (Base1), индуцирующую образование Aβ [77]. При болезни Альцгеймера наблюдается также нарушение аутофагии (процесс, при котором внутренние компоненты клетки доставляются внутрь ее лизосом или вакуолей и подвергаются в них деградации). Одной из причин нарушения аутофагии при AD является мутация пресенилина 1 (PS1), или снижение PS1 в человеческом мозге с возрастом. Посредством CRISPR/Cas9 из клеток ИПСК человека были получены нервные стволовые клетки (NSC) с дефицитом PS1, в которых наблюдалось снижение аутофагии. Было показано, что что дефицит PS1 вызывает подавление аутофагии в человеческих NSC через ингибирование регуляторного каскада ERK/CREB [18].
Для лучшего понимания молекулярного механизма продукции Aβ, разработан метод генетического скрининга на основе системы CRISPR/Cas9, что позволило идентифицировать новый негативный регулятор продукции Aβ кальций и интегрин-связывающий белок 1 (CIB1) [17]. Более того, обнаружено, что уровень мРНК CIB1 снижен на ранней стадии БА. Таким образом, CIB1 может служить маркером для диагностики ранней стадии заболевания.
Рис. 6.
Использование технологий CRISPR/Cas9 для подавления экспрессии генов, ассоциирующихся с болезнью Альцгеймера. APOE4, Base1 – гены, повышенная экспрессия которых наблюдается при болезни Альцгеймера. R7L10/RNP CAS9 – технология CRISPR/Cas9 основанная на слиянии амфифильного пептида R7L10 c RNP CAS9.
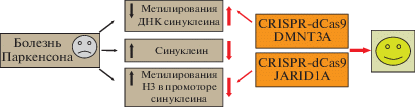
Болезнь Паркинсона относится к нейродегенеративным заболеваниям, которые прогрессируют с возрастом. В основе заболевания лежит избирательная потеря дофаминергических нейронов. Патологическим признаком БП являются повышенные уровни α-синуклеина (SNCA) и его агрегация в тельцах Леви [111]. Альфа синуклеин, кодирующийся геном SNCA, находится в пресинапсе и участвует в высвобождении медиатора, а также модулирует процессы репарации ДНК. Физиологические уровни SNCA необходимы для поддержания функции нейронов. В последние годы было показано, что ключевую роль в болезни Паркинсона играет эпигенетическая дерегуляция α-синуклеина. Причем в мозге БП наблюдается как изменение метилирования ДНК в интроне 1 SNCA [45], так и нарушение метилирования гистона H3 в позиции K4me3 на промоторе SNCA [34], что влияет на транскрипцию SNCA. Через систему CRISPR-dCas9-DMNT3A было индуцировано гиперметилирование ДНК в интроне 1 SNCA дофаминергических нейронов, полученных из клеток ИПСК пациента с БП, что подавило экспрессию SNCA и восстановило жизнеспособность клеток [45]. Технологии CRISPR/Cas9 для подавления экспрессии генов, ассоциирующихся с болезнью Паркинсона обобщены на рис. 7. С другой стороны, в посмертном головном мозге пациентов с БП на промоторе SNCA черной субстанции наблюдается значительное повышение содержание гистона H3K4me3 – эта эпигенетическая модификация приводит к индукции экспрессии генов и, соответственно, увеличивает содержание SNCA [34]. Для снижения экспрессии SNCA была разработана система деметилирования гистона H3K4me3 на основе CRISPR/dCas9, в которой каталитический домен деметилазы JARID1A был направлен на промотор SNCA. Применение CRISPR/dCas9 SunTag-JARID1A значительно снижало содержание H3K4me3 на промоторе SNCA и одновременно снижало уровень α-синуклеина как в линии нейрональных клеток SH-SY5Y, так и в идиопатических дофаминергических нейронах, происходящих от ИПСК БП. Таким образом, эти два эпигенетических подхода с применением CRISPR/dCas9, направленные на модификации ДНК и гистонов и снижение синтеза SNCA, обладают значительным потенциалом для терапии БП.
Рис. 7.
Использование технологий CRISPR/Cas9 для регуляции экспрессии генов, ассоциирующихся с болезнью Паркинсона. При болезни Паркинсона наблюдается изменение метилирования ДНК в гене SNCA и нарушение метилирования гистона H3 на промоторе SNCA. Через манипуляции с метилированием посредством CRISPR-технологии возможно значительное снижение экспрессии SNCA и замедление развития заболевания.
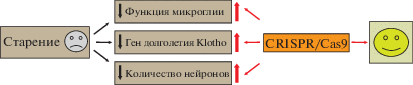
Старение. Хорошо известно, что с возрастом часто происходит ухудшение способностей к обучению, нарушается концентрации внимания, память и некоторые другие психоневрологические характеристики. Важную роль в метаболизме мозга играет микроглия, которая обеспечивает фагоцитарную очистку белковых агрегатов и клеточного мусора. Эта функция ухудшается при старении и нейродегенеративных заболеваниях. Скрининги нокаутов CRISPR/Cas9 идентифицировали негативный регулятор фагоцитоза – рецептор В-клеток CD22, который активировался в стареющей микроглии [79]. Авторы показали, что ингибирование CD22 in vivo способствует удалению остатков миелина, олигомеров β-амилоида и фибрилл α-синуклеина. Долгосрочная блокада функции CD22 антителами перепрограммирует микроглию в исходное состояние и улучшает когнитивные функции у старых мышей. Технологии CRISPR/Cas9 для подавления экспрессии генов, ассоциирующихся со старением обобщены на рис. 8. Полученные данные важны для стратегии восстановления гомеостаза в стареющем мозге. Кроме того, комплекс CRISPR-dCas9 успешно использовали для улучшения когнитивных способностей при старении через транскрипционную активацию гена долголетия Klotho, количество которого снижается с возрастом [15, 118]. Белок Klotho способствует выживанию нейронов и ремиелинизации демиелинизированных аксонов [15, 113]. Сверхэкспрессия Klotho увеличивает продолжительность жизни у мышей примерно на треть, а активация гена Klotho может потенциально облегчить симптомы и/или предотвратить прогрессирование когнитивных нарушений, связанных с нормальным старением и нейродегенеративными заболеваниями, такими как болезнь Альцгеймера и рассеянный склероз, а также невропатологиями, особенно в пожилых, подверженных стрессу группах населения [25, 68, 109, 118]. Таким образом, повышение уровня Klotho посредством CRISPR-dCas9 имеет большой терапевтический потенциал для улучшения когнитивных функций. Важную роль в процессах старения играют теломеры и теломеразы [10]. Укорочение теломер связано с клеточным старением. Благодаря использованию CRISPR/Cas для модификации теломер и теломераз, а также генов, которые на них влияют, в последние годы был сделан огромный шаг вперед для перспектив использования этих молекул в качестве важнейших мишеней для увеличения продолжительности и качества жизни [10]. С целью моделирования старения, в клеточной линии нейробластомы SH-SY5Y теломеры мгновенно удаляли при помощи системы CRISPR-Cas9, нацеленных на повторы теломер, что приводило к дисфункции митохондрий и снижению жизнеспособности клеток. Кроме того, наблюдались изменения в уровнях белков, ассоциированных с болезнью Паркинсона, а экспрессия α-синуклеина на фоне удаления теломер приводила к усилению агрегации белка, что свидетельствует о положительном прямом взаимодействии между старением и патогенезом БП [47].
Рис. 8.
Использование технологий CRISPR/Cas9 для регуляции экспрессии генов, ассоциирующихся со старением. С возрастом наблюдается снижение функций микроглии, понижается экспрессия гена долголетия Klotho и снижается количество нейронов. С помощью CRISPR-технологий возможно замедление этих процессов.
Болезнь Хантингтона [HD] является моногенным нейродегенеративным заболеванием, в настоящее время неизлечимым. Заболевание вызвано аномальным повторением тринуклеотидного повтора CAG в экзоне 1 гена хантингтина [HTT], что приводит к продукции мутантного белка, который образует включения и избирательно разрушает нейроны в полосатом теле и других прилегающих структурах. Чем больше количество копий CAG, тем раньше болезнь проявится. Было показано, что нуклеаза Cas9 из Staphylococcus aureus (небольшой ортолог Cas9), может быть упакована вместе с направляющей РНК в единый вектор AAV и использована для блокады экспрессии мутантного гена HTT путем стимулирования мутаций, вызывающих сдвиг рамки считывания. Введение этой конструкции в полосатое тело мышей R6/2 (модель HD) вызывало выключение мутантного гена HTT и приводило к уменьшению нейрональных включений почти на 50% [27]. Кроме того, экспансия микросателлитных повторов в ДНК продуцируют создание патогенных видов РНК. В связи с этим, разрабатываются технологии для нацеливания на эти РНК для диагностических и терапевтических целей. Одна из них, программируемая система CRISPR-RCas9, способна визуализировать и устранять токсичные РНК с устранением характерных признаков заболевания [7]. С другой стороны, постоянное подавление экспрессии мутантного HTT с помощью CRISPR/Cas9 в полосатом теле мышей с нокаутом HD140Q, экспрессирующих мутантный HTT человека, эффективно истощало агрегаты HTT, ослабляло раннюю невропатологию и уменьшало двигательный дефицит [114]. Была создана линия ИПСК из мононуклеарных клеток крови пациента с HD с 38 повторами CAG в экзоне 1 HTT. Линия ИПСК сохранила мутацию, вызывающую заболевание, и выразила маркеры плюрипотентности. Данная линия представляет собой перспективную модель для исследования болезни [66]. Таким образом, редактирование генов, опосредованное CRISPR/Cas9 и использование линий ИПСК потенциально может быть использовано для лечения HD и других нейродегенеративных расстройств, а также устранения нейрональной токсичности во взрослом мозге.
Когнитивные нарушения и нарушения аутистического спектра. Данные нарушения часто связаны с дисфункцией метилирования в гене FMR1, кодирующем белок FMRP (fragile X mental retardation protein), или мутациями ядерного белка MeCP2 [53, 112]. Так, при синдроме ломкой Х-хромосомы (FXS), связанном с экспансией повторов CGG и их гиперметилированием, в гене FMR1 наблюдается снижение содержания FMRP, вызывающее нарушение трансляции мРНК, участвующих в регуляции синаптической пластичности, что приводит к умственной отсталости и аутизму. При FXS наблюдается также ненормально высокое усиление сигналинга, индуцируемого метаботропным рецептором глутамата (mGluR5). Применение системы CRISPR-Gold, нацеленной на подавление гена mGluR5 в полосатом теле мышей с нокаутом FMRP (модель аутизма) позволило снизить уровень mGluR5 на 40–50% и улучшить некоторые поведенческие характеристики без появления иммунного ответа [53]. Повышенное внимание исследователей вызывает также ген MECP2, связанный с Х хромосомой, мутации в котором вызывают расстройства аутистического спектра, включая синдром Ретта [33]. Ген MECP2 кодирует белок MeCP2, который регулирует экспрессию генов через связывание с метилированными кодонами CGG геномной ДНК. Метилирование ДНК является важной эпигенетической меткой с широкими физиологическими функциями, включая развитие нейронов. Соответственно, дисфункция MeCP2 приводит к значительным физиологическим нарушениям. При дисфункции MeCP2 наблюдается нарушение двигательной функции и координации, тревожное поведение, сложности с обучением, нарушение сна и потеря болевой чувствительности. С появлением технологий CRISPR/Cas появилась надежда на возможность досконального изучения функций MeCP2, с дальнейшей терапией аутистических расстройств, связанных с его мутацией. С помощью CRISPR/Cas была создана линия мышей с делецией в домене AT-hook 1 MECP2, самцы которой воспроизводят отдельные фенотипы, наблюдаемые у людей с заболеваниями аутистического спектра, в том числе тревожное поведение и дефицит обучения [112]. У этих мышей в коре головного мозга происходит экспрессия мутантного Mecp2 и изменена экспрессия нескольких генов, которые специфически экспрессируются в тормозных нейронах мозга. Авторы полагают, что домен AT-hook 1 является критическим для функции белка MeCP2.
Известно, что 36% мутаций в MeCP2, вызывающих синдром Ретта, определяются однонуклеотидными заменами G > A или C > T [94]. Таким образом, замена гуанизина на аденозин потенциально может восстановить функцию MeCP2. В качестве подхода, Sinnamon и коллеги использовали редактирование оснований на уровне мРНК у мышей MECP2317G > A (R106Q) с синдромом Ретта. Для редактирования оснований использовали AAV систему, экспрессирующую каталитический домен аденозиндезаминазы, действующей на РНК, и гид, направленный на MeCP2. Через 1 мес. в трех разных популяциях нейронов гиппокампа 50% РНК MeCP2 перекодировалось, а локализация белка MeCP2 в гетерохроматине восстанавливалась до 50% от уровней дикого типа [94]. Таким образом, авторы полагают, что программируемое редактирование РНК может быть использовано для исправления мутаций в мышиных моделях неврологических заболеваний. Интересно, что пациенты с мутацией MECP2 часто демонстрируют нарушение сна, в связи с этим MeCP2 предложен и в качестве терапевтической мишени для нормализации сна [103].
С использованием системы CRISPR-опосредованного скрининга Perturb-Seq проведена функциональная оценка 35 генов риска расстройства аутистического спектра/задержки нервного развития (ASD/ND) в головном мозге in vivo [42]. Для этого были произведены CRISPR-индуцированные нокауты этих генов в развивающемся мозге мыши в утробе матери, с последующим одноклеточным секвенированием РНК нарушенных клеток в постнатальном мозге. В результате было показано, как различные мутации влияют на типы и состояния клеток в развивающемся организме, и были выявлены нейрональные и глиальные аномалии, связанные с генами риска аутизма.
Шизофрения. Генетически сложное полигенное заболевание, которое часто связано с когнитивными нарушениями и широко распространено в популяции. Чтобы обнаружить механизмы, лежащие в основе полигенных заболеваний, важно понять функции генов, связанных с заболеванием in vivo, и определить приоритетность генов-кандидатов для дальнейшего анализа. Удобным объектом для подобных исследований являются рыбки данио, которые широко используются как модельные системы для раскрытия онтогенетических и поведенческих функций генов человека [37]. Для определения нейробиологических функций 132 генов, связанных с шизофренией при помощи Cas9, были созданы 132 мутанта данио, в результате установлены функции многих из них, в том числе определена важная роль фактора транскрипции znf536 в развитии нейронов переднего мозга, участвующих в социальном поведении и стрессе [101]. Благодаря использованию CRISPR-опосредованных технологий, также были обнаружены синергетические эффекты распространенных вариантов риска шизофрении [90].
Перспективы. Основные трудности применения для терапии когнитивных нарушений в первую очередь связаны с недостатком фундаментальных знаний молекулярных механизмов когнитивных функций, а развитие CRISPR-технологий в ближайшем будущем обещает значительно пополнить эти пробелы. Кроме того, основным препятствием для разработки эффективных методов лечения нейродегенеративных заболеваний является неполное понимание лежащих в их основе клеточных механизмов. Надежды в этой области связаны с успехами в разработках генетических скринингов нейронов, полученных из индуцированных плюрипотентных стволовых клеток человека [ИПСК] с использованием CRISPR-интерференции/активации. Для выяснения вклада различных генов в выживаемость, дифференцировку, регуляцию транскрипции и морфологию клеток, в 2019 году была разработана платформа интерференции CRISPRi для устойчивого нокдауна 2300 эндогенных генов в нейронах, полученных из ИПСК [102]. Проведя CRISPRi- зависимый скрининг, авторы выявили нейрон-специфические гены и гены, которые улучшали выживаемость нейронов после нокдауна, а также гены, которые способствовали долголетию нейронов. С применением этих методов, ученые надеются понять, почему одни типы нейронов в зависимости от нейродегенеративных заболеваний избирательно уязвимы для нейродегенерации, в то время как другие устойчивы [44]. ИПСК в настоящее время широко применяются для восстановления функциональности поврежденной ткани путем замены поврежденных клеток конкурентными, а для также моделирования нейродегенеративных заболеваний с целью разработки новых методов лечения, открытия биомаркеров, разработки и тестирования новых лекарств. Выражаются оптимистичные надежды, что с помощью ИПСК удастся осуществить терапию таких нейродегенеративных заболеваний, как болезни Паркинсона и Альцгеймера [105].
Неполными являются и наши знания о механизмах дифференцировки нервных клеток, и, соответственно, о заболеваниях, связанных с неправильным развитием. На основе метода GESTALTЗ, в котором использовано CRISPR/Cas9-зависимое редактирование генома для постепенного ввода и накопления мутаций в штрих-коде ДНК в течение нескольких раундов деления клетки, что маркирует клетки-потомки [70], разработан scGESTALT. Этот подход при помощи одноклеточных последовательностей РНК (scRNA-seq) и штрих-кодов CRISPR-Cas9 позволяет реконструировать клональные отношения между сотнями типов клеток, образующихся во время развития, и может способствовать пониманию молекулярной идентичности и истории происхождения тысяч клеток во время нормального и патологического развития любых организмов [83]. Кроме того, для определения причинной связи между генотипами и фенотипами нейронов разработана стратегия, основанная на лазерной микродиссекции отдельных нейронов, трансфицированных CRISPR/Cas с дальнейшим секвенированием продуктов ПЦР, содержащих геномную область CRISPR/Cas-мишени в единичных эталонных нейронах [96].
В улучшении нуждаются также технологии доставки CRISPR систем в ЦНС in vivo. Так внутричерепное введение вирусных векторов, применяемое в настоящее время, захватывает территории не более нескольких кубических миллиметров. Оптимизм вызывают недавно разработанные AAV векторы с модифицированными капсидами, которые могут преодолевать гематоэнцефалический барьер, и, соответственно, могут быть введены системно [14]. Успешно ведутся и работы по улучшению как точности и специфичности нацеливания CRISPR, так и повышению эффективности редактирования генома. Недавно была разработана стратегия CRISPR-chrom, увеличивающая эффективность модификации генома в несколько раз через слияние SpCas9 с хроматин-модулирующими пептидами, полученными из белков HMGN1 и HMGB1, гистона H1 и комплексов ремоделирования хроматина [23]. Большими перспективами обладает также система CRISPR-FokI dCas9 [fdCas9], в которой каталитический домен эндонуклеазы FokI связан с dCas9 и парой направляющих sgRNA. Димеризация доменов FokI генерирует двухцепочечные разрывы ДНК, что активирует механизм репарации ДНК и приводит к эффективному редактированию генома [87]. Успех стратегий генной инженерии для медицины также будет зависеть от развития безопасных, иммуно-инертных и направленных на определенные типы клеток систем доставки. Надежда здесь связана с невирусными векторами доставки и обнаружением новых ортологов Cas9 из непатогенных видов бактерий [93]. Современные стратегии редактирования генома, их развитие и перспективы применения детально описаны в обзорах [52, 107, 110].
Начались клинические испытания CRISPR/Cas, которые дают первое представление о том, как нацеливание на ДНК и РНК может способствовать лечению многих генетических и эпигенетических заболеваний человека [100]. В настоящее время несколько коммерческих биотехнологических компаний усовершенствуют и внедряют технологии редактирования и модификации генома in vivo и разрабатывают методы лечения расстройств ЦНС [64]. Скорость развития в этой области – одна из самых высоких в науке. Предполагается, что следующие 5–10 лет будут важнейшими для понимания того, удастся ли вылечить некоторые трудноизлечимые неврологические заболевания с помощью редактирования генома.
Таким образом, CRISPR/CAS технологии, позволяющие модифицировать практически любой ген, либо изменять его экспрессию адаптированные для использования в ЦНС, а также трансгенные и CRISPR-экспрессирующие животные позволят в ближайшем будущем значительно пополнить наши знания о молекулярно-клеточных механизмах функционирования мозга. Нас могут ожидать открытия как в области молекулярных механизмов формирования долговременной памяти, так и в разработке новых подходов к лечению заболеваний, связанных с когнитивными нарушениями.
Список литературы
Гринкевич Л.Н. Эпигенетика и формирование долговременной памяти // Рос. физиол. журн. им. И.М. Сеченова. 2012. Т. 98. № 5. С. 553.
Гринкевич Л.Н. Роль микроРНК в обучении и долговременной памяти // Вавиловский журнал генетики и селекции // 2020. Т. 24. № 8. С. 885. https://doi.org/10.18699/VJ20.687
Дюжикова Н.А., Скоморохова Е.Б., Вайдо А.И. Эпигенетические механизмы формирования постстрессорных состояний // Усп. физиол. наук. 2015. Т. 46. № 1. С. 47.
Aquino-Jarquin G. Emerging Role of CRISPR/Cas9 Technology for MicroRNAs Editing in Cancer Research // Cancer Res. 2017. V. 77. № 24. P. 6812. https://doi.org/10.1158/0008-5472.CAN-17-2142
Bäck S., Necarsulmer J., Whitaker L.R. et al. Neuron-specific genome modification in the adult rat brain using CRISPR-Cas9 transgenic rats // Neuron. 2019. V.102. P. 105.
Bassuk A.G., Zheng A., Li Y., Tsang S.H., Mahajan V.B. Precision Medicine: Genetic Repair of Retinitis Pigmentosa in Patient-Derived Stem Cells // Sci. Rep. 2016. V. 6. № 19969. https://doi.org/10.1038/srep19969
Batra R., Nelles D.A., Pirie E. et al. Elimination of Toxic Microsatellite Repeat Expansion RNA by RNA-Targeting Cas9 // Cell. 2017. V. 170. № 5. P. 899. https://doi.org/10.1016/j.cell.2017.07.010
Behm M., Öhman M. RNA editing: a contributor to neuronal dynamics in the mammalian brain // Trends Genet. 2016. V. 32. P.165.
Bohnsack J., Patel V.K., Morrow A.L. Ethanol Exposure Regulates Gabra1 Expression via Histone Deacetylation at the Promoter in Cultured Cortical Neurons // J. Pharmacol. Exp. Ther. 2017. V. 363. № 1. P. 1. https://doi.org/10.1124/jpet.117.242446
Brane A.C., Tollefsbol T.O. Targeting Telomeres and Telomerase: Studies in Aging and Disease Utilizing CRISPR/Cas9 Technology // Cells. 2019. V. 8. № 2. P. 186. https://doi.org/10.3390/cells8020186
Bruch R., Baaske J., Chatelle C. et al. CRISPR/Cas13a-Powered Electrochemical Microfluidic Biosensor for Nucleic Acid Amplification-Free miRNA Diagnostics // Adv. Mater. 2019. V. 31. № 51. e1905311. https://doi.org/10.1002/adma.201905311
Bruch R., Johnston M., Kling A. et al. CRISPR-powered electrochemical microfluidic multiplexed biosensor for target amplification-free miRNA diagnostics // Biosens Bioelectron. 2021. V. 177. P. 112887. https://doi.org/10.1016/j.bios.2020.112887
Campa C.C., Weisbach N.R., Santinha A.J., Incarnato D., Platt R.J. Multiplexed genome engineering by Cas12a and CRISPR arrays encoded on single transcripts // Nat. Methods 2019. V. 16. P. 887.
Chan K.Y., Jang M.J., Yoo B.B. et al. Engineered AAVs for efficient noninvasive gene delivery to the central and peripheral nervous systems // Nat. Neurosci. 2017. V. 20. P. 1172.
Chen C.D., Zeldich E., Li Y., Yuste A., Abraham C.R. Activation of the Anti-Aging and Cognition-Enhancing Gene Klotho by CRISPR dCas9 Transcriptional Effector Complex // J. Mol. Neurosci. 2018. V. 64. № 2. P. 175. https://doi.org/10.1007/s12031-017-1011-0
Chen L.F., Lin Y.T., Gallegos D.A. et al. Enhancer histone acetylation modulates transcriptional bursting dynamics of neuronal activity-inducible genes // Cell Rep. 2019. V. 26. P. 1174.
Chiu Y.W., Hori Y., Ebinuma I. et al. Identification of calcium and integrin-binding protein 1 as a novel regulator of production of amyloid beta peptide using CRISPR/Cas9-based screening system // FASEB J. 2020. V. 34. № 6. P. 7661. https://doi.org/10.1096/fj.201902966RR
Chong C.M., Ke M., Tan Y. et al. Presenilin 1 deficiency suppresses autophagy in human neural stem cells through reducing γ-secretase-independent ERK/CREB signaling // Cell Death Dis. 2018. V. 9. № 9. P. 879. https://doi.org/10.1038/s41419-018-0945-7
Cong L., Ran F.A., Cox D. et al. Multiplex genome engineering using CRISPR/Cas systems // Science. 2013. V. 339. № 6121. P. 819. https://doi.org/10.1126/science.1231143
Davis K.M., Pattanayak V., Thompson D.B., Zuris J.A., Liu D.R. Small molecule-triggered Cas9 protein with improved genome editing specificity // Nat. Chem. Biol. 2015. V.11. P. 316.
Day J.J., Kennedy A.J., Sweatt J.D. DNA methylation and its implications and accessibility for neuropsychiatric therapeutics //Annu. Rev. Pharmacol. Toxicol. 2015. V. 55. P. 591. https://doi.org/10.1146/annurev-pharmtox-010814-124527
de Solis C.A., Ho A., Holehonnur R., Ploski J.E. The development of a viral mediated CRISPR/Cas9 system with doxycycline dependent gRNA expression for inducible in vitro and in vivo genome editing // Front. Mol. Neurosci. 2016. 9:70.
Ding X., Seebeck T., Feng Y. et al. Improving CRISPR-Cas9 Genome Editing Efficiency by Fusion with Chromatin-Modulating Peptides // CRISPR J. 2019. V. 2. P. 51. https://doi.org/10.1089/crispr.2018.0036
Dow L.E., Fisher J., O’Rourke K.P. et al. Inducible in vivo genome editing with CRISPR-Cas9 // Nat. Biotechnol. 2015. V. 33. P. 390. https://doi.org/10.1038/nbt.3155
Dubal D.B., Yokoyama J.S., Zhu L. et al. Life extension factor klotho enhances cognition // Cell Rep. 2014. V. 7. № 4. P.1065. https://doi.org/10.1016/j.celrep.2014.03.076
Ehrlich M. DNA hypermethylation in disease: mechanisms and clinical relevance // Epigenetics. 2019. V. 14. №12. P. 1141. https://doi.org/10.1080/15592294.2019.1638701
Ekman F.K., Ojala D.S., Adil M.M. et al. CRISPR-Cas9-mediated genome editing increases lifespan and improves motor deficits in a huntington’s disease mouse model // Mol. Ther. Nucleic Acids. 2019. V. 17. P. 829.
Ellenbroek B., Youn J. Rodent models in neuroscience research: is it a rat race? // Dis. Model. Mech. 2016. V. 9. P. 1079.
Feng J., Zhou Y., Campbell S.L. et al. Dnmt1 and Dnmt3a maintain DNA methylation and regulate synaptic function in adult forebrain neurons // Nat. Neurosci. 2010. V. 13. № 4. P. 423. https://doi.org/10.1038/nn.2514
Gao Y., Hisey E., Bradshaw T.W.A. et al. Plug-and-Play Protein Modification Using Homology-Independent Universal Genome Engineering // Neuron. 2019. V. 103. № 4. P. 583. doi: V. 13.https://doi.org/10.1016/j.neuron.2019.05.047
Garza J.C., Qi X., Gjeluci K. et al. Disruption of the psychiatric risk gene Ankyrin 3 enhances microtubule dynamics through GSK3/CRMP2 signaling // Transl. Psychiatry. 2018. V. 8. № 1. P. 135. https://doi.org/10.1038/s41398-018-0182-y
Gilbert L.A., Larson M.H., Morsut L. et al. Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes // Cell. 2013. V. 154. № 2. P. 442. https://doi.org/10.1016/j.cell.2013.06.044
Good K.V., Vincent J.B., Ausió J. MeCP2: The Genetic Driver of Rett Syndrome Epigenetics // Front. Genet. 2021. V. 12. P. 620859. https://doi.org/10.3389/fgene.2021.620859
Guhathakurta S., Kim J., Adams L. et al. Targeted attenuation of elevated histone marks at SNCA alleviates alpha-synuclein in Parkinson’s disease // EMBO Mol. Med. 2021. V. 13. № 2. e12188. https://doi.org/10.15252/emmm.202012188
Hilton I.B., D’Ippolito A.M., Vockley C.M. et al. Epigenome editing by a CRISPR Cas9-based acetyltransferase activates genes from promoters and enhancers // Nat. Biotechnol. 2015. V. 33. P. 510.
Hirosawa M., Fujita Y., Parr C.J.C. et al. Cell-type-specific genome editing with a microRNA-responsive CRISPR-Cas9 switch // Nucleic Acids Res. 2017. V. 45. № 13. e118. https://doi.org/10.1093/nar/gkx309
Hoffman E.J., Turner K., Fernandez J.M. et al. Estrogens Suppress a Behavioral Phenotype in Zebrafish Mutants of the Autism Risk Gene, CNTNAP2 // Neuron. 2016. V. 89. P. 725.
Hoffmann M.D., Aschenbrenner S., Grosse S. et al. Cell-specific CRISPR-Cas9 activation by microRNA-dependent expression of anti-CRISPR protein // Nucleic Acids Res. 2019. V. 47. № 13. e75. https://doi.org/10.1093/nar/gkz271
Huang T.P., Newby G.A., Liu D.R. Precision genome editing using cytosine and adenine base editors in mammalian cells // Nat. Protoc. 2021. V. 16. № 2. P. 1089. https://doi.org/10.1038/s41596-020-00450-9
Incontro S., Asensio C.S., Edwards R.H., Nicoll R.A. Efficient, complete deletion of synaptic proteins using CRISPR // Neuron. 2014. V. 83. № 5. P. 1051. https://doi.org/10.1016/j.neuron.2014.07.043
Jansen R., Embden J.D., Gaastra W., Schouls L.M. Identification of genes that are associated with DNA repeats in prokaryotes // Mol. Microbiol. 2002. V. 43. № 6. P. 1565. https://doi.org/10.1046/j.1365-2958.2002.02839.x
Jin X., Simmons S.K., Guo A. et al. In vivo Perturb-Seq reveals neuronal and glial abnormalities associated with autism risk genes // Science. 2020. V. 370. №6520: eaaz6063. https://doi.org/10.1126/science.aaz6063
Jinek M., Chylinski K., Fonfara I. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity // Science. 2012. V. 337. P. 816.
Kampmann M. A CRISPR Approach to Neurodegenerative Diseases // Trends mol. med. 2017. V. 23. P. 483. https://doi.org/10.1016/j.molmed.2017.04.003
Kantor B., Tagliafierro L., Gu J. et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD // Mol. Ther. 2018. V. 26. № 11. P. 2638. https://doi.org/10.1016/j.ymthe.2018.08.019
Kim H., Ham S., Jo M. et al. CRISPR-Cas9 Mediated Telomere Removal Leads to Mitochondrial Stress and Protein Aggregation // Int. J. Mol. Sci. 2017. V. 18. P. 2093. https://doi.org/10.3390/ijms18102093
Kim S., Kaang B.K. Epigenetic regulation and chromatin remodeling in learning and memory // Exp. Mol. Med. 2017. V. 49. № 1:e281. https://doi.org/10.1038/emm.2016.140
Konermann S., Brigham M.D., Trevino A.E. et al. Optical control of mammalian endogenous transcription and epigenetic states // Nature. 2013. V. 500. № 7463. P. 472–476.
Konermann S., Lotfy P., Brideau N.J. et al. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors // Cell. 2018. V. 173. № 3. P. 665. https://doi.org/10.1016/j.cell.2018.02.033
Kumar N., Stanford W., de Solis C. et al. The development of an AAV-based CRISPR SaCas9 genome editing system that can be delivered to neurons in vivo and regulated via doxycycline and Cre-recombinase // Front. Mol. Neurosci. 2018. V. 11. P.413.
Kuscu C., Mammadov R., Czikora A. et al. Temporal and Spatial Epigenome Editing Allows Precise Gene Regulation in Mammalian Cells // J. Mol. Biol. 2019. V. 431. № 1. P. 111. doi: 48https://doi.org/10.1016/j.jmb.2018.08.001
Lau C.H., Tin C., Suh Y. CRISPR-based strategies for targeted transgene knock-in and gene correction // Fac. Rev. 2020. V. 9. P. 20. https://doi.org/10.12703/r/9-20
Lee B., Lee K., Panda S. et al. Nanoparticle delivery of CRISPR into the brain rescues a mouse model of fragile X syndrome from exaggerated repetitive behaviors // Nat. Biomed. Eng. 2018. V. 2. P. 497.
Lei Y., Zhang X., Su J. et al. Targeted DNA methylation in vivo using an engineered dCas9-MQ1 fusion protein // Nat. Commun. 2017. V. 8. P.16026.
Liang P., Xu Y., Zhang X. et al. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes // Protein Cell. 2015. V. 6. № 5. P. 363. https://doi.org/10.1007/s13238-015-0153-5
Lin Y.T., Seo J., Gao F. et al. APOE4 Causes Widespread Molecular and Cellular Alterations Associated with Alzheimer’s Disease Phenotypes in Human iPSC-Derived Brain Cell Types // Neuron. 2018. V. 98. № 6. P. 1141. https://doi.org/10.1016/j.neuron.2018.05.008
Liu E.Y., Cali C.P., Lee E.B. RNA metabolism in neurodegenerative disease // Dis. Model. Mech. 2017. V. 10. № 5. P. 509. doi.org/https://doi.org/10.1242/dmm.028613
Liu K.I., Ramli M.N., Woo C.W. et al. A chemical-inducible CRISPR-Cas9 system for rapid control of genome editing // Nat. Chem. Biol. 2016. V. 12. № 11. P. 980. https://doi.org/10.1038/nchembio.2179
Liu K.I., Ramli M.N.B., Sutrisnoh N.B., Tan M.H. Rapid Control of Genome Editing in Human Cells by Chemical-Inducible CRISPR-Cas Systems // Methods Mol. Biol. 2018. V. 1772. P. 267. https://doi.org/10.1007/978-1-4939-7795-6_15
Liu Q., He D., Xie L. Prediction of off-target specificity and cell-specific fitness of CRISPR-Cas System using attention boosted deep learning and network-based gene feature // PLoS Comput. Biol. 2019. V. 15. № 10. e1007480. https://doi.org/10.1371/journal.pcbi.1007480
Liu X.S., Jaenisch R. Editing the Epigenome to Tackle Brain Disorders // Trends Neurosci. 2019. V. 42. № 12. P. 861. https://doi.org/10.1016/j.tins.2019.10.003
Liu X.S., Wu H., Ji X. et al. Editing DNA Methylation in the Mammalian Genome // Cell. 2016. V. 167. № 1. P. 233. https://doi.org/10.1016/j.cell.2016.08.056
Liu X.S., Wu H., Krzisch M. et al. Rescue of fragile X syndrome neurons by DNA methylation editing of the FMR1 gene // Cell. 2018. V. 172. № 5. P. 979. https://doi.org/10.1016/j.cell.2018.01.012
Lubroth P., Colasante G., Lignani G. In vivo Genome Editing Therapeutic Approaches for Neurological Disorders: Where Are We in the Translational Pipeline? // Front. Neurosci. 2021. V. 15. № 632522. https://doi.org/10.3389/fnins.2021.632522
Ma Y., Zhang L., Huang X. Building Cre Knockin Rat Lines Using CRISPR/Cas9 // Methods Mol. Biol. 2017. V. 1642. P. 37. https://doi.org/10.1007/978-1-4939-7169-5_3
Malakhova A.A., Grigor’eva E.V., Malankhanova T.B. et al. Generation of induced pluripotent stem cell line ICGi018-A from peripheral blood mononuclear cells of a patient with Huntington’s disease // Stem. Cell Res. 2020. V. 44:101743. https://doi.org/10.1016/j.scr.2020.101743
Mali P., Yang L., Esvelt K.M. et al. RNA-guided human genome engineering via Cas9 // Science. 2013. V. 339. P. 823.
Masso A., Sanchez A., Bosch A., Gimenez-Llort L., Chillon M. Secreted alphaKlotho isoform protects against age-dependent memory deficits // Mol. Psychiatry. 2018. V. 23. № 9. P. 1937. https://doi.org/10.1038/mp.2017.211
McGraw C.M., Samaco R.C., Zoghbi H.Y. Adult neural function requires MeCP2 // Science. 2011. V. 333. № 6039. P. 186. https://doi.org/10.1126/science.1206593
McKenna A., Findlay G.M., Gagnon J.A. et al. Whole-organism lineage tracing by combinatorial and cumulative genome editing // Science. 2016. V. 353. № 6298:aaf7907. https://doi.org/10.1126/science.aaf7907
Mikuni T., Nishiyama J., Sun Y., Kamasawa N., Yasuda R. High-throughput, high-resolution mapping of protein localization in mammalian brain by in vivo genome editing // Cell. 2016. V. 165. № 7. P. 1803. https://doi.org/10.1016/j.cell.2016.04.044
Mojica F.J., Díez-Villaseñor C., García-Martínez J., Soria E.J. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements // Mol. Evol. 2005. V. 60. № 2. P. 174. https://doi.org/10.1007/s00239-004-0046-3
Mojica F.J.M., Díez-Villaseñor C., García-Martínez J., Almendros C. Short motif sequences determine the targets of the prokaryotic CRISPR defence system // Microbiology (Reading). 2009. V. 155 (Pt 3). P. 733. https://doi.org/10.1099/mic.0.023960-0
Moretti P., Levenson J.M., Battaglia F. et al. Learning and memory and synaptic plasticity are impaired in a mouse model of Rett syndrome // J. Neurosci. 2006. V. 26. № 1. P. 319. doi: 05.2006https://doi.org/10.1523/JNEUROSCI.2623
Moses C., Hodgetts S.I., Nugent F. et al. Transcriptional repression of PTEN in neural cells using CRISPR/dCas9 epigenetic editing // Sci. Rep. 2020. V. 10. № 1. P. 11393. https://doi.org/10.1038/s41598-020-68257-y
Nishiyama J., Mikuni T., Yasuda R. Virus-Mediated Genome Editing via Homology-Directed Repair in Mitotic and Postmitotic Cells in Mammalian Brain // Neuron. 2017. V. 96. № 4. P. 755. https://doi.org/10.1016/j.neuron.2017.10.004
Park H., Oh J., Shim G. et al. In vivo neuronal gene editing via CRISPR-Cas9 amphiphilic nanocomplexes alleviates deficits in mouse models of Alzheimer’s disease // Nat. Neurosci. 2019. V. 22. P. 524.
Persico A.M., Napolioni V. Autism genetics // Behav. Brain Res. 2013. V. 25. P. 95.
Pluvinage J.V., Haney M.S., Smith B.A.H. et al. CD22 blockade restores homeostatic microglial phagocytosis in ageing brains // Nature. 2019. V. 568. № 7751. P. 187. https://doi.org/10.1038/s41586-019-1088-4
Port F., Starostecka M., Boutros M. Multiplexed conditional genome editing with Cas12a in Drosophila. Multiplexed conditional genome editing with Cas12a in Drosophila // Proc. Natl. Acad. Sci. U S A. 2020. V. 117. № 37. P. 22890. https://doi.org/10.1073/pnas.2004655117
Qi L.S., Larson M.H., Gilbert L.A. et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression // Cell. 2013. V. 152. P. 1173.
Qiu P.Y., Jiang J., Liu Z. et al. BMAL1 knockout macaque monkeys display reduced sleep and psychiatric disorders // Natl. Sci. Rev. 2019. V. 6. № 1. P. 87. https://doi.org/10.1093/nsr/nwz002
Raj B., Wagner D.E., McKenna A. et al. Simultaneous single-cell profiling of lineages and cell types in the vertebrate brain // Nat. Biotechnol. 2018. V. № 36. P. 442.
Rees H.A., Liu D.R. Base editing: precision chemistry on the genome and transcriptome of living cells // Nat. Rev. Genet. 2018. V. 19. P. 770.
Remy S., Chenouard V., Tesson L. et al. Generation of gene-edited rats by delivery of CRISPR/Cas9 protein and donor DNA into intact zygotes using electroporation // Sci. Rep. 2017. V. 7. № 1. P. 16554. https://doi.org/10.1038/s41598-017-16328-y
Rousseau B.A., Hou Z., Gramelspacher M.J., Zhang Y. Programmable RNA Cleavage and Recognition by a Natural CRISPR-Cas9 System from Neisseria meningitidis // Mol. Cell. 2018. V. 69. № 5. P. 906. https://doi.org/10.1016/j.molcel.2018.01.025
Saifaldeen M., Al-Ansari D.E., Ramotar D., Aouida M. CRISPR FokI Dead Cas9 System: Principles and Applications in Genome Engineering // Cells. 2020. V. 9. № 11. P. 2518. https://doi.org/10.3390/cells9112518
Sandoval A. Jr., Elahi H., Ploski J.E. Genetically Engineering the Nervous System with CRISPR-Cas // eNeuro. 2020. V. 7. № 2. https://doi.org/10.1523/ENEURO.0419-19.2020
Savell K.E., Bach S.V., Zipperly M.E. et al. A Neuron-Optimized CRISPR/dCas9 Activation System for Robust and Specific Gene Regulation // eNeuro. 2019. V. 6. № 1. https://doi.org/10.1523/ENEURO.0495-18.2019
Schrode N., Ho S.M, Yamamuro K. et al. Synergistic effects of common schizophrenia risk variants // Nat. Genet. 2019. V. 51. № 10. P.1475.
Schwank G., Koo Bon-Kyoung, Sasselli V. et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients // Cell Stem. Cell. 2013. V. 13. № 6. P. 653. https://doi.org/10.1016/j.stem.2013.11.002
Sgro A., Blancafort P. Epigenome engineering new technologies for precision medicine // Nucleic Acids Res. 2020. V. 48. № 22. P.12453. https://doi.org/10.1093/nar/gkaa1000
Shalaby K., Aouida M., El-Agnaf O. Tissue-Specific Delivery of CRISPR Therapeutics: Strategies and Mechanisms of Non-Viral Vectors // Int. J. Mol. Sci. 2020. V. 21. № 19. P. 7353. https://doi.org/10.3390/ijms21197353
Sinnamon J.R., Kim S.Y., Fisk J.R. et al. In Vivo Repair of a Protein Underlying a Neurological Disorder by Programmable RNA Editing // Cell Rep. 2020. V. 32. № 2. 107878. https://doi.org/10.1016/j.celrep.2020.107878
Staahl B.T, Benekareddy M., Coulon-Bainier C. et al. Efficient genome editing in the mouse brain by local delivery of engineered Cas9 ribonucleoprotein complexes // Nat. Biotechnol. 2017. V. 35. P. 431.
Steinecke A., Kurabayashi N., Hayano Y., Ishino Y., Taniguchi H. In Vivo Single-Cell Genotyping of Mouse Cortical Neurons Transfected with CRISPR/Cas9 // Cell Rep. 2019. V. 28. № 2. P. 325. https://doi.org/10.1016/j.celrep.2019.06.038
Sun W., Ji W., Hall J. M. et al. Self-assembled DNA nanoclews for the efficient delivery of CRISPR-Cas9 for genome editing // Angew Chem. Int. Ed Engl. 2015. V. 54. № 41. P. 12029. https://doi.org/10.1002/anie.201506030
Swartjes T., Staals R.H.J., van der Oost J. Editor’s cut: DNA cleavage by CRISPR RNA-guided nucleases Cas9 and Cas12a // Biochem. Soc. Trans. 2020. V. 48. № 1. P. 207. https://doi.org/10.1042/BST20190563
Swiech L., Heidenreich M., Banerjee A. et al. In vivo interrogation of gene function in the mammalian brain using CRISPR-Cas9 // Nat. Biotechnol. 2015. V. 33. № 1. P. 102. https://doi.org/10.1038/nbt.3055
Tay L.S., Palmer N., Panwala R., Chew W.L., Mali P. Translating CRISPR-Cas Therapeutics: Approaches and Challenges // CRISPR J. 2020. V. 4. P. 253. https://doi.org/10.1089/crispr.2020.0025
Thyme S.B., Pieper L.M., Li E.H. et al. Phenotypic landscape of schizophrenia-associated genes defines candidates and their shared functions // Cell. 2019. V. 177. P. 478.
Tian R., Gachechiladze M.A., Ludwig C.H. et al. CRISPR Interference-Based Platform for Multimodal Genetic Screens in Human iPSC-Derived Neurons // Neuron. 2019. V. 104. № 2. P. 239. https://doi.org/10.1016/j.neuron.2019.07.014
Tsuchiya Y., Minami Y., Umemura Y. et al. Disruption of MeCP2 attenuates circadian rhythm in CRISPR/Cas9-based Rett syndrome model mouse //Genes Cells. 2015. V. 20. № 12. P. 992. https://doi.org/10.1111/gtc.12305
Uezu A., Kanak D.J., Bradshaw T.W. et al. Identification of an elaborate complex mediating postsynaptic inhibition // Science. 2016. V. 353. № 6304. P.1123. https://doi.org/10.1126/science.aag0821
Valadez-Barba V., Cota-Coronado A., Hernández-Pérez O.R. et al. iPSC for modeling neurodegenerative disorders // Regen. Ther. 2020. V. 15. P. 332. https://doi.org/10.1016/j.reth.2020.11.006
Vermilyea S.C., Babinski A., Tran N., et al. In Vitro CRISPR/Cas9-Directed Gene Editing to Model LRRK2 G2019S Parkinson’s Disease in Common Marmosets // Sci. Rep. 2020. V. 10. № 1. P. 3447. https://doi.org/10.1038/s41598-020-60273-2
Wang D., Zhang F., Gao G. CRISPR-Based Therapeutic Genome Editing: Strategies and In Vivo Delivery by AAV Vectors // Cell. 2020. V. 181. № 1. P. 136. https://doi.org/10.1016/j.cell.2020.03.023
Willems J., de Jong A.P.H., Scheefhals N. et al. ORANGE: A CRISPR/Cas9-based genome editing toolbox for epitope tagging of endogenous proteins in neurons // PLoS Biol. 2020. V. 18. № 4. e3000665. https://doi.org/10.1371/journal.pbio.3000665
Wolf E.J., Chen C.D., Zhao X., et al. Klotho, PTSD, and advanced epigenetic age in cortical tissue // Neuropsychopharmacology. 2021. V. 46. № 4. P. 721. https://doi.org/10.1038/s41386-020-00884-5
Wu Q., Shou J. Toward precise CRISPR DNA fragment editing and predictable 3D genome engineering // J Mol. Cell Biol. 2021. V. 12. № 11. P. 828. https://doi.org/10.1093/jmcb/mjaa060
Wu Y.Y., Kuo H.C. Functional roles and networks of non-coding RNAs in the pathogenesis of neurodegenerative diseases // J. Biomed. Sci. 2020. V. 27. № 1. 49. https://doi.org/10.1186/s12929-020-00636-z
Xu M., Song P., Huang W. et al. Disruption of AT-hook 1 domain in MeCP2 protein caused behavioral abnormality in mice // Biochim. Biophys. Acta Mol. Basis Dis. 2018. V. 1864. № 2. P. 347. https://doi.org/10.1016/j.bbadis.2017.10.022
Xu Y., Sun Z. Molecular basis of Klotho: from gene to function in aging // Endocr. Rev. 2015. V. 36. № 2. P. 174. https://doi.org/10.1210/er.2013-1079
Yang S., Chang R., Yang H. et al. CRISPR/Cas9-mediated gene editing ameliorates neurotoxicity in mouse model of Huntington’s disease // J. Clin. Invest. 2017. V. 127. P. 2719.
Yao X., Wang X., Hu X. et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9 // Cell Res. 2017. V. 27. №6. P. 801.
Yeo N.C., Chavez A., Lance-Byrne A. et al. An enhanced CRISPR repressor for targeted mammalian gene regulation // Nat. Methods. 2018. V. 15. P. 611.
Yim Y.Y., Teague C.D., Nestler E.J. In vivo locus-specific editing of the neuroepigenome // Nat. Rev. Neurosci. 2020. V. 21. № 9. P. 471. https://doi.org/10.1038/s41583-020-0334-y
Zeng C.Y., Yang T.T., Zhou H.J. et al. Lentiviral vector-mediated overexpression of Klotho in the brain improves Alzheimer’s disease-like pathology and cognitive deficits in mice // Neurobiol. Aging. 2019. V. 78. P. 18. https://doi.org/10.1016/j.neurobiolaging.2019.02.003
Zhao J., Li B., Ma J., Jin W., Ma X. Photoactivatable RNA N[6]-Methyladenosine Editing with CRISPR-Cas13 // Small. 2020. V. 30. e1907301. https://doi.org/10.1002/smll.201907301
Zheng Y., Shen W., Zhang J. et al. CRISPR interference based specific and efficient gene inactivation in the brain // Nat. Neurosci. 2018. V. 21. P. 447.
Zhou H., Liu J., Zhou C. et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice // Nat. Neurosci. 2018. 21:440–446
Zhou H., Su J., Hu X. et al. Glia-to-Neuron Conversion by CRISPR-CasRx Alleviates Symptoms of Neurological Disease in Mice // Cell. 2020. V. 181. № 3. P. 590. https://doi.org/10.1016/j.cell.2020.03.024
Zhou X.X., Zou X., Chung H.K. et al. A single-chain photoswitchable CRISPR-Cas9 architecture for light-inducible gene editing and transcription // ACS Chem. Biol. 2018. V. 13. P. 443.
Zuo E., Cai Y.J., Li K. et al. One-step generation of complete gene knockout mice and monkeys by CRISPR/Cas9-mediated gene editing with multiple sgRNAs // Cell Res. 2017. V. 27. №7. P. 933. https://doi.org/10.1038/cr.2017.81
Дополнительные материалы отсутствуют.
Инструменты
Успехи физиологических наук