Биоорганическая химия, 2022, T. 48, № 6, стр. 721-731
Поиск опухолеспецифических промоторов in silico
1 ФГБУН “Институт биоорганической химии им. М.М. Шемякина и Ю.А. Овчинникова” РАН
117997 Москва, ул. Миклухо-Маклая, 16/10, Россия
* E-mail: kachkine@yandex.ru
Поступила в редакцию 06.04.2022
После доработки 18.05.2022
Принята к публикации 20.05.2022
- EDN: ONBVJI
- DOI: 10.31857/S0132342322060124
Аннотация
Ранее мы продемонстрировали опухолеспецифическую активность нескольких природных и химерных промоторов человека. В данной работе исследованы профили узнавания транскрипционных факторов и нуклеотидные мотивы в опухолеспецифических промоторах. Был идентифицирован ряд профилей, которые чаще присутствуют в опухолеспецифических промоторах, чем в промоторах генов домашнего хозяйства. Проведен поиск новых предполагаемых регуляторных связей промоторов с факторами транскрипции. Выявлен нуклеотидный мотив из 44 пар нуклеотидов, характерный для опухолеспецифических промоторов, но не для промоторов генов домашнего хозяйства. Среди 29 598 промоторов человека из базы данных промоторов EPDnew выявлен ряд промоторов с этим мотивом, гены которых ассоциированы с неблагоприятным прогнозом при различных опухолях. Мы предполагаем, что некоторые из этих промоторов могут обладать опухолеспецифической активностью. Также было обнаружено близкое сходство по нуклеотидным мотивам между промоторами генов BIRC5 и MCM2. Результаты исследования могут помочь в понимании особенностей транскрипции генов в опухолях, а также в поиске природных опухолеспецифических промоторов или в создании искусственных промоторов для генной терапии рака и разработки противоопухолевых векторных вакцин.
ВВЕДЕНИЕ
Функционирование эукариотического промотора – сложный процесс, включающий взаимодействие цис- и транс-регуляторных элементов промотора с факторами транскрипции (ФТ), ферментами транскрипционного комплекса и регуляторными РНК [1]. Существенную роль в регуляции транскрипции играют эпигенетические механизмы, такие как конформационные изменения хроматина, метилирование ДНК, модификации гистонов [2], а также физико-химические процессы, связанные с разделением фаз [3]. Однако в основе всей регуляции активности промоторов лежит связывание ФТ со специфическими последовательностями в ДНК и взаимодействие между факторами.
Всего в настоящее время известно более 1600 ФТ человека, их число постоянно растет, но для значительной части из них (более 500 факторов) неизвестны последовательности сайтов связывания с ДНК [4]. Существенная черта сайтов связывания ФТ – их вырожденность и перекрестное сродство разных ФТ к одной и той же последовательности ДНК [5]. Это обеспечивает вероятностную и конкурентную регуляцию транскрипции.
Особенности транскрипции генов наряду с перестройкой метаболизма в трансформированных клетках определяют способности опухолей расти, избегая контроля со стороны окружающих тканей и организма, поэтому изучение этих процессов имеет теоретическое и практическое значение для онкологии. Некоторые из известных ФТ рассматривают в качестве диагностических маркеров опухолей или их метастазов. Так, CDX2 – чувствительный маркер колоректальных аденокарцином, TTF1 – значимый маркер для аденокарцином легкого, PAX8 может служить маркером гинекологических опухолей и т.д. [6]. Поэтому изучение ФТ, в частности присутствия их профилей узнавания в промоторах, остается актуальным.
В настоящее время накопленные данные о структуре промоторов уже открывают большие перспективы для использования промоторов в практической медицине, например, в составе генно-инженерных векторов для генной терапии опухолей [7, 8]. Однако выбор оптимальных промоторов, а также поиск новых промоторов для генно-инженерных терапевтических векторов остаются нетривиальными задачами. В последнее время большой интерес к промоторам возобновился в связи с необходимостью создания вакцинных векторов для профилактики или терапии инфекционных заболеваний и рака [9, 10].
Ранее мы клонировали ряд промоторов генов, регулирующих пролиферацию клеток, которые проявляют опухолеспецифическую активность [11, 12] (табл. 1). Целью данной работы стал поиск особенностей этих промоторов, а также полученных из них химерных промоторов [13], которые обусловливают их повышенную активность в клетках опухолей. Для этого мы сравнили эти промоторы с набором промоторов генов домашнего хозяйства по составу известных профилей узнавания ФТ, а также провели поиск нуклеотидных мотивов de novo в промоторах.
Таблица 1.
Промоторы, исследованные в данной работе
| Промотор | Индекс EPDnew |
|---|---|
| Опухолеспецифические промоторы* | |
| BIRC5 | FP024000 |
| CDC6 | FP023205 |
| CKS1B | FP001855 |
| MCM2 | FP005950 |
| PLK1 | FP021553 |
| POLD1 | FP026208 |
| TERT | FP007749 |
| Промотор PCNA* | |
| PCNA | FP026732 |
| Химерные промоторы | |
| CH2-CH26 | [13] |
| Промоторы генов домашнего хозяйства | |
| ACTB | FP010679 |
| ALAS1 | FP005471 |
| B2M | FP020416 |
| GAPDH | FP017185 |
| GUSB | FP011089, FP011090 |
| HMBS | FP016933, FP016934 |
| HPRT1 | FP029346, FP029347 |
| PGK1 | FP029028 |
| PPIA1 | FP011011 |
| RPL13A | FP026153 |
| RPLP0 | FP018538, FP018539 |
| SDHA | FP007723 |
| TBP | FP010603, FP010604 |
| TFRC | FP006502 |
| TUBB | FP009374 |
| YWHAZ | FP012696, FP012697, FP012698 |
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Профили, преобладающие в опухолеспецифических промоторах. Ранее мы показали, что относительно короткие (размером в сотни нуклеотидов) промоторы ряда генов (табл. 1), участвующих в регуляции пролиферации, обладают опухолеспецифической активностью [11, 12]. Известно, что в исследованных промоторах наиболее важные регуляторные элементы сосредоточены на участке приблизительно до –500 п.н. от точки начала транскрипции (TSS) [12, 14, 15]. Поэтому мы выбрали из базы эукариотических промоторов EPDnew семь последовательностей ДНК с координатами [‒499; +100] по отношению к точке начала транскрипции, которые соответствовали ранее исследованным опухолеспецифическим промоторам. Поскольку для некоторых из этих генов известно более одного промотора, мы отобрали только те промоторы, которые были исследованы в прямых экспериментах (табл. 1). Для сравнения мы использовали 23 промотора генов домашнего хозяйства такой же длины. С помощью программы CiiiDER [16] и базы профилей транскрипционных факторов JASPAR (http://jaspar.genereg.net/) [5] мы провели сравнение (enrichment) опухолеспецифических промоторов с промоторами генов домашнего хозяйства по составу профилей узнавания ФТ (рис. S1 в дополнительных материалах). При этом мы сопоставляли число промоторов в обеих группах, содержащих соответствующие профили ФТ; результат оценивали по критерию Манна–Уитни. Полные результаты сравнения промоторов приведены в табл. S1 в дополнительных материалах. В табл. 2 и S2 (см. дополнительные материалы) приведены профили ФТ, которые статистически значимо (p < 0.05) преобладают в опухолеспецифических промоторах по сравнению с промоторами генов домашнего хозяйства (далее – условно опухолеспецифические ФТ).
Таблица 2.
Факторы транскрипции, профили которых преобладают в опухолеспецифических промоторах по сравнению с промоторами генов домашнего хозяйства
| Фактор транскрипции | Номенклатура JASPAR | Класс | Семейство | Содержатся в опухолеспецифических промоторах |
|---|---|---|---|---|
| RUNX2* | MA0511.2 | Runt domain factors | Runt-related factors | POLD1, BIRC5, PLK1, CDC6, CKS1B |
| Creb3l2* | MA0608.1 | Basic leucine zipper factors (bZIP) | CREB-related factors | MCM2, PLK1, CDC6, TERT |
| SREBF2* | MA0596.1 | Basic helix-loop-helix factors (bHLH) | bHLH-ZIP factors | MCM2, POLD1, BIRC5, PLK1, CKS1B, TERT |
| ETS2* | MA1484.1 | Tryptophan cluster factors | Ets-related factors | MCM2, PLK1, CDC6, CKS1B, TERT |
| CENPB | MA0637.1 | CENP-B | MCM2, CDC6, CKS1B | |
| HEY1 | MA0823.1 | Basic helix-loop-helix factors (bHLH) | Hairy-related factors | MCM2, PLK1, TERT |
| HEY2 | MA0649.1 | Basic helix-loop-helix factors (bHLH) | Hairy-related factors | MCM2, PLK1, TERT |
| RARA::RXRG | MA1149.1 | Nuclear receptors with C4 zinc fingers::Nuclear receptors with C4 zinc fingers | Thyroid hormone receptor-related factors (NR1)::RXR-related receptors (NR2) | MCM2, POLD1, CKS1B |
| SREBF1 (var. 2) | MA0829.2 | Basic helix-loop-helix factors (bHLH) | bHLH-ZIP factors | MCM2, BIRC5, PLK1 |
| ZNF75D* | MA1601.1 | C2H2 zinc finger factors | More than 3 adjacent zinc finger factors | MCM2, POLD1, PLK1, CDC6, CKS1B, TERT |
| Zfx* | MA0146.2 | C2H2 zinc finger factors | More than 3 adjacent zinc finger factors | MCM2, POLD1, BIRC5, CDC6, CKS1B, TERT |
| HOXD11 | MA0908.1 | Homeo domain factors | HOX-related factors | POLD1, CKS1B |
| KLF11 | MA1512.1 | C2H2 zinc finger factors | Three-zinc finger Kruppel-related factors | POLD1, BIRC5 |
| MEF2A | MA0052.4 | MADS box factors | Regulators of differentiation | MCM2, BIRC5 |
| RORB | MA1150.1 | Nuclear receptors with C4 zinc fingers | Thyroid hormone receptor-related factors (NR1) | MCM2, POLD1 |
| SIX1 | MA1118.1 | Homeo domain factors | HD-SINE factors | PLK1, CKS1B |
| Stat6 | MA0520.1 | STAT domain factors | STAT factors | MCM2, CKS1B |
Примечание: представлены факторы с p < 0.05 по критерию Манна–Уитни для числа опухолеспецифических промоторов относительно числа промоторов генов домашнего хозяйства, содержащих данный профиль. Полные результаты сравнения приведены в табл. S1 в дополнительных материалах. Профили узнавания ФТ представлены в табл. S2 и S4 в дополнительных материалах. * Профили, найденные в четырех и более опухолеспецифических промоторах.
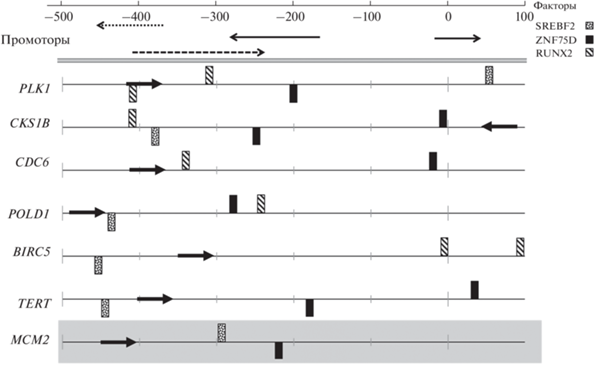
Из семи промоторов более других насыщены профилями ФТ этой группы промоторы MCM2 (13 профилей), CKS1B (10 профилей) и PLK1 (9 профилей) (табл. S3 в дополнительных материалах). Более чем в половине опухолеспецифических промоторов найдены профили узнавания факторов SREBF2, ZNF75D, Zfx (каждый в шести промоторах), RUNX2 и ETS2 (каждый в пяти промоторах), Creb3l2 (в четырех промоторах) (табл. 2). Для трех ФТ удалось выявить преимущественные зоны расположения в промоторах (рис. 1). Так, профили SREBF2 в четырех из семи промоторов расположены в области [–458; –384] относительно TSS на некодирующей цепи ДНК. Профили ZNF75D в четырех промоторах занимают область [–253; –184] относительно TSS также на некодирующей цепи ДНК и в трех промоторах вблизи TSS [–24; +30] на кодирующей цепи. Профили RUNX2 в четырех промоторах располагаются на кодирующей цепи в области [–414; –247].
Рис. 1.
Расположение профилей узнавания факторов транскрипции SREBF2, ZNF75D и RUNX2 в промоторах. Точки начала транскрипции (0) указаны в соответствии с базой эукариотических промоторов EPDnew. Жирные стрелки – Мотив 1.
Взаимодействие факторов транскрипции с промоторами. C помощью ресурса Pathway Сommons [17] мы построили сеть прямых регуляторных взаимодействий выявленных ФТ с продуктами исследованных опухолеспецифических генов (рис. 2). На схеме присутствует антиген пролиферирующих клеток PCNA, поскольку он взаимодействует с продуктами генов, промоторы которых мы исследовали. PCNA участвует в регуляции пролиферации клеток и обладает выраженной опухолеспецифической экспрессией [18]. Однако клонированный нами промотор PCNA [19] был активен как в опухолевых, так и в нормальных клетках [20], поэтому изначально он не был включен в дифференциальный анализ промоторов. В настоящей работе промотор PCNA (FP026732) рассматривается как неспецифический.
Рис. 2.
Прямые регуляторные взаимодействия между продуктами исследованных генов (в черных кружках) и факторами транскрипции (Pathway Commons): (а) – для условно опухолеспецифических факторов транскрипции; (б) – для условно неспецифических факторов транскрипции. Штрихпунктирные стрелки – регуляция экспрессии, штриховые стрелки – модификации, сплошная стрелка – контроль транспорта, штриховые линии без стрелок – связывание в комплекс.
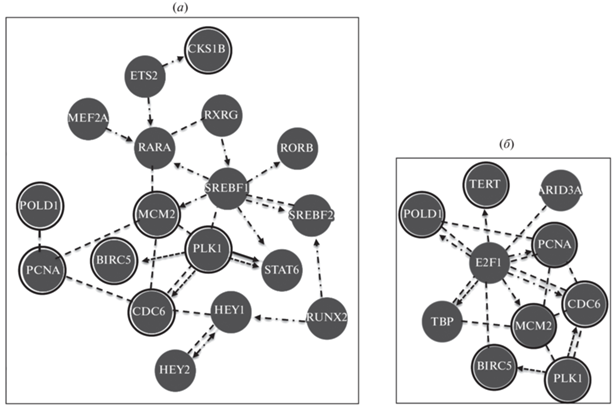
Как видно на рис. 2а, MCM2 и PLK1 занимают центральное положение в данной регуляторной сети, что согласуется с наличием в их промоторах большого количества профилей условно опухолеспецифических ФТ. Факторы Creb3l2, CENPB, SIX1, ZNF75D, Zfx, HOXD11 и KLF11 в регуляторную сеть не вошли, хотя их профили узнавания присутствуют в опухолеспецифических промоторах значительно чаще, чем в промоторах генов домашнего хозяйства. Прямых данных о том, участвуют ли эти семь ФТ в регуляции исследованных промоторов, нет. Однако их отбор не кажется случайным, поскольку эти ФТ, согласно базе данных GEPIA2, демонстрируют дифференциальную экспрессию либо ассоциированы с определенным прогнозом в ряде опухолей (табл. S2 в дополнительных материалах) и, следовательно, могут играть важные роли в канцерогенезе.
Неспецифические факторы транскрипции. Дополнительно мы определили профили ФТ, которые чаще встречаются в промоторах генов домашнего хозяйства по сравнению с опухолеспецифическими промоторами (табл. S4 и S5 в дополнительных материалах, далее – условно неспецифические факторы). Следует отметить, что эти факторы также проявляют дифференциальную экспрессию и могут служить маркерами прогноза при некоторых опухолях. В то же время шесть из семи ФТ этой группы участвуют в развитии и дифференцировке клеток различного типа (Mafb [21], Arid3a [22], MEIS3 [23], BHLHA15 [24], BSX [25], E2F1 [26]). Фактор TBP имеет отношение к общим механизмам транскрипции [27]. Согласно Pathway Commons, только TBP и E2F1 напрямую взаимодействуют с продуктами исследованных опухолеспецифических генов (рис. 2б).
Поиск нуклеотидных мотивов de novo. Для улучшения поиска регуляторных последовательностей ДНК, характерных для опухолеспецифических промоторов, мы провели поиск мотивов de novo с помощью программы MEME Suite [28].
Мы провели дискриминационный поиск нуклеотидных мотивов в семи опухолеспецифических промоторах по отношению к промоторам генов домашнего хозяйства. Таким способом был выявлен Мотив 1 из 44 п.н., который присутствует во всех опухолеспецифических промоторах (рис. S2 в дополнительных материалах). Согласно тесту GOMO, функции Мотива 1 связаны с активностью обонятельных рецепторов, сенсорным восприятием запаха и сигнальным путем рецепторного белка, связанного с G-белком (G-protein coupled receptor protein signaling pathway). В шести из семи исследуемых промоторов Мотив 1 расположен приблизительно в области [–490; –210] по отношению к TSS. В промоторе CKS1B Мотив 1 расположен в области [–47; –4] в обратном направлении по отношению к ориентации промотора. Это может быть связано с двунаправленной природой промотора CKS1B/SHC1. Можно предположить, что Мотив 1 состоит из трех более коротких субмотивов. Поэтому мы проанализировали эти субмотивы отдельно, используя программу Tomtom из MEME Suite и базу данных ФТ позвоночных (табл. S6 в дополнительных материалах).
Так, субмотив 1a содержит профили узнавания энхансерных факторов миоцитов MEF2, которые могут активировать гены, индуцированные факторами роста и стрессом, и участвуют как в супрессии, так и в прогрессии рака в различных условиях [29]; профиль фактора forkhead E1, ассоциированного с раком щитовидной железы [30], и профиль транскрипционного фактора POU homeobox, который относится к главным (master) регуляторам мелкоклеточного рака легких [31]. Фактор NR2E1 также важен в канцерогенезе, т.к. он, в частности, связан с метастазированием рака молочной железы [32]. PHOX2B был определен как ключевой регулятор дифференцировки и поддержания стволовых свойств нейробластомы [33]. Sp100 представляет собой компонент ядерных телец промиелоцитарной лейкемии (PML) [34].
Субмотив 1b содержит профиль узнавания ретиноидных X-рецепторов (RXRs) и рецепторов ретиноевой кислоты (RARs) – ядерных рецепторов, которые опосредуют биологические эффекты ретиноидов, связанные с развитием опухолей [35]. Активация гена DMRT1 обнаружена при герминогенной неоплазии яичка [36]. Интерферон-регулирующий фактор 3 (IFN3) вовлечен в такие важные процессы, как противораковый иммунитет и устойчивость к некоторым бактериальным и вирусным инфекциям [37].
Субмотив 1c включает последовательность, отвечающую на cAMP (cAMP-response element) и способную связывать ФТ семейства CREB3, которые регулируют пролиферацию и миграцию раковых клеток [38], а также профиль фактора ATF6, участвующего в ответе на разворачивание белков (unfolded protein response), причем сверхэкспрессия ATF6 коррелирует с агрессивностью опухолей [39]. ZNF135 относится к мишеням регуляции дифференциально экспрессирующимися микроРНК в карциноме носоглотки [40].
Конечно, в составе Мотива 1 могут быть выявлены профили узнавания других ФТ, кроме перечисленных, а также неизвестных ФТ, по причине вырожденности. Тем не менее наши данные позволяют предположить, что Мотив 1 может быть характерен и для других опухолеспецифических промоторов.
Мы попытались найти другие промоторы, содержащие Мотив 1. Для этого мы просканировали 29 598 промоторов человека из базы данных промоторов EPDnew (все в координатах [–499; +100] относительно TSS) с помощью программы FIMO из пакета MEME Suite. Были найдены 4733 последовательности в обеих ориентациях с p < 0.0001, включая все семь исследуемых промоторов. Десять промоторов, содержащих наиболее близкие к Мотиву 1 последовательности, представлены в табл. 3. По данным The Human Protein Atlas, девять (90%) соответствующих им генов – прогностические маркеры опухолей (в основном, неблагоприятные). Примечательно, что расположение Мотива 1 в приведенных промоторах относительно TSS примерно соответствует таковому в семи исследованных опухолеспецифических промоторах.
Таблица 3.
Десять промоторов, наиболее близких к Мотиву 1, из базы данных EPDnew
| № | Индекс EPDnew | Промотор гена | Старт | Стоп | Цепь | Оценка (score) | p-Value | q-Value | Прогностическое значение в опухолях* |
|---|---|---|---|---|---|---|---|---|---|
| 1 | FP024000 | BIRC5_1 | –357 | –314 | + | 48.9817 | 2,22E-19 | 7,32E-12 | Рак почки (–), печени (–), легких (–) |
| 2 | FP020135 | CDCA4_2 | –437 | –394 | – | 48.4404 | 5,93E-19 | 9,78E-12 | Рак почки (–), печени (–), поджелудочной железы (–), легких (–) |
| 3 | FP014177 | WDR37_1 | –423 | –380 | + | 48.0183 | 1,24E-18 | 1,35E-11 | Рак поджелудочной железы (+), щитовидной железы (–), головы и шеи (+) |
| 4 | FP025645 | YIF1B_2 | –487 | –444 | – | 47.6055 | 2,45E-18 | 1,35E-11 | Рак печени (–), поджелудочной железы (+) |
| 5 | FP027891 | RSPH14_1 | –478 | –435 | – | 47.6055 | 2,45E-18 | 1,35E-11 | Рак почки (+) |
| 6 | FP011458 | SRRT_2 | –403 | –360 | + | 47.6055 | 2,45E-18 | 1,35E-11 | Рак почки (–), печени (–) |
| 7 | FP003432 | PLEK_3 | –470 | –427 | + | 47.4495 | 3,21E-18 | 1,35E-11 | Рак почки (–) |
| 8 | FP013453 | S1PR3_2 | –448 | –405 | – | 47.4404 | 3,28E-18 | 1,35E-11 | Рак почки (–), толстой и прямой кишки (–), молочной железы (+) |
| 9 | FP026025 | C5AR1_1 | –482 | –439 | – | 47.1927 | 4,74E-18 | 1,62E-11 | Рак почки (–), шейки матки (+), яичка (–), яичника (–) |
| 10 | FP009477 | MSH5-SAPCD1_5 | –300 | –257 | + | 47.156 | 5,2E-18 | 1,62E-11 | НП |
Примечание: старт, стоп – положение Мотива 1 относительно точки начала транскрипции; score – оценка соответствия последовательности промотора заданному мотиву; p-value – вероятность того, что случайная последовательность такой же длины соответствует заданному мотиву с таким же или лучшим результатом; q-value – коэффициент ложных обнаружений, если встречаемость мотива принимается значимой (FIMO). * Прогностические маркеры в опухолях согласно базе данных The Human Protein Atlas: (+) – благоприятный прогноз, (–) – неблагоприятный прогноз, НП – нет связи с прогнозом.
Наиболее близкими к Мотиву 1 из семи исследованных промоторов оказались промоторы MCM2 и BIRC5. Отдельный анализ мотивов de novo в этих двух промоторах с помощью MEME Suite выявил консенсусный Мотив 2 длиной 184 п.н. в координатах [–579; –396] для MCM2 и [–496; –303] для BIRC5 относительно TSS (рис. S3 в дополнительных материалах). Интересно, что область сходства между промоторами MCM2 и BIRC5 заметно шире, она охватывает 296 п.н. и имеет одинаковый порядок более коротких мотивов. Мотив, похожий на Мотив 2, может быть выявлен и в других пяти промоторах с помощью FIMO, но с очень низкими оценками (данные не приведены). Поэтому мы считаем такое сходство особенностью промоторов MCM2 и BIRC5.
Анализ химерных промоторов. Мы исследовали химерные промоторы, которые обладали большей активностью в клетках эпидермоидной карциномы A431, чем в нормальных фибробластах (рис. S4 в дополнительных материалах) [13]. C помощью программы CiiiDER мы провели поиск условно опухолеспецифических и неспецифических профилей ФТ в химерных промоторах, а также в неспецифических промоторах CMV и PCNA. Профили Creb3l2, ETS2, HEY1, HEY2 и SREBF1 (var. 2) обнаружены только в химерных промоторах, обладающих повышенной активностью в клетках А431 (CH2, CH20, CH26, рис. S4 в дополнительных материалах), но отсутствуют в неспецифических промоторах CH10, CMV, PCNA (табл. S7 в дополнительных материалах). Все профили условно неспецифических ФТ, преобладающие в промоторах генов домашнего хозяйства (табл. S5 в дополнительных материалах), встречаются как минимум в одном из трех неспецифических промоторов (табл. S8 в дополнительных материалах).
Хотя это наблюдение вряд ли можно оценить статистически, мы склонны рассматривать его как тенденцию, которая подтверждает наши выводы, но требует дополнительной проверки. Только два химерных промотора, CH8 и CH16, содержат Мотив 1 длиной 44 п.н. в составе промоторного фрагмента POLD1 (данные не представлены).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Накопленные данные о структуре и функциях промоторов уже открывают большие перспективы для применения промоторов в практической медицине, например, в составе генно-инженерных векторов для генной терапии опухолей [7, 8]. Завершены клинические исследования генно-терапевтических конструкций, содержащих промоторы вирусов и человека (ClinicalTrials.gov Identifier: NCT01455259, NCT00891748, NCT00197522, NCT00051480; см. обзор Ginn et al. [8] и др.). Кроме генной терапии рака, природные промоторы могут использоваться в генно-инженерных векторах для лечения других заболеваний, в промышленности и различных биотехнологических процессах. Большой интерес к промоторам возобновился в последнее время в связи с необходимостью создания вакцинных векторов для профилактики или терапии инфекционных заболеваний и рака [9, 10]. Разрабатываются подходы с конструированием гибридных промоторов [41, 42] и химерных промоторов [13]. Поэтому исследование первичной структуры промоторов, в частности опухолеспецифических промоторов, остается актуальным.
В данной работе мы исследовали семь опухолеспецифических промоторов (табл. 1) по содержанию профилей узнавания известных ФТ. Несмотря на то что в регуляции транскрипции участвуют как прилежащие к TSS последовательности ДНК, так и отдаленные, было показано, что определенные регуляторные функции сохраняются в относительно коротких проксимальных промоторах, представляющих собой минимальные (core) промоторы с прилегающими цис-регуляторными элементами, общей длиной в несколько сотен нуклеотидов [43]. Это справедливо и для исследованных промоторов, в которых многие важные регуляторные элементы сосредоточены на участке приблизительно до –500 п.н. от TSS [12, 14, 15]. Такой размер промоторов удобен для генно-инженерных манипуляций с ними.
В ходе сравнения промоторов мы выявили 17 профилей узнавания ФТ, более частых в опухолеспецифических промоторах, чем в промоторах генов домашнего хозяйства (условно опухолеспецифические ФТ, табл. 2). В табл. S2 (см. дополнительные материалы) приведены профили узнавания данных факторов в соответствии с базой данных JASPAR [5]. Профили факторов SREBF2, ZNF75D, Zfx, RUNX2, ETS2 и Creb3l2 встречались более чем в половине опухолеспецифических промоторов. Остальные из 17 ФТ встречались в некоторых опухолеспецифических промоторах, но ни в одном из 23 промоторов группы сравнения. Нам также удалось определить преимущественные зоны расположения профилей узнавания факторов SREBF2, ZNF75D и RUNX2 в опухолеспецифических промоторах (рис. 1). Следует отметить, что промоторы CKS1B и PLK1, содержащие, соответственно, 10 и 9 профилей ФТ этой группы (табл. S9 в дополнительных материалах), ранее показали наибольшую опухолевую специфичность по сравнению с другими промоторами [12].
Ряд опухолеспецифических промоторов содержит профили узнавания ФТ, об участии которых в регуляции данных промоторов известно недостаточно. К таким факторам относятся Creb3l2, CENPB, SIX1, ZNF75D, HOXD11, Zfx и KLF11.
Возможно, наши результаты послужат доводом в пользу изучения роли данных ФТ в регуляции соответствующих генов.
Также мы выявили семь профилей ФТ, более представленных в промоторах генов домашнего хозяйства, чем в исследуемых промоторах (табл. S4 и S5 в дополнительных материалах). Шесть из семи ФТ этой группы участвуют в развитии и дифференцировке клеток различного типа (см. выше). Дифференцировку клеток часто рассматривают как процесс, обратный опухолевой трансформации, а также предлагают использовать для терапии опухолей [44]. Наши наблюдения указывают на неслучайность отбора ФТ по признаку неспецифичности в данном случае. В то же время дифференциальная экспрессия этих ФТ в некоторых опухолях и их связь с прогнозом (табл. S4 в дополнительных материалах) свидетельствуют о необходимости более глубокого изучения этого вопроса.
Очевидно, что связывание транскрипционного фактора с ДНК зависит не только от последовательности нуклеотидов, но и от многих других параметров – ориентации профиля, соседних последовательностей, взаимодействия данного ФТ с другими регуляторными молекулами и последовательностями и т.д. [43]. Кроме того, условно опухолеспецифические ФТ могут иметь отношение не столько к опухолевой трансформации клеток, сколько к пролиферативным функциям генов, и это различие следует изучить в каждом конкретном случае. Тем не менее полученные данные дают возможность более прицельно исследовать регуляцию промоторов в прямых экспериментах.
С помощью дискриминационного поиска мотивов de novo мы обнаружили последовательность из 44 п.н. (Мотив 1), которая присутствует во всех семи опухолеспецифических промоторах, но не характерна для промоторов генов домашнего хозяйства. В базе данных EPDnew мы нашли группу промоторов с таким мотивом, причем соответствующие им гены – это прогностические маркеры опухолей. Мы полагаем, что по крайней мере некоторые из этих промоторов могут обладать опухолеспецифической активностью.
Мы показали, что два промотора – BIRC5 и MCM2 – содержат очень близкий мотив из 184 п.н. Более того, область сходства между этими двумя промоторами распространяется на 296 п.н. Мы предполагаем, что такое сходство – скорее особое свойство этих двух промоторов, связанное с их регуляцией, чем типичная общая черта опухолеспецифических промоторов. Хотя это наблюдение представляет интерес, оно выходит за рамки нашего исследования.
Отчасти проверкой наших выводов может служить исследование профилей ФТ в химерных промоторах. Пять из 17 профилей условно опухолеспецифических ФТ, а именно профили Creb3l2, ETS2, HEY1, HEY2 и SREBF1 (var. 2), прошли селекцию на клетках A431 и были отобраны в химерных промоторах по признаку опухолевой специфичности [13]. Только два химерных промотора, CH8 и CH16, содержат Мотив 1 длиной 44 п.н., который был найден в семи нативных опухолеспецифических промоторах. В совокупности с другими данными это может означать, что наличие Мотива 1 в промоторе – достаточное, но не необходимое условие для опухолевой специфичности. Очевидно, эти вопросы требуют более глубокого изучения.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Последовательности промоторов человека получены из базы данных EPDnew (Eucaryotic Promoter Database, https://epd.epfl.ch/EPDnew_database.php) [45] в координатах [–499; +100] по отношению к точке начала транскрипции (TSS) (табл. 1). Для поиска и сравнения профилей ФТ в промоторах, поиска мотивов de novo использовали программы CiiiDER [16], MEME Suite [28] (https://meme-suite.org/meme/) с параметрами по умолчанию и базы профилей узнавания транскрипционных факторов JASPAR (http://jaspar.genereg.net/) [5] и Jolma, 2013 [46]. Различия групп промоторов по частоте профилей ФТ оценивали по статистическому критерию Манна–Уитни, при этом в первую очередь учитывали показатели “significance score” и “gene р-value” (CiiiDER). Экспрессия ФТ в опухолях приведена по базе данных GEPIA2 (http://gepia2.cancer-pku.cn/). Прогностическое значение ФТ в опухолях определяли по базе данных The Human Protein Atlas (https://www.proteinatlas.org/) [47]. Для построения сетей регуляторных взаимодействий использовали ресурс Pathway Commons (http://www.pathwaycommons.org/) [17].
ЗАКЛЮЧЕНИЕ
Показано, что исследованные опухолеспецифические промоторы отличаются от неспецифических промоторов генов домашнего хозяйства присутствием профилей узнавания 17 факторов транскрипции, а также мотива длиной 44 п.н., которые могут быть перспективными объектами исследования опухолеспецифической регуляции экспрессии генов. Присутствие данных последовательностей в каком-либо неизвестном промоторе может указывать на его опухолевую специфичность. Мы полагаем, что наши результаты могут способствовать выбору перспективных природных промоторов для использования в генно-инженерных противоопухолевых конструкциях, таких как векторы с генами-убийцами (killer genes) или векторные вакцины. Из семи исследованных промоторов более других насыщены профилями условно опухолеспецифических ФТ промоторы MCM2, CKS1B и PLK1, что согласуется с данными о значительной опухолевой специфичности этих промоторов [12]. Практическое значение могут иметь и некоторые химерные промоторы, полученные нами ранее [13].
Необходимо подчеркнуть, что выводы данной работы сделаны на основании теоретического анализа нуклеотидной последовательности промоторов. Мы рассматривали профили узнавания ФТ только как нуклеотидные мотивы с определенной вариабельностью. Поэтому все заключения относятся только к присутствию таких последовательностей в промоторах. Действительная роль факторов транскрипции в опухолеспецифической регуляции промоторов должна быть исследована в прямых экспериментах. Мы надеемся, что наша работа поможет лучше понять механизмы опухолеспецифической транскрипции генов и откроет новые возможности для создания искусственных промоторов и генно-инженерных векторов, применяемых в генной терапии опухолей.
Список литературы
Zabidi M.A., Stark A. // Trends Genet. 2016. V. 32. P. 801–814. https://doi.org/10.1016/j.tig.2016.10.003
Corso-Diaz X., Jaeger C., Chaitankar V., Swaroop A. // Prog. Retin. Eye Res. 2018. V. 65. P. 1–27. https://doi.org/10.1016/j.preteyeres.2018.03.002
Gurumurthy A., Shen Y., Gunn E.M., Bungert J. // BioEssays. 2019. V. 41. P. e1800164. https://doi.org/10.1002/bies.201800164
Lambert S.A., Jolma A., Campitelli L.F., Das P.K., Yin Y., Albu M., Chen X., Taipale J., Hughes T.R., Weirauch M.T. // Cell. 2018. V. 172. P. 650–665. https://doi.org/10.1016/j.cell.2018.01.029
Castro-Mondragon J.A., Riudavets-Puig R., Rauluseviciute I., Berhanu Lemma R., Turchi L., Blanc-Mathieu R., Lucas J., Boddie P., Khan A., Manosalva Pérez N., Fornes O., Leung T.Y., Aguirre A., Hammal F., Schmelter D., Baranasic D., Ballester B., Sandelin A., Lenhard B., Vandepoele K., Wasserman W.W., Parcy F., Mathelier A. // Nucleic Acids Res. 2022. V. 50. P. D165–D173. https://doi.org/10.1093/nar/gkab1113
Moll R. // Der Pathologe. 2018. V. 39. P. 285–290. https://doi.org/10.1007/s00292-018-0518-7
Alekseenko I.V., Snezhkov E.V., Chernov I.P., Pleshkan V.V., Potapov V.K., Sass A.V., Monastyrskaya G.S., Kopantzev E.P., Vinogradova T.V., Khramtsov Y.V., Ulasov A.V., Rosenkranz A.A., Sobolev A.S., Bezborodova O.A., Plyutinskaya A.D., Nemtsova E.R., Yakubovskaya R.I., Sverdlov E.D. // J. Transl. Med. 2015. V. 13. P. 78. https://doi.org/10.1186/s12967-015-0433-0
Ginn S.L., Amaya A.K., Alexander I.E., Edelstein M., Abedi M.R. // J. Gene Med. 2018. V. 20. P. e3015. https://doi.org/10.1002/jgm.3015
Watanabe M., Nishikawaji Y., Kawakami H., Kosai K.I. // Viruses. 2021. V. 13. P. 2502. https://doi.org/10.3390/v13122502
Morse M.A., Gwin W.R., 3rd, Mitchell D.A. // Target Oncol. 2021. V. 16. P. 121–152. https://doi.org/10.1007/s11523-020-00788-w
Alekseenko I.V., Pleshkan V.V., Kopantzev E.P., Stukacheva E.A., Chernov I.P., Vinogradova T.V., Sverdlov E.D. // PLoS One. 2012. V. 7. P. e46474. https://doi.org/10.1371/journal.pone.0046474
Kashkin K., Chernov I., Stukacheva E., Monastyrskaya G., Uspenskaya N., Kopantzev E., Sverdlov E. // J. Cell. Biochem. 2015. V. 116. P. 299–309. https://doi.org/10.1002/jcb.24968
Kashkin K.N., Chernov I.P., Didych D.A., Sverdlov E.D. // BioTechniques. 2017. V. 63. P. 107–116. https://doi.org/10.2144/000114586
Takakura M., Kyo S., Kanaya T., Hirano H., Takeda J., Yutsudo M., Inoue M. // Cancer Res. 1999. V. 59. P. 551–557.
Xu R., Zhang P., Huang J., Ge S., Lu J., Qian G. // Biochem. Biophys. Res. Commun. 2007. V. 356. P. 286–292. https://doi.org/10.1016/j.bbrc.2007.02.140
Gearing L.J., Cumming H.E., Chapman R., Finkel A.M., Woodhouse I.B., Luu K., Gould J.A., Forster S.C., Hertzog P.J. // PLoS One. 2019. V. 14. P. e0215495. https://doi.org/10.1371/journal.pone.0215495
Rodchenkov I., Babur O., Luna A., Aksoy B.A., Wong J.V., Fong D., Franz M., Siper M.C., Cheung M., Wrana M., Mistry H., Mosier L., Dlin J., Wen Q., O’Callaghan C., Li W., Elder G., Smith P. T., Dallago C., Cerami E., Gross B., Dogrusoz U., Demir E., Bader G.D., Sander C. // Nucleic Acids Res. 2020. V. 48. P. D489–D497. https://doi.org/10.1093/nar/gkz946
Tang Z., Kang B., Li C., Chen T., Zhang Z. // Nucleic Acids Res. 2019. V. 47. P. W556–W560. https://doi.org/10.1093/nar/gkz430
Свердлов Е.Д., Алексеенко И.В., Безбородова О.А., Виноградова Т.В., Кашкин К.Н., Копанцев Е.П., Монастырская Г.С., Немцова Е.Р., Успенская Н.Я., Чернов И.П., Якубовская Р.И. // Патент RU 2551784 С1, 2015.
Кондратьева Л.Г., Кашкин К.Н., Чернов И.П., Стукачева Е.А., Дидыч Д.А., Копанцев Е.П., Свердлов Е.Д. // Молекулярная генетика, микробиология и вирусология. 2017. Т. 3. С. 89–92. [Kondratyeva L.G., Kashkin K.N., Chernov I.P., Stukacheva E.A., Dydich D.A., Kopantzev E.P., Sverdlov E.D. // Mol. Genet. Microbiol. Virol. 2017. V. 3. P. 137–140]. https://doi.org/10.3103/S089141681703003X
Miyai M., Hamada M., Moriguchi T., Hiruma J., Kamitani-Kawamoto A., Watanabe H., Hara-Chikuma M., Takahashi K., Takahashi S., Kataoka K. // J. Invest. Dermatol. 2016. V. 136. P. 1848–1857. https://doi.org/10.1016/j.jid.2016.05.088
Popowski M., Lee B.K., Rhee C., Iyer V.R., Tucker H.O. // J. Stem Cell Ther. Transplant. 2017. V. 1. P. 52–62. https://doi.org/10.29328/journal.jsctt.1001005
Elkouby Y.M., Polevoy H., Gutkovich Y.E., Michaelov A., Frank D. // Development. 2012. V. 139. P. 1487–1497. https://doi.org/10.1242/dev.072934
Dekaney C.M., King S., Sheahan B., Cortes J.E. // Cell. Mol. Gastroenterol. Hepatol. 2019. V. 8. P. 549–560. https://doi.org/10.1016/j.jcmgh.2019.07.003
Schredelseker T., Driever W. // Development. 2018. V. 145. P. dev163477. https://doi.org/10.1242/dev.163477
Xu X., Zhang T., Zhang M., Li L., Deng G., Lu Z., Zhang Z., Du Y., Feng Y., Feng X., Peng X., Chen F. // Cell. Signal. 2022. V. 93. P. 110280. https://doi.org/10.1016/j.cellsig.2022.110280
Kramm K., Engel C., Grohmann D. // Biochem. Soc. Trans. 2019. V. 47. P. 411–423. https://doi.org/10.1042/bst20180623
Bailey T.L., Johnson J., Grant C.E., Noble W.S. // Nucleic Acids Res. 2015. V. 43. P. W39–W49. https://doi.org/10.1093/nar/gkv416
Di Giorgio E., Hancock W.W., Brancolini C. // Biochim. Biophys. Acta Rev. Cancer. 2018. V. 1870. P. 261–273. https://doi.org/10.1016/j.bbcan.2018.05.007
Credendino S.C., Moccia C., Amendola E., D’Avino G., Di Guida L., Clery E., Greco A., Bellevicine C., Brunetti A., De Felice M., De Vita G. // Int. J. Mol. Sci. 2020. V. 22. P. 25. https://doi.org/10.3390/ijms22010025
Huang Y.H., Klingbeil O., He X.Y., Wu X.S., Arun G., Lu B., Somerville T.D.D., Milazzo J.P., Wilkinson J.E., Demerdash O.E., Spector D.L., Egeblad M., Shi J., Vakoc C.R. // Genes Dev. 2018. V. 32. P. 915–928. https://doi.org/10.1101/gad.314815.118
Park S.B., Hwang K.T., Chung C.K., Roy D., Yoo C. // Clin. Exp. Metastasis. 2020. V. 37. P. 657–674. https://doi.org/10.1007/s10585-020-10060-0
Yang L., Ke X.X., Xuan F., Tan J., Hou J., Wang M., Cui H., Zhang Y. // Cancer Biother. Radiopharm. 2016. V. 31. P. 44–51. https://doi.org/10.1089/cbr.2015.1952
Guion L.G., Sapp M. // Front Cell. Infect. Microbiol. 2020. V. 10. P. 35. https://doi.org/10.3389/fcimb.2020.00035
le Maire A., Alvarez S., Shankaranarayanan P., Lera A.R., Bourguet W., Gronemeyer H. // Curr. Top. Med. Chem. 2012. V. 12. P. 505–527. https://doi.org/10.2174/156802612799436687
Al-Hussain T., Bakshi N., Akhtar M. // Adv. Anat. Pathol. 2015. V. 22. P. 202–212. https://doi.org/10.1097/pap.0000000000000066
Petro T.M. // J. Immunol. 2020. V. 205. P. 1981–1989. https://doi.org/10.4049/jimmunol.2000462
Kim H.C., Choi K.C., Choi H.K., Kang H.B., Kim M.J., Lee Y.H., Lee O.H., Lee J., Kim Y.J., Jun W., Jeong J.W., Yoon H.G. // Cell. Mol. Life Sci. 2010. V. 67. P. 3499–3510. https://doi.org/10.1007/s00018-010-0388-5
Urra H., Dufey E., Avril T., Chevet E., Hetz C. // Trends Cancer. 2016. V. 2. P. 252–262. https://doi.org/10.1016/j.trecan.2016.03.007
Xi T., Zhang G. // Pathol. Res. Pract. 2017. V. 213. P. 83–88. https://doi.org/10.1016/j.prp.2016.12.007
Алексеенко И.В., Плешкан В.В., Сасс А.В., Филюкова О.Б., Снежков Е.В., Свердлов Е.Д. // Доклады Российской академии наук (Биохимия, Биофизика). 2018. Т. 480. С. 609–612. [Alekseenko I.V., Pleshkan V.V., Sass A.V., Filyukova O.B., Snezhkov E.V., Sverdlov E.D. // Dokl. Biochem. Biophys. 2018. V. 480. P. 158–161.] https://doi.org/10.1134/s1607672918030092
Farokhimanesh S., Rahbarizadeh F., Rasaee M.J., Kamali A., Mashkani B. // Biotechnol. Prog. 2010. V. 26. P. 505–511.
Haberle V., Stark A. // Nat. Rev. Mol. Cell Biol. 2018. V. 19. P. 621–637. https://doi.org/10.1038/s41580-018-0028-8
Prasad S., Ramachandran S., Gupta N., Kaushik I., Srivastava S.K. // Biochim. Biophys. Acta Mol. Basis Dis. 2020. V. 1866. P. 165424. https://doi.org/10.1016/j.bbadis.2019.02.019
Dreos R., Ambrosini G., Groux R., Cavin Perier R., Bucher P. // Nucleic Acids Res. 2017. V. 45. P. D51–D55. https://doi.org/10.1093/nar/gkw1069
Jolma A., Yan J., Whitington T., Toivonen J., Nitta K.R., Rastas P., Morgunova E., Enge M., Taipale M., Wei G., Palin K., Vaquerizas J.M., Vincentelli R., Luscombe N.M., Hughes T.R., Lemaire P., Ukkonen E., Kivioja T., Taipale J. // Cell. 2013. V. 152. P. 327–339. https://doi.org/10.1016/j.cell.2012.12.009
Sjöstedt E., Zhong W., Fagerberg L., Karlsson M., Mitsios N., Adori C., Oksvold P., Edfors F., Limiszewska A., Hikmet F., Huang J., Du Y., Lin L., Dong Z., Yang L., Liu X., Jiang H., Xu X., Wang J., Yang H., Bolund L., Mardinoglu A., Zhang C., von Feilitzen K., Lindskog C., Pontén F., Luo Y., Hökfelt T., Uhlén M., Mulder J. // Science. 2020. V. 367. P. eaay5947. https://doi.org/10.1126/science.aay5947
Дополнительные материалы
- скачать ESM_1.csv
- Приложение 1.
Таблица S1.
- скачать ESM_2.docx
- Приложение 2.
Рис. S1. - Рис. S4.
Таблица S2. - Таблица S8.
Инструменты
Биоорганическая химия


