Биоорганическая химия, 2023, T. 49, № 1, стр. 23-31
Белки холодового шока бактерий как инструмент адаптации к стрессу
А. С. Григоров 1, *, Т. Л. Ажикина 1
1 ФГБУН “Институт биоорганической химии им. М.М. Шемякина и Ю.А. Овчинникова” РАН
117997 Москва, ул. Миклухо-Маклая, 16/10, Россия
* E-mail: artgrigorov@gmail.com
Поступила в редакцию 20.06.2022
После доработки 26.07.2022
Принята к публикации 12.08.2022
- EDN: GFEUFH
- DOI: 10.31857/S0132342323010104
Аннотация
Бактерии выработали ряд механизмов, позволяющих справляться со стрессом и приспосабливаться к изменяющимся условиям окружающей среды. Семейство бактериальных белков, содержащих функциональный домен холодового шока, – высококонсервативные белки, связывающие нуклеиновые кислоты, модулирующие транскрипцию и посттранскрипционные события у бактерий. Для многих бактерий показано, что белки холодового шока регулируют экспрессию различных генов, вовлеченных в процессы вирулентности и устойчивости бактерий к стрессам. В обзоре освещены новые данные о механизмах действия и роли белков холодового шока в регуляции экспрессии у внутриклеточных бактериальных патогенов.
СОДЕРЖАНИЕ
ВВЕДЕНИЕ........................................................23
БЕЛКИ ХОЛОДОВОГО ШОКА КАК РЕГУЛЯТОРЫ ЭКСПРЕССИИ ГЕНОВ...........24
БЕЛКИ ХОЛОДОВОГО ШОКА И ВИРУЛЕНТНОСТЬ........................................27
БЕЛКИ ХОЛОДОВОГО ШОКА – ФУНКЦИОНАЛЬНЫЕ ГОМОЛОГИ Hfq?.......29
ЗАКЛЮЧЕНИЕ..................................................29
ВВЕДЕНИЕ
РНК-связывающие белки (RBP) – один из важных компонентов, участвующих в регуляции транскрипции у бактерий и позволяющих бактериям адаптироваться к стрессовым и меняющимся условиям. Один из самых известных примеров бактериальных РНК-связывающих белков – белок-шаперон Hfq, главная функция которого – опосредование взаимодействия между мРНК и малыми некодирующими РНК (нкРНК) бактерий [1, 2]. Ранее считалось, что это основной механизм, благодаря которому происходит взаимодействие нкРНК с их мишенями, однако позже стало понятно, что в разных видах бактерий роль Hfq могут играть и другие белки, например, обнаруженный недавно белок ProQ [3, 4]. Другая хорошо изученная категория RBP, регулирующих адаптивные реакции бактерий, – это белки холодового шока (cold shock protein, CSP). CSP представляют собой семейство небольших белков, высококонсервативных как по последовательности, так и по структуре. Эти ДНК- и РНК-связывающие шапероны содержат домены холодового шока (CSD) [5] и широко распространены среди всех живых организмов.
Структура CSD достаточно проста. Он имеет небольшой размер (67–73 а.о.) и представляет собой β-бочонок, состоящий из пяти антипараллельных β-тяжей (рис. 1). Связывание нуклеиновых кислот происходит за счет стэкинга между нуклеотидными основаниями и ароматическими аминокислотными остатками, расположенными на положительно заряженной поверхности, образованной β-цепями [6, 7]. Такие взаимодействия объясняют малую селективность связывания.
CSP наиболее широко изучены у Escherichia coli, именно для кишечной палочки впервые было показано накопление одного из белков (названного позднее CspA) до 13% от тотальной белковой массы при холодовом стрессе [9]. Всего у E. coli описаны девять белков холодового шока, экспрессия четырех из них (CspA, CspB, CspG, CspI) индуцируется низкими температурами [8], одного (CspD) – голоданием и в ранней стационарной фазе, два экспрессируются на постоянном уровне (CspC и CspE) [10]. Триггеры экспрессии CspF и CspH до сих пор неизвестны.
Гомологичные белки классифицируются как CSP у многих других видов бактерий. Yu et al. на основании филогенетического расстояния между нуклеотидными последовательностями кластеризовали гены CSP эубактерий на 5 различных клад и 12 субклад [11]. Проведенный эволюционный анализ показал, что общий предок генов белков холодового шока существовал >3.5 млрд лет назад, что указывает на исключительно важную роль этих белков в развитии жизни.
В последнее время широко обсуждаются другие роли CSP, не связанные с адаптацией к холодовому стрессу. Показано участие этих белков в процессах формирования биопленок, контроле подвижности бактерий и механизмах вирулентности у патогенных микроорганизмов. Активно исследуется вклад некоторых бактериальных CSP в механизмы инфекции у таких патогенов, как Salmonella enterica, Listeria monocytogenes и Staphylococcus aureus (примеры будут рассмотрены ниже).
БЕЛКИ ХОЛОДОВОГО ШОКА КАК РЕГУЛЯТОРЫ ЭКСПРЕССИИ ГЕНОВ
CspA действует как шаперон, предотвращая формирование вторичных структур РНК при низких температурах [12]. В работе Rennella et al. [7] сообщается, что CspA E. coli предпочтительно связывает одноцепочечные богатые пиримидиновыми нуклеотидами последовательности мРНК, но также взаимодействует с двуцепочечными участками РНК. CspA активирует транскрипцию как минимум двух генов, участвующих в адаптации к низким температурам, – hns [13] и gyrA [14]. CspA может непосредственно участвовать в транскрипции генов как белок-антитерминатор, предотвращая преждевременную терминацию транскриптов [15]. CspA также подавляет собственную экспрессию, образую петлю обратной связи [16]. Образованные при низких температурах высокоструктурированные участки РНК дестабилизируются в результате связывания с CSP, что может способствовать их деградации с помощью РНКаз [12].
В табл. 1 схематично представлены основные молекулярные процессы с бактериальными CSP в качестве ключевых участников.
Таблица 1.
Белки холодового шока бактерий как регуляторы экспрессии генов у бактерий
| Процесс | Cхема | Примеры |
|---|---|---|
| Активация транскрипции за счет связывания с регуляторными участками ДНК | 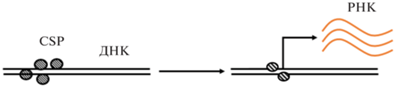 |
Xanthomonas oryzae pv. oryzae [17], Escherichia coli [14, 18] |
| Транскрипционная антитерминация | 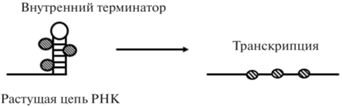 |
Escherichia coli [15], Thermotoga maritima [19] |
| Модуляция трансляции путем дестабилизации двухцепочечных участков РНК в регуляторных
и кодирующих районах (как при низких температурах, так и в нормальных условиях) |
 |
Staphylococcus aureus [20], Escherichia coli [21], Salmonella enterica [22] |
| Неселективное ингибирование трансляции при низких температурах и высоких концентрациях CSP за счет неспецифического связывания мРНК | 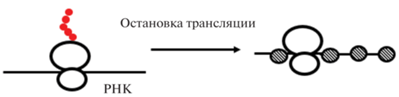 |
Эксперименты in vitro [23] |
| Ускорение терминации транскрипции |  |
Bacillus subtilis [24] |
| Регуляция деградации РНК за счет конформационных изменений, приводящих к формированию или экранированию участков гидролиза РНКаз |  |
Salmonella enterica [25], Staphylococcus aureus [20], Escherichia coli [12] |
| Взаимодействие с нкРНК |  |
Staphylococcus aureus [20], Bacteroides spp. [26] |
| Взаимодействие с деградосомой | 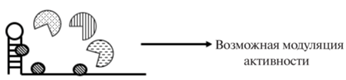 |
Mycobacterium tuberculosis [27], Escherichia coli [28] |
БЕЛКИ ХОЛОДОВОГО ШОКА И ВИРУЛЕНТНОСТЬ
Роль белков холодового шока у бактерий с момента их открытия связывалась с адаптацией к изменяющимся условиям внешней среды и вирулентностью. Для Listeria monocytogenes, патогенной для человека бактерии, которая продуцирует три CSP (CspA, CspB и CspD), было показано, что экспрессия этих белков необходима для выживания в условиях пониженной температуры, окислительного и осмотического стресса [29, 30]. Делеция гена cspD приводит к сильному снижению вирулентных свойств штамма (аттенуации) в условиях холода. Делеция cspA полностью останавливает бактериальный рост при холодовом стрессе. CspD также участвует в адаптации бактерий к условиям осмотического стресса [30]. Однако экспрессия белков холодового шока необходима и в оптимальных для роста L. monocytogenes условиях как в культуре in vitro, так и при инвазии клеток хозяина, т.к. они регулируют формирование биопленок и биосинтез белков, входящих в состав жгутиков [31]. У штамма, содержащего делецию генов трех белков ΔcspABD, а также у мутантных штаммов ΔcspBD, ΔcspAD и ΔcspAB было выявлено снижение количества мРНК и соответствующих продуктов генов hly (кодирует формирующий поры фактор вирулентности листериолизин О, LLO), mpl (металлопротеаза) и plcA (фосфолипаза С). Перечисленные белки – факторы, опосредующие выход L. monocytogens из фагосом [32–34].
Также в мутантных штаммах практически отсутствовал белок ActA при повышении уровня мРНК actA по сравнению со штаммом дикого типа, что указывает на участие CSP в посттранскрипционной регуляции этого гена. ActA – многофункциональный вирулентный фактор. С одной стороны, ActA рекрутирует комплексы полимеризации актина, что способствует актин-опосредованной подвижности бактерии [35, 36]. С другой, этот белок ускоряет агрегацию бактерий и участвует в процессах персистирования внутри хозяина и трансмиссии [37].
Фенотипически делеция генов csp сопровождалась аттенуацией штаммов при заражении клеток моноцитов человека THP-1 и эмбрионов Danio rerio, частичной или полной потерей жгутика и снижением подвижности бактерий (у тройного мутанта подвижность вообще не наблюдалась) [31].
Важный фактор хронического бессимптомного носительства Salmonella enterica – колонизация желчного пузыря сальмонеллами. Для адаптации к высоким концентрациям желчи сальмонелла либо образует биопленки на поверхности желчных камней, либо поражает эпителий желчного пузыря [38]. Ray et al. [39] показали, что синтез CspE активируется во время стресса, вызванного солями желчных кислот, а мутантный штамм S. enterica ΔcspE проявляет дозозависимую чувствительность к солям желчных кислот. Авторы выявили новый транскрипционный фактор, кодируемый геном yciF, который активируется в ответ на желчный стресс в присутствии CspE. Эта регуляция обусловлена способностью CspE стимулировать образование одноцепочечной РНК, тем самым защищая мРНК yciF от деградации, которая может быть опосредована РНКазой Е и PNPазой. Показано, что YciF подавляет синтез поринов OmpD, OmpF и OmpC, тем самым снижая проницаемость клеточной стенки и адаптируя бактерии к стрессовым условиям.
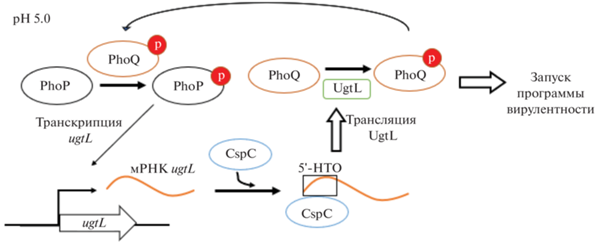
В исследовании белков холодового шока S. enterica был применен транскриптомный подход и проведен сравнительный анализ всех взаимодействующих с белком РНК (таргетомов) разных паралогов Csp [25]. Было показано, что белки CspC и CspE связывают большое число транскриптов, функциональный анализ которых выявил обогащение по категориям генов, связанных с ответом на различные стрессы и вирулентностью. При делеции генов сspC и сspE наблюдалось изменение экспрессии многих генов, входящих в РНК-таргетом этих белков (в том числе генов, связанных с бактериальной подвижностью). Штамм S. enterica, нокаутированный по генам сspC/сspE, становился полностью авирулентным в модели инфекции при заражении мышей линии BALB/c. При этом Choi et al. показали, что несмотря на высокую степень гомологии белков CspC и CspЕ, а также сильно перекрывающийся список их РНК-мишеней, существуют условия, в которых необходим только один из этих белков [22]. Так, при умеренно кислом значении pH, в том числе внутри макрофагов, CspC участвует в активации двухкомпонентной системы PhoP/PhoQ, которая запускает программу вирулентности S. enterica. CspC связывается с лидерной последовательностью мРНК гена ugtL, освобождая последовательность Шайна–Дальгарно, что приводит к инициации трансляции. UgtL критически важен для аутофосфорилирования PhoQ, при его отсутствии не активируется вся система (рис. 2). Авторы также показали, что при делеции гена cspC наблюдается уменьшение вирулентности штамма, что проявляется в снижении способности этого штамма колонизировать внутренние органы при заражении им мышей линии C3H/HeN [22].
Рис. 2.
Схематичное изображение запуска PhoP/PhoQ-системы S. enterica в условиях умеренно кислого pH. CspC усиливает трансляцию гена ugtL, продукт которого необходим для эффективного аутофосфорилирования PhoQ. Адаптировано из статьи Choi et al. [22].
Анализ таргетома белка холодового шока CspA S. aureus также продемонстрировал тот факт, что подавляющее большинство РНК-мишеней вовлечено в реакцию адаптации бактерии в ответ на различные стрессы [20]. Кроме того, в таргетоме CspA найдены транскрипты генов, кодирующие белки, которые участвуют в катаболизме аминокислот, синтезе нуклеозидов и углеводов. При этом CspA способен как положительно влиять на экспрессию своих РНК-мишеней, так и негативно. Интересно, что до сих пор не очень понятно, каким образом CspA узнает свои мишени – консенсусных мотивов в нуклеотидной последовательности РНК обнаружить не удалось, а связывание белка может происходить с любым местом транскрипта.
В бактериях S. aureus CspA (MsaB) связывается с промоторной областью cap-оперона in vitro и активирует экспрессию генов, вовлеченных в образование капсульного полисахарида, что чрезвычайно важно для выживания бактерий в клетках хозяина [40]. CspA, совместно с транскрипционным фактором σβ и белками стафилоксантинового оперона (crtOPQMN), также входит в сложную регуляторную сеть. Стафилоксантин, каротиноидный пигмент, действующий как антиоксидант, – это основной фактор вирулентности золотистого стафилококка, позволяющий бактерии уклоняться от иммунной защиты инфицированного организма. Делеция гена сspA приводит к снижению синтеза стафилоксантина и понижению уровня экспрессии фактора σβ, что снижает уровень резистентности штамма к окислительному стрессу [20]. CspA связывается с мРНК σβ и активирует его экспрессию, но также способен связываться с промотором оперона crtOPQMN [41]. Таким образом, связываясь как с РНК, так и с ДНК, CspA регулирует экспрессию генов, участвующих в различных процессах адаптации, и контролирует патогенность золотистого стафилококка.
Делеция гена cspA бактерии Brucella melitensis вызывает сильные транскриптомные изменения и приводит к аттенуации штамма как in vitro, так и in vivo [42]. В частности, делеция гена сspA вызывает снижение экспрессии ряда генов вирулентности: генов системы секреции IV типа, входящих в virB-оперон, и некоторых генов жгутика [43]. Штамм, нокаутированный по гену сspA, обладает сниженной скоростью роста на минимальной среде, что говорит о важной роли этого гена в регуляции бактериального метаболизма.
Необычен для большинства белков холодового шока механизм работы белка CspA Xanthomonas oryzae pv. oryzae – патогена, вызывающего бактериальный ожог у риса. Wu et al. показали [17], что CspA напрямую подавляет экспрессию двух генов, один из которых кодирует белок хемотаксиса (PXO_RS11830), а другой – белок биосинтеза глюкана (PXO_RS01060), связываясь с последовательностью 5'-CCAAT-3' в области их промоторов. Делеция гена cspA значительно снижает вирулентность штамма, которая восстанавливается только при делеции генов, находящихся под контролем CspA [17].
У Mycobacterium tuberculosis CspA не исследовался так подробно, и сходство его функций и регуляции с гомологами остаются под вопросом. Тем не менее, учитывая важную роль гомологов CspA в других бактериях, есть все основания полагать, что и у M. tuberculosis этот белок выступает глобальным регулятором, контролирующим большое число процессов. В 1999 г. Weldingh и Andersen обнаружили CspA (Rv3648c) в супернатанте культуры M. tuberculosis [44]. В этой же работе было установлено, что CspA вызывает гиперчувствительность замедленного типа.
В одном из последних исследований CspA M. tuberculosis было показано, что этот белок менее структурирован и обладает наименьшей термостабильностью по сравнению с гомологами у других бактерий. Тем не менее белок функционально активен и способен связывать случайные последовательности олигонуклеотидов [45]. CspB M. tuberculosis еще менее изучен, однако интересен тот факт, что он больше похож на Y-белок эукариот, чем на бактериальные белки холодового шока [46].
CspA1 патогенной бактерии Pseudomonas plecoglossicida, экспрессия которого значительно возрастает в условиях холода [47], также управляет экспрессией сигма-фактора sigX, участвующего в процессах вирулентности при пониженной температуре, за счет связывания с промоторной областью его гена [48]. Показано, что гены cspA1 и sigX необходимы для внутриклеточного выживания P. plecoglossicida, а их делеция ухудшает способность бактерии уходить от иммунного ответа. В регулоне sigX находятся гены, модулирующие подвижность бактерии и образование биопленок, делеция sigX приводит к значительному снижению вирулентности [48].
Вовлеченность белков холодового шока в процессы вирулентности была показана и на примере CspD3 бактерии Ralstonia solanacearum, возбудителя бурой гнили картофеля [49]. Продемонстрировано, что CspD3 не участвует в адаптации к холодовому стрессу и формировании биопленок, но его делеция приводит к увеличенной подвижности и существенной аттенуации штамма при заражении табака. Молекулярный механизм, по которому действует CspD3, до сих пор не определен, но было показано, что в штамме, нокаутированном по гену cspD3, снижается экспрессия генов, ассоциированных с вирулентностью по сравнению со штаммом дикого типа. В свою очередь, это может говорить о роли CspD3 как положительного регулятора вирулентности [49].
БЕЛКИ ХОЛОДОВОГО ШОКА – ФУНКЦИОНАЛЬНЫЕ ГОМОЛОГИ Hfq?
Одна из важнейших систем адаптации бактерий к стрессам – регуляция транскрипции малыми нкРНК. Для большинства бактерий такие взаимодействия между мРНК и малыми нкРНК бактерий опосредованы белком-шапероном Hfq или недавно обнаруженным белком ProQ. Однако у некоторых бактерий (все виды микобактерий, Helicobacter pilori) до настоящего момента не выявлены гомологи Hfq, а также не предложены другие РНК-шапероны со схожим механизмом действия. Вопрос необходимости белка-шаперона для посттранскрипционной регуляции экспрессии с помощью нкРНК в таких бактериях остается открытым. Возможно, высокое содержание гуанина и цитозина в геномах микобактерий облегчает взаимодействие между малыми нкРНК и их мишенями, что позволяет обходиться без РНК-шаперона. Тем не менее в последнее время CspA рассматривается как возможный кандидат на эту роль. Он экспрессируется на высоком уровне и достаточно равномерно в разных стрессовых ситуациях. Аминокислотная последовательность CspA M. tuberculosis на 43% идентична домену холодового шока эукариотических белков семейства Y-бокс, которые связываются с нуклеиновыми кислотами и принимают участие в регуляции транскрипции и трансляции [12].
CspA также упоминается в недавнем обзоре микобактериальных малых РНК [50], где отмечается, что этот белок хорошо подходит на роль РНК-шаперона с большим спектром мишеней благодаря своей способности дестабилизировать высокоструктурированные участки РНК, которые особенно характерны для организмов, геном которых обогащен гуанином и цитозином. Интересен факт совместной экспрессии гена cspA с нкРНК ncRv3648c.
В 2021 г. был проведен биоинформатический анализ геномов Bacteroides spp., выступающих основным компонентом микробиоты толстого кишечника млекопитающих [51]. В геномах бактероидов не были выявлены гомологи РНК-связывающих белков Hfq, ProQ и CsrA, однако обнаружен гомолог белка холодового шока BT_1884. Гиперэкспрессия этого белка привела к изменению количеств транскриптов двух нкРНК, что позволило авторам статьи предположить, что BT_1884 может выполнять роль РНК-шаперона [26].
ЗАКЛЮЧЕНИЕ
Белки холодового шока были открыты более 30 лет назад как регуляторы устойчивости бактерий к понижению температуры. Однако в настоящее время очевидно, что их функция не ограничивается приспособлением бактерий к холоду, они – одни из важнейших регуляторов внутриклеточных процессов. Активность многих белков холодового шока не зависит от понижения температуры, а связана с адаптацией к различным стрессам, включающим в том числе и защитные механизмы эукариотических клеток. CSP адаптируют бактериальную клетку к меняющимся условиям, взаимодействуя с ДНК, РНК (как матричными, так и некодирующими), а также, возможно, с другими белками. Воздействие на регуляторный аппарат бактерий может представлять не только научный, но и практический интерес, поскольку позволяет модулировать скорость роста бактерий в широком спектре условий.
Список литературы
Link T.M., Valentin-Hansen P., Brennana R.G. // Proc. Natl. Acad. Sci. USA. 2009. V. 106. P. 19292–19297. https://doi.org/10.1073/PNAS.0908744106
Vogel J., Luisi B.F. // Nat. Rev. Microbiol. 2011. V. 9. P. 578–589. https://doi.org/10.1038/NRMICRO2615
Smirnov A., Förstner K.U., Holmqvist E., Otto A., Günster R., Becher D., Reinhardt R., Vogel J. // Proc. Natl. Acad. Sci. USA. 2016. V. 113. P. 11591–11596. https://doi.org/10.1073/pnas.1609981113
Smirnov A., Wang C., Drewry L.L., Vogel J. // EMBO J. 2017. V. 36. P. 1029–1045. https://doi.org/10.15252/EMBJ.201696127
Ermolenko D.N., Makhatadze G.I. // Cell. Mol. Life Sci. 2002. V. 59. P. 1902–1913. https://doi.org/10.1007/PL00012513
Schindelin H., Jiang W., Inouye M., Heinemann U. // Proc. Natl. Acad. Sci. USA. 1994. V. 91. P. 5119–5123. https://doi.org/10.1073/PNAS.91.11.5119
Rennella E., Sára T., Juen M., Wunderlich C., Imbert L., Solyom Z., Favier A., Ayala I., Weinhäupl K., Schanda P., Konrat R., Kreutz C., Brutscher B. // Nucleic Acids Res. 2017. V. 45. P. 4255–4268. https://doi.org/10.1093/NAR/GKX044
Horn G., Hofweber R., Kremer W., Kalbitzer H.R. // Cell. Mol. Life Sci. 2007. V. 64. P. 1457–1470. https://doi.org/10.1007/S00018-007-6388-4
Goldstein J., Pollitt N.S., Inouye M. // Proc. Natl. Acad. Sci. USA. 1990. V. 87. P. 283–287. https://doi.org/10.1073/PNAS.87.1.283
Yamanaka K., Inouye M. // J. Bacteriology. 1997. V. 179. P. 5126–5130. https://doi.org/10.1128/JB.179.16.5126-5130.1997
Yu T., Keto-Timonen R., Jiang X., Virtanen J.P., Korkeala H. // Int. J. Mol. Sci. 2019. V. 20. P. 4059. https://doi.org/10.3390/IJMS20164059
Jiang W., Hou Y., Inouye M. // J. Biol. Chem. 1997. V. 272. P. 196–202. https://doi.org/10.1074/JBC.272.1.196
Giangrossi M., Gualerzi C.O., Pon C.L. // Biochimie. 2001. V. 83. P. 251–259. https://doi.org/10.1016/S0300-9084(01)01233-0
Jones P.G., Krah R., Tafuri S.R., Wolffe A.P. // J. Bacteriol. 1992. V. 174. P. 5798–5802. https://doi.org/10.1128/JB.174.18.5798-5802.1992
Bae W., Xia B., Inouye M., Severinov K. // Proc. Natl. Acad. Sci. USA. 2000. V. 97. P. 7784–7789. https://doi.org/10.1073/pnas.97.14.7784
Bae W., Jones P.G., Inouye M. // J. Bacteriology. 1997. V. 179. P. 7081–7088. https://doi.org/10.1128/JB.179.22.7081-7088.1997
Wu L., Ma L., Li X., Huang Z., Gao X. // Mol. Plant Pathol. 2019. V. 20. P. 382–391. https://doi.org/10.1111/MPP.12763
la Teana A., Brandi A., Falconi M., Spurio R., Pon C.L., Gualerzi C.O. // Proc. Natl. Acad. Sci. USA. 1991. V. 88. P. 10907–10911. https://doi.org/10.1073/PNAS.88.23.10907
Hwang J., Severinov K., Phadtare S., Inouye M. // Gen. Cells. 2003. V. 8. P. 801–810. https://doi.org/10.1046/j.1365-2443.2003.00675.x
Caballero C.J., Menendez-Gil P., Catalan-Moreno A., Vergara-Irigaray M., García B., Segura V., Irurzun N., Villanueva M., de Los Mozos I.R., Solano C., Lasa I., Toledo-Arana A. // Nucleic. Acids Res. 2018. V. 46. P. 1345. https://doi.org/10.1093/NAR/GKX1284
Zhang Y., Burkhardt D.H., Rouskin S., Li G.W., Weissman J.S., Gross C.A. // Mol. Cell. 2018. V. 70. P. 274.e7–286.e7. https://doi.org/10.1016/J.MOLCEL.2018.02.035
Choi J., Salvail H., Groisman E.A. // Nucleic. Acids Res. 2021. V. 49. P. 11614–11628. https://doi.org/10.1093/NAR/GKAB992
Hofweber R., Horn G., Langmann T., Balbach J., Kremer W., Schmitz G., Kalbitzer H.R. // FEBS J. 2005. V. 272. P. 4691–4702. https://doi.org/10.1111/J.1742-4658.2005.04885.X
Faßhauer P., Busche T., Kalinowski J., Mäder U., Poehlein A., Daniel R., Stülke J. // Microorganisms. 2021. V. 9. P. 1434. https://doi.org/10.3390/microorganisms9071434
Michaux C., Holmqvist E., Vasicek E., Sharan M., Barquist L., Westermann A.J., Gunn J.S., Vogel J. // Proc. Natl. Acad. Sci. USA. 2017. V. 114. P. 6824–6829. https://doi.org/10.1073/pnas.1620772114
Prezza G., Ryan D., Mädler G., Reichardt S., Barquist L., Westermann A.J. // Mol. Microbiol. 2022. V. 117. P. 67–85. https://doi.org/10.1111/MMI.14793
Płociński P., Macios M., Houghton J., Niemiec E., Płocińska R., Brzostek A., Słomka M., Dziadek J., Young D., Dziembowski A. // Nucl. Acids Res. 2019. V. 47. P. 5892–5905. https://doi.org/10.1093/NAR/GKZ251
Feng Y., Huang H., Liao J., Cohen S.N. // J. Biol. Chem. 2001. V. 276. P. 31651–31656. https://doi.org/10.1074/JBC.M102855200
Loepfe C., Raimann E., Stephan R., Tasara T. // Foodborne Pathogens Dis. 2010. V. 7. P. 775–783. https://doi.org/10.1089/FPD.2009.0458
Schmid B., Klumpp J., Raimann E., Loessner M.J., Stephan R., Tasara T. // Appl. Environ. Microbiol. 2009. V. 75. P. 1621–1627. https://doi.org/10.1128/AEM.02154-08
Eshwar A.K., Guldimann C., Oevermann A., Tasara T. // Front. Cell. Infect. Microbiol. 2017. V. 7. P. 453. https://doi.org/10.3389/fcimb.2017.00453
Portnoy D.A., Suzanne Jacks P., Hinrichs D.J. // J. Exp. Med. 1988. V. 167. P. 1459–1471. https://doi.org/10.1084/JEM.167.4.1459
Slepkov E.R., Bitar A.P., Marquis H. // Biochem. J. 2010. V. 432. P. 557–566. https://doi.org/10.1042/BJ20100557
Glomski I.J., Gedde M.M., Tsang A.W., Swanson J.A., Portnoy D.A. // J. Cell. Biol. 2002. V. 156. P. 1029–1038. https://doi.org/10.1083/JCB.200201081
Kocks C., Gouin E., Tabouret M., Berche P., Ohayon H., Cossart P. // Cell. 1992. V. 68. P. 521–531. https://doi.org/10.1016/0092-8674(92)90188-I
Welch M.D., Iwamatsu A., Mitchison T.J. // Nature. 1997. V. 385. P. 265–269. https://doi.org/10.1038/385265a0
Travier L., Guadagnini S., Gouin E., Dufour A., Chenal-Francisque V., Cossart P., Olivo-Marin J.C., Ghigo J.M., Disson O., Lecuit M. // PLoS Pathog. 2013. V. 9. P. e1003131. https://doi.org/10.1371/JOURNAL.PPAT.1003131
Crawford R.W., Rosales-Reyes R., Ramírez-Aguilar M.D.L.L., Chapa-Azuela O., Alpuche-Aranda C., Gunn J.S. // Proc. Natl. Acad. Sci. USA. 2010. V. 107. P. 4353–4358. https://doi.org/10.1073/pnas.1000862107
Ray S., da Costa R., Das M., Nandi D. // J. Biol. Chem. 2019. V. 294. P. 9084–9099. https://doi.org/10.1074/JBC.RA119.008209
Batte J.L., Samanta D., Elasri M.O. // Microbiology (Reading). 2016. V. 162. P. 575–589. https://doi.org/10.1099/MIC.0.000243
Pandey S., Sahukhal G.S., Elasri M.O. // J. Bacteriol. 2019. V. 201. https://doi.org/10.1128/JB.00417-19
Wang Z., Wang S., Wu Q. // FEMS Microbiol. Lett. 2014. V. 354. P. 27–36. https://doi.org/10.1111/1574-6968.12430
Wang Z., Liu W., Wu T., Bie P., Wu Q. // Sci. China Life Sci. 2016. V. 59. P. 417–424. https://doi.org/10.1007/S11427-015-4981-6
Weldingh K., Andersen P. // FEMS Immunol. Med. Microbiol. 1999. V. 23. P. 159–164. https://doi.org/10.1111/J.1574-695X.1999.TB01235.X
D’Auria G., Esposito C., Falcigno L., Calvanese L., Iaccarino E., Ruggiero A., Pedone C., Pedone E., Berisio R. // Biochem. Biophys. Res. Commun. 2010. V. 402. P. 693–698. https://doi.org/10.1016/J.BBRC.2010.10.086
Kumar A., Alam A., Tripathi D., Rani M., Khatoon H., Pandey S., Ehtesham N.Z., Hasnain S.E. // Semin. Cell Dev. Biol. 2018. V. 84. P. 147–157. https://doi.org/10.1016/J.SEMCDB.2018.01.003
Huang L., Liu W., Jiang Q., Zuo Y., Su Y., Zhao L., Qin Y., Yan Q. // Front. Cell. Infect. Microbiol. 2018. V. 8. P. 207. https://doi.org/10.3389/FCIMB.2018.00207/BIBTEX
Huang L., Zhao L., Qi W., Xu X., Zhang J., Zhang J., Yan Q. // Aquaculture. 2020. V. 518. P. 734861. https://doi.org/10.1016/J.AQUACULTURE.2019.734861
Liu Y., Tan X., Cheng H., Gong J., Zhang Y., Wang D., Ding W. // Microb. Pathog. 2020. V. 142. P. 104091. https://doi.org/10.1016/J.MICPATH.2020.104091
Schwenk S., Arnvig K.B. // Pathogens Dis. 2018. V. 76. P. 35. https://doi.org/10.1093/FEMSPD/FTY035
Wexler A.G., Goodman A.L. // Nat. Microbiol. 2017. V. 2. P. 1–11. https://doi.org/10.1038/nmicrobiol.2017.26
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия



